Method Article
Análisis poblacional y unicelular de la persistencia antibiótica en Escherichia coli
En este artículo
Resumen
La persistencia de antibióticos describe la capacidad de pequeñas subpoblaciones dentro de una población isogénica sensible para tolerar transitoriamente altas dosis de antibióticos bactericidas. El presente protocolo combina enfoques para caracterizar el fenotipo de persistencia antibiótica a nivel molecular y celular después de exponer a Escherichia coli a dosis letales de ofloxacino.
Resumen
La persistencia de antibióticos se refiere a la capacidad de pequeñas subpoblaciones bacterianas para tolerar transitoriamente altas dosis de antibióticos bactericidas. Tras el tratamiento con antibióticos bactericidas, la mayor parte de la población bacteriana muere rápidamente. Esta primera fase rápida de matanza es seguida por una disminución sustancial en la tasa de muerte a medida que las células persistentes permanecen viables. Clásicamente, la persistencia se determina a nivel poblacional mediante ensayos de tiempo/muerte realizados con altas dosis de antibióticos y para tiempos de exposición definidos. Si bien este método proporciona información sobre el nivel de células persistentes y la cinética de muerte, no refleja la heterogeneidad intrínseca de célula a célula subyacente al fenómeno de persistencia. El protocolo descrito aquí combina ensayos clásicos de tiempo/muerte con análisis de células individuales utilizando microscopía de fluorescencia en tiempo real. Mediante el uso de reporteros fluorescentes apropiados, las imágenes de microscopía de células vivas pueden proporcionar información sobre los efectos del antibiótico en los procesos celulares, como la replicación y segregación cromosómica, el alargamiento celular y la división celular. La combinación de análisis poblacional y unicelular permite la caracterización molecular y celular del fenotipo de persistencia.
Introducción
Este protocolo tiene como objetivo analizar el fenotipo de persistencia bacteriana en respuesta al tratamiento antibiótico específico a nivel unicelular y poblacional. La persistencia describe la capacidad de pequeñas subpoblaciones dentro de una población isogénica para soportar altas dosis de antibióticos bactericidas (fluoroquinolonas, aminoglucósidos, β-lactámicos, etc.), siendo la concentración inhibitoria mínima (CMI) de las llamadas células persistentes idéntica a la del grueso de la población. La dinámica de muerte bifásica, al medir la supervivencia bacteriana a lo largo del tiempo en presencia de un antibiótico, revela la presencia de células transitoriamente tolerantes a los medicamentos, con una rápida erradicación inicial de las células no persistentes, seguida de una tasa de eliminación mucho más lenta de las células persistentes. Tras la eliminación de antibióticos, estas células dan lugar a una población genéticamente idéntica que muestra una dinámica de muerte similar cuando se trata con el mismo antibiótico 1,2. A diferencia de la persistencia, la resistencia a los antibióticos se define a nivel poblacional y generalmente es una consecuencia de mutaciones de novo o de la transferencia horizontal de genes de un plásmido que confiere resistencia3. Si bien las mutaciones responsables de la resistencia se localizan principalmente en el objetivo del fármaco o en las regiones promotoras de las bombas de eflujo del fármaco, los genes que alteran la frecuencia de persistencia identificados por los enfoques de análisis mutante dirigido y de todo el genoma han demostrado ser numerosos y diversos 2,3,4,5,6,7,8 . Por lo tanto, es probable que las células bacterianas puedan entrar en el estado persistente a través de múltiples vías 9,10,11, y se necesitan enfoques para investigar el fenómeno de persistencia a nivel unicelular para caracterizar la fisiología de estas células persistentes.
El reciente desarrollo de herramientas microfluídicas utilizadas en combinación con microscopía de fluorescencia ha allanado el camino para la caracterización del fenotipo de persistencia y ha destacado el papel de los procesos celulares clave, como la replicación cromosómica12, la reparación del ADN 13 y la división celular14, en la formación de células persistentes. En este artículo, describimos un enfoque integrado que combina ensayos microbiológicos clásicos con imágenes vivas de una sola célula para caracterizar las células persistentes generadas en cultivos de Escherichia coli de crecimiento exponencial tratados con una alta dosis de ofloxacina. El protocolo descrito aquí se puede aplicar para estudiar el fenómeno de persistencia antibiótica en otras especies bacterianas, como Bacillus subtilis15, o condiciones (por ejemplo, persistencia de antibióticos después del tratamiento con β-lactámicos 16) y puede modificarse fácilmente para investigar los muchos fenómenos que involucran heterogeneidad fenotípica17,18,19 . Además, la configuración descrita en este documento se puede combinar con otros reporteros fluorescentes para investigar distintos parámetros celulares de interés, como los niveles intracelulares de pH20 o ATP21 a nivel de una sola célula, que potencialmente pueden producir nuevos conocimientos sobre el fenómeno de persistencia de antibióticos.
Protocolo
NOTA: Utilice cristalería de cultivo estéril, puntas de pipeta y medio de cultivo. Aquí, las células de E. coli se cultivaron en un medio químicamente definido de baja autofluorescencia (ver Tabla de materiales). Las inoculaciones se realizaron en presencia de un quemador Bunsen para minimizar el riesgo de contaminación.
1. Cultivo celular y curva de crecimiento
- Rayar la cepa de interés de una cepa de glicerol congelada en una placa de agar Luria-Bertaini (LB) (suplementada con un antibiótico selectivo, si es necesario), e incubar a 37 °C durante la noche (entre 15 h y 19 h) para obtener colonias individuales.
NOTA: El experimento aquí presentado utiliza dos cepas, E. coli K-12 MG1655 correspondiente a la cepa wt y la cepa isogénica MG1655 hupA-mCherry 22. Esta última cepa expresa la subunidad α marcada con fluorescencia de la proteína asociada al nucleoide HU. El reportero hupA-mCherry está integrado en el locus nativo de hupA. HU-mCherry sirve como un proxy para seguir la dinámica nucleoide en células vivas a medida que se une al ADN de una manera no específica. - Inocular 5 ml de medio (aquí, medio a base de ácido propanosulfónico [MOPS] 3-[N-morfolino]; Tabla 1, Tabla 2 y Tabla 3) suplementado con glucosa al 0,4% y un antibiótico selectivo (si es necesario) con una colonia aislada en un tubo de vidrio (≥25 mL), y colocar el tubo en una incubadora agitadora a 37 °C y 180 rotaciones por minuto (rpm) durante la noche (entre 15 h y 19 h). Alternativamente, se pueden usar tubos de plástico, vidrio o matraces de plástico (≥ 25 ml) en lugar de tubos de vidrio.
NOTA: El medio MOPS suplementado con glicerol a una concentración final de 0,4% se utilizó a lo largo de los experimentos descritos en este trabajo, excepto para los cultivos nocturnos que se realizaron en MOPS glucosa 0,4%. El tiempo de generación de E. coli en MOPS suplementado con glucosa es más corto que en MOPS glycerol. El uso de glucosa MOPS 0.4% en lugar de glicerol MOPS 0.4% en este paso asegura que las células alcancen la fase estacionaria dentro de las 19 h. También se pueden utilizar otros medios de crecimiento, como M9 o medio definido rico (RDM) complementado con distintas fuentes de carbono. Sin embargo, cabe señalar que la tasa de crecimiento y la frecuencia de persistencia son diferentes según el medio y/o el carbono utilizado23. - A la mañana siguiente, centrifugar 1 ml de cultivo a 2.300 x g durante 3 min, desechar el sobrenadante y resuspender suavemente el pellet en el mismo volumen de solución salina tamponada con fosfato (PBS). Mida la densidad óptica a 600 nm (OD 600 nm) y calcule el volumen necesario para un ODinicial de 600 nm de 0,01 en un volumen final de 2 mL.
- Coloque 2 ml de medio de glicerol MOPS al 0,4% en un pocillo de una placa de 24 pocillos de fondo transparente e inocule con el volumen de cultivo calculado durante la noche. Coloque la placa de 24 pocillos en un lector automático de microplacas (consulte la Tabla de materiales) para monitorear el OD600 nm durante 24 h. Ajuste el lector de microplacas para medir el OD600 nm cada 15 min a una temperatura de 37 °C y con alta rotación orbital (140 rpm).
NOTA: Si la cepa utilizada en el experimento codifica un reportero fluorescente, asegúrese de que su tasa de crecimiento sea comparable a la del peso para evitar cualquier artefacto en los experimentos posteriores, ya que la tasa de crecimiento afecta la frecuencia de persistencia de antibióticos23. Debido a las limitaciones de detección a densidades celulares muy bajas y los posibles efectos específicos de la cepa en la relación OD600 nm/unidades formadoras de colonias (UFC), se recomienda evaluar la cinética de crecimiento monitorizando la UFC-1 cuando se trabaja con cepas no caracterizadas.
2. Determinación de la concentración inhibitoria mínima de los antibióticos
NOTA: La concentración inhibitoria mínima (CMI) se define como la dosis más baja de antibiótico en la que no se observa crecimiento bacteriano. La determinación de la CMI debe realizarse para cada antibiótico y cepa. En los experimentos descritos aquí, se utilizó el antibiótico fluoroquinolona ofloxacino (OFX). La determinación de la CMI permite confirmar que la solución antibiótica se ha preparado correctamente, que el antibiótico está activo y que las cepas son igualmente sensibles al antibiótico. Aquí, se realizó el método de dilución de agar publicado para determinar el CMI a OFX de las diferentes cepas utilizadas24. La CMI de un antibiótico dado para una cepa bacteriana dada también puede determinarse a través del método de dilución del caldo24.
- Preparación de las placas para la determinación de MIC
- Preparar una solución madre maestra para el antibiótico utilizado en los experimentos disolviendo 5 mg de OFX en 1 ml de agua ultrapura. Añadir 20 μL de HCl al 37% para aumentar la solubilidad del OFX.
- Derretir 100 ml de agar LB estéril y mantenerlo a 55 °C para evitar la solidificación. Preparar seis matraces de vidrio pequeños (25 ml) y pipetear 5 ml de medio de agar LB líquido en cada matraz con una pipeta estéril.
- Diluir 10 μL de la solución madre maestra de 5 mg·mL-1 OFX en 90 μL de agua ultrapura (dilución 1:10 de la cepa maestra). Añadir 0 μL, 2 μL, 4 μL, 6 μL, 8 μL o 10 μL de la solución OFX de 500 μg·mL-1 a cada uno de los seis matraces de vidrio que contengan 5 ml de medio de agar LB para generar medio de agar LB con concentraciones finales de 0 μg·mL−1, 0,02 μg·mL−1, 0,04 μg·mL−1, 0,06 μg·mL−1, 0,08 μg·mL−1 y 0,1 μg·mL-1 OFX, respectivamente. Mezclar la solución girando los matraces varias veces.
NOTA: Si se conoce la CMI del antibiótico de interés, el rango de concentraciones debe alcanzar de abajo a por encima de la CMI real. Si se desconoce la CIM, se recomienda un amplio rango de concentraciones con una serie de dilución log2 . Asegúrese de que el medio de agar LB se haya enfriado antes de agregar el antibiótico, ya que las altas temperaturas pueden inactivarlo. Sin embargo, es importante no dejar que el medio de agar LB se solidifique antes de agregar el antibiótico, ya que esto podría conducir a la distribución no homogénea del antibiótico en el medio de agar LB. - Verter 5 ml de cada uno de los seis medios de agar LB preparados en la etapa 2.1.3 de forma que aumente la dosis en una placa de cultivo de 6 pocillos utilizando una pipeta estéril. Deje que el agar se enfríe hasta que se solidifique y seque la placa antes de usarla.
NOTA: Prepare la solución antibiótica y la placa de cultivo el día en que se realice el ensayo.
- Ensayo de determinación de CMI
- Inocular 5 ml de medio LB con una colonia aislada en un tubo de vidrio (≥25 mL) y colocar el tubo de vidrio en una incubadora agitadora a 37 °C y 180 rpm durante la noche (entre 15 h y 19 h).
- A la mañana siguiente, medir el OD600 nm y diluir el cultivo en un tubo de ensayo hasta una densidad celular final de 1 x 107 UFC·mL-1 en PBS. Para las cepas ensayadas aquí, 1 x 107 UFC·mL−1 corresponde a un OD600 nm de 0,0125.
NOTA: Si no se conoce la correlación entre la UFC-mL-1 y la OD 600 nm, deberá establecerse la curva de crecimiento determinada por las mediciones de UFC y OD 600 nm para calcular el factor de correlación entre la UFC·mL-1 y la OD600 nm. - Colocar 2 μL del cultivo previamente diluido en cada pocillo de la placa seca de 6 pocillos. Deje secar las manchas antes de colocar la placa en una incubadora a 37 °C durante la noche (entre 15 h y 19 h).
- Al día siguiente, cuente las colonias formadas en cada pozo. La CMI corresponde al pocillo con la concentración mínima de antibiótico donde no se detecta crecimiento bacteriano.
3. Ensayo puntual
NOTA: El método de ensayo puntual es un enfoque cualitativo que permite la estimación del número de células viables (células capaces de generar colonias después del estrés antibiótico). El ensayo puntual se realiza antes del ensayo de tiempo de muerte para proporcionar información sobre la viabilidad de la cepa utilizada en las condiciones ensayadas y para informar sobre las diluciones necesarias durante el ensayo de delimitación temporal (ver sección 4).
- Para preparar las placas de agar LB para ensayos puntuales, vierta 50 ml de agar LB en una placa de Petri cuadrada (144 cm2). Prepare una placa de Petri cuadrada por punto de tiempo. Deje que el agar LB se solidifique y seque las placas antes de usarlas.
- Inocular 5 mL de medio (glucosa MOPS 0,4%) con una colonia aislada en un tubo de vidrio (≥25 mL), y colocar el tubo en una incubadora agitadora a 37 °C y 180 rpm durante la noche (entre 15 h y 19 h).
- Al día siguiente, medir el OD 600 nm y diluir el cultivo en un medio fresco ajustado a la temperatura (37 °C, glicerol MOPS 0,4%) en un tubo de vidrio (≥25 ml) hasta un OD finalde 600 nm de ~0,001. Dejar crecer el cultivo durante la noche en una incubadora a 37 °C a 180 rpm (entre 15 h y 19 h).
- Al día siguiente, medir el OD600 nm e incubar el cultivo hasta un OD finalde 600 nm de 0,3.
- Durante la incubación, prepare placas de 96 pocillos colocando 90 μL de solución de MgSO4 0,01 M en todos los pocillos excepto en los pocillos de la primera fila (fila A). Las placas de 96 pocillos se utilizarán para una dilución en serie de 10 veces en el paso 3.7.
NOTA: En este experimento, dos cepas (wt y hupA-mCherry strain) se probaron por triplicado en siete puntos de tiempo diferentes. Como una placa comprende 12 columnas, una placa se puede usar para dos puntos de tiempo y, por lo tanto, se prepararon un total de cuatro placas. - A una OD 600 nm de 0,3, extraer200 μL de cada cultivo bacteriano. Estas muestras corresponden al punto de tiempo t0 (no tratado) previo al tratamiento antibiótico y permiten la determinación de la UFC-1 antes del tratamiento antibiótico.
- Centrifugar las muestras a 2.300 x g durante 3 min. Durante el tiempo de centrifugación, añadir la concentración deseada de OFX al cultivo líquido y continuar incubando a 37 °C mientras agita.
NOTA: Aquí, OFX se utilizó a una concentración de 5 μg·mL-1 (correspondiente a la CMI multiplicada 83 veces). En un estudio previo, esta concentración fue utilizada para caracterizar el fenómeno de persistencia bajo exposición a OFX25. La concentración de antibióticos utilizada para los ensayos de tiempo/muerte puede variar según el antibiótico, el medio de cultivo y el estado de crecimiento bacteriano. - Después de la centrifugación, resuspender el pellet celular en 200 μL de solución de 0,01 M MgSO4 . Introducir 100 μL en el pocillo vacío de la placa de 96 pocillos preparada en el paso 3.5. Realizar una dilución transfiriendo 10 μL del pozo de la fila A al pozo de la fila B que contiene 90 μL de 0,01 M deMgSO4. Continuar las diluciones seriadas transfiriendo 10 μL del pocillo de la fila B al pozo de la fila C. Repita hasta alcanzar una dilución de 10−7 (siendo cada transferencia una dilución de 10 veces).
NOTA: En este experimento, se probaron seis cultivos (tres para la cepa wt y tres para la cepa hupA-mCherry ) para cada punto de tiempo. De acuerdo con el protocolo, se utiliza una pipeta multicanal para realizar las diluciones en serie para las seis cepas al mismo tiempo. Para minimizar el error técnico, las puntas de la pipeta se cambian entre cada transferencia. - Colocar 10 μL de cada dilución en las placas de agar LB preparadas en la etapa 3.1.
NOTA: Se utiliza una pipeta multicanal para detectar simultáneamente la misma dilución (por ejemplo, una dilución 10−4 ) para los seis cultivos. Durante el manchado, se debe utilizar el primer paso de la pipeta multicanal sin empujar el segundo tope, ya que esto puede provocar la dispensación de microgotas sobre la placa. - En los momentos pertinentes después de la adición del antibiótico, extraer 200 μL del cultivo y realizar diluciones seriadas como se describe en el paso 3.8. Detectar 10 μL de cada dilución, tal como se describe en la etapa 3.9.
NOTA: Para el experimento descrito aquí, se recolectaron siete puntos de tiempo (t0, t1h, t2h, t3h, t4h, t5h, t6h). Para las cepas que exhiben una mayor susceptibilidad a los antibióticos en comparación con el peso, se pueden realizar múltiples lavados en MgSO4 0.01 M para eliminar los antibióticos residuales. - Incubar las placas a 37 °C durante la noche (entre 15 h y 19 h). Al día siguiente, cuente el número de colonias en las dos diluciones más altas para las cuales se pueden detectar colonias. Idealmente, las manchas en las placas de agar para estas diluciones contienen entre 3 y 30 colonias que permiten la determinación precisa de la UFC-1 para cada muestra.
- Calcule la razón de supervivencia dividiendo la UFC·mL-1 calculada de cada punto temporal por la UFC·mL-1 de la población inicial en t0.
4. Ensayos de tiempo muerto
NOTA: Si bien los ensayos puntuales son un método fácil de usar para estimar la tasa de supervivencia de una cepa dada para un antibiótico dado, los ensayos de muerte en el tiempo dan una tasa de supervivencia de mayor resolución y se realizan para cuantificar con precisión la viabilidad bacteriana. El perfil de la curva de muerte se puede utilizar para determinar si una cepa bacteriana dada es sensible, tolerante o resistente al antibiótico en una condición dada. Además, los ensayos de tiempo de muerte permiten determinar el tiempo de exposición a antibióticos necesario para detectar el fenómeno de persistencia (comienzo de la segunda pendiente de la curva de muerte bifásica), así como la frecuencia de persistencia.
- Prepare las placas de agar LB para el ensayo de recubrimiento de muerte temporal. Vierta 25 ml de agar LB en una placa de Petri (±57 cm2). Prepare al menos dos placas de Petri por punto de tiempo (se emplatan dos diluciones por punto de tiempo por cepa).
- Deje que el agar LB se solidifique y seque las placas antes de agregar de cinco a ocho perlas de vidrio estériles a cada placa. Invierta y etiquete las placas de acuerdo con la tensión / condición / punto de tiempo.
NOTA: Las perlas de vidrio permiten la propagación de bacterias en las placas de agar durante los pasos 4.7 y 4.9. Alternativamente, las células se pueden dispersar en la placa de agar utilizando un esparcidor. - Para cada muestra, preparar tubos de vidrio de dilución seriada de 10 veces que contengan 900 μL de solución de 0,01 MMgSO4 . El número de tubos de vidrio de dilución por muestra que debe prepararse corresponde a las diluciones necesarias para detectar colonias en el ensayo puntual.
- Inocular 5 mL de medio (glucosa MOPS 0,4%) con una colonia aislada en un tubo de vidrio (≥25 mL), y colocar el tubo en una incubadora agitadora a 37 °C y 180 rpm durante la noche (entre 15 h y 19 h).
- Después de 16 h de crecimiento, medir el OD 600 nm y diluir el cultivo en un medio fresco ajustado a la temperatura (37 °C, glicerol MOPS 0,4%) en un tubo de vidrio hasta un OD final de600 nm de ~0,001. Dejar crecer el cultivo durante la noche en una incubadora a 37 °C a 180 rpm (entre 15 h y 19 h).
- Al día siguiente, medir el OD600 nm e incubar el cultivo hasta un OD finalde 600 nm de 0,3.
- A una DO 600 nm de 0,3, extraer 100 μL del cultivo, diluir según los datos obtenidos en el ensayo puntual (a una DO 600 nm de 0,3 y sin antibiótico, una dilución 10−5 suele dar 200-300 colonias), y una placa de100 μL sobre las placas de agar LB preparadas en los pasos 4.1-4.2. Agite suavemente las placas para evitar que las perlas entren en contacto con los bordes del plato, ya que esto puede conducir a una propagación no homogénea de las células bacterianas en el medio de agar LB. Esta primera muestra previa al tratamiento antibiótico corresponde al punto de tiempo t0 (UFC·mL-1 antes del tratamiento antibiótico).
- Añadir la concentración deseada de OFX, y continuar incubando a 37 °C mientras agita.
NOTA: Aquí, OFX se utilizó a una concentración de 5 μg·mL-1 (correspondiente a la CMI multiplicada 83 veces). - En los momentos pertinentes después de la adición del antibiótico, extraer 100 μL del cultivo, diluir de acuerdo con los datos obtenidos en el ensayo puntual, y la placa 100 μL en las placas de agar LB preparadas en los pasos 4.1-4.2. Colocar las celdas en placa como se describe en el paso 4.7.
NOTA: Para el experimento descrito aquí, se recolectaron siete puntos de tiempo (t0, t1h, t2h, t3h, t4h, t5h, t6h). Para las cepas que exhiben una mayor susceptibilidad a los antibióticos en comparación con el peso, se pueden realizar múltiples lavados en MgSO4 0.01 M para eliminar los antibióticos residuales. - Incubar las placas a 37 °C durante la noche (entre 15 h y 19 h). Al día siguiente, cuente el número de colonias en las dos diluciones más altas para las cuales se pueden detectar colonias. Idealmente, las placas deben contener 30-300 colonias para permitir una determinación precisa de la UFC-mL-1 en cada muestra.
- Calcule la razón de supervivencia normalizando la UFC-mL-1 en cada punto temporal por la UFC-mL-1 en t0. Trazar el log10 normalizado CFU·mL-1 en función del tiempo.
5. Microscopía microfluídica de lapso de tiempo
NOTA: La siguiente sección describe la preparación de la placa microfluídica, así como el procedimiento de adquisición de imágenes de lapso de tiempo y análisis de imágenes. El objetivo de este experimento es observar y analizar el fenotipo de persistencia en el tratamiento antibiótico a nivel unicelular. Los datos recopilados durante este experimento se pueden utilizar para generar una amplia gama de resultados dependiendo de la pregunta abordada y / o los reporteros fluorescentes utilizados durante el experimento. En el experimento aquí descrito, se realizó un análisis cuantitativo de la longitud celular y la fluorescencia HU-mCherry22, que refleja la organización nucleoide en células persistentes y no persistentes.
- Cultivo celular bacteriano para microscopía de lapso de tiempo microfluídico
- Inocular 5 ml de medio (glicerol MOPS 0,4%, suplementado con un antibiótico selectivo si es necesario) con una colonia aislada en un tubo de vidrio (≥25 mL), y colocar el tubo en una incubadora de agitación a 37 °C y 180 rpm durante la noche (entre 15 h y 19 h).
- Al día siguiente, medir el OD 600 nm y diluir el cultivo en un medio fresco ajustado a la temperatura (37 °C, glicerol MOPS 0.4%) en un tubo de vidrio hasta un OD final600 nm de ~0.001. Dejar crecer el cultivo durante la noche (entre 15 h y 19 h) en una incubadora de agitación a 37 °C y 180 rpm para obtener un cultivo de fase exponencial temprana al día siguiente.
- Preparación de la placa microfluídica y microscopía de lapso de tiempo
NOTA: Los experimentos microfluídicos se pueden realizar en dispositivos microfluídicos disponibles comercialmente (como se describe aquí) o en sistemas microfluídicos producidos internamente.- Retire la solución de conservación (si está presente) de cada pocillo de la placa microfluídica y reemplácela con un medio de cultivo fresco.
NOTA: Si la placa microfluídica contiene un pozo de salida de desechos, la solución de conservación del pozo de salida debe eliminarse pero no reemplazarse por el medio. - Selle la placa microfluídica con el sistema de colector haciendo clic en el botón Sello o a través del software microfluídico (primero seleccione Herramienta, seguido de Placa de sellado).
NOTA: Para sellar la placa, se debe aplicar una presión uniforme a la placa y al colector apretando manualmente la placa contra el colector. Si se realiza correctamente, la nota "sellado" debería aparecer en la interfaz ONIX2. Es importante no aplicar ninguna presión sobre el portaobjetos de vidrio para evitar cualquier riesgo potencial de rotura del colector. - Una vez sellado, realice una primera secuencia de cebado (haga clic en Ejecutar secuencia de cebado líquido en la interfaz del software microfluídico).
NOTA: La secuencia de cebado líquido de ejecución corresponde a 5 min de perfusión a 6,9 kPa para los pozos 1-5, seguido de 5 min de perfusión a 6,9 kPa para el pozo 8, y una ronda final de perfusión para el pozo 6 durante 5 min a 6,9 kPa. La secuencia de cebado líquido de ejecución permite la eliminación de la solución de conservación que aún puede estar presente en los canales que conectan los diferentes pozos. - Incubar la placa en un gabinete controlado termostáticamente del microscopio a la temperatura deseada (aquí, 37 °C) durante un mínimo de 2 h antes del inicio de la microscopía.
- Inicie una segunda secuencia de cebado líquido de ejecución antes de comenzar el experimento.
- Selle la placa microfluídica haciendo clic en Sellar en la interfaz del software microfluídico. Sustituir el medio en el pozo 1 y el pozo 2 por 200 μL de medio fresco, en el pocillo 3 por 200 μL de medio fresco que contenga el antibiótico (aquí, OFX a 5 μg·mL−1), en el pocillo 4 y en el pozo 5 por 200 μL de medio fresco, en el pozo 6 con 200 μL de medio fresco, y en el pocillo 8 con 200 μL de la muestra de cultivo (a partir de la etapa 5.1.2) diluido a un OD600 nm de 0,01 en el medio fresco.
- Selle la placa microfluídica como se describe en el paso 5.2.2 y coloque la placa en el objetivo del microscopio dentro del gabinete del microscopio.
NOTA: Asegúrese de colocar una gota de aceite de inmersión en el objetivo del microscopio antes de colocar la placa microfluídica. - En el software microfluídico, haga clic en Cell Loading (Carga de celdas ) para permitir que la célula se cargue en la placa microfluídica.
NOTA: La etapa de carga celular comprende 15 s de perfusión a 13,8 kPa para el pozo 8, seguido de 15 s de perfusión a 27,6 kPa para el pozo 6 y el pozo 8, y una ronda final de perfusión para el pozo 6 para 30 s a 6,9 kPa. La densidad de las células en la placa microfluídica es crítica para el experimento. La primera parte de este protocolo microfluídico consiste en cultivar bacterias durante 6 h en un medio fresco antes del tratamiento antibiótico. Después de 6 h de crecimiento, la densidad celular tiene que ser suficiente para detectar las células persistentes raras (en las condiciones utilizadas en este estudio, las células persistentes generan a una frecuencia de 10-4). Si la densidad celular es demasiado alta, es difícil distinguir las células individuales, lo que impide un análisis preciso de una sola célula. Como la tasa de crecimiento depende directamente del medio, la densidad de las células en los campos de microscopía debe evaluarse antes de iniciar el experimento. - Establezca un enfoque óptimo utilizando el modo de luz transmitida y seleccione varias regiones de interés (ROI) donde se observe un número de celda apropiado (hasta 300 celdas por campo).
NOTA: Seleccione al menos 40 ROI para asegurarse de que se obtengan imágenes de las células persistentes raras. - En el software microfluídico, haga clic en Crear un protocolo. Programe la inyección de medio fresco a 6,9 kPa durante 6 h (pocillo 1-2), seguida de la inyección del medio que contiene el antibiótico a 6,9 kPa durante 6 h (pocillo 3) y, finalmente, la inyección de medio fresco a 6,9 kPa durante 20 h (pocillos 4-5).
NOTA: Un cultivo bacteriano diluido a un OD600 nm de 0.01 permite que las bacterias crezcan en la cámara microfluídica durante 6 h, asegurando que las células estén en la fase de crecimiento exponencial. Dependiendo del número de células introducidas en el dispositivo microfluídico durante la etapa de carga (ver paso 5.2.8), la duración de la fase de crecimiento se puede adaptar para obtener hasta 300 células por ROI. Como la persistencia es un fenómeno raro, aumentar el número de células por ROI mejora la posibilidad de observar células persistentes. Sin embargo, el número de células no debe exceder las 300 células por ROI, ya que esto hace que el análisis de una sola célula sea tedioso. - Realice imágenes de microscopía en modo de lapso de tiempo con un fotograma cada 15 minutos utilizando luz transmitida y la fuente de luz de excitación para el reportero fluorescente. Aquí, se utilizó una fuente de luz de excitación de 560 nm para la señal mCherry (LED de 580 nm al 10% de potencia con filtro 00 [530-585 ex, 615LP em, Zeiss] y exposición de 100 ms para mCherry). El software Zen3.2 compatible con Zeiss se utilizó para imágenes celulares.
- Retire la solución de conservación (si está presente) de cada pocillo de la placa microfluídica y reemplácela con un medio de cultivo fresco.
- Análisis de imágenes
NOTA: La apertura y visualización de las imágenes de microscopía se realiza con el software de código abierto ImageJ/Fiji (https://fiji.sc/)26. El análisis cuantitativo de imágenes se realiza utilizando el software de código abierto ImageJ/Fiji y el plugin gratuito MicrobeJ (https://microbej.com)27. En este protocolo, se utilizó la versión MicrobeJ 5.13I(14).- Abra el software ImageJ/Fiji en el ordenador y arrastre las imágenes de microscopía time-lapse hyperstack a la barra de carga de Fiji. Utilice Image > Color > Make Composite para fusionar los diferentes canales de la hiperpila. Si los canales del experimento de lapso de tiempo no corresponden al color deseado (por ejemplo, el contraste de fase se muestra en rojo en lugar de gris), use Imagen > color > Organizar canales para aplicar el color apropiado a los canales.
- Abra el complemento MicrobeJ y detecte las células bacterianas utilizando la interfaz de edición manual. Elimine las celdas detectadas automáticamente y describa manualmente las celdas persistentes de interés fotograma a fotograma.
NOTA: Se pueden utilizar diferentes configuraciones para detectar automáticamente celdas individuales. La detección manual se utilizó aquí ya que las células persistentes analizadas forman filamentos largos, que rara vez se detectan correctamente mediante detección automática. - Después de la detección, utilice el icono Resultado en la interfaz de edición manual de MicrobeJ para generar una tabla ResultJ. Guarde el archivo ResultJ y utilice la tabla ResultJ para obtener información sobre los diferentes parámetros de interés del análisis de celda única. En el caso de este protocolo, se exportó la fluorescencia media de la intensidad de HU-mCherry, la longitud celular y el área celular de células individuales.
Resultados
Como se describió anteriormente, las cepas utilizadas para el análisis fenotípico unicelular de células persistentes se caracterizaron en medio de glicerol MOPS al 0,4%. El monitoreo del OD600nm a lo largo del tiempo no mostró diferencias entre las cepas wt y hupA-mCherry (Figura 1). Esto indica que la expresión de la proteína de fusión HU-mCherry no afectó el crecimiento en estas condiciones. Las células bacterianas de ambas cepas inoculadas inicialmente a un OD600 nm de 0,01 alcanzaron la fase exponencial ±8 h después de la inoculación.
La CMI de OFX fue determinada por métodos estandarizados (aquí, dilución serial de agar)24. La CMI se define como la concentración mínima en la que no se detecta un crecimiento visible. Se determinó que la CMI de OFX para ambas cepas era de 0,06 μg·mL−1, lo que indica que la fusión hupA-mCherry no tuvo ningún efecto sobre la sensibilidad a OFX en comparación con la cepa isogénica wt (Figura 2).
Además, determinamos el efecto de un tratamiento letal con OFX (CMI de 83 veces) sobre la viabilidad de ambas cepas utilizadas en este estudio. A medida que el recuento de células viables disminuye con el tiempo con la exposición a OXX, las diluciones de los cultivos bacterianos deben ajustarse adecuadamente para alcanzar de 30 a 300 colonias por placa. Para determinar las diluciones apropiadas a lo largo del tiempo, se realizó un ensayo puntual, donde se colocaron 10 μL de 0 a 10−7 diluciones seriadas de 10 veces en placas de Petri cuadradas utilizando una pipeta multicanal. Las diluciones apropiadas fueron aquellas en las que los clones aislados eran visibles (por ejemplo, en t0 = 10−5, t1h = 10−4/10−3, t4h = 10−2/10−1) (Figura 3).
Si bien el ensayo puntual es un método fácil para obtener información sobre la cinética de la matanza mediada por OFX, no logra determinar con precisión la dinámica de muerte. Cuando se monitorizó la viabilidad de las células de crecimiento exponencial tratadas con OFX mediante el ensayo de muerte temporal, se observó una curva bifásica típica (Figura 4). La primera pendiente de la curva refleja la rápida matanza de la población no persistente (línea discontinua roja). En las condiciones probadas aquí, hasta el 99,9% de las células no pudieron formar colonias después de 3 h en presencia de OFX. Esta primera fase de matanza es seguida por una segunda fase, que muestra una tasa de mortalidad más lenta (línea discontinua azul), que revela la presencia de células persistentes tolerantes a los medicamentos. En las condiciones probadas, la fase persistente comenzó alrededor de 3 h después de la adición de OFX, destacando la necesidad de exponer las células a OFX durante más de 3 h para investigar los fenotipos persistentes. Es importante destacar que la curva de muerte del tiempo muestra que la proteína de fusión hupA-mCherry no tuvo ningún efecto sobre la cinética de muerte del tiempo. La cepa que codifica la fusión fluorescente traslacional puede, por lo tanto, usarse para monitorear las células persistentes utilizando microscopía de fluorescencia.
Además, investigamos el fenómeno de persistencia a nivel unicelular. Para ello, se introdujo la cepa hupA-mCherry en una placa microfluídica, lo que permitió el cambio de las condiciones del medio (aquí, crecimiento, tratamiento y recuperación) mientras se realizaba microscopía time-lapse en un ROI dado. Durante el primer paso del experimento microfluídico, las células introducidas en el dispositivo microfluídico se perfundieron con medio de crecimiento (glicerol MOPS 0.4%) y se dividieron con un tiempo de generación de ~ 2 h (Figura 5 y Figura 6). Esta primera fase de crecimiento indica que las células eran viables y se dividían activamente antes del tratamiento con OFX.
Después de esta primera fase de crecimiento, las células se perfundieron con medio de crecimiento suplementado con 5 μg·mL−1 OFX durante 6 h. Tan pronto como el antibiótico llegó a las células, la división celular se bloqueó (Figura 5 y Figura 6). Después de 6 h de tratamiento con OFX, las células se perfundieron con medio fresco. Mientras que la gran mayoría de las células no pudieron reanudar el crecimiento (Figura 5 y Figura 6), una pequeña subpoblación de bacterias fue capaz de alargar y generar células filamentosas25. Estas células, que fueron capaces de dividirse y generar células hijas viables después del tratamiento OFX, se pueden definir como las células persistentes.
Como esta configuración permite la visualización de las células persistentes antes, durante y después del tratamiento, no solo proporciona información sobre el fenotipo persistente durante la fase de recuperación, sino también sobre el estado fisiológico de las células persistentes antes del tratamiento (Figura 6). En las condiciones probadas, las células persistentes se dividieron de manera similar a las células no persistentes antes del tratamiento OFX, lo que indica que las células persistentes observadas no se originaron en una subpoblación latente (Figura 6)25.
El análisis de la longitud celular de las células persistentes durante la fase de recuperación reveló que cada filamento tenía una tasa específica de elongación. La longitud celular alcanzada por cada persistente antes de la primera división difería de un persistente a otro. Del mismo modo, el momento del evento de primera división fue muy heterogéneo (Figura 6). El filamento persistente en división generó múltiples células hijas, que comenzaron a crecer y dividirse, en su mayor parte, de manera similar a las células no tratadas (Figura 7). La división sucesiva del filamento dio lugar a una disminución progresiva de la longitud celular, dando lugar finalmente a células hijas con una longitud celular similar a la anterior al tratamiento con OFX (Figura 6 y Figura 7B). La gran mayoría de las células no pudieron inducir la filamentación después de la eliminación de OFX. Esta gran población celular corresponde a las células muertas (Figura 5 y Figura 6).
La fusión fluorescente de la proteína HU asociada a nucleoides permite visualizar la dinámica del nucleoide22. El análisis de la intensidad total de fluorescencia de HU-mCherry dentro de la célula se puede utilizar como un proxy para el contenido de ADN22,25. Durante la fase de crecimiento (antes del tratamiento con OXX), la intensidad total de fluorescencia de mCherry varió, reflejando la dinámica de replicación y segregación cromosómica durante el ciclo celular (Figura 8). Después de la adición de OXX, la fluorescencia mCherry aumentó en la célula media, indicativo de compactación de nucleoides, que se ha demostrado que es inducida por la formación de roturas de ADN de doble cadena28 (Figura 5). Las roturas de ADN de doble cadena son consecuencia del mecanismo de acción de OFX, que corrompe las topoisomerasas tipo II ADN-girasa y topoisomerasa IV29,30. En E. coli, la ADN-girasa es el objetivo principal de OFX29,30. Al unir su objetivo en un paso crítico del mecanismo de paso de doble cadena, OFX inhibe la relegación de las hebras de ADN escindidas, lo que finalmente conduce a la liberación de roturas de ADN de doble cadena30. Como se describió anteriormente, las células persistentes para el tratamiento OFX comenzaron a filamentarse durante la recuperación25 (Figura 6). El aumento en la longitud celular se correlacionó con un aumento en la intensidad total de fluorescencia mCherry, lo que refleja el reinicio de la replicación y un aumento en la abundancia de nucleoides en el filamento25 (Figura 7a y Figura 8). Para las células muertas, la intensidad total de fluorescencia mCherry se mantuvo estable durante el tratamiento y durante la fase de recuperación, lo que indica que estas células no pudieron replicar sus cromosomas después de la eliminación de OFX (Figura 8). También se muestra un video microfluídico (Video 1) de células HU-mCherry de E. coli antes, durante y después del tratamiento con ofloxacina.
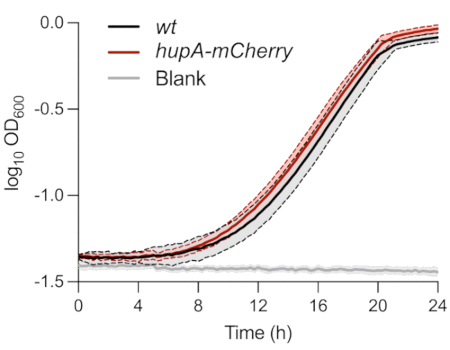
Figura 1: Monitorización del crecimiento de cepas de wt y hupA-mCherry E. coli. Monitorización de densidad óptica (OD600 nm) de wt (negro) y hupA-mCherry (rojo). Los matices y las líneas discontinuas indican las desviaciones estándar de los triplicados biológicos. Haga clic aquí para ver una versión más grande de esta figura.
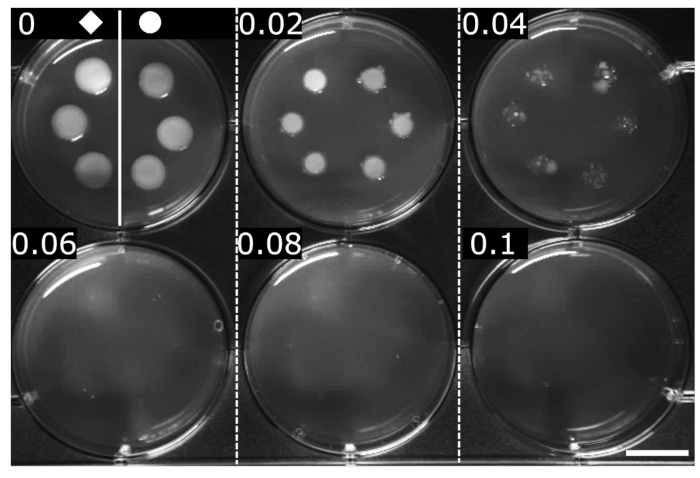
Figura 2: Determinación de la CMI de OFX para las cepas de wt y hupA-mCherry E. coli. El wt () y el hupA-mCherry (●) se cultivaron en medio LB, y se detectaron 2 μL en diluciones seriadas de agar LB que contenía OFX (♦ concentración indicada en cada panel en μg·mL−1). La inhibición del crecimiento es visible a un mínimo de 0,06 μg·mL−1. La figura es un experimento representativo de triplicados biológicos. Barra de escala = 1 cm. Haga clic aquí para ver una versión más grande de esta figura.
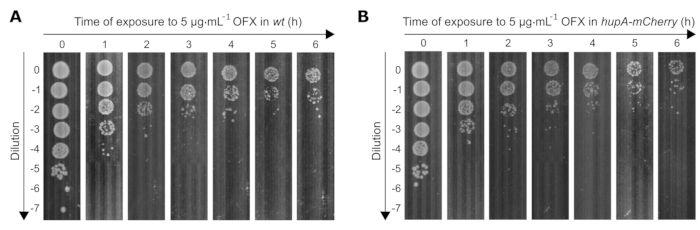
Figura 3: Ensayo puntual de cepas de wt y hupA-mCherry E. coli tras la exposición a OFX. Las cepas (A) wt y (B) hupA-mCherry se cultivaron en glicerol MOPS al 0,4% como se describe en el protocolo (sección 3), y las células de crecimiento exponencial (OD600 nm = 0,3) se trataron con 5 μg·mL−1 OFX. T0 corresponde al punto de tiempo antes de la adición de OFX. T1, T2, T3, T4, T5 y T6 corresponden a 1-6 h después de la adición de OXX. La figura es un experimento representativo de triplicados biológicos. Haga clic aquí para ver una versión más grande de esta figura.
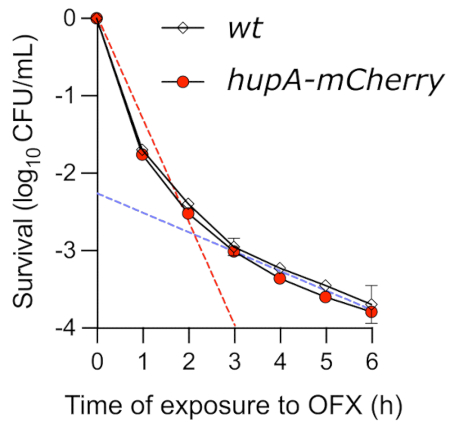
Figura 4: Ensayo de muerte temporal de cepas de wt y hupA-mCherry E. coli tras la exposición a OFX. Las cepas wt () y hupA-mCherry (●) se cultivaron en glicerol MOPS al 0,4% como se describe en el protocolo (sección 4), y las células de crecimiento exponencial (♦ OD600 nm = 0,3) se trataron con 5 μg·mL−1 OFX. Las líneas discontinuas indican la primera fase de muerte "rápida" (roja) y la segunda fase de muerte "lenta" (azul), correspondientes a las subpoblaciones sensibles y persistentes (obtenidas por regresión lineal entre T0 y T2, así como entre T3 y T6, respectivamente). Las barras de error indican las desviaciones estándar de los triplicados biológicos. Haga clic aquí para ver una versión más grande de esta figura.
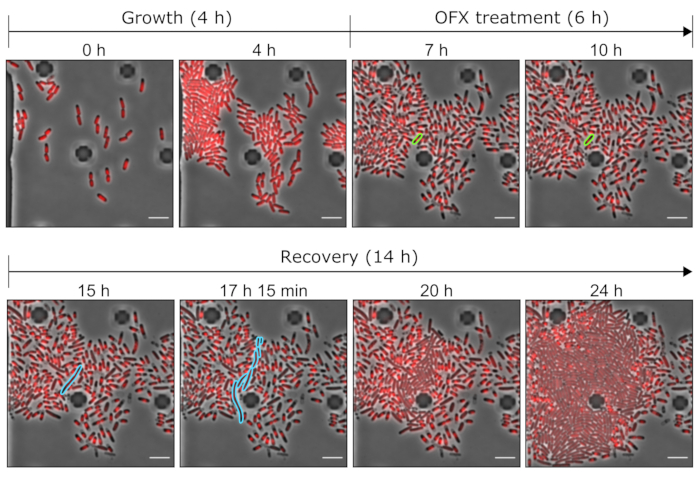
Figura 5: Imágenes representativas del persistidor OFX y células muertas utilizando herramientas microfluídicas. Imágenes representativas de microscopía que muestran los puntos de tiempo relevantes del experimento microfluídico realizado con la cepa hupA-mCherry (contraste de fase en gris, señal HU-mCherry en rojo). Las células que expresan el hupA-mCherry marcado se cultivaron en una placa microfluídica (aquí, 4 h), seguida de un desafío OFX (5 μg·mL−1). Después de 6 h en presencia de OFX, las células se perfundieron con medio fresco, permitiendo que las células persistentes se recuperaran. La célula persistente y sus células progenie durante el tratamiento OFX y después de la eliminación de OFX se resaltan en verde y azul, respectivamente. Los puntos de tiempo correspondientes se indican en cada panel. Barra de escala = 5 μm. Haga clic aquí para ver una versión más grande de esta figura.
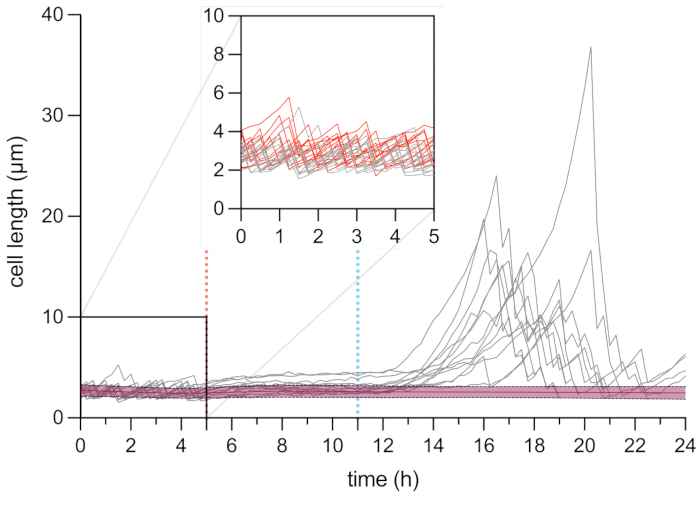
Figura 6: Análisis de lapso de tiempo de microscopía de la longitud de las células persistentes y muertas. Análisis de la longitud celular de células muertas (en rojo, n = 109) y células persistentes (en gris, n = 13). El inicio del tratamiento con OFX (5 μg·mL−1) se indica mediante la línea discontinua roja (5 h), y la eliminación de OFX se indica mediante la línea discontinua azul. El recuadro corresponde a la fase de crecimiento antes de la adición de OFX. Los experimentos se realizaron por triplicado. Los tonos y las líneas discontinuas indican las desviaciones estándar para la población de células muertas (n = 109). Haga clic aquí para ver una versión más grande de esta figura.
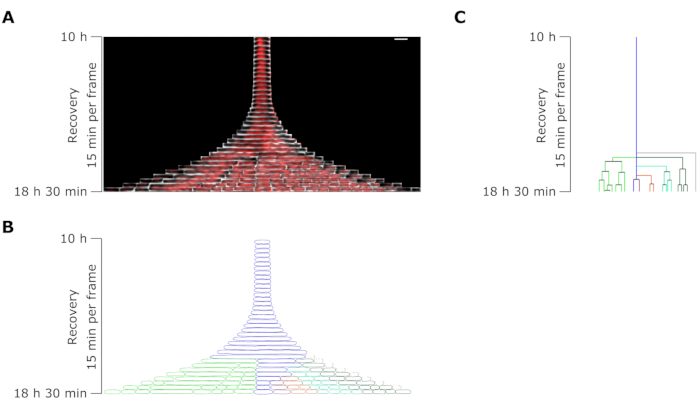
Figura 7: Análisis de lapso de tiempo de microscopía de un persistente representativo a OFX. (A) Cimograma de un persistente OFX representativo y sus células hijas generadas por divisiones de filamentos durante 8.5 h después de la eliminación de OFX (18.5 h después del comienzo del experimento microfluídico, que comprende 4 h de crecimiento, 6 h de tratamiento con OFX de 5 μg·mL−1 y 8.5 h de recuperación después de la eliminación de OFX). Un fotograma corresponde a 15 min. Barra de escala = 5 μm. (B) Máscara generada a partir del cimógrafo persistente en A. La celda persistente monitoreada se indica con un contorno azul, y las celdas hijas se resaltan en colores distintos. (C) Representación esquemática del linaje celular persistente generado a partir de B. El código de colores es idéntico al B. Haga clic aquí para ver una versión más grande de esta figura.
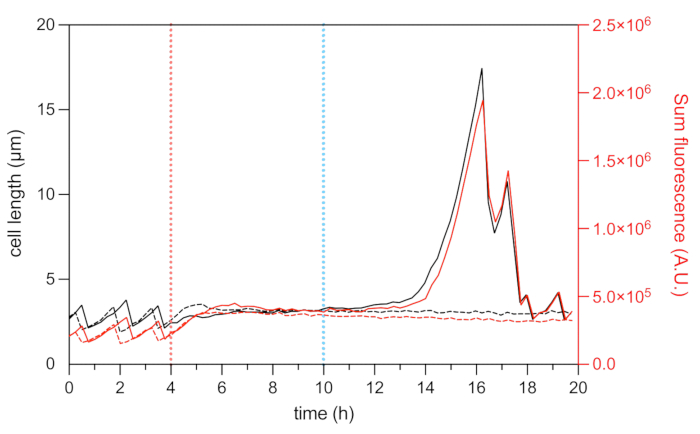
Figura 8: Longitud celular y análisis de fluorescencia mCherry de células persistentes y muertas representativas. Análisis de la longitud celular (eje izquierdo) y la intensidad total de fluorescencia HU-mCherry (eje derecho, mostrado en unidades arbitrarias) de un persistente representativo (líneas sólidas negras y rojas) y una célula muerta representativa (línea negra y roja discontinua) durante el experimento de lapso de tiempo microfluídico. El inicio del tratamiento con OFX (5 μg·mL−1) se indica mediante la línea discontinua roja y la eliminación del OFX mediante la línea discontinua azul. Haga clic aquí para ver una versión más grande de esta figura.
Video 1: Video microfluídico de células HU-mCherry de E. coli antes, durante y después del tratamiento con ofloxacina. Imágenes microfluídicas de lapso de tiempo que muestran células HU-mCherry. Las células se cultivaron durante 4 h en glicerol MOPS 0,4%. Después de 6 h de tratamiento con OFX (5 μg·mL−1), el medio libre de antibióticos se perfundió en la placa microfluídica para permitir que las células persistentes se recuperaran. Barra de escala = 5 μm. Se indica el tiempo (en min). Las fases de crecimiento y recuperación están indicadas por "MOPS- Gly. 0,4%" y el tratamiento OFX por "OFX 5 μg/mL". Haga clic aquí para descargar este video.
| 10x MOPS | |||
| Solución de stock | Volumen de solución madre para 1 L de 10x MOPS | Concentración final en base MOPS 10x | |
| Ácido MOPS | 1 M (ajustado a pH 7,4 usando KOH) | 400 ml | 0,4 m |
| Tricine | 1 M (ajustado a pH 7,4 usando KOH) | 40 ml | 0,04 m |
| FeSO4.7H2O | 0,01 m | 10 ml | 0,0001 M |
| NH4Cl | 1,9 m | 50 ml | 0,095 M |
| K2SO4 | 0,276 millones | 10 ml | 0,00276 millones |
| CaCl2.2H 2O | 0,0005 M | 10 ml | 0,000005 M |
| MgCl 2.6H2O | 0,528 M | 10 ml | 0,00528 millones |
| NaCl | Añadir directamente 29.2 g | 0,5 m | |
| Agua destilada | 460 ml | ||
| Micronutrientes 1000x (ver Tabla 2) | 10 ml | ||
Tabla 1: Composición de 10x MOPS.
| Micronutrientes 1000x | ||
| Concentración en Micronutrimentos 1000x solución madre | Concentración final en base MOPS 10x | |
| (NH4)6Mo7O24.4H2O | 0,000003 m | 0,00000003 M |
| H 3BO3 | 0,0004 M | 0,000004 M |
| CoCl2.6H 2O | 0,00003 millones | 0,0000003 m |
| CuSO4.5H 2O | 0,00001 millones | 0,0000001 m |
| MnCl2.4H 2O | 0,00008 millones | 0,0000008 M |
| ZnSO.7H2O | 0,00001 millones | 0,0000001M |
Tabla 2: Composición de 1.000x micronutrientes.
| MOPS glucosa 0.4% o glicerol MOPS 0.4% | |||
| Solución de stock | Volumen para 1 L MOPS glucosa 0,4 % o glicerol MOPS 0,4 % | Concentración final en glucosa MOPS 0.4% o glicerol MOPS 0.4% | |
| 10x MOPS | véase el cuadro 1. | 100 ml | |
| K2HPO4 | 0,132 m | 10 ml | 0,00132 millones |
| Glucosa (para glucosa MOPS 0,4%) | 20% (20 g en 100 ml de agua destilada) | 20 ml | 0.40% |
| Glicerol (para glicerol MOPS 0,4%) | ≤99% | 4 ml | 0.40% |
| Agua destilada | 870 ml para MOPS glucosa 0.4% o 886 ml para MOPS glicerol 0.4% | ||
Tabla 3: Composición de glucosa MOPS 0,4% y glicerol MOPS 0,4%.
Discusión
El protocolo presentado en este trabajo permite analizar el fenotipo de persistencia observado en respuesta al tratamiento antibiótico a nivel poblacional y unicelular. Los experimentos se realizaron con la cepa MG1655 de E. coli, que se cultivó en un medio químicamente definido (glicerol MOPS 0,4%). Se llevaron a cabo ensayos de muerte temporal y experimentos de microscopía en cultivos de fase exponencial. Utilizamos OFX, una fluoroquinolona, a una concentración de 5 μg·mL−1 para revelar las células persistentes. Los enfoques aquí descritos pueden ser aplicados a otros antibióticos bactericidas, como β-lactámicos, aminoglucósidos o compuestos antimicrobianos31. En consecuencia, se pueden usar otras cepas bacterianas, medios o condiciones de crecimiento. El monitoreo de diferentes fusiones fluorescentes en una configuración similar a la descrita aquí puede ser útil para seguir procesos celulares como la replicación del ADN32, la reparación del ADN25,33 y la división celular34 antes, durante y después del tratamiento con antibióticos. Del mismo modo, los reporteros fluorescentes pueden ser explotados para investigar distintos aspectos de la fisiología celular, como los niveles intracelularesde pH 35, ATP 36 o ROS37. Alternativamente a las fusiones fluorescentes, también se podrían aplicar tintes químicos. Por ejemplo, la fusión hupA-mCherry podría ser reemplazada por 4',6-diamidino-2-fenilindol (DAPI), un colorante fluorescente que tiñe el ADN38. Sin embargo, se debe evitar la realización de microscopía de lapso de tiempo junto con tales tintes fluorescentes, ya que estas técnicas de tinción pueden perturbar la dinámica del ciclo celular durante los experimentos de lapso de tiempo. Alternativamente, tales experimentos pueden ser reemplazados por análisis de curso temporal de imágenes instantáneas en puntos de tiempo relevantes.
Si bien estos reporteros fluorescentes son útiles, no se debe descuidar la cantidad de información que se puede extraer a través del análisis de imágenes de contraste de fase. Aquí, monitoreamos la evolución de la longitud celular a lo largo del crecimiento, el tratamiento OFX y las etapas de recuperación. Otros parámetros basados en imágenes de contraste de fase, como el ancho de la célula, la intensidad del contraste de fase y las curvaturas de las células bacterianas, también se pueden extraer con facilidad utilizando un software adecuado, como MicrobeJ27.
En resumen, el procedimiento descrito aquí puede ser aplicado a otras condiciones y especies bacterianas para monitorear las respuestas celulares a ambientes cambiantes o factores estresantes18,19. Mediante el uso de otros reporteros fluorescentes (reporteros transcripcionales y traslacionales, colorantes químicos) en combinación con un análisis de población, como citometría de flujo / FACS, se pueden abordar preguntas interesantes en un marco multiescala.
Divulgaciones
Los autores declaran que no hay intereses contrapuestos.
Agradecimientos
El trabajo en el laboratorio de Van Melderen está respaldado por las acciones ARC 2018-2023, el Fonds National de la Recherche Scientifique (FNRS CDR J.0182.21F). T.O. es apoyado por una beca ULB. T.S. cuenta con el apoyo de una beca FRIA (FNRS). J.C. cuenta con el apoyo de una beca postdoctoral "chargé de recherches" (FNRS).
Materiales
| Name | Company | Catalog Number | Comments |
| Axio Observer | Zeiss | Inverted fluorescence microscope | |
| CaCl2.2H2O | Merck | 1.02382.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CellASIC ONIX Microfluidic System | Merck | CAX2-S0000 | Microfluidic system |
| CellASIC ONIX2 FG | Merck | ONIX2 1.0.1 | Microfluidic software |
| CellASIC ONIX2 Manifold Basic | Merck | CAX2-MBC20 | Manifold system |
| CoCl2.6H2O | Merck | 1.02539.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CuSO4.5H2O | Merck | 1.02790.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| D-(+)-glucose | Sigma Aldrich | G7021-1KG | Carbon source for MOPS glucose 0.4% growth medium |
| E. coli K-12 MG1655 CF1648 (fnr+) | BE10 | wt reference strain, lab strain | |
| E. coli K-12 MG1655 CF1648 (fnr+) hupA-mCherry::FRT-kan-FRT | BE16 | HU-mCherry fusion integrated via P1 transduction at the native locus, lab strain | |
| FeSO4.7H2O | VWR Chemicals | 24244.232 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Fiji | ImageJ | https://fiji.sc/ | Image software; Schindelin et al. if used in publication |
| Glycerol | Merck | 56815 | Carbon source for MOPS glycerol 0.4% growth medium |
| Greiner CELLSTAR® multiwell culture plate | Merck | M8812-100EA | 24-well clear flat bottom 2ml volume culture plate for automated plate reader |
| H3BO3 | Sigma-Aldrich | B6768-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Hamamatsu ORCA Flash 4.0 digital camera | Hamamatsu | C13440-20CU | Digital Image Acqusition |
| K2HPO4 | Merck | 1.05099.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| KOH | Merck | 1.05029.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| K2SO4 | Merck | 1.05153.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Luria-Broth agar medium | Invitrogen | 22700041 | Growth medium for plating assay |
| Luria-Broth medium | Invitrogen | 12780029 | Growth medium for MIC determination |
| MgCl2.6H2O | Merck | 1.05832.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MgSO4 | Merck | 7487-88-9 | Dilution solution for bacterial survival assay used at 10mM |
| MicrobeJ | Imagej/Fiji plugin | https://www.microbej.com/ | Microscopy image analysis plugin. Ducret et al.for in publication; Detection settings: For bacteria : Area (μm2): 0,1-max; Length (μm): 0,5-max; Width (μm): 0,6-max; Range (μm): 0,5-max; Angularity (rad): 0-0,3; 0-max for all other parameters. |
| Microfluidic Plates CellASIC ONIX | Merck | B04A-03-5PK | Plate for microfluidic system |
| MnCl2.4H2O | Merck | 1.05927.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MOPS, Free Acid ULTROL® Grade | Merck | 475898-500GM | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NaCl | SIgma-Aldrich | S5886-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| (NH4)6Mo7O24.4H2O | Merck | 1.01180.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NH4Cl | Merck | 1.01145.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Ofloxacin | Merck | 82419-36-1 | Fluoroquinolone antibiotic used to treat the bacterial cells |
| Phosphate Buffered Saline (PBS) pH 7.4, 1x | Merck | P3813 | Dilution buffer |
| GENESYS 10S UV-Visible Spectrophotometer | Thermofisher Scientific | 840-208200 | UV-Visible Spectrophotometer, Single/Six Cell Holder with PrinterMeasurement of optical density OD600nm |
| SoftMax Pro | Molecular Devices | Microplate reader software | |
| SpectraMax i3x | Molecular Devices | Microplate reader | |
| N-[Tris(hydroxyméthyl)-méthyl]-glycine | Merck | 1.08602.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Zeiss® immersion Oil 518F | Zeiss | Immersion oil to increase resolution of microscope | |
| Zen3.2 Pro | Zeiss | Microscopic image acquisition and processing software | |
| ZnSO4.7H2O | Merck | 1.08883.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
Referencias
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nature Reviews Microbiology. 17 (7), 441-448 (2019).
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nature Reviews Microbiology. 14 (5), 320-330 (2016).
- Pribis, J. P., Zhai, Y., Hastings, P. J., Rosenberg, S. M. Stress-induced mutagenesis, gambler cells, and stealth targeting antibiotic-induced evolution. mBio. 13 (3), 01074 (2022).
- Molina-Quiroz, R. C., Lazinski, D. W., Camilli, A., Levy, S. B. Transposon-sequencing analysis unveils novel genes involved in the generation of persister cells in uropathogenic Escherichia coli. Antimicrobial Agents and Chemotherapy. 60 (11), 6907-6910 (2016).
- Cameron, D. R., Shan, Y., Zalis, E. A., Isabella, V., Lewis, K. A genetic determinant of persister cell formation in bacterial pathogens. Journal of Bacteriology. 200 (17), 00303-00318 (2018).
- Cui, P., et al. Identification of genes involved in bacteriostatic antibiotic-induced persister formation. Frontiers in Microbiology. 9, 413 (2018).
- Li, T., et al. Identification of novel genes involved in Escherichia coli persistence to tosufloxacin. Frontiers in Cellular and Infection Microbiology. 10, 581986 (2020).
- Verstraeten, N., et al. Obg and membrane depolarization are part of a microbial bet-hedging strategy that leads to antibiotic tolerance. Molecular Cell. 59 (1), 9-21 (2015).
- Hofsteenge, N., van Nimwegen, E., Silander, O. K. Quantitative analysis of persister fractions suggests different mechanisms of formation among environmental isolates of E. coli. BMC Microbiology. 13 (1), 25 (2013).
- Huemer, M., Mairpady Shambat, S., Brugger, S. D., Zinkernagel, A. S. Antibiotic resistance and persistence-Implications for human health and treatment perspectives. EMBO Reports. 21 (12), 51034 (2020).
- Wilmaerts, D., Windels, E. M., Verstraeten, N., Michiels, J. General mechanisms leading to persister formation and awakening. Trends in Genetics. 35 (6), 401-411 (2019).
- Murawski, A. M., Brynildsen, M. P. Ploidy is an important determinant of fluoroquinolone persister survival. Current Biology. 31 (10), 2039-2050 (2021).
- Lemma, A. S., Soto-Echevarria, N., Brynildsen, M. P. Fluoroquinolone persistence in Escherichia coli requires DNA repair despite differing between starving populations. Microorganisms. 10 (2), 286 (2022).
- Balaban, N. Q., Merrin, J., Chait, R., Kowalik, L., Leibler, S. Bacterial persistence as a phenotypic switch. Science. 305 (5690), 1622-1625 (2004).
- Morawska, L. P., Kuipers, O. P. Antibiotic tolerance in environmentally stressed Bacillus subtilis: Physical barriers and induction of a viable but nonculturable state. microLife. 3, (2022).
- Windels, E. M., et al. Enrichment of persisters enabled by a ß-lactam-induced filamentation method reveals their stochastic single-cell awakening. Communications Biology. 2 (1), 426 (2019).
- Leygeber, M., et al. Analyzing microbial population heterogeneity-Expanding the toolbox of microfluidic single-cell cultivations. Journal of Molecular Biology. 431 (23), 4569-4588 (2019).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli growth rate to osmotic shock. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7807-7812 (2014).
- Shi, H., et al. Starvation induces shrinkage of the bacterial cytoplasm. Proceedings of the National Academy of Sciences of the United States of America. 118 (24), 2104686118 (2021).
- Goode, O., et al. Persister Escherichia coli cells have a lower intracellular pH than susceptible cells but maintain their pH in response to antibiotic treatment. mBio. 12 (4), 00909-00921 (2021).
- Manuse, S., et al. Bacterial persisters are a stochastically formed subpopulation of low-energy cells. PLoS Biology. 19 (4), 3001194 (2021).
- Fisher, J. K., et al. Four-dimensional imaging of E. coli nucleoid organization and dynamics in living cells. Cell. 153 (4), 882-895 (2013).
- Cabral, D., Wurster, J., Belenky, P. Antibiotic persistence as a metabolic adaptation: Stress, metabolism, the host, and new directions. Pharmaceuticals. 11 (1), 14 (2018).
- Wiegand, I., Hilpert, K., Hancock, R. E. W. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nature Protocols. 3 (2), 163-175 (2008).
- Goormaghtigh, F., Van Melderen, L. Single-cell imaging and characterization of Escherichia coli persister cells to ofloxacin in exponential cultures. Science Advances. 5 (6), (2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ducret, A., Quardokus, E. M., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nature Microbiology. 1 (7), 16077 (2016).
- Odsbu, I., Skarstad, K. DNA compaction in the early part of the SOS response is dependent on RecN and RecA. Microbiology. 160 (5), 872-882 (2014).
- Pham, T. D. M., Ziora, Z. M., Blaskovich, M. A. T. Quinolone antibiotics. MedChemComm. 10 (10), 1719-1739 (2019).
- Drlica, K., et al. Quinolones: Action and resistance updated. Current Topics in Medicinal Chemistry. 9 (11), 981-998 (2009).
- Kohanski, M. A., Dwyer, D. J., Collins, J. J. How antibiotics kill bacteria: From targets to networks. Nature Reviews Microbiology. 8 (6), 423-435 (2010).
- Reyes-Lamothe, R., Sherratt, D. J., Leake, M. C. Stoichiometry and architecture of active DNA replication machinery in Escherichia coli. Science. 328 (5977), 498-501 (2010).
- Lesterlin, C., Ball, G., Schermelleh, L., Sherratt, D. J. RecA bundles mediate homology pairing between distant sisters during DNA break repair. Nature. 506 (7487), 249-253 (2014).
- Alexeeva, S., Gadella, T. W. J., Verheul, J., Verhoeven, G. S., Den Blaauwen, T. Direct interactions of early and late assembling division proteins in Escherichia coli cells resolved by FRET: Bacterial division studied by spectral FRET. Molecular Microbiology. 77 (2), 384-398 (2010).
- Miesenböck, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Yaginuma, H., et al. Diversity in ATP concentrations in a single bacterial cell population revealed by quantitative single-cell imaging. Scientific Reports. 4, 6522 (2014).
- Ermakova, Y. G., et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide. Nature Communications. 5, 5222 (2014).
- Kapuscinski, J. DAPI: A DNA-specific fluorescent probe. Biotechnic & Histochemistry. 70 (5), 220-233 (1995).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados