Method Article
Analyse de population et de cellules uniques de la persistance des antibiotiques chez Escherichia coli
Dans cet article
Résumé
La persistance des antibiotiques décrit la capacité de petites sous-populations au sein d’une population isogénique sensible à tolérer transitoirement de fortes doses d’antibiotiques bactéricides. Le présent protocole combine des approches pour caractériser le phénotype de persistance des antibiotiques aux niveaux moléculaire et cellulaire après exposition d’Escherichia coli à des doses létales d’ofloxacine.
Résumé
La persistance des antibiotiques fait référence à la capacité de petites sous-populations bactériennes à tolérer transitoirement de fortes doses d’antibiotiques bactéricides. Lors d’un traitement antibiotique bactéricide, la majeure partie de la population bactérienne est rapidement tuée. Cette première phase rapide de destruction est suivie d’une diminution substantielle du taux de destruction car les cellules persistantes restent viables. Classiquement, la persistance est déterminée au niveau de la population par des tests de temps/destruction effectués avec de fortes doses d’antibiotiques et pour des durées d’exposition définies. Bien que cette méthode fournisse des informations sur le niveau de cellules persistantes et la cinétique de destruction, elle ne reflète pas l’hétérogénéité intrinsèque de cellule à cellule sous-jacente au phénomène de persistance. Le protocole décrit ici combine des tests classiques temps/destruction avec l’analyse unicellulaire utilisant la microscopie à fluorescence en temps réel. En utilisant des rapporteurs fluorescents appropriés, l’imagerie microscopique des cellules vivantes peut fournir des informations sur les effets de l’antibiotique sur les processus cellulaires, tels que la réplication et la ségrégation chromosomiques, l’élongation cellulaire et la division cellulaire. La combinaison de l’analyse de population et de cellules uniques permet de caractériser moléculairement et cellulairement le phénotype de persistance.
Introduction
Ce protocole vise à analyser le phénotype de persistance bactérienne en réponse à un traitement antibiotique spécifique au niveau de la cellule unique et de la population. La persistance décrit la capacité de petites sous-populations au sein d’une population isogénique à supporter de fortes doses d’antibiotiques bactéricides (fluoroquinolones, aminoglycosides, β-lactamines, etc.), la concentration minimale inhibitrice (CMI) des cellules dites persistantes étant identique à celle de la majeure partie de la population. La dynamique de destruction biphasique, lors de la mesure de la survie bactérienne au fil du temps en présence d’un antibiotique, révèle la présence de cellules tolérantes transitoirement aux médicaments, avec une éradication rapide initiale des cellules non persistantes, suivie d’un taux de destruction beaucoup plus lent des cellules persistantes. Lors de l’élimination des antibiotiques, ces cellules donnent naissance à une population génétiquement identique qui présente une dynamique de destruction similaire lorsqu’elle est traitée avec le même antibiotique 1,2. Contrairement à la persistance, la résistance aux antibiotiques est définie au niveau de la population et est généralement la conséquence de mutations de novo ou du transfert horizontal de gènes d’un plasmide conférant une résistance3. Alors que les mutations responsables de la résistance sont principalement localisées dans la cible du médicament ou dans les régions promotrices des pompes d’efflux du médicament, les gènes modifiant la fréquence de persistance identifiés par des approches d’analyse des mutants ciblées et à l’échelle du génome se sont révélés nombreux et diversifiés 2,3,4,5,6,7,8 . Par conséquent, il est probable que les cellules bactériennes puissent entrer dans l’état persistant par de multiples voies 9,10,11, et des approches pour étudier le phénomène de persistance au niveau de la cellule unique sont nécessaires pour caractériser la physiologie de ces cellules persistantes.
Le développement récent d’outils microfluidiques utilisés en combinaison avec la microscopie à fluorescence a ouvert la voie à la caractérisation du phénotype de persistance et a mis en évidence le rôle de processus cellulaires clés, tels que la réplication chromosomique12, la réparation de l’ADN13 et la division cellulaire14, dans la formation de cellules persistantes. Dans cet article, nous décrivons une approche intégrée combinant des tests de microbiologie classiques avec l’imagerie vivante unicellulaire pour caractériser les cellules persistantes générées dans des cultures d’Escherichia coli à croissance exponentielle traitées avec une forte dose d’ofloxacine. Le protocole décrit ici peut être appliqué pour étudier le phénomène de persistance des antibiotiques chez d’autres espèces bactériennes, telles que Bacillus subtilis15, ou des conditions (par exemple, la persistance des antibiotiques après un traitement β-lactamines 16) et peut facilement être modifié pour étudier les nombreux phénomènes impliquant une hétérogénéité phénotypique17,18,19 . En outre, la configuration décrite dans cet article peut être combinée avec d’autres rapporteurs fluorescents pour étudier des paramètres cellulaires distincts d’intérêt, tels que les niveaux intracellulaires de pH20 ou d’ATP21 au niveau de la cellule unique, ce qui peut potentiellement produire de nouvelles informations sur le phénomène de persistance des antibiotiques.
Protocole
REMARQUE : Utilisez de la verrerie de culture stérile, des pointes de pipette et un milieu de croissance. Ici, les cellules d’E. coli ont été cultivées dans un milieu chimiquement défini à faible autofluorescence (voir le tableau des matériaux). Les inoculations ont été effectuées en présence d’un brûleur Bunsen afin de minimiser le risque de contamination.
1. Culture cellulaire et courbe de croissance
- Étaler la souche d’intérêt d’un stock de glycérol congelé sur une plaque de gélose Luria-Bertaini (LB) (complétée par un antibiotique sélectif, si nécessaire) et incuber à 37 °C pendant une nuit (entre 15 h et 19 h) pour obtenir des colonies uniques.
NOTE: L’expérience présentée ici utilise deux souches, E. coli K-12 MG1655 correspondant à la souche poids et la souche isogénique MG1655 hupA-mCherry 22. Cette dernière souche exprime la α-sous-unité marquée par fluorescence de la protéine associée aux nucléoïdes HU. Le rapporteur hupA-mCherry est intégré au lieu natif de hupA. HU-mCherry sert de proxy pour suivre la dynamique nucléoïde dans les cellules vivantes lorsqu’il se lie à l’ADN d’une manière non spécifique. - Inoculer 5 mL de milieu (ici, milieu à base d’acide 3-[N-morpholino]propanesulfonique [MOPS]; Tableau 1, Tableau 2 et Tableau 3) complété par du glucose à 0,4 % et un antibiotique sélectif (si nécessaire) avec une colonie isolée dans un tube de verre (≥25 mL), et placer le tube dans un incubateur à agitation réglé à 37 °C et 180 rotations par minute (tr/min) pendant la nuit (entre 15 h et 19 h). Alternativement, des tubes en plastique, en verre ou des flacons en plastique (≥ 25 mL) peuvent être utilisés à la place des tubes en verre.
NOTE: Un milieu MOPS supplémenté en glycérol à une concentration finale de 0,4% a été utilisé tout au long des expériences décrites dans cet article, à l’exception des cultures de nuit qui ont été effectuées dans MOPS glucose 0,4%. Le temps de génération d’E. coli dans MOPS supplémenté en glucose est plus court que celui dans MOPS glycérol. L’utilisation de MOPS glucose 0,4% au lieu de MOPS glycérol 0,4% à cette étape garantit que les cellules atteignent la phase stationnaire dans les 19 heures. D’autres milieux de croissance, tels que M9 ou milieu riche défini (RDM) complété par des sources de carbone distinctes, peuvent également être utilisés. Il faut cependant noter que le taux de croissance et la fréquence de persistance sont différents selon le milieu et/ou le carbone utilisé23. - Le lendemain matin, centrifuger 1 mL de culture à 2 300 x g pendant 3 min, jeter le surnageant et remettre délicatement la pastille en suspension dans le même volume de solution saline tamponnée au phosphate (PBS). Mesurer la densité optique à 600 nm (DO 600 nm) et calculer le volume nécessaire pour une DOinitiale de 600 nm de 0,01 dans un volume final de 2 mL.
- Placer 2 mL de milieu de glycérol MOPS à 0,4 % dans un puits d’une plaque à fond clair de 24 puits et inoculer avec le volume de culture calculé pendant la nuit. Placer la plaque de 24 puits dans un lecteur de microplaques automatisé (voir le tableau des matériaux) pour surveiller la DO600 nm pendant 24 h. Réglez le lecteur de microplaques pour mesurer la DO600 nm toutes les 15 minutes à une température de 37 °C et avec une rotation orbitale élevée (140 tr/min).
NOTE: Si la souche utilisée dans l’expérience code un rapporteur fluorescent, assurez-vous que son taux de croissance est comparable à celui du poids pour éviter tout artefact dans les expériences ultérieures, car le taux de croissance affecte la fréquence de persistance des antibiotiques23. En raison des limites de détection à très faibles densités cellulaires et des effets potentiels spécifiques à la souche sur la relation OD600 nm/unités formant colonies (UFC), il est recommandé d’évaluer la cinétique de croissance en surveillant la UFC-mL-1 lorsque l’on travaille avec des souches non caractérisées.
2. Détermination de la concentration minimale inhibitrice des antibiotiques
NOTE: La concentration minimale inhibitrice (CMI) est définie comme la dose la plus faible d’antibiotique à laquelle aucune croissance bactérienne n’est observée. La détermination de la CMI doit être effectuée pour chaque antibiotique et souche. Dans les expériences décrites ici, l’antibiotique fluoroquinolone ofloxacine (OFX) a été utilisé. La détermination de la CMI permet de confirmer que la solution antibiotique a été correctement préparée, que l’antibiotique est actif et que les souches sont également sensibles à l’antibiotique. Ici, la méthode de dilution de gélose publiée a été réalisée pour déterminer la CMI à OFX des différentes souches utilisées24. La CMI d’un antibiotique donné pour une souche bactérienne donnée peut également être déterminée par la méthode de dilution du bouillon24.
- Préparation des tôles pour la détermination de la CMI
- Préparer une solution mère pour l’antibiotique utilisé dans les expériences en dissolvant 5 mg d’OFX dans 1 mL d’eau ultrapure. Ajouter 20 μL de HCl à 37% pour augmenter la solubilité de l’OFX.
- Faire fondre 100 mL de gélose LB stérile et la conserver à 55 °C pour éviter la solidification. Préparer six petites fioles en verre (25 mL) et pipeter 5 mL de gélose LB liquide dans chaque fiole à l’aide d’une pipette stérile.
- Diluer 10 μL de la solution mère OFX à 5 mg·mL-1 dans 90 μL d’eau ultrapure (dilution 1:10 du stock principal). Ajouter 0 μL, 2 μL, 4 μL, 6 μL, 8 μL ou 10 μL de la solution OFX de 500 μg·mL-1 à chacun des six flacons en verre contenant 5 mL de milieu gélosé LB pour générer un milieu gélosé LB avec des concentrations finales de 0 μg·mL−1, 0,02 μg·mL−1, 0,04 μg·mL−1, 0,06 μg·mL−1, 0,08 μg·mL−1 et 0,1 μg·mL-1 OFX, respectivement. Mélangez la solution en tournant les flacons plusieurs fois.
REMARQUE : Si la CMI de l’antibiotique d’intérêt est connue, la plage de concentrations devrait aller de bas en bas à au-dessus de la CMI réelle. Si la CMI est inconnue, une large gamme de concentrations avec une série de dilutions logarithmique2 est recommandée. Assurez-vous que le milieu gélosé LB a refroidi avant d’ajouter l’antibiotique, car des températures élevées peuvent l’inactiver. Néanmoins, il est important de ne pas laisser le milieu gélosé LB se solidifier avant d’ajouter l’antibiotique, car cela pourrait conduire à une distribution non homogène de l’antibiotique dans le milieu gélosé LB. - Verser 5 mL de chacun des six milieux gélose LB préparés à l’étape 2.1.3 de manière croissante dans une plaque de culture à 6 puits à l’aide d’une pipette stérile. Laisser la gélose refroidir jusqu’à solidification et sécher la plaque avant utilisation.
NOTE: Préparez la solution antibiotique et la plaque de culture le jour où le test est effectué.
- Dosage de détermination de la CMI
- Inoculer 5 mL de milieu LB avec une colonie isolée dans un tube de verre (≥25 mL) et placer le tube de verre dans un incubateur à agitation réglé à 37 °C et 180 tr/min pendant la nuit (entre 15 h et 19 h).
- Le lendemain matin, mesurer la DO600 nm et diluer la culture dans un tube à essai jusqu’à obtenir une densité cellulaire finale de 1 x 107 UFC·mL-1 dans du PBS. Pour les souches testées ici, 1 x 107 UFC·mL−1 correspond à une DO600 nm de 0,0125.
NOTE: Si la corrélation entre l’UFC·mL-1 et la DO 600 nm nm n’est pas connue, la courbe de croissance déterminée par les mesures UFC et OD 600 nm doit être établie pour calculer le facteur de corrélation entre l’UFC·mL-1 et DO600 nm. - Repérez 2 μL de la culture préalablement diluée sur chaque puits de la plaque séchée à 6 puits. Laisser sécher les taches avant de placer la plaque dans un incubateur à 37 °C pendant la nuit (entre 15 h et 19 h).
- Le lendemain, comptez les colonies formées dans chaque puits. La CMI correspond au puits avec la concentration minimale d’antibiotique où aucune croissance bactérienne n’est détectée.
3. Essai ponctuel
NOTE: La méthode de dosage ponctuel est une approche qualitative qui permet d’estimer le nombre de cellules viables (cellules capables de générer des colonies après un stress antibiotique). Le test ponctuel est effectué avant le test time-kill afin de fournir des informations sur la viabilité de la souche utilisée dans les conditions testées et d’informer sur les dilutions nécessaires pendant l’essai time-kill (voir rubrique 4).
- Pour préparer les plaques de gélose LB pour les dosages localisés, verser 50 ml de gélose LB dans une boîte de Petri carrée (144 cm2). Préparez une boîte de Petri carrée par point temporel. Laissez la gélose LB se solidifier et séchez les plaques avant utilisation.
- Inoculer 5 mL de milieu (glucose MOPS 0,4 %) avec une colonie isolée dans un tube de verre (≥25 mL) et placer le tube dans un incubateur à agitation réglé à 37 °C et 180 tr/min pendant la nuit (entre 15 h et 19 h).
- Le lendemain, mesurer la DO600 nm et diluer la culture dans un milieu frais ajusté en température (37 °C, MOPS glycérol 0,4%) dans un tube de verre (≥25 mL) jusqu’à une DO finale de600 nm de ~0,001 . Laisser pousser la culture pendant la nuit dans un incubateur à 37 °C à 180 rpm (entre 15 h et 19 h).
- Le lendemain, mesurer la DO 600 nm et incuber la culture à une DOfinale de 600 nm de 0,3.
- Pendant l’incubation, préparer des plaques de 96 puits en plaçant 90 μL de solution de MgSO4 0,01 M dans chaque puits, sauf les puits de la première rangée (rangée A). Les plaques à 96 puits seront utilisées pour une dilution en série 10 fois à l’étape 3.7.
NOTE: Dans cette expérience, deux souches (wt et hupA-mCherry ) ont été testées en triple à sept moments différents. Comme une plaque comprend 12 colonnes, une plaque peut être utilisée pour deux points temporels et, par conséquent, un total de quatre plaques ont été préparées. - À une DO de600 nm de 0,3, prélever 200 μL de chaque culture bactérienne. Ces échantillons correspondent au point de temps t0 (non traité) avant le traitement antibiotique et permettent de déterminer l’UFC·mL-1 avant le traitement antibiotique.
- Centrifuger les échantillons à 2 300 x g pendant 3 min. Pendant le temps de centrifugation, ajouter la concentration souhaitée d’OFX à la culture liquide et continuer à incuber à 37 °C en agitant.
NOTE: Ici, OFX a été utilisé à une concentration de 5 μg·mL-1 (correspondant à la CMI multipliée par 83). Dans une étude antérieure, cette concentration a été utilisée pour caractériser le phénomène de persistance sous exposition à OFX25. La concentration d’antibiotiques utilisée pour les tests de temps/destruction peut varier en fonction de l’antibiotique, du milieu de culture et de l’état de croissance bactérienne. - Après centrifugation, remettre en suspension la pastille de la cellule dans 200 μL de solution 0,01 M deMgSO4 . Placer 100 μL dans le puits vide de la plaque de 96 puits préparée à l’étape 3.5. Effectuer une dilution en transférant 10 μL du puits de la rangée A dans le puits de la rangée B contenant 90 μL de 0,01 M deMgSO4. Poursuivre les dilutions en série en transférant 10 μL du puits de la rangée B dans le puits de la rangée C. Répéter jusqu’à obtention d’une dilution de 10−7 (chaque transfert étant une dilution de 10 fois).
NOTE: Dans cette expérience, six cultures (trois pour la souche wt et trois pour la souche hupA-mCherry ) ont été testées pour chaque point temporel. Selon le protocole, une pipette à canal multiple est utilisée pour effectuer les dilutions en série pour les six souches en même temps. Pour minimiser l’erreur technique, les pointes des pipettes sont changées entre chaque transfert. - Placer 10 μL de chaque dilution sur les plaques de gélose LB préparées à l’étape 3.1.
NOTA: Une pipette multicanal est utilisée pour repérer simultanément la même dilution (par exemple, une dilution 10−4 ) pour les six cultures. Pendant le repérage, la première butée de la pipette multicanal doit être utilisée sans appuyer sur la deuxième butée, car cela peut entraîner la distribution de microgouttelettes sur la plaque. - Aux moments pertinents après l’ajout d’antibiotiques, prélever 200 μL de la culture et effectuer des dilutions en série comme décrit à l’étape 3.8. Repérer 10 μL de chaque dilution comme décrit à l’étape 3.9.
NOTE: Pour l’expérience décrite ici, sept points temporels ont été collectés (t0, t1h, t2h, t3h, t4h, t5h, t6h). Pour les souches présentant une sensibilité accrue aux antibiotiques par rapport au poids, plusieurs lavages dans MgSO4 0,01 M peuvent être effectués pour éliminer les antibiotiques résiduels. - Incuber les plaques à 37 °C pendant la nuit (entre 15 h et 19 h). Le lendemain, compter le nombre de colonies aux deux dilutions les plus élevées pour lesquelles des colonies peuvent être détectées. Idéalement, les taches sur les plaques de gélose pour ces dilutions contiennent entre 3 et 30 colonies permettant la détermination précise de l’UFC·mL-1 pour chaque échantillon.
- Calculer le rapport de survie en divisant l’UFC·mL-1 calculée de chaque point temporel par l’UFC·mL-1 de la population initiale à t0.
4. Tests time-kill
REMARQUE: Alors que les tests ponctuels sont une méthode facile à utiliser pour estimer le taux de survie d’une souche donnée pour un antibiotique donné, les tests time-kill donnent un taux de survie à plus haute résolution et sont effectués pour quantifier avec précision la viabilité bactérienne. Le profil de la courbe de destruction peut être utilisé pour déterminer si une souche bactérienne donnée est sensible, tolérante ou résistante à l’antibiotique dans une condition donnée. De plus, les tests time-kill permettent de déterminer le temps d’exposition aux antibiotiques nécessaire pour détecter le phénomène de persistance (début de la deuxième pente de la courbe de destruction biphasique) ainsi que la fréquence de persistance.
- Préparer les plaques de gélose LB pour le test de placage time-killing. Verser 25 ml de gélose LB dans une boîte de Petri (±57 cm2). Préparer au moins deux boîtes de Petri par point de temps (deux dilutions par point de temps et par souche sont plaquées).
- Laissez la gélose LB se solidifier et séchez les assiettes avant d’ajouter cinq à huit billes de verre stériles à chaque plaque. Retourner et étiqueter les plaques en fonction de la déformation/condition/point temporel.
NOTE: Les billes de verre permettent la propagation des bactéries sur les plaques de gélose au cours des étapes 4.7 et 4.9. Alternativement, les cellules peuvent être dispersées sur la plaque de gélose à l’aide d’un épandeur. - Pour chaque échantillon, préparer 10 tubes en verre de dilution en série contenant 900 μL de solution 0,01 M deMgSO4 . Le nombre de tubes de verre de dilution par échantillon à préparer correspond aux dilutions nécessaires pour détecter les colonies dans le test localisé.
- Inoculer 5 mL de milieu (glucose MOPS 0,4 %) avec une colonie isolée dans un tube de verre (≥25 mL) et placer le tube dans un incubateur à agitation réglé à 37 °C et 180 tr/min pendant la nuit (entre 15 h et 19 h).
- Après 16 h de croissance, mesurer la DO 600 nm et diluer la culture dans un milieu frais ajusté en température (37 °C, MOPS glycérol 0,4 %) dans un tube de verre jusqu’à obtenir une DO finale de600 nm de ~0,001. Laisser pousser la culture pendant la nuit dans un incubateur à 37 °C à 180 rpm (entre 15 h et 19 h).
- Le lendemain, mesurer la DO 600 nm et incuber la culture à une DOfinale de 600 nm de 0,3.
- A une DO 600 nm de 0,3, prélever 100 μL de la culture, diluer selon les données obtenues dans le spot (à une DO600 nm de 0,3 et sans antibiotique, une dilution 10−5 donne généralement 200-300 colonies), et plaquer100 μL sur les plaques de gélose LB préparées aux étapes 4.1-4.2. Secouez doucement les assiettes pour éviter que les billes ne touchent les bords de la boîte, car cela peut entraîner une propagation non homogène des cellules bactériennes sur le milieu gélosé LB. Ce premier échantillon avant le traitement antibiotique correspond au point de temps t0 (UFC·mL-1 avant traitement antibiotique).
- Ajouter la concentration désirée d’OFX et continuer à incuber à 37 °C en agitant.
NOTE: Ici, OFX a été utilisé à une concentration de 5 μg·mL-1 (correspondant à la CMI multipliée par 83). - Aux moments pertinents après l’ajout d’antibiotiques, prélever 100 μL de la culture, diluer selon les données obtenues dans le dosage local, et plaquer 100 μL sur les plaques de gélose LB préparées aux étapes 4.1-4.2. Plaquez les cellules comme décrit à l’étape 4.7.
NOTE: Pour l’expérience décrite ici, sept points temporels ont été collectés (t0, t1h, t2h, t3h, t4h, t5h, t6h). Pour les souches présentant une sensibilité accrue aux antibiotiques par rapport au poids, plusieurs lavages dans MgSO4 0,01 M peuvent être effectués pour éliminer les antibiotiques résiduels. - Incuber les plaques à 37 °C pendant la nuit (entre 15 h et 19 h). Le lendemain, compter le nombre de colonies aux deux dilutions les plus élevées pour lesquelles des colonies peuvent être détectées. Idéalement, les plaques devraient contenir 30 à 300 colonies pour permettre une détermination précise de l’UFC·mL-1 dans chaque échantillon.
- Calculer le rapport de survie en normalisant l’UFC·mL-1 à chaque point temporel par l’UFC·mL-1 à t0. Tracer le logarithme10 UFC·mL-1 normalisé en fonction du temps.
5. Imagerie microfluidique en microscopie accélérée
REMARQUE: La section suivante décrit la préparation de la plaque microfluidique ainsi que la procédure d’acquisition d’images et d’analyse d’images en accéléré. Le but de cette expérience est d’observer et d’analyser le phénotype de persistance lors d’un traitement antibiotique au niveau unicellulaire. Les données recueillies au cours de cette expérience peuvent être utilisées pour générer un large éventail de résultats en fonction de la question abordée et/ou des rapporteurs fluorescents utilisés pendant l’expérience. Dans l’expérience décrite ici, une analyse quantitative de la longueur de la cellule et de la fluorescence HU-mCherry22, reflétant l’organisation nucléoïde dans les cellules persistantes et non persistantes, a été réalisée.
- Culture cellulaire bactérienne pour microscopie accélérée microfluidique
- Inoculer 5 mL de milieu (MOPS glycérol 0,4 %, complété par un antibiotique sélectif si nécessaire) avec une colonie isolée dans un tube de verre (≥25 mL), et placer le tube dans un incubateur à agitation réglé à 37 °C et 180 tr/min pendant la nuit (entre 15 h et 19 h).
- Le lendemain, mesurer la DO 600 nm et diluer la culture dans un milieu frais ajusté en température (37 °C, MOPS glycérol 0,4%) dans un tube en verre jusqu’à obtenir une DO finale de600 nm de ~0,001. Laisser pousser la culture pendant la nuit (entre 15 h et 19 h) dans un incubateur à agitation à 37 °C et 180 rpm pour obtenir une culture précoce en phase exponentielle le lendemain.
- Préparation de la plaque microfluidique et imagerie microscopique time-lapse
REMARQUE: Les expériences microfluidiques peuvent être effectuées dans des dispositifs microfluidiques disponibles dans le commerce (comme décrit ici) ou dans des systèmes microfluidiques produits en interne.- Retirer la solution de conservation (si présente) de chaque puits de la plaque microfluidique et la remplacer par un milieu de culture frais.
NOTA : Si la plaque microfluidique contient un puits de sortie d’eaux usées, la solution de conservation du puits de sortie doit être retirée mais non remplacée par le milieu. - Scellez la plaque microfluidique avec le système collecteur en cliquant sur le bouton Seal ou via le logiciel microfluidique (sélectionnez d’abord Outil, puis Plaque d’étanchéité).
REMARQUE: Pour sceller la plaque, une pression uniforme doit être appliquée à la plaque et au collecteur en pressant manuellement la plaque contre le collecteur. Si cela est effectué correctement, la note « scellé » devrait apparaître sur l’interface ONIX2. Il est important de ne pas appliquer de pression sur la lame de verre pour éviter tout risque potentiel de rupture du collecteur. - Une fois scellé, effectuez une première séquence d’amorçage (cliquez sur Run Liquid Priming Sequence sur l’interface du logiciel microfluidique).
REMARQUE : La séquence d’amorçage liquide d’exécution correspond à 5 min de perfusion à 6,9 kPa pour les puits 1 à 5, suivies de 5 min de perfusion à 6,9 kPa pour le puits 8 et d’une dernière ronde de perfusion pour le puits 6 pendant 5 min à 6,9 kPa. La séquence d’amorçage du liquide Run permet d’éliminer la solution de conservation qui peut encore être présente dans les canaux reliant les différents puits. - Incuber la plaque dans une armoire thermostatique du microscope à la température souhaitée (ici, 37 °C) pendant au moins 2 h avant le début de l’imagerie microscopique.
- Commencez une deuxième séquence d’amorçage liquide avant de commencer l’expérience .
- Scellez la plaque microfluidique en cliquant sur Seal off sur l’interface du logiciel microfluidique. Remplacer le milieu dans le puits 1 et le puits 2 par 200 μL de milieu frais, dans le puits 3 par 200 μL de milieu frais contenant l’antibiotique (ici, OFX à 5 μg·mL−1), dans le puits 4 et le puits 5 avec 200 μL de milieu frais, dans le puits 6 avec 200 μL de milieu frais, et dans le puits 8 avec 200 μL de l’échantillon de culture (à partir de l’étape 5.1.2) dilué à une DO600 nm de 0,01 dans le milieu frais.
- Scellez la plaque microfluidique comme décrit à l’étape 5.2.2 et placez la plaque sur l’objectif du microscope à l’intérieur de l’armoire de microscope.
REMARQUE: Assurez-vous de placer une goutte d’huile d’immersion sur l’objectif du microscope avant de placer la plaque microfluidique. - Dans le logiciel microfluidique, cliquez sur Chargement de cellule pour permettre le chargement de la cellule dans la plaque microfluidique.
NOTA : L’étape de chargement des cellules comprend 15 s de perfusion à 13,8 kPa pour le puits 8, suivies de 15 s de perfusion à 27,6 kPa pour le puits 6 et le puits 8, et d’une dernière ronde de perfusion pour le puits 6 pendant 30 s à 6,9 kPa. La densité des cellules dans la plaque microfluidique est critique pour l’expérience. La première partie de ce protocole microfluidique consiste à cultiver des bactéries pendant 6 h dans un milieu frais avant le traitement antibiotique. Après 6 h de croissance, la densité cellulaire doit être suffisante pour détecter les cellules persistantes rares (dans les conditions utilisées dans cette étude, les cellules persistantes génèrent à une fréquence de 10−4). Si la densité cellulaire est trop élevée, il est difficile de distinguer les cellules individuelles, ce qui empêche une analyse précise d’une seule cellule. Comme le taux de croissance dépend directement du milieu, la densité des cellules dans les champs de microscopie doit être évaluée avant de lancer l’expérience. - Réglez une mise au point optimale à l’aide du mode de lumière transmise et sélectionnez plusieurs régions d’intérêt (ROI) où un nombre de cellules approprié est observé (jusqu’à 300 cellules par champ).
REMARQUE: Sélectionnez au moins 40 ROI pour vous assurer que les cellules persistantes rares sont imagées. - Sur le logiciel microfluidique, cliquez sur Créer un protocole. Programmer l’injection de milieu frais à 6,9 kPa pendant 6 h (puits 1-2), suivie de l’injection du milieu contenant l’antibiotique à 6,9 kPa pendant 6 h (puits 3), et, enfin, l’injection du milieu frais à 6,9 kPa pendant 20 h (puits 4-5).
NOTE: Une culture bactérienne diluée à une OD600 nm de 0,01 permet aux bactéries de se développer dans la chambre microfluidique pendant 6 h, assurant que les cellules sont dans la phase de croissance exponentielle. En fonction du nombre de cellules introduites dans le dispositif microfluidique lors de l’étape de chargement (voir étape 5.2.8), la durée de la phase de croissance peut être adaptée pour obtenir jusqu’à 300 cellules par ROI. La persistance étant un phénomène rare, l’augmentation du nombre de cellules par ROI améliore la possibilité d’observer les cellules persistantes. Le nombre de cellules ne doit cependant pas dépasser 300 cellules par retour sur investissement, car cela rend l’analyse unicellulaire fastidieuse. - Effectuez l’imagerie microscopique en mode time-lapse avec une image toutes les 15 minutes en utilisant la lumière transmise et la source lumineuse d’excitation pour le rapporteur fluorescent. Ici, une source lumineuse d’excitation de 560 nm pour le signal mCherry a été utilisée (LED 580 nm à 10% de puissance avec filtre 00 [530-585 ex, 615LP em, Zeiss] et exposition de 100 ms pour mCherry). Le logiciel Zen3.2 compatible Zeiss a été utilisé pour l’imagerie cellulaire.
- Retirer la solution de conservation (si présente) de chaque puits de la plaque microfluidique et la remplacer par un milieu de culture frais.
- Analyse d’images
NOTE: L’ouverture et la visualisation des images de microscopie sont effectuées avec le logiciel open source ImageJ/Fiji (https://fiji.sc/)26. L’analyse quantitative des images est effectuée à l’aide du logiciel open source ImageJ/Fiji et du plugin gratuit MicrobeJ (https://microbej.com)27. Dans ce protocole, la version MicrobeJ 5.13I(14) a été utilisée.- Ouvrez le logiciel ImageJ/Fiji sur l’ordinateur et faites glisser les images de microscopie time-lapse hyperstack dans la barre de chargement Fiji. Utilisez Image > Color > Make Composite pour fusionner les différents canaux de l’hyperpile. Si les couches de l’expérience accélérée ne correspondent pas à la couleur souhaitée (par exemple, le contraste de phase est affiché en rouge au lieu de gris), utilisez Image > Couleur > Organiser les couches pour appliquer la couleur appropriée aux couches.
- Ouvrez le plugin MicrobeJ et détectez les cellules bactériennes à l’aide de l’interface d’édition manuelle. Supprimez les cellules détectées automatiquement et délimitez manuellement les cellules persistantes d’intérêt image par image.
REMARQUE: Différents paramètres peuvent être utilisés pour détecter automatiquement des cellules individuelles. La détection manuelle a été utilisée ici car les cellules persistantes analysées forment de longs filaments, qui sont rarement détectés correctement à l’aide de la détection automatique. - Après la détection, utilisez l’icône Résultat dans l’interface d’édition manuelle MicrobeJ pour générer une table ResultJ. Enregistrez le fichier ResultJ et utilisez la table ResultJ pour obtenir des informations sur les différents paramètres d’intérêt de l’analyse unicellulaire. Dans le cas de ce protocole, la fluorescence moyenne de l’intensité HU-mCherry, la longueur de la cellule et la surface cellulaire des cellules individuelles ont été exportées.
Résultats
Comme décrit ci-dessus, les souches utilisées pour l’analyse phénotypique unicellulaire des cellules persistantes ont été caractérisées dans le milieu de glycérol MOPS à 0,4%. La surveillance de la DO600 nm au fil du temps n’a montré aucune différence entre les souches en poids et hupA-mCherry (Figure 1). Cela indique que l’expression de la protéine de fusion HU-mCherry n’a pas eu d’impact sur la croissance dans ces conditions. Les cellules bactériennes des deux souches initialement inoculées à une DO600 nm de 0,01 ont atteint la phase exponentielle ±8 h après l’inoculation.
La CMI de l’OFX a été déterminée par des méthodes normalisées (ici, dilution de gélose en série)24. La CMI est définie comme la concentration minimale où aucune croissance visible n’est détectée. La CMI de l’OFX pour les deux souches a été établie à 0,06 μg·mL−1, ce qui indique que la fusion hupA-mCherry n’a eu aucun effet sur la sensibilité à l’OFX par rapport à la souche isogénique en poids (Figure 2).
Nous avons en outre déterminé l’effet d’un traitement OFX mortel (CMI 83 fois) sur la viabilité des deux souches utilisées dans cette étude. Comme le nombre de cellules viables diminue avec le temps avec l’exposition à l’OFX, les dilutions des cultures bactériennes doivent être ajustées de manière appropriée pour atteindre 30 à 300 colonies par plaque. Pour déterminer les dilutions appropriées au fil du temps, un essai ponctuel a été effectué, où 10 μL de 0 à 10−7 dilutions en série de 10 fois ont été placées sur des boîtes de Petri carrées à l’aide d’une pipette multicanal. Les dilutions appropriées étaient celles où des clones isolés étaient visibles (par exemple, à t0 = 10−5, t1h = 10−4/10−3, t4h = 10−2/10−1) (figure 3).
Bien que le test ponctuel soit une méthode facile pour mieux comprendre la cinétique de la mise à mort médiée par OFX, il ne parvient pas à déterminer avec précision la dynamique de mise à mort. Lorsque la viabilité des cellules à croissance exponentielle traitées avec OFX a été surveillée par le test time-kill, une courbe biphasique typique a été observée (Figure 4). La première pente de la courbe reflète la mise à mort rapide de la population non persistante (ligne pointillée rouge). Dans les conditions testées ici, jusqu’à 99,9% des cellules ont été incapables de former des colonies après 3 h en présence d’OFX. Cette première phase de mise à mort est suivie d’une deuxième phase, montrant un taux de destruction plus lent (ligne pointillée bleue), qui révèle la présence de cellules persistantes tolérantes aux médicaments. Dans les conditions testées, la phase persistante a commencé environ 3 h après l’ajout d’OFX, soulignant la nécessité d’exposer les cellules à l’OFX pendant plus de 3 heures pour étudier les phénotypes persistants. Il est important de noter que la courbe time-kill montre que la protéine de fusion hupA-mCherry n’a eu aucun effet sur la cinétique time-kill. La souche codant pour la fusion fluorescente translationnelle peut donc être utilisée pour surveiller les cellules persistantes à l’aide de la microscopie à fluorescence.
Nous avons ensuite étudié le phénomène de persistance au niveau de la cellule unique. Pour ce faire, la souche hupA-mCherry a été introduite dans une plaque microfluidique, ce qui a permis de changer les conditions du milieu (ici, croissance, traitement et récupération) tout en effectuant une microscopie time-lapse sur un retour sur investissement donné. Au cours de la première étape de l’expérience microfluidique, les cellules introduites dans le dispositif microfluidique ont été perfusées avec un milieu de croissance (glycérol MOPS 0,4%) et divisées avec un temps de génération de ~2 h (Figure 5 et Figure 6). Cette première phase de croissance indique que les cellules étaient viables et se divisent activement avant le traitement OFX.
Après cette première phase de croissance, les cellules ont été perfusées avec un milieu de croissance supplémenté en 5 μg·mL−1 OFX pendant 6 h. Dès que l’antibiotique a atteint les cellules, la division cellulaire a été bloquée (Figure 5 et Figure 6). Après 6 h de traitement OFX, les cellules ont été perfusées avec du milieu frais. Alors que la grande majorité des cellules étaient incapables de reprendre leur croissance (Figure 5 et Figure 6), une petite sous-population de bactéries était capable de s’allonger et de générer des cellules filamenteuses25. Ces cellules, qui ont pu se diviser et générer des cellules filles viables après le traitement OFX, peuvent être définies comme les cellules persistantes.
Comme cette configuration permet de visualiser les cellules persistantes avant, pendant et après le traitement, elle fournit non seulement des informations sur le phénotype persistant pendant la phase de récupération, mais aussi sur l’état physiologique des cellules persistantes avant le traitement (Figure 6). Dans les conditions testées, les cellules persistantes se sont divisées de manière similaire aux cellules non persistantes avant le traitement OFX, ce qui indique que les cellules persistantes observées ne provenaient pas d’une sous-population dormante (Figure 6)25.
L’analyse de la longueur cellulaire des cellules persistantes pendant la phase de récupération a révélé que chaque filament avait un taux d’allongement spécifique. La longueur cellulaire atteinte par chaque persistant avant la première division différait d’un persistant à l’autre. De même, le moment de l’événement de première division était très hétérogène (Figure 6). Le filament persistant en division a généré plusieurs cellules filles, qui ont commencé à croître et à se diviser, pour la plupart, de la même manière que les cellules non traitées (Figure 7). La division successive du filament a ensuite entraîné une diminution progressive de la longueur de la cellule, donnant finalement naissance à des cellules filles de longueur cellulaire similaire à celle d’avant le traitement OFX (Figure 6 et Figure 7B). La grande majorité des cellules étaient incapables d’induire la filamentation après l’élimination de l’OFX. Cette grande population cellulaire correspond aux cellules mortes (Figure 5 et Figure 6).
La fusion fluorescente de la protéine HU associée au nucléoïde permet de visualiser la dynamique du nucléoïde22. L’analyse de l’intensité de fluorescence totale de HU-mCherry dans la cellule peut être utilisée comme approximation de la teneur en ADN22,25. Pendant la phase de croissance (avant le traitement OFX), l’intensité totale de fluorescence mCherry variait, reflétant la dynamique de la réplication et de la ségrégation chromosomiques au cours du cycle cellulaire (Figure 8). Après l’ajout d’OFX, la fluorescence mCherry a augmenté au niveau de la cellule médiane, ce qui indique un compactage nucléoïde, qui s’est avéré induit par la formation de cassures d’ADN double brin28 (Figure 5). Les cassures d’ADN double brin sont une conséquence du mécanisme d’action de l’OFX, qui corrompt les topoisomérases de type II ADNA-gyrase et topoisomérase IV29,30. Chez E. coli, l’ADN-gyrase est la cible principale de l’OFX29,30. En liant sa cible à une étape critique du mécanisme de passage double brin, OFX inhibe la relégation des brins d’ADN clivés, conduisant finalement à la libération de cassures d’ADN double brin30. Comme décrit ci-dessus, les cellules persistantes au traitement OFX ont commencé à filamenter pendant la récupération25 (Figure 6). L’augmentation de la longueur de la cellule est corrélée à une augmentation de l’intensité totale de fluorescence mCherry, ce qui reflète le redémarrage de la réplication et une augmentation de l’abondance des nucléoïdes dans le filament25 (Figure 7a et Figure 8). Pour les cellules mortes, l’intensité totale de fluorescence mCherry est restée stable pendant le traitement et pendant la phase de récupération, indiquant que ces cellules étaient incapables de répliquer leurs chromosomes après l’élimination de l’OFX (Figure 8). Une vidéo microfluidique (vidéo 1) de cellules E. coli HU-mCherry avant, pendant et après le traitement à l’ofloxacine est également montrée.
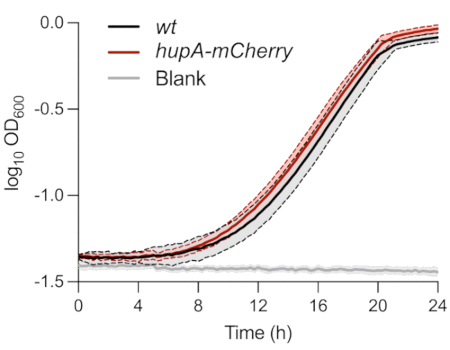
Figure 1 : Surveillance de la croissance des souches d’E. coli en poids et hupA-mCherry. Surveillance de la densité optique (OD600 nm) de poids (noir) et hupA-mCherry (rouge). Les nuances et les lignes pointillées indiquent les écarts-types des triplicates biologiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
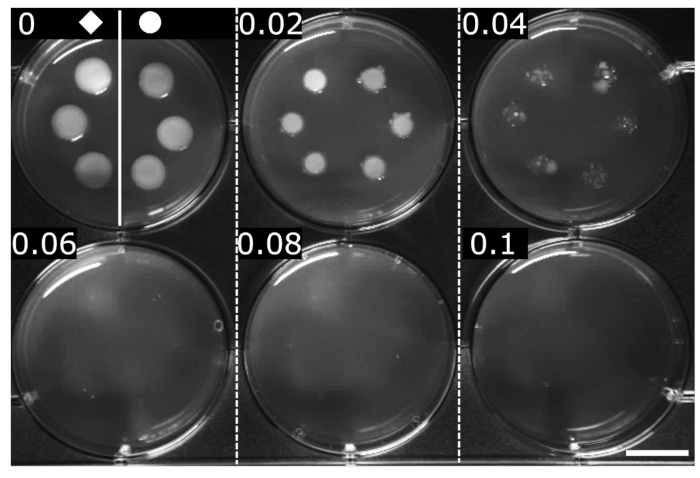
Figure 2 : Détermination de la CMI de l’OFX pour les souches d’E. coli en poids et hupA-mCherry. Le poids () et le hupA-mCherry (●) ont été cultivés en milieu LB, et 2 μL ont été repérés sur des dilutions en série de gélose LB contenant de l’OFX (♦ concentration indiquée dans chaque panel en μg·mL−1). L’inhibition de la croissance est visible à un minimum de 0,06 μg·mL−1. La figure est une expérience représentative de triplicates biologiques. Barre d’échelle = 1 cm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
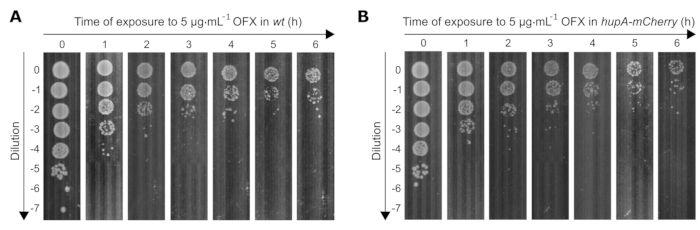
Figure 3 : Essai ponctuel des souches d’E. coli en poids et hupA-mCherry lors de l’exposition à l’OFX. Les souches (A) wt et (B) hupA-mCherry ont été cultivées dans du glycérol MOPS à 0,4% comme décrit dans le protocole (rubrique 3), et les cellules à croissance exponentielle (OD600 nm = 0,3) ont été traitées avec 5 μg·mL−1 OFX. T0 correspond au point de temps avant l’ajout d’OFX. T1, T2, T3, T4, T5 et T6 correspondent à 1-6 h après l’ajout d’OFX. La figure est une expérience représentative de triplicates biologiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
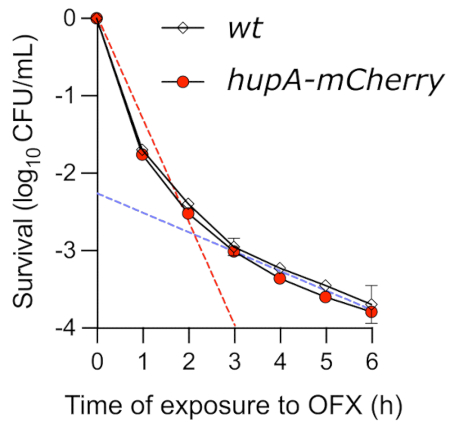
Figure 4 : Essai de destruction temporelle des souches d’E. coli wt et hupA-mCherry lors de l’exposition à l’OFX. Les souches wt () et hupA-mCherry (●) ont été cultivées dans du glycérol MOPS à 0,4% comme décrit dans le protocole (rubrique 4), et les cellules à croissance exponentielle (♦ OD600 nm = 0,3) ont été traitées avec 5 μg·mL−1 OFX. Les lignes pointillées indiquent la première phase de mise à mort « rapide » (rouge) et la deuxième phase de mise à mort « lente » (bleue), correspondant aux sous-populations sensibles et persistantes (obtenues par régression linéaire entre T0 et T2, ainsi qu’entre T3 et T6, respectivement). Les barres d’erreur indiquent les écarts-types des triplicates biologiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
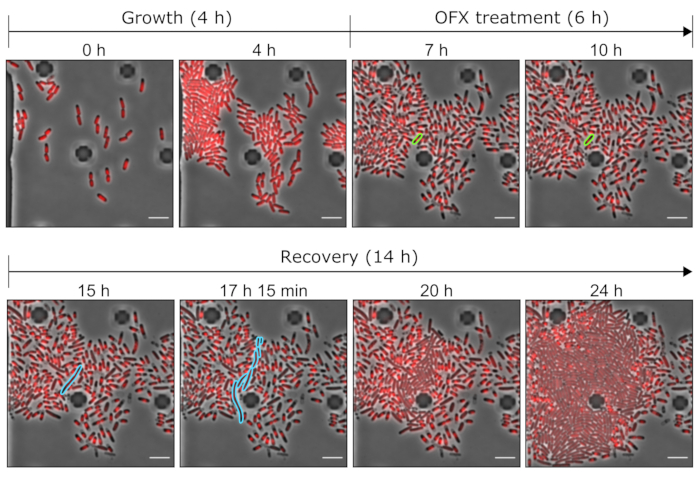
Figure 5 : Images représentatives du persistant OFX et des cellules mortes à l’aide d’outils microfluidiques. Images de microscopie représentatives montrant les points temporels pertinents de l’expérience microfluidique réalisée avec la souche hupA-mCherry (contraste de phase en gris, signal HU-mCherry en rouge). Les cellules exprimant le hupA-mCherry marqué ont été cultivées dans une plaque microfluidique (ici, 4 h), suivie d’une provocation OFX (5 μg·mL−1). Après 6 h en présence d’OFX, les cellules ont été perfusées avec un milieu frais, permettant aux cellules persistantes de récupérer. La cellule persistante et ses cellules de descendance pendant le traitement OFX et après l’élimination de l’OFX sont surlignées en vert et en bleu, respectivement. Les points de temps correspondants sont indiqués sur chaque panneau. Barre d’échelle = 5 μm. Veuillez cliquer ici pour voir une version agrandie de cette figure.
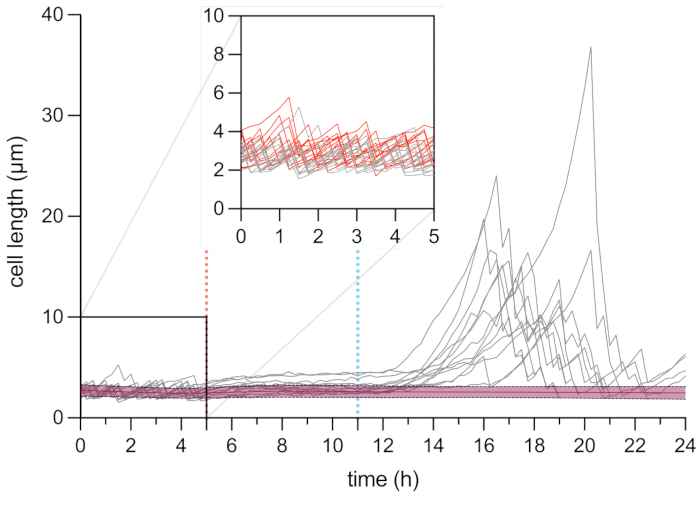
Figure 6 : Analyse en accéléré microscopique de la longueur des cellules persistantes et mortes. Analyse de la longueur cellulaire des cellules mortes (en rouge, n = 109) et des cellules persistantes (en gris, n = 13). Le début du traitement OFX (5 μg·mL−1) est indiqué par la ligne pointillée rouge (5 h), et l’élimination de l’OFX est indiquée par la ligne pointillée bleue. L’encart correspond à la phase de croissance avant l’ajout d’OFX. Les expériences ont été réalisées en trois exemplaires. Les nuances et les lignes pointillées indiquent les écarts-types pour la population de cellules mortes (n = 109). Veuillez cliquer ici pour voir une version agrandie de cette figure.
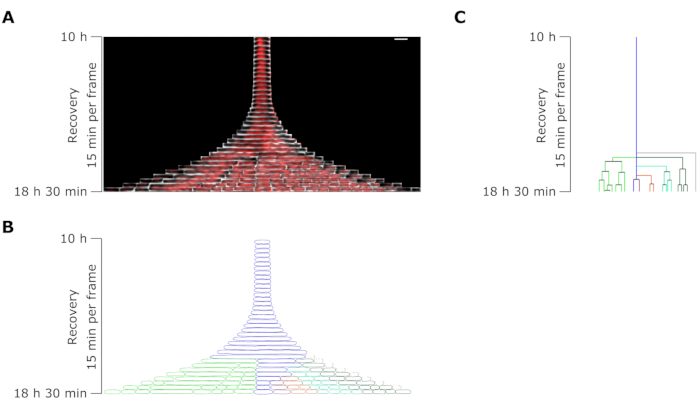
Figure 7 : Analyse en accéléré par microscopie d’un persistant représentatif de l’OFX. (A) Kymographe d’un persistant OFX représentatif et de ses cellules filles généré par divisions filamentaires pendant 8,5 h après l’élimination de l’OFX (18,5 h après le début de l’expérience microfluidique, comprenant 4 h de croissance, 6 h de traitement OFX de 5 μg·mL−1 et 8,5 h de récupération après élimination de l’OFX). Une image correspond à 15 min. Barre d’échelle = 5 μm. (B) Masque généré à partir du kymographe persistant dans A. La cellule persistante surveillée est indiquée par un contour bleu et les cellules filles sont surlignées dans des couleurs distinctes. (C) Représentation schématique de la lignée cellulaire persistante générée à partir de B. Le code couleur est identique à B. Veuillez cliquer ici pour voir une version agrandie de cette figure.
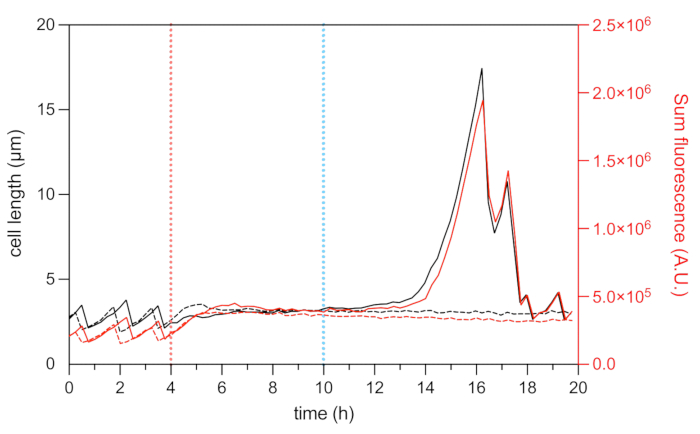
Figure 8 : Longueur cellulaire et analyse de fluorescence mCherry des cellules persistantes et mortes représentatives. Analyse de la longueur de la cellule (axe de gauche) et de l’intensité totale de fluorescence HU-mCherry (axe droit, en unités arbitraires) d’un persistant représentatif (lignes noires et rouges continues) et d’une cellule morte représentative (ligne noire et rouge pointillée) au cours de l’expérience de time-lapse microfluidique. Le début du traitement OFX (5 μg·mL−1) est indiqué par la ligne pointillée rouge et l’élimination de l’OFX par la ligne pointillée bleue. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Vidéo 1 : Vidéo microfluidique des cellules d’E. coli HU-mCherry avant, pendant et après le traitement à l’ofloxacine. Imagerie microfluidique time-lapse montrant des cellules HU-mCherry. Les cellules ont été cultivées pendant 4 h dans MOPS glycérol 0,4%. Après 6 h de traitement OFX (5 μg·mL−1), le milieu sans antibiotique a été perfusé dans la plaque microfluidique pour permettre aux cellules persistantes de récupérer. Barre d’échelle = 5 μm. Le temps (en min) est indiqué. Les phases de croissance et de récupération sont indiquées par « MOPS- Gly. 0,4% » et le traitement OFX par « OFX 5 μg/mL ». Veuillez cliquer ici pour télécharger cette vidéo.
| 10x MOPS | |||
| Solution mère | Volume de solution mère pour 1 L de 10x MOPS | Concentration finale dans 10x base MOPS | |
| Acide MOPS | 1 M (ajusté à pH 7,4 avec KOH) | 400 mL | 0,4 M |
| Tricine | 1 M (ajusté à pH 7,4 avec KOH) | 40 mL | 0,04 M |
| FeSO4.7H2O | 0,01 M | 10 mL | 0,0001 M |
| NH4Cl | 1,9 M | 50 mL | 0,095 M |
| K2SO4 | 0,276 M | 10 mL | 0,00276 M |
| CaCl2.2H 2O | 0,0005 M | 10 mL | 0,000005 M |
| MgCl 2.6H2O | 0,528 M | 10 mL | 0,00528 M |
| NaCl | Ajouter directement 29,2 g | 0,5 M | |
| Eau distillée | 460 mL | ||
| Micronutriments 1000x (voir tableau 2) | 10 mL | ||
Tableau 1 : Composition de 10x MOPS.
| Micronutriments 1000x | ||
| Concentration dans la solution mère de micronutriments 1000x | Concentration finale dans 10x base MOPS | |
| (NH4)6Mo7O24.4H2O | 0,000003 M | 0,00000003 M |
| H3 BO3 | 0,0004 M | 0,000004 M |
| CoCl2.6H 2O | 0,00003 M | 0,0000003 M |
| CuSO4.5H 2O | 0,00001 M | 0,0000001 M |
| MnCl2.4H 2O | 0,00008 M | 0,0000008 M |
| ZnSO.7H2O | 0,00001 M | 0,0000001M |
Tableau 2 : Composition de 1 000x micronutriments.
| MOPS glucose 0,4% ou MOPS glycérol 0,4% | |||
| Solution mère | Volume pour 1 L de glucose MOPS 0,4 % ou MOPS glycérol 0,4 % | Concentration finale en glucose MOPS 0,4% ou glycère MOPS 0,4% | |
| 10x MOPS | voir tableau 1 | 100 mL | |
| K2HPO4 | 0,132 M | 10 mL | 0,00132 M |
| Glucose (pour MOPS glucose 0,4%) | 20 % (20 g dans 100 ml d’eau distillée) | 20 mL | 0.40% |
| Glycérol (pour MOPS glycérol 0,4%) | ≤99 % | 4 mL | 0.40% |
| Eau distillée | 870 mL pour la glycémie MOPS 0,4 % ou 886 mL pour le glycérol MOPS 0,4 % | ||
Tableau 3 : Composition du glucose MOPS 0,4% et du glycérol MOPS 0,4%.
Discussion
Le protocole présenté dans cet article permet l’analyse du phénotype de persistance observé en réponse à un traitement antibiotique à l’échelle de la population et de la cellule unique. Les expériences ont été réalisées avec la souche E. coli MG1655, qui a été cultivée dans un milieu chimiquement défini (glycérol MOPS 0,4%). Des essais de destruction temporelle et des expériences de microscopie ont été réalisés sur des cultures en phase exponentielle. Nous avons utilisé OFX, une fluoroquinolone, à une concentration de 5 μg·mL−1 pour révéler les cellules persistantes. Les approches décrites ici peuvent être appliquées à d’autres antibiotiques bactéricides, tels que les β-lactamines, les aminosides ou les composés antimicrobiens31. En conséquence, d’autres souches bactériennes, milieux ou conditions de croissance peuvent être utilisés. La surveillance de différentes fusions fluorescentes dans une configuration similaire à celle décrite ici peut être utile pour suivre des processus cellulaires tels que la réplication de l’ADN 32, la réparation de l’ADN25,33 et la division cellulaire34 avant, pendant et après le traitement antibiotique. De même, les rapporteurs fluorescents peuvent être exploités pour étudier des aspects distincts de la physiologie cellulaire, tels que les niveaux intracellulaires de pH35, d’ATP 36 ou de ROS37. Alternativement aux fusions fluorescentes, des colorants chimiques pourraient également être appliqués. Par exemple, la fusion hupA-mCherry pourrait être remplacée par le 4',6-diamidino-2-phénylindole (DAPI), un colorant fluorescent qui colore l’ADN38. La réalisation de microscopies time-lapse couplées à de tels colorants fluorescents doit cependant être évitée, car ces techniques de coloration peuvent perturber la dynamique du cycle cellulaire pendant les expériences en accéléré. Alternativement, de telles expériences peuvent être remplacées par des analyses de parcours temporels de l’imagerie instantanée à des points temporels pertinents.
Bien que ces rapporteurs fluorescents soient utiles, la quantité d’informations pouvant être extraites par l’analyse d’images à contraste de phase ne doit pas être négligée. Ici, nous avons surveillé l’évolution de la longueur des cellules tout au long de la croissance, du traitement OFX et des étapes de récupération. D’autres paramètres basés sur des images de contraste de phase, tels que la largeur des cellules, l’intensité du contraste de phase et les courbures des cellules bactériennes, peuvent également être extraits facilement à l’aide d’un logiciel adéquat, tel que MicrobeJ27.
En résumé, la procédure décrite ici peut être appliquée à d’autres conditions et espèces bactériennes pour surveiller les réponses cellulaires aux environnements changeants ou aux facteurs de stress18,19. En utilisant d’autres rapporteurs fluorescents (rapporteurs transcriptionnels et translationnels, colorant chimique) en combinaison avec une analyse de population, telle que la cytométrie en flux / FACS, des questions intéressantes peuvent être abordées dans un cadre multi-échelle.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent.
Remerciements
Les travaux du laboratoire Van Melderen sont soutenus par les actions ARC 2018-2023, le Fonds National de la Recherche Scientifique (FNRS CDR J.0182.21F). T.O. est soutenu par une bourse de l’ULB. T.S. est soutenu par une bourse FRIA (FNRS). J.C. est soutenu par un post-doctorat chargé de recherches (FNRS).
matériels
| Name | Company | Catalog Number | Comments |
| Axio Observer | Zeiss | Inverted fluorescence microscope | |
| CaCl2.2H2O | Merck | 1.02382.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CellASIC ONIX Microfluidic System | Merck | CAX2-S0000 | Microfluidic system |
| CellASIC ONIX2 FG | Merck | ONIX2 1.0.1 | Microfluidic software |
| CellASIC ONIX2 Manifold Basic | Merck | CAX2-MBC20 | Manifold system |
| CoCl2.6H2O | Merck | 1.02539.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CuSO4.5H2O | Merck | 1.02790.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| D-(+)-glucose | Sigma Aldrich | G7021-1KG | Carbon source for MOPS glucose 0.4% growth medium |
| E. coli K-12 MG1655 CF1648 (fnr+) | BE10 | wt reference strain, lab strain | |
| E. coli K-12 MG1655 CF1648 (fnr+) hupA-mCherry::FRT-kan-FRT | BE16 | HU-mCherry fusion integrated via P1 transduction at the native locus, lab strain | |
| FeSO4.7H2O | VWR Chemicals | 24244.232 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Fiji | ImageJ | https://fiji.sc/ | Image software; Schindelin et al. if used in publication |
| Glycerol | Merck | 56815 | Carbon source for MOPS glycerol 0.4% growth medium |
| Greiner CELLSTAR® multiwell culture plate | Merck | M8812-100EA | 24-well clear flat bottom 2ml volume culture plate for automated plate reader |
| H3BO3 | Sigma-Aldrich | B6768-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Hamamatsu ORCA Flash 4.0 digital camera | Hamamatsu | C13440-20CU | Digital Image Acqusition |
| K2HPO4 | Merck | 1.05099.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| KOH | Merck | 1.05029.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| K2SO4 | Merck | 1.05153.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Luria-Broth agar medium | Invitrogen | 22700041 | Growth medium for plating assay |
| Luria-Broth medium | Invitrogen | 12780029 | Growth medium for MIC determination |
| MgCl2.6H2O | Merck | 1.05832.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MgSO4 | Merck | 7487-88-9 | Dilution solution for bacterial survival assay used at 10mM |
| MicrobeJ | Imagej/Fiji plugin | https://www.microbej.com/ | Microscopy image analysis plugin. Ducret et al.for in publication; Detection settings: For bacteria : Area (μm2): 0,1-max; Length (μm): 0,5-max; Width (μm): 0,6-max; Range (μm): 0,5-max; Angularity (rad): 0-0,3; 0-max for all other parameters. |
| Microfluidic Plates CellASIC ONIX | Merck | B04A-03-5PK | Plate for microfluidic system |
| MnCl2.4H2O | Merck | 1.05927.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MOPS, Free Acid ULTROL® Grade | Merck | 475898-500GM | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NaCl | SIgma-Aldrich | S5886-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| (NH4)6Mo7O24.4H2O | Merck | 1.01180.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NH4Cl | Merck | 1.01145.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Ofloxacin | Merck | 82419-36-1 | Fluoroquinolone antibiotic used to treat the bacterial cells |
| Phosphate Buffered Saline (PBS) pH 7.4, 1x | Merck | P3813 | Dilution buffer |
| GENESYS 10S UV-Visible Spectrophotometer | Thermofisher Scientific | 840-208200 | UV-Visible Spectrophotometer, Single/Six Cell Holder with PrinterMeasurement of optical density OD600nm |
| SoftMax Pro | Molecular Devices | Microplate reader software | |
| SpectraMax i3x | Molecular Devices | Microplate reader | |
| N-[Tris(hydroxyméthyl)-méthyl]-glycine | Merck | 1.08602.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Zeiss® immersion Oil 518F | Zeiss | Immersion oil to increase resolution of microscope | |
| Zen3.2 Pro | Zeiss | Microscopic image acquisition and processing software | |
| ZnSO4.7H2O | Merck | 1.08883.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
Références
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nature Reviews Microbiology. 17 (7), 441-448 (2019).
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nature Reviews Microbiology. 14 (5), 320-330 (2016).
- Pribis, J. P., Zhai, Y., Hastings, P. J., Rosenberg, S. M. Stress-induced mutagenesis, gambler cells, and stealth targeting antibiotic-induced evolution. mBio. 13 (3), 01074 (2022).
- Molina-Quiroz, R. C., Lazinski, D. W., Camilli, A., Levy, S. B. Transposon-sequencing analysis unveils novel genes involved in the generation of persister cells in uropathogenic Escherichia coli. Antimicrobial Agents and Chemotherapy. 60 (11), 6907-6910 (2016).
- Cameron, D. R., Shan, Y., Zalis, E. A., Isabella, V., Lewis, K. A genetic determinant of persister cell formation in bacterial pathogens. Journal of Bacteriology. 200 (17), 00303-00318 (2018).
- Cui, P., et al. Identification of genes involved in bacteriostatic antibiotic-induced persister formation. Frontiers in Microbiology. 9, 413 (2018).
- Li, T., et al. Identification of novel genes involved in Escherichia coli persistence to tosufloxacin. Frontiers in Cellular and Infection Microbiology. 10, 581986 (2020).
- Verstraeten, N., et al. Obg and membrane depolarization are part of a microbial bet-hedging strategy that leads to antibiotic tolerance. Molecular Cell. 59 (1), 9-21 (2015).
- Hofsteenge, N., van Nimwegen, E., Silander, O. K. Quantitative analysis of persister fractions suggests different mechanisms of formation among environmental isolates of E. coli. BMC Microbiology. 13 (1), 25 (2013).
- Huemer, M., Mairpady Shambat, S., Brugger, S. D., Zinkernagel, A. S. Antibiotic resistance and persistence-Implications for human health and treatment perspectives. EMBO Reports. 21 (12), 51034 (2020).
- Wilmaerts, D., Windels, E. M., Verstraeten, N., Michiels, J. General mechanisms leading to persister formation and awakening. Trends in Genetics. 35 (6), 401-411 (2019).
- Murawski, A. M., Brynildsen, M. P. Ploidy is an important determinant of fluoroquinolone persister survival. Current Biology. 31 (10), 2039-2050 (2021).
- Lemma, A. S., Soto-Echevarria, N., Brynildsen, M. P. Fluoroquinolone persistence in Escherichia coli requires DNA repair despite differing between starving populations. Microorganisms. 10 (2), 286 (2022).
- Balaban, N. Q., Merrin, J., Chait, R., Kowalik, L., Leibler, S. Bacterial persistence as a phenotypic switch. Science. 305 (5690), 1622-1625 (2004).
- Morawska, L. P., Kuipers, O. P. Antibiotic tolerance in environmentally stressed Bacillus subtilis: Physical barriers and induction of a viable but nonculturable state. microLife. 3, (2022).
- Windels, E. M., et al. Enrichment of persisters enabled by a ß-lactam-induced filamentation method reveals their stochastic single-cell awakening. Communications Biology. 2 (1), 426 (2019).
- Leygeber, M., et al. Analyzing microbial population heterogeneity-Expanding the toolbox of microfluidic single-cell cultivations. Journal of Molecular Biology. 431 (23), 4569-4588 (2019).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli growth rate to osmotic shock. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7807-7812 (2014).
- Shi, H., et al. Starvation induces shrinkage of the bacterial cytoplasm. Proceedings of the National Academy of Sciences of the United States of America. 118 (24), 2104686118 (2021).
- Goode, O., et al. Persister Escherichia coli cells have a lower intracellular pH than susceptible cells but maintain their pH in response to antibiotic treatment. mBio. 12 (4), 00909-00921 (2021).
- Manuse, S., et al. Bacterial persisters are a stochastically formed subpopulation of low-energy cells. PLoS Biology. 19 (4), 3001194 (2021).
- Fisher, J. K., et al. Four-dimensional imaging of E. coli nucleoid organization and dynamics in living cells. Cell. 153 (4), 882-895 (2013).
- Cabral, D., Wurster, J., Belenky, P. Antibiotic persistence as a metabolic adaptation: Stress, metabolism, the host, and new directions. Pharmaceuticals. 11 (1), 14 (2018).
- Wiegand, I., Hilpert, K., Hancock, R. E. W. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nature Protocols. 3 (2), 163-175 (2008).
- Goormaghtigh, F., Van Melderen, L. Single-cell imaging and characterization of Escherichia coli persister cells to ofloxacin in exponential cultures. Science Advances. 5 (6), (2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ducret, A., Quardokus, E. M., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nature Microbiology. 1 (7), 16077 (2016).
- Odsbu, I., Skarstad, K. DNA compaction in the early part of the SOS response is dependent on RecN and RecA. Microbiology. 160 (5), 872-882 (2014).
- Pham, T. D. M., Ziora, Z. M., Blaskovich, M. A. T. Quinolone antibiotics. MedChemComm. 10 (10), 1719-1739 (2019).
- Drlica, K., et al. Quinolones: Action and resistance updated. Current Topics in Medicinal Chemistry. 9 (11), 981-998 (2009).
- Kohanski, M. A., Dwyer, D. J., Collins, J. J. How antibiotics kill bacteria: From targets to networks. Nature Reviews Microbiology. 8 (6), 423-435 (2010).
- Reyes-Lamothe, R., Sherratt, D. J., Leake, M. C. Stoichiometry and architecture of active DNA replication machinery in Escherichia coli. Science. 328 (5977), 498-501 (2010).
- Lesterlin, C., Ball, G., Schermelleh, L., Sherratt, D. J. RecA bundles mediate homology pairing between distant sisters during DNA break repair. Nature. 506 (7487), 249-253 (2014).
- Alexeeva, S., Gadella, T. W. J., Verheul, J., Verhoeven, G. S., Den Blaauwen, T. Direct interactions of early and late assembling division proteins in Escherichia coli cells resolved by FRET: Bacterial division studied by spectral FRET. Molecular Microbiology. 77 (2), 384-398 (2010).
- Miesenböck, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Yaginuma, H., et al. Diversity in ATP concentrations in a single bacterial cell population revealed by quantitative single-cell imaging. Scientific Reports. 4, 6522 (2014).
- Ermakova, Y. G., et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide. Nature Communications. 5, 5222 (2014).
- Kapuscinski, J. DAPI: A DNA-specific fluorescent probe. Biotechnic & Histochemistry. 70 (5), 220-233 (1995).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon