Method Article
Análise Populacional e Unicelular da Persistência de Antibióticos em Escherichia coli
Neste Artigo
Resumo
A persistência de antibióticos descreve a capacidade de pequenas subpopulações dentro de uma população isogênica sensível de tolerar transitoriamente altas doses de antibióticos bactericidas. O presente protocolo combina abordagens para caracterizar o fenótipo de persistência do antibiótico em nível molecular e celular após exposição de Escherichia coli a doses letais de ofloxacina.
Resumo
A persistência de antibióticos refere-se à capacidade de pequenas subpopulações bacterianas tolerarem transitoriamente altas doses de antibióticos bactericidas. Após o tratamento com antibióticos bactericidas, a maior parte da população bacteriana é rapidamente morta. Esta primeira fase rápida de matança é seguida por uma diminuição substancial na taxa de matança, uma vez que as células persistentes permanecem viáveis. Classicamente, a persistência é determinada em nível populacional por ensaios de tempo/morte realizados com altas doses de antibióticos e por tempos de exposição definidos. Embora este método forneça informações sobre o nível de células persistentes e a cinética de morte, ele falha em refletir a heterogeneidade intrínseca célula-célula subjacente ao fenômeno de persistência. O protocolo aqui descrito combina ensaios clássicos de tempo/morte com análise de célula única usando microscopia de fluorescência em tempo real. Usando repórteres fluorescentes apropriados, a imagem de microscopia de células vivas pode fornecer informações sobre os efeitos do antibiótico em processos celulares, como replicação e segregação cromossômica, alongamento celular e divisão celular. A combinação de análise populacional e unicelular permite a caracterização molecular e celular do fenótipo de persistência.
Introdução
Este protocolo tem como objetivo analisar o fenótipo de persistência bacteriana em resposta ao tratamento antibiótico específico em nível unicelular e populacional. A persistência descreve a capacidade de pequenas subpopulações dentro de uma população isogênica suportarem altas doses de antibióticos bactericidas (fluoroquinolonas, aminoglicosídeos, β-lactâmicos, etc.), sendo a concentração inibitória mínima (CIM) das chamadas células persistentes idêntica à da maior parte da população. A dinâmica de morte bifásica, ao medir a sobrevivência bacteriana ao longo do tempo na presença de um antibiótico, revela a presença de células transitoriamente tolerantes a drogas, com uma rápida erradicação inicial das células não persistentes, seguida por uma taxa de morte muito mais lenta das células persistentes. Após a remoção do antibiótico, essas células dão origem a uma população geneticamente idêntica que apresenta dinâmica de morte semelhante quando tratadas com o mesmo antibiótico 1,2. Em contraste com a persistência, a resistência aos antibióticos é definida em nível populacional e geralmente é consequência de mutações de novo ou da transferência horizontal de genes de um plasmídeo que confere resistência3. Enquanto as mutações responsáveis pela resistência estão localizadas principalmente no alvo do fármaco ou nas regiões promotoras das bombas de efluxo do fármaco, genes que alteram a frequência de persistência identificados por abordagens de análise genômica ampla e mutante direcionada têm se mostrado numerosos e diversos 2,3,4,5,6,7,8 . Portanto, é provável que as células bacterianas possam entrar no estado persistente através de múltiplas vias9,10,11, e abordagens para investigar o fenômeno de persistência em nível unicelular são necessárias para caracterizar a fisiologia dessas células persistentes.
O recente desenvolvimento de ferramentas microfluídicas usadas em combinação com a microscopia de fluorescência abriu caminho para a caracterização do fenótipo de persistência e destacou o papel de processos celulares chave, como replicação cromossômica 12, reparo de DNA13 e divisão celular14, na formação celular persistente. Neste artigo, descrevemos uma abordagem integrada combinando ensaios clássicos de microbiologia com imagens vivas unicelulares para caracterizar células persistentes geradas em culturas de Escherichia coli em crescimento exponencial tratadas com uma alta dose de ofloxacina. O protocolo aqui descrito pode ser aplicado para estudar o fenômeno da persistência do antibiótico em outras espécies bacterianas, como Bacillus subtilis15, ou condições (por exemplo, persistência do antibiótico após tratamento com β-lactâmicos 16) e pode ser facilmente modificado para investigar os diversos fenômenos que envolvem a heterogeneidade fenotípica17,18,19 . Além disso, a configuração descrita neste artigo pode ser combinada com outros repórteres fluorescentes para investigar parâmetros celulares distintos de interesse, como níveis intracelulares de pH20 ou ATP21 em nível de célula única, o que pode potencialmente produzir novos insights sobre o fenômeno de persistência do antibiótico.
Protocolo
NOTA: Use vidro de cultura estéril, pontas de pipeta e meio de crescimento. Aqui, células de E. coli foram cultivadas em um meio quimicamente definido de baixa autofluorescência (ver Tabela de Materiais). As inoculações foram realizadas na presença de um queimador de Bunsen para minimizar o risco de contaminação.
1. Cultura celular e curva de crescimento
- Esticar a cepa de interesse de um estoque congelado de glicerol em uma placa de ágar Luria-Bertaini (LB) (suplementada com um antibiótico seletivo, se necessário) e incubar a 37 °C durante a noite (entre 15 h e 19 h) para obter colônias únicas.
NOTA: O experimento aqui apresentado utiliza duas linhagens, E. coli K-12 MG1655 correspondente à cepa wt e a linhagem isogênica MG1655 hupA-mCherry 22. Esta última cepa expressa a subunidade α marcada com fluorescência da proteína associada ao nucleóide HU. O repórter hupA-mCherry está integrado ao locus nativo do hupA. HU-mCherry serve como um proxy para acompanhar a dinâmica nucleóide em células vivas à medida que se liga ao DNA de uma maneira não-específica. - Inocular 5 mL de meio (aqui, meio à base de ácido propanossulfônico 3-[N-morfolino] [MOPS]; Tabela 1, Tabela 2 e Tabela 3) suplementado com glicose a 0,4% e antibiótico seletivo (se necessário) com colônia isolada em tubo de vidro (≥25 mL), e colocar o tubo em incubadora de agitação regulada para 37 °C e 180 rotações por minuto (rpm) durante a noite (entre 15 h e 19 h). Alternativamente, tubos de plástico, vidro ou frascos de plástico (≥ 25 mL) podem ser usados em vez de tubos de vidro.
OBS: O meio MOPS suplementado com glicerol na concentração final de 0,4% foi utilizado durante todos os experimentos descritos neste trabalho, com exceção das culturas noturnas que foram realizadas em MOPS glicose 0,4%. O tempo de geração de E. coli em MOPS suplementado com glicose é menor do que em MOPS glicerol. O uso de MOPS glicose 0,4% em vez de MOPS glicerol 0,4% nesta etapa garante que as células atinjam a fase estacionária dentro de 19 h. Outros meios de crescimento, como M9 ou meio rico definido (RDM) suplementado com fontes de carbono distintas, também podem ser usados. Deve-se, no entanto, notar que a taxa de crescimento e a frequência de persistência são diferentes dependendo do meio e/ou do carbono utilizado23. - Na manhã seguinte, centrifugar 1 mL de cultura a 2.300 x g por 3 min, descartar o sobrenadante e ressuspender suavemente o pellet no mesmo volume de solução salina tamponada com fosfato (PBS). Medir a densidade óptica a 600 nm (OD 600 nm) e calcular o volume necessário para um ODinicial de 600 nm de 0,01 em um volume final de 2 mL.
- Colocar 2 mL de meio MOPS glicerol 0,4% em um poço de uma placa de fundo claro de 24 poços e inocular com o volume de cultura noturno calculado. Coloque a placa de 24 poços em um leitor de microplacas automatizado (consulte Tabela de Materiais) para monitorar o OD600 nm por 24 horas. Ajuste o leitor de microplacas para medir o OD600 nm a cada 15 min a uma temperatura de 37 °C e com alta rotação orbital (140 rpm).
NOTA: Se a cepa usada no experimento codificar um repórter fluorescente, certifique-se de que sua taxa de crescimento seja comparável à do wt para evitar qualquer artefato nos experimentos subsequentes, pois a taxa de crescimento afeta a frequência de persistência do antibiótico23. Devido às limitações de detecção em densidades celulares muito baixas e aos potenciais efeitos específicos da cepa na relação OD600 nm/unidades formadoras de colônias (UFC), recomenda-se avaliar a cinética de crescimento monitorando a UFC·mL-1 quando se trabalha com cepas não caracterizadas.
2. Determinação da concentração inibitória mínima dos antibióticos
NOTA: A concentração inibitória mínima (CIM) é definida como a dose mais baixa de antibiótico na qual não se observa crescimento bacteriano. A determinação da CIM precisa ser realizada para cada antibiótico e cepa. Nos experimentos aqui descritos, foi utilizado o antibiótico fluoroquinolona ofloxacina (OFX). A determinação da CIM permite confirmar que a solução antibiótica foi corretamente preparada, que o antibiótico está ativo e que as cepas são igualmente sensíveis ao antibiótico. Aqui, o método de diluição em ágar publicado foi realizado para determinar a CIM para OFX das diferentes cepas utilizadas24. A CIM de um determinado antibiótico para uma determinada cepa bacteriana também pode ser determinada pelo método de diluição em caldo24.
- Preparação das placas para determinação da CIM
- Preparar uma solução-mãe mestra para o antibiótico utilizado nas experiências, dissolvendo 5 mg de OFX em 1 ml de água ultrapura. Adicionar 20 μL de HCl a 37% para aumentar a solubilidade do OFX.
- Derreter 100 mL de ágar LB estéril e mantê-lo a 55 °C para evitar a solidificação. Preparar seis pequenos frascos de vidro (25 mL) e pipetar 5 mL de meio líquido de ágar LB em cada frasco usando uma pipeta estéril.
- Diluir 10 μL da solução-mãe OFX de 5 mg·mL-1 em 90 μL de água ultrapura (diluição de 1:10 da matéria-prima principal). Adicionar 0 μL, 2 μL, 4 μL, 6 μL, 8 μL ou 10 μL da solução OFX de 500 μg·mL-1 a cada um dos seis frascos de vidro contendo 5 mL de meio de ágar LB para gerar meio de ágar LB com concentrações finais de 0 μg·mL−1, 0,02 μg·mL−1, 0,04 μg·mL−1, 0,06 μg·mL−1, 0,08 μg·mL−1 e 0,1 μg·mL-1 OFX, respectivamente. Misture a solução girando várias vezes os frascos.
NOTA: Se a CIM do antibiótico de interesse for conhecida, o intervalo de concentrações deve ir de baixo para acima da CIM real. Se a CIM for desconhecida, recomenda-se uma grande gama de concentrações com uma série de diluição log2 . Certifique-se de que o meio de ágar LB tenha esfriado antes de adicionar o antibiótico, pois altas temperaturas podem inativá-lo. No entanto, é importante não deixar o meio de ágar LB solidificar antes da adição do antibiótico, pois isso poderia levar à distribuição não homogênea do antibiótico no meio de ágar LB. - Deitar 5 ml de cada um dos seis meios de ágar LB preparados no passo 2.1.3 de forma doseadora crescente para uma placa de cultura de 6 poços utilizando uma pipeta estéril. Deixe o ágar esfriar até a solidificação e seque a placa antes de usar.
NOTA: Preparar a solução antibiótica e a placa de cultura no dia em que o ensaio for realizado.
- Ensaio de determinação da CIM
- Inocular 5 mL de meio LB com uma colônia isolada em um tubo de vidro (≥25 mL) e colocar o tubo de vidro em uma incubadora de agitação regulada para 37 °C e 180 rpm durante a noite (entre 15 h e 19 h).
- Na manhã seguinte, medir o OD600 nm e diluir a cultura em um tubo de ensaio até uma densidade celular final de 1 x 107 UFC·mL-1 em PBS. Para as cepas testadas aqui, 1 x 107 UFC·mL−1 corresponde a um OD600 nm de 0,0125.
NOTA: Se a correlação entre UFC·mL-1 e OD 600 nm não for conhecida, a curva de crescimento determinada pelas medidas de UFC e OD600 nm deve ser estabelecida para calcular o fator de correlação entre UFC·mL-1 e OD600 nm. - Coloque 2 μL da cultura previamente diluída em cada poço da placa seca de 6 poços. Deixe as manchas secarem antes de colocar a placa em uma incubadora a 37 °C durante a noite (entre 15 h e 19 h).
- No dia seguinte, conte as colônias formadas em cada poço. A CIM corresponde ao poço com concentração mínima de antibiótico onde não é detectado crescimento bacteriano.
3. Ensaio pontual
NOTA: O método spot assay é uma abordagem qualitativa que permite estimar o número de células viáveis (células capazes de gerar colônias após estresse antibiótico). O ensaio pontual é realizado antes do ensaio de time-kill para fornecer informações sobre a viabilidade da estirpe utilizada nas condições testadas e para informar sobre as diluições necessárias durante o ensaio time-kill (ver secção 4).
- Para preparar as placas de ágar LB para ensaios pontuais, despeje 50 mL de ágar LB em uma placa de Petri quadrada (144 cm2). Prepare uma placa de Petri quadrada por ponto de tempo. Deixe o ágar LB solidificar e seque as placas antes de usar.
- Inocular 5 mL de meio (MOPS glicose 0,4%) com uma colônia isolada em um tubo de vidro (≥25 mL) e colocar o tubo em uma incubadora de agitação regulada para 37 °C e 180 rpm durante a noite (entre 15 h e 19 h).
- No dia seguinte, medir o OD 600 nm e diluir a cultura em meio fresco ajustado à temperatura (37 °C, MOPS glicerol 0,4%) em um tubo de vidro (≥25 mL) para um OD final de600 nm de ~0,001. Deixe a cultura crescer durante a noite em uma incubadora a 37 °C a 180 rpm (entre 15 h e 19 h).
- No dia seguinte, medir o OD600 nm, e incubar a cultura para um ODfinal 600 nm de 0,3.
- Durante a incubação, prepare placas de 96 poços colocando 90 μL de solução de MgSO4 0,01 M em todos os poços, exceto nos poços da primeira fileira (fileira A). As placas de 96 poços serão usadas para diluição seriada de 10 vezes na etapa 3.7.
NOTA: Neste experimento, duas linhagens (wt e hupA-mCherry ) foram testadas em triplicata em sete momentos diferentes. Como uma placa é composta por 12 colunas, uma placa pode ser usada para dois pontos de tempo e, assim, um total de quatro placas foram preparadas. - A um OD600 nm de 0,3, retirar 200 μL de cada cultura bacteriana. Essas amostras correspondem ao tempo t0 (não tratado) anterior ao tratamento com antibiótico e permitem a determinação da UFC·mL-1 antes do tratamento com antibiótico.
- Centrifugar as amostras a 2.300 x g por 3 min. Durante o tempo de centrifugação, adicione a concentração desejada de OFX à cultura líquida e continue a incubar a 37 °C enquanto agita.
NOTA: Aqui, OFX foi usado na concentração de 5 μg·mL-1 (correspondendo à CIM multiplicada por 83 vezes). Em estudo anterior, essa concentração foi utilizada para caracterizar o fenômeno de persistência sob exposição OFX25. A concentração do antibiótico utilizado para os ensaios de tempo/morte pode variar dependendo do antibiótico, do meio de cultura e do estado de crescimento bacteriano. - Após centrifugação, ressuspender o pellet de células em 200 μL de solução de MgSO4 0,01 M. Colocar 100 μL no poço vazio da placa de 96 poços preparada na etapa 3.5. Efectuar uma diluição transferindo 10 μL do poço da linha A para o poço da linha B contendo 90 μL de 0,01 M de MgSO4. Continue as diluições em série transferindo 10 μL do poço na linha B para o poço na linha C. Repita até atingir uma diluição de 10−7 (com cada transferência sendo uma diluição de 10 vezes).
NOTA: Neste experimento, seis culturas (três para a linhagem wt e três para a cepa hupA-mCherry ) foram testadas para cada momento. De acordo com o protocolo, uma pipeta de canal muti é usada para realizar as diluições seriadas para as seis cepas ao mesmo tempo. Para minimizar o erro técnico, as pontas da pipeta são trocadas entre cada transferência. - Colocar 10 μL de cada diluição nas placas de ágar LB preparadas na etapa 3.1.
NOTA: Uma pipeta multicanal é usada para detectar simultaneamente a mesma diluição (por exemplo, uma diluição de 10−4 ) para as seis culturas. Durante a mancha, a primeira parada da pipeta multicanal deve ser usada sem empurrar a segunda parada, pois isso pode resultar em microgotículas sendo dispensadas na placa. - Em momentos relevantes após a adição do antibiótico, retirar 200 μL da cultura e realizar diluições seriadas conforme descrito na etapa 3.8. Identificar 10 μL de cada diluição, tal como descrito no passo 3.9.
OBS: Para o experimento aqui descrito, foram coletados sete momentos (t0, t1h, t2h, t3h, t4h, t5h, t6h). Para cepas que apresentam maior suscetibilidade a antibióticos em relação ao peso em peso, múltiplas lavagens em MgSO4 0,01 M podem ser realizadas para remover antibióticos residuais. - Incubar as placas a 37 °C durante a noite (entre 15 h e 19 h). No dia seguinte, conte o número de colônias nas duas diluições mais altas para as quais as colônias podem ser detectadas. Idealmente, os pontos nas placas de ágar para essas diluições contêm entre 3 a 30 colônias, permitindo a determinação precisa do UFC·mL-1 para cada amostra.
- Calcular a razão de sobrevivência dividindo a UFC·mL-1 calculada de cada ponto de tempo pela UFC·mL-1 da população inicial em t0.
4. Ensaios de tempo-morte
NOTA: Embora os ensaios pontuais sejam um método fácil de usar para estimar a taxa de sobrevivência de uma determinada cepa para um determinado antibiótico, os ensaios de morte por tempo fornecem uma taxa de sobrevivência de maior resolução e são realizados para quantificar com precisão a viabilidade bacteriana. O perfil da curva de morte pode ser usado para determinar se uma determinada cepa bacteriana é sensível, tolerante ou resistente ao antibiótico em uma determinada condição. Além disso, ensaios de time-kill permitem determinar o tempo de exposição ao antibiótico necessário para detectar o fenômeno de persistência (início da segunda inclinação da curva de morte bifásica), bem como a frequência de persistência.
- Prepare placas de ágar LB para o ensaio de time-kill plating. Despeje 25 mL de ágar LB em uma placa de Petri (±57 cm2). Prepare pelo menos duas placas de Petri por ponto de tempo (duas diluições por ponto de tempo por cepa são chapeadas).
- Deixe o ágar LB solidificar e seque as placas antes de adicionar cinco a oito esferas de vidro estéreis a cada placa. Inverter e rotular as placas de acordo com a deformação/condição/ponto.
NOTA: As esferas de vidro permitem a propagação de bactérias nas placas de ágar durante as etapas 4.7 e 4.9. Alternativamente, as células podem ser dispersas na placa de ágar usando um espalhador. - Para cada amostra, preparar tubos de vidro de diluição em série 10 vezes contendo 900 μL de solução de MgSO4 0,01 M. O número de tubos de vidro de diluição por amostra que precisa ser preparada corresponde às diluições necessárias para detectar colônias no ensaio local.
- Inocular 5 mL de meio (MOPS glicose 0,4%) com uma colônia isolada em um tubo de vidro (≥25 mL) e colocar o tubo em uma incubadora de agitação regulada para 37 °C e 180 rpm durante a noite (entre 15 h e 19 h).
- Após 16 h de crescimento, medir o OD 600 nm e diluir a cultura em meio fresco ajustado à temperatura (37 °C, MOPS glicerol 0,4%) em um tubo de vidro para um ODfinal de 600 nm de ~0,001. Deixe a cultura crescer durante a noite em uma incubadora a 37 °C a 180 rpm (entre 15 h e 19 h).
- No dia seguinte, medir o OD600 nm, e incubar a cultura para um ODfinal 600 nm de 0,3.
- A um OD 600 nm de 0,3, retirar 100 μL da cultura, diluir de acordo com os dados obtidos no ensaio spot (a um OD600 nm de 0,3 e sem antibiótico, uma diluição de 10−5 geralmente dá 200-300 colônias) e placa100 μL nas placas de ágar LB preparadas nas etapas 4.1-4.2. Agite suavemente as placas para evitar que as contas entrem em contato com as bordas da placa, pois isso pode levar a uma disseminação não homogênea das células bacterianas no meio de ágar LB. Essa primeira amostra prévia ao tratamento com antibiótico corresponde ao tempo t0 (UFC·mL-1 antes do tratamento com antibiótico).
- Adicione a concentração desejada de OFX e continue a incubar a 37 °C enquanto agita.
NOTA: Aqui, OFX foi usado na concentração de 5 μg·mL-1 (correspondendo à CIM multiplicada por 83 vezes). - Em momentos relevantes após a adição do antibiótico, retirar 100 μL da cultura, diluir de acordo com os dados obtidos no ensaio spot e placa 100 μL nas placas de ágar LB preparadas nas etapas 4.1-4.2. Plaquear as células conforme descrito no passo 4.7.
OBS: Para o experimento aqui descrito, foram coletados sete momentos (t0, t1h, t2h, t3h, t4h, t5h, t6h). Para cepas que apresentam maior suscetibilidade a antibióticos em relação ao peso em peso, múltiplas lavagens em MgSO4 0,01 M podem ser realizadas para remover antibióticos residuais. - Incubar as placas a 37 °C durante a noite (entre 15 h e 19 h). No dia seguinte, conte o número de colônias nas duas diluições mais altas para as quais as colônias podem ser detectadas. Idealmente, as placas devem conter 30-300 colônias para permitir uma determinação precisa da UFC·mL-1 em cada amostra.
- Calcular a razão de sobrevivência normalizando a UFC·mL-1 em cada ponto de tempo pela UFC·mL-1 em t0. Plotar o log10 normalizado CFU·mL-1 em função do tempo.
5. Microscopia microfluídica time-lapse
NOTA: A seção a seguir descreve a preparação da placa microfluídica, bem como a aquisição de imagens em time-lapse e o procedimento de análise de imagens. O objetivo deste experimento é observar e analisar o fenótipo de persistência ao tratamento antibiótico em nível unicelular. Os dados coletados durante este experimento podem ser usados para gerar uma ampla gama de resultados, dependendo da pergunta abordada e/ou dos repórteres fluorescentes usados durante o experimento. No experimento aqui descrito, foi realizada análise quantitativa do comprimento celular e fluorescência HU-mCherry22, refletindo a organização nucleóide em células persistentes e não persistentes.
- Cultura de células bacterianas para microscopia microfluídica time-lapse
- Inocular 5 mL de meio (MOPS glicerol 0,4%, suplementado com um antibiótico seletivo se necessário) com uma colônia isolada em um tubo de vidro (≥25 mL) e colocar o tubo em uma incubadora de agitação regulada para 37 °C e 180 rpm durante a noite (entre 15 h e 19 h).
- No dia seguinte, medir o OD 600 nm e diluir a cultura em meio fresco ajustado à temperatura (37 °C, MOPS glicerol 0,4%) em um tubo de vidro para um OD final de600 nm de ~0,001. Deixe a cultura crescer durante a noite (entre 15 h e 19 h) em uma incubadora de agitação a 37 °C e 180 rpm para obter uma cultura de fase exponencial precoce no dia seguinte.
- Preparo da placa microfluídica e imagens de microscopia time-lapse
NOTA: Experimentos microfluídicos podem ser realizados em dispositivos microfluídicos comercialmente disponíveis (conforme descrito aqui) ou em sistemas microfluídicos produzidos internamente.- Remova a solução de conservação (se presente) de cada poço da placa microfluídica e substitua-a por um meio de cultura fresco.
NOTA: Se a placa microfluídica contiver um poço de saída de resíduos, a solução de conservação do poço de saída deve ser removida, mas não substituída pelo meio. - Sele a placa microfluídica com o sistema coletor clicando no botão Seal ou através do software microfluídico (primeiro selecione Tool, seguido por Seal Plate).
NOTA: Para selar a placa, uma pressão uniforme deve ser aplicada à placa e ao coletor, apertando manualmente a placa contra o coletor. Se executada corretamente, a nota "lacrada" deve aparecer na interface ONIX2. É importante não aplicar nenhuma pressão sobre a lâmina de vidro para evitar qualquer risco potencial de quebrar o coletor. - Uma vez selado, execute uma primeira sequência de priming (clique em Run Liquid Priming Sequence na interface do software microfluídico).
NOTA: A sequência Run Liquid Priming corresponde a 5 min de perfusão a 6,9 kPa para os poços 1-5, seguida de 5 min de perfusão a 6,9 kPa para o poço 8 e uma rodada final de perfusão para o poço 6 por 5 min a 6,9 kPa. O Run Liquid Priming Sequence permite a remoção da solução de conservação que ainda pode estar presente nos canais que conectam os diferentes poços. - Incubar a placa em um gabinete do microscópio controlado por termostato na temperatura desejada (aqui, 37 °C) por um mínimo de 2 h antes do início da imagem da microscopia.
- Inicie uma segunda sequência de escorvamento líquido antes de iniciar o experimento.
- Isole a placa microfluídica clicando em Seal off na interface do software microfluídico. Substitua o meio no poço 1 e no poço 2 por 200 μL de meio fresco, no poço 3 por 200 μL de meio fresco contendo o antibiótico (aqui, OFX a 5 μg·mL−1), no poço 4 e no poço 5 por 200 μL de meio fresco, no poço 6 por 200 μL de meio fresco e no poço 8 por 200 μL da amostra de cultura (a partir do passo 5.1.2) diluídos a um ODde 600 nm de 0,01 no meio fresco.
- Selar a placa microfluídica, conforme descrito na etapa 5.2.2, e colocá-la na objetiva do microscópio dentro do gabinete do microscópio.
NOTA: Certifique-se de colocar uma gota de óleo de imersão na objetiva do microscópio antes de colocar a placa microfluídica. - No software microfluídico, clique em Cell Loading para permitir o carregamento da célula na placa microfluídica.
NOTA: A etapa de Carregamento Celular compreende 15 s de perfusão a 13,8 kPa para o poço 8, seguida de 15 s de perfusão a 27,6 kPa para o poço 6 e poço 8, e uma rodada final de perfusão para o poço 6 por 30 s a 6,9 kPa. A densidade das células na placa microfluídica é crítica para o experimento. A primeira parte deste protocolo microfluídico consiste no cultivo de bactérias por 6 h em meio fresco antes do tratamento com antibiótico. Após 6 h de crescimento, a densidade celular deve ser suficiente para detectar as células persistentes raras (nas condições usadas neste estudo, as células persistentes geram a uma frequência de 10−4). Se a densidade celular é muito alta, é difícil distinguir as células individuais, o que impede a análise precisa de uma única célula. Como a taxa de crescimento é diretamente dependente do meio, a densidade de células nos campos de microscopia deve ser avaliada antes do início do experimento. - Defina um foco ideal usando o modo de luz transmitida e selecione várias regiões de interesse (ROIs) onde um número de célula apropriado é observado (até 300 células por campo).
NOTA: Selecione pelo menos 40 ROIs para certificar-se de que as células persistentes raras são imageadas. - No software microfluídico, clique em Criar um Protocolo. Programar a injeção de meio fresco a 6,9 kPa por 6 h (poço 1-2), seguida da injeção do meio contendo o antibiótico a 6,9 kPa por 6 h (poço 3) e, por fim, a injeção de meio fresco a 6,9 kPa por 20 h (poços 4-5).
NOTA: Uma cultura bacteriana diluída a um OD600 nm de 0,01 permite que as bactérias cresçam na câmara microfluídica por 6 h, garantindo que as células estejam na fase de crescimento exponencial. Dependendo do número de células introduzidas no dispositivo microfluídico durante a etapa de carregamento (ver passo 5.2.8), a duração da fase de crescimento pode ser adaptada para obter até 300 células por ROI. Como a persistência é um fenômeno raro, o aumento do número de células por ROI melhora a possibilidade de observar células persistentes. O número de células não deve, no entanto, exceder 300 células por ROI, pois isso torna a análise de célula única tediosa. - Execute imagens de microscopia no modo time-lapse com um quadro a cada 15 min usando a luz transmitida e a fonte de luz de excitação para o repórter fluorescente. Aqui, uma fonte de luz de excitação de 560 nm para o sinal mCherry foi usada (LED de 580 nm a 10% de potência com filtro 00 [530-585 ex, 615LP em, Zeiss] e exposição de 100 ms para mCherry). O software Zen3.2 compatível com Zeiss foi usado para imagens celulares.
- Remova a solução de conservação (se presente) de cada poço da placa microfluídica e substitua-a por um meio de cultura fresco.
- Análise das imagens
NOTA: A abertura e visualização das imagens de microscopia são realizadas com o software open-source ImageJ/Fiji (https://fiji.sc/)26. A análise quantitativa das imagens é realizada utilizando o software open-source ImageJ/Fiji e o plugin livre MicrobeJ (https://microbej.com)27. Nesse protocolo, foi utilizada a versão MicrobeJ 5.13I(14).- Abra o software ImageJ/Fiji no computador e arraste as imagens de microscopia de lapso de tempo da hiperpilha para a barra de carregamento de Fiji. Use Image > Color > Make Composite para fundir os diferentes canais da hiperstack. Se os canais do experimento de lapso de tempo não corresponderem à cor desejada (por exemplo, o contraste de fase é mostrado em vermelho em vez de cinza), use Imagem > Cor > Organizar Canais para aplicar a cor apropriada aos canais.
- Abra o plugin MicrobeJ e detecte as células bacterianas usando a interface de edição manual. Exclua as células detectadas automaticamente e contorne manualmente as células persistentes de interesse quadro a quadro.
Observação : configurações diferentes podem ser usadas para detectar automaticamente células individuais. A detecção manual foi usada aqui, pois as células persistentes analisadas formam filamentos longos, que raramente são detectados corretamente usando detecção automática. - Após a detecção, use o ícone Resultado na interface de edição manual do MicrobeJ para gerar uma tabela ResultJ. Salve o arquivo ResultJ e use a tabela ResultJ para obter informações sobre diferentes parâmetros de interesse da análise de célula única. No caso deste protocolo, a fluorescência média da intensidade HU-mCherry, o comprimento celular e a área celular de células individuais foram exportados.
Resultados
Como descrito acima, as cepas utilizadas para a análise fenotípica unicelular de células persistentes foram caracterizadas em meio MOPS glicerol 0,4%. O monitoramento da OD600nm ao longo do tempo não mostrou diferença entre as linhagens wt e hupA-mCherry (Figura 1). Isso indica que a expressão da proteína de fusão HU-mCherry não influenciou o crescimento nessas condições. As células bacterianas de ambas as linhagens inicialmente inoculadas a uma OD600 nm de 0,01 atingiram a fase exponencial ±8 h após a inoculação.
A CIM de OFX foi determinada por métodos padronizados (aqui, diluição seriada em ágar)24. A CIM é definida como a concentração mínima em que não é detectado crescimento visível. A CIM de OFX para ambas as cepas foi determinada em 0,06 μg·mL−1, indicando que a fusão hupA-mCherry não teve efeito sobre a sensibilidade à OFX em comparação com a linhagem em peso isogênica (Figura 2).
Determinamos ainda o efeito de um tratamento OFX letal (CIM 83 vezes) sobre a viabilidade de ambas as cepas utilizadas neste estudo. Como a contagem de células viáveis diminui ao longo do tempo com a exposição ao OFX, as diluições das culturas bacterianas precisam ser ajustadas adequadamente para atingir 30 a 300 colônias por placa. Para determinar as diluições apropriadas ao longo do tempo, um ensaio spot foi realizado, onde 10 μL de 0 a 10−7 diluições seriadas de 10 vezes foram colocadas em placas de Petri quadradas usando uma pipeta multicanal. As diluições apropriadas foram aquelas em que os clones isolados eram visíveis (por exemplo, em t0 = 10−5, t1h = 10−4/10−3, t4h = 10−2/10−1) (Figura 3).
Embora o ensaio pontual seja um método fácil para obter informações sobre a cinética da matança mediada por OFX, ele não consegue determinar com precisão a dinâmica de matança. Quando a viabilidade de células de crescimento exponencial tratadas com OFX foi monitorada pelo ensaio time-kill, uma curva bifásica típica foi observada (Figura 4). A primeira inclinação da curva reflete a rápida matança da população não persistente (linha tracejada vermelha). Nas condições testadas, até 99,9% das células foram incapazes de formar colônias após 3 h na presença de OFX. Esta primeira fase de matança é seguida por uma segunda fase, mostrando uma taxa de mortalidade mais lenta (linha tracejada azul), que revela a presença de células persistentes tolerantes a drogas. Nas condições testadas, a fase persistente iniciou-se por volta de 3 h após a adição de OFX, evidenciando a necessidade de exposição das células à OFX por mais de 3 h para investigar os fenótipos persistentes. É importante ressaltar que a curva time-kill mostra que a proteína de fusão hupA-mCherry não teve efeito sobre a cinética time-kill . A cepa que codifica a fusão fluorescente translacional pode, portanto, ser usada para monitorar as células persistentes usando microscopia de fluorescência.
Passamos a investigar o fenômeno da persistência em nível unicelular. Para isso, a cepa hupA-mCherry foi introduzida em uma placa microfluídica, o que permitiu a mudança das condições do meio (aqui, crescimento, tratamento e recuperação) durante a realização da microscopia de lapso de tempo em uma dada ROI. Durante a primeira etapa do experimento microfluídico, as células introduzidas no dispositivo microfluídico foram perfundidas com meio de cultura (MOPS glicerol 0,4%) e divididas com um tempo de geração de ~2 h (Figura 5 e Figura 6). Esta primeira fase de crescimento indica que as células estavam viáveis e se dividindo ativamente antes do tratamento com OFX.
Após essa primeira fase de crescimento, as células foram perfundidas com meio de cultura suplementado com 5 μg·mL−1 de OFX por 6 h. Assim que o antibiótico atingia as células, a divisão celular era bloqueada (Figura 5 e Figura 6). Após 6 h de tratamento com OFX, as células foram perfundidas com meio fresco. Enquanto a grande maioria das células não conseguiu retomar o crescimento (Figura 5 e Figura 6), uma pequena subpopulação de bactérias foi capaz de alongar e gerar células filamentosas25. Essas células, que foram capazes de se dividir e gerar células-filhas viáveis após o tratamento com OFX, podem ser definidas como células persistentes.
Como essa configuração permite a visualização das células persistentes antes, durante e após o tratamento, ela não apenas fornece informações sobre o fenótipo persistente durante a fase de recuperação, mas também sobre o estado fisiológico das células persistentes antes do tratamento (Figura 6). Nas condições testadas, as células persistentes dividiram-se de forma semelhante às células não persistentes antes do tratamento com OFX, indicando que as células persistentes observadas não se originaram de uma subpopulação dormente (Figura 6)25.
A análise do comprimento celular das células persistentes durante a fase de recuperação revelou que cada filamento apresentou uma taxa específica de alongamento. O comprimento celular atingido por cada persistente antes da primeira divisão diferiu de um persistente para outro. Da mesma forma, o momento do evento da primeira divisão foi altamente heterogêneo (Figura 6). O filamento persistente em divisão gerou múltiplas células filhas, que começaram a crescer e se dividir, em sua maioria, de forma semelhante às células não tratadas (Figura 7). A divisão sucessiva do filamento resultou, então, em uma diminuição progressiva do comprimento celular, dando origem a células-filhas com comprimento celular semelhante ao anterior ao tratamento com OFX (Figura 6 e Figura 7B). A grande maioria das células foi incapaz de induzir filamentação após a remoção de OFX. Essa grande população celular corresponde às células mortas (Figura 5 e Figura 6).
A fusão fluorescente da proteína HU associada ao nucleóide permite a visualização da dinâmica do nucleóide22. A análise da intensidade total da fluorescência de HU-mCherry dentro da célula pode ser utilizada como proxy do conteúdo deDNA22,25. Durante a fase de crescimento (antes do tratamento com OFX), a intensidade total de fluorescência do mCherry variou, refletindo a dinâmica de replicação e segregação cromossômica durante o ciclo celular (Figura 8). Após a adição de OFX, a fluorescência mCherry aumentou na célula média, indicando compactação nucleoide, que demonstrou ser induzida pela formação de quebras de DNA de fita dupla28 (Figura 5). Quebras de DNA de fita dupla são consequência do mecanismo de ação da OFX, que corrompe as topoisomerases tipo II DNA-girase e topoisomerase IV29,30. Em E. coli, a DNA-girase é o alvo primário da OFX29,30. Ao ligar seu alvo em uma etapa crítica do mecanismo de passagem de fita dupla, o OFX inibe o rebaixamento das cadeias de DNA clivadas, levando à liberação de quebras de DNA de fita dupla30. Como descrito acima, as células persistentes ao tratamento com OFX começaram a filamentar durante a recuperação25 (Figura 6). O aumento do comprimento celular correlacionou-se com um aumento na intensidade total da fluorescência mCherry, o que reflete o reinício da replicação e um aumento na abundância nucleóide no filamento25 (Figura 7a e Figura 8). Para as células mortas, a intensidade total da fluorescência do mCherry manteve-se estável durante o tratamento e durante a fase de recuperação, indicando que essas células não conseguiram replicar seus cromossomos após a remoção da OFX (Figura 8). Vídeo microfluídico (Vídeo 1) de células HU-mCherry de E. coli antes, durante e após o tratamento com ofloxacina também é mostrado.
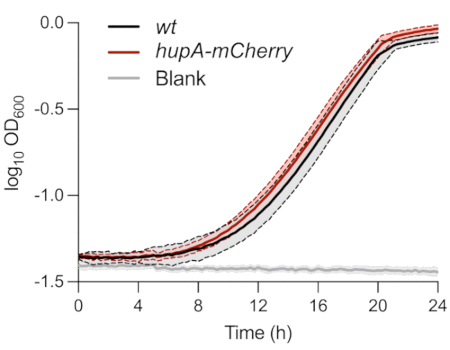
Figura 1: Monitoramento do crescimento de cepas de E. coli wt e hupA-mCherry. Monitoramento óptico de densidade (OD600 nm) de wt (preto) e hupA-mCherry (vermelho). As tonalidades e linhas tracejadas indicam os desvios-padrão das triplicatas biológicas. Clique aqui para ver uma versão maior desta figura.
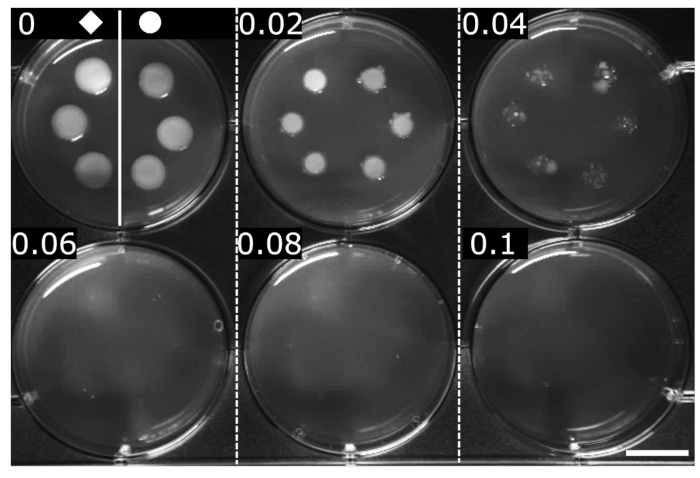
Figura 2: Determinação da CIM de OFX para as estirpes de E. coli em peso e hupA-mCherry. O wt () e o hupA-mCherry (●) foram cultivados em meio LB, e 2 μL foram detectados em diluições seriadas de ágar LB contendo OFX (♦ concentração indicada em cada painel em μg·mL−1). A inibição do crescimento é visível a um mínimo de 0,06 μg·mL−1. A figura é um experimento representativo de triplicatas biológicas. Barra de escala = 1 cm. Clique aqui para ver uma versão maior desta figura.
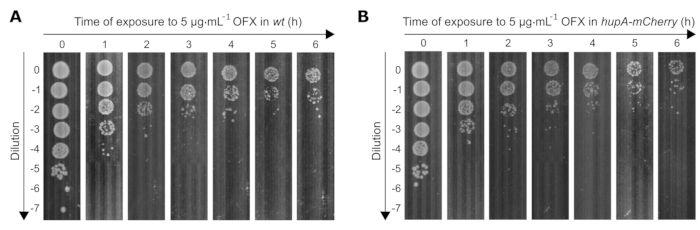
Figura 3: Ensaio pontual de cepas de E. coli wt e hupA-mCherry após exposição à OFX. As cepas (A) em peso e (B) hupA-mCherry foram cultivadas em glicerol MOPS 0,4%, conforme descrito no protocolo (seção 3), e as células de crescimento exponencial (OD600 nm = 0,3) foram tratadas com 5 μg·mL−1 OFX. T0 corresponde ao ponto de tempo antes da adição de OFX. T1, T2, T3, T4, T5 e T6 correspondem a 1-6 h após a adição de OFX. A figura é um experimento representativo de triplicatas biológicas. Clique aqui para ver uma versão maior desta figura.
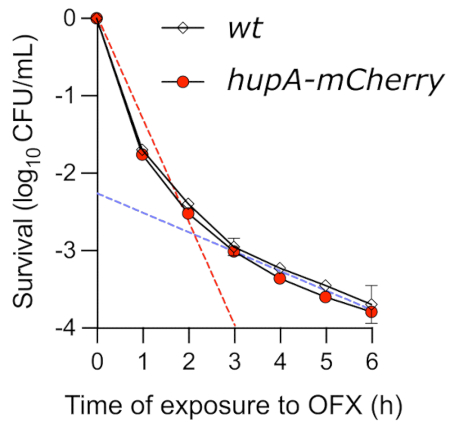
Figura 4: Ensaio Time-kill de cepas wt e hupA-mCherry E. coli após exposição à OFX. As cepas wt () e hupA-mCherry (●) foram cultivadas em glicerol MOPS 0,4%, conforme descrito no protocolo (seção 4), e as células de crescimento exponencial (♦ OD600 nm = 0,3) foram tratadas com 5 μg·mL−1 OFX. As linhas tracejadas indicam a primeira fase de morte "rápida" (vermelha) e a segunda fase de morte "lenta" (azul), correspondendo às subpopulações sensível e persistente (obtidas por regressão linear entre T0 e T2, bem como entre T3 e T6, respectivamente). As barras de erro indicam os desvios-padrão das triplicatas biológicas. Clique aqui para ver uma versão maior desta figura.
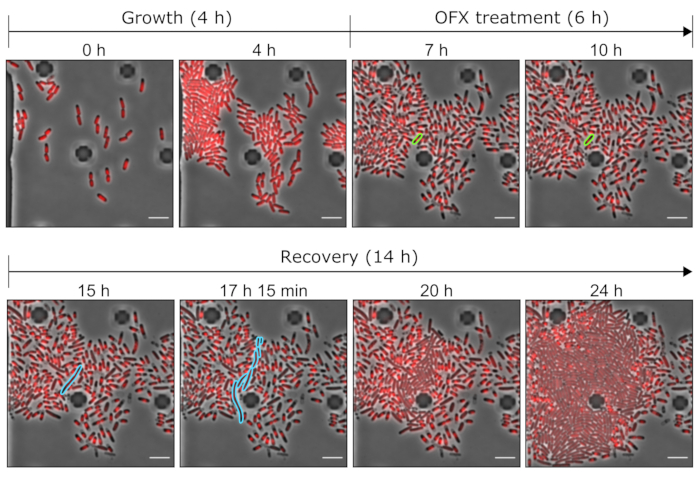
Figura 5: Imagens representativas do persistente OFX e das células mortas usando ferramentas microfluídicas. Imagens de microscopia representativas mostrando os pontos de tempo relevantes do experimento microfluídico realizado com a cepa hupA-mCherry (contraste de fase em cinza, sinal HU-mCherry em vermelho). As células que expressam o hupA-mCherry marcado foram cultivadas em uma placa microfluídica (aqui, 4 h), seguida por um desafio OFX (5 μg·mL−1). Após 6 h na presença de OFX, as células foram perfundidas com meio fresco, permitindo que as células persistentes se recuperassem. A célula persistente e suas células de progênie durante o tratamento com OFX e após a remoção de OFX são destacadas em verde e azul, respectivamente. Os pontos de tempo correspondentes são indicados em cada painel. Barra de escala = 5 μm. Clique aqui para ver uma versão maior desta figura.
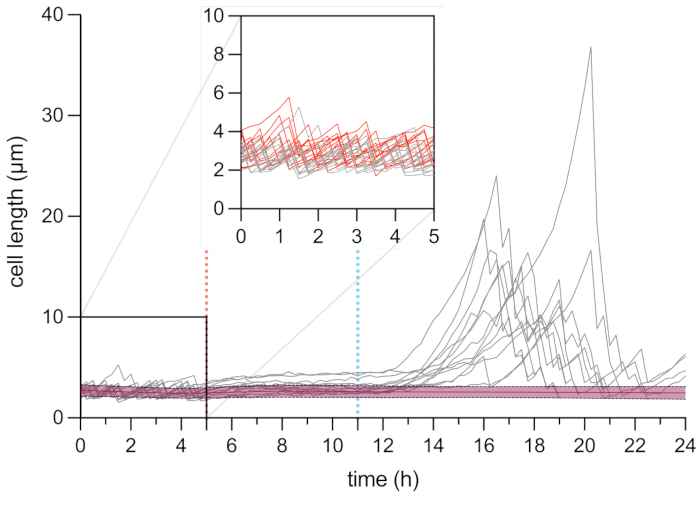
Figura 6: Análise microscópica time-lapse do comprimento das células persistentes e mortas. Análise do comprimento celular de células mortas (em vermelho, n = 109) e células persistentes (em cinza, n = 13). O início do tratamento com OFX (5 μg·mL−1) é indicado pela linha tracejada vermelha (5 h), e a remoção da OFX é indicada pela linha tracejada azul. O inset corresponde à fase de crescimento antes da adição de OFX. Os experimentos foram realizados em triplicata. As tonalidades e linhas tracejadas indicam os desvios padrão para a população de células mortas (n = 109). Clique aqui para ver uma versão maior desta figura.
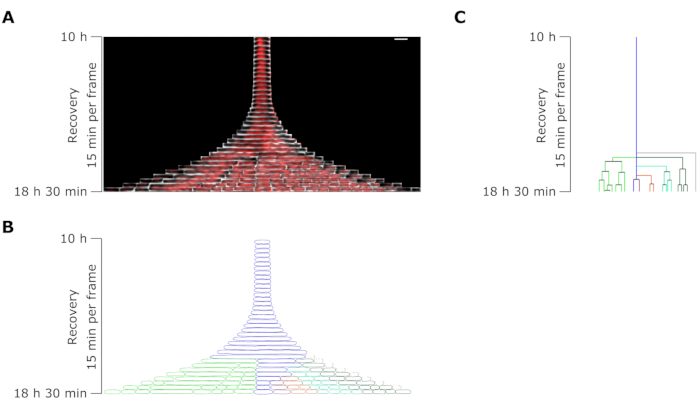
Figura 7: Análise de time-lapse de microscopia de um persistente representativo para OFX. (A) Quimógrafo de um persistente OFX representativo e suas células filhas geradas por divisões filamentares durante 8,5 h após a remoção de OFX (18,5 h após o início do experimento microfluídico, compreendendo 4 h de crescimento, 6 h de 5 μg·mL−1 de tratamento com OFX e 8,5 h de recuperação após remoção de OFX). Um quadro corresponde a 15 min. Barra de escala = 5 μm. (B) Máscara gerada a partir do quimógrafo persistente em A. A célula persistente monitorada é indicada com um contorno azul, e as células filhas são realçadas em cores distintas. (C) Representação esquemática da linhagem celular persistente gerada a partir de B. A codificação de cores é idêntica a B. Clique aqui para ver uma versão maior desta figura.
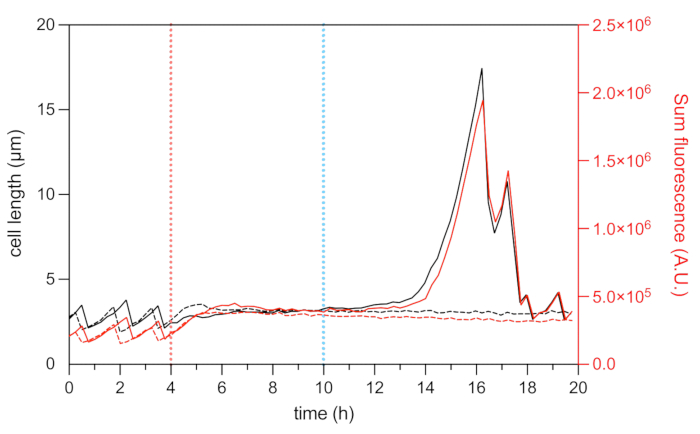
Figura 8: Comprimento celular e análise de fluorescência mCherry de células persistentes e mortas representativas. Análise do comprimento celular (eixo esquerdo) e da intensidade total de fluorescência HU-mCherry (eixo direito, mostrado em unidades arbitrárias) de um persistente representativo (linhas sólidas pretas e vermelhas) e de uma célula morta representativa (linha tracejada preta e vermelha) durante o experimento de lapso de tempo microfluídico. O início do tratamento com OFX (5 μg·mL−1) é indicado pela linha tracejada vermelha e a remoção da OFX pela linha tracejada azul. Clique aqui para ver uma versão maior desta figura.
Vídeo 1: Vídeo microfluídico de células de E. coli HU-mCherry antes, durante e após o tratamento com ofloxacina. Imagem microfluídica de lapso de tempo mostrando células HU-mCherry. As células foram cultivadas por 4 h em MOPS glicerol 0,4%. Após 6 h de tratamento com OFX (5 μg·mL−1), o meio livre de antibiótico foi perfundido na placa microfluídica para permitir a recuperação das células persistentes. Barra de escala = 5 μm. O tempo (em min) é indicado. As fases de crescimento e recuperação são indicadas por "MOPS- Gly. 0,4%" e o tratamento com OFX por "OFX 5 μg/mL". Clique aqui para baixar este vídeo.
| 10x MOPS | |||
| Solução de estoque | Volume de solução-mãe para 1 L de 10x MOPS | Concentração final em base MOPS 10x | |
| Ácido MOPS | 1 M (ajustado para pH 7,4 usando KOH) | 400 mL | 0.4 milh |
| Tricina | 1 M (ajustado para pH 7,4 usando KOH) | 40 mL | 0.04 milh |
| FeSO4.7H2O | 0.01 milh | 10 mL | 0.0001 milh |
| NH4Cl | 1.9 milh | 50 mL | 0.095 milh |
| K2SO4 | 0.276 milh | 10 mL | 0.00276 milh |
| CaCl2.2H 2O | 0.0005 milh | 10 mL | 0.000005 milh |
| MgCl 2.6H2O | 0.528 milh | 10 mL | 0.00528 milh |
| NaCl | adicionar diretamente 29.2 g | 0.5 milh | |
| Água destilada | 460 mL | ||
| Micronutrimentos 1000x (ver Tabela 2) | 10 mL | ||
Tabela 1: Composição de MOPS 10x.
| Micronutrientes 1000x | ||
| Concentração em Micronutriments 1000x solução estoque | Concentração final em base MOPS 10x | |
| (NH4)6Mo7O24.4H2O | 0.000003 milh | 0.00000003 milh |
| H 3BO3 | 0.0004 milh | 0.000004 milh |
| CoCl2.6H 2O | 0.00003 milh | 0.0000003 milh |
| CuSO4.5H 2O | 0.00001 milh | 0.0000001 milh |
| MnCl2.4H 2O | 0.00008 milh | 0.0000008 milh |
| ZnSO.7H2O | 0.00001 milh | 0,0000001M |
Tabela 2: Composição de 1.000x micronutrientes.
| MOPS glicose 0,4% ou MOPS glicerol 0,4% | |||
| Solução de estoque | Volume para 1 L MOPS glicose 0,4 % ou MOPS glicerol 0,4 % | Concentração final em MOPS glicose 0,4% ou MOPS glicerl 0,4% | |
| 10x MOPS | ver Tabela 1 | 100 mL | |
| K2HPO4 | 0.132 milh | 10 mL | 0.00132 milh |
| Glicose (para MOPS glicose 0,4%) | 20% (20 g em 100 mL de água destilada) | 20 mL | 0.40% |
| Glicerol (para MOPS glicerol 0,4%) | ≤99% | 4 mL | 0.40% |
| Água destilada | 870 mL para MOPS glicose 0,4% ou 886 mL para MOPS glicerol 0,4% | ||
Tabela 3: Composição da MOPS glicose 0,4% e MOPS glicerol 0,4%.
Discussão
O protocolo apresentado neste trabalho permite a análise do fenótipo de persistência observado em resposta ao tratamento com antibióticos em nível populacional e unicelular. Os experimentos foram realizados com a cepa MG1655 de E. coli, cultivada em meio quimicamente definido (glicerol MOPS 0,4%). Ensaios de time-kill e experimentos de microscopia foram realizados em culturas de fase exponencial. Usamos OFX, uma fluoroquinolona, na concentração de 5 μg·mL−1 para revelar as células persistentes. As abordagens aqui descritas podem ser aplicadas a outros antibióticos bactericidas, como β-lactâmicos, aminoglicosídeos ou compostos antimicrobianos31. Assim, outras cepas bacterianas, meios ou condições de crescimento podem ser usados. O monitoramento de diferentes fusões fluorescentes em uma configuração semelhante à descrita aqui pode ser útil para acompanhar processos celulares como replicação de DNA32, reparo de DNA25,33 e divisão celular34 antes, durante e após o tratamento com antibióticos. Da mesma forma, repórteres fluorescentes podem ser explorados para investigar aspectos distintos da fisiologia celular, como níveis intracelulares de pH35, ATP 36 ou ROS37. Alternativamente às fusões fluorescentes, corantes químicos também podem ser aplicados. Por exemplo, a fusão hupA-mCherry poderia ser substituída por 4',6-diamidino-2-fenilindol (DAPI), um corante fluorescente que cora o DNA38. A realização de microscopia de lapso de tempo acoplada a tais corantes fluorescentes deve, no entanto, ser evitada, uma vez que essas técnicas de coloração podem perturbar a dinâmica do ciclo celular durante experimentos de lapso de tempo. Alternativamente, tais experimentos podem ser substituídos por análises de curso temporal de imagens instantâneas em pontos de tempo relevantes.
Embora esses repórteres fluorescentes sejam úteis, a quantidade de informações que podem ser extraídas através da análise de imagens de contraste de fase não deve ser negligenciada. Aqui, monitoramos a evolução do comprimento celular ao longo dos estágios de crescimento, tratamento com OFX e recuperação. Outros parâmetros baseados em imagens de contraste de fase, como largura celular, intensidade de contraste de fase e curvaturas das células bacterianas, também podem ser extraídos com facilidade usando software adequado, como o MicrobeJ27.
Em resumo, o procedimento aqui descrito pode ser aplicado a outras condições e espécies bacterianas para monitorar respostas celulares a ambientes em mudança ou estressores18,19. Usando outros repórteres fluorescentes (repórteres transcricionais e translacionais, corante químico) em combinação com uma análise populacional, como citometria de fluxo/FACS, questões interessantes podem ser abordadas em uma estrutura multiescala.
Divulgações
Os autores declaram não haver interesses concorrentes.
Agradecimentos
O trabalho no laboratório Van Melderen é apoiado pelas ações ARC 2018-2023, o Fonds National de la Recherche Scientifique (FNRS CDR J.0182.21F). T.O. é apoiado por uma bolsa da ULB. T.S. é apoiado por uma bolsa FRIA (FNRS). J.C. é apoiado por uma bolsa de pós-doutorado "chargé de recherches" (FNRS).
Materiais
| Name | Company | Catalog Number | Comments |
| Axio Observer | Zeiss | Inverted fluorescence microscope | |
| CaCl2.2H2O | Merck | 1.02382.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CellASIC ONIX Microfluidic System | Merck | CAX2-S0000 | Microfluidic system |
| CellASIC ONIX2 FG | Merck | ONIX2 1.0.1 | Microfluidic software |
| CellASIC ONIX2 Manifold Basic | Merck | CAX2-MBC20 | Manifold system |
| CoCl2.6H2O | Merck | 1.02539.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CuSO4.5H2O | Merck | 1.02790.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| D-(+)-glucose | Sigma Aldrich | G7021-1KG | Carbon source for MOPS glucose 0.4% growth medium |
| E. coli K-12 MG1655 CF1648 (fnr+) | BE10 | wt reference strain, lab strain | |
| E. coli K-12 MG1655 CF1648 (fnr+) hupA-mCherry::FRT-kan-FRT | BE16 | HU-mCherry fusion integrated via P1 transduction at the native locus, lab strain | |
| FeSO4.7H2O | VWR Chemicals | 24244.232 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Fiji | ImageJ | https://fiji.sc/ | Image software; Schindelin et al. if used in publication |
| Glycerol | Merck | 56815 | Carbon source for MOPS glycerol 0.4% growth medium |
| Greiner CELLSTAR® multiwell culture plate | Merck | M8812-100EA | 24-well clear flat bottom 2ml volume culture plate for automated plate reader |
| H3BO3 | Sigma-Aldrich | B6768-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Hamamatsu ORCA Flash 4.0 digital camera | Hamamatsu | C13440-20CU | Digital Image Acqusition |
| K2HPO4 | Merck | 1.05099.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| KOH | Merck | 1.05029.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| K2SO4 | Merck | 1.05153.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Luria-Broth agar medium | Invitrogen | 22700041 | Growth medium for plating assay |
| Luria-Broth medium | Invitrogen | 12780029 | Growth medium for MIC determination |
| MgCl2.6H2O | Merck | 1.05832.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MgSO4 | Merck | 7487-88-9 | Dilution solution for bacterial survival assay used at 10mM |
| MicrobeJ | Imagej/Fiji plugin | https://www.microbej.com/ | Microscopy image analysis plugin. Ducret et al.for in publication; Detection settings: For bacteria : Area (μm2): 0,1-max; Length (μm): 0,5-max; Width (μm): 0,6-max; Range (μm): 0,5-max; Angularity (rad): 0-0,3; 0-max for all other parameters. |
| Microfluidic Plates CellASIC ONIX | Merck | B04A-03-5PK | Plate for microfluidic system |
| MnCl2.4H2O | Merck | 1.05927.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MOPS, Free Acid ULTROL® Grade | Merck | 475898-500GM | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NaCl | SIgma-Aldrich | S5886-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| (NH4)6Mo7O24.4H2O | Merck | 1.01180.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NH4Cl | Merck | 1.01145.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Ofloxacin | Merck | 82419-36-1 | Fluoroquinolone antibiotic used to treat the bacterial cells |
| Phosphate Buffered Saline (PBS) pH 7.4, 1x | Merck | P3813 | Dilution buffer |
| GENESYS 10S UV-Visible Spectrophotometer | Thermofisher Scientific | 840-208200 | UV-Visible Spectrophotometer, Single/Six Cell Holder with PrinterMeasurement of optical density OD600nm |
| SoftMax Pro | Molecular Devices | Microplate reader software | |
| SpectraMax i3x | Molecular Devices | Microplate reader | |
| N-[Tris(hydroxyméthyl)-méthyl]-glycine | Merck | 1.08602.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Zeiss® immersion Oil 518F | Zeiss | Immersion oil to increase resolution of microscope | |
| Zen3.2 Pro | Zeiss | Microscopic image acquisition and processing software | |
| ZnSO4.7H2O | Merck | 1.08883.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
Referências
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nature Reviews Microbiology. 17 (7), 441-448 (2019).
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nature Reviews Microbiology. 14 (5), 320-330 (2016).
- Pribis, J. P., Zhai, Y., Hastings, P. J., Rosenberg, S. M. Stress-induced mutagenesis, gambler cells, and stealth targeting antibiotic-induced evolution. mBio. 13 (3), 01074 (2022).
- Molina-Quiroz, R. C., Lazinski, D. W., Camilli, A., Levy, S. B. Transposon-sequencing analysis unveils novel genes involved in the generation of persister cells in uropathogenic Escherichia coli. Antimicrobial Agents and Chemotherapy. 60 (11), 6907-6910 (2016).
- Cameron, D. R., Shan, Y., Zalis, E. A., Isabella, V., Lewis, K. A genetic determinant of persister cell formation in bacterial pathogens. Journal of Bacteriology. 200 (17), 00303-00318 (2018).
- Cui, P., et al. Identification of genes involved in bacteriostatic antibiotic-induced persister formation. Frontiers in Microbiology. 9, 413 (2018).
- Li, T., et al. Identification of novel genes involved in Escherichia coli persistence to tosufloxacin. Frontiers in Cellular and Infection Microbiology. 10, 581986 (2020).
- Verstraeten, N., et al. Obg and membrane depolarization are part of a microbial bet-hedging strategy that leads to antibiotic tolerance. Molecular Cell. 59 (1), 9-21 (2015).
- Hofsteenge, N., van Nimwegen, E., Silander, O. K. Quantitative analysis of persister fractions suggests different mechanisms of formation among environmental isolates of E. coli. BMC Microbiology. 13 (1), 25 (2013).
- Huemer, M., Mairpady Shambat, S., Brugger, S. D., Zinkernagel, A. S. Antibiotic resistance and persistence-Implications for human health and treatment perspectives. EMBO Reports. 21 (12), 51034 (2020).
- Wilmaerts, D., Windels, E. M., Verstraeten, N., Michiels, J. General mechanisms leading to persister formation and awakening. Trends in Genetics. 35 (6), 401-411 (2019).
- Murawski, A. M., Brynildsen, M. P. Ploidy is an important determinant of fluoroquinolone persister survival. Current Biology. 31 (10), 2039-2050 (2021).
- Lemma, A. S., Soto-Echevarria, N., Brynildsen, M. P. Fluoroquinolone persistence in Escherichia coli requires DNA repair despite differing between starving populations. Microorganisms. 10 (2), 286 (2022).
- Balaban, N. Q., Merrin, J., Chait, R., Kowalik, L., Leibler, S. Bacterial persistence as a phenotypic switch. Science. 305 (5690), 1622-1625 (2004).
- Morawska, L. P., Kuipers, O. P. Antibiotic tolerance in environmentally stressed Bacillus subtilis: Physical barriers and induction of a viable but nonculturable state. microLife. 3, (2022).
- Windels, E. M., et al. Enrichment of persisters enabled by a ß-lactam-induced filamentation method reveals their stochastic single-cell awakening. Communications Biology. 2 (1), 426 (2019).
- Leygeber, M., et al. Analyzing microbial population heterogeneity-Expanding the toolbox of microfluidic single-cell cultivations. Journal of Molecular Biology. 431 (23), 4569-4588 (2019).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli growth rate to osmotic shock. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7807-7812 (2014).
- Shi, H., et al. Starvation induces shrinkage of the bacterial cytoplasm. Proceedings of the National Academy of Sciences of the United States of America. 118 (24), 2104686118 (2021).
- Goode, O., et al. Persister Escherichia coli cells have a lower intracellular pH than susceptible cells but maintain their pH in response to antibiotic treatment. mBio. 12 (4), 00909-00921 (2021).
- Manuse, S., et al. Bacterial persisters are a stochastically formed subpopulation of low-energy cells. PLoS Biology. 19 (4), 3001194 (2021).
- Fisher, J. K., et al. Four-dimensional imaging of E. coli nucleoid organization and dynamics in living cells. Cell. 153 (4), 882-895 (2013).
- Cabral, D., Wurster, J., Belenky, P. Antibiotic persistence as a metabolic adaptation: Stress, metabolism, the host, and new directions. Pharmaceuticals. 11 (1), 14 (2018).
- Wiegand, I., Hilpert, K., Hancock, R. E. W. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nature Protocols. 3 (2), 163-175 (2008).
- Goormaghtigh, F., Van Melderen, L. Single-cell imaging and characterization of Escherichia coli persister cells to ofloxacin in exponential cultures. Science Advances. 5 (6), (2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ducret, A., Quardokus, E. M., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nature Microbiology. 1 (7), 16077 (2016).
- Odsbu, I., Skarstad, K. DNA compaction in the early part of the SOS response is dependent on RecN and RecA. Microbiology. 160 (5), 872-882 (2014).
- Pham, T. D. M., Ziora, Z. M., Blaskovich, M. A. T. Quinolone antibiotics. MedChemComm. 10 (10), 1719-1739 (2019).
- Drlica, K., et al. Quinolones: Action and resistance updated. Current Topics in Medicinal Chemistry. 9 (11), 981-998 (2009).
- Kohanski, M. A., Dwyer, D. J., Collins, J. J. How antibiotics kill bacteria: From targets to networks. Nature Reviews Microbiology. 8 (6), 423-435 (2010).
- Reyes-Lamothe, R., Sherratt, D. J., Leake, M. C. Stoichiometry and architecture of active DNA replication machinery in Escherichia coli. Science. 328 (5977), 498-501 (2010).
- Lesterlin, C., Ball, G., Schermelleh, L., Sherratt, D. J. RecA bundles mediate homology pairing between distant sisters during DNA break repair. Nature. 506 (7487), 249-253 (2014).
- Alexeeva, S., Gadella, T. W. J., Verheul, J., Verhoeven, G. S., Den Blaauwen, T. Direct interactions of early and late assembling division proteins in Escherichia coli cells resolved by FRET: Bacterial division studied by spectral FRET. Molecular Microbiology. 77 (2), 384-398 (2010).
- Miesenböck, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Yaginuma, H., et al. Diversity in ATP concentrations in a single bacterial cell population revealed by quantitative single-cell imaging. Scientific Reports. 4, 6522 (2014).
- Ermakova, Y. G., et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide. Nature Communications. 5, 5222 (2014).
- Kapuscinski, J. DAPI: A DNA-specific fluorescent probe. Biotechnic & Histochemistry. 70 (5), 220-233 (1995).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados