Method Article
대장균의 항생제 지속성에 대한 개체군 및 단일 세포 분석
요약
항생제 지속성은 민감한 동종 집단 내의 작은 하위 집단이 고용량의 살균 항생제를 일시적으로 견딜 수 있는 능력을 설명합니다. 현재 프로토콜은 대장균 을 치사량의 오플록사신에 노출시킨 후 분자 및 세포 수준에서 항생제 지속성 표현형을 특성화하기 위한 접근 방식을 결합합니다.
초록
항생제 지속성은 고용량의 살균성 항생제를 일시적으로 견딜 수 있는 작은 박테리아 하위 집단의 능력을 나타냅니다. 살균 항생제 처리시 박테리아 집단의 대부분이 빠르게 죽습니다. 이 첫 번째 빠른 살상 단계는 지속 세포가 생존 할 수 있기 때문에 살상 비율이 크게 감소합니다. 일반적으로 지속성은 고용량의 항생제로 수행된 시간/사멸 분석과 정의된 노출 시간에 의해 모집단 수준에서 결정됩니다. 이 방법은 지속성 세포의 수준과 사멸 동역학에 대한 정보를 제공하지만 지속성 현상의 기저에 있는 고유한 세포 간 이질성을 반영하지 못합니다. 여기에 설명된 프로토콜은 실시간 형광 현미경을 사용하는 단일 세포 분석과 고전적인 시간/사멸 분석을 결합합니다. 적절한 형광 리포터를 사용하여 살아있는 세포의 현미경 이미징은 염색체 복제 및 분리, 세포 신장 및 세포 분열과 같은 세포 과정에 대한 항생제의 영향에 관한 정보를 제공할 수 있습니다. 집단과 단일 세포 분석을 결합하면 지속성 표현형의 분자 및 세포 특성 분석이 가능합니다.
서문
이 프로토콜은 단일 세포 및 집단 수준에서 특정 항생제 치료에 대한 반응으로 박테리아 지속성 표현형을 분석하는 것을 목표로 합니다. 지속성은 동종 집단 내의 작은 하위 집단이 고용량의 살균 항생제(플루오로퀴놀론, 아미노글리코사이드, β-락탐 등)를 견딜 수 있는 능력을 설명하며, 소위 지속성 세포의 최소 억제 농도(MIC)는 대부분의 집단과 동일합니다. 항생제가 있는 상태에서 시간 경과에 따른 박테리아 생존을 측정할 때 이상성 사멸 역학은 일시적으로 약물 내성 세포의 존재를 나타내며, 비지속성 세포의 초기 빠른 박멸과 지속성 세포의 사멸 속도가 훨씬 느립니다. 항생제 제거 시, 이 세포들은 동일한 항생제로 치료했을 때 유사한 살상 역학을 나타내는 유전적으로 동일한 집단을 생성한다 1,2. 지속성과 대조적으로, 항생제 내성은 집단 수준에서 정의되며 일반적으로 새로운 돌연변이 또는 내성 부여 플라스미드3의 수평 유전자 전달의 결과입니다. 내성을 담당하는 돌연변이는 대부분 약물의 표적 또는 약물 유출 펌프의 프로모터 영역에 위치하지만, 게놈 전체 및 표적 돌연변이 분석 접근법에 의해 확인된 지속성 빈도를 변경하는 유전자는 다양하고 다양한 것으로 입증되었습니다 2,3,4,5,6,7,8 . 따라서 박테리아 세포는 여러 경로 9,10,11을 통해 지속성 상태로 들어갈 수 있으며 이러한 지속성 세포의 생리학을 특성화하기 위해서는 단일 세포 수준에서 지속성 현상을 조사하기 위한 접근 방식이 필요합니다.
형광 현미경과 함께 사용되는 미세유체 도구의 최근 개발은 지속성 표현형의 특성화를 위한 길을 열었고 지속성 세포 형성에서 염색체 복제(염색체 복제) 12, DNA 복구(DNA repair) 13 및 세포 분열(cell division)14과 같은 주요 세포 과정의 역할을 강조했습니다. 이 논문에서 우리는 고용량의 오플록사신으로 처리된 기하급수적으로 성장하는 대장균 배양에서 생성된 지속성 세포를 특성화하기 위해 고전적인 미생물학 분석과 단일 세포 라이브 이미징을 결합한 통합 접근 방식을 설명합니다. 여기에 기술된 프로토콜은 바실러스 서브틸리스(Bacillus subtilis)15와 같은 다른 박테리아 종에서의 항생제 지속성 현상 또는 조건(예를 들어, β-락탐 처리 후 항생제 지속성 16)을 연구하는 데 적용될 수 있으며, 표현형 이질성17,18,19과 관련된 많은 현상을 조사하기 위해 쉽게 수정될 수 있다 . 또한, 이 논문에 설명된 설정은 다른 형광 리포터와 결합하여 단일 세포 수준에서 pH20 또는 ATP21의 세포 내 수준과 같은 관심 있는 뚜렷한 세포 매개변수를 조사할 수 있으며, 이는 잠재적으로 항생제 지속성 현상에 대한 새로운 통찰력을 생성할 수 있습니다.
프로토콜
알림: 멸균 배양 유리 제품, 피펫 팁 및 성장 배지를 사용하십시오. 여기서, 대장균 세포는 화학적으로 정의된 저자가형광 배지에서 성장하였다( 재료 표 참조). 오염 위험을 최소화하기 위해 분젠 버너가 있는 상태에서 접종을 실시했습니다.
1. 세포 배양 및 성장 곡선
- Luria-Bertaini(LB) 한천 플레이트(필요한 경우 선택적 항생제가 보충됨)에 냉동 글리세롤 스톡에서 관심 균주를 줄무늬로 만들고 37°C에서 하룻밤(15시간에서 19시간 사이) 배양하여 단일 콜로니를 얻습니다.
참고: 여기에 제시된 실험은 wt 균주에 해당하는 E. coli K-12 MG1655와 동종 MG1655 hupA-mCherry 균주22의 두 가지 균주를 사용합니다. 후자의 균주는 HU 뉴클레오이드 관련 단백질의 형광 태깅된 α-서브유닛을 발현한다. hupA-mCherry 리포터는 hupA의 기본 위치에 통합됩니다. HU-mCherry는 비특이적 방식으로 DNA에 결합하기 때문에 살아있는 세포의 뉴클레오이드 역학을 추적하는 프록시 역할을 합니다. - 5 mL의 배지(여기서, 3-[N-morpholino]propanesulfonic acid-based medium[MOPS]; 표 1, 표 2 및 표 3)에서 분리된 콜로니와 함께 글루코스 0.4% 및 선택적 항생제 (필요한 경우)를 보충한 유리 튜브 (≥25 mL)에 넣고, 튜브를 37°C 및 분당 180회전(rpm)으로 설정된 진탕 배양기에 하룻밤 동안(15 h 및 19 h 사이) 두었다. 또는 유리관 대신 플라스틱 튜브, 유리 또는 플라스틱 플라스크(≥ 25mL)를 사용할 수 있습니다.
참고: 0.4%의 최종 농도로 글리세롤이 보충된 MOPS 배지는 MOPS 포도당 0.4%에서 수행된 하룻밤 배양을 제외하고 이 논문에 설명된 실험 전반에 걸쳐 사용되었습니다. 포도당이 보충된 MOPS에서 대장균 의 생성 시간은 MOPS 글리세롤보다 짧습니다. 이 단계에서 MOPS 글리세롤 0.4% 대신 MOPS 포도당 0.4%를 사용하면 세포가 19시간 이내에 정지 상태에 도달합니다. 뚜렷한 탄소원이 보충된 M9 또는 RDM(Rich Defined Medium)과 같은 다른 성장 배지도 사용할 수 있습니다. 그러나, 성장률 및 지속성 빈도는 사용된 배지 및/또는 탄소에 따라 다르다는 점에 유의해야 한다23. - 다음날 아침, 2,300 x g 에서 3분 동안 배양물 1mL를 원심분리하고, 상청액을 버리고, 펠릿을 동일한 부피의 인산염 완충 식염수(PBS)에 부드럽게 재현탁합니다. 600 nm(OD600 nm)에서 광학 밀도를 측정하고, 2 mL의 최종 부피에서 0.01의 초기 OD600 nm 에 필요한 부피를 계산한다.
- 0.4% MOPS 글리세롤 배지 2mL를 투명한 바닥 24웰 플레이트의 웰에 넣고 계산된 하룻밤 배양 부피를 접종합니다. 24웰 플레이트를 자동화된 마이크로플레이트 리더( 재료 표 참조)에 넣어 24시간 동안 OD600nm 를 모니터링합니다. 마이크로플레이트 리더를 37°C의 온도와 높은 궤도 회전(140rpm)에서 15분마다 OD600nm 를 측정하도록 설정합니다.
참고: 실험에 사용된 균주가 형광 리포터를 인코딩하는 경우, 성장률이 항생제 지속성 빈도에 영향을 미치므로 후속 실험에서 아티팩트를 피하기 위해 성장률이 wt 의 성장률과 비슷한지 확인하십시오23. 매우 낮은 세포 밀도에서의 검출 한계와 OD600nm/집락 형성 단위(CFU) 관계에 대한 잠재적인 균주별 영향으로 인해 특성화되지 않은 균주로 작업할 때 CFU·mL-1 을 모니터링하여 성장 역학을 평가하는 것이 좋습니다.
2. 항생제의 최소 억제 농도 결정
참고: 최소 억제 농도(MIC)는 박테리아 성장이 관찰되지 않는 가장 낮은 용량의 항생제로 정의됩니다. MIC의 결정은 각 항생제 및 균주에 대해 수행되어야 합니다. 여기에 기술된 실험에서, 플루오로퀴놀론 항생제 오플록사신(OFX)을 사용하였다. MIC의 결정은 항생제 용액이 올바르게 준비되었는지, 항생제가 활성인지, 균주가 항생제에 동등하게 민감한지 확인할 수 있습니다. 여기서, 공개된 한천 희석법은 사용된24개의 다른 균주의 OFX에 대한 MIC를 확인하기 위해 수행되었다. 주어진 박테리아 균주에 대한 주어진 항생제의 MIC는 또한 브로스 희석 방법24를 통해 결정될 수 있다.
- MIC 결정을위한 플레이트 준비
- 초순수 1mL에 OFX 5mg을 용해시켜 실험에 사용된 항생제의 마스터 원액을 준비합니다. OFX의 용해도를 높이기 위해 20 μL의 37% HCl을 첨가합니다.
- 멸균 LB 한천 100mL를 녹이고 응고를 방지하기 위해 55°C에서 보관합니다. 6개의 작은 유리 플라스크(25 mL)를 준비하고, 멸균 피펫을 사용하여 5 mL의 액체 LB 한천 배지를 각 플라스크에 피펫팅한다.
- 10 μL의 5 mg·mL-1 OFX 마스터 스톡 용액을 90 μL의 초순수에 희석합니다(마스터 스톡의 1:10 희석). 5 mL의 LB 한천 배지가 들어있는 6 개의 유리 플라스크 각각에 500 μg·mL-1 OFX 용액의 0 μL, 2 μL, 4 μL, 6 μL, 8 μL 또는 10 μL를 첨가하여 최종 농도가 0 μg·mL-1, 0.02 μg·mL-1, 0.04 μg·mL-1, 각각 0.06 μg·mL-1, 0.08 μg·mL-1 및 0.1 μg·mL-1 OFX입니다. 플라스크를 여러 번 회전시켜 용액을 혼합하십시오.
참고: 관심 항생제의 MIC를 알고 있는 경우 농도 범위는 실제 MIC의 아래에서 위로 도달해야 합니다. MIC를 알 수 없는 경우 log2 희석 시리즈를 사용한 광범위한 농도가 권장됩니다. 고온에서 항생제를 비활성화할 수 있으므로 항생제를 추가하기 전에 LB 한천 배지가 냉각되었는지 확인하십시오. 그럼에도 불구하고 항생제를 첨가하기 전에 LB 한천 배지가 응고되지 않도록 하는 것이 중요한데, 이는 LB 한천 배지에서 항생제의 비균질한 분포로 이어질 수 있기 때문입니다. - 단계 2.1.3에서 제조된 6개의 LB 한천 배지를 각각 5 mL씩 용량 증량 방식으로 멸균 피펫을 이용하여 6-웰 배양 플레이트에 붓는다. 한천이 응고될 때까지 식히고 사용하기 전에 플레이트를 건조시키십시오.
참고: 항생제 용액을 준비하고 분석이 수행되는 날 배양 접시를 준비합니다.
- MIC 측정 분석
- 분리된 콜로니와 함께 5 mL의 LB 배지를 유리관(≥25 mL)에 접종하고, 유리관을 37°C 및 180rpm으로 설정된 진탕 인큐베이터에 하룻밤 동안(15시간 내지 19시간 사이) 넣는다.
- 다음날 아침, OD600 nm를 측정하고, 배양액을 시험관에 희석하여 PBS에서 1 x 107 CFU·mL-1 의 최종 세포 밀도로 한다. 여기에서 테스트한 균주의 경우 1 x 107 CFU·mL-1 은 0.0125의 OD600nm 에 해당합니다.
참고: CFU·mL-1과 OD 600nm 사이의 상관관계를 알 수 없는 경우 CFU·mL-1과 OD 600nm 사이의 상관 계수를 계산하기 위해 CFU 및 OD600nm 측정에 의해 결정된 성장 곡선을 설정해야 합니다. - 미리 희석된 배양액 2 μL를 건조된 6-웰 플레이트의 모든 웰에 스팟합니다. 플레이트를 37°C의 인큐베이터에 하룻밤(15시간에서 19시간 사이) 넣기 전에 반점을 건조시키십시오.
- 다음날, 각 우물에 형성된 식민지를 세십시오. MIC는 박테리아 성장이 감지되지 않는 항생제의 최소 농도를 가진 우물에 해당합니다.
3. 스팟 분석
참고: 스팟 분석 방법은 생존 가능한 세포(항생제 스트레스 후 콜로니를 생성할 수 있는 세포)의 수를 추정할 수 있는 정성적 접근 방식입니다. 스팟 분석은 테스트된 조건에서 사용된 균주의 생존력에 대한 통찰력을 제공하고 타임 킬 분석 중에 필요한 희석에 대해 알리기 위해 타임 킬 분석 전에 수행됩니다(섹션 4 참조).
- 스팟 분석을 위해 LB 한천 플레이트를 준비하려면 LB 한천 50mL를 정사각형 페트리 접시(144cm2)에 붓습니다. 시점당 하나의 정사각형 페트리 접시를 준비합니다. LB 한천을 굳히고 사용하기 전에 플레이트를 건조시킵니다.
- 유리관(≥25mL)에 분리된 콜로니와 함께 배지(MOPS 포도당 0.4%)의 5mL를 접종하고, 튜브를 37°C 및 180rpm으로 설정된 진탕 배양기에 밤새(15시간에서 19시간 사이) 넣습니다.
- 다음날, OD 600 nm를 측정하고, 배양액을 신선한 온도 조절 배지(37°C, MOPS glycerol 0.4%)로 유리관(≥25 mL)에 넣어 ~0.001의 최종OD600 nm로 희석하였다. 배양물을 37°C의 180rpm(15시간에서 19시간 사이)의 인큐베이터에서 밤새 성장시킵니다.
- 다음날에, OD600 nm를 측정하고, 배양물을 0.3의 최종OD600 nm 로 배양한다.
- 인큐베이션하는 동안, 첫 번째 행(행 A)의 웰을 제외한 모든 웰에 0.01 MMgSO4 용액 90 μL를 배치하여 96-웰 플레이트를 준비한다. 96웰 플레이트는 3.7단계에서 10배 연속 희석에 사용됩니다.
참고: 이 실험에서는 두 가지 균주(wt 및 hupA-mCherry 균주)를 7개의 서로 다른 시점에서 세 번 테스트했습니다. 하나의 플레이트가 12개의 컬럼으로 구성되어 있기 때문에 하나의 플레이트를 2개의 시점에 사용할 수 있으므로 총 4개의 플레이트를 준비했습니다. - 0.3의 OD600 nm 에서 각 박테리아 배양 물 200 μL를 회수합니다. 이 샘플은 항생제 치료 전의 t0(미처리) 시점에 해당하며 항생제 치료 전 CFU·mL-1 을 측정할 수 있습니다.
- 시료를 2,300 x g 에서 3분 동안 원심분리합니다. 원심분리 시간 동안, 원하는 농도의 OFX를 액체 배양물에 첨가하고, 진탕하면서 37°C에서 계속 배양한다.
참고: 여기서 OFX는 5μg·mL-1 (MIC를 83배 곱한 값에 해당)의 농도로 사용되었습니다. 이전 연구에서, 이 농도는 OFX 노출25 하에서 지속성 현상을 특성화하는 데 사용되었다. 시간/사멸 분석에 사용되는 항생제 농도는 항생제, 배양 배지 및 박테리아 성장 상태에 따라 달라질 수 있습니다. - 원심분리 후, 세포 펠릿을 0.01 MMgSO4 용액 200 μL에 재현탁시킨다. 단계 3.5에서 제조한 96-웰 플레이트의 빈 웰에 100 μL를 넣는다. 10 μL의 웰을 행 A의 웰에 0.01 MMgSO4의 90 μL를 함유하는 행 B의 웰로 옮겨 희석을 수행한다. B행의 웰 10μL를 C행의 웰로 옮겨 연속 희석을 계속합니다. 10-7 의 희석에 도달할 때까지 반복합니다(모든 전송은 10배 희석됨).
참고: 이 실험에서는 모든 시점에 대해 6개의 배양물( wt 균주에 대해 3개, hupA-mCherry 균주에 대해 3개)을 테스트했습니다. 프로토콜에 따르면, 다중 채널 피펫은 동시에 6 개의 균주에 대한 연속 희석을 수행하는 데 사용됩니다. 기술적 오류를 최소화하기 위해 파이펫 팁은 이송할 때마다 교체됩니다. - 각 희석액 10μL를 단계 3.1에서 제조된 LB 한천 플레이트에 스팟합니다.
참고: 다중 채널 피펫을 사용하여 6개 배양에 대해 동일한 희석액(예: 10-4 희석액)을 동시에 검출합니다. 스팟팅하는 동안 두 번째 스톱을 누르지 않고 다중 채널 피펫의 첫 번째 스톱을 사용해야 하며, 이로 인해 미세 방울이 플레이트에 분배될 수 있습니다. - 항생제 첨가 후 관련 시점에서 배양액 200μL를 회수하고 3.8단계에 설명된 대로 연속 희석을 수행합니다. 3.9단계에 설명된 대로 각 희석액 10μL를 스팟합니다.
참고: 여기에 설명된 실험에 대해 7개의 시점(t0, t1h, t2h, t3h, t4h, t5h, t6h)을 수집했습니다. 중량에 비해 증가된 항생제 감수성을 나타내는 균주의 경우 , 잔류 항생제를 제거하기 위해MgSO4 0.01 M에서 다중 세척을 수행할 수 있습니다. - 플레이트를 37°C에서 하룻밤(15시간에서 19시간 사이)에 배양합니다. 다음날, 콜로니를 검출 할 수있는 두 개의 가장 높은 희석액에서 콜로니의 수를 세십시오. 이상적으로, 이러한 희석을 위한 한천 플레이트의 반점에는 3-30개의 콜로니가 포함되어 있어 각 샘플에 대한 CFU·mL-1 을 정확하게 측정할 수 있습니다.
- 각 시점의 계산된 CFU·mL-1을 t0에서 초기 모집단의 CFU·mL-1로 나누어 생존율을 계산합니다.
4. 타임 킬 어세이
참고: 스팟 분석은 주어진 항생제에 대해 주어진 균주의 생존율을 추정하는 데 사용하기 쉬운 방법이지만, 시간 사멸 분석은 더 높은 분해능 생존율을 제공하고 박테리아 생존율을 정확하게 정량화하기 위해 수행됩니다. 사멸 곡선의 프로파일은 주어진 박테리아 균주가 주어진 조건에서 항생제에 민감한지, 내성이 있는지 또는 내성이 있는지를 결정하는 데 사용할 수 있습니다. 또한, 시간 사멸 분석을 통해 지속성 현상(이상성 사멸 곡선의 두 번째 기울기 시작)과 지속성 빈도를 감지하는 데 필요한 항생제 노출 시간을 결정할 수 있습니다.
- 타임 킬 도금 분석을 위해 LB 한천 플레이트를 준비합니다. LB 한천 25mL를 페트리 접시 (±57cm2)에 붓습니다. 시점당 최소 2개의 페트리 접시를 준비합니다(균주당 시점당 2개의 희석액이 도금됨).
- LB 한천을 고형화시키고 각 플레이트에 5-8 개의 멸균 유리 비드를 추가하기 전에 플레이트를 건조시킵니다. 변형률/조건/시점에 따라 플레이트를 뒤집고 라벨을 붙입니다.
알림: 유리 구슬을 사용하면 4.7단계와 4.9단계에서 한천 플레이트에 박테리아가 퍼질 수 있습니다. 대안적으로, 세포는 스프레더를 사용하여 한천 플레이트 상에 분산될 수 있다. - 각 샘플에 대해 900μL의 0.01M MgSO4 용액을 포함하는 10배 연속 희석 유리관을 준비합니다. 준비해야 하는 샘플당 희석 유리관의 수는 스팟 분석에서 콜로니를 검출하는 데 필요한 희석액과 일치합니다.
- 유리관(≥25mL)에 분리된 콜로니와 함께 배지(MOPS 포도당 0.4%)의 5mL를 접종하고, 튜브를 37°C 및 180rpm으로 설정된 진탕 배양기에 밤새(15시간에서 19시간 사이) 넣습니다.
- 16시간 성장 후, OD 600 nm를 측정하고, 배양물을 신선한 온도 조절 배지(37°C, MOPS 글리세롤 0.4%)로 유리관에 넣어 ~0.001의 최종 OD600 nm로 희석하였다. 배양물을 37°C의 180rpm(15시간에서 19시간 사이)의 인큐베이터에서 밤새 성장시킵니다.
- 다음날에, OD600 nm를 측정하고, 배양물을 0.3의 최종OD600 nm 로 배양한다.
- 0.3의 OD 600 nm에서, 배양 물 100 μL을 회수하고, 스팟 분석에서 얻은 데이터에 따라 희석한다 (0.3의 OD600 nm에서 항생제없이, 10-5 희석은 보통 200-300 콜로니를 제공한다), 4.1-4.2 단계에서 제조 된 LB 한천 플레이트에 100 μL를 플레이트한다. 비드가 접시 가장자리에 닿지 않도록 플레이트를 부드럽게 흔들면 LB 한천 배지에서 박테리아 세포가 균질하지 않게 퍼질 수 있습니다. 항생제 치료 전의 이 첫 번째 샘플은 t0 시점(항생제 치료 전 CFU·mL-1)에 해당합니다.
- 원하는 농도의 OFX를 첨가하고, 진탕하면서 37°C에서 계속 배양한다.
참고: 여기서 OFX는 5μg·mL-1 (MIC를 83배 곱한 값에 해당)의 농도로 사용되었습니다. - 항생제 첨가 후 관련 시점에서 배양액 100 μL를 회수하고 스팟 분석에서 얻은 데이터에 따라 희석하고 4.1-4.2 단계에서 제조된 LB 한천 플레이트에 100 μL를 플레이트합니다. 단계 4.7에 설명된 대로 세포를 플레이트합니다.
참고: 여기에 설명된 실험에 대해 7개의 시점(t0, t1h, t2h, t3h, t4h, t5h, t6h)을 수집했습니다. 중량에 비해 증가된 항생제 감수성을 나타내는 균주의 경우, 잔류 항생제를 제거하기 위해MgSO4 0.01 M에서 다중 세척을 수행할 수 있습니다. - 플레이트를 37°C에서 하룻밤(15시간에서 19시간 사이)에 배양합니다. 다음날, 콜로니를 검출 할 수있는 두 개의 가장 높은 희석액에서 콜로니의 수를 세십시오. 이상적으로, 플레이트는 각 샘플에서 CFU·mL-1 을 정확하게 측정할 수 있도록 30-300개의 콜로니를 포함해야 합니다.
- 각 시점에서 CFU·mL-1을 t0에서 CFU·mL-1로 정규화하여 생존율을 계산합니다. 정규화된 로그10 CFU·mL-1을 시간 함수로 플로팅합니다.
5. 미세유체 타임랩스 현미경 이미징
참고: 다음 섹션에서는 미세유체 플레이트의 준비와 타임랩스 이미지 획득 및 이미지 분석 절차에 대해 설명합니다. 이 실험의 목적은 단일 세포 수준에서 항생제 치료 시 지속성 표현형을 관찰하고 분석하는 것입니다. 이 실험 중에 수집된 데이터는 해결된 질문 및/또는 실험 중에 사용된 형광 리포터에 따라 광범위한 결과를 생성하는 데 사용할 수 있습니다. 여기에 기술된 실험에서, 지속세포 및 비지속세포에서 핵체 조직을 반영하는 HU-mCherry 형광22의 정량적 분석을 수행하였다.
- 미세유체 타임랩스 현미경을 위한 박테리아 세포 배양
- 유리관(≥25mL)에 분리된 콜로니를 배지(MOPS 글리세롤 0.4%, 필요한 경우 선택적 항생제로 보충됨) 5mL를 접종하고 튜브를 37°C 및 180rpm으로 설정된 진탕 배양기에 하룻밤 동안(15시간에서 19시간 사이) 놓습니다.
- 다음날, OD 600 nm를 측정하고, 배양액을 신선한 온도 조절 배지(37°C, MOPS 글리세롤 0.4%)에서 ~0.001의 최종OD600 nm로 유리관에 희석하였다. 배양물을 37°C 및 180rpm의 진탕 인큐베이터에서 하룻밤(15시간 내지 19시간 사이)에 성장시켜 다음날 초기 지수상 배양액을 얻었다.
- 미세유체 플레이트 및 타임랩스 현미경 이미징의 준비
참고: 미세유체 실험은 상업적으로 이용 가능한 미세유체 장치(여기에 설명된 대로) 또는 사내에서 생산된 미세유체 시스템에서 수행할 수 있습니다.- 미세유체 플레이트의 모든 웰에서 보존 용액(존재하는 경우)을 제거하고 새로운 배양 배지로 교체합니다.
알림: 미세유체판에 폐기물 배출구 웰이 포함되어 있는 경우 배출구 웰의 보존 용액을 제거해야 하지만 매체로 교체해서는 안 됩니다. - Seal 버튼을 클릭하거나 미세 유체 소프트웨어를 통해 매니폴드 시스템으로 미세유체 플레이트를 밀봉합니다(먼저 Tool을 선택한 다음 Seal Plate를 선택).
알림: 플레이트를 밀봉하려면 플레이트를 매니폴드에 수동으로 압착하여 플레이트와 매니폴드에 균일한 압력을 가해야 합니다. 올바르게 수행되면 ONIX2 인터페이스에 "sealed"라는 메모가 나타나야 합니다. 매니폴드가 파손될 수 있는 잠재적인 위험을 피하기 위해 유리 슬라이드에 압력을 가하지 않는 것이 중요합니다. - 밀봉되면 첫 번째 프라이밍 시퀀스를 수행합니다(미세유체 소프트웨어 인터페이스에서 Run Liquid Priming Sequence 클릭).
알림: 액체 프라이밍 시퀀스 실행 은 웰 5-6.9에 대해 1kPa에서 5분의 관류, 5우물에 대해 6.9kPa에서 8분의 관류, 6kPa에서 5분 동안 웰 6.9에 대한 최종 관류 라운드에 해당합니다. Run Liquid Priming Sequence 를 사용하면 서로 다른 웰을 연결하는 채널에 여전히 존재할 수 있는 보존 용액을 제거할 수 있습니다. - 현미경 이미징을 시작하기 전에 최소 2시간 동안 원하는 온도(여기서는 37°C)에서 온도 조절 장치로 제어되는 현미경 캐비닛에서 플레이트를 배양합니다.
- 실험을 시작하기 전에 두 번째 Run Liquid Priming Sequence 를 시작합니다.
- microfluidic 소프트웨어 인터페이스에서 Seal off(씰 오프)를 클릭하여 미세유체 플레이트를 밀봉합니다. 웰 1과 웰 2의 배지를 200 μL의 신선한 배지로, 웰 3을 항생제가 들어있는 신선한 배지 200 μL로 교체하십시오 (여기서는 5 μg·mL-1의 OFX), 웰 4 및 웰 5는 200 μL의 신선한 배지로, 웰 6에는 200 μL의 신선한 배지로, 그리고 웰 8에는 OD 600 nm로 희석 된 배양 샘플200 μL의 배양 샘플 (5.1.2 단계에서) 신선한 배지에서 0.01.
- 5.2.2 단계에 설명된 대로 미세유체 플레이트를 밀봉하고 플레이트를 현미경 캐비닛 내부의 현미경 대물렌즈에 놓습니다.
알림: 미세유체 플레이트를 배치하기 전에 현미경 대물렌즈에 침지 오일 한 방울을 떨어뜨려야 합니다. - 미세유체 소프트웨어에서 Cell Loading 을 클릭하여 세포가 미세유체 플레이트에 로딩되도록 합니다.
참고: 세포 로딩 단계는 웰 8에 대해 13.8kPa에서 15초의 관류, 웰 6 및 웰 8에 대해 27.6kPa에서 15초의 관류, 6.9kPa에서 30초 동안 웰 6에 대한 최종 관류 라운드로 구성됩니다. 미세유체 플레이트의 세포 밀도는 실험에 매우 중요합니다. 이 미세유체 프로토콜의 첫 번째 부분은 항생제 치료 전에 신선한 배지에서 6시간 동안 박테리아를 성장시키는 것으로 구성됩니다. 6시간의 성장 후, 세포 밀도는 희귀한 지속세포 세포를 검출하기에 충분해야 합니다(이 연구에 사용된 조건에서 지속세포 세포는 10-4의 빈도로 생성됨). 세포 밀도가 너무 높으면 개별 세포를 구별하기 어려워 정확한 단일 세포 분석이 불가능합니다. 성장률은 배지에 직접적으로 의존하기 때문에 실험을 시작하기 전에 현미경 분야의 세포 밀도를 평가해야 합니다. - 투과광 모드를 사용하여 최적의 초점을 설정하고 적절한 셀 번호가 관찰되는 여러 관심 영역(ROI)을 선택합니다(필드당 최대 300개 셀).
참고: 최소 40개의 ROI를 선택하여 희귀 지속형 셀이 이미지화되도록 합니다. - microfluidic 소프트웨어에서 Create a Protocol(프로토콜 생성)을 클릭합니다. 6 시간 동안 6.9 kPa (웰 1-2)에서 신선한 배지를 주입 한 다음, 6 시간 동안 6.9 kPa에서 항생제가 포함 된 배지를 주입 (웰 3)하고, 마지막으로 6.9 kPa에서 20 시간 동안 신선한 배지를 주입합니다 (웰 4-5).
참고: 0.01의 OD600nm 로 희석된 박테리아 배양은 박테리아가 6시간 동안 미세유체 챔버에서 성장할 수 있도록 하여 세포가 기하급수적인 성장 단계에 있도록 합니다. 로딩 단계(단계 5.2.8 참조) 동안 마이크로유체 장치에 도입된 세포의 수에 따라, 성장 단계의 지속 기간은 ROI 당 최대 300개의 세포를 얻도록 적응될 수 있다. 지속성은 드문 현상이므로 ROI당 세포 수를 늘리면 지속성 세포를 관찰할 가능성이 높아집니다. 그러나 세포 수는 단일 세포 분석을 지루하게 만들기 때문에 ROI당 300개의 세포를 초과해서는 안 됩니다. - 타임랩스 모드에서 투과광과 형광 리포터용 여기 광원을 사용하여 15분마다 한 프레임으로 현미경 이미징을 수행합니다. 여기서, mCherry 신호에 대한 560 nm 여기 광원을 사용하였다 (필터 00 [530-585 ex, 615LP em, Zeiss] 및 mCherry에 대한 100 ms 노출을 갖는 10% 전력에서 580 nm LED). Zeiss 호환 Zen3.2 소프트웨어는 세포 이미징에 사용되었습니다.
- 미세유체 플레이트의 모든 웰에서 보존 용액(존재하는 경우)을 제거하고 새로운 배양 배지로 교체합니다.
- 이미지 분석
참고: 현미경 이미지의 열기 및 시각화는 오픈 소스 ImageJ/Fiji 소프트웨어(https://fiji.sc/)26을 사용하여 수행됩니다. 정량적 이미지 분석은 오픈 소스 ImageJ/Fiji 소프트웨어와 무료 MicrobeJ 플러그인(https://microbej.com)27을 사용하여 수행됩니다. 이 프로토콜에서는 MicrobeJ 5.13I(14) 버전이 사용되었습니다.- 컴퓨터에서 ImageJ/Fiji 소프트웨어를 열고 하이퍼스택 타임랩스 현미경 이미지를 Fiji 로딩 바에 드래그합니다. Image > Color > Make Composite 를 사용하여 하이퍼스택의 여러 채널을 융합합니다. 타임랩스 실험의 채널이 원하는 색상과 일치하지 않는 경우(예: 위상차가 회색 대신 빨간색으로 표시되는 경우) 이미지 > 색상 > 채널 정렬 을 사용하여 채널에 적절한 색상을 적용합니다.
- MicrobeJ 플러그인을 열고 수동 편집 인터페이스를 사용하여 박테리아 세포를 감지합니다. 자동으로 감지된 셀을 삭제하고 관심 있는 지속형 셀을 프레임별로 수동으로 간략하게 설명합니다.
알림: 개별 셀을 자동으로 감지하기 위해 다른 설정을 사용할 수 있습니다. 분석된 지속세포가 긴 필라멘트를 형성하기 때문에 수동 검출이 사용되었으며, 자동 검출을 사용하여 올바르게 검출되는 경우는 거의 없습니다. - 감지 후 MicrobeJ 수동 편집 인터페이스의 결과 아이콘을 사용하여 ResultJ 테이블을 생성합니다. ResultJ 파일을 저장하고 ResultJ 테이블을 사용하여 단일 세포 분석에서 관심 있는 다양한 파라미터에 대한 통찰력을 얻을 수 있습니다. 이 프로토콜의 경우, HU-mCherry 강도의 평균 형광, 세포 길이 및 개별 세포의 세포 면적을 내보냈습니다.
결과
상술한 바와 같이, 단일세포 표현형 분석에 사용된 균주는 지속성 세포의 MOPS 글리세롤 0.4% 배지에서 특성화되었다. 시간 경과에 따른 OD600nm 의 모니터링은 wt 및 hupA-mCherry 균주 간에 차이가 없음을 보여주었습니다(그림 1). 이는 HU-mCherry 융합 단백질의 발현이 이러한 조건에서 성장에 영향을 미치지 않았음을 나타냅니다. 0.01의OD600 nm 에서 초기에 접종된 두 균주의 박테리아 세포는 접종 후 ±8시간 후에 지수기에 도달했습니다.
OFX의 MIC는 표준화된 방법(여기서는 직렬 한천 희석)에 의해 결정되었습니다(24). MIC는 눈에 보이는 성장이 감지되지 않는 최소 농도로 정의됩니다. 두 균주에 대한 OFX의 MIC는 0.06 μg·mL-1로 결정되었으며, 이는 hupA-mCherry 융합이 동종 wt 균주와 비교하여 OFX에 대한 민감도에 영향을 미치지 않았음을 나타냅니다(그림 2).
우리는 또한 이 연구에 사용된 두 균주의 생존력에 대한 치명적인 OFX 처리(83배 MIC)의 효과를 결정했습니다. OFX 노출로 생존 가능한 세포 수가 시간이 지남에 따라 감소함에 따라 박테리아 배양의 희석액은 플레이트당 30-300개의 콜로니에 도달하도록 적절하게 조정되어야 합니다. 시간 경과에 따른 적절한 희석액을 결정하기 위해, 스팟 분석을 수행했으며, 여기서 0 내지 10-7 개의 연속 10 배 희석액 10 μL를 다중 채널 피펫을 사용하여 정사각형 페트리 접시에 배치했습니다. 적절한 희석은 분리된 클론이 보이는 경우(예: t0 = 10-5, t1h = 10-4/10-3, t4h = 10-2/10-1)였습니다(그림 3).
스팟 분석은 OFX 매개 사멸의 동역학에 대한 통찰력을 얻는 쉬운 방법이지만 사멸 역학을 정확하게 결정하지는 못합니다. OFX로 처리된 기하급수적으로 성장하는 세포의 생존율을 time-kill 분석에 의해 모니터링했을 때, 전형적인 이상성 곡선이 관찰되었다(도 4). 곡선의 첫 번째 기울기는 비지속적 개체군(빨간색 점선)의 급속한 사멸을 반영합니다. 여기에서 테스트된 조건에서, 세포의 최대 99.9%는 OFX의 존재 하에서 3시간 후에 콜로니를 형성할 수 없었다. 이 첫 번째 살상 단계 다음에는 두 번째 단계가 이어지며, 이는 더 느린 사멸 속도(파란색 점선)를 보여 약물 내성 지속 세포의 존재를 나타냅니다. 테스트된 조건에서 지속기 단계는 OFX 첨가 후 약 3시간 후에 시작되어 지속적 표현형을 조사하기 위해 세포를 3시간 이상 OFX에 노출시켜야 할 필요성을 강조했습니다. 중요하게도, 타임-킬 곡선은 hupA-mCherry 융합 단백질이 타임-킬 동역학에 영향을 미치지 않는다는 것을 보여줍니다. 따라서 번역 형광 융합을 코딩하는 균주는 형광 현미경을 사용하여 지속성 세포를 모니터링하는 데 사용할 수 있습니다.
우리는 단일 세포 수준에서 지속성 현상을 조사하기 위해 계속 진행했습니다. 이를 위해 hupA-mCherry 균주를 미세유체 플레이트에 도입하여 주어진 ROI에 대해 타임랩스 현미경을 수행하는 동안 배지 조건(여기서는 성장, 처리 및 회복)의 변화를 허용했습니다. 미세유체 실험의 첫 번째 단계에서 미세유체 장치에 도입된 세포를 성장 배지(MOPS 글리세롤 0.4%)로 관류하고 ~2h의 생성 시간으로 분할했습니다(그림 5 및 그림 6). 이 첫 번째 성장 단계는 세포가 OFX 치료 전에 생존 가능하고 활발하게 분열했음을 나타냅니다.
이 첫 번째 성장 단계 후, 세포는 6시간 동안 5μg·mL-1 OFX로 보충된 성장 배지로 관류되었습니다. 항생제가 세포에 도달하자마자 세포 분열이 차단되었습니다(그림 5 및 그림 6). 6시간의 OFX 처리 후, 세포를 새로운 배지로 관류시켰다. 대다수의 세포가 성장을 재개할 수 없었지만(그림 5 및 그림 6), 소수의 박테리아 소집단은 필라멘트 세포를 늘리고 생성할 수 있었다25. OFX 처리 후 생존 가능한 딸 세포를 분열하고 생성할 수 있었던 이러한 세포는 지속성 세포로 정의할 수 있습니다.
이 설정을 통해 처리 전, 처리 중 및 처리 후 지속생존세포 시각화가 가능하므로 회복 단계 중 지속생존기 표현형에 대한 정보뿐만 아니라 처리 전 지속생존세포 세포의 생리학적 상태에 대한 정보도 제공합니다(그림 6). 테스트된 조건에서 지속세포 세포는 OFX 처리 전에 비지속세포와 유사하게 분열되었으며, 이는 관찰된 지속세포가 휴면 하위 집단에서 유래하지 않았음을 나타냅니다(그림 6)25.
회복 단계 동안 지속자 세포의 세포 길이 분석은 각 필라멘트가 특정 연신율을 갖는 것으로 나타났습니다. 첫 번째 분열 전에 각 지속자가 도달한 세포 길이는 지속자마다 달랐습니다. 마찬가지로, 첫 번째 디비전 이벤트의 타이밍은 매우 이질적이었습니다(그림 6). 분열하는 지속적 필라멘트는 여러 개의 딸 세포를 생성했으며, 이 딸 세포는 대부분 처리되지 않은 세포와 유사하게 성장하고 분열하기 시작했습니다(그림 7). 필라멘트의 연속적인 분열은 세포 길이의 점진적인 감소를 초래하여 궁극적으로 OFX 처리 전과 유사한 세포 길이를 가진 딸 세포를 생성했습니다(그림 6 및 그림 7B). 대다수의 세포는 OFX 제거 후 필라멘트화를 유도할 수 없었습니다. 이 큰 세포 집단은 죽은 세포에 해당합니다(그림 5 및 그림 6).
뉴클레오이드 관련 단백질 HU의 형광 융합은 뉴클레오이드22의 역학을 시각화할 수 있습니다. 세포 내 HU-mCherry의 총 형광 강도 분석은 DNA 함량22,25에 대한 프록시로 사용할 수 있습니다. 성장 단계(OFX 처리 전) 동안 총 mCherry 형광 강도는 세포 주기 동안 염색체 복제 및 분리의 역학을 반영하여 다양했습니다(그림 8). OFX 첨가 후, mCherry 형광은 중간 세포에서 증가하여 핵체 압축을 나타내며, 이는 이중 가닥 DNA 파괴28의 형성에 의해 유도되는 것으로 나타났습니다(그림 5). 이중 가닥 DNA 절단은 유형 II 토포이소머라제 DNA-자이라제 및 토포이소머라제 IV29,30을 손상시키는 OFX의 작용 메커니즘의 결과입니다. 대장균에서 DNA-자이라제는 OFX29,30의 주요 표적입니다. 이중 가닥 통과 메커니즘의 중요한 단계에서 표적을 결합함으로써 OFX는 절단된 DNA 가닥의 강등을 억제하여 궁극적으로 이중 가닥 DNA 절단의 방출로 이어집니다30. 전술한 바와 같이, OFX 처리에 대한 지속세포는 회복 동안 필라멘트를 시작하였다25(도 6). 세포 길이의 증가는 총 mCherry 형광 강도의 증가와 상관관계가 있으며, 이는 복제 재시작 및 필라멘트25의 뉴클레오이드 존재비의 증가를 반영합니다(그림 7a 및 그림 8). 죽은 세포의 경우, 총 mCherry 형광 강도는 치료 중 및 회복 단계 동안 안정적으로 유지되었으며, 이는 이러한 세포가 OFX 제거 후 염색체를 복제할 수 없음을 나타냅니다(그림 8). 오플록사신 처리 전, 도중 및 후의 대장균 HU-mCherry 세포의 미세유체 비디오(비디오 1)도 표시됩니다.
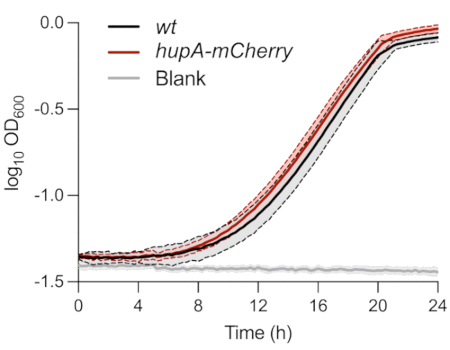
그림 1: wt 및 hupA-mCherry E. coli 균주의 성장 모니터링. wt(검정색) 및 hupA-mCherry(빨간색)의 광학 밀도 모니터링(OD600nm). 음영과 점선은 생물학적 삼중체의 표준 편차를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
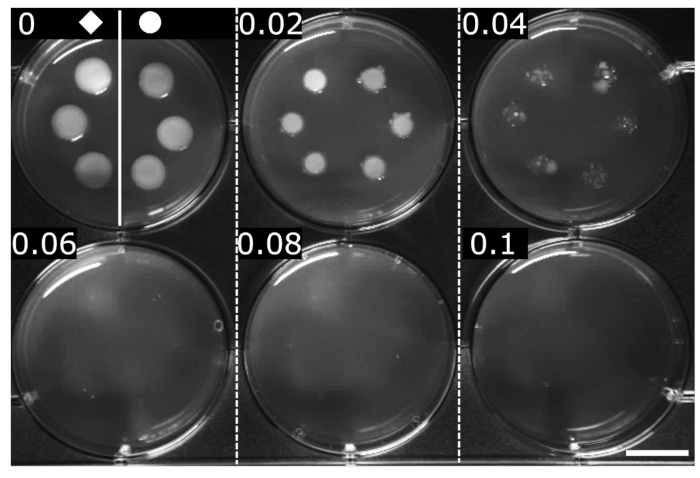
그림 2: wt 및 hupA-mCherry E. coli 균주에 대한 OFX의 MIC 측정. wt (♦) 및 hupA-mCherry (●)는 LB 배지에서 성장시켰고, 2μL는 OFX 함유 LB 한천(각 패널에 μg·mL-1로 표시된 농도)의 연속 희석액에서 발견되었습니다. 성장 억제는 최소 0.06 μg·mL-1에서 볼 수 있습니다. 이 그림은 생물학적 삼중의 대표적인 실험입니다. 스케일 바 = 1cm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
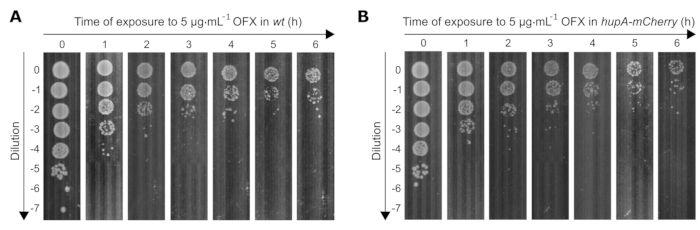
그림 3: OFX 에 노출된 wt 및 hupA-mCherry E. coli 균주의 스팟 분석. (A) wt 및 (B) hupA-mCherry 균주는 프로토콜(섹션 3)에 설명된 대로 MOPS 글리세롤 0.4%에서 성장시켰고, 지수적으로 성장하는 세포(OD600nm = 0.3)는 5μg·mL-1 OFX로 처리되었습니다. T0는 OFX가 추가되기 전의 시점에 해당합니다. T1, T2, T3, T4, T5 및 T6은 OFX 추가 후 1-6시간에 해당합니다. 이 그림은 생물학적 삼중의 대표적인 실험입니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
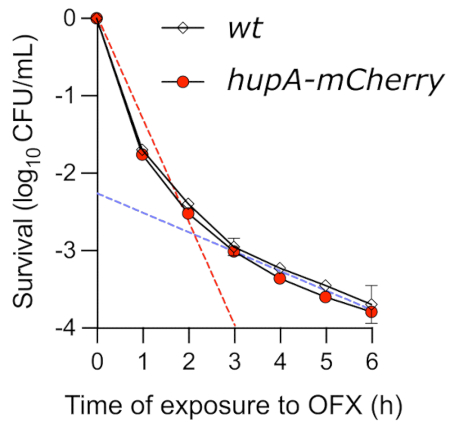
그림 4: OFX 에 노출된 wt 및 hupA-mCherry E. coli 균주의 시간 사멸 분석. wt (♦) 및 hupA-mCherry (●) 균주는 프로토콜(섹션 4)에 설명된 대로 MOPS 글리세롤 0.4%에서 성장시켰고, 지수적으로 성장하는 세포(OD600nm = 0.3)를 5μg·mL-1 OFX로 처리했습니다. 점선은 첫 번째 "빠른"(빨간색) 사멸 단계와 두 번째 "느린"(파란색) 사멸 단계를 나타내며, 민감하고 지속적인 하위 모집단에 해당합니다(각각 T0와 T2 사이, T3과 T6 사이의 선형 회귀에 의해 얻어짐). 오차 막대는 생물학적 삼중체의 표준 편차를 나타냅니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
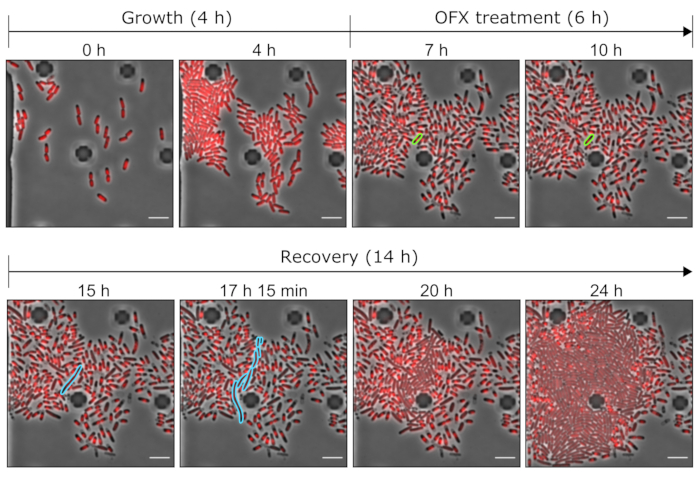
그림 5: 미세유체 도구를 사용한 OFX persister 및 dead 세포의 대표 이미지. hupA-mCherry 균주로 수행된 미세유체 실험의 관련 시점을 보여주는 대표적인 현미경 이미지(회색의 위상차, 빨간색의 HU-mCherry 신호). 태그된 hupA-mCherry 를 발현하는 세포는 미세유체 플레이트(여기서는 4시간)에서 성장한 후 OFX 챌린지(5μg·mL-1)를 수행했습니다. OFX의 존재 하에 6시간 후, 세포를 새로운 배지로 관류하여 지속기 세포가 회복되도록 했습니다. OFX 처리 중 및 OFX 제거 후의 지속세포 및 그 자손 세포는 각각 녹색 및 파란색으로 강조 표시됩니다. 해당 시점이 각 패널에 표시됩니다. 스케일 바 = 5 μm. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
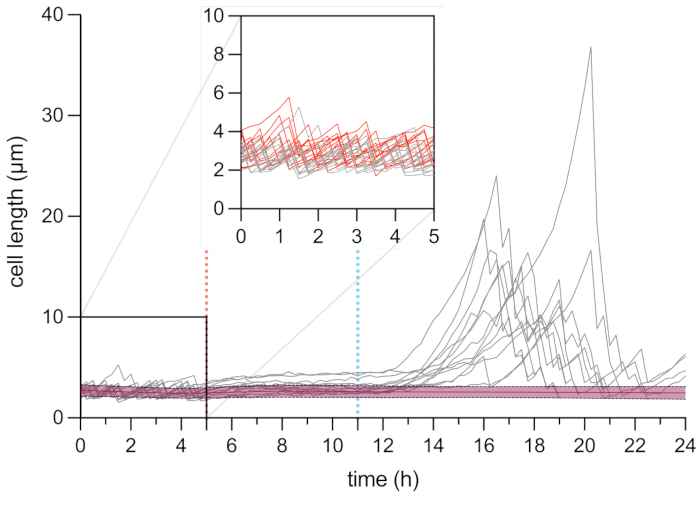
그림 6: 지속세포와 죽은 세포의 길이에 대한 현미경 타임랩스 분석. 죽은 세포(빨간색, n = 109) 및 지속 세포(회색, n = 13)의 세포 길이 분석. OFX 치료의 시작(5μg·mL-1)은 빨간색 점선(5시간)으로 표시되고 OFX 제거는 파란색 점선으로 표시됩니다. 삽입물은 OFX 첨가 전의 성장 단계에 해당합니다. 실험은 삼중으로 수행되었다. 음영과 점선은 죽은 세포 집단에 대한 표준 편차를 나타냅니다(n = 109). 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
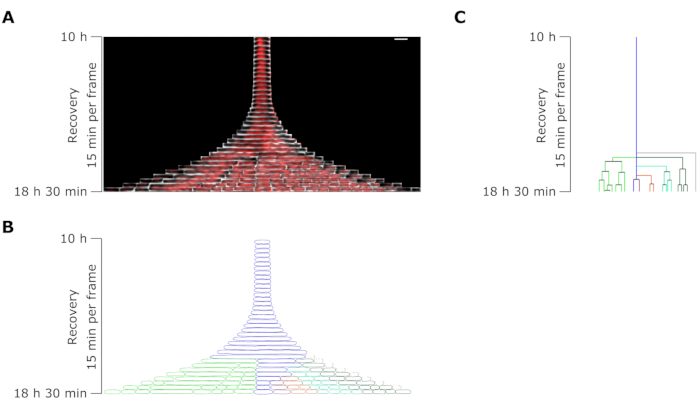
그림 7: OFX에 대한 대표적인 지속자의 현미경 타임랩스 분석 . (A) OFX 제거 후 8.5시간 동안 필라멘트 분열에 의해 생성된 대표적인 OFX 지속기 및 그 딸 세포의 키모그래프(미세유체 실험 시작 후 18.5시간, 4시간의 성장, 6시간의 5μg·mL-1 OFX 처리 및 8.5시간의 OFX 제거 후 회복). 한 프레임은 15분에 해당합니다. 스케일 바 = 5 μm. (B) A의 퍼시서 키모그래프에서 생성된 마스크. 모니터링되는 지속자 셀은 파란색 윤곽선으로 표시되고 딸 셀은 고유한 색상으로 강조 표시됩니다. (C) B로부터 생성된 지속세포 계통의 개략도. 색상 코딩은 B와 동일합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
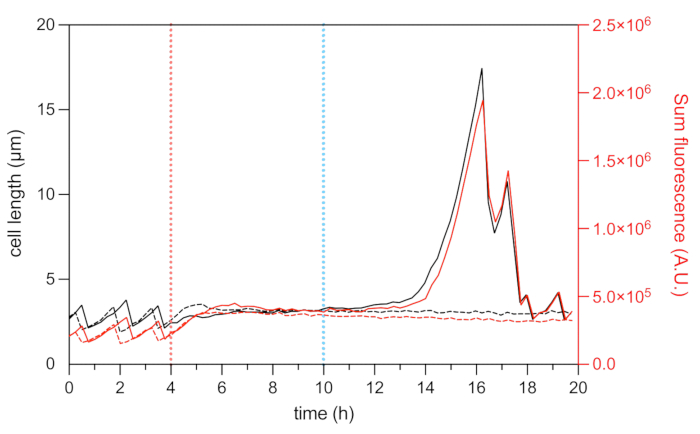
그림 8: 대표적인 지속세포(persister cell)와 사멸(dead cell)의 세포 길이 및 mCherry 형광 분석. 미세유체 타임랩스 실험 동안 대표적인 지속세포(검은색 및 빨간색 실선)와 대표 데드 셀(검은색 및 빨간색 점선)의 세포 길이(왼쪽 축) 및 총 HU-mCherry 형광 강도(오른쪽 축, 임의의 단위로 표시)를 분석했습니다. OFX 치료(5μg·mL-1)의 시작은 빨간색 점선으로 표시되고 OFX 제거는 파란색 점선으로 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
비디오 1: 오플록사신 치료 전, 도중 및 후의 대장균 HU-mCherry 세포의 미세유체 비디오. HU-mCherry 세포를 보여주는 미세유체 타임랩스 이미징. 세포를 0.4%의 글리세롤 중에서 4시간 동안 MOPS 성장시켰다. 6시간의 OFX 처리(5μg·mL-1) 후, 항생제가 없는 배지를 미세유체 플레이트에 관류하여 지속 세포가 회복될 수 있도록 했습니다. 스케일 바 = 5 μm. 시간(분)이 표시됩니다. 성장 및 회복 단계는 "MOPS-Gly. 0.4%" 및 "OFX 5μg/mL"에 의한 OFX 처리. 이 비디오를 다운로드하려면 여기를 클릭하십시오.
| 걸레 10개 | |||
| 원액 | 10x MOPS 1L에 대한 스톡 용액의 부피 | 10x MOPS 베이스의 최종 농축 | |
| 걸레산 | 1 M (KOH를 사용하여 pH 7.4로 조정) | 400 밀리리터 | 0.4 미터 |
| 트리신 | 1 M (KOH를 사용하여 pH 7.4로 조정) | 40 밀리리터 | 0.04 미터 |
| FeSO4.7H2O | 0.01 미터 | 10 mL | 0.0001 미터 |
| NH4Cl | 1.9 미터 | 50 밀리리터 | 0.095 미터 |
| K2그래서4 | 0.276 미터 | 10 mL | 0.00276 미터 |
| CaCl2.2H 2O | 0.0005 미터 | 10 mL | 0.000005 미터 |
| 마그네슘 2.6H2O | 0.528 미터 | 10 mL | 0.00528 미터 |
| 염화나트륨(NaCl) | 29.2 g을 직접 첨가하십시오 | 0.5 미터 | |
| 증류수 | 460 mL의 | ||
| 미량 영양소 1000x (표 2 참조) | 10 mL | ||
표 1: 10x MOPS의 구성.
| 미량 영양소 1000배 | ||
| 미량 영양소 농도 1000x 원액 | 10x MOPS 베이스의 최종 농축 | |
| (NH4)6Mo7O24.4H2O | 0.000003 미터 | 0.00000003 미터 |
| H3 보3 | 0.0004 미터 | 0.000004 미터 |
| CoCl2.6H 2O | 0.00003 미터 | 0.0000003 미터 |
| CuSO4.5H 2O | 0.00001 미터 | 0.0000001 미터 |
| MnCl 2.4H2O | 0.00008 미터 | 0.0000008 미터 |
| ZnSO.7H2O | 0.00001 미터 | 0.0000001엠 |
표 2: 1,000x 미량 영양소의 구성.
| MOPS 포도당 0.4 % 또는 MOPS 글리세롤 0.4 % | |||
| 원액 | 1 L MOPS 포도당 0.4 % 또는 MOPS 글리세롤 0.4 %의 부피 | MOPS 포도당 0.4 % 또는 MOPS 글리세 0.4 %의 최종 농도 | |
| 걸레 10개 | 표 1 참조 | 100 밀리리터 | |
| K2HPO4 | 0.132 미터 | 10 mL | 0.00132 미터 |
| 포도당 (MOPS 포도당 0.4 %) | 20%(증류수 100mL에 20g) | 20 밀리리터 | 0.40% |
| 글리세롤(MOPS 글리세롤용 0.4%) | ≤99% | 4 밀리리터 | 0.40% |
| 증류수 | MOPS 포도당 0.4%의 경우 870mL 또는 MOPS 글리세롤 0.4%의 경우 886mL | ||
표 3: MOPS 포도당 0.4% 및 MOPS 글리세롤 0.4%의 조성.
토론
이 논문에 제시된 프로토콜은 집단 및 단일 세포 수준에서 항생제 치료에 대한 반응으로 관찰된 지속성 표현형을 분석할 수 있습니다. 실험은 화학적으로 정의된 배지(MOPS 글리세롤 0.4%)에서 성장한 대장균 MG1655 균주로 수행되었습니다. Time-kill assay 및 현미경 실험은 지수 위상 배양에서 수행되었습니다. 우리는 5μg·mL-1 농도의 플루오로퀴놀론인 OFX를 사용하여 지속 세포를 밝혔습니다. 여기에 기술된 접근법은 다른 살균성 항생제, 예컨대 β-락탐, 아미노글리코시드, 또는 항미생물 화합물(31)에 적용될 수 있다. 따라서, 다른 박테리아 균주, 배지 또는 성장 조건이 사용될 수 있다. 여기에 기술된 것과 유사한 설정에서 상이한 형광 융합체를 모니터링하는 것은 항생제 치료 전, 도중 및 후에 DNA 복제(32), DNA 복구(25, 33) 및 세포 분열(34)과 같은 세포 과정을 따르는 데 유용할 수 있다. 유사하게, 형광 리포터는 세포 내 pH35, ATP 36 또는 ROS37 수준과 같은 세포 생리학의 뚜렷한 측면을 조사하기 위해 이용될 수 있습니다. 형광 융합 대신 화학 염료도 사용할 수 있습니다. 예를 들어, hupA-mCherry 융합은 DNA38을 염색하는 형광 염료인 4',6-diamidino-2-phenylindole(DAPI)로 대체될 수 있습니다. 그러나 이러한 형광 염료와 결합된 타임랩스 현미경을 수행하는 것은 이러한 염색 기술이 타임랩스 실험 중에 세포 주기의 역학을 교란시킬 수 있으므로 피해야 합니다. 대안적으로, 이러한 실험은 관련 시점들에서 스냅-샷 이미징의 시간-경과 분석에 의해 대체될 수 있다.
이러한 형광 리포터가 도움이 되기는 하지만, 위상차 이미지 분석을 통해 추출할 수 있는 정보의 양을 무시해서는 안 됩니다. 여기에서 우리는 성장, OFX 치료 및 회복 단계 전반에 걸쳐 세포 길이 진화를 모니터링했습니다. 박테리아 세포의 세포 폭, 위상차 강도 및 곡률과 같은 위상차 이미지를 기반으로 하는 다른 매개변수도 MicrobeJ27과 같은 적절한 소프트웨어를 사용하여 쉽게 추출할 수 있습니다.
요약하면, 여기에 기술된 절차는 변화하는 환경 또는 스트레스 요인에 대한 세포 반응을 모니터링하기 위해 다른 조건 및 박테리아 종에 적용될 수 있다18, 19. 다른 형광 리포터(전사 및 번역 리포터, 화학 염료)를 유세포 분석/FACS와 같은 모집단 분석과 함께 사용하면 다중 스케일 프레임워크에서 흥미로운 질문을 해결할 수 있습니다.
공개
저자는 경쟁 이익을 선언하지 않습니다.
감사의 말
Van Melderen 연구실에서의 작업은 ARC 조치 2018-2023, Fonds National de la Recherche Scientifique(FNRS CDR J.0182.21F)의 지원을 받습니다. TO는 ULB 펠로우십의 지원을 받습니다. TS는 FRIA 펠로우십(FNRS)의 지원을 받습니다. JC는 박사후 연구원 "chargé de recherches"(FNRS)의 지원을 받습니다.
자료
| Name | Company | Catalog Number | Comments |
| Axio Observer | Zeiss | Inverted fluorescence microscope | |
| CaCl2.2H2O | Merck | 1.02382.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CellASIC ONIX Microfluidic System | Merck | CAX2-S0000 | Microfluidic system |
| CellASIC ONIX2 FG | Merck | ONIX2 1.0.1 | Microfluidic software |
| CellASIC ONIX2 Manifold Basic | Merck | CAX2-MBC20 | Manifold system |
| CoCl2.6H2O | Merck | 1.02539.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CuSO4.5H2O | Merck | 1.02790.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| D-(+)-glucose | Sigma Aldrich | G7021-1KG | Carbon source for MOPS glucose 0.4% growth medium |
| E. coli K-12 MG1655 CF1648 (fnr+) | BE10 | wt reference strain, lab strain | |
| E. coli K-12 MG1655 CF1648 (fnr+) hupA-mCherry::FRT-kan-FRT | BE16 | HU-mCherry fusion integrated via P1 transduction at the native locus, lab strain | |
| FeSO4.7H2O | VWR Chemicals | 24244.232 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Fiji | ImageJ | https://fiji.sc/ | Image software; Schindelin et al. if used in publication |
| Glycerol | Merck | 56815 | Carbon source for MOPS glycerol 0.4% growth medium |
| Greiner CELLSTAR® multiwell culture plate | Merck | M8812-100EA | 24-well clear flat bottom 2ml volume culture plate for automated plate reader |
| H3BO3 | Sigma-Aldrich | B6768-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Hamamatsu ORCA Flash 4.0 digital camera | Hamamatsu | C13440-20CU | Digital Image Acqusition |
| K2HPO4 | Merck | 1.05099.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| KOH | Merck | 1.05029.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| K2SO4 | Merck | 1.05153.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Luria-Broth agar medium | Invitrogen | 22700041 | Growth medium for plating assay |
| Luria-Broth medium | Invitrogen | 12780029 | Growth medium for MIC determination |
| MgCl2.6H2O | Merck | 1.05832.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MgSO4 | Merck | 7487-88-9 | Dilution solution for bacterial survival assay used at 10mM |
| MicrobeJ | Imagej/Fiji plugin | https://www.microbej.com/ | Microscopy image analysis plugin. Ducret et al.for in publication; Detection settings: For bacteria : Area (μm2): 0,1-max; Length (μm): 0,5-max; Width (μm): 0,6-max; Range (μm): 0,5-max; Angularity (rad): 0-0,3; 0-max for all other parameters. |
| Microfluidic Plates CellASIC ONIX | Merck | B04A-03-5PK | Plate for microfluidic system |
| MnCl2.4H2O | Merck | 1.05927.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MOPS, Free Acid ULTROL® Grade | Merck | 475898-500GM | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NaCl | SIgma-Aldrich | S5886-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| (NH4)6Mo7O24.4H2O | Merck | 1.01180.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NH4Cl | Merck | 1.01145.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Ofloxacin | Merck | 82419-36-1 | Fluoroquinolone antibiotic used to treat the bacterial cells |
| Phosphate Buffered Saline (PBS) pH 7.4, 1x | Merck | P3813 | Dilution buffer |
| GENESYS 10S UV-Visible Spectrophotometer | Thermofisher Scientific | 840-208200 | UV-Visible Spectrophotometer, Single/Six Cell Holder with PrinterMeasurement of optical density OD600nm |
| SoftMax Pro | Molecular Devices | Microplate reader software | |
| SpectraMax i3x | Molecular Devices | Microplate reader | |
| N-[Tris(hydroxyméthyl)-méthyl]-glycine | Merck | 1.08602.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Zeiss® immersion Oil 518F | Zeiss | Immersion oil to increase resolution of microscope | |
| Zen3.2 Pro | Zeiss | Microscopic image acquisition and processing software | |
| ZnSO4.7H2O | Merck | 1.08883.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
참고문헌
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nature Reviews Microbiology. 17 (7), 441-448 (2019).
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nature Reviews Microbiology. 14 (5), 320-330 (2016).
- Pribis, J. P., Zhai, Y., Hastings, P. J., Rosenberg, S. M. Stress-induced mutagenesis, gambler cells, and stealth targeting antibiotic-induced evolution. mBio. 13 (3), 01074 (2022).
- Molina-Quiroz, R. C., Lazinski, D. W., Camilli, A., Levy, S. B. Transposon-sequencing analysis unveils novel genes involved in the generation of persister cells in uropathogenic Escherichia coli. Antimicrobial Agents and Chemotherapy. 60 (11), 6907-6910 (2016).
- Cameron, D. R., Shan, Y., Zalis, E. A., Isabella, V., Lewis, K. A genetic determinant of persister cell formation in bacterial pathogens. Journal of Bacteriology. 200 (17), 00303-00318 (2018).
- Cui, P., et al. Identification of genes involved in bacteriostatic antibiotic-induced persister formation. Frontiers in Microbiology. 9, 413 (2018).
- Li, T., et al. Identification of novel genes involved in Escherichia coli persistence to tosufloxacin. Frontiers in Cellular and Infection Microbiology. 10, 581986 (2020).
- Verstraeten, N., et al. Obg and membrane depolarization are part of a microbial bet-hedging strategy that leads to antibiotic tolerance. Molecular Cell. 59 (1), 9-21 (2015).
- Hofsteenge, N., van Nimwegen, E., Silander, O. K. Quantitative analysis of persister fractions suggests different mechanisms of formation among environmental isolates of E. coli. BMC Microbiology. 13 (1), 25 (2013).
- Huemer, M., Mairpady Shambat, S., Brugger, S. D., Zinkernagel, A. S. Antibiotic resistance and persistence-Implications for human health and treatment perspectives. EMBO Reports. 21 (12), 51034 (2020).
- Wilmaerts, D., Windels, E. M., Verstraeten, N., Michiels, J. General mechanisms leading to persister formation and awakening. Trends in Genetics. 35 (6), 401-411 (2019).
- Murawski, A. M., Brynildsen, M. P. Ploidy is an important determinant of fluoroquinolone persister survival. Current Biology. 31 (10), 2039-2050 (2021).
- Lemma, A. S., Soto-Echevarria, N., Brynildsen, M. P. Fluoroquinolone persistence in Escherichia coli requires DNA repair despite differing between starving populations. Microorganisms. 10 (2), 286 (2022).
- Balaban, N. Q., Merrin, J., Chait, R., Kowalik, L., Leibler, S. Bacterial persistence as a phenotypic switch. Science. 305 (5690), 1622-1625 (2004).
- Morawska, L. P., Kuipers, O. P. Antibiotic tolerance in environmentally stressed Bacillus subtilis: Physical barriers and induction of a viable but nonculturable state. microLife. 3, (2022).
- Windels, E. M., et al. Enrichment of persisters enabled by a ß-lactam-induced filamentation method reveals their stochastic single-cell awakening. Communications Biology. 2 (1), 426 (2019).
- Leygeber, M., et al. Analyzing microbial population heterogeneity-Expanding the toolbox of microfluidic single-cell cultivations. Journal of Molecular Biology. 431 (23), 4569-4588 (2019).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli growth rate to osmotic shock. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7807-7812 (2014).
- Shi, H., et al. Starvation induces shrinkage of the bacterial cytoplasm. Proceedings of the National Academy of Sciences of the United States of America. 118 (24), 2104686118 (2021).
- Goode, O., et al. Persister Escherichia coli cells have a lower intracellular pH than susceptible cells but maintain their pH in response to antibiotic treatment. mBio. 12 (4), 00909-00921 (2021).
- Manuse, S., et al. Bacterial persisters are a stochastically formed subpopulation of low-energy cells. PLoS Biology. 19 (4), 3001194 (2021).
- Fisher, J. K., et al. Four-dimensional imaging of E. coli nucleoid organization and dynamics in living cells. Cell. 153 (4), 882-895 (2013).
- Cabral, D., Wurster, J., Belenky, P. Antibiotic persistence as a metabolic adaptation: Stress, metabolism, the host, and new directions. Pharmaceuticals. 11 (1), 14 (2018).
- Wiegand, I., Hilpert, K., Hancock, R. E. W. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nature Protocols. 3 (2), 163-175 (2008).
- Goormaghtigh, F., Van Melderen, L. Single-cell imaging and characterization of Escherichia coli persister cells to ofloxacin in exponential cultures. Science Advances. 5 (6), (2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ducret, A., Quardokus, E. M., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nature Microbiology. 1 (7), 16077 (2016).
- Odsbu, I., Skarstad, K. DNA compaction in the early part of the SOS response is dependent on RecN and RecA. Microbiology. 160 (5), 872-882 (2014).
- Pham, T. D. M., Ziora, Z. M., Blaskovich, M. A. T. Quinolone antibiotics. MedChemComm. 10 (10), 1719-1739 (2019).
- Drlica, K., et al. Quinolones: Action and resistance updated. Current Topics in Medicinal Chemistry. 9 (11), 981-998 (2009).
- Kohanski, M. A., Dwyer, D. J., Collins, J. J. How antibiotics kill bacteria: From targets to networks. Nature Reviews Microbiology. 8 (6), 423-435 (2010).
- Reyes-Lamothe, R., Sherratt, D. J., Leake, M. C. Stoichiometry and architecture of active DNA replication machinery in Escherichia coli. Science. 328 (5977), 498-501 (2010).
- Lesterlin, C., Ball, G., Schermelleh, L., Sherratt, D. J. RecA bundles mediate homology pairing between distant sisters during DNA break repair. Nature. 506 (7487), 249-253 (2014).
- Alexeeva, S., Gadella, T. W. J., Verheul, J., Verhoeven, G. S., Den Blaauwen, T. Direct interactions of early and late assembling division proteins in Escherichia coli cells resolved by FRET: Bacterial division studied by spectral FRET. Molecular Microbiology. 77 (2), 384-398 (2010).
- Miesenböck, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Yaginuma, H., et al. Diversity in ATP concentrations in a single bacterial cell population revealed by quantitative single-cell imaging. Scientific Reports. 4, 6522 (2014).
- Ermakova, Y. G., et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide. Nature Communications. 5, 5222 (2014).
- Kapuscinski, J. DAPI: A DNA-specific fluorescent probe. Biotechnic & Histochemistry. 70 (5), 220-233 (1995).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유