Method Article
Populations- und Einzelzellanalyse der Antibiotikapersistenz in Escherichia coli
In diesem Artikel
Zusammenfassung
Die Antibiotikapersistenz beschreibt die Fähigkeit kleiner Subpopulationen innerhalb einer empfindlichen isogenen Population, hohe Dosen bakterizider Antibiotika vorübergehend zu tolerieren. Das vorliegende Protokoll kombiniert Ansätze zur Charakterisierung des Antibiotika-Persistenz-Phänotyps auf molekularer und zellulärer Ebene, nachdem Escherichia coli tödlichen Dosen von Ofloxacin ausgesetzt wurde.
Zusammenfassung
Die Persistenz von Antibiotika bezieht sich auf die Fähigkeit kleiner bakterieller Subpopulationen, hohe Dosen bakterizider Antibiotika vorübergehend zu tolerieren. Nach einer bakteriziden Antibiotikabehandlung wird der Großteil der Bakterienpopulation schnell abgetötet. Auf diese erste schnelle Phase der Abtötung folgt eine deutliche Abnahme der Abtötungsrate, da die Persisterzellen lebensfähig bleiben. Klassischerweise wird die Persistenz auf Populationsebene durch Time/Kill-Assays bestimmt, die mit hohen Antibiotikadosen und für definierte Expositionszeiten durchgeführt werden. Diese Methode liefert zwar Informationen über den Gehalt an Persisterzellen und die Abtötungskinetik, spiegelt aber nicht die intrinsische Zell-zu-Zell-Heterogenität wider, die dem Persistenzphänomen zugrunde liegt. Das hier beschriebene Protokoll kombiniert klassische Time/Kill-Assays mit der Einzelzellanalyse mittels Echtzeit-Fluoreszenzmikroskopie. Durch die Verwendung geeigneter Fluoreszenzreporter kann die mikroskopische Bildgebung lebender Zellen Aufschluss über die Auswirkungen des Antibiotikums auf zelluläre Prozesse wie Chromosomenreplikation und -segregation, Zellverlängerung und Zellteilung geben. Die Kombination von Populations- und Einzelzellanalyse ermöglicht die molekulare und zelluläre Charakterisierung des Persistenzphänotyps.
Einleitung
Dieses Protokoll zielt darauf ab, den bakteriellen Persistenzphänotyp als Reaktion auf eine spezifische Antibiotikabehandlung auf Einzelzell- und Populationsebene zu analysieren. Persistenz beschreibt die Fähigkeit kleiner Subpopulationen innerhalb einer isogenen Population, hohe Dosen bakterizider Antibiotika (Fluorchinolone, Aminoglykoside, β-Lactame usw.) zu ertragen, wobei die minimale Hemmkonzentration (MHK) der sogenannten Persisterzellen identisch mit der des Großteils der Bevölkerung ist. Die biphasische Abtötungsdynamik zeigt bei der Messung des bakteriellen Überlebens im Laufe der Zeit in Gegenwart eines Antibiotikums das Vorhandensein vorübergehender medikamententoleranter Zellen, mit einer anfänglichen schnellen Eradikation der Nicht-Persister-Zellen, gefolgt von einer viel langsameren Abtötungsrate der Persisterzellen. Nach Entfernung des Antibiotikums führen diese Zellen zu einer genetisch identischen Population, die eine ähnliche Abtötungsdynamik aufweist, wenn sie mit demselben Antibiotikum behandeltwird 1,2. Im Gegensatz zur Persistenz wird die Antibiotikaresistenz auf Populationsebene definiert und ist in der Regel entweder eine Folge von de novo Mutationen oder dem horizontalen Gentransfer eines resistenzverleihenden Plasmids3. Während die Mutationen, die für die Resistenz verantwortlich sind, meist im Zielmolekül des Wirkstoffs oder in den Promotorregionen der Wirkstoff-Effluxpumpen lokalisiert sind, haben sich Gene, die die Persistenzhäufigkeit verändern, die durch genomweite und gezielte Mutantenanalyseansätze identifiziert wurden, als zahlreich und vielfältig erwiesen 2,3,4,5,6,7,8 . Daher ist es wahrscheinlich, dass Bakterienzellen über mehrere Wege in den Persisterzustand eintreten können 9,10,11, und es sind Ansätze zur Untersuchung des Persistenzphänomens auf Einzelzellebene erforderlich, um die Physiologie dieser Persisterzellen zu charakterisieren.
Die jüngste Entwicklung mikrofluidischer Werkzeuge, die in Kombination mit der Fluoreszenzmikroskopie verwendet werden, hat den Weg für die Charakterisierung des Persistenzphänotyps geebnet und die Rolle wichtiger zellulärer Prozesse wie Chromosomenreplikation12, DNA-Reparatur 13 und Zellteilung14 bei der Bildung von Persisterzellen hervorgehoben. In dieser Arbeit beschreiben wir einen integrierten Ansatz, der klassische mikrobiologische Assays mit Einzelzell-Live-Bildgebung kombiniert, um Persisterzellen zu charakterisieren, die in exponentiell wachsenden Escherichia coli-Kulturen erzeugt wurden, die mit einer hohen Dosis Ofloxacin behandelt wurden. Das hier beschriebene Protokoll kann angewendet werden, um das Phänomen der Antibiotikapersistenz bei anderen Bakterienarten, wie z. B. Bacillus subtilis15, oder Erkrankungen (z. B. Antibiotikapersistenz nach β-Lactam-Behandlung 16) zu untersuchen, und kann leicht modifiziert werden, um die vielen Phänomene zu untersuchen, die mit phänotypischer Heterogenität verbunden sind17,18,19 . Darüber hinaus kann der in dieser Arbeit beschriebene Aufbau mit anderen fluoreszierenden Reportern kombiniert werden, um bestimmte zelluläre Parameter von Interesse zu untersuchen, wie z. B. intrazelluläre Konzentrationen von pH20 oder ATP21 auf Einzelzellebene, was möglicherweise neue Einblicke in das Phänomen der Antibiotikapersistenz liefern könnte.
Protokoll
Anmerkungen: Verwenden Sie sterile Kulturgläser, Pipettenspitzen und Nährmedium. Hier wurden E. coli-Zellen in einem chemisch definierten Medium mit niedriger Autofluoreszenz gezüchtet (siehe Materialtabelle). Die Impfungen wurden in Anwesenheit eines Bunsenbrenners durchgeführt, um das Risiko einer Kontamination zu minimieren.
1. Zellkultur und Wachstumskurve
- Streichen Sie den interessierenden Stamm aus einem gefrorenen Glycerinvorrat auf eine Luria-Bertaini (LB)-Agarplatte (ggf. ergänzt mit einem selektiven Antibiotikum) und inkubieren Sie ihn bei 37 °C über Nacht (zwischen 15 h und 19 h), um einzelne Kolonien zu erhalten.
HINWEIS: Das hier vorgestellte Experiment verwendet zwei Stämme, E. coli K-12 MG1655, die dem wt-Stamm entsprechen, und den isogenen MG1655 hupA-mCherry-Stamm 22. Letzterer Stamm exprimiert die fluoreszenzmarkierte α-Untereinheit des HU-Nukleoid-assoziierten Proteins. Der hupA-mCherry-Reporter ist am nativen Ort von hupA integriert. HU-mCherry dient als Proxy, um die Nukleoiddynamik in lebenden Zellen zu verfolgen, da es auf unspezifische Weise an DNA bindet. - Inokulieren Sie 5 mL Medium (hier 3-[N-Morpholino]propansulfonsäure-basiertes Medium [MOPS]; Tabelle 1, Tabelle 2 und Tabelle 3) mit Glukose 0,4 % und einem selektiven Antibiotikum (falls erforderlich) mit einer isolierten Kolonie in einem Glasröhrchen (≥25 ml) ergänzt und das Röhrchen über Nacht (zwischen 15 h und 19 h) in einen Schüttelbrutschrank mit 37 °C und 180 Umdrehungen pro Minute (U/min) geben. Alternativ können anstelle von Glasröhrchen Kunststoffröhrchen, Glas oder Kunststoffkolben (≥ 25 ml) verwendet werden.
HINWEIS: MOPS-Medium, das mit Glycerin in einer Endkonzentration von 0,4 % angereichert war, wurde in allen in diesem Artikel beschriebenen Experimenten verwendet, mit Ausnahme der Nachtkulturen, die in MOPS-Glukose 0,4 % durchgeführt wurden. Die Generationszeit von E. coli in MOPS, das mit Glukose angereichert ist, ist kürzer als die in MOPS-Glycerin. Die Verwendung von MOPS-Glukose 0,4 % anstelle von MOPS-Glycerin 0,4 % in diesem Schritt stellt sicher, dass die Zellen innerhalb von 19 h die stationäre Phase erreichen. Andere Nährmedien, wie z. B. M9 oder ein reichhaltig definiertes Medium (FDM), das mit unterschiedlichen Kohlenstoffquellen ergänzt wird, können ebenfalls verwendet werden. Es ist jedoch zu beachten, dass die Wachstumsrate und die Persistenzhäufigkeit je nach Medium und/oder verwendetem Kohlenstoff unterschiedlich sind23. - Zentrifugieren Sie am nächsten Morgen 1 ml Kultur bei 2.300 x g für 3 Minuten, verwerfen Sie den Überstand und resuspendieren Sie das Pellet vorsichtig in der gleichen Menge phosphatgepufferter Kochsalzlösung (PBS). Messen Sie die optische Dichte bei 600 nm (OD600 nm) und berechnen Sie das Volumen, das für einen anfänglichen OD von600 nm von 0,01 in einem Endvolumen von 2 ml benötigt wird.
- Geben Sie 2 ml MOPS-Glycerin-0,4%-Medium in eine Vertiefung einer durchsichtigen 24-Well-Platte und beimpfen Sie mit dem berechneten Kulturvolumen über Nacht. Legen Sie die 24-Well-Platte in einen automatisierten Mikroplatten-Reader (siehe Materialtabelle), um den OD600 nm für 24 Stunden zu überwachen. Stellen Sie den Mikroplatten-Reader so ein, dass er den OD600 nm alle 15 Minuten bei einer Temperatur von 37 °C und mit hoher Orbitalrotation (140 U/min) misst.
HINWEIS: Wenn der im Experiment verwendete Stamm für einen fluoreszierenden Reporter kodiert, stellen Sie sicher, dass seine Wachstumsrate mit der des wt vergleichbar ist, um Artefakte in den nachfolgenden Experimenten zu vermeiden, da die Wachstumsrate die Persistenzfrequenz von Antibiotikabeeinflusst 23. Aufgrund der Detektionsgrenzen bei sehr niedrigen Zelldichten und der möglichen stammspezifischen Auswirkungen auf die Beziehung zwischen OD600 nm und koloniebildenden Einheiten (KBE) wird empfohlen, die Wachstumskinetik durch die Überwachung der CFU·mL-1 zu bewerten, wenn mit uncharakterisierten Stämmen gearbeitet wird.
2. Bestimmung der minimalen Hemmkonzentration der Antibiotika
Anmerkungen: Die minimale Hemmkonzentration (MHK) ist definiert als die niedrigste Dosis eines Antibiotikums, bei der kein Bakterienwachstum beobachtet wird. Die Bestimmung der MHK muss für jedes Antibiotikum und jeden Stamm durchgeführt werden. In den hier beschriebenen Experimenten wurde das Fluorchinolon-Antibiotikum Ofloxacin (OFX) eingesetzt. Die Bestimmung der MHK ermöglicht die Bestätigung, dass die Antibiotikalösung korrekt hergestellt wurde, das Antibiotikum aktiv ist und die Stämme gleichermaßen empfindlich auf das Antibiotikum reagieren. Hier wurde die veröffentlichte Agar-Verdünnungsmethode durchgeführt, um das MHK zu OFX der verschiedenen verwendeten Stämme zu bestimmen24. Die MHK eines gegebenen Antibiotikums für einen gegebenen Bakterienstamm kann auch über das Bouillonverdünnungsverfahren24 bestimmt werden.
- Vorbereitung der Platten für die MHK-Bestimmung
- Bereiten Sie eine Stammlösung für das in den Experimenten verwendete Antibiotikum vor, indem Sie 5 mg OFX in 1 ml Reinstwasser auflösen. Fügen Sie 20 μl 37% HCl hinzu, um die Löslichkeit des OFX zu erhöhen.
- Schmelzen Sie 100 ml sterilen LB-Agar und halten Sie ihn bei 55 °C, um eine Erstarrung zu vermeiden. Bereiten Sie sechs kleine Glaskolben (25 ml) vor und pipettieren Sie mit einer sterilen Pipette 5 ml flüssiges LB-Agar-Agarmedium in jeden Kolben.
- Verdünnen Sie 10 μL der 5 mg·mL-1 OFX-Stammstofflösung in 90 μL Reinstwasser (1:10 Verdünnung des Stammstoffs). 0 μl, 2 μl, 4 μl, 6 μl, 8 μl oder 10 μl der 500 μg·mL-1 OFX-Lösung in jeden der sechs Glaskolben mit 5 ml LB-Agar-Agar-Medium geben, um LB-Agar-Medium mit Endkonzentrationen von 0 μg·mL−1, 0,02 μg·mL−1, 0,04 μg·mL−1 zu erzeugen, 0,06 μg·mL−1, 0,08 μg·mL−1 bzw. 0,1 μg·mL-1 OFX. Mischen Sie die Lösung, indem Sie die Kolben mehrmals drehen.
HINWEIS: Wenn die MHK des betreffenden Antibiotikums bekannt ist, sollte der Konzentrationsbereich von unterhalb bis oberhalb der tatsächlichen MHK reichen. Wenn das MHK unbekannt ist, wird ein großer Konzentrationsbereich mit einer log-2-Verdünnungsreihe empfohlen. Stellen Sie sicher, dass das LB-Agar-Medium abgekühlt ist, bevor Sie das Antibiotikum hinzufügen, da hohe Temperaturen es inaktivieren können. Dennoch ist es wichtig, das LB-Agar-Medium vor der Zugabe des Antibiotikums nicht erstarren zu lassen, da dies zu einer inhomogenen Verteilung des Antibiotikums im LB-Agar-Medium führen könnte. - Von jedem der sechs in Schritt 2.1.3 hergestellten sechs LB-Agarmedien wird dosiserhöhend mit einer sterilen Pipette 5 ml in eine 6-Well-Kulturplatte gegossen. Lassen Sie den Agar abkühlen, bis er fest ist, und trocknen Sie die Platte vor der Verwendung.
Anmerkungen: Bereiten Sie die Antibiotikalösung und die Kulturplatte am Tag der Durchführung des Assays vor.
- Assay zur MHK-Bestimmung
- Beimpfen Sie 5 ml LB-Medium mit einer isolierten Kolonie in einem Glasröhrchen (≥25 ml) und stellen Sie das Glasröhrchen über Nacht (zwischen 15 h und 19 h) in einen Schüttelinkubator, der auf 37 °C und 180 U/min eingestellt ist.
- Am nächsten Morgen wird die OD600 nm gemessen und die Kultur in einem Reagenzglas auf eine endgültige Zelldichte von 1 x 107 KBE·mL-1 in PBS verdünnt. Für die hier getesteten Stämme entspricht 1 x 107 KBE ·mL−1 einem OD600 nm von 0,0125.
HINWEIS: Wenn die Korrelation zwischen CFU·mL-1 und OD 600 nm nicht bekannt ist, sollte die durch KBE- und OD 600 nm-Messungen ermittelte Wachstumskurve ermittelt werden, um den Korrelationsfaktor zwischen KBE·mL-1 und OD600 nm zu berechnen. - Sprühen Sie 2 μl der zuvor verdünnten Kultur auf jede Vertiefung der getrockneten 6-Well-Platte. Lassen Sie die Flecken trocknen, bevor Sie die Platte über Nacht (zwischen 15 h und 19 h) bei 37 °C in einen Inkubator geben.
- Zählen Sie am nächsten Tag die Kolonien, die sich in jedem Brunnen gebildet haben. Das MHK entspricht dem Bohrloch mit der minimalen Antibiotikakonzentration, bei dem kein Bakterienwachstum festgestellt wird.
3. Stichprobenartiger Test
HINWEIS: Die Spot-Assay-Methode ist ein qualitativer Ansatz, der die Abschätzung der Anzahl lebensfähiger Zellen (Zellen, die in der Lage sind, nach antibiotischem Stress Kolonien zu bilden) ermöglicht. Der Spot-Assay wird vor dem Time-Kill-Assay durchgeführt, um Einblicke in die Lebensfähigkeit des verwendeten Stammes unter den getesteten Bedingungen zu geben und um über die Verdünnungen zu informieren, die während des Time-Kill-Assays erforderlich sind (siehe Abschnitt 4).
- Um die LB-Agarplatten für Spot-Assays vorzubereiten, gießen Sie 50 ml LB-Agar in eine quadratische Petrischale (144cm2). Bereiten Sie eine quadratische Petrischale pro Zeitpunkt vor. Lassen Sie das LB-Agar fest werden und trocknen Sie die Platten vor der Verwendung.
- Beimpfen Sie 5 ml Medium (MOPS-Glukose 0,4 %) mit einer isolierten Kolonie in einem Glasröhrchen (≥25 ml) und legen Sie das Röhrchen über Nacht (zwischen 15 h und 19 h) in einen Schüttelinkubator, der auf 37 °C und 180 U/min eingestellt ist.
- Messen Sie am nächsten Tag den OD 600 nm und verdünnen Sie die Kultur in einem Glasröhrchen (≥25 ml) in frischem, temperaturangepasstem Medium (37 °C, MOPS-Glycerin 0,4 %) auf einen endgültigen ODvon 600 nm von ~0,001. Lassen Sie die Kultur über Nacht in einem Inkubator bei 37 °C bei 180 U/min (zwischen 15 h und 19 h) wachsen.
- Am nächsten Tag wird der OD 600 nm gemessen und die Kultur auf einen endgültigen OD von600 nm von 0,3 inkubiert.
- Bereiten Sie während der Inkubation 96-Well-Platten vor, indem Sie 90 μl 0,01 MMgSO4-Lösung in jede Vertiefung mit Ausnahme der Vertiefungen der ersten Reihe (Reihe A) geben. Die 96-Well-Platten werden für die 10-fache serielle Verdünnung in Schritt 3.7 verwendet.
HINWEIS: In diesem Experiment wurden zwei Stämme (wt und hupA-mCherry-Stamm ) zu sieben verschiedenen Zeitpunkten in dreifacher Ausführung getestet. Da eine Platte aus 12 Säulen besteht, kann eine Platte für zwei Zeitpunkte verwendet werden, so dass insgesamt vier Platten hergestellt wurden. - Bei einem ODvon 600 nm von 0,3 werden 200 μl jeder Bakterienkultur entnommen. Diese Proben entsprechen dem t0 (unbehandelten) Zeitpunkt vor der Antibiotikabehandlung und ermöglichen die Bestimmung der KBE·mL-1 vor der Antibiotikabehandlung.
- Zentrifugieren Sie die Proben bei 2.300 x g für 3 min. Während der Zentrifugationszeit die gewünschte OFX-Konzentration in die Flüssigkultur geben und bei 37 °C unter Schütteln weiter inkubieren.
HINWEIS: Hier wurde OFX in einer Konzentration von 5 μg·mL-1 (entsprechend dem 83-fachen MHK multipliziert) eingesetzt. In einer früheren Studie wurde diese Konzentration verwendet, um das Persistenzphänomen unter OFX-Exposition zu charakterisieren25. Die Antibiotikakonzentration, die für die Time/Kill-Assays verwendet wird, kann je nach Antibiotikum, Nährmedium und Bakterienwachstum variieren. - Nach der Zentrifugation wird das Zellpellet in 200 μl 0,01 MMgSO4-Lösung resuspendiert. Geben Sie 100 μl in die leere Vertiefung der in Schritt 3.5 vorbereiteten 96-Well-Platte. Führen Sie eine Verdünnung durch, indem Sie 10 μl der Vertiefung in Reihe A in die Vertiefung in Reihe B überführen, die 90 μl 0,01 MMgSO4 enthält. Setzen Sie die seriellen Verdünnungen fort, indem Sie 10 μl der Vertiefung in Reihe B in die Vertiefung in Reihe C überführen. Wiederholen Sie den Vorgang, bis eine Verdünnung von 10−7 erreicht ist (wobei jeder Transfer eine 10-fache Verdünnung ist).
HINWEIS: In diesem Experiment wurden sechs Kulturen (drei für den wt-Stamm und drei für den hupA-mCherry-Stamm ) für jeden Zeitpunkt getestet. Dem Protokoll zufolge wird eine Mehrkanalpipette verwendet, um die seriellen Verdünnungen für die sechs Stämme gleichzeitig durchzuführen. Um den technischen Fehler zu minimieren, werden die Pipettenspitzen zwischen jedem Transfer gewechselt. - 10 μl jeder Verdünnung werden auf die in Schritt 3.1 hergestellten LB-Agarplatten aufgetragen.
Anmerkungen: Eine Mehrkanalpipette wird verwendet, um gleichzeitig die gleiche Verdünnung (z. B. eine 10− 4-Verdünnung) für die sechs Kulturen zu erkennen. Während der Fleckung sollte der erste Anschlag der Mehrkanalpipette verwendet werden, ohne den zweiten Anschlag zu drücken, da dies dazu führen kann, dass Mikrotröpfchen auf die Platte abgegeben werden. - Zu relevanten Zeitpunkten nach der Zugabe von Antibiotika sind 200 μl der Kultur zu entnehmen und serielle Verdünnungen durchzuführen, wie in Schritt 3.8 beschrieben. Spott 10 μl jeder Verdünnung, wie in Schritt 3.9 beschrieben.
HINWEIS: Für das hier beschriebene Experiment wurden sieben Zeitpunkte gesammelt (t0, t1h, t2h, t3h, t4h, t5h, t6h). Bei Stämmen, die im Vergleich zur wt eine erhöhte Antibiotikaempfindlichkeit aufweisen, können mehrere Waschgänge in MgSO4 0,01 M durchgeführt werden, um Antibiotikareste zu entfernen. - Inkubieren Sie die Platten bei 37 °C über Nacht (zwischen 15 h und 19 h). Zählen Sie am nächsten Tag die Anzahl der Völker bei den beiden höchsten Verdünnungen, für die Kolonien nachgewiesen werden können. Idealerweise enthalten die Flecken auf den Agarplatten für diese Verdünnungen zwischen 3 und 30 Kolonien, was eine genaue Bestimmung des KBE·mL-1 für jede Probe ermöglicht.
- Berechnen Sie die Überlebensrate, indem Sie die berechnete KBE·mL-1 jedes Zeitpunkts durch die KBE ·mL-1 der Ausgangspopulation bei t0 dividieren.
4. Time-Kill-Assays
HINWEIS: Während Spot-Assays eine einfach anzuwendende Methode sind, um die Überlebensrate eines bestimmten Stammes für ein bestimmtes Antibiotikum abzuschätzen, liefern Time-Kill-Assays eine höher aufgelöste Überlebensrate und werden durchgeführt, um die bakterielle Lebensfähigkeit genau zu quantifizieren. Anhand des Profils der Abtötungskurve kann festgestellt werden, ob ein bestimmter Bakterienstamm unter bestimmten Bedingungen empfindlich, tolerant oder resistent gegen das Antibiotikum ist. Darüber hinaus ermöglichen Time-Kill-Assays die Bestimmung der Zeit der Antibiotika-Exposition, die zum Nachweis des Persistenzphänomens (Beginn der zweiten Steigung der biphasischen Abtötungskurve) sowie der Persistenzhäufigkeit erforderlich ist.
- Bereiten Sie LB-Agar-Agarplatten für den Time-Kill-Plating-Assay vor. Gießen Sie 25 ml LB-Agar in eine Petrischale (±57 cm2). Bereiten Sie mindestens zwei Petrischalen pro Zeitpunkt vor (zwei Verdünnungen pro Zeitpunkt und Stamm werden plattiert).
- Lassen Sie das LB-Agar fest werden und trocknen Sie die Platten, bevor Sie fünf bis acht sterile Glasperlen auf jede Platte geben. Invertieren und beschriften Sie die Platten entsprechend der Dehnung/Bedingung/dem Zeitpunkt.
Anmerkungen: Glasperlen ermöglichen die Ausbreitung von Bakterien auf den Agarplatten in Schritt 4.7 und Schritt 4.9. Alternativ können die Zellen mit einem Spreizer auf der Agarplatte dispergiert werden. - Für jede Probe bereiten Sie 10-fach serielle Verdünnungsglasröhrchen mit 900 μl 0,01 MMgSO4-Lösung vor. Die Anzahl der Verdünnungsglasröhrchen pro Probe, die präpariert werden muss, entspricht den Verdünnungen, die zum Nachweis von Kolonien im Spot-Assay erforderlich sind.
- Beimpfen Sie 5 ml Medium (MOPS-Glukose 0,4 %) mit einer isolierten Kolonie in einem Glasröhrchen (≥25 ml) und legen Sie das Röhrchen über Nacht (zwischen 15 h und 19 h) in einen Schüttelinkubator, der auf 37 °C und 180 U/min eingestellt ist.
- Nach 16 h Wachstum wird der OD 600 nm gemessen und die Kultur in einem Glasröhrchen in frisches, temperaturangepasstes Medium (37 °C, MOPS-Glycerin 0,4 %) auf einen endgültigen ODvon 600 nm von ~0,001 verdünnt. Lassen Sie die Kultur über Nacht in einem Inkubator bei 37 °C bei 180 U/min (zwischen 15 h und 19 h) wachsen.
- Am nächsten Tag wird der OD 600 nm gemessen und die Kultur auf einen endgültigen OD von600 nm von 0,3 inkubiert.
- Bei einem OD von 600 nm von 0,3 werden 100 μl der Kultur entnommen, gemäß den im Spot-Assay erhaltenen Daten verdünnt (bei einer OD von 600 nm von 0,3 und ohne Antibiotikum ergibt eine Verdünnung von 10−5 normalerweise 200-300 Kolonien) und100 μl auf die in den Schritten 4.1-4.2 hergestellten LB-Agarplatten auftragen. Schütteln Sie die Teller vorsichtig, um zu vermeiden, dass die Kügelchen die Ränder der Schale berühren, da dies zu einer inhomogenen Ausbreitung der Bakterienzellen auf dem LB-Agarmedium führen kann. Diese erste Probe vor der Antibiotikabehandlung entspricht dem t0-Zeitpunkt (KBE·ml-1 vor der Antibiotikabehandlung).
- Fügen Sie die gewünschte Konzentration von OFX hinzu und inkubieren Sie bei 37 °C unter Schütteln.
HINWEIS: Hier wurde OFX in einer Konzentration von 5 μg·mL-1 (entsprechend dem 83-fachen MHK multipliziert) eingesetzt. - Zu relevanten Zeitpunkten nach der Zugabe von Antibiotika werden 100 μl der Kultur entnommen, gemäß den im Spot-Assay erhaltenen Daten verdünnt und 100 μl auf die in den Schritten 4.1-4.2 hergestellten LB-Agarplatten aufgetragen. Platten Sie die Zellen wie in Schritt 4.7 beschrieben.
HINWEIS: Für das hier beschriebene Experiment wurden sieben Zeitpunkte gesammelt (t0, t1h, t2h, t3h, t4h, t5h, t6h). Bei Stämmen, die im Vergleich zur wt eine erhöhte Antibiotikaempfindlichkeit aufweisen, können mehrere Waschgänge in MgSO4 0,01 M durchgeführt werden, um Antibiotikareste zu entfernen. - Inkubieren Sie die Platten bei 37 °C über Nacht (zwischen 15 h und 19 h). Zählen Sie am nächsten Tag die Anzahl der Völker bei den beiden höchsten Verdünnungen, für die Kolonien nachgewiesen werden können. Idealerweise sollten die Platten 30-300 Kolonien enthalten, um eine genaue Bestimmung des KBE·mL-1 in jeder Probe zu ermöglichen.
- Berechnen Sie die Überlebensrate, indem Sie die KBE ·mL-1 zu jedem Zeitpunkt um die KBE ·mL-1 bei t0 normalisieren. Zeichnen Sie die logarithmische10 normalisierte KBE·mL-1 als Funktion der Zeit auf.
5. Mikrofluidische Zeitraffer-Mikroskopie-Bildgebung
HINWEIS: Im folgenden Abschnitt werden die Vorbereitung der mikrofluidischen Platte sowie das Verfahren zur Zeitrafferaufnahme und Bildanalyse beschrieben. Das Ziel dieses Experiments ist es, den Persistenzphänotyp nach Antibiotikabehandlung auf Einzelzellebene zu beobachten und zu analysieren. Die während dieses Experiments gesammelten Daten können verwendet werden, um eine breite Palette von Ergebnissen zu generieren, abhängig von der behandelten Fragestellung und/oder den während des Experiments verwendeten fluoreszierenden Reportern. In dem hier beschriebenen Experiment wurde eine quantitative Analyse der Zelllänge und der HU-mCherry-Fluoreszenz22 durchgeführt, die die Nukleoidorganisation in Persister- und Non-Persister-Zellen widerspiegelt.
- Bakterielle Zellkultur für die mikrofluidische Zeitraffermikroskopie
- Beimpfen Sie 5 ml Medium (MOPS-Glycerin 0,4 %, bei Bedarf mit einem selektiven Antibiotikum) mit einer isolierten Kolonie in einem Glasröhrchen (≥25 ml) und legen Sie das Röhrchen über Nacht (zwischen 15 h und 19 h) in einen Schüttelbrutschkasten, der auf 37 °C und 180 U/min eingestellt ist.
- Am nächsten Tag wird der OD 600 nm gemessen und die Kultur in einem frischen, temperaturangepassten Medium (37 °C, MOPS-Glycerin 0,4 %) in einem Glasröhrchen auf einen endgültigen ODvon 600 nm von ~0,001 verdünnt. Lassen Sie die Kultur über Nacht (zwischen 15 h und 19 h) in einem Schüttelinkubator bei 37 °C und 180 U/min wachsen, um am nächsten Tag eine frühe Exponentialphasenkultur zu erhalten.
- Präparation der Mikrofluidikplatte und Zeitraffer-Mikroskopie-Bildgebung
HINWEIS: Mikrofluidische Experimente können in handelsüblichen mikrofluidischen Geräten (wie hier beschrieben) oder in selbst hergestellten mikrofluidischen Systemen durchgeführt werden.- Entfernen Sie die Konservierungslösung (falls vorhanden) aus jeder Vertiefung der mikrofluidischen Platte und ersetzen Sie sie durch ein frisches Nährmedium.
Anmerkungen: Wenn die mikrofluidische Platte eine Abfallauslassvertiefung enthält, sollte die Konservierungslösung der Auslasswanne entfernt, aber nicht durch das Medium ersetzt werden. - Versiegeln Sie die mikrofluidische Platte mit dem Verteilersystem, indem Sie auf die Schaltfläche Versiegeln oder über die Mikrofluidik-Software klicken (wählen Sie zuerst Tool, dann Seal Plate).
Anmerkungen: Um die Platte abzudichten, sollte ein gleichmäßiger Druck auf die Platte und den Verteiler ausgeübt werden, indem die Platte manuell gegen den Verteiler gedrückt wird. Bei korrekter Ausführung sollte der Hinweis "versiegelt" auf der ONIX2-Schnittstelle erscheinen. Es ist wichtig, keinen Druck auf den Glasschieber auszuüben, um ein mögliches Risiko eines Bruchs des Verteilers zu vermeiden. - Führen Sie nach dem Versiegeln eine erste Priming-Sequenz durch (klicken Sie auf Run Liquid Priming Sequence auf der mikrofluidischen Softwareoberfläche).
HINWEIS: Die Run Liquid Priming Sequence entspricht 5 Minuten Perfusion bei 6,9 kPa für Wells 1-5, gefolgt von 5 Minuten Perfusion bei 6,9 kPa für Well 8 und einer abschließenden Perfusionsrunde für Well 6 für 5 min bei 6,9 kPa. Die Run Liquid Priming-Sequenz ermöglicht die Entfernung der Konservierungslösung, die möglicherweise noch in den Kanälen vorhanden ist, die die verschiedenen Vertiefungen verbinden. - Inkubieren Sie die Platte in einem thermostatisch gesteuerten Schrank des Mikroskops bei der gewünschten Temperatur (hier 37 °C) für mindestens 2 h vor Beginn der mikroskopischen Bildgebung.
- Starten Sie eine zweite Run Liquid Priming-Sequenz , bevor Sie mit dem Experiment beginnen.
- Versiegeln Sie die mikrofluidische Platte, indem Sie auf der mikrofluidischen Softwareschnittstelle auf Abdichten klicken. Ersetzen Sie das Medium in Vertiefung 1 und Vertiefung 2 durch 200 μl frisches Medium, in Vertiefung 3 durch 200 μl frisches Medium, das das Antibiotikum enthält (hier OFX bei 5 μg·mL−1), in Vertiefung 4 und Vertiefung 5 durch 200 μl Frischmedium, in Vertiefung 6 mit 200 μl frischem Medium und in Vertiefung 8 mit 200 μl der Kulturprobe (aus Schritt 5.1.2), verdünnt aufOD 600 nm von 0,01 im frischen Medium.
- Verschließen Sie die mikrofluidische Platte wie in Schritt 5.2.2 beschrieben und setzen Sie die Platte auf das Mikroskopobjektiv im Mikroskopschrank.
Anmerkungen: Stellen Sie sicher, dass Sie einen Tropfen Immersionsöl auf das Mikroskopobjektiv geben, bevor Sie die mikrofluidische Platte platzieren. - Klicken Sie in der Mikrofluidik-Software auf Cell Loading , um das Laden der Zellen in die Mikrofluidikplatte zu ermöglichen.
HINWEIS : Der Zellladeschritt umfasst 15 s Perfusion bei 13,8 kPa für Well 8, gefolgt von 15 s Perfusion bei 27,6 kPa für Well 6 und Well 8 und einer abschließenden Perfusionsrunde für Well 6 für 30 s bei 6,9 kPa. Die Dichte der Zellen in der mikrofluidischen Platte ist entscheidend für das Experiment. Der erste Teil dieses mikrofluidischen Protokolls besteht darin, Bakterien vor der Antibiotikabehandlung 6 Stunden lang in einem frischen Medium zu züchten. Nach 6 h Wachstum muss die Zelldichte ausreichend sein, um die seltenen Persisterzellen zu erkennen (unter den in dieser Studie verwendeten Bedingungen erzeugen sich die Persisterzellen mit einer Frequenz von 10−4). Ist die Zelldichte zu hoch, ist es schwierig, die einzelnen Zellen zu unterscheiden, was eine präzise Einzelzellanalyse verhindert. Da die Wachstumsrate direkt vom Medium abhängt, sollte die Dichte der Zellen in den Mikroskopiefeldern vor Beginn des Experiments beurteilt werden. - Stellen Sie im Durchlichtmodus einen optimalen Fokus ein und wählen Sie mehrere Regions of Interest (ROIs) aus, in denen eine geeignete Zellzahl beobachtet wird (bis zu 300 Zellen pro Feld).
HINWEIS: Wählen Sie mindestens 40 ROIs aus, um sicherzustellen, dass die seltenen Persisterzellen abgebildet werden. - Klicken Sie in der Mikrofluidik-Software auf Protokoll erstellen. Programmieren Sie die Injektion von frischem Medium bei 6,9 kPa für 6 h (Well 1-2), gefolgt von der Injektion des Mediums, das das Antibiotikum enthält, bei 6,9 kPa für 6 h (Well 3) und schließlich der Injektion von frischem Medium bei 6,9 kPa für 20 h (Wells 4-5).
HINWEIS: Eine Bakterienkultur, die auf einen OD von600 nm von 0,01 verdünnt ist, ermöglicht es den Bakterien, 6 Stunden lang in der mikrofluidischen Kammer zu wachsen, wodurch sichergestellt wird, dass sich die Zellen in der exponentiellen Wachstumsphase befinden. Abhängig von der Anzahl der Zellen, die während des Ladeschritts in das mikrofluidische Gerät eingebracht werden (siehe Schritt 5.2.8), kann die Dauer der Wachstumsphase angepasst werden, um bis zu 300 Zellen pro ROI zu erhalten. Da Persistenz ein seltenes Phänomen ist, verbessert eine Erhöhung der Anzahl der Zellen pro ROI die Möglichkeit, Persisterzellen zu beobachten. Die Zellzahl sollte jedoch 300 Zellen pro ROI nicht überschreiten, da dies die Einzelzellanalyse mühsam macht. - Führen Sie Mikroskopieaufnahmen im Zeitraffermodus mit einem Bild alle 15 Minuten durch, indem Sie das Durchlicht und die Anregungslichtquelle für den Fluoreszenzreporter verwenden. Hier wurde eine 560 nm Anregungslichtquelle für das mCherry-Signal verwendet (580 nm LED bei 10% Leistung mit Filter 00 [530-585 ex, 615LP em, Zeiss] und 100 ms Belichtung für mCherry). Für die Zellbildgebung kam die Zeiss-kompatible Software Zen3.2 zum Einsatz.
- Entfernen Sie die Konservierungslösung (falls vorhanden) aus jeder Vertiefung der mikrofluidischen Platte und ersetzen Sie sie durch ein frisches Nährmedium.
- Bildanalyse
HINWEIS: Das Öffnen und Visualisieren der Mikroskopiebilder erfolgt mit der Open-Source-Software ImageJ/Fiji (https://fiji.sc/)26. Die quantitative Bildanalyse wird mit der Open-Source-Software ImageJ/Fiji und dem kostenlosen MicrobeJ-Plugin (https://microbej.com)27 durchgeführt. In diesem Protokoll wurde die Version MicrobeJ 5.13I(14) verwendet.- Öffnen Sie die ImageJ/Fiji-Software auf dem Computer und ziehen Sie die Hyperstack-Zeitraffer-Mikroskopiebilder in die Fidschi-Ladeleiste. Verwenden Sie Image > Color > Make Composite , um die verschiedenen Kanäle des Hyperstacks zu verschmelzen. Wenn die Kanäle des Zeitraffer-Experiments nicht der gewünschten Farbe entsprechen (z. B. der Phasenkontrast wird in Rot statt in Grau dargestellt), verwenden Sie Bild > Farbe > Kanäle anordnen , um die entsprechende Farbe auf die Kanäle anzuwenden.
- Öffnen Sie das MicrobeJ-Plugin und erkennen Sie die Bakterienzellen mithilfe der manuellen Bearbeitungsoberfläche. Löschen Sie die automatisch erkannten Zellen, und umreißen Sie die gewünschten Persisterzellen manuell Bild für Bild.
HINWEIS: Es können verschiedene Einstellungen verwendet werden, um einzelne Zellen automatisch zu erkennen. Hier kam die manuelle Detektion zum Einsatz, da die analysierten Persisterzellen lange Filamente bilden, die mit der automatischen Detektion nur selten korrekt detektiert werden. - Verwenden Sie nach der Erkennung das Ergebnissymbol in der manuellen Bearbeitungsoberfläche von MicrobeJ, um eine ResultJ-Tabelle zu generieren. Speichern Sie die ResultJ-Datei, und verwenden Sie die ResultJ-Tabelle, um Einblicke in verschiedene Parameter zu erhalten, die für die Einzelzellenanalyse von Interesse sind. Bei diesem Protokoll wurden die mittlere Fluoreszenz der HU-mCherry-Intensität, die Zelllänge und die Zellfläche der einzelnen Zellen exportiert.
Ergebnisse
Wie oben beschrieben, wurden die Stämme, die für die einzelzellige phänotypische Analyse von Persisterzellen verwendet wurden, in MOPS-Glycerin-0,4%-Medium charakterisiert. Die Überwachung des OD600nm über die Zeit zeigte keinen Unterschied zwischen den wt - und hupA-mCherry-Stämmen (Abbildung 1). Dies deutet darauf hin, dass die Expression des HU-mCherry-Fusionsproteins unter diesen Bedingungen keinen Einfluss auf das Wachstum hatte. Die Bakterienzellen beider Stämme, die initial bei einem OD600 nm von 0,01 inokuliert wurden, erreichten die exponentielle Phase ±8 h nach der Inokulation.
Das MHK von OFX wurde durch standardisierte Methoden (hier serielle Agar-Verdünnung) bestimmt24. MHK ist definiert als die minimale Konzentration, bei der kein sichtbares Wachstum festgestellt wird. Die MHK von OFX für beide Stämme wurde mit 0,06 μg·mL−1 bestimmt, was darauf hindeutet, dass die hupA-mCherry-Fusion keinen Einfluss auf die Sensitivität gegenüber OFX im Vergleich zum isogenen wt-Stamm hatte (Abbildung 2).
Des Weiteren untersuchten wir die Wirkung einer letalen OFX-Behandlung (83-fache MHK) auf die Lebensfähigkeit beider in dieser Studie verwendeter Stämme. Da die Anzahl der lebensfähigen Zellen mit der Zeit bei OFX-Exposition abnimmt, müssen die Verdünnungen der Bakterienkulturen entsprechend angepasst werden, um 30 bis 300 Kolonien pro Platte zu erreichen. Um die geeigneten Verdünnungen über die Zeit zu bestimmen, wurde ein Spot-Assay durchgeführt, bei dem 10 μL von 0 bis 10−7 seriellen 10-fachen Verdünnungen mit einer Mehrkanalpipette auf quadratische Petrischalen gegeben wurden. Die geeigneten Verdünnungen waren diejenigen, bei denen isolierte Klone sichtbar waren (z. B. bei t0 = 10−5, t1h = 10−4/10−3, t4h = 10−2/10−1) (Abbildung 3).
Der Spot-Assay ist zwar eine einfache Methode, um Einblicke in die Kinetik der OFX-vermittelten Tötung zu gewinnen, aber er kann die Tötungsdynamik nicht genau bestimmen. Als die Viabilität exponentiell wachsender Zellen, die mit OFX behandelt wurden, mit dem Time-Kill-Assay überwacht wurde, wurde eine typische biphasische Kurve beobachtet (Abbildung 4). Die erste Steigung der Kurve spiegelt das schnelle Absterben der Nicht-Persister-Population wider (rote gestrichelte Linie). Unter den hier getesteten Bedingungen waren bis zu 99,9% der Zellen nach 3 h in Gegenwart von OFX nicht in der Lage, Kolonien zu bilden. Auf diese erste Phase der Abtötung folgt eine zweite Phase, die eine langsamere Abtötungsrate zeigt (blaue gestrichelte Linie), die das Vorhandensein von medikamententoleranten Persisterzellen verrät. Unter den getesteten Bedingungen begann die Persisterphase etwa 3 h nach der OFX-Zugabe, was die Notwendigkeit unterstreicht, die Zellen länger als 3 h OFX auszusetzen, um die Persister-Phänotypen zu untersuchen. Wichtig ist, dass die Time-Kill-Kurve zeigt, dass das hupA-mCherry-Fusionsprotein keinen Einfluss auf die Time-Kill-Kinetik hatte. Der Stamm, der für die translationale Fluoreszenzfusion kodiert, kann daher verwendet werden, um die Persisterzellen mit Hilfe der Fluoreszenzmikroskopie zu überwachen.
Des Weiteren untersuchten wir das Persistenzphänomen auf Einzelzellebene. Zu diesem Zweck wurde der hupA-mCherry-Stamm in eine mikrofluidische Platte eingebracht, die die Änderung der Mediumbedingungen (hier Wachstum, Behandlung und Erholung) ermöglichte, während Zeitraffermikroskopie bei einem bestimmten ROI durchgeführt wurde. Im ersten Schritt des mikrofluidischen Experiments wurden die in das mikrofluidische Gerät eingebrachten Zellen mit Wachstumsmedium (MOPS-Glycerin 0,4%) perfundiert und mit einer Generationszeit von ~2 h geteilt (Abbildung 5 und Abbildung 6). Diese erste Wachstumsphase deutet darauf hin, dass die Zellen vor der OFX-Behandlung lebensfähig waren und sich aktiv teilten.
Nach dieser ersten Wachstumsphase wurden die Zellen für 6 h mit Wachstumsmedium perfundiert, das mit 5 μg·mL−1 OFX angereichert war. Sobald das Antibiotikum die Zellen erreichte, wurde die Zellteilung blockiert (Abbildung 5 und Abbildung 6). Nach 6 h OFX-Behandlung wurden die Zellen mit frischem Medium perfundiert. Während die überwiegende Mehrheit der Zellen nicht in der Lage war, ihr Wachstum wieder aufzunehmen (Abbildung 5 und Abbildung 6), war eine kleine Subpopulation von Bakterien in der Lage, sich zu verlängern und fadenförmige Zellen zu bilden25. Diese Zellen, die sich nach der OFX-Behandlung teilen und lebensfähige Tochterzellen bilden konnten, können als Persisterzellen definiert werden.
Da dieser Aufbau die Visualisierung der Persisterzellen vor, während und nach der Behandlung ermöglicht, liefert er nicht nur Informationen über den Persister-Phänotyp während der Erholungsphase, sondern auch über den physiologischen Zustand der Persisterzellen vor der Behandlung (Abbildung 6). Unter den getesteten Bedingungen teilten sich die Persisterzellen vor der OFX-Behandlung ähnlich wie Nicht-Persister-Zellen, was darauf hindeutet, dass die beobachteten Persisterzellen nicht aus einer ruhenden Subpopulation stammten (Abbildung 6)25.
Die Zelllängenanalyse von Persisterzellen während der Erholungsphase ergab, dass jedes Filament eine bestimmte Elongationsrate aufwies. Die Zelllänge, die jeder Persister vor der ersten Teilung erreichte, unterschied sich von Persister zu Persister. Ebenso war der Zeitpunkt der Erstligaveranstaltung sehr heterogen (Abbildung 6). Das sich teilende Persisterfilament erzeugte mehrere Tochterzellen, die größtenteils ähnlich wie unbehandelte Zellen zu wachsen und sich zu teilen begannen (Abbildung 7). Die sukzessive Teilung des Filaments führte dann zu einer fortschreitenden Abnahme der Zelllänge, was schließlich zu Tochterzellen mit ähnlicher Zelllänge wie vor der OFX-Behandlung führte (Abbildung 6 und Abbildung 7B). Die überwiegende Mehrheit der Zellen war nach der Entfernung von OFX nicht in der Lage, eine Filamentation zu induzieren. Diese große Zellpopulation entspricht den toten Zellen (Abbildung 5 und Abbildung 6).
Die Fluoreszenzfusion des Nukleoid-assoziierten Proteins HU ermöglicht die Visualisierung der Dynamik des Nukleoids22. Die Analyse der Gesamtfluoreszenzintensität von HU-mCherry innerhalb der Zelle kann als Proxy für den DNA-Gehalt22,25 verwendet werden. Während der Wachstumsphase (vor der OFX-Behandlung) variierte die gesamte mCherry-Fluoreszenzintensität, was die Dynamik der Chromosomenreplikation und -trennung während des Zellzyklus widerspiegelt (Abbildung 8). Nach OFX-Zugabe nahm die mCherry-Fluoreszenz in der Mitte der Zelle zu, was auf eine Nukleoidverdichtung hinweist, die nachweislich durch die Bildung von Doppelstrang-DNA-Brüchen induziert wird28 (Abbildung 5). Doppelsträngige DNA-Brüche sind eine Folge des Wirkmechanismus von OFX, der die Typ-II-Topoisomerasen DNA-Gyrase und Topoisomerase IV korrumpiert29,30. In E. coli ist die DNA-Gyrase das primäre Ziel von OFX29,30. Durch die Bindung seines Ziels an einen kritischen Schritt des Doppelstrang-Passage-Mechanismus hemmt OFX den Abstieg der gespaltenen DNA-Stränge, was letztendlich zur Freisetzung von Doppelstrang-DNA-Brüchenführt 30. Wie oben beschrieben, begannen die Persisterzellen der OFX-Behandlung während der Erholung zu filamenten25 (Abbildung 6). Die Zunahme der Zelllänge korrelierte mit einer Zunahme der gesamten mCherry-Fluoreszenzintensität, was einen Neustart der Replikation und eine Zunahme der Nukleoidhäufigkeit im Filament25 widerspiegelt (Abbildung 7a und Abbildung 8). Bei toten Zellen blieb die gesamte mCherry-Fluoreszenzintensität während der Behandlung und während der Erholungsphase stabil, was darauf hindeutet, dass diese Zellen nach der OFX-Entfernung nicht in der Lage waren, ihre Chromosomen zu replizieren (Abbildung 8). Ein mikrofluidisches Video (Video 1) von E. coli HU-mCherry-Zellen vor, während und nach der Behandlung mit Ofloxacin wird ebenfalls gezeigt.
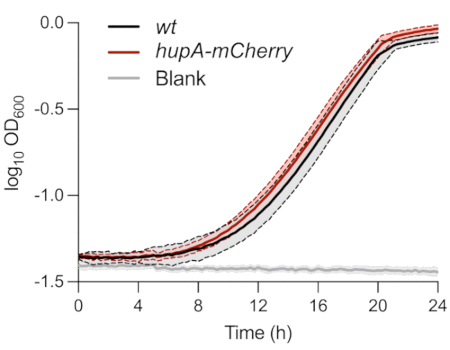
Abbildung 1: Wachstumsüberwachung von wt und hupA-mCherry E. coli Stämmen. Optische Dichteüberwachung (OD600 nm) von wt (schwarz) und hupA-mCherry (rot). Die Schattierungen und gestrichelten Linien zeigen die Standardabweichungen der biologischen Triplikate an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
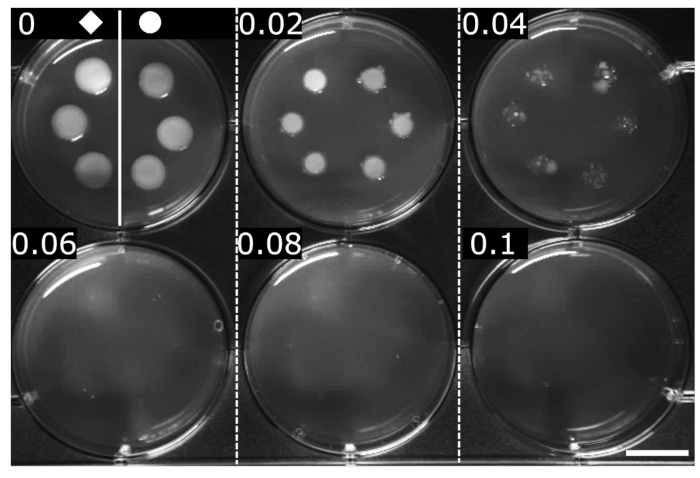
Abbildung 2: Bestimmung des MHK von OFX für die Stämme wt und hupA-mCherry E. coli. wt () und hupA-mCherry (●) wurden in LB-Medium gezüchtet, und 2 μL wurden auf seriellen Verdünnungen von OFX-haltigem LB-Agar entdeckt (♦ Konzentration in jedem Panel in μg·mL−1 angegeben). Die Wachstumshemmung ist bei einem Minimum von 0,06 μg·mL−1 sichtbar. Die Abbildung ist ein repräsentatives Experiment biologischer Triplikate. Maßstabsleiste = 1 cm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
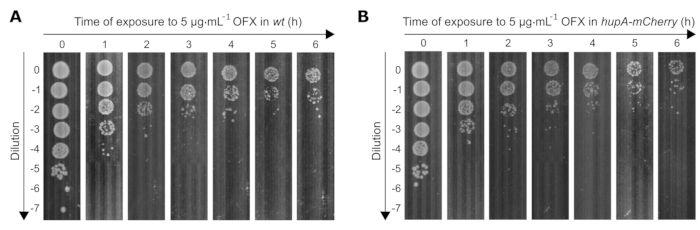
Abbildung 3: Spot-Assay von wt - und hupA-mCherry E. coli-Stämmen nach OFX-Exposition. Die Stämme (A) wt und (B) hupA-mCherry wurden wie im Protokoll (Abschnitt 3) beschrieben in MOPS-Glycerin 0,4 % gezüchtet, und die exponentiell wachsenden Zellen (OD600 nm = 0,3) wurden mit 5 μg·mL−1 OFX behandelt. T0 entspricht dem Zeitpunkt vor der Addition von OFX. T1, T2, T3, T4, T5 und T6 entsprechen 1-6 h nach der OFX-Zugabe. Die Abbildung ist ein repräsentatives Experiment biologischer Triplikate. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
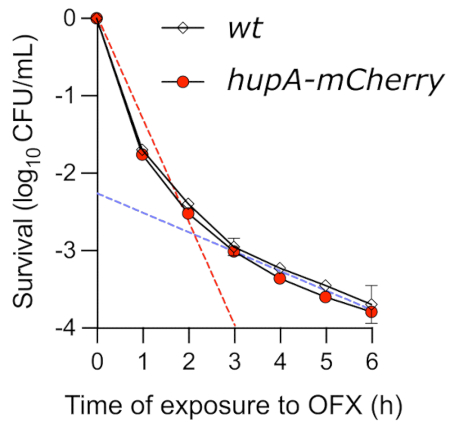
Abbildung 4: Time-Kill-Assay von wt - und hupA-mCherry E . coli-Stämmen bei Exposition gegenüber OFX. Die Stämme wt () und hupA-mCherry (●) wurden in MOPS Glycerin 0,4 % gezüchtet, wie im Protokoll (Abschnitt 4) beschrieben, und die exponentiell wachsenden Zellen (♦ OD600 nm = 0,3) wurden mit 5 μg·mL−1 OFX behandelt. Die gestrichelten Linien zeigen die erste "schnelle" (rote) Tötungsphase und die zweite "langsame" (blaue) Tötungsphase, die den sensiblen und persistenten Subpopulationen entspricht (erhalten durch lineare Regression zwischen T0 und T2 bzw. zwischen T3 und T6). Die Fehlerbalken zeigen die Standardabweichungen der biologischen Triplikate an. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
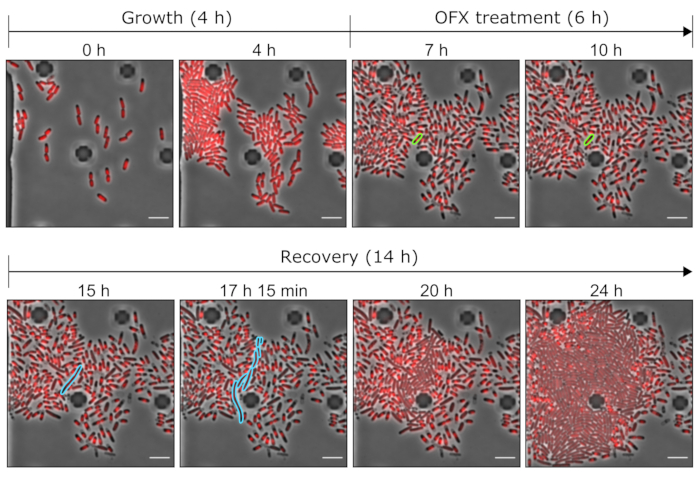
Abbildung 5: Repräsentative Bilder des OFX-Persisters und der toten Zellen mit mikrofluidischen Werkzeugen. Repräsentative Mikroskopieaufnahmen, die die relevanten Zeitpunkte des mikrofluidischen Experiments mit dem hupA-mCherry-Stamm zeigen (Phasenkontrast in grau, HU-mCherry-Signal in rot). Die Zellen, die das markierte hupA-mCherry exprimieren, wurden in einer mikrofluidischen Platte gezüchtet (hier 4 h), gefolgt von einer OFX-Challenge (5 μg·mL−1). Nach 6 h in Gegenwart von OFX wurden die Zellen mit frischem Medium durchblutet, so dass sich die Persisterzellen erholen konnten. Die Persisterzelle und ihre Nachkommen werden während der OFX-Behandlung und nach der OFX-Entfernung grün bzw. blau hervorgehoben. Die entsprechenden Zeitpunkte sind auf jeder Tafel angegeben. Maßstabsbalken = 5 μm. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
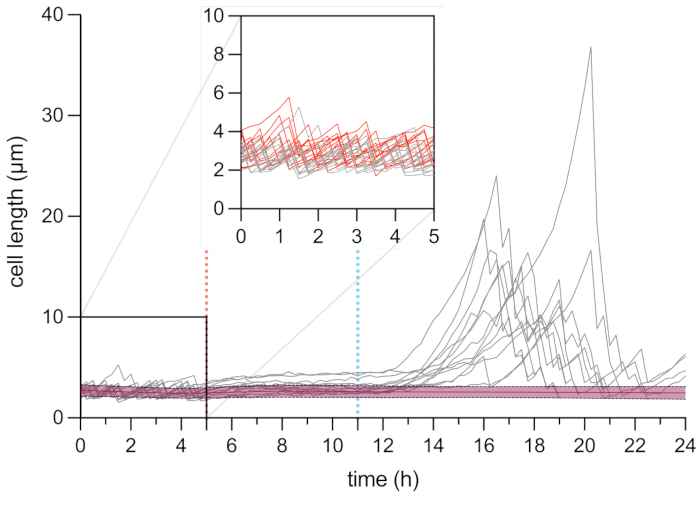
Abbildung 6: Mikroskopische Zeitrafferanalyse der Länge des Persisters und der toten Zellen. Zelllängenanalyse von toten Zellen (in rot, n = 109) und Persisterzellen (in grau, n = 13). Der Beginn der OFX-Behandlung (5 μg·mL−1) wird durch die rot gestrichelte Linie (5 h) und die OFX-Entfernung durch die blau gestrichelte Linie angezeigt. Der Einsatz entspricht der Wachstumsphase vor der OFX-Zugabe. Die Experimente wurden in dreifacher Ausführung durchgeführt. Die Schattierungen und gestrichelten Linien geben die Standardabweichungen für die Population toter Zellen an (n = 109). Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
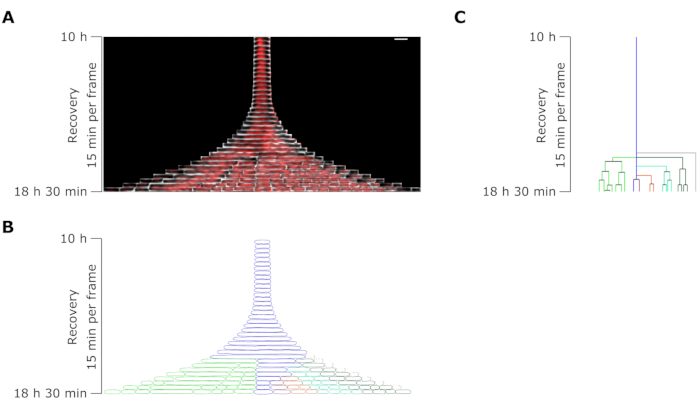
Abbildung 7: Mikroskopische Zeitrafferanalyse eines repräsentativen Persisters zu OFX. (A) Kymograph eines repräsentativen OFX-Persisters und seiner Tochterzellen, die durch Filamentteilungen während 8,5 h nach OFX-Entfernung erzeugt wurden (18,5 h nach Beginn des mikrofluidischen Experiments, bestehend aus 4 h Wachstum, 6 h 5 μg·mL−1 OFX-Behandlung und 8,5 h Erholung nach OFX-Entfernung). Ein Bild entspricht 15 min. Maßstabsbalken = 5 μm. (B) Maske, die aus dem Persister-Kymographen in A generiert wurde. Die überwachte Persisterzelle wird mit einem blauen Umriss gekennzeichnet, und die Tochterzellen werden farblich hervorgehoben. (C) Schematische Darstellung der aus B generierten Persisterzelllinie. Die Farbcodierung ist identisch mit B. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
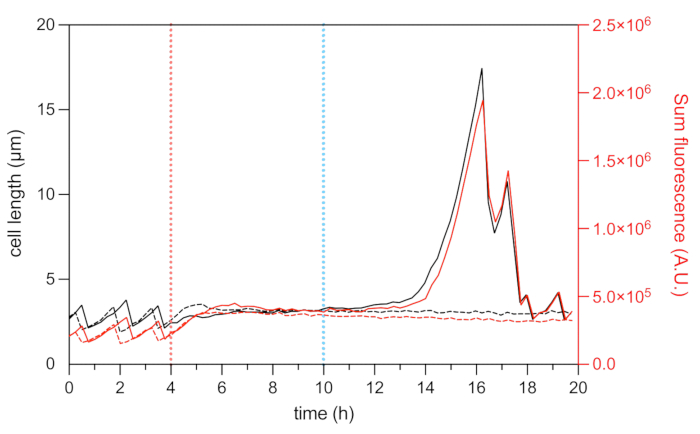
Abbildung 8: Zelllängen- und mCherry-Fluoreszenzanalyse von repräsentativen Persister- und toten Zellen. Analyse der Zelllänge (linke Achse) und der gesamten HU-mCherry-Fluoreszenzintensität (rechte Achse, dargestellt in beliebigen Einheiten) eines repräsentativen Persisters (durchgezogene schwarze und rote Linien) und einer repräsentativen toten Zelle (gestrichelte schwarze und rote Linie) während des mikrofluidischen Zeitrafferexperiments. Der Beginn der OFX-Behandlung (5 μg·mL−1) wird durch die rote gestrichelte Linie und die OFX-Entfernung durch die blaue gestrichelte Linie angezeigt. Bitte klicken Sie hier, um eine größere Version dieser Abbildung zu sehen.
Video 1: Mikrofluidisches Video von E. coli HU-mCherry-Zellen vor, während und nach der Behandlung mit Ofloxacin. Mikrofluidische Zeitrafferaufnahmen mit HU-mCherry-Zellen. Die Zellen wurden für 4 h in MOPS Glycerin 0,4% gezüchtet. Nach 6 h OFX-Behandlung (5 μg·mL−1) wurde das antibiotikafreie Medium in der Mikrofluidikplatte perfundiert, damit sich die Persisterzellen erholen konnten. Maßstabsbalken = 5 μm. Die Zeit (in min) wird angezeigt. Die Wachstums- und Erholungsphasen werden durch "MOPS-Gly. 0,4%" und die OFX-Behandlung mit "OFX 5 μg/mL". Bitte klicken Sie hier, um dieses Video herunterzuladen.
| 10x MOPS | |||
| Lösung aus dem Bestand | Volumen der Stammlösung für 1 L 10x MOPS | Endkonzentration in 10x MOPS-Base | |
| MOPS-Säure | 1 M (mit KOH auf pH 7,4 eingestellt) | 400 ml | 0,4 Mio. |
| Tricin | 1 M (mit KOH auf pH 7,4 eingestellt) | 40 ml | 0,04 M |
| FeSO4.7H2O | 0,01 M | 10 ml | 0,0001 M |
| NH4Cl | 1,9 Mio. | 50 ml | 0,095 Mio. |
| K2SO4 | 0.276 M | 10 ml | 0,00276 M |
| CaCl2.2H 2O | 0.0005 M | 10 ml | 0.000005 M |
| MgCl 2,6H2O | 0,528 M | 10 ml | 0,00528 m |
| NaCl | Direkt hinzufügen 29,2 g | 0,5 M | |
| Destilliertes Wasser | 460 mL | ||
| Mikronährstoffe 1000x (siehe Tabelle 2) | 10 ml | ||
Tabelle 1: Zusammensetzung von 10x MOPS.
| Mikronährstoffe 1000x | ||
| Konzentration in Micronutriments 1000x Stammlösung | Endkonzentration in 10x MOPS-Base | |
| (NH4)6Mo7O24.4H2O | 0.000003 M | 0.00000003 M |
| H3 BO3 | 0,0004 M | 0.000004 M |
| CoCl2.6H 2O | 0.00003 M | 0.0000003 M |
| CuSO4,5H 2Ø | 0,00001 M | 0,0000001 M |
| MnCl2,4H 2Ø | 0.00008 M | 0.0000008 M |
| ZnSO.7H2O | 0,00001 M | 0.00000001M |
Tabelle 2: Zusammensetzung von 1.000x Mikronährstoffen.
| MOPS-Glukose 0,4 % oder MOPS-Glycerin 0,4 % | |||
| Lösung aus dem Bestand | Volumen für 1 L MOPS Glucose 0,4 % oder MOPS Glycerin 0,4 % | Endkonzentration in MOPS-Glukose 0,4 % oder MOPS-Glycerin 0,4 % | |
| 10x MOPS | siehe Tabelle 1 | 100 ml | |
| K2HPO4 | 0,132 Mio. | 10 ml | 0.00132 M |
| Glukose (für MOPS-Glukose 0,4%) | 20% (20 g in 100 ml destilliertem Wasser) | 20 ml | 0.40% |
| Glycerin (für MOPS-Glycerin 0,4%) | ≤99% | 4 ml | 0.40% |
| Destilliertes Wasser | 870 ml für MOPS-Glukose 0,4 % oder 886 ml für MOPS-Glycerin 0,4 % | ||
Tabelle 3: Zusammensetzung von MOPS-Glukose 0,4 % und MOPS-Glycerin 0,4 %.
Diskussion
Das in dieser Arbeit vorgestellte Protokoll ermöglicht die Analyse des Persistenzphänotyps, der als Reaktion auf eine Antibiotikabehandlung auf Populations- und Einzelzellebene beobachtet wird. Die Experimente wurden mit dem E. coli MG1655-Stamm durchgeführt, der in einem chemisch definierten Medium (MOPS-Glycerin 0,4%) gezüchtet wurde. Time-Kill-Assays und Mikroskopie-Experimente wurden an Exponentialphasenkulturen durchgeführt. Wir verwendeten OFX, ein Fluorchinolon, in einer Konzentration von 5 μg·mL−1, um die Persisterzellen sichtbar zu machen. Die hier beschriebenen Ansätze können auf andere bakterizide Antibiotika angewendet werden, wie z. B. β-Lactame, Aminoglykoside oder antimikrobielle Verbindungen31. Dementsprechend können andere Bakterienstämme, Medien oder Wachstumsbedingungen verwendet werden. Die Überwachung verschiedener Fluoreszenzfusionen in einem ähnlichen Aufbau wie dem hier beschriebenen kann nützlich sein, um zelluläre Prozesse wie DNA-Replikation32, DNA-Reparatur25, 33 und Zellteilung34 vor, während und nach der Antibiotikabehandlung zu verfolgen. In ähnlicher Weise können fluoreszierende Reporter genutzt werden, um verschiedene Aspekte der Zellphysiologie zu untersuchen, wie z. B. den intrazellulären pH-Wertvon 35, ATP36 oder ROS37. Alternativ zu fluoreszierenden Fusionen könnten auch chemische Farbstoffe eingesetzt werden. Zum Beispiel könnte die hupA-mCherry-Fusion durch 4',6-Diamidino-2-phenylindol (DAPI) ersetzt werden, einen Fluoreszenzfarbstoff, der DNA38 färbt. Die Durchführung von Zeitraffermikroskopie in Verbindung mit solchen Fluoreszenzfarbstoffen sollte jedoch vermieden werden, da diese Färbetechniken die Dynamik des Zellzyklus während Zeitrafferexperimenten stören können. Alternativ können solche Experimente durch Zeitverlaufsanalysen von Schnappschüssen zu relevanten Zeitpunkten ersetzt werden.
Während solche fluoreszierenden Reporter hilfreich sind, sollte die Menge an Informationen, die durch die Analyse von Phasenkontrastbildern extrahiert werden können, nicht vernachlässigt werden. Hier haben wir die Entwicklung der Zelllänge während des Wachstums, der OFX-Behandlung und der Erholungsphase beobachtet. Andere Parameter, die auf Phasenkontrastbildern basieren, wie z. B. Zellbreite, Phasenkontrastintensität und Krümmungen der Bakterienzellen, können ebenfalls problemlos mit geeigneter Software wie MicrobeJ27 extrahiert werden.
Zusammenfassend lässt sich sagen, dass das hier beschriebene Verfahren auf andere Erkrankungen und Bakterienarten angewendet werden kann, um zelluläre Reaktionen auf sich verändernde Umgebungen oder Stressoren zu überwachen18,19. Durch die Verwendung anderer Fluoreszenzreporter (transkriptionelle und translationale Reporter, chemischer Farbstoff) in Kombination mit einer Populationsanalyse, wie z.B. Durchflusszytometrie/FACS, können interessante Fragestellungen in einem Multiskalenrahmen bearbeitet werden.
Offenlegungen
Die Autoren erklären keine Interessenkonflikte.
Danksagungen
Die Arbeit im Van Melderen-Labor wird durch die ARC-Aktionen 2018-2023, den Fonds National de la Recherche Scientifique (FNRS CDR J.0182.21F), unterstützt. T.O. wird durch ein ULB-Stipendium unterstützt. T.S. wird durch ein FRIA-Stipendium (FNRS) unterstützt. J.C. wird durch ein Postdoktoranden-Stipendium "chargé de recherches" (FNRS) unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Axio Observer | Zeiss | Inverted fluorescence microscope | |
| CaCl2.2H2O | Merck | 1.02382.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CellASIC ONIX Microfluidic System | Merck | CAX2-S0000 | Microfluidic system |
| CellASIC ONIX2 FG | Merck | ONIX2 1.0.1 | Microfluidic software |
| CellASIC ONIX2 Manifold Basic | Merck | CAX2-MBC20 | Manifold system |
| CoCl2.6H2O | Merck | 1.02539.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| CuSO4.5H2O | Merck | 1.02790.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| D-(+)-glucose | Sigma Aldrich | G7021-1KG | Carbon source for MOPS glucose 0.4% growth medium |
| E. coli K-12 MG1655 CF1648 (fnr+) | BE10 | wt reference strain, lab strain | |
| E. coli K-12 MG1655 CF1648 (fnr+) hupA-mCherry::FRT-kan-FRT | BE16 | HU-mCherry fusion integrated via P1 transduction at the native locus, lab strain | |
| FeSO4.7H2O | VWR Chemicals | 24244.232 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Fiji | ImageJ | https://fiji.sc/ | Image software; Schindelin et al. if used in publication |
| Glycerol | Merck | 56815 | Carbon source for MOPS glycerol 0.4% growth medium |
| Greiner CELLSTAR® multiwell culture plate | Merck | M8812-100EA | 24-well clear flat bottom 2ml volume culture plate for automated plate reader |
| H3BO3 | Sigma-Aldrich | B6768-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Hamamatsu ORCA Flash 4.0 digital camera | Hamamatsu | C13440-20CU | Digital Image Acqusition |
| K2HPO4 | Merck | 1.05099.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| KOH | Merck | 1.05029.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| K2SO4 | Merck | 1.05153.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Luria-Broth agar medium | Invitrogen | 22700041 | Growth medium for plating assay |
| Luria-Broth medium | Invitrogen | 12780029 | Growth medium for MIC determination |
| MgCl2.6H2O | Merck | 1.05832.1000 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MgSO4 | Merck | 7487-88-9 | Dilution solution for bacterial survival assay used at 10mM |
| MicrobeJ | Imagej/Fiji plugin | https://www.microbej.com/ | Microscopy image analysis plugin. Ducret et al.for in publication; Detection settings: For bacteria : Area (μm2): 0,1-max; Length (μm): 0,5-max; Width (μm): 0,6-max; Range (μm): 0,5-max; Angularity (rad): 0-0,3; 0-max for all other parameters. |
| Microfluidic Plates CellASIC ONIX | Merck | B04A-03-5PK | Plate for microfluidic system |
| MnCl2.4H2O | Merck | 1.05927.0100 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| MOPS, Free Acid ULTROL® Grade | Merck | 475898-500GM | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NaCl | SIgma-Aldrich | S5886-1KG | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| (NH4)6Mo7O24.4H2O | Merck | 1.01180.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| NH4Cl | Merck | 1.01145.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Ofloxacin | Merck | 82419-36-1 | Fluoroquinolone antibiotic used to treat the bacterial cells |
| Phosphate Buffered Saline (PBS) pH 7.4, 1x | Merck | P3813 | Dilution buffer |
| GENESYS 10S UV-Visible Spectrophotometer | Thermofisher Scientific | 840-208200 | UV-Visible Spectrophotometer, Single/Six Cell Holder with PrinterMeasurement of optical density OD600nm |
| SoftMax Pro | Molecular Devices | Microplate reader software | |
| SpectraMax i3x | Molecular Devices | Microplate reader | |
| N-[Tris(hydroxyméthyl)-méthyl]-glycine | Merck | 1.08602.0250 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
| Zeiss® immersion Oil 518F | Zeiss | Immersion oil to increase resolution of microscope | |
| Zen3.2 Pro | Zeiss | Microscopic image acquisition and processing software | |
| ZnSO4.7H2O | Merck | 1.08883.0500 | For MOPS glucose 0.4% and MOPS glycerol 0.4% growth medium |
Referenzen
- Balaban, N. Q., et al. Definitions and guidelines for research on antibiotic persistence. Nature Reviews Microbiology. 17 (7), 441-448 (2019).
- Brauner, A., Fridman, O., Gefen, O., Balaban, N. Q. Distinguishing between resistance, tolerance and persistence to antibiotic treatment. Nature Reviews Microbiology. 14 (5), 320-330 (2016).
- Pribis, J. P., Zhai, Y., Hastings, P. J., Rosenberg, S. M. Stress-induced mutagenesis, gambler cells, and stealth targeting antibiotic-induced evolution. mBio. 13 (3), 01074 (2022).
- Molina-Quiroz, R. C., Lazinski, D. W., Camilli, A., Levy, S. B. Transposon-sequencing analysis unveils novel genes involved in the generation of persister cells in uropathogenic Escherichia coli. Antimicrobial Agents and Chemotherapy. 60 (11), 6907-6910 (2016).
- Cameron, D. R., Shan, Y., Zalis, E. A., Isabella, V., Lewis, K. A genetic determinant of persister cell formation in bacterial pathogens. Journal of Bacteriology. 200 (17), 00303-00318 (2018).
- Cui, P., et al. Identification of genes involved in bacteriostatic antibiotic-induced persister formation. Frontiers in Microbiology. 9, 413 (2018).
- Li, T., et al. Identification of novel genes involved in Escherichia coli persistence to tosufloxacin. Frontiers in Cellular and Infection Microbiology. 10, 581986 (2020).
- Verstraeten, N., et al. Obg and membrane depolarization are part of a microbial bet-hedging strategy that leads to antibiotic tolerance. Molecular Cell. 59 (1), 9-21 (2015).
- Hofsteenge, N., van Nimwegen, E., Silander, O. K. Quantitative analysis of persister fractions suggests different mechanisms of formation among environmental isolates of E. coli. BMC Microbiology. 13 (1), 25 (2013).
- Huemer, M., Mairpady Shambat, S., Brugger, S. D., Zinkernagel, A. S. Antibiotic resistance and persistence-Implications for human health and treatment perspectives. EMBO Reports. 21 (12), 51034 (2020).
- Wilmaerts, D., Windels, E. M., Verstraeten, N., Michiels, J. General mechanisms leading to persister formation and awakening. Trends in Genetics. 35 (6), 401-411 (2019).
- Murawski, A. M., Brynildsen, M. P. Ploidy is an important determinant of fluoroquinolone persister survival. Current Biology. 31 (10), 2039-2050 (2021).
- Lemma, A. S., Soto-Echevarria, N., Brynildsen, M. P. Fluoroquinolone persistence in Escherichia coli requires DNA repair despite differing between starving populations. Microorganisms. 10 (2), 286 (2022).
- Balaban, N. Q., Merrin, J., Chait, R., Kowalik, L., Leibler, S. Bacterial persistence as a phenotypic switch. Science. 305 (5690), 1622-1625 (2004).
- Morawska, L. P., Kuipers, O. P. Antibiotic tolerance in environmentally stressed Bacillus subtilis: Physical barriers and induction of a viable but nonculturable state. microLife. 3, (2022).
- Windels, E. M., et al. Enrichment of persisters enabled by a ß-lactam-induced filamentation method reveals their stochastic single-cell awakening. Communications Biology. 2 (1), 426 (2019).
- Leygeber, M., et al. Analyzing microbial population heterogeneity-Expanding the toolbox of microfluidic single-cell cultivations. Journal of Molecular Biology. 431 (23), 4569-4588 (2019).
- Rojas, E., Theriot, J. A., Huang, K. C. Response of Escherichia coli growth rate to osmotic shock. Proceedings of the National Academy of Sciences of the United States of America. 111 (21), 7807-7812 (2014).
- Shi, H., et al. Starvation induces shrinkage of the bacterial cytoplasm. Proceedings of the National Academy of Sciences of the United States of America. 118 (24), 2104686118 (2021).
- Goode, O., et al. Persister Escherichia coli cells have a lower intracellular pH than susceptible cells but maintain their pH in response to antibiotic treatment. mBio. 12 (4), 00909-00921 (2021).
- Manuse, S., et al. Bacterial persisters are a stochastically formed subpopulation of low-energy cells. PLoS Biology. 19 (4), 3001194 (2021).
- Fisher, J. K., et al. Four-dimensional imaging of E. coli nucleoid organization and dynamics in living cells. Cell. 153 (4), 882-895 (2013).
- Cabral, D., Wurster, J., Belenky, P. Antibiotic persistence as a metabolic adaptation: Stress, metabolism, the host, and new directions. Pharmaceuticals. 11 (1), 14 (2018).
- Wiegand, I., Hilpert, K., Hancock, R. E. W. Agar and broth dilution methods to determine the minimal inhibitory concentration (MIC) of antimicrobial substances. Nature Protocols. 3 (2), 163-175 (2008).
- Goormaghtigh, F., Van Melderen, L. Single-cell imaging and characterization of Escherichia coli persister cells to ofloxacin in exponential cultures. Science Advances. 5 (6), (2019).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Ducret, A., Quardokus, E. M., Brun, Y. V. MicrobeJ, a tool for high throughput bacterial cell detection and quantitative analysis. Nature Microbiology. 1 (7), 16077 (2016).
- Odsbu, I., Skarstad, K. DNA compaction in the early part of the SOS response is dependent on RecN and RecA. Microbiology. 160 (5), 872-882 (2014).
- Pham, T. D. M., Ziora, Z. M., Blaskovich, M. A. T. Quinolone antibiotics. MedChemComm. 10 (10), 1719-1739 (2019).
- Drlica, K., et al. Quinolones: Action and resistance updated. Current Topics in Medicinal Chemistry. 9 (11), 981-998 (2009).
- Kohanski, M. A., Dwyer, D. J., Collins, J. J. How antibiotics kill bacteria: From targets to networks. Nature Reviews Microbiology. 8 (6), 423-435 (2010).
- Reyes-Lamothe, R., Sherratt, D. J., Leake, M. C. Stoichiometry and architecture of active DNA replication machinery in Escherichia coli. Science. 328 (5977), 498-501 (2010).
- Lesterlin, C., Ball, G., Schermelleh, L., Sherratt, D. J. RecA bundles mediate homology pairing between distant sisters during DNA break repair. Nature. 506 (7487), 249-253 (2014).
- Alexeeva, S., Gadella, T. W. J., Verheul, J., Verhoeven, G. S., Den Blaauwen, T. Direct interactions of early and late assembling division proteins in Escherichia coli cells resolved by FRET: Bacterial division studied by spectral FRET. Molecular Microbiology. 77 (2), 384-398 (2010).
- Miesenböck, G., De Angelis, D. A., Rothman, J. E. Visualizing secretion and synaptic transmission with pH-sensitive green fluorescent proteins. Nature. 394 (6689), 192-195 (1998).
- Yaginuma, H., et al. Diversity in ATP concentrations in a single bacterial cell population revealed by quantitative single-cell imaging. Scientific Reports. 4, 6522 (2014).
- Ermakova, Y. G., et al. Red fluorescent genetically encoded indicator for intracellular hydrogen peroxide. Nature Communications. 5, 5222 (2014).
- Kapuscinski, J. DAPI: A DNA-specific fluorescent probe. Biotechnic & Histochemistry. 70 (5), 220-233 (1995).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten