Method Article
Aislamiento de núcleos de tejido hepático congelado rápidamente para multiómica unicelular
En este artículo
Resumen
Aquí, presentamos un protocolo para aislar núcleos de tejidos hepáticos archivados congelados por flash para RNA-seq de núcleo único, ATAC-seq y multiómica articular (RNA-seq y ATAC-seq).
Resumen
El hígado es un tejido complejo y heterogéneo responsable de llevar a cabo muchas funciones fisiológicas críticas, como el mantenimiento de la homeostasis energética y el metabolismo de los xenobióticos, entre otras. Estas tareas se realizan a través de una estrecha coordinación entre las células hepáticas parenquimatosas y no parenquimatosas. Además, varias actividades metabólicas se limitan a áreas específicas del lóbulo hepático, un fenómeno llamado zonificación hepática. Los avances recientes en las tecnologías de secuenciación de células individuales han permitido a los investigadores investigar la heterogeneidad tisular a una resolución de una sola célula. En muchos tejidos complejos, incluido el hígado, los protocolos de disociación enzimática y/o mecánica severa pueden afectar negativamente la viabilidad o la calidad de las suspensiones unicelulares necesarias para caracterizar exhaustivamente este órgano en salud y enfermedad.
Este documento describe un protocolo robusto y reproducible para aislar núcleos de tejidos hepáticos congelados y archivados. Este método produce núcleos de alta calidad que son compatibles con los enfoques ómicos de una sola célula aguas abajo, incluido el ARN-seq de un solo núcleo, el ensayo para la cromatina accesible a transposasa con secuenciación de alto rendimiento (ATAC-seq), así como las ómicas multimodales (ARN-seq conjunto y ATAC-seq). Este método se ha utilizado con éxito para el aislamiento de núcleos de muestras de hígado congelado de humanos y no humanos sanos y enfermos. Este enfoque permite el aislamiento imparcial de todos los principales tipos de células en el hígado y, por lo tanto, ofrece una metodología sólida para estudiar el hígado a la resolución de una sola célula.
Introducción
La genómica unicelular se está convirtiendo rápidamente en una metodología esencial para estudiar la función hepática y evaluar el impacto de la heterogeneidad celular en condiciones de salud y enfermedad1. El rápido desarrollo de la "multiómica" para la medición simultánea de diferentes capas de información y la expansión paralela de tuberías computacionales robustas está allanando el camino para el descubrimiento de tipos y subtipos celulares previamente desconocidos en el hígado normal y enfermo2.
La posibilidad de explorar biobancos y muestras congeladas archivadas ha aumentado significativamente las oportunidades para revisar y descubrir el papel de las células no parenquimatosas 3,4,5 e investigar el papel de los hepatocitos poliploides durante el envejecimiento y en enfermedades crónicas 6,7,8,9 . Por lo tanto, este documento describe un protocolo de aislamiento de núcleo único robusto y reproducible para hígados archivados congelados (FF) que es compatible con la secuenciación de ARN de núcleo único aguas abajo y la secuenciación ATAC, así como con ómicas multimodales (ARN-seq conjunto y ATAC-seq) (Figura 1).
Este flujo de trabajo permite la investigación del transcriptoma y la accesibilidad a la cromatina de todos los tipos celulares en el hígado, independientemente del tamaño o la fragilidad celular, en protocolos de disociación enzimática. Se puede realizar con pequeñas secciones de tejido (15-30 mg o 5-10 mm3) de muestras humanas preciosas o ratones transgénicos. La determinación de la alta pureza del aislamiento de los núcleos incluye la cuantificación y medición del tamaño nuclear, que podría correlacionarse con el aumento del tamaño celular y la senescencia 10,11, y esta pureza es relevante para el análisis tanto de la ploidía de hepatocitos 12 como de los mecanismos transcripcionales dependientes del tamaño celular11,13,14,15 . Además, los núcleos aislados de hígados congelados retienen información valiosa sobre la zonificación hepática. El flujo de trabajo y la recolección de tejidos permiten la validación de datos genómicos unicelulares o análisis complementarios adicionales, como inmunohistoquímica o transcriptómica espacial del mismo tejido y del mismo individuo. Por lo tanto, este enfoque se puede aplicar a múltiples afecciones de enfermedad hepática y organismos modelo de manera sistemática y confiable.
Protocolo
Todos los experimentos con animales se llevaron a cabo de conformidad con la legislación alemana de bienestar animal y las regulaciones del Gobierno de Alta Baviera. El alojamiento de los animales fue aprobado de acuerdo con el §11 de la Ley alemana de bienestar animal y realizado de acuerdo con la Directiva 2010/63 / UE.
1. Preparación del tejido
- Sacrificar un ratón macho C57BL / 6J de 3 meses a 22 meses de edad por dislocación cervical. Coloque al animal en una tabla de disección, asegure las extremidades con alfileres y desinfecte el abdomen con etanol al 70%.
- Realizar necropsia según lo recomendado por Treuting et al.16.
- Abra el abdomen hasta la caja torácica, visualice el hígado y use fórceps para extraer cuidadosamente el hígado sin perforar los lóbulos.
- Extraiga el hígado intacto sosteniendo el diafragma con fórceps y retirando el tejido de conexión con tijeras.
- Lave el órgano en solución salina tamponada con fosfato frío (PBS), séquelo con una toalla de papel limpia y corte los lóbulos hepáticos en varios pedazos para diferentes propósitos: FF para aislamiento de un solo núcleo y multiómica; fijación de paraformaldehído (PFA al 10%) para la incorporación de parafina; y/o incrustado en un compuesto de temperatura óptima de corte (OCT) para análisis histológicos adicionales (Figura 1A).
- Alícuota las piezas hepáticas en crioviales o tubos de tapón de rosca de 5 ml, e inmediatamente congelar rápidamente en nitrógeno líquido. Almacene las muestras de hígado congeladas a -80 °C para experimentos multiómicos de núcleo único aguas abajo (Figura 1B, C).
NOTA: El tejido criopreservado se puede almacenar de forma segura a -80 °C durante varios años y siempre debe transportarse en hielo seco a -80 °C antes de su uso.
2. Aislamiento de núcleos
- Limpieza de sobremesa y preparación de los buffers y consumibles
- Limpie los bancos de trabajo y las pipetas con una solución de descontaminación de etanol y RNasa al 70%, o utilice mesas y materiales dedicados libres de RNasa.
- Preenfríe tanto el cucharón oscilante como las centrífugas de ángulo fijo, los tubos de 1,5 ml/2 ml y las placas de pocillos múltiples a 4 °C, como se detalla a continuación.
- Preenfríe las pinzas de un solo uso sin RNasa utilizadas para la manipulación de tejidos, un bisturí estéril desechable y una placa de Petri en hielo seco (-60 °C).
- Enfriar previamente el homogeneizador de vidrio Dounce y los morteros sobre hielo (4 °C). Coloque cada mortero (A y B) en un tubo de 5 ml para evitar el contacto directo con el hielo y la posible contaminación por RNasa.
- Prepare una solución diluyente de medio de iodixanol (IDM) (Tabla 1A) y úsela para hacer diluciones al 50% y 29% del medio de gradiente de densidad de material de iodixanol al 60% (Tabla 1B y Tabla 1C, respectivamente).
- Preenfríe todos los tubos. Para cada muestra que se va a procesar, prepare los siguientes tubos:
- Prepare tres tubos de unión de ADN bajo de 1,5 ml (uno para el homogeneizado de tejido filtrado, un segundo que contenga 250 μL de una dilución al 50% de solución de iodixanol y un tercero para la suspensión de núcleos limpios).
- Prepare un tubo de fondo redondo de 2 ml que contenga 500 μL de una dilución al 29% de solución de iodixanol para la separación por gradiente de densidad.
- Preparar un tubo cónico de 15 ml para el medio de aislamiento de núcleos-2 (NIM-2) y el tampón de homogeneización (HB).
- Preparar el aislamiento de núcleos medio-1 (NIM-1) (Tabla 1D), y utilizarlo para realizar el NIM-2 (Tabla 1E) y, posteriormente, el HB (Tabla 1F). Agregue ambos inhibidores de la ARNasa justo antes de usar, como se describe a continuación en el protocolo.
- Prepare el tampón de almacenamiento de núcleos (NSB) como se describe en la Tabla 1G. Agregue un inhibidor de la ARNasa recombinante justo antes de usar. Agregue un inhibidor de ARNasa basado en proteínas en el NSB antes de usarlo para la clasificación de FACS (opcional).
- Prepare un vaso de precipitados de 500 ml con agua estéril para remojar los homogeneizadores y morteros Dounce después de la homogeneización del tejido para la limpieza y mantenimiento óptimos de los homogeneizadores.
- Homogeneización tisular
- Corte una pieza de tejido de 20-30 mg (o 5 mm3) con un bisturí preenfriado dentro de la placa de Petri en hielo seco. A continuación, transfiera inmediatamente la placa de Petri a hielo húmedo (4 °C). Agregue 1 ml de HB y, con el bisturí frío, pique el tejido tanto como sea posible para permitir que se aspire fácilmente con una punta de orificio ancho de 1 ml.
NOTA: Utilice siempre puntas de orificio ancho para todas las transferencias de tejido/núcleo. Como alternativa, se pueden cortar puntas de 1 ml con un bisturí estéril sobre una cubierta de plástico estéril para generar orificios anchos (Figura 2A). - Recoja la suspensión de tejido y transfiérala a un homogeneizador de vidrio de 2 ml preenfriado (Figura 2B).
- Lave la placa de Petri con 0.5-1 ml adicionales de HB y recoja todas las piezas de tejido restantes mientras mantiene todo en hielo.
- Lentamente y con cuidado, haz cinco golpes con el mortero suelto A sobre hielo. Evite crear burbujas usando movimientos de torsión de la mano de mortero al tirar de ella hacia arriba y hacia abajo. Asegúrese de que el mortero se mueva cuidadosamente de la parte superior a la inferior del homogeneizador con cada golpe.
- Posteriormente, realice 10-15 movimientos lentos con mortero B apretado sobre hielo . Evita crear burbujas.
NOTA: Se recomienda inspeccionar visualmente los núcleos bajo el microscopio después de 10 golpes con mortero B para determinar si se necesitan más golpes. Para ello, utilice tinción con azul de tripano (proporción 1:1 de tripano a muestra) y un hemocitómetro manual (p. ej., mezcle 10 μL de azul de tripano con 10 μL de suspensión de núcleos y use 10 μL de la mezcla para examinarla bajo el microscopio; Figura 2C). - Filtre el homogeneizado a través de un filtro celular de 50 μm mientras lo transfiere a un tubo preenfriado de 1,5 ml. Use más de un filtro y/o tubo para homogeneizados que contengan una gran cantidad de grumos de tejido conectivo.
- Enjuague el homogeneizador y los filtros utilizados con 0.5-1 ml adicionales de HB para recoger completamente todo el homogeneizado del tejido. Proceda a la centrifugación por gradiente de densidad.
- Corte una pieza de tejido de 20-30 mg (o 5 mm3) con un bisturí preenfriado dentro de la placa de Petri en hielo seco. A continuación, transfiera inmediatamente la placa de Petri a hielo húmedo (4 °C). Agregue 1 ml de HB y, con el bisturí frío, pique el tejido tanto como sea posible para permitir que se aspire fácilmente con una punta de orificio ancho de 1 ml.
- Centrifugación por gradiente de densidad
- Centrifugar el homogeneizado filtrado en una centrífuga preenfriada de ángulo fijo a 1.000 × g durante 8 min a 4 °C.
- Mientras la muestra está girando, prepare un tubo de 1,5 ml que contenga 250 μL de dilución de iodixanol al 50% y un tubo de 2 ml que contenga 500 μL de dilución de iodixanol al 29%. Mantenga ambos tubos en hielo.
- Después de la centrifugación, aspirar el sobrenadante sin alterar el pellet con una bomba de vacío.
NOTA: El uso de pipeteo manual comprometerá la calidad de la suspensión final de los núcleos. - Añadir 250 μL de HB al pellet utilizando la punta de pipeta de orificio ancho de 1 ml y resuspender muy lentamente.
- Transfiera 250 μL de la suspensión de núcleos a un tubo preenfriado de 1,5 ml que contenga 250 μL de dilución de iodixanol al 50%, y mezcle suave pero completamente para generar una suspensión de iodixanol/núcleos al 25%.
- Transfiera 500 μL de la suspensión de yododinaxanol/núcleos al 25% a un tubo preenfriado de 2 ml que contenga 500 μL de dilución de iodixanol al 29%.
NOTA: Los 500 μL de la suspensión de iodixanol/núcleos al 25% deben depositarse suavemente sobre los 500 μL de solución de iodixanol al 29%, de modo que las mezclas de iodixanol al 25%/29% muestren una clara separación de fases. Utilice el lado de la pared del tubo con la punta de la pipeta colocada en un ángulo de 45° para crear esta interfaz de degradado. A partir de entonces, el tubo debe manipularse suavemente para no alterar este gradiente. - Centrifugar el tubo en una centrífuga de cucharón oscilante preenfriada a 12.500 g durante 20 minutos con el freno apagado.
- Justo antes de completar el paso de centrifugación, agregue los inhibidores de ARNasa al tampón NSB cuando proceda a las tuberías scRNA-seq (consulte la Tabla 1G).
- Después de la centrifugación, aspirar el sobrenadante sin alterar el pellet con una bomba de vacío.
NOTA: El uso de pipeteo manual comprometerá la calidad de la suspensión final de los núcleos. - Con la punta de la pipeta de orificio ancho de 1 ml, resuspenda suavemente el pellet en 100-300 μL de NSB y transfiera la suspensión de núcleos a un tubo limpio y preenfriado de 1,5 ml.
- Contar los núcleos usando solución de azul de tripano (proporción 1:1 de tripano a muestra) y un hemocitómetro manual (p. ej., mezclar 10 μL de azul de tripano con 10 μL de suspensión de núcleos, y usar 10 μL de la mezcla para el recuento; Figura 2D).
- Utilice inmediatamente la suspensión de núcleos obtenida para el ensayo genómico de núcleo único.
NOTA: La suspensión de núcleos en NSB se puede almacenar refrigerada a 4 °C durante un máximo de 1 semana para su posterior análisis por citometría de flujo y/o flujo de imágenes, pero no para snRNA-seq o snATAC-seq.
3. Clasificación de núcleos para el perfil de ploidía de hepatocitos o enfoques de secuenciación bien basados
- Para la clasificación celular basada en citometría de flujo, filtrar la suspensión de núcleos a través de un filtro de 50 μm en un tubo FACS preenfriado de 5 ml.
- Utilice un clasificador de citometría de flujo equipado con una boquilla de 100 μm. Cargue el tubo FACS en el clasificador y obtenga una vista previa de la muestra.
- Configure la estrategia de compuerta para la clasificación de núcleos, comenzando con una puerta de dispersión trazando el área de dispersión hacia adelante frente al área de dispersión lateral (FSC-A / SSC-A), seguida de Hoechst-Height versus Hoechst-Area (puerta de núcleos), y luego Hoechst-Width versus Hoechst-Area (puerta singlets). Visualice el perfil de ploidía de los núcleos en el histograma del área de Hoechst.
NOTA: Para una mejor resolución máxima, visualice el canal de Hoechst (450/50) en la escala lineal (Figura 3A). - Para clasificar en placas de 96/384 pocillos, ajuste el retardo de gotas y optimice la alineación de la placa utilizando el método colorimétrico con sustrato de bencidina peroxidasa de rábano picante (TMB-HRP), como se describió anteriormente6.
- Ajuste tanto el enfriamiento de la muestra como el soporte de la placa para que se enfríen a 4 °C, con la rotación de la muestra encendida a 300 rpm.
- Ordenar los núcleos individuales a una concentración de muestra de ~1 x 105 núcleos/ml y con un caudal de 200-500 eventos/s.
4. Inspección visual y cuantificación de parámetros nucleares mediante citometría de imagen (opcional)
- Cargue un tubo de 0,5 ml que contenga 50 μL de la suspensión de núcleos a una concentración de 2 × 107 núcleos/ml.
- Configure una puerta de todos los eventos basada en la relación de aspecto frente al canal de fluorescencia de Hoechst para visualizar el perfil de ploidía de los núcleos (Figura 3B).
- Adquiera la muestra con un aumento de 40x utilizando campo claro (BF) y el canal de fluorescencia de Hoechst.
- Inspeccione y cuantifique núcleos 2n y 4n utilizando la medición de campo claro y la intensidad de fluorescencia de Hoechst con software de citometría de imágenes (Figura 3C).
5. Construcción y secuenciación de bibliotecas de ARN-seq, ATAC-seq o multioma de un solo núcleo
- Para enfoques snRNA-seq basados en gotas, cargue la suspensión de núcleos purificados directamente en el dispositivo microfluídico para la partición paralela automatizada y el código de barras molecular17.
- Después de completar la ejecución microfluídica en el dispositivo de partición de una sola célula, recoja los núcleos encapsulados en perlas de gel, incube y limpie como se describe anteriormente en las pautas del fabricante17.
- Realice 11 ciclos de reacción en cadena de la polimerasa (PCR) para la preamplificación del ADNc utilizando el siguiente programa: 3 min a 98 °C, (15 s a 98 °C, 20 s a 63 °C y 1 min a 72 °C) x 11, 1 min a 72 °C y mantener a 4 °C. Continúe con una reparación final y un paso de cola en A y la ligadura del adaptador, según lo indicado por el fabricante17. Para la posterior construcción final de la biblioteca de expresión génica, realice 10 ciclos de PCR utilizando el siguiente programa: 45 s a 98 °C, (20 s a 98 °C, 30 s a 54 °C, 20 s a 72 °C) x 10, 1 min a 72 °C y mantener a 4 °C.
- Secuencie las bibliotecas obtenidas a una profundidad de lectura de ~20,000-50,000 lecturas medias por núcleo.
- Para la secuenciación basada en gotitas de multiómica articular (ARN + ATAC), incubar los núcleos hepáticos en tampón de lisis durante 5 minutos y luego etiquetarlos durante 1 h, como se describió anteriormente18.
- Cargue los núcleos etiquetados directamente en un dispositivo microfluídico para la partición paralela automatizada y el código de barras molecular.
- Una vez completada la ejecución microfluídica, recoja los núcleos encapsulados en perlas de gel, incube y limpie como lo describe el fabricante18.
- Realice seis ciclos de PCR para el paso de preamplificación del ADNc utilizando el siguiente programa: 5 min a 72 °C, 3 min a 98 °C, (20 s a 98 °C, 30 s a 63 °C, 1 min a 72 °C) x 6, 1 min a 72 °C y mantener a 4 °C.
- Tomar 35 μL de la muestra preamplificada y realizar una amplificación de ADNc de la siguiente manera: 3 min a 98 °C, (15 s a 98 °C, 20 s a 63 °C, 1 min a 72 °C) x 6, 1 min a 72 °C y mantener a 4 °C. Continúe con la reparación final y el paso de cola en A y la ligadura del adaptador, según lo indicado por el fabricante18 . Para la posterior PCR de indexación final de la muestra, realice 15 ciclos de PCR de la siguiente manera: 45 s a 98 °C, (20 s a 98 °C, 30 s a 54 °C, 20 s a 72 °C) x 15, 1 min a 72 °C y mantener a 4 °C.
- Para la construcción de bibliotecas ATAC, use 40 μL y amplíe durante seis ciclos de PCR para la indexación de muestras utilizando el siguiente programa: 45 s a 98 °C, (20 s a 98 °C, 30 s a 67 °C, 20 s a 72 °C) x 6, 1 min a 72 °C y mantener a 4 °C.
- Secuenciar las bibliotecas de expresión génica multioma obtenidas a una profundidad de lectura mínima de 20.000 pares de lectura por núcleo y las bibliotecas ATAC multiome a una profundidad de lectura mínima de 25.000 pares de lectura por celda, según lo recomendado por el fabricante18.
Resultados
Este flujo de trabajo para el aislamiento de un solo núcleo a partir de muestras de hígado congeladas está diseñado para la multiómica de un solo núcleo y se basa en tres pasos principales, que se pueden resumir como i) recolección de muestras para el análisis paralelo de heterogeneidad celular y arquitectura tisular, ii) suspensión de un solo núcleo y iii) multiómica de un solo núcleo (Figura 1 ). Los hígados extraídos se diseccionan de ratones sacrificados y se cortan en pedazos para su inspección histológica para la incorporación de parafina, la criosección o ambas. Otras piezas cortadas se congelan inmediatamente en nitrógeno líquido para el aislamiento de un solo núcleo aguas abajo para análisis multiómicos. Este sistema de recolección de tejidos permite al usuario validar aún más los datos ómicos de un solo núcleo en secciones de tejido del mismo individuo, complementando así el conjunto de datos con transcriptómica espacial o con análisis inmunohistoquímicos, si es necesario.
El examen microscópico de los núcleos extraídos de hígados congelados con el método descrito aquí muestra que el paso de centrifugación del gradiente de densidad facilita enormemente la eliminación de restos celulares y tisulares no deseados (Figura 2C, D). Además, esta metodología preserva todos los niveles de ploidía, que pueden ser validados y cuantificados por análisis citométricos (Figura 3).
Para validar aún más el rendimiento de este protocolo, realizamos snRNA-seq basado en gotas en núcleos no clasificados y ordenados por FACS y analizamos los datos siguiendo la tubería de Seurat. En resumen, los núcleos únicos extraídos se prepararon para snRNA-seq basado en gotitas como se describió anteriormente19. Para el snRNA-seq de núcleos 2n y 4n o niveles superiores de ploidía, se utilizaron 11 ciclos para el paso de amplificación del ADNc y 10 ciclos para la construcción final de la biblioteca de expresión génica. Las bibliotecas resultantes se secuenciaron a una profundidad de lectura de ~ 25,000-39,000 lecturas medias por núcleo. Las lecturas de núcleos únicos obtenidos se mapearon al genoma de ratón GRCm39 / mm39. Al ejecutar la canalización de preprocesamiento, se agregó el comando -- include-intron para determinar la inclusión y cuantificación del ARN mensajero no empalmado (ARNm) presente en los núcleos. El algoritmo EmptyDrops incorporado dentro del alineador filtró y eliminó las gotas vacías.
El paquete R Seurat (versión 4.1.1) se utilizó para calcular métricas de control de calidad (QC) utilizando la matriz de recuento de identificador molecular único (UMI) como lo genera la canalización de análisis snRNA-seq. Se eliminaron los recuentos con menos de 100 características (genes) y menos de 10 células. Los núcleos se filtraron de acuerdo con los umbrales de control de calidad identificados: número mínimo de genes = 200 y número máximo de genes = 8.000, fracción mitocondrial <1% y fracción ribosómica <2%. Los 3.000 genes altamente variables (HVG) principales se utilizaron para el análisis de componentes principales (PCA), como se implementó en Seurat. La agrupación basada en gráficos se realizó después de ingresar las 15 dimensiones principales de PC resultantes del análisis PCA. Para agrupar las celdas, aplicamos la técnica de optimización de modularidad (algoritmo de Lovaina) con un parámetro de resolución establecido en 0,5. Para visualizar y explorar este conjunto de datos, ejecutamos la reducción dimensional no lineal, a saber, Uniform Manifold Approximation and Projection (UMAP). La identidad de cada conglomerado fue asignada con base en el conocimiento previo de los genes marcadores 6,20,21.
La Figura 4A muestra métricas de alta calidad a partir de los datos obtenidos utilizando este método de extracción de núcleos. UMAP representa el número de recuentos en todos los núcleos y los principales tipos de células que se pueden identificar con confianza solo con el transcriptoma nuclear y la secuenciación relativamente superficial (~ 25,000-40,000 lecturas medias por célula) (Figura 4B). Este enfoque permite la investigación de factores de transcripción específicos del hígado, como Hnf4a, Mlxipl y Ppara, así como genes diana posteriores implicados en el metabolismo de los xenobióticos (es decir, Cyp2e1 y Cyp2f2) (Figura 4C). Cabe destacar que los núcleos extraídos retuvieron información crítica sobre la zonificación hepática, como lo demuestra el patrón complementario de genes distintivos como el Cyp2e1 pericentral y el periportal Cyp2f2 (Figura 4C).
Además, evaluamos la compatibilidad de los núcleos no clasificados extraídos utilizando este método con ensayos multiómicos más nuevos para el perfil simultáneo del paisaje epigenómico (ATAC) y la expresión génica (ARN) en los mismos núcleos únicos18. Optimizamos el tiempo de incubación de la lisis de los núcleos a 5 min antes de la transposición. Las bibliotecas de secuenciación se construyeron ejecutando seis ciclos de PCR para la preamplificación de ADNc. De la muestra preamplificada, se tomaron 35 μL y se amplificaron aún más mediante 15 ciclos de PCR para la indexación de la muestra para construir la biblioteca de expresión génica. Un electroferograma representativo de la traza de ADNc se muestra en la Figura 5A (arriba). Para la construcción de las bibliotecas ATAC, se utilizaron 40 μL de muestra preamplificada y se ejecutaron durante seis ciclos de PCR adicionales para la indexación de la muestra, con el electroferograma representativo de la traza de ADN que se muestra en la Figura 5A (abajo). La biblioteca de expresión génica (ARN) resultante se secuenció a una profundidad de 44.600 lecturas por núcleo y la biblioteca ATAC a una profundidad de 43.500 lecturas por núcleo (Figura 5B).
Similar al protocolo de secuenciación basado en gotas de modalidad única descrito anteriormente, el mapeo de lectura, la alineación, la eliminación de gotas vacías y el recuento de fragmentos se realizaron siguiendo las pautas estándar, como se describió anteriormente, utilizando el genoma de referencia GRCm39 / mm3918. Utilizando los paquetes Seurat y Signac22, realizamos un análisis de "vecino más cercano ponderado" (WNN) para múltiples mediciones de ambas modalidades (ARN + ATAC) (Figura 5C), lo que nos permitió identificar y anotar los tipos de células hepáticas mayores y menores sin sesgos prominentes debido al tamaño de la célula o la fragilidad nuclear (Figura 5D). La tubería, según lo publicado por el laboratorio Satija22,23, incluye pasos estándar de control de calidad -preprocesamiento y reducción dimensional- realizados en ambos ensayos de forma independiente. Para tener una buena representación de la combinación ponderada de las modalidades RNA-seq y ATAC-seq, se trazó el gráfico WNN y se utilizó para la visualización, agrupamiento y anotación UMAP basado en genes marcadores previamente identificados 6,20,21. De manera similar a los ensayos que utilizan modalidad única, también detectamos reguladores transcripcionales aguas arriba (Hnf4a, Ppara, Mlxipl) y genes distintivos de la zonificación hepática (Hamp, Cyp2e1 y Cyp2f2) (Figura 5E).
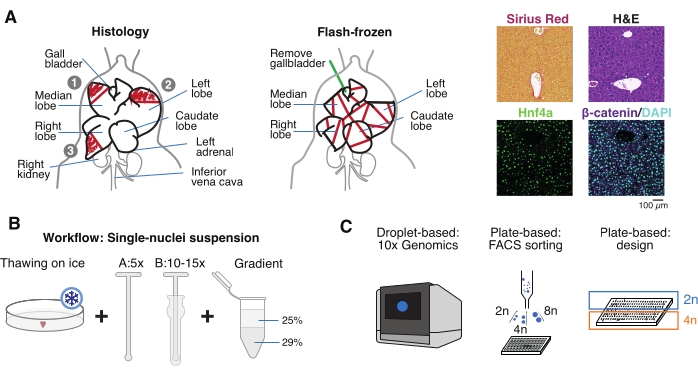
Figura 1: Descripción general experimental, flujo de trabajo y aplicaciones genómicas unicelulares. (A) Representación ilustrativa del muestreo de tejido para histología (izquierda, se seleccionan tres secciones para la incorporación de parafina y/o criosección), recolección de tejido congelado rápidamente para genómica unicelular (centro) y análisis representativos de inmunohistoquímica e inmunofluorescencia (derecha); barra de escala = 100 μm. (B) Pasos críticos para suspensiones de un solo núcleo de alta calidad. (C) Las suspensiones de núcleos pueden cargarse en un chip de cromo 10x o utilizarse para la clasificación de FACS y los enfoques basados en placas. Abreviaturas: H&E = hematoxilina y eosina; DAPI = 4',6-diamidino-2-fenilindol; FACS = clasificación celular activada por fluorescencia. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Disección hepática y homogeneización del tejido en vidrio. (A) Hígado murino representativo de un ratón C57BL6/J de 3 meses de edad (izquierda); La sección hepática utilizada para los aislamientos de un solo núcleo antes (medio) y después de picar el tejido con el bisturí (derecha). Barras de escala = 1 cm. (B) Imágenes ilustrativas de 2 ml Homogeneización del vidrio de rebote antes de los trazos con mortero "suelto" A (izquierda) y después del mortero "apretado" B (derecha). Monitorización de la homogeneización tisular mediante un hemocitómetro (C) antes de la centrifugación por gradiente y (D) después de la centrifugación por gradiente. Haga clic aquí para ver una versión más grande de esta figura.
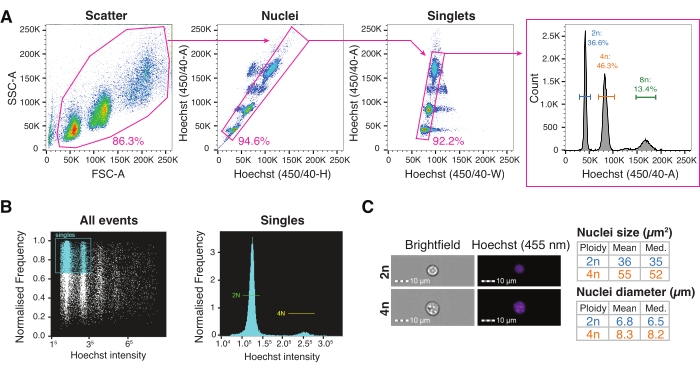
Figura 3: Clasificación celular activada por fluorescencia y citometría de flujo de imágenes para la caracterización de núcleos de alto rendimiento . (A) Estrategia de compuerta para la clasificación de FACS de un solo núcleo en una placa para el interrogatorio de diferentes niveles de ploidía. Puerta de dispersión basada en el área de dispersión hacia adelante versus el área de dispersión lateral configurada para excluir residuos; puerta de núcleos basada en Hoechst-Height versus Hoechst-Area que incorpora múltiples poblaciones de núcleos; puerta de singletes basada en Hoechst-Width versus Hoechst-A establecido para la discriminación de dobletes; el histograma Hoechst-A permite la visualización del perfil de ploidía de los núcleos. (B) Cuantificación por citometría representativa de imágenes de todos los eventos (izquierda) y eventos únicos (derecha), mostrando núcleos diploides y tetraploides. (C) Imágenes de campo claro y Hoechst de núcleos 2n y 4n y su cuantificación mediante citometría de imagen. Abreviaturas: FACS = clasificación celular activada por fluorescencia; FSC-A = área de dispersión hacia adelante; SSC-A = área de dispersión lateral; Hoechst-H = altura de Hoechst; Hoechst-A = Área de Hoechst; Hoechst-W = Hoechst-Width. Haga clic aquí para ver una versión más grande de esta figura.
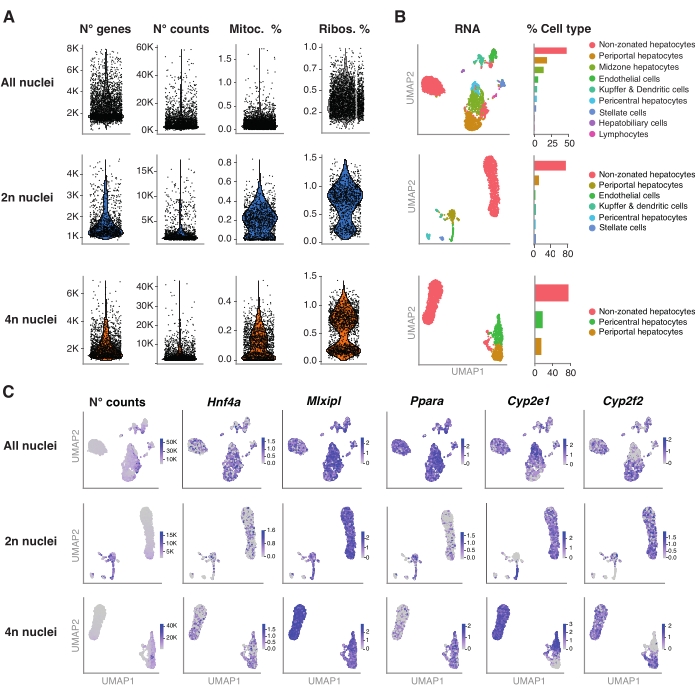
Figura 4: Caracterización profunda de la ploidía de hepatocitos con snRNA-seq . (A) Gráficos de violín que muestran el número de genes, recuentos, porcentaje de genes mitocondriales y porcentaje de genes ribosómicos detectados. (B) UMAP que demuestra los tipos celulares detectados utilizando snRNA-seq (izquierda), con la cuantificación expresada en porcentaje de núcleos (derecha). (C) UMAP ilustra el número de recuentos e indica la expresión de genes específicos de hepatocitos. Todos los núcleos (fila superior), 2n núcleos (fila central) y 4n núcleos (fila inferior). Abreviaturas: snRNA-seq = RNA-seq de núcleo único; UMAP = Aproximación y proyección uniforme de la variedad; mitoc. = genes mitocondriales; Ribos. = genes ribosómicos. Haga clic aquí para ver una versión más grande de esta figura.
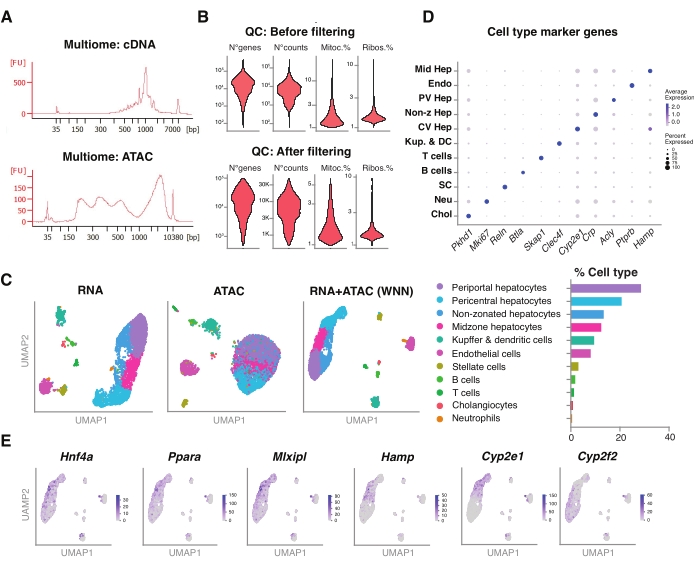
Figura 5: Control de calidad y análisis de multiómica (conjunto RNA-seq y ATAC-seq) a partir de hígados jóvenes congelados y archivados . (A) Trazas electroforéticas automáticas representativas obtenidas después de la tubería multiómica que muestran el producto de peso molecular después de la síntesis de ADNc y ATAC. (B) Diagrama de violín que muestra el número de recuentos para ATAC-seq, RNA-seq, el porcentaje de genes mitocondriales y el porcentaje de genes ribosómicos antes y después del filtrado. (C) UMAP muestra la expresión génica de RNA-seq (izquierda), ATAC-seq (centro) y modalidades conjuntas-RNA-seq y ATAC-seq (derecha). (D) Diferentes tipos de células se anotan en diferentes colores, con la expresión de genes específicos de hepatocitos indicados. (E) Gráfico de características que muestra la expresión específica de conglomerados de los genes indicados en los tipos de células indicados. Abreviaturas: ATAC-seq = ensayo para cromatina accesible a transposasa con secuenciación de alto rendimiento; Hep media = hepatocitos zonales medios; Endo = células endoteliales; PV Hep = hepatocitos periportales; Hep no z = hepatocitos no zonificados; CV Hep = hepatocitos pericentrales; Kup & DC = Kupffer & células dendríticas; SC = células estrelladas, Neu = neutrófilos; Chol = colangiocitos; mitoc. = genes mitocondriales; Ribos. = genes ribosómicos. Haga clic aquí para ver una versión más grande de esta figura.
| Reactivo | Acción | 10 ml | 15 ml | 50 ml | ||
| (A ) Medio de iodixanol (IDM) | ||||||
| 250 mM de sacarosa | 1M | 12.5 mL | ||||
| 150 mM KCl | 2M | 3,75 ml | ||||
| 30 mM MgCl2 | 1M | 1,5 ml | ||||
| Tampón Tris de 60 mM pH 8.0 | 1M | 3 ml | ||||
| Agua ultrapura libre de RNasa | 29,25 ml | |||||
| B) 50 % IDM | ||||||
| Iodixanol | 60% | 12.5 mL | ||||
| IDM | 2,5 ml | |||||
| (C) 29% IDM | ||||||
| Iodixanol | 60% | 7,25 ml | ||||
| IDM | 7,75 ml | |||||
| d ) Aislamiento de núcleos medio-1 (NIM-1) | ||||||
| 250 mM de sacarosa | 1M | 12.5 mL | ||||
| KCl de 25 mM | 2M | 0,625 ml | ||||
| 5 mM MgCl2 | 1M | 0,25 ml | ||||
| Tampón Tris de 10 mM pH 8.0 | 1M | 0.5 mL | ||||
| Agua ultrapura sin Rnase | 36.125 mL | |||||
| e ) Aislamiento de núcleos medio-2 (NIM-2) | ||||||
| Búfer NIM-1 | 9,99 ml | |||||
| Ditiotreitol (TDT) | 1 mM | 0,01 ml | ||||
| Tableta de inhibidor de la proteasa (sin EDTA) | 1 | 1 tableta | ||||
| f ) Tampón de homogeneización (HB) | ||||||
| Búfer NIM-2 | 9.697 mL | |||||
| Inhibidor de la RNasa recombinante | 40 U/μL | 0.1 mL | ||||
| Inhibidor de la ARNasa basado en proteínas (SUPERase•IN) | 20 U/μL | 0.1 mL | ||||
| 0.1% Tritón-X | 10% | 0.1 mL | ||||
| 3 μg/ml Hoechst 33342 | 10 mg/ml | 0.003 mL | ||||
| g ) Tampón de almacenamiento de núcleos (NSB) | ||||||
| 166.5 mM de sacarosa | 1M | 1.665 mL | ||||
| 5 mM MgCl2 | 1M | 0,05 ml | ||||
| 10 mM Tris pH 8.0 | 1M | 0.1 mL | ||||
| Inhibidor de la RNasa recombinante | 40 U/μL | 0.1 mL | ||||
| Inhibidor de la RNasa basado en proteínas (SUPERase•IN) * | 20 U/μL | 0.1 mL | ||||
| Agua ultrapura libre de RNasa | 8.085 mL | |||||
| * Opcional (solo para clasificación FACS) | ||||||
Tabla 1: Recetas de soluciones. (A) Preparación de medio de iodixanol (IDM); (B) dilución al 50% de solución de iodixanol; (C) Dilución al 29% de solución de iodixanol. (D) Preparación de núcleos de aislamiento medio-1 (NIM-1). (E) Preparación de núcleos de aislamiento medio-2 (NIM-2). (F) Preparación del tampón de homogeneización (HB). (G) Preparación del tampón de almacenamiento de núcleos (NSB).
Discusión
La disección de la composición celular del hígado por RNA-seq de una sola célula o de un solo núcleo proporciona una comprensión más profunda del desarrollo y la progresión de la enfermedad hepática 3,4,5,24. El aislamiento unicelular de hígados consume mucho tiempo y requiere protocolos que involucran disociación mecánica o enzimática severa25,26,27. Es ampliamente aceptado que cada tejido requiere una evaluación sistemática para determinar el protocolo óptimo de disociación tisular, así como un método de almacenamiento adecuado para capturar tipos de células frágiles o núcleos28. Dependiendo de la disponibilidad de tejido, la enfermedad de interés, la etapa de desarrollo o el organismo modelo, la preparación de una suspensión de un solo núcleo para el procesamiento posterior podría ser una metodología más adecuada que el uso de suspensiones unicelulares. Es importante destacar que, en el hígado, scRNA-seq y snRNA-seq han mostrado una alta correlación entre el ARNm nuclear y citoplasmático, sugiriendo que ambos enfoques presentan información complementaria 2,3,4,6,29.
Este documento proporciona un aislamiento estandarizado, robusto y reproducible de un solo núcleo a partir de muestras de hígado congeladas y archivadas de ratones y otras especies, incluidos humanos y macacos. Este método se puede utilizar para ratones de tipo salvaje alimentados con comida y una dieta alta en grasas (HFD) y para modelos de fibrosis hepática en ratones utilizando enfoques genómicos de núcleo único basados en placas y gotitas6. Este método se basa en el protocolo descrito originalmente para el tejido cerebral por Krishnaswami et al.30 con modificaciones adicionales adaptadas para el hígado congelado rápidamente. La homogeneización óptima libera la mayoría de los núcleos del tejido sin afectar negativamente la integridad de la membrana nuclear. Sin embargo, la sobreduplicación puede dañar los núcleos frágiles y disminuir su calidad general. Los hígados jóvenes y / o grasos generalmente requieren solo 5 golpes con mortero A y 10 golpes con mortero B, mientras que los hígados viejos y / o fibróticos pueden requerir 15 golpes con mortero B pero no más. No se recomienda, por lo tanto, realizar más trazos más allá de los números aquí indicados. La sobreduplicación podría afectar negativamente la calidad de la suspensión de un solo núcleo y aumentar la cantidad de ARN ambiental. Posteriormente, esto podría llevar a la necesidad de realizar pasos adicionales de filtrado computacional durante los análisis de datos posteriores.
El protocolo presentado aquí es versátil y se puede ajustar a diferentes afecciones hepáticas en ratones jóvenes (3 meses) y viejos (24 meses). Dado que encontramos que una sección más grande del hígado es necesaria para los tejidos viejos, HFD y fibróticos, el tamaño del tejido disponible para el procesamiento puede representar una limitación para algunos usuarios con cantidades más pequeñas de material biológico inicial. Sin embargo, la purificación por gradiente es muy recomendable para el procesamiento inmediato de muestras con ensayos genómicos basados en gotas. Si los núcleos necesitan ser FACS ordenados en placas de 96/384 pocillos para ensayos bien basados, se puede omitir la purificación por gradiente. Alentamos a los usuarios a realizar la purificación del gradiente si hay suficiente muestra de tejido para obtener la concentración de núcleos recomendada para la clasificación de FACS (es decir, ~ 1 × 105 núcleos / ml).
El hígado se caracteriza por la naturaleza poliploide de los hepatocitos9, pero el papel de la ploidía de los hepatocitos en la fisiología normal y la enfermedad aún no está claro. Existe un creciente cuerpo de evidencia que indica que la ploidía proporciona variabilidad genómica31, y es bien sabido que la ploidía aumenta con la edad 32,33. Sin embargo, el enriquecimiento de los hepatocitos tetraploides mononucleados también se asocia clínicamente con un mal pronóstico en el carcinoma hepatocelular humano (CHC)34. Del mismo modo, los cambios en los niveles de ploidía de hepatocitos están relacionados con enfermedades hepáticas crónicas relacionadas con el envejecimiento, como la enfermedad del hígado graso no alcohólico (NAFLD)35,36,37. La ploidía es la condición de poseer más de dos copias del genoma, que se puede explorar teñiendo el contenido del genoma con un tinte de ADN como Hoechst38. El colorante Hoechst, que se agrega al HB antes de la extracción, etiqueta todos los núcleos durante el protocolo de aislamiento. Esto permite la distinción entre núcleos diploides y poliploides en función de su contenido de ADN cuando son excitados por un láser UV (350 nm) o violeta (450 nm) en un instrumento de citometría de flujo. Con la estrategia de compuerta mostrada, se pueden investigar niveles 2n, 4n, 8n y más altos de ploidía de hepatocitos en hígados congelados y archivados para comprender mejor el papel de la heterogeneidad celular en la función tisular1 (Figura 3A). Además, la morfología nuclear, incluyendo el tamaño y el volumen, se puede cuantificar utilizando citometría de flujo de imágenes para correlacionar los cambios en el tamaño del núcleo con los cambios en el número total de recuentos o el número de genes dependiendo del nivel de ploidía (Figura 3B, C).
La medición ómica multimodal ofrece la oportunidad de investigar varias capas de organización genómica simultáneamente. El enfoque multiómico conjunto ARN + ATAC permite la investigación de reguladores aguas arriba y genes metabólicos aguas abajo, proporcionando un enfoque integral para estudiar las redes transcripcionales y la arquitectura de la cromatina asociada con la función hepática a la resolución de una sola célula. Además, con los avances en los métodos computacionales que pueden explicar la escasez de datos y la reducción en los costos de secuenciación, la multiómica de una sola célula es pionera en la evaluación de múltiples modalidades de la misma célula. Este protocolo de aislamiento de núcleo único es compatible con la evaluación individual y conjunta de conjuntos de datos de expresión y cromatina. Hemos utilizado pipelines estándar establecidos por Stuart et al.23 (paquete Signac) para ilustrar la calidad de los datos, mientras que varios métodos computacionales disponibles y alternativos pueden ser fácilmente adoptados para análisis posteriores23,39,40,41.
En general, la multiómica de un solo núcleo permite la investigación de tejidos hepáticos de primates bioarchivados, de ratón FF, humanos y no humanos utilizando una cantidad muy pequeña de material de muestra inicial mediante la implementación del protocolo de extracción de núcleos presentado aquí. Esta herramienta invaluable permitirá a los biólogos hepáticos interrogar tanto la expresión génica como la accesibilidad a la cromatina en el contexto de diversas patologías hepáticas. Además, varios niveles de ploidía de hepatocitos y el ajuste resultante de la expresión génica dependiendo de su ubicación en el lóbulo hepático podrían revelar su papel en las patologías hepáticas. Por lo tanto, anticipamos que la investigación de la heterogeneidad celular proporcionará nuevas oportunidades para el desarrollo de la medicina de precisión y las intervenciones dirigidas contra enfermedades como el CHC y la NAFLD.
Divulgaciones
Los autores declaran que no tienen conflictos de intereses.
Agradecimientos
Esta investigación fue apoyada por el Helmholtz Pioneer Campus (M.S., K.Y., C.P.M.-J.) y el Instituto de Biología Computacional (C. T.-L.). Esta investigación también fue apoyada por AMED bajo el número de subvención JP20jm0610035 (C.P.M.-J.). Agradecemos el apoyo de Core Genomics en HMGU (I. de la Rosa) y Bioinformatics (T. Walzthoeni), en particular Xavier Pastor por su capacitación y orientación. Agradecemos a A. Feuchtinger, U. Buchholz, J. Bushe y a todos los demás miembros del personal de la instalación central de Patología y Análisis de Tejidos de HMGU por su apoyo técnico y científico, así como a J. Zorn, R. Erdelen, D. Würzinger, miembros del personal de E-Streifen, así como a la instalación central de Servicios para Animales de Laboratorio por su apoyo científico y discusión en curso. Estamos agradecidos con Core Facility Cell Analysis en TranslaTUM (R. Mishra) y Luminex, A DiaSorin Company (P. Rein). Agradecemos al Dr. I Deligiannis por su apoyo técnico. El Dr. M. Hartman, el Dr. A. Schröder y la Sra. A. Barden (Helmholtz Pioneer Campus) fueron fundamentales para su apoyo legal, administrativo y administrativo.
Materiales
| Name | Company | Catalog Number | Comments |
| 10% Tween 20 - 5 mL | Bio-Rad | 1662404 | |
| 2100 Bioanalyzer Instrument | Agilent | G2939BA | |
| Adhesive PCR film | Thermo Fisher Scientific | AB0558 | |
| Bioanalyzer High Sensitivity DNA Analysis | Agilent | 5067-4626 | |
| C1000 Touch Thermal Cycler | Bio-Rad | 1851196 | |
| Cell Sorter | For fluoresence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. | ||
| Centrifuge 5430R | Eppendorf | 5428000619 | Use chilled at 4 °C |
| Chromium Controller & Next GEM Accessory Kit | 10X Genomics | 1000204 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | 1000127 | |
| Chromium Next GEM Chip J Single cell kit, 16 reactions | 10X Genomics | 1000230 | |
| Chromium Next GEM Single Cell 3' Kit v3.1, 4 reactions | 10X Genomics | 1000269 | |
| Chromium Next GEM Single Cell Multiome ATAC + Gene Expression Reagent Bundle, 4 reactions | 10X Genomics | 1000285 | |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Sigma Aldrich | 11873580001 | |
| Dithiothreitol (DTT) 1 M solution | Sigma Aldrich | 646563 | Make 1 mM stock solution and use at 1 µM final concentration. |
| DNA AWAY Surface Decontaminant | Thermo Fisher Scientific | 10223471 | Wipe surfaces and pipettes before start of experiment |
| DNA LoBind Tubes, 1.5 mL | Thermo Fisher Scientific | 16628742 | |
| DNA LoBind Tubes, 2.0 mL | Thermo Fisher Scientific | 16638742 | |
| Elution Buffer (EB) - 250 mL | Qiagen | 19086 | |
| Eppendorf ThermoMixer C | Thermo Fisher Scientific | 13527550 | |
| Eppendorf ThermoMixer C Accessory: Smartblock | Thermo Fisher Scientific | 13518470 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade - 100 mL | Thermo Fisher Scientific | 10517694 | |
| Filters 50 µm, sterile | SYSMEX PARTEC - CELLTRICS | 04-004-2327 | Adjust filter diameter according with tissue and nuclei size |
| Glycerin (Glycerol), 50% (v/v) - 1 L | Ricca Chemical Company | 3290-32 | |
| Hard-Shell, 384-Well PCR Plates, thin wall, skirted, clear/white | Bio-Rad | HSP3905 | |
| Herenz Heinz ABS Forceps | Thermo Fisher Scientific | 1131884 | |
| Hoechst 33342, Trihydrochloride, Trihydrate, 10 mg/mL Solution in Water | Invitrogen | H3570 | Light-sensitive |
| Imaging Flow Cytometer | For imaging flow cytometry analysis, e.g. Luminex Amnis ImageStream | ||
| Invitrogen TE Buffer - 100 mL | Thermo Fisher Scientific | 11568846 | |
| KCl (2 M), RNase-free, 100 mL | Thermo Fisher Scientific | AM9640G | |
| MgCl2 (1 M), 100 mL | Thermo Fisher Scientific | AM9530G | |
| MicroAmp 8-Cap Strip, clear-300 strips | Thermo Fisher Scientific | 10209104 | |
| MicroAmp 8-Tube Strip, 0.2 mL-125 strips | Thermo Fisher Scientific | 10733087 | |
| Mörser 2 mL DOUNCE | Wagner & Munz GmbH | 9651632 | RNase zap and rinse with MillQ before use |
| MyFuge 12 Mini MicroCentrifuge C1012 | Benchmark Scientific | C1012 | Or any other strip and tube mini centrifuge |
| Neubauer Hemocytometer | OMNILAB LABORZENTRUM | 5435293 | Visualize and count nuclei under microscope |
| Nuclease-Free Water (not DEPC-Treated) | Thermo Fisher Scientific | AM9937 | |
| OptiPrep Density Gradient Medium | Sigma Aldrich | D1556 | |
| Pipette tips RT LTS 1000 µL, Wide-O | Mettler Toledo | 30389218 | |
| Pistill "A" 2 mL | Wagner & Munz GmbH | 9651621 | RNase zap and rinse with MillQ before use |
| Pistill "B" 2 mL | Wagner & Munz GmbH | 9651627 | RNase zap and rinse with MillQ before use |
| Polypropylene 15 mL Centrifuge Tube | Thermo Fisher Scientific | 10579691 | |
| Polystyrene Petri dish, 60 mm x 15 mm | Thermo Fisher Scientific | 10634141 | |
| Polystyrene Round-Bottom 5 mL FACS Tubes | Thermo Fisher Scientific | 10100151 | |
| Protector RNase inhibitor - 2,000 U | Sigma Aldrich | 3335399001 | Keep in -20 °C until use |
| Protein-based RNase Inhibitor SUPERase•In (20 U/μL) | Thermo Fisher Scientific | AM2696 | Keep in -20 °C until use |
| Recombinant RNase Inhibitor | Clontech Takara | 2313B | Keep in -20 °C until use |
| RNAse free microfuge tubes - 0.5 mL | Thermo Fisher Scientific | AM12450 | |
| RNaseZap RNase Decontamination Solution | Thermo Fisher Scientific | AM9780 | Wipe surfaces and pipettes before start of experiment |
| SPRIselect - 60 mL | Beckman Coulter | B23318 | Aliquot and store in 4 °C |
| Sucrose, 500 g | Sigma Aldrich | S0389-500G | Make a 1 M stock solution |
| Swann-Morton Sterile Disposable Stainless Steel Scalpels | Thermo Fisher Scientific | 11798343 | |
| Tris-HCI (1M), pH 8.0 | Invitrogen | 15568025 | |
| Triton X-100, 98%, 100 mL | Thermo Fisher Scientific | 10671652 | Make 10% stock solution. Keep at 4 °C and protect from light. |
| Trypan Blue Solution, 0.4% | Thermo Fisher Scientific | 11538886 | |
| Vortex- Mixer | VWR | 444-1372 | Or any other type of vortex |
Referencias
- Kamies, R., Martinez-Jimenez, C. P. Advances of single-cell genomics and epigenomics in human disease: where are we now. Mamm Genome. 31 (5-6), 170-180 (2020).
- Ramachandran, P., Matchett, K. P., Dobie, R., Wilson-Kanamori, J. R., Henderson, N. C. Single-cell technologies in hepatology: New insights into liver biology and disease pathogenesis. Nature Reviews Gastroenterology & Hepatology. 17 (8), 457-472 (2020).
- Ramachandran, P., et al. Resolving the fibrotic niche of human liver cirrhosis at single-cell level. Nature. 575 (7783), 512-518 (2019).
- Andrews, T. S., et al. Single-cell, single-nucleus, and spatial RNA sequencing of the human liver identifies cholangiocyte and mesenchymal heterogeneity. Hepatology Communications. 6 (4), 821-840 (2022).
- Xiong, X., et al. Landscape of intercellular crosstalk in healthy and NASH liver revealed by single-cell secretome gene analysis. Molecular Cell. 75 (3), 644-660 (2019).
- Richter, M. L., et al. Single-nucleus RNA-seq2 reveals functional crosstalk between liver zonation and ploidy. Nature Communications. 12 (1), 4264 (2021).
- Matsumoto, T., Wakefield, L., Tarlow, B. D., Grompe, M. In vivo lineage tracing of polyploid hepatocytes reveals extensive proliferation during liver regeneration. Cell Stem Cell. 26 (1), 34-47 (2020).
- Chen, F., et al. Broad distribution of hepatocyte proliferation in liver homeostasis and regeneration. Cell Stem Cell. 26 (1), 27-33 (2020).
- Donne, R., Saroul-Ainama, M., Cordier, P., Celton-Morizur, S., Desdouets, C. Polyploidy in liver development, homeostasis and disease. Nature Reviews. Gastroenterology & Hepatology. 17 (7), 391-405 (2020).
- Lengefeld, J., et al. Cell size is a determinant of stem cell potential during aging. Science Advances. 7 (46), 0271 (2021).
- Lanz, M. C., et al. Increasing cell size remodels the proteome and promotes senescence. Mol Cell. 82 (17), 3255-3269 (2022).
- Kim, J. Y., et al. PIDDosome-SCAP crosstalk controls high-fructose-diet-dependent transition from simple steatosis to steatohepatitis. Cell Metabolism. 34 (10), 1548-1560 (2022).
- Padovan-Merhar, O., et al. Single mammalian cells compensate for differences in cellular volume and DNA copy number through independent global transcriptional mechanisms. Molecular Cell. 58 (2), 339-352 (2015).
- Miettinen, T. P., et al. Identification of transcriptional and metabolic programs related to mammalian cell size. Current Biology. 24 (6), 598-608 (2014).
- Vargas-Garcia, C. A., Ghusinga, K. R., Singh, A. Cell size control and gene expression homeostasis in single-cells. Current Opinion in Systems Biology. 8, 109-116 (2018).
- Knoblaugh, S. E., Randolph-Habecker, J. . Necropsy and histology. In Comparative Anatomy and Histology: A Mouse, Rat, and Human Atlas (Second Edition). , (2018).
- Chromium Next GEM Single Cell 3 Reagent Kits v3.1 User Guide. Document number CG000204 (Rev D). 10x Genomics Available from: https://www.10xgenomics.com/support/single-cell-gene-expression/documentation/steps/library-prep/chromium-single-cell-3-reagent-kits-user-guide-v-3-1-chemistry (2019)
- Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide. Document number CG000338 (Rev D). 10x Genomics Available from: https://www.10xgenomics.com/support/single-cell-multiome-atac-plus-gene-expression/documentation/steps/library-prep/chromium-next-gem-single-cell-multiome-atac-plus-gene-expression-reagent-kits-user-guide (2021)
- MacParland, S. A., et al. Single cell RNA sequencing of human liver reveals distinct intrahepatic macrophage populations. Nature Communications. 9 (1), 4383 (2018).
- Martinez-Jimenez, C. P., Kyrmizi, I., Cardot, P., Gonzalez, F. J., Talianidis, I. Hepatocyte nuclear factor 4alpha coordinates a transcription factor network regulating hepatic fatty acid metabolism. Molecular and Cell Biology. 30 (3), 565-577 (2010).
- Schmidt, D., et al. Five-vertebrate ChIP-seq reveals the evolutionary dynamics of transcription factor binding. Science. 328 (5981), 1036-1040 (2010).
- Hao, Y., et al. Integrated analysis of multimodal single-cell data. Cell. 184 (13), 3573-3587 (2021).
- Stuart, T., Srivastava, A., Madad, S., Lareau, C. A., Satija, R. Single-cell chromatin state analysis with Signac. Nature Methods. 18 (11), 1333-1341 (2021).
- Sampaziotis, F., et al. Cholangiocyte organoids can repair bile ducts after transplantation in the human liver. Science. 371 (6531), 839-846 (2021).
- Gomez-Lechon, M. J., Donato, M. T., Castell, J. V., Jover, R. Human hepatocytes as a tool for studying toxicity and drug metabolism. Current Drug Metabolism. 4 (4), 292-312 (2003).
- Gomez-Lechon, M. J., Donato, M. T., Castell, J. V., Jover, R. Human hepatocytes in primary culture: The choice to investigate drug metabolism in man. Current Drug Metabolism. 5 (5), 443-462 (2004).
- vanden Brink, S. C., et al. Single-cell sequencing reveals dissociation-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Denisenko, E., et al. Systematic assessment of tissue dissociation and storage biases in single-cell and single-nucleus RNA-seq workflows. Genome Biology. 21 (1), 130 (2020).
- Nault, R., Fader, K. A., Bhattacharya, S., Zacharewski, T. R. Single-nuclei RNA sequencing assessment of the hepatic effects of 2,3,7,8-tetrachlorodibenzo-p-dioxin. Cellular and Molecular Gastroenterology and Hepatology. 11 (1), 147-159 (2021).
- Krishnaswami, S. R., et al. Using single nuclei for RNA-seq to capture the transcriptome of postmortem neurons. Nature Protocols. 11 (3), 499-524 (2016).
- Duncan, A. W., et al. Aneuploidy as a mechanism for stress-induced liver adaptation. Journal of Clinical Investigation. 122 (9), 3307-3315 (2012).
- Kreutz, C., et al. Hepatocyte ploidy is a diversity factor for liver homeostasis. Frontiers in Physiology. 8, 862 (2017).
- Hunt, N. J., Kang, S. W. S., Lockwood, G. P., Le Couteur, D. G., Cogger, V. C. Hallmarks of aging in the liver. Computational and Structural Biotechnology Journal. 17, 1151-1161 (2019).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. 69 (2), 355-364 (2019).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Schwartz-Arad, D., Zajicek, G., Bartfeld, E. The streaming liver IV: DNA content of the hepatocyte increases with its age. Liver. 9 (2), 93-99 (1989).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv. B, Cell Pathology Including Molecular Pathology. 64 (6), 387-393 (1993).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Granja, J. M., et al. ArchR is a scalable software package for integrative single-cell chromatin accessibility analysis. Nature Genetics. 53 (3), 403-411 (2021).
- Bredikhin, D., Kats, I., Stegle, O. MUON: Multimodal omics analysis framework. Genome Biology. 23 (1), 42 (2022).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nature Methods. 19 (2), 179-186 (2022).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados