Method Article
Isolamento de núcleos de tecido hepático congelado por flash para multiômica unicelular
Neste Artigo
Resumo
Aqui, apresentamos um protocolo para isolar núcleos de tecidos hepáticos arquivados congelados por flash para RNA-seq, ATAC-seq de núcleo único e multiômica articular (RNA-seq e ATAC-seq).
Resumo
O fígado é um tecido complexo e heterogêneo responsável por realizar muitas funções fisiológicas críticas, como a manutenção da homeostase energética e o metabolismo dos xenobióticos, entre outras. Essas tarefas são realizadas através de uma coordenação estreita entre as células parenquimatosas hepáticas e não parenquimatosas. Além disso, várias atividades metabólicas estão confinadas a áreas específicas do lóbulo hepático - um fenômeno chamado zonação hepática. Avanços recentes em tecnologias de sequenciamento de célula única capacitaram os pesquisadores a investigar a heterogeneidade tecidual em uma resolução de célula única. Em muitos tecidos complexos, incluindo o fígado, protocolos rígidos de dissociação enzimática e/ou mecânica podem afetar negativamente a viabilidade ou a qualidade das suspensões unicelulares necessárias para caracterizar de forma abrangente esse órgão na saúde e na doença.
Este trabalho descreve um protocolo robusto e reprodutível para isolar núcleos de tecidos hepáticos congelados e arquivados. Este método produz núcleos de alta qualidade que são compatíveis com abordagens ômicas unicelulares a jusante, incluindo RNA-seq de núcleo único, ensaio para cromatina acessível à transposase com sequenciamento de alto rendimento (ATAC-seq), bem como ômica multimodal (RNA-seq articular e ATAC-seq). Este método tem sido usado com sucesso para o isolamento de núcleos de amostras de fígado congelado de humanos humanos, ratos e não humanos saudáveis e doentes. Essa abordagem permite o isolamento imparcial de todos os principais tipos de células no fígado e, portanto, oferece uma metodologia robusta para estudar o fígado na resolução de célula única.
Introdução
A genômica unicelular está rapidamente se tornando uma metodologia essencial para estudar a função hepática e avaliar o impacto da heterogeneidade celular nas condições de saúde e doença1. O rápido desenvolvimento de "multiômicas" para a medição simultânea de diferentes camadas de informação e a expansão paralela de pipelines computacionais robustos estão abrindo caminho para a descoberta de tipos e subtipos celulares anteriormente desconhecidos no fígado normal e doente2.
A possibilidade de explorar biobancos e amostras congeladas arquivadas aumentou significativamente as oportunidades de revisitar e descobrir o papel das células não parenquimatosas 3,4,5 e investigar o papel dos hepatócitos poliploides durante o envelhecimento e em doenças crônicas 6,7,8,9 . Portanto, este trabalho descreve um protocolo de isolamento de núcleo único robusto e reprodutível para fígados arquivados congelados por flash (FF) que é compatível com o sequenciamento de RNA de núcleo único a jusante e o sequenciamento ATAC, bem como com a ômica multimodal (RNA-seq articular e ATAC-seq) (Figura 1).
Este fluxo de trabalho permite a investigação do transcriptoma e a acessibilidade da cromatina de todos os tipos de células no fígado, independentemente do tamanho ou fragilidade celular, em protocolos de dissociação enzimática. Pode ser realizado com pequenas seções de tecido (15-30 mg ou 5-10 mm3) de amostras humanas preciosas ou camundongos transgênicos. A determinação da alta pureza do isolamento dos núcleos inclui a quantificação e a mensuração do tamanho nuclear, o que pode se correlacionar com o aumento do tamanho e da senescência celular 10,11, e essa pureza é relevante tanto para a análise da ploidia dos hepatócitos 12 quanto dos mecanismos transcricionais dependentes do tamanho celular11,13,14,15 . Além disso, núcleos isolados de fígados congelados retêm informações valiosas sobre a zonação hepática. O fluxo de trabalho e a coleta de tecidos permitem a validação de dados genômicos unicelulares ou análises complementares adicionais, como imuno-histoquímica ou transcriptômica espacial do mesmo tecido e do mesmo indivíduo. Portanto, essa abordagem pode ser aplicada a várias condições de doença hepática e organismos modelo de forma sistemática e confiável.
Protocolo
Todas as experiências com animais foram realizadas em conformidade com a Legislação Alemã de Bem-Estar Animal e os regulamentos do Governo da Alta Baviera. O alojamento de animais foi aprovado de acordo com o § 11 da Lei Alemã de Bem-Estar Animal e realizado de acordo com a Diretiva 2010/63/UE.
1. Preparação de tecidos
- Sacrificar um rato C57BL/6J macho de 3 meses a 22 meses de idade por luxação cervical. Coloque o animal em uma tábua de dissecação, prenda as extremidades com alfinetes e desinfete o abdômen com etanol a 70%.
- Realizar necropsia conforme preconizado por Treuting et al.16.
- Abra o abdômen até a caixa torácica, visualize o fígado e use fórceps para remover cuidadosamente o fígado sem perfurar os lóbulos.
- Extraia o fígado intacto segurando o diafragma com fórceps e removendo o tecido de conexão com uma tesoura.
- Lave o órgão em solução salina tamponada com fosfato frio (PBS), seque-o em uma toalha de papel limpa e corte os lóbulos do fígado em vários pedaços para diferentes fins: FF para isolamento de núcleo único e multiômico; fixo de paraformaldeído (10% PFA) para incorporação de parafina; e/ou incorporado em composto de temperatura de corte ideal (OCT) para análises histológicas adicionais (Figura 1A).
- Aliquot os pedaços de fígado em criovios ou tubos de tampa de rosca de 5 mL e congele imediatamente em nitrogênio líquido. Armazenar as amostras de fígado congelado a -80 °C para experimentos multiômicos de núcleo único a jusante (Figura 1B, C).
NOTA: O tecido criopreservado pode ser armazenado com segurança a -80 °C por vários anos e deve sempre ser transportado em gelo seco a -80 °C antes do uso.
2. Isolamento dos núcleos
- Limpeza e preparação de bancadas dos amortecedores e consumíveis
- Limpe as bancadas e pipetas de trabalho com etanol a 70% e solução de descontaminação de RNase ou use bancadas e materiais dedicados sem RNase.
- Pré-arrefeça a caçamba oscilante e as centrífugas de ângulo fixo, tubos de 1,5 mL/2 mL e placas de vários poços a 4 °C, conforme detalhado abaixo.
- Pré-resfrie as pinças descartáveis e de uso único sem RNase usadas para manuseio de tecidos, um bisturi estéril descartável e uma placa de Petri em gelo seco (-60 °C).
- Pré-arrefecer o homogeneizador de vidro Dounce e pilões sobre gelo (4 °C). Coloque cada pilão (A e B) em um tubo de 5 mL para evitar o contato direto com o gelo e a potencial contaminação por RNase.
- Preparar uma solução diluente de meio iodixanol (IDM) (Tabela 1A) e usá-la para fazer diluições de 50% e 29% do meio gradiente de densidade de estoque de iodixanol a 60% (Tabela 1B e Tabela 1C, respectivamente).
- Pré-arrefecer todos os tubos. Para cada amostra que será processada, prepare os seguintes tubos:
- Preparar três tubos de ligação de baixo ADN de 1,5 ml (um para o homogeneizado de tecido filtrado, um segundo contendo 250 μL de uma diluição a 50% de solução de iodixanol e um terceiro para a suspensão de núcleos limpos).
- Preparar um tubo de fundo redondo de 2 ml contendo 500 μL de diluição a 29% de solução de iodixanol para a separação do gradiente de densidade.
- Preparar um tubo cônico de 15 mL para o meio de isolamento dos núcleos-2 (NIM-2) e tampão de homogeneização (HB).
- Preparar o meio de isolamento dos núcleos-1 (NIM-1) (Tabela 1D) e utilizá-lo para fazer o NIM-2 (Tabela 1E) e, posteriormente, o HB (Tabela 1F). Adicione ambos os inibidores de RNAse imediatamente antes do uso, conforme descrito abaixo no protocolo.
- Prepare o buffer de armazenamento de núcleos (NSB) conforme descrito na Tabela 1G. Adicione o inibidor de RNAse recombinante imediatamente antes do uso. Adicione o inibidor de RNAse à base de proteínas ao NSB antes de usar para classificação FACS (opcional).
- Prepare um copo de 500 mL com água estéril para mergulhar os homogeneizadores e pilões Dounce após a homogeneização do tecido para a limpeza e manutenção ideais dos homogeneizadores.
- Homogeneização tecidual
- Corte um pedaço de tecido de 20-30 mg (ou 5 mm3) com um bisturi pré-resfriado dentro da placa de Petri em gelo seco. Em seguida, transfira imediatamente a placa de Petri para o gelo molhado (4 °C). Adicione 1 mL de HB e, usando o bisturi frio, pice o tecido o máximo possível para permitir que ele seja facilmente aspirado com uma ponta de orifício largo de 1 mL.
NOTA: Sempre use pontas de orifício largo para todas as transferências de tecidos/núcleos. Como alternativa, pontas de 1 mL podem ser cortadas com bisturi estéril em uma tampa plástica estéril para gerar orifícios largos (Figura 2A). - Coletar a suspensão tecidual e transferi-la para um homogeneizador de rejeição de vidro pré-resfriado de 2 mL (Figura 2B).
- Lave a placa de Petri com um adicional de 0,5-1 mL de HB e colete todos os pedaços de tecido restantes, mantendo tudo no gelo.
- Lenta e cuidadosamente faça cinco golpes com pilão solto A no gelo. Evite criar bolhas usando movimentos de torção do pilão ao puxá-lo para cima e para baixo. Certifique-se de que o pilão se move cuidadosamente de cima para baixo do homogeneizador a cada braçada.
- Posteriormente, execute 10-15 movimentos lentos com pilão apertado B no gelo. Evite criar bolhas.
NOTA: Recomenda-se inspecionar visualmente os núcleos sob o microscópio após 10 golpes com pilão B para verificar se mais derrames são necessários. Faça isso usando a coloração azul de tripano (proporção de 1:1 de tripano para amostra) e um hemocitômetro manual (por exemplo, misture 10 μL de azul de tripano com 10 μL de suspensão de núcleos e use 10 μL da mistura para exame ao microscópio; Figura 2C). - Filtre o homogeneizado através de um filtro de células de 50 μm enquanto o transfere para um tubo pré-refrigerado de 1,5 mL. Use mais de um filtro e/ou tubo para homogeneizados contendo uma grande quantidade de aglomerados de tecido conjuntivo.
- Enxaguar o homogeneizador e os filtros utilizados com um adicional de 0,5-1 ml de HB para recolher minuciosamente todo o homogeneizado tecidual. Prossiga para a centrifugação do gradiente de densidade.
- Corte um pedaço de tecido de 20-30 mg (ou 5 mm3) com um bisturi pré-resfriado dentro da placa de Petri em gelo seco. Em seguida, transfira imediatamente a placa de Petri para o gelo molhado (4 °C). Adicione 1 mL de HB e, usando o bisturi frio, pice o tecido o máximo possível para permitir que ele seja facilmente aspirado com uma ponta de orifício largo de 1 mL.
- Centrifugação por gradiente de densidade
- Centrifugar o homogeneizado filtrado numa centrífuga pré-refrigerada de ângulo fixo a 1.000 × g durante 8 min a 4 °C.
- Enquanto a amostra estiver girando, prepare um tubo de 1,5 mL contendo 250 μL de diluição de iodixanol a 50% e um tubo de 2 mL contendo 500 μL de diluição de iodixanol a 29%. Mantenha ambos os tubos no gelo.
- Após a centrifugação, aspirar o sobrenadante sem perturbar o pellet usando uma bomba de vácuo.
NOTA: O uso da pipetagem manual comprometerá a qualidade da suspensão final dos núcleos. - Adicione 250 μL de HB ao pellet usando a ponta da pipeta de orifício largo de 1 mL e ressuspenda muito lentamente.
- Transfira 250 μL da suspensão de núcleos para um tubo pré-resfriado de 1,5 mL contendo 250 μL de diluição de iodixanol a 50% e misture suavemente, mas completamente, para gerar uma suspensão de iodixanol / núcleos a 25%.
- Transferir 500 μL da suspensão de iodixanol/núcleos a 25% para um tubo de 2 ml pré-refrigerado contendo 500 μL de diluição de iodixanol a 29%.
NOTA: Os 500 μL da suspensão de iodixanol/núcleos a 25% devem ser depositados suavemente sobre os 500 μL de solução de iodixanol a 29%, de modo a que as misturas de iodixanol a 25%/29% apresentem uma separação clara de fases. Use o lado da parede do tubo com a ponta da pipeta posicionada em um ângulo de 45° para criar essa interface de gradiente. A partir de então, o tubo deve ser manuseado suavemente para não perturbar esse gradiente. - Centrifugar o tubo em uma centrífuga de balde oscilante pré-refrigerada a 12.500 g por 20 minutos com o freio ajustado para OFF.
- Imediatamente antes de a etapa de centrifugação ser concluída, adicione os inibidores de RNAse ao tampão NSB ao prosseguir para as tubulações scRNA-seq (ver Tabela 1G).
- Após a centrifugação, aspirar o sobrenadante sem perturbar o pellet usando uma bomba de vácuo.
NOTA: O uso da pipetagem manual comprometerá a qualidade da suspensão final dos núcleos. - Usando a ponta da pipeta de orifício largo de 1 mL, ressuspenda suavemente o pellet em 100-300 μL de NSB e transfira a suspensão dos núcleos para um tubo limpo e pré-resfriado de 1,5 mL.
- Conte os núcleos usando solução azul de tripano (proporção de 1:1 de tripano para amostra) e um hemocitômetro manual (por exemplo, misture 10 μL de azul de tripano com 10 μL de suspensão de núcleos e use 10 μL da mistura para contagem; Figura 2D).
- Utilizar a suspensão de núcleos obtida imediatamente para o ensaio genómico de núcleo único.
NOTA: A suspensão de núcleos em NSB pode ser armazenada refrigerada a 4 °C por até 1 semana para análise adicional por citometria de fluxo e/ou de fluxo de imagem, mas não para snRNA-seq ou snATAC-seq.
3. Classificação de núcleos para perfil de ploidia de hepatócitos ou abordagens de sequenciamento bem baseadas
- Para a classificação celular baseada em citometria de fluxo, filtre a suspensão de núcleos através de um filtro de 50 μm em um tubo FACS pré-resfriado de 5 mL.
- Use um classificador de citometria de fluxo equipado com um bocal de 100 μm. Carregue o tubo FACS no classificador e visualize a amostra.
- Configure a estratégia de bloqueio para a classificação de núcleos, começando com uma porta de dispersão plotando a área de dispersão para a frente versus a área de dispersão lateral (FSC-A/SSC-A), seguida por Hoechst-Height versus Hoechst-Area (porta de núcleos) e, em seguida, Hoechst-Width versus Hoechst-Area (porta singlet). Visualize o perfil de ploidia dos núcleos no histograma Hoechst-Area.
NOTA: Para uma melhor resolução de pico, visualize o canal Hoechst (450/50) na escala linear (Figura 3A). - Para a classificação em placas de 96/384 poços, defina o atraso da gotícula e otimize o alinhamento da placa usando o método colorimétrico com substrato benzidina peroxidase de rábano (TMB-HRP), conforme descrito anteriormente6.
- Regule o arrefecimento da amostra e o suporte da placa para arrefecer a 4 °C, com a rotação da amostra ligada a 300 rpm.
- Classifique os núcleos únicos em uma concentração de amostra de ~1 x 105 núcleos/mL e com a taxa de fluxo em 200-500 eventos/s.
4. Inspeção visual e quantificação de parâmetros nucleares por citometria de imagem (opcional)
- Carregar um tubo de 0,5 ml contendo 50 μL da suspensão de núcleos a uma concentração de 2 × 107 núcleos/ml.
- Configure uma porta de todos os eventos com base na proporção em relação ao canal de fluorescência de Hoechst para visualizar o perfil de ploidia dos núcleos (Figura 3B).
- Adquira a amostra a uma ampliação de 40x utilizando o campo brilhante (BF) e o canal de fluorescência Hoechst.
- Inspecionar e quantificar núcleos 2n e 4n utilizando a medição de campo brilhante e a intensidade de fluorescência de Hoechst com software de citometria de imagem (Figura 3C).
5. Construção e sequenciamento de bibliotecas de RNA-seq, ATAC-seq ou multioma de núcleo único
- Para abordagens snRNA-seq baseadas em gotículas, carregue a suspensão de núcleos purificados diretamente no dispositivo microfluídico para particionamento paralelo automatizado e código de barras molecular17.
- Após a conclusão da corrida microfluídica no dispositivo de particionamento de célula única, colete os núcleos encapsulados em esferas de gel, incube e limpe conforme descrito anteriormente nas diretrizes do fabricante17.
- Realizar 11 ciclos de reação em cadeia da polimerase (PCR) para a pré-amplificação do cDNA usando o seguinte programa: 3 min a 98 °C, (15 s a 98 °C, 20 s a 63 °C e 1 min a 72 °C) x 11, 1 min a 72 °C e manter a 4 °C. Continue com um reparo final e uma ligadura de retailing e adaptador, conforme indicado pelo fabricante17. Para a construção final subsequente da biblioteca de expressão gênica, realizar 10 ciclos de PCR usando o seguinte programa: 45 s a 98 °C, (20 s a 98 °C, 30 s a 54 °C, 20 s a 72 °C) x 10, 1 min a 72 °C e segure a 4 °C.
- Sequencie as bibliotecas obtidas a uma profundidade de leitura de ~20.000-50.000 leituras médias por núcleo.
- Para o sequenciamento baseado em gotículas multiômicas articulares (RNA + ATAC), incubar os núcleos hepáticos em tampão de lise por 5 min e, em seguida, marcá-los por 1 h, conforme descrito anteriormente18.
- Carregue os núcleos marcados diretamente em um dispositivo microfluídico para particionamento paralelo automatizado e código de barras molecular.
- Após a conclusão da corrida microfluídica, colete os núcleos encapsulados em grânulos de gel, incube e limpe conforme descrito pelo fabricante18.
- Realizar seis ciclos de PCR para a etapa de pré-amplificação do cDNA usando o seguinte programa: 5 min a 72 °C, 3 min a 98 °C, (20 s a 98 °C, 30 s a 63 °C, 1 min a 72 °C) x 6, 1 min a 72 °C e manter a 4 °C.
- Colher 35 μL da amostra pré-amplificada e efectuar uma amplificação do cDNA da seguinte forma: 3 min a 98 °C, (15 s a 98 °C, 20 s a 63 °C, 1 min a 72 °C) x 6, 1 min a 72 °C e manter a 4 °C. Continue com o reparo final e a etapa de cauda A e ligadura do adaptador, conforme indicado pelo fabricante18 . Para a PCR de indexação final da amostra subsequente, efectuar 15 ciclos de PCR da seguinte forma: 45 s a 98 °C, (20 s a 98 °C, 30 s a 54 °C, 20 s a 72 °C) x 15, 1 min a 72 °C e manter a 4 °C.
- Para a construção de bibliotecas ATAC, use 40 μL e amplie por seis ciclos de PCR para indexação de amostras usando o seguinte programa: 45 s a 98 °C, (20 s a 98 °C, 30 s a 67 °C, 20 s a 72 °C) x 6, 1 min a 72 °C e segure a 4 °C.
- Sequenciar as bibliotecas de expressão gênica multioma obtidas a uma profundidade mínima de leitura de 20.000 pares de leitura por núcleo e as bibliotecas ATAC multioma a uma profundidade mínima de leitura de 25.000 pares de leitura por célula, conforme recomendado pelo fabricante18.
Resultados
Esse fluxo de trabalho para isolamento de núcleo único de amostras de fígado congeladas é adaptado para multiômica de núcleo único e depende de três etapas principais, que podem ser resumidas como i) coleta de amostras para a análise paralela da heterogeneidade celular e arquitetura tecidual, ii) suspensão de núcleo único e iii) multiômica de núcleo único (Figura 1 ). Os fígados extraídos são dissecados de camundongos eutanasiados e cortados em pedaços para inspeção histológica para incorporação de parafina, criossecção ou ambos. Outras peças cortadas são imediatamente congeladas em nitrogênio líquido para isolamento a jusante, de núcleo único para análises multiômicas. Este sistema de coleta de tecidos permite que o usuário valide ainda mais os dados de ômica de núcleo único em seções de tecido do mesmo indivíduo, complementando assim o conjunto de dados com transcriptômica espacial ou com análises imuno-histoquímicas, se necessário.
O exame microscópico dos núcleos extraídos de fígados congelados com o método aqui descrito mostra que a etapa de centrifugação do gradiente de densidade facilita sobremaneira a remoção de detritos celulares e teciduais indesejados (Figura 2C,D). Além disso, essa metodologia preserva todos os níveis de ploidia, que podem ser validados e quantificados por análises citométricas (Figura 3).
Para validar ainda mais o desempenho deste protocolo, realizamos snRNA-seq baseado em gotículas em núcleos não classificados e classificados por FACS e analisamos os dados seguindo o pipeline de Seurat. Em resumo, os núcleos únicos extraídos foram preparados para snRNA-seq à base de gotículas, conforme descrito anteriormente19. Para o snRNA-seq de núcleos 2n e 4n ou níveis superiores de ploidia, foram utilizados 11 ciclos para a etapa de amplificação do cDNA e 10 ciclos para a construção da biblioteca final de expressão gênica. As bibliotecas resultantes foram sequenciadas a uma profundidade de leitura de ~25.000-39.000 leituras médias por núcleo. As leituras de núcleos únicos obtidas foram mapeadas para o genoma de camundongos GRCm39/mm39. Ao executar o pipeline de pré-processamento, o comando - -include-intron foi adicionado para verificar a inclusão e quantificação do RNA mensageiro não emendado (mRNA) presente nos núcleos. O algoritmo EmptyDrops incorporado no alinhador filtrou e removeu as gotículas vazias.
O pacote R Seurat (versão 4.1.1) foi usado para calcular métricas de controle de qualidade (QC) usando a matriz de contagem de identificador molecular único (UMI) como saída pelo pipeline de análise snRNA-seq. Contagens com menos de 100 características (genes) e menos de 10 células foram removidas. Os núcleos foram filtrados de acordo com os limiares de QC identificados: número mínimo de genes = 200 e número máximo de genes = 8.000, fração mitocondrial <1% e fração ribossomal <2%. Os 3.000 principais genes altamente variáveis (HVGs) foram usados para a análise de componentes principais (PCA), conforme implementado no Seurat. O agrupamento baseado em gráficos foi realizado após a inserção das 15 principais dimensões de PC resultantes da análise da ACP. Para agrupar as células, aplicou-se a técnica de otimização de modularidade (algoritmo de Louvain) com um parâmetro de resolução definido como 0,5. Para visualizar e explorar esse conjunto de dados, executamos a redução dimensional não linear, ou seja, Uniform Manifold Approximation and Projection (UMAP). A identidade de cada cluster foi atribuída com base no conhecimento prévio dos genes marcadores 6,20,21.
A Figura 4A mostra métricas de alta qualidade a partir dos dados obtidos por meio desse método de extração de núcleos. O UMAP representa o número de contagens em todos os núcleos e os principais tipos de células que podem ser identificados com confiança apenas com o transcriptoma nuclear e sequenciamento relativamente superficial (~25.000-40.000 leituras médias por célula) (Figura 4B). Essa abordagem permite a investigação de fatores de transcrição específicos do fígado, como Hnf4a, Mlxipl e Ppara, bem como genes-alvo a jusante envolvidos no metabolismo de xenobióticos (ou seja, Cyp2e1 e Cyp2f2) (Figura 4C). Vale ressaltar que os núcleos extraídos mantiveram informações críticas sobre a zonação hepática, como mostra o padrão complementar de genes característicos, como o Cyp2e1 pericentral e o Cyp2f2 periportal (Figura 4C).
Avaliamos ainda a compatibilidade dos núcleos não classificados extraídos usando esse método com ensaios multiômicos mais recentes para o perfil simultâneo da paisagem epigenômica (ATAC) e da expressão gênica (RNA) nos mesmos núcleos únicos18. Otimizamos o tempo de incubação da lise dos núcleos para 5 min antes da transposição. As bibliotecas de sequenciamento foram construídas por meio da execução de seis ciclos de PCR para a pré-amplificação do cDNA. Da amostra pré-amplificada, 35 μL foram retirados e posteriormente amplificados por 15 ciclos de PCR para indexação da amostra para construção da biblioteca de expressão gênica. Um eletroferograma representativo do traço de cDNA é mostrado na Figura 5A (acima). Para a construção das bibliotecas ATAC, foram utilizados 40 μL de amostra pré-amplificada e executados por mais seis ciclos de PCR para indexação da amostra, com o eletroferograma representativo do traço de DNA mostrado na Figura 5A (abaixo). A biblioteca de expressão gênica (RNA) resultante foi sequenciada a uma profundidade de 44.600 leituras por núcleo e a biblioteca ATAC a uma profundidade de 43.500 leituras por núcleo (Figura 5B).
Semelhante ao descrito acima, o protocolo de sequenciamento baseado em gotículas de modalidade única, mapeamento de leitura, alinhamento, remoção de gotas vazias e contagem de fragmentos foram realizados seguindo diretrizes padrão, conforme descrito anteriormente, usando o genoma de referência GRCm39/mm3918. Utilizando os pacotes Seurat e Signac22, realizamos uma análise de "vizinho mais próximo ponderado" (WNN) para múltiplas medidas de ambas as modalidades (RNA + ATAC) (Figura 5C), o que nos permitiu identificar e anotar os tipos de células hepáticas maiores e menores, sem vieses proeminentes devido ao tamanho da célula ou fragilidade nuclear (Figura 5D). O pipeline, conforme publicado pelo laboratório Satija22,23, inclui etapas padrão de QC - pré-processamento e redução dimensional - feitas em ambos os ensaios de forma independente. Para se ter uma boa representação da combinação ponderada das modalidades RNA-seq e ATAC-seq, o gráfico WNN foi plotado e utilizado para visualização, agrupamento e anotação do UMAP com base em genes marcadores previamente identificados 6,20,21. Semelhante aos ensaios usando a modalidade única, também detectamos reguladores transcricionais a montante (Hnf4a, Ppara, Mlxipl) e genes característicos da zonação hepática (Hamp, Cyp2e1 e Cyp2f2) (Figura 5E).
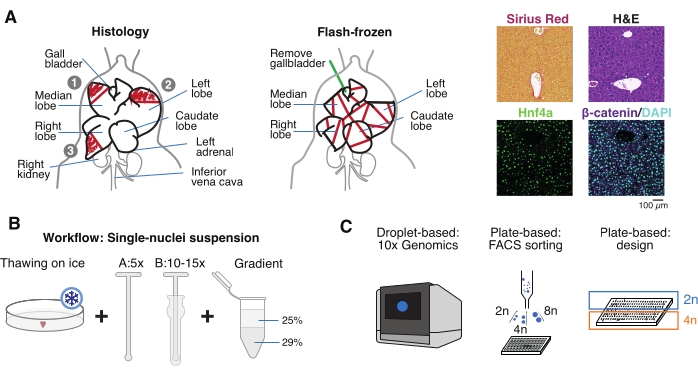
Figura 1: Visão geral experimental, fluxo de trabalho e aplicações genômicas de célula única. (A) Representação ilustrativa da amostragem de tecidos para histologia (à esquerda, três seções são selecionadas para incorporação de parafina e/ou criosecção), coleta de tecido congelado por flash para genômica de célula única (meio) e análises representativas de imunohistoquímica e imunofluorescência (direita); barra de escala = 100 μm. (B) Etapas críticas para suspensões de núcleo único de alta qualidade. (C) As suspensões de núcleos podem ser carregadas em um chip de cromo 10x ou usadas para classificação FACS e abordagens baseadas em placas. Abreviaturas: H&E = hematoxilina e eosina; DAPI = 4',6-diamidino-2-fenilindol; FACS = classificação celular ativada por fluorescência. Por favor, clique aqui para ver uma versão maior desta figura.

Figura 2: Dissecção hepática e homogeneização do tecido de vidro de rejeição. (A) Fígado murino representativo de um rato C57BL6/J de 3 meses de idade (à esquerda); a secção hepática utilizada para os isolamentos de núcleo único antes (meio) e depois de picar o tecido com o bisturi (à direita). Barras de escala = 1 cm. (B) Imagens ilustrativas de 2 mL de homogeneização Dounce-glass antes de golpes com pilão "solto" A (esquerda) e após pilão "apertado" B (direita). Monitorização da homogeneização tecidual utilizando hemocitômetro (C) antes da centrifugação do gradiente e (D) após a centrifugação do gradiente. Por favor, clique aqui para ver uma versão maior desta figura.
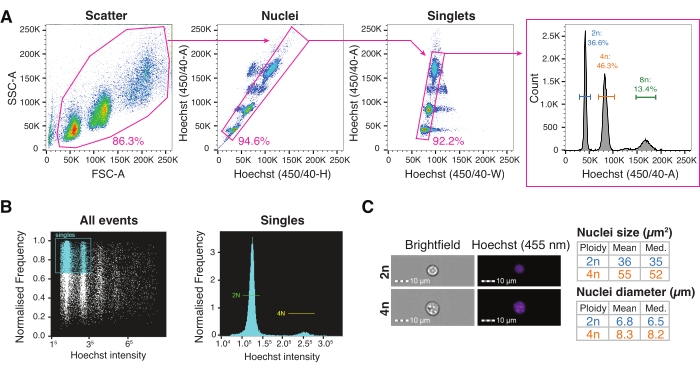
Figura 3: Classificação celular ativada por fluorescência e citometria de fluxo de imagem para caracterização de núcleos de alto rendimento . (A) Estratégia de gating para classificação FACS de núcleo único em uma placa para a interrogação de diferentes níveis de ploidia. Porta de dispersão com base na área de dispersão para a frente versus a área de dispersão lateral definida para excluir detritos; porta de núcleos com base em Hoechst-Height versus Hoechst-Area incorporando múltiplas populações de núcleos; porta singlets baseada no conjunto Hoechst-Width versus Hoechst-A para discriminação dupla; o histograma Hoechst-A permite a visualização do perfil ploidial dos núcleos. (B) Quantificação representativa da citometria de imagem de todos os eventos (esquerda) e única (direita), mostrando núcleos diploide e tetraploide. (C) Imagens de Brightfield e Hoechst de núcleos 2n e 4n e sua quantificação usando citometria de imagem. Abreviaturas: FACS = classificação celular ativada por fluorescência; FSC-A = área de dispersão para a frente; SSC-A = área de dispersão lateral; Hoechst-H = Hoechst-Altura; Hoechst-A = Hoechst-Área; Hoechst-W = Hoechst-Largura. Por favor, clique aqui para ver uma versão maior desta figura.
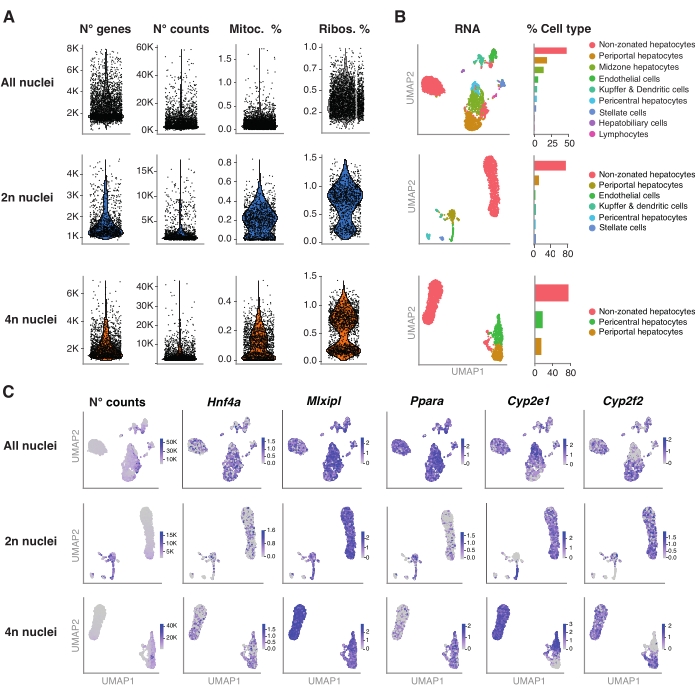
Figura 4: Caracterização profunda da ploidia de hepatócitos com snRNA-seq . (A) Gráficos de violino mostrando o número de genes, contagens, porcentagem de genes mitocondriais e porcentagem de genes ribossômicos detectados. (B) UMAP demonstrando os tipos celulares detectados usando snRNA-seq (esquerda), com a quantificação expressa em porcentagem de núcleos (direita). (C) O UMAP ilustra o número de contagens e indica a expressão de genes específicos de hepatócitos. Todos os núcleos (linha superior), 2n núcleos (linha do meio) e 4n núcleos (linha inferior). Abreviaturas: snRNA-seq = RNA-seq de núcleo único; UMAP = Aproximação e Projeção Uniforme da Variedade; mitoc. = genes mitocondriais; Ribos. = genes ribossomais. Por favor, clique aqui para ver uma versão maior desta figura.
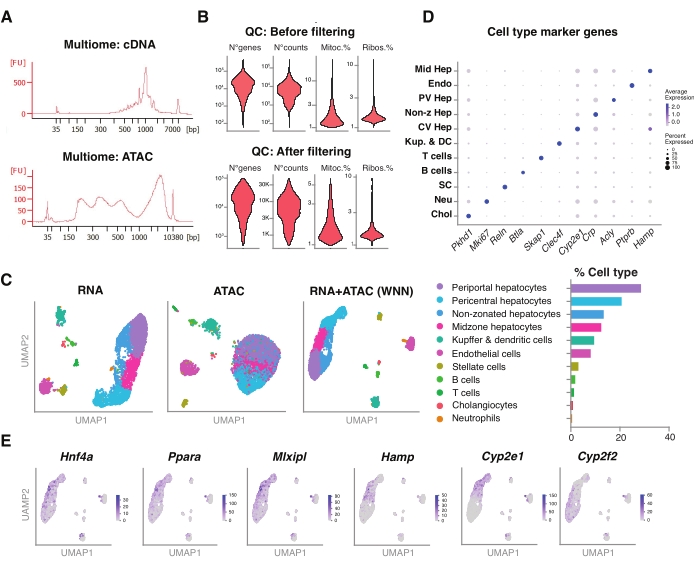
Figura 5: Controle de qualidade e análise de multiômica (RNA-seq articular e ATAC-seq) de fígados jovens congelados e arquivados. (A) Traços eletroforéticos automáticos representativos obtidos após o pipeline multiômico mostrando o produto de peso molecular após a síntese de cDNA e ATAC. (B) Gráfico para violino demonstrando o número de contagens para ATAC-seq, RNA-seq, a porcentagem de genes mitocondriais e a porcentagem de genes ribossômicos antes e após a filtragem. (C) O UMAP mostra a expressão gênica das modalidades RNA-seq (esquerda), ATAC-seq (meio) e articulares-RNA-seq e ATAC-seq (direita). (D) Diferentes tipos de células são anotados em cores diferentes, com a expressão de genes específicos de hepatócitos indicados. (E) Gráfico de características mostrando a expressão específica do cluster dos genes indicados nos tipos de células indicados. Abreviaturas: ATAC-seq = ensaio para cromatina acessível à transposase com sequenciamento de alto rendimento; Hep média = hepatócitos zonais médios; Endo = células endoteliais; Phepatite PV = hepatócitos periportais; Hep não-z = hepatócitos não zonados; Hep CV = hepatócitos pericentrais; Kup & DC = Kupffer & células dendríticas; SC = células estreladas, Neu = neutrófilos; Cola = colangiócitos; mitoc. = genes mitocondriais; Ribos. = genes ribossomais. Por favor, clique aqui para ver uma versão maior desta figura.
| Reagente | Estoque | 10 mL | 15 mL | 50 mL | ||
| (A ) Iodixanol Médio (IDM) | ||||||
| 250 mM Sacarose | 1 milhão | 12,5 mL | ||||
| 150 mM KCl | 2 milhões | 3,75 mL | ||||
| 30 mM MgCl2 | 1 milhão | 1,5 mL | ||||
| Tampão Tris de 60 mM pH 8,0 | 1 milhão | 3 mL | ||||
| Água ultrapura livre de RNase | 29,25 mL | |||||
| B) 50 % IDM | ||||||
| Iodixanol | 60% | 12,5 mL | ||||
| IDM | 2,5 mL | |||||
| (C) 29% IDM | ||||||
| Iodixanol | 60% | 7,25 mL | ||||
| IDM | 7,75 mL | |||||
| (D ) Núcleo de Isolamento Médio-1 (NIM-1) | ||||||
| 250 mM Sacarose | 1 milhão | 12,5 mL | ||||
| 25 mM KCl | 2 milhões | 0,625 mL | ||||
| 5 mM MgCl2 | 1 milhão | 0,25 ml | ||||
| Tampão Tris de 10 mM pH 8,0 | 1 milhão | 0,5 ml | ||||
| Água ultrapura sem Rnase | 36,125 mL | |||||
| (E ) Núcleo de Isolamento Médio-2 (NIM-2) | ||||||
| Buffer NIM-1 | 9,99 mL | |||||
| Dithiothreitol (TDT) | 1 mM | 0,01 ml | ||||
| Comprimido inibidor de protease (livre de EDTA) | 1 | 1 Tablet | ||||
| (F ) Buffer de homogeneização (HB) | ||||||
| Buffer NIM-2 | 9,697 mL | |||||
| Inibidor da RNase Recombinante | 40 U/μL | 0,1 ml | ||||
| Inibidor de RNAse à base de proteínas (SUPERase•IN) | 20 U/μL | 0,1 ml | ||||
| 0,1% Tritão-X | 10% | 0,1 ml | ||||
| 3 μg/ml Hoechst 33342 | 10 mg/mL | 0,003 mL | ||||
| (G ) Buffer de armazenamento de núcleos (NSB) | ||||||
| 166.5 mM Sacarose | 1 milhão | 1,665 mL | ||||
| 5 mM MgCl2 | 1 milhão | 0,05 ml | ||||
| 10 mM Tris pH 8,0 | 1 milhão | 0,1 ml | ||||
| Inibidor da RNase Recombinante | 40 U/μL | 0,1 ml | ||||
| Inibidor da RNase à base de proteínas (SUPERase•IN) * | 20 U/μL | 0,1 ml | ||||
| Água ultrapura livre de RNase | 8,085 mL | |||||
| * Opcional (apenas para classificação FACS) | ||||||
Tabela 1: Receitas de solução. (A) Preparação do meio iodixanol (IDM); (B) diluição a 50% da solução de iodixanol; (C) Diluição a 29% da solução de iodixanol. (D) Preparação do meio de isolamento de núcleos-1 (NIM-1). (E) Preparação do meio de isolamento de núcleos-2 (NIM-2). (F) Preparação do tampão de homogeneização (HB). (G) Preparação do tampão de armazenamento de núcleos (NSB).
Discussão
A dissecação da composição celular do fígado por RNA-seq de célula única ou núcleo único proporciona uma compreensão mais profunda do desenvolvimento e progressão da doença hepática 3,4,5,24. O isolamento unicelular do fígado é demorado e requer protocolos que envolvam dissociação mecânica ou enzimática severa25,26,27. É amplamente aceito que todo tecido requer uma avaliação sistemática para determinar o protocolo de dissociação tecidual ideal, bem como um método de armazenamento adequado para capturar tipos celulares frágeis ou núcleos28. Dependendo da disponibilidade tecidual, doença de interesse, estágio de desenvolvimento ou organismo modelo, a preparação de uma suspensão de núcleo único para processamento a jusante pode ser uma metodologia mais adequada do que usar suspensões de célula única. É importante ressaltar que, no fígado, scRNA-seq e snRNA-seq têm mostrado alta correlação entre mRNA nuclear e citoplasmático, sugerindo que ambas as abordagens apresentam informações complementares 2,3,4,6,29.
Este artigo fornece um isolamento padronizado, robusto e reprodutível de núcleo único de amostras de fígado congeladas e arquivadas de camundongos e outras espécies, incluindo humanos e macacos. Esse método pode ser usado para camundongos do tipo selvagem alimentados com ração e dieta rica em gordura (HFD) e para modelos de fibrose hepática em camundongos usando abordagens genômicas de núcleo único à base de placas e gotículas6. Esse método baseia-se no protocolo descrito originalmente para o tecido cerebral por Krishnaswami et al.30 com modificações adicionais adaptadas para o fígado congelado por flash. A homogeneização ideal libera a maioria dos núcleos do tecido sem afetar negativamente a integridade da membrana nuclear. O exagero, no entanto, pode danificar os núcleos frágeis e diminuir sua qualidade geral. Fígados jovens e / ou gordurosos geralmente requerem apenas 5 derrames com pilão A e 10 derrames com pilão B, enquanto fígados velhos e / ou fibróticos podem exigir 15 derrames com pilão B, mas não mais. Não é recomendado, portanto, realizar mais traços além dos números aqui indicados. O excesso de douncing pode afetar negativamente a qualidade da suspensão de núcleo único e aumentar a quantidade de RNA ambiente. Posteriormente, isso pode levar à necessidade de executar etapas adicionais de filtragem computacional durante as análises de dados a jusante.
O protocolo aqui apresentado é versátil e pode ser ajustado a diferentes condições hepáticas em camundongos jovens (3 meses) e idosos (24 meses). Como descobrimos que uma seção maior do fígado é necessária para tecidos velhos, HFD e fibróticos, o tamanho do tecido disponível para processamento pode representar uma limitação para alguns usuários com quantidades menores de material biológico inicial. No entanto, a purificação por gradiente é altamente recomendada para o processamento imediato da amostra com ensaios genômicos baseados em gotículas. Se os núcleos precisarem ser classificados em FACS em placas de 96/384 poços para ensaios bem fundamentados, a purificação do gradiente pode ser omitida. Encorajamos os usuários ainda a realizar a purificação do gradiente se houver amostra de tecido suficiente para obter a concentração recomendada de núcleos para a classificação FACS (ou seja, ~ 1 × 105 núcleos / mL).
O fígado é caracterizado pela natureza poliploide dos hepatócitos9, mas o papel da ploidia dos hepatócitos na fisiologia e na doença normais ainda não está claro. Há um crescente corpo de evidências indicando que a ploidia fornece variabilidade genômica31, e é bem sabido que a ploidia aumenta com a idadede 32,33 anos. O enriquecimento de hepatócitos tetraploides mononucleados, no entanto, também está clinicamente associado a mau prognóstico no carcinoma hepatocelular humano (CHC)34. Da mesma forma, alterações nos níveis de ploidia de hepatócitos estão ligadas a doenças hepáticas crônicas relacionadas ao envelhecimento, como a doença hepática gordurosa não alcoólica (DHGNA)35,36,37. A ploidia é a condição de possuir mais de duas cópias do genoma, que podem ser exploradas pela coloração do conteúdo do genoma com um corante de DNA, como o Hoechst38. O corante Hoechst, que é adicionado ao HB antes da extração, marca todos os núcleos durante o protocolo de isolamento. Isso permite a distinção entre núcleos diploide e poliploide com base em seu conteúdo de DNA quando excitado por um laser UV (350 nm) ou violeta (450 nm) em um instrumento de citometria de fluxo. Com a estratégia de gating apresentada, 2n, 4n, 8n e níveis mais elevados de ploidia de hepatócitos podem ser investigados em fígados congelados e arquivados para melhor compreender o papel da heterogeneidade celular na função tecidual1 (Figura 3A). Além disso, a morfologia nuclear, incluindo o tamanho e o volume, pode ser quantificada usando citometria de fluxo de imagem para correlacionar mudanças no tamanho do núcleo com mudanças no número total de contagens ou número de genes, dependendo do nível de ploidia (Figura 3B,C).
A medição ômica multimodal oferece a oportunidade de investigar várias camadas de organização genômica simultaneamente. A abordagem multiômica conjunta RNA + ATAC permite a investigação de reguladores a montante e genes metabólicos a jusante, fornecendo uma abordagem abrangente para o estudo de redes transcricionais e a arquitetura da cromatina associada à função hepática na resolução de célula única. Além disso, com os avanços nos métodos computacionais que podem explicar a esparsidade dos dados e a redução nos custos de sequenciamento, a multiômica de célula única é pioneira na avaliação de múltiplas modalidades a partir de uma mesma célula. Este protocolo de isolamento de núcleo único é compatível com a avaliação individual e conjunta de conjuntos de dados de expressão e cromatina. Utilizamos pipelines padrão estabelecidos por Stuart et al.23 (pacote Signac) para ilustrar a qualidade dos dados, enquanto vários métodos computacionais disponíveis e alternativos podem ser facilmente adotados para análises a jusante23,39,40,41.
No geral, a multiômica de núcleo único permite a investigação de tecidos hepáticos de camundongos com FF bioarquivados, humanos e não humanos usando uma quantidade muito pequena de material de amostra inicial, implementando o protocolo de extração de núcleo apresentado aqui. Esta ferramenta inestimável capacitará os biólogos do fígado a interrogar a expressão gênica e a acessibilidade da cromatina no contexto de várias patologias hepáticas. Além disso, vários níveis de ploidia de hepatócitos e o ajuste resultante da expressão gênica dependente de sua localização no lóbulo hepático podem revelar seu papel nas patologias hepáticas. Portanto, antecipamos que a investigação da heterogeneidade celular proporcionará novas oportunidades para o desenvolvimento da medicina de precisão e intervenções direcionadas contra doenças como CHC e DHGNA.
Divulgações
Os autores declaram que não têm conflitos de interesse.
Agradecimentos
Esta pesquisa foi apoiada pelo Helmholtz Pioneer Campus (M.S., K.Y., C.P.M.-J.) e pelo Institute of Computational Biology (C. T.-L.). Esta pesquisa também foi apoiada pela AMED sob o número de concessão JP20jm0610035 (C.P.M.-J.). Agradecemos ao apoio do Core Genomics no HMGU (I. de la Rosa) e Bioinformática (T. Walzthoeni), em particular Xavier Pastor pelo treinamento e orientação. Agradecemos a A. Feuchtinger, U. Buchholz, J. Bushe e todos os outros membros da equipe da HMGU Pathology and Tissue Analytic core facility por seu apoio técnico e científico, bem como a J. Zorn, R. Erdelen, D. Würzinger, membros da equipe da E-Streifen, bem como à instalação central de Serviços de Animais de Laboratório por seu apoio científico e discussão contínuos. Somos gratos à Core Facility Cell Analysis da TranslaTUM (R. Mishra) e à Luminex, A DiaSorin Company (P. Rein). Agradecemos ao Dr. I Deligiannis pelo seu apoio técnico. O Dr. M. Hartman, o Dr. A. Schröder e a Sra. A. Barden (Helmholtz Pioneer Campus) foram fundamentais para o seu apoio jurídico, gerencial e administrativo.
Materiais
| Name | Company | Catalog Number | Comments |
| 10% Tween 20 - 5 mL | Bio-Rad | 1662404 | |
| 2100 Bioanalyzer Instrument | Agilent | G2939BA | |
| Adhesive PCR film | Thermo Fisher Scientific | AB0558 | |
| Bioanalyzer High Sensitivity DNA Analysis | Agilent | 5067-4626 | |
| C1000 Touch Thermal Cycler | Bio-Rad | 1851196 | |
| Cell Sorter | For fluoresence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. | ||
| Centrifuge 5430R | Eppendorf | 5428000619 | Use chilled at 4 °C |
| Chromium Controller & Next GEM Accessory Kit | 10X Genomics | 1000204 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | 1000127 | |
| Chromium Next GEM Chip J Single cell kit, 16 reactions | 10X Genomics | 1000230 | |
| Chromium Next GEM Single Cell 3' Kit v3.1, 4 reactions | 10X Genomics | 1000269 | |
| Chromium Next GEM Single Cell Multiome ATAC + Gene Expression Reagent Bundle, 4 reactions | 10X Genomics | 1000285 | |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Sigma Aldrich | 11873580001 | |
| Dithiothreitol (DTT) 1 M solution | Sigma Aldrich | 646563 | Make 1 mM stock solution and use at 1 µM final concentration. |
| DNA AWAY Surface Decontaminant | Thermo Fisher Scientific | 10223471 | Wipe surfaces and pipettes before start of experiment |
| DNA LoBind Tubes, 1.5 mL | Thermo Fisher Scientific | 16628742 | |
| DNA LoBind Tubes, 2.0 mL | Thermo Fisher Scientific | 16638742 | |
| Elution Buffer (EB) - 250 mL | Qiagen | 19086 | |
| Eppendorf ThermoMixer C | Thermo Fisher Scientific | 13527550 | |
| Eppendorf ThermoMixer C Accessory: Smartblock | Thermo Fisher Scientific | 13518470 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade - 100 mL | Thermo Fisher Scientific | 10517694 | |
| Filters 50 µm, sterile | SYSMEX PARTEC - CELLTRICS | 04-004-2327 | Adjust filter diameter according with tissue and nuclei size |
| Glycerin (Glycerol), 50% (v/v) - 1 L | Ricca Chemical Company | 3290-32 | |
| Hard-Shell, 384-Well PCR Plates, thin wall, skirted, clear/white | Bio-Rad | HSP3905 | |
| Herenz Heinz ABS Forceps | Thermo Fisher Scientific | 1131884 | |
| Hoechst 33342, Trihydrochloride, Trihydrate, 10 mg/mL Solution in Water | Invitrogen | H3570 | Light-sensitive |
| Imaging Flow Cytometer | For imaging flow cytometry analysis, e.g. Luminex Amnis ImageStream | ||
| Invitrogen TE Buffer - 100 mL | Thermo Fisher Scientific | 11568846 | |
| KCl (2 M), RNase-free, 100 mL | Thermo Fisher Scientific | AM9640G | |
| MgCl2 (1 M), 100 mL | Thermo Fisher Scientific | AM9530G | |
| MicroAmp 8-Cap Strip, clear-300 strips | Thermo Fisher Scientific | 10209104 | |
| MicroAmp 8-Tube Strip, 0.2 mL-125 strips | Thermo Fisher Scientific | 10733087 | |
| Mörser 2 mL DOUNCE | Wagner & Munz GmbH | 9651632 | RNase zap and rinse with MillQ before use |
| MyFuge 12 Mini MicroCentrifuge C1012 | Benchmark Scientific | C1012 | Or any other strip and tube mini centrifuge |
| Neubauer Hemocytometer | OMNILAB LABORZENTRUM | 5435293 | Visualize and count nuclei under microscope |
| Nuclease-Free Water (not DEPC-Treated) | Thermo Fisher Scientific | AM9937 | |
| OptiPrep Density Gradient Medium | Sigma Aldrich | D1556 | |
| Pipette tips RT LTS 1000 µL, Wide-O | Mettler Toledo | 30389218 | |
| Pistill "A" 2 mL | Wagner & Munz GmbH | 9651621 | RNase zap and rinse with MillQ before use |
| Pistill "B" 2 mL | Wagner & Munz GmbH | 9651627 | RNase zap and rinse with MillQ before use |
| Polypropylene 15 mL Centrifuge Tube | Thermo Fisher Scientific | 10579691 | |
| Polystyrene Petri dish, 60 mm x 15 mm | Thermo Fisher Scientific | 10634141 | |
| Polystyrene Round-Bottom 5 mL FACS Tubes | Thermo Fisher Scientific | 10100151 | |
| Protector RNase inhibitor - 2,000 U | Sigma Aldrich | 3335399001 | Keep in -20 °C until use |
| Protein-based RNase Inhibitor SUPERase•In (20 U/μL) | Thermo Fisher Scientific | AM2696 | Keep in -20 °C until use |
| Recombinant RNase Inhibitor | Clontech Takara | 2313B | Keep in -20 °C until use |
| RNAse free microfuge tubes - 0.5 mL | Thermo Fisher Scientific | AM12450 | |
| RNaseZap RNase Decontamination Solution | Thermo Fisher Scientific | AM9780 | Wipe surfaces and pipettes before start of experiment |
| SPRIselect - 60 mL | Beckman Coulter | B23318 | Aliquot and store in 4 °C |
| Sucrose, 500 g | Sigma Aldrich | S0389-500G | Make a 1 M stock solution |
| Swann-Morton Sterile Disposable Stainless Steel Scalpels | Thermo Fisher Scientific | 11798343 | |
| Tris-HCI (1M), pH 8.0 | Invitrogen | 15568025 | |
| Triton X-100, 98%, 100 mL | Thermo Fisher Scientific | 10671652 | Make 10% stock solution. Keep at 4 °C and protect from light. |
| Trypan Blue Solution, 0.4% | Thermo Fisher Scientific | 11538886 | |
| Vortex- Mixer | VWR | 444-1372 | Or any other type of vortex |
Referências
- Kamies, R., Martinez-Jimenez, C. P. Advances of single-cell genomics and epigenomics in human disease: where are we now. Mamm Genome. 31 (5-6), 170-180 (2020).
- Ramachandran, P., Matchett, K. P., Dobie, R., Wilson-Kanamori, J. R., Henderson, N. C. Single-cell technologies in hepatology: New insights into liver biology and disease pathogenesis. Nature Reviews Gastroenterology & Hepatology. 17 (8), 457-472 (2020).
- Ramachandran, P., et al. Resolving the fibrotic niche of human liver cirrhosis at single-cell level. Nature. 575 (7783), 512-518 (2019).
- Andrews, T. S., et al. Single-cell, single-nucleus, and spatial RNA sequencing of the human liver identifies cholangiocyte and mesenchymal heterogeneity. Hepatology Communications. 6 (4), 821-840 (2022).
- Xiong, X., et al. Landscape of intercellular crosstalk in healthy and NASH liver revealed by single-cell secretome gene analysis. Molecular Cell. 75 (3), 644-660 (2019).
- Richter, M. L., et al. Single-nucleus RNA-seq2 reveals functional crosstalk between liver zonation and ploidy. Nature Communications. 12 (1), 4264 (2021).
- Matsumoto, T., Wakefield, L., Tarlow, B. D., Grompe, M. In vivo lineage tracing of polyploid hepatocytes reveals extensive proliferation during liver regeneration. Cell Stem Cell. 26 (1), 34-47 (2020).
- Chen, F., et al. Broad distribution of hepatocyte proliferation in liver homeostasis and regeneration. Cell Stem Cell. 26 (1), 27-33 (2020).
- Donne, R., Saroul-Ainama, M., Cordier, P., Celton-Morizur, S., Desdouets, C. Polyploidy in liver development, homeostasis and disease. Nature Reviews. Gastroenterology & Hepatology. 17 (7), 391-405 (2020).
- Lengefeld, J., et al. Cell size is a determinant of stem cell potential during aging. Science Advances. 7 (46), 0271 (2021).
- Lanz, M. C., et al. Increasing cell size remodels the proteome and promotes senescence. Mol Cell. 82 (17), 3255-3269 (2022).
- Kim, J. Y., et al. PIDDosome-SCAP crosstalk controls high-fructose-diet-dependent transition from simple steatosis to steatohepatitis. Cell Metabolism. 34 (10), 1548-1560 (2022).
- Padovan-Merhar, O., et al. Single mammalian cells compensate for differences in cellular volume and DNA copy number through independent global transcriptional mechanisms. Molecular Cell. 58 (2), 339-352 (2015).
- Miettinen, T. P., et al. Identification of transcriptional and metabolic programs related to mammalian cell size. Current Biology. 24 (6), 598-608 (2014).
- Vargas-Garcia, C. A., Ghusinga, K. R., Singh, A. Cell size control and gene expression homeostasis in single-cells. Current Opinion in Systems Biology. 8, 109-116 (2018).
- Knoblaugh, S. E., Randolph-Habecker, J. . Necropsy and histology. In Comparative Anatomy and Histology: A Mouse, Rat, and Human Atlas (Second Edition). , (2018).
- Chromium Next GEM Single Cell 3 Reagent Kits v3.1 User Guide. Document number CG000204 (Rev D). 10x Genomics Available from: https://www.10xgenomics.com/support/single-cell-gene-expression/documentation/steps/library-prep/chromium-single-cell-3-reagent-kits-user-guide-v-3-1-chemistry (2019)
- Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide. Document number CG000338 (Rev D). 10x Genomics Available from: https://www.10xgenomics.com/support/single-cell-multiome-atac-plus-gene-expression/documentation/steps/library-prep/chromium-next-gem-single-cell-multiome-atac-plus-gene-expression-reagent-kits-user-guide (2021)
- MacParland, S. A., et al. Single cell RNA sequencing of human liver reveals distinct intrahepatic macrophage populations. Nature Communications. 9 (1), 4383 (2018).
- Martinez-Jimenez, C. P., Kyrmizi, I., Cardot, P., Gonzalez, F. J., Talianidis, I. Hepatocyte nuclear factor 4alpha coordinates a transcription factor network regulating hepatic fatty acid metabolism. Molecular and Cell Biology. 30 (3), 565-577 (2010).
- Schmidt, D., et al. Five-vertebrate ChIP-seq reveals the evolutionary dynamics of transcription factor binding. Science. 328 (5981), 1036-1040 (2010).
- Hao, Y., et al. Integrated analysis of multimodal single-cell data. Cell. 184 (13), 3573-3587 (2021).
- Stuart, T., Srivastava, A., Madad, S., Lareau, C. A., Satija, R. Single-cell chromatin state analysis with Signac. Nature Methods. 18 (11), 1333-1341 (2021).
- Sampaziotis, F., et al. Cholangiocyte organoids can repair bile ducts after transplantation in the human liver. Science. 371 (6531), 839-846 (2021).
- Gomez-Lechon, M. J., Donato, M. T., Castell, J. V., Jover, R. Human hepatocytes as a tool for studying toxicity and drug metabolism. Current Drug Metabolism. 4 (4), 292-312 (2003).
- Gomez-Lechon, M. J., Donato, M. T., Castell, J. V., Jover, R. Human hepatocytes in primary culture: The choice to investigate drug metabolism in man. Current Drug Metabolism. 5 (5), 443-462 (2004).
- vanden Brink, S. C., et al. Single-cell sequencing reveals dissociation-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Denisenko, E., et al. Systematic assessment of tissue dissociation and storage biases in single-cell and single-nucleus RNA-seq workflows. Genome Biology. 21 (1), 130 (2020).
- Nault, R., Fader, K. A., Bhattacharya, S., Zacharewski, T. R. Single-nuclei RNA sequencing assessment of the hepatic effects of 2,3,7,8-tetrachlorodibenzo-p-dioxin. Cellular and Molecular Gastroenterology and Hepatology. 11 (1), 147-159 (2021).
- Krishnaswami, S. R., et al. Using single nuclei for RNA-seq to capture the transcriptome of postmortem neurons. Nature Protocols. 11 (3), 499-524 (2016).
- Duncan, A. W., et al. Aneuploidy as a mechanism for stress-induced liver adaptation. Journal of Clinical Investigation. 122 (9), 3307-3315 (2012).
- Kreutz, C., et al. Hepatocyte ploidy is a diversity factor for liver homeostasis. Frontiers in Physiology. 8, 862 (2017).
- Hunt, N. J., Kang, S. W. S., Lockwood, G. P., Le Couteur, D. G., Cogger, V. C. Hallmarks of aging in the liver. Computational and Structural Biotechnology Journal. 17, 1151-1161 (2019).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. 69 (2), 355-364 (2019).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Schwartz-Arad, D., Zajicek, G., Bartfeld, E. The streaming liver IV: DNA content of the hepatocyte increases with its age. Liver. 9 (2), 93-99 (1989).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv. B, Cell Pathology Including Molecular Pathology. 64 (6), 387-393 (1993).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Granja, J. M., et al. ArchR is a scalable software package for integrative single-cell chromatin accessibility analysis. Nature Genetics. 53 (3), 403-411 (2021).
- Bredikhin, D., Kats, I., Stegle, O. MUON: Multimodal omics analysis framework. Genome Biology. 23 (1), 42 (2022).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nature Methods. 19 (2), 179-186 (2022).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados