Method Article
בידוד גרעינים מרקמת כבד קפואה עבור מולטיומיה חד-תאית
In This Article
Summary
כאן אנו מציגים פרוטוקול לבידוד גרעינים מרקמות כבד מוקפאות בהבזק, בארכיון עבור RNA-seq, ATAC-seq ומולטיומיקה של מפרקים (RNA-seq ו-ATAC-seq).
Abstract
הכבד הוא רקמה מורכבת והטרוגנית האחראית על ביצוע פונקציות פיזיולוגיות קריטיות רבות, כגון שמירה על הומאוסטזיס אנרגיה ומטבוליזם של קסנוביוטיקה, בין היתר. משימות אלה מבוצעות באמצעות תיאום הדוק בין תאים פרנכימליים בכבד לבין תאים שאינם פרנכימליים. בנוסף, פעילויות מטבוליות שונות מוגבלות לאזורים ספציפיים של אונת הכבד - תופעה הנקראת ייעוד כבד. ההתקדמות האחרונה בטכנולוגיות ריצוף של תאים בודדים אפשרה לחוקרים לחקור הטרוגניות של רקמות ברזולוציה של תא יחיד. ברקמות מורכבות רבות, כולל הכבד, פרוטוקולי דיסוציאציה אנזימטיים ו/או מכניים קשים יכולים להשפיע לרעה על הכדאיות או על האיכות של התרחיפים החד-תאיים הדרושים לאפיון מקיף של איבר זה בבריאות ובמחלות.
מאמר זה מתאר פרוטוקול חזק וניתן לשחזור לבידוד גרעינים מרקמות כבד קפואות בארכיון. שיטה זו מניבה גרעינים באיכות גבוהה התואמים לגישות אומיקה חד-תאית במורד הזרם, כולל RNA-seq בעל גרעין יחיד, בדיקה לכרומטין נגיש לטרנספוזאז עם ריצוף בתפוקה גבוהה (ATAC-seq), כמו גם אומיקה רב-מודאלית (RNA-seq משותפת ו-ATAC-seq). שיטה זו שימשה בהצלחה לבידוד גרעינים מדגימות כבד קפואות של בני אדם, עכברים ופרימטים שאינם בני אדם בריאים וחולים. גישה זו מאפשרת בידוד בלתי משוחד של כל סוגי התאים העיקריים בכבד, ולכן מציעה מתודולוגיה איתנה לחקר הכבד ברזולוציה של תא בודד.
Introduction
גנומיקה חד-תאית הופכת במהירות למתודולוגיה חיונית לחקר תפקוד הכבד ולהערכת ההשפעה של הטרוגניות תאית במצבי בריאות ומחלות1. ההתפתחות המהירה של "מולטיומיקה" למדידה סימולטנית של שכבות מידע שונות וההתרחבות המקבילה של צינורות חישוביים חזקים סוללת את הדרך לגילוי סוגי תאים ותת-סוגים שלא היו ידועים קודם לכן בכבד הרגיל והחולה2.
האפשרות לחקור ביו-בנקים ודגימות קפואות בארכיון הגדילה באופן משמעותי את ההזדמנויות לבחון מחדש ולגלות את תפקידם של תאים שאינם פרנכימליים 3,4,5 ולחקור את תפקידם של הפטוציטים פוליפלואידים במהלך ההזדקנות ובמחלות כרוניות 6,7,8,9 . לכן, מאמר זה מתאר פרוטוקול בידוד חזק וניתן לשחזור של גרעין יחיד עבור כבדים ארכיוניים קפואים (FF) התואם לריצוף RNA בגרעין יחיד במורד הזרם ולריצוף ATAC, כמו גם לאומיקה רב-מודאלית (RNA-seq משותפת ו-ATAC-seq) (איור 1).
תהליך עבודה זה מאפשר לחקור את התעתיק ואת נגישות הכרומטין של כל סוגי התאים בכבד, ללא תלות בגודל התא או בשבריריותו, בפרוטוקולי דיסוציאציה אנזימטית. זה יכול להתבצע עם קטעי רקמות קטנות (15-30 מ"ג או 5-10 מ"מ3) מדגימות אנושיות יקרות ערך או עכברים מהונדסים. קביעת הטוהר הגבוה של בידוד הגרעינים כוללת כימות ומדידה של גודל הגרעין, אשר עשוי להיות בקורלציה עם הגדלת גודל התא וסנסנציה 10,11, וטוהר זה רלוונטי לניתוח הן של פלואידית הפטוציטים 12 והן של מנגנוני שעתוק תלויי גודל התא11,13,14,15 . בנוסף, גרעינים שבודדו מכבדים קפואים שומרים מידע רב ערך על ייעוד הכבד. זרימת העבודה ואיסוף הרקמות מאפשרים אימות של נתוני גנומיקה חד-תאית או ניתוחים משלימים נוספים, כגון אימונוהיסטוכימיה או תעתיק מרחבי מאותה רקמה ומאותו אדם. לכן, גישה זו יכולה להיות מיושמת על מחלות כבד מרובות ואורגניזמים מודל באופן שיטתי ואמין.
Protocol
כל הניסויים בבעלי חיים בוצעו בהתאם לחקיקה הגרמנית לרווחת בעלי חיים ולתקנות ממשלת בוואריה עילית. דיור לבעלי חיים אושר על פי §11 של החוק הגרמני לרווחת בעלי חיים ובוצע בהתאם להנחיה 2010/63/EU.
1. הכנת רקמות
- להקריב עכבר C57BL/6J זכר בן 3 חודשים עד 22 חודשים על ידי נקע צוואר הרחם. הניחו את החיה על קרש ניתוח, אבטחו את הגפיים עם סיכות וחטאו את הבטן עם 70% אתנול.
- בצע נמק כפי שהומלץ על ידי Treuting et al.16.
- פתח את הבטן עד לצלעות, דמיינו את הכבד, והשתמשו במלקחיים כדי להסיר בזהירות את הכבד מבלי לחדור את האונות.
- חלץ את הכבד השלם על ידי החזקת הסרעפת עם מלקחיים והסרת הרקמה המחברת עם מספריים.
- שוטפים את האיבר בתמיסת מלח קרה עם פוספטים (PBS), מייבשים אותו על מגבת נייר נקייה וחותכים את אונות הכבד למספר חלקים למטרות שונות: FF לבידוד גרעין יחיד ומולטיומיה; פרפורמלדהיד קבוע (10% PFA) להטמעת פרפין; ו/או מוטמע בתרכובת טמפרטורת חיתוך אופטימלית (OCT) לצורך ניתוחים היסטולוגיים נוספים (איור 1A).
- Aliquot את חתיכות הכבד לתוך cryovials או 5 מ"ל בורג מכסה צינורות, ומיד הבזק להקפיא חנקן נוזלי. אחסנו את דגימות הכבד הקפואות בטמפרטורה של −80 מעלות צלזיוס עבור ניסויי מולטיומיקה של גרעין יחיד במורד הזרם (איור 1B, C).
הערה: ניתן לאחסן בבטחה את הרקמה בהקפאה בטמפרטורה של 80°C- למשך מספר שנים, ויש לשנע אותה תמיד על קרח יבש בטמפרטורה של 80°C- לפני השימוש.
2. בידוד גרעינים
- ניקוי ספסלים והכנת המאגרים והחומרים המתכלים
- נקו את משטחי הספסלים והפיפטות העובדים עם 70% אתנול ותמיסת RNase לפירוק, או השתמשו במשטחי ספסל וחומרים ייעודיים ללא RNase.
- קדם הן את הדלי המתנדנד והן את הצנטריפוגות בעלות הזווית הקבועה, צינורות 1.5 מ"ל/2 מ"ל, ולוחות מרובי בארות ל-4 מעלות צלזיוס, כמפורט להלן.
- הכינו מראש את הפינצטה החד-פעמית נטולת ה-RNase המשמשת לטיפול ברקמות, אזמל סטרילי חד פעמי וצלחת פטרי על קרח יבש (−60 מעלות צלזיוס).
- Prechill את ההומוגנייזר זכוכית Dounce ומזיקים על קרח (4 °C (4 °F). הכניסו כל מזיק (A ו-B) לתוך צינור של 5 מ"ל כדי למנוע מגע ישיר עם הקרח וזיהום RNase פוטנציאלי.
- הכינו תמיסה מדוללת של מדיום יודיקסנול (IDM) (טבלה 1A), והשתמשו בה כדי לבצע דילולים של 50% ו-29% מהמדיום ההדרגתי של צפיפות מלאי יודיקסנול של 60% (טבלה 1B וטבלה 1C, בהתאמה).
- Prechill כל הצינורות. עבור כל דגימה כי הוא הולך להיות מעובד, להכין את הצינורות הבאים:
- הכינו שלושה צינורות קשירה בעלי DNA נמוך של 1.5 מ"ל (אחד עבור הומוגנט הרקמה המסוננת, השני המכיל 250 μL של דילול 50% של תמיסת יודיקסנול, ושלישית עבור תרחיף הגרעינים הנקיים).
- הכן צינור אחד בעל תחתית עגולה של 2 מ"ל המכיל 500 μL של דילול של 29% של תמיסת יודיקסנול להפרדת גרדיאנט הצפיפות.
- הכינו צינור חרוטי אחד של 15 מ"ל לבידוד גרעינים בינוני-2 (NIM-2) ומאגר הומוגניזציה (HB).
- הכן את בידוד הגרעינים בינוני-1 (NIM-1) (טבלה 1D), והשתמש בו כדי ליצור את NIM-2 (טבלה 1E) ולאחר מכן, את HB (טבלה 1F). הוסיפו את שני מעכבי ה-RNAse ממש לפני השימוש, כפי שמתואר להלן בפרוטוקול.
- הכן את מאגר אחסון הגרעינים (NSB) כמתואר בטבלה 1G. יש להוסיף מעכב RNAse רקומביננטי ממש לפני השימוש. הוסיפו מעכב RNAse מבוסס חלבון ל-NSB לפני השימוש במיון FACS (אופציונלי).
- הכינו של 500 מ"ל עם מים סטריליים כדי להשרות את ההומוגנייזרים והמזיקים של Dounce לאחר הומוגניזציה של הרקמות לניקוי ותחזוקה אופטימליים של ההומוגנייזרים.
- הומוגניזציה של רקמות
- חותכים חתיכת רקמה של 20-30 מ"ג (או 5 מ"מ3) עם אזמל מוכן מראש בתוך צלחת פטרי על קרח יבש. לאחר מכן, מיד להעביר את צלחת פטרי לקרח רטוב (4 מעלות צלזיוס). הוסף 1 מ"ל של HB, ובאמצעות אזמל קר, לטחון את הרקמה ככל האפשר כדי לאפשר לו להיות שואף בקלות עם קצה 1 מ"ל רחב פתח.
הערה: השתמש תמיד בטיפים רחבים לכל העברות הרקמות/גרעינים. כחלופה, ניתן לחתוך קצוות של 1 מ"ל עם אזמל סטרילי על כיסוי פלסטיק סטרילי כדי ליצור פתחים רחבים (איור 2A). - אספו את תרחיף הרקמות, והעבירו אותו לזכוכית Dounce הומוגנייזר של 2 מ"ל (איור 2B).
- שטפו את צלחת הפטרי עם תוספת של 0.5-1 מ"ל של HB, ואספו את כל חתיכות הרקמה הנותרות תוך שמירה על הכל על קרח.
- לאט ובזהירות לעשות חמש משיכות עם מזיק A רופף על קרח. הימנעו מיצירת בועות על ידי שימוש בתנועות פיתול של המזיק בעת משיכתו למעלה ולמטה. ודא כי המזיק נע בזהירות מהחלק העליון לתחתית של ההומוגנייזר עם כל שבץ.
- לאחר מכן, בצע 10-15 משיכות איטיות עם מזיק B הדוק על קרח. הימנעו מיצירת בועות.
הערה: מומלץ לבדוק באופן חזותי את הגרעינים מתחת למיקרוסקופ לאחר 10 פעימות עם מזיק B כדי לוודא אם יש צורך במשיכות נוספות. עשו זאת על ידי שימוש בכתמים כחולים של טריפאן (יחס של 1:1 בין טריפאן לדגימה) ובהמוציטומטר ידני (למשל, לערבב 10 μL של טריפאן כחול עם 10 μL של תרחיף גרעינים, ולהשתמש ב-10 μL של התערובת לבדיקה תחת המיקרוסקופ; איור 2C). - מסננים את ההומוגנט דרך מסננת תאים בקוטר 50 מיקרומטר תוך כדי העברתו לצינור 1.5 מ"ל מוכן מראש. יש להשתמש ביותר ממסנן ו/או צינור אחד עבור הומוג'נטים המכילים כמות גבוהה של גושי רקמת חיבור.
- שטפו את ההומוגנייזר ואת המסננים המשמשים עם 0.5-1 מ"ל נוספים של HB כדי לאסוף ביסודיות את כל ההומוגנט של הרקמות. המשך לצנטריפוגה של שיפוע הצפיפות.
- חותכים חתיכת רקמה של 20-30 מ"ג (או 5 מ"מ3) עם אזמל מוכן מראש בתוך צלחת פטרי על קרח יבש. לאחר מכן, מיד להעביר את צלחת פטרי לקרח רטוב (4 מעלות צלזיוס). הוסף 1 מ"ל של HB, ובאמצעות אזמל קר, לטחון את הרקמה ככל האפשר כדי לאפשר לו להיות שואף בקלות עם קצה 1 מ"ל רחב פתח.
- צנטריפוגה הדרגתית של צפיפות
- צנטריפוגה ההומוגנית המסוננת בצנטריפוגה קבועה בזווית קבועה ב-1,000 × גרם למשך 8 דקות ב-4 מעלות צלזיוס.
- בזמן שהדגימה מסתובבת, הכינו שפופרת של 1.5 מ"ל המכילה 250 μL של דילול 50% יודיקסנול וצינור אחד של 2 מ"ל המכיל 500 μL של 29% דילול יודיקסנול. שמור את שני הצינורות על קרח.
- לאחר צנטריפוגה, שאפו את הסופר-נטנט מבלי להפריע לכדור באמצעות משאבת ואקום.
הערה: שימוש בצנרת ידנית יפגע באיכות מתלי הגרעינים הסופיים. - הוסיפו 250 μL של HB לכדור באמצעות קצה פיפטה רחב של 1 מ"ל, ושתו באיטיות רבה.
- מעבירים 250 מיקרוליטר מתרחיף הגרעינים לתוך צינור 1.5 מ"ל קדמי המכיל 250 μL של 50% דילול יודיקסנול, ומערבבים בעדינות אך ביסודיות כדי ליצור תרחיף 25% יודיקסנול/גרעינים.
- העבר 500 μL מתרחיף 25% יודיקסנול/גרעינים לתוך צינור 2 מ"ל מוכן מראש המכיל 500 μL של 29% דילול יודיקסנול.
הערה: יש להפקיד בעדינות את 500 μL של תרחיף 25% יודיקסנול/גרעינים על גבי תמיסת 500 μL של 29% יודיקסנול, כך שתערובות 25%/29% יודיקסנול מראות הפרדת פאזה ברורה. השתמש בצד של קיר הצינור כאשר קצה הפיפטה ממוקם בזווית של 45° כדי ליצור ממשק הדרגתי זה. מכאן ואילך, יש לטפל בצינור בעדינות כדי לא להפריע לשיפוע זה. - צנטריפוגה את הצינור בצנטריפוגת דלי מתנדנדת מראש ב 12,500 גרם במשך 20 דקות עם הבלם מוגדר OFF.
- רגע לפני השלמת שלב הצנטריפוגה, הוסיפו את מעכבי ה-RNAse למאגר ה-NSB כאשר אתם ממשיכים לצינורות scRNA-seq (ראו טבלה 1G).
- לאחר צנטריפוגה, שאפו את הסופר-נטנט מבלי להפריע לכדור באמצעות משאבת ואקום.
הערה: שימוש בצנרת ידנית יפגע באיכות מתלי הגרעינים הסופיים. - באמצעות קצה פיפטה רחב של 1 מ"ל, יש לתלות בעדינות את הכדור ב-100-300 מיקרון של NSB, ולהעביר את תלייה הגרעינים לצינור נקי ומצונן מראש של 1.5 מ"ל.
- לספור את הגרעינים באמצעות תמיסת טריפאן כחולה (יחס של 1:1 בין טריפאן לדגימה) והמוציטומטר ידני (למשל, לערבב 10 μL של כחול טריפאן עם 10 μL של תרחיף גרעינים, ולהשתמש 10 μL של התערובת לספירה; איור 2D).
- השתמש בהשעיית הגרעינים המתקבלת באופן מיידי לבדיקת הגנומיקה של גרעין יחיד.
הערה: ניתן לאחסן את תרחיף הגרעינים ב-NSB בקירור בטמפרטורה של 4 מעלות צלזיוס למשך עד שבוע אחד לצורך ניתוח נוסף על ידי ציטומטריה של זרימה ו/או הדמיה, אך לא עבור snRNA-seq או snATAC-seq.
3. מיון גרעינים עבור פרופיל פלואידי הפטוציטים או גישות ריצוף מבוססות היטב
- למיון תאים מבוסס ציטומטריה של זרימה, סנן את תרחיף הגרעינים דרך מסנן של 50 מיקרומטר לתוך צינור FACS של 5 מ"ל מראש.
- השתמש סדרן ציטומטריה זרימה מצויד זרבובית 100 מיקרומטר. טען את צינור FACS על הממיין והצג תצוגה מקדימה של הדגימה.
- הגדר את אסטרטגיית ההטיה למיון הגרעינים, החל משער פיזור על ידי התוויית אזור הפיזור הקדמי לעומת אזור הפיזור הצדדי (FSC-A/SSC-A), לאחר מכן Hoechst-Height לעומת Hoechst-Area (שער גרעינים), ולאחר מכן Hoechst-Width לעומת Hoechst-Area (שער סינגלט). דמיינו את פרופיל הגרעינים בהיסטוגרמה של אזור Hoechst.
הערה: לקבלת רזולוציית שיא טובה יותר, דמיינו את ערוץ Hoechst (450/50) בסולם הליניארי (איור 3A). - למיון ללוחות 96/384-well, הגדר את השהיית הטיפות, ומטב את יישור הלוחות בשיטה colorimetric עם בנזידין מצע-חזרת peroxidase (TMB-HRP), כפי שתואר קודםלכן 6.
- הגדר הן את קירור הדגימה והן את מחזיק הצלחת לקירור בטמפרטורה של 4 מעלות צלזיוס, כאשר סיבוב הדגימה מופעל ב-300 סל"ד.
- מיין את הגרעינים הבודדים בריכוז דגימה של ~ 1 x 105 גרעינים / מ"ל ועם קצב הזרימה ב 200-500 אירועים / שנייה.
4. בדיקה חזותית וכימות של פרמטרים גרעיניים על ידי הדמיה ציטומטריה (אופציונלי)
- טען צינור של 0.5 מ"ל המכיל 50 μL של תרחיף הגרעינים בריכוז של 2 × 107 גרעינים למ"ל.
- הגדר שער לכל האירועים בהתבסס על יחס הגובה-רוחב לעומת ערוץ הפלואורסצנציה של Hoechst כדי להמחיש את פרופיל הגרעינים (איור 3B).
- קבל את הדגימה בהגדלה של 40x באמצעות שדה בהיר (BF) וערוץ הפלואורסצנציה Hoechst.
- בדוק וכמת גרעיני 2n ו-4n באמצעות מדידת שדה בהיר ועוצמת פלואורסצנציה של Hoechst באמצעות תוכנת הדמיה ציטומטרית (איור 3C).
5. גרעין יחיד RNA-seq, ATAC-seq, או בנייה וריצוף של ספרייה רב-ערוצית
- עבור גישות snRNA-seq המבוססות על טיפות, טען את תרחיף הגרעינים המטוהרים ישירות לתוך ההתקן המיקרופלואידי למחיצות מקביליות אוטומטיות וברקוד מולקולרי17.
- לאחר השלמת הריצה המיקרופלואידית בהתקן החלוקה החד-תאית, אספו את הגרעינים עטופים בג'ל חרוזים, דגרו ונקו כפי שתואר קודם לכן בהנחיות היצרן17.
- בצע 11 מחזורי תגובת שרשרת פולימראז (PCR) עבור קדם-הגברה של cDNA באמצעות התוכנית הבאה: 3 דקות ב-98 °C, (15 שניות ב-98°C, 20 שניות ב-63°C, ודקה אחת ב-72°C) x 11, דקה אחת ב-72°C, והחזקה ב-4°C. המשך עם תיקון קצה ושלב זנב A וקשירת מתאם, כפי שצוין על ידי היצרן17. לבניית ספריית ביטוי הגנים הסופית שלאחר מכן, בצע 10 מחזורי PCR באמצעות התוכנית הבאה: 45 שניות ב- 98 °C, (20 שניות ב- 98 ° C, 30 שניות ב- 54 ° C, 20 שניות ב- 72 ° C) x 10, דקה אחת ב- 72 ° C, והחזק ב- 4 ° C.
- רצף את הספריות המתקבלות לעומק קריאה של ~20,000-50,000 קריאות ממוצעות לכל גרעין.
- עבור ריצוף מבוסס טיפות של מולטיומיקה משותפת (RNA + ATAC), יש לדגום את גרעיני הכבד במאגר הליזיס למשך 5 דקות, ולאחר מכן לתייג אותם למשך שעה אחת, כפי שתואר קודםלכן 18.
- טען את הגרעינים המתויגים ישירות לתוך התקן מיקרופלואידי למחיצות מקביליות אוטומטיות וברקוד מולקולרי.
- לאחר השלמת הריצה המיקרופלואידית, אספו את הגרעינים עטופים בחרוזי ג'ל, דגירה וניקוי כפי שתואר על ידי היצרן18.
- בצע שישה מחזורי PCR לשלב ההגברה של cDNA באמצעות התוכנית הבאה: 5 דקות ב- 72 °C, 3 דקות ב- 98 °C, (20 שניות ב- 98 °C, 30 שניות ב- 63 ° C, דקה אחת ב- 72 ° C) x 6, דקה אחת ב- 72 ° C, והחזק ב- 4 ° C.
- קח 35 μL של הדגימה preamplified, ולבצע הגברה cDNA כדלקמן: 3 דקות ב 98 °C, (15 שניות ב 98 °C, 20 שניות ב 63 °C, 1 דקות ב 72 °C) x 6, 1 דקות ב 72 °C, ולהחזיק ב 4 °C. המשך עם תיקון הקצה ושלב A-tailing וקשירת המתאם, כפי שצוין על ידי היצרן18 . עבור האינדקס הסופי הבא של PCR, בצע 15 מחזורי PCR כדלקמן: 45 שניות ב- 98 °C, (20 שניות ב- 98 ° C, 30 שניות ב- 54 ° C, 20 שניות ב- 72 ° C) x 15, דקה אחת ב- 72 ° C, והחזק ב- 4 ° C.
- לבניית ספריות ATAC, השתמש ב- 40 μL, והגבר עבור שישה מחזורי PCR לאינדקס דגימות באמצעות התוכנית הבאה: 45 שניות ב- 98 ° C, (20 שניות ב- 98 ° C, 30 שניות ב- 67 ° C, 20 שניות ב- 72 ° C) x 6, דקה אחת ב- 72 ° C, והחזק ב- 4 ° C.
- רצף ספריות ביטוי הגנים הרב-תכליתיים המתקבלים לעומק קריאה מינימלי של 20,000 זוגות קריאה לכל גרעין וספריות ATAC מרובות לעומק קריאה מינימלי של 25,000 זוגות קריאה לכל תא, לפי המלצת היצרן18.
תוצאות
תהליך עבודה זה לבידוד גרעין יחיד מדגימות כבד קפואות מותאם למולטיומיקה של גרעין יחיד ומסתמך על שלושה שלבים עיקריים, שניתן לסכם אותם כ- i) איסוף דגימות לניתוח מקביל של הטרוגניות תאית וארכיטקטורת רקמות, ב) תרחיף של גרעין יחיד, ו- iii) מולטיומיקה של גרעין יחיד (איור 1 ). הכבדים המופקים מנותחים מעכברים מורדמים ונחתכים לחתיכות לבדיקה היסטולוגית לצורך הטמעת פרפין, קריוקציה או שניהם. חתיכות חתוכות אחרות מוקפאות מיד בחנקן נוזלי לבידוד במורד הזרם, בגרעין יחיד לניתוחי מולטיומיקה. מערכת זו של איסוף רקמות מאפשרת למשתמש לאמת עוד יותר את נתוני הגרעין הבודד על מקטעי רקמות מאותו אדם, ובכך להשלים את מערך הנתונים עם תעתיק מרחבי או עם ניתוחי אימונוהיסטוכימיה, במידת הצורך.
הבדיקה המיקרוסקופית של הגרעינים המופקים מכבדים קפואים בשיטה המתוארת כאן מראה ששלב הצנטריפוגה של שיפוע הצפיפות מקל מאוד על הסרת פסולת תאית ורקמות לא רצויה (איור 2C,D). יתר על כן, מתודולוגיה זו משמרת את כל רמות הפלואידיות, הניתנות לאימות ולכימות על ידי ניתוחים ציטומטריים (איור 3).
כדי לאמת עוד יותר את הביצועים של פרוטוקול זה, ערכנו snRNA-seq מבוסס טיפות על גרעינים לא ממוינים וממוינים FACS וניתחנו את הנתונים בעקבות צינור Seurat. בקצרה, הגרעינים הבודדים שחולצו הוכנו עבור snRNA-seq מבוסס טיפות כפי שתואר קודםלכן 19. עבור snRNA-seq של גרעיני 2n ו-4n או רמות גבוהות יותר של פלואידיה, 11 מחזורים שימשו לשלב ההגברה של cDNA ו-10 מחזורים לבניית ספריית ביטוי הגנים הסופית. הספריות שהתקבלו רוצפו לעומק קריאה של ~25,000-39,000 קריאות ממוצעות לכל גרעין. קריאות הגרעינים הבודדים שהתקבלו מופו לגנום העכבר GRCm39/mm39. בעת הפעלת צינור העיבוד המקדים, נוספה הפקודה -- include-intron כדי לוודא את ההכללה והכימות של RNA שליח לא מחובר (mRNA) הנמצא בגרעינים. אלגוריתם EmptyDrops ששולב בתוך הקשתית סינן והסיר את הטיפות הריקות.
חבילת R Seurat (גרסה 4.1.1) שימשה לחישוב מדדי בקרת איכות (QC) באמצעות מטריצת ספירת המזהה המולקולרי הייחודית (UMI) כפי שהופקה על ידי צינור ניתוח snRNA-seq. ספירות עם פחות מ-100 תכונות (גנים) ופחות מ-10 תאים הוסרו. הגרעינים סוננו לפי ספי QC שזוהו: מספר מינימלי של גנים = 200 ומספר מקסימלי של גנים = 8,000, שבר מיטוכונדריאלי <1% ושבר ריבוזומלי <2%. 3,000 הגנים המשתנים ביותר (HVGs) שימשו לניתוח הרכיבים העיקריים (PCA), כפי שהם מיושמים ב-Seurat. אשכולות מבוססי גרף בוצעו לאחר הזנת 15 ממדי המחשב המובילים כתוצאה מניתוח PCA. כדי לקבץ את התאים, יישמנו את טכניקת מיטוב המודולריות (אלגוריתם Louvain) עם פרמטר רזולוציה המוגדר ל-0.5. כדי להמחיש ולחקור מערך נתונים זה, הרצנו הפחתה ממדית לא ליניארית, כלומר קירוב והקרנה של סעפת אחידה (UMAP). הזהות של כל אשכול הוקצתה על סמך ידע מוקדם של הגנים של הסמן 6,20,21.
איור 4A מציג מדדים איכותיים מהנתונים שהתקבלו בשיטה זו של מיצוי גרעינים. UMAP מייצג את מספר הספירות בכל הגרעינים וסוגי התאים העיקריים שניתן לזהות בביטחון רק עם תעתיק גרעיני וריצוף רדוד יחסית (~25,000-40,000 קריאות ממוצעות לתא) (איור 4B). גישה זו מאפשרת לחקור גורמי שעתוק ספציפיים לכבד כגון Hnf4a, Mlxipl ו-Ppara, כמו גם גני מטרה במורד הזרם המעורבים בחילוף החומרים של קסנוביוטיקה (כלומר, Cyp2e1 ו-Cyp2f2)(איור 4C). יש לציין כי הגרעינים שחולצו שמרו מידע קריטי על ייעוד הכבד, כפי שמוצג על ידי התבנית המשלימה של גנים סימן היכר כגון Cyp2e1 pericentral ו- periportal Cyp2f2 (איור 4C).
בנוסף, הערכנו את התאימות של הגרעינים הלא ממוינים שחולצו בשיטה זו עם מבחני מולטיומיקה חדשים יותר ליצירת פרופיל סימולטני של הנוף האפיגנומי (ATAC) וביטוי הגנים (RNA) באותם גרעינים בודדים18. ביצענו אופטימיזציה של זמן הדגירה של הגרעינים ל-5 דקות לפני הטרנספוזיציה. ספריות הריצוף נבנו על ידי הרצת שישה מחזורי PCR עבור הקדם-אמפליפיקציה של cDNA. מהדגימה הקדם-מוגברת נלקחו 35 μL והוגברה עוד יותר על ידי 15 מחזורי PCR לאינדקס הדגימה לבניית ספריית ביטוי הגנים. אלקטרופרוגרמה מייצגת של עקבות ה-cDNA מוצגת באיור 5A (למעלה). לבניית ספריות ATAC, נעשה שימוש ב-40 μL של דגימה קדם-אמפלית והופעלו במשך שישה מחזורי PCR נוספים לאינדקס הדגימה, כאשר האלקטרופרוגרמה המייצגת של עקבות הדנ"א מוצגת באיור 5A (למטה). ספריית ביטוי הגנים (RNA) שנוצרה רוצפה לעומק של 44,600 קריאות לכל גרעין וספריית ATAC לעומק של 43,500 קריאות לכל גרעין (איור 5B).
בדומה לפרוטוקול ריצוף מבוסס טיפות שתואר לעיל, מיפוי קריאה, יישור, הסרת טיפות ריקות וספירת מקטעים בוצעו בהתאם להנחיות סטנדרטיות, כפי שתואר קודם לכן, תוך שימוש בגנום הייחוס GRCm39/mm3918. באמצעות חבילות Seurat ו-Signac22, ביצענו ניתוח "שכנה קרובה משוקללת" (WNN) עבור מדידות מרובות של שני האופנים (RNA + ATAC) (איור 5C), מה שאיפשר לנו לזהות ולבאר הן את סוגי תאי הכבד הגדולים והן את סוגי תאי הכבד הקטנים ללא הטיות בולטות עקב גודל התא או שבריריות גרעינית (איור 5D). הצינור, כפי שפורסם על ידי מעבדת Satija22,23, כולל שלבי QC סטנדרטיים - עיבוד מקדים והפחתה ממדית - נעשה על שני המבחנים באופן עצמאי. כדי לקבל ייצוג טוב של השילוב המשוקלל של אופני ה-RNA-seq וה-ATAC-seq, גרף ה-WNN שרטט ושימש להדמיה, אשכולות וביאור של UMAP בהתבסס על גנים של סמניםשזוהו בעבר 6,20,21. בדומה למבחנים המשתמשים בשיטה יחידה, זיהינו גם מווסתי שעתוק במעלה הזרם (Hnf4a, Ppara, Mlxipl) וגנים סימן היכר של אזור הכבד (Hamp, Cyp2e1 ו-Cyp2f2) (איור 5E).
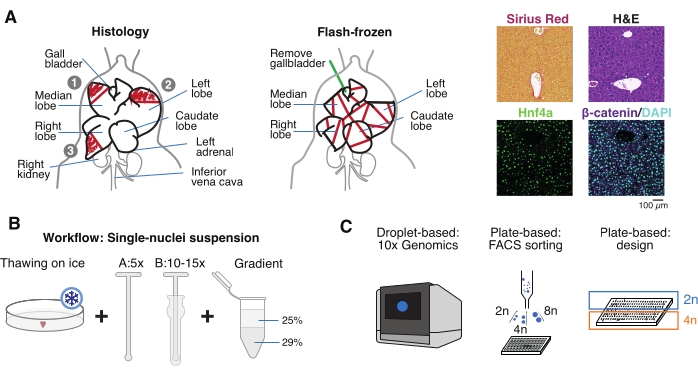
איור 1: סקירה כללית של ניסויים, זרימת עבודה ויישומים גנומיים של תאים בודדים. (A) ייצוג המחשה של דגימת רקמות להיסטולוגיה (משמאל, שלושה חלקים נבחרים להטמעת פרפין ו/או קריוסקציה), איסוף רקמות קפואות הבזק לגנומיקה חד-תאית (באמצע), וניתוחי אימונוהיסטוכימיה ואימונופלואורסצנציה מייצגים (מימין); סרגל קנה מידה = 100 מיקרומטר. (B) שלבים קריטיים עבור מתלים איכותיים בעלי גרעין יחיד. (C) ניתן לטעון את מתלי הגרעינים על שבב כרום פי 10 או להשתמש בהם למיון FACS ולגישות מבוססות צלחת. קיצורים: H&E = המטוקסילין ואאוזין; DAPI = 4',6-diamidino-2-phenylindole; FACS = מיון תאים המופעל על ידי פלואורסצנציה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.

איור 2: דיסקציה של הכבד והומוגניות של רקמת זכוכית Dounce. (A) כבד מורין מייצג מעכבר C57BL6/J בן 3 חודשים (משמאל); קטע הכבד המשמש לבידודים של גרעין יחיד לפני (אמצע) ואחרי טחינת הרקמה עם האזמל (מימין). פסי קנה מידה = 1 ס"מ. (B) תמונות אילוסטרציה של הומוגניזציה של 2 מ"ל מזכוכית Dounce לפני משיכות עם מזיק "רופף" A (משמאל) ואחרי מזיק "הדוק" B (מימין). ניטור הומוגניזציה של רקמות באמצעות המוציטומטר (C) לפני צנטריפוגה הדרגתית ו- (D) לאחר צנטריפוגה הדרגתית. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
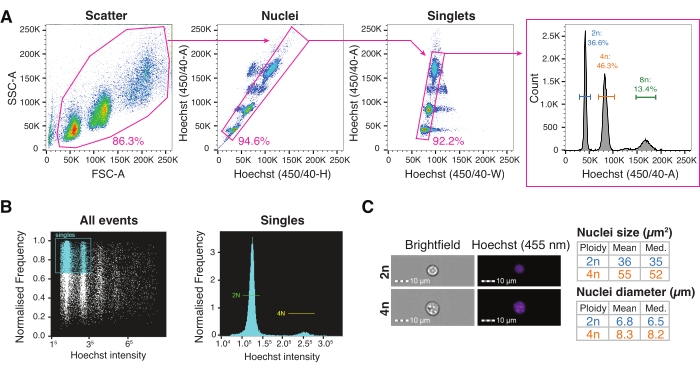
איור 3: מיון תאים המופעלים על ידי פלואורסצנציה וציטומטריה של זרימת הדמיה לאפיון גרעינים בעלי תפוקה גבוהה . (A) אסטרטגיית Gating למיון FACS בעל גרעין יחיד לצלחת לחקירת רמות שונות של פלואידיה. שער פיזור המבוסס על אזור הפיזור הקדמי לעומת אזור הפיזור הצדדי שנקבע כדי לא לכלול פסולת; שער גרעינים המבוסס על Hoechst-Height לעומת Hoechst-Area המאגד אוכלוסיות גרעינים מרובות; שער סינגלטס המבוסס על Hoechst-Width לעומת Hoechst-A שנקבע לאפליה כפולה; ההיסטוגרמה של Hoechst-A מאפשרת הדמיה של פרופיל הגרעינים. (B) כימות ציטומטריה של הדמיה מייצגת של כל האירועים (משמאל) ואירועים בודדים (מימין), המציגה גרעינים דיפלואידים וטטרפלואידים. (C) תמונות ברייטפילד והוכסט של גרעיני 2n ו-4n וכימותם באמצעות ציטומטריה של הדמיה. קיצורים: FACS = מיון תאים המופעל על ידי פלואורסצנציה; FSC-A = אזור פיזור קדימה; SSC-A = אזור פיזור צדדי; Hoechst-H = הוכסט-גובה; Hoechst-A = Hoechst-Area; Hoechst-W = Hoechst-Width. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
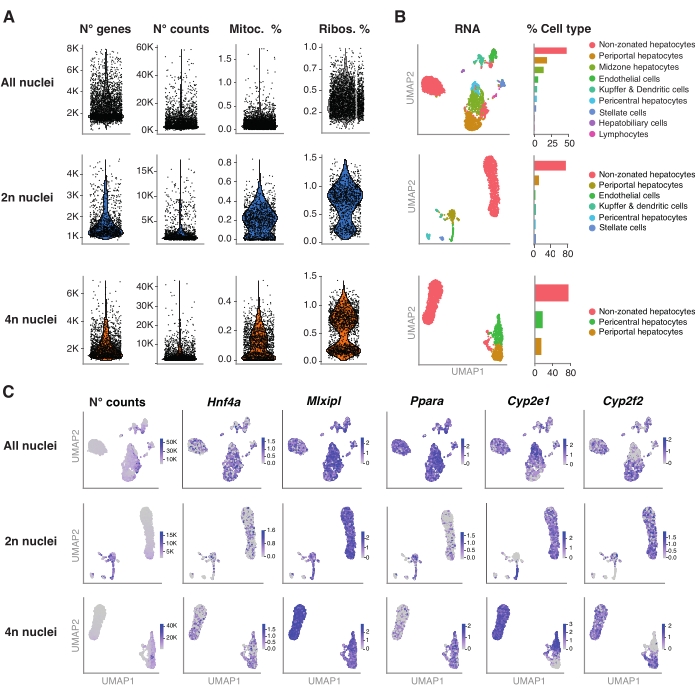
איור 4: אפיון עמוק של פלואידית הפטוציטים עם snRNA-seq . (A) עלילות כינור המציגות את מספר הגנים, הספירות, אחוז הגנים המיטוכונדריאליים ואחוז הגנים הריבוזומליים שזוהו. (B) UMAP מדגים את סוגי התאים שזוהו באמצעות snRNA-seq (משמאל), כאשר הכימות מבוטא באחוז הגרעינים (מימין). (C) UMAP ממחיש את מספר הספירות ומציין את הביטוי של גנים ספציפיים להפטוציטים. כל הגרעינים (שורה עליונה), 2n גרעינים (שורה אמצעית) ו-4n גרעינים (שורה תחתונה). קיצורים: snRNA-seq = גרעין יחיד RNA-seq; UMAP = קירוב והקרנה של סעפת אחידה; מיטוקא. = גנים מיטוכונדריאליים; ריבוס. = גנים ריבוזומליים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
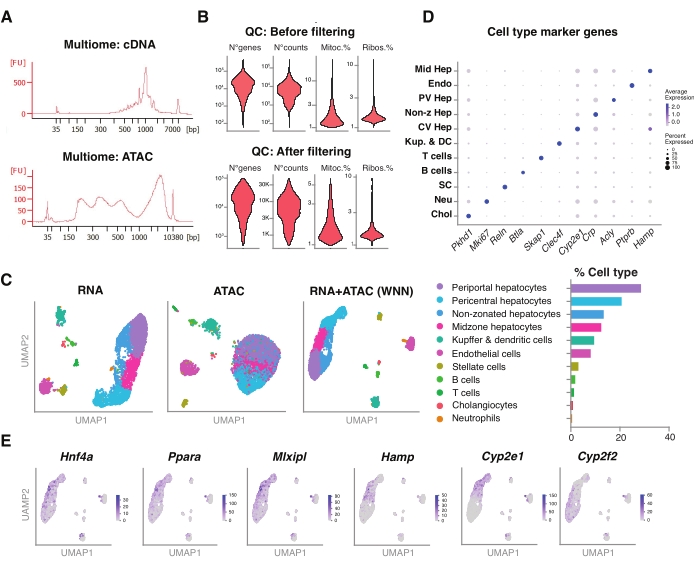
איור 5: בקרת איכות וניתוח של מולטיומיקה (RNA-seq משותף ו-ATAC-seq) מכבדים צעירים קפואים בארכיון. (A) עקבות אלקטרופורטיים אוטומטיים מייצגים המתקבלים לאחר הצינור המולטיומי המציג את תוצר המשקל המולקולרי לאחר סינתזת cDNA ו- ATAC. (B) עלילת כינור המדגימה את מספר הספירות עבור ATAC-seq, RNA-seq, אחוז הגנים המיטוכונדריאליים ואחוז הגנים הריבוזומליים לפני ואחרי הסינון. (C) UMAP מראה את ביטוי הגנים מ-RNA-seq (משמאל), ATAC-seq (באמצע) ומפרקים - RNA-seq ו- ATAC-seq (מימין). (D) סוגי תאים שונים מבוארים בצבעים שונים, עם ביטוי של גנים ספציפיים להפטוציטים. (E) עלילת תכונה המציגה את הביטוי הספציפי לאשכולות של הגנים המצוינים בסוגי התאים שצוינו. קיצורים: ATAC-seq = בדיקה לכרומטין נגיש לטרנספוזאז עם ריצוף בתפוקה גבוהה; אמצע Hep = הפטוציטים באמצע האזור; אנדו = תאי אנדותל; PV Hep = הפטוציטים פריפורטליים; Non-z Hep = הפטוציטים שאינם מיועדים; CV Hep = הפטוציטים פריצנטריים; קופ & DC = קופפר ותאים דנדריטיים; SC = תאים כוכביים, Neu = נויטרופילים; צ'ול = כולנגיוציטים; מיטוקא. = גנים מיטוכונדריאליים; ריבוס. = גנים ריבוזומליים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
| מגיב | מניות | 10 מ"ל | 15 מ"ל | 50 מ"ל | ||
| (A ) יודיקסנול בינוני (IDM) | ||||||
| 250 מ' סוכרוז | 1 דקות | 12.5 מ"ל | ||||
| 150 מ"מ קק"ל | 2 דקות | 3.75 מ"ל | ||||
| 30 מ"מ MgCl2 | 1 דקות | 1.5 מ"ל | ||||
| 60 מ"מ מאגר טריס pH 8.0 | 1 דקות | 3 מ"ל | ||||
| מים נטולי RNase אולטרה-טהורים | 29.25 מ"ל | |||||
| (ב) 50% IDM | ||||||
| יודיקסנול | 60% | 12.5 מ"ל | ||||
| IDM | 2.5 מ"ל | |||||
| (C) 29% IDM | ||||||
| יודיקסנול | 60% | 7.25 מ"ל | ||||
| IDM | 7.75 מ"ל | |||||
| (D ) בידוד גרעינים בינוני-1 (NIM-1) | ||||||
| 250 מ' סוכרוז | 1 דקות | 12.5 מ"ל | ||||
| 25 מ"מ צלזיוס | 2 דקות | 0.625 מ"ל | ||||
| 5 מ"מ MgCl2 | 1 דקות | 0.25 מ"ל | ||||
| 10 מ"מ מאגר טריס pH 8.0 | 1 דקות | 0.5 מ"ל | ||||
| מים אולטרה-טהורים ללא רנאז | 36.125 מ"ל | |||||
| (E ) בידוד גרעינים בינוני-2 (NIM-2) | ||||||
| מאגר NIM-1 | 9.99 מ"ל | |||||
| דיתיותרייטול (DTT) | 1 מ"מ | 0.01 מ"ל | ||||
| טבליה מעכבת פרוטאז (ללא EDTA) | 1 | 1 טבליה | ||||
| (F ) מאגר הומוגניזציה (HB) | ||||||
| מאגר NIM-2 | 9.697 מ"ל | |||||
| מעכב RNase רקומביננטי | 40 U/μL | 0.1 מ"ל | ||||
| מעכב RNAse מבוסס חלבון (SUPERase•IN) | 20 U/μL | 0.1 מ"ל | ||||
| 0.1% טריטון-איקס | 10% | 0.1 מ"ל | ||||
| 3 מיקרוגרם / מ"ל Hoechst 33342 | 10 מ"ג/מ"ל | 0.003 מ"ל | ||||
| (G ) מאגר אחסון גרעינים (NSB) | ||||||
| 166.5 מ"מ סוכרוז | 1 דקות | 1.665 מ"ל | ||||
| 5 מ"מ MgCl2 | 1 דקות | 0.05 מ"ל | ||||
| 10 מ"מ טריס pH 8.0 | 1 דקות | 0.1 מ"ל | ||||
| מעכב RNase רקומביננטי | 40 U/μL | 0.1 מ"ל | ||||
| מעכב RNase על בסיס חלבון (SUPERase•IN) * | 20 U/μL | 0.1 מ"ל | ||||
| מים נטולי RNase אולטרה-טהורים | 8.085 מ"ל | |||||
| * אופציונלי (למיון FACS בלבד) | ||||||
טבלה 1: מתכוני פתרונות. (A) הכנת מדיום יודיקסנול (IDM); (B) דילול של 50% של תמיסת יודיקסנול; (C) דילול של 29% של תמיסת יודיקסנול. (D) הכנת בידוד גרעינים בינוני-1 (NIM-1). (E) הכנת בידוד גרעינים בינוני-2 (NIM-2). (F) הכנת חיץ הומוגניזציה (HB). (G) הכנת מאגר אחסון גרעינים (NSB).
Discussion
ניתוח ההרכב התאי של הכבד על ידי RNA-seq חד-תאי או חד-גרעיני מספק הבנה מעמיקה יותר של התפתחות מחלות כבד והתקדמותן 3,4,5,24. בידוד חד-תאי מכבדים גוזל זמן רב ודורש פרוטוקולים הכוללים דיסוציאציה מכנית או אנזימטית קשה25,26,27. מקובל כי כל רקמה דורשת הערכה שיטתית כדי לקבוע את פרוטוקול הדיסוציאציה האופטימלי של הרקמה, כמו גם שיטת אחסון מתאימה ללכידת סוגי תאים שבירים או גרעינים28. בהתאם לזמינות הרקמות, למחלה המעניינת, לשלב ההתפתחותי או לאורגניזם המודל, הכנת תרחיף בעל גרעין יחיד לעיבוד במורד הזרם עשויה להיות מתודולוגיה מתאימה יותר מאשר שימוש במתלים חד-תאיים. חשוב לציין שבכבד, scRNA-seq ו-snRNA-seq הראו מתאם גבוה בין mRNA גרעיני וציטופלסמי, מה שמרמז על כך ששתי הגישות מציגות מידע משלים 2,3,4,6,29.
מאמר זה מספק בידוד סטנדרטי, חזק וניתן לשחזור של גרעין יחיד מדגימות כבד קפואות בארכיון מעכברים וממינים אחרים, כולל בני אדם וקופי מקוק. ניתן להשתמש בשיטה זו עבור עכברים מסוג בר הניזונים מצ'או ותזונה עתירת שומן (HFD) ולמודלים של עכברים של פיברוזיס בכבד תוך שימוש הן בגישות גנומיות מבוססות צלחת והן בגישות גנומיות מבוססות טיפות6. שיטה זו מסתמכת על הפרוטוקול שתואר במקור עבור רקמת המוח על ידי Krishnaswami et al.30 עם שינויים נוספים המותאמים לכבד קפוא פלאש. הומוגניזציה אופטימלית משחררת את רוב הגרעינים מהרקמה מבלי להשפיע לרעה על שלמות קרום הגרעין. עם זאת, שטיפת יתר עלולה לפגוע בגרעינים השבריריים ולפגוע באיכותם הכללית. כבדים צעירים ו/או שומניים זקוקים בדרך כלל ל-5 פעימות בלבד עם מזיק A ו-10 פעימות עם מזיק B, בעוד כבדים ישנים ו/או פיברוטיים עשויים להזדקק ל-15 פעימות עם מזיק B אך לא יותר. לא מומלץ, אם כן, לבצע משיכות נוספות מעבר למספרים שצוינו כאן. שטיפת יתר עלולה להשפיע לרעה על איכות ההשעיה בעלת הגרעין הבודד ולהגדיל את כמות הרנ"א הסביבתי. לאחר מכן, הדבר עשוי להוביל לצורך בביצוע שלבי סינון חישוביים נוספים במהלך ניתוחי הנתונים במורד הזרם.
הפרוטוקול המוצג כאן הוא רב-תכליתי וניתן להתאים אותו למצבי כבד שונים בעכברים צעירים (3 חודשים) ומבוגרים (24 חודשים). מאחר שמצאנו שחלק גדול יותר של הכבד נחוץ לרקמות ישנות, HFD ופיברוטיות, גודל הרקמה הזמין לעיבוד יכול להוות מגבלה עבור חלק מהמשתמשים עם כמויות קטנות יותר של חומר ביולוגי התחלתי. עם זאת, טיהור הדרגתי מומלץ מאוד לעיבוד דגימה מיידי עם בדיקות גנומיות מבוססות טיפות. אם הגרעינים צריכים להיות ממוינים ל-FACS ללוחות 96/384-well עבור מבחנים מבוססים היטב, ניתן להשמיט טיהור הדרגתי. אנו ממליצים למשתמשים עדיין לבצע את טיהור השיפוע אם יש מספיק דגימת רקמה כדי להשיג את ריכוז הגרעינים המומלץ למיון FACS (כלומר, ~1 × 105 גרעינים למ"ל).
הכבד מאופיין באופי הפוליפלואידי של הפטוציטים9, אך תפקידה של פלואידית הפטוציטים בפיזיולוגיה ומחלה רגילה עדיין לא ברור. יש יותר ויותר עדויות המצביעות על כך שפלואידיה מספקת שונות גנומית31, וידוע כי פלואידיה עולה עם גיל32,33. עם זאת, העשרה של הפטוציטים טטרפלואידים חד-גרעיניים קשורה קלינית גם לפרוגנוזה גרועה בקרצינומה הפטוצלולרית אנושית (HCC)34. באופן דומה, שינויים ברמות פלואידיה של הפטוציטים קשורים למחלות כבד כרוניות הקשורות להזדקנות, כגון מחלת כבד שומני שאינה אלכוהולית (NAFLD)35,36,37. פלואידי הוא מצב של החזקת יותר משני עותקים של הגנום, אשר ניתן לחקור על ידי צביעת תוכן הגנום עם צבע DNA כגון Hoechst38. צבע Hoechst, שנוסף ל-HB לפני המיצוי, מסמן את כל הגרעינים במהלך פרוטוקול הבידוד. זה מאפשר את ההבחנה בין גרעינים דיפלואידים ופוליפלואידים בהתבסס על תכולת הדנ"א שלהם כאשר הם מתרגשים על ידי לייזר UV (350 ננומטר) או סגול (450 ננומטר) במכשיר ציטומטריה של זרימה. בעזרת אסטרטגיית ה-gating המוצגת לראווה, ניתן לחקור רמות 2n, 4n, 8n ורמות גבוהות יותר של פלואידית הפטוציטים בכבדים קפואים בארכיון כדי להבין טוב יותר את התפקיד של הטרוגניות תאית בתפקוד רקמות1 (איור 3A). יתר על כן, ניתן לכמת את המורפולוגיה הגרעינית, כולל הגודל והנפח, באמצעות ציטומטריה של זרימת הדמיה כדי לתאם שינויים בגודל הגרעין עם שינויים במספר הכולל של ספירות או במספר הגנים בהתאם לרמת הפלואידים (איור 3B,C).
מדידת אומיקה רב-מודאלית מציעה את ההזדמנות לחקור מספר שכבות של ארגון גנומי בו זמנית. הגישה המולטיומית המשותפת RNA + ATAC מאפשרת לחקור את הרגולטורים במעלה הזרם וגנים מטבוליים במורד הזרם, ומספקת גישה מקיפה לחקר רשתות שעתוק וארכיטקטורת הכרומטין הקשורה לתפקוד הכבד ברזולוציה של תא בודד. יתר על כן, עם ההתקדמות בשיטות חישוביות שיכולות להסביר את דלילות הנתונים ואת הירידה בעלויות הריצוף, מולטיומיקה חד-תאית היא חלוצה בהערכה של מספר אופנים מאותו תא. פרוטוקול בידוד זה בעל גרעין יחיד תואם להערכה פרטנית ומשותפת של מערכי נתונים של ביטוי וכרומטין. השתמשנו בצינורות סטנדרטיים שנקבעו על ידי Stuart et al.23 (חבילת Signac) כדי להמחיש את איכות הנתונים, בעוד שניתן לאמץ בקלות מספר שיטות חישוביות זמינות וחלופיות לניתוחים במורד הזרם23,39,40,41.
באופן כללי, מולטיומיקה של גרעין יחיד מאפשרת לחקור רקמות כבד של פרימטים בארכיון ביולוגי, עכברי FF, בני אדם ולא אנושיים תוך שימוש בכמות קטנה מאוד של חומר דגימה התחלתי על ידי יישום פרוטוקול מיצוי הגרעין המוצג כאן. כלי רב ערך זה יסמיך ביולוגים של הכבד לחקור הן את ביטוי הגנים והן את נגישות הכרומטין בהקשר של פתולוגיות כבד שונות. בנוסף, רמות שונות של פלואידית הפטוציטים וההתאמה הנובעת מכך של ביטוי גנים בהתאם למיקומם באונה הכבד עשויות לחשוף את תפקידם בפאתולוגיות הכבד. לכן, אנו צופים כי חקירת ההטרוגניות התאית תספק הזדמנויות חדשות לפיתוח רפואה מדויקת והתערבויות ממוקדות נגד מחלות כגון HCC ו- NAFLD.
Disclosures
המחברים מצהירים כי אין להם ניגודי עניינים.
Acknowledgements
מחקר זה נתמך על ידי קמפוס חלוץ הלמהולץ (M.S., K.Y., C.P.M.-J.) והמכון לביולוגיה חישובית (C. T.-L.). מחקר זה נתמך גם על ידי AMED תחת מענק מספר JP20jm0610035 (C.P.M.-J.). אנו מודים לגנומיקה המרכזית ב-HMGU (I. de la Rosa) ובביואינפורמטיקה (T. Walzthoeni), ובמיוחד ל-Xavier Pastor על הכשרה והדרכה. אנו מודים ל- A. Feuchtinger, U. Buchholz, J. Bushe, ולכל שאר אנשי הצוות ממתקן הליבה הפתולוגי והאנליטי של רקמות HMGU על תמיכתם הטכנית והמדעית, כמו גם, J. Zorn, R. Erdelen, D. Würzinger, אנשי הצוות של E-Streifen, כמו גם למתקן הליבה של שירותי חיות המעבדה על התמיכה והדיון המדעיים המתמשכים שלהם. אנו אסירי תודה לניתוח תאי מתקן הליבה ב- TranslaTUM (ר 'מישרה), ו- Luminex, חברת DiaSorin (פ' ריין). אנו מודים לד"ר אי דליגיאניס על תמיכתו הטכנית. ד"ר מ. הרטמן, ד"ר א. שרדר וגב' א. בארדן (קמפוס חלוץ הלמהולץ) היו הבסיס לתמיכתם המשפטית, הניהולית והאדמיניסטרטיבית.
Materials
| Name | Company | Catalog Number | Comments |
| 10% Tween 20 - 5 mL | Bio-Rad | 1662404 | |
| 2100 Bioanalyzer Instrument | Agilent | G2939BA | |
| Adhesive PCR film | Thermo Fisher Scientific | AB0558 | |
| Bioanalyzer High Sensitivity DNA Analysis | Agilent | 5067-4626 | |
| C1000 Touch Thermal Cycler | Bio-Rad | 1851196 | |
| Cell Sorter | For fluoresence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. | ||
| Centrifuge 5430R | Eppendorf | 5428000619 | Use chilled at 4 °C |
| Chromium Controller & Next GEM Accessory Kit | 10X Genomics | 1000204 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | 1000127 | |
| Chromium Next GEM Chip J Single cell kit, 16 reactions | 10X Genomics | 1000230 | |
| Chromium Next GEM Single Cell 3' Kit v3.1, 4 reactions | 10X Genomics | 1000269 | |
| Chromium Next GEM Single Cell Multiome ATAC + Gene Expression Reagent Bundle, 4 reactions | 10X Genomics | 1000285 | |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Sigma Aldrich | 11873580001 | |
| Dithiothreitol (DTT) 1 M solution | Sigma Aldrich | 646563 | Make 1 mM stock solution and use at 1 µM final concentration. |
| DNA AWAY Surface Decontaminant | Thermo Fisher Scientific | 10223471 | Wipe surfaces and pipettes before start of experiment |
| DNA LoBind Tubes, 1.5 mL | Thermo Fisher Scientific | 16628742 | |
| DNA LoBind Tubes, 2.0 mL | Thermo Fisher Scientific | 16638742 | |
| Elution Buffer (EB) - 250 mL | Qiagen | 19086 | |
| Eppendorf ThermoMixer C | Thermo Fisher Scientific | 13527550 | |
| Eppendorf ThermoMixer C Accessory: Smartblock | Thermo Fisher Scientific | 13518470 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade - 100 mL | Thermo Fisher Scientific | 10517694 | |
| Filters 50 µm, sterile | SYSMEX PARTEC - CELLTRICS | 04-004-2327 | Adjust filter diameter according with tissue and nuclei size |
| Glycerin (Glycerol), 50% (v/v) - 1 L | Ricca Chemical Company | 3290-32 | |
| Hard-Shell, 384-Well PCR Plates, thin wall, skirted, clear/white | Bio-Rad | HSP3905 | |
| Herenz Heinz ABS Forceps | Thermo Fisher Scientific | 1131884 | |
| Hoechst 33342, Trihydrochloride, Trihydrate, 10 mg/mL Solution in Water | Invitrogen | H3570 | Light-sensitive |
| Imaging Flow Cytometer | For imaging flow cytometry analysis, e.g. Luminex Amnis ImageStream | ||
| Invitrogen TE Buffer - 100 mL | Thermo Fisher Scientific | 11568846 | |
| KCl (2 M), RNase-free, 100 mL | Thermo Fisher Scientific | AM9640G | |
| MgCl2 (1 M), 100 mL | Thermo Fisher Scientific | AM9530G | |
| MicroAmp 8-Cap Strip, clear-300 strips | Thermo Fisher Scientific | 10209104 | |
| MicroAmp 8-Tube Strip, 0.2 mL-125 strips | Thermo Fisher Scientific | 10733087 | |
| Mörser 2 mL DOUNCE | Wagner & Munz GmbH | 9651632 | RNase zap and rinse with MillQ before use |
| MyFuge 12 Mini MicroCentrifuge C1012 | Benchmark Scientific | C1012 | Or any other strip and tube mini centrifuge |
| Neubauer Hemocytometer | OMNILAB LABORZENTRUM | 5435293 | Visualize and count nuclei under microscope |
| Nuclease-Free Water (not DEPC-Treated) | Thermo Fisher Scientific | AM9937 | |
| OptiPrep Density Gradient Medium | Sigma Aldrich | D1556 | |
| Pipette tips RT LTS 1000 µL, Wide-O | Mettler Toledo | 30389218 | |
| Pistill "A" 2 mL | Wagner & Munz GmbH | 9651621 | RNase zap and rinse with MillQ before use |
| Pistill "B" 2 mL | Wagner & Munz GmbH | 9651627 | RNase zap and rinse with MillQ before use |
| Polypropylene 15 mL Centrifuge Tube | Thermo Fisher Scientific | 10579691 | |
| Polystyrene Petri dish, 60 mm x 15 mm | Thermo Fisher Scientific | 10634141 | |
| Polystyrene Round-Bottom 5 mL FACS Tubes | Thermo Fisher Scientific | 10100151 | |
| Protector RNase inhibitor - 2,000 U | Sigma Aldrich | 3335399001 | Keep in -20 °C until use |
| Protein-based RNase Inhibitor SUPERase•In (20 U/μL) | Thermo Fisher Scientific | AM2696 | Keep in -20 °C until use |
| Recombinant RNase Inhibitor | Clontech Takara | 2313B | Keep in -20 °C until use |
| RNAse free microfuge tubes - 0.5 mL | Thermo Fisher Scientific | AM12450 | |
| RNaseZap RNase Decontamination Solution | Thermo Fisher Scientific | AM9780 | Wipe surfaces and pipettes before start of experiment |
| SPRIselect - 60 mL | Beckman Coulter | B23318 | Aliquot and store in 4 °C |
| Sucrose, 500 g | Sigma Aldrich | S0389-500G | Make a 1 M stock solution |
| Swann-Morton Sterile Disposable Stainless Steel Scalpels | Thermo Fisher Scientific | 11798343 | |
| Tris-HCI (1M), pH 8.0 | Invitrogen | 15568025 | |
| Triton X-100, 98%, 100 mL | Thermo Fisher Scientific | 10671652 | Make 10% stock solution. Keep at 4 °C and protect from light. |
| Trypan Blue Solution, 0.4% | Thermo Fisher Scientific | 11538886 | |
| Vortex- Mixer | VWR | 444-1372 | Or any other type of vortex |
References
- Kamies, R., Martinez-Jimenez, C. P. Advances of single-cell genomics and epigenomics in human disease: where are we now. Mamm Genome. 31 (5-6), 170-180 (2020).
- Ramachandran, P., Matchett, K. P., Dobie, R., Wilson-Kanamori, J. R., Henderson, N. C. Single-cell technologies in hepatology: New insights into liver biology and disease pathogenesis. Nature Reviews Gastroenterology & Hepatology. 17 (8), 457-472 (2020).
- Ramachandran, P., et al. Resolving the fibrotic niche of human liver cirrhosis at single-cell level. Nature. 575 (7783), 512-518 (2019).
- Andrews, T. S., et al. Single-cell, single-nucleus, and spatial RNA sequencing of the human liver identifies cholangiocyte and mesenchymal heterogeneity. Hepatology Communications. 6 (4), 821-840 (2022).
- Xiong, X., et al. Landscape of intercellular crosstalk in healthy and NASH liver revealed by single-cell secretome gene analysis. Molecular Cell. 75 (3), 644-660 (2019).
- Richter, M. L., et al. Single-nucleus RNA-seq2 reveals functional crosstalk between liver zonation and ploidy. Nature Communications. 12 (1), 4264 (2021).
- Matsumoto, T., Wakefield, L., Tarlow, B. D., Grompe, M. In vivo lineage tracing of polyploid hepatocytes reveals extensive proliferation during liver regeneration. Cell Stem Cell. 26 (1), 34-47 (2020).
- Chen, F., et al. Broad distribution of hepatocyte proliferation in liver homeostasis and regeneration. Cell Stem Cell. 26 (1), 27-33 (2020).
- Donne, R., Saroul-Ainama, M., Cordier, P., Celton-Morizur, S., Desdouets, C. Polyploidy in liver development, homeostasis and disease. Nature Reviews. Gastroenterology & Hepatology. 17 (7), 391-405 (2020).
- Lengefeld, J., et al. Cell size is a determinant of stem cell potential during aging. Science Advances. 7 (46), 0271 (2021).
- Lanz, M. C., et al. Increasing cell size remodels the proteome and promotes senescence. Mol Cell. 82 (17), 3255-3269 (2022).
- Kim, J. Y., et al. PIDDosome-SCAP crosstalk controls high-fructose-diet-dependent transition from simple steatosis to steatohepatitis. Cell Metabolism. 34 (10), 1548-1560 (2022).
- Padovan-Merhar, O., et al. Single mammalian cells compensate for differences in cellular volume and DNA copy number through independent global transcriptional mechanisms. Molecular Cell. 58 (2), 339-352 (2015).
- Miettinen, T. P., et al. Identification of transcriptional and metabolic programs related to mammalian cell size. Current Biology. 24 (6), 598-608 (2014).
- Vargas-Garcia, C. A., Ghusinga, K. R., Singh, A. Cell size control and gene expression homeostasis in single-cells. Current Opinion in Systems Biology. 8, 109-116 (2018).
- Knoblaugh, S. E., Randolph-Habecker, J. . Necropsy and histology. In Comparative Anatomy and Histology: A Mouse, Rat, and Human Atlas (Second Edition). , (2018).
- Chromium Next GEM Single Cell 3 Reagent Kits v3.1 User Guide. Document number CG000204 (Rev D). 10x Genomics Available from: https://www.10xgenomics.com/support/single-cell-gene-expression/documentation/steps/library-prep/chromium-single-cell-3-reagent-kits-user-guide-v-3-1-chemistry (2019)
- Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide. Document number CG000338 (Rev D). 10x Genomics Available from: https://www.10xgenomics.com/support/single-cell-multiome-atac-plus-gene-expression/documentation/steps/library-prep/chromium-next-gem-single-cell-multiome-atac-plus-gene-expression-reagent-kits-user-guide (2021)
- MacParland, S. A., et al. Single cell RNA sequencing of human liver reveals distinct intrahepatic macrophage populations. Nature Communications. 9 (1), 4383 (2018).
- Martinez-Jimenez, C. P., Kyrmizi, I., Cardot, P., Gonzalez, F. J., Talianidis, I. Hepatocyte nuclear factor 4alpha coordinates a transcription factor network regulating hepatic fatty acid metabolism. Molecular and Cell Biology. 30 (3), 565-577 (2010).
- Schmidt, D., et al. Five-vertebrate ChIP-seq reveals the evolutionary dynamics of transcription factor binding. Science. 328 (5981), 1036-1040 (2010).
- Hao, Y., et al. Integrated analysis of multimodal single-cell data. Cell. 184 (13), 3573-3587 (2021).
- Stuart, T., Srivastava, A., Madad, S., Lareau, C. A., Satija, R. Single-cell chromatin state analysis with Signac. Nature Methods. 18 (11), 1333-1341 (2021).
- Sampaziotis, F., et al. Cholangiocyte organoids can repair bile ducts after transplantation in the human liver. Science. 371 (6531), 839-846 (2021).
- Gomez-Lechon, M. J., Donato, M. T., Castell, J. V., Jover, R. Human hepatocytes as a tool for studying toxicity and drug metabolism. Current Drug Metabolism. 4 (4), 292-312 (2003).
- Gomez-Lechon, M. J., Donato, M. T., Castell, J. V., Jover, R. Human hepatocytes in primary culture: The choice to investigate drug metabolism in man. Current Drug Metabolism. 5 (5), 443-462 (2004).
- vanden Brink, S. C., et al. Single-cell sequencing reveals dissociation-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Denisenko, E., et al. Systematic assessment of tissue dissociation and storage biases in single-cell and single-nucleus RNA-seq workflows. Genome Biology. 21 (1), 130 (2020).
- Nault, R., Fader, K. A., Bhattacharya, S., Zacharewski, T. R. Single-nuclei RNA sequencing assessment of the hepatic effects of 2,3,7,8-tetrachlorodibenzo-p-dioxin. Cellular and Molecular Gastroenterology and Hepatology. 11 (1), 147-159 (2021).
- Krishnaswami, S. R., et al. Using single nuclei for RNA-seq to capture the transcriptome of postmortem neurons. Nature Protocols. 11 (3), 499-524 (2016).
- Duncan, A. W., et al. Aneuploidy as a mechanism for stress-induced liver adaptation. Journal of Clinical Investigation. 122 (9), 3307-3315 (2012).
- Kreutz, C., et al. Hepatocyte ploidy is a diversity factor for liver homeostasis. Frontiers in Physiology. 8, 862 (2017).
- Hunt, N. J., Kang, S. W. S., Lockwood, G. P., Le Couteur, D. G., Cogger, V. C. Hallmarks of aging in the liver. Computational and Structural Biotechnology Journal. 17, 1151-1161 (2019).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. 69 (2), 355-364 (2019).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Schwartz-Arad, D., Zajicek, G., Bartfeld, E. The streaming liver IV: DNA content of the hepatocyte increases with its age. Liver. 9 (2), 93-99 (1989).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv. B, Cell Pathology Including Molecular Pathology. 64 (6), 387-393 (1993).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Granja, J. M., et al. ArchR is a scalable software package for integrative single-cell chromatin accessibility analysis. Nature Genetics. 53 (3), 403-411 (2021).
- Bredikhin, D., Kats, I., Stegle, O. MUON: Multimodal omics analysis framework. Genome Biology. 23 (1), 42 (2022).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nature Methods. 19 (2), 179-186 (2022).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved