Method Article
シングルセルマルチオミクスのための瞬間凍結肝臓組織からの核の単離
要約
ここでは、一核RNA-seq、ATAC-seq、およびジョイントマルチオミクス(RNA-seqおよびATAC-seq)について、瞬間凍結されたアーカイブされた肝臓組織から核を単離するためのプロトコルを紹介します。
要約
肝臓は、とりわけエネルギー恒常性の維持および生体異物の代謝など、多くの重要な生理学的機能を実行する責任がある複雑で不均一な組織である。これらの作業は、肝実質細胞と非実質細胞の間の緊密な調整によって実行されます。さらに、さまざまな代謝活動が肝小葉の特定の領域に限定されています-肝臓ゾーニングと呼ばれる現象。シングルセルシーケンシング技術の最近の進歩により、研究者はシングルセル分解能で組織の不均一性を調査できるようになりました。肝臓を含む多くの複雑な組織では、過酷な酵素的および/または機械的解離プロトコルが、健康と疾患においてこの臓器を包括的に特徴付けるために必要な単一細胞懸濁液の生存率または品質に悪影響を与える可能性があります。
この論文では、凍結されアーカイブされた肝臓組織から核を単離するための堅牢で再現性のあるプロトコルについて説明します。この方法では、一核RNA-seq、ハイスループットシーケンシングによるトランスポゼアクセス可能なクロマチンのアッセイ(ATAC-seq)、マルチモーダルオミクス(ジョイントRNA-seqおよびATAC-seq)など、下流のシングルセルオミクスアプローチと互換性のある高品質の核が得られます。この方法は、健康および病気のヒト、マウス、および非ヒト霊長類の凍結肝臓サンプルからの核の単離に成功しています。このアプローチは、肝臓のすべての主要な細胞タイプの偏りのない単離を可能にし、したがって、単一細胞分解能で肝臓を研究するための堅牢な方法論を提供します。
概要
シングルセルゲノミクスは、肝機能を研究し、健康と病状における細胞の不均一性の影響を評価するために不可欠な方法論に急速になりつつあります1。異なる情報層を同時に測定するための「マルチオミクス」の急速な発展と、堅牢な計算パイプラインの並行拡張は、正常および病気の肝臓におけるこれまで知られていなかった細胞型とサブタイプの発見への道を開いています2。
バイオバンクとアーカイブされた凍結サンプルを探索する可能性は、非実質細胞の役割を再検討して発見する機会を大幅に増加させました3,4,5、および老化中および慢性疾患における倍数体肝細胞の役割を調査する6,7,8,9 .したがって、この論文では、ダウンストリームの一核RNAシーケンシングおよびATACシーケンシング、およびマルチモーダルオミクス(ジョイントRNA-seqおよびATAC-seq)と互換性のある、瞬間凍結(FF)アーカイブ肝臓用の堅牢で再現性のある単核分離プロトコルについて説明します(図1)。
このワークフローにより、酵素解離プロトコルにおいて、細胞サイズや脆弱性に関係なく、肝臓のすべての細胞タイプのトランスクリプトームとクロマチンアクセシビリティを調査できます。貴重なヒトサンプルまたはトランスジェニックマウスからの小さな組織切片(15〜30 mgまたは5〜10 mm3)で実行できます。核単離の高純度を決定することは、核サイズの定量化および測定を含み、これは細胞サイズおよび老化の増加と相関する可能性がある10,11、そしてこの純度は肝細胞倍数性12および細胞サイズ依存性転写機構の両方の分析に関連している11,13,14,15。.さらに、凍結肝臓から分離された核は、肝臓のゾーニングに関する貴重な情報を保持します。ワークフローと組織収集により、単一細胞ゲノミクスデータの検証や、同じ組織および同じ個体からの免疫組織化学や空間トランスクリプトミクスなどのさらなる補完的な分析が可能になります。したがって、このアプローチは、複数の肝疾患状態およびモデル生物に体系的かつ確実に適用することができる。
プロトコル
すべての動物実験は、ドイツの動物福祉法およびオーバーバイエルン州政府の規則に従って実施されました。動物の飼育は、ドイツ動物福祉法の§11に従って承認され、指令2010/63 / EUに従って実施されました。
1.ティッシュの準備
- 生後3ヶ月から22ヶ月の雄C57BL/6Jマウスを頸部脱臼により屠殺する。動物を解剖板の上に置き、四肢をピンで固定し、70%エタノールで腹部を消毒します。
- Treuting et al.16で推奨されているように剖検を行う。
- 腹部を胸郭まで開き、肝臓を視覚化し、鉗子を使用して小葉を刺さずに慎重に肝臓を取り除きます。
- 横隔膜を鉗子で保持し、ハサミで接続組織を除去することにより、無傷の肝臓を抽出します。
- 臓器を冷たいリン酸緩衝生理食塩水(PBS)で洗い、清潔なペーパータオルで軽くたたいて乾かし、さまざまな目的のために肝小葉をいくつかの部分に切ります:単核分離およびマルチオミクス用のFF。パラフィン包埋用のパラホルムアルデヒド固定(10%PFA)。および/またはさらなる組織学的分析のために最適な切削温度(OCT)コンパウンドに埋め込まれています(図1A)。
- 肝臓片をクライオバイアルまたは5 mLスクリューキャップチューブに分注し、すぐに液体窒素で瞬間凍結します。凍結肝臓サンプルを-80°Cで保存し、下流の単核マルチオミクス実験を行います(図1B、C)。
注意: 凍結保存された組織は、-80°Cで数年間安全に保管でき、使用前に必ず-80°Cのドライアイスで輸送する必要があります。
2. 原子核の分離
- ベンチトップ洗浄とバッファーと消耗品の準備
- 作業用ベンチトップとピペットを70%エタノールとRNase除染溶液で洗浄するか、専用のRNaseフリーのベンチトップと材料を使用してください。
- 以下に詳述するように、スイングバケットと固定角度遠心分離機、1.5 mL/2 mLチューブ、およびマルチウェルプレートの両方を4°Cにプレチルします。
- ティッシュハンドリングに使用されるRNaseフリーの使い捨てピンセット、使い捨て滅菌メス、およびドライアイス(-60°C)のペトリ皿をプレチルします。
- ダウンスガラスホモジナイザーと乳棒を氷上で予冷します(4°C)。各乳棒(A および B)を5mLチューブに入れて、氷との直接接触および潜在的なRNase汚染を避けます。
- ヨージキサノール培地(IDM)の希釈溶液を調製し(表1A)、それを使用して、60%ヨージキサノールストック密度勾配培地(それぞれ表1B および 表1C)の50%および29%希釈を行う。
- すべてのチューブを事前に冷やします。処理するサンプルごとに、次のチューブを準備します。
- 3本の1.5 mL低DNA結合チューブを準備します(1本はろ過した組織ホモジネート用、もう1本目は250 μLの50%希釈ヨージキサノール溶液、もう1本目はクリーンな核懸濁液用)。
- 密度勾配分離用のヨージキサノール溶液の29%希釈液500 μLを含む2 mL丸底チューブを1本準備します。
- 核分離媒体-2(NIM-2)およびホモジナイズバッファー(HB)用の15 mLコニカルチューブを1本用意します。
- 核分離媒体-1(NIM-1)を準備し(表1D)、それを使用してNIM-2(表1E)、続いてHB(表1F)を作成します。両方のRNAse阻害剤は、以下のプロトコルに記載されているように、使用直前に追加してください。
- 表1Gに記載されるように核貯蔵バッファー(NSB)を調製する。使用直前に組換えRNAse阻害剤を添加します。FACSソーティングに使用する前に、タンパク質ベースのRNAse阻害剤をNSBに追加します(オプション)。
- ホモジナイザーの最適な洗浄とメンテナンスのために、組織の均質化に続いてDounceホモジナイザーと乳棒を浸すために、滅菌水で500 mLビーカーを準備します。
- 組織の均質化
- ドライアイスのペトリ皿の内側にプレチルメスで20〜30 mg(または5 mm3)のティッシュ片を切り取ります。その後、直ちにシャーレを湿氷(4°C)に移す。1 mLのHBを加え、冷たいメスを使用して、1 mLの幅の開口部の先端で簡単に吸引できるように、組織をできるだけ細かく刻みます。
注意: すべての組織/核の移動には常に広いオリフィスチップを使用してください。別の方法として、1 mLのチップを滅菌プラスチックカバー上の滅菌メスで切断して、広いオリフィスを生成することができます(図2A)。 - 組織懸濁液を収集し、プレチルド2 mLガラスDounceホモジナイザーに移します(図2B)。
- 追加の0.5〜1 mLのHBでペトリ皿を洗い、すべてを氷上に保ちながら残りの組織片をすべて集めます。
- 氷の上で緩い乳棒 A でゆっくりと慎重に5ストロークを行います。乳棒を上下に引っ張るときに乳棒のねじれ運動を使用して泡を作成することは避けてください。乳棒がストロークごとにホモジナイザーの上部から下部に注意深く移動することを確認します。
- その後、氷上でタイトな乳棒 B で10〜15回のスローストロークを実行します。泡の作成は避けてください。
注:乳棒 B で10ストロークした後、顕微鏡で核を目視検査して、さらにストロークが必要かどうかを確認することをお勧めします。これを行うには、トリパンブルー染色(トリパンとサンプルの比率が1:1)と手動血球計算盤(例:.、10 μLのトリパンブルーを10 μLの核懸濁液と混合し、顕微鏡下での検査に10 μLの混合物を使用します。 図2C)。 - ホモジネートを50 μmのセルストレーナーでろ過し、プレチルした1.5 mLチューブに移します。結合組織の塊を大量に含むホモジネートには、複数のフィルターやチューブを使用してください。
- ホモジナイザーと使用したフィルターを追加の0.5〜1 mLのHBですすぎ、すべての組織ホモジネートを完全に収集します。密度勾配遠心分離に進みます。
- ドライアイスのペトリ皿の内側にプレチルメスで20〜30 mg(または5 mm3)のティッシュ片を切り取ります。その後、直ちにシャーレを湿氷(4°C)に移す。1 mLのHBを加え、冷たいメスを使用して、1 mLの幅の開口部の先端で簡単に吸引できるように、組織をできるだけ細かく刻みます。
- 密度勾配遠心分離
- ろ過したホモジネートを、プレチルした固定角遠心分離機で1,000 × g で4°Cで8分間遠心分離します。
- サンプルを回転させながら、250 μLの50%ヨージキサノール希釈液を含む1.5 mLチューブと、500 μLの29%ヨージキサノール希釈液を含む1本の2 mLチューブを準備します。両方のチューブを氷の上に置いてください。
- 遠心分離後、真空ポンプを用いてペレットを乱すことなく上清を吸引する。
注意: 手動ピペッティングを使用すると、最終的な核懸濁液の品質が低下します。 - 1 mLのワイドオリフィスピペットチップを使用して250 μLのHBをペレットに加え、非常にゆっくりと再懸濁します。
- 250 μLの核懸濁液を、250 μLの50%ヨージキサノール希釈液を含むプレチルド1.5 mLチューブに移し、穏やかに、しかし完全に混合して、25%ヨージキサノール/核懸濁液を生成します。
- 500 μLの25%ヨージキサノール/核懸濁液を、500 μLの29%ヨージキサノール希釈液を含むプレチルド2 mLチューブに移します。
注:500 μLの25%ヨージキサノール/核懸濁液は、25%/29%ヨージキサノール混合物が明確な相分離を示すように、500 μLの29%ヨージキサノール溶液の上に穏やかに堆積させる必要があります。.ピペットチップを45°の角度で配置したチューブ壁の側面を使用して、このグラジエントインターフェースを作成します。それ以降、チューブはこの勾配を乱さないように穏やかに取り扱う必要があります。 - ブレーキをオフにした状態で、12,500gのプレチルスイングバケット遠心分離機でチューブを20分間遠心分離します。
- 遠心分離ステップが完了する直前に、scRNA-seqパイプラインに進む際にRNAse阻害剤をNSBバッファーに追加します( 表1Gを参照)。
- 遠心分離後、真空ポンプを用いてペレットを乱すことなく上清を吸引する。
注意: 手動ピペッティングを使用すると、最終的な核懸濁液の品質が低下します。 - 1 mLのワイドオリフィスピペットチップを使用して、ペレットを100〜300 μLのNSBに静かに再懸濁し、核懸濁液を清潔でプレチルした1.5 mLチューブに移します。
- トリパンブルー溶液(トリパンとサンプルの比率1:1)と手動血球計算盤(例:.、10 μLのトリパンブルーと10 μLの核懸濁液を混合し、10 μLの混合物を使用してカウントします。 図2D)。
- 得られた核懸濁液を直ちに単核ゲノミクスアッセイに使用します。
注:NSB中の核懸濁液は、フローおよび/またはイメージングフローサイトメトリーによるさらなる分析のために、4°Cで最大1週間冷蔵保存できますが、snRNA-seqまたはsnATAC-seqではできません。
3. 肝細胞倍数性プロファイリングまたはウェルベースシーケンシングアプローチのための核ソーティング
- フローサイトメトリーベースのセルソーティングでは、核懸濁液を50 μmフィルターを通してプレチルド5 mL FACSチューブにろ過します。
- 100 μmノズルを備えたフローサイトメトリーソーターを使用してください。FACSチューブをソーターにロードし、サンプルをプレビューします。
- 前方散乱領域対側散乱領域(FSC-A/SSC-A)をプロットしてスキャッターゲートから始め、次にヘキスト高さ対ヘキスト領域(核ゲート)、次にヘキスト幅対ヘキスト領域(シングレットゲート)をプロットして、核ソートのゲーティング戦略を設定します。ヘキスト面積ヒストグラム上の核倍数性プロファイルを視覚化します。
注意: ピーク分解能を向上させるには、ヘキストチャネル(450/50)を線形スケールで視覚化します(図3A)。 - 96/384ウェルプレートにソーティングするには、液滴遅延を設定し、前述のようにベンジジン基質-西洋ワサビペルオキシダーゼ(TMB-HRP)を用いた比色法を使用してプレートアライメントを最適化します6。
- サンプル冷却とプレートホルダーの両方を4°Cで冷却するように設定し、サンプル回転を300rpmでオンにします。
- 単一核を~1 x 105 核/mLのサンプル濃度で、200-500イベント/秒の流速でソートします。
4. イメージングサイトメトリーによる核内パラメータの目視検査と定量(オプション)
- 50 μLの核懸濁液を2 ×10 7 核/mLの濃度で含む0.5 mLチューブをロードします。
- アスペクト比とヘキスト蛍光チャンネルに基づいて 全イベント ゲートを設定し、核倍数性プロファイルを視覚化します(図3B)。
- 明視野(BF)とヘキスト蛍光チャンネルを使用して、40倍の倍率でサンプルを取得します。
- 明視野測定とイメージングサイトメトリーソフトウェアによるヘキスト蛍光強度を使用して、2nおよび4n核を検査および定量します(図3C)。
5. 単一核RNA-seq、ATAC-seq、またはマルチオームライブラリの構築とシーケンシング
- 液滴ベースのsnRNA-seqアプローチでは、精製された核懸濁液をマイクロ流体デバイスに直接ロードして、自動並列分配と分子バーコーディングを行います17。
- 単一細胞分配装置でマイクロ流体ランが完了した後、ゲルビーズ封入核を収集し、インキュベートし、製造業者のガイドライン17で前述したように洗浄する。
- 次のプログラムを使用して、cDNAプリアンプのポリメラーゼ連鎖反応(PCR)サイクルを11回実行します:98°Cで3分(98°Cで15秒、63°Cで20秒、72°Cで1分)x 11、72°Cで1分、4°Cで保持します。 製造元17で示されているように、エンドリペアとAテーリングステップとアダプター結紮を続行します。その後の最終的な遺伝子発現ライブラリー構築のために、次のプログラムを使用して10回のPCRサイクルを実行します:98°Cで45秒、(98°Cで20秒、54°Cで30秒、72°Cで20秒)x 10、72°Cで1分、4°Cで保持します。
- 得られたライブラリを、核あたり~20,000-50,000平均リードの読み取り深度までシーケンスします。
- 関節マルチオミクス(RNA+ATAC)液滴ベースのシーケンシングでは、肝核を溶解バッファー中で5分間インキュベートし、次いで1時間タグ付けします(前述の18)。
- タグ付き核をマイクロ流体デバイスに直接ロードして、自動並列分配と分子バーコーディングを行います。
- マイクロ流体ランが完了した後、ゲルビーズ封入核を収集し、インキュベートし、製造業者18によって記載されるように洗浄する。
- 次のプログラムを使用して、cDNAプリアンプステップの6つのPCRサイクルを実行します:72°Cで5分、98°Cで3分、(98°Cで20秒、63°Cで30秒、72°Cで1分)x 6、72°Cで1分、4°Cで保持します。
- プリアンプサンプルを35 μL採取し、以下のようにcDNA増幅を行う:98°Cで3分、(98°Cで15秒、63°Cで20秒、72°Cで1分)×6、72°Cで1分、4°Cで保持する。 製造元18 が示すように、エンドリペアとAテーリングステップとアダプター結紮を続行します。その後の最終サンプルインデックスPCRでは、98°Cで45秒(98°Cで20秒、54°Cで30秒、72°Cで20秒)x 15、72°Cで1分、4°Cで保持するように15回のPCRサイクルを実行します。
- ATACライブラリの構築には、40 μLを使用し、次のプログラムを使用してサンプルインデックス作成用の6つのPCRサイクルで増幅します:98°Cで45秒(98°Cで20秒、67°Cで30秒、72°Cで20秒)x 6、72°Cで1分、4°Cで保持します。
- 得られたマルチオーム遺伝子発現ライブラリを、核あたり20,000リードペアの最小リード深さに配列決定し、マルチオームATACライブラリを細胞あたり25,000リードペアの最小リード深さに配列決定します(製造元18)。
結果
凍結肝臓サンプルからの単核単離のワークフローは、単核マルチオミクス用に調整されており、i)細胞の不均一性と組織構造の並行解析のためのサンプル収集、ii)単核懸濁液、およびiii)単核マルチオミクス(図1).抽出された肝臓は安楽死させたマウスから解剖され、パラフィン包埋、凍結切片、またはその両方の組織学的検査のために断片に切断されます。他の切断片は、マルチオミクス分析のために下流の単核単離のために液体窒素中で直ちに瞬間凍結されます。この組織採取システムにより、ユーザーは同じ個体の組織切片の単一核オミクスデータをさらに検証することができ、必要に応じて空間トランスクリプトミクスまたは免疫組織化学分析でデータセットを補完することができます。
ここで説明した方法で凍結肝臓から抽出した核の顕微鏡検査は、密度勾配遠心分離ステップが不要な細胞および組織の破片の除去を大幅に促進することを示しています(図2C、D)。さらに、この方法論では、サイトメトリー分析によって検証および定量化できるすべてのレベルの倍数性を保持します(図3)。
このプロトコルの性能をさらに検証するために、未選別およびFACS選別された核に対して液滴ベースのsnRNA-seqを実施し、Seuratパイプラインに従ってデータを分析しました。簡単に説明すると、抽出された単一核は、前述の19のように液滴ベースのsnRNA−seqのために調製された。2nおよび4n核以上の倍数性のsnRNA-seqについては、cDNA増幅ステップに11サイクル、最終的な遺伝子発現ライブラリ構築に10サイクルを使用しました。得られたライブラリは、核あたり~25,000-39,000平均リードの読み取り深度に配列決定されました。得られた単一核リードは、GRCm39/mm39マウスゲノムにマッピングされました。前処理パイプラインを実行するときに、核内に存在するスプライスされていないメッセンジャーRNA(mRNA)の包含と定量を確認するために、コマンド- -include-intron が追加されました。アライナー内に組み込まれた EmptyDrops アルゴリズムは、空の液滴をろ過して除去しました。
RパッケージSeurat(バージョン4.1.1)は、snRNA-seq分析パイプラインによって出力された一意の分子識別子(UMI)カウントマトリックスを使用して品質管理(QC)メトリックを計算するために使用されました。100未満の特徴(遺伝子)および10未満の細胞を有するカウントは除去された。核は、同定されたQC閾値に従ってフィルタリングされた:遺伝子の最小数= 200および遺伝子の最大数= 8,000、ミトコンドリア画分<1%およびリボソーム画分<2%。上位3,000の高可変遺伝子(HVG)は、スーラで実装された主成分分析(PCA)に使用されました。グラフベースのクラスタリングは、PCA分析の結果得られた上位15個のPCディメンションを入力した後に実行されました。セルをクラスター化するために、解像度パラメーターを0.5に設定してモジュール性最適化手法(Louvain アルゴリズム)を適用しました。このデータセットを視覚化して探索するために、非線形次元削減、つまり一様多様体近似および投影(UMAP)を実行しました。各クラスターの同一性は、マーカー遺伝子6、20、21の事前知識に基づいて割り当てた。
図4Aは、この核抽出方法を用いて得られたデータからの高品質の指標を示す。UMAPは、核トランスクリプトームと比較的浅いシーケンシング(細胞あたり~25,000-40,000平均リード)でのみ確実に識別できるすべての核と主要な細胞タイプのカウント数を表します(図4B)。このアプローチにより、Hnf4a、Mlxipl、Pparaなどの肝臓特異的転写因子や、生体異物の代謝に関与する下流の標的遺伝子(すなわち、Cyp2e1およびCyp2f2)の調査が可能になります(図4C)。 注目すべきことに、抽出された核は、中心周囲Cyp2e1や門脈周囲Cyp2f2などの特徴的な遺伝子の相補パターンによって示されるように、肝臓のゾーニングに関する重要な情報を保持していました(図4C)。
さらに、この方法を使用して抽出された未分類の核と、同じ単一核内のエピゲノムランドスケープ(ATAC)と遺伝子発現(RNA)の同時プロファイリングのための新しいマルチオミクスアッセイとの適合性を評価しました18。核溶解インキュベーション時間を転位の5分前に最適化しました。シーケンシングライブラリは、cDNAプリアンプのために6つのPCRサイクルを実行することによって構築されました。プリアンプサンプルから35 μLを採取し、サンプルインデックス作成のために15 PCRサイクルでさらに増幅して、遺伝子発現ライブラリーを構築しました。cDNAトレースの代表的なエレクトロフェログラムを 図5A (上)に示す。ATACライブラリの構築には、40 μLのプリアンプサンプルを使用し、サンプルインデックス作成のためにさらに6回のPCRサイクルを実行し、DNAトレースの代表的な電気泳動図を 図5A (下)に示します。得られた遺伝子発現ライブラリー(RNA)を核当たり44,600リードの深さまで配列決定し、ATACライブラリーを核当たり43,500リードの深さまで配列決定した(図5B)。
上記のシングルモダリティ、液滴ベースのシーケンシングプロトコルと同様に、GRCm39/mm39参照ゲノム18を使用して、前述のように、リードマッピング、アライメント、空のドロップ除去、およびフラグメントカウントを実行しました。SeuratおよびSignacパッケージ22を使用して、両方のモダリティ(RNA+ATAC)の複数の測定に対して「加重最近傍」(WNN)分析を実行し(図5C)、細胞サイズまたは核の脆弱性による顕著なバイアスなしに、主要肝細胞タイプとマイナー肝細胞タイプの両方を識別して注釈を付けることができました(図5D)。Satijaラボ22,23によって公開されているパイプラインには、両方のアッセイで独立して行われる標準的なQCステップ(前処理と寸法削減)が含まれています。RNA-seqとATAC-seqモダリティの重み付けされた組み合わせを適切に表現するために、WNNグラフをプロットし、以前に同定されたマーカー遺伝子6、20、21に基づいてUMAPの視覚化、クラスタリング、および注釈付けに使用しました。シングルモダリティを使用したアッセイと同様に、上流の転写調節因子(Hnf4a、Ppara、Mlxipl)および肝ゾーニングの特徴的な遺伝子(Hamp、Cyp2e1、およびCyp2f2)も検出しました(図5E)。
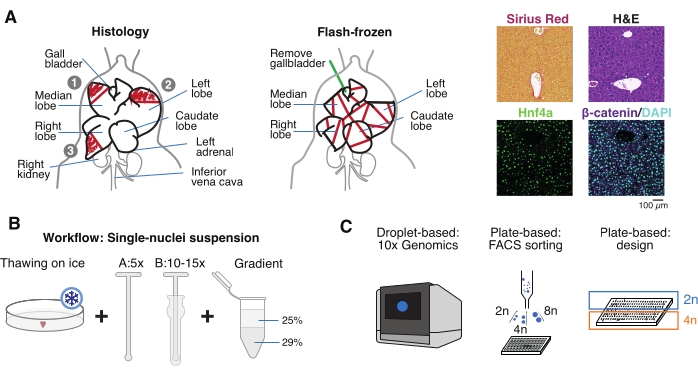
図1:実験の概要、ワークフロー、および単一細胞ゲノムアプリケーション。 (A)組織学のための組織サンプリングの例示的表現(左、パラフィン包埋および/または凍結切片のために3つのセクションが選択されています)、単一細胞ゲノミクスのための瞬間凍結組織収集(中央)、および代表的な免疫組織化学および免疫蛍光分析(右)。スケールバー = 100 μm。 (B)高品質の単核懸濁液のための重要なステップ。(C)核懸濁液は、10倍クロムチップに装填するか、FACS選別およびプレートベースのアプローチに使用できます。略語:H&E =ヘマトキシリンとエオジン;DAPI = 4',6-ジアミジノ-2-フェニルインドール;FACS = 蛍光活性化細胞選別。 この図の拡大版を表示するには、ここをクリックしてください。

図2:肝解離とダウンスガラス組織の均質化。 (A)生後3ヶ月のC57BL6/Jマウスの代表的なマウス肝臓(左)。メスで組織を細切する前(中央)と切開後の単核分離に使用した肝臓切片(右)。スケールバー= 1 cm。 (B)「緩い」乳棒 A (左)と「堅い」乳棒 B (右)によるストローク前の2 mLのダウンスガラス均質化の実例画像。血球計算盤を用いて組織ホモジナイズをモニタリングする(C)勾配遠心分離前および(D)勾配遠心分離後。 この図の拡大版を表示するには、ここをクリックしてください。
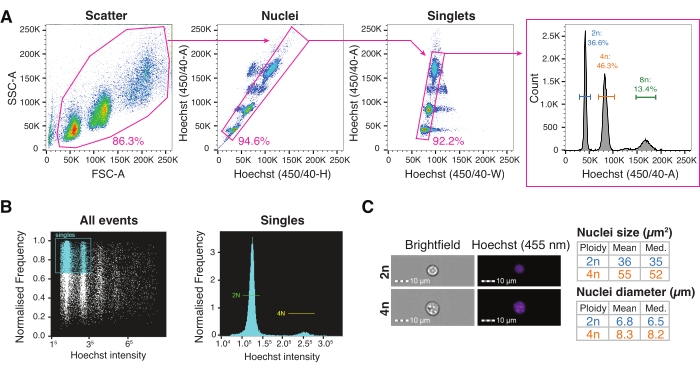
図3:ハイスループット核の特性評価のための蛍光活性化細胞ソーティングおよびイメージングフローサイトメトリー 。 (A)異なるレベルの倍数性の調査のためのプレートへの単核FACSソーティングのためのゲーティング戦略。前方散乱領域とデブリを除外するように設定された側面散乱領域に基づく散布ゲート。複数の核集団を組み込んだヘキスト高さ対ヘキスト面積に基づく核ゲート。ダブレット識別のためのヘキスト幅対ヘキストA集合に基づくシングレットゲート。Hoechst-Aヒストグラムは、核倍数性プロファイルの視覚化を可能にします。(B)すべての事象(左)および単一事象(右)の代表的なイメージングサイトメトリー定量、二倍体および四倍体核を示す。(C)2nおよび4n核の明視野およびヘキスト画像とイメージングサイトメトリーを用いたそれらの定量。略語:FACS =蛍光活性化細胞選別;FSC-A = 前方散乱領域;SSC-A = 側方散乱領域;ヘキスト-H = ヘキスト-高さ;ヘキスト-A = ヘキスト-エリア;ヘキスト-W = ヘキスト幅。 この図の拡大版を表示するには、ここをクリックしてください。
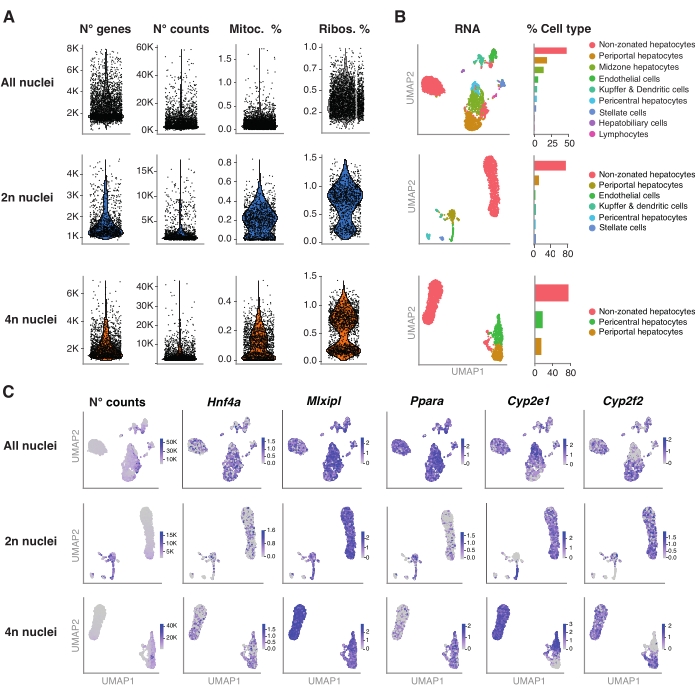
図4:snRNA-seqによる肝細胞倍数性の詳細な特性評価 。 (A)遺伝子の数、数、ミトコンドリア遺伝子の割合、および検出されたリボソーム遺伝子の割合を示すバイオリンプロット。(B)snRNA-seqを用いて検出された細胞型を示すUMAP(左)、定量を核の割合で表す(右)。(C)UMAPは、肝細胞特異的遺伝子のカウント数を示し、発現を示した。すべての原子核(上段)、2n個の原子核(中段)、および4n個の原子核(下段)。略語:snRNA-seq = 一核RNA-seq;UMAP = 一様多様体の近似と射影;ミトック。=ミトコンドリア遺伝子;リボ。=リボソーム遺伝子。 この図の拡大版を表示するには、ここをクリックしてください。
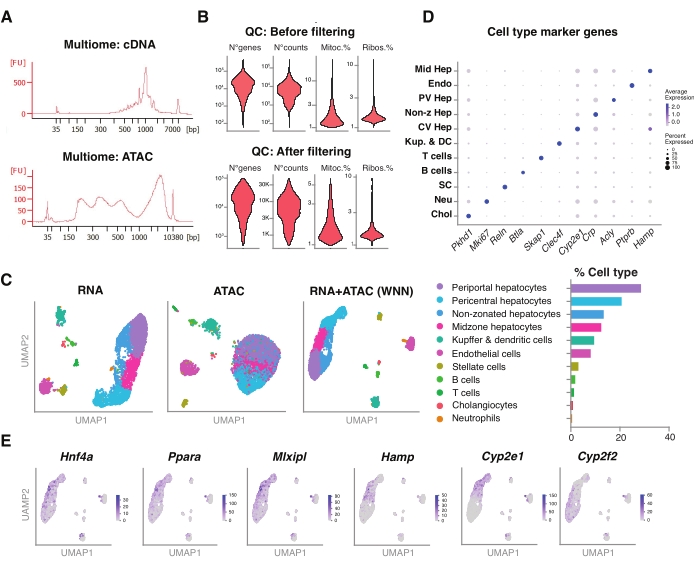
図5:凍結されアーカイブされた若い肝臓からのマルチオミクス(ジョイントRNA-seqおよびATAC-seq)の品質管理と分析 。 (a)マルチオミックパイプラインの後に得られた代表的な自動電気泳動痕跡、cDNA合成後の分子量積およびATACを示す。(B)ATAC-seq、RNA-seq、ミトコンドリア遺伝子の割合、およびフィルタリング前後のリボソーム遺伝子の割合のカウント数を示すバイオリンプロット。(C)UMAPは、RNA-seq(左)、ATAC-seq(中央)、および関節モダリティ-RNA-seqおよびATAC-seq(右)からの遺伝子発現を示す。(D)異なる細胞タイプは、示された肝細胞特異的遺伝子の発現とともに、異なる色で注釈が付けられます。(e)示された細胞型における示された遺伝子のクラスター特異的発現を示す特徴プロット。略語:ATAC-seq = ハイスループットシーケンシングによるトランスポザーゼアクセス可能なクロマチンのアッセイ。ミッドヘップ=ミッドゾーン肝細胞;遠藤=内皮細胞;PV Hep =門脈周囲肝細胞;非zHep =非ゾーニング肝細胞;CVヘップ=中心周囲肝細胞;Kup & DC = Kupffer & 樹状細胞;SC =星細胞、ノイ=好中球;チョル=胆管細胞;ミトック。=ミトコンドリア遺伝子;リボ。=リボソーム遺伝子。 この図の拡大版を表示するには、ここをクリックしてください。
| 試薬 | 株式 | 10ミリリットル | 15ミリリットル | 50ミリリットル | ||
| (A)ヨージキサノール培地(IDM) | ||||||
| 250 mM ショ糖 | 1M | 12.5ミリリットル | ||||
| 150 ミリリットル KCl | 2M | 3.75ミリリットル | ||||
| 30 mM マグネシウムCl2 | 1M | 1.5ミリリットル | ||||
| 60 mM トリス緩衝液 pH 8.0 | 1M | 3ミリリットル | ||||
| 超高純度RNaseフリーウォーター | 29.25 ミリリットル | |||||
| (B) 50 % IDM | ||||||
| ヨージキサノール | 60% | 12.5ミリリットル | ||||
| ティッカー | 2.5ミリリットル | |||||
| (C) 29% IDM | ||||||
| ヨージキサノール | 60% | 7.25ミリリットル | ||||
| ティッカー | 7.75ミリリットル | |||||
| (D ) 核分離媒体-1 (NIM-1) | ||||||
| 250 mM ショ糖 | 1M | 12.5ミリリットル | ||||
| 25 ミリリットル KCl | 2M | 0.625 ミリリットル | ||||
| 5 mM マグネシウムCl2 | 1M | 0.25 ミリリットル | ||||
| 10 mM トリス緩衝液 pH 8.0 | 1M | 0.5ミリリットル | ||||
| 超高純度ルナーゼフリー水 | 36.125 ミリリットル | |||||
| (E ) 核分離媒体-2 (NIM-2) | ||||||
| NIM-1 バッファー | 9.99 ミリリットル | |||||
| ジチオスレイトール(DTT) | 1ミリリットル | 0.01ミリリットル | ||||
| プロテアーゼ阻害剤錠(EDTAフリー) | 1 | 1錠 | ||||
| (F ) ホモジナイズバッファー(HB) | ||||||
| NIM-2 バッファー | 9.697 ミリリットル | |||||
| 組換えRNase阻害剤 | 40 U/μL | 0.1ミリリットル | ||||
| タンパク質ベースのRNAse阻害剤(スーパーアーゼ•IN) | 20 U/μL | 0.1ミリリットル | ||||
| 0.1% トリトン-X | 10% | 0.1ミリリットル | ||||
| 3 μg/mL ヘキスト 33342 | 10ミリグラム/ミリリットル | 0.003ミリリットル | ||||
| (G ) 核貯蔵バッファー (NSB) | ||||||
| 166.5 mM スクロース | 1M | 1.665ミリリットル | ||||
| 5 mM マグネシウムCl2 | 1M | 0.05ミリリットル | ||||
| 10 mM トリス pH 8.0 | 1M | 0.1ミリリットル | ||||
| 組換えRNase阻害剤 | 40 U/μL | 0.1ミリリットル | ||||
| タンパク質ベースのRNase阻害剤(スーパーアーゼ•IN)* | 20 U/μL | 0.1ミリリットル | ||||
| 超高純度RNaseフリーウォーター | 8.085 ミリリットル | |||||
| * オプション (FACS ソートのみ) | ||||||
表1:ソリューションレシピ。 (a)ヨージキサノール培地(IDM)の調製;(b)ヨージキサノール溶液の50%希釈;(c)ヨージキサノール溶液の29%希釈。(d)核分離培地-1(NIM-1)の調製。(e)核分離培地-2(NIM-2)の調製。(f)ホモジナイズバッファー(HB)の調製。(g)核貯蔵バッファー(NSB)の調製。
ディスカッション
単一細胞または単核RNA-seqによって肝臓の細胞組成を解剖することで、肝疾患の発症と進行をより深く理解することができます3、4、5、24。肝臓からの単一細胞単離は時間がかかり、過酷な機械的または酵素的解離を伴うプロトコルが必要です25,26,27。すべての組織が最適な組織解離プロトコルを決定するための体系的な評価、および脆弱な細胞型または核を捕捉するための適切な保存方法を必要とすることは広く受け入れられています28。組織の入手可能性、関心のある疾患、発生段階、またはモデル生物によっては、下流処理用の単核懸濁液の調製が、単一細胞懸濁液を使用するよりも適切な方法論である可能性があります。重要なことに、肝臓では、scRNA-seqとsnRNA-seqが核内mRNAと細胞質mRNAの間に高い相関を示しており、両方のアプローチが相補的な情報を提示することを示唆しています2,3,4,6,29。
この論文は、マウスおよびヒトやマカクを含む他の種からの凍結およびアーカイブされた肝臓サンプルからの標準化された堅牢で再現性のある単核単離を提供します。この方法は、固形飼料と高脂肪食(HFD)を与えられた野生型マウス、およびウェルプレートベースおよび液滴ベースの単核ゲノムアプローチの両方を使用した肝線維症のマウスモデルに使用できます6。この方法は、Krishnaswami et al.30によって脳組織について最初に記述されたプロトコルに依存しており、瞬間凍結肝臓用に調整された追加の変更が加えられています。最適な均質化は、核膜の完全性に悪影響を与えることなく、組織からほとんどの核を解放します。ただし、過度に跳ね返ると、壊れやすい原子核が損傷し、全体的な品質が低下する可能性があります。若年および/または脂肪肝は通常、乳棒Aで5ストローク、乳棒Bで10ストロークのみを必要としますが、老人および/または線維性肝臓は乳棒Bで15ストロークを必要とする場合がありますが、それ以上は必要ありません。したがって、ここに示されている数を超えてストロークを実行することはお勧めしません。過剰に跳ね返ると、単核懸濁液の品質に悪影響を及ぼし、周囲RNAの量を増加させる可能性があります。その後、これにより、ダウンストリームデータ分析中に追加の計算フィルタリング手順を実行する必要が生じる可能性があります。
ここで紹介するプロトコルは用途が広く、若齢(3か月)および老齢(24か月)のマウスのさまざまな肝臓の状態に合わせて調整できます。肝臓のより大きな部分が古い組織、HFD、および線維性組織に必要であることがわかったため、処理に利用できる組織のサイズは、出発生物材料の量が少ない一部のユーザーにとって制限をもたらす可能性があります。ただし、液滴ベースのゲノムアッセイによる即時サンプル処理には、グラジエント精製を強くお勧めします。ウェルベースのアッセイのために核を96ウェル/384ウェルプレートにFACSソーティングする必要がある場合は、グラジエント精製を省略できます。FACSソーティングに推奨される核濃度(~1 ×10 5 nuclei/mL)を得るのに十分な組織サンプルがある場合は、グラジエント精製を実行することをお勧めします。
肝臓は肝細胞の倍数体の性質によって特徴付けられる9が、正常な生理機能および疾患における肝細胞倍数性の役割はまだ明らかではない。倍数性がゲノムの多様性を提供することを示す証拠が増えており31、倍数性が32,33歳とともに増加することはよく知られています。しかし、単核四倍体肝細胞の濃縮は、ヒト肝細胞癌(HCC)の予後不良と臨床的にも関連しています34。同様に、肝細胞の倍数性レベルの変化は、非アルコール性脂肪性肝疾患(NAFLD)などの加齢関連の慢性肝疾患に関連しています35,36,37。倍数性は、ゲノムのコピーを2つ以上所有している状態であり、ゲノムの内容をHoechst38などのDNA色素で染色することで探索できます。抽出前にHBに添加されるヘキスト色素は、単離プロトコル中にすべての核を標識します。これにより、フローサイトメトリー装置でUV(350 nm)または紫色(450 nm)レーザーで励起したときのDNA含有量に基づいて、二倍体核と倍数体核を区別することができます。紹介されたゲーティング戦略により、2n、4n、8n、およびそれ以上のレベルにある肝細胞の倍数性を凍結されたアーカイブされた肝臓で調査して、組織機能における細胞の不均一性の役割をよりよく理解することができます1(図3A)。さらに、サイズと体積を含む核形態をイメージングフローサイトメトリーを使用して定量し、核サイズの変化と、倍数性レベルに応じた総数数または遺伝子数の変化を相関させることができます(図3B、C)。
マルチモーダルオミクス測定は、ゲノム構成の複数の層を同時に調査する機会を提供します。共同RNA + ATACマルチオミクスアプローチは、上流の調節因子と下流の代謝遺伝子の調査を可能にし、単一細胞分解能で肝機能に関連する転写ネットワークとクロマチン構造を研究するための包括的なアプローチを提供します。さらに、データのスパース性とシーケンシングコストの削減を説明できる計算方法の進歩により、シングルセルマルチオミクスは、同じセルからの複数のモダリティの評価の先駆者です。この単核分離プロトコルは、発現およびクロマチンデータセットの個別および共同評価と互換性があります。我々は、Stuart et al.23(Signacパッケージ)によって確立された標準パイプラインを使用してデータの品質を説明しましたが、いくつかの利用可能な代替計算方法は、ダウンストリーム分析に簡単に採用できます23、39、40、41。
全体として、単核マルチオミクスは、ここに示されている核抽出プロトコルを実装することにより、非常に少量の出発サンプル材料を使用して、バイオアーカイブされたFFマウス、ヒト、および非ヒト霊長類の肝臓組織の調査を可能にします。この非常に貴重なツールは、肝臓生物学者がさまざまな肝臓の病状の文脈で遺伝子発現とクロマチンアクセシビリティの両方を調査することを可能にします。さらに、さまざまなレベルの肝細胞倍数性と、その結果としての肝小葉内の位置に応じた遺伝子発現の調整により、肝臓の病状におけるそれらの役割が明らかになる可能性があります。したがって、細胞の不均一性の調査は、HCCやNAFLDなどの疾患に対する精密医療と標的介入の開発のための新しい機会を提供すると期待しています。
開示事項
著者は、利益相反がないことを宣言します。
謝辞
この研究は、ヘルムホルツパイオニアキャンパス(M.S.、K.Y.、C.P.M.-J.)および計算生物学研究所(C.T.-L.)の支援を受けました。この研究は、AMEDの助成金番号JP20jm0610035(C.P.M.-J.)の支援も受けています。HMGU(I. de la Rosa)のコアゲノミクスとBioinformatics(T. Walzthoeni)のサポート、特にトレーニングとガイダンスを提供してくれたザビエル牧師に感謝します。A.フォイヒティンガー、U.ブッフホルツ、J.ブッシュ、およびHMGU病理学および組織分析コア施設の他のすべてのスタッフの技術的および科学的サポート、およびE-StreifenのスタッフであるJ.ゾーン、R.エルデレン、D.ヴュルツィンガー、および実験動物サービスのコア施設の継続的な科学的サポートと議論に感謝します。TranslaTUM(R. Mishra)とIlluminationex, A DiaSorin Company(P. Rein)のコアファシリティセルアナリシスに感謝しています。I Deligiannis博士の技術サポートに感謝します。M.ハートマン博士、A.シュレーダー博士、A.バーデン氏(ヘルムホルツパイオニアキャンパス)は、法的、管理的、および管理上のサポートの基本でした。
資料
| Name | Company | Catalog Number | Comments |
| 10% Tween 20 - 5 mL | Bio-Rad | 1662404 | |
| 2100 Bioanalyzer Instrument | Agilent | G2939BA | |
| Adhesive PCR film | Thermo Fisher Scientific | AB0558 | |
| Bioanalyzer High Sensitivity DNA Analysis | Agilent | 5067-4626 | |
| C1000 Touch Thermal Cycler | Bio-Rad | 1851196 | |
| Cell Sorter | For fluoresence-activated cell sorting (FACS); e.g. BD FACSAria Cell Sorter. | ||
| Centrifuge 5430R | Eppendorf | 5428000619 | Use chilled at 4 °C |
| Chromium Controller & Next GEM Accessory Kit | 10X Genomics | 1000204 | |
| Chromium Next GEM Chip G Single Cell Kit | 10X Genomics | 1000127 | |
| Chromium Next GEM Chip J Single cell kit, 16 reactions | 10X Genomics | 1000230 | |
| Chromium Next GEM Single Cell 3' Kit v3.1, 4 reactions | 10X Genomics | 1000269 | |
| Chromium Next GEM Single Cell Multiome ATAC + Gene Expression Reagent Bundle, 4 reactions | 10X Genomics | 1000285 | |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Sigma Aldrich | 11873580001 | |
| Dithiothreitol (DTT) 1 M solution | Sigma Aldrich | 646563 | Make 1 mM stock solution and use at 1 µM final concentration. |
| DNA AWAY Surface Decontaminant | Thermo Fisher Scientific | 10223471 | Wipe surfaces and pipettes before start of experiment |
| DNA LoBind Tubes, 1.5 mL | Thermo Fisher Scientific | 16628742 | |
| DNA LoBind Tubes, 2.0 mL | Thermo Fisher Scientific | 16638742 | |
| Elution Buffer (EB) - 250 mL | Qiagen | 19086 | |
| Eppendorf ThermoMixer C | Thermo Fisher Scientific | 13527550 | |
| Eppendorf ThermoMixer C Accessory: Smartblock | Thermo Fisher Scientific | 13518470 | |
| Ethanol, Absolute (200 Proof), Molecular Biology Grade - 100 mL | Thermo Fisher Scientific | 10517694 | |
| Filters 50 µm, sterile | SYSMEX PARTEC - CELLTRICS | 04-004-2327 | Adjust filter diameter according with tissue and nuclei size |
| Glycerin (Glycerol), 50% (v/v) - 1 L | Ricca Chemical Company | 3290-32 | |
| Hard-Shell, 384-Well PCR Plates, thin wall, skirted, clear/white | Bio-Rad | HSP3905 | |
| Herenz Heinz ABS Forceps | Thermo Fisher Scientific | 1131884 | |
| Hoechst 33342, Trihydrochloride, Trihydrate, 10 mg/mL Solution in Water | Invitrogen | H3570 | Light-sensitive |
| Imaging Flow Cytometer | For imaging flow cytometry analysis, e.g. Luminex Amnis ImageStream | ||
| Invitrogen TE Buffer - 100 mL | Thermo Fisher Scientific | 11568846 | |
| KCl (2 M), RNase-free, 100 mL | Thermo Fisher Scientific | AM9640G | |
| MgCl2 (1 M), 100 mL | Thermo Fisher Scientific | AM9530G | |
| MicroAmp 8-Cap Strip, clear-300 strips | Thermo Fisher Scientific | 10209104 | |
| MicroAmp 8-Tube Strip, 0.2 mL-125 strips | Thermo Fisher Scientific | 10733087 | |
| Mörser 2 mL DOUNCE | Wagner & Munz GmbH | 9651632 | RNase zap and rinse with MillQ before use |
| MyFuge 12 Mini MicroCentrifuge C1012 | Benchmark Scientific | C1012 | Or any other strip and tube mini centrifuge |
| Neubauer Hemocytometer | OMNILAB LABORZENTRUM | 5435293 | Visualize and count nuclei under microscope |
| Nuclease-Free Water (not DEPC-Treated) | Thermo Fisher Scientific | AM9937 | |
| OptiPrep Density Gradient Medium | Sigma Aldrich | D1556 | |
| Pipette tips RT LTS 1000 µL, Wide-O | Mettler Toledo | 30389218 | |
| Pistill "A" 2 mL | Wagner & Munz GmbH | 9651621 | RNase zap and rinse with MillQ before use |
| Pistill "B" 2 mL | Wagner & Munz GmbH | 9651627 | RNase zap and rinse with MillQ before use |
| Polypropylene 15 mL Centrifuge Tube | Thermo Fisher Scientific | 10579691 | |
| Polystyrene Petri dish, 60 mm x 15 mm | Thermo Fisher Scientific | 10634141 | |
| Polystyrene Round-Bottom 5 mL FACS Tubes | Thermo Fisher Scientific | 10100151 | |
| Protector RNase inhibitor - 2,000 U | Sigma Aldrich | 3335399001 | Keep in -20 °C until use |
| Protein-based RNase Inhibitor SUPERase•In (20 U/μL) | Thermo Fisher Scientific | AM2696 | Keep in -20 °C until use |
| Recombinant RNase Inhibitor | Clontech Takara | 2313B | Keep in -20 °C until use |
| RNAse free microfuge tubes - 0.5 mL | Thermo Fisher Scientific | AM12450 | |
| RNaseZap RNase Decontamination Solution | Thermo Fisher Scientific | AM9780 | Wipe surfaces and pipettes before start of experiment |
| SPRIselect - 60 mL | Beckman Coulter | B23318 | Aliquot and store in 4 °C |
| Sucrose, 500 g | Sigma Aldrich | S0389-500G | Make a 1 M stock solution |
| Swann-Morton Sterile Disposable Stainless Steel Scalpels | Thermo Fisher Scientific | 11798343 | |
| Tris-HCI (1M), pH 8.0 | Invitrogen | 15568025 | |
| Triton X-100, 98%, 100 mL | Thermo Fisher Scientific | 10671652 | Make 10% stock solution. Keep at 4 °C and protect from light. |
| Trypan Blue Solution, 0.4% | Thermo Fisher Scientific | 11538886 | |
| Vortex- Mixer | VWR | 444-1372 | Or any other type of vortex |
参考文献
- Kamies, R., Martinez-Jimenez, C. P. Advances of single-cell genomics and epigenomics in human disease: where are we now. Mamm Genome. 31 (5-6), 170-180 (2020).
- Ramachandran, P., Matchett, K. P., Dobie, R., Wilson-Kanamori, J. R., Henderson, N. C. Single-cell technologies in hepatology: New insights into liver biology and disease pathogenesis. Nature Reviews Gastroenterology & Hepatology. 17 (8), 457-472 (2020).
- Ramachandran, P., et al. Resolving the fibrotic niche of human liver cirrhosis at single-cell level. Nature. 575 (7783), 512-518 (2019).
- Andrews, T. S., et al. Single-cell, single-nucleus, and spatial RNA sequencing of the human liver identifies cholangiocyte and mesenchymal heterogeneity. Hepatology Communications. 6 (4), 821-840 (2022).
- Xiong, X., et al. Landscape of intercellular crosstalk in healthy and NASH liver revealed by single-cell secretome gene analysis. Molecular Cell. 75 (3), 644-660 (2019).
- Richter, M. L., et al. Single-nucleus RNA-seq2 reveals functional crosstalk between liver zonation and ploidy. Nature Communications. 12 (1), 4264 (2021).
- Matsumoto, T., Wakefield, L., Tarlow, B. D., Grompe, M. In vivo lineage tracing of polyploid hepatocytes reveals extensive proliferation during liver regeneration. Cell Stem Cell. 26 (1), 34-47 (2020).
- Chen, F., et al. Broad distribution of hepatocyte proliferation in liver homeostasis and regeneration. Cell Stem Cell. 26 (1), 27-33 (2020).
- Donne, R., Saroul-Ainama, M., Cordier, P., Celton-Morizur, S., Desdouets, C. Polyploidy in liver development, homeostasis and disease. Nature Reviews. Gastroenterology & Hepatology. 17 (7), 391-405 (2020).
- Lengefeld, J., et al. Cell size is a determinant of stem cell potential during aging. Science Advances. 7 (46), 0271 (2021).
- Lanz, M. C., et al. Increasing cell size remodels the proteome and promotes senescence. Mol Cell. 82 (17), 3255-3269 (2022).
- Kim, J. Y., et al. PIDDosome-SCAP crosstalk controls high-fructose-diet-dependent transition from simple steatosis to steatohepatitis. Cell Metabolism. 34 (10), 1548-1560 (2022).
- Padovan-Merhar, O., et al. Single mammalian cells compensate for differences in cellular volume and DNA copy number through independent global transcriptional mechanisms. Molecular Cell. 58 (2), 339-352 (2015).
- Miettinen, T. P., et al. Identification of transcriptional and metabolic programs related to mammalian cell size. Current Biology. 24 (6), 598-608 (2014).
- Vargas-Garcia, C. A., Ghusinga, K. R., Singh, A. Cell size control and gene expression homeostasis in single-cells. Current Opinion in Systems Biology. 8, 109-116 (2018).
- Knoblaugh, S. E., Randolph-Habecker, J. . Necropsy and histology. In Comparative Anatomy and Histology: A Mouse, Rat, and Human Atlas (Second Edition). , (2018).
- Chromium Next GEM Single Cell 3 Reagent Kits v3.1 User Guide. Document number CG000204 (Rev D). 10x Genomics Available from: https://www.10xgenomics.com/support/single-cell-gene-expression/documentation/steps/library-prep/chromium-single-cell-3-reagent-kits-user-guide-v-3-1-chemistry (2019)
- Chromium Next GEM Single Cell Multiome ATAC + Gene Expression User Guide. Document number CG000338 (Rev D). 10x Genomics Available from: https://www.10xgenomics.com/support/single-cell-multiome-atac-plus-gene-expression/documentation/steps/library-prep/chromium-next-gem-single-cell-multiome-atac-plus-gene-expression-reagent-kits-user-guide (2021)
- MacParland, S. A., et al. Single cell RNA sequencing of human liver reveals distinct intrahepatic macrophage populations. Nature Communications. 9 (1), 4383 (2018).
- Martinez-Jimenez, C. P., Kyrmizi, I., Cardot, P., Gonzalez, F. J., Talianidis, I. Hepatocyte nuclear factor 4alpha coordinates a transcription factor network regulating hepatic fatty acid metabolism. Molecular and Cell Biology. 30 (3), 565-577 (2010).
- Schmidt, D., et al. Five-vertebrate ChIP-seq reveals the evolutionary dynamics of transcription factor binding. Science. 328 (5981), 1036-1040 (2010).
- Hao, Y., et al. Integrated analysis of multimodal single-cell data. Cell. 184 (13), 3573-3587 (2021).
- Stuart, T., Srivastava, A., Madad, S., Lareau, C. A., Satija, R. Single-cell chromatin state analysis with Signac. Nature Methods. 18 (11), 1333-1341 (2021).
- Sampaziotis, F., et al. Cholangiocyte organoids can repair bile ducts after transplantation in the human liver. Science. 371 (6531), 839-846 (2021).
- Gomez-Lechon, M. J., Donato, M. T., Castell, J. V., Jover, R. Human hepatocytes as a tool for studying toxicity and drug metabolism. Current Drug Metabolism. 4 (4), 292-312 (2003).
- Gomez-Lechon, M. J., Donato, M. T., Castell, J. V., Jover, R. Human hepatocytes in primary culture: The choice to investigate drug metabolism in man. Current Drug Metabolism. 5 (5), 443-462 (2004).
- vanden Brink, S. C., et al. Single-cell sequencing reveals dissociation-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Denisenko, E., et al. Systematic assessment of tissue dissociation and storage biases in single-cell and single-nucleus RNA-seq workflows. Genome Biology. 21 (1), 130 (2020).
- Nault, R., Fader, K. A., Bhattacharya, S., Zacharewski, T. R. Single-nuclei RNA sequencing assessment of the hepatic effects of 2,3,7,8-tetrachlorodibenzo-p-dioxin. Cellular and Molecular Gastroenterology and Hepatology. 11 (1), 147-159 (2021).
- Krishnaswami, S. R., et al. Using single nuclei for RNA-seq to capture the transcriptome of postmortem neurons. Nature Protocols. 11 (3), 499-524 (2016).
- Duncan, A. W., et al. Aneuploidy as a mechanism for stress-induced liver adaptation. Journal of Clinical Investigation. 122 (9), 3307-3315 (2012).
- Kreutz, C., et al. Hepatocyte ploidy is a diversity factor for liver homeostasis. Frontiers in Physiology. 8, 862 (2017).
- Hunt, N. J., Kang, S. W. S., Lockwood, G. P., Le Couteur, D. G., Cogger, V. C. Hallmarks of aging in the liver. Computational and Structural Biotechnology Journal. 17, 1151-1161 (2019).
- Bou-Nader, M., et al. Polyploidy spectrum: a new marker in HCC classification. Gut. 69 (2), 355-364 (2019).
- Gentric, G., et al. Oxidative stress promotes pathologic polyploidization in nonalcoholic fatty liver disease. Journal of Clinical Investigation. 125 (3), 981-992 (2015).
- Schwartz-Arad, D., Zajicek, G., Bartfeld, E. The streaming liver IV: DNA content of the hepatocyte increases with its age. Liver. 9 (2), 93-99 (1989).
- Kudryavtsev, B. N., Kudryavtseva, M. V., Sakuta, G. A., Stein, G. I. Human hepatocyte polyploidization kinetics in the course of life cycle. Virchows Archiv. B, Cell Pathology Including Molecular Pathology. 64 (6), 387-393 (1993).
- Duncan, A. W., et al. The ploidy conveyor of mature hepatocytes as a source of genetic variation. Nature. 467 (7316), 707-710 (2010).
- Granja, J. M., et al. ArchR is a scalable software package for integrative single-cell chromatin accessibility analysis. Nature Genetics. 53 (3), 403-411 (2021).
- Bredikhin, D., Kats, I., Stegle, O. MUON: Multimodal omics analysis framework. Genome Biology. 23 (1), 42 (2022).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nature Methods. 19 (2), 179-186 (2022).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved