Method Article
Un análisis extraíble cuantitativo optimizado de proteínas de unión al ARN utilizando ARN biotinilado corto
En este artículo
Resumen
Aquí, presentamos un método in vitro optimizado para descubrir, cuantificar y validar interactores de proteínas de secuencias específicas de ARN, utilizando extracto de proteína total de células humanas, perlas de estreptavidina recubiertas con ARN biotinilado y análisis de espectrometría de masas.
Resumen
Las interacciones proteína-ARN regulan la expresión génica y las funciones celulares a nivel transcripcional y posttranscripcional. Por esta razón, la identificación de los socios de unión de un ARN de interés sigue siendo de gran importancia para revelar los mecanismos detrás de muchos procesos celulares. Sin embargo, las moléculas de ARN pueden interactuar transitoria y dinámicamente con algunas proteínas de unión al ARN (RBP), especialmente con las no canónicas. Por lo tanto, se necesitan en gran medida métodos mejorados para aislar e identificar esas prácticas comerciales restrictivas.
Para identificar las proteínas asociadas de una secuencia de ARN conocida de manera eficiente y cuantitativa, desarrollamos un método basado en la extracción y caracterización de todas las proteínas que interactúan, a partir del extracto de proteína total celular. Optimizamos la extracción de proteínas utilizando ARN biotinilado precargado en perlas recubiertas de estreptavidina. Como prueba de concepto, empleamos una secuencia corta de ARN conocida por unirse a la proteína TDP-43 asociada a la neurodegeneración y un control negativo de una composición de nucleótidos diferente pero de la misma longitud. Después de bloquear las perlas con ARNt de levadura, cargamos las secuencias de ARN biotiniladas en las perlas de estreptavidina y las incubamos con el extracto de proteína total de las células HEK 293T. Después de la incubación y varios pasos de lavado para eliminar los aglutinantes inespecíficos, eluimos las proteínas que interactúan con una solución con alto contenido de sal, compatible con los reactivos de cuantificación de proteínas más utilizados y con la preparación de muestras para espectrometría de masas. Cuantificamos el enriquecimiento de TDP-43 en el pull-down realizado con el aglutinante de ARN conocido en comparación con el control negativo por espectrometría de masas. Utilizamos la misma técnica para verificar las interacciones selectivas de otras proteínas predichas computacionalmente como aglutinantes únicos de nuestro ARN de interés o del control. Finalmente, validamos el protocolo por western blot mediante la detección de TDP-43 con un anticuerpo apropiado.
Este protocolo permitirá el estudio de las proteínas asociadas de un ARN de interés en condiciones cercanas a las fisiológicas, ayudando a descubrir interacciones proteína-ARN únicas e impredecibles.
Introducción
Las proteínas de unión al ARN (RBP) han surgido como actores cruciales en la regulación de genes transcripcionales y post-transcripcionales, ya que están involucradas en procesos como el empalme de ARNm, localización celular de ARN, traducción, modificación y degradación 1,2,3. Tales interacciones entre las dos macromoléculas están altamente coordinadas, equilibradas con precisión y esenciales para la formación de centros funcionales y de procesamiento. Las variaciones o desregulaciones dentro de estos centros tienen el potencial de interrumpir las redes de proteína-ARN finamente reguladas y se asocian cada vez más con una variedad de enfermedades humanas, incluyendo el cáncer 4,5 y los trastornos neurodegenerativos 6,7,8. Las interacciones entre las moléculas de ARN y sus socios de unión a proteínas pueden ser estables y fáciles de validar experimentalmente, o altamente dinámicas, transitorias y más difíciles de caracterizar.
En los últimos años, se han realizado intensos esfuerzos para comprender estas interacciones. Entre los métodos más establecidos, los ensayos de extracción de proteínas (PD) son probablemente los enfoques más apreciados y comúnmente utilizados para desentrañar los principales actores que constituyen complejos de ribonucleoproteínas (RNP) y otras redes de interacción proteína-ARN 3,9,10. Las DP incluyen un amplio paraguas de técnicas informativas, como la inmunoprecipitación del ARN (RIP)11,12 o de la proteína (CLIP)13,14 de interés. Algunos de estos protocolos de ARN-PD emplean un ARN conocido como cebo para las proteínas15, con mayor frecuencia aprovechando las etiquetas de alta afinidad como la biotina. En este caso, los socios de interacción de un ARN biotinilado se pueden detectar anclando el ARN en perlas recubiertas de estreptavidina, lo que permite un aislamiento eficiente de las RNP. Las principales limitaciones de estos enfoques suelen ser el diseño de las sondas biotiniladas y la prueba de su capacidad para unirse a proteínas diana. Para ello, podría ser útil basarse en los datos publicados de CLIP de la proteína de interés, si están disponibles, ya que revelan, con alta precisión, las regiones cortas de ARN que corresponden a picos de interacciones con la proteína diana13,16. Estas mismas regiones podrían usarse para desarrollar sondas para PD. Un método alternativo para diseñar tales cebos de ARN podría ser la evolución sistemática de ligandos por enriquecimiento exponencial (SELEX)17, que permiten el diseño de aptámeros a través de la selección in vitro, a partir de una biblioteca aleatoria completa y a través de una serie de ciclos de optimización impulsados por PCR. Sin embargo, SELEX es complejo y requiere mucho tiempo, y los resultados finales dependen en gran medida de la biblioteca inicial. Para seleccionar el cebo de ARN a utilizar en el protocolo aquí presentado, se aprovechó otro enfoque, consistente en utilizar un cebo de ARN diseñado de novo mediante la potencia computacional del algoritmo catRAPID, que predice la unión preferencial de una proteína dada hacia ciertas secuencias de ARN18,19,20.
El protocolo presentado aquí es una versión de un ARN-PD optimizado para eluyer socios proteicos específicos en condiciones casi fisiológicas, sin el uso de detergente, agentes desnaturalizantes o altas temperaturas. Se basa en perlas nano-superparamagnéticas recubiertas covalentemente con estreptavidina altamente purificada y el uso de un ARN biotinilado específico diseñado in silico como cebo. Este protocolo proporciona un método rápido y eficiente para aislar los socios de unión de moléculas de ARN biotiniladas en condiciones nativas, ofreciendo el potencial para una amplia gama de aplicaciones posteriores. Para probar este protocolo, se utilizó una secuencia de aptámero de ARN monocatenario de 10 nucleótidos, previamente diseñada para unirse a la proteína TAR de unión al ADN 43 (TDP-43) con alta afinidad y especificidad20. A partir de los lisados celulares HEK 293T, los interactores del aptámero de ARN biotinilado se identificaron mediante análisis de espectrometría de masas realizado en muestras separadas del cebo de ARN utilizando un tampón hipertónico. Este análisis confirmó la identificación y cuantificación exitosa de TDP-43 como aglutinante preferido.
Este protocolo permite la identificación exitosa de interactores de proteínas utilizando solo un oligonucleótido de ARN sintetizado in vitro corto. Además, el uso de aptámeros de ARN diseñados in silico como sondas de DP21,22 garantiza la especificidad de los objetivos a costos significativamente reducidos.
Protocolo
1. Métodos y materiales generales
- Preparar el medio adecuado para el cultivo celular de mamífero elegido y calentarlo previamente a 37 °C durante 20 minutos antes de su uso.
- Prepare el material requerido con anticipación, como se describe en la Tabla de materiales. Cristalería en autoclave, artículos de plástico y existencias reguladoras.
- Prepare los búferes como se describe en la Tabla 1. Ajustar el pH de las soluciones madre utilizando HCl o NaOH concentrado antes de diluir los componentes a sus volúmenes finales.
2. Preparación de líneas celulares de mamíferos
- Cultive células HEK 293T en el medio Eagle Modificado (DMEM) de Dulbecco suplementado con suero bovino fetal al 10% (FBS) y solución de penicilina/estreptomicina de 100 μg/ml. Incubarlos a 37 °C en una incubadora humidificada suministrada con 5% deCO2. Dividir rutinariamente las células.
- Antes de desprender, enjuague las células con suficiente solución salina tamponada con fosfato (PBS) para cubrir la superficie de crecimiento.
- Retire el PBS y agregue una capa ultrafina de solución de tripsina-EDTA.
- Incubar las células a 37 °C en una incubadora humidificada provista de 5% deCO2 durante 5 min, o hasta que las células se desprendan (deben verse dispersas bajo observación microscópica).
- Diluir la solución de tripsina-EDTA diez veces añadiendo DMEM completo para inactivarla y contar las células.
- Placa 1.5 x 105 células/mL en placas de 6 pocillos, considerando dos pocillos/condición a probar.
- Incubar las células a 37 °C durante 48 h en una incubadora humidificada con 5% deCO2.
NOTA: Consulte las instrucciones del fabricante para el tipo de medio y suplemento apto para la línea celular. Además, la cantidad y el tiempo de incubación de la tripsina-EDTA dependen de la línea celular. Algunos tipos de células crecen más rápido / más lento de lo que se informó en este protocolo; Por lo tanto, la concentración de siembra debe probarse de antemano.
3. Cosecha total de proteínas
- Retire el medio de los pocillos donde crecen las células.
- Lave cada pocillo de las placas de 6 pocillos con 1 ml de PBS.
- Deseche el PBS.
- Mueva las placas sobre hielo y continúe con el paso 3.5 o congele las placas secas a -80 °C para facilitar la lisis.
- Agregue 200 μL de tampón de lisis a cada pocillo.
- Use un raspador de células para separar y romper las células.
- Transfiera el extracto celular derivado de dos pocillos al mismo tubo de 1,5 ml.
- Coloque el tubo que contiene el extracto de proteína en hielo durante 30 minutos.
- Centrifugar la célula lisa a 17.000 x g durante 15 min a 4 °C.
- Transfiera cada sobrenadante a un tubo preenfriado.
NOTA: Se recomienda un total de 106-10 7 celdas para cada condición de EP. La lisis celular y la recolección de proteínas deben realizarse con tampones helados. Se deben agregar inhibidores de la proteasa al tampón de lisis para prevenir la degradación de proteínas.
4. Determinación de la concentración de proteínas
- Preparar el reactivo Bradford, según lo indicado por el productor, diluyéndolo cinco veces endH2O.
- Distribuir 1 ml de reactivo en una cubeta de 1 cm, añadir 1 μL de la muestra y mezclar por inversión.
- Incubar en la oscuridad a temperatura ambiente durante 5-10 min.
- Lea la absorbancia a 595 nm.
- Calcular el volumen de extracto proteico correspondiente a 1,5 mg de proteínas y llevar todas las muestras a un volumen final de 600 μL utilizando tampón de lisis.
- Mantenga las muestras en hielo hasta que las use.
NOTA: Se puede utilizar cualquier otro método de determinación de la concentración de proteínas, siguiendo las recomendaciones de compatibilidad de tampón. En cualquier caso, el tampón de lisis debe usarse como un espacio en blanco. Muchos reactivos no son compatibles con el ditiothreitol (DTT). Se recomienda añadir TDT u otros agentes reductores sólo después de la cuantificación de proteínas (DTT a una concentración final de 1 mM).
5. Preparación de cuentas
- Mezcle las cuentas en su búfer de almacenamiento moviendo el tubo.
- Calcule 100 μL de medio de suspensión/muestra y coloque el volumen en una rejilla magnética.
- Lavado de cuentas
- Retire la solución de almacenamiento y lave las perlas agregando 1 ml de tampón/tubo de lisis e invierta manualmente.
- Retire el búfer con el bastidor magnético.
- Repita el paso de lavado.
- Agregue un volumen de tampón de lisis igual al volumen inicial del medio de suspensión, mezcle moviendo el tubo y dispense el medio uniformemente en tantos tubos de 1,5 ml como muestras.
- Bloqueo de cuentas
- Retirar el tampón utilizando la rejilla magnética y añadir 600 μL de una solución de 0,25 mg/ml de ARNt de levadura preparada en tampón de lisis.
- Incubar durante 1 h a temperatura ambiente en una rueda giratoria.
- Retire la solución de ARNt utilizando la rejilla magnética.
- Añadir 600 μL de tampón de lisis y lavar mezclando manualmente.
- Repita el paso de lavado y deseche el tampón.
6. Carga de cuentas
- Preparar 200 μg de oligonucleótido de ARN en 600 μL de tampón de lisis para cada tubo que contenga el medio inicial de suspensión de 100 μL (ahora perlas bloqueadas).
- Añadir el oligo a las perlas e incubar durante 1 h a temperatura ambiente mientras giramos.
- Retire la solución, agregue 600 μL de tampón de lisis y lave las perlas dos veces girando los tubos durante 5 minutos a temperatura ambiente.
- Deseche el búfer.
NOTA: Nunca mueva las cuentas, sino que mueva en su lugar. Limite el número de pasos de pipeteo, a menos que sea necesario. Cuando sea posible, use puntas cortadas de 1 ml. La cantidad de medio de lodo de perlas / muestra depende de la capacidad de unión de las perlas y la cantidad inicial de las proteínas totales. Si se predice que el oligo de ARN tiene una cantidad significativa de estructura secundaria, recomendamos desnaturalizarlo primero a 80 °C durante 10 min y luego enfriarlo lentamente a temperatura ambiente o reconstruirlo incubándolo a 30 °C durante 1 h. Se sugiere recuperar el oligo de ARN después de la carga del cordón y determinar la concentración restante, con el fin de optimizar la cantidad requerida para la carga y evaluar la posibilidad de reutilizar el ARN.
7. Unión a proteínas en perlas
NOTA: A partir de ahora, cuando sea posible, realice los pasos a 4 °C.
- Tomar un volumen al 5% de la solución de proteína de 600 μL y mantenerlo como INPUT (IN) para su posterior análisis (1,5 mg de proteínas se disuelven en 600 μL, por lo que el 5% corresponde a 30 μL y 75 μg de proteínas).
- Añadir la mezcla de proteínas restante a cada tubo de perlas cargadas y dejar girar lentamente durante la noche a 4 °C.
8. Lavado de aglutinantes inespecíficos
- Retire la fracción no unida utilizando la rejilla magnética. Ahorre el 5% del volumen y etiquételo como FLOWTHROUGH (FT) (el volumen no unido es de aproximadamente 600 μL, por lo que nuevamente mantenga 30 μL para un análisis posterior).
- Añadir 1 ml de tampón de lavado 1 a las perlas y dejar girar durante 5 min a 4 °C.
- Deseche el búfer.
- Repita los pasos 8.2 y 8.3.
- Añadir 1 ml de tampón de lavado 2 a las perlas y dejar girar durante 5 min a 4 °C.
- Deseche el sobrenadante.
9. Eliminación de aglutinantes específicos
- Añadir 100 μL de tampón de elución 1 o tampón de elución 2 a las perlas.
- Mezclar manualmente moviendo e incubar durante 5 min a temperatura ambiente.
- Colocar los tubos en un termomezclador y agitar vigorosamente durante 5 min a 95 °C.
- Coloque el tubo en la rejilla magnética y recoja la fracción eluyida en un tubo limpio.
- Gire rápidamente las perlas con una centrífuga de banco para maximizar la recuperación del eluido.
- Ahorre el 5% del volumen total de ELADO (EL) para análisis adicionales (el volumen total es de 100 μL, por lo que separe 5 μL en otro tubo).
- Si es necesario, la concentración de proteína se puede determinar como en la sección 4, utilizando el tampón de elución como un espacio en blanco.
NOTA: Se recomienda abstenerse de añadir TDT al tampón de elución hasta que se haya determinado la concentración de proteínas. Si no se requiere cuantificación de proteínas, o si se dispone de kits de cuantificación de proteínas compatibles con agentes reductores, se puede agregar 1 mM de TDT al tampón de elución desde el principio. En este protocolo, se probaron tanto el tampón de elución 1 (que contiene 1 M NaCl) como el tampón de elución 2 (con 2 M NaCl). No se observó ninguna diferencia en la eficiencia de elución de la proteína diana con el aumento de la fuerza iónica, pero se recomienda probar ambas condiciones antes de establecer el tampón más apropiado. Si una alta presencia de sal en el tampón de elución representa una limitación para un análisis posterior, la cantidad muy baja de detergentes en el tampón de elución permite el intercambio de tampón. Como alternativa, el eluido se puede diluir para alcanzar la concentración de sal deseada.
10. Identificación de captadores de proteínas por espectrometría de masas
- Precipitación de acetona
- Concentrar las proteínas eluyidas diluyéndolas cuatro veces en acetona fría (-20 °C).
- Vortex e incubar el tubo a -20 °C durante la noche.
- Girar a 17.000 x g durante 30 min a 4 °C.
- Retire suavemente el sobrenadante y deje que la acetona se evapore hasta que el pellet se haya secado por completo.
- Digestión de proteínas en solución
- Disuelva el pellet de proteína añadiendo 50 μL de tampón de desnaturalización.
- Añadir TDT a una concentración final de 5 mM, permitiendo la reducción de proteínas durante 30 min a 55 °C.
- Enfriar las muestras a temperatura ambiente y proceder con la reacción de alquilación de proteínas, añadiendo yodoacetamida (IAA) a una concentración de 10 mM durante 15 min.
- Digerir las proteínas utilizando una enzima adecuada (tripsina, LysC) e incubar las muestras durante la noche a 37 °C.
- Detenga la digestión agregando 1 μL de ácido trifluoroacético (TFA) al 10%.
- Limpie y concentre los péptidos en una microcolumna de fase C18 invertida personalizada, como se describió anteriormente19.
- Péptidos de eluido de la punta C18 con tampón B.
- Retire el componente orgánico utilizando una centrífuga al vacío y resuspenda los péptidos en 5 μL de ácido fórmico al 0,1% para su posterior análisis.
NOTA: Alternativamente, la digestión de proteínas se puede realizar "en perlas" inmediatamente después de lavar los aglutinantes no específicos (pasos 8.1-8.6), lo que hace que el protocolo sea más rápido. Sin embargo, es aconsejable probar la eficiencia de la enzima que trabaja "en perlas" proteínas inmovilizadas en comparación con el estándar en la digestión en solución, para garantizar la cobertura óptima de la secuencia de proteínas experimentales.
- Cromatografía líquida-espectrometría de masas en tándem (LC-MS/MS)
- Enchufar la columna analítica (fase estacionaria C18) y mantenerla a 45 °C durante el tiempo de funcionamiento.
- Conecte la columna a la salida de una válvula rotativa de seis puertos de la bomba LC a través de un accesorio capilar hermético para los dedos (20 μm x 550 mm) en una configuración de una sola columna.
- Ajuste la configuración de LC de la siguiente manera:
- Cargue los péptidos a presión controlada (980 bar) en el tampón A.
- Aplique un gradiente de tampón B del 5% al 20% a 300 nL/min durante 59 min, seguido de un gradiente B del 20%-30% durante 15 min y un gradiente B del 30%-65% durante 5 min.
- Agregue un paso de lavado aumentando la concentración de tampón B hasta un 95% durante 5 minutos más un paso isocrático de 5 minutos al 95% de tampón B.
- Opere el espectrómetro de masas en modo de adquisición dependiente de datos (DDA) para cambiar automáticamente entre eventos MS y MSMS.
- Defina un recuento de bucles igual a 15 utilizando un valor objetivo de control automático de ganancia (AGC) de 3 x 106 y 1 x 105 para los eventos MS y MSMS, respectivamente.
- Establezca el tiempo máximo permitido de acumulación de iones en 20 ms para MS con una resolución de 60 K y 100 ms para MSMS con una resolución de 15 K.
- Realice un experimento de fragmentación de alta disociación de colisión (HCD) utilizando una energía de colisión normalizada del 28%, con un tiempo de exclusión dinámica de 20 s.
- Opere los parámetros de origen de la siguiente manera:
Voltaje de pulverización: 1.7 kV
Voltaje capilar: 275 °C
Ni una funda ni gas auxiliar utilizado
NOTA: En este protocolo, el análisis de espectrometría de masas (MS) de cromatografía líquida de ultra alto rendimiento (UHPLC) se ha realizado específicamente utilizando una configuración de columna única LC, acoplada a un instrumento híbrido de órbita de triple cuadrupolo (Tabla de materiales). Se pueden utilizar otros sistemas LCMS, pero se recomienda una adaptación de los parámetros.
- Análisis de datos
- Utilice el botón Cargar para importar los archivos sin procesar.
- Defina los nombres de los experimentos haciendo clic en el botón Establecer experimento .
- Introduzca la sección de parámetros específicos del grupo para especificar todos los parámetros relacionados con la identificación:
Enzima utilizada para la digestión: Tripsina/P
Escotes perdidos: hasta tres
Modificación corregida: Carbamidometilación
Modificación variable: N-acetil (proteína), oxidación (M) - Cargue un archivo FASTA actualizado, disponible en bases de datos públicas como UniprotKB.
- Especifique las reglas de análisis correctas según el origen de la base de datos elegida.
- Defina un valor de tasa de descubrimiento falso (FDR) de parentesco = 1 para proteínas y péptidos.
- Agregue la opción de cuantificación sin etiqueta (LFQ) en la pestaña Cuantificación sin etiqueta.
- Mantenga el recuento mínimo de la relación LFQ en dos.
NOTA: Aquí, describimos el análisis de datos utilizando el software MaxQuant24 y Perseus25 para realizar la cuantificación de proteínas y el posterior análisis estadístico, respectivamente. Sin embargo, el análisis de datos se puede realizar con cualquier otra bioinformática disponible comercialmente o gratuita. FDR se estima utilizando un enfoque basado en bases de datos de señuelos de destino26. Péptido y proteína FDR igual a 0.01 significa que se espera que los péptidos y proteínas identificados contengan 1% de falsos positivos.
- Análisis estadístico
- Cargue el archivo .txt grupos de proteínas para realizar análisis estadísticos a nivel de proteína.
- Defina los valores LFQ como columnas principales.
- Elimine "reverso" y "contaminantes" filtrando las filas basadas en la columna categórica.
- Utilice las filas de anotación categórica para agrupar las diferentes condiciones experimentales.
- Reducir la matriz de datos, seleccionando el número de valores válidos en cada uno de los grupos definidos previamente.
- Seleccione la prueba estadística que mejor se adapte a las condiciones experimentales (es decir, prueba t, ANOVA de prueba de muestra múltiple).
- Determine un límite para sugerencias significativas con un cálculo basado en FDR. Normalmente, tanto 0,01 como 0,05 se aceptan como umbrales para el valor de p ajustado.
- Visualice los resultados de los análisis diferenciales basados en estadísticas de la prueba t utilizando una representación de diagrama de volcán.
- Exporte la matriz final en formato .txt para su posterior edición de la tabla de resultados final.
NOTA: La carpeta de configuración contiene un archivo FASTA con proteínas como las queratinas que se consideran contaminantes comunes en los experimentos proteómicos globales, que se marcan con un + en la tabla de salida. En el presente estudio, teniendo dos condiciones de muestra, se utiliza una prueba t para el análisis estadístico.
11. Validación de resultados por Western blot
- Preparación de muestras
- Agregue el volumen apropiado de 4x búfer de carga de muestras a cada alícuota de IN, FT y EL.
- Hervir las muestras durante 5 min a 95°C.
- Centrifugado rápido para recuperar la muestra evaporada de la parte superior de los tubos.
- SDS-PAGE y transferencia de gel
- Cargue las muestras en un gel de poliacrilamida desnaturalizante al 4% -12%.
- Ejecute el gel con el búfer de ejecución MES SDS durante 1,5 h a 120 V.
- Transfiera el gel sobre una membrana de nitrocelulosa con un casete de transferencia semiseco, siguiendo las instrucciones del fabricante. Recomendamos una transferencia de 10 minutos a 15 V.
- Inmunodetección
- Bloquear la membrana con albúmina sérica bovina (BSA) al 10% durante 1 h a temperatura ambiente mientras agita suavemente.
- Agregue el anticuerpo primario preparado en BSA al 5% en TBST, de acuerdo con las instrucciones del fabricante. Dejar toda la noche a 4 °C o durante 1 h a temperatura ambiente bajo agitación suave.
- Lave la membrana tres veces con TBST, cada vez durante 5 minutos.
- Añadir el anticuerpo secundario preparado en TBST durante 1 h a temperatura ambiente bajo agitación.
- Lave la membrana tres veces con TBST, cada vez durante 5 minutos.
- Visualice los resultados utilizando un generador de imágenes de borrones.
NOTA: Para detectar TDP-43, un aglutinante conocido de nuestro ARN, se utilizó un anticuerpo monoclonal de conejo recombinante y se dejó con la membrana durante la noche a 4 °C. Como anticuerpo secundario, se utilizó la peroxidasa de rábano picante (HRP) IgG anti-conejo, pero también funcionaría un anticuerpo secundario fluorescente. Para visualizar los anticuerpos en la membrana, la membrana se incubó con el sustrato Clarity Western ECL durante 1 minuto, antes de obtener imágenes con el sistema de imágenes ChemiDoc.
Resultados
Para verificar la validez del protocolo propuesto, los experimentos de DP presentados aquí se realizaron con un aptámero de ARN biotinilado diseñado in silico para unirse específicamente a TDP-4320. Este ARN se une a su diana proteica con alta afinidad de unión (Kd = 90 nM)20. Aquí, este ARN, de secuencia 5'-CGGUGUUGCU-3', se conoce con el nombre de "+ARN". Como control negativo, se utilizó la secuencia complementaria inversa de +ARN, que aquí se llama "-ARN". Su secuencia es 5'-AGCAACACCG-3'. -El ARN muestra una afinidad de unión significativamente menor hacia TDP-43 (Kd = 1,5 μM)19. Para el propósito del protocolo descrito aquí, estos oligonucleótidos de ARN se han comprado conjugados a una molécula de biotina, para permitir la unión a las perlas de estreptavidina. +RNA se compró con un TEG de biotina en su extremo 3', que incluye un espaciador de trietilenglicol de 15 átomos entre la biotina y el grupo fosfato del ácido nucleico; -El ARN en cambio tenía una biotina en su extremo 5', conjugada al ácido nucleico a través de un enlazador amino-C6. Sin embargo, si el diseño del cebo de ARN es robusto, y siempre que no haya interferencia estructural o química entre el enlazador y el ARN, se podrían emplear otras posiciones para la conjugación de biotina y otras longitudes de enlazador.
Conocer la identidad de la proteína principal que se encuentra unida a la sonda +RNA después de la DP permitió la validación del protocolo mediante la identificación de TDP-43 en el eluido, utilizando espectrometría de masas (MS) y western blot (WB) (Figura 1).
El análisis de MS se llevó a cabo en cuatro réplicas de DP realizadas utilizando +ARN o -ARN (Figura 2). La identificación de los interactomas de +RNA y -RNA está más allá del alcance de este protocolo, sin embargo, se informan algunos resultados que validan la precisión del protocolo. Cabe destacar que el trazado de las proteínas significativamente enriquecidas en un diagrama de volcán reveló que el contenido total de proteínas y las proteínas enriquecidas eluidas de +ARN fue significativamente mayor que lo que se recuperó de -ARN (Figura 2). Esto significa que, a pesar de tener la misma longitud y contenido estructural (lineal), el +ARN puede establecer un mayor número de interacciones específicas, que se retienen hasta la etapa de elución con alta sal. Es probable que el -ARN establezca un mayor número de contactos no específicos que se interrumpen durante los pasos de lavado. Como era de esperar, TDP-43 fue identificado como un interactor único de +RNA20; la cuantificación promedio libre de etiqueta (LFQ) para las cuatro réplicas de PD realizadas con +ARN es de 31,96 ± 0,56, mientras que la proteína no se identifica entre los interactores de -ARN. Además, entre todos los interactianos únicos de +ARN, se encontró que TDP-43 es la proteína más abundantemente enriquecida.
Para validar aún más el protocolo, se utilizó el algoritmo interno catRAPID 18,19 para predecir computacionalmente qué otras proteínas se unirían específicamente a +RNA o -RNA. En particular, las puntuaciones de interacción para +RNA y -RNA con las proteínas que componen el proteoma humano se calcularon utilizando la característica de 'propensión a la interacción' catRAPID, como se definió en nuestro trabajo anterior27. Entre las proteínas puntuadas con alta confianza, se predijo que HNRNPH3 se uniera selectivamente a +ARN (+ puntuación de interacción ARN = 1.01; puntuación de interacción -ARN = 0.21) y PCBP2 para interactuar específicamente con -ARN (+ puntuación de interacción ARN = -0.5; puntuación de interacción -ARN = 0.31) (Figura 3A). Además, se predijo que la proteína RBM41 era promiscua para ambos oligonucleótidos de ARN (+ puntuación de interacción ARN = 0,4; puntuación de interacción -ARN = 0,39) (Figura 3A). El análisis de MS confirmó la presencia de HNRNPH3 y PCBP2 en la PD de +RNA y -RNA respectivamente, mientras que RBM41 se encontró interactuando con ambos (Figura 3B).
WB se utilizó para detectar la presencia de TDP-43 para confirmar aún más los resultados y durante la optimización del protocolo (Figura 4). En el procedimiento descrito aquí, se recolectaron diferentes muestras en diferentes etapas. La muestra de entrada (IN) consistió en el total de proteínas diluidas en tampón de lisis. El flujo a través (FT) se obtuvo después de una incubación nocturna de las proteínas totales con las perlas de estreptavidina pre-recubiertas con el ARN biotinilado, que representa la fracción de proteínas que no se unieron al ARN. Finalmente, el eluido (EL) contenía todas las proteínas que reconocían específicamente el ARN investigado, ya que entre el FT y el EL tres pasos de lavado con 150 mM de sal y 0,1% de tritón-X deberían haber eliminado las interacciones más débiles.
Para cada réplica, la misma cantidad (5% v/v) de IN, FT y EL se ejecutó en paralelo en una SDS-PAGE y se tiñó con un anticuerpo anti-TDP-43 (Figura 4). En el caso del +ARN, se observó la banda de TDP-43 en IN y en EL, lo que indica que la proteína, presente desde el principio en el extracto proteico total, es retenida por +ARN durante los pasos de lavado y solo se eluye al final con un tampón de sal alto. TDP-43 también estaba presente en IN para -RNA, sin embargo, la banda correspondiente a la proteína también es visible en FT, lo que indica que este ARN no se une a TDP-43. La ausencia de la banda TDP-43 en EL confirma este resultado.
Durante la optimización del protocolo, la elución de las proteínas específicamente unidas a las secuencias de ARN se probó tanto con un tampón de elución que contenía 1 M NaCl (EB1) como con un tampón de elución completo con 2 M NaCl (EB2) (Figura 4). Los eluidos obtenidos con cualquiera de los EB se compararon en una SDS-PAGE y se borraron con el anticuerpo anti-TDP-43. Las imágenes obtenidas se analizaron con ImageJ28 para cuantificar cualquier diferencia en la cantidad de TDP-43 eluyente con los dos tampones. En general, no se observaron diferencias significativas, y concluimos que, dentro de estos ensayos, 1 M de sal es suficiente para interrumpir incluso las interacciones proteína-ARN más fuertes.
En general, los resultados reportados aquí para EM y WBs demuestran que este protocolo es eficiente para capturar los interactores de proteínas de un ARN dado de una manera específica, y que permite la elución en tampones compatibles con el análisis posterior.
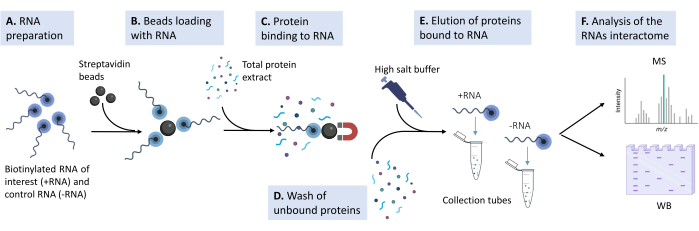
Figura 1: Croquis de la tubería experimental utilizada en el protocolo propuesto . (A) El oligonucleótido de ARN biotinilado se prepara en tampón de lisis a la concentración apropiada. (B) Las perlas magnéticas de estreptavidina se lavan, se bloquean con ARNt de levadura y se cargan con el ARN biotinilado. (C) El extracto proteico total derivado de líneas celulares de mamíferos cultivados se agrega a la mezcla de perlas y ARN. (D) Se realizan múltiples lavados para eliminar interacciones no específicas. (E) Los interactores proteicos específicos se separan del ARN con una solución hipertónica. (F) La identidad de los interactianos es revelada por espectrometría de masas, y los casos específicos son validados por Western blot. Haga clic aquí para ver una versión más grande de esta figura.
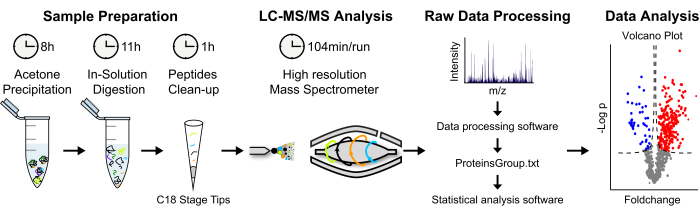
Figura 2: Estrategia analítica para la cuantificación de proteínas basadas en EM sin etiquetado . (A) Las proteínas eludidas se precipitan en acetona fría durante la noche. Luego, las proteínas se desnaturalizan y se realiza una digestión en solución. Los péptidos proteolíticos se concentran y desalan. (B) Los péptidos se analizan a través de LC-MS / MS utilizando un "enfoque de escopeta". (C) El procesamiento y análisis de datos brutos se realiza utilizando el software MaxQuant y Perseus, respectivamente. (D) Las proteínas enriquecidas estadísticamente significativas se muestran en un diagrama de volcán. Haga clic aquí para ver una versión más grande de esta figura.
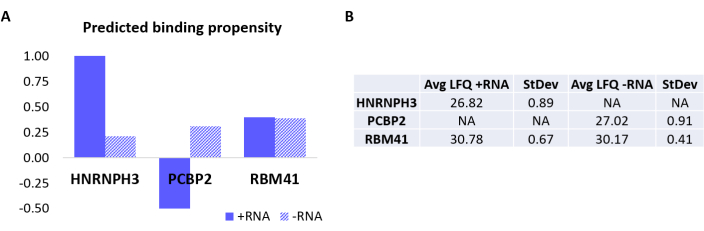
Figura 3: Correlación entre las propensiones de interacción predichas y las interacciones determinadas experimentalmente de +ARN y -ARN. (A) catRAPID puntuaciones de interacción en relación con HNRNPH3, PCBP2 y RBM41, lo que indica la unión preferencial de HNRNPH3 para +RNA y de PCBP2 para -RNA, mientras que se predice que RBM41 se une indiscriminadamente a ambas secuencias de ARN. (B) Promedios de cuantificación sin etiquetas determinados por análisis de espectrometría de masas a partir de las extracciones realizadas con +ARN y -ARN. El análisis confirma que HNRNPH3 solo se une al +ARN, el PCBP2 solo se une al ARN y RBM41 se une a ambos por igual. Haga clic aquí para ver una versión más grande de esta figura.
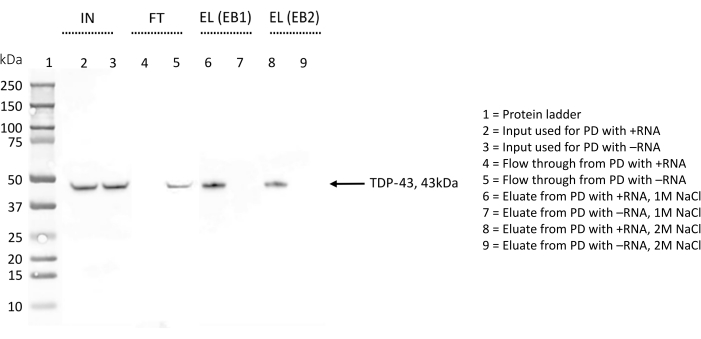
Figura 4: Validación de Western blot de la presencia/ausencia de TDP-43 entre los interactianos de secuencias de ARN elegidas. La membrana WB ha sido tratada con anticuerpos anti-TDP-43. IN = entrada; FT = flujo continuo; EL (EB1) = elución con tampón de elución 1; EL (EB2) = elución con tampón de elución 2; el signo "+" indica muestras derivadas del pull-down realizado con +RNA; el signo "-" indica muestras derivadas del pull-down realizado con -RNA; El carril 1 contiene una escalera de proteínas. TDP-43 se indica con una flecha. El WB indica que TDP-43 se encuentra entre los interactores de +ARN pero no entre los interactores de ARN-ARN. Haga clic aquí para ver una versión más grande de esta figura.
| Nombre del búfer | Composición | |||||
| 10x Tranfer buffer | 250 mM tris, 1.92 M glicina, 1% SDS, 20% metanol. Diluir 10 veces antes del uso | PD | ||||
| 20X MES SDS ejecutando buffer | 1 M MES, 1 M tris, 2% SDS, 20 mM EDTA. Ajuste el pH a 7.3. Diluir 20 veces antes del uso | |||||
| 4x Búfer de carga de muestras | 0,25 M Tris base, 0,28 M SDS, 40% glicerol, 20% 2-mercapto-etanol, 4 mg/ml de azul de bromfenol | |||||
| Tampón de elución 1 | 20 mM fosfato pH 7,5, 1 M NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM TDT (añadir después de la cuantificación) | |||||
| Tampón de elución 2 | 20 mM fosfato pH 7,5, 2 M NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM TDT (añadir después de la cuantificación) | |||||
| Tampón de lisis | 10 mM Tris-HCl pH 7,4, 150 mM NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM DTT e inhibidores de la proteasa | |||||
| Solución salina tamponada con Tris con Tween-20 | 1 M Tris-HCl pH 7.4, 3 M NaCl, 2.0% Tween-20 | |||||
| Tampón de lavado 1 | 10 mM Tris-HCl pH 7.4, 150 mM NaCl, 0.5 mM EDTA, 0.1 % Triton TM X-100, 1 mM DTT e inhibidores de la proteasa | |||||
| Tampón de lavado 2 | 25 mM Hepes pH 8, 150 mM NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM DTT e inhibidores de la proteasa | |||||
| Búfer A | 0,1% de ácido fórmico | SRA. | ||||
| Búfer B | 60% acetonitrilo, 0,1% ácido fórmico | |||||
| Tampón de desnaturalización | 8M de urea, 50 mM de Tris-HCl | |||||
Tabla 1: Tampones PD y MS. Nombres y composición de los tampones utilizados para los experimentos de extracción (PD) o para el análisis de espectrometría de masas (MS).
Discusión
Este trabajo informa de la optimización de un protocolo de DP realizado con oligonucleótidos de ARN biotinilados para capturar sus interactores de proteínas. El protocolo descrito aquí es fácil de realizar, requiere poco material y produce resultados altamente confiables. Es importante destacar que los aspectos más novedosos de este protocolo consisten en el uso de un cebo de ARN diseñado completamente in silico y específico para la proteína diana, y la elución de todas las proteínas unidas al cebo de ARN interrumpiendo directamente sus interacciones con una solución con alto contenido de sal, en lugar de disociar la estreptavidina de la biotina con detergente y tratamiento a alta temperatura.
Este protocolo aprovecha la fuerza del enlace entre biotina y estreptavidina29,30. De acuerdo con las perlas de estreptavidina elegidas, la carga del ARN biotinilado debe probarse y cuantificarse antes de continuar. Además, el plegamiento tridimensional del ARN podría afectar la eficiencia de carga en las perlas, ya que podría limitar la exposición de la biotina a la estreptavidina. El bloqueo de las perlas con ARNt no biotinilado mejora la limpieza de los resultados al limitar las interacciones no específicas con las perlas. El búfer de carga y el tampón de elución deben elegirse en función de las aplicaciones posteriores. Aquí, se propusieron condiciones muy suaves, adecuadas para la mayoría de las aplicaciones y desarrolladas para preservar posibles complejos de proteínas. Sin embargo, este método es altamente adaptable; el usuario puede elegir cualquier línea celular y cualquier tamaño de ARN, y podría decidir repetir el protocolo después del plegamiento / despliegue del ARN para determinar el efecto de la estructura en las propiedades de unión.
Otro aspecto original de este protocolo es el uso de herramientas de predicción in silico para asegurar la exactitud de los resultados20. Saber de antemano qué proteínas deben identificarse como interactores del ARN de interés da la ventaja sin precedentes de validar los aspectos técnicos del protocolo. Por ejemplo, utilizando un simple análisis WB, es posible verificar la presencia de una proteína diana conocida en las muestras derivadas de los diferentes pasos del protocolo antes de proceder con el análisis de MS, que requiere instrumentación especializada y es más costoso. Además, recientemente se informó de un método para utilizar catRAPID20, un algoritmo interno de predicción de proteína-ARN, para diseñar ARN de novo específico para una proteína diana. Hasta hace poco, la única tubería disponible para diseñar aptámeros de ADN/ARN para una proteína diana era el enfoque SELEX (evolución sistemática de ligandos por enriquecimiento exponencial)31. El método in silico permite un diseño mucho más rápido y rentable de aptámeros de ARN.
Las principales limitaciones de este método están asociadas con la necesidad de trabajar en tampones y herramientas libres de nucleasas. Además, si se considera necesario confirmar in vitro la unión entre un ARN diseñado de novo y una proteína diana antes de la EP, la proteína debe producirse y purificarse y determinarse la unión con enfoques biofísicos. Esta es una limitación que se comparte con la producción de anticuerpos monoclonales.
A pesar de estos problemas menores, los métodos confiables para mapear las interacciones ARN-proteína, como el que se presenta aquí, pueden acercar a los científicos a revelar redes macromoleculares y actores principales complejos de muchos mecanismos fisiológicos y patológicos, como los involucrados en el cáncer, las miocardiopatías, la diabetes, las infecciones microbianas y los trastornos genéticos y neurodegenerativos.
Divulgaciones
Los autores no tienen intereses financieros contrapuestos u otros conflictos de intereses.
Agradecimientos
Los autores desean agradecer al grupo de investigación del Prof. Tartaglia y al Dr. Cuomo por el apoyo ofrecido. E.Z. recibió fondos de la beca MINDED del programa de investigación e innovación Horizonte 2020 de la Unión Europea bajo el acuerdo de subvención Marie Sklodowska-Curie No. 754490.
Materiales
| Name | Company | Catalog Number | Comments |
| 6-well tissue culture plates | VWR | 10861-554 | CELLS |
| Cell scrapers | BIOSIGMA | 10153 | CELLS |
| Dulbecco′s Modified Eagle′s Medium (DMEM) | Thermo Fisher Scientfic | 11995065 | CELLS |
| Fetal Bovine Serum, qualified, heat inactivated, Brazil | Thermo Fisher Scientfic | 10500064 | CELLS |
| Phosphate Buffer Saline (PBS, Waltham, MA) | Thermo Fisher Scientfic | 14190169 | CELLS |
| Trypsin (0.25%), phenol red | Thermo Fisher Scientfic | 15050065 | CELLS |
| Anti-rabbit IgG horseradish peroxidase (HRP) | Cellsignal | 7070 | PD |
| Biotinylated RNA | Eurofins | Custom RNA oligonucleotides | PD |
| Bovine serum albumin | Sigma-Aldrich | A9418 | PD |
| Clarity Western ECL Substrate, 500 ml | Biorad | 1705061 | PD |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Merck - Sigma Aldrich | 5056489001 | PD |
| NuPAGE 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel, 10-well | Invitrogen | NP0321BOX | PD |
| Recombinant anti-TDP43 antibody | Abcam | ab109535 | PD |
| Ribonucleic acid, transfer from baker's yeast (S. cerevisiae) | Merck - Sigma Aldrich | R5636-1ML | PD |
| Streptavidin Mag Sepharose | Merck - Sigma Aldrich | GE28-9857-99 | PD |
| Trans-Blot Turbo RTA Mini 0.2 µm PVDF Transfer Kit | Biorad | 1704272 | PD |
| Acetone | Thermo Fisher Scientfic | 022928.K2 | MS |
| C18 cartridge | Thermo Fisher Scientfic | 13-110-018 | MS |
| Dithiothreitol (DTT) | Thermo Fisher Scientfic | 20290 | MS |
| EASY-Spray HPLC Columns | Thermo Scientific | ES902 | MS |
| iodoacetamide (IAA) | Sigma Aldrich S.r.l. | I6125 | MS |
| Lys-C/Trypsin | Promega | V5073 | MS |
| Trifluoroacetic acid (TFA) | Thermo Fisher Scientfic | 28904 | MS |
| Urea | Thermo Fisher Scientfic | J75826.A7 | MS |
| Equipment | |||
| ChemiDoc imaging system | Bio-Rad | CELLS | |
| Dyna Mag -2 , Magnetic rack | Invitrogen | CELLS | |
| Forma Series 3 water jacketed C02 incubator | Thermo Scientific | PD | |
| PROTEAN II xi cell , power supply for PAGE applications | Bio-Rad | PD | |
| Rotating wheel, rotator SB3 | Stuart | PD | |
| Water bath set at 37 °C | VWR | PD | |
| XCell SureLock Mini-Cell electrophoresis system | ThermoFisher Scientific | MS | |
| Easy-nLC 1200 UHPLC | Thermo Scientific | MS | |
| Q exactive Mass Spectrometer | Thermo Scientific | MS | |
| Software | Version | ||
| MaxQuant | 2.0.3.0 | MS | |
| Perseus | 1.6.14.0 | MS |
Referencias
- Gebauer, F., Schwarzl, T., Valcárcel, J., Hentze, M. W. RNA-binding proteins in human genetic disease. Nature Reviews Genetics. 22 (3), 185-198 (2021).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Cooper, T. A., Wan, L., Dreyfuss, G. RNA and disease. Cell. 136 (4), 777-793 (2009).
- Qin, H., et al. RNA-binding proteins in tumor progression. Journal of Hematology & Oncology. 13 (1), 90 (2020).
- Nussbacher, J. K., Tabet, R., Yeo, G. W., Lagier-Tourenne, C. Disruption of RNA metabolism in neurological diseases and emerging therapeutic interventions. Neuron. 102 (2), 294-320 (2019).
- Duan, R., Sharma, S., Xia, Q., Garber, K., Jin, P. Towards understanding RNA-mediated neurological disorders. Journal of Genetics and Genomics. 41 (9), 473-484 (2014).
- Maziuk, B., Ballance, H. I., Wolozin, B. Dysregulation of RNA binding protein aggregation in neurodegenerative disorders. Frontiers in Molecular Neuroscience. 10, 89 (2017).
- Zielinski, J., et al. In vivo identification of ribonucleoprotein-RNA interactions. Proceedings of the National Academy of Sciences. 103 (5), 1557-1562 (2006).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Weidmann, C. A., Mustoe, A. M., Jariwala, P. B., Calabrese, J. M., Weeks, K. M. Analysis of RNA-protein networks with RNP-MaP defines functional hubs on RNA. Nature Biotechnology. 39 (3), 347-356 (2021).
- Graindorge, A., et al. In-cell identification and measurement of RNA-protein interactions. Nature Communications. 10 (1), 5317 (2019).
- Ule, J., Hwang, H. W., Darnell, R. B. The future of cross-linking and immunoprecipitation (CLIP). Cold Spring Harbor Perspectives in Biology. 10 (8), 032243 (2018).
- Ascano, M., Hafner, M., Cekan, P., Gerstberger, S., Tuschl, T. Identification of RNA-protein interaction networks using PAR-CLIP. Wiley Interdisciplinary Reviews. RNA. 3 (2), 159-177 (2012).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Sugimoto, Y., et al. Analysis of CLIP and iCLIP methods for nucleotide-resolution studies of protein-RNA interactions. Genome Biology. 13 (8), (2012).
- Bayat, P., et al. SELEX methods on the road to protein targeting with nucleic acid aptamers. Biochimie. 154, 132-155 (2018).
- Armaos, A., Colantoni, A., Proietti, G., Rupert, J., Tartaglia, G. G. CatRAPID omics v2.0: Going deeper and wider in the prediction of protein-RNA interactions. Nucleic Acids Research. 49, 72-79 (2021).
- Agostini, F., et al. CatRAPID omics: A web server for large-scale prediction of protein-RNA interactions. Bioinformatics. 29 (22), 2928-2930 (2013).
- Zacco, E., et al. Probing TDP-43 condensation using an in silico designed aptamer. Nature Communications. 13 (1), 3306 (2022).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Zhang, Y., Lai, B. S., Juhas, M. Recent advances in aptamer discovery and applications. Molecules. 24 (5), 941 (2019).
- UniProt Consortium. UniProt: the Universal Protein Knowledgebase in 2023. Nucleic Acids Research. 51, 523-531 (2023).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Bellucci, M., Agostini, F., Masin, M., Tartaglia, G. G. Predicting protein associations with long noncoding RNAs. Nature Methods. 8 (6), 444-445 (2011).
- Gallo-Oller, G., Ordoñez, R., Dotor, J. A new background subtraction method for Western blot densitometry band quantification through image analysis software. Journal of Immunological Methods. 457, 1-5 (2018).
- Weissinger, R., Heinold, L., Akram, S., Jansen, R. P., Hermesh, O. RNA proximity labeling: A new detection tool for RNA-protein interactions. Molecules. 26 (8), 2270 (2021).
- Hirsch, J. D., et al. Easily reversible desthiobiotin binding to streptavidin, avidin, and other biotin-binding proteins: Uses for protein labeling, detection, and isolation. Analytical Biochemistry. 308 (2), 343-357 (2002).
- Sefah, K., Shangguan, D., Xiong, X., O'Donoghue, M. B., Tan, W. Development of DNA aptamers using cell-SELEX. Nature Protocols. 5 (6), 1169-1185 (2010).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados