Method Article
Un'analisi pull-down quantitativa ottimizzata delle proteine leganti l'RNA utilizzando RNA biotinilato corto
In questo articolo
Riepilogo
Qui, presentiamo un metodo in vitro ottimizzato per scoprire, quantificare e convalidare gli interattori proteici di specifiche sequenze di RNA, utilizzando estratto proteico totale da cellule umane, perle di streptavidina rivestite con RNA biotinilato e analisi di spettrometria di massa.
Abstract
Le interazioni proteina-RNA regolano l'espressione genica e le funzioni cellulari a livello trascrizionale e post-trascrizionale. Per questo motivo, identificare i partner di legame di un RNA di interesse rimane di grande importanza per svelare i meccanismi alla base di molti processi cellulari. Tuttavia, le molecole di RNA potrebbero interagire transitoriamente e dinamicamente con alcune proteine leganti l'RNA (RBP), specialmente con quelle non canoniche. Pertanto, sono estremamente necessari metodi migliori per isolare e identificare tali RBP.
Per identificare i partner proteici di una sequenza di RNA nota in modo efficiente e quantitativo, abbiamo sviluppato un metodo basato sul pull-down e la caratterizzazione di tutte le proteine interagenti, a partire dall'estratto proteico totale cellulare. Abbiamo ottimizzato il pull-down proteico utilizzando RNA biotinilato precaricato su perle rivestite di streptavidina. Come prova di concetto, abbiamo impiegato una breve sequenza di RNA nota per legare la proteina associata alla neurodegenerazione TDP-43 e un controllo negativo di una diversa composizione nucleotidica ma della stessa lunghezza. Dopo aver bloccato le perle con tRNA di lievito, abbiamo caricato le sequenze di RNA biotinilato sulle perle di streptavidina e le abbiamo incubate con l'estratto proteico totale dalle cellule HEK 293T. Dopo l'incubazione e diverse fasi di lavaggio per rimuovere i leganti non specifici, abbiamo eluito le proteine interagenti con una soluzione ad alto contenuto di sale, compatibile con i reagenti di quantificazione proteica più comunemente usati e con la preparazione del campione per la spettrometria di massa. Abbiamo quantificato l'arricchimento di TDP-43 nel pull-down eseguito con il legante RNA noto rispetto al controllo negativo mediante spettrometria di massa. Abbiamo usato la stessa tecnica per verificare le interazioni selettive di altre proteine computazionalmente previste come leganti unici del nostro RNA di interesse o del controllo. Infine, abbiamo convalidato il protocollo mediante western blot attraverso la rilevazione di TDP-43 con un anticorpo appropriato.
Questo protocollo consentirà lo studio dei partner proteici di un RNA di interesse in condizioni quasi fisiologiche, aiutando a scoprire interazioni proteina-RNA uniche e imprevedibili.
Introduzione
Le proteine leganti l'RNA (RBP) sono emerse come attori cruciali nella regolazione genica trascrizionale e post-trascrizionale, poiché sono coinvolte in processi come lo splicing dell'mRNA, la localizzazione cellulare dell'RNA, la traduzione, la modifica e la degradazione 1,2,3. Tali interazioni tra le due macromolecole sono altamente coordinate, bilanciate con precisione ed essenziali per la formazione di hub funzionali e di elaborazione. Variazioni o disregolazioni all'interno di questi hub hanno il potenziale di interrompere le reti proteina-RNA finemente regolate e sono sempre più associate a una varietà di malattie umane, tra cui il cancro 4,5 e le malattie neurodegenerative 6,7,8. Le interazioni tra le molecole di RNA e i loro partner leganti le proteine possono essere stabili e facili da convalidare sperimentalmente, o altamente dinamiche, transitorie e più difficili da caratterizzare.
Negli ultimi anni, sono stati intrapresi intensi sforzi per comprendere queste interazioni. Tra i metodi più consolidati, i saggi di pull-down proteico (PD) sono probabilmente gli approcci più apprezzati e comunemente usati per svelare i principali attori che costituiscono i complessi ribonucleoproteici (RNP) e altre reti di interazione proteina-RNA 3,9,10. I PD includono un ampio ombrello di tecniche informative, come l'immunoprecipitazione dell'RNA (RIP)11,12 o della proteina (CLIP)13,14 di interesse. Alcuni di questi protocolli RNA-PD impiegano un RNA noto come esca per le proteine15, più frequentemente sfruttando tag ad alta affinità come la biotina. In questo caso, i partner di interazione di un RNA biotinilato possono essere rilevati ancorando l'RNA su perle rivestite di streptavidina, consentendo un isolamento efficiente degli RNP. I principali limiti di questi approcci sono solitamente la progettazione delle sonde biotinilate e la verifica della loro capacità di legare le proteine bersaglio. A tal fine, potrebbe essere utile basarsi sui dati CLIP pubblicati della proteina di interesse, se disponibili, poiché rivelano, con alta precisione, le brevi regioni di RNA che corrispondono ai picchi di interazioni con la proteina bersaglio13,16. Queste stesse regioni potrebbero essere utilizzate per sviluppare sonde per PD. Un metodo alternativo per progettare tali esche a RNA potrebbe essere l'evoluzione sistematica dei ligandi mediante arricchimento esponenziale (SELEX)17, che consente la progettazione di aptameri attraverso la selezione in vitro, a partire da una libreria randomizzata completa e attraverso una serie di cicli di ottimizzazione guidati dalla PCR. Tuttavia, SELEX è complesso e richiede molto tempo e i risultati finali dipendono fortemente dalla libreria iniziale. Per selezionare l'esca a RNA da utilizzare nel protocollo qui presentato, è stato sfruttato un altro approccio, consistente nell'utilizzare un'esca a RNA progettata de novo mediante la potenza computazionale dell'algoritmo catRAPID, che prevede il legame preferenziale di una data proteina verso determinate sequenze di RNA18,19,20.
Il protocollo qui introdotto è una versione di un RNA-PD ottimizzato per eluire specifici partner proteici in condizioni quasi fisiologiche, senza l'uso di detergenti, agenti denaturanti o alte temperature. Si basa su perle nano-superparamagnetiche rivestite covalentemente con streptavidina altamente purificata e sull'uso di uno specifico RNA biotinilato progettato in silico come esca. Questo protocollo fornisce un metodo rapido ed efficiente per isolare i partner di legame delle molecole di RNA biotinilato in condizioni native, offrendo il potenziale per una vasta gamma di applicazioni a valle. Per testare questo protocollo, è stata utilizzata una sequenza di aptameri di RNA a singolo filamento a 10 nucleotidi, precedentemente progettata per legare la proteina TAR DNA-binding protein 43 (TDP-43) con alta affinità e specificità,20. A partire dai lisati delle cellule HEK 293T, gli interattori dell'aptamero di RNA biotinilato sono stati identificati mediante analisi di spettrometria di massa eseguite su campioni staccati dall'esca di RNA utilizzando un tampone ipertonico. Questa analisi ha confermato l'identificazione e la quantificazione con successo del TDP-43 come legante preferito.
Questo protocollo consente di identificare con successo gli interattori proteici utilizzando solo un breve oligonucleotide di RNA sintetizzato in vitro. Inoltre, l'uso di aptameri di RNA progettati in silico come sonde PD21,22 garantisce specificità per i bersagli a costi significativamente ridotti.
Protocollo
1. Metodi generali e materiale
- Preparare il terreno appropriato per la coltura cellulare di mammifero scelta e preriscaldarlo a 37 °C per 20 minuti prima dell'uso.
- Preparare in anticipo il materiale richiesto, come descritto nella tabella dei materiali. Vetreria in autoclave, articoli in plastica e buffer stock.
- Preparare i buffer come descritto nella tabella 1. Regolare il pH delle soluzioni madre utilizzando HCl concentrato o NaOH prima di diluire i componenti ai loro volumi finali.
2. Preparazione della linea cellulare di mammifero
- Coltivare cellule HEK 293T nel Modified Eagle Medium (DMEM) di Dulbecco integrato con siero bovino fetale al 10% (FBS) e 100 μg / mL di soluzione di penicillina / streptomicina. Incubarli a 37 °C in un incubatore umidificato alimentato con il 5% di CO2. Dividere regolarmente le celle.
- Prima di staccare, sciacquare le cellule con una soluzione salina tamponata con fosfato (PBS) sufficiente a coprire la superficie di crescita.
- Rimuovere il PBS e aggiungere uno strato ultrasottile di soluzione tripsina-EDTA.
- Incubare le cellule a 37 °C in un incubatore umidificato dotato di CO2 al 5% per 5 minuti, o fino a quando le cellule non sono staccate (dovrebbero apparire disperse sotto osservazione microscopica).
- Diluire la soluzione di tripsina-EDTA dieci volte aggiungendo DMEM completo per inattivarla e contare le cellule.
- Piastra 1,5 x 105 celle / ml in piastre a 6 pozzetti, considerando due pozzetti / condizione da testare.
- Incubare le celle a 37 °C per 48 ore in un incubatore umidificato con il 5% di CO2.
NOTA: Controllare le istruzioni del produttore per il tipo di mezzo e supplemento adatto per la linea cellulare. Inoltre, la quantità e il tempo di incubazione della tripsina-EDTA dipendono dalla linea cellulare. Alcuni tipi di cellule crescono più velocemente / più lentamente di quanto riportato in questo protocollo; Pertanto, la concentrazione della semina dovrebbe essere testata in anticipo.
3. Raccolta totale di proteine
- Rimuovere il mezzo dai pozzetti in cui crescono le cellule.
- Lavare ogni pozzetto delle piastre a 6 pozzetti con 1 mL di PBS.
- Scartare il PBS.
- Spostare le piastre sul ghiaccio e procedere con il punto 3.5 o congelare le piastre asciutte a -80 °C per facilitare la lisi.
- Aggiungere 200 μL di tampone di lisi a ciascun pozzetto.
- Utilizzare un raschietto cellulare per staccare e rompere le celle.
- Trasferire l'estratto cellulare derivante da due pozzetti nello stesso tubo da 1,5 ml.
- Posizionare il tubo contenente l'estratto proteico sul ghiaccio per 30 minuti.
- Centrifugare i lisati cellulari a 17.000 x g per 15 minuti a 4 °C.
- Trasferire ogni surnatante in un tubo pre-raffreddato.
NOTA: Si raccomanda un totale di 106-10 7 cellule per ogni condizione PD. La lisi cellulare e la raccolta delle proteine devono essere eseguite con tamponi ghiacciati. Gli inibitori della proteasi devono essere aggiunti al tampone di lisi per prevenire la degradazione delle proteine.
4. Determinazione della concentrazione proteica
- Preparare il reattivo Bradford, come indicato dal produttore, diluendolo cinque volte in dH2O.
- Distribuire 1 mL di reagente in una cuvetta da 1 cm, aggiungere 1 μL del campione e miscelare per inversione.
- Incubare al buio a temperatura ambiente per 5-10 min.
- Leggere l'assorbanza a 595 nm.
- Calcolare il volume di estratto proteico corrispondente a 1,5 mg di proteine e portare tutti i campioni ad un volume finale di 600 μL utilizzando tampone di lisi.
- Conservare i campioni sul ghiaccio fino all'uso.
NOTA: È possibile utilizzare qualsiasi altro metodo di determinazione della concentrazione proteica, seguendo le raccomandazioni di compatibilità del tampone. In ogni caso, il buffer di lisi deve essere usato come uno spazio vuoto. Molti reagenti non sono compatibili con il ditiotreitolo (DTT). Si raccomanda di aggiungere DTT o altri agenti riducenti solo dopo la quantificazione delle proteine (DTT a una concentrazione finale di 1 mM).
5. Preparazione delle perline
- Mescolare le perline nel loro buffer di stoccaggio facendo scorrere il tubo.
- Calcolare 100 μL di mezzo/campione di liquame e posizionare il volume in un rack magnetico.
- Lavaggio delle perline
- Rimuovere la soluzione di conservazione e lavare le perle aggiungendo 1 mL di tampone/tubo di lisi e capovolgerlo manualmente.
- Rimuovere il buffer utilizzando il rack magnetico.
- Ripetere la fase di lavaggio.
- Aggiungere un volume di tampone di lisi pari al volume iniziale del mezzo liquame, mescolare facendo scorrere il tubo ed erogare uniformemente il mezzo in tante provette da 1,5 mL quanti sono i campioni.
- Blocco delle perline
- Rimuovere il tampone utilizzando il rack magnetico e aggiungere 600 μL di una soluzione 0,25 mg/mL di tRNA di lievito preparata in tampone di lisi.
- Incubare per 1 ora a temperatura ambiente su una ruota rotante.
- Rimuovere la soluzione di tRNA utilizzando il rack magnetico.
- Aggiungere 600 μL di tampone di lisi e lavare mescolando manualmente.
- Ripetere la fase di lavaggio ed eliminare il tampone.
6. Caricamento delle perline
- Preparare 200 μg di oligonucleotide di RNA in 600 μL di tampone di lisi per ogni provetta contenente il mezzo di liquame iniziale da 100 μL (ora sfere bloccate).
- Aggiungere l'oligo alle perle e incubare per 1 ora a temperatura ambiente durante la rotazione.
- Rimuovere la soluzione, aggiungere 600 μL di tampone di lisi e lavare le perle due volte ruotando i tubi per 5 minuti a temperatura ambiente.
- Eliminare il buffer.
NOTA: Non vortice mai le perline, ma sfoglia invece. Limitare il numero di passaggi di pipettaggio, a meno che non sia necessario. Quando possibile, utilizzare il taglio, 1 mL di punte. La quantità di liquame di perline / campione dipende dalla capacità di legame delle perle e dalla quantità iniziale delle proteine totali. Se si prevede che l'oligo RNA abbia una quantità significativa di struttura secondaria, si consiglia di denaturarlo prima a 80 °C per 10 minuti e poi raffreddarlo lentamente a temperatura ambiente o ripiegarlo incubandolo a 30 °C per 1 ora. Si suggerisce di recuperare l'oligo di RNA dopo il caricamento delle perle e determinare la concentrazione residua, al fine di ottimizzare la quantità necessaria per il carico e valutare la possibilità di riutilizzare l'RNA.
7. Legame proteico sulle perle
NOTA: D'ora in poi, quando possibile, eseguire i passaggi a 4 °C.
- Prelevare un volume del 5% dalla soluzione proteica da 600 μL e tenerlo come INPUT (IN) per ulteriori analisi (1,5 mg di proteine vengono sciolti in 600 μL, quindi il 5% corrisponde a 30 μL e 75 μg di proteine).
- Aggiungere la miscela proteica rimanente a ciascun tubo di perline caricate e lasciare ruotare lentamente per una notte a 4 °C.
8. Lavaggio di leganti non specifici
- Rimuovere la frazione non legata utilizzando il rack magnetico. Risparmiare il 5% del volume ed etichettarlo come FLOWTHROUGH (FT) (il volume non legato è di circa 600 μL, quindi di nuovo conservare 30 μL per ulteriori analisi).
- Aggiungere 1 mL di tampone di lavaggio 1 alle perline e lasciarlo ruotare per 5 minuti a 4 °C.
- Eliminare il buffer.
- Ripetere i passaggi 8.2 e 8.3.
- Aggiungere 1 mL di tampone di lavaggio 2 alle perline e lasciarlo ruotare per 5 minuti a 4 °C.
- Scartare il surnatante.
9. Eluizione di leganti specifici
- Aggiungere 100 μL di tampone di eluizione 1 o tampone di eluizione 2 alle perle.
- Mescolare manualmente sfiorando e incubare per 5 minuti a temperatura ambiente.
- Mettere i tubi in un termomiscelatore e agitare vigorosamente per 5 minuti a 95 °C.
- Posizionare il tubo nel rack magnetico e raccogliere la frazione eluita in un tubo pulito.
- Girare rapidamente le perle con una centrifuga da banco per massimizzare il recupero dell'eluato.
- Risparmiare il 5% del volume totale di ELUATE (EL) per ulteriori analisi (il volume totale è di 100 μL, quindi separare 5 μL in un'altra provetta).
- Se necessario, la concentrazione proteica può essere determinata come nel paragrafo 4, usando il tampone di eluizione come bianco.
NOTA: Si raccomanda di astenersi dall'aggiungere DTT al tampone di eluizione fino a quando non sia stata determinata la concentrazione proteica. Se la quantificazione delle proteine non è richiesta, o se sono disponibili kit di quantificazione delle proteine compatibili con agenti riducenti, è possibile aggiungere 1 mM DTT al tampone di eluizione fin dall'inizio. In questo protocollo, sono stati testati sia il buffer di eluizione 1 (contenente 1 M NaCl) che il buffer di eluizione 2 (con 2 M NaCl). Nessuna differenza nell'efficienza di eluizione della proteina bersaglio è stata osservata con un aumento della forza ionica, ma si raccomanda di testare entrambe le condizioni prima di stabilire il tampone più appropriato. Se un'elevata presenza di sale nel tampone di eluizione rappresenta una limitazione per ulteriori analisi, la quantità molto bassa di detergenti nel tampone di eluizione consente lo scambio tampone. In alternativa, l'eluato può essere diluito per raggiungere la concentrazione salina desiderata.
10. Identificazione di leganti proteici mediante spettrometria di massa
- Precipitazione di acetone
- Concentrare le proteine eluite diluendole quattro volte in acetone freddo (-20 °C).
- Vortice e incubare il tubo a -20 °C durante la notte.
- Centrifugare a 17.000 x g per 30 minuti a 4 °C.
- Rimuovere delicatamente il surnatante e lasciare evaporare l'acetone fino a quando il pellet non si è completamente asciugato.
- Digestione delle proteine in soluzione
- Sciogliere il pellet proteico aggiungendo 50 μL di tampone di denaturazione.
- Aggiungere DTT a una concentrazione finale di 5 mM, consentendo la riduzione delle proteine per 30 minuti a 55 °C.
- Raffreddare i campioni a temperatura ambiente e procedere con la reazione di alchilazione proteica, aggiungendo iodoacetamide (IAA) ad una concentrazione di 10 mM per 15 min.
- Digerire le proteine utilizzando un enzima adatto (tripsina, LysC) e incubare i campioni per una notte a 37 °C.
- Interrompere la digestione aggiungendo 1 μL di acido trifluoroacetico al 10% (TFA).
- Pulire e concentrare i peptidi su una micro-colonna C18 a fase invertita personalizzata, come descritto in precedenza19.
- Eluire i peptidi dalla punta C18 con tampone B.
- Rimuovere il componente organico utilizzando una centrifuga sottovuoto e risospendere i peptidi in 5 μL di acido formico allo 0,1% per ulteriori analisi.
NOTA: In alternativa, la digestione delle proteine può essere eseguita "on-beads" immediatamente dopo il lavaggio di leganti non specifici (passaggi 8.1-8.6), rendendo così il protocollo più veloce. Tuttavia, è consigliabile testare l'efficienza dell'enzima lavorando proteine immobilizzate "on-beads" rispetto allo standard nella digestione in soluzione, al fine di garantire la copertura ottimale della sequenza proteica sperimentale.
- Cromatografia liquida-spettrometria di massa tandem (LC-MS/MS)
- Collegare la colonna analitica (fase stazionaria C18) e mantenerla a 45 °C durante la corsa.
- Collegare la colonna all'uscita di una valvola rotativa a sei vie della pompa LC attraverso un raccordo capillare a tenuta di dita (20 μm x 550 mm) in una configurazione a colonna singola.
- Regolare le impostazioni LC come segue:
- Caricare i peptidi a pressione controllata (980 bar) nel tampone A.
- Applicare un gradiente Buffer B del 5%-20% a 300 nL/min su 59 minuti, seguito da un gradiente Buffer B del 20%-30% su 15 min e un gradiente Buffer B del 30%-65% su 5 min.
- Aggiungere una fase di lavaggio aumentando la concentrazione di Buffer B fino al 95% su 5 min più una fase isocratica di 5 min al 95% Buffer B.
- Utilizzare lo spettrometro di massa in modalità DDA (Data-Dependent Acquisition) per passare automaticamente tra eventi MS e MSMS.
- Definire un numero di loop pari a 15 utilizzando un valore target di controllo automatico del guadagno (AGC) di 3 x 106 e 1 x 105 rispettivamente per gli eventi MS e MSMS.
- Impostare il tempo massimo di accumulo di ioni consentito su 20 ms per MS con una risoluzione di 60 K e 100 ms per MSMS con una risoluzione di 15 K.
- Eseguire un esperimento di frammentazione ad alta dissociazione delle collisioni (HCD) utilizzando un'energia di collisione normalizzata del 28%, con un tempo di esclusione dinamica di 20 s.
- Gestire i parametri di origine come segue:
Tensione di spruzzo: 1,7 kV
Tensione capillare: 275 °C
Né guaina né gas ausiliario utilizzati
NOTA: In questo protocollo, l'analisi spettrometrica di massa (MS) per cromatografia liquida ad altissime prestazioni (UHPLC) è stata eseguita specificamente utilizzando una configurazione LC a colonna singola, accoppiata a uno strumento orbitrap ibrido a triplo quadrupolo (Table of Materials). È possibile utilizzare altri sistemi LCMS, ma si raccomanda un adattamento dei parametri.
- Analisi dei dati
- Utilizzate il pulsante Carica per importare i file raw.
- Definisci i nomi degli esperimenti facendo clic sul pulsante Imposta esperimento .
- Accedere alla sezione dei parametri specifici del gruppo per specificare tutti i parametri relativi all'identificazione:
Enzima utilizzato per la digestione: Tripsina/P
Scollature mancate: fino a tre
Modifica fissa: Carbamidomethylation
Modifica variabile: N-acetil (Proteina), Ossidazione (M) - Caricare un file FASTA aggiornato, disponibile da database pubblici come UniprotKB.
- Specificare le regole di analisi corrette in base all'origine del database scelto.
- Definire un valore FDR (Parentage False Discovery Rate) = 1 sia per le proteine che per i peptidi.
- Aggiungere l'opzione Quantificazione senza etichetta (LFQ) nella scheda Quantificazione senza etichetta.
- Mantenere il conteggio minimo del rapporto LFQ a due.
NOTA: Qui descriviamo l'analisi dei dati utilizzando MaxQuant24 e il software Perseus25 per eseguire rispettivamente la quantificazione delle proteine e la successiva analisi statistica. Tuttavia, l'analisi dei dati può essere eseguita con qualsiasi altro bioinformatico disponibile in commercio o gratuito. FDR è stimato utilizzando un approccio basato su database target-decoy26. FDR peptidico e proteico pari a 0,01 significa che i peptidi e le proteine identificati dovrebbero contenere l'1% di falsi positivi.
- Analisi statistica
- Caricare il file proteingroups.txt per eseguire analisi statistiche a livello proteico.
- Definire i valori LFQ come colonne principali.
- Rimuovere "reverse" e "contaminanti" filtrando le righe in base alla colonna categoriale.
- Utilizzare le righe di annotazione categoriche per raggruppare le diverse condizioni sperimentali.
- Ridurre la matrice di dati, selezionando il numero di valori validi in ciascuno dei gruppi definiti in precedenza.
- Selezionare il test statistico che meglio si adatta alle condizioni sperimentali (ad esempio, t-test, test a campione multiplo ANOVA).
- Determinare un cut-off per suggerimenti significativi con un calcolo basato su FDR. In genere, sia 0,01 che 0,05 sono accettati come soglie per il valore p regolato.
- Visualizza i risultati delle analisi differenziali basate sulle statistiche del test t utilizzando una rappresentazione del grafico del vulcano.
- Esportare la matrice finale in .txt formato per ulteriori modifiche della tabella dei risultati finali.
NOTA: la cartella di configurazione contiene un file FASTA con proteine come le cheratine che sono considerate contaminanti comuni negli esperimenti di proteomica globale, che sono contrassegnati con un + nella tabella di output. Nel presente studio, avendo due condizioni campione, viene utilizzato un t-test per l'analisi statistica.
11. Convalida dei risultati mediante western blot
- Preparazione del campione
- Aggiungere il volume appropriato di buffer di caricamento del campione 4x a ciascuna aliquota di IN, FT ed EL.
- Far bollire i campioni per 5 minuti a 95°C.
- Centrifuga rapida per recuperare il campione evaporato dalla parte superiore dei tubi.
- SDS-PAGE e trasferimento gel
- Caricare i campioni su un gel di poliacrilammide denaturante al 4% -12%.
- Far funzionare il gel con tampone MES SDS per 1,5 ore a 120 V.
- Trasferire il gel su una membrana di nitrocellulosa con una cassetta di trasferimento semi-secca, seguendo le istruzioni del produttore. Si consiglia un trasferimento di 10 minuti a 15 V.
- Immunorilevazione
- Bloccare la membrana con albumina sierica bovina (BSA) al 10% per 1 ora a temperatura ambiente agitando delicatamente.
- Aggiungere l'anticorpo primario preparato in 5% BSA in TBST, secondo le istruzioni del produttore. Lasciare per una notte a 4 °C o per 1 ora a temperatura ambiente sotto leggero agitazione.
- Lavare la membrana tre volte con TBST, ogni volta per 5 minuti.
- Aggiungere l'anticorpo secondario preparato in TBST per 1 ora a temperatura ambiente sotto agitazione.
- Lavare la membrana tre volte con TBST, ogni volta per 5 minuti.
- Visualizza i risultati utilizzando un imager blot.
NOTA: Per rilevare TDP-43, è stato utilizzato un legante noto del nostro RNA, l'anticorpo monoclonale di coniglio ricombinante che è stato lasciato con la membrana per una notte a 4 °C. Come anticorpo secondario, è stata utilizzata la perossidasi di rafano IgG anti-coniglio (HRP), ma anche un anticorpo secondario fluorescente funzionerebbe. Per visualizzare gli anticorpi sulla membrana, la membrana è stata incubata con il substrato Clarity Western ECL per 1 minuto, prima di eseguire l'imaging con il sistema di imaging ChemiDoc.
Risultati
Per verificare la validità del protocollo proposto, gli esperimenti PD qui presentati sono stati eseguiti con un aptamero di RNA biotinilato progettato in silico per legare specificamente TDP-4320. Questo RNA lega il suo bersaglio proteico con un'elevata affinità di legame (Kd = 90 nM)20. Qui, questo RNA, di sequenza 5'-CGGUGUUGCU-3', è indicato con il nome "+RNA". Come controllo negativo, è stata utilizzata la sequenza complementare inversa di +RNA, che è qui chiamata "-RNA". La sua sequenza è 5'-AGCAACACCG-3'. -L'RNA mostra un'affinità di legame significativamente inferiore verso TDP-43 (Kd = 1,5 μM)19. Ai fini del protocollo qui descritto, questi oligonucleotidi di RNA sono stati acquistati coniugati ad una molecola di biotina, per consentire il legame alle perle di streptavidina. +RNA è stato acquistato con una biotina-TEG alla sua estremità 3', che include uno spaziatore di glicole trietilenico a 15 atomi tra la biotina e il gruppo fosfato dell'acido nucleico; -RNA invece aveva una biotina alla sua estremità 5', coniugata all'acido nucleico tramite un linker ammino-C6. Tuttavia, se il design dell'esca RNA è robusto, e fintanto che non vi è alcuna interferenza strutturale o chimica tra il linker e l'RNA, potrebbero essere impiegate altre posizioni per la coniugazione della biotina e altre lunghezze del linker.
Conoscere l'identità della proteina principale da trovare legata alla sonda +RNA dopo il PD ha permesso la validazione del protocollo mediante identificazione di TDP-43 nell'eluato, utilizzando sia la spettrometria di massa (MS) che il western blot (WB) (Figura 1).
L'analisi della SM è stata effettuata su quattro repliche di PD eseguite utilizzando +RNA o -RNA (Figura 2). L'identificazione degli interattomi di +RNA e -RNA va oltre lo scopo di questo protocollo, tuttavia vengono riportati alcuni risultati che convalidano l'accuratezza del protocollo. Da notare, tracciare le proteine significativamente arricchite in un grafico vulcanico ha rivelato che il contenuto proteico totale e le proteine arricchite eluite da +RNA erano significativamente più alte di quelle recuperate da -RNA (Figura 2). Ciò significa che, pur avendo la stessa lunghezza e contenuto strutturale (lineare), +RNA può stabilire un numero maggiore di interazioni specifiche, che vengono mantenute fino alla fase di eluizione con alto contenuto di sale. È probabile che -RNA stabilisca invece un numero maggiore di contatti non specifici che vengono interrotti durante le fasi di lavaggio. Come previsto, TDP-43 è stato identificato come un unico interagente di +RNA20; la quantificazione media label-free (LFQ) per le quattro repliche PD eseguite con +RNA è 31,96 ± 0,56, mentre la proteina non è identificata tra gli interattori di -RNA. Inoltre, tra tutti gli interattori unici di +RNA, TDP-43 è risultata essere la proteina più abbondantemente arricchita.
Per convalidare ulteriormente il protocollo, l'algoritmo interno catRAPID18,19 è stato utilizzato per prevedere computazionalmente quali altre proteine si legherebbero specificamente +RNA o -RNA. In particolare, i punteggi di interazione per +RNA e -RNA con le proteine che compongono il proteoma umano sono stati calcolati utilizzando la funzione catRAPID "propensione all'interazione", come definito nel nostro precedente lavoro27. Tra le proteine valutate con elevata confidenza, è stato previsto che HNRNPH3 si leghi selettivamente +RNA (+RNA interaction score = 1,01; -RNA interaction score = 0,21) e PCBP2 interagisca specificamente con -RNA (+RNA interaction score = -0.5; -RNA interaction score = 0.31) (Figura 3A). Inoltre, è stato previsto che la proteina RBM41 sia promiscua per entrambi gli oligonucleotidi dell'RNA (+RNA interaction score = 0.4; -RNA interaction score = 0.39) (Figura 3A). L'analisi della SM ha infatti confermato la presenza di HNRNPH3 e PCBP2 nella PD rispettivamente di +RNA e -RNA, mentre RBM41 è stato trovato interagire con entrambi (Figura 3B).
WB è stato utilizzato per rilevare la presenza di TDP-43 per confermare ulteriormente i risultati e durante l'ottimizzazione del protocollo (Figura 4). Nella procedura qui descritta, sono stati raccolti campioni diversi in fasi diverse. Il campione di input (IN) era costituito dalle proteine totali diluite nel tampone di lisi. Il flowthrough (FT) è stato ottenuto dopo un'incubazione notturna delle proteine totali con le perle di streptavidina pre-rivestite con l'RNA biotinilato, che rappresenta la frazione di proteine che non legano l'RNA. Infine, l'eluato (EL) conteneva tutte le proteine che riconoscevano specificamente l'RNA in esame, poiché tra le fasi FT e EL tre fasi di lavaggio con 150 mM di sale e 0,1% di tritone-X avrebbero dovuto rimuovere le interazioni più deboli.
Per ogni replica, la stessa quantità (5% v/v) di IN, FT ed EL è stata eseguita in parallelo su una SDS-PAGE e colorata con un anticorpo anti-TDP-43 (Figura 4). Nel caso di +RNA, la banda di TDP-43 è stata osservata in IN e in EL, indicando che la proteina, presente fin dall'inizio nell'estratto proteico totale, viene trattenuta da +RNA durante le fasi di lavaggio e viene eluita solo alla fine con un tampone salino elevato. TDP-43 era presente anche in IN per -RNA, tuttavia la banda corrispondente alla proteina è visibile anche in FT, indicando che questo RNA non lega TDP-43. L'assenza della banda TDP-43 in EL conferma questo risultato.
Durante l'ottimizzazione del protocollo, l'eluizione delle proteine specificamente legate alle sequenze di RNA è stata sondata sia con un tampone di eluizione contenente 1 M NaCl (EB1) sia con un tampone di eluizione completo di 2 M NaCl (EB2) (Figura 4). Gli eluati ottenuti con entrambi gli EB sono stati confrontati su una SDS-PAGE e violati con l'anticorpo anti-TDP-43. Le immagini ottenute sono state poi analizzate con ImageJ28 per quantificare qualsiasi differenza nella quantità di TDP-43 eluita con i due buffer. Nel complesso, non è stata osservata alcuna differenza significativa e abbiamo concluso che, all'interno di questi test, 1 M di sale è sufficiente per interrompere anche le interazioni proteina-RNA più forti.
Nel complesso, i risultati qui riportati per SM e WB dimostrano che questo protocollo è efficiente nel catturare gli interattori proteici di un dato RNA in un modo specifico e che consente l'eluizione in tamponi compatibili con l'analisi a valle.
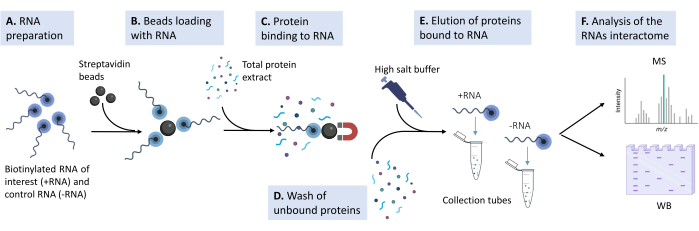
Figura 1: Schizzo della pipeline sperimentale utilizzata nel protocollo proposto . (A) L'oligonucleotide di RNA biotinilato viene preparato in tampone di lisi alla concentrazione appropriata. (B) Le perle magnetiche di streptavidina vengono lavate, bloccate con tRNA di lievito e caricate con l'RNA biotinilato. (C) L'estratto proteico totale derivato da linee cellulari di mammifero in coltura viene aggiunto alla miscela perline-RNA. (D) Vengono eseguiti più lavaggi per rimuovere interazioni non specifiche. (E) Gli interattori proteici specifici sono staccati dall'RNA con una soluzione ipertonica. (F) L'identità degli interattori è rivelata dalla spettrometria di massa e casi specifici sono convalidati dal western blot. Fare clic qui per visualizzare una versione ingrandita di questa figura.
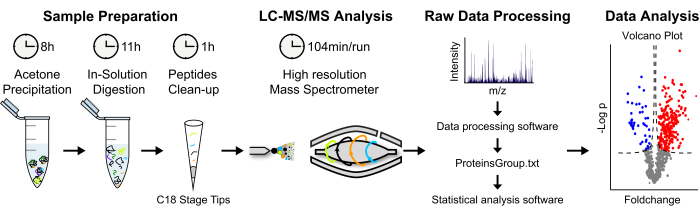
Figura 2: Strategia analitica per la quantificazione delle proteine basate su SM label-free . (A) Le proteine eluite vengono precipitate in acetone freddo durante la notte. Le proteine vengono quindi denaturate e viene eseguita una digestione in soluzione. I peptidi proteolitici sono concentrati e dissalati. (B) I peptidi vengono analizzati tramite LC-MS/MS utilizzando un "approccio shotgun". (C) L'elaborazione e l'analisi dei dati grezzi vengono eseguite utilizzando rispettivamente i software MaxQuant e Perseus. (D) Le proteine arricchite statisticamente significative sono visualizzate in un grafico vulcanico. Fare clic qui per visualizzare una versione ingrandita di questa figura.
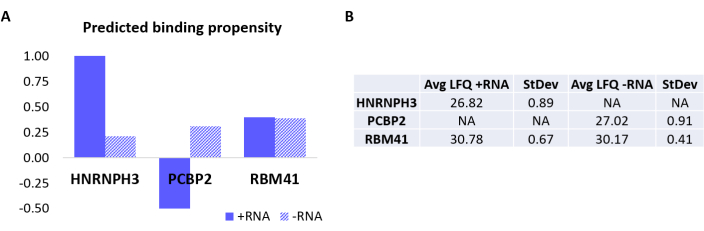
Figura 3: Correlazione tra le propensioni all'interazione previste e le interazioni determinate sperimentalmente di +RNA e -RNA. (A) punteggidi interazione RAPID di cat relativi a HNRNPH3, PCBP2 e RBM41, indicando il legame preferenziale di HNRNPH3 per +RNA e di PCBP2 per -RNA, mentre si prevede che RBM41 leghi indiscriminatamente entrambe le sequenze di RNA. (B) Medie di quantificazione label-free determinate mediante analisi di spettrometria di massa dai pull-down eseguiti con +RNA e -RNA. L'analisi conferma che HNRNPH3 lega esclusivamente +RNA, PCBP2 lega esclusivamente -RNA e RBM41 lega entrambi allo stesso modo. Fare clic qui per visualizzare una versione ingrandita di questa figura.
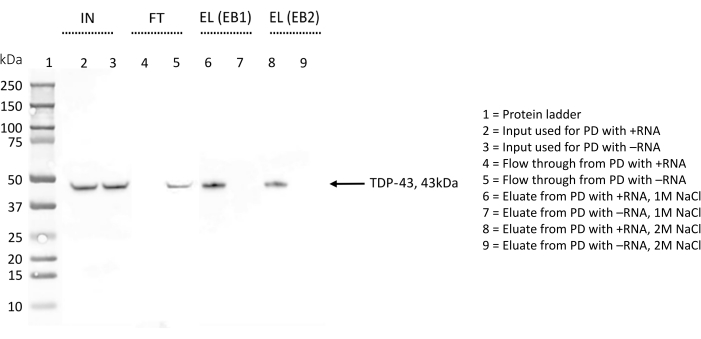
Figura 4: Validazione Western blot della presenza/assenza di TDP-43 tra gli interattori delle sequenze di RNA scelte. La membrana WB è stata trattata con anticorpi anti-TDP-43. IN = input; FT = flusso continuo; EL (EB1) = eluizione con buffer di eluizione 1; EL (EB2) = eluizione con tampone di eluizione 2; il segno "+" indica campioni derivati dal pull-down eseguito con +RNA; il segno "-" indica campioni derivati dal pull-down eseguito con -RNA; La corsia 1 contiene una scala proteica. TDP-43 è indicato da una freccia. Il WB indica che TDP-43 si trova tra gli interattori +RNA ma non tra gli interattori -RNA. Fare clic qui per visualizzare una versione ingrandita di questa figura.
| Nome buffer | Composizione | |||||
| Buffer di trasferimento 10x | 250 mM tris, 1,92 M glicina, 1% SDS, 20% metanolo. Diluire 10 volte prima dell'uso | PD | ||||
| Buffer di esecuzione 20X MES SDS | 1 M MES, 1 M tris, 2% SDS, 20 mM EDTA. Regolare il pH a 7,3. Diluire 20 volte prima dell'uso | |||||
| 4x buffer di caricamento del campione | 0,25 M Tris base, 0,28 M SDS, 40% glicerolo, 20% 2-mercapto-etanolo, 4 mg/ml blu di bromofenolo | |||||
| Tampone di eluizione 1 | 20 mM fosfato pH 7,5, 1 M NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM DTT (da aggiungere previa quantificazione) | |||||
| Buffer di eluizione 2 | 20 mM fosfato pH 7,5, 2 M NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM DTT (da aggiungere previa quantificazione) | |||||
| Tampone di lisi | 10 mM Tris-HCl pH 7,4, 150 mM NaCl, 0,5 mM EDTA, 0,1% Triton X-100, 1 mM DTT e inibitori della proteasi | |||||
| Soluzione salina tris-tamponata con Tween-20 | 1 M Tris-HCl pH 7,4, 3 M NaCl, 2,0% Tween-20 | |||||
| Tampone di lavaggio 1 | 10 mM Tris-HCl pH 7,4, 150 mM NaCl, 0,5 mM EDTA, 0,1 % Triton TM X-100, 1 mM DTT e inibitori della proteasi | |||||
| Tampone di lavaggio 2 | 25 mM Hepes pH 8, 150 mM NaCl, 0,5 mM EDTA, 0,1% Triton X-100, 1 mM DTT e inibitori della proteasi | |||||
| Buffer A | 0,1% acido formico | MS | ||||
| Buffer B | 60% acetonitrile, 0,1% acido formico | |||||
| Tampone di denaturazione | 8M urea, 50 mM Tris-HCl | |||||
Tabella 1: tamponi PD e MS. Nomi e composizione dei tamponi utilizzati per gli esperimenti di pull-down (PD) o per l'analisi di spettrometria di massa (MS).
Discussione
Questo lavoro riporta l'ottimizzazione di un protocollo PD eseguito con oligonucleotidi di RNA biotinilato per catturare i loro interattori proteici. Il protocollo qui descritto è semplice da eseguire, richiede poco materiale e produce risultati altamente affidabili. È importante sottolineare che gli aspetti più innovativi di questo protocollo consistono nell'uso di un'esca a RNA progettata completamente in silico e specifica per il bersaglio proteico, e l'eluizione di tutte le proteine legate all'esca di RNA interrompendo direttamente le loro interazioni con una soluzione ad alto contenuto di sale, piuttosto che dissociando la streptavidina dalla biotina con detergente e trattamento ad alta temperatura.
Questo protocollo sfrutta la forza del legame tra biotina e streptavidina29,30. Secondo le sfere di streptavidina scelte, il carico dell'RNA biotinilato deve essere testato e quantificato prima di procedere. Inoltre, il ripiegamento tridimensionale dell'RNA potrebbe influenzare l'efficienza di caricamento sulle perline, poiché potrebbe limitare l'esposizione della biotina alla streptavidina. Bloccare le perle con tRNA non biotinilato migliora la pulizia dei risultati limitando le interazioni non specifiche con le perle. Il buffer di carico e il buffer di eluizione devono essere scelti a seconda delle applicazioni a valle. Qui sono state proposte condizioni molto miti, adatte alla maggior parte delle applicazioni e sviluppate per preservare potenziali complessi proteici. Questo metodo è tuttavia altamente adattabile; l'utente può scegliere qualsiasi linea cellulare e qualsiasi dimensione dell'RNA, e potrebbe decidere di ripetere il protocollo dopo il folding/unfolding dell'RNA per determinare l'effetto della struttura sulle proprietà di legame.
Un altro aspetto originale di questo protocollo è l'uso di strumenti di previsione in silico per garantire la correttezza dei risultati20. Sapere in anticipo quali proteine devono essere identificate come interattori dell'RNA di interesse dà il vantaggio senza precedenti di validare gli aspetti tecnici del protocollo. Ad esempio, utilizzando una semplice analisi WB, è possibile verificare la presenza di un bersaglio proteico noto nei campioni derivati dai diversi passaggi del protocollo prima di procedere con l'analisi MS, che richiede strumentazione specializzata ed è più costosa. Inoltre, è stato recentemente riportato un metodo per utilizzare catRAPID20, un algoritmo interno di predizione proteina-RNA, per progettare RNA de novo specifico per una proteina bersaglio. Fino a poco tempo fa, l'unica pipeline disponibile per progettare aptameri DNA/RNA per una proteina bersaglio era l'approccio SELEX (systematic evolution of ligands by exponential enrichment)31. Il metodo in silico consente una progettazione molto più rapida ed economica degli aptameri di RNA.
Le principali limitazioni di questo metodo sono associate alla necessità di lavorare in buffer e strumenti privi di nucleasi. Inoltre, se si ritiene necessario confermare in vitro il legame tra un RNA progettato de novo e una proteina bersaglio prima della PD, la proteina deve essere prodotta e purificata e il legame determinato con approcci biofisici. Questa è una limitazione condivisa con la produzione di anticorpi monoclonali.
Nonostante questi problemi minori, metodi affidabili per mappare le interazioni RNA-proteina, come quello qui presentato, possono avvicinare gli scienziati a svelare reti macromolecolari e attori principali complessi di molti meccanismi fisiologici e patologici, come quelli coinvolti nel cancro, cardiomiopatie, diabete, infezioni microbiche e disturbi genetici e neurodegenerativi.
Divulgazioni
Gli autori non hanno interessi finanziari concorrenti o altri conflitti di interesse.
Riconoscimenti
Gli autori ringraziano il gruppo di ricerca del Prof. Tartaglia e del Dr. Cuomo per il supporto offerto. E.Z. ha ricevuto finanziamenti dalla borsa di studio MINDED del programma di ricerca e innovazione Horizon 2020 dell'Unione Europea nell'ambito dell'accordo di sovvenzione Marie Sklodowska-Curie n. 754490.
Materiali
| Name | Company | Catalog Number | Comments |
| 6-well tissue culture plates | VWR | 10861-554 | CELLS |
| Cell scrapers | BIOSIGMA | 10153 | CELLS |
| Dulbecco′s Modified Eagle′s Medium (DMEM) | Thermo Fisher Scientfic | 11995065 | CELLS |
| Fetal Bovine Serum, qualified, heat inactivated, Brazil | Thermo Fisher Scientfic | 10500064 | CELLS |
| Phosphate Buffer Saline (PBS, Waltham, MA) | Thermo Fisher Scientfic | 14190169 | CELLS |
| Trypsin (0.25%), phenol red | Thermo Fisher Scientfic | 15050065 | CELLS |
| Anti-rabbit IgG horseradish peroxidase (HRP) | Cellsignal | 7070 | PD |
| Biotinylated RNA | Eurofins | Custom RNA oligonucleotides | PD |
| Bovine serum albumin | Sigma-Aldrich | A9418 | PD |
| Clarity Western ECL Substrate, 500 ml | Biorad | 1705061 | PD |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Merck - Sigma Aldrich | 5056489001 | PD |
| NuPAGE 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel, 10-well | Invitrogen | NP0321BOX | PD |
| Recombinant anti-TDP43 antibody | Abcam | ab109535 | PD |
| Ribonucleic acid, transfer from baker's yeast (S. cerevisiae) | Merck - Sigma Aldrich | R5636-1ML | PD |
| Streptavidin Mag Sepharose | Merck - Sigma Aldrich | GE28-9857-99 | PD |
| Trans-Blot Turbo RTA Mini 0.2 µm PVDF Transfer Kit | Biorad | 1704272 | PD |
| Acetone | Thermo Fisher Scientfic | 022928.K2 | MS |
| C18 cartridge | Thermo Fisher Scientfic | 13-110-018 | MS |
| Dithiothreitol (DTT) | Thermo Fisher Scientfic | 20290 | MS |
| EASY-Spray HPLC Columns | Thermo Scientific | ES902 | MS |
| iodoacetamide (IAA) | Sigma Aldrich S.r.l. | I6125 | MS |
| Lys-C/Trypsin | Promega | V5073 | MS |
| Trifluoroacetic acid (TFA) | Thermo Fisher Scientfic | 28904 | MS |
| Urea | Thermo Fisher Scientfic | J75826.A7 | MS |
| Equipment | |||
| ChemiDoc imaging system | Bio-Rad | CELLS | |
| Dyna Mag -2 , Magnetic rack | Invitrogen | CELLS | |
| Forma Series 3 water jacketed C02 incubator | Thermo Scientific | PD | |
| PROTEAN II xi cell , power supply for PAGE applications | Bio-Rad | PD | |
| Rotating wheel, rotator SB3 | Stuart | PD | |
| Water bath set at 37 °C | VWR | PD | |
| XCell SureLock Mini-Cell electrophoresis system | ThermoFisher Scientific | MS | |
| Easy-nLC 1200 UHPLC | Thermo Scientific | MS | |
| Q exactive Mass Spectrometer | Thermo Scientific | MS | |
| Software | Version | ||
| MaxQuant | 2.0.3.0 | MS | |
| Perseus | 1.6.14.0 | MS |
Riferimenti
- Gebauer, F., Schwarzl, T., Valcárcel, J., Hentze, M. W. RNA-binding proteins in human genetic disease. Nature Reviews Genetics. 22 (3), 185-198 (2021).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Cooper, T. A., Wan, L., Dreyfuss, G. RNA and disease. Cell. 136 (4), 777-793 (2009).
- Qin, H., et al. RNA-binding proteins in tumor progression. Journal of Hematology & Oncology. 13 (1), 90 (2020).
- Nussbacher, J. K., Tabet, R., Yeo, G. W., Lagier-Tourenne, C. Disruption of RNA metabolism in neurological diseases and emerging therapeutic interventions. Neuron. 102 (2), 294-320 (2019).
- Duan, R., Sharma, S., Xia, Q., Garber, K., Jin, P. Towards understanding RNA-mediated neurological disorders. Journal of Genetics and Genomics. 41 (9), 473-484 (2014).
- Maziuk, B., Ballance, H. I., Wolozin, B. Dysregulation of RNA binding protein aggregation in neurodegenerative disorders. Frontiers in Molecular Neuroscience. 10, 89 (2017).
- Zielinski, J., et al. In vivo identification of ribonucleoprotein-RNA interactions. Proceedings of the National Academy of Sciences. 103 (5), 1557-1562 (2006).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Weidmann, C. A., Mustoe, A. M., Jariwala, P. B., Calabrese, J. M., Weeks, K. M. Analysis of RNA-protein networks with RNP-MaP defines functional hubs on RNA. Nature Biotechnology. 39 (3), 347-356 (2021).
- Graindorge, A., et al. In-cell identification and measurement of RNA-protein interactions. Nature Communications. 10 (1), 5317 (2019).
- Ule, J., Hwang, H. W., Darnell, R. B. The future of cross-linking and immunoprecipitation (CLIP). Cold Spring Harbor Perspectives in Biology. 10 (8), 032243 (2018).
- Ascano, M., Hafner, M., Cekan, P., Gerstberger, S., Tuschl, T. Identification of RNA-protein interaction networks using PAR-CLIP. Wiley Interdisciplinary Reviews. RNA. 3 (2), 159-177 (2012).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Sugimoto, Y., et al. Analysis of CLIP and iCLIP methods for nucleotide-resolution studies of protein-RNA interactions. Genome Biology. 13 (8), (2012).
- Bayat, P., et al. SELEX methods on the road to protein targeting with nucleic acid aptamers. Biochimie. 154, 132-155 (2018).
- Armaos, A., Colantoni, A., Proietti, G., Rupert, J., Tartaglia, G. G. CatRAPID omics v2.0: Going deeper and wider in the prediction of protein-RNA interactions. Nucleic Acids Research. 49, 72-79 (2021).
- Agostini, F., et al. CatRAPID omics: A web server for large-scale prediction of protein-RNA interactions. Bioinformatics. 29 (22), 2928-2930 (2013).
- Zacco, E., et al. Probing TDP-43 condensation using an in silico designed aptamer. Nature Communications. 13 (1), 3306 (2022).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Zhang, Y., Lai, B. S., Juhas, M. Recent advances in aptamer discovery and applications. Molecules. 24 (5), 941 (2019).
- UniProt Consortium. UniProt: the Universal Protein Knowledgebase in 2023. Nucleic Acids Research. 51, 523-531 (2023).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Bellucci, M., Agostini, F., Masin, M., Tartaglia, G. G. Predicting protein associations with long noncoding RNAs. Nature Methods. 8 (6), 444-445 (2011).
- Gallo-Oller, G., Ordoñez, R., Dotor, J. A new background subtraction method for Western blot densitometry band quantification through image analysis software. Journal of Immunological Methods. 457, 1-5 (2018).
- Weissinger, R., Heinold, L., Akram, S., Jansen, R. P., Hermesh, O. RNA proximity labeling: A new detection tool for RNA-protein interactions. Molecules. 26 (8), 2270 (2021).
- Hirsch, J. D., et al. Easily reversible desthiobiotin binding to streptavidin, avidin, and other biotin-binding proteins: Uses for protein labeling, detection, and isolation. Analytical Biochemistry. 308 (2), 343-357 (2002).
- Sefah, K., Shangguan, D., Xiong, X., O'Donoghue, M. B., Tan, W. Development of DNA aptamers using cell-SELEX. Nature Protocols. 5 (6), 1169-1185 (2010).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon