Method Article
짧은 비오티닐화 RNA를 사용한 RNA 결합 단백질의 최적화된 정량적 풀다운 분석
요약
여기에서 우리는 인간 세포의 총 단백질 추출물, 비오티닐화 RNA로 코팅된 스트렙타비딘 비드 및 질량 분석 분석을 사용하여 특정 RNA 서열의 단백질 상호작용자를 발견, 정량화 및 검증하기 위한 최적화된 시험관 내 방법을 제시합니다.
초록
단백질-RNA 상호작용은 전사 및 전사 후 수준에서 유전자 발현 및 세포 기능을 조절합니다. 이러한 이유로 관심 RNA의 결합 파트너를 식별하는 것은 많은 세포 과정의 메커니즘을 밝히는 데 매우 중요합니다. 그러나 RNA 분자는 일부 RNA 결합 단백질(RBP), 특히 비표준 단백질과 일시적이고 동적으로 상호 작용할 수 있습니다. 따라서, 이러한 RBP를 분리하고 식별하기 위한 개선된 방법이 절실히 필요하다.
알려진 RNA 서열의 단백질 파트너를 효율적이고 정량적으로 식별하기 위해 세포 총 단백질 추출물에서 시작하여 모든 상호 작용하는 단백질의 풀다운 및 특성 분석을 기반으로 하는 방법을 개발했습니다. 우리는 스트렙타비딘 코팅 비드에 미리 로드된 비오티닐화 RNA를 사용하여 단백질 풀다운을 최적화했습니다. 개념 증명으로, 우리는 신경 퇴행 관련 단백질 TDP-43과 다른 뉴클레오티드 조성의 음성 대조군에 결합하는 것으로 알려진 짧은 RNA 서열을 사용했지만 길이는 동일했습니다. 효모 tRNA로 비드를 차단한 후, 스트렙타비딘 비드에 비오티닐화된 RNA 서열을 로딩하고 HEK 293T 세포에서 추출한 총 단백질 추출물과 함께 배양했습니다. 비특이적 결합제를 제거하기 위한 배양 및 여러 세척 단계 후, 가장 일반적으로 사용되는 단백질 정량 시약 및 질량 분석을 위한 시료 전처리와 호환되는 고염 용액으로 상호 작용하는 단백질을 용리했습니다. 우리는 질량 분석법에 의해 음성 대조군과 비교하여 알려진 RNA 결합제로 수행된 풀다운에서 TDP-43의 농축을 정량화했습니다. 우리는 동일한 기술을 사용하여 관심 RNA 또는 대조군의 고유한 결합체로 계산적으로 예측된 다른 단백질의 선택적 상호 작용을 확인했습니다. 마지막으로, 우리는 적절한 항체를 가진 TDP-43의 검출을 통해 웨스턴 블롯으로 프로토콜을 검증했습니다.
이 프로토콜을 사용하면 생리학적 조건에 가까운 조건에서 관심 RNA의 단백질 파트너를 연구할 수 있어 독특하고 예측할 수 없는 단백질-RNA 상호작용을 발견하는 데 도움이 됩니다.
서문
RNA 결합 단백질(RBP)은 mRNA 스플라이싱, RNA 세포 국소화, 번역, 변형 및 분해와 같은 과정에 관여하기 때문에 전사 및 전사 후 유전자 조절에서 중요한 역할을 하는 것으로 나타났습니다 1,2,3. 두 거대분자 사이의 이러한 상호 작용은 고도로 조정되고 정밀하게 균형을 이루며 기능 및 처리 허브의 형성에 필수적입니다. 이러한 허브 내의 변이 또는 조절 장애는 미세하게 조절된 단백질-RNA 네트워크를 방해할 가능성이 있으며 암4,5 및 신경퇴행성 장애 6,7,8을 포함한 다양한 인간 질병과 점점 더 연관되고 있습니다. RNA 분자와 단백질 결합 파트너 간의 상호 작용은 안정적이고 실험적으로 검증하기 쉬울 수도 있고, 매우 역동적이고 일시적이며 특성화하기가 더 어려울 수도 있습니다.
최근 몇 년 동안 이러한 상호 작용을 이해하기 위해 집중적인 노력이 기울여졌습니다. 가장 확립된 방법 중에서, 단백질 풀다운 분석(PD)은 아마도 리보핵단백질(RNP) 복합체 및 기타 단백질-RNA 상호작용 네트워크를 구성하는 주요 역할을 밝히기 위해 가장 높이 평가되고 일반적으로 사용되는 접근법일 것입니다 3,9,10. PD에는 관심 있는 RNA(RIP)11,12 또는 단백질(CLIP)13,14의 면역침전과 같은 광범위한 정보 기술이 포함됩니다. 이들 RNA-PD 프로토콜 중 일부는 단백질15에 대한 미끼로서 알려진 RNA를 사용하며, 가장 빈번하게는 비오틴과 같은 고친화성 태그를 이용한다. 이 경우, 비오티닐화된 RNA의 상호작용 파트너는 스트렙타비딘 코팅 비드에 RNA를 고정하여 RNP의 효율적인 분리를 가능하게 함으로써 검출될 수 있습니다. 이러한 접근법의 주요 한계는 일반적으로 비오티닐화 프로브의 설계와 표적 단백질에 결합하는 능력의 테스트입니다. 이러한 목적을 위해, 표적 단백질13,16과의 상호 작용 피크에 해당하는 짧은 RNA 영역을 높은 정밀도로 나타내기 때문에 관심 단백질의 공개된 CLIP 데이터에 의존하는 것이 유용할 수 있습니다. 이 동일한 영역은 PD용 프로브를 개발하는 데 사용될 수 있습니다. 이러한 RNA 미끼를 설계하는 또 다른 방법은 지수 농축(SELEX)17에 의한 리간드의 체계적인 진화일 수 있으며, 이는 포괄적인 무작위 라이브러리에서 시작하여 일련의 PCR 기반 최적화 주기를 통해 시험관 내 선택을 통해 앱타머를 설계할 수 있습니다. 그러나 SELEX는 복잡하고 시간이 많이 걸리며 최종 결과는 초기 라이브러리에 크게 의존합니다. 여기에 제시된 프로토콜에서 사용할 RNA 미끼를 선택하기 위해, 특정 RNA 서열18,19,20에 대한 주어진 단백질의 우선적 결합을 예측하는 catRAPID 알고리즘의 계산 능력에 의해 새로 설계된 RNA 미끼를 사용하는 것으로 구성된 또 다른 접근법이 이용되었습니다.
여기에 소개된 프로토콜은 세제, 변성제 또는 고온을 사용하지 않고 생리학적 조건에 가까운 조건에서 특정 단백질 파트너를 용리하도록 최적화된 RNA-PD 버전입니다. 그것은 고도로 정제된 스트렙타비딘으로 공유 코팅된 나노 초상자성 비드와 특정 인 실리코 로 설계된 비오티닐화 RNA를 미끼로 사용합니다. 이 프로토콜은 기본 조건에서 비오티닐화 RNA 분자의 결합 파트너를 분리하는 빠르고 효율적인 방법을 제공하여 광범위한 다운스트림 응용 분야에 대한 잠재력을 제공합니다. 이 프로토콜을 테스트하기 위해 이전에 높은 친화력과 특이성으로 단백질 TAR DNA 결합 단백질 43(TDP-43)에 결합하도록 설계된 10-뉴클레오티드 단일 가닥 RNA 앱타머 서열을 사용했습니다20. HEK 293T 세포 용해물에서 시작하여 비오티닐화 RNA 앱타머의 상호작용자는 고장성 완충액을 사용하여 RNA 미끼에서 분리된 샘플에 대해 수행된 질량 분석 분석을 통해 확인되었습니다. 이 분석은 TDP-43의 바람직한 결합제로서의 성공적인 식별 및 정량화를 확인시켜 주었다.
이 프로토콜은 짧은 시험관 내 합성 RNA 올리고뉴클레오티드만을 사용하여 단백질 상호작용자의 성공적인 식별을 가능하게 합니다. 또한, 인실리코(in silico) 설계된 RNA 앱타머를 PD 프로브(21,22)로 사용하면 상당히 감소된 비용으로 표적에 대한 특이성을 보장할 수 있습니다.
프로토콜
1. 일반적인 방법 및 재료
- 선택한 포유류 세포 배양을 위한 적절한 배지를 준비하고 사용하기 전에 37°C에서 20분 동안 예열합니다.
- 재료 표에 설명 된대로 필요한 재료를 미리 준비하십시오. 오토클레이브 유리 제품, 플라스틱 제품 및 완충 스톡.
- 표 1에 기재된 바와 같이 버퍼를 준비한다. 성분을 최종 부피로 희석하기 전에 농축된 HCl 또는 NaOH를 사용하여 원액의 pH를 조정합니다.
2. 포유류 세포주 준비
- 10% 소 태아 혈청(FBS)과 100μg/mL 페니실린/스트렙토마이신 용액이 보충된 Dulbecco's Modified Eagle Medium(DMEM)에서 HEK 293T 세포를 성장시킵니다. 5%CO2가 공급되는 가습 배양기에서 37°C에서 배양한다. 정기적으로 세포를 분할하십시오.
- 분리하기 전에 성장하는 표면을 덮을 수 있을 만큼 충분한 인산염 완충 식염수(PBS) 용액으로 세포를 헹굽니다.
- PBS를 제거하고 트립신-EDTA 용액의 초박형 층을 추가합니다.
- 세포를 37°C에서 5분 동안 또는 세포가 분리될 때까지 5%CO2 가 제공된 가습 인큐베이터에서 배양합니다(현미경 관찰 하에서 분산된 것처럼 보여야 함).
- 트립신-EDTA 용액을 완전한 DMEM을 추가하여 10배로 희석하여 비활성화하고 세포를 계산합니다.
- 6웰 플레이트에 1.5 x 105 cells/mL를 플레이트하고 테스트할 2개의 웰/조건을 고려합니다.
- 세포를 5%CO2를 갖는 가습된 인큐베이터에서 48시간 동안 37°C에서 인큐베이션한다.
참고: 배지 유형에 대한 제조업체 지침을 확인하고 세포주에 적합한 보충제를 확인하십시오. 또한, 트립신-EDTA의 양과 배양 시간은 세포주에 따라 다릅니다. 일부 세포 유형은 이 프로토콜에서 보고된 것보다 더 빠르거나 느리게 성장합니다. 따라서 파종 농도를 미리 테스트해야합니다.
3. 총 단백질 수확량
- 세포가 자라는 우물에서 배지를 제거하십시오.
- 6-웰 플레이트의 각 웰을 1 mL의 PBS로 세척한다.
- PBS를 폐기합니다.
- 플레이트를 얼음 위로 옮기고 3.5단계를 진행하거나 용해를 용이하게 하기 위해 건조 플레이트를 -80°C에서 동결합니다.
- 각 웰에 200μL의 용해 완충액을 추가합니다.
- 셀 스크레이퍼를 사용하여 셀을 분리하고 부수십시오.
- 두 개의 웰에서 파생된 세포 추출물을 동일한 1.5mL 튜브로 옮깁니다.
- 단백질 추출물이 들어 있는 튜브를 얼음 위에 30분 동안 놓습니다.
- 세포 용해물을 4°C에서 15분 동안 17,000 x g 로 원심분리합니다.
- 각 상층액을 미리 냉각된 튜브로 옮깁니다.
알림: 각 PD 조건에 대해 총 10개의6-10 7 개의 셀이 권장됩니다. 세포 용해 및 단백질 수확은 얼음처럼 차가운 완충액으로 수행해야 합니다. 단백질 분해를 방지하기 위해 용해 완충액에 프로테아제 억제제를 첨가해야 합니다.
4. 단백질 농도 결정
- Bradford 시약을 생산자가 표시한대로 dH2O에서 5 배 희석하여 준비합니다.
- 1cm 큐벳에 1mL의 시약을 분배하고 1μL의 샘플을 추가하고 반전으로 혼합합니다.
- 실온에서 5-10 분 동안 어둠 속에서 배양하십시오.
- 595nm에서 흡광도를 읽습니다.
- 단백질 1.5mg에 해당하는 단백질 추출물의 부피를 계산하고 용해 완충액을 사용하여 모든 샘플을 최종 부피 600μL로 가져옵니다.
- 사용할 때까지 샘플을 얼음 위에 보관하십시오.
참고: 완충액 호환성 권장 사항에 따라 다른 단백질 농도 측정 방법을 사용할 수 있습니다. 어떤 경우든 용해 버퍼는 공백으로 사용해야 합니다. 많은 시약은 디티오트레이톨(DTT)과 호환되지 않습니다. 단백질 정량 (DTT를 최종 농도 1mM까지) 후에 만 DTT 또는 기타 환원제를 첨가하는 것이 좋습니다.
5. 비드 준비
- 튜브를 튕겨 저장 버퍼에 있는 비드를 혼합합니다.
- 100μL의 슬러리 매체/시료를 계산하고 부피를 마그네틱 랙에 넣습니다.
- 비드 워시
- 저장 용액을 제거하고 용해 완충액/튜브 1mL를 추가하여 비드를 세척하고 수동으로 뒤집습니다.
- 마그네틱 랙을 사용하여 버퍼를 제거합니다.
- 세척 단계를 반복하십시오.
- 슬러리 배지의 초기 부피와 동일한 부피의 용해 완충액을 추가하고, 튜브를 튕겨서 혼합하고, 샘플이 있는 만큼의 1.5mL 튜브에 배지를 균일하게 분주합니다.
- 비드 블로킹
- 마그네틱 랙을 사용하여 완충액을 제거하고 용해 완충액에 준비된 효모 tRNA의 0.25mg/mL 용액 600μL를 추가합니다.
- 회전하는 바퀴에서 실온에서 1 시간 동안 배양하십시오.
- 마그네틱 랙을 사용하여 tRNA 용액을 제거합니다.
- 600 μL의 용해 완충액을 첨가하고 수동으로 혼합하여 세척합니다.
- 세척 단계를 반복하고 버퍼를 버립니다.
6. 비드 로딩
- 초기 100 μL 슬러리 배지(현재 차단된 비드)를 포함하는 각 튜브에 대해 600 μL의 용해 완충액에 200 μg의 RNA 올리고뉴클레오티드를 준비합니다.
- 올리고를 비드에 넣고 회전하면서 실온에서 1시간 동안 배양합니다.
- 용액을 제거하고, 600 μL의 용해 완충액을 첨가하고, 실온에서 5분 동안 튜브를 회전시켜 비드를 두 번 세척한다.
- 버퍼를 삭제합니다.
알림: 구슬을 휘두르지 말고 대신 튕기십시오. 필요한 경우가 아니면 피펫팅 단계의 수를 제한하십시오. 가능하면 절단 1mL 팁을 사용하십시오. 비드 슬러리 매질/샘플의 양은 비드의 결합 능력과 총 단백질의 시작 양에 따라 달라집니다. RNA 올리고가 상당한 양의 2차 구조를 가질 것으로 예상되는 경우 먼저 80°C에서 10분 동안 변성시킨 다음 실온에서 천천히 냉각하거나 30°C에서 1시간 동안 배양하여 리폴딩하는 것이 좋습니다. 로딩에 필요한 양을 최적화하고 RNA 재사용 가능성을 평가하기 위해 비드 로딩 후 RNA 올리고를 회수하고 남은 농도를 결정하는 것이 좋습니다.
7. 구슬에 단백질 결합
알림: 지금부터 가능하면 4°C에서 단계를 수행하십시오.
- 600 μL 단백질 용액에서 5 % 부피를 취하여 추가 분석을 위해 INPUT (IN)으로 유지합니다 (1.5 mg의 단백질은 600 μL에 용해되므로 5 %는 30 μL 및 75 μg의 단백질에 해당).
- 나머지 단백질 혼합물을 로드된 비드의 각 튜브에 추가하고 4°C에서 밤새 천천히 회전시킵니다.
8. 비특이적 바인더의 세척
- 마그네틱 랙을 사용하여 결합되지 않은 부분을 제거합니다. 부피의 5%를 절약하고 FLOWTHROUGH(FT)로 레이블을 지정합니다(결합되지 않은 부피는 약 600μL이므로 추가 분석을 위해 다시 30μL를 유지).
- 세척 완충액 1 1 mL를 비드에 넣고 4°C에서 5분 동안 회전시킵니다.
- 버퍼를 삭제합니다.
- 8.2단계와 8.3단계를 반복합니다.
- 세척 완충액 2 1 mL를 비드에 첨가하고 4°C에서 5분 동안 회전시킨다.
- 상청액을 버리십시오.
9. 특정 바인더의 용출
- 100μL의 용출 완충액 1 또는 용출 완충액 2를 비드에 추가합니다.
- 가볍게 치면서 수동으로 혼합하고 실온에서 5분 동안 배양합니다.
- 튜브를 열혼합기에 넣고 95°C에서 5분 동안 세게 흔듭니다.
- 튜브를 마그네틱 랙에 넣고 용출된 분획을 깨끗한 튜브에 수집합니다.
- 벤치 원심분리기로 비드를 빠르게 회전시켜 용리액의 회수율을 극대화합니다.
- 추가 분석을 위해 총 ELUATE(EL) 부피의 5%를 절약합니다(총 부피는 100μL이므로 5μL를 다른 튜브에 분리).
- 필요한 경우, 단백질 농도는 블랭크로서 용출 완충액을 사용하여 섹션 4에서와 같이 결정될 수 있다.
참고: 단백질 농도가 결정될 때까지 용출 완충액에 DTT를 추가하지 않는 것이 좋습니다. 단백질 정량이 필요하지 않거나 환원제 호환 단백질 정량 키트를 사용할 수 있는 경우 처음부터 1mM DTT를 용리 버퍼에 추가할 수 있습니다. 이 프로토콜에서는 용출 완충액 1(1M NaCl 함유)과 용출 완충액 2(2M NaCl 포함)를 모두 테스트했습니다. 증가된 이온 강도에 따라 표적 단백질의 용출 효율에는 차이가 관찰되지 않았지만 가장 적절한 완충액을 설정하기 전에 두 조건을 모두 테스트하는 것이 좋습니다. 용출 완충액에 염분이 많이 존재하여 추가 분석에 한계가 있는 경우, 용출 완충액에 있는 세제의 양이 매우 적기 때문에 완충액 교환이 가능합니다. 대안으로, 용출액은 원하는 염 농도에 도달하도록 희석될 수 있다.
10. 질량분석법에 의한 단백질 결합제 동정
- 아세톤 침전
- 용출된 단백질을 저온(-20°C) 아세톤에 4배 희석하여 농축합니다.
- 튜브를 -20°C에서 밤새 소용돌이 배양합니다.
- 4°C에서 17,000 x g 로 30분 동안 회전합니다.
- 상층액을 부드럽게 제거하고 펠릿이 완전히 마를 때까지 아세톤이 증발하도록 합니다.
- 용액 내 단백질 분해
- 변성 완충액 50μL를 첨가하여 단백질 펠릿을 용해시킨다.
- DTT를 최종 농도 5mM에 첨가하여 55°C에서 30분 동안 단백질을 환원시킵니다.
- 샘플을 실온에서 냉각하고 단백질 알킬화 반응을 진행하여 요오드아세트아미드(IAA)를 10mM 농도로 15분 동안 첨가합니다.
- 적절한 효소(트립신, LysC)를 사용하여 단백질을 분해하고 샘플을 37°C에서 밤새 배양합니다.
- 10% 트리플루오로아세트산(TFA) 1μL를 추가하여 소화를 중지합니다.
- 펩티드를 세척하고 이전에 기술된 바와 같이 맞춤형 역상 C18 마이크로-컬럼 상에서 농축한다19.
- 버퍼 B를 사용하여 C18 팁에서 펩타이드를 용리합니다.
- 진공 원심분리기를 사용하여 유기 성분을 제거하고 추가 분석을 위해 0.1% 포름산 5μL에 펩타이드를 재현탁합니다.
참고: 대안적으로, 단백질 분해는 비특이적 결합제를 세척한 직후 "비드에서" 수행될 수 있으므로(단계 8.1-8.6), 따라서 프로토콜이 더 빨라집니다. 그러나 최적의 실험 단백질 서열 범위를 보장하기 위해 용액 분해에서 표준과 비교하여 "비드" 고정 단백질을 작동하는 효소의 효율성을 테스트하는 것이 좋습니다.
- 액체 크로마토그래피-탠덤 질량분석법(LC-MS/MS)
- 분석 컬럼(C18-고정상)을 연결하고 실행 시간 동안 45°C로 유지합니다.
- 단일 컬럼 구성에서 캐필러리 핑거 타이트 피팅(20μm x 550mm)을 통해 컬럼을 LC 펌프의 6포트 로터리 밸브 출구에 연결합니다.
- 다음과 같이 LC 설정을 조정합니다.
- 조절된 압력(980bar) 하에서 펩타이드를 버퍼 A에 로드합니다.
- 59분 동안 300nL/min에서 5%-20% 버퍼 B 그래디언트를 적용한 다음, 15분 동안 20%-30% 버퍼 B 그래디언트를, 5분 동안 30%-65% 버퍼 B 그래디언트를 적용합니다.
- 5분에 걸쳐 버퍼 B의 농도를 최대 95%까지 증가시키고 95% 버퍼 B에서 5분 등용매 단계를 추가하여 세척 단계를 추가합니다.
- DDA(Data-Dependent Acquisition) 모드에서 질량분석기를 작동하여 MS와 MSMS 이벤트 사이를 자동으로 전환합니다.
- MS 및 MSMS이벤트에 대해 각각 3 x 106 및 1 x 10 5의 자동 이득 제어(AGC) 목표 값을 사용하여 루프 수를 15로 정의합니다.
- 최대 허용 이온 축적 시간을 분해능이 60K인 MS의 경우 20ms로, 분해능이 15K인 MSMS의 경우 100ms로 설정합니다.
- 20초의 동적 배제 시간으로 28%의 정규화된 충돌 에너지를 사용하여 높은 충돌 해리(HCD) 단편화 실험을 수행합니다.
- 소스 파라미터를 다음과 같이 작동하십시오.
스프레이 전압: 1.7 kV
모세관 전압: 275 °C
외피나 보조 가스가 사용되지 않음
참고: 이 프로토콜에서 초고성능 액체 크로마토그래피(UHPLC) 질량 분석(MS) 분석은 하이브리드 삼중 사중극자 orbitrap 기기(재료 표)에 결합된 LC 단일 컬럼 설정을 사용하여 특별히 수행되었습니다. 다른 LCMS 시스템을 사용할 수 있지만 파라미터를 조정하는 것이 좋습니다.
- 데이터 분석
- 로드 버튼을 사용하여 원시 파일을 가져옵니다.
- 실험 설정 버튼을 클릭하여 실험 이름을 정의합니다.
- group-specific parameters(그룹별 매개변수) 섹션을 입력하여 식별과 관련된 모든 매개변수를 지정합니다.
소화에 사용되는 효소: 트립신/P
놓친 분열 : 최대 3 개
고정 수정: 카르바미도메틸화
가변 변형: N-아세틸(단백질), 산화(M) - UniprotKB와 같은 공용 데이터베이스에서 사용할 수 있는 업데이트된 FASTA 파일을 업로드합니다.
- 선택한 데이터베이스의 원본에 따라 올바른 구문 분석 규칙을 지정합니다.
- 단백질과 펩타이드 모두에 대해 혈통 거짓 발견률(FDR) 값 = 1을 정의합니다.
- 라벨 프리 정량화(Label Free Quantification) 탭에 라벨 프리 정량화(LFQ) 옵션을 추가합니다.
- 최소 LFQ 비율 수를 2로 유지합니다.
참고: 여기서는 MaxQuant24 및 Perseus 소프트웨어25 를 사용하여 각각 단백질 정량화 및 후속 통계 분석을 수행하는 데이터 분석에 대해 설명합니다. 그러나, 데이터 분석은 임의의 다른 상업적으로 이용가능한 또는 자유로운 생물정보학으로 수행될 수 있다. FDR은 타겟-디코이 데이터베이스-기반 접근법(26)을 사용하여 추정된다. 0.01과 같은 펩타이드 및 단백질 FDR은 확인된 펩타이드 및 단백질이 1%의 위양성을 함유할 것으로 예상됨을 의미한다.
- 통계 분석
- proteingroups.txt 파일을 불러와 단백질 수준에서 통계 분석을 수행합니다.
- LFQ 값을 기본 열로 정의합니다.
- categorical형 열을 기준으로 행을 필터링하여 "reverse" 및 "contaminants"를 제거합니다.
- 범주형 주석 행을 사용하여 다양한 실험 조건을 그룹화할 수 있습니다.
- 데이터 행렬을 줄여 이전에 정의된 각 그룹에서 유효한 값의 수를 선택합니다.
- 실험 조건에 더 적합한 통계 검정(예: t-검정, 다중 표본 검정 ANOVA)을 선택합니다.
- FDR 기반 계산을 사용하여 유의미한 힌트에 대한 구분값을 결정합니다. 일반적으로 0.01과 0.05는 모두 수정된 p 값에 대한 임계값으로 허용됩니다.
- 화산 플롯 표현을 사용하여 t-검정 통계량을 기반으로 차등 분석 결과를 시각화합니다.
- 최종 결과 테이블을 추가로 편집할 수 있도록 최종 행렬을 .txt 형식으로 내보냅니다.
참고: 구성 폴더에는 글로벌 단백질체 실험에서 일반적인 오염 물질로 간주되는 케라틴과 같은 단백질이 포함된 FASTA 파일이 포함되어 있으며 출력 표에 +로 플래그가 지정되어 있습니다. 본 연구에서는 두 가지 샘플 조건을 가지고 통계 분석을 위해 t-검정을 사용합니다.
11. 웨스턴 블롯에 의한 결과 검증
- 시료 전처리
- 적절한 부피의 4x 샘플 로딩 버퍼를 IN, FT 및 EL의 각 부분 표본에 추가합니다.
- 샘플을 95°C에서 5분 동안 끓입니다.
- 튜브 상단에서 증발된 샘플을 회수하기 위한 빠른 회전.
- SDS-PAGE 및 젤 전사
- 4%-12% 변성 폴리아크릴아미드 겔에 샘플을 로드합니다.
- 120V에서 1.5시간 동안 MES SDS 실행 버퍼로 젤을 실행합니다.
- 니트로 셀룰로오스 멤브레인에 젤을 반건식 이송 카세트로 옮기고, 제조사 지침에 따라. 15V에서 10분 전송하는 것이 좋습니다.
- 면역 검출
- 부드럽게 교반하면서 실온에서 10% 소 혈청 알부민(BSA)으로 막을 차단합니다.
- 제조업체의 지침에 따라 TBST에 5% BSA로 준비된 1차 항체를 추가합니다. 4 °C에서 밤새 또는 실온에서 1시간 동안 부드럽게 교반합니다.
- 멤브레인을 TBST로 세 번, 매번 5분 동안 세척합니다.
- TBST에서 제조된 2차 항체를 상온에서 1시간 동안 교반 하에 첨가한다.
- 멤브레인을 TBST로 세 번, 매번 5분 동안 세척합니다.
- 블롯 이미저를 사용하여 결과를 시각화합니다.
참고: 당사 RNA의 알려진 결합제인 TDP-43을 검출하기 위해 재조합 토끼 단클론 항체를 사용하고 4°C에서 밤새 멤브레인과 함께 두었습니다. 2차 항체로는 항토끼 IgG 양고추냉이 과산화효소(HRP)가 사용되었지만 형광 2차 항체도 효과가 있었습니다. 멤브레인에 있는 항체를 시각화하기 위해 멤브레인을 Clarity Western ECL 기질과 함께 1분 동안 배양한 후 ChemiDoc 이미징 시스템으로 이미징했습니다.
결과
제안된 프로토콜의 타당성을 검증하기 위해, 여기에 제시된 PD 실험은 TDP-4320에 특이적으로 결합하도록 설계된 비오티닐화 RNA 앱타머로 수행되었다. 이 RNA는 높은 결합 친화력(Kd = 90 nM)으로 단백질 표적에 결합합니다.20. 여기서, 서열 5'-CGGUGUUGCU-3'의 이 RNA는 "+RNA"라는 이름으로 지칭된다. 음성 대조군으로는 +RNA의 역상보적 서열(여기서는 "-RNA"라고 함)을 사용했습니다. 그 서열은 5'-AGCAACACCG-3'입니다. -RNA는 TDP-43(Kd = 1.5μM)에 대해 상당히 낮은 결합 친화도를 나타냅니다(19). 본원에 기재된 프로토콜의 목적을 위해, 이들 RNA 올리고뉴클레오티드는 스트렙타비딘 비드에 결합할 수 있도록 비오틴 분자에 접합된 것을 구입하였다. +RNA는 비오틴과 핵산의 인산기 사이에 15원자 트리에틸렌 글리콜 스페이서를 포함하는 3' 말단에 비오틴-TEG와 함께 구입했습니다. -RNA는 대신 5' 말단에 비오틴을 가지고 있으며 아미노-C6 링커를 통해 핵산에 접합되었습니다. 그러나 RNA 미끼의 설계가 견고하고 링커와 RNA 사이에 구조적 또는 화학적 간섭이 없는 한 비오틴 접합 및 기타 링커 길이를 위한 다른 위치를 사용할 수 있습니다.
PD 후 +RNA 프로브에 결합된 주요 단백질의 정체를 알면 질량분석법(MS)과 웨스턴 블롯(WB)을 모두 사용하여 용리액에서 TDP-43을 식별하여 프로토콜을 검증할 수 있습니다(그림 1).
MS 분석은 +RNA 또는 -RNA를 사용하여 수행된 4개의 PD 복제에 대해 수행되었습니다(그림 2). +RNA와 -RNA의 상호작용체 식별은 이 프로토콜의 범위를 벗어나지만 프로토콜의 정확성을 검증하는 일부 결과가 보고됩니다. 주목할 점은, 화산 플롯에서 유의하게 농축된 단백질을 플로팅한 결과, +RNA에서 용출된 총 단백질 함량과 농축 단백질이 -RNA에서 회수된 것보다 훨씬 더 높다는 것을 알 수 있었습니다(그림 2). 이는 동일한 길이와 구조적 함량(선형)을 가짐에도 불구하고 +RNA가 더 많은 수의 특정 상호작용을 확립할 수 있음을 의미하며, 이는 높은 염분으로 용리 단계까지 유지됩니다. 대신 -RNA는 세척 단계 동안 중단되는 더 많은 수의 비특이적 접촉을 설정할 가능성이 있습니다. 예상대로 TDP-43은 +RNA20의 독특한 상호작용자로 확인되었습니다. +RNA로 수행된 4개의 PD 복제에 대한 평균 무표지 정량화(LFQ)는 31.96 ± 0.56인 반면, 단백질은 -RNA의 상호작용자들 사이에서 확인되지 않습니다. 또한, +RNA의 모든 독특한 상호작용체 중에서 TDP-43이 가장 풍부하게 농축된 단백질인 것으로 밝혀졌습니다.
프로토콜을 추가로 검증하기 위해 사내 알고리즘 catRAPID18,19를 사용하여 +RNA 또는 -RNA에 특이적으로 결합하는 다른 단백질을 계산적으로 예측했습니다. 특히, +RNA 및 -RNA와 인간 프로테옴을 구성하는 단백질의 상호작용 점수는 이전 연구27에서 정의된 바와 같이 catRAPID '상호작용 성향' 기능을 사용하여 계산되었습니다. 높은 신뢰도로 점수가 매겨진 단백질 중 HNRNPH3는 +RNA(+RNA 상호작용 점수 = 1.01; -RNA 상호작용 점수 = 0.21) 및 PCBP2가 -RNA와 특이적으로 상호작용하는 것으로 예측되었다(+RNA 상호작용 점수 = -0.5; -RNA 상호작용 점수 = 0.31)(도 3A). 또한, 단백질 RBM41은 RNA 올리고뉴클레오티드(+RNA 상호작용 점수 = 0.4; -RNA 상호작용 점수 = 0.39) 모두에 대해 난잡한 것으로 예측되었다(도 3A). MS 분석은 실제로 각각 +RNA 및 -RNA의 PD에서 HNRNPH3 및 PCBP2의 존재를 확인한 반면, RBM41은 둘 다와 상호작용하는 것으로 나타났습니다(그림 3B).
WB는 TDP-43의 존재를 감지하여 결과를 추가로 확인하고 프로토콜 최적화하는 동안 사용되었습니다(그림 4). 여기에 설명된 절차에서 서로 다른 샘플을 서로 다른 단계에서 수집했습니다. 투입 샘플(IN)은 용해 완충액에 희석된 총 단백질로 구성되었다. 플로우 스루(FT)는 RNA에 결합하지 않은 단백질의 분획을 나타내는 비오티닐화된 RNA로 미리 코팅된 스트렙타비딘 비드를 사용하여 전체 단백질을 하룻밤 동안 배양한 후 얻었습니다. 마지막으로, 용출액(EL)은 FT와 EL 단계 사이에 150mM 염과 0.1% triton-X를 사용한 3개의 세척 단계가 가장 약한 상호작용을 제거했어야 했기 때문에 조사 중인 RNA를 특이적으로 인식하는 모든 단백질을 포함했습니다.
각 복제에 대해 동일한 양(5% v/v)의 IN, FT 및 EL을 SDS-PAGE에서 병렬로 실행하고 항-TDP-43 항체로 염색했습니다(그림 4). +RNA의 경우, TDP-43의 띠가 IN과 EL에서 관찰되었는데, 이는 전체 단백질 추출물에서 처음부터 존재하는 단백질이 세척 단계 동안 +RNA에 의해 유지되고 마지막에만 고염 완충액으로 용리됨을 나타냅니다. TDP-43은 또한 -RNA에 대해 IN에 존재했지만, 단백질에 해당하는 밴드는 FT에서도 볼 수 있으며, 이는 이 RNA가 TDP-43에 결합하지 않음을 나타냅니다. EL에 TDP-43 대역이 없으면 이 결과가 확인됩니다.
프로토콜을 최적화하는 동안 RNA 서열에 특이적으로 결합된 단백질의 용출은 1M NaCl(EB1)을 포함하는 용출 완충액과 2M NaCl(EB2)을 포함하는 완전한 용출 완충액을 사용하여 모두 조사되었습니다(그림 4). EB로 얻은 용출액을 SDS-PAGE에서 비교하고 항-TDP-43 항체로 블롯팅했습니다. 이어서, 얻어진 이미지를 ImageJ28 로 분석하여 2개의 완충액으로 용리된 TDP-43 양의 차이를 정량화하였다. 전반적으로, 유의미한 차이는 관찰되지 않았으며, 우리는 이러한 분석 내에서 1M 염이 가장 강력한 단백질-RNA 상호작용을 방해하기에 충분하다는 결론을 내렸습니다.
전반적으로, MS 및 WB에 대해 여기에 보고된 결과는 이 프로토콜이 특정 방식으로 주어진 RNA의 단백질 상호작용자를 캡처하는 데 효율적이며 다운스트림 분석과 호환되는 완충액에서 용리를 가능하게 한다는 것을 보여줍니다.
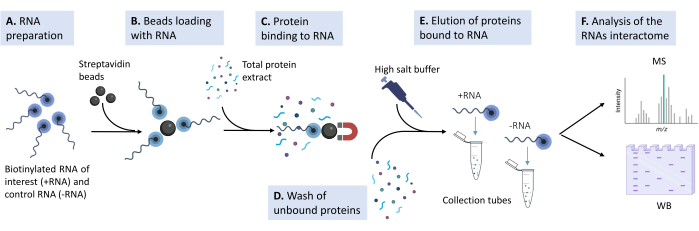
그림 1: 제안된 프로토콜에 사용된 실험 파이프라인의 스케치 . (A) 비오티닐화 RNA 올리고뉴클레오티드는 적절한 농도의 용해 완충액에서 제조된다. (B) 마그네틱 스트렙타비딘 비드를 세척하고, 효모 tRNA로 차단하고, 비오티닐화된 RNA를 로딩합니다. (c) 배양된 포유동물 세포주로부터 유래된 총 단백질 추출물을 비드-RNA 혼합물에 첨가한다. (D) 비특이적 상호 작용을 제거하기 위해 여러 번 세척합니다. (E) 특정 단백질 상호작용자는 고장성 용액으로 RNA에서 분리됩니다. (F) 상호 작용자의 신원은 질량 분석법에 의해 밝혀지고, 특정 사례는 웨스턴 블롯에 의해 검증됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
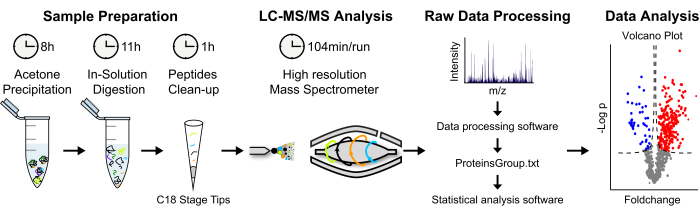
그림 2: 무표지 MS 기반 단백질 정량화를 위한 분석 전략 . (A) 용출된 단백질은 하룻밤 동안 차가운 아세톤에 침전됩니다. 그런 다음 단백질이 변성되고 용액 내 분해가 수행됩니다. 단백질 분해 펩타이드는 농축되고 탈염됩니다. (B) 펩타이드는 "샷건 접근법"을 사용하여 LC-MS/MS를 통해 분석됩니다. (C) 원시 데이터 처리 및 분석은 각각 MaxQuant 및 Perseus 소프트웨어를 사용하여 수행됩니다. (D) 통계적으로 유의미한 농축 단백질은 화산 플롯에 표시됩니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
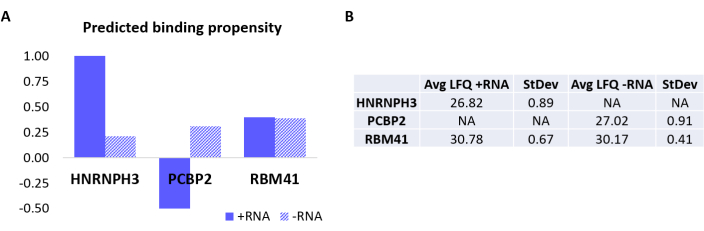
그림 3: 예측된 상호작용 성향과 실험적으로 결정된 +RNA 및 -RNA의 상호작용 간의 상관관계. (A) HNRNPH3, PCBP2 및 RBM41에 대한 고양이RAPID 상호작용 점수로, +RNA에 대한 HNRNPH3 및 -RNA에 대한 PCBP2의 우선적인 결합을 나타내는 반면, RBM41은 두 RNA 서열 모두에 무차별적으로 결합할 것으로 예측됩니다. (B) +RNA 및 -RNA로 수행된 풀다운에서 질량 분석 분석에 의해 결정된 무표지 정량 평균. 분석 결과 HNRNPH3는 +RNA에만 결합하고, PCBP2는 -RNA에만 결합하고, RBM41은 둘 다 동등하게 결합합니다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
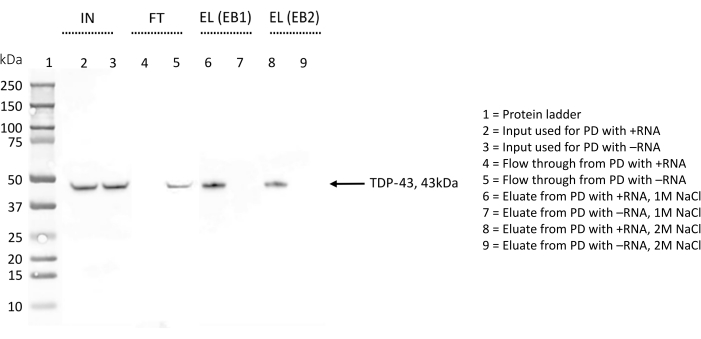
그림 4: 선택된 RNA 서열의 상호작용자들 사이에서 TDP-43의 존재/부재에 대한 웨스턴 블롯 검증. WB 멤브레인은 항-TDP-43 항체로 처리되었습니다. IN = 입력; FT = 플로우 스루; EL (EB1) = 용출 완충액 1을 사용한 용출; EL (EB2) = 용출 완충액 2를 사용한 용출; "+" 기호는 +RNA로 수행된 풀다운에서 파생된 샘플을 나타냅니다. 부호 "-"는 -RNA로 수행 된 풀다운에서 파생 된 샘플을 나타냅니다. 레인 1에는 단백질 사다리가 있습니다. TDP-43은 화살표로 표시됩니다. WB는 TDP-43이 +RNA 상호작용자들 사이에서는 발견되지만 -RNA 상호작용자들 사이에서는 발견되지 않는다는 것을 나타낸다. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
| 버퍼 이름 | 구성 | |||||
| 10x 전송 버퍼 | 250 mM 트리스, 1.92 M 글리신, 1% SDS, 20% 메탄올. 사용 전에 10배 희석하십시오 | 증권 시세 표시기 | ||||
| 20X MES SDS 러닝 버퍼 | 1M MES, 1M 트리스, 2% SDS, 20m EDTA. pH를 7.3으로 조정합니다. 사용 전에 20배 희석하십시오 | |||||
| 4x 시료 로딩 버퍼 | 0.25M 트리스 염기, 0.28M SDS, 40% 글리세롤, 20% 2-메르캅토-에탄올, 4mg/ml 브롬페놀 블루 | |||||
| 용출 버퍼 1 | 20 mM 인산염 pH 7.5, 1 M NaCl, 0.5 mM EDTA, 0.1 % Triton X-100, 1 mM DTT (정량 후 첨가) | |||||
| 용출 버퍼 2 | 20 mM 인산염 pH 7.5, 2 M NaCl, 0.5 mM EDTA, 0.1 % Triton X-100, 1 mM DTT (정량 후 첨가) | |||||
| 용해 버퍼 | 10 mM Tris-HCl pH 7.4, 150 mM NaCl, 0.5 mM EDTA, 0.1 % Triton X-100, 1 mM DTT 및 프로테아제 억제제 | |||||
| Tween-20이 포함된 트리스 완충 식염수 | 1 M Tris-HCl pH 7.4, 3 M NaCl, 2.0% 트윈-20 | |||||
| 세척 버퍼 1 | 10 mM Tris-HCl pH 7.4, 150 mM NaCl, 0.5 mM EDTA, 0.1 % TritonTM X-100, 1 mM DTT 및 프로테아제 억제제 | |||||
| 세척 버퍼 2 | 25 mM Hepes pH 8, 150 mM NaCl, 0.5 mM EDTA, 0.1 % Triton X-100, 1 mM DTT 및 프로테아제 억제제 | |||||
| 버퍼 A | 포름산 0.1% | 미시시피 | ||||
| 버퍼 B | 60% 아세토니트릴, 0.1% 포름산 | |||||
| 변성 버퍼 | 8M 요소, 50mM Tris-HCl | |||||
표 1: PD 및 MS 버퍼. 풀다운 실험(PD) 또는 질량 분석 분석(MS)에 사용되는 완충액의 이름 및 구성.
토론
이 연구는 단백질 상호작용자를 포획하기 위해 비오티닐화 RNA 올리고뉴클레오티드로 수행된 PD 프로토콜의 최적화를 보고합니다. 여기에 설명된 프로토콜은 수행이 간단하고 재료가 거의 필요하지 않으며 매우 신뢰할 수 있는 결과를 생성합니다. 중요하게도, 이 프로토콜의 가장 새로운 측면은 단백질 표적에 특이적으로 완전히 인실리코(in silico )로 설계된 RNA 미끼를 사용하고, 세제 및 고온 처리로 비오틴에서 스트렙타비딘을 분리하는 대신 고염 용액과의 상호 작용을 직접 방해하여 RNA 미끼에 결합된 모든 단백질을 용출하는 것입니다.
이 프로토콜은 비오틴과 스트렙타비딘 사이의 결합 강도를 이용합니다29,30. 선택된 스트렙타비딘 비드에 따르면, 비오티닐화 RNA의 로딩은 진행하기 전에 테스트되고 정량화되어야 합니다. 또한 RNA 3차원 접힘은 스트렙타비딘에 대한 비오틴의 노출을 제한할 수 있기 때문에 비드의 로딩 효율에 영향을 미칠 수 있습니다. 비오티닐화 tRNA로 비드를 차단하면 비드와의 비특이적 상호작용을 제한하여 결과의 청정도를 향상시킬 수 있습니다. 로딩 버퍼와 용출 버퍼는 다운스트림 응용 분야에 따라 선택해야 합니다. 여기서, 대부분의 용도에 적합하고 잠재적인 단백질 복합체를 보존하기 위해 개발된 매우 온화한 조건이 제안되었다. 그러나 이 방법은 적응력이 뛰어납니다. 사용자는 모든 세포주와 RNA 크기를 선택할 수 있으며 RNA를 접거나 펼친 후 프로토콜을 반복하여 결합 특성에 대한 구조의 영향을 결정할 수 있습니다.
이 프로토콜의 또 다른 본래의 측면은 결과의 정확성을 보장하기 위해 인실리코(in silico) 예측 도구를 사용하는 것이다(20). 관심 RNA의 상호 작용자로 식별되어야 하는 단백질을 미리 알면 프로토콜의 기술적 측면을 검증할 수 있는 전례 없는 이점을 얻을 수 있습니다. 예를 들어, 간단한 WB 분석을 사용하면 MS 분석을 진행하기 전에 프로토콜의 여러 단계에서 파생된 샘플에서 알려진 단백질 표적의 존재를 확인할 수 있으며, 이는 특수 기기가 필요하고 비용이 더 많이 듭니다. 또한, 표적 단백질에 특이적인 de novo RNA를 설계하기 위해 자체 단백질-RNA 예측 알고리즘인 catRAPID20을 이용하는 방법이 최근 보고되었다. 최근까지 표적 단백질에 대한 DNA/RNA 앱타머를 설계하는 데 사용할 수 있는 유일한 파이프라인은 SELEX(지수 농축에 의한 리간드의 체계적인 진화) 접근 방식31이었습니다. 인실리코(in silico) 방법을 사용하면 RNA 앱타머를 훨씬 빠르고 비용 효율적으로 설계할 수 있습니다.
이 방법의 주요 한계는 뉴클레아제가 없는 버퍼 및 도구에서 작업해야 하는 필요성과 관련이 있습니다. 또한, de novo-designed RNA와 PD 이전에 표적 단백질 사이의 결합을 시험관 내에서 확인하는 것이 필요하다고 생각되는 경우, 단백질을 생산 및 정제하고 생물물리학적 접근법으로 결합을 결정해야 합니다. 이것은 단클론 항체의 생산과 공유되는 한계입니다.
이러한 사소한 문제에도 불구하고 여기에 제시된 것과 같은 RNA-단백질 상호 작용을 매핑하는 신뢰할 수 있는 방법을 통해 과학자들은 암, 심근병증, 당뇨병, 미생물 감염, 유전 및 신경퇴행성 장애와 관련된 것과 같은 많은 생리학적 및 병리학적 메커니즘의 거대분자 네트워크와 복잡한 주요 행위자를 밝히는 데 더 가까이 다가갈 수 있습니다.
공개
저자는 경쟁적인 재정적 이해 관계나 기타 이해 상충이 없습니다.
감사의 말
저자는 Tartaglia 교수와 Cuomo 박사의 연구 그룹에 감사의 말을 전합니다. E.Z.는 Marie Sklodowska-Curie 보조금 계약 번호 754490에 따라 유럽 연합의 Horizon 2020 연구 및 혁신 프로그램의 MINDED 펠로우십으로부터 자금을 받았습니다.
자료
| Name | Company | Catalog Number | Comments |
| 6-well tissue culture plates | VWR | 10861-554 | CELLS |
| Cell scrapers | BIOSIGMA | 10153 | CELLS |
| Dulbecco′s Modified Eagle′s Medium (DMEM) | Thermo Fisher Scientfic | 11995065 | CELLS |
| Fetal Bovine Serum, qualified, heat inactivated, Brazil | Thermo Fisher Scientfic | 10500064 | CELLS |
| Phosphate Buffer Saline (PBS, Waltham, MA) | Thermo Fisher Scientfic | 14190169 | CELLS |
| Trypsin (0.25%), phenol red | Thermo Fisher Scientfic | 15050065 | CELLS |
| Anti-rabbit IgG horseradish peroxidase (HRP) | Cellsignal | 7070 | PD |
| Biotinylated RNA | Eurofins | Custom RNA oligonucleotides | PD |
| Bovine serum albumin | Sigma-Aldrich | A9418 | PD |
| Clarity Western ECL Substrate, 500 ml | Biorad | 1705061 | PD |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Merck - Sigma Aldrich | 5056489001 | PD |
| NuPAGE 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel, 10-well | Invitrogen | NP0321BOX | PD |
| Recombinant anti-TDP43 antibody | Abcam | ab109535 | PD |
| Ribonucleic acid, transfer from baker's yeast (S. cerevisiae) | Merck - Sigma Aldrich | R5636-1ML | PD |
| Streptavidin Mag Sepharose | Merck - Sigma Aldrich | GE28-9857-99 | PD |
| Trans-Blot Turbo RTA Mini 0.2 µm PVDF Transfer Kit | Biorad | 1704272 | PD |
| Acetone | Thermo Fisher Scientfic | 022928.K2 | MS |
| C18 cartridge | Thermo Fisher Scientfic | 13-110-018 | MS |
| Dithiothreitol (DTT) | Thermo Fisher Scientfic | 20290 | MS |
| EASY-Spray HPLC Columns | Thermo Scientific | ES902 | MS |
| iodoacetamide (IAA) | Sigma Aldrich S.r.l. | I6125 | MS |
| Lys-C/Trypsin | Promega | V5073 | MS |
| Trifluoroacetic acid (TFA) | Thermo Fisher Scientfic | 28904 | MS |
| Urea | Thermo Fisher Scientfic | J75826.A7 | MS |
| Equipment | |||
| ChemiDoc imaging system | Bio-Rad | CELLS | |
| Dyna Mag -2 , Magnetic rack | Invitrogen | CELLS | |
| Forma Series 3 water jacketed C02 incubator | Thermo Scientific | PD | |
| PROTEAN II xi cell , power supply for PAGE applications | Bio-Rad | PD | |
| Rotating wheel, rotator SB3 | Stuart | PD | |
| Water bath set at 37 °C | VWR | PD | |
| XCell SureLock Mini-Cell electrophoresis system | ThermoFisher Scientific | MS | |
| Easy-nLC 1200 UHPLC | Thermo Scientific | MS | |
| Q exactive Mass Spectrometer | Thermo Scientific | MS | |
| Software | Version | ||
| MaxQuant | 2.0.3.0 | MS | |
| Perseus | 1.6.14.0 | MS |
참고문헌
- Gebauer, F., Schwarzl, T., Valcárcel, J., Hentze, M. W. RNA-binding proteins in human genetic disease. Nature Reviews Genetics. 22 (3), 185-198 (2021).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Cooper, T. A., Wan, L., Dreyfuss, G. RNA and disease. Cell. 136 (4), 777-793 (2009).
- Qin, H., et al. RNA-binding proteins in tumor progression. Journal of Hematology & Oncology. 13 (1), 90 (2020).
- Nussbacher, J. K., Tabet, R., Yeo, G. W., Lagier-Tourenne, C. Disruption of RNA metabolism in neurological diseases and emerging therapeutic interventions. Neuron. 102 (2), 294-320 (2019).
- Duan, R., Sharma, S., Xia, Q., Garber, K., Jin, P. Towards understanding RNA-mediated neurological disorders. Journal of Genetics and Genomics. 41 (9), 473-484 (2014).
- Maziuk, B., Ballance, H. I., Wolozin, B. Dysregulation of RNA binding protein aggregation in neurodegenerative disorders. Frontiers in Molecular Neuroscience. 10, 89 (2017).
- Zielinski, J., et al. In vivo identification of ribonucleoprotein-RNA interactions. Proceedings of the National Academy of Sciences. 103 (5), 1557-1562 (2006).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Weidmann, C. A., Mustoe, A. M., Jariwala, P. B., Calabrese, J. M., Weeks, K. M. Analysis of RNA-protein networks with RNP-MaP defines functional hubs on RNA. Nature Biotechnology. 39 (3), 347-356 (2021).
- Graindorge, A., et al. In-cell identification and measurement of RNA-protein interactions. Nature Communications. 10 (1), 5317 (2019).
- Ule, J., Hwang, H. W., Darnell, R. B. The future of cross-linking and immunoprecipitation (CLIP). Cold Spring Harbor Perspectives in Biology. 10 (8), 032243 (2018).
- Ascano, M., Hafner, M., Cekan, P., Gerstberger, S., Tuschl, T. Identification of RNA-protein interaction networks using PAR-CLIP. Wiley Interdisciplinary Reviews. RNA. 3 (2), 159-177 (2012).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Sugimoto, Y., et al. Analysis of CLIP and iCLIP methods for nucleotide-resolution studies of protein-RNA interactions. Genome Biology. 13 (8), (2012).
- Bayat, P., et al. SELEX methods on the road to protein targeting with nucleic acid aptamers. Biochimie. 154, 132-155 (2018).
- Armaos, A., Colantoni, A., Proietti, G., Rupert, J., Tartaglia, G. G. CatRAPID omics v2.0: Going deeper and wider in the prediction of protein-RNA interactions. Nucleic Acids Research. 49, 72-79 (2021).
- Agostini, F., et al. CatRAPID omics: A web server for large-scale prediction of protein-RNA interactions. Bioinformatics. 29 (22), 2928-2930 (2013).
- Zacco, E., et al. Probing TDP-43 condensation using an in silico designed aptamer. Nature Communications. 13 (1), 3306 (2022).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Zhang, Y., Lai, B. S., Juhas, M. Recent advances in aptamer discovery and applications. Molecules. 24 (5), 941 (2019).
- UniProt Consortium. UniProt: the Universal Protein Knowledgebase in 2023. Nucleic Acids Research. 51, 523-531 (2023).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Bellucci, M., Agostini, F., Masin, M., Tartaglia, G. G. Predicting protein associations with long noncoding RNAs. Nature Methods. 8 (6), 444-445 (2011).
- Gallo-Oller, G., Ordoñez, R., Dotor, J. A new background subtraction method for Western blot densitometry band quantification through image analysis software. Journal of Immunological Methods. 457, 1-5 (2018).
- Weissinger, R., Heinold, L., Akram, S., Jansen, R. P., Hermesh, O. RNA proximity labeling: A new detection tool for RNA-protein interactions. Molecules. 26 (8), 2270 (2021).
- Hirsch, J. D., et al. Easily reversible desthiobiotin binding to streptavidin, avidin, and other biotin-binding proteins: Uses for protein labeling, detection, and isolation. Analytical Biochemistry. 308 (2), 343-357 (2002).
- Sefah, K., Shangguan, D., Xiong, X., O'Donoghue, M. B., Tan, W. Development of DNA aptamers using cell-SELEX. Nature Protocols. 5 (6), 1169-1185 (2010).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유