Method Article
Une analyse quantitative optimisée des protéines de liaison à l’ARN à l’aide d’ARN biotinylé court
Dans cet article
Résumé
Ici, nous présentons une méthode in vitro optimisée pour découvrir, quantifier et valider les interacteurs protéiques de séquences d’ARN spécifiques, en utilisant l’extrait protéique total de cellules humaines, des billes de streptavidine recouvertes d’ARN biotinylé et une analyse par spectrométrie de masse.
Résumé
Les interactions protéine-ARN régulent l’expression des gènes et les fonctions cellulaires aux niveaux transcriptionnel et post-transcriptionnel. Pour cette raison, l’identification des partenaires de liaison d’un ARN d’intérêt reste d’une grande importance pour dévoiler les mécanismes derrière de nombreux processus cellulaires. Cependant, les molécules d’ARN peuvent interagir de manière transitoire et dynamique avec certaines protéines de liaison à l’ARN (RBP), en particulier avec les protéines non canoniques. Il est donc absolument nécessaire d’améliorer les méthodes permettant d’isoler et d’identifier ces pratiques commerciales restrictives.
Pour identifier efficacement et quantitativement les partenaires protéiques d’une séquence d’ARN connue, nous avons développé une méthode basée sur l’extraction et la caractérisation de toutes les protéines en interaction, à partir de l’extrait de protéine totale cellulaire. Nous avons optimisé l’extraction des protéines en utilisant de l’ARN biotinylé préchargé sur des billes enrobées de streptavidine. Comme preuve de concept, nous avons utilisé une courte séquence d’ARN connue pour lier la protéine TDP-43 associée à la neurodégénérescence et un contrôle négatif d’une composition nucléotidique différente mais de la même longueur. Après avoir bloqué les billes avec de l’ARNt de levure, nous avons chargé les séquences d’ARN biotinylées sur les billes de streptavidine et les avons incubées avec l’extrait protéique total des cellules HEK 293T. Après incubation et plusieurs étapes de lavage pour éliminer les liants non spécifiques, nous avons élué les protéines en interaction avec une solution à haute teneur en sel, compatible avec les réactifs de quantification des protéines les plus couramment utilisés et avec la préparation des échantillons pour la spectrométrie de masse. Nous avons quantifié l’enrichissement de TDP-43 dans le pull-down effectué avec le liant à ARN connu par rapport au contrôle négatif par spectrométrie de masse. Nous avons utilisé la même technique pour vérifier les interactions sélectives d’autres protéines prédites par calcul comme étant des liants uniques de notre ARN d’intérêt ou du contrôle. Enfin, nous avons validé le protocole par transfert Western via la détection de TDP-43 avec un anticorps approprié.
Ce protocole permettra l’étude des partenaires protéiques d’un ARN d’intérêt dans des conditions quasi physiologiques, aidant à découvrir des interactions protéine-ARN uniques et imprévues.
Introduction
Les protéines de liaison à l’ARN (RBP) sont devenues des acteurs cruciaux dans la régulation transcriptionnelle et post-transcriptionnelle des gènes, car elles sont impliquées dans des processus tels que l’épissage de l’ARNm, la localisation cellulaire de l’ARN, la traduction, la modification et la dégradation 1,2,3. De telles interactions entre les deux macromolécules sont hautement coordonnées, précisément équilibrées et essentielles à la formation de centres fonctionnels et de traitement. Les variations ou les dérégulations au sein de ces centres ont le potentiel de perturber les réseaux protéine-ARN finement régulés et sont de plus en plus associés à une variété de maladies humaines, y compris le cancer 4,5 et les troubles neurodégénératifs 6,7,8. Les interactions entre les molécules d’ARN et leurs partenaires de liaison aux protéines peuvent être soit stables et faciles à valider expérimentalement, soit très dynamiques, transitoires et plus difficiles à caractériser.
Ces dernières années, des efforts intensifs ont été entrepris pour comprendre ces interactions. Parmi les méthodes les plus établies, les tests de pull-down () de protéines sont probablement les approches les plus appréciées et les plus couramment utilisées pour démêler les principaux acteurs constituant les complexes ribonucléoprotéines (RNP) et autres réseaux d’interaction protéine-ARN 3,9,10. Les MP comprennent un large éventail de techniques informatives, telles que l’immunoprécipitation de l’ARN (RIP)11,12 ou de la protéine (CLIP)13,14 d’intérêt. Certains de ces protocoles ARN-utilisent un ARN connu comme appât pour les protéines15, le plus souvent en tirant parti des marqueurs de haute affinité tels que la biotine. Dans ce cas, les partenaires d’interaction d’un ARN biotinylé peuvent être détectés en ancrant l’ARN sur des billes enrobées de streptavidine, permettant une isolation efficace des RNP. Les principales limites de ces approches sont généralement la conception des sondes biotinylées et le test de leur capacité à lier les protéines cibles. À cette fin, il pourrait être utile de s’appuyer sur les données CLIP publiées de la protéine d’intérêt, si elles sont disponibles, car elles révèlent, avec une grande précision, les courtes régions d’ARN qui correspondent aux pics d’interactions avec la protéine cible13,16. Ces mêmes régions pourraient être utilisées pour développer des sondes pour les DP. Une autre méthode pour concevoir de tels appâts à ARN pourrait être l’évolution systématique des ligands par enrichissement exponentiel (SELEX)17, qui permet la conception d’aptamères par sélection in vitro, à partir d’une bibliothèque randomisée complète et via une série de cycles d’optimisation pilotés par PCR. Cependant, SELEX est complexe et prend beaucoup de temps, et les résultats finaux dépendent fortement de la bibliothèque initiale. Pour sélectionner l’appât à ARN à utiliser dans le protocole présenté ici, une autre approche a été exploitée, consistant à utiliser un appât à ARN conçu de novo au moyen de la puissance de calcul de l’algorithme catRAPID, qui prédit la liaison préférentielle d’une protéine donnée vers certaines séquences d’ARN18,19,20.
Le protocole présenté ici est une version d’un ARN-optimisé pour éluer des partenaires protéiques spécifiques dans des conditions quasi physiologiques, sans utiliser de détergent, d’agents dénaturants ou de températures élevées. Il repose sur des billes nano-superparamagnétiques recouvertes de streptavidine hautement purifiée et l’utilisation d’un ARN biotinylé spécifique conçu in silico comme appât. Ce protocole fournit une méthode rapide et efficace pour isoler les partenaires de liaison des molécules d’ARN biotinylées dans des conditions natives, offrant le potentiel pour un large éventail d’applications en aval. Pour tester ce protocole, une séquence d’aptamère d’ARN simple brin de 10 nucléotides, précédemment conçue pour lier la protéine TAR DNA-binding protein 43 (TDP-43) avec une affinité et une spécificité élevées, a été utilisée20. À partir de lysats cellulaires HEK 293T, les interacteurs de l’aptamère à ARN biotinylé ont été identifiés au moyen d’une analyse par spectrométrie de masse réalisée sur des échantillons détachés de l’appât à ARN à l’aide d’un tampon hypertonique. Cette analyse a confirmé l’identification et la quantification réussies de TDP-43 comme liant préféré.
Ce protocole permet l’identification réussie d’interacteurs protéiques en utilisant seulement un court oligonucléotide à ARN synthétisé in vitro. De plus, l’utilisation d’aptamères à ARN conçus in silico comme sondes21,22 garantit la spécificité des cibles à des coûts considérablement réduits.
Protocole
1. Méthodes générales et matériel
- Préparer le milieu approprié pour la culture de cellules de mammifères choisie et le préchauffer à 37 °C pendant 20 minutes avant utilisation.
- Préparez le matériel requis à l’avance, tel que décrit dans le tableau des matériaux. Verrerie autoclave, articles en plastique et stocks tampons.
- Préparez les tampons comme décrit dans le tableau 1. Ajuster le pH des solutions mères en utilisant du HCl concentré ou du NaOH avant de diluer les composants jusqu’à leur volume final.
2. Préparation de lignées cellulaires de mammifères
- Cultiver des cellules HEK 293T dans le milieu Eagle modifié (DMEM) de Dulbecco supplémenté avec 10% de sérum bovin fœtal (FBS) et 100 μg/mL de solution de pénicilline/streptomycine. Incuber à 37 °C dans un incubateur humidifié alimenté avec 5% de CO2. Divisez régulièrement les cellules.
- Avant de détacher, rincer les cellules avec suffisamment de solution saline tamponnée au phosphate (PBS) pour couvrir la surface de croissance.
- Retirez le PBS et ajoutez une couche ultra-fine de solution trypsine-EDTA.
- Incuber les cellules à 37 °C dans un incubateur humidifié muni de 5% de CO2 pendant 5 min, ou jusqu’à ce que les cellules soient détachées (elles doivent sembler dispersées sous observation microscopique).
- Diluer la solution trypsine-EDTA dix fois en ajoutant du DMEM complet pour l’inactiver et compter les cellules.
- Plaque 1,5 x 105 cellules/ml dans des plaques à 6 puits, en tenant compte de deux puits/condition à tester.
- Incuber les cellules à 37 °C pendant 48 h dans un incubateur humidifié à 5% de CO2.
REMARQUE: Vérifiez les instructions du fabricant pour le type de milieu et de supplément apte à la lignée cellulaire. En outre, la quantité et le temps d’incubation de trypsine-EDTA dépendent de la lignée cellulaire. Certains types de cellules se développent plus rapidement / plus lentement que ce qui a été rapporté dans ce protocole; Par conséquent, la concentration de semis doit être testée au préalable.
3. Récolte totale de protéines
- Retirez le milieu des puits où les cellules se développent.
- Lavez chaque puits des plaques à 6 puits avec 1 mL de PBS.
- Jetez le PBS.
- Déplacer les plaques sur de la glace et passer à l’étape 3.5 ou congeler les plaques sèches à -80 °C pour faciliter la lyse.
- Ajouter 200 μL de tampon de lyse à chaque puits.
- Utilisez un grattoir de cellules pour détacher et casser les cellules.
- Transférer l’extrait cellulaire provenant de deux puits dans le même tube de 1,5 mL.
- Placer le tube contenant l’extrait de protéine sur de la glace pendant 30 min.
- Centrifuger les lysats cellulaires à 17 000 x g pendant 15 min à 4 °C.
- Transférer chaque surnageant dans un tube pré-refroidi.
REMARQUE: Un total de 106-10 7 cellules pour chaque condition de DP sont recommandés. La lyse cellulaire et la récolte de protéines doivent être effectuées avec des tampons glacés. Des inhibiteurs de la protéase doivent être ajoutés au tampon de lyse pour prévenir la dégradation des protéines.
4. Détermination de la concentration en protéines
- Préparer le réactif Bradford, comme indiqué par le producteur, en le diluant cinq fois dansdH2O.
- Répartir 1 mL de réactif dans une cuvette de 1 cm, ajouter 1 μL de l’échantillon et mélanger par inversion.
- Incuber dans l’obscurité à température ambiante pendant 5-10 min.
- Lire l’absorbance à 595 nm.
- Calculer le volume d’extrait protéique correspondant à 1,5 mg de protéines et porter tous les échantillons à un volume final de 600 μL à l’aide d’un tampon de lyse.
- Conservez les échantillons sur la glace jusqu’à utilisation.
REMARQUE: Toute autre méthode de détermination de la concentration en protéines peut être utilisée, conformément aux recommandations de compatibilité tampon. Dans tous les cas, le tampon de lyse doit être utilisé comme un blanc. De nombreux réactifs ne sont pas compatibles avec le dithiothréitol (DTT). Il est recommandé d’ajouter du DTT ou d’autres agents réducteurs uniquement après quantification des protéines (DTT à une concentration finale de 1 mM).
5. Préparation des perles
- Mélangez les perles dans leur tampon de stockage en agitant le tube.
- Calculer 100 μL de milieu de boue/échantillon et placer le volume dans une grille magnétique.
- Lavage des perles
- Retirez la solution de stockage et lavez les billes en ajoutant 1 mL de tampon/tube de lyse et retournez-le manuellement.
- Retirez la mémoire tampon à l’aide du rack magnétique.
- Répétez l’étape de lavage.
- Ajouter un volume de tampon de lyse égal au volume initial du milieu de boue, mélanger en agitant le tube et distribuer le milieu uniformément dans autant de tubes de 1,5 mL qu’il y a d’échantillons.
- Blocage des billes
- Retirer le tampon à l’aide de la grille magnétique et ajouter 600 μL d’une solution à 0,25 mg/mL d’ARNt de levure préparée dans un tampon de lyse.
- Incuber pendant 1 h à température ambiante sur une roue tournante.
- Retirez la solution d’ARNt à l’aide du rack magnétique.
- Ajouter 600 μL de tampon de lyse et laver en mélangeant manuellement.
- Répétez l’étape de lavage et jetez le tampon.
6. Chargement du cordon
- Préparer 200 μg d’ARN oligonucléotide dans 600 μL de tampon de lyse pour chaque tube contenant le milieu de suspension initial de 100 μL (maintenant des billes bloquées).
- Ajouter l’oligo aux billes et incuber pendant 1 h à température ambiante en tournant.
- Retirer la solution, ajouter 600 μL de tampon de lyse et laver les billes deux fois en tournant les tubes pendant 5 min à température ambiante.
- Supprimez la mémoire tampon.
REMARQUE: Ne jamais vortex les perles, mais effleurer à la place. Limitez le nombre d’étapes de pipetage, sauf si nécessaire. Dans la mesure du possible, utilisez des pointes coupées de 1 mL. La quantité de milieu de boue ou d’échantillon dépend de la capacité de liaison des billes et de la quantité initiale de protéines totales. Si l’on prévoit que l’oligo ARN a une structure secondaire importante, nous recommandons de le dénaturer d’abord à 80 °C pendant 10 minutes, puis de le refroidir lentement à température ambiante ou de le remodeler en l’incubant à 30 °C pendant 1 h. Il est suggéré de récupérer l’oligo ARN après chargement des billes et de déterminer la concentration restante, afin d’optimiser la quantité nécessaire pour la charge et d’évaluer la possibilité de réutiliser l’ARN.
7. Liaison aux protéines sur les billes
REMARQUE: À partir de maintenant, lorsque cela est possible, effectuez les étapes à 4 ° C.
- Prélever un volume de 5% de la solution protéique de 600 μL et le conserver comme INPUT (IN) pour une analyse plus approfondie (1,5 mg de protéines sont dissous dans 600 μL, donc 5% correspond à 30 μL et 75 μg de protéines).
- Ajouter le reste du mélange de protéines à chaque tube de billes chargées et laisser tourner lentement pendant la nuit à 4 °C.
8. Lavage des liants non spécifiques
- Retirez la fraction non liée à l’aide du rack magnétique. Économisez 5% du volume et étiquetez-le comme FLOWTHROUGH (FT) (le volume non lié est d’environ 600 μL, donc gardez à nouveau 30 μL pour une analyse plus approfondie).
- Ajouter 1 mL de tampon de lavage 1 aux billes et laisser tourner pendant 5 min à 4 °C.
- Supprimez la mémoire tampon.
- Répétez les étapes 8.2 et 8.3.
- Ajouter 1 ml de tampon de lavage 2 aux billes et laisser tourner pendant 5 min à 4 °C.
- Jetez le surnageant.
9. Élution de liants spécifiques
- Ajouter 100 μL de tampon d’élution 1 ou de tampon d’élution 2 aux billes.
- Mélanger manuellement en agitant et incuber pendant 5 min à température ambiante.
- Placer les tubes dans un thermomélangeur et agiter vigoureusement pendant 5 min à 95 °C.
- Placez le tube dans le rack magnétique et recueillez la fraction éluée dans un tube propre.
- Faites tourner rapidement les billes avec une centrifugeuse de banc pour maximiser la récupération de l’éluat.
- Économisez 5 % du volume total d’ELUATE (EL) pour d’autres analyses (le volume total est de 100 μL, séparez donc 5 μL dans un autre tube).
- Si nécessaire, la concentration en protéines peut être déterminée comme dans la section 4, en utilisant un tampon d’élution comme blanc.
NOTE: Il est recommandé de s’abstenir d’ajouter du DTT au tampon d’élution jusqu’à ce que la concentration en protéines ait été déterminée. Si la quantification des protéines n’est pas nécessaire, ou si des kits de quantification des protéines compatibles avec les agents réducteurs sont disponibles, 1 mM de TNT peut être ajouté au tampon d’élution dès le départ. Dans ce protocole, le tampon d’élution 1 (contenant 1 M NaCl) et le tampon d’élution 2 (avec 2 M NaCl) ont été testés. Aucune différence dans l’efficacité d’élution de la protéine cible n’a été observée avec une force ionique accrue, mais il est recommandé de tester les deux conditions avant d’établir le tampon le plus approprié. Si une forte présence de sel dans le tampon d’élution représente une limite pour une analyse plus approfondie, la très faible quantité de détergents dans le tampon d’élution permet un échange tampon. Comme alternative, l’éluat peut être dilué pour atteindre la concentration de sel souhaitée.
10. Identification des liants protéiques par spectrométrie de masse
- Précipitation de l’acétone
- Concentrer les protéines éluées en les diluant quatre fois dans de l’acétone froide (-20 °C).
- Vortex et incuber le tube à -20 °C pendant une nuit.
- Essorer à 17 000 x g pendant 30 min à 4 °C.
- Retirez délicatement le surnageant et laissez l’acétone s’évaporer jusqu’à ce que la pastille soit complètement sèche.
- Digestion des protéines en solution
- Dissoudre la pastille de protéine en ajoutant 50 μL de tampon de dénaturation.
- Ajouter le DTT à une concentration finale de 5 mM, permettant une réduction des protéines pendant 30 min à 55 °C.
- Refroidir les échantillons à température ambiante et procéder à la réaction d’alkylation des protéines, en ajoutant de l’iodoacétamide (IAA) à une concentration de 10 mM pendant 15 min.
- Digérer les protéines à l’aide d’une enzyme appropriée (trypsine, LysC) et incuber les échantillons pendant une nuit à 37 °C.
- Arrêtez la digestion en ajoutant 1 μL d’acide trifluoroacétique (TFA) à 10%.
- Nettoyez et concentrez les peptides sur une micro-colonne C18 en phase inversée personnalisée, comme décrit précédemment19.
- Peptides élutés de la pointe C18 avec tampon B.
- Retirer le composant organique à l’aide d’une centrifugeuse sous vide et remettre les peptides en suspension dans 5 μL d’acide formique à 0,1% pour une analyse plus approfondie.
REMARQUE: Alternativement, la digestion des protéines peut être effectuée « sur des perles » immédiatement après le lavage des liants non spécifiques (étapes 8.1-8.6), rendant ainsi le protocole plus rapide. Cependant, il est conseillé de tester l’efficacité de l’enzyme travaillant « sur les billes » des protéines immobilisées par rapport à la digestion standard en solution, afin de garantir la couverture optimale de la séquence protéique expérimentale.
- Chromatographie liquide-spectrométrie de masse en tandem (LC-MS/MS)
- Branchez la colonne d’analyse (phase stationnaire C18) et maintenez-la à 45 °C pendant la durée de l’exécution.
- Connectez la colonne à la sortie d’une vanne rotative à six orifices de la pompe LC à l’aide d’un raccord capillaire serré au doigt (20 μm x 550 mm) dans une configuration à une colonne.
- Ajustez les paramètres LC comme suit :
- Charger les peptides sous pression contrôlée (980 bar) dans le tampon A.
- Appliquez un gradient B de tampon B de 5 % à 20 % à 300 nL/min pendant 59 min, suivi d’un gradient B de tampon de 20 % à 30 % sur 15 min et d’un gradient B de tampon B de 30 % à 65 % sur 5 min.
- Ajouter une étape de lavage en augmentant la concentration du tampon B jusqu’à 95 % sur 5 min plus une étape isocratique de 5 min à 95 % du tampon B.
- Utilisez le spectromètre de masse en mode d’acquisition dépendante des données (DDA) pour basculer automatiquement entre les événements MS et MSMS.
- Définissez un nombre de boucles égal à 15 à l’aide d’une valeur cible de contrôle automatique de gain (AGC) de 3 x 106 et 1 x 105 pour les événements MS et MSMS, respectivement.
- Réglez le temps d’accumulation d’ions maximal autorisé à 20 ms pour MS avec une résolution de 60 K, et à 100 ms pour MSMS avec une résolution de 15 K.
- Effectuer une expérience de fragmentation par dissociation par collision élevée (HCD) en utilisant une énergie de collision normalisée de 28 %, avec un temps d’exclusion dynamique de 20 s.
- Utilisez les paramètres source comme suit :
Tension de pulvérisation : 1,7 kV
Tension capillaire : 275 °C
Ni gaine ni gaz auxiliaire utilisés
REMARQUE: Dans ce protocole, l’analyse par chromatographie liquide à ultra-haute performance (UHPLC) par spectrométrie de masse (MS) a été spécifiquement réalisée à l’aide d’une configuration LC à colonne unique, couplée à un instrument hybride triple quadripolaire orbitrap (Table of Materials). D’autres systèmes LCMS peuvent être utilisés, mais une adaptation des paramètres est recommandée.
- Analyse des données
- Utilisez le bouton Charger pour importer les fichiers bruts.
- Définissez les noms des expériences en cliquant sur le bouton Définir l’expérience .
- Entrez dans la section des paramètres spécifiques au groupe pour spécifier tous les paramètres liés à l’identification :
Enzyme utilisée pour la digestion : Trypsine/P
Clivages manqués : jusqu’à trois
Modification fixe: Carbamidométhylation
Modification variable: N-acétyle (protéine), oxydation (M) - Téléchargez un fichier FASTA mis à jour, disponible à partir de bases de données publiques telles que UniprotKB.
- Spécifiez les règles d’analyse correctes en fonction de la source de la base de données choisie.
- Définissez une valeur de taux de fausse découverte (FDR) = 1 pour les protéines et les peptides.
- Ajoutez l’option Quantification sans étiquette (LFQ) dans l’onglet Quantification sans étiquette.
- Maintenir le nombre minimum de LFQ à deux.
REMARQUE: Ici, nous décrivons l’analyse des données à l’aide des logiciels MaxQuant24 et Perseus25 pour effectuer la quantification des protéines et l’analyse statistique ultérieure, respectivement. Cependant, l’analyse des données peut être effectuée avec toute autre bioinformatique disponible dans le commerce ou gratuite. Le FDR est estimé à l’aide d’une approche fondée sur une base de données cible-leurre26. Un FDR peptidique et protéique égal à 0,01 signifie que les peptides et les protéines identifiés devraient contenir 1% de faux positifs.
- Analyse statistique
- Chargez le fichier protéine.txt pour effectuer une analyse statistique au niveau de la protéine.
- Définissez les valeurs LFQ en tant que colonnes principales.
- Supprimez « inverser » et « contaminants » en filtrant les lignes en fonction de la colonne catégorielle.
- Utilisez les lignes d’annotation catégorielles pour regrouper les différentes conditions expérimentales.
- Réduisez la matrice de données, en sélectionnant le nombre de valeurs valides dans chacun des groupes définis précédemment.
- Sélectionnez le test statistique qui convient le mieux aux conditions expérimentales (c.-à-d. test t, test ANOVA à échantillons multiples).
- Déterminez un seuil pour les indices significatifs à l’aide d’un calcul basé sur le FDR. En règle générale, 0,01 et 0,05 sont acceptés comme seuils pour la valeur p ajustée.
- Visualisez les résultats des analyses différentielles basées sur les statistiques du test t à l’aide d’une représentation de parcelle volcanique.
- Exportez la matrice finale dans .txt format pour une modification ultérieure du tableau de résultats finaux.
Remarque : le dossier de configuration contient un fichier FASTA avec des protéines telles que les kératines qui sont considérées comme des contaminants courants dans les expériences protéomiques globales, qui sont marquées d’un + dans la table de sortie. Dans la présente étude, ayant deux conditions d’échantillonnage, un test t est utilisé pour l’analyse statistique.
11. Validation des résultats par transfert Western
- Préparation des échantillons
- Ajouter le volume approprié de tampon de chargement d’échantillon 4x à chaque aliquote de IN, FT et EL.
- Faire bouillir les échantillons pendant 5 min à 95°C.
- Rotation rapide pour récupérer l’échantillon évaporé du haut des tubes.
- SDS-PAGE et transfert de gel
- Charger les échantillons sur un gel de polyacrylamide dénaturant à 4% -12%.
- Exécutez le gel avec le tampon de fonctionnement MES SDS pendant 1,5 h à 120 V.
- Transférer le gel sur une membrane de nitrocellulose avec une cassette de transfert semi-sèche, en suivant les instructions du fabricant. Nous recommandons un transfert de 10 min à 15 V.
- Immunodétection
- Bloquer la membrane avec 10% d’albumine sérique bovine (BSA) pendant 1 h à température ambiante tout en agitant doucement.
- Ajouter l’anticorps primaire préparé dans 5% BSA dans TBST, selon les instructions du fabricant. Laisser reposer toute la nuit à 4 °C ou pendant 1 h à température ambiante sous agitation douce.
- Lavez la membrane trois fois avec TBST, chaque fois pendant 5 min.
- Ajouter l’anticorps secondaire préparé dans TBST pendant 1 h à température ambiante sous agitation.
- Lavez la membrane trois fois avec TBST, chaque fois pendant 5 min.
- Visualisez les résultats à l’aide d’un imageur de transfert.
REMARQUE: Pour détecter TDP-43, un liant connu de notre ARN, un anticorps monoclonal recombinant de lapin a été utilisé et laissé avec la membrane pendant la nuit à 4 ° C. En tant qu’anticorps secondaire, la peroxydase de raifort (HRP) IgG anti-lapin a été utilisée, mais un anticorps secondaire fluorescent fonctionnerait également. Pour visualiser les anticorps sur la membrane, la membrane a été incubée avec le substrat ECL Clarity Western pendant 1 min, avant d’être associée au système d’imagerie ChemiDoc.
Résultats
Pour vérifier la validité du protocole proposé, les expériences présentées ici ont été réalisées avec un aptamère d’ARN biotinylé conçu in silico pour se lier spécifiquement à TDP-4320. Cet ARN se lie à sa cible protéique avec une affinité de liaison élevée (Kd = 90 nM)20. Ici, cet ARN, de la séquence 5'-CGGUGUUGCU-3', est désigné par le nom de « +ARN ». En tant que contrôle négatif, la séquence complémentaire inverse de +ARN, appelée ici « -ARN », a été utilisée. Sa séquence est 5'-AGCAACACCG-3'. -L’ARN montre une affinité de liaison significativement plus faible envers TDP-43 (Kd = 1,5 μM)19. Pour les besoins du protocole décrit ici, ces oligonucléotides d’ARN ont été achetés conjugués à une molécule de biotine, pour permettre la liaison aux billes de streptavidine. +ARN a été acheté avec un biotine-TEG à son extrémité 3', qui comprend un espaceur de triéthylèneglycol à 15 atomes entre la biotine et le groupe phosphate de l’acide nucléique; -L’ARN avait plutôt une biotine à son extrémité 5', conjuguée à l’acide nucléique via un agent de liaison amino-C6. Cependant, si la conception de l’appât à ARN est robuste et tant qu’il n’y a pas d’interférence structurelle ou chimique entre l’agent de liaison et l’ARN, d’autres positions pour la conjugaison de la biotine et d’autres longueurs de liaison pourraient être utilisées.
Connaître l’identité de la protéine principale liée à la sonde +ARN après la a permis de valider le protocole par identification de TDP-43 dans l’éluat, en utilisant à la fois la spectrométrie de masse (MS) et le transfert Western (WB) (Figure 1).
L’analyse de la SM a été réalisée sur quatre répétitions de effectuées à l’aide de +ARN ou d’ARN-ARN (Figure 2). L’identification des interactomes de +ARN et -ARN dépasse le cadre de ce protocole, mais certains résultats qui valident l’exactitude du protocole sont rapportés. Il convient de noter que le tracé des protéines significativement enrichies dans un graphique de volcan a révélé que la teneur totale en protéines et les protéines enrichies éluées à partir de +ARN étaient significativement plus élevées que ce qui a été récupéré de l’ARN (Figure 2). Cela signifie que, bien qu’ayant la même longueur et le même contenu structurel (linéaire), +ARN peut établir un plus grand nombre d’interactions spécifiques, qui sont conservées jusqu’à l’étape d’élution avec un sel élevé. Il est probable que l’ARN établit plutôt un nombre plus élevé de contacts non spécifiques qui sont perturbés pendant les étapes de lavage. Comme prévu, TDP-43 a été identifié comme un interacteur unique de +ARN20; la quantification moyenne sans marquage (LFQ) pour les quatre réplications réalisées avec +ARN est de 31,96 ± 0,56, tandis que la protéine n’est pas identifiée parmi les interacteurs de l’ARN-. De plus, parmi tous les interacteurs uniques de +ARN, TDP-43 s’est avéré être la protéine la plus richement enrichie.
Pour valider davantage le protocole, l’algorithme interne catRAPID18,19 a été utilisé pour prédire par calcul quelles autres protéines se lieraient spécifiquement à +ARN ou -ARN. En particulier, les scores d’interaction pour +ARN et ARN-ARN avec les protéines composant le protéome humain ont été calculés à l’aide de la fonction de « propension à l’interaction » de catRAPID, telle que définie dans nos travaux précédents27. Parmi les protéines notées avec un degré de confiance élevé, HNRNPH3 devait se lier sélectivement +ARN (+score d’interaction ARN = 1,01; score d’interaction ARN = 0,21) et PCBP2 pour interagir spécifiquement avec -ARN (+score d’interaction ARN = -0,5; -score d’interaction ARN = 0,31) (Figure 3A). De plus, la protéine RBM41 a été prédite comme étant une promiscuité pour les deux oligonucléotides d’ARN (+score d’interaction ARN = 0,4; score d’interaction ARN = 0,39) (Figure 3A). L’analyse MS a en effet confirmé la présence de HNRNPH3 et de PCBP2 dans la de +ARN et -ARN respectivement, tandis que RBM41 a été trouvé en interaction avec les deux (Figure 3B).
WB a été utilisé pour détecter la présence de TDP-43 afin de confirmer davantage les résultats et lors de l’optimisation du protocole (Figure 4). Dans la procédure décrite ici, différents échantillons ont été prélevés à différentes étapes. L’échantillon d’entrée (IN) était constitué des protéines totales diluées dans un tampon de lyse. Le flux continu (FT) a été obtenu après une nuit d’incubation des protéines totales avec les billes de streptavidine pré-enrobées avec l’ARN biotinylé, représentant la fraction de protéines qui ne se sont pas liées à l’ARN. Enfin, l’éluat (EL) contenait toutes les protéines qui reconnaissaient spécifiquement l’ARN étudié, car entre les étapes FT et EL, trois étapes de lavage avec 150 mM de sel et 0,1% de triton-X auraient dû éliminer les interactions les plus faibles.
Pour chaque répétition, la même quantité (5 % v/v) d’IN, FT et EL a été administrée en parallèle sur une SDS-PAGE et colorée avec un anticorps anti-TDP-43 (Figure 4). Dans le cas de +ARN, la bande de TDP-43 a été observée dans IN et dans EL, indiquant que la protéine, présente dès le départ dans l’extrait protéique total, est retenue par +ARN pendant les étapes de lavage et n’est éluée qu’à la fin avec un tampon salin élevé. TDP-43 était également présent dans IN pour -ARN, mais la bande correspondant à la protéine est également visible dans FT, indiquant que cet ARN ne se lie pas à TDP-43. L’absence de la bande TDP-43 dans EL confirme ce résultat.
Au cours de l’optimisation du protocole, l’élution des protéines spécifiquement liées aux séquences d’ARN a été sondée à la fois avec un tampon d’élution contenant 1 M NaCl (EB1) et avec un tampon d’élution complet avec 2 M NaCl (EB2) (Figure 4). Les éluats obtenus avec l’un ou l’autre EB ont été comparés sur une SDS-PAGE et épongés avec l’anticorps anti-TDP-43. Les images obtenues ont ensuite été analysées avec ImageJ28 pour quantifier toute différence de quantité de TDP-43 éluée avec les deux tampons. Dans l’ensemble, aucune différence significative n’a été observée et nous avons conclu que, dans ces essais, 1 M de sel est suffisant pour perturber même les interactions protéine-ARN les plus fortes.
Dans l’ensemble, les résultats rapportés ici pour la SEP et les WB démontrent que ce protocole est efficace pour capturer les interacteurs protéiques d’un ARN donné d’une manière spécifique, et qu’il permet l’élution dans des tampons compatibles avec l’analyse en aval.
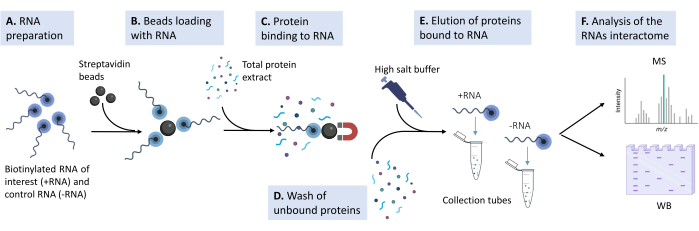
Figure 1 : Croquis du pipeline expérimental utilisé dans le protocole proposé. (A) L’oligonucléotide ARN biotinylé est préparé dans un tampon de lyse à la concentration appropriée. (B) Les billes magnétiques de streptavidine sont lavées, bloquées par de l’ARNt de levure et chargées d’ARN biotinylé. (C) Un extrait protéique total dérivé de lignées cellulaires de mammifères cultivées est ajouté au mélange perles-ARN. (D) Plusieurs lavages sont effectués pour éliminer les interactions non spécifiques. (E) Les interacteurs protéiques spécifiques sont détachés de l’ARN avec une solution hypertonique. (F) L’identité des interacteurs est révélée par spectrométrie de masse, et les cas spécifiques sont validés par transfert Western. Veuillez cliquer ici pour voir une version agrandie de cette figure.
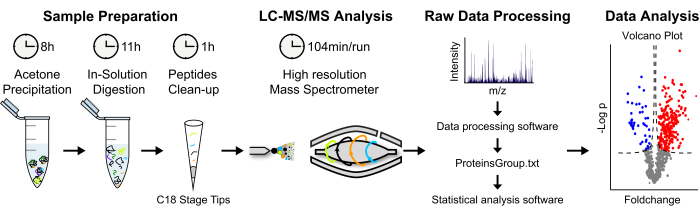
Figure 2 : Stratégie analytique pour la quantification des protéines MS sans marquage. (A) Les protéines éluées sont précipitées dans de l’acétone froide pendant une nuit. Les protéines sont ensuite dénaturées et une digestion en solution est effectuée. Les peptides protéolytiques sont concentrés et dessalés. (B) Les peptides sont analysés via LC-MS/MS en utilisant une « approche shotgun ». (C) Le traitement et l’analyse des données brutes sont effectués à l’aide des logiciels MaxQuant et Perseus, respectivement. (D) Des protéines enrichies statistiquement significatives sont affichées dans un graphique volcanique. Veuillez cliquer ici pour voir une version agrandie de cette figure.
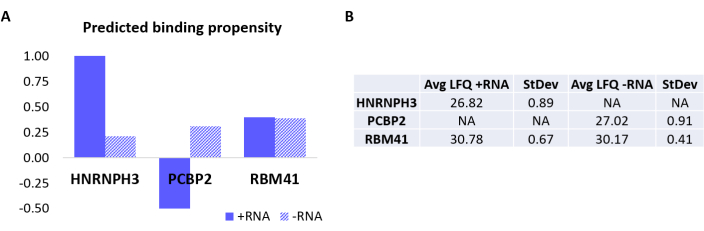
Figure 3 : Corrélation entre les propensions aux interactions prédites et les interactions déterminées expérimentalement de +ARN et ARN-ARN. (A) les scores d’interaction RAPID cat par rapport à HNRNPH3, PCBP2 et RBM41, indiquant une liaison préférentielle de HNRNPH3 pour +ARN et de PCBP2 pour -ARN, tandis que RBM41 devrait lier indistinctement lesdeux séquences d’ARN. (B) Moyennes de quantification sans marquage déterminées par analyse par spectrométrie de masse à partir des pull-downs effectuées avec +ARN et ARN-ARN. L’analyse confirme que HNRNPH3 se lie uniquement +ARN, PCBP2 se lie uniquement -ARN et RBM41 lie les deux de manière égale. Veuillez cliquer ici pour voir une version agrandie de cette figure.
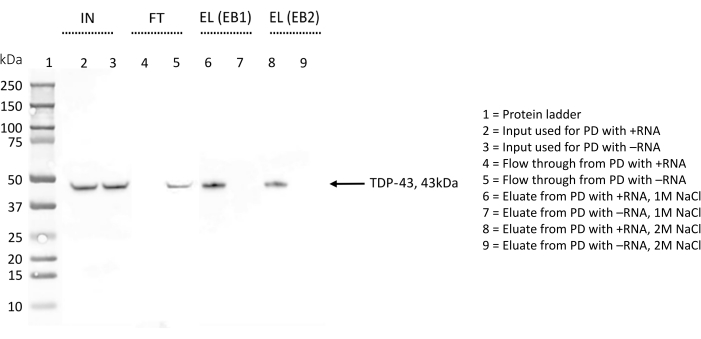
Figure 4 : Validation par transfert Western de la présence/absence de TDP-43 parmi les interacteurs des séquences d’ARN choisies. La membrane WB a été traitée avec un anticorps anti-TDP-43. IN = entrée; FT = accréditive; EL (EB1) = élution avec tampon d’élution 1; EL (EB2) = élution avec tampon d’élution 2; le signe « + » indique des échantillons dérivés du pull-down effectué avec +ARN; le signe « - » indique des échantillons provenant du pull-down effectué avec -ARN; La voie 1 contient une échelle à protéines. TDP-43 est indiqué par une flèche. La WB indique que TDP-43 se trouve parmi les interacteurs +ARN mais pas parmi les interacteurs -ARN. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| Nom de la mémoire tampon | Composition | |||||
| 10x tampon de transfert | 250 mM de tris, 1,92 M de glycine, 1% de SDS, 20% de méthanol. Diluer 10 fois l’utilisation antérieure | DP | ||||
| 20X MESS SDS en cours d’exécution Buffer | 1 M MES, 1 M tris, 2% SDS, 20 mM EDTA. Ajuster le pH à 7,3. Diluer 20 fois l’utilisation antérieure | |||||
| 4x Tampon de chargement d’échantillon | 0,25 M Tris base, FDS 0,28 M, 40% glycérol, 20% 2-mercapto-éthanol, 4 mg/ml de bleu de bromphénol | |||||
| Tampon d’élution 1 | 20 mM phosphate pH 7,5, 1 M NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM TNT (à ajouter après quantification) | |||||
| Tampon d’élution 2 | 20 mM phosphate pH 7,5, 2 M NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM de TNT (à ajouter après quantification) | |||||
| Tampon de lyse | 10 mM Tris-HCl pH 7,4, 150 mM NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM DTT et inhibiteurs de protéase | |||||
| Solution saline tamponnée Tris avec Tween-20 | 1 M Tris-HCl pH 7,4, 3 M NaCl, 2,0% Tween-20 | |||||
| Tampon de lavage 1 | 10 mM Tris-HCl pH 7,4, 150 mM NaCl, 0,5 mM EDTA, 0,1 % Triton TM X-100, 1 mM DTT et inhibiteurs de protéase | |||||
| Tampon de lavage 2 | Hepes pH 8 25 mM, NaCl 150 mM, 0,5 mM EDTA, 0,1 % Triton X-100, DTT 1 mM et inhibiteurs de la protéase | |||||
| Tampon A | 0,1% d’acide formique | MS | ||||
| Tampon B | 60 % d’acétonitrile, 0,1 % d’acide formique | |||||
| Tampon de dénaturation | 8M d’urée, 50 mM de Tris-HCl | |||||
Tableau 1 : Tampons de DP et de SM. Noms et composition des tampons utilisés pour les expériences de pull-down () ou pour l’analyse par spectrométrie de masse (MS).
Discussion
Ce travail rapporte l’optimisation d’un protocole de réalisé avec des oligonucléotides d’ARN biotinylés pour capturer leurs interacteurs protéiques. Le protocole décrit ici est simple à réaliser, nécessite peu de matériel et produit des résultats très fiables. Il est important de noter que les aspects les plus novateurs de ce protocole consistent en l’utilisation d’un appât à ARN conçu entièrement in silico et spécifique à la cible protéique, et l’élution de toutes les protéines liées à l’appât à ARN en perturbant directement leurs interactions avec une solution à haute teneur en sel, plutôt qu’en dissociant la streptavidine de la biotine avec un détergent et un traitement à haute température.
Ce protocole tire parti de la force de la liaison entre la biotine et la streptavidine 29,30. Selon les billes de streptavidine choisies, la charge de l’ARN biotinylé doit être testée et quantifiée avant de procéder. En outre, le repliement tridimensionnel de l’ARN pourrait affecter l’efficacité de chargement sur les billes, car il pourrait limiter l’exposition de la biotine à la streptavidine. Le blocage des billes avec de l’ARNt non biotinylé améliore la propreté des résultats en limitant les interactions non spécifiques avec les billes. Le tampon de chargement et le tampon d’élution doivent être choisis en fonction des applications en aval. Ici, des conditions très douces, adaptées à la majorité des applications et développées pour préserver les complexes protéiques potentiels, ont été proposées. Cette méthode est cependant très adaptable; l’utilisateur peut choisir n’importe quelle lignée cellulaire et n’importe quelle taille d’ARN, et peut décider de répéter le protocole après le repliement / dépliage de l’ARN pour déterminer l’effet de la structure sur les propriétés de liaison.
Un autre aspect original de ce protocole est l’utilisation d’outils de prédiction in silico pour assurer l’exactitude des résultats20. Savoir à l’avance quelles protéines doivent être identifiées comme interacteurs de l’ARN d’intérêt donne l’avantage sans précédent de valider les aspects techniques du protocole. Par exemple, à l’aide d’une simple analyse WB, il est possible de vérifier la présence d’une cible protéique connue dans les échantillons issus des différentes étapes du protocole avant de procéder à l’analyse MS, qui nécessite une instrumentation spécialisée et est plus coûteuse. En outre, une méthode permettant d’utiliser le chatRAPID20, un algorithme interne de prédiction protéine-ARN, pour concevoir de novo l’ARN spécifique d’une protéine cible a récemment été rapportée. Jusqu’à récemment, le seul pipeline disponible pour concevoir des aptamères ADN/ARN pour une protéine cible était l’approche SELEX (systematic evolution of ligands by exponential enrichment)31. La méthode in silico permet une conception beaucoup plus rapide et rentable des aptamères d’ARN.
Les principales limites de cette méthode sont associées à la nécessité de travailler dans des tampons et des outils sans nucléase. En outre, s’il est jugé nécessaire de confirmer in vitro la liaison entre un ARN conçu de novo et une protéine cible avant la MP, la protéine doit être produite et purifiée et la liaison déterminée par des approches biophysiques. C’est une limitation qui est partagée avec la production d’anticorps monoclonaux.
Malgré ces problèmes mineurs, des méthodes fiables pour cartographier les interactions ARN-protéine, comme celle présentée ici, peuvent rapprocher les scientifiques pour dévoiler des réseaux macromoléculaires et des acteurs principaux complexes de nombreux mécanismes physiologiques et pathologiques, tels que ceux impliqués dans le cancer, les cardiomyopathies, le diabète, les infections microbiennes et les troubles génétiques et neurodégénératifs.
Déclarations de divulgation
Les auteurs n’ont pas d’intérêts financiers concurrents ou d’autres conflits d’intérêts.
Remerciements
Les auteurs tiennent à remercier le groupe de recherche du professeur Tartaglia et du Dr Cuomo pour le soutien offert. E.Z. a reçu un financement de la bourse MINDED du programme de recherche et d’innovation Horizon 2020 de l’Union européenne dans le cadre de la convention de subvention Marie Sklodowska-Curie n ° 754490.
matériels
| Name | Company | Catalog Number | Comments |
| 6-well tissue culture plates | VWR | 10861-554 | CELLS |
| Cell scrapers | BIOSIGMA | 10153 | CELLS |
| Dulbecco′s Modified Eagle′s Medium (DMEM) | Thermo Fisher Scientfic | 11995065 | CELLS |
| Fetal Bovine Serum, qualified, heat inactivated, Brazil | Thermo Fisher Scientfic | 10500064 | CELLS |
| Phosphate Buffer Saline (PBS, Waltham, MA) | Thermo Fisher Scientfic | 14190169 | CELLS |
| Trypsin (0.25%), phenol red | Thermo Fisher Scientfic | 15050065 | CELLS |
| Anti-rabbit IgG horseradish peroxidase (HRP) | Cellsignal | 7070 | PD |
| Biotinylated RNA | Eurofins | Custom RNA oligonucleotides | PD |
| Bovine serum albumin | Sigma-Aldrich | A9418 | PD |
| Clarity Western ECL Substrate, 500 ml | Biorad | 1705061 | PD |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Merck - Sigma Aldrich | 5056489001 | PD |
| NuPAGE 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel, 10-well | Invitrogen | NP0321BOX | PD |
| Recombinant anti-TDP43 antibody | Abcam | ab109535 | PD |
| Ribonucleic acid, transfer from baker's yeast (S. cerevisiae) | Merck - Sigma Aldrich | R5636-1ML | PD |
| Streptavidin Mag Sepharose | Merck - Sigma Aldrich | GE28-9857-99 | PD |
| Trans-Blot Turbo RTA Mini 0.2 µm PVDF Transfer Kit | Biorad | 1704272 | PD |
| Acetone | Thermo Fisher Scientfic | 022928.K2 | MS |
| C18 cartridge | Thermo Fisher Scientfic | 13-110-018 | MS |
| Dithiothreitol (DTT) | Thermo Fisher Scientfic | 20290 | MS |
| EASY-Spray HPLC Columns | Thermo Scientific | ES902 | MS |
| iodoacetamide (IAA) | Sigma Aldrich S.r.l. | I6125 | MS |
| Lys-C/Trypsin | Promega | V5073 | MS |
| Trifluoroacetic acid (TFA) | Thermo Fisher Scientfic | 28904 | MS |
| Urea | Thermo Fisher Scientfic | J75826.A7 | MS |
| Equipment | |||
| ChemiDoc imaging system | Bio-Rad | CELLS | |
| Dyna Mag -2 , Magnetic rack | Invitrogen | CELLS | |
| Forma Series 3 water jacketed C02 incubator | Thermo Scientific | PD | |
| PROTEAN II xi cell , power supply for PAGE applications | Bio-Rad | PD | |
| Rotating wheel, rotator SB3 | Stuart | PD | |
| Water bath set at 37 °C | VWR | PD | |
| XCell SureLock Mini-Cell electrophoresis system | ThermoFisher Scientific | MS | |
| Easy-nLC 1200 UHPLC | Thermo Scientific | MS | |
| Q exactive Mass Spectrometer | Thermo Scientific | MS | |
| Software | Version | ||
| MaxQuant | 2.0.3.0 | MS | |
| Perseus | 1.6.14.0 | MS |
Références
- Gebauer, F., Schwarzl, T., Valcárcel, J., Hentze, M. W. RNA-binding proteins in human genetic disease. Nature Reviews Genetics. 22 (3), 185-198 (2021).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Cooper, T. A., Wan, L., Dreyfuss, G. RNA and disease. Cell. 136 (4), 777-793 (2009).
- Qin, H., et al. RNA-binding proteins in tumor progression. Journal of Hematology & Oncology. 13 (1), 90 (2020).
- Nussbacher, J. K., Tabet, R., Yeo, G. W., Lagier-Tourenne, C. Disruption of RNA metabolism in neurological diseases and emerging therapeutic interventions. Neuron. 102 (2), 294-320 (2019).
- Duan, R., Sharma, S., Xia, Q., Garber, K., Jin, P. Towards understanding RNA-mediated neurological disorders. Journal of Genetics and Genomics. 41 (9), 473-484 (2014).
- Maziuk, B., Ballance, H. I., Wolozin, B. Dysregulation of RNA binding protein aggregation in neurodegenerative disorders. Frontiers in Molecular Neuroscience. 10, 89 (2017).
- Zielinski, J., et al. In vivo identification of ribonucleoprotein-RNA interactions. Proceedings of the National Academy of Sciences. 103 (5), 1557-1562 (2006).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Weidmann, C. A., Mustoe, A. M., Jariwala, P. B., Calabrese, J. M., Weeks, K. M. Analysis of RNA-protein networks with RNP-MaP defines functional hubs on RNA. Nature Biotechnology. 39 (3), 347-356 (2021).
- Graindorge, A., et al. In-cell identification and measurement of RNA-protein interactions. Nature Communications. 10 (1), 5317 (2019).
- Ule, J., Hwang, H. W., Darnell, R. B. The future of cross-linking and immunoprecipitation (CLIP). Cold Spring Harbor Perspectives in Biology. 10 (8), 032243 (2018).
- Ascano, M., Hafner, M., Cekan, P., Gerstberger, S., Tuschl, T. Identification of RNA-protein interaction networks using PAR-CLIP. Wiley Interdisciplinary Reviews. RNA. 3 (2), 159-177 (2012).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Sugimoto, Y., et al. Analysis of CLIP and iCLIP methods for nucleotide-resolution studies of protein-RNA interactions. Genome Biology. 13 (8), (2012).
- Bayat, P., et al. SELEX methods on the road to protein targeting with nucleic acid aptamers. Biochimie. 154, 132-155 (2018).
- Armaos, A., Colantoni, A., Proietti, G., Rupert, J., Tartaglia, G. G. CatRAPID omics v2.0: Going deeper and wider in the prediction of protein-RNA interactions. Nucleic Acids Research. 49, 72-79 (2021).
- Agostini, F., et al. CatRAPID omics: A web server for large-scale prediction of protein-RNA interactions. Bioinformatics. 29 (22), 2928-2930 (2013).
- Zacco, E., et al. Probing TDP-43 condensation using an in silico designed aptamer. Nature Communications. 13 (1), 3306 (2022).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Zhang, Y., Lai, B. S., Juhas, M. Recent advances in aptamer discovery and applications. Molecules. 24 (5), 941 (2019).
- UniProt Consortium. UniProt: the Universal Protein Knowledgebase in 2023. Nucleic Acids Research. 51, 523-531 (2023).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Bellucci, M., Agostini, F., Masin, M., Tartaglia, G. G. Predicting protein associations with long noncoding RNAs. Nature Methods. 8 (6), 444-445 (2011).
- Gallo-Oller, G., Ordoñez, R., Dotor, J. A new background subtraction method for Western blot densitometry band quantification through image analysis software. Journal of Immunological Methods. 457, 1-5 (2018).
- Weissinger, R., Heinold, L., Akram, S., Jansen, R. P., Hermesh, O. RNA proximity labeling: A new detection tool for RNA-protein interactions. Molecules. 26 (8), 2270 (2021).
- Hirsch, J. D., et al. Easily reversible desthiobiotin binding to streptavidin, avidin, and other biotin-binding proteins: Uses for protein labeling, detection, and isolation. Analytical Biochemistry. 308 (2), 343-357 (2002).
- Sefah, K., Shangguan, D., Xiong, X., O'Donoghue, M. B., Tan, W. Development of DNA aptamers using cell-SELEX. Nature Protocols. 5 (6), 1169-1185 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon