Method Article
ניתוח כמותי אופטימלי של חלבונים קושרי RNA באמצעות RNA קצר ביוטינילציה
In This Article
Summary
כאן, אנו מציגים שיטה מותאמת במבחנה לחשיפה, כימות ואימות של אינטראקטורים חלבוניים של רצפי RNA ספציפיים, תוך שימוש בתמצית חלבון כוללת מתאים אנושיים, חרוזי סטרפטווידין המצופים ב-RNA ביוטינילציה וניתוח ספקטרומטריית מסות.
Abstract
אינטראקציות חלבון-רנ"א מווסתות את ביטוי הגנים ואת תפקודי התא ברמת השעתוק ואחרי השעתוק. מסיבה זו, זיהוי השותפים הקושרים של רנ"א מעניין נותר בעל חשיבות גבוהה לחשיפת המנגנונים העומדים מאחורי תהליכים תאיים רבים. עם זאת, מולקולות RNA עשויות לקיים אינטראקציה ארעית ודינמית עם חלבונים קושרי RNA מסוימים (RBPs), במיוחד עם חלבונים שאינם קנוניים. לפיכך, שיטות משופרות לבודד ולזהות RBPs כאלה נחוצים מאוד.
כדי לזהות את השותפים החלבוניים של רצף RNA ידוע בצורה יעילה וכמותית, פיתחנו שיטה המבוססת על משיכה ואפיון של כל החלבונים המקיימים אינטראקציה, החל מתמצית החלבון הכולל בתאים. ביצענו אופטימיזציה של משיכת החלבון כלפי מטה באמצעות RNA ביוטינילציה שהוטען מראש על חרוזים מצופים סטרפטווידין. כהוכחת היתכנות, השתמשנו ברצף רנ"א קצר הידוע כקושר את החלבון TDP-43 הקשור לניוון עצבי ובקרה שלילית של הרכב נוקלאוטידים שונה אך באותו אורך. לאחר חסימת החרוזים עם tRNA שמרים, העמסנו את רצפי הרנ"א הביוטינילטים על חרוזי הסטרפטאבידין ודגרנו עליהם עם תמצית החלבון הכוללת מתאי HEK 293T. לאחר הדגירה ומספר שלבי שטיפה להסרת חומרים מקשרים לא ספציפיים, דיללנו את החלבונים המקיימים אינטראקציה בתמיסה עתירת מלח, התואמת לריאגנטים הנפוצים ביותר לכימות חלבונים ולהכנת דגימות לספקטרומטריית מסות. כימתנו את ההעשרה של TDP-43 במשיכה למטה שבוצעה עם קושר הרנ"א הידוע בהשוואה לבקרה השלילית על ידי ספקטרומטריית מסות. השתמשנו באותה טכניקה כדי לאמת את האינטראקציות הסלקטיביות של חלבונים אחרים שנחזו באופן חישובי להיות קלסרים ייחודיים של הרנ"א שלנו שמעניין או של הבקרה. לבסוף, תיקפנו את הפרוטוקול על ידי Western Blot באמצעות זיהוי של TDP-43 עם נוגדן מתאים.
פרוטוקול זה יאפשר לחקור את שותפי החלבונים של RNA בעל עניין במצבים כמעט פיזיולוגיים, ויסייע לחשוף אינטראקציות חלבון-רנ"א ייחודיות ובלתי צפויות.
Introduction
חלבונים קושרי RNA (RBPs) התגלו כשחקנים מכריעים בבקרת גנים שעתוק ופוסט-שעתוק, שכן הם מעורבים בתהליכים כגון שחבור mRNA, לוקליזציה של תאי RNA, תרגום, שינוי ופירוק 1,2,3. אינטראקציות כאלה בין שתי המקרומולקולות מתואמות מאוד, מאוזנות במדויק וחיוניות ליצירת רכזות פונקציונליות ועיבוד. לווריאציות או לליקויים בתוך מרכזים אלה יש פוטנציאל לשבש את רשתות החלבון-רנ"א המווסתות דק, והן קשורות יותר ויותר למגוון מחלות אנושיות, כולל סרטן 4,5 והפרעות נוירודגנרטיביות 6,7,8. יחסי הגומלין בין מולקולות RNA לבין שותפיהן הקושרים חלבונים יכולים להיות יציבים וקלים לאימות ניסיוני, או דינמיים מאוד, חולפים וקשים יותר לאפיון.
בשנים האחרונות נעשו מאמצים אינטנסיביים להבין אינטראקציות אלה. בין השיטות המבוססות ביותר, מבחני משיכת חלבונים (PDs) הם כנראה הגישות המוערכות והנפוצות ביותר לפענוח השחקנים העיקריים המרכיבים קומפלקסים של ריבונוקלאו פרוטאין (RNP) ורשתות אינטראקציה חלבון-רנ"א אחרות 3,9,10. PDs כוללים מטריה רחבה של טכניקות אינפורמטיביות, כגון משקעים חיסוניים של RNA (RIP)11,12 או חלבון (CLIP)13,14 של עניין. חלק מפרוטוקולי RNA-PD אלה משתמשים ב-RNA ידוע כפיתיון לחלבונים15, לרוב על ידי ניצול תגי זיקה גבוהה כגון ביוטין. במקרה זה, ניתן לזהות את שותפי האינטראקציה של RNA ביוטינילציה על ידי עיגון הרנ"א על חרוזים מצופים סטרפטווידין, מה שמאפשר בידוד יעיל של ה-RNPs. המגבלות העיקריות של גישות אלה הן בדרך כלל התכנון של בדיקות biotinylated ובדיקת היכולת שלהם לקשור חלבוני מטרה. לשם כך, זה יכול להיות שימושי להסתמך על נתוני CLIP שפורסמו של החלבון המעניין, אם זמינים, שכן הם חושפים, בדיוק גבוה, את אזורי הרנ"א הקצרים המתאימים לשיאים של אינטראקציות עם חלבון המטרה13,16. אותם אזורים יכולים לשמש לפיתוח בדיקות עבור PDs. שיטה חלופית לתכנון פיתיונות רנ"א כאלה עשויה להיות אבולוציה שיטתית של ליגנדות על ידי העשרה מעריכית (SELEX)17, המאפשרות תכנון של אפטמרים באמצעות בחירה חוץ גופית, החל מספרייה אקראית מקיפה ודרך סדרה של מחזורי אופטימיזציה מונחי PCR. עם זאת, SELEX הוא מורכב וגוזל זמן, והתוצאות הסופיות תלויות מאוד בספרייה הראשונית. כדי לבחור את פיתיון הרנ"א לשימוש בפרוטוקול המוצג כאן, נוצלה גישה נוספת, המורכבת משימוש בפיתיון RNA שתוכנן דה נובו באמצעות כוח החישוב של חתולהאלגוריתם RAPID, המנבא את הקישור המועדף של חלבון נתון כלפי רצפי RNA מסוימים18,19,20.
הפרוטוקול שהוצג כאן הוא גרסה של RNA-PD המותאמת לנטרול שותפים חלבוניים ספציפיים בתנאים כמעט פיזיולוגיים, ללא שימוש בחומרי ניקוי, חומרי דנטורינג או טמפרטורות גבוהות. הוא מסתמך על חרוזים ננו-על-פאראמגנטיים המצופים באופן קוולנטי בסטרפטאבידין מטוהר מאוד ועל שימוש ב-RNA ספציפי בסיליקו המתוכנן כפיתיון. פרוטוקול זה מספק שיטה מהירה ויעילה לבידוד השותפים הקושרים של מולקולות RNA ביוטיניליות בתנאים טבעיים, ומציע פוטנציאל למגוון רחב של יישומים במורד הזרם. כדי לבחון פרוטוקול זה, נעשה שימוש ברצף אפטמר RNA חד-גדילי בן 10 נוקלאוטידים, שתוכנן בעבר לקשור את החלבון TAR DNA binding protein 43 (TDP-43) בעל זיקה וספציפיות גבוהות,20. החל מליזטים של תאי HEK 293T, זוהו האינטראקטורים של אפטמר RNA ביוטינילציה באמצעות ניתוח ספקטרומטריית מסה שבוצע על דגימות שנותקו מפיתיון הרנ"א באמצעות חיץ היפרטוני. ניתוח זה אישר את הזיהוי והכימות המוצלחים של TDP-43 כקלסר מועדף.
פרוטוקול זה מאפשר זיהוי מוצלח של אינטראקטורים חלבוניים באמצעות אוליגונוקלאוטיד RNA מסונתז במבחנה קצר בלבד. יתר על כן, השימוש בסיליקו מתוכנן RNA aptamers כמו PD בדיקות21,22 מבטיח ספציפיות עבור המטרות בעלויות מופחתות באופן משמעותי.
Protocol
1. שיטות וחומרים כלליים
- הכינו את המדיום המתאים לתרבית תאי היונקים שנבחרה וחממו אותה מראש ב-37°C למשך 20 דקות לפני השימוש.
- הכינו מראש את החומר הנדרש, כמתואר בטבלת החומרים. כלי זכוכית, כלי פלסטיק ומלאי חיץ Autoclave.
- הכן את המאגרים כמתואר בטבלה 1. התאם את ה- pH של תמיסות המלאי באמצעות HCl מרוכז או NaOH לפני דילול הרכיבים לנפחים הסופיים שלהם.
2. הכנת קו תאי יונקים
- לגדל תאי HEK 293T במדיום הנשר המעובד (DMEM) של Dulbecco בתוספת 10% נסיוב בקר עוברי (FBS) ותמיסת פניצילין/סטרפטומיצין 100 מיקרוגרם/מ"ל. לדגור אותם ב 37 ° C באינקובטור לח מסופק עם 5% CO2. לפצל את התאים באופן שגרתי.
- לפני הניתוק, שטפו את התאים בתמיסת מלח חוצצת פוספט (PBS) מספקת כדי לכסות את פני הגידול.
- הסר את PBS והוסף שכבה דקה במיוחד של תמיסת טריפסין-EDTA.
- לדגור על התאים בטמפרטורה של 37°C באינקובטור לח המסופק עם 5%CO2 למשך 5 דקות, או עד שהתאים מנותקים (הם צריכים להיראות מפוזרים תחת תצפית מיקרוסקופית).
- לדלל את תמיסת טריפסין-EDTA פי עשרה על ידי הוספת DMEM מלא כדי להשבית אותו ולספור את התאים.
- צלחת 1.5 x 105 תאים / מ"ל בלוחות 6 בארות, בהתחשב בשתי בארות / מצב לבדיקה.
- לדגור על התאים ב 37 ° C במשך 48 שעות באינקובטור לח עם 5% CO2.
הערה: בדוק את הוראות היצרן עבור סוג המדיום והתוסף המתאים לקו התא. כמו כן, כמות וזמן הדגירה של טריפסין-EDTA תלויים בקו התא. סוגי תאים מסוימים גדלים מהר יותר/לאט יותר ממה שדווח בפרוטוקול זה; לכן, ריכוז הזריעה צריך להיבדק מראש.
3. קציר חלבון כולל
- הסר מדיום מהבארות שבהן גדלים תאים.
- לשטוף כל באר של צלחות 6 באר עם 1 מ"ל של PBS.
- השליכו את ה-PBS.
- הזיזו את הצלחות על קרח והמשיכו לשלב 3.5 או הקפיאו את הצלחות היבשות בטמפרטורה של -80°C כדי להקל על הליזה.
- הוסף 200 μL של חיץ ליזיס לכל באר.
- השתמש במגרד תאים כדי לנתק ולשבור את התאים.
- מעבירים את תמצית התא הנובעת משתי בארות לאותו צינור של 1.5 מ"ל.
- הניחו את הצינורית המכילה את תמצית החלבון על קרח למשך 30 דקות.
- צנטריפוגה התא lysates ב 17,000 x גרם במשך 15 דקות ב 4 ° C.
- מעבירים כל סופרנאטנט לצינור מקורר מראש.
הערה: מומלץ לכלול 106-10 7 תאים עבור כל תנאי PD. ליזה של תאים וקציר חלבונים צריכים להתבצע עם מאגרים קרים כקרח. יש להוסיף מעכבי פרוטאז למאגר הליזה כדי למנוע פירוק חלבון.
4. קביעת ריכוז חלבון
- הכן מגיב ברדפורד, כפי שצוין על ידי המפיק, על ידי דילול אותו פי חמישה ב dH2O.
- להפיץ 1 מ"ל של מגיב בקובט 1 ס"מ, להוסיף 1 μL של המדגם, ומערבבים על ידי היפוך.
- יש לדגור בחושך בטמפרטורת החדר למשך 5-10 דקות.
- קרא את הספיגה ב 595 ננומטר.
- חשב את נפח תמצית החלבון המתאים ל -1.5 מ"ג חלבונים והביא את כל הדגימות לנפח סופי של 600 מיקרוליטר באמצעות חיץ ליזיס.
- יש לשמור את הדגימות על קרח עד לשימוש.
הערה: ניתן להשתמש בכל שיטה אחרת לקביעת ריכוז חלבון, בהתאם להמלצות תאימות חיץ. בכל מקרה, יש להשתמש במאגר הליזיס כריק. ריאגנטים רבים אינם תואמים dithiothreitol (DTT). מומלץ להוסיף DTT או חומרים מחזרים אחרים רק לאחר כימות חלבונים (DTT לריכוז סופי של 1 מילימול).
5. הכנת חרוזים
- ערבבו את החרוזים במאגר האחסון שלהם על ידי הזזת הצינור.
- חשב 100 μL של מדיום slurry / מדגם ומקם את עוצמת הקול במדף מגנטי.
- שטיפת חרוזים
- הסר את פתרון האחסון ושטוף את החרוזים על ידי הוספת 1 מ"ל של חיץ/צינור ליזיס והפוך אותו ידנית.
- הסר את המאגר באמצעות המדף המגנטי.
- חזרו על שלב הכביסה.
- הוסף נפח של חיץ ליזיס השווה לנפח ההתחלתי של מדיום הבוצה, ערבב על ידי הזזת הצינור, וחלק את התווך באופן אחיד לתוך צינורות 1.5 מ"ל ככל שיש דגימות.
- חסימת חרוזים
- הסר את המאגר באמצעות המדף המגנטי והוסף 600 μL של תמיסת 0.25 מ"ג/מ"ל של tRNA שמרים שהוכנה במאגר ליזיס.
- יש לדגור במשך שעה בטמפרטורת החדר על גלגל מסתובב.
- הסר את תמיסת ה-tRNA באמצעות המדף המגנטי.
- יש להוסיף 600 μL של חיץ ליזיס ולשטוף על ידי ערבוב ידני.
- חזרו על שלב הכביסה והשליכו את החוצץ.
6. טעינת חרוזים
- הכינו 200 מיקרוגרם של RNA oligonucleotide ב-600 μL של חיץ ליזה עבור כל צינור המכיל את התווך הראשוני של 100 μL slurry (כעת חרוזים חסומים).
- מוסיפים את האוליגו לחרוזים ודגרים במשך שעה בטמפרטורת החדר תוך כדי סיבוב.
- הסר את התמיסה, הוסף 600 μL של חיץ ליזיס, ושטוף את החרוזים פעמיים על ידי סיבוב הצינורות במשך 5 דקות בטמפרטורת החדר.
- מחק את המאגר.
הערה: לעולם אל תערבל את החרוזים, אלא החלק במקום. הגבל את מספר שלבי הצנרת, אלא אם יש צורך. במידת האפשר, השתמש לחתוך 1 מ"ל קצוות. כמות מדיום החרוזים/מדגם תלויה ביכולת הקישור של החרוזים ובכמות ההתחלתית של סך החלבונים. אם אוליגו הרנ"א צפוי להיות בעל כמות משמעותית של מבנה משני, אנו ממליצים תחילה לפרק אותו בטמפרטורה של 80 מעלות צלזיוס למשך 10 דקות ולאחר מכן לקרר אותו לאט בטמפרטורת החדר או לדגור אותו מחדש על ידי דגירה ב-30 מעלות צלזיוס למשך שעה אחת. מוצע לשחזר את אוליגו הרנ"א לאחר העמסת חרוזים ולקבוע את הריכוז שנותר, על מנת לייעל את הכמות הנדרשת להעמסה ולהעריך את האפשרות לשימוש חוזר ב-RNA.
7. קשירת חלבון על חרוזים
הערה: מעתה והלאה, במידת האפשר, בצע את השלבים ב- 4 °C.
- קח נפח של 5% מתמיסת החלבון 600 μL ושמור אותו כקלט (IN) לניתוח נוסף (1.5 מ"ג של חלבונים מומסים ב 600 μL, כך 5% מתאים 30 μL ו 75 מיקרוגרם של חלבונים).
- הוסיפו את תערובת החלבון הנותרת לכל צינור של חרוזים עמוסים והשאירו סיבוב איטי למשך הלילה בטמפרטורה של 4°C.
8. שטיפת קלסרים לא ספציפיים
- הסר את המקטע הלא מאוגד באמצעות המדף המגנטי. שמור 5% מהנפח ותייג אותו כ- FLOWTHROUGH (FT) (הנפח הלא מאוגד הוא כ- 600 μL, אז שוב שמור 30 μL לניתוח נוסף).
- הוסף 1 מ"ל של חיץ כביסה 1 לחרוזים ולהשאיר אותו מסתובב במשך 5 דקות ב 4 ° C.
- מחק את המאגר.
- חזור על שלבים 8.2 ו- 8.3.
- הוסיפו 1 מ"ל של חיץ כביסה 2 לחרוזים והשאירו אותו מסתובב במשך 5 דקות ב-4°C.
- השליכו את הסופרנטנט.
9. ביטול קלסרים ספציפיים
- הוסף 100 μL של חיץ elution 1 או חיץ elution 2 לחרוזים.
- מערבבים ידנית על ידי תנועה ודגירה במשך 5 דקות בטמפרטורת החדר.
- הכניסו את הצינורות לתרמימיקסר ונערו נמרצות במשך 5 דקות בחום של 95°C.
- הכניסו את הצינור למדף המגנטי ואספו את החלק המדולל לתוך צינור נקי.
- סובב במהירות את החרוזים עם צנטריפוגת ספסל כדי למקסם את התאוששות הפליטה.
- שמור 5% מנפח ELUATE (EL) הכולל לניתוחים נוספים (הנפח הכולל הוא 100 μL, לכן יש להפריד 5 μL לצינור אחר).
- במידת הצורך, ניתן לקבוע את ריכוז החלבון כמו בסעיף 4, על ידי שימוש בחיץ אלוציה כריק.
הערה: מומלץ להימנע מהוספת DTT למאגר האלוציה עד לקביעת ריכוז החלבון. אם אין צורך בכימות חלבון, או אם קיימות ערכות כימות חלבון תואמות חומרי חיזור, ניתן להוסיף DTT של 1 mM למאגר ה-elution מההתחלה. בפרוטוקול זה נבדקו הן חיץ אלוציה 1 (המכיל 1 M NaCl) והן חיץ אלוציה 2 (עם 2 M NaCl). לא נצפה הבדל ביעילות האלוציה של חלבון המטרה עם חוזק יוני מוגבר, אך מומלץ לבדוק את שני התנאים לפני קביעת החיץ המתאים ביותר. אם נוכחות גבוהה של מלח במאגר האלוטיון מהווה מגבלה לניתוח נוסף, הכמות הנמוכה מאוד של דטרגנטים במאגר האלוטיון מאפשרת החלפת חיץ. כחלופה, ניתן לדלל את האלואט כדי להגיע לריכוז המלח הרצוי.
10. זיהוי קושרי חלבונים על ידי ספקטרומטריית מסות
- משקעים אצטון
- רכז את החלבונים המדוללים על ידי דילול אותו פי ארבעה באצטון קר (-20 ° C).
- מערבלים ודגרים על הצינור ב -20 מעלות צלזיוס למשך הלילה.
- סחור ב-17,000 x גרם במשך 30 דקות ב-4°C.
- מוציאים בעדינות את הסופרנאטנט ומאפשרים לאצטון להתאדות עד שהגלולה מתייבשת לחלוטין.
- עיכול חלבונים בתמיסה
- להמיס את גלולת החלבון על ידי הוספת 50 μL של מאגר denaturation.
- הוסף DTT לריכוז סופי של 5 mM, המאפשר הפחתת חלבון במשך 30 דקות ב 55 ° C.
- קררו את הדגימות בטמפרטורת החדר והמשיכו בתגובת האלקילציה של החלבון, תוך הוספת יודואצטמיד (IAA) בריכוז של 10 מילימטר למשך 15 דקות.
- לעכל את החלבונים באמצעות אנזים מתאים (טריפסין, LysC) ולדגור את הדגימות במשך הלילה ב 37 ° C.
- עצור את העיכול על ידי הוספת 1 μL של 10% חומצה trifluoroacetic (TFA).
- נקו ורכזו את הפפטידים במיקרו-עמודה C18 הפוכה בהתאמה אישית, כפי שתואר קודם לכן19.
- פפטידים אלוטים מקצה C18 עם חיץ B.
- הסר את הרכיב האורגני באמצעות צנטריפוגת ואקום והשהה מחדש את הפפטידים ב 5 μL של 0.1% חומצה פורמית לניתוח נוסף.
הערה: לחלופין, ניתן לבצע עיכול חלבונים "על חרוזים" מיד לאחר שטיפת חומרים מקשרים לא ספציפיים (שלבים 8.1-8.6), ובכך להפוך את הפרוטוקול למהיר יותר. עם זאת, מומלץ לבדוק את יעילות האנזים הפועל "על חרוזים" חלבונים משותקים בהשוואה לסטנדרט בעיכול תמיסה, על מנת להבטיח כיסוי אופטימלי של רצף החלבונים הניסיוני.
- כרומטוגרפיה נוזלית-ספקטרומטריית מסה טנדם (LC-MS/MS)
- חבר את העמודה האנליטית (שלב C18-נייח) ושמור אותה על 45 ° C במהלך הריצה.
- חבר את העמוד לשקע של שסתום סיבובי בעל שש יציאות של משאבת LC באמצעות התאמה קפילרית הדוקה לאצבע (20 מיקרומטר x 550 מ"מ) בתצורת עמודה אחת.
- התאם את הגדרות LC באופן הבא:
- טען את הפפטידים בלחץ מבוקר (980 בר) בחיץ A.
- החל מעבר צבע של 5%-20% Buffer B ב- 300 nL/min במשך 59 דקות, ולאחר מכן שיפוע Buffer B של 20%-30% במשך 15 דקות ומעבר צבע של 30%-65% Buffer B במשך 5 דקות.
- הוסף שלב שטיפה על ידי הגדלת הריכוז של Buffer B עד 95% במשך 5 דקות בתוספת צעד איזוקרטי של 5 דקות ב- 95% Buffer B.
- הפעל את ספקטרומטר המסות במצב רכישה תלוית נתונים (DDA) כדי לעבור באופן אוטומטי בין אירועי MS ו- MSMS.
- הגדר ספירת לולאה שווה ל- 15 באמצעות ערך יעד אוטומטי של בקרת רווח (AGC) של 3 x 106 ו- 1 x 105 עבור אירועי MS ו- MSMS, בהתאמה.
- הגדר את זמן צבירת היונים המרבי המותר ל- 20 אלפיות השנייה עבור MS עם רזולוציה של 60 K, ו- 100 אלפיות השנייה עבור MSMS עם רזולוציה של 15 K.
- בצע ניסוי פיצול דיסוציאציה של התנגשות גבוהה (HCD) באמצעות אנרגיית התנגשות מנורמלת של 28%, עם זמן הרחקה דינמי של 20 שניות.
- הפעל את פרמטרי המקור באופן הבא:
מתח התזה: 1.7 קילו וולט
מתח נימי: 275 °C
לא נעשה שימוש בנדן או בגז עזר
הערה: בפרוטוקול זה, ניתוח ספקטרומטרי מסה (MS) של כרומטוגרפיה נוזלית בעלת ביצועים גבוהים במיוחד (UHPLC) בוצע במיוחד באמצעות מערך LC של עמודה אחת, המוצמד למכשיר אורביטרפ משולש מרובע היברידי (טבלה של חומרים). ניתן להשתמש במערכות LCMS אחרות, אך מומלץ לבצע התאמה של פרמטרים.
- ניתוח נתונים
- השתמש בלחצן טען כדי לייבא את הקבצים הגולמיים.
- הגדר את שמות הניסויים בלחיצה על הלחצן Set Experiment .
- הזן את מקטע הפרמטרים הספציפיים לקבוצה כדי לציין את כל הפרמטרים הקשורים לזיהוי:
אנזים המשמש לעיכול: טריפסין/P
מחשופים שהוחמצו: עד שלושה
שינוי קבוע: carbamidomethylation
שינוי משתנה: N-אצטיל (חלבון), חמצון (M) - העלה קובץ FASTA מעודכן, הזמין ממסדי נתונים ציבוריים כגון UniprotKB.
- ציין את כללי הניתוח הנכונים בהתאם למקור מסד הנתונים שנבחר.
- הגדר ערך שיעור גילוי שווא של הורות (FDR) = 1 הן עבור חלבונים והן עבור פפטידים.
- הוסף את האפשרות כימות ללא תוויות (LFQ) בכרטיסיה כימות ללא תווית.
- שמור על ספירת יחס LFQ מינימלית על שתיים.
הערה: כאן, אנו מתארים ניתוח נתונים באמצעות MaxQuant24 ותוכנת Perseus25 לביצוע כימות חלבונים וניתוח סטטיסטי עוקב, בהתאמה. עם זאת, ניתוח נתונים יכול להתבצע עם כל ביואינפורמטיקה אחרת זמינה מסחרית או חופשית. FDR מוערך באמצעות גישה מבוססת מסד נתונים של הטעיית מטרה26. פפטיד וחלבון FDR השווים ל-0.01 פירושם שפפטידים וחלבונים שזוהו צפויים להכיל 1% מהתוצאות החיוביות הכוזבות.
- ניתוח סטטיסטי
- טען את הקובץ .txt קבוצות החלבונים לביצוע ניתוח סטטיסטי ברמת החלבון.
- הגדר את ערכי LFQ כעמודות ראשיות.
- הסר "הפוך" ו"מזהמים" על-ידי סינון שורות בהתבסס על העמודה הקטגורית.
- השתמש בשורות הביאור הקטגוריאלי כדי לקבץ את תנאי הניסוי השונים.
- הקטן את מטריצת הנתונים, תוך בחירת מספר הערכים החוקיים בכל אחת מהקבוצות שהוגדרו קודם לכן.
- בחר את המבחן הסטטיסטי המתאים יותר לתנאי הניסוי (כלומר, מבחן t, מבחן מדגמי מרובים ANOVA).
- קבע חיתוך עבור רמזים משמעותיים באמצעות חישוב מבוסס FDR. בדרך כלל, הן 0.01 והן 0.05 מתקבלים כערכי סף עבור ערך p המתואם.
- דמיינו את התוצאות של ניתוחים דיפרנציאליים המבוססים על סטטיסטיקות של מבחן t באמצעות ייצוג חלקת הר געש.
- ייצא את המטריצה הסופית בפורמט .txt לעריכה נוספת של טבלת התוצאות הסופיות.
הערה: תיקיית התצורה מכילה קובץ FASTA עם חלבונים כגון קרטינים הנחשבים למזהמים נפוצים בניסויים פרוטאומיים גלובליים, המסומנים בסימן + בטבלת הפלט. במחקר הנוכחי, שיש שני תנאי מדגם, מבחן t משמש לניתוח סטטיסטי.
11. אימות תוצאות על ידי כתם מערבי
- הכנת דוגמאות
- הוסף את הנפח המתאים של מאגר טעינת דגימה 4x לכל aliquot של IN, FT ו- EL.
- מרתיחים את הדגימות במשך 5 דקות בטמפרטורה של 95 מעלות צלזיוס.
- סיבוב מהיר כדי לשחזר דגימה מתאדה מהחלק העליון של הצינורות.
- העברת SDS-PAGE וג'ל
- יש להעמיס דגימות על ג'ל פוליאקרילאמיד דנטורינג 4%-12%.
- הפעל את הג'ל עם מאגר הפעלה MES SDS למשך 1.5 שעות ב- 120 V.
- מעבירים את הג'ל על קרום ניטרוצלולוז עם קסטת העברה חצי יבשה, בהתאם להוראות היצרן. אנו ממליצים על העברה של 10 דקות ב- 15 V.
- זיהוי חיסוני
- חסמו את הממברנה עם אלבומין 10% בסרום בקר (BSA) למשך שעה אחת בטמפרטורת החדר תוך כדי עצבנות עדינה.
- הוסף את הנוגדן הראשוני שהוכן ב- 5% BSA ב- TBST, בהתאם להוראות היצרן. יש להשאיר למשך הלילה בטמפרטורה של 4°C צלזיוס או למשך שעה אחת בטמפרטורת החדר תחת תסיסה עדינה.
- שטפו את הממברנה שלוש פעמים עם TBST, בכל פעם במשך 5 דקות.
- מוסיפים את הנוגדן המשני שהוכן ב-TBST למשך שעה אחת בטמפרטורת החדר תחת תסיסה.
- שטפו את הממברנה שלוש פעמים עם TBST, בכל פעם במשך 5 דקות.
- הצג את התוצאות באופן חזותי באמצעות מכונת imager של כתם.
הערה: לגילוי TDP-43, קושר ידוע של הרנ"א שלנו, נעשה שימוש בנוגדן חד-שבטי רקומביננטי של ארנב והושאר עם הממברנה למשך הלילה בטמפרטורה של 4°C. כנוגדן משני, נעשה שימוש בנוגדן נגד ארנב מסוג IgG horseradish peroxidase (HRP), אך נוגדן משני פלואורסצנטי יעבוד גם כן. כדי להמחיש את הנוגדנים על הממברנה, הממברנה הודגרה עם מצע Clarity Western ECL במשך דקה אחת, לפני הדמיה עם מערכת ההדמיה ChemiDoc.
תוצאות
כדי לאמת את תוקפו של הפרוטוקול המוצע, ניסויי PD המוצגים כאן בוצעו עם אפטמר RNA ביוטינילציה שתוכנן בסיליקו כדי לקשור באופן ספציפי TDP-4320. רנ"א זה קושר את יעד החלבון שלו בזיקה גבוהה (Kd = 90 nM)20. כאן, RNA זה, מרצף 5'-CGGUGUUGCU-3', מכונה בשם "+RNA". כבקרה שלילית, נעשה שימוש ברצף המשלים ההפוך של +RNA, שנקרא כאן "-RNA". הרצף שלו הוא 5'-AGCAACACCG-3'. -RNA מראה זיקה נמוכה משמעותית כלפי TDP-43 (Kd = 1.5 μM)19. לצורך הפרוטוקול המתואר כאן, אוליגונוקלאוטידים אלה של RNA נרכשו כשהם מצומדים למולקולת ביוטין, כדי לאפשר קשירה לחרוזי הסטרפטאבידין. +RNA נרכש עם ביוטין-TEG בקצה 3' שלו, הכולל מרווח טריאתילן גליקול בן 15 אטומים בין הביוטין לקבוצת הפוספטים של חומצת הגרעין; - במקום זאת היה לרנ"א ביוטין בקצה ה-5' שלו, מצומד לחומצת הגרעין באמצעות קישור אמינו-C6. עם זאת, אם התכנון של פיתיון הרנ"א חזק, וכל עוד אין הפרעה מבנית או כימית בין המקשר לבין הרנ"א, ניתן להשתמש במיקומים אחרים עבור צימוד הביוטין ואורכי קישור אחרים.
ידיעת זהותו של החלבון העיקרי שנמצא קשור לגשושית +RNA לאחר PD אפשרה את אימות הפרוטוקול על-ידי זיהוי TDP-43 ב-eluate, הן באמצעות ספקטרומטריית מסה (MS) והן באמצעות כתם מערבי (WB) (איור 1).
ניתוח טרשת נפוצה בוצע על ארבעה שכפול PD שבוצעו באמצעות +RNA או -RNA (איור 2). זיהוי האינטראקטומים של +RNA ו-RNA הוא מעבר להיקף של פרוטוקול זה, אולם כמה תוצאות המאמתות את דיוק הפרוטוקול מדווחות. שימו לב, שרטוט החלבונים המועשרים באופן משמעותי בחלקת הר געש גילה שתכולת החלבונים הכוללת והחלבונים המועשרים שנדחקו מ-+RNA הייתה גבוהה משמעותית ממה שהופק מ-RNA (איור 2). משמעות הדבר היא שלמרות שיש לו אורך ותוכן מבני זהה (ליניארי), +RNA יכול ליצור מספר גבוה יותר של אינטראקציות ספציפיות, אשר נשמרות עד שלב האלוציה עם מלח גבוה. סביר להניח כי -RNA במקום זאת יוצר מספר גבוה יותר של מגעים לא ספציפיים אשר משובשים במהלך שלבי הכביסה. כצפוי, TDP-43 זוהה כאינטראקטור ייחודי של +RNA20; הכימות הממוצע ללא תוויות (LFQ) עבור ארבעת העתקי PD המבוצעים עם +RNA הוא 31.96 ± 0.56, בעוד שהחלבון אינו מזוהה בין האינטראקטורים של -RNA. בנוסף, מבין כל האינטראקטורים הייחודיים של +RNA, TDP-43 נמצא כחלבון המועשר ביותר בשפע.
כדי לאמת עוד יותר את הפרוטוקול, האלגוריתם הפנימי RAPID18,19 שימש לחיזוי חישובי אילו חלבונים אחרים יקשרו באופן ספציפי +RNA או -RNA. בפרט, ציוני אינטראקציה עבור +RNA ו-RNA עם החלבונים המרכיבים את הפרוטאום האנושי חושבו באמצעות תכונת 'נטיית האינטראקציה' של catRAPID, כפי שהוגדרה בעבודתנו הקודמת27. מבין החלבונים שקיבלו ציון ביטחון גבוה, HNRNPH3 נחזה להיקשר באופן סלקטיבי +RNA (+RNA interaction score = 1.01; -RNA interaction score = 0.21) ו-PCBP2 כדי לקיים אינטראקציה ספציפית עם -RNA (+RNA interaction score = -0.5; -RNA interaction score = 0.31) (איור 3A). נוסף על כך, החלבון RBM41 נחזה להיות מופקר עבור שני אוליגונוקלאוטידים של RNA (+ציון אינטראקציה של RNA = 0.4; -ציון אינטראקציה של RNA = 0.39) (איור 3A). ניתוח הטרשת הנפוצה אכן אישר את נוכחותם של HNRNPH3 ו-PCBP2 ב-PD של +RNA ו-RNA בהתאמה, בעוד ש-RBM41 נמצא באינטראקציה עם שניהם (איור 3B).
WB שימש כדי לזהות נוכחות של TDP-43 כדי לאשר עוד יותר את התוצאות ובמהלך אופטימיזציה של פרוטוקול (איור 4). בהליך המתואר כאן נאספו דגימות שונות בשלבים שונים. דגימת הקלט (IN) כללה את סך החלבונים המדוללים במאגר ליזיס. הזרימה (FT) התקבלה לאחר דגירה של לילה של סך החלבונים עם חרוזי סטרפטאבידין מצופים מראש ב-RNA ביוטינילציה, המייצג את חלק החלבונים שלא קשרו את הרנ"א. לבסוף, האלואט (EL) הכיל את כל החלבונים שזיהו באופן ספציפי את הרנ"א הנחקר, שכן בין שלבי FT ו-EL שלושה שלבי שטיפה עם מלח 150 מילימטר ו-0.1% טריטון-X היו אמורים להסיר את האינטראקציות החלשות ביותר.
עבור כל שכפול, אותה כמות (5% v/v) של IN, FT ו-EL רצה במקביל על SDS-PAGE והוכתמה בנוגדן אנטי-TDP-43 (איור 4). במקרה של +RNA, הרצועה של TDP-43 נצפתה ב-IN וב-EL, מה שמצביע על כך שהחלבון, שנמצא מלכתחילה בתמצית החלבון הכוללת, נשמר על ידי +RNA במהלך שלבי השטיפה והוא מדולל רק בסוף עם חיץ מלח גבוה. TDP-43 היה קיים גם ב-IN עבור -RNA, אולם הרצועה המתאימה לחלבון נראית גם ב-FT, מה שמצביע על כך ש-RNA זה אינו נקשר ל-TDP-43. היעדר רצועת TDP-43 ב- EL מאשר תוצאה זו.
במהלך האופטימיזציה של הפרוטוקול, נבדקה האלוציציה של החלבונים הקשורים באופן ספציפי לרצפי הרנ"א הן באמצעות חיץ אלוציה המכיל 1 M NaCl (EB1) והן באמצעות חיץ אלוציה המכיל 2 M NaCl (EB2) (איור 4). אלוטים שהתקבלו עם EB הושוו על SDS-PAGE ונמחקו עם נוגדן anti-TDP-43. התמונות שהתקבלו נותחו לאחר מכן עם ImageJ28 כדי לכמת כל הבדל בכמות TDP-43 המדוללת עם שני המאגרים. בסך הכל, לא נצפה הבדל משמעותי, והסקנו כי בתוך בדיקות אלה, מלח 1M מספיק כדי לשבש אפילו את אינטראקציות חלבון-RNA החזקות ביותר.
בסך הכל, התוצאות המדווחות כאן עבור MS ו- WBs מראות כי פרוטוקול זה יעיל בלכידת אינטראקטורים חלבוניים של RNA נתון באופן מסוים, וכי הוא מאפשר את האלוציה במאגרים התואמים לאנליזה במורד הזרם.
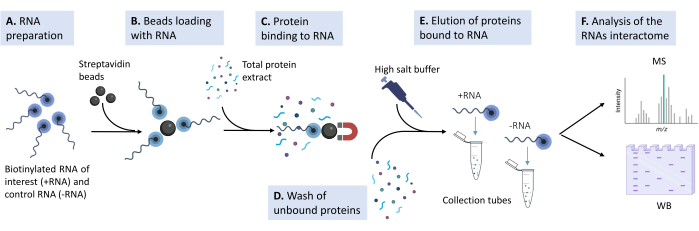
איור 1: שרטוט של צינור הניסוי ששימש בפרוטוקול המוצע . (A) אוליגונוקלאוטיד RNA ביוטינילציה מוכן בחיץ ליזה בריכוז המתאים. (B) חרוזי סטרפטווידין מגנטיים נשטפים, נחסמים ב-tRNA שמרים ועמוסים ב-RNA ביוטינילציה. (C) תמצית חלבון כוללת שמקורה בקווי תאים של יונקים בתרבית מתווספת לתערובת החרוזים-רנ"א. (D) שטיפות מרובות מבוצעות כדי להסיר אינטראקציות לא ספציפיות. (E) האינטראקטורים החלבוניים הספציפיים מנותקים מהרנ"א בתמיסה היפרטונית. (F) זהות האינטראקטורים נחשפת על ידי ספקטרומטריית מסות, ומקרים ספציפיים מאומתים על ידי כתם מערבי. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
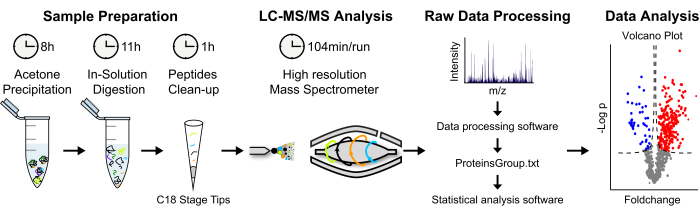
איור 2: אסטרטגיה אנליטית לכימות חלבונים ללא תוויות המבוססות על טרשת נפוצה . (A) חלבונים שעברו פליטה מואצים באצטון קר למשך הלילה. חלבונים עוברים דנטורציה, ומתבצע עיכול בתמיסה. פפטידים מפרקי חלבון מרוכזים ומתפלים. (B) פפטידים מנותחים באמצעות LC-MS/MS באמצעות "גישת רובה ציד". (C) עיבוד וניתוח נתונים גולמיים מתבצע באמצעות תוכנות MaxQuant ו-Perseus, בהתאמה. (D) חלבונים מועשרים מובהקים סטטיסטית מוצגים בחלקת הר געש. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
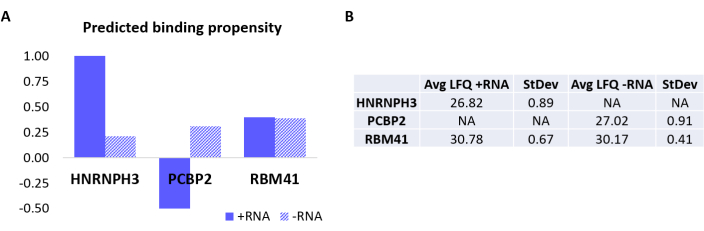
איור 3: מתאם בין נטיות אינטראקציה חזויות לבין אינטראקציות שנקבעו בניסוי של +RNA ו-RNA. (A) ציוני אינטראקציה מהירה של חתולים ביחס ל-HNRNPH3, PCBP2 ו-RBM41, המצביעים על קשירה מועדפת שלHNRNPH3 עבור +RNA ושל PCBP2 עבור -RNA, בעוד ש-RBM41 צפוי לקשור ללא הבחנה את שני רצפי ה-RNA. (B) ממוצעי כימות ללא תוויות שנקבעו על ידי ניתוח ספקטרומטריית מסות מהמשיכות שבוצעו עם +RNA ו-RNA. הניתוח מאשר כי HNRNPH3 קושר אך ורק +RNA, PCBP2 קושר אך ורק -RNA, ו- RBM41 קושר את שניהם באופן שווה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
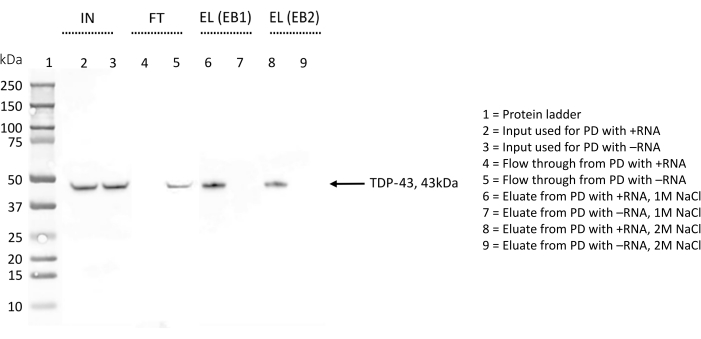
איור 4: אימות כתם מערבי של נוכחות/היעדר TDP-43 בקרב האינטראקטורים של רצפי רנ"א נבחרים. קרום WB טופל בנוגדן נגד TDP-43. IN = קלט; FT = זרימה; EL (EB1) = אלוציה עם חיץ אלוציה 1; EL (EB2) = אלוציה עם חיץ אלוציה 2; הסימן "+" מציין דגימות הנגזרות מהרשימה הנפתחת שבוצעה עם +RNA; הסימן "-" מציין דגימות הנגזרות מהמשיכה למטה שבוצעה עם -RNA; נתיב 1 מכיל סולם חלבונים. TDP-43 מסומן בחץ. ה-WB מציין כי TDP-43 נמצא בקרב +RNA interactors אך לא בקרב -RNA interactors. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
| שם מאגר | הרכב | |||||
| 10x מאגר Tranfer | 250 מ"מ תריס, 1.92 מ' גליצין, 1% SDS, 20% מתנול. לדלל 10 קיפולים לפני השימוש | משטרת | ||||
| מאגר פועל 20X MES SDS | 1 M MES, 1 M tris, 2% SDS, 20 mM EDTA. התאם את ה- pH ל- 7.3. לדלל 20 קיפולים לפני השימוש | |||||
| 4x מאגר טעינה לדוגמה | 0.25 M בסיס תריס, 0.28 M SDS, 40% גליצרול, 20% 2-מרקפטו-אתנול, 4 מ"ג/מ"ל ברומפנול כחול | |||||
| חיץ Elution 1 | 20 mM פוספט pH 7.5, 1 M NaCl, 0.5 mM EDTA, 0.1% Triton X-100, 1 mM DTT (יתווסף לאחר כימות) | |||||
| חיץ Elution 2 | 20 mM פוספט pH 7.5, 2 M NaCl, 0.5 mM EDTA, 0.1% Triton X-100, 1 mM DTT (יתווסף לאחר כימות) | |||||
| חיץ ליזיס | 10 mM Tris-HCl pH 7.4, 150 mM NaCl, 0.5 mM EDTA, 0.1% Triton X-100, 1 mM DTT ומעכבי פרוטאז | |||||
| מלח חוצץ טריס עם Tween-20 | 1 M Tris-HCl pH 7.4, 3 M NaCl, 2.0% Tween-20 | |||||
| חיץ כביסה 1 | 10 mM Tris-HCl pH 7.4, 150 mM NaCl, 0.5 mM EDTA, 0.1% Triton TM X-100, 1 mM DTT ומעכבי פרוטאז | |||||
| חיץ כביסה 2 | 25 mM Hepes pH 8, 150 mM NaCl, 0.5 mM EDTA, 0.1% Triton X-100, 1 mM DTT ומעכבי פרוטאז | |||||
| חיץ א' | 0.1% חומצה פורמית | מ.ס. | ||||
| חיץ B | 60% אצטוניטריל, 0.1% חומצה פורמית | |||||
| מאגר דנטורציה | 8M אוריאה, 50 mM Tris-HCl | |||||
טבלה 1: מאגרי PD ו- MS. שמות והרכב המאגרים המשמשים לניסויי משיכה כלפי מטה (PD) או לניתוח ספקטרומטריית מסות (MS).
Discussion
עבודה זו מדווחת על אופטימיזציה של פרוטוקול PD המבוצע עם אוליגונוקלאוטידים של RNA ביוטינילציה כדי ללכוד את האינטראקטורים החלבוניים שלהם. הפרוטוקול המתואר כאן פשוט לביצוע, דורש מעט חומר ומפיק תוצאות אמינות ביותר. חשוב לציין, ההיבטים החדשניים ביותר של פרוטוקול זה כוללים את השימוש בפיתיון RNA המתוכנן במלואו בסיליקו וספציפי למטרת החלבון, ופליטת כל החלבונים הקשורים לפיתיון הרנ"א על ידי שיבוש ישיר של האינטראקציות שלהם עם תמיסת מלח גבוהה, במקום על ידי ניתוק הסטרפטאבידין מהביוטין עם חומר ניקוי וטיפול בטמפרטורה גבוהה.
פרוטוקול זה מנצל את חוזק הקשר בין ביוטין לסטרפטאווידין29,30. על פי חרוזי הסטרפטאבידין שנבחרו, יש לבדוק ולכמת את טעינת הרנ"א הביוטינילציה לפני שתמשיך. כמו כן, הקיפול התלת-ממדי של הרנ"א עשוי להשפיע על יעילות ההעמסה של החרוזים, שכן הוא עשוי להגביל את חשיפת הביוטין לסטרפטווידין. חסימת החרוזים עם tRNA לא ביוטינילציה משפרת את ניקיון התוצאות על ידי הגבלת אינטראקציות לא ספציפיות עם החרוזים. יש לבחור את מאגר ההעמסה ואת מאגר האלוציה בהתאם ליישומים במורד הזרם. כאן הוצעו תנאים קלים מאוד, המתאימים לרוב היישומים ופותחו כדי לשמר קומפלקסים חלבוניים פוטנציאליים. עם זאת, שיטה זו ניתנת להתאמה גבוהה; המשתמש יכול לבחור כל קו תא וכל גודל RNA, ויכול להחליט לחזור על הפרוטוקול לאחר קיפול/פתיחת הרנ"א כדי לקבוע את השפעת המבנה על תכונות הקשירה.
היבט מקורי נוסף של פרוטוקול זה הוא השימוש בכלי חיזוי סיליקו כדי להבטיח את נכונות התוצאות20. הידיעה מראש אילו חלבונים צריכים להיות מזוהים כאינטראקטורים של הרנ"א המעניין נותנת את היתרון חסר התקדים של אימות ההיבטים הטכניים של הפרוטוקול. לדוגמה, באמצעות ניתוח WB פשוט, ניתן לאמת את נוכחותו של יעד חלבון ידוע בדגימות הנגזרות מהשלבים השונים של הפרוטוקול לפני שתמשיך בניתוח טרשת נפוצה, הדורש מכשור מיוחד ויקר יותר. בנוסף, לאחרונה דווח על שיטה לשימוש ב-cat RAPID20, אלגוריתם חיזוי חלבון-רנ"א פנימי, כדי לתכנן RNA דה נובו ספציפי לחלבון מטרה. עד לאחרונה, הצינור הזמין היחיד לתכנון APTAMERS של DNA/RNA עבור חלבון מטרה היה גישת SELEX (אבולוציה שיטתית של ליגנדים על ידי העשרה מעריכית)31. שיטת in silico מאפשרת תכנון הרבה יותר מהיר וחסכוני של APTAMERS RNA.
המגבלות העיקריות של שיטה זו קשורות לצורך לעבוד במאגרים וכלים ללא נוקלאז. יתר על כן, אם יש צורך לאשר במבחנה את הקישור בין RNA מתוכנן דה נובו לבין חלבון מטרה לפני PD, החלבון צריך להיות מיוצר ומטוהר והקישור נקבע בגישות ביופיזיקליות. זוהי מגבלה המשותפת לייצור נוגדנים חד שבטיים.
למרות בעיות שוליות אלה, שיטות אמינות למיפוי אינטראקציות RNA-חלבון, כמו זו המוצגת כאן, יכולות לקרב מדענים לחשיפת רשתות מקרומולקולריות ושחקנים ראשיים מורכבים של מנגנונים פיזיולוגיים ופתולוגיים רבים, כגון אלה המעורבים בסרטן, קרדיומיופתיה, סוכרת, זיהומים מיקרוביאליים והפרעות גנטיות וניווניות.
Disclosures
למחברים אין אינטרסים כלכליים מתחרים או ניגודי עניינים אחרים.
Acknowledgements
המחברים רוצים להודות לקבוצת המחקר של פרופ' טרטליה וד"ר קואומו על התמיכה המוצעת. א.ז. קיבלה מימון ממלגת MINDED של תוכנית המחקר והחדשנות Horizon 2020 של האיחוד האירופי במסגרת הסכם המענק מס' 754490 ע"ש מארי סקלודובסקה-קירי.
Materials
| Name | Company | Catalog Number | Comments |
| 6-well tissue culture plates | VWR | 10861-554 | CELLS |
| Cell scrapers | BIOSIGMA | 10153 | CELLS |
| Dulbecco′s Modified Eagle′s Medium (DMEM) | Thermo Fisher Scientfic | 11995065 | CELLS |
| Fetal Bovine Serum, qualified, heat inactivated, Brazil | Thermo Fisher Scientfic | 10500064 | CELLS |
| Phosphate Buffer Saline (PBS, Waltham, MA) | Thermo Fisher Scientfic | 14190169 | CELLS |
| Trypsin (0.25%), phenol red | Thermo Fisher Scientfic | 15050065 | CELLS |
| Anti-rabbit IgG horseradish peroxidase (HRP) | Cellsignal | 7070 | PD |
| Biotinylated RNA | Eurofins | Custom RNA oligonucleotides | PD |
| Bovine serum albumin | Sigma-Aldrich | A9418 | PD |
| Clarity Western ECL Substrate, 500 ml | Biorad | 1705061 | PD |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Merck - Sigma Aldrich | 5056489001 | PD |
| NuPAGE 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel, 10-well | Invitrogen | NP0321BOX | PD |
| Recombinant anti-TDP43 antibody | Abcam | ab109535 | PD |
| Ribonucleic acid, transfer from baker's yeast (S. cerevisiae) | Merck - Sigma Aldrich | R5636-1ML | PD |
| Streptavidin Mag Sepharose | Merck - Sigma Aldrich | GE28-9857-99 | PD |
| Trans-Blot Turbo RTA Mini 0.2 µm PVDF Transfer Kit | Biorad | 1704272 | PD |
| Acetone | Thermo Fisher Scientfic | 022928.K2 | MS |
| C18 cartridge | Thermo Fisher Scientfic | 13-110-018 | MS |
| Dithiothreitol (DTT) | Thermo Fisher Scientfic | 20290 | MS |
| EASY-Spray HPLC Columns | Thermo Scientific | ES902 | MS |
| iodoacetamide (IAA) | Sigma Aldrich S.r.l. | I6125 | MS |
| Lys-C/Trypsin | Promega | V5073 | MS |
| Trifluoroacetic acid (TFA) | Thermo Fisher Scientfic | 28904 | MS |
| Urea | Thermo Fisher Scientfic | J75826.A7 | MS |
| Equipment | |||
| ChemiDoc imaging system | Bio-Rad | CELLS | |
| Dyna Mag -2 , Magnetic rack | Invitrogen | CELLS | |
| Forma Series 3 water jacketed C02 incubator | Thermo Scientific | PD | |
| PROTEAN II xi cell , power supply for PAGE applications | Bio-Rad | PD | |
| Rotating wheel, rotator SB3 | Stuart | PD | |
| Water bath set at 37 °C | VWR | PD | |
| XCell SureLock Mini-Cell electrophoresis system | ThermoFisher Scientific | MS | |
| Easy-nLC 1200 UHPLC | Thermo Scientific | MS | |
| Q exactive Mass Spectrometer | Thermo Scientific | MS | |
| Software | Version | ||
| MaxQuant | 2.0.3.0 | MS | |
| Perseus | 1.6.14.0 | MS |
References
- Gebauer, F., Schwarzl, T., Valcárcel, J., Hentze, M. W. RNA-binding proteins in human genetic disease. Nature Reviews Genetics. 22 (3), 185-198 (2021).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Cooper, T. A., Wan, L., Dreyfuss, G. RNA and disease. Cell. 136 (4), 777-793 (2009).
- Qin, H., et al. RNA-binding proteins in tumor progression. Journal of Hematology & Oncology. 13 (1), 90 (2020).
- Nussbacher, J. K., Tabet, R., Yeo, G. W., Lagier-Tourenne, C. Disruption of RNA metabolism in neurological diseases and emerging therapeutic interventions. Neuron. 102 (2), 294-320 (2019).
- Duan, R., Sharma, S., Xia, Q., Garber, K., Jin, P. Towards understanding RNA-mediated neurological disorders. Journal of Genetics and Genomics. 41 (9), 473-484 (2014).
- Maziuk, B., Ballance, H. I., Wolozin, B. Dysregulation of RNA binding protein aggregation in neurodegenerative disorders. Frontiers in Molecular Neuroscience. 10, 89 (2017).
- Zielinski, J., et al. In vivo identification of ribonucleoprotein-RNA interactions. Proceedings of the National Academy of Sciences. 103 (5), 1557-1562 (2006).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Weidmann, C. A., Mustoe, A. M., Jariwala, P. B., Calabrese, J. M., Weeks, K. M. Analysis of RNA-protein networks with RNP-MaP defines functional hubs on RNA. Nature Biotechnology. 39 (3), 347-356 (2021).
- Graindorge, A., et al. In-cell identification and measurement of RNA-protein interactions. Nature Communications. 10 (1), 5317 (2019).
- Ule, J., Hwang, H. W., Darnell, R. B. The future of cross-linking and immunoprecipitation (CLIP). Cold Spring Harbor Perspectives in Biology. 10 (8), 032243 (2018).
- Ascano, M., Hafner, M., Cekan, P., Gerstberger, S., Tuschl, T. Identification of RNA-protein interaction networks using PAR-CLIP. Wiley Interdisciplinary Reviews. RNA. 3 (2), 159-177 (2012).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Sugimoto, Y., et al. Analysis of CLIP and iCLIP methods for nucleotide-resolution studies of protein-RNA interactions. Genome Biology. 13 (8), (2012).
- Bayat, P., et al. SELEX methods on the road to protein targeting with nucleic acid aptamers. Biochimie. 154, 132-155 (2018).
- Armaos, A., Colantoni, A., Proietti, G., Rupert, J., Tartaglia, G. G. CatRAPID omics v2.0: Going deeper and wider in the prediction of protein-RNA interactions. Nucleic Acids Research. 49, 72-79 (2021).
- Agostini, F., et al. CatRAPID omics: A web server for large-scale prediction of protein-RNA interactions. Bioinformatics. 29 (22), 2928-2930 (2013).
- Zacco, E., et al. Probing TDP-43 condensation using an in silico designed aptamer. Nature Communications. 13 (1), 3306 (2022).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Zhang, Y., Lai, B. S., Juhas, M. Recent advances in aptamer discovery and applications. Molecules. 24 (5), 941 (2019).
- UniProt Consortium. UniProt: the Universal Protein Knowledgebase in 2023. Nucleic Acids Research. 51, 523-531 (2023).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Bellucci, M., Agostini, F., Masin, M., Tartaglia, G. G. Predicting protein associations with long noncoding RNAs. Nature Methods. 8 (6), 444-445 (2011).
- Gallo-Oller, G., Ordoñez, R., Dotor, J. A new background subtraction method for Western blot densitometry band quantification through image analysis software. Journal of Immunological Methods. 457, 1-5 (2018).
- Weissinger, R., Heinold, L., Akram, S., Jansen, R. P., Hermesh, O. RNA proximity labeling: A new detection tool for RNA-protein interactions. Molecules. 26 (8), 2270 (2021).
- Hirsch, J. D., et al. Easily reversible desthiobiotin binding to streptavidin, avidin, and other biotin-binding proteins: Uses for protein labeling, detection, and isolation. Analytical Biochemistry. 308 (2), 343-357 (2002).
- Sefah, K., Shangguan, D., Xiong, X., O'Donoghue, M. B., Tan, W. Development of DNA aptamers using cell-SELEX. Nature Protocols. 5 (6), 1169-1185 (2010).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved