Method Article
Uma Análise Pull-Down Quantitativa Otimizada de Proteínas Ligadoras de RNA Usando RNA Biotinilado Curto
Neste Artigo
Resumo
Aqui, apresentamos um método in vitro otimizado para descobrir, quantificar e validar interatores proteicos de sequências específicas de RNA, usando extrato proteico total de células humanas, esferas de estreptavidina revestidas com RNA biotinilado e análise por espectrometria de massas.
Resumo
As interações proteína-RNA regulam a expressão gênica e as funções celulares em níveis transcricionais e pós-transcricionais. Por esta razão, identificar os parceiros de ligação de um RNA de interesse permanece de grande importância para desvendar os mecanismos por trás de muitos processos celulares. No entanto, moléculas de RNA podem interagir transitória e dinamicamente com algumas proteínas ligadoras de RNA (RBPs), especialmente com outras não-canônicas. Portanto, métodos aprimorados para isolar e identificar tais RBPs são extremamente necessários.
Para identificar os parceiros proteicos de uma sequência de RNA conhecida de forma eficiente e quantitativa, desenvolvemos um método baseado no pull-down e caracterização de todas as proteínas interagentes, a partir do extrato proteico total celular. Otimizamos o pull-down da proteína usando RNA biotinilado pré-carregado em esferas revestidas com estreptavidina. Como prova de conceito, empregamos uma sequência curta de RNA conhecida por se ligar à proteína TDP-43 associada à neurodegeneração e um controle negativo de uma composição de nucleotídeos diferente, mas com o mesmo comprimento. Depois de bloquear as esferas com tRNA de levedura, carregamos as sequências de RNA biotinilado nas esferas de estreptavidina e as incubamos com o extrato proteico total das células HEK 293T. Após incubação e várias etapas de lavagem para remoção de ligantes inespecíficos, eluímos as proteínas interativas com uma solução com alto teor de sal, compatível com os reagentes de quantificação de proteínas mais comumente usados e com a preparação da amostra para espectrometria de massas. Nós quantificamos o enriquecimento de TDP-43 no pull-down realizado com o ligante de RNA conhecido comparado ao controle negativo por espectrometria de massas. Usamos a mesma técnica para verificar as interações seletivas de outras proteínas previstas computacionalmente como ligantes únicas de nosso RNA de interesse ou do controle. Finalmente, validamos o protocolo por western blot através da detecção de TDP-43 com um anticorpo apropriado.
Este protocolo permitirá o estudo dos parceiros proteicos de um RNA de interesse em condições quase fisiológicas, ajudando a descobrir interacções proteína-RNA únicas e imprevisíveis.
Introdução
As proteínas ligadoras de RNA (RBPs) têm emergido como atores cruciais na regulação gênica transcricional e pós-transcricional, uma vez que estão envolvidas em processos como splicing de RNAm, localização, tradução, modificação e degradação celular de RNA1,2,3. Tais interações entre as duas macromoléculas são altamente coordenadas, precisamente balanceadas e essenciais para a formação de hubs funcionais e de processamento. Variações ou desregulações dentro desses centros têm o potencial de interromper as redes proteína-RNA finamente reguladas e estão cada vez mais associadas a uma variedade de doenças humanas, incluindo câncer 4,5 e doenças neurodegenerativas 6,7,8. As interações entre moléculas de RNA e seus parceiros de ligação a proteínas podem ser estáveis e fáceis de validar experimentalmente, ou altamente dinâmicas, transitórias e mais difíceis de caracterizar.
Nos últimos anos, esforços intensivos têm sido empreendidos para compreender essas interações. Dentre os métodos mais estabelecidos, os ensaios pull-down de proteínas (DPs) são provavelmente as abordagens mais apreciadas e comumente utilizadas para desvendar os principais atores constituintes dos complexos ribonucleoprotéicos (RNP) e outras redes de interação proteína-RNA3,9,10. As DPs incluem um amplo escopo de técnicas informativas, como a imunoprecipitação do RNA (RIP)11,12 ou da proteína (CLIP)13,14 de interesse. Alguns desses protocolos de RNA-PD empregam um RNA conhecido como isca para proteínas15, mais frequentemente aproveitando etiquetas de alta afinidade, como a biotina. Neste caso, os parceiros de interação de um RNA biotinilado podem ser detectados pela ancoragem do RNA em esferas revestidas com estreptavidina, permitindo o isolamento eficiente das RNPs. As principais limitações dessas abordagens são geralmente o planejamento das sondas biotiniladas e o teste de sua capacidade de se ligar a proteínas-alvo. Para isso, poderia ser útil contar com dados publicados do CLIP da proteína de interesse, se disponíveis, uma vez que eles revelam, com alta precisão, as regiões curtas do RNA que correspondem a picos de interações com a proteína-alvo13,16. Essas mesmas regiões poderiam ser usadas para desenvolver sondas para PDs. Um método alternativo para projetar tais iscas de RNA pode ser a evolução sistemática de ligantes por enriquecimento exponencial (SELEX)17, que possibilitam o planejamento de aptâmeros através de seleção in vitro, a partir de uma biblioteca randomizada abrangente e através de uma série de ciclos de otimização orientados por PCR. No entanto, o SELEX é complexo e demorado, e os resultados finais são altamente dependentes da biblioteca inicial. Para selecionar a isca de RNA a ser utilizada no protocolo aqui apresentado, outra abordagem foi explorada, consistindo na utilização de uma isca de RNA projetada de novo por meio do poder computacional do algoritmo catRAPID, que prevê a ligação preferencial de uma dada proteína a determinadas sequências deRNA18,19,20.
O protocolo aqui apresentado é uma versão de um RNA-PD otimizado para eluir parceiros proteicos específicos em condições quase fisiológicas, sem o uso de detergente, agentes desnaturantes ou altas temperaturas. Ele se baseia em esferas nano-superparamagnéticas revestidas covalentemente com estreptavidina altamente purificada e o uso de um RNA biotinilado específico in silico projetado como isca. Este protocolo fornece um método rápido e eficiente para isolar os parceiros de ligação de moléculas de RNA biotinilado em condições nativas, oferecendo o potencial para uma ampla gama de aplicações a jusante. Para testar esse protocolo, foi utilizada uma sequência de aptâmeros de RNA de fita simples de 10 nucleotídeos, previamente projetada para se ligar à proteína TAR DNA-binding protein 43 (TDP-43) com alta afinidade e especificidade20. A partir de lisados celulares HEK 293T, os interatores do aptâmero de RNA biotinilado foram identificados por meio de análise por espectrometria de massa realizada em amostras destacadas da isca de RNA usando um tampão hipertônico. Esta análise confirmou o sucesso na identificação e quantificação do TDP-43 como ligante preferencial.
Este protocolo permite a identificação bem sucedida de interatores proteicos usando apenas um oligonucleotídeo de RNA sintetizado in vitro e curto. Além disso, o uso de aptâmeros de RNA projetados in silico como sondas de PD21,22 garante especificidade para os alvos a custos significativamente reduzidos.
Protocolo
1. Métodos gerais e material
- Preparar o meio adequado para a cultura de células de mamíferos escolhida e pré-aquecê-lo a 37 °C durante 20 minutos antes da utilização.
- Prepare o material necessário com antecedência, conforme descrito na Tabela de Materiais. Autoclave de vidro, plasticware e estoques tampão.
- Prepare os buffers conforme descrito na Tabela 1. Ajustar o pH das soluções-mãe utilizando HCl ou NaOH concentrado antes de diluir os componentes até aos seus volumes finais.
2. Preparação de linhagens celulares de mamíferos
- Cultivar células HEK 293T em meio Dulbecco's Modified Eagle Medium (DMEM) suplementado com 10% de soro fetal bovino (FBS) e 100 μg/mL de solução de penicilina/estreptomicina. Incubá-los a 37 °C em estufa umidificada abastecida com 5% de CO2. Rotineiramente dividir as células.
- Antes de se desprender, lave as células com solução salina tamponada com fosfato (PBS) suficiente para cobrir a superfície de crescimento.
- Remova o PBS e adicione uma camada ultrafina de solução de tripsina-EDTA.
- Incubar as células a 37 °C em uma incubadora umidificada provida de CO2 a 5% por 5 min, ou até que as células estejam destacadas (elas devem parecer dispersas sob observação microscópica).
- Diluir a solução de tripsina-EDTA dez vezes adicionando DMEM completo para inativá-la e contar as células.
- Placa 1,5 x 105 células/mL em placas de 6 poços, considerando dois poços/condição a testar.
- Incubar as células a 37 °C durante 48 h numa estufa humidificada com 5% de CO2.
NOTA: Verifique as instruções do fabricante para o tipo de meio e suplemento apto para a linha celular. Além disso, a quantidade e o tempo de incubação da tripsina-EDTA dependem da linhagem celular. Alguns tipos celulares crescem mais rápido/mais lentamente do que o relatado neste protocolo; assim, a concentração de semeadura deve ser testada previamente.
3. Colheita total de proteínas
- Remova o meio dos poços onde as células estão crescendo.
- Lave cada poço das placas de 6 poços com 1 mL de PBS.
- Descarte o PBS.
- Mova as placas no gelo e prossiga com o passo 3.5 ou congele as placas secas a -80 °C para facilitar a lise.
- Adicionar 200 μL de tampão de lise a cada poço.
- Use um raspador de células para desprender e quebrar as células.
- Transfira o extrato celular derivado de dois poços para o mesmo tubo de 1,5 mL.
- Coloque o tubo contendo o extrato proteico no gelo por 30 min.
- Centrifugar os lisados celulares a 17.000 x g por 15 min a 4 °C.
- Transfira cada sobrenadante para um tubo pré-resfriado.
NOTA: Um total de 106-10 7 células para cada condição de DP são recomendados. A lise celular e a colheita de proteínas devem ser realizadas com tampões gelados. Inibidores de protease devem ser adicionados ao tampão de lise para evitar a degradação de proteínas.
4. Determinação da concentração de proteínas
- Preparar o reagente de Bradford, conforme indicado pelo produtor, diluindo-o cinco vezes em dH2O.
- Distribuir 1 mL de reagente em uma cubeta de 1 cm, adicionar 1 μL da amostra e misturar por inversão.
- Incubar no escuro à temperatura ambiente durante 5-10 min.
- Leia a absorbância a 595 nm.
- Calcular o volume de extracto proteico correspondente a 1,5 mg de proteínas e levar todas as amostras a um volume final de 600 μL utilizando tampão de lise.
- Mantenha as amostras no gelo até o uso.
NOTA: Qualquer outro método de determinação da concentração de proteínas pode ser usado, seguindo as recomendações de compatibilidade de buffer. Em qualquer caso, o tampão de lise deve ser usado como um branco. Muitos reagentes não são compatíveis com ditiotreitol (TDT). Recomenda-se adicionar TDT ou outros agentes redutores somente após a quantificação da proteína (TDT para uma concentração final de 1 mM).
5. Preparação de contas
- Misture as contas em seu buffer de armazenamento mexendo o tubo.
- Calcular 100 μL de meio de lama/amostra e colocar o volume num rack magnético.
- Lavagem de contas
- Retire a solução de armazenamento e lave as esferas adicionando 1 mL de tampão de lise/tubo e inverta-o manualmente.
- Remova o buffer usando o rack magnético.
- Repita a etapa de lavagem.
- Adicione um volume de tampão de lise igual ao volume inicial do meio de lama, misture agitando o tubo e distribua o meio uniformemente em tantos tubos de 1,5 mL quantos forem as amostras.
- Bloqueio de contas
- Remova o tampão usando o rack magnético e adicione 600 μL de uma solução de 0,25 mg/mL de tRNA de levedura preparada em tampão de lise.
- Incubar durante 1 h à temperatura ambiente numa roda giratória.
- Remova a solução de tRNA usando o rack magnético.
- Adicionar 600 μL de tampão de lise e lavar misturando manualmente.
- Repita a etapa de lavagem e descarte o tampão.
6. Carregamento do talão
- Preparar 200 μg de oligonucleotídeo de ARN em 600 μL de tampão de lise para cada tubo contendo o meio de lama inicial de 100 μL (agora contas bloqueadas).
- Adicione o oligo às contas e incube por 1 h à temperatura ambiente durante a rotação.
- Retire a solução, adicione 600 μL de tampão de lise e lave as contas duas vezes girando os tubos durante 5 minutos à temperatura ambiente.
- Descarte o buffer.
NOTA: Nunca vomite as contas, mas mexa em vez disso. Limite o número de etapas de pipetagem, a menos que necessário. Quando possível, use pontas cortadas de 1 mL. A quantidade de meio de lama/amostra depende da capacidade de ligação das esferas e da quantidade inicial das proteínas totais. Se se prevê que o RNA oligo tenha uma quantidade significativa de estrutura secundária, recomendamos primeiro desnaturá-lo a 80 °C por 10 min e, em seguida, resfriá-lo lentamente à temperatura ambiente ou refold ele incubando-o a 30 °C por 1 h. Sugere-se recuperar o RNA oligo após o carregamento do talão e determinar a concentração restante, a fim de otimizar a quantidade necessária para o carregamento e avaliar a possibilidade de reutilização do RNA.
7. Ligação às proteínas nas esferas
NOTA: A partir de agora, quando possível, execute as etapas a 4 °C.
- Tome um volume de 5% da solução proteica de 600 μL e mantenha-o como INPUT (IN) para análise posterior (1,5 mg de proteínas são dissolvidos em 600 μL, de modo que 5% corresponde a 30 μL e 75 μg de proteínas).
- Adicione a mistura de proteínas restante a cada tubo de contas carregadas e deixe girar lentamente durante a noite a 4 °C.
8. Lavagem de aglutinantes inespecíficos
- Remova a fração não acoplada usando o rack magnético. Economize 5% do volume e rotule-o como FLOWTHROUGH (FT) (o volume não ligado é de cerca de 600 μL, portanto, mantenha novamente 30 μL para análise posterior).
- Adicionar 1 ml de tampão de lavagem 1 às contas e deixá-lo a girar durante 5 min a 4 °C.
- Descarte o buffer.
- Repita as etapas 8.2 e 8.3.
- Adicionar 1 ml de tampão de lavagem 2 às contas e deixá-lo a rodar durante 5 minutos a 4 °C.
- Descarte o sobrenadante.
9. Eluição de ligantes específicos
- Adicionar 100 μL de tampão de eluição 1 ou tampão de eluição 2 às contas.
- Misture manualmente agitando e incube por 5 min à temperatura ambiente.
- Coloque os tubos numa misturadeira e agite vigorosamente durante 5 min a 95 °C.
- Coloque o tubo no rack magnético e colete a fração eluída em um tubo limpo.
- Gire rapidamente as contas com uma centrífuga de bancada para maximizar a recuperação do eluato.
- Economize 5% do volume total de ELUATE (EL) para análises adicionais (o volume total é de 100 μL, então separe 5 μL em outro tubo).
- Se necessário, a concentração de proteínas pode ser determinada como na secção 4, utilizando tampão de eluição como um espaço em branco.
NOTA: Recomenda-se abster-se de adicionar TDT ao tampão de eluição até que a concentração de proteínas tenha sido determinada. Se a quantificação de proteínas não for necessária, ou se estiverem disponíveis kits de quantificação de proteínas compatíveis com agentes redutores, 1 mM de TDT pode ser adicionado ao tampão de eluição desde o início. Nesse protocolo, tanto o tampão de eluição 1 (contendo NaCl 1M) quanto o tampão de eluição 2 (com NaCl 2M) foram testados. Nenhuma diferença na eficiência de eluição da proteína-alvo foi observada com o aumento da força iônica, mas recomenda-se testar ambas as condições antes de estabelecer o tampão mais adequado. Se uma alta presença de sal no tampão de eluição representa uma limitação para uma análise posterior, a quantidade muito baixa de detergentes no tampão de eluição permite a troca de tampão. Como alternativa, o eluato pode ser diluído para atingir a concentração de sal desejada.
10. Identificação de ligantes de proteínas por espectrometria de massas
- Precipitação de acetona
- Concentrar as proteínas eluídas diluindo-as quatro vezes em acetona fria (-20 °C).
- Vórtice e incube o tubo a -20 °C durante a noite.
- Gire a 17.000 x g por 30 min a 4 °C.
- Retire suavemente o sobrenadante e deixe a acetona evaporar até que o pellet tenha secado completamente.
- Digestão de proteínas em solução
- Dissolver o pellet de proteína adicionando 50 μL de tampão de desnaturação.
- Adicionar TDT a uma concentração final de 5 mM, permitindo a redução da proteína durante 30 min a 55 °C.
- Resfriar as amostras à temperatura ambiente e prosseguir com a reação de alquilação proteica, adicionando iodoacetamida (IAA) a uma concentração de 10 mM por 15 min.
- Digerir as proteínas utilizando uma enzima adequada (tripsina, LysC) e incubar as amostras durante a noite a 37 °C.
- Pare a digestão adicionando 1 μL de ácido trifluoroacético (TFA) a 10%.
- Limpar e concentrar os peptídeos em uma microcoluna C18 de fase reversa personalizada, conforme descrito anteriormente19.
- Eluir peptídeos da ponta C18 com Buffer B.
- Remova o componente orgânico usando uma centrífuga a vácuo e ressuspenda os peptídeos em 5 μL de ácido fórmico a 0,1% para análise posterior.
NOTA: Alternativamente, a digestão de proteínas pode ser realizada "em contas" imediatamente após a lavagem de ligantes inespecíficos (passos 8.1-8.6), tornando o protocolo mais rápido. No entanto, é aconselhável testar a eficiência da enzima trabalhando "on-beads" proteínas imobilizadas em comparação com o padrão na digestão em solução, a fim de garantir a cobertura ótima da sequência experimental de proteínas.
- Cromatografia líquida-espectrometria de massas em tandem (LC-MS/MS)
- Ligar a coluna analítica (fase C18-estacionária) e mantê-la a 45 °C durante o tempo de corrida.
- Conecte a coluna à saída de uma válvula rotativa de seis portas da bomba LC através de um encaixe capilar estanque (20 μm x 550 mm) em uma configuração de coluna única.
- Ajuste as configurações de LC da seguinte maneira:
- Carregar os peptídeos sob pressão controlada (980 bar) no tampão A.
- Aplique um gradiente de Buffer B de 5%-20% a 300 nL/min ao longo de 59 min, seguido por um gradiente de Buffer B de 20%-30% ao longo de 15 min e um gradiente de Buffer B de 30%-65% ao longo de 5 min.
- Adicione uma etapa de lavagem aumentando a concentração de Buffer B até 95% ao longo de 5 min mais uma etapa isocrática de 5 min a 95% Buffer B.
- Opere o espectrômetro de massa no modo de aquisição dependente de dados (DDA) para alternar automaticamente entre eventos MS e MSMS.
- Defina uma contagem de loop igual a 15 usando um valor de destino de controle de ganho automático (AGC) de 3 x 106 e 1 x 105 para os eventos MS e MSMS, respectivamente.
- Defina o tempo máximo permitido de acumulação de íons para 20 ms para MS com resolução de 60 K e 100 ms para MSMS com resolução de 15 K.
- Realizar um experimento de fragmentação de alta dissociação de colisão (HCD) usando uma energia de colisão normalizada de 28%, com um tempo de exclusão dinâmica de 20 s.
- Opere os parâmetros de origem da seguinte maneira:
Tensão de pulverização: 1,7 kV
Tensão capilar: 275 °C
Nem bainha nem gás auxiliar utilizados
NOTA: Neste protocolo, a análise por cromatografia líquida de ultra-alta eficiência (UHPLC) por espectrometria de massas (MS) foi especificamente realizada usando uma configuração de coluna única LC, acoplada a um instrumento orbitrap híbrido triplo quadrupolo (Tabela de Materiais). Outros sistemas LCMS podem ser usados, mas uma adaptação de parâmetros é recomendada.
- Análise de dados
- Use o botão Carregar para importar os arquivos brutos.
- Defina os nomes dos experimentos clicando no botão Definir experimento .
- Insira a seção de parâmetros específicos do grupo para especificar todos os parâmetros relacionados à identificação:
Enzima usada para digestão: Tripsina/P
Clivagens perdidas: até três
Modificação fixa: Carbamidometilação
Modificação variável: N-acetil (Proteína), Oxidação (M) - Carregue um arquivo FASTA atualizado, disponível em bancos de dados públicos, como UniprotKB.
- Especifique as regras de análise corretas de acordo com a origem do banco de dados escolhido.
- Defina um valor de taxa de falsa descoberta (FDR) = 1 para proteínas e peptídeos.
- Adicione a opção de quantificação sem rótulo (LFQ) na guia Quantificação sem rótulo.
- Mantenha a contagem mínima da razão LFQ em dois.
NOTA: Descrevemos aqui a análise dos dados utilizando os softwares MaxQuant24 e Perseus25 para realizar a quantificação de proteínas e posterior análise estatística, respectivamente. No entanto, a análise dos dados pode ser realizada com qualquer outra bioinformática disponível comercialmente ou livre. O FDR é estimado usando uma abordagem baseada em banco de dados de chamarizde destino 26. Peptídeo e proteína FDR igual a 0,01 significa que peptídeos e proteínas identificados devem conter 1% de falsos positivos.
- Análise estatística
- Carregue o arquivo .txt grupos de proteínas para realizar a análise estatística no nível de proteína.
- Defina os valores LFQ como colunas principais.
- Remova "reverso" e "contaminantes" filtrando linhas com base na coluna categórica.
- Use as linhas de anotação categórica para agrupar as diferentes condições experimentais.
- Reduza a matriz de dados, selecionando o número de valores válidos em cada um dos grupos definidos anteriormente.
- Selecione o teste estatístico que melhor se adapta às condições experimentais (ou seja, teste t, teste de amostras múltiplas ANOVA).
- Determine um ponto de corte para dicas significativas com um cálculo baseado em FDR. Normalmente, tanto 0,01 quanto 0,05 são aceitos como limiares para o valor de p ajustado.
- Visualize os resultados de análises diferenciais baseadas em estatísticas de teste t usando uma representação de gráfico de vulcão.
- Exporte a matriz final em .txt formato para posterior edição da tabela de resultados finais.
Observação : A pasta de configuração contém um arquivo FASTA com proteínas como queratinas que são consideradas contaminantes comuns em experimentos proteômicos globais, que são sinalizados com um + na tabela de saída. No presente estudo, com duas condições amostrais, é utilizado o teste t para a análise estatística.
11. Validação dos resultados por western blot
- Preparo da amostra
- Adicione o volume apropriado de buffer de carregamento de amostra 4x a cada alíquota de IN, FT e EL.
- Ferva as amostras por 5 min a 95°C.
- Rotação rápida para recuperar a amostra evaporada do topo dos tubos.
- SDS-PAGE e transferência de gel
- Carregar amostras em um gel de poliacrilamida desnaturante 4%-12%.
- Execute o gel com o buffer de funcionamento MES SDS por 1,5 h a 120 V.
- Transfira o gel em uma membrana de nitrocelulose com um de transferência semi-seco, seguindo as instruções do fabricante. Recomendamos uma transferência de 10 minutos a 15 V.
- Imunodetecção
- Bloquear a membrana com albumina de soro bovino (BSA) a 10% durante 1 h à temperatura ambiente, agitando suavemente.
- Adicionar o anticorpo primário preparado em BSA a 5% em TBST, de acordo com as instruções do fabricante. Deixar durante a noite a 4 °C ou durante 1 h à temperatura ambiente sob agitação suave.
- Lave a membrana três vezes com TBST, cada vez por 5 min.
- Adicionar o anticorpo secundário preparado em TBST durante 1 h à temperatura ambiente sob agitação.
- Lave a membrana três vezes com TBST, cada vez por 5 min.
- Visualize os resultados usando um blot imager.
NOTA: Para detectar TDP-43, um ligante conhecido de nosso RNA, o anticorpo monoclonal de coelho recombinante foi usado e deixado com a membrana durante a noite a 4 °C. Como anticorpo secundário, foi utilizada a anti-peroxidase de raiz forte IgG de coelho (HRP), mas um anticorpo secundário fluorescente também funcionaria. Para visualizar os anticorpos na membrana, a membrana foi incubada com o substrato Clarity Western ECL por 1 min, antes da obtenção de imagens com o sistema de imagem ChemiDoc.
Resultados
Para verificar a validade do protocolo proposto, os experimentos de DP aqui apresentados foram realizados com um aptâmero de RNA biotinilado projetado in silico para ligar especificamente o TDP-4320. Esse RNA liga-se ao seu alvo proteico com alta afinidade de ligação (Kd = 90 nM)20. Aqui, este RNA, da sequência 5'-CGGUGUUGCU-3', é referido com o nome "+RNA". Como controle negativo, foi utilizada a sequência complementar reversa de +RNA, aqui denominada "-RNA". Sua sequência é 5'-AGCAACACCG-3'. -RNA mostra uma afinidade de ligação significativamente menor ao TDP-43 (Kd = 1,5 μM)19. Para o propósito do protocolo aqui descrito, esses oligonucleotídeos de RNA foram comprados conjugados a uma molécula de biotina, para permitir a ligação às esferas de estreptavidina. O +RNA foi comprado com um TEG de biotina em sua extremidade 3', que inclui um espaçador de trietilenoglicol de 15 átomos entre a biotina e o grupo fosfato do ácido nucleico; -RNA, em vez disso, tinha uma biotina em sua extremidade 5', conjugada ao ácido nucleico através de um ligante amino-C6. No entanto, se o projeto da isca de RNA for robusto, e desde que não haja interferência estrutural ou química entre o ligante e o RNA, outras posições para a conjugação de biotina e outros comprimentos de ligante poderiam ser empregadas.
O conhecimento da identidade da principal proteína a ser encontrada ligada à sonda de +RNA após a DP possibilitou a validação do protocolo pela identificação de TDP-43 no eluato, utilizando espectrometria de massas (MS) e western blot (WB) (Figura 1).
A análise de MS foi realizada em quatro réplicas de PD realizadas usando +RNA ou -RNA (Figura 2). A identificação dos interatomizadores de +RNA e -RNA está fora do escopo deste protocolo, porém alguns resultados que validam a acurácia do protocolo são relatados. Digno de nota, plotar as proteínas significativamente enriquecidas em um gráfico de vulcão revelou que o conteúdo de proteína total e as proteínas enriquecidas eluídas de +RNA foi significativamente maior do que o que foi recuperado de -RNA (Figura 2). Isso significa que, apesar de ter o mesmo comprimento e conteúdo estrutural (linear), o +RNA pode estabelecer um número maior de interações específicas, que são retidas até a etapa de eluição com sal alto. É provável que o -RNA, em vez disso, estabeleça um número maior de contatos inespecíficos que são interrompidos durante as etapas de lavagem. Como esperado, o TDP-43 foi identificado como um interator único do +RNA20; a quantificação label-free média (LFQ) para as quatro réplicas de PD realizadas com +RNA é de 31,96 ± 0,56, enquanto a proteína não é identificada entre os interatores do -RNA. Além disso, entre todos os interatores únicos de +RNA, TDP-43 foi encontrado para ser a proteína mais abundantemente enriquecida.
Para validar ainda mais o protocolo, o algoritmo interno catRAPID18,19 foi usado para prever computacionalmente quais outras proteínas se ligariam especificamente a +RNA ou -RNA. Em particular, os escores de interação para +RNA e -RNA com as proteínas que compõem o proteoma humano foram calculados usando a característica catRAPID 'interaction propensity', conforme definido em nosso trabalho anterior27. Entre as proteínas pontuadas com alta confiança, HNRNPH3 foi previsto para ligar seletivamente +RNA (+escore de interação RNA = 1,01; -RNA interaction score = 0,21) e PCBP2 para interagir especificamente com -RNA (+RNA interaction score = -0,5; -RNA interaction score = 0,31) (Figura 3A). Além disso, a proteína RBM41 foi prevista como promíscua para ambos os oligonucleotídeos de RNA (+escore de interação de RNA = 0,4; escore de interação -RNA = 0,39) (Figura 3A). A análise de MS confirmou a presença de HNRNPH3 e PCBP2 na PD de +RNA e -RNA, respectivamente, enquanto RBM41 foi encontrado interagindo com ambos (Figura 3B).
O WB foi utilizado para detectar a presença de TDP-43 para confirmar ainda mais os resultados e durante a otimização do protocolo (Figura 4). No procedimento aqui descrito, diferentes amostras foram coletadas em diferentes estágios. A amostra de entrada (IN) consistiu das proteínas totais diluídas em tampão de lise. O flowthrough (FT) foi obtido após uma incubação noturna das proteínas totais com as esferas de estreptavidina pré-revestidas com o RNA biotinilado, representando a fração de proteínas que não se ligaram ao RNA. Finalmente, o eluato (EL) continha todas as proteínas que reconheciam especificamente o RNA sob investigação, uma vez que entre as etapas FT e EL três etapas de lavagem com sal 150 mM e triton-X 0,1% deveriam ter removido as interações mais fracas.
Para cada repetição, a mesma quantidade (5% v/v) de IN, FT e EL foi executada em paralelo em um SDS-PAGE e corada com um anticorpo anti-TDP-43 (Figura 4). No caso do +RNA, a banda de TDP-43 foi observada no IN e no EL, indicando que a proteína, presente desde o início no extrato proteico total, é retida pelo +RNA durante as etapas de lavagem e só é eluída ao final com um tampão salino alto. O TDP-43 também esteve presente na IN para -RNA, porém a banda correspondente à proteína também é visível na FT, indicando que este RNA não se liga ao TDP-43. A ausência da banda TDP-43 na EL confirma esse resultado.
Durante a otimização do protocolo, a eluição das proteínas ligadas especificamente às sequências de RNA foi sondada tanto com um tampão de eluição contendo 1 M NaCl (EB1) quanto com um tampão de eluição completo com 2 M NaCl (EB2) (Figura 4). Os eluato obtidos com ambos os EB foram comparados em SDS-PAGE e borrados com o anticorpo anti-TDP-43. As imagens obtidas foram então analisadas com o ImageJ28 para quantificar qualquer diferença na quantidade de TDP-43 eluída com os dois tampões. No geral, nenhuma diferença significativa foi observada, e concluímos que, dentro desses ensaios, o sal de 1 M é suficiente para interromper até mesmo as interações proteína-RNA mais fortes.
De modo geral, os resultados aqui relatados para MS e WBs demonstram que este protocolo é eficiente em capturar os interatores proteicos de um dado RNA de maneira específica, e que possibilita a eluição em tampões compatíveis com a análise a jusante.
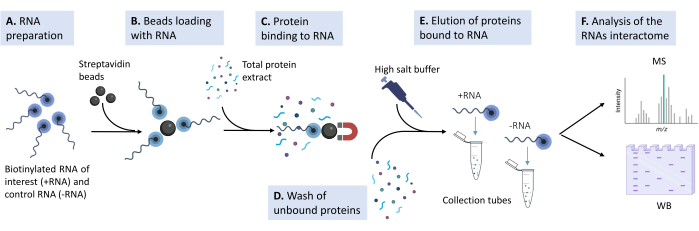
Figura 1: Croqui do pipeline experimental utilizado no protocolo proposto . (A) O oligonucleotídeo de RNA biotinilado é preparado em tampão de lise na concentração apropriada. (B) As esferas magnéticas de estreptavidina são lavadas, bloqueadas com tRNA de levedura e carregadas com o RNA biotinilado. (C) Extrato proteico total derivado de linhagens celulares de mamíferos cultivadas é adicionado à mistura de contas e RNA. (D) Múltiplas lavagens são realizadas para remover interações inespecíficas. (E) Os interatores proteicos específicos são destacados do RNA com uma solução hipertônica. (F) A identidade dos interatores é revelada por espectrometria de massas, e casos específicos são validados por western blot. Clique aqui para ver uma versão maior desta figura.
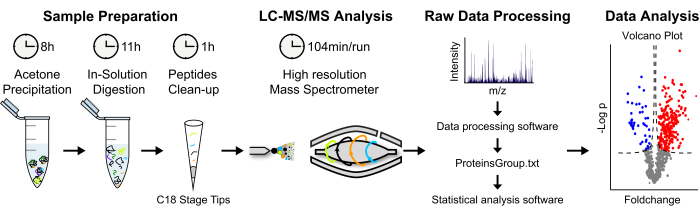
Figura 2: Estratégia analítica para quantificação de proteínas à base de MS sem rótulo . (A) As proteínas eluídas são precipitadas em acetona fria durante a noite. As proteínas são então desnaturadas e uma digestão em solução é realizada. Os peptídeos proteolíticos são concentrados e dessalinizados. (B) Os peptídeos são analisados via LC-MS/MS usando uma "abordagem shotgun". (C) O processamento e a análise dos dados brutos são realizados utilizando os softwares MaxQuant e Perseus, respectivamente. (D) Proteínas enriquecidas estatisticamente significativas são exibidas em um gráfico de vulcão. Clique aqui para ver uma versão maior desta figura.
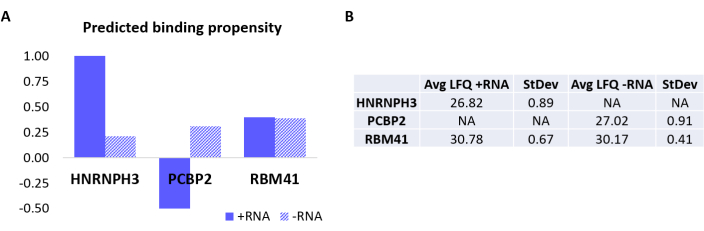
Figura 3: Correlação entre propensões de interação previstas e interações determinadas experimentalmente de +RNA e -RNA. (A) escores de interação RAPID do gatoem relação a HNRNPH3, PCBP2 e RBM41, indicando ligação preferencial de HNRNPH3 para +RNA e de PCBP2 para -RNA, enquanto RBM41 é previsto para ligar indiscriminadamente ambas as sequências de RNA. (B) Médias de quantificação label-free determinadas por espectrometria de massa a partir dos pull-downs realizados com +RNA e -RNA. A análise confirma que HNRNPH3 se liga unicamente a +RNA, PCBP2 liga somente -RNA, e RBM41 se liga a ambos igualmente. Clique aqui para ver uma versão maior desta figura.
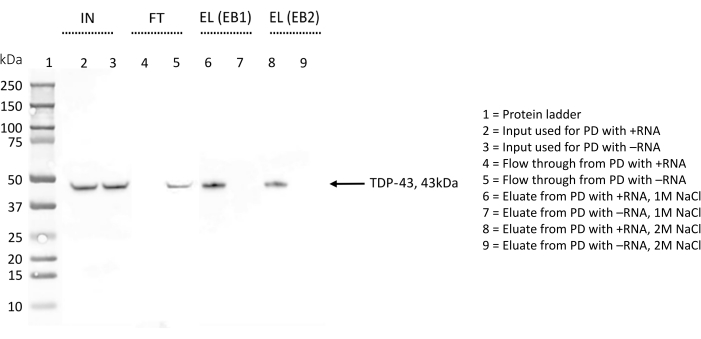
Figura 4: Validação por Western blot da presença/ausência de TDP-43 entre os interatores das sequências de RNA escolhidas. A membrana WB foi tratada com o anticorpo anti-TDP-43. IN = entrada; FT = fluxo-through; EL (EB1) = eluição com tampão de eluição 1; EL (EB2) = eluição com tampão de eluição 2; o sinal "+" indica amostras derivadas do pull-down realizado com +RNA; o sinal "-" indica amostras derivadas do pull-down realizado com -RNA; A faixa 1 contém uma escada de proteínas. O TDP-43 é indicado por uma seta. O WB indica que o TDP-43 é encontrado entre os interatores de +RNA, mas não entre os interatores de -RNA. Clique aqui para ver uma versão maior desta figura.
| Nome do buffer | Composição | |||||
| 10x buffer de transferência | Tris 250 mM, glicina 1,92 M, SDS 1%, metanol 20%. Diluir 10 vezes antes do uso | PD | ||||
| 20X MES SDS buffer de execução | 1 M MES, 1 M tris, 2% SDS, 20 mM EDTA. Ajuste o pH para 7,3. Diluir 20 vezes antes da utilização | |||||
| 4x Buffer de carregamento de amostra | 0,25 M Tris base, 0,28 M SDS, 40% glicerol, 20% 2-mercapto-etanol, 4 mg/ml azul de bromfenol | |||||
| Tampão de eluição 1 | 20 mM fosfato pH 7,5, 1 M NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM TDT (a adicionar após quantificação) | |||||
| Buffer de eluição 2 | 20 mM fosfato pH 7,5, 2 M NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM TDT (a adicionar após quantificação) | |||||
| Tampão de lise | Tris-HCl 10 mM pH 7,4, NaCl 150 mM, EDTA 0,5 mM, Triton X-100 a 0,1 %, TDT 1 mM e inibidores de protease | |||||
| Solução salina tamponada com Tween-20 | 1 M Tris-HCl pH 7,4, 3 M NaCl, 2,0% Tween-20 | |||||
| Tampão de lavagem 1 | Tris-HCl 10 mM pH 7,4, NaCl 150 mM, EDTA 0,5 mM, Triton TM X-100 a 0,1 %, TDT 1 mM e inibidores de protease | |||||
| Tampão de lavagem 2 | 25 mM Hepes pH 8, 150 mM NaCl, 0,5 mM EDTA, 0,1 % Triton X-100, 1 mM TDT e inibidores de protease | |||||
| Buffer A | 0,1% de ácido fórmico | MS | ||||
| Amortecedor B | 60% acetonitrila, 0,1% ácido fórmico | |||||
| Tampão de desnaturação | 8M de ureia, 50 mM de Tris-HCl | |||||
Tabela 1: Buffers PD e MS. Nomes e composição dos tampões utilizados para as experiências pull-down (PD) ou para a análise por espectrometria de massas (MS).
Discussão
Este trabalho relata a otimização de um protocolo de DP realizado com oligonucleotídeos de RNA biotinilado para capturar seus interatores proteicos. O protocolo aqui descrito é simples de executar, requer pouco material e produz resultados altamente confiáveis. É importante ressaltar que os aspectos mais novos deste protocolo consistem no uso de uma isca de RNA projetada totalmente in silico e específica para o alvo proteico, e na eluição de todas as proteínas ligadas à isca de RNA, interrompendo diretamente suas interações com uma solução de alto teor de sal, em vez de dissociar a estreptavidina da biotina com detergente e tratamento de alta temperatura.
Esse protocolo aproveita a força da ligação entre biotina e estreptavidina29,30. De acordo com as esferas de estreptavidina escolhidas, o carregamento do RNA biotinilado deve ser testado e quantificado antes de prosseguir. Além disso, o dobramento tridimensional do RNA pode afetar a eficiência de carregamento nas contas, uma vez que pode limitar a exposição da biotina à estreptavidina. O bloqueio das esferas com RNAt não biotinilado melhora a limpeza dos resultados, limitando as interações inespecíficas com as contas. O buffer de carregamento e o buffer de eluição devem ser escolhidos dependendo das aplicações a jusante. Aqui, condições muito amenas, adequadas à maioria das aplicações e desenvolvidas para preservar potenciais complexos proteicos, foram propostas. Este método é, no entanto, altamente adaptável; o usuário pode escolher qualquer linha celular e qualquer tamanho de RNA, e pode decidir repetir o protocolo após o dobramento/desdobramento do RNA para determinar o efeito da estrutura nas propriedades de ligação.
Outro aspecto original desse protocolo é o uso de ferramentas de predição in silico para garantir a correção dos resultados20. Saber antecipadamente quais proteínas devem ser identificadas como interatores do RNA de interesse dá a vantagem inédita de validar os aspectos técnicos do protocolo. Por exemplo, usando uma análise WB simples, é possível verificar a presença de um alvo proteico conhecido nas amostras derivadas das diferentes etapas do protocolo antes de prosseguir com a análise de EM, que requer instrumentação especializada e é mais dispendiosa. Além disso, um método para usar o gatoRAPID20, um algoritmo interno de predição de proteína-RNA, para projetar de novo RNA específico para uma proteína-alvo foi recentemente relatado. Até recentemente, o único pipeline disponível para projetar aptâmeros de DNA/RNA para uma proteína-alvo era a abordagem SELEX (systematic evolution of ligands by exponencial enrichment)31. O método in silico permite um projeto muito mais rápido e econômico de aptâmeros de RNA.
As principais limitações deste método estão associadas à necessidade de trabalhar em buffers e ferramentas livres de nucleases. Além disso, se for considerado necessário confirmar in vitro a ligação entre um RNA projetado de novo e uma proteína-alvo prévia à DP, a proteína precisa ser produzida e purificada e a ligação determinada com abordagens biofísicas. Essa é uma limitação compartilhada com a produção de anticorpos monoclonais.
Apesar dessas pequenas questões, métodos confiáveis para mapear interações RNA-proteína, como o aqui apresentado, podem aproximar os cientistas para desvendar redes macromoleculares e atores principais complexos de muitos mecanismos fisiológicos e patológicos, como os envolvidos em câncer, cardiomiopatias, diabetes, infecções microbianas e doenças genéticas e neurodegenerativas.
Divulgações
Os autores não têm interesses financeiros concorrentes ou outros conflitos de interesse.
Agradecimentos
Os autores gostariam de agradecer ao grupo de pesquisa do Prof. Tartaglia e do Dr. Cuomo pelo apoio oferecido. E.Z. recebeu financiamento da bolsa MINDED do programa de pesquisa e inovação Horizonte 2020 da União Europeia sob o acordo de subvenção Marie Sklodowska-Curie nº 754490.
Materiais
| Name | Company | Catalog Number | Comments |
| 6-well tissue culture plates | VWR | 10861-554 | CELLS |
| Cell scrapers | BIOSIGMA | 10153 | CELLS |
| Dulbecco′s Modified Eagle′s Medium (DMEM) | Thermo Fisher Scientfic | 11995065 | CELLS |
| Fetal Bovine Serum, qualified, heat inactivated, Brazil | Thermo Fisher Scientfic | 10500064 | CELLS |
| Phosphate Buffer Saline (PBS, Waltham, MA) | Thermo Fisher Scientfic | 14190169 | CELLS |
| Trypsin (0.25%), phenol red | Thermo Fisher Scientfic | 15050065 | CELLS |
| Anti-rabbit IgG horseradish peroxidase (HRP) | Cellsignal | 7070 | PD |
| Biotinylated RNA | Eurofins | Custom RNA oligonucleotides | PD |
| Bovine serum albumin | Sigma-Aldrich | A9418 | PD |
| Clarity Western ECL Substrate, 500 ml | Biorad | 1705061 | PD |
| cOmplete, EDTA-free Protease Inhibitor Cocktail | Merck - Sigma Aldrich | 5056489001 | PD |
| NuPAGE 4 to 12%, Bis-Tris, 1.0 mm, Mini Protein Gel, 10-well | Invitrogen | NP0321BOX | PD |
| Recombinant anti-TDP43 antibody | Abcam | ab109535 | PD |
| Ribonucleic acid, transfer from baker's yeast (S. cerevisiae) | Merck - Sigma Aldrich | R5636-1ML | PD |
| Streptavidin Mag Sepharose | Merck - Sigma Aldrich | GE28-9857-99 | PD |
| Trans-Blot Turbo RTA Mini 0.2 µm PVDF Transfer Kit | Biorad | 1704272 | PD |
| Acetone | Thermo Fisher Scientfic | 022928.K2 | MS |
| C18 cartridge | Thermo Fisher Scientfic | 13-110-018 | MS |
| Dithiothreitol (DTT) | Thermo Fisher Scientfic | 20290 | MS |
| EASY-Spray HPLC Columns | Thermo Scientific | ES902 | MS |
| iodoacetamide (IAA) | Sigma Aldrich S.r.l. | I6125 | MS |
| Lys-C/Trypsin | Promega | V5073 | MS |
| Trifluoroacetic acid (TFA) | Thermo Fisher Scientfic | 28904 | MS |
| Urea | Thermo Fisher Scientfic | J75826.A7 | MS |
| Equipment | |||
| ChemiDoc imaging system | Bio-Rad | CELLS | |
| Dyna Mag -2 , Magnetic rack | Invitrogen | CELLS | |
| Forma Series 3 water jacketed C02 incubator | Thermo Scientific | PD | |
| PROTEAN II xi cell , power supply for PAGE applications | Bio-Rad | PD | |
| Rotating wheel, rotator SB3 | Stuart | PD | |
| Water bath set at 37 °C | VWR | PD | |
| XCell SureLock Mini-Cell electrophoresis system | ThermoFisher Scientific | MS | |
| Easy-nLC 1200 UHPLC | Thermo Scientific | MS | |
| Q exactive Mass Spectrometer | Thermo Scientific | MS | |
| Software | Version | ||
| MaxQuant | 2.0.3.0 | MS | |
| Perseus | 1.6.14.0 | MS |
Referências
- Gebauer, F., Schwarzl, T., Valcárcel, J., Hentze, M. W. RNA-binding proteins in human genetic disease. Nature Reviews Genetics. 22 (3), 185-198 (2021).
- Hentze, M. W., Castello, A., Schwarzl, T., Preiss, T. A brave new world of RNA-binding proteins. Nature Reviews Molecular Cell Biology. 19 (5), 327-341 (2018).
- Castello, A., et al. Insights into RNA biology from an atlas of mammalian mRNA-binding proteins. Cell. 149 (6), 1393-1406 (2012).
- Cooper, T. A., Wan, L., Dreyfuss, G. RNA and disease. Cell. 136 (4), 777-793 (2009).
- Qin, H., et al. RNA-binding proteins in tumor progression. Journal of Hematology & Oncology. 13 (1), 90 (2020).
- Nussbacher, J. K., Tabet, R., Yeo, G. W., Lagier-Tourenne, C. Disruption of RNA metabolism in neurological diseases and emerging therapeutic interventions. Neuron. 102 (2), 294-320 (2019).
- Duan, R., Sharma, S., Xia, Q., Garber, K., Jin, P. Towards understanding RNA-mediated neurological disorders. Journal of Genetics and Genomics. 41 (9), 473-484 (2014).
- Maziuk, B., Ballance, H. I., Wolozin, B. Dysregulation of RNA binding protein aggregation in neurodegenerative disorders. Frontiers in Molecular Neuroscience. 10, 89 (2017).
- Zielinski, J., et al. In vivo identification of ribonucleoprotein-RNA interactions. Proceedings of the National Academy of Sciences. 103 (5), 1557-1562 (2006).
- Armaos, A., Zacco, E., Sanchez de Groot, N., Tartaglia, G. G. RNA-protein interactions: Central players in coordination of regulatory networks. BioEssays. 43 (2), 2000118 (2021).
- Weidmann, C. A., Mustoe, A. M., Jariwala, P. B., Calabrese, J. M., Weeks, K. M. Analysis of RNA-protein networks with RNP-MaP defines functional hubs on RNA. Nature Biotechnology. 39 (3), 347-356 (2021).
- Graindorge, A., et al. In-cell identification and measurement of RNA-protein interactions. Nature Communications. 10 (1), 5317 (2019).
- Ule, J., Hwang, H. W., Darnell, R. B. The future of cross-linking and immunoprecipitation (CLIP). Cold Spring Harbor Perspectives in Biology. 10 (8), 032243 (2018).
- Ascano, M., Hafner, M., Cekan, P., Gerstberger, S., Tuschl, T. Identification of RNA-protein interaction networks using PAR-CLIP. Wiley Interdisciplinary Reviews. RNA. 3 (2), 159-177 (2012).
- McHugh, C. A., Russell, P., Guttman, M. Methods for comprehensive experimental identification of RNA-protein interactions. Genome Biology. 15 (1), 203 (2014).
- Sugimoto, Y., et al. Analysis of CLIP and iCLIP methods for nucleotide-resolution studies of protein-RNA interactions. Genome Biology. 13 (8), (2012).
- Bayat, P., et al. SELEX methods on the road to protein targeting with nucleic acid aptamers. Biochimie. 154, 132-155 (2018).
- Armaos, A., Colantoni, A., Proietti, G., Rupert, J., Tartaglia, G. G. CatRAPID omics v2.0: Going deeper and wider in the prediction of protein-RNA interactions. Nucleic Acids Research. 49, 72-79 (2021).
- Agostini, F., et al. CatRAPID omics: A web server for large-scale prediction of protein-RNA interactions. Bioinformatics. 29 (22), 2928-2930 (2013).
- Zacco, E., et al. Probing TDP-43 condensation using an in silico designed aptamer. Nature Communications. 13 (1), 3306 (2022).
- Leppek, K., Stoecklin, G. An optimized streptavidin-binding RNA aptamer for purification of ribonucleoprotein complexes identifies novel ARE-binding proteins. Nucleic Acids Research. 42 (2), 13 (2014).
- Zhang, Y., Lai, B. S., Juhas, M. Recent advances in aptamer discovery and applications. Molecules. 24 (5), 941 (2019).
- UniProt Consortium. UniProt: the Universal Protein Knowledgebase in 2023. Nucleic Acids Research. 51, 523-531 (2023).
- Tyanova, S., Temu, T., Cox, J. The MaxQuant computational platform for mass spectrometry-based shotgun proteomics. Nature Protocols. 11 (12), 2301-2319 (2016).
- Tyanova, S., et al. The Perseus computational platform for comprehensive analysis of (prote)omics data. Nature Methods. 13 (9), 731-740 (2016).
- Rappsilber, J., Mann, M., Ishihama, Y. Protocol for micro-purification, enrichment, pre-fractionation and storage of peptides for proteomics using StageTips. Nature Protocols. 2 (8), 1896-1906 (2007).
- Bellucci, M., Agostini, F., Masin, M., Tartaglia, G. G. Predicting protein associations with long noncoding RNAs. Nature Methods. 8 (6), 444-445 (2011).
- Gallo-Oller, G., Ordoñez, R., Dotor, J. A new background subtraction method for Western blot densitometry band quantification through image analysis software. Journal of Immunological Methods. 457, 1-5 (2018).
- Weissinger, R., Heinold, L., Akram, S., Jansen, R. P., Hermesh, O. RNA proximity labeling: A new detection tool for RNA-protein interactions. Molecules. 26 (8), 2270 (2021).
- Hirsch, J. D., et al. Easily reversible desthiobiotin binding to streptavidin, avidin, and other biotin-binding proteins: Uses for protein labeling, detection, and isolation. Analytical Biochemistry. 308 (2), 343-357 (2002).
- Sefah, K., Shangguan, D., Xiong, X., O'Donoghue, M. B., Tan, W. Development of DNA aptamers using cell-SELEX. Nature Protocols. 5 (6), 1169-1185 (2010).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados