Method Article
Localización ultraestructural de LC3 endógena mediante microscopía óptica correlativa en sección
En este artículo
Resumen
Aquí, presentamos un protocolo para la microscopía correlativa de luz-electrónica optimizada en sección basada en el marcaje endógeno y fluorescente como herramienta para investigar la localización de proteínas raras en relación con la ultraestructura celular. El poder de este enfoque se demuestra mediante la localización ultraestructural de LC3 endógena en células hambrientas sin tratamiento con bafilomycina.
Resumen
La visualización de orgánulos autofágicos a nivel ultraestructural mediante microscopía electrónica (EM) es esencial para establecer su identidad y revelar detalles que son importantes para comprender el proceso autofágico. Sin embargo, los métodos de EM a menudo carecen de información molecular, lo que obstruye la correlación de la información ultraestructural obtenida por EM con la localización basada en microscopía de fluorescencia de proteínas específicas de autofagia. Además, la rareza de los autofagosomas en condiciones celulares inalteradas dificulta la investigación por parte de la EM, que requiere un gran aumento y, por lo tanto, proporciona un campo de visión limitado.
En respuesta a ambos desafíos, se aplicó un método de microscopía óptica y electrónica correlativa en sección (CLEM) basado en el marcaje fluorescente para correlacionar un marcador autofagosomal común, LC3, con la ultraestructura EM. El método se utilizó para cribar rápidamente las células en microscopía de fluorescencia para el marcaje de LC3 en combinación con otros marcadores relevantes. Posteriormente, CLEM identificó las características ultraestructurales subyacentes de puntos seleccionados marcados con LC3. El método se aplicó a células hambrientas sin añadir inhibidores de la acidificación lisosomal.
En estas condiciones, la LC3 se encontró predominantemente en los autofagosomas y raramente en los autolisosomas, en los que la LC3 se degrada rápidamente. Estos datos muestran tanto la viabilidad como la sensibilidad de este enfoque, demostrando que CLEM se puede utilizar para proporcionar información ultraestructural sobre la autofagia mediada por LC3 en condiciones nativas, sin tratamientos farmacológicos ni alteraciones genéticas. En general, este método presenta una herramienta valiosa para los estudios de localización ultraestructural de proteínas de autofagia y otros antígenos escasos al unir la microscopía óptica con los datos de EM.
Introducción
La autofagia es un proceso clave para la eliminación y el reciclaje de proteínas y orgánulos citoplasmáticos. El proceso de macroautofagia (en lo sucesivo denominado autofagia) implica la formación de orgánulos de doble membrana, los autofagosomas, que permiten a las células encerrar moléculas citoplasmáticas y orgánulos para su degradación lisosomal. La autofagia ocurre a nivel basal en la mayoría de las células y se regula al alza en respuesta a las condiciones celulares, como la inanición o el estrés celular. La autofagia se produce de una manera específica del sustrato, dirigiéndose a estructuras o proteínas específicas para su degradación, o como un proceso a granel no selectivo que abarca partes del citosol. En la autofagia selectiva, los autofagosomas se forman por la conjugación de proteínas de la familia Atg8 (proteínas asociadas a microtúbulos 1A/B, cadena ligera 3A/B/C [LC3] y GABARAPs) a membranas derivadas del reciclaje de endosomas, el Golgi y/o el retículo endoplásmico (RE)1. LC3 reconoce la carga autofágica en el citosol directamente o a través de adaptadores de autofagia selectiva como P62/SQSTM. Las nuevas membranas autofágicas pueden conjugarse con LC3, expandirse y fusionarse para formar una membrana doble completa que encierra la carga, llamada autofagosoma. El autofagosoma madura y finalmente se fusiona con un endosoma o lisosoma, tras lo cual la carga autofágica y los adaptadores se degradan2.
Los estudios sobre la formación, maduración y fusión de autofagosomas a menudo hacen uso de tecnologías de microscopía óptica. La microscopía de fluorescencia de LC3 se utiliza generalmente para evaluar el número y la localización celular de los autofagosomas en diferentes condiciones. Además, al acoplar LC3 a GFP sensible al pH y RFP estable al pH en una llamada sonda en tándem, se puede medir el flujo general de autofagia en células vivas en función de la pérdida de fluorescencia de GFP3. Estos enfoques son herramientas valiosas para que los investigadores comprendan el papel y el mecanismo de la autofagia en diferentes condiciones. Otra herramienta invaluable es la microscopía electrónica (EM), que revela la ultraestructura de orgánulos autofágicos en diferentes etapas de la autofagia 4,5,6,7,8. Hasta la fecha, la EM sigue siendo el método de elección para identificar las etapas precisas de la formación de autofagosomas mediante la discriminación de diferentes membranas autofágicas por morfología: fagóforo (doble membrana no completamente cerrada), autofagosoma (doble membrana cerrada alrededor de la carga citosólica) y autolisosoma (pérdida [parcial] de la membrana autofágica interna). Sin embargo, la morfología sin información molecular puede ser propensa a la identificación errónea o a la ambigüedad. Immuno-EM es el método más completo para la caracterización molecular simultánea y la clasificación morfológica de orgánulos autofágicos. Por ejemplo, el marcaje inmunogold de LC3 en criosecciones descongeladas permite la localización ultraestructural de LC3 y la identificación precisa de orgánulos marcados con LC39.
Un inconveniente de la EM es el pequeño campo de visión que conlleva el gran aumento necesario para observar la fina ultraestructura de las membranas autofágicas y, en el caso de la inmuno-EM, para localizar la etiqueta que marca la proteína de interés. Debido a su escasez y bajos niveles de proteínas, esto generalmente dificulta el análisis cuantitativo de EM de los autofagosomas. Para aumentar el número de autofagosomas, las células a menudo se privan de alimentos y se tratan con bafilomycin A1 (BafA1), un inhibidor de la acidificación y degradación lisosomal. Sin el tratamiento con BafA1, la búsqueda de autofagosomas por EM requiere mucho tiempo, debido a la escasez de estos orgánulos. El método presentado en este manuscrito aborda este problema a través del marcaje fluorescente y la obtención de imágenes de LC3 endógena en criosecciones descongeladas en un microscopio de fluorescencia antes de la preparación adicional para la EM. A continuación, las imágenes fluorescentes guían la búsqueda de estructuras marcadas con LC3 en el EM. Después de la recolección, las imágenes EM se correlacionan con las imágenes de fluorescencia para agregar información molecular (la presencia de LC3) a la ultraestructura de la célula. Este método de "CLEM en sección" aumenta en gran medida la capacidad de encontrar estructuras marcadas con LC3, especialmente en condiciones no tratadas, para su posterior identificación y clasificación por EM.
Este método se aplicó a células HEPG210 derivadas de hepatoblastoma que no estaban saturadas para encontrar autofagosomas en condiciones inalteradas (es decir, no se utilizó BafA1). Se encontraron relativamente pocos puntos fluorescentes (menos de uno por perfil celular en una sección de 90 nm), lo que concuerda con el alto recambio de la CL311. Esta escasez de puntos de LC3 enfatizó el valor de CLEM; mediante la selección de regiones con varios puntos fluorescentes para la obtención de imágenes en el ME, se encontraron orgánulos positivos para LC3 y se caracterizaron de una manera mucho más efectiva que a través de la inmuno-EM convencional. Esto reveló que la mayoría de los orgánulos LC3 positivos eran autofagosomas, definidos por su morfología, lo que contrasta con los resultados obtenidos en células tratadas con BafA1, donde los autolisosomas son más comunes9. Estos datos muestran que con CLEM en sección, la autofagia se puede estudiar a nivel ultraestructural sin necesidad de inhibir el flujo autofágico.
Protocolo
1. Preparación de herramientas y reactivos
NOTA: Para conocer los reactivos, tampones y soluciones necesarios, consulte el Archivo complementario 1 o 12 para obtener más información. Para obtener detalles relacionados con todos los materiales, reactivos, equipos y software utilizados en este protocolo, consulte la Tabla de materiales.
- Fijadores
- Prepare un tampón de fosfato (PB) de 0,2 M o un tampón de 0,2 M PIPES, HEPES, EGTA, MgSO4 (PHEM), como se describe en el Archivo Suplementario 1, para utilizarlo como base para soluciones fijadoras.
NOTA: Los fijadores se tamponan rutinariamente en tampón 0,1 M PB o PHEM para amortiguar la acidificación causada por la reacción del aldehído con el material biológico. - Dado que la calidad del paraformaldehído (PFA) es clave para la fijación fiable de la ultraestructura de las muestras, utilice PFA de grado EM. Para seguir este protocolo, use soluciones madre al 16%, preparadas a partir de gránulos de PFA de alta calidad (consulte el Archivo Suplementario 1).
PRECAUCIÓN: El paraformaldehído es un producto químico peligroso (indicaciones de peligro H228, H301, H302, H311, H314, H315, H317, H318, H331, H332, H335, H341, H350). Cuando trabaje con PFA, use equipo de protección (guantes, bata de laboratorio y gafas protectoras) y trabaje con una capucha para productos químicos. Los residuos que contienen PFA deben recogerse y eliminarse de acuerdo con las directrices y reglamentos de los institutos. - Combine 10 ml de PB 0,2 M, 5 ml de PFA al 16 % (en agua desmineralizada [dH 2 O]) y 5 ml de dH2O para preparar una solución fijadora de PFA al 4 %.
- Opcional: La adición de glutaraldehído (GA) al 0,02%-0,5% a la solución fijadora del paso 1.1.3 mejora la preservación de la ultraestructura, pero reduce la antigenicidad de la muestra frente a muchos anticuerpos.
NOTA: Cuando se desee la fijación de GA, utilice GA de grado EM de un proveedor adecuado.
PRECAUCIÓN: El glutaraldehído es un producto químico peligroso (indicaciones de peligro H301, H302, H314, H317, H330, H332, H334, H335, H400, H411). Trabaje con una capucha química y use equipo de protección (guantes, bata de laboratorio y gafas protectoras) cuando manipule AG. Los desechos que contienen AG deben recolectarse y eliminarse de acuerdo con las pautas y regulaciones de los institutos.
- Prepare un tampón de fosfato (PB) de 0,2 M o un tampón de 0,2 M PIPES, HEPES, EGTA, MgSO4 (PHEM), como se describe en el Archivo Suplementario 1, para utilizarlo como base para soluciones fijadoras.
- Herramientas y materiales
- Rasque la superficie de los pasadores portamuestras de aluminio y soniquelos en etanol 3 x 10 min para eliminar los restos de metal y garantizar una adherencia óptima cuando se monten bloques de celdas incrustados en gelatina en los pines.
- Utilice un recipiente de almacenamiento adecuado para almacenar los pasadores portamuestras de aluminio con sus muestras en nitrógeno líquido (LN2).
- Haz un manipulador colocando un solo cabello o pestaña en el extremo de una brocheta de madera con esmalte de uñas.
- Haz un bucle de recogida. Dobla un alambre de acero inoxidable de 0,3 mm de grosor alrededor de una barra redonda de 3 mm de diámetro y retuerce los extremos, formando un bucle en un extremo. Inserte los extremos retorcidos en la punta de una pipeta. Inserta una brocheta de madera desde el otro extremo y pégala con pegamento o resina.
NOTA: Un lazo de captación también está disponible comercialmente (consulte la Tabla de materiales). - Para preparar los bucles de secado de rejilla, siga los mismos pasos para hacer bucles de recogida: forme un alambre de acero inoxidable en un bucle de 4 mm y fíjelo en una punta de pipeta grande con pegamento o resina.
- Cubra las rejillas con una película delgada de soporte como formvar (protocolo en el Archivo Suplementario 1). Antes de usar, cubra las rejillas con una fina capa de carbón.
NOTA: Las rejillas listas para usar están disponibles comercialmente (consulte la Tabla de materiales). Las rejillas recubiertas de Formvar se pueden almacenar indefinidamente a temperatura ambiente (RT); Las rejillas recubiertas de carbono pueden almacenarse durante varios meses en RT. - Prepare portaobjetos de vidrio limpios y cubreobjetos grandes (24 mm x 24 mm es ideal con portaobjetos de vidrio de 25 mm de ancho), como en 13.
2. Fijación y preparación de la muestra
- Fijación
- Utilice el fijador preparado en el paso 1.1.3 (PFA al 4% en 0,1 M PB). Para líneas celulares adherentes, cultivar 1-5 × 10 6 células en placas de6 cm. Añadir el fijador al medio de cultivo en una proporción de 1:1 e incubar la muestra durante 5 min a RT. Luego, reemplace la mezcla de fijador medio con fijador solo e incube durante 2 h en RT.
NOTA: Los recuentos exactos de células, la confluencia y las condiciones de cultivo pueden variar según el sistema modelo utilizado. - Almacenar las muestras durante la noche o durante un máximo de 3-4 semanas en PFA al 0,5% en 0,1 M PB a 4 °C.
NOTA: Se puede agregar AG a la fijación (ver paso 1.1.4) y la longitud de fijación se puede cambiar para encontrar un equilibrio óptimo entre la preservación de la morfología y la antigenicidad, que difiere según la muestra y el etiquetado. Para obtener más información, consulte14.
- Utilice el fijador preparado en el paso 1.1.3 (PFA al 4% en 0,1 M PB). Para líneas celulares adherentes, cultivar 1-5 × 10 6 células en placas de6 cm. Añadir el fijador al medio de cultivo en una proporción de 1:1 e incubar la muestra durante 5 min a RT. Luego, reemplace la mezcla de fijador medio con fijador solo e incube durante 2 h en RT.
- Incrustación de muestras
- Lave el plato con celdas fijas 3 veces con PBS en RT. Luego, reemplácelo con PBS que contenga 0.15% de glicina e incube durante 10 minutos a RT.
- Sustituir el PBS que contiene glicina al 0,15% por gelatina al 1% en PBS precalentado a 37 °C, y raspar y transferir las células en gelatina al 1% a un tubo de microcentrífuga. Granular las células a 6.000 × g durante 1 min a RT en una microcentrífuga. A continuación, retire la gelatina al 1% sin alterar el gránulo y añada gelatina al 12% calentada a 37 °C. Vuelva a suspender el gránulo celular pipeteando suavemente hacia arriba y hacia abajo con puntas de pipeta o pipetas Pasteur de vidrio precalentadas a 37 °C.
- Incubar a 37 °C durante 10 min; A continuación, pegue las células a 6.000 × g durante 1 min. Solidifica la gelatina en hielo durante 30 min.
- Para eliminar las células incrustadas en gelatina del tubo, corte el extremo del tubo que contiene el gránulo del resto del tubo con una cuchilla de afeitar. Luego, perpendicular al primer corte, corte el extremo del tubo con el gránulo de celda por la mitad.
- Incubar las dos mitades del extremo del tubo que contienen el gránulo celular incrustado en gelatina en sacarosa 2,3 M durante 10 min a 4 °C. Esto hace que las mitades de los gránulos celulares incrustados en gelatina se encojan ligeramente y se desprendan del tubo de plástico.
NOTA: Los gránulos celulares incluidos en gelatina deben mantenerse a 4 °C o helados tanto como sea posible para evitar que la sacarosa 2,3 M se vuelva demasiado viscosa y la gelatina demasiado blanda. Durante la manipulación de los gránulos de células incrustados en gelatina en los siguientes pasos, trabaje con una sola muestra a la vez y mantenga las demás en hielo, o trabaje en una habitación fría (~4 °C). Evite el sobrecalentamiento de los gránulos celulares incrustados en gelatina por la luz solar, las lámparas de microscopio calientes u otras fuentes de calor. - Retire las mitades del tubo con el gránulo celular incrustado en gelatina de la sacarosa 2,3 M. Luego, retire las mitades de gránulos celulares incrustados en gelatina de las mitades del tubo de plástico con unas pinzas. Corta manualmente el gránulo en bloques de un tamaño adecuado (~1 mm3) con una cuchilla de afeitar. Utilice un microscopio de disección estereoscópica para ampliar el sujeto durante el corte.
- Infundir los bloques celulares incrustados en gelatina con sacarosa 2,3 M durante 3-16 h, girando de extremo a extremo en un rotor a 4 °C.
- Coloque un bloque de celdas incrustado en gelatina en un pasador portamuestras de aluminio (consulte el paso 1.2.1). Deje suficiente sacarosa 2,3 M alrededor de los bordes del bloque para que forme un "collar" delgado entre el bloque y el pasador. Evite demasiada sacarosa 2,3 M cubriendo la parte superior del bloque. Congele a presión y almacene en LN2.
3. Seccionamiento
- Recorte (véase también12)
- Tome un alfiler con un bloque de células incrustadas en gelatina del almacenamiento de LN2 y colóquelo dentro de un conjunto de criomicrótomo a -80 °C.
- Recorta la parte frontal del bloque para aplanar su superficie y obtener secciones de ~250 nm. Sumerja el asa de 3 mm en una solución de recogida (sacarosa 1:1 2,3 M y metilcelulosa al 2%), inserte el asa en la cámara criogénica del micrótomo y espere hasta que comience a formarse hielo en la gota (normalmente 5-7 s). Luego, recoja inmediatamente la sección presionando rápida pero suavemente la gota contra ellos. Retire el asa de la cámara criogénica, espere hasta que la gota se haya descongelado por completo y presione la gota sobre un portaobjetos de vidrio.
- Compruebe la orientación de la célula mediante la tinción con azul de toluidina de las secciones.
- Coloque una gota de solución de azul de toluidina (ver Archivo Suplementario 1) encima de las secciones en un portaobjetos de vidrio y seque en una placa calefactora a 80 °C hasta que los bordes de la gota estén secos.
- Retire el portaobjetos de vidrio de la placa calefactora y enjuague suavemente el azul de toluidina con dH2O, recogiéndolo en un contenedor de residuos adecuado.
- Seque el portaobjetos de vidrio y verifique la orientación de la celda en las secciones a través de un microscopio óptico simple de sobremesa.
- Recorte los lados del bloque seccionando 50-100 μm en el lado de la cara del bloque de muestra con la esquina de la cuchilla. Recorte cuatro lados de la cara del bloque de muestra girando el portamuestras 90° después de recortar cada lado para crear un rectángulo sobresaliente de ~250 μm x 375 μm. Seleccione el área sobresaliente en función de la orientación de la celda determinada en el paso anterior.
- Seccionamiento y recogida
- Enfriar el criomicrótomo a -100 °C. Corta una cinta desde el rectángulo que sobresale, las secciones tienen un grosor de 70-90 nm y un brillo plateado y dorado. Guíe las secciones lejos del filo de la navaja de diamante con un pelo en un palo (ver sección 1.2.3) para crear una cinta larga (2-5 mm).
- Una vez que se haya formado una cinta adecuada, deje de seccionar para recoger la cinta. Sumerja el asa de recogida de 3 mm en sacarosa 2,3 M y metilcelulosa al 2% mezclada 1:1, inserte el asa en la cámara criogénica del micrótomo y espere hasta que la gota comience a congelarse (normalmente 5-7 s). Luego, recoja inmediatamente las secciones presionando rápida pero suavemente la gota contra ellas. Retire el asa de la cámara criogénica, espere hasta que la gota se haya descongelado por completo y presione la gota sobre una rejilla preparada (paso 1.2.6).
NOTA: Las rejillas con secciones pueden almacenarse a 4 °C durante varios meses.
4. Etiquetado y microscopía óptica
- Etiquetado
- Coloque las rejillas con secciones (Figura 1A) con la sección hacia abajo en ~ 1 ml de PBS en un plato pequeño o placa de pocillos múltiples. Incubar a 37 °C durante 30 min.
NOTA: Este paso elimina la gelatina que se encuentra entre las células; La gelatina no es necesaria después del corte e interfiere con el protocolo restante. - Procese las rejillas con la sección hacia abajo en gotas de ~75 μL en parafilm (consulte la Figura 1B). Comience con PBS + lavados de glicina al 0,15 % (3 x 2 min) en RT. A continuación, incubar las rejillas con 0,1% de albúmina sérica bovina (BSA)-c + 0,5% de gelatina de piel de pescado (FSG) en PBS durante 10 min a RT como paso de bloqueo. Diluir los anticuerpos primarios en BSA-c al 0,1% + FSG al 0,5% en PBS e incubar las rejillas en gotas de ~10 μL de esta solución durante 1 h a RT (Figura 1C).
- Lave las rejillas en 0.1% BSA en PBS 5x en RT. A continuación, diluir los anticuerpos secundarios y el 4',6-diamidino-2-fenilindol (DAPI; 10 μg/mL) en BSA-c al 0,1% + FSG al 0,5% en PBS e incubar las rejillas en gotas ~10 μL de esta solución durante 30+ min a RT (Figura 1C). Lave las rejillas en PBS 5x en RT.
NOTA: Opcionalmente, un anticuerpo secundario puede marcarse con partículas de oro coloidal de 5, 10, 15 o 20 nm conjugadas con la proteína A (PAG) para la localización de la proteína de interés en la ME. Si lo desea, incubar las rejillas con PAG durante 20 min en RT después del paso 4.1.3. A continuación, lavar 5 veces con PBS en RT. Evite el uso simultáneo de múltiples anticuerpos primarios y tome nota de la reactividad de PAG a las IgG de diferentes especies para evitar reacciones cruzadas no deseadas. Para obtener más información, consulte12.
- Coloque las rejillas con secciones (Figura 1A) con la sección hacia abajo en ~ 1 ml de PBS en un plato pequeño o placa de pocillos múltiples. Incubar a 37 °C durante 30 min.
- Montaje de muestras para microscopía óptica
- Sumerja las rejillas en glicerol al 50 % en dH 2 O2x 5 min a RT. Intercala las rejillas entre un portaobjetos de vidrio y un cubreobjetos en glicerol al 50 %, una rejilla por cubreobjetos, con secciones orientadas hacia el cubreobjetos (Figura 1D).
NOTA: La calidad del etiquetado puede deteriorarse cuando las rejillas se mantienen montadas en glicerol al 50% durante más de 30 minutos. Por lo tanto, se recomienda montar e crear imágenes de dos o tres rejillas a la vez y dejar las demás en la solución de etiquetado secundario.
- Sumerja las rejillas en glicerol al 50 % en dH 2 O2x 5 min a RT. Intercala las rejillas entre un portaobjetos de vidrio y un cubreobjetos en glicerol al 50 %, una rejilla por cubreobjetos, con secciones orientadas hacia el cubreobjetos (Figura 1D).
- Microscopía óptica
- Lleve un portaobjetos de vidrio con rejillas intercaladas a un microscopio de campo amplio con una platina automatizada. Seleccione un objetivo de aceite de gran aumento (63x o 100x). Cree un conjunto de mosaicos de imagen de (parte de) la cinta de secciones (Figura 1E).
NOTA: Algunos anticuerpos secundarios pueden formar agregados fluorescentes en las rejillas, especialmente alrededor de pliegues o desgarros en las secciones. Además, algunos tipos de células y tejidos contienen estructuras autofluorescentes. Si se esperan tales problemas, se recomienda incluir una rejilla de control negativa no incubada con anticuerpos primarios.
- Lleve un portaobjetos de vidrio con rejillas intercaladas a un microscopio de campo amplio con una platina automatizada. Seleccione un objetivo de aceite de gran aumento (63x o 100x). Cree un conjunto de mosaicos de imagen de (parte de) la cinta de secciones (Figura 1E).
- Desmontaje y contraste EM
- Agregue 10 μL de dH2O al costado del sándwich de cubreobjetos de vidrio y espere a que la acción capilar llene la interfaz del sándwich de cubreobjetos de vidrio. Retire con cuidado el cubreobjetos sin mezclar el aceite de inmersión en el glicerol. Recupere las rejillas con pinzas y sumérjalas en dH2O 3x a RT para eliminar el glicerol al 50%.
NOTA: El aceite puede interferir con la tinción de uranilo y deteriorar el contraste EM. - Seque cuidadosamente la parte posterior de la rejilla con papel de seda sin pelusa.
NOTA: Si la muestra también fue marcada con partículas de oro coloidal, realice los siguientes pasos: coloque la rejilla con las secciones con la sección hacia abajo sobre las gotas de PBS y lave 2 veces en RT. Postfijo en AG al 1% durante 5 min en RT (consulte la nota de precaución en 1.1.4). Lavar en PBS 2 veces en RT. - Coloque las rejillas con la sección hacia abajo sobre gotas de dH2O y lave 8 veces a RT.
- Para teñir las secciones para contraste en EM, incubar con acetato de uranilo (UA), pH 7, durante 5 min a RT (Figura 1F).
- Antes de colocar las rejillas, enfríe la UA:metilcelulosa, pH 4, colocando gotas en parafilm sobre una placa de metal sobre hielo. A continuación, lavar las rejillas con UA:metilcelulosa, pH 4, 2x e incubar con UA:metilcelulosa helada, pH 4, durante 10 min (Figura 1F).
PRECAUCIÓN: El acetato de uranilo es un producto químico peligroso (indicaciones de peligro H300, H330, H373, H411). En los pasos que requieran UA, trabaje con una capucha química y use equipo de protección (bata de laboratorio, guantes y gafas protectoras). Recolectar y eliminar los residuos que contengan AU de acuerdo con las pautas y regulaciones de los institutos. - Realice un bucle de secado de la rejilla insertando un bucle de secado de rejilla en la gota de UA:metilcelulosa debajo de la rejilla y levantándola suavemente hasta que la rejilla se desprenda de la gota12. Seque el exceso de UA:metilcelulosa tocando el bucle en un ángulo de ~60° (secciones hacia abajo) sobre papel de filtro sin pelusa (consulte la Tabla de materiales) y arrastrándolo lentamente a lo largo del papel hasta que no se absorba más UA:metilcelulosa. A continuación, coloque el bucle con la rejilla en una rejilla adecuada y déjelo secar durante >10 min a RT (Figura 1G).
- Agregue 10 μL de dH2O al costado del sándwich de cubreobjetos de vidrio y espere a que la acción capilar llene la interfaz del sándwich de cubreobjetos de vidrio. Retire con cuidado el cubreobjetos sin mezclar el aceite de inmersión en el glicerol. Recupere las rejillas con pinzas y sumérjalas en dH2O 3x a RT para eliminar el glicerol al 50%.
5. EM
- Utilice la visión general obtenida por microscopía óptica para localizar una región de interés (ROI) para obtener imágenes en el microscopio electrónico de transmisión (TEM; Figura 1H). Anote el ROI en el conjunto de datos de microscopía óptica. Una vez seleccionada una región, obtenga un conjunto de mosaicos de imágenes con un aumento de 20.000x-50.000x en el TEM. Reconstruir el conjunto de mosaicos de imágenes en el software de posprocesamiento15,16.
6. Correlación y análisis
- Cargue el conjunto de datos de microscopía óptica y EM en un software de procesamiento de imágenes adecuado, como ImageJ/Fiji17, el complemento ec-CLEM en Icy18 o Photoshop. Recorte y gire el conjunto de datos de microscopía óptica para que coincida con el conjunto de mosaicos EM.
- Realizar la correlación basada en la señal DAPI en fluorescencia y contornos nucleares en EM (Figura 1I). Cambie las imágenes para superponerlas con precisión y realice la correlación manual con precisión. Para que el enfoque sea más exacto, aplique la correlación basada en puntos de referencia a través, por ejemplo, del complemento ec-CLEM en Icy o el complemento BigWarp en ImageJ, para correlacionar las imágenes mediante la selección manual de los puntos correspondientes. Se dispone de un protocolo detallado, paso a paso, para la correlación con ec-CLEM19.
NOTA: Este enfoque también funciona bien con el uso de sondas fiduciales bimodales20,21.
- Realizar la correlación basada en la señal DAPI en fluorescencia y contornos nucleares en EM (Figura 1I). Cambie las imágenes para superponerlas con precisión y realice la correlación manual con precisión. Para que el enfoque sea más exacto, aplique la correlación basada en puntos de referencia a través, por ejemplo, del complemento ec-CLEM en Icy o el complemento BigWarp en ImageJ, para correlacionar las imágenes mediante la selección manual de los puntos correspondientes. Se dispone de un protocolo detallado, paso a paso, para la correlación con ec-CLEM19.
- Analice las imágenes correlacionadas seleccionando ROI basadas en la señal fluorescente en un programa adecuado (por ejemplo, ImageJ). Para el análisis cuantitativo, cree una colección de ROI para todos los orgánulos marcados. A continuación, inspeccione la ultraestructura correspondiente de los ROI individuales y clasifíquelos en función de los elementos morfológicos.
Resultados
De Maziere et al.9 han publicado recientemente un protocolo inmuno-EM optimizado para el marcaje inmuno-oro de LC3 en criosecciones ultrafinas9. Este estudio incluyó condiciones de inanición sin BafA1, en las que la LC3 estaba presente, pero era relativamente rara y difícil de encontrar por EM. En un estudio separado se introdujo un método CLEM en sección, que utiliza la sensibilidad del marcaje de fluorescencia para visualizar proteínas endógenas relativamente raras y de baja expresión y correlacionarlas con la ultraestructura EM14. En este caso, estos dos enfoques se combinan mediante el uso del protocolo de etiquetado LC3 optimizado como parte de un enfoque CLEM.
Las células HEPG2, células derivadas del hígado con niveles relativamente altos de autofagia basal22, se sometieron a inanición en un medio mínimo (solución salina equilibrada de Earle [EBSS]) durante 2 h antes de la fijación en PFA al 4%. A esto le siguió la preparación de la muestra mediante el método Tokuyasu de criosección ultrafina (secciones 1-3; ver Ranura y Geuze12), que es altamente compatible con CLEM en la sección14,23. Las criosecciones descongeladas se marcaron con fluorescencia (sección 4 del protocolo y Figura 1) utilizando el anticuerpo primario anti-LC3 de ratón9. Además, se utilizó anti-LAMP1 de conejo para indicar endolisosomas, seguido de anticuerpos secundarios anti-ratón AlexaFluor488 y anti-conejo AlexaFluor568. Las rejillas se intercalaron entre un cubreobjetos y un portaobjetos de vidrio y se tomaron imágenes en RT en un microscopio de campo amplio (objetivo de aceite de 100x 1,47 NA, cámara sCMOS).
Una ventaja del marcaje por fluorescencia de secciones delgadas sobre el IF convencional de célula entera es el aumento de la resolución en Z, ya que el espesor físico de la sección es de 60-90 nm. Con esta resolución Z mejorada, el marcaje de fluorescencia de LC3 y LAMP1 en secciones delgadas revela muy poca colocalización (Figura 2A). En las células tratadas con inhibidores lisosomales, como BafA1, se produce una alta colocalización, ya que la LC3 encerrada en lisosomales permanece sin degradar9. En las células no tratadas, la LC3 se degrada rápidamente al entrar en contacto con lisosomas positivos para LAMP1 enzimáticamente activos y, por lo tanto, la colocalización es rara en estas condiciones. En general, se observó menos de un punctum LC3 por perfil celular. Esto indica que incluso en condiciones de inanición, el recambio de autofagosomas es rápido, lo que mantiene bajos los números de autofagosomas. También destaca la importancia de utilizar CLEM para encontrar las raras estructuras marcadas con LC3, utilizando el gran campo de visión proporcionado por la microscopía óptica. Además, la mayor sensibilidad del marcaje de fluorescencia en comparación con el marcaje de oro permite la identificación de más orgánulos positivos para LC3 que en la inmuno-EM convencional, lo que ayuda aún más a su caracterización.
Después de adquirir un mosaico completo de la cinta de secciones, las cuadrículas se recuperaron del microscopio y se tiñeron posteriormente para EM utilizando UA y el método de bucle de salida (pasos de protocolo 4.4-4.6; Figura 1F,G). Este método de "bucle de salida" garantiza que una fina capa de UA:metilcelulosa permanezca en la rejilla, lo que crea el contraste deseado en el EM. El grosor de la capa depende de la velocidad y el ángulo con el que la UA:metilcelulosa se seca en el papel de filtro. Arrastrar el bucle demasiado rápido puede dejar demasiada UA:metilcelulosa en la cuadrícula y oscurecer la apariencia de las secciones en EM. Arrastrar demasiado despacio puede extraer demasiada UA:metilcelulosa, lo que da como resultado muy poca tinción y mala morfología, y corre el riesgo de que la rejilla se salga del bucle. La coloración "mancha de aceite" (Figura 1G) en rejillas secas indica un espesor adecuado de la capa de UA:metilcelulosa.
Después del bucle y el secado, las rejillas se visualizaron en un TEM a ROI seleccionadas por fluorescencia. Los conjuntos de datos de FI y EM se correlacionaron superponiendo la señal DAPI a los contornos de los núcleos visibles en el ME, generando una imagen integrada que contiene información de ambas modalidades.
Encontrar la misma área en EM que la seleccionada en IF puede ser un desafío. Por lo tanto, se recomienda tener a mano una imagen general del conjunto de mosaicos de FI mientras se busca en el EM. Los usuarios deben buscar características reconocibles en ambas modalidades, como pliegues o rasgaduras en las secciones, barras de cuadrícula o disposición de núcleos. También es importante tener en cuenta que la muestra puede aparecer rotada y reflejada en EM. Se pueden utilizar "cuadrículas de búsqueda" con características específicas para identificar áreas para facilitar la correlación (consulte la Tabla de materiales).
La correlación de los orgánulos LC3 positivos con la ultraestructura EM reveló que los diferentes puntos representaban distintas etapas de autofagia (Figura 2B). Aunque la preservación de la ultraestructura autofagosómica es un desafío en las criosecciones, se observaron con frecuencia orgánulos con contenido citoplasmático y membranas dobles (Figura 2C, flechas en orgánulos 1-5; Figura suplementaria S1), que son características morfológicas definitorias de los autofagosomas. Curiosamente, los puntos fluorescentes bastante débiles fueron identificados por EM como autolisosomas LC3 positivos (Figura 2C, orgánulo 6; el contenido autofágico está marcado con *), caracterizados por un contenido denso y vesículas intraluminales. Esto mostró que cantidades muy pequeñas de LC3 son visibles en el FI de las criosecciones ultrafinas, e indicó que, a pesar del entorno degradativo, algunas LC3 son detectables en autolisosomas en estado estacionario. Sin embargo, la mayoría de los punchas positivos para LC3 representaban autofagosomas, mientras que los autolisosomas eran muy raros. Esto contrasta con las células tratadas con BafA1, que acumulan principalmente autolisosomas y no autofagosomas9.
En resumen, este protocolo describe un método CLEM en sección para vincular la información molecular obtenida por microscopía de fluorescencia con la ultraestructura de EM. Este método aumenta la sensibilidad de la inmuno-EM, ya que solo se utilizan fluoróforos para el marcaje y estos generalmente producen más señal que las sondas EM. El método es especialmente adecuado para el uso de criosecciones ultrafinas, en las que se pueden obtener altos niveles de fluorescencia específica sobre una tinción de fondo insignificante. Mediante el uso de la fluorescencia para detectar estructuras o eventos raros y correlacionar los ROI seleccionados con los EM, el tiempo de operación de EM y los costos asociados pueden reducirse considerablemente. La sensibilidad y viabilidad del método queda demostrada por la visualización de LC3 en células no tratadas y hambrientas, mostrando que LC3 se asocia predominantemente a autofagosomas en estas condiciones, con niveles muy bajos visibles en autolisosomas.
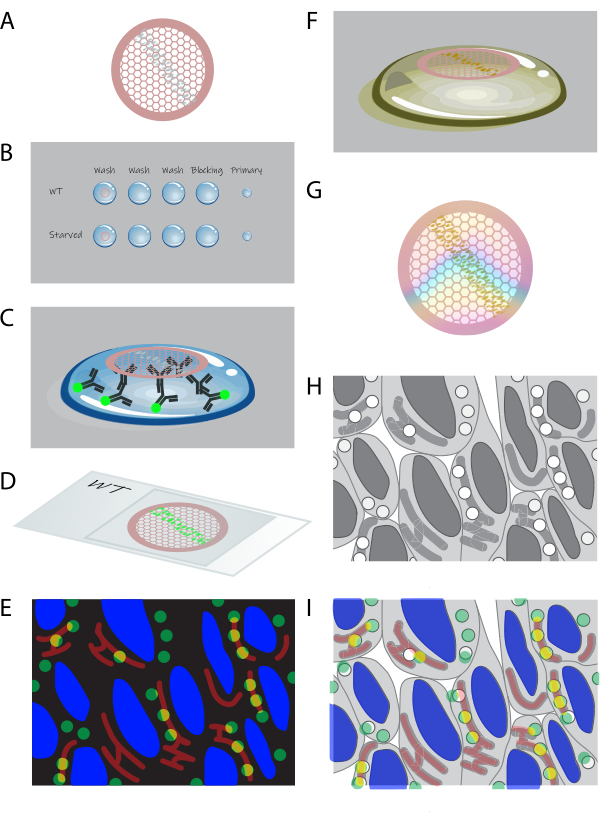
Figura 1: Vista general esquemática del CLEM en sección . (A) Las criosecciones de células incluidas en gelatina se recogen en una rejilla de cobre recubierta de formvar. (B) Las rejillas se procesan de sección hacia abajo en gotas de las soluciones apropiadas. (C) Las rejillas están marcadas con anticuerpos primarios y secundarios fluorescentes. (D) Las rejillas se intercalan entre un cubreobjetos y un portaobjetos de vidrio en glicerol al 50%. (E) Las imágenes de fluorescencia se recogen en un microscopio de campo amplio. (F) Las rejillas se recuperan del portaobjetos de vidrio y se procesan posteriormente mediante tinción de uranilo para EM. (G) Después del secado, las rejillas pueden ser fotografiadas por TEM. (H) El mosaico de imágenes TEM de gran aumento se adquiere de un área seleccionada a partir de datos de fluorescencia. (I) Las imágenes de la microscopía de fluorescencia y la microscopía electromagnética se correlacionan y se superponen. Haga clic aquí para ver una versión más grande de esta figura.
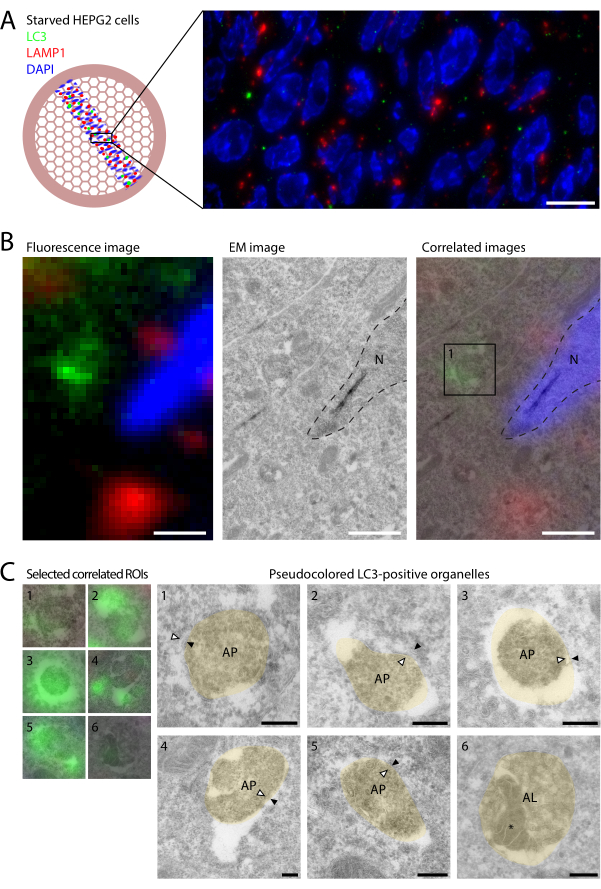
Figura 2: CLEM de LC3 y LAMP1 en células HEPG2 hambrientas. Las células HEPG2 se privaron de presencia durante 2 h en EBSS antes de la fijación con PFA al 4% durante 2 h. (A) Las imágenes de FI de LC3 (verde) y LAMP1 (rojo) en secciones revelan relativamente pocos puntos de LC3 y poca colocalización con LAMP1. (B) Vincular la información molecular de IF (panel izquierdo) con la información ultraestructural obtenida en EM (panel central) mediante la superposición de las dos modalidades de imagen basadas en DAPI y contornos nucleares (líneas discontinuas, panel derecho). La ultraestructura de los compartimentos individuales marcados con LC3, como se ejemplifica en el recuadro 1 (panel derecho), se muestra en C. (C) Ultraestructura de los compartimentos positivos para LC3. Las imágenes CLEM se muestran a la izquierda y las imágenes EM pseudocoloreadas (beige) a la derecha (las imágenes EM sin color se muestran en la Figura Suplementaria S1). Las membranas autofagosomales internas y externas están indicadas por puntas de flecha blancas y negras, respectivamente. El contenido autofágico dentro del autolisosoma en el ejemplo 6 se indica con *. Barras de escala = 10 μm (A), 1 μm (B), 200 nm (C). Haga clic aquí para ver una versión más grande de esta figura.
Figura suplementaria S1: Imágenes EM sin color de orgánulos LC3 positivos. (De la A a la F) Imágenes EM sin colorear de ejemplos pseudocoloreados 1-6 que se muestran en la Figura 2C. Los orgánulos se seleccionaron mediante fluorescencia LC3, como se describe en la Figura 2. Las membranas autofagosomales internas y externas están indicadas por puntas de flecha blancas y negras, respectivamente. El contenido autofágico dentro del autolisosoma en el ejemplo 6 se indica con *. Barras de escala = 200 nm. Abreviaturas: AL = autolisosoma; AP = autofagosoma; M = mitocondria. Haga clic aquí para descargar este archivo.
Archivo complementario 1: Tampones y soluciones utilizadas en este estudio. Este archivo complementario contiene las recetas y protocolos necesarios para fabricar los tampones y soluciones utilizados en este estudio. Haga clic aquí para descargar este archivo.
Discusión
El método presentado aquí aprovecha los avances recientes en CLEM en sección basado en criosección: la alta sensibilidad del etiquetado de FI y la correlación precisa (error de <100 nm) entre FM y EM14,24. Esto da como resultado un método con la sensibilidad para etiquetar fluorescentemente proteínas endógenas escasas y la capacidad de superponerlas con alta precisión a la ultraestructura EM. Por lo tanto, este método evita la necesidad de (sobre)expresión de proteínas marcadas exógenamente y el uso de marcadores EM menos sensibles. La viabilidad del método se demuestra mediante ejemplos de CLEM en LC3 endógena en células inexistentes, sin el uso de inhibidores lisosomales.
Las criosecciones descongeladas obtenidas con el método Tokuyasu son muestras ideales para inmuno-EM, ya que a diferencia de las secciones de resina, son permeables a los anticuerpos. Combinado con una fijación suave y procedimientos de contraste, esto generalmente produce una excelente eficiencia de etiquetado sobre otros métodos sin comprometer la ultraestructura detallada, y visualiza excelentemente las membranas celulares 12,25,26. Además, las criosecciones son altamente compatibles con la microscopía de fluorescencia, lo que las convierte en sustratos valiosos para CLEM. Tanto el marcaje clásico de inmunogold como el CLEM en criosecciones han proporcionado información fundamental para comprender la organización subcelular 14,27,28,29,30.
En la actualidad, las aplicaciones de CLEM en criosecciones descongeladas son cada vez más frecuentes, como resultado de los continuos desarrollos y optimizaciones 14,20,24,31,32,33,34 que han mejorado la calidad, aplicabilidad y precisión del abordaje. Ahora, mediante la correlación precisa de grandes conjuntos de imágenes de FI y EM, la técnica facilita el cribado de la ultraestructura de los componentes celulares endógenos marcados con fluorescencia 14,32,33. Esta es una ventaja con respecto a la inmuno-EM clásica, donde la búsqueda de estructuras marcadas con oro generalmente requiere un gran aumento y, por lo tanto, es más laboriosa y requiere más tiempo. Es por esta razón que la localización de LC3 en la ultraestructura se beneficia enormemente de CLEM. Los orgánulos LC3 positivos son comunes cuando se bloquea el aclaramiento autofágico (es decir, cuando las células se tratan con BafA1 o agentes que aumentan el pH), mientras que los orgánulos autofágicos se eliminan rápidamente en células inalteradas o hambrientas, lo que resulta en niveles muy bajos de estado estacionario. En tales condiciones, encontrar orgánulos marcados con LC3 utilizando inmuno-EM clásica puede ser un desafío, y CLEM ofrece una clara ventaja.
Anteriormente, CLEM en secciones de resina se aplicaba en estudios que utilizaban la expresión ectópica de LC3-GFP o una sonda en tándem LC3-GFP-RFP 35,36,37,38,39. En estos estudios, se realizaron imágenes de fluorescencia antes de la inclusión o directamente en secciones de resina acrílica40, y las muestras se analizaron posteriormente mediante EM. Hay varias ventajas de la incrustación de resina; La ultraestructura autofagosomal está generalmente bien conservada, especialmente si el material está congelado a alta presión40. Además, el contraste del material incrustado en resina teñido de metales pesados es generalmente más pronunciado que el de las criosecciones teñidas con uranilo. Las secciones incrustadas en resina son compatibles con los métodos volumétricos de EM, como la tomografía de matrices, FIB-SEM o SEM de cara de bloque en serie, mientras que las criosecciones no lo son. En los abordajes que realizan imágenes antes de la inclusión, las imágenes de células vivas son una opción41 que no está disponible en CLEM en criosecciones. La ventaja clave de CLEM en criosecciones sobre estas alternativas es la alta señal de FI, lo que permite la inmunolocalización de proteínas raras sin necesidad de permeabilización o sobreexpresión de la membrana. Esto evita la posible extracción de membranas, la sobreexpresión de artefactos42 y la modificación genética del sujeto, lo que, combinado con la posibilidad de correlacionar grandes áreas en FI y EM, lo convierte en una excelente herramienta para estudiar la LC3 y la autofagia.
Aquí, la aplicación de CLEM en sección a células HEPG2 hambrientas reveló que LC3 se localizaba predominantemente en estructuras identificadas como autofagosomas. Además, se encontraron algunas manchas débilmente fluorescentes en los autolisosomas. Esto contrasta directamente con las células tratadas con BafA19 y refleja la rápida degradación de las proteínas autofagosomales una vez que el autofagosoma se fusiona con los lisosomas. En general, los datos demostraron que el CLEM de criosecciones descongeladas puede proporcionar información sobre la autofagia mediada por LC3 en condiciones nativas. Los datos también ponen de manifiesto la sensibilidad de la tecnología, ya que la LC3 se detectó incluso en autolisosomas que contienen solo niveles bajos de epítopos de LC3 intactos. La aplicación adicional de esta técnica mediante la obtención de imágenes de LC3 en diferentes modelos y condiciones mejorará nuestra comprensión de la autofagia y otros procesos biológicos mediados por LC3, como la fagocitosis asociada a LC3 o la conjugación de ATG8 en membranas individuales.
Más allá de la autofagia, el CLEM en sección se puede aplicar a otros eventos o estructuras raras, como la división celular, la infección, los tipos de células raras en los tejidos, los cinetocoros, los cilios primarios o los orgánulos específicos del tipo celular. La detección efectiva del tema de interés por parte de IF puede facilitar en gran medida el estudio ultraestructural de estas rarezas. Además, se demostró14 que la técnica puede ser utilizada para localizar proteínas de una manera más sensible que la inmuno-EM clásica. El ajuste de la longitud de fijación puede ampliar aún más esta sensibilidad, lo que permite la localización ultraestructural de proteínas muy poco abundantes o poco antigénicas. Por último, el método CLEM en sección facilita la selección rápida de un número cuantitativo de orgánulos, lo que facilita un análisis más robusto de la distribución ultraestructural de una proteína determinada.
CLEM en criosecciones requiere el equipo y la experiencia para la criosección. En los grupos con acceso a estas herramientas (p. ej., criomicrótomos), la implementación de CLEM en sección es sencilla y solo requiere la disponibilidad de un microscopio automatizado de campo amplio, una configuración a la que la mayoría de los laboratorios tienen acceso. Además, el método está disponible en instalaciones de ME en todo el mundo. Dado que el CLEM en sección combina la aplicación de los métodos IF y EM establecidos, el método se adapta fácilmente y puede combinarse con, por ejemplo, la tomografía 20,33,43, el volumen de sección en serie EM de un número limitado de secciones 44 o la microscopía de superresolución 45. Esta versatilidad del método admite aplicaciones a una amplia gama de cuestiones biológicas.
Divulgaciones
Los autores declaran que no existen conflictos de intereses.
Agradecimientos
Agradecemos a nuestros colegas del Centro de Medicina Molecular del Centro Médico Universitario de Utrecht por sus fructíferas discusiones y comentarios. Agradecemos a los colegas pasados y presentes del laboratorio de Klumperman por realizar mejoras continuas en nuestras tecnologías de microscopía. La infraestructura EM utilizada para este trabajo forma parte del programa de investigación National Roadmap for Large-Scale Research Infrastructure (NEMI) financiado por el Consejo de Investigación Holandés (NWO), número de proyecto 184.034.014 a JK.
Materiales
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Antibody donkey anti-mouse Alexa Fluor 488 | Life Technologies | #A21202 | use 1:250 |
| Antibody donkey anti-rabbit Alexa Fluor 568 | Life Technologies | A#10042 | use 1:250 |
| Antibody mouse anti-LC3 | Cosmo Bio | CTB-LC3-2-IC | use 1:100 |
| Antibody rabbit anti-LAMP1 | Cell Signaling | 9091 | use 1:250 |
| Bovine serum Albumin, fraction V | Sigma-Aldrich | A-9647 | |
| BSA-c | Aurion | 900.099 | |
| BSA-conjugated gold | Cell Microscopy Core, UMC Utrecht | BSAG 5 nm | |
| Water-free Chloroform | Merck | 1.02447.0500 | |
| DAPI | Invitrogen | 10184322 | Use at end concentration of 10 µg/ml |
| EGTA | Sigma-Aldrich | E4378 | |
| Fish-skin Gelatin | Sigma-Aldrich | G7765 | |
| Food-grade gelatin | Merck | G1890 | |
| Formvar, Vinylec E | SPI | 02492-RA | |
| Gluteraldehyde | Serva | 23115.01 | See CAUTION note |
| Glycerol | Boom | MBAK 7044.1000 | |
| Glycine | Merck | 1042010250 | |
| HEPES | Sigma-Aldrich | H3375 | |
| Methylcellulose, 25 centipoises | Sigma-Aldrich | M-6385 | |
| MgSO4 | Riedel-de Haen | 12142 | |
| Na2HPO4 (PB component A) | Merck | 106580-0500 | |
| NaBH4 | Merck | 806373 | |
| NaH2PO4 (PB component B) | Merck | 106346 | |
| NH4OH | Sigma-Aldrich | 221228-0025 | |
| Oxalic acid | Merck | 100495 | |
| Paraformaldehyde prills | Sigma-Aldrich | 441244 | See CAUTION note |
| PIPES | Merck | 110220 | |
| Protein-A conjugated gold | Cell Microscopy Core, UMC Utrecht | PAG 5, 10, 15 or 20 nm | |
| Sucrose D(+) | VWR | 27483294 | |
| Uranyl acetate | SPI | 020624-AB | See CAUTION note |
| Tools and consumables | |||
| Pick-up loop | Electron Microscopy Sciences | 70944 | |
| Filter paper, qualitative, medium-fast | LLG | 6.242 668 | |
| Finder grids | Ted Pella | G100F1 | |
| Grids | Cell Microscopy Core, UMC Utrecht | CU 100 mesh | |
| Microscopes | |||
| Leica Thunder widefield microscope | Leica | Components: 100x, 1.47 NA TIRF objective; Photometrics prime 95B sCMOS camera; LAS X software; | |
| Leica UC7 ultracryomicrotome | Leica | ||
| Tecnai T12 | FEI | Components: Veleta VEL-FEI-TEC12-TEM camera; SerialEM software | |
| Software | |||
| ec-CLEM in icy | open source | Paul-Gilloteaux et al., 2017 | |
| Fiji | open source | Schindelin et al., 2012 | |
| IMOD | open source | Mastronarde et al., 2017 | |
| Photoshop | Adobe | ||
| SerialEM | open source | Mastronarde et al., 2018 |
Referencias
- Hu, Y., Reggiori, F. Molecular regulation of autophagosome formation. Biochemical Society Transactions. 50 (1), 55-69 (2022).
- Reggiori, F., Ungermann, C. Autophagosome maturation and fusion. Journal of Molecular Biology. 429 (4), 486-496 (2017).
- Kimura, S., Noda, T., Yoshimori, T. Dissection of the autophagosome maturation process by a novel reporter protein, tandem fluorescent-tagged LC3. Autophagy. 3 (5), 452-460 (2007).
- Baba, M., Takeshige, K., Baba, N., Ohsumi, Y. Ultrastructural analysis of the autophagic process in yeast: detection of autophagosomes and their characterization. The Journal of Cell Biology. 124 (6), 903-913 (1994).
- Takeshige, K., Baba, M., Tsuboi, S., Noda, T., Ohsumi, Y. Autophagy in yeast demonstrated with proteinase-deficient mutants and conditions for its induction. The Journal of Cell Biology. 119 (2), 301-311 (1992).
- De Duve, C., Wattiaux, R. Functions of lysosomes. Annual Review of Physiology. 28, 435-492 (1966).
- Arstila, A. U., Trump, B. F. Studies on cellular autophagocytosis. The formation of autophagic vacuoles in the liver after glucagon administration. The American Journal of Pathology. 53 (5), 687-733 (1968).
- Eskelinen, E. L., Reggiori, F., Baba, M., Kovács, A. L., Seglen, P. O. Seeing is believing: The impact of electron microscopy on autophagy research. Autophagy. 7 (9), 935-956 (2011).
- De Mazière, A., et al. An optimized protocol for immuno-electron microscopy of endogenous LC3. Autophagy. 18 (12), 3004-3022 (2022).
- López-Terrada, D., Cheung, S. W., Finegold, M. J., Knowles, B. B. Hep G2 is a hepatoblastoma-derived cell line. Human Pathology. 40 (10), 1512-1515 (2009).
- Tanida, I., Minematsu-Ikeguchi, N., Ueno, T., Kominami, E. Lysosomal turnover, but not a cellular level, of endogenous LC3 is a marker for autophagy. Autophagy. 1 (2), 84-91 (2005).
- Slot, J. W., Geuze, H. J. Cryosectioning and immunolabeling. Nature Protocols. 2 (10), 2480-2491 (2007).
- Waterman-Storer, C. M. Microtubule/organelle motility assays. Current Protocols in Cell Biology. 13 (1), (2001).
- vander Beek, J., de Heus, C., Liv, N., Klumperman, J. Quantitative correlative microscopy reveals the ultrastructural distribution of endogenous endosomal proteins. The Journal of Cell Biology. 221 (1), e202106044(2022).
- Mastronarde, D. N. Advanced data acquisition from electron microscopes with SerialEM. Microscopy and Microanalysis. 24, 864-865 (2018).
- Mastronarde, D. N., Held, S. R. Automated tilt series alignment and tomographic reconstruction in IMOD. Journal of Structural Biology. 197 (2), 102-113 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Paul-Gilloteaux, P., et al. EC-CLEM: Flexible multidimensional registration software for correlative microscopies. Nature Methods. 14 (2), 102-103 (2017).
- Heiligenstein, X., Paul-Gilloteaux, P., Raposo, G., Salamero, J. eC-CLEM: A multidimension, multimodel software to correlate intermodal images with a focus on light and electron microscopy. Methods in Cell Biology. 140, 335-352 (2017).
- Fermie, J., et al. Bimodal endocytic probe for three-dimensional correlative light and electron microscopy. Cell Reports Methods. 2 (5), 100220(2022).
- Fokkema, J., et al. Fluorescently labelled silica coated gold nanoparticles as fiducial markers for correlative light and electron microscopy. Scientific Reports. 8 (1), 13625(2018).
- Czaja, M. J., et al. Functions of autophagy in normal and diseased liver. Autophagy. 9 (8), 1131(2013).
- Robinson, J. M., Takizawa, T., Pombo, A., Cook, P. R. Correlative fluorescence and electron microscopy on ultrathin cryosections: Bridging the resolution gap. Journal of Histochemistry and Cytochemistry. 49 (7), 803-808 (2001).
- Mohammadian, S., et al. High accuracy, fiducial marker-based image registration of correlative microscopy images. Scientific Reports. 9 (1), 3211(2019).
- Tokuyasu, K. T. A study of positive staining of ultrathin frozen sections. Journal of Ultrasructure Research. 63 (3), 287-307 (1978).
- Slot, J. W., Geuze, H. J. A new method of preparing gold probes for multiple-labeling cytochemistry. European Journal of Cell Biology. 38 (1), 87-93 (1985).
- Klumperman, J., Raposo, G. The complex ultrastructure of the endolysosomal system. Cold Spring Harbor Perspectives in Biology. 6 (10), a016857(2014).
- Geuze, H. J., Slot, J. W., Strous, G. J., Lodish, H. F., Schwartz, A. L. Intracellular site of asialoglycoprotein receptor-ligand uncoupling: Double-label immunoelectron microscopy during receptor-mediated endocytosis. Cell. 32 (1), 277-287 (1983).
- Biazik, J., Ylä-Anttila, P., Vihinen, H., Jokitalo, E., Eskelinen, E. L. Ultrastructural relationship of the phagophore with surrounding organelles. Autophagy. 11 (3), 439-451 (2015).
- Fahimi, H. D., Reich, D., Völkl, A., Baumgart, E. Contributions of the immunogold technique to investigation of the biology of peroxisomes. Histochemistry and Cell Biology. 106 (1), 105-114 (1996).
- Vicidomini, G., et al. A novel approach for correlative light electron microscopy analysis. Microscopy Research and Technique. 73 (3), 215-224 (2010).
- Vicidomini, G., et al. High data output and automated 3D correlative light-electron microscopy method. Traffic. 9 (11), 1828-1838 (2008).
- Cortese, K., et al. 3D HDO-CLEM: cellular compartment analysis by correlative light-electron microscopy on cryosection. Methods in Cell Biology. 111, 95-115 (2012).
- van Rijnsoever, C., Oorschot, V., Klumperman, J. Correlative light-electron microscopy (CLEM) combining live-cell imaging and immunolabeling of ultrathin cryosections. Nature Methods. 5 (11), 973-980 (2008).
- Razi, M., Chan, E. Y. W., Tooze, S. A. Early endosomes and endosomal coatomer are required for autophagy. The Journal of Cell Biology. 185 (2), 305-321 (2009).
- Ligeon, L. A., Barois, N., Werkmeister, E., Bongiovanni, A., Lafont, F. Structured illumination microscopy and correlative microscopy to study autophagy. Methods. 75, 61-68 (2015).
- Biazik, J., Vihinen, H., Anwar, T., Jokitalo, E., Eskelinen, E. L. The versatile electron microscope: An ultrastructural overview of autophagy. Methods. 75, 44-53 (2015).
- Gudmundsson, S., Kahlhofer, J., Baylac, N., Kallio, K., Eskelinen, E. L. Correlative light and electron microscopy of autophagosomes. Methods in Molecular Biology. 1880, 199-209 (2019).
- Kriel, J., et al. Correlative light and electron microscopy (CLEM): bringing together the best of both worlds to study neuronal autophagy. Imaging and Quantifying Neuronal Autophagy. 171, 135-147 (2022).
- Largeau, C., Legouis, R. Correlative light and electron microscopy to analyze LC3 proteins in Caenorhabditis elegans embryo. Methods in Molecular Biology. 1880, 281-293 (2019).
- Fermie, J., et al. Single organelle dynamics linked to 3D structure by correlative live-cell imaging and 3D electron microscopy. Traffic. 19 (5), 354-369 (2018).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: Caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Ladinsky, M. S., Howell, K. E. Electron tomography of immunolabeled cryosections. Methods in Cell Biology. 79, 543-558 (2007).
- Oorschot, V., Lindsey, B. W., Kaslin, J., Ramm, G. TEM, SEM, and STEM-based immuno-CLEM workflows offer complementary advantages. Scientific Reports. 11 (1), 899(2021).
- Franke, C., et al. Correlative single-molecule localization microscopy and electron tomography reveals endosome nanoscale domains. Traffic. 20 (8), 601-617 (2019).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados