Method Article
Localizzazione ultrastrutturale di LC3 endogena mediante microscopia elettronico-ottica correlativa in sezione
In questo articolo
Riepilogo
Qui, presentiamo un protocollo per la microscopia elettronico-leggera correlativa ottimizzata in sezione basata sulla marcatura endogena e fluorescente come strumento per studiare la localizzazione di proteine rare in relazione all'ultrastruttura cellulare. La potenza di questo approccio è dimostrata dalla localizzazione ultrastrutturale di LC3 endogena in cellule affamate senza trattamento con bafilomicina.
Abstract
La visualizzazione degli organelli autofagici a livello ultrastrutturale mediante microscopia elettronica (EM) è essenziale per stabilire la loro identità e rivelare dettagli importanti per la comprensione del processo autofagico. Tuttavia, i metodi EM spesso mancano di informazioni molecolari, ostacolando la correlazione delle informazioni ultrastrutturali ottenute dall'EM con la localizzazione basata sulla microscopia a fluorescenza di specifiche proteine dell'autofagia. Inoltre, la rarità degli autofagosomi in condizioni cellulari inalterate ostacola l'indagine da parte dell'EM, che richiede un elevato ingrandimento, e quindi fornisce un campo visivo limitato.
In risposta a entrambe le sfide, è stato applicato un metodo di microscopia elettronica correlativa alla luce (CLEM) in sezione basato sulla marcatura fluorescente per correlare un marcatore autofagosomico comune, LC3, all'ultrastruttura EM. Il metodo è stato utilizzato per eseguire rapidamente lo screening delle cellule al microscopio a fluorescenza per la marcatura LC3 in combinazione con altri marcatori pertinenti. Successivamente, le caratteristiche ultrastrutturali sottostanti di punti selezionati marcati con LC3 sono state identificate mediante CLEM. Il metodo è stato applicato alle cellule affamate senza l'aggiunta di inibitori dell'acidificazione lisosomiale.
In queste condizioni, LC3 è stata trovata prevalentemente sugli autofagosomi e raramente negli autolisosomi, in cui LC3 è rapidamente degradata. Questi dati mostrano sia la fattibilità che la sensibilità di questo approccio, dimostrando che CLEM può essere utilizzato per fornire approfondimenti ultrastrutturali sull'autofagia mediata da LC3 in condizioni native, senza trattamenti farmacologici o alterazioni genetiche. Nel complesso, questo metodo rappresenta uno strumento prezioso per gli studi di localizzazione ultrastrutturale delle proteine dell'autofagia e di altri antigeni scarsi, collegando la microscopia ottica ai dati EM.
Introduzione
L'autofagia è un processo chiave per la clearance e il riciclaggio delle proteine citoplasmatiche e degli organelli. Il processo di macro-autofagia (di seguito chiamato autofagia) comporta la formazione di organelli a doppia membrana, autofagosomi, che consentono alle cellule di racchiudere molecole citoplasmatiche e organelli per la degradazione lisosomiale. L'autofagia si verifica a livello basale nella maggior parte delle cellule ed è sovraregolata in risposta a condizioni cellulari, come la fame o lo stress cellulare. L'autofagia si verifica in modo specifico del substrato, mirando a strutture o proteine specifiche per la degradazione, o come un processo di massa non selettivo che comprende parti del citosol. Nell'autofagia selettiva, gli autofagosomi sono formati dalla coniugazione di proteine della famiglia Atg8 (proteine associate ai microtubuli 1A/B a catena leggera 3A/B/C [LC3] e GABARAPs) a membrane derivate dal riciclaggio degli endosomi, del Golgi e/o del reticolo endoplasmatico (ER)1. LC3 riconosce il carico autofagico nel citosol direttamente o tramite adattatori selettivi per l'autofagia come P62/SQSTM. Le nuove membrane autofagiche possono quindi essere coniugate a LC3, espandersi e fondersi per formare una doppia membrana completa che racchiude il carico, chiamato autofagosoma. L'autofagosoma matura e alla fine si fonde con un endosoma o un lisosoma, dopodiché il carico autofagico e gli adattatori vengono degradati2.
Gli studi sulla formazione, la maturazione e la fusione degli autofagosomi spesso si avvalgono di tecnologie di microscopia ottica. La microscopia a fluorescenza di LC3 è generalmente utilizzata per valutare il numero e la localizzazione cellulare degli autofagosomi in diverse condizioni. Inoltre, accoppiando LC3 a GFP sensibile al pH e RFP stabile a pH in una cosiddetta sonda tandem, il flusso complessivo di autofagia può essere misurato nelle cellule vive in funzione della perdita di fluorescenza GFP3. Questi approcci sono strumenti preziosi per i ricercatori per comprendere il ruolo e il meccanismo dell'autofagia in diverse condizioni. Un altro strumento prezioso è la microscopia elettronica (EM), che rivela l'ultrastruttura degli organelli autofagici in diversi stadi dell'autofagia 4,5,6,7,8. Ad oggi, l'EM è ancora il metodo di scelta per identificare gli stadi precisi della formazione dell'autofagosoma discriminando diverse membrane autofagiche in base alla morfologia: fagoforo (doppia membrana non completamente chiusa), autofagosoma (doppia membrana chiusa attorno al carico citosolico) e autolisosoma (perdita [parziale] della membrana autofagica interna). La morfologia senza informazioni molecolari, tuttavia, può essere soggetta a errori di identificazione o ambiguità. Immuno-EM è il metodo più completo per la caratterizzazione molecolare simultanea e la classificazione morfologica degli organelli autofagici. Ad esempio, la marcatura immunogold di LC3 su criosezioni scongelate consente la localizzazione ultrastrutturale di LC3 e l'identificazione precisa degli organelli marcati con LC39.
Uno svantaggio dell'EM è il piccolo campo visivo che viene fornito con l'elevato ingrandimento richiesto per osservare la fine ultrastruttura delle membrane autofagiche e, nel caso dell'immuno-EM, per individuare l'etichetta che contrassegna la proteina di interesse. A causa della loro scarsità e dei bassi livelli di proteine, questo generalmente ostacola l'analisi quantitativa EM degli autofagosomi. Per aumentare il numero di autofagosomi, le cellule vengono spesso affamate e trattate con bafilomicina A1 (BafA1), un inibitore dell'acidificazione e della degradazione lisosomiale. Senza il trattamento con BafA1, la ricerca di autofagosomi da parte dell'EM è dispendiosa in termini di tempo, a causa della scarsità di questi organelli. Il metodo presentato in questo manoscritto affronta questo problema attraverso la marcatura fluorescente e l'imaging di LC3 endogena su criosezioni scongelate in un microscopio a fluorescenza prima di un'ulteriore preparazione per EM. Le immagini fluorescenti guidano quindi la ricerca di strutture marcate con LC3 nell'EM. Dopo la raccolta, le immagini EM vengono correlate con le immagini di fluorescenza per aggiungere informazioni molecolari - la presenza di LC3 - all'ultrastruttura della cellula. Questo metodo "on-section CLEM" aumenta notevolmente la capacità di trovare strutture marcate con LC3, specialmente in condizioni non trattate, per la successiva identificazione e classificazione mediante EM.
Questo metodo è stato applicato a cellule HEPG210 derivate da epatoblastoma affamate per trovare autofagosomi in condizioni inalterate (cioè non è stato utilizzato BafA1). Sono stati trovati relativamente pochi punti fluorescenti (meno di uno per profilo cellulare in una sezione di 90 nm), il che è in accordo con l'elevato turnover di LC311. Questa scarsità di LC3-puncta ha enfatizzato il valore di CLEM; selezionando regioni con diversi punti fluorescenti per l'imaging nell'EM, gli organelli LC3-positivi sono stati trovati e caratterizzati in modo molto più efficace rispetto all'immuno-EM convenzionale. Ciò ha rivelato che la maggior parte degli organelli LC3-positivi erano autofagosomi, come definito dalla loro morfologia, il che è in contrasto con i risultati ottenuti nelle cellule trattate con BafA1, dove gli autolisosomi sono più comuni9. Questi dati mostrano che con CLEM in sezione, l'autofagia può essere studiata a livello ultrastrutturale senza la necessità di inibire il flusso autofagico.
Protocollo
1. Preparazione di strumenti e reagenti
NOTA: per i reagenti, i tamponi e le soluzioni necessari, vedere File supplementare 1 o 12 per ulteriori informazioni. Per i dettagli relativi a tutti i materiali, i reagenti, le apparecchiature e il software utilizzati in questo protocollo, vedere la Tabella dei materiali.
- Fissativi
- Preparare un tampone fosfato (PB) da 0,2 M o un tampone da 0,2 M PIPES, HEPES, EGTA, MgSO4 (PHEM), come descritto nel file supplementare 1, da utilizzare come base per soluzioni fissative.
NOTA: I fissativi vengono regolarmente tamponati in tampone PB o PHEM da 0,1 M per tamponare l'acidificazione causata dalla reazione dell'aldeide con il materiale biologico. - Poiché la qualità della paraformaldeide (PFA) è fondamentale per una fissazione affidabile dell'ultrastruttura dei campioni, utilizzare PFA di grado EM. Per seguire questo protocollo, utilizzare soluzioni madre al 16%, preparate con prill PFA di alta qualità (vedi Scheda supplementare 1).
ATTENZIONE: La paraformaldeide è una sostanza chimica pericolosa (indicazioni di pericolo H228, H301, H302, H311, H314, H315, H317, H318, H331, H332, H335, H341, H350). Quando si lavora con PFA, indossare dispositivi di protezione (guanti, camice da laboratorio e occhiali protettivi) e lavorare in una cappa chimica. I rifiuti che contengono PFA devono essere raccolti e smaltiti secondo le linee guida e i regolamenti degli istituti. - Combinare 10 mL di PBA 0,2 M, 5 mL di PFA al 16% (in acqua demineralizzata [dH 2 O]) e5 mL di dH2O per preparare una soluzione fissativa di PFA al 4%.
- Opzionale: l'aggiunta di glutaraldeide (GA) allo 0,02%-0,5% alla soluzione fissativa del passaggio 1.1.3 migliora la conservazione dell'ultrastruttura, ma riduce l'antigenicità del campione nei confronti di molti anticorpi.
NOTA: Quando si desidera il fissaggio GA, utilizzare GA di grado EM di un fornitore adatto.
ATTENZIONE: La glutaraldeide è una sostanza chimica pericolosa (indicazioni di pericolo H301, H302, H314, H317, H330, H332, H334, H335, H400, H411). Lavorare in una cappa chimica e indossare dispositivi di protezione (guanti, camice da laboratorio e occhiali protettivi) quando si manipola GA. I rifiuti contenenti GA devono essere raccolti e smaltiti secondo le linee guida e i regolamenti degli istituti.
- Preparare un tampone fosfato (PB) da 0,2 M o un tampone da 0,2 M PIPES, HEPES, EGTA, MgSO4 (PHEM), come descritto nel file supplementare 1, da utilizzare come base per soluzioni fissative.
- Strumenti e materiali
- Grattare la superficie dei perni portacampioni in alluminio e sonicarli in etanolo 3 x 10 minuti per rimuovere i residui metallici e garantire un'aderenza ottimale quando i blocchi di cellule incorporati in gelatina sono montati sui perni.
- Utilizzare un contenitore di stoccaggio adatto a conservare i perni portacampioni in alluminio con i loro campioni in azoto liquido (LN2).
- Crea un manipolatore apponendo un singolo capello o ciglia all'estremità di uno spiedino di legno usando lo smalto.
- Crea un ciclo di pickup. Piegare un filo di acciaio inossidabile di 0,3 mm di spessore attorno a una barra tonda di 3 mm di diametro e attorcigliare le estremità insieme, formando un anello su un'estremità. Inserire le estremità attorcigliate in un puntale per pipetta. Inserire uno spiedino di legno dall'altra estremità e fissarlo con colla o resina.
NOTA: È disponibile in commercio anche un anello di prelievo (vedere la tabella dei materiali). - Per preparare i circuiti di asciugatura a griglia, seguire gli stessi passaggi per realizzare gli anelli di prelievo: formare un filo di acciaio inossidabile in un anello di 4 mm e fissarlo su un puntale di pipetta grande con colla o resina.
- Rivestire le griglie utilizzando una pellicola di supporto sottile come formvar (protocollo nel file supplementare 1). Prima dell'uso, rivestire le griglie con un sottile strato di carbonio.
NOTA: Le griglie pronte all'uso sono disponibili in commercio (vedi Tabella dei materiali). Le griglie rivestite in Formvar possono essere conservate a tempo indeterminato a temperatura ambiente (RT); Le griglie rivestite in carbonio possono essere conservate per diversi mesi presso RT. - Preparare vetrini puliti e vetrini coprioggetti di grandi dimensioni (24 mm x 24 mm è l'ideale con vetrini larghi 25 mm), come in 13.
2. Fissazione e preparazione del campione
- Fissazione
- Utilizzare il fissativo preparato al punto 1.1.3 (4% PFA in 0,1 M PB). Per le linee cellulari aderenti, coltivare 1-5 × 10 6 cellule in piastre da6 cm. Aggiungere il fissativo al terreno di coltura in rapporto 1:1 e incubare il campione per 5 minuti a RT. Quindi, sostituire la miscela media-fissativa con solo fissativo e incubare per 2 ore a RT.
NOTA: Il numero esatto di cellule, la confluenza e le condizioni di coltura possono variare a seconda del sistema modello utilizzato. - Conservare i campioni per una notte o per un massimo di 3-4 settimane in PFA allo 0,5% in 0,1 M PB a 4 °C.
NOTA: GA può essere aggiunto alla fissazione (vedere il passaggio 1.1.4) e la lunghezza della fissazione può essere modificata per trovare un equilibrio ottimale tra la conservazione della morfologia e l'antigenicità, che differisce a seconda del campione e dell'etichettatura. Per ulteriori informazioni, vedere14.
- Utilizzare il fissativo preparato al punto 1.1.3 (4% PFA in 0,1 M PB). Per le linee cellulari aderenti, coltivare 1-5 × 10 6 cellule in piastre da6 cm. Aggiungere il fissativo al terreno di coltura in rapporto 1:1 e incubare il campione per 5 minuti a RT. Quindi, sostituire la miscela media-fissativa con solo fissativo e incubare per 2 ore a RT.
- Incorporamento di campioni
- Lavare il piatto con celle fisse 3 volte con PBS a RT. Quindi, sostituire con PBS contenente lo 0,15% di glicina e incubare per 10 minuti a RT.
- Sostituire il PBS contenente lo 0,15% di glicina con l'1% di gelatina nel PBS preriscaldato a 37 °C, quindi raschiare e trasferire le cellule in gelatina all'1% in una provetta per microcentrifuga. Pellet le cellule a 6.000 × g per 1 minuto a RT in una microcentrifuga. Quindi, rimuovere la gelatina all'1% senza disturbare il pellet e aggiungere la gelatina al 12% riscaldata a 37 °C. Risospendere il pellet cellulare pipettando delicatamente su e giù con puntali per pipette o pipette Pasteur in vetro preriscaldate a 37 °C.
- Incubare a 37 °C per 10 min; Quindi, pellettizzare le cellule a 6.000 × g per 1 minuto. Solidificare la gelatina con ghiaccio per 30 min.
- Per rimuovere le cellule incorporate nella gelatina dal tubo, tagliare l'estremità del tubo contenente il pellet dal resto del tubo con una lama di rasoio. Quindi, perpendicolarmente al primo taglio, tagliare a metà l'estremità del tubo con il pellet cellulare.
- Incubare le due metà delle estremità del tubo contenenti il pellet cellulare incorporato nella gelatina in saccarosio 2,3 M per 10 minuti a 4 °C. Ciò fa sì che le metà del pellet cellulare incorporato nella gelatina si restringano leggermente e si stacchino dal tubo di plastica.
NOTA: I pellet cellulari incorporati nella gelatina devono essere mantenuti a 4 °C o il più possibile ghiacciati per evitare che il saccarosio 2,3 M diventi troppo viscoso e la gelatina troppo morbida. Durante la manipolazione dei pellet cellulari incorporati nella gelatina nelle fasi successive, lavorare con un solo campione alla volta e mantenere gli altri in ghiaccio, oppure lavorare in una stanza fredda (~4 °C). Evitare il surriscaldamento dei pellet cellulari incorporati nella gelatina a causa della luce solare, delle lampade calde del microscopio o di altre fonti di calore. - Rimuovere le metà del tubo con il pellet cellulare incorporato nella gelatina dal saccarosio 2,3 M. Quindi, rimuovere le metà del pellet cellulare incorporato nella gelatina dalle metà del tubo di plastica con una pinzetta. Tagliare manualmente il pellet in blocchi di dimensioni adeguate (~1 mm3) con una lama di rasoio. Utilizzare uno stereomicroscopio da dissezione per ingrandire il soggetto durante il taglio.
- Infondere i blocchi cellulari incorporati nella gelatina con saccarosio 2,3 M per 3-16 ore, girando da un capo all'altro in un rotore a 4 °C.
- Posizionare un blocco di cellule incorporato nella gelatina su un perno portacampioni in alluminio (vedere il passaggio 1.2.1). Lasciare una quantità sufficiente di saccarosio 2,3 M intorno ai bordi del blocco in modo che formi un sottile "collare" tra il blocco e il perno. Evitare che troppo saccarosio 2,3 M ricopra la parte superiore del blocco. Congelare a scatto e conservare in LN2.
3. Sezionamento
- Rifilatura (vedi anche12)
- Prelevare uno spillo con un blocco di cellule incorporate nella gelatina dalla conservazione di LN2 e posizionarlo all'interno di un criomicrotomo impostato a -80 °C.
- Tagliare la parte anteriore del blocco per appiattirne la superficie e ottenere sezioni di ~250 nm. Immergere l'ansa da 3 mm nella soluzione di prelievo (saccarosio 1:1 2,3 M e metilcellulosa al 2%), inserire l'ansa nella criocamera del microtomo e attendere che inizi a formarsi ghiaccio nella gocciolina (in genere 5-7 s). Quindi, prelevare immediatamente la sezione premendo rapidamente ma delicatamente la gocciolina contro di essi. Rimuovere l'anello dalla criocamera, attendere che la gocciolina si sia completamente scongelata e premere la goccia su un vetrino.
- Controllare l'orientamento delle cellule colorando le sezioni con il blu di toluidina.
- Mettere una goccia di soluzione di blu di toluidina (vedere Scheda supplementare 1) sopra le sezioni su un vetrino e asciugare su una piastra riscaldante a 80 °C fino a quando i bordi della goccia non sono asciutti.
- Rimuovere il vetrino dalla piastra riscaldante e sciacquare delicatamente il blu di toluidina con dH2O, raccogliendolo in un apposito contenitore per rifiuti.
- Asciugare il vetrino e controllare l'orientamento delle cellule nelle sezioni attraverso un semplice microscopio ottico da banco.
- Tagliare i lati del blocco sezionando 50-100 μm sul lato della faccia del blocco campione con l'angolo del coltello. Tagliare quattro lati della faccia del blocco del campione ruotando il portacampioni di 90° dopo aver tagliato ciascun lato per creare un rettangolo sporgente di ~250 μm x 375 μm. Selezionate l'area sporgente in base all'orientamento della cella determinato nel passo precedente.
- Sezionamento e prelievo
- Raffreddare il criomicrotomo a -100 °C. Sezionare un nastro dal rettangolo sporgente, le sezioni hanno uno spessore di 70-90 nm e una lucentezza argentea-dorata. Allontanare le sezioni dal bordo del coltello diamantato con un capello su un bastoncino (vedere la sezione 1.2.3) per creare un nastro lungo (2-5 mm).
- Una volta formato un nastro adatto, interrompere il sezionamento per prelevare il nastro. Immergere l'anello di prelievo da 3 mm in saccarosio 2,3 M e metilcellulosa al 2% mescolati 1:1, inserire l'anello nella criocamera del microtomo e attendere che la gocciolina inizi a congelarsi (in genere 5-7 secondi). Quindi, raccogli immediatamente le sezioni premendo rapidamente ma delicatamente la goccia contro di esse. Rimuovere l'anello dalla criocamera, attendere che la gocciolina si sia completamente scongelata e premere la goccia su una griglia preparata (passaggio 1.2.6).
NOTA: Le griglie con sezioni possono essere conservate a 4 °C per diversi mesi.
4. Etichettatura e microscopia ottica
- Etichettatura
- Posizionare le griglie con le sezioni (Figura 1A) rivolte verso il basso su ~1 mL di PBS in una piccola piastra o in una piastra a più pozzetti. Incubare a 37 °C per 30 min.
NOTA: Questo passaggio rimuove la gelatina che si trova tra le celle; La gelatina non è necessaria dopo il sezionamento e interferisce con il protocollo rimanente. - Trattare le griglie con il lato della sezione rivolto verso il basso su goccioline da ~75 μL sul parafilm (vedere la Figura 1B). Iniziare con lavaggi PBS + glicina allo 0,15% (3 x 2 min) a RT. Quindi, incubare le griglie con albumina sierica bovina (BSA)-c allo 0,1% + gelatina di pelle di pesce (FSG) allo 0,5% in PBS per 10 minuti a RT come fase di blocco. Diluire gli anticorpi primari in BSA-c allo 0,1% + FSG allo 0,5% in PBS e incubare le griglie su goccioline di ~10 μL di questa soluzione per 1 ora a RT (Figura 1C).
- Lavare le griglie allo 0,1% BSA in PBS 5x a RT. Quindi, diluire gli anticorpi secondari e il 4',6-diamidino-2-fenilindolo (DAPI; 10 μg/mL) in 0,1% BSA-c + 0,5% FSG in PBS e incubare le griglie su goccioline ~10 μL di questa soluzione per 30+ minuti a RT (Figura 1C). Lavare le griglie in PBS 5x a RT.
NOTA: Opzionalmente, un anticorpo secondario può essere marcato con particelle di oro colloidale da 5, 10, 15 o 20 nm coniugate alla proteina A (PAG) per la localizzazione della proteina di interesse nell'EM. Se lo si desidera, incubare le griglie con PAG per 20 minuti a RT dopo il passaggio 4.1.3. Quindi, lavare 5 volte con PBS a RT. Evitare l'uso simultaneo di più anticorpi primari e prendere nota della reattività del PAG alle IgG di specie diverse per prevenire reazioni crociate indesiderate. Per ulteriori informazioni, vedere12.
- Posizionare le griglie con le sezioni (Figura 1A) rivolte verso il basso su ~1 mL di PBS in una piccola piastra o in una piastra a più pozzetti. Incubare a 37 °C per 30 min.
- Montaggio di campioni per microscopia ottica
- Immergere le griglie in glicerolo al 50% in dH 2 O2x 5 min a RT. Inserire le griglie tra un vetrino e un vetrino coprioggetto in glicerolo al 50%, una griglia per vetrino, con le sezioni rivolte verso il vetrino coprioggetto (Figura 1D).
NOTA: La qualità dell'etichettatura può deteriorarsi quando le griglie vengono mantenute montate in glicerolo al 50% per più di 30 minuti. Si consiglia quindi di montare e fotografare due o tre griglie alla volta e lasciare le altre sulla soluzione di etichettatura secondaria.
- Immergere le griglie in glicerolo al 50% in dH 2 O2x 5 min a RT. Inserire le griglie tra un vetrino e un vetrino coprioggetto in glicerolo al 50%, una griglia per vetrino, con le sezioni rivolte verso il vetrino coprioggetto (Figura 1D).
- Microscopia ottica
- Portare un vetrino con griglie a sandwich su un microscopio a campo largo con un tavolino automatizzato. Selezionare un obiettivo per olio ad alto ingrandimento (63x o 100x). Creare un set di riquadri immagine di (parte di) la barra multifunzione di sezioni (Figura 1E).
NOTA: Alcuni anticorpi secondari possono formare aggregati fluorescenti sulle griglie, specialmente intorno a pieghe o lacerazioni nelle sezioni. Inoltre, alcuni tipi di cellule e tessuti contengono strutture autofluorescenti. Se si prevedono tali problemi, si consiglia di includere una griglia di controllo negativo non incubata con anticorpi primari.
- Portare un vetrino con griglie a sandwich su un microscopio a campo largo con un tavolino automatizzato. Selezionare un obiettivo per olio ad alto ingrandimento (63x o 100x). Creare un set di riquadri immagine di (parte di) la barra multifunzione di sezioni (Figura 1E).
- Smontaggio e contrasto EM
- Aggiungere 10 μL di dH2O sul lato del sandwich vetrino-vetrino coprioggetti e attendere che l'azione capillare riempia l'interfaccia vetro-vetrino. Rimuovere con cautela il vetrino coprioggetti senza mescolare l'olio da immersione nel glicerolo. Recupera le griglie con una pinzetta e immergi in dH2O 3x a RT per lavare via il glicerolo al 50%.
NOTA: L'olio può interferire con la colorazione dell'uranile e deteriorare il contrasto EM. - Asciugare accuratamente il retro della griglia con carta velina priva di lanugine.
NOTA: Se il campione è stato etichettato anche con particelle di oro colloidale, eseguire i seguenti passaggi: posizionare la griglia con le sezioni con il lato della sezione rivolto verso il basso sulle goccioline di PBS e lavare 2 volte a RT. Postfix all'1% GA per 5 minuti a RT (vedere la nota di attenzione al punto 1.1.4). Lavare in PBS 2 volte a RT. - Posizionare le griglie con il lato rivolto verso il basso sulle goccioline di dH2O e lavare 8 volte a RT.
- Per colorare le sezioni per il contrasto in EM, incubare con acetato di uranile (UA), pH 7, per 5 minuti a RT (Figura 1F).
- Prima di posizionare le griglie, raffreddare l'UA:metilcellulosa, pH 4, posizionando le goccioline sul parafilm su una piastra metallica su ghiaccio. Quindi, lavare le griglie con UA:metilcellulosa ghiacciata, pH 4, 2x e incubare con UA:metilcellulosa ghiacciata, pH 4, per 10 minuti (Figura 1F).
ATTENZIONE: L'acetato di uranile è una sostanza chimica pericolosa (indicazioni di pericolo H300, H330, H373, H411). Nei passaggi che richiedono UA, lavorare in una cappa chimica e indossare dispositivi di protezione (camice da laboratorio, guanti e occhiali protettivi). Raccogliere e smaltire i rifiuti contenenti UA secondo le linee guida e i regolamenti degli istituti. - Avvolgere le griglie inserendo un anello di asciugatura della griglia nella goccia di metilcellulosa UA sotto la griglia e sollevandola delicatamente fino a quando la griglia non viene estratta dalla gocciolina12. Tamponare l'eccesso di UA:metilcellulosa toccando l'anello con un angolo di ~60° (sezioni rivolte verso il basso) su carta da filtro priva di lanugine (vedere la tabella dei materiali) e trascinandolo lentamente lungo la carta fino a quando non viene assorbito più UA:metilcellulosa. Quindi, posizionare l'anello con la griglia in una griglia adatta e lasciare asciugare per >10 minuti a RT (Figura 1G).
- Aggiungere 10 μL di dH2O sul lato del sandwich vetrino-vetrino coprioggetti e attendere che l'azione capillare riempia l'interfaccia vetro-vetrino. Rimuovere con cautela il vetrino coprioggetti senza mescolare l'olio da immersione nel glicerolo. Recupera le griglie con una pinzetta e immergi in dH2O 3x a RT per lavare via il glicerolo al 50%.
5. EM
- Utilizzare la panoramica ottenuta dalla microscopia ottica per individuare una regione di interesse (ROI) per l'imaging nel microscopio elettronico a trasmissione (TEM; Figura 1H). Annota il ROI sul set di dati di microscopia ottica. Una volta selezionata un'area, ottenere un set di riquadri immagine con un ingrandimento di 20.000x-50.000x nel TEM. Ricostruire il tileset di immagini nel software di post-elaborazione15,16.
6. Correlazione e analisi
- Caricare la microscopia ottica e il set di dati EM in un software di elaborazione delle immagini adatto, come ImageJ/Fiji17, il plug-in ec-CLEM in Icy18 o Photoshop. Ritaglia e ruota il set di dati della microscopia ottica in modo che corrisponda al tileset EM.
- Eseguire la correlazione basata sul segnale DAPI in fluorescenza e sui contorni nucleari in EM (Figura 1I). Sposta le immagini per sovrapporle con precisione ed eseguire la correlazione manuale in modo accurato. Per rendere l'approccio più preciso, applica la correlazione basata sui punti di riferimento attraverso, ad esempio, il plug-in ec-CLEM in Icy o il plug-in BigWarp in ImageJ, per correlare le immagini attraverso la selezione manuale dei punti corrispondenti. È disponibile un protocollo dettagliato e passo-passo per la correlazione con ec-CLEM19.
NOTA: Questo approccio funziona bene anche con l'uso di sonde fiduciali bimodali20,21.
- Eseguire la correlazione basata sul segnale DAPI in fluorescenza e sui contorni nucleari in EM (Figura 1I). Sposta le immagini per sovrapporle con precisione ed eseguire la correlazione manuale in modo accurato. Per rendere l'approccio più preciso, applica la correlazione basata sui punti di riferimento attraverso, ad esempio, il plug-in ec-CLEM in Icy o il plug-in BigWarp in ImageJ, per correlare le immagini attraverso la selezione manuale dei punti corrispondenti. È disponibile un protocollo dettagliato e passo-passo per la correlazione con ec-CLEM19.
- Analizzare le immagini correlate selezionando i ROI in base al segnale fluorescente in un programma adatto (ad esempio, ImageJ). Per l'analisi quantitativa, creare una raccolta di ROI per tutti gli organelli etichettati. Quindi, ispezionare l'ultrastruttura corrispondente delle singole ROI e classificarle in base agli elementi morfologici.
Risultati
Un protocollo immuno-EM ottimizzato per la marcatura immuno-gold di LC3 su criosezioni ultrasottili è stato recentemente pubblicato da De Maziere et al.9. Questo studio ha incluso condizioni di fame senza BafA1, in cui LC3 era presente ma relativamente raro e difficile da trovare nei campi elettromagnetici. In uno studio separato è stato introdotto un metodo CLEM in sezione, che utilizza la sensibilità della marcatura a fluorescenza per visualizzare proteine endogene relativamente rare e a bassa espressione e correlarle all'ultrastruttura EM14. In questo caso, questi due approcci sono combinati dall'uso del protocollo di etichettatura LC3 ottimizzato come parte di un approccio CLEM.
Le cellule HEPG2, cellule derivate dal fegato con livelli relativamente elevati di autofagia basale22, sono state affamate in un terreno minimo (soluzione salina bilanciata di Earle [EBSS]) per 2 ore prima della fissazione in PFA al 4%. A ciò è seguita la preparazione del campione con il metodo Tokuyasu di criosezione ultrasottile (sezioni 1-3; vedi Slot e Geuze12), che è altamente compatibile con CLEM 14,23 in sezione. Le criosezioni scongelate sono state marcate in fluorescenza (sezione 4 del protocollo e Figura 1) utilizzando l'anticorpo primario anti-LC3 di topo9. Inoltre, l'anti-LAMP1 di coniglio è stato utilizzato per indicare gli endo-lisosomi, seguito dagli anticorpi secondari anti-topo AlexaFluor488 e anti-coniglio AlexaFluor568. Le griglie sono state inserite tra un vetrino coprioggetto e un vetrino e sono state riprese a RT su un microscopio a campo largo (obiettivo a olio 100x 1,47 NA, fotocamera sCMOS).
Un vantaggio della marcatura a fluorescenza di sezioni sottili rispetto all'IF convenzionale a cellula intera è la maggiore risoluzione in Z, poiché lo spessore fisico della sezione è di 60-90 nm. Con questa migliore risoluzione Z, la marcatura della fluorescenza di LC3 e LAMP1 su sezioni sottili rivela pochissima colocalizzazione (Figura 2A). Nelle cellule trattate con inibitori lisosomiali, come BafA1, si verifica un'elevata colocalizzazione, poiché la LC3 racchiusa nei lisosomi rimane inalterata9. Nelle cellule non trattate, LC3 si degrada rapidamente al contatto con lisosomi enzimaticamente attivi, LAMP1-positivi, e quindi la co-localizzazione è rara in queste condizioni. In generale, è stato osservato meno di un punto LC3 per profilo cellulare. Ciò indica che anche in condizioni di fame, il turnover degli autofagosomi è rapido, mantenendo basso il numero di autofagosomi. Evidenzia inoltre l'importanza di utilizzare CLEM per trovare le rare strutture marcate con LC3, utilizzando l'ampio campo visivo fornito dalla microscopia ottica. Inoltre, la maggiore sensibilità della marcatura a fluorescenza rispetto alla marcatura in oro consente l'identificazione di un maggior numero di organelli positivi per LC3 rispetto all'immuno-EM convenzionale, favorendo ulteriormente la loro caratterizzazione.
Dopo aver acquisito un tileset completo del nastro di sezioni, le griglie sono state recuperate dal microscopio e post-colorate per EM utilizzando UA e il metodo loop-out (passaggi del protocollo 4.4-4.6; Figura 1F,G). Questo metodo "loop-out" assicura che un sottile strato di UA:metilcellulosa rimanga sulla griglia, creando il contrasto desiderato nell'EM. Lo spessore dello strato dipende dalla velocità e dall'angolo con cui l'UA:metilcellulosa viene cancellata sulla carta da filtro. Trascinare il loop troppo velocemente può lasciare troppa UA:metilcellulosa sulla griglia e scurire l'aspetto delle sezioni in EM. Un trascinamento troppo lento può allontanare troppa UA:metilcellulosa, con conseguente scarsa colorazione e scarsa morfologia, e rischia che la griglia non esca dal circuito. La colorazione "chiazza d'olio" (Figura 1G) su griglie asciutte indica uno spessore dello strato UA:metilcellulosa adatto.
Dopo il loop-out e l'essiccazione, le griglie sono state visualizzate in un TEM a ROI selezionati mediante fluorescenza. I set di dati IF ed EM sono stati correlati sovrapponendo il segnale DAPI ai contorni dei nuclei visibili nell'EM, generando un'immagine integrata contenente informazioni di entrambe le modalità.
Trovare la stessa area in EM selezionata in IF può essere difficile. Si consiglia quindi di tenere a portata di mano un'immagine panoramica del tileset IF durante la ricerca nell'EM. Gli utenti dovrebbero cercare caratteristiche riconoscibili in entrambe le modalità, come pieghe o strappi nelle sezioni, barre della griglia o disposizione dei nuclei. È anche importante tenere presente che il campione può apparire ruotato e specchiato in EM. Le "griglie di ricerca" con caratteristiche specifiche per identificare le aree possono essere utilizzate per facilitare la correlazione (vedi Tabella dei materiali).
La correlazione degli organelli LC3-positivi con l'ultrastruttura EM ha rivelato che i diversi punti rappresentavano stadi distinti di autofagia (Figura 2B). Sebbene la conservazione dell'ultrastruttura autofagosomica sia impegnativa nelle criosezioni, sono stati frequentemente osservati organelli con contenuto citoplasmatico e doppie membrane (Figura 2C, frecce negli organelli 1-5; Figura supplementare S1), che definiscono le caratteristiche morfologiche degli autofagosomi. È interessante notare che macchie fluorescenti piuttosto deboli sono state identificate da EM come autolisosomi LC3-positivi (Figura 2C, organello 6; il contenuto autofagico è contrassegnato *), caratterizzati da contenuto denso e vescicole intraluminali. Ciò ha mostrato che quantità molto piccole di LC3 sono visibili nell'IF di criosezioni ultrasottili e ha indicato che, nonostante l'ambiente degradativo, alcune LC3 sono rilevabili negli autolisosomi allo stato stazionario. Tuttavia, la maggior parte dei punti LC3-positivi rappresentava autofagosomi, mentre gli autolisosomi erano molto rari. Ciò è in contrasto con le cellule trattate con BafA1, che accumulano principalmente autolisosomi e non autofagosomi9.
In sintesi, questo protocollo descrive un metodo CLEM in sezione per collegare le informazioni molecolari ottenute dalla microscopia a fluorescenza all'ultrastruttura dell'EM. Questo metodo aumenta la sensibilità dell'immuno-EM, poiché per la marcatura vengono utilizzati solo fluorofori che generalmente producono più segnale rispetto alle sonde EM. Il metodo è particolarmente adatto per l'utilizzo di criosezioni ultrasottili, in cui è possibile ottenere alti livelli di fluorescenza specifica su una colorazione di fondo trascurabile. Utilizzando la fluorescenza per lo screening di strutture o eventi rari e correlando le ROI selezionate ai campi elettromagnetici, è possibile ridurre notevolmente il tempo di funzionamento dei mercati emergenti e i costi associati. La sensibilità e la fattibilità del metodo è dimostrata dalla visualizzazione di LC3 in cellule non trattate e affamate, mostrando che LC3 si associa prevalentemente agli autofagosomi in queste condizioni, con livelli molto bassi visibili negli autolisosomi.
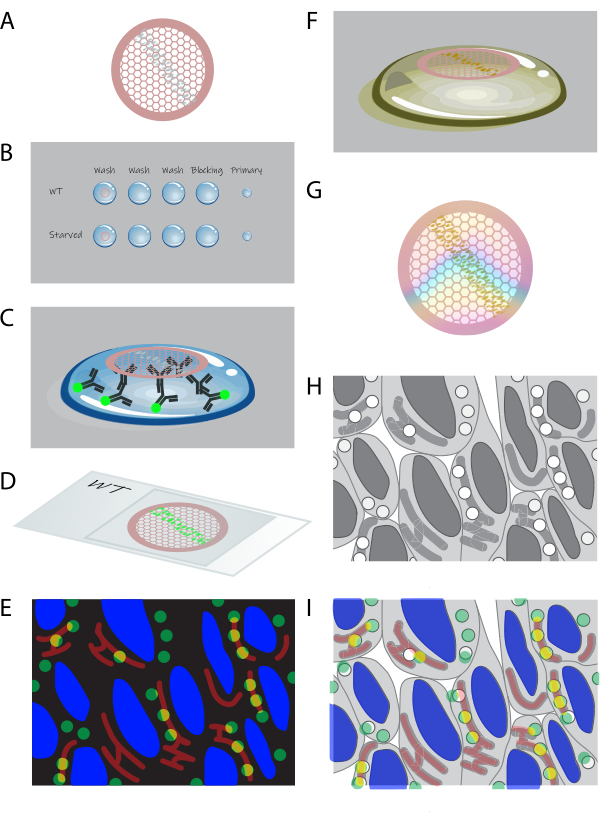
Figura 1: Panoramica schematica del CLEM in sezione . (A) Le criosezioni da cellule incorporate in gelatina sono raccolte su una griglia di rame rivestita di formvar. (B) Le griglie vengono lavorate sezionalmente verso il basso su goccioline delle soluzioni appropriate. (C) Le griglie sono marcate con anticorpi primari e secondari fluorescenti. (D) Le griglie sono inserite tra un vetrino coprioggetto e un vetrino in glicerolo al 50%. (E) Le immagini di fluorescenza sono raccolte in un microscopio a campo largo. (F) Le griglie vengono recuperate dal vetrino e ulteriormente lavorate mediante colorazione con uranile per EM. (G) Dopo l'asciugatura, le griglie possono essere visualizzate tramite TEM. (H) Il tileset di immagini TEM ad alto ingrandimento viene acquisito da un'area selezionata dai dati di fluorescenza. (I) Le immagini della microscopia a fluorescenza e dell'EM sono correlate e sovrapposte. Fare clic qui per visualizzare una versione più grande di questa figura.
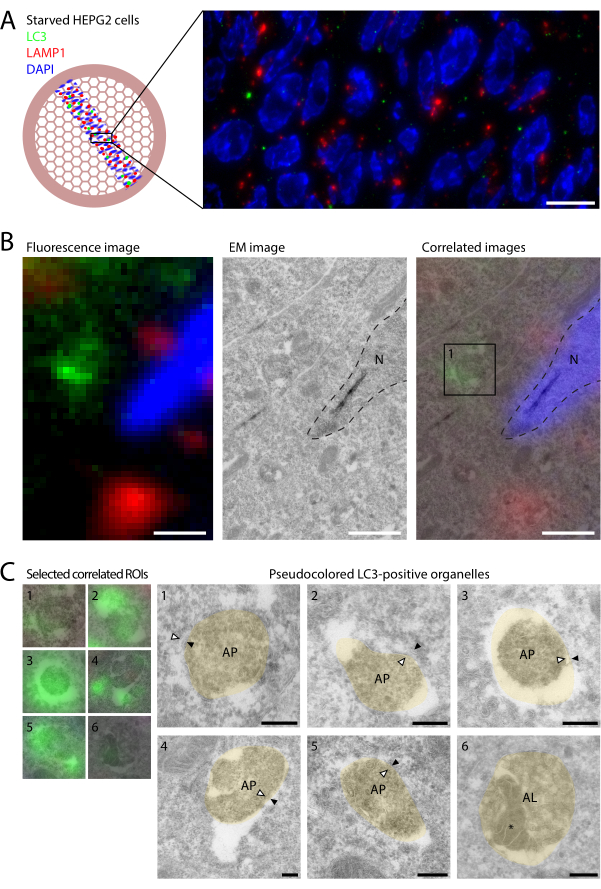
Figura 2: CLEM di LC3 e LAMP1 in celle HEPG2 affamate. Le cellule HEPG2 sono state affamate per 2 ore in EBSS prima della fissazione con PFA al 4% per 2 ore. (A) L'imaging IF di LC3 (verde) e LAMP1 (rosso) sulle sezioni rivela relativamente pochi punti LC3 e poca colocalizzazione con LAMP1. (B) Collegare le informazioni molecolari da IF (pannello di sinistra) alle informazioni ultrastrutturali ottenute in EM (pannello centrale) sovrapponendo le due modalità di imaging basate su DAPI e contorni nucleari (linee tratteggiate, pannello di destra). L'ultrastruttura dei singoli compartimenti marcati LC3, come esemplificato dal riquadro 1 (pannello di destra), è mostrata in C. (C) Ultrastruttura dei compartimenti LC3-positivi. Le immagini CLEM sono mostrate a sinistra e le immagini EM pseudocolorate (beige) a destra (le immagini EM non colorate sono mostrate nella Figura supplementare S1). Le membrane autofagosomiali interne ed esterne sono indicate rispettivamente da punte di freccia bianche e nere. Il contenuto autofagico all'interno dell'autolisosoma nell'esempio 6 è indicato da *. Barre di scala = 10 μm (A), 1 μm (B), 200 nm (C). Fare clic qui per visualizzare una versione più grande di questa figura.
Figura supplementare S1: Immagini EM non colorate di organelli LC3-positivi. (A-F) Immagini EM non colorate degli esempi pseudocolorati 1-6 mostrate nella Figura 2C. Gli organelli sono stati selezionati mediante fluorescenza LC3, come descritto nella Figura 2. Le membrane autofagosomiali interne ed esterne sono indicate rispettivamente da punte di freccia bianche e nere. Il contenuto autofagico all'interno dell'autolisosoma nell'esempio 6 è indicato da *. Barre di scala = 200 nm. Abbreviazioni: AL = autolisosoma; AP = autofagosoma; M = mitocondrio. Fare clic qui per scaricare il file.
File supplementare 1: Tamponi e soluzioni utilizzati in questo studio. Questo file supplementare contiene le ricette e i protocolli necessari per realizzare i tamponi e le soluzioni utilizzate in questo studio. Fare clic qui per scaricare il file.
Discussione
Il metodo qui presentato sfrutta i recenti progressi nel CLEM in sezione basato sulla criosezione, l'elevata sensibilità della marcatura IF e l'accurata correlazione (errore di <100 nm) tra FM e EM14,24. Ciò si traduce in un metodo con la sensibilità di marcare in modo fluorescente proteine endogene scarse e la capacità di sovrapporle con elevata precisione all'ultrastruttura EM. Pertanto, questo metodo evita la necessità di (sovra)espressione di proteine marcate esogenamente e l'uso di etichette EM meno sensibili. La fattibilità del metodo è dimostrata da esempi di CLEM su LC3 endogena in cellule affamate, senza l'uso di inibitori lisosomiali.
Le criosezioni scongelate ottenute con il metodo Tokuyasu sono campioni ideali per l'immuno-EM, poiché a differenza delle sezioni in resina, sono permeabili agli anticorpi. In combinazione con una lieve fissazione e procedure di contrasto, questo generalmente produce un'eccellente efficienza di marcatura rispetto ad altri metodi senza compromettere l'ultrastruttura dettagliata e visualizza in modo eccellente le membrane cellulari 12,25,26. Inoltre, le criosezioni sono altamente compatibili con la microscopia a fluorescenza, il che le rende substrati preziosi per CLEM. Sia la marcatura classica dell'immunogold che il CLEM sulle criosezioni hanno fornito informazioni fondamentali nella comprensione dell'organizzazione subcellulare 14,27,28,29,30.
Attualmente, le applicazioni di CLEM su criosezioni scongelate stanno diventando sempre più diffuse, a seguito di continui sviluppi e ottimizzazioni 14,20,24,31,32,33,34 che hanno migliorato la qualità, l'applicabilità e l'accuratezza dell'approccio. Ora, grazie all'accurata correlazione di grandi set di immagini IF ed EM, la tecnica facilita lo screening per l'ultrastruttura dei componenti cellulari endogeni marcati con fluorescenza 14,32,33. Questo è un vantaggio rispetto al classico immuno-EM, in cui la ricerca di strutture marcate con oro richiede in genere un elevato ingrandimento ed è, quindi, più laboriosa e dispendiosa in termini di tempo. È per questo motivo che la localizzazione di LC3 nell'ultrastruttura trae grande beneficio da CLEM. Gli organelli LC3-positivi sono comuni quando la clearance autofagica è bloccata (cioè quando le cellule sono trattate con BafA1 o agenti che aumentano il pH), mentre gli organelli autofagici vengono rapidamente eliminati nelle cellule inalterate o affamate, con conseguenti livelli di stato stazionario molto bassi. In tali condizioni, trovare organelli marcati con LC3 utilizzando l'immuno-EM classico può essere difficile e CLEM offre un chiaro vantaggio.
In precedenza, CLEM su sezioni di resina è stato applicato in studi che utilizzavano l'espressione ectopica di LC3-GFP o una sonda tandem LC3-GFP-RFP 35,36,37,38,39. In questi studi, l'imaging a fluorescenza è stato eseguito prima dell'inclusione o direttamente nelle sezioni di resina acrilica40 e i campioni sono stati successivamente sottoposti a screening mediante EM. Ci sono diversi vantaggi dell'inclusione in resina; L'ultrastruttura autofagosomiale è generalmente ben conservata, soprattutto se il materiale è congelato ad alta pressione40. Inoltre, il contrasto del materiale incorporato in resina colorato con metalli pesanti è generalmente più pronunciato rispetto a quello delle criosezioni colorate con uranile. Le sezioni incorporate in resina sono compatibili con i metodi EM volumetrici, come la tomografia array, FIB-SEM o SEM seriale blockface, mentre le criosezioni non lo sono. Negli approcci che eseguono l'imaging prima dell'inclusione, l'imaging di cellule vive è un'opzione41 che non è disponibile in CLEM sulle criosezioni. Il vantaggio principale di CLEM sulle criosezioni rispetto a queste alternative è l'elevato segnale IF, che consente l'immunolocalizzazione di proteine rare senza la necessità di permeabilizzazione o sovraespressione della membrana. Ciò evita la potenziale estrazione di membrana, artefatti di sovraespressione42 e modificazioni genetiche del soggetto, il che, combinato con la possibilità di correlare ampie aree in IF ed EM, lo rende un ottimo strumento per lo studio di LC3 e autofagia.
Qui, l'applicazione di CLEM in sezione a cellule HEPG2 affamate ha rivelato che LC3 prevalentemente localizzato in strutture identificate come autofagosomi. Inoltre, sono state trovate alcune macchie debolmente fluorescenti negli autolisosomi. Ciò è in diretto contrasto con le cellule trattate con BafA19 e riflette la rapida degradazione delle proteine autofagosomiali una volta che l'autofagosoma si fonde con i lisosomi. Nel complesso, i dati hanno dimostrato che il CLEM delle criosezioni scongelate può fornire informazioni sull'autofagia mediata da LC3 in condizioni native. I dati evidenziano anche la sensibilità della tecnologia, dal momento che LC3 è stata rilevata anche in autolisosomi che contengono solo bassi livelli di epitopi LC3 intatti. Un'ulteriore applicazione di questa tecnica mediante l'imaging di LC3 in diversi modelli e condizioni migliorerà la nostra comprensione dell'autofagia e di altri processi biologici mediati da LC3, come la fagocitosi associata a LC3 o la coniugazione di ATG8 a singole membrane.
Oltre all'autofagia, il CLEM in sezione può essere applicato ad altri eventi o strutture rare, come la divisione cellulare, l'infezione, i tipi di cellule rare nei tessuti, i cinetocori, le ciglia primarie o gli organelli specifici del tipo di cellula. Uno screening efficace per l'argomento di interesse da parte dell'IF può facilitare notevolmente lo studio ultrastrutturale di queste rarità. Inoltre, èstato dimostrato che la tecnica può essere utilizzata per localizzare le proteine in modo più sensibile rispetto all'immuno-EM classico. La regolazione della lunghezza di fissazione può estendere ulteriormente questa sensibilità, consentendo la localizzazione ultrastrutturale di proteine molto poco abbondanti o scarsamente antigeniche. Infine, il metodo CLEM in sezione facilita la selezione rapida di un numero quantitativo di organelli, facilitando un'analisi più robusta della distribuzione ultrastrutturale di una data proteina.
CLEM sulle criosezioni richiede l'attrezzatura e l'esperienza per la criosezione. Nei gruppi con accesso a questi strumenti (ad esempio, criomicrotomi), l'implementazione di CLEM in sezione è semplice e richiede solo la disponibilità di un microscopio automatico a campo largo, una configurazione a cui la maggior parte dei laboratori ha accesso. Inoltre, il metodo è disponibile nelle strutture EM di tutto il mondo. Poiché il CLEM in sezione combina l'applicazione di metodi IF ed EM consolidati, il metodo è facilmente adattabile e può essere combinato, ad esempio, con la tomografia 20,33,43, il volume EM della sezione seriale di un numero limitato di sezioni 44 o la microscopia a super-risoluzione 45. Questa versatilità del metodo supporta le applicazioni a un'ampia gamma di questioni biologiche.
Divulgazioni
Gli autori dichiarano che non sussistono conflitti di interesse.
Riconoscimenti
Ringraziamo i nostri colleghi del Centro di Medicina Molecolare dell'University Medical Center di Utrecht per le fruttuose discussioni e feedback. Ringraziamo i colleghi passati e presenti del laboratorio Klumperman per i continui miglioramenti apportati alle nostre tecnologie di microscopia. L'infrastruttura EM utilizzata per questo lavoro fa parte del programma di ricerca National Roadmap for Large-Scale Research Infrastructure (NEMI) finanziato dal Dutch Research Council (NWO), numero di progetto 184.034.014 a JK.
Materiali
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Antibody donkey anti-mouse Alexa Fluor 488 | Life Technologies | #A21202 | use 1:250 |
| Antibody donkey anti-rabbit Alexa Fluor 568 | Life Technologies | A#10042 | use 1:250 |
| Antibody mouse anti-LC3 | Cosmo Bio | CTB-LC3-2-IC | use 1:100 |
| Antibody rabbit anti-LAMP1 | Cell Signaling | 9091 | use 1:250 |
| Bovine serum Albumin, fraction V | Sigma-Aldrich | A-9647 | |
| BSA-c | Aurion | 900.099 | |
| BSA-conjugated gold | Cell Microscopy Core, UMC Utrecht | BSAG 5 nm | |
| Water-free Chloroform | Merck | 1.02447.0500 | |
| DAPI | Invitrogen | 10184322 | Use at end concentration of 10 µg/ml |
| EGTA | Sigma-Aldrich | E4378 | |
| Fish-skin Gelatin | Sigma-Aldrich | G7765 | |
| Food-grade gelatin | Merck | G1890 | |
| Formvar, Vinylec E | SPI | 02492-RA | |
| Gluteraldehyde | Serva | 23115.01 | See CAUTION note |
| Glycerol | Boom | MBAK 7044.1000 | |
| Glycine | Merck | 1042010250 | |
| HEPES | Sigma-Aldrich | H3375 | |
| Methylcellulose, 25 centipoises | Sigma-Aldrich | M-6385 | |
| MgSO4 | Riedel-de Haen | 12142 | |
| Na2HPO4 (PB component A) | Merck | 106580-0500 | |
| NaBH4 | Merck | 806373 | |
| NaH2PO4 (PB component B) | Merck | 106346 | |
| NH4OH | Sigma-Aldrich | 221228-0025 | |
| Oxalic acid | Merck | 100495 | |
| Paraformaldehyde prills | Sigma-Aldrich | 441244 | See CAUTION note |
| PIPES | Merck | 110220 | |
| Protein-A conjugated gold | Cell Microscopy Core, UMC Utrecht | PAG 5, 10, 15 or 20 nm | |
| Sucrose D(+) | VWR | 27483294 | |
| Uranyl acetate | SPI | 020624-AB | See CAUTION note |
| Tools and consumables | |||
| Pick-up loop | Electron Microscopy Sciences | 70944 | |
| Filter paper, qualitative, medium-fast | LLG | 6.242 668 | |
| Finder grids | Ted Pella | G100F1 | |
| Grids | Cell Microscopy Core, UMC Utrecht | CU 100 mesh | |
| Microscopes | |||
| Leica Thunder widefield microscope | Leica | Components: 100x, 1.47 NA TIRF objective; Photometrics prime 95B sCMOS camera; LAS X software; | |
| Leica UC7 ultracryomicrotome | Leica | ||
| Tecnai T12 | FEI | Components: Veleta VEL-FEI-TEC12-TEM camera; SerialEM software | |
| Software | |||
| ec-CLEM in icy | open source | Paul-Gilloteaux et al., 2017 | |
| Fiji | open source | Schindelin et al., 2012 | |
| IMOD | open source | Mastronarde et al., 2017 | |
| Photoshop | Adobe | ||
| SerialEM | open source | Mastronarde et al., 2018 |
Riferimenti
- Hu, Y., Reggiori, F. Molecular regulation of autophagosome formation. Biochemical Society Transactions. 50 (1), 55-69 (2022).
- Reggiori, F., Ungermann, C. Autophagosome maturation and fusion. Journal of Molecular Biology. 429 (4), 486-496 (2017).
- Kimura, S., Noda, T., Yoshimori, T. Dissection of the autophagosome maturation process by a novel reporter protein, tandem fluorescent-tagged LC3. Autophagy. 3 (5), 452-460 (2007).
- Baba, M., Takeshige, K., Baba, N., Ohsumi, Y. Ultrastructural analysis of the autophagic process in yeast: detection of autophagosomes and their characterization. The Journal of Cell Biology. 124 (6), 903-913 (1994).
- Takeshige, K., Baba, M., Tsuboi, S., Noda, T., Ohsumi, Y. Autophagy in yeast demonstrated with proteinase-deficient mutants and conditions for its induction. The Journal of Cell Biology. 119 (2), 301-311 (1992).
- De Duve, C., Wattiaux, R. Functions of lysosomes. Annual Review of Physiology. 28, 435-492 (1966).
- Arstila, A. U., Trump, B. F. Studies on cellular autophagocytosis. The formation of autophagic vacuoles in the liver after glucagon administration. The American Journal of Pathology. 53 (5), 687-733 (1968).
- Eskelinen, E. L., Reggiori, F., Baba, M., Kovács, A. L., Seglen, P. O. Seeing is believing: The impact of electron microscopy on autophagy research. Autophagy. 7 (9), 935-956 (2011).
- De Mazière, A., et al. An optimized protocol for immuno-electron microscopy of endogenous LC3. Autophagy. 18 (12), 3004-3022 (2022).
- López-Terrada, D., Cheung, S. W., Finegold, M. J., Knowles, B. B. Hep G2 is a hepatoblastoma-derived cell line. Human Pathology. 40 (10), 1512-1515 (2009).
- Tanida, I., Minematsu-Ikeguchi, N., Ueno, T., Kominami, E. Lysosomal turnover, but not a cellular level, of endogenous LC3 is a marker for autophagy. Autophagy. 1 (2), 84-91 (2005).
- Slot, J. W., Geuze, H. J. Cryosectioning and immunolabeling. Nature Protocols. 2 (10), 2480-2491 (2007).
- Waterman-Storer, C. M. Microtubule/organelle motility assays. Current Protocols in Cell Biology. 13 (1), (2001).
- vander Beek, J., de Heus, C., Liv, N., Klumperman, J. Quantitative correlative microscopy reveals the ultrastructural distribution of endogenous endosomal proteins. The Journal of Cell Biology. 221 (1), e202106044(2022).
- Mastronarde, D. N. Advanced data acquisition from electron microscopes with SerialEM. Microscopy and Microanalysis. 24, 864-865 (2018).
- Mastronarde, D. N., Held, S. R. Automated tilt series alignment and tomographic reconstruction in IMOD. Journal of Structural Biology. 197 (2), 102-113 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Paul-Gilloteaux, P., et al. EC-CLEM: Flexible multidimensional registration software for correlative microscopies. Nature Methods. 14 (2), 102-103 (2017).
- Heiligenstein, X., Paul-Gilloteaux, P., Raposo, G., Salamero, J. eC-CLEM: A multidimension, multimodel software to correlate intermodal images with a focus on light and electron microscopy. Methods in Cell Biology. 140, 335-352 (2017).
- Fermie, J., et al. Bimodal endocytic probe for three-dimensional correlative light and electron microscopy. Cell Reports Methods. 2 (5), 100220(2022).
- Fokkema, J., et al. Fluorescently labelled silica coated gold nanoparticles as fiducial markers for correlative light and electron microscopy. Scientific Reports. 8 (1), 13625(2018).
- Czaja, M. J., et al. Functions of autophagy in normal and diseased liver. Autophagy. 9 (8), 1131(2013).
- Robinson, J. M., Takizawa, T., Pombo, A., Cook, P. R. Correlative fluorescence and electron microscopy on ultrathin cryosections: Bridging the resolution gap. Journal of Histochemistry and Cytochemistry. 49 (7), 803-808 (2001).
- Mohammadian, S., et al. High accuracy, fiducial marker-based image registration of correlative microscopy images. Scientific Reports. 9 (1), 3211(2019).
- Tokuyasu, K. T. A study of positive staining of ultrathin frozen sections. Journal of Ultrasructure Research. 63 (3), 287-307 (1978).
- Slot, J. W., Geuze, H. J. A new method of preparing gold probes for multiple-labeling cytochemistry. European Journal of Cell Biology. 38 (1), 87-93 (1985).
- Klumperman, J., Raposo, G. The complex ultrastructure of the endolysosomal system. Cold Spring Harbor Perspectives in Biology. 6 (10), a016857(2014).
- Geuze, H. J., Slot, J. W., Strous, G. J., Lodish, H. F., Schwartz, A. L. Intracellular site of asialoglycoprotein receptor-ligand uncoupling: Double-label immunoelectron microscopy during receptor-mediated endocytosis. Cell. 32 (1), 277-287 (1983).
- Biazik, J., Ylä-Anttila, P., Vihinen, H., Jokitalo, E., Eskelinen, E. L. Ultrastructural relationship of the phagophore with surrounding organelles. Autophagy. 11 (3), 439-451 (2015).
- Fahimi, H. D., Reich, D., Völkl, A., Baumgart, E. Contributions of the immunogold technique to investigation of the biology of peroxisomes. Histochemistry and Cell Biology. 106 (1), 105-114 (1996).
- Vicidomini, G., et al. A novel approach for correlative light electron microscopy analysis. Microscopy Research and Technique. 73 (3), 215-224 (2010).
- Vicidomini, G., et al. High data output and automated 3D correlative light-electron microscopy method. Traffic. 9 (11), 1828-1838 (2008).
- Cortese, K., et al. 3D HDO-CLEM: cellular compartment analysis by correlative light-electron microscopy on cryosection. Methods in Cell Biology. 111, 95-115 (2012).
- van Rijnsoever, C., Oorschot, V., Klumperman, J. Correlative light-electron microscopy (CLEM) combining live-cell imaging and immunolabeling of ultrathin cryosections. Nature Methods. 5 (11), 973-980 (2008).
- Razi, M., Chan, E. Y. W., Tooze, S. A. Early endosomes and endosomal coatomer are required for autophagy. The Journal of Cell Biology. 185 (2), 305-321 (2009).
- Ligeon, L. A., Barois, N., Werkmeister, E., Bongiovanni, A., Lafont, F. Structured illumination microscopy and correlative microscopy to study autophagy. Methods. 75, 61-68 (2015).
- Biazik, J., Vihinen, H., Anwar, T., Jokitalo, E., Eskelinen, E. L. The versatile electron microscope: An ultrastructural overview of autophagy. Methods. 75, 44-53 (2015).
- Gudmundsson, S., Kahlhofer, J., Baylac, N., Kallio, K., Eskelinen, E. L. Correlative light and electron microscopy of autophagosomes. Methods in Molecular Biology. 1880, 199-209 (2019).
- Kriel, J., et al. Correlative light and electron microscopy (CLEM): bringing together the best of both worlds to study neuronal autophagy. Imaging and Quantifying Neuronal Autophagy. 171, 135-147 (2022).
- Largeau, C., Legouis, R. Correlative light and electron microscopy to analyze LC3 proteins in Caenorhabditis elegans embryo. Methods in Molecular Biology. 1880, 281-293 (2019).
- Fermie, J., et al. Single organelle dynamics linked to 3D structure by correlative live-cell imaging and 3D electron microscopy. Traffic. 19 (5), 354-369 (2018).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: Caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Ladinsky, M. S., Howell, K. E. Electron tomography of immunolabeled cryosections. Methods in Cell Biology. 79, 543-558 (2007).
- Oorschot, V., Lindsey, B. W., Kaslin, J., Ramm, G. TEM, SEM, and STEM-based immuno-CLEM workflows offer complementary advantages. Scientific Reports. 11 (1), 899(2021).
- Franke, C., et al. Correlative single-molecule localization microscopy and electron tomography reveals endosome nanoscale domains. Traffic. 20 (8), 601-617 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon