Method Article
Ultrastrukturelle Lokalisierung von endogenem LC3 mittels korrelativer Lichtelektronenmikroskopie
In diesem Artikel
Zusammenfassung
In dieser Arbeit stellen wir ein Protokoll für eine optimierte korrelative Lichtelektronenmikroskopie vor, die auf endogener, fluoreszierender Markierung basiert, um die Lokalisierung seltener Proteine in Bezug auf die zelluläre Ultrastruktur zu untersuchen. Die Leistungsfähigkeit dieses Ansatzes wird durch die ultrastrukturelle Lokalisierung von endogenem LC3 in ausgehungerten Zellen ohne Bafilomycin-Behandlung demonstriert.
Zusammenfassung
Die Visualisierung autophagischer Organellen auf ultrastruktureller Ebene durch Elektronenmikroskopie (EM) ist unerlässlich, um ihre Identität festzustellen und Details zu enthüllen, die für das Verständnis des autophagischen Prozesses wichtig sind. EM-Methoden fehlen jedoch oft molekulare Informationen, was die Korrelation der durch EM gewonnenen ultrastrukturellen Informationen mit der fluoreszenzmikroskopischen Lokalisierung spezifischer Autophagieproteine behindert. Darüber hinaus erschwert die Seltenheit von Autophagosomen unter unveränderten zellulären Bedingungen die Untersuchung mittels EM, die eine hohe Vergrößerung erfordert und daher ein begrenztes Sichtfeld bietet.
Als Antwort auf beide Herausforderungen wurde eine auf Fluoreszenzmarkierung basierende Methode der korrelativen Lichtelektronenmikroskopie (CLEM) angewendet, um einen gemeinsamen autophagosomalen Marker, LC3, mit der EM-Ultrastruktur zu korrelieren. Die Methode wurde verwendet, um Zellen in der Fluoreszenzmikroskopie für die LC3-Markierung in Kombination mit anderen relevanten Markern schnell zu screenen. Anschließend wurden die zugrundeliegenden ultrastrukturellen Merkmale ausgewählter LC3-markierter Spots mittels CLEM identifiziert. Die Methode wurde auf ausgehungerte Zellen angewendet, ohne dass Inhibitoren der lysosomalen Ansäuerung zugesetzt wurden.
Unter diesen Bedingungen wurde LC3 überwiegend auf Autophagosomen und selten in Autolysosomen gefunden, in denen LC3 schnell abgebaut wird. Diese Daten zeigen sowohl die Machbarkeit als auch die Sensitivität dieses Ansatzes und zeigen, dass CLEM verwendet werden kann, um ultrastrukturelle Einblicke in die LC3-vermittelte Autophagie unter nativen Bedingungen zu liefern - ohne medikamentöse Behandlungen oder genetische Veränderungen. Insgesamt stellt diese Methode ein wertvolles Werkzeug für ultrastrukturelle Lokalisierungsstudien von Autophagieproteinen und anderen seltenen Antigenen dar, indem sie die Lichtmikroskopie mit EM-Daten verbindet.
Einleitung
Autophagie ist ein Schlüsselprozess für die Beseitigung und das Recycling von zytoplasmatischen Proteinen und Organellen. Der Prozess der Makroautophagie (im Folgenden Autophagie genannt) beinhaltet die Bildung von Doppelmembranorganellen, Autophagosomen, die es den Zellen ermöglichen, zytoplasmatische Moleküle und Organellen für den lysosomalen Abbau einzuschließen. Autophagie findet in den meisten Zellen auf basaler Ebene statt und wird als Reaktion auf zelluläre Bedingungen wie Hunger oder zellulären Stress hochreguliert. Die Autophagie erfolgt entweder substratspezifisch, indem sie auf bestimmte Strukturen oder Proteine abzielt, um sie abzubauen, oder als nicht-selektiver Massenprozess, der Teile des Zytosols umfasst. Bei der selektiven Autophagie werden Autophagosomen durch die Konjugation von Proteinen der Atg8-Familie (Mikrotubuli-assoziierte Proteine 1A/B-Leichtkette 3A/B/C [LC3] und GABARAPs) mit Membranen gebildet, die aus Recycling-Endosomen, dem Golgi und/oder dem endoplasmatischen Retikulum (ER)1 stammen. LC3 erkennt autophagische Fracht im Zytosol direkt oder über selektive Autophagie-Adapter wie P62/SQSTM. Neue autophagische Membranen können dann mit LC3 konjugiert werden, sich ausdehnen und verschmelzen, um eine vollständige Doppelmembran zu bilden, die die Fracht umschließt - das sogenannte Autophagosom. Das Autophagosom reift heran und verschmilzt schließlich mit einem Endosom oder Lysosom, woraufhin die autophagische Fracht und die Adapter abgebaut werden2.
Studien zur Autophagosomenbildung, -reifung und -fusion nutzen häufig lichtmikroskopische Technologien. Die Fluoreszenzmikroskopie von LC3 wird im Allgemeinen verwendet, um die Anzahl und zelluläre Lokalisation von Autophagosomen unter verschiedenen Bedingungen zu beurteilen. Darüber hinaus kann durch die Kopplung von LC3 an pH-sensitives GFP und pH-stabiles RFP in einer sogenannten Tandemsonde der Gesamtfluss der Autophagie in lebenden Zellen in Abhängigkeit vom GFP-Fluoreszenzverlustgemessen werden 3. Diese Ansätze sind wertvolle Werkzeuge für Forscher, um die Rolle und den Mechanismus der Autophagie unter verschiedenen Bedingungen zu verstehen. Ein weiteres unschätzbares Werkzeug ist die Elektronenmikroskopie (EM), die die Ultrastruktur von autophagischen Organellen in verschiedenen Stadien der Autophagie aufdeckt 4,5,6,7,8. Bis heute ist EM immer noch die Methode der Wahl, um die genauen Stadien der Autophagosomenbildung zu identifizieren, indem verschiedene autophagische Membranen nach ihrer Morphologie unterschieden werden: Phagophor (Doppelmembran nicht vollständig geschlossen), Autophagosom (geschlossene Doppelmembran um zytosolische Fracht) und Autolysosom ([teilweiser] Verlust der inneren autophagischen Membran). Eine Morphologie ohne molekulare Informationen kann jedoch anfällig für Fehlidentifikationen oder Mehrdeutigkeiten sein. Immuno-EM ist die umfassendste Methode zur gleichzeitigen molekularen Charakterisierung und morphologischen Klassifikation autophagischer Organellen. Zum Beispiel ermöglicht die Immunogold-Markierung von LC3 auf aufgetauten Kryoschnitten die ultrastrukturelle Lokalisierung von LC3 und die präzise Identifizierung von LC3-markierten Organellen9.
Ein Nachteil der EM ist das kleine Sichtfeld, das mit der hohen Vergrößerung einhergeht, die erforderlich ist, um die feine Ultrastruktur der autophagischen Membranen zu beobachten und im Falle der Immun-EM die Markierung zu finden, die das gewünschte Protein markiert. Aufgrund ihrer Knappheit und ihres geringen Proteingehalts erschwert dies in der Regel die quantitative EM-Analyse von Autophagosomen. Um die Anzahl der Autophagosomen zu erhöhen, werden die Zellen oft ausgehungert und mit Bafilomycin A1 (BafA1), einem Inhibitor der lysosomalen Ansäuerung und des Abbaus, behandelt. Ohne BafA1-Behandlung ist die Suche nach Autophagosomen mittels EM aufgrund der Knappheit dieser Organellen zeitintensiv. Die in diesem Manuskript vorgestellte Methode befasst sich mit diesem Problem durch Fluoreszenzmarkierung und Bildgebung von endogenem LC3 auf aufgetauten Kryoschnitten in einem Fluoreszenzmikroskop vor der weiteren Vorbereitung auf EM. Die Fluoreszenzbilder leiten dann die Suche nach LC3-markierten Strukturen in der EM. Nach der Entnahme werden die EM-Bilder mit den Fluoreszenzbildern korreliert, um der Ultrastruktur der Zelle molekulare Informationen – das Vorhandensein von LC3 – hinzuzufügen. Diese "On-Section-CLEM"-Methode erhöht die Fähigkeit, LC3-markierte Strukturen zu finden, insbesondere unter unbehandelten Bedingungen, für die anschließende Identifizierung und Klassifizierung durch EM.
Diese Methode wurde auf ausgehungerte Hepatoblastom-abgeleitete HEPG210-Zellen angewendet, um Autophagosomen unter unveränderten Bedingungen (d.h. es wurde kein BafA1 verwendet) zu finden. Es wurden relativ wenige fluoreszierende Punkte (weniger als eine pro Zellprofil in einem 90-nm-Schnitt) gefunden, was mit dem hohen Umsatz von LC311 übereinstimmt. Diese Spärlichkeit von LC3-puncta unterstrich den Wert von CLEM; Durch die Auswahl von Regionen mit mehreren fluoreszierenden Punkten für die Bildgebung in der EM wurden LC3-positive Organellen gefunden und auf eine wesentlich effektivere Weise charakterisiert als durch konventionelle Immun-EM. Dabei zeigte sich, dass es sich bei der Mehrzahl der LC3-positiven Organellen um Autophagosomen handelte, die durch ihre Morphologie definiert sind, was im Gegensatz zu den Ergebnissen steht, die in BafA1-behandelten Zellen erzielt wurden, wo Autolysosomen häufiger vorkommen9. Diese Daten zeigen, dass mit On-Section-CLEM die Autophagie auf ultrastruktureller Ebene untersucht werden kann, ohne dass der autophagische Fluss gehemmt werden muss.
Protokoll
1. Vorbereitung von Werkzeugen und Reagenzien
HINWEIS: Weitere Informationen zu den erforderlichen Reagenzien, Puffern und Lösungen finden Sie in der Zusatzdatei 1 oder 12 . Einzelheiten zu allen Materialien, Reagenzien, Geräten und Software, die in diesem Protokoll verwendet werden, finden Sie in der Materialtabelle.
- Fixiermittel
- Bereiten Sie 0,2 M Phosphatpuffer (PB) oder 0,2 M PIPES, HEPES, EGTA, MgSO4 (PHEM)-Puffer vor, wie in Ergänzungsdatei 1 beschrieben, um ihn als Basis für Fixierlösungen zu verwenden.
HINWEIS: Fixiermittel werden routinemäßig in 0,1 M PB- oder PHEM-Puffer gepuffert, um gegen Versauerung zu puffern, die durch die Aldehydreaktion mit dem biologischen Material verursacht wird. - Da die Qualität des Paraformaldehyds (PFA) der Schlüssel zu einer zuverlässigen Fixierung der Ultrastruktur der Proben ist, sollten Sie PFA in EM-Qualität verwenden. Um diesem Protokoll zu folgen, verwenden Sie 16%ige Stammlösungen, die aus hochwertigen PFA-Prills hergestellt werden (siehe Ergänzungsdatei 1).
ACHTUNG: Paraformaldehyd ist eine gefährliche Chemikalie (Gefahrenhinweise H228, H301, H302, H311, H314, H315, H317, H318, H331, H332, H335, H341, H350). Tragen Sie bei der Arbeit mit PFA Schutzausrüstung (Handschuhe, Laborkittel und Schutzbrille) und arbeiten Sie mit einer Chemikalienhaube. Abfälle, die PFA enthalten, sollten gemäß den Richtlinien und Vorschriften der Institute gesammelt und entsorgt werden. - Kombinieren Sie 10 ml 0,2 M PB, 5 ml 16 % PFA (in demineralisiertem Wasser [dH2O]) und 5 mldH2O, um eine Fixierlösung von 4 % PFA herzustellen.
- Optional: Die Zugabe von 0,02 % bis 0,5 % Glutaraldehyd (GA) zur Fixierlösung aus Schritt 1.1.3 verbessert die Erhaltung der Ultrastruktur, verringert jedoch die Antigenität der Probe gegenüber vielen Antikörpern.
HINWEIS: Wenn eine GA-Fixierung gewünscht ist, verwenden Sie EM-GA von einem geeigneten Lieferanten.
VORSICHT: Glutaraldehyd ist eine gefährliche Chemikalie (Gefahrenhinweise H301, H302, H314, H317, H330, H332, H334, H335, H400, H411). Arbeiten Sie in einer Chemikalienhaube und tragen Sie Schutzausrüstung (Handschuhe, Laborkittel und Schutzbrille), wenn Sie GA manipulieren. GA-haltige Abfälle sollten gemäß den Richtlinien und Vorschriften des Instituts gesammelt und entsorgt werden.
- Bereiten Sie 0,2 M Phosphatpuffer (PB) oder 0,2 M PIPES, HEPES, EGTA, MgSO4 (PHEM)-Puffer vor, wie in Ergänzungsdatei 1 beschrieben, um ihn als Basis für Fixierlösungen zu verwenden.
- Werkzeuge und Materialien
- Kratzen Sie die Oberfläche der Aluminium-Probenhalterstifte an und beschallen Sie sie 3 x 10 Minuten in Ethanol, um Metallreste zu entfernen und eine optimale Haftung zu gewährleisten, wenn in Gelatine eingebettete Zellblöcke auf den Stiften montiert sind.
- Verwenden Sie einen Aufbewahrungskanister, der geeignet ist, die Aluminium-Probenhalterstifte mit ihren Proben in flüssigem Stickstoff (LN2) zu lagern.
- Stellen Sie einen Manipulator her, indem Sie ein einzelnes Haar oder eine einzelne Wimper mit Nagellack am Ende eines Holzspießes anbringen.
- Mache eine Tonabnehmerschleife. Biegen Sie einen 0,3 mm dicken Edelstahldraht um eine Rundstange mit einem Durchmesser von 3 mm und verdrehen Sie die Enden zusammen, sodass an einem Ende eine Schlaufe entsteht. Führen Sie die verdrehten Enden in eine Pipettenspitze ein. Stecken Sie einen Holzspieß vom anderen Ende und befestigen Sie ihn mit Kleber oder Harz.
HINWEIS: Eine Tonabnehmerschleife ist auch im Handel erhältlich (siehe Materialtabelle). - Um die Gittertrocknungsschleifen vorzubereiten, gehen Sie wie folgt vor, um Tonabnehmerschlaufen herzustellen: Einen Edelstahldraht in einer 4-mm-Schlaufe formen und mit Klebstoff oder Harz auf eine große Pipettenspitze aufkleben.
- Beschichten Sie die Gitter mit einer dünnen Stützfolie wie z. B. formvar (Protokoll in Supplemental File 1). Beschichten Sie die Gitter vor dem Gebrauch mit einer dünnen Schicht Kohlenstoff.
HINWEIS: Gebrauchsfertige Gitter sind im Handel erhältlich (siehe Materialtabelle). Formvar-beschichtete Gitter können unbegrenzt bei Raumtemperatur (RT) gelagert werden; Carbonbeschichtete Gitter können bei RT mehrere Monate gelagert werden. - Bereiten Sie saubere Objektträger und große Deckgläser vor (24 mm x 24 mm ist ideal für 25 mm breite Objektträger), wie in 13.
2. Fixierung und Probenvorbereitung
- Fixierung
- Verwenden Sie das in Schritt 1.1.3 vorbereitete Fixiermittel (4 % PFA in 0,1 M PB). Für adhärente Zelllinien kultivieren Sie 1-5 × 106 Zellen in 6-cm-Schalen. Geben Sie das Fixiermittel im Verhältnis 1:1 in das Nährmedium und inkubieren Sie die Probe 5 Minuten lang bei RT. Ersetzen Sie dann das Medium-Fixiermittel-Gemisch durch nur Fixiermittel und inkubieren Sie 2 h bei RT.
HINWEIS: Die genaue Zellzahl, Konfluenz und Kulturbedingungen können je nach verwendetem Modellsystem variieren. - Lagern Sie die Proben über Nacht oder bis zu 3-4 Wochen in 0,5 % PFA in 0,1 M PB bei 4 °C.
HINWEIS: GA kann der Fixierung hinzugefügt werden (siehe Schritt 1.1.4) und die Fixationslänge kann geändert werden, um ein optimales Gleichgewicht zwischen der Erhaltung der Morphologie und der Antigenität zu finden, die je nach Probe und Markierung unterschiedlich ist. Weitere Informationen finden Sie unter14.
- Verwenden Sie das in Schritt 1.1.3 vorbereitete Fixiermittel (4 % PFA in 0,1 M PB). Für adhärente Zelllinien kultivieren Sie 1-5 × 106 Zellen in 6-cm-Schalen. Geben Sie das Fixiermittel im Verhältnis 1:1 in das Nährmedium und inkubieren Sie die Probe 5 Minuten lang bei RT. Ersetzen Sie dann das Medium-Fixiermittel-Gemisch durch nur Fixiermittel und inkubieren Sie 2 h bei RT.
- Beispiel-Einbettung
- Spülen Sie das Geschirr mit festen Zellen 3x mit PBS bei RT. Ersetzen Sie dann PBS mit 0,15 % Glycin und inkubieren Sie 10 Minuten lang bei RT.
- Ersetzen Sie die PBS mit 0,15 % Glycin durch 1 % Gelatine in PBS, das auf 37 °C vorgewärmt ist, und schaben Sie die Zellen ab und überführen Sie sie in 1 % Gelatine in ein Mikrozentrifugenröhrchen. Die Zellen werden bei 6.000 × g für 1 min bei RT in einer Mikrozentrifuge pelletiert. Dann wird die 1%ige Gelatine entfernt, ohne das Pellet zu stören, und 12%ige auf 37 °C erwärmte Gelatine hinzugefügt. Resuspendieren Sie das Zellpellet durch vorsichtiges Auf- und Abpipettieren mit Pipettenspitzen oder auf 37 °C vorgewärmten Pasteurpipetten aus Glas.
- 10 min bei 37 °C inkubieren; Anschließend werden die Zellen 1 Minute lang bei 6.000 × g pelletiert. Die Gelatine auf Eis 30 Min. verfestigen.
- Um die in Gelatine eingebetteten Zellen aus dem Röhrchen zu entfernen, schneiden Sie das Röhrchenende, das das Pellet enthält, mit einer Rasierklinge vom Rest des Röhrchens ab. Schneiden Sie dann senkrecht zum ersten Schnitt das Röhrchenende mit dem Zellpellet in zwei Hälften.
- Die beiden Hälften des Röhrchenends, die das in Gelatine eingebettete Zellpellet enthalten, werden 10 Minuten lang bei 4 °C in 2,3 M Saccharose inkubiert. Dies führt dazu, dass die in Gelatine eingebetteten Zellpellethälften leicht schrumpfen und sich aus dem Kunststoffrohr lösen.
HINWEIS: Die in Gelatine eingebetteten Zellpellets sollten so weit wie möglich bei 4 °C oder eiskalt aufbewahrt werden, um zu vermeiden, dass die 2,3 M Saccharose zu zähflüssig und die Gelatine zu weich wird. Bei der Manipulation der in Gelatine eingebetteten Zellpellets in den nächsten Schritten arbeiten Sie jeweils nur mit einer Probe und lassen Sie die anderen auf Eis oder arbeiten Sie in einem kalten (~4 °C) Raum. Vermeiden Sie eine Überhitzung der in Gelatine eingebetteten Zellpellets durch Sonnenlicht, heiße Mikroskoplampen oder andere Wärmequellen. - Entfernen Sie die Röhrchenhälften mit dem in Gelatine eingebetteten Zellpellet aus der 2,3 M Saccharose. Entfernen Sie dann die in Gelatine eingebetteten Zellpellethälften mit einer Pinzette aus den Kunststoffröhrchenhälften. Schneiden Sie das Pellet mit einer Rasierklinge manuell in Blöcke geeigneter Größe (~1 mm3). Verwenden Sie ein Stereo-Präpariermikroskop, um das Motiv während des Schneidens zu vergrößern.
- Die in Gelatine eingebetteten Zellblöcke werden 3-16 h lang mit 2,3 M Saccharose infundiert und in einem Rotor bei 4 °C umgedreht.
- Platzieren Sie einen in Gelatine eingebetteten Zellblock auf einem Probenhalterstift aus Aluminium (siehe Schritt 1.2.1). Lassen Sie so viel 2,3 M Saccharose an den Rändern des Blocks stehen, dass sie einen dünnen "Kragen" zwischen dem Block und dem Stift bildet. Vermeiden Sie, dass zu viel 2,3 M Saccharose die Oberseite des Blocks bedeckt. Einfrieren und in LN2 lagern.
3. Schneiden
- Trimmen (siehe auch12)
- Nehmen Sie einen Stift mit einem Block aus in Gelatine eingebetteten Zellen aus der LN2-Lagerung und legen Sie ihn in ein Kryomikrotom, das auf -80 °C eingestellt ist.
- Schneiden Sie die Vorderseite des Blocks ab, um seine Oberfläche abzuflachen und ~250 nm Abschnitte zu erhalten. Tauchen Sie die 3-mm-Schleife in eine Tonabnehmerlösung (1:1 2,3 M Saccharose und 2 % Methylcellulose), führen Sie die Schleife in die Kältekammer des Mikrotoms ein und warten Sie, bis sich Eis im Tröpfchen bildet (typischerweise 5-7 s). Nehmen Sie dann sofort den Abschnitt auf, indem Sie den Tropfen schnell, aber vorsichtig dagegen drücken. Entfernen Sie die Schlaufe aus der Kältekammer, warten Sie, bis der Tropfen vollständig aufgetaut ist, und drücken Sie den Tropfen auf einen Objektträger.
- Überprüfen Sie die Zellorientierung durch Toluidinblau-Färbung der Abschnitte.
- Geben Sie einen Tropfen Toluidinblaulösung (siehe Ergänzungsdatei 1) auf die Abschnitte eines Objektträgers und trocknen Sie ihn auf einer Heizplatte bei 80 °C, bis die Ränder des Tropfens trocken sind.
- Nehmen Sie den Objektträger von der Heizplatte ab und spülen Sie das Toluidinblau vorsichtig mit dH2O ab und sammeln Sie es in einem geeigneten Abfallbehälter.
- Trocknen Sie den Objektträger und überprüfen Sie die Zellausrichtung in den Abschnitten mit einem einfachen Tischlichtmikroskop.
- Schneiden Sie die Seiten des Blocks ab, indem Sie 50-100 μm in die Seite der Probenblockfläche mit der Messerecke schneiden. Schneiden Sie vier Seiten der Probenblockfläche ab, indem Sie den Probenhalter nach dem Trimmen jeder Seite um 90° drehen, um ein hervorstehendes Rechteck von ~250 μm x 375 μm zu erhalten. Wählen Sie den hervorstehenden Bereich basierend auf der im vorherigen Schritt ermittelten Zellenausrichtung aus.
- Schneiden und Aufnehmen
- Das Kryomikrotom auf -100 °C abkühlen. Schneiden Sie ein Band aus dem hervorstehenden Rechteck ab, dessen Abschnitte 70-90 nm dick und silbrig-golden schimmern. Führen Sie die Abschnitte mit einem Stäbchenhaar von der Schneide des Diamantmessers weg (siehe Abschnitt 1.2.3), um ein langes Band (2-5 mm) zu erhalten.
- Sobald sich ein geeignetes Band gebildet hat, hören Sie auf zu schneiden, um das Band aufzunehmen. Tauchen Sie die 3-mm-Tonabnehmerschleife in 2,3 M Saccharose und 2 % Methylcellulose, die 1:1 gemischt sind, führen Sie die Schleife in die Kältekammer des Mikrotoms ein und warten Sie, bis das Tröpfchen zu gefrieren beginnt (typischerweise 5-7 s). Nehmen Sie dann die Abschnitte sofort auf, indem Sie den Tropfen schnell, aber vorsichtig dagegen drücken. Entfernen Sie die Schlaufe aus der Kältekammer, warten Sie, bis das Tröpfchen vollständig aufgetaut ist, und drücken Sie das Tröpfchen auf ein vorbereitetes Gitter (Schritt 1.2.6).
HINWEIS: Gitter mit Querschnitten können bei 4 °C mehrere Monate gelagert werden.
4. Markierung und Lichtmikroskopie
- Kennzeichnung
- Legen Sie die Gitter mit den Abschnitten (Abbildung 1A) mit der Schnittseite nach unten auf ~1 ml PBS in eine kleine Schale oder eine Multi-Well-Platte. Bei 37 °C 30 min inkubieren.
HINWEIS: In diesem Schritt wird die Gelatine entfernt, die sich zwischen den Zellen befindet. Gelatine wird nach dem Schneiden nicht benötigt und stört das restliche Protokoll. - Verarbeiten Sie die Gitter mit der Schnittseite nach unten auf ~75 μl-Tröpfchen auf Parafilm (siehe Abbildung 1B). Beginnen Sie mit PBS + 0,15% Glycin-Waschungen (3 x 2 min) bei RT. Dann werden die Gitter mit 0,1 % Rinderserumalbumin (BSA)-c + 0,5 % Fischhautgelatine (FSG) in PBS für 10 Minuten bei RT als Blockierungsschritt inkubiert. Verdünnen Sie die Primärantikörper in 0,1 % BSA-c + 0,5 % FSG in PBS und inkubieren Sie die Gitter auf ~10 μl-Tröpfchen dieser Lösung für 1 h bei RT (Abbildung 1C).
- Waschen Sie die Gitter in 0,1 % BSA in PBS 5x bei RT. Anschließend werden sekundäre Antikörper und 4',6-Diamidino-2-phenylindol (DAPI; 10 μg/ml) in 0,1 % BSA-c + 0,5 % FSG in PBS verdünnt und die Gitter auf ~10 μl-Tröpfchen dieser Lösung für 30+ min bei RT inkubiert (Abbildung 1C). Waschen Sie die Gitter in PBS 5x bei RT.
HINWEIS: Optional kann ein sekundärer Antikörper mit 5, 10, 15 oder 20 nm kolloidalen Goldpartikeln markiert werden, die mit Protein A (PAG) konjugiert sind, um das Protein von Interesse in EM zu lokalisieren. Wenn dies gewünscht ist, inkubieren Sie die Gitter mit PAG für 20 min bei RT nach Schritt 4.1.3. Waschen Sie dann 5x mit PBS bei RT. Vermeiden Sie die gleichzeitige Verwendung mehrerer Primärantikörper und achten Sie auf die Reaktivität von PAG auf die IgGs verschiedener Spezies, um unerwünschte Kreuzreaktionen zu vermeiden. Weitere Informationen finden Sie unter12.
- Legen Sie die Gitter mit den Abschnitten (Abbildung 1A) mit der Schnittseite nach unten auf ~1 ml PBS in eine kleine Schale oder eine Multi-Well-Platte. Bei 37 °C 30 min inkubieren.
- Einbetten von Proben für die Lichtmikroskopie
- Tauchen Sie die Gitter in 50 % Glycerin in dH 2 O2x 5 min bei RT. Legen Sie die Gitter zwischen einem Objektträger und einem Deckglas in 50 % Glycerin, ein Gitter pro Deckglas, mit Abschnitten zum Deckglas (Abbildung 1D).
HINWEIS: Die Qualität der Etikettierung kann sich verschlechtern, wenn die Gitter länger als 30 Minuten in 50 % Glycerin montiert sind. Es wird daher empfohlen, zwei oder drei Gitter gleichzeitig zu montieren und abzubilden und die anderen auf der sekundären Beschriftungslösung zu belassen.
- Tauchen Sie die Gitter in 50 % Glycerin in dH 2 O2x 5 min bei RT. Legen Sie die Gitter zwischen einem Objektträger und einem Deckglas in 50 % Glycerin, ein Gitter pro Deckglas, mit Abschnitten zum Deckglas (Abbildung 1D).
- Lichtmikroskopie
- Bringen Sie einen Objektträger mit Sandwichgittern zu einem Weitfeldmikroskop mit einem automatisierten Tisch. Wählen Sie ein Ölobjektiv mit hoher Vergrößerung (63-fach oder 100-fach). Erstellen Sie ein Bildkachelset (eines Teils davon) des Menübands mit Abschnitten (Abbildung 1E).
HINWEIS: Einige sekundäre Antikörper können fluoreszierende Aggregate auf Gittern bilden, insbesondere um Falten oder Risse in den Abschnitten. Darüber hinaus enthalten einige Zelltypen und Gewebe autofluoreszierende Strukturen. Wenn solche Probleme zu erwarten sind, wird empfohlen, ein Negativkontrollgitter beizufügen, das nicht mit primären Antikörpern inkubiert wurde.
- Bringen Sie einen Objektträger mit Sandwichgittern zu einem Weitfeldmikroskop mit einem automatisierten Tisch. Wählen Sie ein Ölobjektiv mit hoher Vergrößerung (63-fach oder 100-fach). Erstellen Sie ein Bildkachelset (eines Teils davon) des Menübands mit Abschnitten (Abbildung 1E).
- Abmontage und EM-Kontrastierung
- Geben Sie 10 μl dH2O an die Seite des Glasträger-Deckglas-Sandwiches und warten Sie, bis die Kapillarwirkung die Glas-Deckglas-Sandwich-Schnittstelle gefüllt hat. Entfernen Sie vorsichtig das Deckglas, ohne Immersionsöl in das Glycerin zu mischen. Holen Sie die Gitter mit einer Pinzette heraus und tauchen Sie sie in dH2O 3x bei RT, um das 50%ige Glycerin abzuwaschen.
HINWEIS: Öl kann die Uranylfärbung stören und den EM-Kontrast verschlechtern. - Trocknen Sie die Rückseite des Gitters vorsichtig mit fusselfreiem Seidenpapier ab.
HINWEIS: Wenn die Probe auch mit kolloidalen Goldpartikeln markiert wurde, führen Sie die folgenden Schritte durch: Legen Sie das Gitter mit Abschnitten mit der Schnittseite nach unten auf PBS-Tröpfchen und waschen Sie es 2x bei RT. Postfix in 1% GA für 5 min bei RT (siehe Warnhinweis unter 1.1.4). 2x bei RT in PBS waschen. - Die Gitter mit der Sektion nach unten auf dH2O Tröpfchen legen und 8x bei RT waschen.
- Um die Kontrastschnitte bei EM zu färben, inkubieren Sie mit Uranylacetat (UA), pH 7, für 5 Minuten bei RT (Abbildung 1F).
- Bevor Sie die Gitter platzieren, kühlen Sie die UA:Methylcellulose, pH 4, ab, indem Sie Tröpfchen auf Parafilm auf eine Metallplatte auf Eis legen. Waschen Sie dann die Gitter mit eiskalter UA:Methylcellulose, pH 4, 2x und inkubieren Sie sie 10 Minuten lang mit eiskaltem UA:Methylcellulose, pH 4 (Abbildung 1F).
ACHTUNG: Uranylacetat ist eine gefährliche Chemikalie (Gefahrenhinweise H300, H330, H373, H411). Bei Schritten, die UA erfordern, arbeiten Sie in einer chemischen Haube und tragen Sie Schutzausrüstung (Laborkittel, Handschuhe und Schutzbrille). Sammeln und entsorgen Sie UA-haltige Abfälle gemäß den Richtlinien und Vorschriften des Instituts. - Schlaufe die Gitter aus, indem du eine Gittertrocknungsschleife in das UA:Methylcellulose-Tröpfchen unterhalb des Gitters einführst und vorsichtig anhebst, bis das Gitter von dem Tropfen12 abgezogen wird. Tupfen Sie die überschüssige UA:Methylcellulose ab, indem Sie die Schlaufe in einem Winkel von ~60° (Abschnitte nach unten) auf fusselfreies Filterpapier (siehe Materialtabelle) berühren und langsam über das Papier ziehen, bis keine UA:Methylcellulose mehr absorbiert wird. Legen Sie dann die Schlaufe mit dem Gitter in ein geeignetes Gestell und lassen Sie sie >10 Minuten bei RT trocknen (Abbildung 1G).
- Geben Sie 10 μl dH2O an die Seite des Glasträger-Deckglas-Sandwiches und warten Sie, bis die Kapillarwirkung die Glas-Deckglas-Sandwich-Schnittstelle gefüllt hat. Entfernen Sie vorsichtig das Deckglas, ohne Immersionsöl in das Glycerin zu mischen. Holen Sie die Gitter mit einer Pinzette heraus und tauchen Sie sie in dH2O 3x bei RT, um das 50%ige Glycerin abzuwaschen.
5. EM
- Nutzen Sie die lichtmikroskopische Übersicht, um eine Region of Interest (ROI) für die Bildgebung im Transmissionselektronenmikroskop (TEM; Abbildung 1H). Kommentieren Sie den ROI für den Lichtmikroskopie-Datensatz. Sobald eine Region ausgewählt ist, erhalten Sie ein Bildkachelset mit 20.000-50.000-facher Vergrößerung im TEM. Rekonstruieren Sie das Bild-Tileset in der Nachbearbeitungssoftware15,16.
6. Korrelation und Analyse
- Laden Sie den Lichtmikroskopie- und EM-Datensatz in eine geeignete Bildbearbeitungssoftware, z. B. ImageJ/Fiji17, das ec-CLEM-Plug-in in Icy18 oder Photoshop. Schneiden Sie das Lichtmikroskopie-Dataset zu und drehen Sie es, damit es mit dem EM-Kachelset übereinstimmt.
- Führen Sie die Korrelation basierend auf dem DAPI-Signal in Fluoreszenz und den Kernkonturen in EM durch (Abbildung 1I). Verschieben Sie die Bilder, um sie präzise zu überlagern und die manuelle Korrelation genau durchzuführen. Um den Ansatz genauer zu machen, wenden Sie eine landmarkenbasierte Korrelation an, z. B. durch das ec-CLEM-Plugin in Icy oder das BigWarp-Plugin in ImageJ, um die Bilder durch manuelle Auswahl entsprechender Punkte zu korrelieren. Ein detailliertes Schritt-für-Schritt-Protokoll für die Korrelation mit ec-CLEM ist verfügbar19.
ANMERKUNG: Dieser Ansatz funktioniert auch gut mit der Verwendung von bimodalen Passermarkensonden20,21.
- Führen Sie die Korrelation basierend auf dem DAPI-Signal in Fluoreszenz und den Kernkonturen in EM durch (Abbildung 1I). Verschieben Sie die Bilder, um sie präzise zu überlagern und die manuelle Korrelation genau durchzuführen. Um den Ansatz genauer zu machen, wenden Sie eine landmarkenbasierte Korrelation an, z. B. durch das ec-CLEM-Plugin in Icy oder das BigWarp-Plugin in ImageJ, um die Bilder durch manuelle Auswahl entsprechender Punkte zu korrelieren. Ein detailliertes Schritt-für-Schritt-Protokoll für die Korrelation mit ec-CLEM ist verfügbar19.
- Analysieren Sie die korrelierten Bilder, indem Sie ROIs basierend auf dem Fluoreszenzsignal in einem geeigneten Programm (z. B. ImageJ) auswählen. Erstellen Sie für die quantitative Analyse eine Sammlung von ROIs für alle gekennzeichneten Organellen. Inspizieren Sie dann die entsprechende Ultrastruktur der einzelnen ROIs und klassifizieren Sie sie anhand morphologischer Elemente.
Ergebnisse
Ein optimiertes Immuno-EM-Protokoll für die Immuno-Gold-Markierung von LC3 auf ultradünnen Kryoschnitten wurde kürzlich von De Maziere et al. veröffentlicht.9. Diese Studie umfasste hungernde Bedingungen ohne BafA1, in denen LC3 zwar vorhanden, aber relativ selten und für EM schwer zu finden war. In einer separaten Studie wurde eine On-Section-CLEM-Methode vorgestellt, die die Sensitivität der Fluoreszenzmarkierung nutzt, um relativ seltene und niedrig exprimierte endogene Proteine sichtbar zu machen und diese mit der EM-Ultrastruktur zu korrelieren14. Hier werden diese beiden Ansätze durch die Verwendung des optimierten LC3-Markierungsprotokolls im Rahmen eines CLEM-Ansatzes kombiniert.
HEPG2-Zellen, aus der Leber stammende Zellen mit relativ hoher basaler Autophagie22, wurden vor der Fixierung in 4% PFA 2 h lang in minimalem Medium (Earle's balanced salt solution [EBSS]) ausgehungert. Darauf folgte die Probenvorbereitung mit der Tokuyasu-Methode der ultradünnen Kryosektion (Abschnitte 1-3; siehe Slot und Geuze12), die sehr gut mit dem On-Sektions-CLEM 14,23 kompatibel ist. Die aufgetauten Kryoschnitte wurden mit einem Maus-Anti-LC3-Primärantikörper9 fluoreszenzmarkiert (Protokollabschnitt 4 und Abbildung 1). Zusätzlich wurde Kaninchen-Anti-LAMP1 verwendet, um Endo-Lysosomen anzuzeigen, gefolgt von Anti-Maus-AlexaFluor488- und Anti-Kaninchen-AlexaFluor568-Sekundärantikörpern. Die Gitter wurden zwischen einem Deckglas und einem Objektträger platziert und mit RT auf einem Weitfeldmikroskop (100x 1,47 NA Ölobjektiv, sCMOS-Kamera) abgebildet.
Ein Vorteil der Fluoreszenzmarkierung von Dünnschliffen gegenüber herkömmlichen Ganzzell-IF ist die erhöhte Auflösung in Z, da die physikalische Dicke des Schnitts 60-90 nm beträgt. Mit dieser verbesserten Z-Auflösung zeigt die Fluoreszenzmarkierung von LC3 und LAMP1 auf Dünnschliffen nur eine sehr geringe Kolokalisation (Abbildung 2A). In Zellen, die mit lysosomalen Inhibitoren wie BafA1 behandelt wurden, kommt es zu einer hohen Kolokalisation, da lysosomal-umschlossenes LC3 nicht abgebaut wird9. In unbehandelten Zellen wird LC3 bei Kontakt mit enzymatisch aktiven, LAMP1-positiven Lysosomen schnell abgebaut, so dass eine Kolokalisation unter diesen Bedingungen selten ist. Im Allgemeinen wurde weniger als ein LC3-Punctum pro Zellprofil beobachtet. Dies deutet darauf hin, dass selbst unter Hungerbedingungen der Umsatz von Autophagosomen schnell abläuft, wodurch die Anzahl der Autophagosomen niedrig bleibt. Es unterstreicht auch, wie wichtig es ist, CLEM zu verwenden, um die seltenen LC3-markierten Strukturen zu finden, indem das große Sichtfeld der Lichtmikroskopie genutzt wird. Darüber hinaus ermöglicht die höhere Sensitivität der Fluoreszenzmarkierung im Vergleich zur Goldmarkierung die Identifizierung von mehr LC3-positiven Organellen als bei der herkömmlichen Immun-EM, was ihre Charakterisierung weiter unterstützt.
Nach der Erfassung eines vollständigen Kachelsatzes des Schnittbandes wurden die Gitter aus dem Mikroskop entnommen und mit UA und der Loop-Out-Methode für EM nachgefärbt (Protokollschritte 4.4-4.6; Abbildung 1F,G). Diese "Loop-Out"-Methode sorgt dafür, dass eine dünne Schicht UA:Methylcellulose auf dem Gitter verbleibt, wodurch der gewünschte Kontrast im EM entsteht. Die Dicke der Schicht hängt von der Geschwindigkeit und dem Winkel ab, mit dem die UA:Methylcellulose auf das Filterpapier abgetupft wird. Wenn Sie die Schleife zu schnell ziehen, kann zu viel UA:Methylcellulose auf dem Raster zurückbleiben und das Erscheinungsbild der Abschnitte in EM verdunkeln. Zu langsames Ziehen kann zu viel UA:Methylcellulose wegziehen, was zu zu wenig Färbung und schlechter Morphologie führt und das Risiko birgt, dass das Gitter aus der Schleife fällt. Die "Ölteppich"-Färbung (Abbildung 1G) auf trockenen Gittern weist auf eine geeignete UA:Methylcellulose-Schichtdicke hin.
Nach dem Loop-Out und der Trocknung wurden die Gitter in einem TEM mit ROIs abgebildet, die durch Fluoreszenz ausgewählt wurden. Die IF- und EM-Datensätze wurden korreliert, indem das DAPI-Signal mit den Umrissen der im EM sichtbaren Kerne überlagert wurde, wodurch ein integriertes Bild mit Informationen beider Modalitäten erzeugt wurde.
Es kann eine Herausforderung sein, den gleichen Bereich in EM zu finden, der in IF ausgewählt wurde. Es empfiehlt sich daher, bei der Suche im EM ein Übersichtsbild des IF-Tilesets griffbereit zu haben. Benutzer sollten in beiden Modalitäten nach erkennbaren Merkmalen suchen, wie z. B. Falten oder Risse in den Abschnitten, Gitterstäben oder der Anordnung der Kerne. Es ist auch wichtig zu bedenken, dass die Probe in EM gedreht und gespiegelt erscheinen kann. "Finder-Raster" mit spezifischen Merkmalen zur Identifizierung von Bereichen können verwendet werden, um die Korrelation zu erleichtern (siehe Materialtabelle).
Die Korrelation der LC3-positiven Organellen mit der EM-Ultrastruktur zeigte, dass die verschiedenen Puncta unterschiedliche Stadien der Autophagie repräsentierten (Abbildung 2B). Obwohl die Erhaltung der autophagosomalen Ultrastruktur in Kryoschnitten eine Herausforderung darstellt, wurden häufig Organellen mit zytoplasmatischem Gehalt und Doppelmembranen beobachtet (Abbildung 2C, Pfeile in Organellen 1-5; Ergänzende Abbildung S1), die morphologische Merkmale von Autophagosomen definieren. Interessanterweise wurden eher schwache fluoreszierende Spots mittels EM als LC3-positive Autolysosomen identifiziert (Abbildung 2C, Organelle 6; autophagischer Gehalt ist markiert *), die sich durch dichten Inhalt und intraluminale Vesikel auszeichnen. Dies zeigte, dass sehr geringe Mengen an LC3 in IF von ultradünnen Kryoschnitten sichtbar sind, und deutete darauf hin, dass trotz des abbauenden Milieus etwas LC3 in stationären Autolysosomen nachweisbar ist. Die Mehrzahl der LC3-positiven Puncta repräsentierte jedoch Autophagosomen, während Autolysosomen sehr selten waren. Dies steht im Gegensatz zu BafA1-behandelten Zellen, die in erster Linie Autolysosomen und keine Autophagosomen akkumulieren9.
Zusammenfassend beschreibt dieses Protokoll eine On-Section-CLEM-Methode zur Verknüpfung von molekularen Informationen, die durch Fluoreszenzmikroskopie gewonnen wurden, mit der Ultrastruktur von EM. Diese Methode erhöht die Sensitivität von Immuno-EM, da nur Fluorophore für die Markierung verwendet werden und diese in der Regel mehr Signal liefern als EM-Sonden. Die Methode eignet sich besonders für den Einsatz von ultradünnen Kryoschnitten, bei denen ein hohes Maß an spezifischer Fluoreszenz über vernachlässigbare Hintergrundfärbungen erzielt werden kann. Durch die Verwendung von Fluoreszenz zum Screening nach seltenen Strukturen oder Ereignissen und die Korrelation ausgewählter ROIs mit EM können die EM-Betriebszeit und die damit verbundenen Kosten erheblich gesenkt werden. Die Sensitivität und Durchführbarkeit der Methode wird durch die Visualisierung von LC3 in unbehandelten, ausgehungerten Zellen demonstriert, die zeigt, dass LC3 unter diesen Bedingungen überwiegend an Autophagosomen assoziiert, wobei in Autolysosomen nur sehr geringe Konzentrationen sichtbar sind.
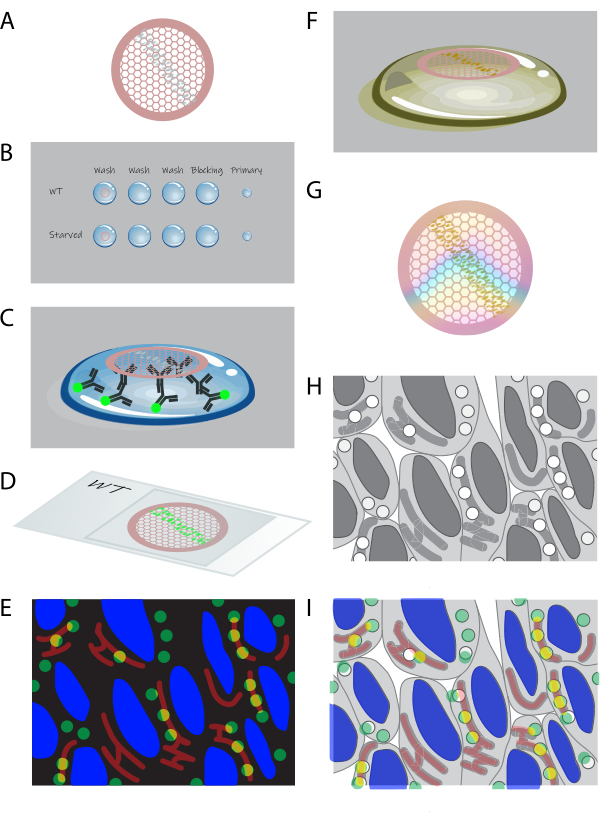
Abbildung 1: Schematische Übersicht über CLEM im Querschnitt . (A) Kryoschnitte von in Gelatine eingebetteten Zellen werden auf einem formvarbeschichteten Kupfergitter gesammelt. (B) Die Gitter werden abschnittsweise auf Tröpfchen der entsprechenden Lösungen verarbeitet. (C) Die Gitter sind mit primären und fluoreszierenden sekundären Antikörpern markiert. (D) Die Gitter werden zwischen einem Deckglas und einem Objektträger aus 50 % Glycerin angeordnet. (E) Fluoreszenzbilder werden in einem Weitfeldmikroskop aufgenommen. (F) Die Gitter werden aus dem Objektträger entnommen und durch Uranylfärbung für EM weiterverarbeitet. (G) Nach dem Trocknen können die Gitter mittels TEM abgebildet werden. (H) TEM-Bildkacheln mit hoher Vergrößerung werden aus einem Bereich aufgenommen, der aus Fluoreszenzdaten ausgewählt wurde. (I) Bilder aus Fluoreszenzmikroskopie und EM werden korreliert und überlagert. Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
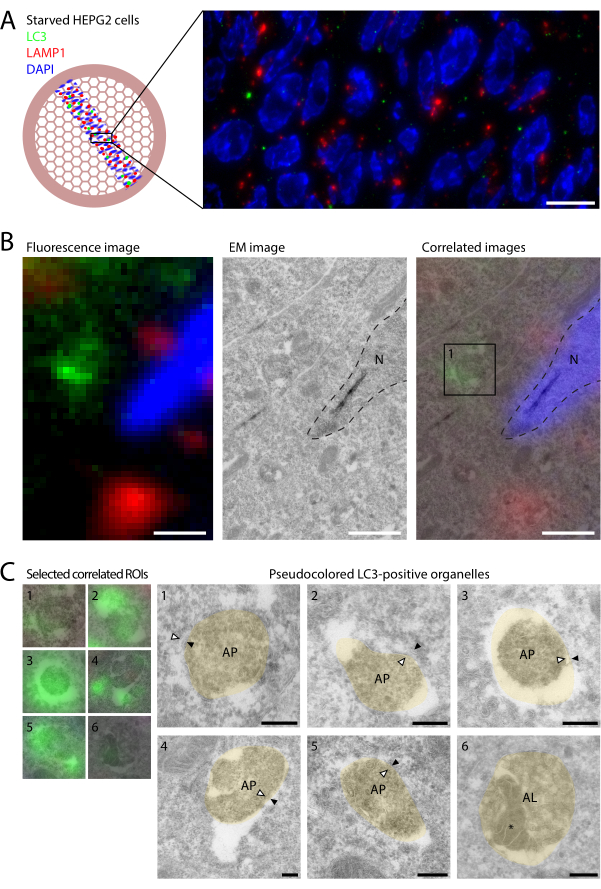
Abbildung 2: CLEM von LC3 und LAMP1 in ausgehungerten HEPG2-Zellen. HEPG2-Zellen wurden vor der Fixierung mit 4% PFA für 2 h in EBSS für 2 h ausgehungert. (A) IF-Bildgebung von LC3 (grün) und LAMP1 (rot) auf Schnitten zeigt relativ wenige LC3-Punkte und eine geringe Kolokalisation mit LAMP1. (B) Verknüpfung der molekularen Informationen von IF (linkes Bild) mit den ultrastrukturellen Informationen, die in EM (mittleres Bild) erhalten wurden, durch Überlagerung der beiden Bildgebungsmodalitäten basierend auf DAPI und Kernkonturen (gestrichelte Linien, rechtes Bild). Die Ultrastruktur der einzelnen LC3-markierten Kompartimente, wie sie in Kasten 1 (rechtes Bild) dargestellt ist, ist in C dargestellt. (C) Ultrastruktur von LC3-positiven Kompartimenten. CLEM-Bilder sind auf der linken Seite und pseudofarbige (beigefarbene) EM-Bilder auf der rechten Seite zu sehen (ungefärbte EM-Bilder sind in der ergänzenden Abbildung S1 dargestellt). Innere und äußere autophagosomale Membranen sind durch weiße bzw. schwarze Pfeilspitzen gekennzeichnet. Der autophagische Gehalt innerhalb des Autolysosoms in Beispiel 6 wird durch * angegeben. Maßstabsbalken = 10 μm (A), 1 μm (B), 200 nm (C). Bitte klicken Sie hier, um eine größere Version dieser Abbildung anzuzeigen.
Ergänzende Abbildung S1: Ungefärbte EM-Bilder von LC3-positiven Organellen. (A-F) Ungefärbte EM-Bilder der pseudofarbigen Beispiele 1-6, die in Abbildung 2C gezeigt sind. Die Organellen wurden durch LC3-Fluoreszenz ausgewählt, wie in Abbildung 2 beschrieben. Innere und äußere autophagosomale Membranen sind durch weiße bzw. schwarze Pfeilspitzen gekennzeichnet. Der autophagische Gehalt innerhalb des Autolysosoms in Beispiel 6 wird durch * angegeben. Maßstabsbalken = 200 nm. Abkürzungen: AL = Autolysosom; AP = Autophagosom; M = Mitochondrium. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Ergänzende Datei 1: In dieser Studie verwendete Puffer und Lösungen. Diese ergänzende Datei enthält die Rezepte und Protokolle, die zur Herstellung der in dieser Studie verwendeten Puffer und Lösungen erforderlich sind. Bitte klicken Sie hier, um diese Datei herunterzuladen.
Diskussion
Die hier vorgestellte Methode nutzt die jüngsten Fortschritte in der Kryosektions-basierten On-Section-CLEM - die hohe Sensitivität der IF-Markierung und die genaue (<100 nm Fehler) Korrelation zwischen FM und EM14,24. Dies führt zu einer Methode mit der Empfindlichkeit, seltene, endogene Proteine fluoreszenzmarkiert zu markieren und diese mit hoher Präzision auf die EM-Ultrastruktur zu überlagern. Somit vermeidet diese Methode die Notwendigkeit einer (Über-)Expression von exogen markierten Proteinen und die Verwendung weniger empfindlicher EM-Markierungen. Die Machbarkeit der Methode wird anhand von CLEM-Beispielen an endogenem LC3 in ausgehungerten Zellen ohne den Einsatz lysosomaler Inhibitoren gezeigt.
Aufgetaute Kryoschnitte, die mit der Tokuyasu-Methode gewonnen wurden, sind ideale Proben für die Immun-EM, da sie im Gegensatz zu Harzschnitten durchlässig für Antikörper sind. In Kombination mit milder Fixierung und kontrastierenden Verfahren führt dies im Allgemeinen zu einer hervorragenden Markierungseffizienz gegenüber anderen Methoden, ohne die detaillierte Ultrastruktur zu beeinträchtigen, und visualisiert Zellmembranen hervorragend 12,25,26. Darüber hinaus sind Kryoschnitte sehr gut kompatibel mit der Fluoreszenzmikroskopie, was sie zu wertvollen Substraten für CLEM macht. Sowohl die klassische Immunogold-Markierung als auch CLEM auf Kryosektionen haben bahnbrechende Einblicke in das Verständnis der subzellulären Organisation geliefert 14,27,28,29,30.
Derzeit werden CLEM auf aufgetaute Kryoschnitte immer häufiger angewendet, was auf kontinuierliche Entwicklungen und Optimierungenzurückzuführen ist 14,20,24,31,32,33,34, die die Qualität, Anwendbarkeit und Genauigkeit des Ansatzes verbessert haben. Durch die genaue Korrelation großer ZF- und EM-Bildkacheln erleichtert die Technik nun das Screening auf die Ultrastruktur fluoreszenzmarkierter endogener zellulärer Komponenten 14,32,33. Dies ist ein Vorteil gegenüber der klassischen Immuno-EM, bei der die Suche nach goldmarkierten Strukturen typischerweise eine hohe Vergrößerung erfordert und daher aufwändiger und zeitintensiver ist. Aus diesem Grund profitiert die Lokalisierung von LC3 in der Ultrastruktur stark von CLEM. LC3-positive Organellen treten häufig auf, wenn die autophagische Clearance blockiert ist (d. h. wenn Zellen mit BafA1 oder pH-erhöhenden Mitteln behandelt werden), während autophagische Organellen in unveränderten oder ausgehungerten Zellen schnell beseitigt werden, was zu sehr niedrigen Steady-State-Werten führt. Unter solchen Bedingungen kann es schwierig sein, LC3-markierte Organellen mit klassischer Immun-EM zu finden, und CLEM bietet einen klaren Vorteil.
Zuvor wurde CLEM auf Harzschnitten in Studien mit ektopischer Expression von LC3-GFP oder einer LC3-GFP-RFP-Tandemsonde angewendet 35,36,37,38,39. In diesen Studien wurde eine Fluoreszenzbildgebung vor dem Einbetten oder direkt in Acrylharzabschnitten40 durchgeführt, und die Proben wurden anschließend durch EM gescreent. Es gibt mehrere Vorteile der Harzeinbettung; Die autophagosomale Ultrastruktur ist im Allgemeinen gut erhalten, insbesondere wenn das Material unter Hochdruck gefroren ist40. Darüber hinaus ist der Kontrast von schwermetallgefärbtem, harzeingebettetem Material im Allgemeinen ausgeprägter als der von uranylgefärbten Kryoschnitten. In Harz eingebettete Schnitte sind mit volumetrischen EM-Methoden wie Array-Tomographie, FIB-REM oder seriellem Blockface-REM kompatibel, Kryoschnitte hingegen nicht. Bei Ansätzen, bei denen vor dem Einbetten eine Bildgebung durchgeführt wird, ist die Bildgebung lebender Zellen eine Option41, die in CLEM für Kryoschnitte nicht verfügbar ist. Der Hauptvorteil von CLEM bei Kryosektionen gegenüber diesen Alternativen ist das hohe IF-Signal, das eine Immunlokalisierung seltener Proteine ermöglicht, ohne dass eine Membranpermeabilisierung oder Überexpression erforderlich ist. Dies vermeidet eine mögliche Membranextraktion, Überexpressionsarefakte42 und genetische Modifikationen des Probanden, was es in Kombination mit der Möglichkeit, große Bereiche in IF und EM zu korrelieren, zu einem hervorragenden Werkzeug für die Untersuchung von LC3 und Autophagie macht.
Hier zeigte die Anwendung von On-Section-CLEM auf ausgehungerte HEPG2-Zellen, dass LC3 hauptsächlich in Strukturen lokalisiert ist, die als Autophagosomen identifiziert wurden. Zusätzlich wurden einige schwach fluoreszierende Flecken in Autolysosomen gefunden. Dies steht in direktem Gegensatz zu Zellen, die mit BafA19 behandelt wurden, und spiegelt den schnellen Abbau autophagosomaler Proteine wider, sobald das Autophagosom mit den Lysosomen fusioniert. Insgesamt zeigten die Daten, dass CLEM von aufgetauten Kryoschnitten Einblicke in die LC3-vermittelte Autophagie unter nativen Bedingungen geben kann. Die Daten unterstreichen auch die Empfindlichkeit der Technologie, da LC3 auch in Autolysosomen nachgewiesen wurde, die nur geringe Mengen an intakten LC3-Epitopen enthalten. Die weitere Anwendung dieser Technik durch die Abbildung von LC3 in verschiedenen Modellen und Bedingungen wird unser Verständnis der Autophagie und anderer LC3-vermittelter biologischer Prozesse, wie z.B. der LC3-assoziierten Phagozytose oder der Konjugation von ATG8 mit einzelnen Membranen, verbessern.
Über die Autophagie hinaus kann CLEM im Schnitt auch auf andere seltene Ereignisse oder Strukturen angewendet werden, wie z. B. Zellteilung, Infektion, seltene Zelltypen in Geweben, Kinetochoren, primäre Zilien oder zelltypspezifische Organellen. Ein effektives Screening des interessierenden Themas durch IF kann die ultrastrukturelle Untersuchung dieser Raritäten erheblich erleichtern. Des Weiteren konnte gezeigt werden, dass mit der Technik Proteine empfindlicher lokalisiertwerden können als mit der klassischen Immun-EM. Die Anpassung der Fixationslänge kann diese Empfindlichkeit weiter verlängern und die ultrastrukturelle Lokalisierung von sehr niedrig oder schlecht antigenen Proteinen ermöglichen. Schließlich erleichtert die On-Section-CLEM-Methode die schnelle Selektion einer quantitativen Anzahl von Organellen und ermöglicht so eine robustere Analyse der ultrastrukturellen Verteilung eines bestimmten Proteins.
CLEM für Kryosektionen erfordert die Ausrüstung und das Fachwissen für die Kryosektion. In Gruppen mit Zugang zu diesen Werkzeugen (z. B. Kryomikrotomen) ist die Implementierung von On-Section-CLEM unkompliziert und erfordert nur die Verfügbarkeit eines automatisierten Weitfeldmikroskops, zu dem die meisten Labore Zugang haben. Darüber hinaus ist die Methode in EM-Einrichtungen weltweit verfügbar. Da die On-Section-CLEM die Anwendung etablierter IF- und EM-Methoden kombiniert, ist die Methode leicht anpassbar und kann beispielsweise mit der Tomographie 20,33,43, der seriellen Schnittvolumen-EM einer begrenzten Anzahl von Abschnitten 44 oder der superauflösenden Mikroskopie 45 kombiniert werden. Diese Vielseitigkeit der Methode unterstützt die Anwendung auf ein breites Spektrum biologischer Fragestellungen.
Offenlegungen
Die Autoren erklären, dass es keine Interessenkonflikte gibt.
Danksagungen
Wir danken unseren Kolleginnen und Kollegen am Zentrum für Molekulare Medizin des Universitätsklinikums Utrecht für die fruchtbaren Gespräche und Rückmeldungen. Wir danken ehemaligen und aktuellen Kollegen des Klumperman-Labors für die kontinuierliche Verbesserung unserer Mikroskopietechnologien. Die für diese Arbeit verwendete EM-Infrastruktur ist Teil des Forschungsprogramms National Roadmap for Large-Scale Research Infrastructure (NEMI), das vom niederländischen Forschungsrat (NWO) finanziert wird, Projektnummer 184.034.014 an JK.
Materialien
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Antibody donkey anti-mouse Alexa Fluor 488 | Life Technologies | #A21202 | use 1:250 |
| Antibody donkey anti-rabbit Alexa Fluor 568 | Life Technologies | A#10042 | use 1:250 |
| Antibody mouse anti-LC3 | Cosmo Bio | CTB-LC3-2-IC | use 1:100 |
| Antibody rabbit anti-LAMP1 | Cell Signaling | 9091 | use 1:250 |
| Bovine serum Albumin, fraction V | Sigma-Aldrich | A-9647 | |
| BSA-c | Aurion | 900.099 | |
| BSA-conjugated gold | Cell Microscopy Core, UMC Utrecht | BSAG 5 nm | |
| Water-free Chloroform | Merck | 1.02447.0500 | |
| DAPI | Invitrogen | 10184322 | Use at end concentration of 10 µg/ml |
| EGTA | Sigma-Aldrich | E4378 | |
| Fish-skin Gelatin | Sigma-Aldrich | G7765 | |
| Food-grade gelatin | Merck | G1890 | |
| Formvar, Vinylec E | SPI | 02492-RA | |
| Gluteraldehyde | Serva | 23115.01 | See CAUTION note |
| Glycerol | Boom | MBAK 7044.1000 | |
| Glycine | Merck | 1042010250 | |
| HEPES | Sigma-Aldrich | H3375 | |
| Methylcellulose, 25 centipoises | Sigma-Aldrich | M-6385 | |
| MgSO4 | Riedel-de Haen | 12142 | |
| Na2HPO4 (PB component A) | Merck | 106580-0500 | |
| NaBH4 | Merck | 806373 | |
| NaH2PO4 (PB component B) | Merck | 106346 | |
| NH4OH | Sigma-Aldrich | 221228-0025 | |
| Oxalic acid | Merck | 100495 | |
| Paraformaldehyde prills | Sigma-Aldrich | 441244 | See CAUTION note |
| PIPES | Merck | 110220 | |
| Protein-A conjugated gold | Cell Microscopy Core, UMC Utrecht | PAG 5, 10, 15 or 20 nm | |
| Sucrose D(+) | VWR | 27483294 | |
| Uranyl acetate | SPI | 020624-AB | See CAUTION note |
| Tools and consumables | |||
| Pick-up loop | Electron Microscopy Sciences | 70944 | |
| Filter paper, qualitative, medium-fast | LLG | 6.242 668 | |
| Finder grids | Ted Pella | G100F1 | |
| Grids | Cell Microscopy Core, UMC Utrecht | CU 100 mesh | |
| Microscopes | |||
| Leica Thunder widefield microscope | Leica | Components: 100x, 1.47 NA TIRF objective; Photometrics prime 95B sCMOS camera; LAS X software; | |
| Leica UC7 ultracryomicrotome | Leica | ||
| Tecnai T12 | FEI | Components: Veleta VEL-FEI-TEC12-TEM camera; SerialEM software | |
| Software | |||
| ec-CLEM in icy | open source | Paul-Gilloteaux et al., 2017 | |
| Fiji | open source | Schindelin et al., 2012 | |
| IMOD | open source | Mastronarde et al., 2017 | |
| Photoshop | Adobe | ||
| SerialEM | open source | Mastronarde et al., 2018 |
Referenzen
- Hu, Y., Reggiori, F. Molecular regulation of autophagosome formation. Biochemical Society Transactions. 50 (1), 55-69 (2022).
- Reggiori, F., Ungermann, C. Autophagosome maturation and fusion. Journal of Molecular Biology. 429 (4), 486-496 (2017).
- Kimura, S., Noda, T., Yoshimori, T. Dissection of the autophagosome maturation process by a novel reporter protein, tandem fluorescent-tagged LC3. Autophagy. 3 (5), 452-460 (2007).
- Baba, M., Takeshige, K., Baba, N., Ohsumi, Y. Ultrastructural analysis of the autophagic process in yeast: detection of autophagosomes and their characterization. The Journal of Cell Biology. 124 (6), 903-913 (1994).
- Takeshige, K., Baba, M., Tsuboi, S., Noda, T., Ohsumi, Y. Autophagy in yeast demonstrated with proteinase-deficient mutants and conditions for its induction. The Journal of Cell Biology. 119 (2), 301-311 (1992).
- De Duve, C., Wattiaux, R. Functions of lysosomes. Annual Review of Physiology. 28, 435-492 (1966).
- Arstila, A. U., Trump, B. F. Studies on cellular autophagocytosis. The formation of autophagic vacuoles in the liver after glucagon administration. The American Journal of Pathology. 53 (5), 687-733 (1968).
- Eskelinen, E. L., Reggiori, F., Baba, M., Kovács, A. L., Seglen, P. O. Seeing is believing: The impact of electron microscopy on autophagy research. Autophagy. 7 (9), 935-956 (2011).
- De Mazière, A., et al. An optimized protocol for immuno-electron microscopy of endogenous LC3. Autophagy. 18 (12), 3004-3022 (2022).
- López-Terrada, D., Cheung, S. W., Finegold, M. J., Knowles, B. B. Hep G2 is a hepatoblastoma-derived cell line. Human Pathology. 40 (10), 1512-1515 (2009).
- Tanida, I., Minematsu-Ikeguchi, N., Ueno, T., Kominami, E. Lysosomal turnover, but not a cellular level, of endogenous LC3 is a marker for autophagy. Autophagy. 1 (2), 84-91 (2005).
- Slot, J. W., Geuze, H. J. Cryosectioning and immunolabeling. Nature Protocols. 2 (10), 2480-2491 (2007).
- Waterman-Storer, C. M. Microtubule/organelle motility assays. Current Protocols in Cell Biology. 13 (1), (2001).
- vander Beek, J., de Heus, C., Liv, N., Klumperman, J. Quantitative correlative microscopy reveals the ultrastructural distribution of endogenous endosomal proteins. The Journal of Cell Biology. 221 (1), e202106044 (2022).
- Mastronarde, D. N. Advanced data acquisition from electron microscopes with SerialEM. Microscopy and Microanalysis. 24, 864-865 (2018).
- Mastronarde, D. N., Held, S. R. Automated tilt series alignment and tomographic reconstruction in IMOD. Journal of Structural Biology. 197 (2), 102-113 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Paul-Gilloteaux, P., et al. EC-CLEM: Flexible multidimensional registration software for correlative microscopies. Nature Methods. 14 (2), 102-103 (2017).
- Heiligenstein, X., Paul-Gilloteaux, P., Raposo, G., Salamero, J. eC-CLEM: A multidimension, multimodel software to correlate intermodal images with a focus on light and electron microscopy. Methods in Cell Biology. 140, 335-352 (2017).
- Fermie, J., et al. Bimodal endocytic probe for three-dimensional correlative light and electron microscopy. Cell Reports Methods. 2 (5), 100220 (2022).
- Fokkema, J., et al. Fluorescently labelled silica coated gold nanoparticles as fiducial markers for correlative light and electron microscopy. Scientific Reports. 8 (1), 13625 (2018).
- Czaja, M. J., et al. Functions of autophagy in normal and diseased liver. Autophagy. 9 (8), 1131 (2013).
- Robinson, J. M., Takizawa, T., Pombo, A., Cook, P. R. Correlative fluorescence and electron microscopy on ultrathin cryosections: Bridging the resolution gap. Journal of Histochemistry and Cytochemistry. 49 (7), 803-808 (2001).
- Mohammadian, S., et al. High accuracy, fiducial marker-based image registration of correlative microscopy images. Scientific Reports. 9 (1), 3211 (2019).
- Tokuyasu, K. T. A study of positive staining of ultrathin frozen sections. Journal of Ultrasructure Research. 63 (3), 287-307 (1978).
- Slot, J. W., Geuze, H. J. A new method of preparing gold probes for multiple-labeling cytochemistry. European Journal of Cell Biology. 38 (1), 87-93 (1985).
- Klumperman, J., Raposo, G. The complex ultrastructure of the endolysosomal system. Cold Spring Harbor Perspectives in Biology. 6 (10), a016857 (2014).
- Geuze, H. J., Slot, J. W., Strous, G. J., Lodish, H. F., Schwartz, A. L. Intracellular site of asialoglycoprotein receptor-ligand uncoupling: Double-label immunoelectron microscopy during receptor-mediated endocytosis. Cell. 32 (1), 277-287 (1983).
- Biazik, J., Ylä-Anttila, P., Vihinen, H., Jokitalo, E., Eskelinen, E. L. Ultrastructural relationship of the phagophore with surrounding organelles. Autophagy. 11 (3), 439-451 (2015).
- Fahimi, H. D., Reich, D., Völkl, A., Baumgart, E. Contributions of the immunogold technique to investigation of the biology of peroxisomes. Histochemistry and Cell Biology. 106 (1), 105-114 (1996).
- Vicidomini, G., et al. A novel approach for correlative light electron microscopy analysis. Microscopy Research and Technique. 73 (3), 215-224 (2010).
- Vicidomini, G., et al. High data output and automated 3D correlative light-electron microscopy method. Traffic. 9 (11), 1828-1838 (2008).
- Cortese, K., et al. 3D HDO-CLEM: cellular compartment analysis by correlative light-electron microscopy on cryosection. Methods in Cell Biology. 111, 95-115 (2012).
- van Rijnsoever, C., Oorschot, V., Klumperman, J. Correlative light-electron microscopy (CLEM) combining live-cell imaging and immunolabeling of ultrathin cryosections. Nature Methods. 5 (11), 973-980 (2008).
- Razi, M., Chan, E. Y. W., Tooze, S. A. Early endosomes and endosomal coatomer are required for autophagy. The Journal of Cell Biology. 185 (2), 305-321 (2009).
- Ligeon, L. A., Barois, N., Werkmeister, E., Bongiovanni, A., Lafont, F. Structured illumination microscopy and correlative microscopy to study autophagy. Methods. 75, 61-68 (2015).
- Biazik, J., Vihinen, H., Anwar, T., Jokitalo, E., Eskelinen, E. L. The versatile electron microscope: An ultrastructural overview of autophagy. Methods. 75, 44-53 (2015).
- Gudmundsson, S., Kahlhofer, J., Baylac, N., Kallio, K., Eskelinen, E. L. Correlative light and electron microscopy of autophagosomes. Methods in Molecular Biology. 1880, 199-209 (2019).
- Kriel, J., et al. Correlative light and electron microscopy (CLEM): bringing together the best of both worlds to study neuronal autophagy. Imaging and Quantifying Neuronal Autophagy. 171, 135-147 (2022).
- Largeau, C., Legouis, R. Correlative light and electron microscopy to analyze LC3 proteins in Caenorhabditis elegans embryo. Methods in Molecular Biology. 1880, 281-293 (2019).
- Fermie, J., et al. Single organelle dynamics linked to 3D structure by correlative live-cell imaging and 3D electron microscopy. Traffic. 19 (5), 354-369 (2018).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: Caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Ladinsky, M. S., Howell, K. E. Electron tomography of immunolabeled cryosections. Methods in Cell Biology. 79, 543-558 (2007).
- Oorschot, V., Lindsey, B. W., Kaslin, J., Ramm, G. TEM, SEM, and STEM-based immuno-CLEM workflows offer complementary advantages. Scientific Reports. 11 (1), 899 (2021).
- Franke, C., et al. Correlative single-molecule localization microscopy and electron tomography reveals endosome nanoscale domains. Traffic. 20 (8), 601-617 (2019).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten