Method Article
Localisation ultrastructurale de la LC3 endogène par microscopie électronique luminescente corrélative sur coupe
Dans cet article
Résumé
Nous présentons ici un protocole de microscopie électronique optique corrélative corrélative sur coupe basé sur le marquage endogène par fluorescence comme outil pour étudier la localisation de protéines rares en relation avec l’ultrastructure cellulaire. La puissance de cette approche est démontrée par la localisation ultrastructurale de la LC3 endogène dans des cellules affamées sans traitement par la bafilomycine.
Résumé
La visualisation des organites autophagiques au niveau ultrastructural par microscopie électronique (EM) est essentielle pour établir leur identité et révéler des détails importants pour la compréhension du processus autophagique. Cependant, les méthodes EM manquent souvent d’informations moléculaires, ce qui entrave la corrélation entre les informations ultrastructurales obtenues par EM et la localisation de protéines d’autophagie spécifiques par microscopie à fluorescence. De plus, la rareté des autophagosomes dans des conditions cellulaires non altérées entrave l’investigation par EM, qui nécessite un fort grossissement, et fournit donc un champ de vision limité.
En réponse à ces deux défis, une méthode de microscopie électronique corrélative en coupe (CLEM) basée sur le marquage fluorescent a été appliquée pour corréler un marqueur autophagosomique commun, LC3, à l’ultrastructure EM. La méthode a été utilisée pour cribler rapidement les cellules en microscopie à fluorescence pour le marquage LC3 en combinaison avec d’autres marqueurs pertinents. Par la suite, les caractéristiques ultrastructurales sous-jacentes de certaines taches marquées par LC3 ont été identifiées par CLEM. La méthode a été appliquée à des cellules affamées sans ajout d’inhibiteurs de l’acidification lysosomale.
Dans ces conditions, la LC3 a été retrouvée principalement sur les autophagosomes et rarement dans les autolysosomes, dans lesquels la LC3 est rapidement dégradée. Ces données montrent à la fois la faisabilité et la sensibilité de cette approche, démontrant que CLEM peut être utilisé pour fournir des informations ultrastructurales sur l’autophagie médiée par la LC3 dans des conditions natives, sans traitements médicamenteux ni altérations génétiques. Dans l’ensemble, cette méthode présente un outil précieux pour les études de localisation ultrastructurale des protéines d’autophagie et d’autres antigènes rares en reliant la microscopie optique aux données EM.
Introduction
L’autophagie est un processus clé pour l’élimination et le recyclage des protéines et des organites cytoplasmiques. Le processus de macro-autophagie (ci-après appelé autophagie) implique la formation d’organites à double membrane, les autophagosomes, qui permettent aux cellules d’enfermer des molécules cytoplasmiques et des organites pour la dégradation lysosomale. L’autophagie se produit au niveau basal dans la plupart des cellules et est régulée à la hausse en réponse à des conditions cellulaires, telles que la famine ou le stress cellulaire. L’autophagie se produit soit d’une manière spécifique au substrat, ciblant des structures ou des protéines spécifiques pour la dégradation, soit sous la forme d’un processus de masse non sélectif englobant des parties du cytosol. Dans l’autophagie sélective, les autophagosomes sont formés par la conjugaison de protéines de la famille Atg8 (protéines associées aux microtubules 1A/B chaîne légère 3A/B/C [LC3] et GABARAPs) à des membranes dérivées du recyclage des endosomes, du Golgi et/ou du réticulum endoplasmique (ER)1. LC3 reconnaît la cargaison autophagique dans le cytosol directement ou via des adaptateurs d’autophagie sélectifs tels que P62/SQSTM. De nouvelles membranes autophagiques peuvent ensuite être conjuguées à LC3, se dilater et fusionner pour former une double membrane complète renfermant la cargaison, appelée autophagosome. L’autophagosome mûrit et finit par fusionner avec un endosome ou un lysosome, après quoi la cargaison autophagique et les adaptateurs sont dégradés2.
Les études sur la formation, la maturation et la fusion des autophagosomes font souvent appel à des technologies de microscopie optique. La microscopie à fluorescence de la LC3 est généralement utilisée pour évaluer le nombre et la localisation cellulaire des autophagosomes dans différentes conditions. De plus, en couplant la LC3 à la GFP sensible au pH et à la RFP stable au pH dans une sonde dite tandem, le flux global d’autophagie peut être mesuré dans les cellules vivantes en fonction de la perte de fluorescence de la GFP3. Ces approches sont des outils précieux pour les chercheurs afin de comprendre le rôle et le mécanisme de l’autophagie dans différentes conditions. Un autre outil précieux est la microscopie électronique (EM), qui révèle l’ultrastructure des organites autophagiques à différents stades de l’autophagie 4,5,6,7,8. À ce jour, l’EM reste la méthode de choix pour identifier les étapes précises de la formation de l’autophagosome en discriminant différentes membranes autophagiques par morphologie : phagophore (double membrane non complètement fermée), autophagosome (double membrane fermée autour de la cargaison cytosolique) et autolysosome (perte [partielle] de la membrane autophagique interne). Cependant, la morphologie sans information moléculaire peut être sujette à des erreurs d’identification ou à des ambiguïtés. L’immuno-EM est la méthode la plus complète pour la caractérisation moléculaire et la classification morphologique simultanées des organites autophagiques. Par exemple, le marquage immunogold de la LC3 sur des cryocoupes décongelées permet la localisation ultrastructurale de la LC3 et l’identification précise des organites marqués LC39.
L’inconvénient de l’EM est le faible champ de vision qui accompagne le fort grossissement nécessaire pour observer l’ultrastructure fine des membranes autophagiques et, dans le cas de l’immuno-EM, pour localiser le marquage qui marque la protéine d’intérêt. En raison de leur rareté et de leur faible teneur en protéines, cela entrave généralement l’analyse EM quantitative des autophagosomes. Pour augmenter le nombre d’autophagosomes, les cellules sont souvent affamées et traitées avec de la bafilomycine A1 (BafA1), un inhibiteur de l’acidification et de la dégradation lysosomales. Sans traitement BafA1, la recherche d’autophagosomes par EM prend beaucoup de temps, en raison de la rareté de ces organites. La méthode présentée dans ce manuscrit aborde cette question par le marquage fluorescent et l’imagerie de la LC3 endogène sur des cryocoupes décongelées dans un microscope à fluorescence avant une préparation ultérieure à l’EM. Les images fluorescentes guident ensuite la recherche de structures marquées LC3 dans l’EM. Après la collecte, les images EM sont corrélées avec les images de fluorescence pour ajouter de l’information moléculaire – la présence de LC3 – à l’ultrastructure de la cellule. Cette méthode « CLEM sur coupe » augmente considérablement la capacité à trouver des structures marquées LC3, en particulier dans des conditions non traitées, pour une identification et une classification ultérieures par EM.
Cette méthode a été appliquée à des cellules HEPG210 dérivées d’hépatoblastomes affamés pour trouver des autophagosomes dans des conditions non modifiées (c’est-à-dire qu’aucun BafA1 n’a été utilisé). Relativement peu de puncta fluorescents (moins d’un par profil cellulaire dans une section de 90 nm) ont été trouvés, ce qui est en accord avec le renouvellement élevé de LC311. Cette rareté de LC3-puncta a mis en évidence la valeur de CLEM ; en sélectionnant des régions avec plusieurs puncta fluorescentes pour l’imagerie dans l’EM, des organites LC3-positifs ont été trouvés et caractérisés d’une manière beaucoup plus efficace que par immuno-EM conventionnelle. Cela a révélé que la majorité des organites LC3 positifs étaient des autophagosomes, tels que définis par leur morphologie, ce qui contraste avec les résultats obtenus dans les cellules traitées par BafA1, où les autolysosomes sont plus fréquents9. Ces données montrent qu’avec le CLEM en coupe, l’autophagie peut être étudiée au niveau ultrastructurel sans qu’il soit nécessaire d’inhiber le flux autophagique.
Protocole
1. Préparation des outils et des réactifs
REMARQUE : Pour connaître les réactifs, tampons et solutions requis, consultez le fichier supplémentaire 1 ou 12 pour plus d’informations. Pour plus de détails sur tous les matériaux, réactifs, équipements et logiciels utilisés dans ce protocole, reportez-vous au tableau des matériaux.
- Fixatifs
- Préparer un tampon phosphate (PB) de 0,2 M ou un tampon de 0,2 M DE TUYAUX, HEPES, EGTA, MgSO4 (PHEM), tel que décrit dans le fichier supplémentaire 1, à utiliser comme base pour les solutions fixatrices.
NOTA : Les fixateurs sont systématiquement tamponnés dans un tampon de 0,1 M PB ou PHEM pour tamponner contre l’acidification causée par la réaction de l’aldéhyde avec le matériel biologique. - Comme la qualité du paraformaldéhyde (PFA) est essentielle à une fixation fiable de l’ultrastructure des échantillons, utilisez du PFA de qualité EM. Pour suivre ce protocole, utilisez des solutions mères à 16 %, préparées à partir de granulés de PFA de haute qualité (voir Fiche supplémentaire 1).
ATTENTION : Le paraformaldéhyde est un produit chimique dangereux (mentions de danger H228, H301, H302, H311, H314, H315, H317, H318, H331, H332, H335, H341, H350). Lorsque vous travaillez avec de l’APF, portez un équipement de protection (gants, blouse de laboratoire et lunettes de protection) et travaillez dans une hotte chimique. Les déchets contenant du PFA doivent être collectés et éliminés conformément aux directives et réglementations des instituts. - Mélanger 10 mL de 0,2 M PB, 5 mL de PFA à 16 % (dans de l’eau déminéralisée [dH 2 O]) et 5 mL de dH2O pour préparer une solution fixatrice à 4 % de PFA.
- Facultatif : L’ajout de 0,02 % à 0,5 % de glutaraldéhyde (GA) à la solution de fixateur de l’étape 1.1.3 améliore la préservation de l’ultrastructure, mais réduit l’antigénicité de l’échantillon vis-à-vis de nombreux anticorps.
REMARQUE : Lorsque la fixation GA est souhaitée, utilisez un GA de qualité EM provenant d’un fournisseur approprié.
ATTENTION : Le glutaraldéhyde est un produit chimique dangereux (mentions de danger H301, H302, H314, H317, H330, H332, H334, H335, H400, H411). Travailler dans une hotte chimique et porter un équipement de protection (gants, blouse de laboratoire et lunettes de protection) lors de la manipulation de l’AG. Les déchets contenant de l’AG doivent être collectés et éliminés conformément aux directives et règlements des instituts.
- Préparer un tampon phosphate (PB) de 0,2 M ou un tampon de 0,2 M DE TUYAUX, HEPES, EGTA, MgSO4 (PHEM), tel que décrit dans le fichier supplémentaire 1, à utiliser comme base pour les solutions fixatrices.
- Outils et matériaux
- Grattez la surface des broches des porte-échantillons en aluminium et sonicez-les dans de l’éthanol 3 x 10 min pour éliminer les restes métalliques et assurer une adhérence optimale lorsque des blocs cellulaires enrobés de gélatine sont montés sur les broches.
- Utilisez une boîte de stockage adaptée pour stocker les goupilles porte-échantillons en aluminium avec leurs échantillons dans de l’azote liquide (LN2).
- Fabriquez un manipulateur en apposant un seul cheveu ou cil au bout d’une brochette en bois à l’aide de vernis à ongles.
- Faites une boucle de ramassage. Pliez un fil d’acier inoxydable de 0,3 mm d’épaisseur autour d’une barre ronde de 3 mm de diamètre et torsadez les extrémités ensemble, formant une boucle à une extrémité. Insérez les extrémités torsadées dans une pointe de pipette. Insérez une brochette en bois à l’autre extrémité et fixez-la avec de la colle ou de la résine.
REMARQUE : Une boucle de captation est également disponible dans le commerce (voir le tableau des matériaux). - Pour préparer les boucles de séchage de la grille, suivez les mêmes étapes que pour la réalisation des boucles de ramassage : formez un fil d’acier inoxydable dans une boucle de 4 mm et fixez-le sur un grand embout de pipette avec de la colle ou de la résine.
- Enduisez les grilles à l’aide d’une fine pellicule de support telle que formvar (protocole dans le fichier supplémentaire 1). Avant utilisation, enduisez les grilles d’une fine couche de carbone.
REMARQUE : Des grilles prêtes à l’emploi sont disponibles dans le commerce (voir le tableau des matériaux). Les grilles revêtues de Formvar peuvent être stockées indéfiniment à température ambiante (RT) ; les grilles revêtues de carbone peuvent être stockées pendant plusieurs mois chez RT. - Préparez des lames de verre propres et de grandes lamelles (24 mm x 24 mm est idéal avec des lames de verre de 25 mm de large), comme en 13.
2. Fixation et préparation de l’échantillon
- Fixation
- Utiliser le fixateur préparé à l’étape 1.1.3 (4 % de PFA dans 0,1 M PB). Pour les lignées cellulaires adhérentes, cultiver 1 à 5 × 10 cellules de 6 dans des boîtes de6 cm. Ajouter le fixateur au milieu de culture dans un rapport de 1 :1 et incuber l’échantillon pendant 5 min à RT. Ensuite, remplacez le mélange milieu-fixateur par un fixateur uniquement et incubez pendant 2 h à RT.
REMARQUE : Le nombre exact de cellules, la confluence et les conditions de culture peuvent varier selon le système modèle utilisé. - Conservez les échantillons pendant la nuit ou jusqu’à 3-4 semaines dans 0,5 % de PFA dans 0,1 M PB à 4 °C.
NOTE : GA peut être ajouté à la fixation (voir étape 1.1.4) et la longueur de fixation peut être modifiée pour trouver un équilibre optimal entre la préservation de la morphologie et l’antigénicité, qui diffère selon le spécimen et le marquage. Pour plus d’informations, reportez-vous àla section 14.
- Utiliser le fixateur préparé à l’étape 1.1.3 (4 % de PFA dans 0,1 M PB). Pour les lignées cellulaires adhérentes, cultiver 1 à 5 × 10 cellules de 6 dans des boîtes de6 cm. Ajouter le fixateur au milieu de culture dans un rapport de 1 :1 et incuber l’échantillon pendant 5 min à RT. Ensuite, remplacez le mélange milieu-fixateur par un fixateur uniquement et incubez pendant 2 h à RT.
- Exemple d’incorporation
- Lavez la vaisselle avec des cellules fixes 3x avec du PBS à RT. Ensuite, remplacez-le par du PBS contenant 0,15 % de glycine et incubez pendant 10 min à RT.
- Remplacer le PBS contenant 0,15 % de glycine par 1 % de gélatine dans du PBS préchauffé à 37 °C, puis gratter et transférer les cellules dans de la gélatine à 1 % dans un tube de microcentrifugation. Pelleter les cellules à 6 000 × g pendant 1 min à RT dans une microcentrifugeuse. Ensuite, retirez le 1 % de gélatine sans déranger la pastille et ajoutez 12 % de gélatine chauffée à 37 °C. Remettre en suspension la pastille cellulaire en pipetant doucement de haut en bas à l’aide d’embouts de pipette ou de pipettes Pasteur en verre préchauffées à 37 °C.
- Incuber à 37 °C pendant 10 min ; Ensuite, granulez les cellules à 6 000 × g pendant 1 min. Solidifier la gélatine sur de la glace pendant 30 min.
- Pour retirer les cellules enrobées de gélatine du tube, coupez l’extrémité du tube contenant la pastille du reste du tube avec une lame de rasoir. Ensuite, perpendiculairement à la première coupe, coupez l’extrémité du tube avec la pastille de cellule en deux.
- Incuber les deux moitiés d’extrémité du tube contenant la pastille cellulaire enrobée de gélatine dans du saccharose 2,3 M pendant 10 min à 4 °C. Cela provoque le rétrécissement léger des moitiés de pastilles cellulaires enrobées de gélatine et leur déloge du tube en plastique.
REMARQUE : Les pastilles cellulaires enrobées de gélatine doivent être conservées à 4 °C ou glacées autant que possible pour éviter que le saccharose de 2,3 M ne devienne trop visqueux et que la gélatine ne devienne trop molle. Lors de la manipulation des pastilles cellulaires enrobées de gélatine dans les étapes suivantes, travaillez avec un seul échantillon à la fois et gardez les autres sur de la glace, ou travaillez dans une pièce froide (~4 °C). Évitez la surchauffe des pastilles cellulaires enrobées de gélatine par la lumière du soleil, les lampes chaudes du microscope ou d’autres sources de chaleur. - Retirez les moitiés du tube avec la pastille de cellules enrobée de gélatine du saccharose 2,3 M. Ensuite, retirez les moitiés de pastilles cellulaires enrobées de gélatine des moitiés de tubes en plastique à l’aide d’une pince à épiler. Coupez manuellement le granulé en blocs d’une taille appropriée (~1 mm3) avec une lame de rasoir. Utilisez un microscope à dissection stéréoscopique pour grossir le sujet pendant le découpage.
- Infuser les blocs cellulaires enrobés de gélatine avec du saccharose de 2,3 M pendant 3 à 16 h, en les tournant d’un bout à l’autre dans un rotor à 4 °C.
- Placez un bloc cellulaire enrobé de gélatine sur une broche porte-échantillon en aluminium (voir étape 1.2.1). Laissez suffisamment de saccharose de 2,3 M sur les bords du bloc pour qu’il forme un mince « collier » entre le bloc et la goupille. Évitez qu’une trop grande quantité de saccharose de 2,3 M recouvre le dessus du bloc. Congeler et stocker dans LN2.
3. Sectionnement
- Rognage (voir aussi12)
- Prenez une épingle contenant un bloc de cellules enrobées de gélatine dans le stockage LN2 et placez-la dans un cryomicrotome réglé à -80 °C.
- Coupez l’avant du bloc pour aplatir sa surface et obtenir des sections de ~250 nm. Trempez l’anse de 3 mm dans une solution de ramassage (saccharose 1 :1 2,3 M et méthylcellulose 2 %), insérez l’anse dans la chambre cryogénique du microtome et attendez que de la glace commence à se former dans la gouttelette (généralement 5 à 7 s). Ensuite, ramassez immédiatement la section en appuyant rapidement mais doucement la gouttelette contre eux. Retirez la boucle de la chambre cryogénique, attendez que la gouttelette soit complètement décongelée et appuyez sur la gouttelette sur une lame de verre.
- Vérifier l’orientation des cellules par coloration au bleu de toluidine des sections.
- Placez une goutte de solution bleue de toluidine (voir Fichier supplémentaire 1) sur les sections d’une lame de verre et séchez sur une plaque chauffante à 80 °C jusqu’à ce que les bords de la goutte soient secs.
- Retirez la lame de verre de la plaque chauffante et rincez délicatement le bleu de toluidine avec dH2O, en le recueillant dans un conteneur à déchets approprié.
- Séchez la lame de verre et vérifiez l’orientation des cellules dans les sections à l’aide d’un simple microscope optique de paillasse.
- Coupez les côtés du bloc en sectionnant 50 à 100 μm sur le côté de la face du bloc d’échantillon avec le coin du couteau. Coupez les quatre côtés de la face du bloc d’échantillon en faisant pivoter le porte-échantillon de 90° après avoir coupé chaque côté pour créer un rectangle saillant de ~250 μm x 375 μm. Sélectionnez la zone saillante en fonction de l’orientation de la cellule déterminée à l’étape précédente.
- Sectionnement et ramassage
- Refroidir le cryomicrotome à -100 °C. Sectionnez un ruban à partir du rectangle saillant, les sections ayant une épaisseur de 70 à 90 nm et un éclat argenté-doré. Éloignez les sections du tranchant du couteau diamanté avec un cheveu sur un bâton (voir section 1.2.3) pour créer un long ruban (2-5 mm).
- Une fois qu’un ruban approprié s’est formé, arrêtez la section pour ramasser le ruban. Trempez la boucle de ramassage de 3 mm dans du saccharose 2,3 M et de la méthylcellulose 2 % mélangée 1 :1, insérez la boucle dans la chambre cryogénique du microtome et attendez que la gouttelette commence à geler (généralement 5 à 7 s). Ensuite, ramassez immédiatement les sections en appuyant rapidement mais doucement la gouttelette contre elles. Retirez la boucle de la chambre cryogénique, attendez que la gouttelette soit complètement décongelée et appuyez sur la gouttelette sur une grille préparée (étape 1.2.6).
REMARQUE : Les grilles avec sections peuvent être stockées à 4 °C pendant plusieurs mois.
4. Marquage et microscopie optique
- Étiquetage
- Placez les grilles avec des sections (Figure 1A) côté section vers le bas sur ~1 mL de PBS dans une petite boîte ou une assiette à plusieurs puits. Incuber à 37 °C pendant 30 min.
REMARQUE : Cette étape enlève la gélatine qui se trouve entre les cellules ; La gélatine n’est pas nécessaire après la coupe et interfère avec le reste du protocole. - Traiter les grilles côté section vers le bas sur des gouttelettes de ~75 μL sur le parafilm (voir la figure 1B). Commencez par des lavages PBS + 0,15 % de glycine (3 x 2 min) à RT. Ensuite, incuber les grilles avec 0,1 % d’albumine sérique bovine (BSA)-c + 0,5 % de gélatine de peau de poisson (FSG) dans du PBS pendant 10 min à RT comme étape de blocage. Diluer les anticorps primaires dans 0,1 % BSA-c + 0,5 % FSG dans le PBS et incuber les grilles sur des gouttelettes de ~10 μL de cette solution pendant 1 h à RT (Figure 1C).
- Lavez les grilles dans 0,1 % de BSA dans PBS 5x à RT. Ensuite, diluer les anticorps secondaires et le 4',6-diamidino-2-phénylindole (DAPI ; 10 μg/mL) dans 0,1 % BSA-c + 0,5 % FSG dans le PBS et incuber les grilles sur des gouttelettes de ~10 μL de cette solution pendant 30+ min à RT (Figure 1C). Lavez les grilles dans PBS 5x à RT.
REMARQUE : En option, un anticorps secondaire peut être marqué avec des particules d’or colloïdal de 5, 10, 15 ou 20 nm conjuguées à la protéine A (PAG) pour la localisation de la protéine d’intérêt dans l’EM. Si vous le souhaitez, incubez les grilles avec PAG pendant 20 min à RT après l’étape 4.1.3. Ensuite, lavez-vous 5 fois avec du PBS à RT. Évitez l’utilisation simultanée de plusieurs anticorps primaires et prenez note de la réactivité du PAG aux IgG de différentes espèces pour éviter les réactions croisées indésirables. Pour plus d’informations, reportez-vous àla section 12.
- Placez les grilles avec des sections (Figure 1A) côté section vers le bas sur ~1 mL de PBS dans une petite boîte ou une assiette à plusieurs puits. Incuber à 37 °C pendant 30 min.
- Montage d’échantillons pour la microscopie optique
- Immergez les grilles dans du glycérol à 50 % en dH 2 O2x 5 min à RT. Placez les grilles en sandwich entre une lame de verre et une lamelle de recouvrement dans du glycérol à 50 %, une grille par lamelle, avec des sections faisant face à la lamelle (Figure 1D).
REMARQUE : La qualité de l’étiquetage peut se détériorer lorsque les grilles sont maintenues montées dans du glycérol à 50% pendant plus de 30 minutes. Il est donc recommandé de monter et d’imager deux ou trois grilles à la fois et de laisser les autres sur la solution d’étiquetage secondaire.
- Immergez les grilles dans du glycérol à 50 % en dH 2 O2x 5 min à RT. Placez les grilles en sandwich entre une lame de verre et une lamelle de recouvrement dans du glycérol à 50 %, une grille par lamelle, avec des sections faisant face à la lamelle (Figure 1D).
- Microscopie optique
- Amenez une lame de verre avec des grilles prises en sandwich vers un microscope à grand champ avec une platine automatisée. Sélectionnez un objectif à huile à fort grossissement (63x ou 100x). Créez un jeu de mosaïques d’images du ruban de sections (Figure 1E) (ou d’une partie de celui-ci) (Figure 1E).
REMARQUE : Certains anticorps secondaires peuvent former des agrégats fluorescents sur les grilles, en particulier autour des plis ou des déchirures dans les sections. De plus, certains types de cellules et de tissus contiennent des structures autofluorescentes. Si de tels problèmes sont attendus, il est conseillé d’inclure une grille de contrôle négative non incubée avec des anticorps primaires.
- Amenez une lame de verre avec des grilles prises en sandwich vers un microscope à grand champ avec une platine automatisée. Sélectionnez un objectif à huile à fort grossissement (63x ou 100x). Créez un jeu de mosaïques d’images du ruban de sections (Figure 1E) (ou d’une partie de celui-ci) (Figure 1E).
- Démontage et contraste EM
- Ajouter 10 μL de dH2O sur le côté du sandwich lamelle en verre et attendre que l’action capillaire remplisse l’interface sandwich lamelle verre-lamelle. Retirez délicatement la lamelle sans mélanger l’huile d’immersion dans le glycérol. Récupérez les grilles à l’aide d’une pince à épiler et plongez-les dans dH2O 3x à RT pour éliminer le glycérol à 50 %.
REMARQUE : L’huile peut interférer avec la coloration de l’uranyle et détériorer le contraste EM. - Séchez soigneusement le dos de la grille avec du papier de soie non pelucheux.
REMARQUE : Si l’échantillon a également été marqué avec des particules d’or colloïdal, effectuez les opérations suivantes : placez la grille avec des sections côté section vers le bas sur des gouttelettes de PBS et lavez 2x à RT. Postfix à 1% GA pendant 5 min à RT (voir la mise en garde au point 1.1.4). Laver en PBS 2x à RT. - Placez les grilles côté section vers le bas sur des gouttelettes de dH2O et lavez 8x à RT.
- Pour colorer les coupes de contraste en EM, incuber avec de l’acétate d’uranyle (UA), pH 7, pendant 5 min à RT (Figure 1F).
- Avant de placer les grilles, refroidir l’UA :méthylcellulose, pH 4, en plaçant des gouttelettes sur du parafilm sur une plaque métallique sur de la glace. Ensuite, lavez les grilles avec de l’UA :méthylcellulose, pH 4, 2x et incubez avec de l’UA :méthylcellulose, pH 4, pendant 10 minutes (Figure 1F).
ATTENTION : L’acétate d’uranyle est un produit chimique dangereux (mentions de danger H300, H330, H373, H411). Dans les étapes qui nécessitent une UA, travaillez dans une hotte chimique et portez un équipement de protection (blouse de laboratoire, gants et lunettes de protection). Collecter et éliminer les déchets contenant de l’AU conformément aux directives et règlements des instituts. - Bouclez les grilles en insérant une boucle de séchage de la grille dans la gouttelette UA :méthylcellulose sous la grille et en la soulevant doucement jusqu’à ce que la grille soit retirée de la gouttelette12. Épongez l’excès d’UA :méthylcellulose en touchant la boucle à un angle de ~60° (sections vers le bas) sur du papier filtre non pelucheux (voir le tableau des matériaux) et en le faisant glisser lentement le long du papier jusqu’à ce qu’il n’y ait plus d’UA :méthylcellulose absorbée. Ensuite, placez la boucle avec la grille dans une grille appropriée et laissez sécher pendant >10 min à RT (Figure 1G).
- Ajouter 10 μL de dH2O sur le côté du sandwich lamelle en verre et attendre que l’action capillaire remplisse l’interface sandwich lamelle verre-lamelle. Retirez délicatement la lamelle sans mélanger l’huile d’immersion dans le glycérol. Récupérez les grilles à l’aide d’une pince à épiler et plongez-les dans dH2O 3x à RT pour éliminer le glycérol à 50 %.
5. EM (en anglais seulement)
- Utilisez la vue d’ensemble obtenue par microscopie optique pour localiser une région d’intérêt (ROI) pour l’imagerie au microscope électronique à transmission (MET ; Graphique 1H). Annotez le retour sur investissement sur le jeu de données de microscopie optique. Une fois qu’une région est sélectionnée, obtenez un ensemble de tuiles d’image à un grossissement de 20 000 x à 50 000x dans le TEM. Reconstruire le jeu de tuiles d’image dans le logiciel de post-traitement15,16.
6. Corrélation et analyse
- Chargez le jeu de données de microscopie optique et d’EM dans un logiciel de traitement d’image approprié, tel que ImageJ/Fiji17, le plug-in ec-CLEM d’Icy18 ou Photoshop. Recadrez et faites pivoter le jeu de données de microscopie optique pour qu’il corresponde au jeu de tuiles EM.
- Effectuer la corrélation basée sur le signal DAPI en fluorescence et les contours nucléaires en EM (Figure 1I). Déplacez les images pour les superposer avec précision et effectuez la corrélation manuelle avec précision. Pour rendre l’approche plus précise, appliquez une corrélation basée sur des points de repère, par exemple, via le plug-in ec-CLEM dans Icy ou le plug-in BigWarp dans ImageJ, pour corréler les images via la sélection manuelle des points correspondants. Un protocole détaillé, étape par étape, pour la corrélation avec ec-CLEM est disponible19.
REMARQUE : Cette approche fonctionne également bien avec l’utilisation de sondes de repère bimodales20,21.
- Effectuer la corrélation basée sur le signal DAPI en fluorescence et les contours nucléaires en EM (Figure 1I). Déplacez les images pour les superposer avec précision et effectuez la corrélation manuelle avec précision. Pour rendre l’approche plus précise, appliquez une corrélation basée sur des points de repère, par exemple, via le plug-in ec-CLEM dans Icy ou le plug-in BigWarp dans ImageJ, pour corréler les images via la sélection manuelle des points correspondants. Un protocole détaillé, étape par étape, pour la corrélation avec ec-CLEM est disponible19.
- Analysez les images corrélées en sélectionnant les ROI en fonction du signal fluorescent dans un programme approprié (par exemple, ImageJ). Pour l’analyse quantitative, créez une collection de ROI pour tous les organites marqués. Ensuite, inspectez l’ultrastructure correspondante des ROI individuels et classez-les en fonction d’éléments morphologiques.
Résultats
Un protocole immuno-EM optimisé pour le marquage immuno-or de la LC3 sur des cryocoupes ultraminces a été récemment publié par De Maziere et al.9. Cette étude a porté sur des conditions de famine sans BafA1, dans lesquelles la CL3 était présente mais relativement rare et difficile à trouver par EM. Une méthode CLEM sur coupe a été introduite dans une étude distincte, qui utilise la sensibilité du marquage par fluorescence pour visualiser des protéines endogènes relativement rares et faiblement exprimées et les corréler à l’ultrastructure EM14. Ici, ces deux approches sont combinées par l’utilisation du protocole de marquage LC3 optimisé dans le cadre d’une approche CLEM.
Les cellules HEPG2, des cellules dérivées du foie avec des niveaux relativement élevés d’autophagie basale22, ont été affamées dans un milieu minimal (solution saline équilibrée d’Earle [EBSS]) pendant 2 h avant la fixation dans 4% de PFA. S’en est suivie la préparation de l’échantillon par la méthode Tokuyasu de cryosection ultramince (sections 1 à 3 ; voir Fente et Geuze12), qui est hautement compatible avec la méthode CLEM 14,23 sur section. Les cryocoupes décongelées ont été marquées par fluorescence (section 4 du protocole et figure 1) à l’aide de l’anticorps primaire anti-LC39 de souris. De plus, l’anticorps anti-LAMP1 du lapin a été utilisé pour indiquer les endo-lysosomes, suivi des anticorps secondaires anti-souris AlexaFluor488 et anti-lapin AlexaFluor568. Des grilles ont été prises en sandwich entre une lamelle de recouvrement et une lame de verre et photographiées à RT sur un microscope à grand champ (objectif à huile 100x 1,47 NA, caméra sCMOS).
L’un des avantages du marquage par fluorescence des lames minces par rapport à l’IF conventionnel des cellules entières est l’augmentation de la résolution en Z, puisque l’épaisseur physique de la section est de 60 à 90 nm. Grâce à cette résolution Z améliorée, le marquage par fluorescence de LC3 et de LAMP1 sur des lames minces révèle très peu de colocalisation (Figure 2A). Dans les cellules traitées avec des inhibiteurs lysosomaux, tels que BafA1, une colocalisation élevée se produit, car la LC3 enfermée dans le lysosome reste non dégradée9. Dans les cellules non traitées, la LC3 est rapidement dégradée au contact de lysosomes enzymatiquement actifs et positifs pour LAMP1, et la colocalisation est donc rare dans ces conditions. En général, moins d’un punctum de LC3 par profil cellulaire a été observé. Cela indique que même dans des conditions de famine, le renouvellement des autophagosomes est rapide, ce qui maintient le nombre d’autophagosomes à un faible niveau. Il souligne également l’importance d’utiliser CLEM pour trouver les structures rares marquées LC3, en utilisant le large champ de vision fourni par la microscopie optique. De plus, la sensibilité plus élevée du marquage par fluorescence par rapport au marquage à l’or permet d’identifier plus d’organites LC3-positifs que dans l’immuno-EM conventionnelle, ce qui facilite encore leur caractérisation.
Après l’acquisition d’un ensemble complet de tuiles du ruban de sections, les grilles ont été extraites du microscope et post-colorées pour l’EM à l’aide de l’UA et de la méthode de bouclage (étapes de protocole 4.4-4.6 ; Graphique 1F, G). Cette méthode de « bouclage » garantit qu’une fine couche d’UA :méthylcellulose reste sur la grille, ce qui crée le contraste souhaité dans l’EM. L’épaisseur de la couche dépend de la vitesse et de l’angle avec lesquels l’UA :méthylcellulose est étalée sur le papier filtre. Si vous faites glisser la boucle trop rapidement, vous risquez de laisser trop d’UA :méthylcellulose sur la grille et d’assombrir l’apparence des sections dans EM. Si vous traînez trop lentement, vous risquez d’évacuer trop d’UA :méthylcellulose, ce qui entraîne trop peu de coloration et une mauvaise morphologie, et risque de faire tomber la grille hors de la boucle. La coloration de la « nappe d’huile » (figure 1G) sur les grilles sèches indique une épaisseur de couche UA :méthylcellulose appropriée.
Après bouclage et séchage, les grilles ont été imagées dans un MET à des retours d’intérêt sélectionnés par fluorescence. Les ensembles de données IF et EM ont été corrélés en superposant le signal DAPI aux contours des noyaux visibles dans l’EM, générant une image intégrée contenant des informations sur les deux modalités.
Il peut être difficile de trouver la même zone dans EM que celle sélectionnée dans IF. Il est donc recommandé de garder une vue d’ensemble de l’ensemble de tuiles IF à portée de main lors de la recherche dans l’EM. Les utilisateurs doivent rechercher des caractéristiques reconnaissables dans les deux modalités, telles que des plis ou des déchirures dans les sections, des barres de grille ou la disposition des noyaux. Il est également important de garder à l’esprit que l’échantillon peut apparaître tourné et reflété dans EM. Des « grilles de recherche » avec des caractéristiques spécifiques pour identifier les zones peuvent être utilisées pour faciliter la corrélation (voir le tableau des matériaux).
La corrélation des organites LC3-positifs à l’ultrastructure EM a révélé que les différents puncta représentaient des stades distincts de l’autophagie (Figure 2B). Bien que la préservation de l’ultrastructure autophagosomale soit difficile dans les cryosections, des organites à contenu cytoplasmique et à double membrane ont été fréquemment observés (Figure 2C, flèches dans les organites 1-5 ; Figure supplémentaire S1), qui définissent les caractéristiques morphologiques des autophagosomes. Il est intéressant de noter que des taches fluorescentes assez faibles ont été identifiées par EM comme des autolysosomes LC3 positifs (Figure 2C, organite 6 ; le contenu autophagique est marqué *), caractérisés par un contenu dense et des vésicules intraluminales. Cela a montré que de très petites quantités de LC3 sont visibles dans les FI des cryosections ultraminces, et a indiqué que malgré le milieu de dégradation, une partie de la LC3 est détectable dans les autolysosomes à l’état d’équilibre. Cependant, la majorité des puncta LC3-positives représentaient des autophagosomes, alors que les autolysosomes étaient très rares. Cela contraste avec les cellules traitées au BafA1, qui accumulent principalement des autolysosomes et non des autophagosomes9.
En résumé, ce protocole décrit une méthode CLEM sur coupe pour relier l’information moléculaire obtenue par microscopie à fluorescence à l’ultrastructure de l’EM. Cette méthode augmente la sensibilité de l’immuno-EM, car seuls les fluorophores sont utilisés pour le marquage et ceux-ci produisent généralement plus de signal que les sondes EM. La méthode est particulièrement adaptée à l’utilisation de cryosections ultraminces, dans lesquelles des niveaux élevés de fluorescence spécifique sur une coloration de fond négligeable peuvent être obtenus. En utilisant la fluorescence pour dépister des structures ou des événements rares et en corrélant les ROI sélectionnés à la ME, le temps d’opération de la ME et les coûts associés peuvent être considérablement réduits. La sensibilité et la faisabilité de la méthode sont démontrées par la visualisation de la LC3 dans des cellules affamées non traitées, montrant que la LC3 s’associe principalement aux autophagosomes dans ces conditions, avec de très faibles niveaux visibles dans les autolysosomes.
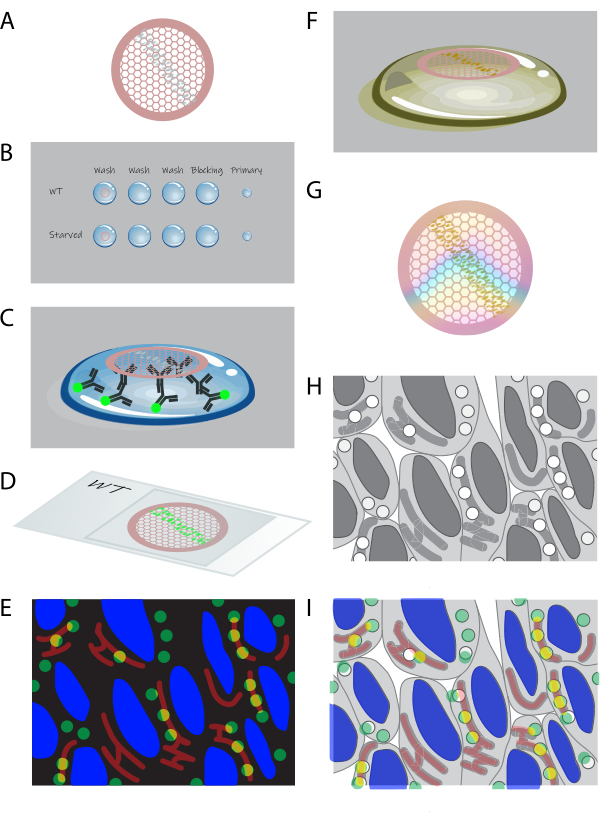
Figure 1 : Vue d’ensemble schématique du CLEM sur la section. (A) Les cryocoupes de cellules enrobées de gélatine sont recueillies sur une grille de cuivre recouverte de formvar. (B) Les grilles sont traitées en coupe sur des gouttelettes des solutions appropriées. (C) Les grilles sont marquées avec des anticorps primaires et secondaires fluorescents. (D) Des grilles sont prises en sandwich entre une lamelle et une lame de verre en glycérol à 50 %. (E) Les images de fluorescence sont recueillies dans un microscope à grand champ. (F) Les grilles sont extraites de la lame de verre et traitées ultérieurement par coloration à l’uranyle pour les EM. (G) Après séchage, les grilles peuvent être imagées par MET. (H) L’ensemble de tuiles d’image TEM à fort grossissement est acquis à partir d’une zone sélectionnée à partir de données de fluorescence. (I) Les images de la microscopie à fluorescence et de l’EM sont corrélées et superposées. Veuillez cliquer ici pour voir une version agrandie de cette figure.
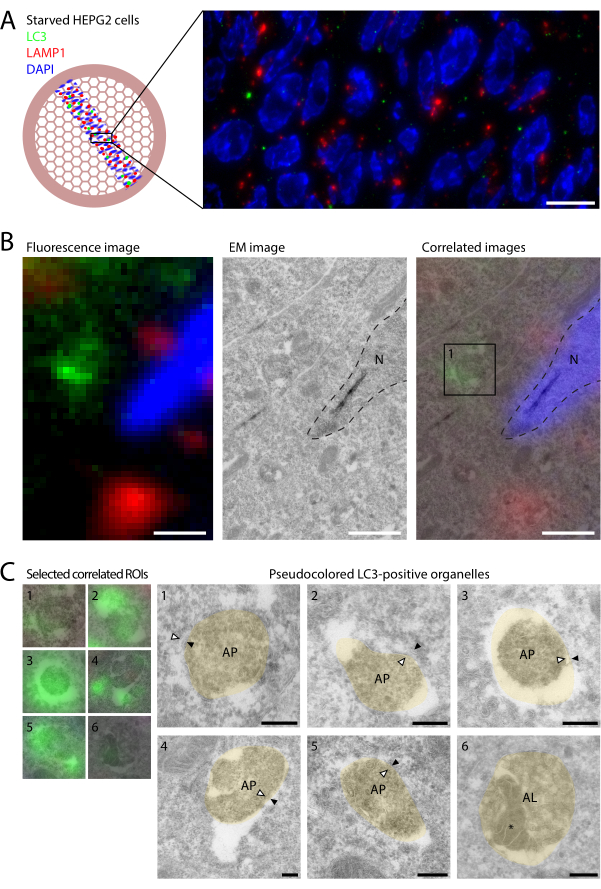
Figure 2 : CLEM de LC3 et LAMP1 dans des cellules HEPG2 affamées. Les cellules HEPG2 ont été affamées pendant 2 h dans l’EBSS avant la fixation avec 4 % de PFA pendant 2 h. (A) L’imagerie IF de LC3 (vert) et LAMP1 (rouge) sur les coupes révèle relativement peu de ponctuations de LC3 et peu de colocalisation avec LAMP1. (B) Relier l’information moléculaire de l’IF (panneau de gauche) à l’information ultrastructurale obtenue en EM (panneau du milieu) en superposant les deux modalités d’imagerie basées sur le DAPI et les contours nucléaires (lignes pointillées, panneau de droite). L’ultrastructure des différents compartiments marqués LC3, illustrée par l’encadré 1 (panneau de droite), est illustrée en C. (C) Ultrastructure des compartiments LC3 positifs. Les images CLEM sont montrées à gauche et les images EM pseudocolorées (beiges) à droite (les images EM non colorées sont montrées dans la figure supplémentaire S1). Les membranes autophagosomiques internes et externes sont indiquées par des pointes de flèches blanches et noires, respectivement. Le contenu autophagique à l’intérieur de l’autolysosome dans l’exemple 6 est indiqué par *. Barres d’échelle = 10 μm (A), 1 μm (B), 200 nm (C). Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire S1 : Images EM non colorées d’organites LC3 positifs. (A-F) Images EM non colorées des exemples pseudo-colorés 1 à 6 illustrés à la figure 2C. Les organites ont été sélectionnés par fluorescence LC3, comme décrit pour la figure 2. Les membranes autophagosomiques internes et externes sont indiquées par des pointes de flèches blanches et noires, respectivement. Le contenu autophagique à l’intérieur de l’autolysosome dans l’exemple 6 est indiqué par *. Barres d’échelle = 200 nm. Abréviations : AL = autolysosome ; AP = autophagosome ; M = mitochondrie. Veuillez cliquer ici pour télécharger ce fichier.
Fichier supplémentaire 1 : Tampons et solutions utilisés dans cette étude. Ce fichier supplémentaire contient les recettes et les protocoles nécessaires à la fabrication des tampons et des solutions utilisés dans cette étude. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
La méthode présentée ici tire parti des progrès récents dans le domaine de la CLEM sur section basée sur la cryosection, c’est-à-dire la haute sensibilité du marquage IF et la corrélation précise (erreur de <100 nm) entre FM et EM14,24. Il en résulte une méthode sensible au marquage fluorescent de protéines endogènes rares et capable de les superposer avec une grande précision à l’ultrastructure EM. Ainsi, cette méthode permet d’éviter la (sur)expression de protéines marquées exogènes et l’utilisation de marquages EM moins sensibles. La faisabilité de la méthode est démontrée par des exemples de CLEM sur la LC3 endogène dans des cellules affamées, sans l’utilisation d’inhibiteurs lysosomaux.
Les cryocoupes décongelées obtenues avec la méthode Tokuyasu sont des échantillons idéaux pour l’immuno-EM, car contrairement aux coupes de résine, elles sont perméables aux anticorps. Combiné à des procédures de fixation et de contraste douces, cela donne généralement une superbe efficacité de marquage par rapport à d’autres méthodes sans compromettre l’ultrastructure détaillée, et visualise parfaitement les membranes cellulaires 12,25,26. De plus, les cryosections sont hautement compatibles avec la microscopie à fluorescence, ce qui en fait des substrats précieux pour CLEM. Le marquage immunogold classique et le CLEM sur les cryocoupes ont fourni des informations déterminantes dans la compréhension de l’organisation subcellulaire 14,27,28,29,30.
À l’heure actuelle, les applications de CLEM sur les cryocoupes décongelées sont de plus en plus répandues, à la suite de développements et d’optimisations continus 14,20,24,31,32,33,34 qui ont amélioré la qualité, l’applicabilité et la précision de l’approche. Aujourd’hui, grâce à une corrélation précise de grands ensembles de tuiles d’images IF et EM, la technique facilite le criblage de l’ultrastructure des composants cellulaires endogènes marqués par fluorescence 14,32,33. Il s’agit d’un avantage par rapport à l’immuno-EM classique, où la recherche de structures marquées à l’or nécessite généralement un fort grossissement et est donc plus laborieuse et prend beaucoup de temps. C’est pour cette raison que la localisation de la LC3 dans l’ultrastructure bénéficie grandement de CLEM. Les organites LC3-positifs sont fréquents lorsque la clairance autophagique est bloquée (c’est-à-dire lorsque les cellules sont traitées avec du BafA1 ou des agents augmentant le pH), tandis que les organites autophagiques sont rapidement éliminés dans les cellules non altérées ou affamées, ce qui entraîne des niveaux d’équilibre très faibles. Dans de telles conditions, il peut être difficile de trouver des organites marqués par LC3 à l’aide de l’immuno-EM classique, et le CLEM offre un net avantage.
Auparavant, le CLEM sur des coupes de résine a été appliqué dans des études utilisant l’expression ectopique de LC3-GFP ou une sonde tandem LC3-GFP-RFP 35,36,37,38,39. Dans ces études, l’imagerie par fluorescence a été réalisée avant l’enrobage ou directement dans les sections de résine acrylique40, et les échantillons ont ensuite été examinés par EM. L’enrobage de résine présente plusieurs avantages ; L’échostructure autophagosomale est généralement bien conservée, surtout si le matériau est congelé à haute pression40. De plus, le contraste des matériaux enrobés de résine colorés aux métaux lourds est généralement plus prononcé que celui des cryocoupes colorées à l’uranyle. Les coupes enrobées de résine sont compatibles avec les méthodes EM volumétriques, telles que la tomographie sur puce, le FIB-MEB ou le MEB en série, alors que les cryosections ne le sont pas. Dans les approches qui effectuent l’imagerie avant l’intégration, l’imagerie de cellules vivantes est une option41 qui n’est pas disponible dans CLEM sur les cryosections. Le principal avantage du CLEM sur les cryosections par rapport à ces alternatives est le signal IF élevé, permettant l’immunolocalisation de protéines rares sans avoir besoin de perméabilisation ou de surexpression membranaire. Cela permet d’éviter les potentielles extractions membranaires, les artefacts de surexpression42 et les modifications génétiques du sujet, ce qui, combiné à la possibilité de corréler de grandes surfaces dans l’IF et l’EM, en fait un excellent outil pour étudier la LC3 et l’autophagie.
Ici, l’application de CLEM sur section à des cellules HEPG2 affamées a révélé que LC3 était principalement localisée à des structures identifiées comme des autophagosomes. De plus, quelques taches faiblement fluorescentes ont été trouvées dans les autolysosomes. Ceci est en contraste direct avec les cellules traitées avec BafA19 et reflète la dégradation rapide des protéines autophagosomiques une fois que l’autophagosome fusionne avec les lysosomes. Dans l’ensemble, les données ont démontré que le CLEM des cryocoupes décongelées peut fournir des informations sur l’autophagie médiée par la LC3 dans des conditions natives. Les données mettent également en évidence la sensibilité de la technologie, puisque la LC3 a été détectée même dans des autolysosomes qui ne contiennent que de faibles niveaux d’épitopes de LC3 intacts. L’application ultérieure de cette technique par imagerie de la LC3 dans différents modèles et conditions améliorera notre compréhension de l’autophagie et d’autres processus biologiques médiés par la LC3, tels que la phagocytose associée à la LC3 ou la conjugaison de l’ATG8 à des membranes individuelles.
Au-delà de l’autophagie, le CLEM sur section peut être appliqué à d’autres événements ou structures rares, tels que la division cellulaire, l’infection, les types de cellules rares dans les tissus, les kinétochores, les cils primaires ou les organites spécifiques au type cellulaire. Un dépistage efficace du sujet d’intérêt par l’IF peut grandement faciliter l’étude ultrastructurale de ces raretés. De plus, il a été démontré14 que la technique peut être utilisée pour localiser des protéines d’une manière plus sensible que l’immuno-EM classique. L’ajustement de la longueur de fixation peut encore étendre cette sensibilité, permettant la localisation ultrastructurale de protéines très peu abondantes ou peu antigéniques. Enfin, la méthode CLEM en coupe facilite la sélection rapide d’un nombre quantitatif d’organites, facilitant une analyse plus robuste de la distribution ultrastructurale d’une protéine donnée.
Le CLEM sur les cryosections nécessite l’équipement et l’expertise nécessaires à la cryosection. Dans les groupes ayant accès à ces outils (par exemple, les cryomicrotomes), la mise en œuvre du CLEM sur section est simple et ne nécessite que la disponibilité d’un microscope à grand champ automatisé, une configuration à laquelle la plupart des laboratoires ont accès. De plus, la méthode est disponible dans les installations EM du monde entier. Étant donné que le CLEM en coupe combine l’application des méthodes IF et EM établies, la méthode est facilement adaptable et peut être combinée, par exemple, avec la tomographie 20,33,43, le volume de coupe en série EM d’un nombre limité de sections 44, ou la microscopie à super-résolution 45. Cette polyvalence de la méthode permet des applications à un large éventail de questions biologiques.
Déclarations de divulgation
Les auteurs déclarent qu’il n’y a pas de conflits d’intérêts.
Remerciements
Nous remercions nos collègues du Centre de médecine moléculaire du Centre médical universitaire d’Utrecht pour leurs discussions et leurs commentaires fructueux. Nous remercions les collègues passés et présents du laboratoire Klumperman pour l’amélioration continue de nos technologies de microscopie. L’infrastructure EM utilisée pour ce travail fait partie du programme de recherche National Roadmap for Large-Scale Research Infrastructure (NEMI) financé par le Conseil néerlandais de la recherche (NWO), numéro de projet 184.034.014 à JK.
matériels
| Name | Company | Catalog Number | Comments |
| Chemicals and reagents | |||
| Antibody donkey anti-mouse Alexa Fluor 488 | Life Technologies | #A21202 | use 1:250 |
| Antibody donkey anti-rabbit Alexa Fluor 568 | Life Technologies | A#10042 | use 1:250 |
| Antibody mouse anti-LC3 | Cosmo Bio | CTB-LC3-2-IC | use 1:100 |
| Antibody rabbit anti-LAMP1 | Cell Signaling | 9091 | use 1:250 |
| Bovine serum Albumin, fraction V | Sigma-Aldrich | A-9647 | |
| BSA-c | Aurion | 900.099 | |
| BSA-conjugated gold | Cell Microscopy Core, UMC Utrecht | BSAG 5 nm | |
| Water-free Chloroform | Merck | 1.02447.0500 | |
| DAPI | Invitrogen | 10184322 | Use at end concentration of 10 µg/ml |
| EGTA | Sigma-Aldrich | E4378 | |
| Fish-skin Gelatin | Sigma-Aldrich | G7765 | |
| Food-grade gelatin | Merck | G1890 | |
| Formvar, Vinylec E | SPI | 02492-RA | |
| Gluteraldehyde | Serva | 23115.01 | See CAUTION note |
| Glycerol | Boom | MBAK 7044.1000 | |
| Glycine | Merck | 1042010250 | |
| HEPES | Sigma-Aldrich | H3375 | |
| Methylcellulose, 25 centipoises | Sigma-Aldrich | M-6385 | |
| MgSO4 | Riedel-de Haen | 12142 | |
| Na2HPO4 (PB component A) | Merck | 106580-0500 | |
| NaBH4 | Merck | 806373 | |
| NaH2PO4 (PB component B) | Merck | 106346 | |
| NH4OH | Sigma-Aldrich | 221228-0025 | |
| Oxalic acid | Merck | 100495 | |
| Paraformaldehyde prills | Sigma-Aldrich | 441244 | See CAUTION note |
| PIPES | Merck | 110220 | |
| Protein-A conjugated gold | Cell Microscopy Core, UMC Utrecht | PAG 5, 10, 15 or 20 nm | |
| Sucrose D(+) | VWR | 27483294 | |
| Uranyl acetate | SPI | 020624-AB | See CAUTION note |
| Tools and consumables | |||
| Pick-up loop | Electron Microscopy Sciences | 70944 | |
| Filter paper, qualitative, medium-fast | LLG | 6.242 668 | |
| Finder grids | Ted Pella | G100F1 | |
| Grids | Cell Microscopy Core, UMC Utrecht | CU 100 mesh | |
| Microscopes | |||
| Leica Thunder widefield microscope | Leica | Components: 100x, 1.47 NA TIRF objective; Photometrics prime 95B sCMOS camera; LAS X software; | |
| Leica UC7 ultracryomicrotome | Leica | ||
| Tecnai T12 | FEI | Components: Veleta VEL-FEI-TEC12-TEM camera; SerialEM software | |
| Software | |||
| ec-CLEM in icy | open source | Paul-Gilloteaux et al., 2017 | |
| Fiji | open source | Schindelin et al., 2012 | |
| IMOD | open source | Mastronarde et al., 2017 | |
| Photoshop | Adobe | ||
| SerialEM | open source | Mastronarde et al., 2018 |
Références
- Hu, Y., Reggiori, F. Molecular regulation of autophagosome formation. Biochemical Society Transactions. 50 (1), 55-69 (2022).
- Reggiori, F., Ungermann, C. Autophagosome maturation and fusion. Journal of Molecular Biology. 429 (4), 486-496 (2017).
- Kimura, S., Noda, T., Yoshimori, T. Dissection of the autophagosome maturation process by a novel reporter protein, tandem fluorescent-tagged LC3. Autophagy. 3 (5), 452-460 (2007).
- Baba, M., Takeshige, K., Baba, N., Ohsumi, Y. Ultrastructural analysis of the autophagic process in yeast: detection of autophagosomes and their characterization. The Journal of Cell Biology. 124 (6), 903-913 (1994).
- Takeshige, K., Baba, M., Tsuboi, S., Noda, T., Ohsumi, Y. Autophagy in yeast demonstrated with proteinase-deficient mutants and conditions for its induction. The Journal of Cell Biology. 119 (2), 301-311 (1992).
- De Duve, C., Wattiaux, R. Functions of lysosomes. Annual Review of Physiology. 28, 435-492 (1966).
- Arstila, A. U., Trump, B. F. Studies on cellular autophagocytosis. The formation of autophagic vacuoles in the liver after glucagon administration. The American Journal of Pathology. 53 (5), 687-733 (1968).
- Eskelinen, E. L., Reggiori, F., Baba, M., Kovács, A. L., Seglen, P. O. Seeing is believing: The impact of electron microscopy on autophagy research. Autophagy. 7 (9), 935-956 (2011).
- De Mazière, A., et al. An optimized protocol for immuno-electron microscopy of endogenous LC3. Autophagy. 18 (12), 3004-3022 (2022).
- López-Terrada, D., Cheung, S. W., Finegold, M. J., Knowles, B. B. Hep G2 is a hepatoblastoma-derived cell line. Human Pathology. 40 (10), 1512-1515 (2009).
- Tanida, I., Minematsu-Ikeguchi, N., Ueno, T., Kominami, E. Lysosomal turnover, but not a cellular level, of endogenous LC3 is a marker for autophagy. Autophagy. 1 (2), 84-91 (2005).
- Slot, J. W., Geuze, H. J. Cryosectioning and immunolabeling. Nature Protocols. 2 (10), 2480-2491 (2007).
- Waterman-Storer, C. M. Microtubule/organelle motility assays. Current Protocols in Cell Biology. 13 (1), (2001).
- vander Beek, J., de Heus, C., Liv, N., Klumperman, J. Quantitative correlative microscopy reveals the ultrastructural distribution of endogenous endosomal proteins. The Journal of Cell Biology. 221 (1), e202106044 (2022).
- Mastronarde, D. N. Advanced data acquisition from electron microscopes with SerialEM. Microscopy and Microanalysis. 24, 864-865 (2018).
- Mastronarde, D. N., Held, S. R. Automated tilt series alignment and tomographic reconstruction in IMOD. Journal of Structural Biology. 197 (2), 102-113 (2017).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Paul-Gilloteaux, P., et al. EC-CLEM: Flexible multidimensional registration software for correlative microscopies. Nature Methods. 14 (2), 102-103 (2017).
- Heiligenstein, X., Paul-Gilloteaux, P., Raposo, G., Salamero, J. eC-CLEM: A multidimension, multimodel software to correlate intermodal images with a focus on light and electron microscopy. Methods in Cell Biology. 140, 335-352 (2017).
- Fermie, J., et al. Bimodal endocytic probe for three-dimensional correlative light and electron microscopy. Cell Reports Methods. 2 (5), 100220 (2022).
- Fokkema, J., et al. Fluorescently labelled silica coated gold nanoparticles as fiducial markers for correlative light and electron microscopy. Scientific Reports. 8 (1), 13625 (2018).
- Czaja, M. J., et al. Functions of autophagy in normal and diseased liver. Autophagy. 9 (8), 1131 (2013).
- Robinson, J. M., Takizawa, T., Pombo, A., Cook, P. R. Correlative fluorescence and electron microscopy on ultrathin cryosections: Bridging the resolution gap. Journal of Histochemistry and Cytochemistry. 49 (7), 803-808 (2001).
- Mohammadian, S., et al. High accuracy, fiducial marker-based image registration of correlative microscopy images. Scientific Reports. 9 (1), 3211 (2019).
- Tokuyasu, K. T. A study of positive staining of ultrathin frozen sections. Journal of Ultrasructure Research. 63 (3), 287-307 (1978).
- Slot, J. W., Geuze, H. J. A new method of preparing gold probes for multiple-labeling cytochemistry. European Journal of Cell Biology. 38 (1), 87-93 (1985).
- Klumperman, J., Raposo, G. The complex ultrastructure of the endolysosomal system. Cold Spring Harbor Perspectives in Biology. 6 (10), a016857 (2014).
- Geuze, H. J., Slot, J. W., Strous, G. J., Lodish, H. F., Schwartz, A. L. Intracellular site of asialoglycoprotein receptor-ligand uncoupling: Double-label immunoelectron microscopy during receptor-mediated endocytosis. Cell. 32 (1), 277-287 (1983).
- Biazik, J., Ylä-Anttila, P., Vihinen, H., Jokitalo, E., Eskelinen, E. L. Ultrastructural relationship of the phagophore with surrounding organelles. Autophagy. 11 (3), 439-451 (2015).
- Fahimi, H. D., Reich, D., Völkl, A., Baumgart, E. Contributions of the immunogold technique to investigation of the biology of peroxisomes. Histochemistry and Cell Biology. 106 (1), 105-114 (1996).
- Vicidomini, G., et al. A novel approach for correlative light electron microscopy analysis. Microscopy Research and Technique. 73 (3), 215-224 (2010).
- Vicidomini, G., et al. High data output and automated 3D correlative light-electron microscopy method. Traffic. 9 (11), 1828-1838 (2008).
- Cortese, K., et al. 3D HDO-CLEM: cellular compartment analysis by correlative light-electron microscopy on cryosection. Methods in Cell Biology. 111, 95-115 (2012).
- van Rijnsoever, C., Oorschot, V., Klumperman, J. Correlative light-electron microscopy (CLEM) combining live-cell imaging and immunolabeling of ultrathin cryosections. Nature Methods. 5 (11), 973-980 (2008).
- Razi, M., Chan, E. Y. W., Tooze, S. A. Early endosomes and endosomal coatomer are required for autophagy. The Journal of Cell Biology. 185 (2), 305-321 (2009).
- Ligeon, L. A., Barois, N., Werkmeister, E., Bongiovanni, A., Lafont, F. Structured illumination microscopy and correlative microscopy to study autophagy. Methods. 75, 61-68 (2015).
- Biazik, J., Vihinen, H., Anwar, T., Jokitalo, E., Eskelinen, E. L. The versatile electron microscope: An ultrastructural overview of autophagy. Methods. 75, 44-53 (2015).
- Gudmundsson, S., Kahlhofer, J., Baylac, N., Kallio, K., Eskelinen, E. L. Correlative light and electron microscopy of autophagosomes. Methods in Molecular Biology. 1880, 199-209 (2019).
- Kriel, J., et al. Correlative light and electron microscopy (CLEM): bringing together the best of both worlds to study neuronal autophagy. Imaging and Quantifying Neuronal Autophagy. 171, 135-147 (2022).
- Largeau, C., Legouis, R. Correlative light and electron microscopy to analyze LC3 proteins in Caenorhabditis elegans embryo. Methods in Molecular Biology. 1880, 281-293 (2019).
- Fermie, J., et al. Single organelle dynamics linked to 3D structure by correlative live-cell imaging and 3D electron microscopy. Traffic. 19 (5), 354-369 (2018).
- Kuma, A., Matsui, M., Mizushima, N. LC3, an autophagosome marker, can be incorporated into protein aggregates independent of autophagy: Caution in the interpretation of LC3 localization. Autophagy. 3 (4), 323-328 (2007).
- Ladinsky, M. S., Howell, K. E. Electron tomography of immunolabeled cryosections. Methods in Cell Biology. 79, 543-558 (2007).
- Oorschot, V., Lindsey, B. W., Kaslin, J., Ramm, G. TEM, SEM, and STEM-based immuno-CLEM workflows offer complementary advantages. Scientific Reports. 11 (1), 899 (2021).
- Franke, C., et al. Correlative single-molecule localization microscopy and electron tomography reveals endosome nanoscale domains. Traffic. 20 (8), 601-617 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon