Method Article
Modelos de ratón de ingeniería genómica somática utilizando microinyección in vivo y electroporación
En este artículo
Resumen
Este protocolo describe la microinyección y la electroporación in vivo para la edición del genoma mediada por CRISPR restringida regionalmente en el oviducto de ratón.
Resumen
Los modelos de ratón genéticamente modificados de línea germinal (G-GEMM) han proporcionado información valiosa sobre la función génica in vivo en el desarrollo, la homeostasis y la enfermedad. Sin embargo, el tiempo y el costo asociados con la creación y el mantenimiento de colonias son altos. Los avances recientes en la edición del genoma mediada por CRISPR han permitido la generación de GEMM somáticos (S-GEMM) al dirigirse directamente a la célula / tejido / órgano de interés.
El oviducto, o trompa de Falopio en humanos, se considera el tejido de origen del cáncer de ovario más común, los carcinomas ováricos serosos de alto grado (HGSC). Las HGSC se inician en la región de la trompa de Falopio distal al útero, ubicada adyacente al ovario, pero no en la trompa de Falopio proximal. Sin embargo, los modelos tradicionales de ratón de HGSC se dirigen a todo el oviducto y, por lo tanto, no recapitulan la condición humana. Presentamos un método de microinyección de solución de ADN, ARN o ribonucleoproteína (RNP) en la luz del oviducto y electroporación in vivo para atacar células epiteliales de la mucosa en regiones restringidas a lo largo del oviducto. Hay varias ventajas de este método para el modelado del cáncer, tales como 1) alta adaptabilidad en la orientación del área / tejido / órgano y región de electroporación, 2) alta flexibilidad en los tipos de células objetivo (flexibilidad celular) cuando se usa en combinación con promotores específicos para la expresión de Cas9, 3) alta flexibilidad en el número de células electroporadas (frecuencia relativamente baja), 4) no se requiere una línea de ratón específica (modelado de enfermedades inmunocompetentes), 5) alta flexibilidad en la combinación de mutaciones genéticas y 6) posibilidad de rastrear células electroporadas cuando se usa en combinación con una línea reportera Cre. Por lo tanto, este método rentable recapitula la iniciación del cáncer humano.
Introducción
La trompa de Falopio, llamada oviducto en ratones, es una estructura tubular que conecta el útero con el ovario. Desempeña un papel esencial en la reproducción de mamíferos, proporcionando el ambiente para la fertilización interna y el desarrollo preimplantacional 1,2. A pesar de su importancia, poco se sabe sobre su función y homeostasis, en parte debido al desarrollo de técnicas de fertilización in vitro que eluden cualquier problema de infertilidad relacionado con este órgano3. Sin embargo, se ha reconocido que las lesiones precancerosas del carcinoma seroso de ovario de alto grado (HGSC), un histotipo agresivo de cáncer de ovario que representa alrededor del 75% de los carcinomas de ovario y el 85% de las muertes relacionadas4, están restringidas al epitelio distal de la trompa de Falopio 5,6,7,8 . Esto indica que no todas las células de nuestro cuerpo son igualmente susceptibles a los insultos oncogénicos, sino que solo las células únicas / susceptibles en cada tejido / órgano se convierten en la célula de origen en el cáncer, denominada flexibilidad celular9. En esta línea, se ha demostrado que las células epiteliales de la trompa distal de Falopio, situadas adyacentes al ovario, son distintas del resto de la trompa10,11. Por lo tanto, los modelos tradicionales de ratón de HGSC, que se dirigen a todas las células en el oviducto, no recapitulan la condición humana. En un estudio reciente, utilizamos una combinación de edición del genoma mediada por CRISPR, electroporación de oviductos in vivo y rastreo de linaje basado en Cre para inducir con éxito HGSC mediante la mutación de cuatro genes supresores de tumores en el oviducto distalde ratón 12,13. Este manuscrito presenta un protocolo paso a paso que describe este procedimiento de microinyección y electroporación in vivo para apuntar al epitelio de la mucosa del oviducto de ratón.
Este método tiene varias ventajas. Se puede adaptar para atacar otros tejidos/órganos, incluido el parénquima de órganos14. Aunque se pueden usar otros enfoques de administración de genes in vivo, como los sistemas lentiviral y adenoviral, para lograr una orientación similar específica de tejidos / órganos, el área de focalización se ajusta más fácilmente utilizando diferentes tamaños de electrodos tipo pinza para la administración basada en electroporación. Dependiendo de la concentración de ADN/ARN/ribonucleoproteínas (RNPs), parámetros de electroporación y tamaño de los electrodos, el número de células electroporadas puede ser alterado. Además, se pueden atacar tipos de células específicas cuando se usan en combinación con promotores para la expresión de Cas9, sin la necesidad absoluta de modelos de ratón genéticamente modificados de línea germinal (G-GEMM). Además, a diferencia de los sistemas de administración viral, la electroporación permite la entrega de plásmidos múltiples en células individuales y menos restricción en el tamaño de ADN de inserción15. El cribado in vivo de mutaciones genéticas también se puede realizar con relativa facilidad debido a esta alta flexibilidad. Además, las células electroporadas pueden ser rastreadas o rastreadas cuando este método se utiliza en combinación con líneas reporteras Cre como Tdtomato o Confetti16,17.
Protocolo
Los animales fueron alojados en jaulas estáticas de microaislamiento con tapas de filtro, ubicadas en una sala dedicada que contiene un gabinete de bioseguridad tipo II. Todo el trabajo con animales se realizó de acuerdo con las pautas institucionales y fue aprobado por el Comité de Cuidado de Animales de la Facultad de Medicina y Ciencias de la Salud de la Universidad McGill (AUP # 7843).
1. Preparación de la aguja de microinyección
- Tire de un tubo capilar de vidrio en una punta afilada utilizando un extractor de micropipetas con el siguiente programa: P = 500, Calor = 576, PULL = 50, VEL = 80, DEL = 70.
- Usando un par de pinzas de punta ultrafina cónicas, corte el extremo puntiagudo del tubo capilar tirado bajo un microscopio de disección para crear una abertura, como se muestra en la Figura 1A, B. Asegúrese de que la abertura no sea demasiado grande, ya que esto dañará el oviducto y evitará la inyección lenta.
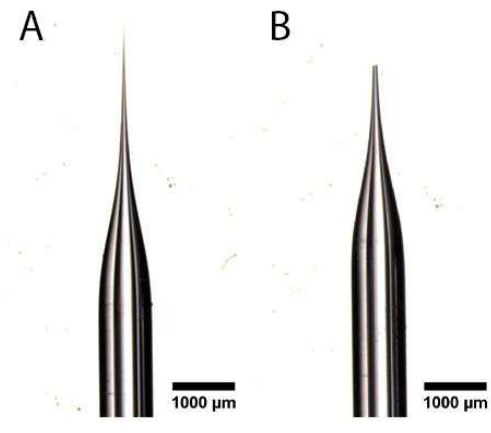
Figura 1: Preparación de la aguja de microinyección. (A,B) Tubo capilar tirado con un extremo puntiagudo (A) que fue cortado para crear una abertura (B). Haga clic aquí para ver una versión más grande de esta figura.
2. Preparación del anestésico para inyección intraperitoneal
- Prepare avertin filtrado y diluido el día de la cirugía. La solución madre de Avertin (2,2,2-tribromoetanol) se prepara a una concentración de 1,6 g/ml en alcohol terc-amílico agitando vigorosamente y se almacena en la oscuridad.
- Diluir la solución madre de avertin con solución salina isotónica al 1,25% v/v hasta una concentración final de 20 mg/ml dentro de una campana extractora. Vortex el avertin diluido y filtrar a través de un filtro estéril de 0,22 μm. Manténgase en la oscuridad.
3. Preparación del área de cirugía
- El área de la cirugía debe ser un ambiente estéril dedicado, preferiblemente un banco limpio o un gabinete de bioseguridad. Organice el equipo de cirugía estéril, el microscopio, el micromanipulador y el electroporador según sea necesario. Un ejemplo de esto se muestra en la Figura 2.
- Caliente la almohadilla térmica / disco y colóquelo debajo de una jaula limpia y totalmente equipada. Esta jaula se utilizará para albergar a los ratones después de la cirugía.
- Vierta 1x solución salina tamponada con fosfato estéril (PBS) en una placa de Petri estéril. Use un par de tijeras limpias para cortar papel absorbente en cuadrados, con dimensiones aproximadas de 1 cm x 1 cm. Deje caer estos trozos de papel absorbente en el PBS 1x para remojarlos.
4. Preparación de la solución y la aguja inyectable
- Prepare la solución inyectable el día de la cirugía. Mezclar el ADN/ARN/RNPs con azul de tripano filtrado hasta una concentración final mínima de 200 ng/μL cada uno y 5%, respectivamente. Guarde este producto a temperatura ambiente antes de la inyección.
NOTA: Use una jeringa de 1 ml para tomar 100% de azul de tripano y filtrar a través de un filtro estéril de 0,22 μm antes de su uso. - Acople la aguja capilar tirada, en adelante llamada aguja de microinyección, al micromanipulador de montaje de abrazadera estabilizado por un soporte magnético (configuración que se muestra en la Figura 2B).
- Pipetear 2 μL de la solución inyectable sobre una placa de Petri estéril. Mientras observa bajo el microscopio, tome lentamente 1-2 μL de esta solución en la aguja de microinyección usando la jeringa a presión de aire conectada al micromanipulador. Evite las burbujas/aspirar burbujas en la aguja de microinyección.

Figura 2: Preparación del área de cirugía. (A) Configuración del área de cirugía dentro de un banco limpio. (B) Configuración del microscopio de disección y del micromanipulador para un individuo diestro. Haga clic aquí para ver una versión más grande de esta figura.
5. Exponer el tracto reproductor femenino
- Anestesiar a una ratona hembra de 6-8 semanas administrando avertin (anestésico) filtrado y diluido por vía intraperitoneal a una dosis de 0,25-0,5 mg/g. Después de esto, administrar carprofeno (analgésico preventivo) por vía subcutánea a una dosis de 5 mg / kg.
NOTA: Otras alternativas que se pueden usar como anestésico son isoflurano o ketamina/xilazina. - Coloque el papel absorbente en una almohadilla / disco limpio y calentado. Luego, coloque el ratón anestesiado sobre este papel absorbente caliente con el lado dorsal hacia arriba. Aplique lubricante ocular en cada ojo para evitar que se seque durante la cirugía.
NOTA: Confirme que la almohadilla térmica / disco está ligeramente caliente al tacto probando con el dorso de la mano. - Pruebe el paro anestésico profundo pellizcando el dedo del pie del ratón anestesiado con un par de fórceps. Retire el pelaje dorsal alrededor del sitio de la posible incisión (ubicación que se muestra en la Figura 3A) y limpie la piel desnuda con antiséptico.
- Use un par de pinzas rectas romas para pellizcar la piel desnuda y crear una incisión de 1 cm de largo a lo largo de la línea media del cuerpo con un par de tijeras romas afiladas estériles. Limpie el área alrededor de la incisión con antiséptico.
- Pellizque y sostenga un lado del sitio del corte con un par de pinzas Adson estériles. Luego, usando un par de pinzas dentadas curvas estériles, separe suavemente la piel de la pared del cuerpo, comenzando en la incisión de la línea media y moviéndose lateralmente.
- Localice la almohadilla de grasa debajo del riñón. Con un par de pinzas rectas y romas estériles, pellizque la pared del cuerpo directamente sobre la almohadilla de grasa. Cree una pequeña incisión en la pared del cuerpo con un par de tijeras de disección estériles de punta afilada, teniendo cuidado de evitar los vasos sanguíneos.
- Mientras sigue pellizcando la pared del cuerpo con pinzas rectas romas, inserte un par de pinzas curvas romas estériles en la incisión y ensanche la incisión creada en la pared del cuerpo.
- Agarre la almohadilla de grasa visible con las pinzas curvas romas y sáquela del orificio para exponer el ovario, el oviducto y el útero.
- Para mantener el tracto reproductivo expuesto, sujete la almohadilla de grasa con una pinza de bulldog estéril, como se muestra en la Figura 3B.
6. Inyección de oviducto in vivo y electroporación
- Coloque cuidadosamente el papel absorbente calentado y el ratón anestesiado con el tracto reproductivo expuesto en el escenario de un microscopio de disección, de modo que se pueda observar el tracto. Un ejemplo de la vista observada bajo el microscopio se muestra en la Figura 3B.
- Ajuste el micromanipulador para que la aguja de inyección con solución también se pueda observar bajo el microscopio.
- Usando un par de pinzas cónicas estériles de punta ultrafina, mantenga estable la región del oviducto que se va a inyectar. La región a inyectar debe estar en línea con la dirección de la aguja de microinyección.
NOTA: Use la abrazadera bulldog que ancla la almohadilla de grasa y el tracto reproductivo para girar / mover suavemente el tracto a una posición óptima, si es necesario. - Ajuste el micromanipulador para perforar el oviducto con la aguja de microinyección, mientras alimenta simultáneamente el oviducto en la aguja utilizando las pinzas de punta ultrafina cónica. Mueva suavemente la aguja de microinyección para confirmar que se ha insertado en el oviducto.
NOTA: Inserte la aguja de microinyección en un segmento recto en lugar de en un punto de inflexión del oviducto enrollado para evitar múltiples punciones y fugas de solución inyectable. - Inyecte lentamente hasta 1 μL de solución en el oviducto, mientras observa el movimiento de la solución azul y la expansión de la luz del oviducto bajo el microscopio. Tenga cuidado de no introducir burbujas en la luz. Las imágenes representativas de oviductos inyectados con 100% de azul de tripano se muestran en la Figura 3C, D.
- Retire la aguja del oviducto. Cubra el área a tratar con un trozo de papel absorbente empapado previamente en PBS estéril 1x (a partir del paso 3.3).
- Agarre el papel preempapado y el área/región a la que se dirigirá con un par de pinzas de electrodos (1 mm, 3 mm o 5 mm, dependiendo del tamaño de la región a la que se dirigirá) y retírese del cuerpo, antes de electroporar utilizando estos ajustes en el generador / electroporador de pulsos: 30 V, tres pulsos, intervalo de 1 s, 50 ms de longitud de pulso, unipolar.
NOTA: La distancia entre los electrodos debe ajustarse de tal manera que los electrodos puedan sujetarse a la región objetivo sin deformar el oviducto. La distancia recomendada es de alrededor de 1 mm. - Después de la electroporación, retire el papel absorbente y coloque el ratón (con el papel absorbente calentado debajo) de nuevo en una almohadilla térmica. Desenganche la pinza bulldog y empuje con cuidado el tracto reproductivo expuesto debajo de la pared del cuerpo. Repita la sección 5 para exponer el tracto reproductivo del otro lado.
NOTA: La electroporación es exitosa independientemente de la etapa estral del ratón, siempre y cuando la luz esté llena con la solución. - Cubra el tracto expuesto con papel absorbente empapado previamente en 1x PBS estéril para evitar que se seque, luego prepare la aguja de microinyección para la segunda inyección repitiendo los pasos 4.2 y 4.3.
NOTA: Reemplace la aguja de microinyección según sea necesario. Evite el secado del tejido expuesto aplicando 1x PBS estéril cuando sea necesario. - Repita los pasos anteriores para inyectar y electropolar el oviducto en el lado opuesto.
NOTA: Vuelva a aplicar el lubricante ocular según sea necesario durante y después de la cirugía. - Después de que ambos oviductos hayan sido electroporados y colocados de nuevo debajo de la pared del cuerpo, suture o grape el sitio de la incisión dorsal. Para la sutura, use un hemostático para agarrar la aguja de las suturas trenzadas de seda (círculo 3/8; calibre: 5-0; tamaño de la aguja: 18 mm; longitud del hilo: 75 cm). Pellizque y sostenga un lado del sitio del corte con un par de pinzas dentadas curvas estériles, luego use el hemostático para manipular la sutura para cerrar la herida.
- Mueva el ratón a una jaula limpia que se coloca encima de una almohadilla/disco caliente (desde el paso 3.2) y supervise hasta que el ratón esté activo.
- Administrar 5 mg/kg de carprofeno por vía subcutánea cada 24 h durante los próximos 3 días y vigilar durante los próximos 10 días.
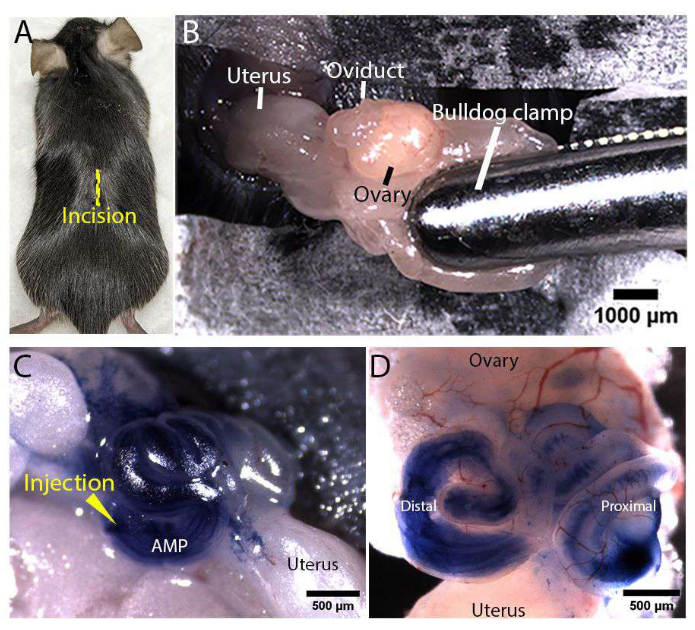
Figura 3: Exposición del aparato reproductor femenino y microinyección . (A) Ubicación de la incisión en la línea media (mostrada como una línea amarilla) en el lado dorsal de un ratón Rosa-LSLtdTomato. (B) Exposición del tracto reproductor femenino. La almohadilla de grasa se sujetó con una abrazadera de bulldog estéril para anclar el tracto y mantenerlo expuesto. (C,D) Imágenes representativas de la inyección de oviducto in vivo . Se insertó una aguja de microinyección en la ampolla distal (marcada como AMP) y se inyectó azul de tripano 100% filtrado en la luz del oviducto mientras el tracto estaba expuesto (C). Imagen representativa de un oviducto disecado, que demuestra que la solución de azul de tripano inyectada se distribuye por toda la luz del oviducto distal y proximal (D). Haga clic aquí para ver una versión más grande de esta figura.
Resultados
La Figura 1 y la Figura 2 muestran la preparación de la aguja de microinyección y la configuración del área de cirugía, respectivamente, para la microinyección in vivo y la electroporación del oviducto de ratón. Durante la cirugía, el tracto reproductor femenino fue expuesto a través de incisiones realizadas en la piel dorsal (Figura 3A) y la pared corporal de un ratón anestesiado Rosa-LSLtdTomato (Gt(ROSA)26Sortm14(CAG-tdTomato)Hze/J)17. Se sujetó una abrazadera de bulldog a la almohadilla de grasa sobre el tracto para mantenerla expuesta y anclada (Figura 3B). Se inyectó una solución inyectable que contenía plásmidos PCS2 CreNLS18 a una concentración de 400 ng/μL en la ampolla (AMP marcado) del oviducto enrollado y se dejó dispersar por toda la luz del oviducto (Figura 3C, D). Aunque se utilizaron plásmidos PCS2 CreNLS para generar resultados representativos en este manuscrito, el método descrito se puede utilizar con ADN/ARN/RNP fácilmente intercambiables en la solución de inyección para la edición de genes mediada por CRISPR/Cas en el oviducto de ratón. Luego, se colocaron electrodos tipo pinza de 1 mm o 3 mm, de modo que el oviducto distal estuviera entre los electrodos para la administración basada en electroporación.
Los oviductos se cosecharon 4 días después de la cirugía y se estiraron mediante la eliminación del mesosalpinx para visualizar la especificidad de la electroporación. Usando electrodos tipo pinza de 1 mm o 3 mm, el área objetivo, marcada con TdTomato, se restringió al epitelio del oviducto distal (Figura 4A, D). El tamaño de esta área objetivo se controló aún más mediante el uso de electrodos de diferentes tamaños. Usando electrodos tipo pinza de 3 mm, apuntamos a un área mucho más grande del AMP (Figura 4B, C), en comparación con apuntar solo a la punta más distal, el INF (Figura 4E, F), utilizando electrodos tipo pinza de 1 mm. Estos oviductos cosechados se fijaron, se trataron con un gradiente de sacarosa y se seccionaron utilizando un criostato para análisis detallados. Las secciones se contrastaron con Hoescht 33342 y faloidina para teñir los núcleos y el citoesqueleto respectivamente, luego se obtuvieron imágenes utilizando un microscopio confocal de barrido puntual. En la región objetivo, las células electroporadas, marcadas por TdTomato, se distribuyeron aleatoriamente entre células no electroporadas (TdTomato-ve) (Figura 4G). Además, las células electroporadas se restringieron al epitelio de la mucosa y no se encontraron en la capa estromal o muscular subyacente (Figura 4G, H). Finalmente, para confirmar la edición génica mediada por CRISPR-Cas, las células TdTomato+ve pueden aislarse mediante clasificación celular activada por fluorescencia (FACS) para el aislamiento de ADN y secuenciación de MiSeq12.
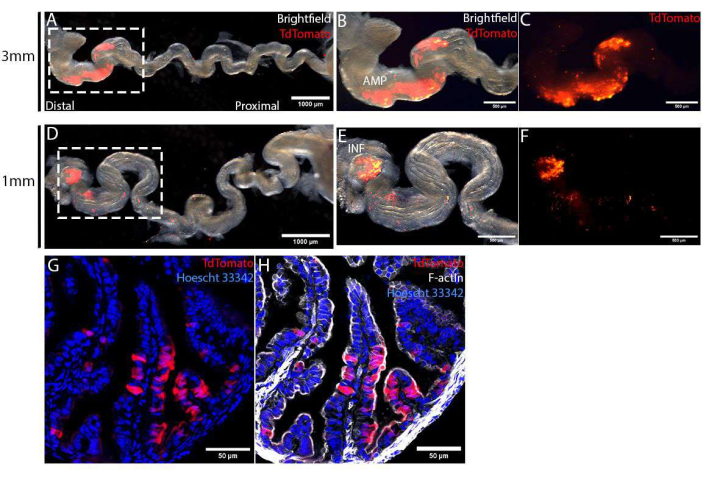
Figura 4: Validación de la inyección y electroporación in vivo exitosa de células epiteliales del oviducto, 4 días después de la cirugía. (A-C) Electroporación utilizando electrodos tipo pinza de 3 mm de tamaño. El oviducto se desenrolló eliminando el mesosalpinx para una mejor visualización de la especificidad de la electroporación (A). El área de electroporación, identificada por la expresión de TdTomato, se restringió al oviducto distal, marcado como AMP (B, C). (D-F) Electroporación mediante electrodos tipo pinza de 1 mm. El oviducto se desenrolló eliminando el mesosalpinx para una mejor visualización de la especificidad de la electroporación (D). El área de electroporación se restringió al infundíbulo (etiquetado INF), la punta más distal del oviducto (E, F). (G,H) Sección transversal del oviducto distal 4 días después de la electroporación con plásmidos PCS2 CreNLS. Las células electroporadas marcadas con TdTomato (G) se restringieron a la monocapa epitelial (H). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
Los pasos cruciales en este protocolo detallado son la microinyección de solución de ADN / ARN / RNP en el lumen del oviducto y el control de la fuerza y el área de electroporación. La fuga de solución de ADN/ARN/RNP durante la microinyección puede causar la transfección de áreas/células no deseadas. Para una electroporación consistente y eficiente, es preferible llenar el lumen del oviducto con la solución (Figura 3C, D). Esto se debe a que el área de electroporación está controlada principalmente por el tamaño y la colocación del electrodo. La electroporación débil reduce el número de células electroporadas, y la electroporación severa puede causar la electroporación de células no deseadas o alterar la estructura del tejido. El número de células electroporadas se puede variar ajustando la concentración de la solución de ADN/ARN/RNP o los parámetros de electroporación. Sin embargo, dado que los altos voltajes / producción de calor durante la electroporación podrían dañar el tejido, estos parámetros deben probarse antes de su uso en ratones vivos / anestesiados. Finalmente, debido a la naturaleza exigente de este procedimiento, se recomienda practicar la exposición del tracto y la microinyección en ratones muertos antes de realizar esta cirugía en ratones vivos / anestesiados.
Se reconoce que múltiples genes están involucrados en la iniciación del cáncer, por lo tanto, recapitular este evento requiere la modificación multialélica de varios genes. Además, también se sabe que no todas las células son igualmente susceptibles a las mutaciones oncogénicas9. El modelado del cáncer, por lo tanto, requiere líneas específicas de ratón Cre para lograr un control estricto de los insultos oncogénicos. Sin embargo, su disponibilidad y especificidad causa varios desafíos, incluyendo efectos en tejidos no dirigidos y letalidad19. Además, los G-GEMM tradicionales incurren en altos costos de mantenimiento y requieren tiempo para la generación de la línea del mouse. El desarrollo de la tecnología de edición del genoma CRISPR / Cas9 y la mejora de la entrega de genes en células somáticas específicas nos ha permitido superar estos problemas. En este protocolo, presentamos un método de administración de ADN/ARN/RNP para la manipulación del genoma somático que puede ser utilizado para el modelado del cáncer, sin la necesidad absoluta de líneas específicas de ratón12. Mediante la inyección de soluciones de ADN/ARN/RNP en el lumen y el uso de diferentes tamaños de electrodos tipo pinza para la administración basada en electroporación, se dirigen áreas restringidas del epitelio de la mucosa del oviducto de ratón (Figura 4A-F). Esto es especialmente útil para modelar el inicio de HGSC que se originan en el extremo distal de la trompa de Falopio.
El método presentado de microinyección y electroporación in vivo es muy versátil para dirigirse a tejidos/órganos con un lumen. El parénquima orgánico también es objetivo utilizando este método14. Los enfoques de administración viral, como los sistemas lentiviral y adenoviral, también se pueden usar para fines similares. Sin embargo, las ventajas de la electroporación sobre los enfoques de administración viral son: 1) administración múltiple de plásmidos en células individuales, 2) sin restricción en el tamaño del inserto15 y 3) fácil control del área y el momento de la administración. Además, la especificidad de la focalización se puede mejorar mediante el uso de promotores específicos del linaje. Esto a veces es difícil en los sistemas virales debido al límite en el empaquetado viral.
Como es la naturaleza de la electroporación, las células epiteliales electroporadas se intercalan aleatoriamente con células sanas sin editar (Figura 4G). Sin embargo, este mosaicismo en células modificadas genéticamente y no modificadas podría ser ventajoso para estudiar la iniciación del cáncer, ya que recapitula la naturaleza esporádica del inicio temprano del cáncer dentro de un microambiente inmunocompetente. La frecuencia de las células electroporadas se puede ajustar variando las concentraciones de la solución de ADN/ARN/RNP y/o los parámetros de electroporación. Utilizando la edición de genes mediada por CRISPR-Cas en combinación con el protocolo presentado, es fácil detectar múltiples genes y generar patrones heterogéneos de mutaciones / combinaciones alélicas en genes específicos; esto es particularmente útil en el modelado de cánceres que se presentan con un número considerable de alteraciones genómicas como HGSCs20,21. Además, mediante la secuenciación de genes dirigidos durante la progresión del cáncer, es posible seguir la evolución clonal in vivo de tumores y metástasis en ratones inmunocompetentes12.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
Los autores agradecen a Katie Teng y al Dr. Matthew J. Ford por proporcionar plásmidos utilizados para generar resultados representativos y optimización de protocolos. K.H. fue apoyado por la beca doctoral Fonds de recherche du Québec - Santé (FRQS), la Fundación Donnor, Delta Kappa Gamma World Fellowship, el Centro de Investigación en Reproducción y Desarrollo (CRRD), Hugh E. Burke, Rolande & Marcel Gosselin y Alexander McFee.
Materiales
| Name | Company | Catalog Number | Comments |
| 0.22 µm sterile filter unit | LifeGene | SF0.22PES | |
| 1 mL syringe | Terumo Medical Corportation | SS-01T | |
| 1 mm tweezer-type electrodes | BEX CO., LTD | LF650P1 | |
| 3 mm tweezer-type electrodes | BEX CO., LTD | LF650P3 | |
| 30G x 1/2 Needle | BD | 305106 | Needle is attached to the 1 mL syringe when using injectable anesthetic or analgesic. |
| Adson forceps | Fisher Scientific | 10-000-451 | |
| Air-pressured syringe | BD | B302995 | Attached to the micromanipulator; as shown in Figure 2B |
| Analgesic | - | - | Carprofen, dose: 5mg/kg. |
| Antiseptic | Cardinal Health | OMEL0000017 | Baxedin: 2% chlorhexidine in 70% isopropyl alcohol |
| Avertin (2,2,2-tribromoethanol) - Anesthetic | Sigma Aldrich | T48402-25G | |
| Clamp mount micromanipulator | AD Instruments | MM-33 | |
| Curved serrated forceps | Fisher Scientific | NC0696845 | |
| Dissecting microscope | Zeiss | SteREO Discovery.V8 | Apochromat S, 0.63X, FWD 107mm. Attached KL 200 LED for top light, labelled as light source in Figure 2B. |
| Eye lubricant | Alcon | - | Systane ointment (Lubricant eye ointment) |
| Glass capillary needles | Sutter Instrument Co. | B100–50-10 | Outside diameter: 1 mm, inside diameter: 0.5 mm, length: 10 cm |
| Hartman hemostats | Fisher Scientific | 50-822-711 | |
| Heating pad/disc | - | - | SnuggleSafe Pet Bed Microwave Heating Pad was used in this protocol. Microwave for 2-5min, and test with back of gloved hand. |
| Magnetic Mount for micromanipulator | AD Instruments | MB-B | Used to keep the clamp mount micromanipulator stable and upright during injection. |
| Micro bulldog clamp | Fisher Scientific | 50-822-230 | 3 cm |
| Micropipette puller | Sutter Instrument Co. | P-97 | P-97 Flaming/Brown type micropipette puller. Program used: P = 500, Heat = 576, PULL = 50, VEL = 80, DEL = 70 |
| Petri dish | Fisher Scientific | 263991 | Autoclaved/sterile glass petridishes can also be used. |
| Phosphate Buffered Saline (PBS) | Bio Basic Inc. | PD8117 | 10X PBS was diluted to 1X with DI water. Autoclave before use. |
| Pulse generator/Electroporator | BEX CO., LTD | CUY21 EDIT II | Electroporation settings: 30 V, 3 pulses, 1 s interval, P. length = 50 ms, unipolar |
| Rosa-LSLtdTomato mice | The Jackson Laboratory | 7914 | Gt(ROSA)26Sortm14(CAG-tdTomato)Hze/J |
| Sharp-blunt dissection scissors | Fisher Scientific | 28251 | |
| Sharp-pointed dissecting scissors | Fisher Scientific | SDI130 | |
| Shaver | - | - | Hair removal cream can also be used. |
| Silk braided sutures | Ethicon | 682G | 3/8 circle, gauge: 5-0, needle size: 18 mm, thread length: 75 cm. Staples can also be used. |
| Tapered ultrafine tip forceps | Fisher Scientific | 12-000-122 | |
| Thin absorbent paper | Kimberly-Clark Professional | 34120 | Kimwipes |
| Trypan blue | STEMCELL Technologies | 7050 | Filtered using 0.22 µm sterile filter before use for microinjection. |
Referencias
- Avilés, M., Coy, P., Rizos, D. The oviduct: A key organ for the success of early reproductive events. Animal Frontiers. 5 (1), 25-31 (2015).
- Li, S., Winuthayanon, W. Oviduct: roles in fertilization and early embryo development. The Journal of Endocrinology. 232 (1), R1-R26 (2017).
- Briceag, I., et al. Fallopian tubes--literature review of anatomy and etiology in female infertility. Journal of Medicine and Life. 8 (2), 129-131 (2015).
- Perets, R., et al. Transformation of the fallopian tube secretory epithelium leads to high-grade serous ovarian cancer in Brca;Tp53;Pten models. Cancer Cell. 24 (6), 751-765 (2013).
- Labidi-Galy, S. I., et al. High grade serous ovarian carcinomas originate in the fallopian tube. Nature Communications. 8 (1), 1093(2017).
- Shaw, P. A., Rouzbahman, M., Pizer, E. S., Pintilie, M., Begley, H. Candidate serous cancer precursors in fallopian tube epithelium of BRCA1/2 mutation carriers. Modern Pathology. 22 (9), 1133-1138 (2009).
- Soong, T. R., et al. Evidence for lineage continuity between early serous proliferations (ESPs) in the Fallopian tube and disseminated high-grade serous carcinomas. The Journal of Pathology. 246 (3), 344-351 (2018).
- Lee, Y., et al. A candidate precursor to serous carcinoma that originates in the distal fallopian tube. The Journal of Pathology. 211 (1), 26-35 (2007).
- Puisieux, A., Pommier, R. M., Morel, A. P., Lavial, F. Cellular pliancy and the multistep process of tumorigenesis. Cancer Cell. 33 (2), 164-172 (2018).
- Harwalkar, K., et al. Anatomical and cellular heterogeneity in the mouse oviduct-its potential roles in reproduction and preimplantation development. Biology of Reproduction. 104 (6), 1249-1261 (2021).
- Ford, M. J., et al. Oviduct epithelial cells constitute two developmentally distinct lineages that are spatially separated along the distal-proximal axis. Cell Reports. 36 (10), 109677(2021).
- Teng, K., et al. Modeling high-grade serous ovarian carcinoma using a combination of in vivo fallopian tube electroporation and CRISPR-Cas9-mediated genome editing. Cancer Research. 81 (20), 5147-5160 (2021).
- Ford, M. J., Yamanaka, Y. Reprogramming mouse oviduct epithelial cells using in vivo electroporation and CRISPR/Cas9-mediated genetic manipulation. Methods in Molecular Biology. 2429, 367-377 (2022).
- Maresch, R., et al. Multiplexed pancreatic genome engineering and cancer induction by transfection-based CRISPR/Cas9 delivery in mice. Nature Communications. 7, 10770(2016).
- Braun, C. J., Adames, A. C., Saur, D., Rad, R. Tutorial: design and execution of CRISPR in vivo screens. Nature Protocols. 17 (9), 1903-1925 (2022).
- Livet, J., et al. Transgenic strategies for combinatorial expression of fluorescent proteins in the nervous system. Nature. 450 (7166), 56-62 (2007).
- Madisen, L., et al. A robust and high-throughput Cre reporting and characterization system for the whole mouse brain. Nature Neuroscience. 13 (1), 133-140 (2010).
- Chazaud, C., Yamanaka, Y., Pawson, T., Rossant, J. Early lineage segregation between epiblast and primitive endoderm in mouse blastocysts through the Grb2-MAPK pathway. Developmental Cell. 10 (5), 615-624 (2006).
- Heyer, J., Kwong, L. N., Lowe, S. W., Chin, L. Non-germline genetically engineered mouse models for translational cancer research. Nature Reviews. Cancer. 10 (7), 470-480 (2010).
- Kroeger Jr, P. T., Drapkin, R. Pathogenesis and heterogeneity of ovarian cancer. Current Opinion in Obstetrics & Gynecology. 29 (1), 26-34 (2017).
- Cancer Genome Atlas Research Network. Integrated genomic analyses of ovarian carcinoma. Nature. 474 (7353), 609-615 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados