Method Article
Tomografía tridimensional de órgano entero de alta resolución de infecciones microbianas
En este artículo
Resumen
Aquí, describimos un procedimiento que permite la detección de bacterias patógenas en todo el órgano durante la infección y la cuantificación de las actividades de los reporteros fluorescentes.
Resumen
La mayoría de las infecciones tienen lugar dentro de tejidos tridimensionales del huésped con una anatomía intrincada y una fisiología del huésped que varía localmente. La posición de las células patógenas dentro de este entorno diverso afecta significativamente sus niveles de estrés, respuestas, destino y contribución a la progresión general de la enfermedad y al fracaso del tratamiento. Sin embargo, debido a las dificultades técnicas para localizar células patógenas de tamaño μm dentro de órganos huésped de tamaño cm, esta área de investigación ha sido relativamente inexplorada. Aquí, presentamos un método para abordar este desafío. Empleamos tomografía seriada de dos fotones y análisis de imágenes mejorado por IA para localizar células individuales de Salmonella en todo el bazo, los lóbulos del hígado y los ganglios linfáticos completos de ratones infectados. Mediante el uso de reporteros fluorescentes y la administración de anticuerpos in vivo , se puede determinar la tasa de replicación de células individuales de Salmonella , su interacción local con células inmunitarias específicas y las respuestas bacterianas a los antibióticos. Estas metodologías abren vías para un examen exhaustivo de las infecciones, su prevención y tratamiento en el contexto de los tejidos tridimensionales.
Introducción
Las infecciones ocurren en tejidos con anatomía compleja y fisiología compartimentada. Los diversos microambientes que coexisten en el tejido infectado pueden determinar el destino de los subconjuntos de patógenos locales y sus contribuciones al resultado general de la enfermedad 1,2,3. Sin embargo, el mapeo 3D exhaustivo de patógenos microbianos en tejidos de tamaño CM sigue siendo un desafío4. La obtención de imágenes del cerebro y otros órganos es un campo de investigación muy activo con estrategias experimentales en constante mejora5, pero muchos métodos aún carecen de la resolución inferior a μm que se requeriría para identificar patógenos bacterianos de tamaño μm con confianza. Por el contrario, la tomografía seriada de dos fotones (STP)6 permite la obtención automática de imágenes multicolor y sin deformaciones de tejidos completos con una resolución en el plano inferior a μm, lo que produce conjuntos de datos volumétricos completos. Este método combina la sección física repetida del tejido utilizando un vibratomo con imágenes intermitentes de dos fotones de las caras de los bloques emergentes con luz infrarroja. La tomografía STP ha sido ampliamente utilizada para mapear axones delgados en el cerebro para establecer mapas de conectividad 7,8,9,10.
La tomografía STP también permite el mapeo 3D de células patógenas microbianas individuales (Salmonella, Toxoplasma) en todos los tejidos infectados11,12 utilizando un tomógrafo. La generación del segundo armónico revela vainas de colágeno alrededor de las arterias y en bandas fibrosas como las trabéculas del bazo, proporcionando así un contexto anatómico. Los anticuerpos fluorescentes inyectados in vivo se pueden utilizar para teñir las células huésped y revelar las interacciones entre las células patógenas individuales y las células inmunitarias infiltrantes, como los neutrófilos. Aquí, se describe la tubería que involucra el procesamiento del tejido, la obtención de imágenes, la unión de mosaicos de imágenes con corrección de iluminación, el apilamiento de imágenes en tres dimensiones y la segmentación mediante herramientas de aprendizaje automático. Esta tubería produce posiciones 3D de patógenos individuales, células y microcolonias dentro de su contexto de huésped. Contar el número de células individuales dentro de las microcolonias sigue siendo difícil debido a los límites de resolución, pero tales números se pueden estimar en función del brillo integrado de la microcolonia. La tubería se puede adaptar fácilmente a otros modelos de infección si se dispone de patógenos recombinantes que expresan GFP o YFP.
Protocolo
Todos los experimentos con animales descritos aquí han sido aprobados por las autoridades (licencia 2239, Kantonales Veterinäramt Basel) y siguen las directrices locales (Tierschutz-Verordnung, Basilea) y la ley suiza de protección animal (Tierschutz-Gesetz).
1. Preparación y almacenamiento de tejidos infectados
- Utilice un modelo de infección de su elección. Aquí, ratones de 10 a 16 semanas de edad de ambos sexos están infectados con ~ 1,000 unidades formadoras de colonias (UFC) por inyección intravenosa o 5 x 107 UFC por administración intragástrica con una aguja de punta redonda. Las cepas de ratón adecuadas incluyen BALB/c y C57BL/6.
NOTA: El mismo enfoque se puede adaptar fácilmente para otras especies hospederas. - Asegúrese de que los patógenos expresen proteínas fluorescentes para permitir su identificación en el tejido no teñido. Los patógenos que expresan GFP.mut213, YPet14, TIMERbac15 o mWasabi16 se pueden visualizar utilizando una excitación de 940 nm17. La salmonela que expresa mCherry18 a niveles fácilmente detectables por citometría de flujo19 podría obtenerse utilizando una excitación de 800 nm, pero tiene una mala relación señal-fondo en el bazo y el hígado infectados, que muestran una autofluorescencia sustancial.
- Asegúrese de que todas las proteínas fluorescentes conserven el >70% de su intensidad de fluorescencia después de la fijación con paraformaldehído a pH neutro basada en la cuantificación con citometría de flujo. Aquí, se utiliza tejido de ratón infectado, a saber, bazo, hígado, ganglios linfáticos y parches de Peyer.
- Teñir los marcadores de superficie de la célula huésped in vivo inyectando anticuerpos marcados con ficoeritrina (PE) 10 min antes de la perfusión. Para teñir los neutrófilos, inyecte por vía intravenosa 4 μg de anti-Ly-6G-PE en 100 μL de PBS. Para teñir los macrófagos de la zona marginal en el bazo, inyectar 4 μg de anti-CD169-PE en 100 μL de PBS (ver Tabla de Materiales).
- Fijar los tejidos infectados mediante perfusión transcárdica estándar20 con 15 mL de tampón de fosfato 50 mM helado (PB; 10 mM NaH2PO4, 40 mMNa2HPO4, pH 7,4), seguido de 35 mL de paraformaldehído al 4% (PFA; fijador) en PB (Figura 1A).
- En resumen, administrar anestesia profunda al animal infectado mediante inyección intraperitoneal de 100 mg/kg de ketamina y 16 mg/kg de xilacina. Asegure una anestesia adecuada confirmando la pérdida del reflejo de pellizco del dedo del pie.
- Desinfecte el pelaje con etanol al 70%, séquelo con una toalla de papel estéril y abra quirúrgicamente el pecho con pinzas y tijeras. Coloque una aguja estéril en el ventrículo izquierdo del corazón, abra la aurícula derecha con tijeras estériles y pase el primer tampón y luego el fijador a través de la aguja, el ventrículo izquierdo y todo el sistema vascular con una bomba. Esto fija todos los tejidos de forma rápida y uniforme y provoca la eutanasia.
- Recoja el tejido después de la perfusión, incube en un volumen 20x de paraformaldehído frío al 4% en tampón PB y colóquelo en un agitador a 4 °C durante la noche (Figura 1B). La obtención de imágenes es posible para tejidos en el rango de tamaño de 0,05 a 2 cm3. Para pañuelos más grandes, córtelos en trozos adecuados.
- Al día siguiente, lave el pañuelo 3 veces durante 10 minutos cada una con tampón PB agitando a 40 rpm a temperatura ambiente para eliminar el PFA.
NOTA: Debido a la toxicidad del PFA, estos pasos deben llevarse a cabo dentro de una campana extractora. Deseche los residuos de PFA de acuerdo con las pautas institucionales. - Retirar el exceso de tampón PB y añadir 20x volumen de tampón de almacenamiento crioprotector21 (876 mM de sacarosa, 0,25 mM de polivinilpirrolidona, 40% (v/v) de etilenglicol, disuelto en 50 mM de tampón PB) a la muestra agitando a 40 rpm a 4 °C durante 6-8 h o hasta que los tejidos se hundan hasta el fondo. Incubar las muestras a -20 °C durante al menos 3 días para garantizar una reducción eficaz de la autofluorescencia tisular11. Las muestras se pueden utilizar hasta 5 años cuando se almacenan a -20 °C.
NOTA: El hundimiento del tejido indica la penetración adecuada del tampón de almacenamiento en el tejido.
2. Incrustación de muestras
- Incruste el tejido en un bloque de agarosa para un corte suave con un vibrátomo. Preactive la agarosa químicamente por oxidación con periodato y luego reduzca con borohidruro para formar reticulación con el tejido para mejorar la estabilidad durante el proceso de corte. Realice los pasos que se describen a continuación.
- Preparar agarosa oxidada al 4% pesando 2,25 g de agarosa y añadir 0,21 g de NaIO4. Añadir 100 mL de PB a la agarosa y NaIO4. Remover a temperatura ambiente en una campana extractora durante 2-3 h con protección contra la luz.
NOTA: Paso crítico: No exceda la agitación durante más de 3 h o la agarosa se polimerizará mal. - Recuperar la agarosa por filtración por membrana en un filtro de vacío estándar con una membrana de 0,2 μm. Elimine el NaIO4 restante lavando 3 veces con 50 ml de tampón PB. Vuelva a suspender la agarosa oxidada en 50 mL de tampón PB.
NOTA: No derrita la agarosa durante esta etapa. La agarosa puede almacenarse a 4 °C durante un máximo de 2-3 semanas protegida de la luz. - Prepare la solución de borohidruro añadiendo 19 g de bórax y 3 g de ácido bórico a 1 L de agua. Revuelva hasta que se disuelva y ajuste el pH a 9-9.5 con 1 M de NaOH. En una placa de agitación en una campana extractora, calentar 100 ml de tampón de borato a 40 °C. Añadir 0,2 g de NaBH4 y remover durante 15-30 min mientras se protege de la luz.
NOTA: La solución de borohidruro se puede almacenar durante varias semanas a temperatura ambiente en la oscuridad. Paso crítico: Al agregar NaBH4 al tampón de borato, se formará gas, por lo tanto, realice este paso dentro de una campana extractora. No realice este paso en un recipiente herméticamente cerrado porque podría provocar una explosión. - Tome el tejido del congelador después de una incubación suficiente para reducir la autofluorescencia (≥ 3 días), elimine el exceso de tampón de almacenamiento de crioprotector y lave el tejido 3 veces durante 10 minutos cada uno con tampón PB agitando a 40 rpm a temperatura ambiente (RT).
- Derretir aproximadamente 20 mL de suspensión de agarosa oxidada (preparada en el paso 1) en un microondas (700 W durante ~30 s), dejar que se enfríe lo suficiente como para manipularla (~45 °C) y verterla en un molde de plástico.
NOTA: Paso crítico: No agregue la agarosa directamente después de calentarla en el microondas sobre la muestra, esto podría llevar a la destrucción del tejido. - Use una pinza para recoger suavemente el pañuelo e insertarlo rápidamente en la parte inferior central de la agarosa. Incubar el molde en RT durante 15 minutos para permitir que la agarosa se polimerice.
- Después de la polimerización de la agarosa, abra el molde de plástico con un bisturí desde sus cuatro esquinas y recoja con cuidado el cubo de agarosa con los dedos enguantados.
- Sumerja la muestra en una solución de borohidruro (preparada en el paso 2) durante 2-3 h a 4 °C para iniciar la reticulación de la agarosa oxidada y el tejido. Esto evita que el tejido se desprenda durante el corte.
NOTA: Paso crítico: La muestra sumergida en borohidruro debe incubarse protegida de la luz a 4 °C. - Lave el cubo de agarosa con tampón PB 2 veces durante 10 min. Gire el cubo de agarosa de modo que el tejido quede en la parte superior y fije el bloque a un portaobjetos magnético (portaobjetos de microscopio con dos imanes unidos a la parte posterior) utilizando unas gotas de adhesivo instantáneo. El pegamento tarda entre 10 y 20 minutos en solidificarse, así que coloque un pañuelo de papel con gotas de tampón PB encima de la agarosa, asegurándose de que la muestra permanezca adecuadamente hidratada en todo momento (Figura 1C).
3. Preparación del micrótomo y montaje del escenario
- Coloque el portaobjetos magnético sobre el metal en el centro de la caja de muestras del tomógrafo.
- Llene la caja con tampón PB y colóquela con cuidado en la platina y apriete el tornillo de la izquierda para asegurar la caja de muestras en la platina del tomógrafo.
NOTA: No apriete demasiado los tornillos, ya que dañará la caja de plástico. - Coloque la cuchilla en el micrótomo y cambie la posición de la muestra con cuidado cerca y debajo del micrótomo. La superficie superior de la agarosa debe estar al mismo nivel que la hoja del micrótomo.
NOTA: Colocarlo demasiado bajo perderá tiempo de corte, mientras que colocarlo demasiado alto eliminará una gran parte del bloque en el primer corte. - Mueva la plataforma para colocar el cubo de agarosa en el centro del objetivo y haga clic repetidamente en el botón Slice en el software del tomógrafo hasta que obtenga una porción intacta de todo el cubo de agarosa.
4. Asignación de la superficie y adquisición de imágenes 2D
- Centre la lente del objetivo en la muestra de tejido en las direcciones x e y.
- Abra el software del láser y encienda el láser en el software. Al escuchar un tic-tac, encienda el interruptor láser y ajuste la longitud de onda a 800 nm.
NOTA: La fuente láser debe calentarse durante al menos 30 minutos, idealmente 1 hora, antes de la toma de imágenes. - Cierre las puertas del gabinete del microscopio, apague todas las fuentes de luz de la habitación, encienda los PMT y ajuste el voltaje a 750 V.
NOTA: Paso crítico: Los PMT son muy sensibles a la luz, manténgalos apagados a menos que las puertas del gabinete estén cerradas y todas las fuentes de luz de la habitación estén apagadas. - Ajuste la configuración del protocolo del software del tomógrafo como se describe y configure el obturador del microscopio en automático y el voltaje en 20 y 3 para V1 y V2, respectivamente.
- Haga clic en el botón 2D en el software del tomógrafo para capturar una instantánea de la muestra.
- Una vez que aparezca una imagen con autofluorescencia visible, ajuste el z-piezo para establecer el plano focal en la superficie del cubo de agarosa. El primer escaneo será a 20 μm bajo la superficie para adquirir una imagen estable de la muestra.
- Si no hay un fondo de autofluorescencia visible, aumente el contraste hasta que aparezca una señal. Puede ocurrir que no haya una señal visible (es decir, debido a la agarosa en la parte superior del tejido). En este caso, ajuste el eje z con el dispositivo piezoeléctrico z hasta que llegue a la superficie de agarosa. Es posible que sea necesario el movimiento manual de la lente si la posición deseada está fuera del alcance del dispositivo z-piezoeléctrico.
- Corte varios cortes de 50 μm después de encontrar la superficie de la muestra y adquiera imágenes 2D después de cada corte para asegurarse de que el plano focal esté configurado correctamente en la superficie de la cara del bloque.
5. Encontrar los bordes de las muestras y ajustar el láser al punto de partida
- En función de las coordenadas xy del área de escaneo láser (4 coordenadas de esquina), limite el área de imagen al tejido con una imagen mínima del bloque de agarosa circundante.
- Apague los PMT, ajuste la longitud de onda del láser a 800 nm, cambie el obturador láser para que se abra. La posición de la luz láser que ingresa al bloque de agarosa es visible como un punto rojo de luz dispersa (Figura 2A, B).
Paso crítico: Tenga especial cuidado con el láser de clase 4. Puede dañar rápidamente los tejidos humanos y los ojos. El personal necesita una capacitación especial para operar un instrumento de este tipo de manera segura. - Utilice el punto láser mientras desplaza la platina para encontrar las coordenadas de los bordes del tejido utilizando la secuencia atrás, derecha, delante e izquierda.
- Vuelva a verificar las coordenadas y coloque el punto láser en la esquina frontal derecha, que es el punto de inicio predeterminado para obtener imágenes del tejido.
- Convierta las coordenadas de la ventana de la consola en tamaños de bloque reconocibles por la máquina, que se definen mediante los pasos xy. Introduzca estas mediciones de pasos en el protocolo del software. Cierre las puertas del armario.
6. 3D escaneo / seccionamiento
- Cambie la longitud de onda a 940 nm para excitar GFP, mWasabi o YPet y PE.
NOTA: El láser es invisible para el ojo humano en esta longitud de onda. Para mejorar la relación señal-fondo para GFP, mWasabi y el componente emisor de verde de TIMERbac, coloque un filtro de paso de banda estrecho de 510/20 nm delante de PMT 2. Esto reduce la interferencia de la autofluorescencia tisular verde-amarilla. - Cambie el obturador del microscopio al modo automático y los ajustes de voltaje del obturador a 20 para V1 y 1,71 para V2.
- Ajuste los siguientes tres parámetros en el software: Espesor de la sección: para la mayoría de los propósitos, establézcalo en 50 μm. Para el hígado, se establece en 30 μm debido a la profundidad de imagen limitada. El número de secciones físicas: esto determina la profundidad total del tejido que se obstruirá en consecuencia, establecida en consecuencia. El número de planos a capturar para cada sección: esto determinará la resolución z. Utilice 5 planos (con una resolución vertical de 10 μm) para la mayoría de los propósitos. Para el hígado use 3 o menos planos.
- Ajuste la ganancia del láser. Esto debe probarse empíricamente para diferentes tejidos y profundidad de escaneo.
- Defina la ruta de acceso y el nombre de la carpeta para almacenar las imágenes adquiridas y proporcione la información adecuada para el metarchivo de imágenes.
- Vuelva a comprobar todos los valores introducidos.
- Haga clic en la configuración de mosaico 3D para iniciar el proceso de escaneo (Figura 1D).
- Una vez completadas las imágenes, recoja cuidadosamente las secciones de tejido del tanque de agua y guárdelas en un tampón de almacenamiento a -20 °C hasta su uso posterior (Figura 1E; Figura 2C,D).
7. Procesamiento de imágenes y análisis de datos
- Descargue los scripts de MATLAB en un equipo Linux desde https://github.com/BumannLab/Li_BumannLab_2020. Copie los scripts en una carpeta, por ejemplo, /home/user/Program/.
NOTA: El complemento Computer Vision Toolbox para MATLAB y Fiji (o ImageJ) debe estar instalado en equipos Linux para que el script sea completamente funcional. - Cose las imágenes de los mosaicos como se describe a continuación.
- Transfiera datos desde el servidor del tomógrafo a la computadora Linux.
- Abra el script de MATLAB StepOneStitchingAndArchive.m y localice la carpeta de origen que contiene los datos sin procesar. Defina las carpetas de origen y destino en el script.
- Cambie a la pestaña Editor y haga clic en Ejecutar. La información de progreso se muestra en la ventana de comandos. El procesamiento incluye la lectura de la información del archivo Mosaic, la generación del índice de mosaico antes de la unión de la imagen, la corrección de la iluminación de fondo con Cidre22 (Figura 3), así como la corrección de la iluminación entre capas ópticas adquiridas a diferentes profundidades. Busque las imágenes unidas en una subcarpeta llamada stitchedImages_100 en la carpeta de origen. Comprima los datos sin procesar para el almacenamiento a largo plazo utilizando el comando tar y guárdelos como un solo archivo con la extensión de nombre de archivo tar.bz2 (Figura 1F).
- Realice el entrenamiento y la segmentación del modelo con la máquina de vectores de soporte. Para entrenar la máquina de vectores de soporte y realizar la segmentación, siga los pasos que se indican a continuación.
NOTA: Las imágenes unidas para cada uno de los tres canales de fluorescencia se almacenan en 3 subcarpetas (denominadas 1, 2 y 3, correspondientes a la fluorescencia roja, naranja y verde) de stitchedImages_100. Cada subcarpeta contiene todas las imágenes unidas del mismo canal.- Obtenga una vista previa de las imágenes unidas. Abra una imagen con el mismo nombre de archivo de cada subcarpeta con Fiji (o ImageJ). Fusiona los tres canales en una imagen a color. Ajuste el brillo de cada canal hasta que se vean señales claras de las bacterias y la autofluorescencia de los tejidos. Tenga en cuenta el nivel de intensidad máxima ajustado de cada canal para el siguiente paso.
- Abra el script de MATLAB StepTwoSegmentationAndAnalysis.m y vaya al código fuente. Defina la carpeta de origen y los nombres de las imágenes para el entrenamiento. Por ejemplo
fuenteD = '/' ;
red_name = [fuenteD '1/section_020_01.tif']; green_name = [fuenteD '2/section_020_01.tif'];
blue_name = [fuenteD '3/section_020_01.tif']; - Vaya a la pestaña Editor y haga clic en Ejecutar.
- Aparecerá un cuadro de diálogo solicitando umbrales de color, rellénelo según la verificación manual previa con Fiji (paso 7.3.1).
- Seleccione las regiones para el fondo y las regiones de interés (es decir, bacterias) de acuerdo con los cuadros de diálogo. Aparecerá gráficos que muestran qué tan bien se ha entrenado el modelo y preguntarán si es necesario agregar más regiones. Agregue más regiones de interés y antecedentes hasta que las bacterias puedan segmentarse claramente (Figura 4A, B).
- Asigne un nombre al modelo y guárdelo. El script cargará automáticamente las imágenes y aplicará un filtro mediano (radio de 2 píxeles).
- El proceso de segmentación se ejecuta automáticamente y la información de progreso se muestra en la ventana de comandos. Observa el progreso de la segmentación. Si la segmentación no funciona bien con el modelo en otras imágenes unidas, repita el entrenamiento del modelo para incluir más regiones bacterianas y de fondo.
- Para ver los resultados de la segmentación, compruebe el archivo denominado Allpositions_filter3D.txt. Este fichero contiene las coordenadas del centro de gravedad de cada bacteria o microcolonia bacteriana y la correspondiente intensidad de fluorescencia integrada (Figura 1G).
- Elimine los artefactos adicionales con la red neuronal convolucional como se describe a continuación.
NOTA: Para algunas bacterias, la segmentación simple con la máquina de vectores de soporte todavía incluye algunos artefactos de imagen. Esto es particularmente cierto para las bacterias que expresan YPet. La segmentación basada en redes neuronales puede eliminar eficazmente estos artefactos (Figura 4C). El complemento Computer Vision Toolbox para MATLAB y Fiji (o ImageJ) debe estar instalado en equipos Windows para que el script sea completamente funcional. Para obtener información sobre la red neuronal subyacente, consulte: https://ch.mathworks.com/help/deeplearning/ug/create-simple-deep-learning-network-for-classification.html- Prepara las imágenes. Convierta archivos tif a archivos jpg con diferentes ajustes de brillo usando Fiji. Identifique con la curación manual al menos 600 bacterias y 600 artefactos y guarde imágenes recortadas de estos objetos en las carpetas adecuadas.
- Abra el script de MATLAB CNNtestV4.m y busque los archivos de origen que contienen las imágenes de objeto del paso 7.4.1.
- Defina la carpeta de imágenes para el entrenamiento. Por ejemplo
digitDatasetPath = archivo completo ('D:\20200602_CNN','fortrainingPixel12Folder2');
Defina el número de imágenes para el entrenamiento, por ejemplo, numTrainFiles = 600. - Vaya a la pestaña Editor y haga clic en Ejecutar. Después del entrenamiento, el archivo de modelo CNN se guarda como gregnet1.mat en la carpeta de origen.
- Defina una carpeta con imágenes de prueba. Por ejemplo
digitDatasetPathTest = fullfile('D:\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
Los resultados se guardan en un archivo YourFile.txt que contiene información sobre el nombre de la imagen y la clasificación como bacteria o artefacto. - Defina la carpeta con imágenes de prueba. Por ejemplo
digitDatasetPathTest = fullfile('D:\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
Los resultados se guardarán en un archivo YourFile.txt, que contiene información sobre el nombre de la imagen y la clasificación como bacteria o artefacto.
- Estime el tamaño de las microcolonias en función de la intensidad de fluorescencia de las bacterias individuales, como se describe a continuación.
NOTA: La resolución espacial del tomógrafo es insuficiente para resolver células bacterianas individuales dentro de microcolonias densamente empaquetadas. Sin embargo, el número de bacterias en cada microcolonia puede estimarse en función de la fluorescencia total de la microcolonia en relación con la fluorescencia de una sola bacteria. La precisión de esta estimación depende de la homogeneidad de la intensidad de la fluorescencia en toda la población bacteriana.- Confirmar la identidad de eventos positivos para GFP en la tomografía STP mediante la tinción de componentes bacterianos como el lipopolisacárido mediante inmunohistoquímica de las secciones recuperadas del tomógrafo (Figura 1H; Figura 5).
- Identifique al menos 30 bacterias individuales y recupere su intensidad de fluorescencia a partir de los resultados de segmentación correspondientes. Utilice el valor de la intensidad mediana como referencia para una sola bacteria. Calcule el número bacteriano de cada microcolonia dividiendo la intensidad de fluorescencia total de la microcolonia por el valor de referencia para una sola bacteria (Figura 1I).
- Realice la reconstrucción 3D con software de visualización como se describe a continuación.
- Preparación de la imagen
- Tome las imágenes del canal azul, que contienen señales de segundo armónico de colágeno que proporcionan una referencia anatómica útil que incluye cápsulas de tejido, arterias y trabéculas. Utilice imágenes segmentadas para bacterias como el2º canal.
- Agrupe las imágenes del 1ercanal 10 veces en los ejes x e y y guarde las imágenes reducidas en una nueva carpeta. Realice 3 réplicas de imágenes reducidas dentro de la misma carpeta. El nombre de las nuevas réplicas debe mantener la misma secuencia. Por ejemplo: section_001_01.tif, replicar 1 con el nombre section_001_01-copy.tif, section_001_01-copy-copy.tif. Aplique estos pasos también a las imágenes del2º canal. La reducción de tamaño produce tamaños de archivo más manejables. Las imágenes triples suavizarán el eje z.
- Combine las imágenes de cada canal para formar pilas de imágenes con Fiji. Haga clic en Imagen > Pilas > Herramientas > Clasificador de pilas > Ordenar por etiquetas.
- Realizar filtrado 3D. Haga clic en Procesar filtros > > filtro 3D y establezca el tamaño del filtro Z en 6. Guarde las pilas de imágenes.
- Realice la visualización 3D como se describe a continuación.
- Abra el paquete de software de visualización en la vista Arena (configuración predeterminada). Utilice el icono de la carpeta de inspección para añadir carpetas a la vista de Arena. Haga doble clic para abrir el archivo *.ims o *.tif (preparado en 7.6.1).
- Con Editar > Propiedades de imagen, ajuste las representaciones de color (LUT) de los diferentes canales en la ventana Ajuste de visualización.
- Haga clic en Avanzado para establecer manualmente los valores mínimos/máximos y un valor para la corrección gamma.
Haga clic en los nombres de los canales para cambiar los nombres y las LUT. - Después de ajustar la apariencia de la imagen, exporte la vista actual con la herramienta Instantánea.
- Utilice el icono Animación para representar los datos 3D como una película. Utilice el puntero de navegación para buscar una perspectiva/vista y zoom y utilice + Añadir para añadir fotogramas clave. Muévase a otra posición y agregue el siguiente fotograma clave. Presione el botón rojo de grabación para crear la película. Guárdelo en la carpeta de destino y el tipo de archivo deseados (Figura 1J).
- Preparación de la imagen
Resultados
El procedimiento descrito permite la detección de células individuales de Salmonella en órganos completos de ratón, como el bazo, el hígado, los ganglios linfáticos mesentéricos y los parches de Peyer11 (Figura 5 y Figura 6). También detecta parásitos Toxoplasma gondii en el cerebro de ratón12. Algunos tejidos infectados, como el hígado, las placas de Peyer y el bazo, emiten una autofluorescencia sustancial en el rango verde-amarillo. La autofluorescencia se ve reforzada por la fijación con paraformaldehído, que es necesaria para preservar la estructura del tejido. La detección de la fluorescencia verde de GFP, mWasabi y el componente verde de TIMERbac contra este fondo de autofluorescencia se mejora colocando un filtro de paso de banda estrecho de 510/20 nm (que transmite la mayoría de las emisiones de GFP pero bloquea una gran parte del espectro de autofluorescencia) frente al fotomultiplicador 2 (que recoge las emisiones verdes) y reduciendo la autofluorescencia tisular almacenando tejidos fijos durante 3 o más días en crioprotector11. Sin embargo, las bacterias aún deben expresar al menos unos pocos miles de copias por célula de GFP u otras proteínas fluorescentes. Por otro lado, se deben evitar los niveles excesivos de proteínas fluorescentes para minimizar los costos de aptitud física que podrían conducir a una virulencia atenuada23.
La segmentación correcta de las bacterias fluorescentes en los tejidos puede confirmarse mediante inmunohistoquímica de las secciones de tejido recuperadas después de la toma de imágenes. Específicamente, los objetos teñidos con un anticuerpo contra componentes bacterianos de la superficie, como el lipopolisacárido, se pueden alinear con la imagen de fluorescencia obtenida por tomografía STP (Figura 5). Es importante tener en cuenta que algunas células de Salmonella teñidas carecen de proteínas fluorescentes y, por lo tanto, de fluorescencia detectable tanto en la microscopía confocal como en la tomografía STP. Estas células son Salmonella que han sido eliminadas por el sistema inmune del huésped, como lo demuestra la clasificación por citometría de flujo y los cultivos de crecimiento de células individuales clasificadas, así como la estrecha correlación entre el número de unidades formadoras de colonias en placas de agar y el número de células fluorescentes de Salmonella según lo determinado por citometría de flujo24. Además, la pérdida de plásmidos puede dar lugar a células viables no fluorescentes y esto debe probarse mediante siembra en medios con y sin antibióticos apropiados correspondientes al marcador de selección en el plásmido. En el caso de los plásmidos derivados de pSC101, la pérdida de plásmidos in vivo es rara19. Para la mayoría de los casetes de expresión cromosómicamente integrados, como sifB::gfp utilizado en11, la pérdida de expresión es indetectable in vivo. Si la segmentación es incoherente con los datos de inmunohistoquímica, es necesario modificar la canalización de segmentación.
La resolución de la tomografía STP es insuficiente para resolver células bacterianas individuales dentro de microcolonias densamente empaquetadas. Sin embargo, la intensidad de fluorescencia total de la microcolonia permite estimar el número de células de Salmonella . Esto requiere una cepa fluorescente con niveles de fluorescencia altamente homogéneos, como Salmonella sifB::gfp11. La combinación del número estimado de células de Salmonella para todas las microcolonias y células individuales produce cargas totales de tejido bacteriano que son consistentes con métodos alternativos como la siembra o la citometría de flujo de homogeneizados de tejido11. La placa y la citometría de flujo no se pueden realizar directamente desde los mismos tejidos porque deben fijarse por perfusión para la tomografía STP. En cambio, tienen que hacerse con animales adicionales que no son fijos. Si la mediana de las cargas bacterianas determinadas por los distintos enfoques difiere en más de 3 veces, la viabilidad de las bacterias fluorescentes podría verse comprometida (en el caso de las unidades formadoras de colonias más bajas) o algunas bacterias podrían haber perdido la construcción del indicador fluorescente (en el caso de las unidades formadoras de colonias más altas). Se requerirá un experimento de control para identificar la fuente de tales discrepancias.
La tomografía STP proporciona la localización de células bacterianas dentro de la estructura 3D de los tejidos infectados. Las segundas señales armónicas del colágeno proporcionan puntos de referencia anatómicos como arterias y trabéculas. Además, las células huésped se pueden teñir in vivo inyectando un anticuerpo a los marcadores de superficie antes de la perfusión (Figura 5 y Figura 6). Esta tinción proporciona puntos de referencia adicionales para los compartimentos tisulares y microambientes específicos, incluidos los focos de inflamación (los marcadores intracelulares y algunos compartimentos con barreras de difusión, como la pulpa blanca esplénica o el cerebro, no son fácilmente accesibles, adecuados para esta tinción in vivo ). Este enfoque reveló que la pulpa blanca esplénica es un compartimento tisular que permite la supervivencia a largo plazo de Salmonella durante la quimioterapia antimicrobiana11.
Finalmente, la replicación de Salmonella a tasas moderadas o lentas puede ser identificada y localizada dentro de la estructura 3D de los tejidos utilizando cepas que expresan un temporizadorbac, un reportero de una sola célula para la tasa de replicación11,15.
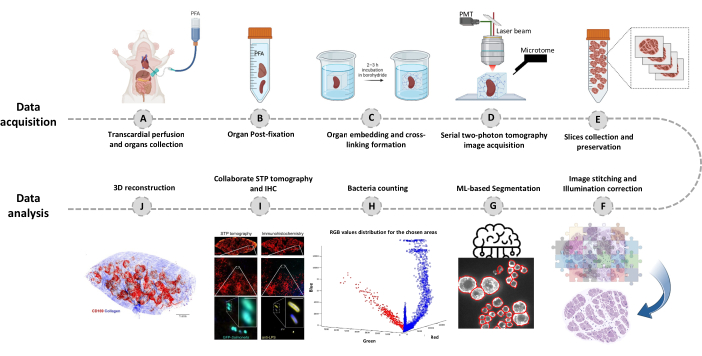
Figura 1: Procedimiento para la obtención de imágenes de todo el órgano mediante tomografía seriada de dos fotones (STP). (A-B) El órgano se extrae después de la perfusión transcardíaca y se almacena durante la noche en paraformaldehído al 4% (PFA) a 4 °C. (C-E) El órgano se incrusta en agarosa oxidada y se reticula, luego el tejido se escanea y se corta mediante tomografía STP. Los cortes se recogen para su posterior inmunohistoquímica. (De Padres a Viernes) Canales de análisis computacional para cuantificar el número de bacterias, confirmar bacterias fluorescentes y reconstrucción 3D de posiciones bacterianas. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Configuración de la tomografía seriada de dos fotones (STP). (A) Tomógrafo que incorpora (B) imágenes de 2 fotones con (C) corte de tejido en serie automatizado. (D) Cortes de tejido recolectados para investigaciones de seguimiento. Haga clic aquí para ver una versión más grande de esta figura.
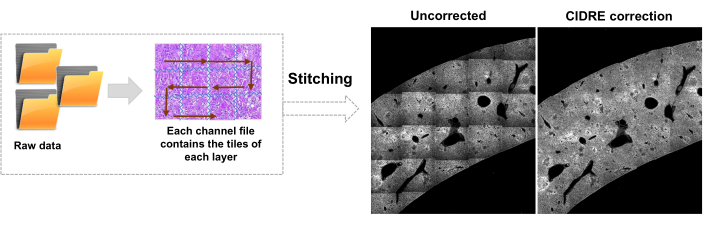
Figura 3: Costura de azulejos y corrección de iluminación. Las baldosas se cosen y la iluminación desigual se corrige mediante distribuciones de intensidad corregidas mediante minimización de energía regularizada (CIDRE). Haga clic aquí para ver una versión más grande de esta figura.
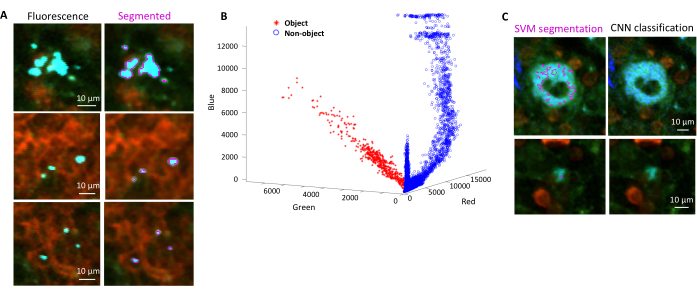
Figura 4: Segmentación de Salmonella que expresa la proteína verde fluorescente (GFP) utilizando una máquina de vectores de soporte (SVM) y una red neuronal convolucional (CNN). (A) Imágenes representativas de objetos GFP segmentados por SVM (izquierda) e imágenes correspondientes (derecha) con regiones segmentadas por SVM (barra de escala: 10 μm). (B) Distribución de valores agrupados rojo-verde-azul (RGB) para las regiones segmentadas. (C) Imágenes representativas de objetos no GFP identificados falsamente por la SVM como bacterias (izquierda). CNN los descarta correctamente como fondo (derecha, barra de escala: 10 μm). Haga clic aquí para ver una versión más grande de esta figura.
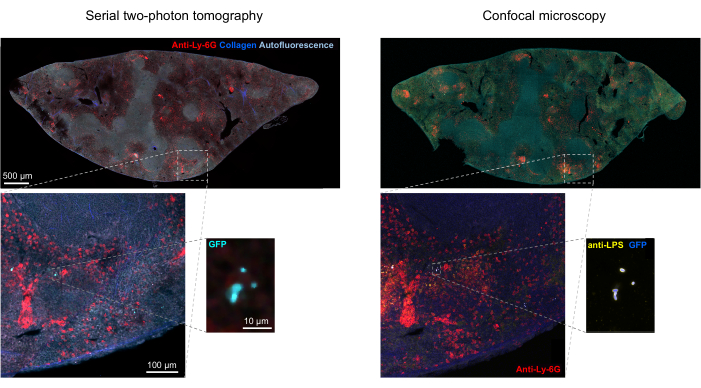
Figura 5: Detección de Salmonella expresando la proteína verde fluorescente (GFP) por tomografía y confirmación por inmunohistoquímica. Imágenes de la misma sección adquiridas por tomografía (izquierda) o microscopía confocal después de la tinción con un anticuerpo contra el lipopolisacárido de Salmonella (derecha). Los neutrófilos (rojos) se tiñeron mediante la inyección in vivo de un anticuerpo anti-Ly-6G marcado con PE antes de la perfusión. Haga clic aquí para ver una versión más grande de esta figura.
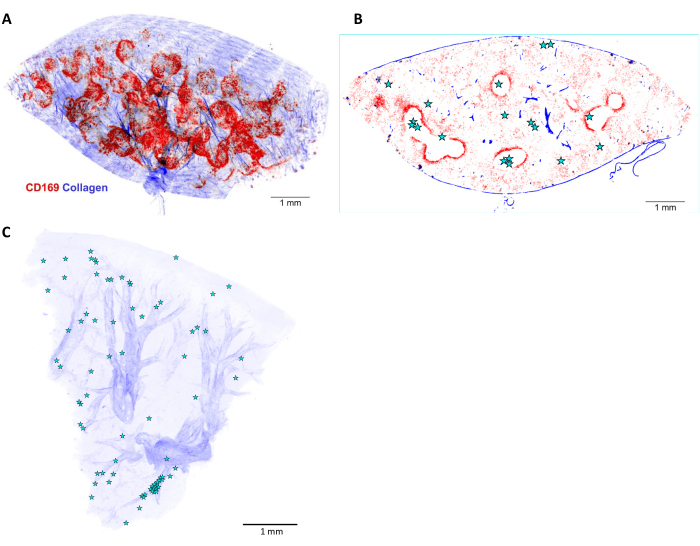
Figura 6: Reconstrucción y localización 3D de Salmonella en bazo de ratón infectado. (A) Reconstrucción tridimensional (3D) de un corte de bazo de 5 mm de espesor y teñido in vivo antes de la perfusión con anticuerpo anti-CD169 (rojo). La señal azul representa el colágeno detectado por segundos armónicos. (B) Un plano óptico de la pila 3D que se muestra en (A). Las posiciones de las células o microcolonias de Salmonella están indicadas por estrellas. (C) Reconstrucción 3D de las posiciones (estrellas) de Salmonella en el hígado infectado. Las arterias son visibles en función de sus vainas de colágeno (azul). Barra de escala: 1 mm. Haga clic aquí para ver una versión más grande de esta figura.
Discusión
El contexto tisular local de los patógenos bacterianos es crucial para determinar los ataques locales del huésped, las adaptaciones bacterianas, el resultado local de las interacciones entre el huésped y el patógeno y la quimioterapia antimicrobiana, y las contribuciones individuales al resultado general de la enfermedad. Obtener imágenes de bacterias del tamaño de un micrómetro en órganos de centímetros ha sido un desafío. La tomografía seriada de dos fotones (STP) proporciona una resolución espacial suficiente para detectar células bacterianas individuales en órganos completos, secciones e imágenes automatizadas, y un rendimiento suficiente (~1 órgano por día)11. Si bien los antígenos del huésped se pueden teñir in vivo, las células patógenas deben expresar proteínas fluorescentes adecuadas para garantizar una detección completa de las células patógenas intracelulares. Los conjuntos de datos resultantes (0,5-1,5 TeraByte por órgano) plantean desafíos sustanciales para las infraestructuras de TI para el análisis y el almacenamiento de datos.
Hay varios pasos críticos en este método. En primer lugar, se requiere una cepa patógena con expresión detectable y homogénea de la proteína fluorescente GFP o YFP. Idealmente, se utiliza un casete de expresión cromosómica25 para minimizar la heterogeneidad de fluorescencia debido a la variación del número de copias del plásmido. Se requiere una intensidad de fluorescencia suficiente, pero deben evitarse niveles excesivos de proteína fluorescente para evitar alteraciones de la aptitud del patógeno23. Los niveles de expresión apropiados se pueden obtener mediante la selección de un promotor apropiado y el ajuste fino del sitio de unión ribosómico25 o de toda la región no traducida 5' (UTR)26. En segundo lugar, la fijación de la perfusión debe implicar un lavado inicial con tampón para eliminar la mayor cantidad posible de eritrocitos de la circulación sanguínea. Esto es particularmente crítico para el bazo y el hígado (aunque la eliminación completa de eritrocitos de estos órganos es difícil). Los eritrocitos restantes absorben la luz en la parte visible del espectro, comprometiendo la calidad de la imagen27. En tercer lugar, el almacenamiento de los tejidos fijados en el crioprotector es fundamental para reducir la autofluorescencia tisular, que es particularmente alta en los tejidos inflamados y puede eclipsar la fluorescencia comparativamente débil de las células patógenas11. En cuarto lugar, la reticulación efectiva del tejido con el bloque de agarosa circundante es fundamental para un corte suave del vibratomo sin que el tejido salte fuera del bloque de agarosa. En quinto lugar, las señales fluorescentes y su identificación como células patógenas deben verificarse de forma independiente utilizando enfoques ortogonales como la tinción con anticuerpos contra componentes patógenos (como el lipopolisacárido para bacterias gramnegativas) y la microscopía confocal de las secciones extraídas del tomógrafo11. Algunos tejidos infectados contienen partículas autofluorescentes con forma similar y espectros de fluorescencia superpuestos que pueden malinterpretarse fácilmente como células patógenas. En sexto lugar, la cantidad de células patógenas dentro de las microcolonias debe compararse con enfoques ortogonales como la microscopía confocal para evaluar la precisión. Las cargas bacterianas generales basadas en estos cálculos deben verificarse mediante la comparación con enfoques ortogonales como la citometría de flujo y la siembra.
Modificaciones importantes del protocolo STP ampliamente utilizado incluyen la colocación de un filtro de paso de banda estrecho de 510/20 nm frente al fotomultiplicador 211, para reducir la interferencia de la autofluorescencia verde-amarilla que es particularmente fuerte en el hígado infectado e inflamado, el bazo y los parches de Peyer. La fuerte autofluorescencia y el aumento de la dispersión de la luz de dichos órganos en comparación con el cerebro (que domina otras aplicaciones de STP) también genera la necesidad de una corrección más efectiva para la iluminación desigual. Como otra modificación, este protocolo emplea el enfoque CIDRE22 para este propósito (Figura 3) y la segmentación de bacterias basada en IA. Finalmente, el preprocesamiento tisular se modificó al incluir una etapa de incubación en crioprotector a -20 °C que reduce la autofluorescencia tisular y, por lo tanto, facilita la detección de células patógenas pequeñas con fluorescencia relativamente débil11.
La solución de problemas puede ser necesaria si no se pueden detectar señales de patógenos, o si la segmentación produce una sensibilidad insuficiente (se pasan por alto demasiadas células patógenas) o una precisión insuficiente (se segmentan demasiadas partículas de fondo como células patógenas). Si la autofluorescencia del tejido de fondo es detectable pero hay muy pocas señales de patógenos, es posible que los patógenos contengan cantidades insuficientes de proteínas fluorescentes. Esto puede ser probado usando microscopía confocal de secciones de tejido del mismo tejido infectado o citometría de flujo de homogeneizados de tejido19,28. Las razones subyacentes podrían ser niveles de expresión insuficientes o inestabilidad del casete de expresión. Las estrategias de mitigación podrían incluir promotores alternativos para impulsar la expresión, la adaptación al codón de los genes que codifican la proteína fluorescente para la especie patógena, el empleo de construcciones episomales con mayor número de copias o la estabilización de casetes de expresión mediante la integración cromosómica o la complementación letal equilibrada29. La elección de la proteína fluorescente también es importante, pero la detección es posible con GFP.mut2, mWasabi, YPet y TIMERbac. Si la segmentación es inexacta, esto podría deberse a una fluorescencia del patógeno demasiado débil que podría abordarse como se describió anteriormente, o a un fondo de autofluorescencia tisular demasiado alto. La perfusión extensiva de la solución de lavado o la incubación prolongada en el tampón de almacenamiento inmediatamente antes de la inclusión en el bloque de agarosa y la tomografía podrían resolver estos problemas. Por último, se requiere un entrenamiento suficiente de la red neuronal para una clasificación precisa, pero un entrenamiento excesivo puede conducir a un sobreajuste que perjudique el rendimiento de las nuevas muestras.
En la actualidad, ningún otro método puede obtener imágenes de órganos enteros con una resolución espacial suficiente en 3D para detectar bacterias individuales. Las mejoras futuras en la limpieza de tejidos y la microscopía de lámina ligera podrían lograr una resolución similar. Esto podría permitir la obtención de imágenes a mayor velocidad y con más canales fluorescentes.
Una limitación importante de STP es la resolución de píxeles en el plano de ~0,5 μm y la resolución vertical de 5 a 10 μm, que es insuficiente para resolver bacterias muy cercanas, por ejemplo, dentro de una microcolonia densamente empaquetada. Sin embargo, es posible recuperar secciones de tejido después de la tomografía para la microscopía confocal secundaria de alta resolución de partes de tejido seleccionadas. Otra limitación de STP es la disponibilidad de solo tres canales de fluorescencia, lo que restringe el número de fluoróforos que se pueden obtener imágenes simultáneamente. Una vez más, el análisis secundario de las secciones de tejido recuperadas con métodos de multiplexación puede revelar la ubicación y la intensidad de muchos más marcadores para partes de tejido seleccionadas. Esta información podría integrarse en la estructura 3D general del tejido circundante según lo determinado con STP.
En conclusión, este protocolo permite investigaciones detalladas de las interacciones huésped-patógeno a nivel local y de todo el órgano. El protocolo debe ser fácilmente adaptable a otros patógenos (siempre que se puedan obtener como cepas fluorescentes), otros órganos y diferentes especies huésped.
Divulgaciones
Los autores no tienen nada que revelar.
Agradecimientos
El trabajo contó con el apoyo de la Fundación Nacional Suiza para la Ciencia 310030_156818, 310030_182315 y NCCR_ 180541 AntiResist (a DB).
Materiales
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agarose Low Melt | Roth | Art. 6351.5 25g | |
| Boric acid | Sigma-Aldrich | 6768-500G | |
| Instant adhesive Loctite 435 | Henkel | ||
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Poly(ethylene glycol) | Sigma-Aldrich | P5413-1kg | |
| Polyvinylpyrrolidone | Sigma-Aldrich | PVP-100G | |
| Sodium borohydride | Sigma-Aldrich | 71321-25g | |
| Sodium hydroxide | Merck | 106453 | |
| Sodium periodate | Sigma-Aldrich | 311448-100G | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640-250G | |
| Sodium phosphate monobasic dihydrate | Sigma-Aldrich | 71500-1KG | |
| Sodium tetraborate | Sigma-Aldrich | 221732-100g | |
| Sucrose | AppliChem | A4734,1000 | |
| Tris-buffered saline (TBS) | Merck | T5912-1L | |
| Triton X-100 | Sigma-Aldrich | 9002-93-1 | |
| Vacuum filtration 500 | TPP | TPP99250 | |
| Equipment | |||
| Blade | Campden Instruments Limited | 01-01-4692 | |
| MAITAI Laser | Spectra-Physics | ||
| Peel away plastic mold | Sigma-Aldrich | E6032-1CS | |
| TissueCyte 1000 tomograph | TissueVision | ||
| Antibody/dyes | |||
| DAPI | Merck | D9542-5MG | |
| Primary antibodies | |||
| anti-LPS Salmonella, rabbit | Sifin | REF TS 1624 | |
| anti-CD169-PE, clone 3D6.112 | Biolegend | 142403 | |
| anti-Ly-6G-PE, clone 1A8 | Biolegend | 127608 | |
| Secondary antibodies | Invitrogen | ||
| chicken anti-rabbit Alexa 647 | Invitrogen | A-21443 | |
| Software | Company | Version | |
| Fiji | Image J | 1.54g or later | |
| MATLAB | MathWorks | 2017b/2018b or later | |
| Orchestrator (tomograph) | TissueVision | ||
| Visualization software Imaris | Oxford Instruments | 9.9.0 or later |
Referencias
- Bjarnsholt, T., et al. The importance of understanding the infectious microenvironment. Lancet Infect Dis. 22 (3), e88-e92 (2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Bumann, D., Cunrath, O. Heterogeneity of Salmonella-host interactions in infected host tissues. Curr Opin Microbiol. 39, 57-63 (2017).
- Hofmann, J., Keppler, S. J. Tissue clearing and 3D imaging - putting immune cells into context. J Cell Sci. 134 (15), jcs258494 (2021).
- Blain, R., et al. A tridimensional atlas of the developing human head. Cell. 186 (26), 5910-5924.e17 (2023).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Meth. 9 (3), 255-258 (2012).
- Oh, S. W., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Kim, Y., et al. Brain-wide maps reveal stereotyped cell-type-based cortical architecture and subcortical sexual dimorphism. Cell. 171 (2), 456-469.e422 (2017).
- Matho, K. S., et al. Genetic dissection of the glutamatergic neuron system in cerebral cortex. Nature. 598 (7879), 182-187 (2021).
- Muñoz-Castañeda, R., et al. Cellular anatomy of the mouse primary motor cortex. Nature. 598 (7879), 159-166 (2021).
- Li, J., et al. Tissue compartmentalization enables Salmonella persistence during chemotherapy. Proc Natl Acad Sci U S A. 118 (51), e2113951118 (2021).
- Dogga, S. K., et al. Importance of aspartyl protease 5 in the establishment of the intracellular niche during acute and chronic infection of Toxoplasma gondii. Mol Microbiol. 118 (6), 601-622 (2022).
- Cormack, B. P., Valdivia, R. H., Falkow, S. FACS-optimized mutants of the green fluorescent protein (GFP). Gene. 173 (1 Spec No), 33-38 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Claudi, B., et al. Phenotypic variation of Salmonella in host tissues delays eradication by antimicrobial chemotherapy. Cell. 158 (4), 722-733 (2014).
- Ai, H. W., Olenych, S. G., Wong, P., Davidson, M. W., Campbell, R. E. Hue-shifted monomeric variants of Clavularia cyan fluorescent protein: identification of the molecular determinants of color and applications in fluorescence imaging. BMC Biol. 6, 13 (2008).
- Drobizhev, M., Makarov, N. S., Tillo, S. E., Hughes, T. E., Rebane, A. Two-photon absorption properties of fluorescent proteins. Nat Methods. 8 (5), 393-399 (2011).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Burton, N. A., et al. Disparate impact of oxidative host defenses determines the fate of Salmonella during systemic infection in mice. Cell Host&Microbe. 15 (1), 72-83 (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio Protoc. 11 (5), e3988 (2021).
- de Olmos, J., Hardy, H., Heimer, L. The afferent connections of the main and the accessory olfactory bulb formations in the rat: an experimental HRP-study. J Comp Neurol. 181 (2), 213-244 (1978).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nat Methods. 12 (5), 404-406 (2015).
- Wendland, M., Bumann, D. Optimization of GFP levels for analyzing Salmonella gene expression during an infection. FEBS Lett. 521 (1-3), 105-108 (2002).
- Barat, S., et al. Immunity to intracellular Salmonella depends on surface-associated antigens. PLoS Pathog. 8 (10), e1002966 (2012).
- Rollenhagen, C., Sorensen, M., Rizos, K., Hurvitz, R., Bumann, D. Antigen selection based on expression levels during infection facilitates vaccine development for an intracellular pathogen. Proc Natl Acad Sci U S A. 101 (23), 8739-8744 (2004).
- Chen, F., Cocaign-Bousquet, M., Girbal, L., Nouaille, S. 5'UTR sequences influence protein levels in Escherichia coli by regulating translation initiation and mRNA stability. Front Microbiol. 13, 1088941 (2022).
- Weissleder, R. A clearer vision for in vivo imaging. Nat Biotechnol. 19 (4), 316-317 (2001).
- Bumann, D. Examination of Salmonella gene expression in an infected mammalian host using the green fluorescent protein and two-colour flow cytometry. Mol.Microbiol. 43 (5), 1269-1283 (2002).
- Nakayama, K., Kelly, S. M., Curtiss III, R. J. B. t. Construction of an Asd+ expression-cloning vector: stable maintenance and high level expression of cloned genes in a Salmonella. vaccine strain. 6 (6), 693-697 (1988).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados