Method Article
Tomographie tridimensionnelle à haute résolution des infections microbiennes
Dans cet article
Résumé
Nous décrivons ici une procédure qui permet de détecter les bactéries pathogènes à l’échelle de l’organe pendant l’infection et de quantifier les activités des rapporteurs fluorescents.
Résumé
La plupart des infections se produisent dans les tissus tridimensionnels de l’hôte avec une anatomie complexe et une physiologie de l’hôte variant localement. Le positionnement des cellules pathogènes dans cet environnement diversifié affecte considérablement leur niveau de stress, leurs réponses, leur destin et leur contribution à la progression globale de la maladie et à l’échec du traitement. Cependant, en raison des difficultés techniques rencontrées pour localiser des cellules pathogènes de taille μm dans des organes hôtes de taille cm, ce domaine de recherche a été relativement inexploré. Nous présentons ici une méthode pour relever ce défi. Nous utilisons la tomographie à deux photons en série et l’analyse d’image améliorée par l’IA pour localiser les cellules individuelles de Salmonella dans l’ensemble de la rate, des lobes hépatiques et des ganglions lymphatiques entiers des souris infectées. À l’aide de rapporteurs fluorescents et de l’administration d’anticorps in vivo , il est possible de déterminer le taux de réplication des cellules uniques de Salmonella , leur interaction locale avec des cellules immunitaires spécifiques et les réponses bactériennes aux antibiotiques. Ces méthodologies ouvrent la voie à un examen complet des infections, de leur prévention et de leur traitement dans le contexte tissulaire tridimensionnel.
Introduction
Les infections se produisent dans des tissus à l’anatomie complexe et à la physiologie compartimentée. Les divers microenvironnements qui coexistent dans les tissus infectés peuvent déterminer le destin des sous-ensembles locaux d’agents pathogènes et leurs contributions à l’issue globale de la maladie 1,2,3. Cependant, la cartographie 3D complète des agents pathogènes microbiens dans des tissus de taille centimétrique reste difficile4. L’imagerie du cerveau et d’autres organes est un domaine de recherche très actif avec des stratégies expérimentales en constante amélioration5, mais de nombreuses méthodes n’ont toujours pas la résolution inférieure au μm qui serait nécessaire pour identifier avec certitude des agents pathogènes bactériens de taille μm. En revanche, la tomographie série à deux photons (STP)6 permet une imagerie multicolore automatisée et sans déformation de tissus entiers avec une résolution dans le plan inférieure à μm, ce qui permet d’obtenir des ensembles de données volumétriques complets. Cette méthode combine une section physique répétée du tissu à l’aide d’un vibratome avec une imagerie intermittente à deux photons des faces émergentes des blocs émergents avec une lumière infrarouge. La tomographie STP a été largement utilisée pour cartographier les axones minces dans le cerveau afin d’établir des cartes de connectivité 7,8,9,10.
La tomographie STP permet également de cartographier en 3D des cellules pathogènes microbiennes individuelles (Salmonella, Toxoplasma) dans l’ensemble des tissus infectés11,12 à l’aide d’un tomographe. La génération de seconde harmonique révèle des gaines de collagène autour des artères et dans des bandes fibreuses telles que les trabécules de la rate, fournissant ainsi un contexte anatomique. Des anticorps fluorescents injectés in vivo peuvent être utilisés pour colorer les cellules hôtes afin de révéler les interactions entre les cellules pathogènes individuelles et les cellules immunitaires infiltrantes telles que les neutrophiles. Ici, le pipeline impliquant le traitement du tissu, l’imagerie, l’assemblage de tuiles d’imagerie avec correction de l’éclairage, l’empilement d’images en trois dimensions et la segmentation à l’aide d’outils d’apprentissage automatique est décrit. Ce pipeline produit des positions 3D d’agents pathogènes individuels, de cellules et de microcolonies dans leur contexte hôte. Le comptage du nombre de cellules individuelles au sein des microcolonies reste difficile en raison des limites de résolution, mais ce nombre peut être estimé sur la base de la luminosité intégrée de la microcolonie. Le pipeline peut être facilement adapté à d’autres modèles d’infection si des agents pathogènes recombinants exprimant la GFP ou le YFP sont disponibles.
Protocole
Toutes les expériences sur les animaux décrites ici ont été approuvées par les autorités (licence 2239, Kantonales Veterinäramt Basel) et suivent les directives locales (Tierschutz-Verordnung, Bâle) et la loi suisse sur la protection des animaux (Tierschutz-Gesetz).
1. Préparation et conservation des tissus infectés
- Utilisez le modèle d’infection de votre choix. Ici, des souris âgées de 10 à 16 semaines des deux sexes sont infectées avec ~1 000 unités formant colonies (UFC) par injection intraveineuse ou 5 x 107 UFC par administration intragastrique avec une aiguille à bout rond. Les souches de souris appropriées comprennent BALB/c et C57BL/6.
REMARQUE : La même approche peut être facilement adaptée à d’autres espèces hôtes. - S’assurer que les agents pathogènes expriment des protéines fluorescentes pour permettre l’identification dans les tissus non colorés. Les agents pathogènes exprimant GFP.mut213, YPet14, TIMERbac15 ou mWasabi16 peuvent tous être visualisés à l’aide d’une excitation de 940 nm17. Les salmonelles exprimant mCherry18 à des niveaux facilement détectables par cytométrie en flux19 ont pu être imagées à l’aide d’une excitation de 800 nm, mais ont de mauvais rapports signal/bruit de fond dans la rate et le foie infectés, qui montrent une autofluorescence substantielle.
- S’assurer que toutes les protéines fluorescentes conservent >70 % de leur intensité de fluorescence après fixation avec du paraformaldéhyde à pH neutre sur la base de la quantification par cytométrie en flux. Ici, des tissus de souris infectés, à savoir la rate, le foie, les ganglions lymphatiques et les patchs de Peyer, sont utilisés.
- Colorer les marqueurs de surface de la cellule hôte in vivo en injectant des anticorps marqués à la phycoérythrine (PE) 10 min avant la perfusion. Pour colorer les neutrophiles, injecter par voie intraveineuse 4 μg d’anti-Ly-6G-PE dans 100 μL de PBS. Pour colorer les macrophages de la zone marginale de la rate, injecter 4 μg d’anti-CD169-PE dans 100 μL de PBS (voir le tableau des matériaux).
- Fixer les tissus infectés par perfusion transcardique standard20 avec 15 mL de tampon phosphate glacé de 50 mM (PB ; 10 mM NaH2PO4, 40 mMNa2HPO4, pH 7,4), suivi de 35 mL de paraformaldéhyde à 4 % (PFA ; fixateur) dans du PB (figure 1A).
- En bref, administrer une anesthésie profonde à l’animal infecté par injection intrapéritonéale de 100 mg/kg de kétamine et de 16 mg/kg de xylazine. Assurez-vous d’une anesthésie appropriée en confirmant la perte du réflexe de pincement des orteils.
- Désinfectez la fourrure avec de l’éthanol à 70 %, séchez-la avec une serviette en papier stérile et ouvrez chirurgicalement la poitrine avec une pince à épiler et des ciseaux. Placez une aiguille stérile dans le ventricule gauche du cœur, ouvrez l’oreillette droite avec des ciseaux stériles, puis faites passer le premier tampon, puis le fixateur à travers l’aiguille, le ventricule gauche et l’ensemble du système vasculaire à l’aide d’une pompe. Cela fixe tous les tissus rapidement et uniformément et provoque l’euthanasie.
- Prélever le tissu après la perfusion, l’incuber dans un volume de 20 fois de paraformaldéhyde froid à 4 % dans un tampon PB et le placer sur un agitateur à 4 °C pendant la nuit (figure 1B). L’imagerie est possible pour les tissus de 0,05 à 2 cm3. Pour les tissus plus gros, coupez-les en morceaux appropriés.
- Le lendemain, lavez le mouchoir 3 fois pendant 10 minutes chacun avec un tampon PB en secouant à 40 tr/min à température ambiante pour éliminer le PFA.
REMARQUE : En raison de la toxicité PFA, ces étapes doivent être effectuées à l’intérieur d’une hotte. Éliminez les déchets PFA conformément aux directives institutionnelles. - Retirer tout excès de tampon PB et ajouter 20 fois le volume de tampon de stockage cryoprotecteur21 (876 mM de saccharose, 0,25 mM de polyvinylpyrrolidone, 40 % (v/v) d’éthylène glycol, dissous dans un tampon de PB de 50 mM) à l’échantillon en agitant à 40 tr/min à 4 °C pendant 6 à 8 h ou jusqu’à ce que les tissus coulent au fond. Incuber les échantillons à -20 °C pendant au moins 3 jours pour assurer une réduction efficace de l’autofluorescence tissulaire11. Les échantillons peuvent être utilisés jusqu’à 5 ans s’ils sont conservés à -20 °C.
REMARQUE : L’enfoncement du tissu indique une bonne pénétration du tampon de stockage dans le tissu.
2. Intégration de l’échantillon
- Incorporez le tissu dans un bloc d’agarose pour une coupe en douceur avec un vibratome. Pré-activer chimiquement l’agarose par oxydation avec du periodate, puis réduire avec du borohydrure pour former une réticulation avec le tissu pour une meilleure stabilité pendant le processus de coupe. Effectuez les étapes décrites ci-dessous.
- Préparez de l’agarose oxydée à 4 % en pesant 2,25 g d’agarose et ajoutez 0,21 g de NaIO4. Ajouter 100 mL de PB à l’agarose et au NaIO4. Remuer à température ambiante dans une hotte pendant 2-3 h à l’abri de la lumière.
REMARQUE : Étape critique : Ne pas dépasser l’agitation pendant plus de 3 h ou l’agarose polymérisera mal. - Récupérer l’agarose par filtration membranaire dans un filtre à vide standard avec une membrane de 0,2 μm. Retirez le NaIO4 restant en lavant 3 fois avec 50 ml de tampon PB. Remettre l’agarose oxydée en suspension dans 50 mL de tampon PB.
REMARQUE : Ne faites pas fondre l’agarose pendant cette étape. L’agarose peut être conservée à 4 °C jusqu’à 2-3 semaines à l’abri de la lumière. - Préparez une solution de borohydrure en ajoutant 19 g de borax et 3 g d’acide borique à 1 L d’eau. Remuer jusqu’à dissolution et ajuster le pH à 9-9,5 avec 1 M de NaOH. Sur une plaque agitatrice dans une hotte, chauffer 100 mL de tampon à borate à 40 °C. Ajouter 0,2 g de NaBH4 et remuer pendant 15-30 min à l’abri de la lumière.
REMARQUE : La solution de borohydrure peut être conservée pendant plusieurs semaines à température ambiante dans l’obscurité. Étape critique : Lors de l’ajout de NaBH4 au tampon de borate, du gaz se forme, effectuez donc cette étape à l’intérieur d’une hotte. Ne faites pas cette étape dans un récipient hermétiquement fermé car cela pourrait entraîner une explosion. - Sortez les tissus du congélateur après une incubation suffisante pour réduire l’autofluorescence (≥ 3 jours), retirez l’excès de tampon de stockage cryoprotecteur et lavez les tissus 3 fois pendant 10 minutes chacun avec un tampon PB en agitant à 40 tr/min à température ambiante (RT).
- Faites fondre environ 20 ml de suspension d’agarose oxydée (préparée à l’étape 1) au micro-ondes (700 W pendant ~30 s), laissez-le refroidir suffisamment pour être manipulé (~45 °C) et versez-le dans un moule en plastique.
REMARQUE : Étape critique : N’ajoutez pas l’agarose directement après l’avoir chauffé au micro-ondes sur l’échantillon, cela pourrait entraîner la destruction du tissu. - À l’aide d’une pince à épiler, prélevez doucement le tissu et insérez-le rapidement dans le centre inférieur de l’agarose. Incuber le moule à RT pendant 15 min pour permettre à l’agarose de polymériser.
- Après la polymérisation de l’agarose, ouvrez le moule en plastique avec un scalpel par ses quatre coins et ramassez soigneusement le cube d’agarose avec des doigts gantés.
- Immerger l’échantillon dans une solution de borohydrure (préparée à l’étape 2) pendant 2 à 3 h à 4 °C pour initier la réticulation de l’agarose oxydée et du tissu. Cela empêche le tissu de se détacher lors de la section.
REMARQUE : Étape critique : L’échantillon immergé dans le borohydrure doit être incubé à l’abri de la lumière à 4 °C. - Lavez le cube d’agarose avec un tampon PB 2x pendant 10 min. Tournez le cube d’agarose de manière à ce que le tissu soit sur la face supérieure et fixez le bloc à une lame magnétique (lame de microscope avec deux aimants fixés à l’arrière) à l’aide de quelques gouttes d’adhésif instantané. Il faut 10 à 20 minutes pour que la colle se solidifie, alors placez un mouchoir en papier avec des gouttes de tampon PB sur l’agarose en veillant à ce que l’échantillon reste suffisamment hydraté tout au long de l’album (Figure 1C).
3. Préparation du microtome et mise en scène
- Placez la lame magnétique sur le métal au centre de la boîte d’échantillons du tomomètre.
- Remplissez la boîte avec du tampon PB et positionnez-la soigneusement sur la platine et serrez la vis sur la gauche pour fixer la boîte d’échantillons sur la platine du tomographe.
REMARQUE : Ne serrez pas trop les vis, sinon cela endommagerait la boîte en plastique. - Placez la lame sur le microtome et déplacez soigneusement la position de l’échantillon près et en dessous du microtome. La surface supérieure de l’agarose doit être au même niveau que la lame du microtome.
REMARQUE : Le positionner trop bas fera perdre du temps de coupe, tandis que le positionner trop haut enlèvera une grande partie du bloc lors de la première coupe. - Déplacez la platine pour positionner le cube d’agarose au centre de l’objectif et cliquez à plusieurs reprises sur le bouton Slice dans le logiciel de tomographie jusqu’à ce que vous obteniez une tranche intacte du cube d’agarose entier.
4. Attribution de la surface et acquisition d’images 2D
- Centrez la lentille de l’objectif sur l’échantillon de tissu dans les directions x et y.
- Ouvrez le logiciel laser et allumez le laser dans le logiciel. Lorsque vous entendez un tic-tac, allumez l’interrupteur laser et ajustez la longueur d’onde à 800 nm.
REMARQUE : La source laser doit être chauffée pendant au moins 30 min, idéalement 1 h, avant l’imagerie. - Fermez les portes de l’armoire du microscope, éteignez toutes les sources lumineuses de la pièce, allumez les PMT et réglez la tension sur 750 V.
REMARQUE : Étape critique : Les PMT sont très sensibles à la lumière, gardez-les éteints à moins que les portes de l’armoire ne soient fermées et que toutes les sources de lumière de la pièce soient éteintes. - Ajustez les paramètres de protocole du logiciel du tomographe comme indiqué et réglez l’obturateur du microscope sur automatique et la tension sur 20 et 3 pour V1 et V2, respectivement.
- Cliquez sur le bouton 2D dans le logiciel de tomographie pour capturer une capture de l’échantillon.
- Une fois qu’une image avec une autofluorescence visible apparaît, ajustez le piézo z pour définir le plan focal sur la surface du cube d’agarose. Le premier balayage se fera à 20 μm sous la surface pour acquérir une image stable de l’échantillon.
- Si aucun arrière-plan d’autofluorescence n’est visible, augmentez le contraste jusqu’à ce qu’un signal apparaisse. Il peut arriver qu’il n’y ait pas de signal visible (c’est-à-dire à cause de l’agarose sur le dessus du tissu). Dans ce cas, ajustez l’axe z à l’aide de l’appareil z-piezo jusqu’à ce qu’il atteigne la surface de l’agarose. Un mouvement manuel de l’objectif peut être nécessaire si la position souhaitée est en dehors de la portée de l’appareil z-piezo.
- Coupez plusieurs tranches de 50 μm après avoir trouvé la surface de l’échantillon et acquérez des images 2D après chaque coupe pour vous assurer que le plan focal est correctement défini sur la surface de la face du bloc.
5. Trouver les bords des échantillons et régler le laser sur le point de départ
- Sur la base des coordonnées xy de la zone de balayage laser (4 coordonnées d’angle), confinez la zone d’imagerie au tissu avec une imagerie minimale du bloc d’agarose environnant.
- Éteignez les PMT, réglez la longueur d’onde du laser sur 800 nm, basculez l’obturateur laser sur l’ouverture. La position de la lumière laser entrant dans le bloc d’agarose est visible sous la forme d’un point rouge de lumière diffusée (Figure 2A,B).
Étape critique : Soyez particulièrement prudent avec le laser de classe 4. Il peut rapidement endommager les tissus humains et les yeux. Le personnel a besoin d’une formation spéciale pour utiliser un tel instrument en toute sécurité. - Utilisez le spot laser tout en déplaçant la platine pour trouver les coordonnées des bords des tissus à l’aide de la séquence arrière, droite, avant et gauche.
- Vérifiez les coordonnées et positionnez le point laser dans le coin avant droit, qui est le point de départ par défaut pour l’imagerie du tissu.
- Convertissez les coordonnées de la fenêtre de la console en tailles de bloc reconnaissables par la machine, qui sont définies par les pas xy. Entrez ces mesures d’étape dans le protocole du logiciel. Fermez les portes de l’armoire.
6. 3D numérisation / sectionnement
- Changez la longueur d’onde à 940 nm pour exciter GFP, mWasabi ou YPet et PE.
REMARQUE : Le laser est invisible à l’œil humain à cette longueur d’onde. Pour améliorer le rapport signal/bruit de fond pour GFP, mWasabi et la composante émettrice verte de TIMERbac, placez un filtre passe-bande étroite de 510/20 nm devant PMT 2. Cela réduit l’interférence par l’autofluorescence des tissus vert-jaune. - Basculez l’obturateur du microscope en mode automatique et les réglages de tension d’obturation sur 20 pour V1 et 1,71 pour V2.
- Ajustez les trois paramètres suivants dans le logiciel : Épaisseur de la section - pour la plupart des usages, réglée sur 50 μm. Pour le foie, régler à 30 μm en raison de la profondeur d’imagerie limitée. Le nombre de coupes physiques - cela détermine la profondeur totale du tissu qui sera imagée, réglée en conséquence. Le nombre de plans à capturer pour chaque section déterminera la résolution z. Utilisez 5 plans (offrant une résolution verticale de 10 μm) pour la plupart des usages. Pour le foie, utilisez 3 plans ou moins.
- Réglez le gain laser. Cela doit être testé empiriquement pour différents tissus et profondeurs de balayage.
- Définissez le chemin d’accès et le nom du dossier pour le stockage des images acquises et fournissez les informations appropriées pour le métafichier d’imagerie.
- Vérifiez toutes les valeurs saisies.
- Cliquez sur le paramètre de mosaïque 3D pour lancer le processus de numérisation (Figure 1D).
- Une fois l’imagerie terminée, prélever soigneusement les coupes de tissu dans le réservoir d’eau et les conserver dans un tampon de stockage à -20 °C jusqu’à ce qu’elles soient utilisées (figure 1E ; Figures 2C, D).
7. Traitement d’images et analyse de données
- Téléchargez les scripts MATLAB sur un ordinateur Linux à partir de https://github.com/BumannLab/Li_BumannLab_2020. Copiez les scripts dans un dossier, par exemple /home/user/Program/.
REMARQUE : l’add-on Computer Vision Toolbox pour MATLAB et Fiji (ou ImageJ) doit être installé sur des ordinateurs Linux pour que le script soit entièrement fonctionnel. - Brodez les images de tuiles comme décrit ci-dessous.
- Transférez les données du serveur de tomographe vers l’ordinateur Linux.
- Ouvrez le script MATLAB StepOneStitchingAndArchive.m et localisez le dossier source contenant les données brutes. Définissez les dossiers source et destination dans le script.
- Passez à l’onglet Editeur et cliquez sur Exécuter. Les informations de progression s’affichent dans la fenêtre de commande. Le traitement comprend la lecture des informations du fichier Mosaic, la génération de l’index des tuiles avant l’assemblage de l’image, la correction de l’éclairage d’arrière-plan avec Cidre22 (Figure 3) ainsi que la correction de l’éclairage entre les couches optiques acquises à différentes profondeurs. Recherchez les images assemblées dans un sous-dossier appelé stitchedImages_100 dans le dossier source. Compressez les données brutes pour un stockage à long terme à l’aide de la commande tar et enregistrez-les en tant que fichier unique avec l’extension de nom de fichier tar.bz2 (Figure 1F).
- Effectuez l’entraînement et la segmentation du modèle avec une machine à vecteurs de support. Pour entraîner la machine à vecteurs de support et effectuer la segmentation, suivez les étapes ci-dessous.
REMARQUE : Les images assemblées pour chacun des trois canaux de fluorescence sont stockées dans 3 sous-dossiers (nommés 1, 2 et 3, correspondant à la fluorescence rouge, orange et verte) de stitchedImages_100. Chaque sous-dossier contient toutes les images assemblées du même canal.- Prévisualisez les images assemblées. Ouvrez une image avec le même nom de fichier à partir de chaque sous-dossier avec Fiji (ou ImageJ). Fusionnez les trois canaux en une seule image couleur. Ajustez la luminosité de chaque canal jusqu’à ce que des signaux clairs soient visibles provenant de bactéries et d’autofluorescence tissulaire. Notez le niveau d’intensité maximal ajusté de chaque canal pour l’étape suivante.
- Ouvrez le script MATLAB StepTwoSegmentationAndAnalysis.m et accédez à source. Définissez le dossier source et les noms des images pour l’entraînement. Par exemple
sourceD = '/' ;
red_name = [sourceD '1/section_020_01.tif'] ; green_name = [sourceD '2/section_020_01.tif'] ;
blue_name = [sourceD '3/section_020_01.tif'] ; - Allez dans l’onglet Éditeur et cliquez sur Exécuter.
- Une boîte de dialogue demandant des seuils de couleur apparaîtra, remplissez-la en fonction de la vérification manuelle précédente avec Fidji (étape 7.3.1).
- Sélectionnez les régions pour l’arrière-plan et les régions d’intérêt (c’est-à-dire les bactéries) en fonction des boîtes de dialogue. Des graphiques s’affichent pour montrer la qualité de l’entraînement du modèle et demander si d’autres régions doivent être ajoutées. Ajoutez d’autres régions d’intérêt et de fond jusqu’à ce que les bactéries puissent être clairement segmentées (Figure 4A,B).
- Nommez et enregistrez le modèle. Le script chargera automatiquement les images et appliquera un filtre médian (rayon 2 pixels).
- Le processus de segmentation s’exécute automatiquement et les informations de progression s’affichent dans la fenêtre de commande. Surveillez l’avancement de la segmentation. Si la segmentation ne fonctionne pas correctement avec le modèle sur d’autres images assemblées, répétez l’entraînement du modèle pour inclure davantage de régions d’arrière-plan et bactériennes.
- Pour voir les résultats de la segmentation, vérifiez le fichier nommé Allpositions_filter3D.txt. Ce fichier contient les coordonnées du centre de gravité de chaque bactérie ou microcolonie bactérienne et l’intensité de fluorescence intégrée correspondante (Figure 1G).
- Supprimez les artefacts supplémentaires à l’aide du réseau neuronal convolutif comme décrit ci-dessous.
REMARQUE : Pour certaines bactéries, une simple segmentation avec la machine à vecteurs de support inclut encore certains artefacts d’imagerie. Cela est particulièrement vrai pour les bactéries exprimant YPet. La segmentation basée sur les réseaux neuronaux peut supprimer efficacement ces artefacts (Figure 4C). Le module complémentaire Computer Vision Toolbox pour MATLAB et Fiji (ou ImageJ) doit être installé sur les ordinateurs Windows pour que le script soit entièrement fonctionnel. Pour plus d’informations sur le réseau neuronal sous-jacent, consultez : https://ch.mathworks.com/help/deeplearning/ug/create-simple-deep-learning-network-for-classification.html- Préparez les images. Convertissez des fichiers tif en fichiers jpg avec différents réglages de luminosité à l’aide de Fidji. Identifiez à l’aide d’une sélection manuelle au moins 600 bactéries et 600 artefacts, ainsi que des images recadrées en toute sécurité de ces objets dans des dossiers appropriés.
- Ouvrez le script MATLAB CNNtestV4.m et naviguez jusqu’aux fichiers sources contenant les images d’objet de l’étape 7.4.1.
- Définissez le dossier d’images pour l’entraînement. Par exemple
digitDatasetPath = fullfile ('D :\20200602_CNN','fortrainingPixel12Folder2') ;
Définissez le nombre d’images pour l’entraînement, par exemple, numTrainFiles = 600. - Allez dans l’onglet Éditeur et cliquez sur Exécuter. Après l’entraînement, le fichier du modèle CNN est enregistré sous le nom gregnet1.mat dans le dossier source.
- Définissez un dossier avec des images de test. Par exemple
digitDatasetPathTest = fullfile('D :\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
Les résultats sont enregistrés dans un fichier YourFile.txt qui contient des informations sur le nom de l’image et la classification en tant que bactéries ou artefacts. - Définissez le dossier avec des images de test. Par exemple
digitDatasetPathTest = fullfile('D :\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
Les résultats seront enregistrés dans un fichier YourFile.txt, qui contient des informations sur le nom de l’image et la classification en tant que bactérie ou artefact.
- Estimez la taille des microcolonies en fonction de l’intensité de fluorescence d’une seule bactérie, comme décrit ci-dessous.
REMARQUE : La résolution spatiale du tomographe est insuffisante pour résoudre les cellules bactériennes individuelles au sein de microcolonies densément tassées. Cependant, le nombre de bactéries dans chaque microcolonie peut être estimé sur la base de la fluorescence totale de la microcolonie par rapport à la fluorescence d’une seule bactérie. Cette précision d’estimation dépend de l’homogénéité de l’intensité de la fluorescence dans la population bactérienne.- Confirmer l’identité des événements positifs à la GFP dans la tomographie STP en colorant les composants bactériens tels que le lipopolysaccharide à l’aide de l’immunohistochimie de coupes récupérées du tomographe (Figure 1H ; Figure 5).
- Identifiez au moins 30 bactéries uniques et récupérez leur intensité de fluorescence à partir des résultats de segmentation correspondants. Utiliser la valeur d’intensité médiane comme référence pour une seule bactérie. Calculez le nombre de bactéries de chaque microcolonie en divisant l’intensité de fluorescence totale de la microcolonie par la valeur de référence d’une seule bactérie (figure 1I).
- Effectuez une reconstruction 3D à l’aide d’un logiciel de visualisation comme décrit ci-dessous.
- Préparation de l’image
- Prenez les images du canal bleu, qui contiennent des signaux de deuxième harmonique de collagène fournissant une référence anatomique utile, y compris les capsules de tissus, les artères et les trabécules. Utilisez des images segmentées pour les bactéries comme 2ème canal.
- Rangez les images du 1ercanal 10 fois sur les axes x et y et enregistrez les images réduites dans un nouveau dossier. Faites 3 répliques d’images réduites dans le même dossier. Le nom des nouvelles réplicats doit conserver la même séquence. Par exemple : section_001_01.tif, répliquez 1 avec le nom section_001_01-copy.tif, section_001_01-copy-copy.tif. Appliquez également ces étapes aux images du 2èmecanal. La réduction des tailles permet d’obtenir des tailles de fichiers plus gérables. Les images triples vont lisser l’axe z.
- Fusionnez les images de chaque canal pour former des piles d’images avec Fidji. Cliquez sur Piles > d’images > Outils > Trieur d’empilements > Trier par étiquettes.
- Effectuez un filtrage 3D. Cliquez sur Traiter les filtres > > Filtre 3D et définissez la taille du filtre Z sur 6. Enregistrez les piles d’images.
- Effectuez une visualisation 3D comme décrit ci-dessous.
- Ouvrez le progiciel de visualisation dans la vue Arena (paramètre par défaut). Utilisez l’icône du dossier de surveillance pour ajouter des dossiers à la vue Arena. Double-cliquez pour ouvrir le fichier *.ims ou *.tif (préparé dans la version 7.6.1).
- À l’aide de l’option Modifier>Propriétés de l’image, ajustez les représentations des couleurs (LUT) des différents canaux dans la fenêtre de réglage de l’affichage.
- Cliquez sur Avancé pour régler manuellement les valeurs min/max et une valeur pour la correction gamma.
Cliquez sur les noms des canaux pour modifier les noms et les LUT. - Après avoir ajusté l’apparence de l’image, exportez la vue actuelle à l’aide de l’outil Instantané.
- Utilisez l’icône Animation pour représenter les données 3D sous la forme d’un film. Utilisez le pointeur de navigation pour trouver une perspective/vue et zoomer et utilisez + Ajouter pour ajouter des images clés. Déplacez-vous vers une autre position et ajoutez l’image clé suivante. Appuyez sur le bouton d’enregistrement rouge pour construire le film. Enregistrez-le dans le dossier de destination et le type de fichier souhaités (Figure 1J).
- Préparation de l’image
Résultats
La procédure décrite permet de détecter des cellules individuelles de Salmonella dans des organes entiers de souris tels que la rate, le foie, les ganglions lymphatiques mésentériques et les plaques de Peyer11 (figures 5 et 6). Il détecte également les parasites Toxoplasma gondii dans le cerveau de la souris12. Certains tissus infectés, notamment le foie, les plaques de Peyer et la rate, émettent une autofluorescence substantielle dans la gamme vert-jaune. L’autofluorescence est encore renforcée par la fixation au paraformaldéhyde, qui est nécessaire pour préserver la structure tissulaire. La détection de la fluorescence verte de la GFP, du mWasabi et de la composante verte de TIMERbac contre ce fond d’autofluorescence est améliorée en plaçant un filtre passe-bande étroite 510/20 nm (transmettant la plupart des émissions GFP mais bloquant une grande partie du spectre d’autofluorescence) devant le photomultiplicateur 2 (qui collecte les émissions vertes) et en réduisant l’autofluorescence tissulaire en stockant les tissus fixés pendant 3 jours ou plus dans le cryoprotecteur11. Cependant, les bactéries devraient toujours exprimer au moins quelques milliers de copies par cellule de GFP ou d’autres protéines fluorescentes. D’autre part, des niveaux excessifs de protéines fluorescentes doivent être évités afin de minimiser les coûts de remise en forme qui pourraient conduire à une virulence atténuée23.
La segmentation correcte des bactéries fluorescentes dans les tissus peut être confirmée par l’immunohistochimie des coupes de tissus récupérées après l’imagerie. Plus précisément, les objets colorés avec un anticorps contre les composants de surface bactériens tels que le lipopolysaccharide peuvent être alignés avec l’image de fluorescence obtenue par tomographie STP (Figure 5). Il est important de noter que certaines cellules colorées de Salmonella sont dépourvues de protéines fluorescentes et donc de fluorescence détectable à la fois en microscopie confocale et en tomographie STP. Ces cellules sont des Salmonella qui ont été tuées par le système immunitaire de l’hôte, comme le démontrent le tri cytométrique en flux et les cultures de croissance à partir de cellules triées uniques, ainsi que la corrélation étroite entre le nombre d’unités formant des colonies sur les plaques de gélose et le nombre de cellules fluorescentes de Salmonella , déterminée par cytométrie en flux24. De plus, la perte de plasmide peut entraîner la formation de cellules viables non fluorescentes, ce qui doit être testé par placage sur des milieux avec et sans antibiotiques appropriés correspondant au marqueur de sélection sur le plasmide. Pour les plasmides dérivés de pSC101, la perte de plasmide in vivo est rare19. Pour la plupart des cassettes d’expression chromosomiquement intégrées telles que sifB ::gfp utilisées dans11, la perte d’expression est indétectable in vivo. Si la segmentation n’est pas cohérente avec les données d’immunohistochimie, le pipeline de segmentation doit être modifié.
La résolution de la tomographie STP est insuffisante pour résoudre les cellules bactériennes individuelles au sein de microcolonies densément tassées. Cependant, l’intensité totale de fluorescence de la microcolonie permet d’estimer le nombre de cellules Salmonella . Cela nécessite une souche fluorescente avec des niveaux de fluorescence très homogènes tels que Salmonella sifB ::gfp11. La combinaison du nombre estimé de cellules de Salmonella pour toutes les microcolonies et les cellules individuelles permet d’obtenir des charges tissulaires bactériennes totales qui sont compatibles avec d’autres méthodes telles que le placage ou la cytométrie en flux des homogénats tissulaires11. Le placage et la cytométrie en flux ne peuvent pas être effectués directement à partir des mêmes tissus car ils doivent être fixés par perfusion pour la tomographie STP. Au lieu de cela, ils doivent être faits avec des animaux supplémentaires qui ne sont pas fixes. Si les charges bactériennes médianes déterminées par les différentes approches diffèrent de plus de 3 fois, la viabilité des bactéries fluorescentes pourrait être compromise (dans le cas d’unités formant des colonies inférieures) ou certaines bactéries pourraient avoir perdu la construction du rapporteur fluorescent (dans le cas d’unités formant des colonies supérieures). Une expérience de contrôle sera nécessaire pour identifier la source de ces écarts.
La tomographie STP permet de localiser les cellules bactériennes dans la structure 3D des tissus infectés. Les signaux de la deuxième harmonique du collagène fournissent des repères anatomiques tels que les artères et les trabécules. De plus, les cellules hôtes peuvent être colorées in vivo en injectant un anticorps aux marqueurs de surface avant la perfusion (Figure 5 et Figure 6). Cette coloration fournit des repères supplémentaires pour les compartiments tissulaires et les microenvironnements spécifiques, y compris les foyers d’inflammation (les marqueurs intracellulaires et certains compartiments avec des barrières de diffusion, tels que la splénique, la pulpe blanche ou le cerveau, ne sont pas facilement accessibles, adaptés à cette coloration in vivo ). Cette approche a révélé que la pulpe splénique blanche est un compartiment tissulaire qui permet la survie à long terme de Salmonella pendant la chimiothérapie antimicrobienne11.
Enfin, la réplication de Salmonella à des taux modérés ou lents peut être identifiée et localisée dans la structure 3D des tissus à l’aide de souches qui expriment timerbac, un rapporteur unicellulaire pour le taux de réplication11,15.
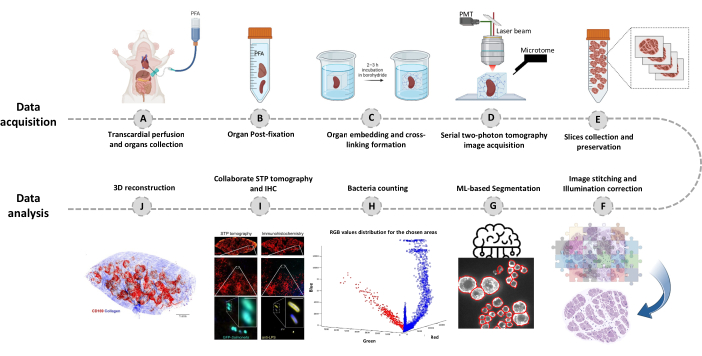
Figure 1 : Procédure d’imagerie de l’organe entier à l’aide de la tomographie à deux photons en série (STP). (A-B) L’organe est prélevé après une perfusion transcardique et stocké pendant la nuit dans du paraformaldéhyde à 4 % (PFA) à 4 °C. (C-E) L’organe est incorporé dans de l’agarose oxydée et réticulé, puis le tissu est scanné et tranché à l’aide de la tomographie STP. Les tranches sont collectées pour l’immunohistochimie ultérieure. (F-J) Pipelines d’analyse computationnelle pour la quantification du nombre de bactéries, la confirmation des bactéries fluorescentes et la reconstruction 3D des positions bactériennes. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Configuration de la tomographie en série à deux photons (STP). (A) Tomographe qui intègre (B) l’imagerie à 2 photons avec (C) la section automatisée des tissus en série. (D) Coupes de tissu collectées pour des investigations de suivi. Veuillez cliquer ici pour voir une version agrandie de cette figure.
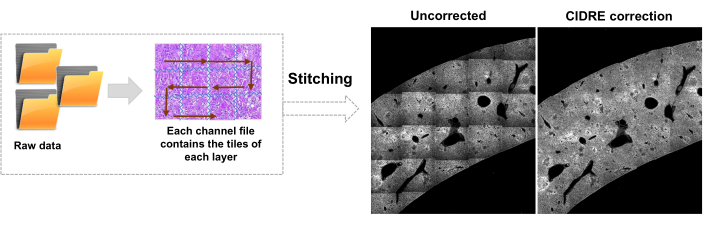
Figure 3 : Couture des carreaux et correction de l’éclairage. Les tuiles sont assemblées et l’éclairage inégal est corrigé à l’aide de distributions d’intensité corrigées à l’aide de la minimisation d’énergie régularisée (CIDRE). Veuillez cliquer ici pour voir une version agrandie de cette figure.
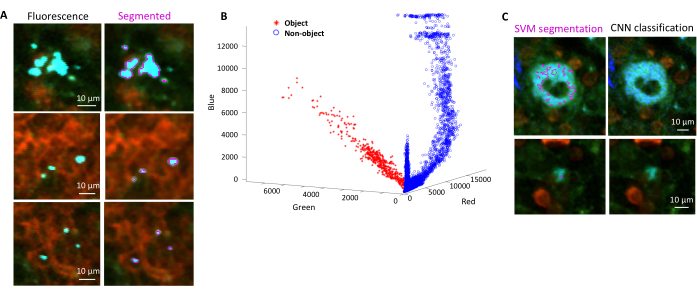
Figure 4 : Segmentation de Salmonella exprimant la protéine fluorescente verte (GFP) à l’aide d’une machine à vecteurs de support (SVM) et d’un réseau neuronal convolutif (CNN). (A) Images représentatives d’objets GFP segmentés par SVM (à gauche) et images correspondantes (à droite) avec régions segmentées par SVM (Barre d’échelle : 10 μm). (B) Distribution des valeurs en grappes rouge-vert-bleu (RVB) pour les régions segmentées. (C) Images représentatives d’objets non-GFP faussement identifiés par la SVM comme des bactéries (à gauche). CNN les rejette correctement en tant qu’arrière-plan (à droite, barre d’échelle : 10 μm). Veuillez cliquer ici pour voir une version agrandie de cette figure.
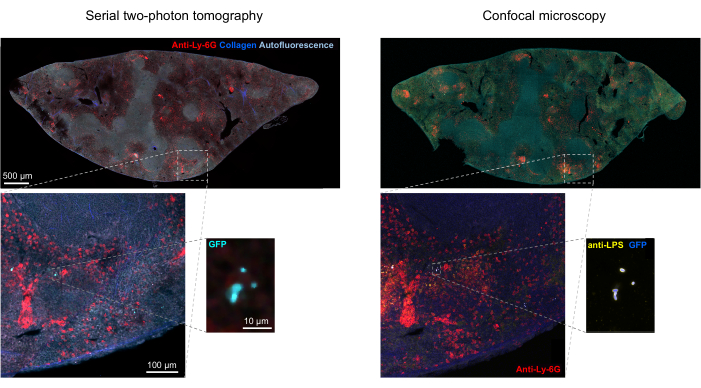
Figure 5 : Détection de Salmonella exprimant la protéine fluorescente verte (GFP) par tomographie et confirmation par immunohistochimie. Images de la même section acquises par tomographie (à gauche) ou microscopie confocale après coloration avec un anticorps contre le lipopolysaccharide de Salmonella (à droite). Les neutrophiles (en rouge) ont été colorés par injection in vivo d’un anticorps anti-Ly-6G marqué en PE avant la perfusion. Veuillez cliquer ici pour voir une version agrandie de cette figure.
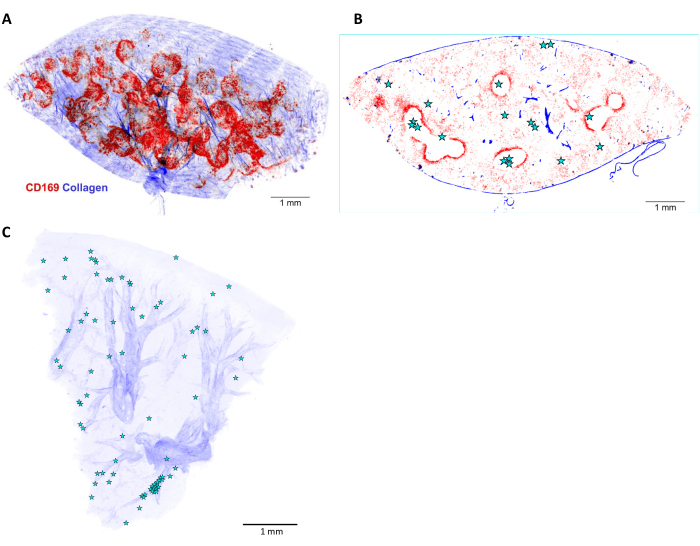
Figure 6 : Reconstruction 3D et localisation de Salmonella dans la rate de souris infectée. (A) Reconstruction tridimensionnelle (3D) d’une tranche de rate de 5 mm d’épaisseur et colorée in vivo avant perfusion avec un anticorps anti-CD169 (rouge). Le signal bleu représente le collagène détecté par les secondes harmoniques. (B) Un plan optique de la pile 3D illustrée en (A). Les positions des cellules ou microcolonies de Salmonella sont indiquées par des étoiles. (C) Reconstruction 3D des positions de Salmonella (étoiles) dans le foie infecté. Les artères sont visibles grâce à leurs gaines de collagène (bleu). Barre d’échelle : 1 mm. Veuillez cliquer ici pour voir une version plus grande de cette figure.
Discussion
Le contexte tissulaire local des agents pathogènes bactériens est crucial pour déterminer les attaques locales de l’hôte, les adaptations bactériennes, l’issue locale des interactions hôte-pathogène et de la chimiothérapie antimicrobienne, et les contributions individuelles à l’issue globale de la maladie. L’imagerie de bactéries de taille micrométrique dans des organes de quelques centimètres a été un défi. La tomographie à deux photons en série (STP) offre une résolution spatiale suffisante pour détecter des cellules bactériennes individuelles dans des organes entiers, une coupe et une imagerie automatisées, et un débit suffisant (~1 organe par jour)11. Bien que les antigènes de l’hôte puissent être colorés in vivo, les cellules pathogènes doivent exprimer des protéines fluorescentes appropriées pour assurer une détection complète des cellules pathogènes intracellulaires. Les ensembles de données qui en résultent (0,5 à 1,5 téraoctet par organe) posent des défis considérables aux infrastructures informatiques pour l’analyse et le stockage des données.
Cette méthode comporte plusieurs étapes critiques. Tout d’abord, une souche pathogène avec une expression détectable et homogène de la protéine fluorescente GFP ou YFP est requise. Idéalement, une cassette d’expression chromosomique25 est utilisée pour minimiser l’hétérogénéité de fluorescence due à la variation du nombre de copies plasmidiques. Une intensité de fluorescence suffisante est requise, mais des niveaux excessifs de protéines fluorescentes doivent être évités pour éviter les troubles de la condition physique de l’agent pathogène23. Des niveaux d’expression appropriés peuvent être obtenus par la sélection d’un promoteur approprié et l’ajustement fin du site de liaison ribosomique25 ou de l’ensemble de la région 5' non traduite (UTR)26. Deuxièmement, la fixation de la perfusion doit impliquer un lavage initial avec un tampon pour éliminer autant d’érythrocytes que possible de la circulation sanguine. Ceci est particulièrement critique pour la rate et le foie (bien que l’élimination complète des érythrocytes de ces organes soit difficile). Les érythrocytes restants absorbent la lumière dans la partie visible du spectre, compromettant la qualité de l’imagerie27. Troisièmement, le stockage des tissus fixés dans le cryoprotecteur est essentiel pour réduire l’autofluorescence des tissus, qui est particulièrement élevée dans les tissus enflammés et peut éclipser la fluorescence relativement faible des cellules pathogènes11. Quatrièmement, la réticulation efficace du tissu avec le bloc d’agarose environnant est essentielle pour une coupe en douceur du vibratome sans que le tissu ne sorte du bloc d’agarose. Cinquièmement, les signaux fluorescents et leur identification en tant que cellules pathogènes doivent être vérifiés indépendamment à l’aide d’approches orthogonales telles que la coloration avec des anticorps contre les composants pathogènes (tels que le lipopolysaccharide pour les bactéries à Gram négatif) et la microscopie confocale des coupes extraites du tomographe11. Certains tissus infectés contiennent des particules auto-fluorescentes de forme similaire et de spectres de fluorescence qui se chevauchent et qui peuvent facilement être interprétées à tort comme des cellules pathogènes. Sixièmement, la quantité de cellules pathogènes dans les microcolonies doit être comparée à des approches orthogonales telles que la microscopie confocale pour évaluer la précision. Les charges bactériennes globales basées sur ces calculs doivent être vérifiées par comparaison avec des approches orthogonales telles que la cytométrie en flux et le placage.
Parmi les modifications importantes apportées au protocole STP largement utilisé, citons la mise en place d’un filtre passe-bande étroit de 510/20 nm devant le photomultiplicateur 211, afin de réduire les interférences de l’autofluorescence vert-jaune qui est particulièrement forte dans le foie, la rate et les plaques de Peyer infectées et enflammées. La forte autofluorescence et l’augmentation de la diffusion de la lumière de ces organes par rapport au cerveau (qui domine les autres applications de STP) génèrent également un besoin de correction plus efficace de l’éclairage inégal. Autre modification, ce protocole utilise l’approche CIDRE22 à cette fin (Figure 3) et la segmentation des bactéries basée sur l’IA. Enfin, le prétraitement tissulaire a été modifié en incluant une étape d’incubation dans un cryoprotecteur à -20 °C, ce qui réduit l’autofluorescence tissulaire et facilite ainsi la détection des petites cellules pathogènes à fluorescence relativement faible11.
Le dépannage peut être nécessaire si aucun signal d’agent pathogène ne peut être détecté, ou si la segmentation donne une sensibilité insuffisante (trop de cellules pathogènes sont manquées) ou une précision insuffisante (trop de particules d’arrière-plan sont segmentées en cellules pathogènes). Si l’autofluorescence des tissus de fond est détectable mais qu’il y a trop peu de signaux pathogènes, les agents pathogènes peuvent contenir des quantités insuffisantes de protéines fluorescentes. Cela peut être testé à l’aide de la microscopie confocale de coupes de tissus du même tissu infecté ou de la cytométrie en flux d’homogénats de tissus19,28. Les raisons sous-jacentes peuvent être des niveaux d’expression insuffisants ou une instabilité de la cassette d’expression. Les stratégies d’atténuation pourraient inclure d’autres promoteurs pour stimuler l’expression, l’adaptation des codons des gènes codant pour la protéine fluorescente pour l’espèce pathogène, l’utilisation de constructions épisomiques avec un nombre de copies plus élevé, ou la stabilisation des cassettes d’expression par intégration chromosomique ou complémentation létale équilibrée29. Le choix de la protéine fluorescente est également important, mais la détection est possible avec GFP.mut2, mWasabi, YPet et TIMERbac. Si la segmentation est inexacte, cela peut être dû à une fluorescence pathogène trop faible qui pourrait être traitée comme décrit ci-dessus, ou à un fond d’autofluorescence tissulaire trop élevé. Une perfusion importante de solution de lavage ou une incubation prolongée dans la mémoire tampon de stockage immédiatement avant l’intégration dans le bloc d’agarose et la tomodensitométrie peuvent résoudre ces problèmes. Enfin, un entraînement suffisant du réseau neuronal est nécessaire pour une classification précise, mais un entraînement excessif peut conduire à un surajustement qui nuit aux performances des nouveaux échantillons.
À l’heure actuelle, aucune autre méthode ne permet d’imager des organes entiers à une résolution spatiale suffisante en 3D pour détecter des bactéries individuelles. Les améliorations futures de l’élimination des tissus et de la microscopie à feuillet de lumière pourraient permettre d’obtenir une résolution similaire. Cela pourrait permettre d’obtenir des images à une vitesse plus élevée et avec plus de canaux fluorescents.
Une limitation importante de la STP est la résolution des pixels dans le plan de ~0,5 μm et la résolution verticale de 5 à 10 μm, ce qui est insuffisant pour résoudre les bactéries situées à proximité, par exemple au sein d’une microcolonie densément emballée. Cependant, il est possible de récupérer des coupes de tissus après la tomodensitométrie pour la microscopie confocale secondaire à haute résolution de parties de tissus sélectionnées. Une autre limitation de STP est la disponibilité de seulement trois canaux de fluorescence, ce qui limite le nombre de fluorophores pouvant être imagés simultanément. Encore une fois, l’analyse secondaire de coupes de tissus récupérées avec des méthodes de multiplexage peut révéler l’emplacement et l’intensité de beaucoup plus de marqueurs pour des parties de tissus sélectionnées. Cette information pourrait être intégrée dans la structure 3D globale du tissu environnant, telle que déterminée avec STP.
En conclusion, ce protocole permet d’étudier en détail les interactions hôte-pathogène au niveau local et au niveau de l’organe entier. Le protocole doit être facilement adaptable à d’autres agents pathogènes (à condition qu’ils puissent être obtenus sous forme de souches fluorescentes), à d’autres organes et à différentes espèces hôtes.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Le travail a été soutenu par le Fonds national suisse de la recherche scientifique 310030_156818, 310030_182315 et NCCR_ 180541 AntiResist (à DB).
matériels
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agarose Low Melt | Roth | Art. 6351.5 25g | |
| Boric acid | Sigma-Aldrich | 6768-500G | |
| Instant adhesive Loctite 435 | Henkel | ||
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Poly(ethylene glycol) | Sigma-Aldrich | P5413-1kg | |
| Polyvinylpyrrolidone | Sigma-Aldrich | PVP-100G | |
| Sodium borohydride | Sigma-Aldrich | 71321-25g | |
| Sodium hydroxide | Merck | 106453 | |
| Sodium periodate | Sigma-Aldrich | 311448-100G | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640-250G | |
| Sodium phosphate monobasic dihydrate | Sigma-Aldrich | 71500-1KG | |
| Sodium tetraborate | Sigma-Aldrich | 221732-100g | |
| Sucrose | AppliChem | A4734,1000 | |
| Tris-buffered saline (TBS) | Merck | T5912-1L | |
| Triton X-100 | Sigma-Aldrich | 9002-93-1 | |
| Vacuum filtration 500 | TPP | TPP99250 | |
| Equipment | |||
| Blade | Campden Instruments Limited | 01-01-4692 | |
| MAITAI Laser | Spectra-Physics | ||
| Peel away plastic mold | Sigma-Aldrich | E6032-1CS | |
| TissueCyte 1000 tomograph | TissueVision | ||
| Antibody/dyes | |||
| DAPI | Merck | D9542-5MG | |
| Primary antibodies | |||
| anti-LPS Salmonella, rabbit | Sifin | REF TS 1624 | |
| anti-CD169-PE, clone 3D6.112 | Biolegend | 142403 | |
| anti-Ly-6G-PE, clone 1A8 | Biolegend | 127608 | |
| Secondary antibodies | Invitrogen | ||
| chicken anti-rabbit Alexa 647 | Invitrogen | A-21443 | |
| Software | Company | Version | |
| Fiji | Image J | 1.54g or later | |
| MATLAB | MathWorks | 2017b/2018b or later | |
| Orchestrator (tomograph) | TissueVision | ||
| Visualization software Imaris | Oxford Instruments | 9.9.0 or later |
Références
- Bjarnsholt, T., et al. The importance of understanding the infectious microenvironment. Lancet Infect Dis. 22 (3), e88-e92 (2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Bumann, D., Cunrath, O. Heterogeneity of Salmonella-host interactions in infected host tissues. Curr Opin Microbiol. 39, 57-63 (2017).
- Hofmann, J., Keppler, S. J. Tissue clearing and 3D imaging - putting immune cells into context. J Cell Sci. 134 (15), jcs258494(2021).
- Blain, R., et al. A tridimensional atlas of the developing human head. Cell. 186 (26), 5910-5924.e17 (2023).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Meth. 9 (3), 255-258 (2012).
- Oh, S. W., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Kim, Y., et al. Brain-wide maps reveal stereotyped cell-type-based cortical architecture and subcortical sexual dimorphism. Cell. 171 (2), 456-469.e422 (2017).
- Matho, K. S., et al. Genetic dissection of the glutamatergic neuron system in cerebral cortex. Nature. 598 (7879), 182-187 (2021).
- Muñoz-Castañeda, R., et al. Cellular anatomy of the mouse primary motor cortex. Nature. 598 (7879), 159-166 (2021).
- Li, J., et al. Tissue compartmentalization enables Salmonella persistence during chemotherapy. Proc Natl Acad Sci U S A. 118 (51), e2113951118(2021).
- Dogga, S. K., et al. Importance of aspartyl protease 5 in the establishment of the intracellular niche during acute and chronic infection of Toxoplasma gondii. Mol Microbiol. 118 (6), 601-622 (2022).
- Cormack, B. P., Valdivia, R. H., Falkow, S. FACS-optimized mutants of the green fluorescent protein (GFP). Gene. 173 (1 Spec No), 33-38 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Claudi, B., et al. Phenotypic variation of Salmonella in host tissues delays eradication by antimicrobial chemotherapy. Cell. 158 (4), 722-733 (2014).
- Ai, H. W., Olenych, S. G., Wong, P., Davidson, M. W., Campbell, R. E. Hue-shifted monomeric variants of Clavularia cyan fluorescent protein: identification of the molecular determinants of color and applications in fluorescence imaging. BMC Biol. 6, 13(2008).
- Drobizhev, M., Makarov, N. S., Tillo, S. E., Hughes, T. E., Rebane, A. Two-photon absorption properties of fluorescent proteins. Nat Methods. 8 (5), 393-399 (2011).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Burton, N. A., et al. Disparate impact of oxidative host defenses determines the fate of Salmonella during systemic infection in mice. Cell Host&Microbe. 15 (1), 72-83 (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio Protoc. 11 (5), e3988(2021).
- de Olmos, J., Hardy, H., Heimer, L. The afferent connections of the main and the accessory olfactory bulb formations in the rat: an experimental HRP-study. J Comp Neurol. 181 (2), 213-244 (1978).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nat Methods. 12 (5), 404-406 (2015).
- Wendland, M., Bumann, D. Optimization of GFP levels for analyzing Salmonella gene expression during an infection. FEBS Lett. 521 (1-3), 105-108 (2002).
- Barat, S., et al. Immunity to intracellular Salmonella depends on surface-associated antigens. PLoS Pathog. 8 (10), e1002966(2012).
- Rollenhagen, C., Sorensen, M., Rizos, K., Hurvitz, R., Bumann, D. Antigen selection based on expression levels during infection facilitates vaccine development for an intracellular pathogen. Proc Natl Acad Sci U S A. 101 (23), 8739-8744 (2004).
- Chen, F., Cocaign-Bousquet, M., Girbal, L., Nouaille, S. 5'UTR sequences influence protein levels in Escherichia coli by regulating translation initiation and mRNA stability. Front Microbiol. 13, 1088941(2022).
- Weissleder, R. A clearer vision for in vivo imaging. Nat Biotechnol. 19 (4), 316-317 (2001).
- Bumann, D. Examination of Salmonella gene expression in an infected mammalian host using the green fluorescent protein and two-colour flow cytometry. Mol.Microbiol. 43 (5), 1269-1283 (2002).
- Nakayama, K., Kelly, S. M., Curtiss III, R. J. B. t Construction of an Asd+ expression-cloning vector: stable maintenance and high level expression of cloned genes in a Salmonella. vaccine strain. 6 (6), 693-697 (1988).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon