Method Article
Tomografia tridimensionale tridimensionale ad alta risoluzione di infezioni microbiche
In questo articolo
Riepilogo
In questo articolo, descriviamo una procedura che consente il rilevamento a livello di organo di batteri patogeni durante l'infezione e la quantificazione delle attività del reporter fluorescente.
Abstract
La maggior parte delle infezioni avviene all'interno di tessuti ospiti tridimensionali con un'anatomia complessa e una fisiologia dell'ospite variabile localmente. Il posizionamento delle cellule patogene all'interno di questo ambiente diversificato influenza in modo significativo i loro livelli di stress, le risposte, il destino e il contributo alla progressione complessiva della malattia e al fallimento del trattamento. Tuttavia, a causa delle difficoltà tecniche nel localizzare cellule patogene di dimensioni μm all'interno di organi ospiti di dimensioni cm, quest'area di ricerca è stata relativamente inesplorata. Qui presentiamo un metodo per affrontare questa sfida. Utilizziamo la tomografia seriale a due fotoni e l'analisi delle immagini potenziata dall'intelligenza artificiale per localizzare le singole cellule di Salmonella in tutta la milza, i lobi epatici e i linfonodi interi dei topi infetti. Utilizzando reporter fluorescenti e la somministrazione di anticorpi in vivo , è possibile determinare il tasso di replicazione delle singole cellule di Salmonella , la loro interazione locale con specifiche cellule immunitarie e le risposte batteriche agli antibiotici. Queste metodologie aprono la strada a un esame completo delle infezioni, della loro prevenzione e del loro trattamento all'interno del contesto tissutale tridimensionale.
Introduzione
Le infezioni si verificano in tessuti con anatomia complessa e fisiologia compartimentalizzata. I diversi microambienti che coesistono nel tessuto infetto possono determinare il destino dei sottogruppi di patogeni locali e il loro contributo all'esito complessivo della malattia 1,2,3. Tuttavia, la mappatura 3D completa dei patogeni microbici in tessuti di dimensioni centimetriche rimane impegnativa4. L'imaging del cervello e di altri organi è un campo di ricerca molto attivo con strategie sperimentali in costante miglioramento5, ma molti metodi mancano ancora della risoluzione inferiore al μm che sarebbe necessaria per identificare con sicurezza i patogeni batterici di dimensioni μm. Al contrario, la tomografia seriale a due fotoni (STP)6 consente l'imaging automatizzato multicolore e privo di deformazioni di interi tessuti con risoluzione in piano inferiore al μm, producendo set di dati volumetrici completi. Questo metodo combina il sezionamento fisico ripetuto del tessuto utilizzando un vibratomo con l'imaging intermittente a due fotoni delle facce emergenti del blocco con luce infrarossa. La tomografia STP è stata ampiamente utilizzata per mappare gli assoni sottili nel cervello per stabilire mappe di connettività 7,8,9,10.
La tomografia STP consente inoltre la mappatura 3D di singole cellule microbiche patogene (Salmonella, Toxoplasma) in tutti i tessuti infetti11,12 utilizzando un tomografo. La generazione della seconda armonica rivela guaine di collagene attorno alle arterie e nelle bande fibrose come le trabecole della milza, fornendo così un contesto anatomico. Gli anticorpi fluorescenti iniettati in vivo possono essere utilizzati per colorare le cellule ospiti per rivelare le interazioni tra le singole cellule patogene e le cellule immunitarie infiltranti come i neutrofili. Qui viene descritta la pipeline che coinvolge l'elaborazione del tessuto, l'imaging, lo stitching di piastrelle di imaging con correzione dell'illuminazione, l'impilamento di immagini in tre dimensioni e la segmentazione utilizzando strumenti di apprendimento automatico. Questa pipeline produce posizioni 3D di singoli agenti patogeni, cellule e microcolonie all'interno del loro contesto ospite. Il conteggio del numero di singole cellule all'interno delle microcolonie rimane difficile a causa dei limiti di risoluzione, ma tali numeri possono essere stimati in base alla luminosità integrata della microcolonia. La pipeline può essere facilmente adattata ad altri modelli di infezione se sono disponibili patogeni ricombinanti che esprimono GFP o YFP.
Protocollo
Tutti gli esperimenti sugli animali qui descritti sono stati approvati dalle autorità (licenza 2239, Kantonales Veterinäramt Basel) e seguono le linee guida locali (Tierschutz-Verordnung, Basilea) e la legge svizzera sulla protezione degli animali (Tierschutz-Gesetz).
1. Preparazione e conservazione dei tessuti infetti
- Utilizzare un modello di infezione a scelta. Qui, topi di età compresa tra 10 e 16 settimane di entrambi i sessi vengono infettati con ~ 1.000 unità formanti colonie (CFU) mediante iniezione endovenosa o 5 x 107 CFU mediante somministrazione intragastrica con un ago a punta tonda. I ceppi di topi adatti includono BALB/c e C57BL/6.
NOTA: Lo stesso approccio può essere facilmente adattato per altre specie ospiti. - Assicurarsi che i patogeni esprimano proteine fluorescenti per consentire l'identificazione nei tessuti non colorati. I patogeni che esprimono GFP.mut213, YPet14, TIMERbac15 o mWasabi16 possono essere visualizzati utilizzando l'eccitazione a 940 nm17. La Salmonella che esprime mCherry18 a livelli facilmente rilevabili mediante citometria a flusso19 potrebbe essere visualizzata utilizzando un'eccitazione di 800 nm, ma ha scarsi rapporti segnale/fondo nella milza e nel fegato infetti, che mostrano una sostanziale autofluorescenza.
- Assicurarsi che tutte le proteine fluorescenti mantengano >70% della loro intensità di fluorescenza dopo la fissazione con paraformaldeide a pH neutro sulla base della quantificazione con citometria a flusso. Qui vengono utilizzati tessuti di topo infetti, vale a dire milza, fegato, linfonodi e cerotti di Peyer.
- Colorare i marcatori della superficie delle cellule ospiti in vivo iniettando anticorpi marcati con ficoeritrina (PE) 10 minuti prima della perfusione. Per colorare i neutrofili, iniettare per via endovenosa 4 μg di anti-Ly-6G-PE in 100 μL di PBS. Per colorare i macrofagi della zona marginale nella milza, iniettare 4 μg di anti-CD169-PE in 100 μL di PBS (vedi Tabella dei materiali).
- Fissare i tessuti infetti attraverso la perfusione transcardica standard20 con 15 mL di tampone fosfato 50 mM ghiacciato (PB; 10 mM NaH2PO4, 40 mMNa2HPO4, pH 7,4), seguito da 35 mL di paraformaldeide al 4% (PFA; fissativo) in PB (Figura 1A).
- In breve, somministrare l'anestesia profonda all'animale infetto mediante iniezione intraperitoneale di 100 mg/kg di ketamina e 16 mg/kg di xilazina. Garantire un'adeguata anestesia confermando la perdita del riflesso di pizzicamento delle dita.
- Disinfetta il pelo usando etanolo al 70%, asciugalo con un tovagliolo di carta sterile e apri chirurgicamente il torace con una pinzetta e delle forbici. Posizionare un ago sterile nel ventricolo sinistro del cuore, aprire l'atrio destro con forbici sterili e far scorrere il primo tampone e poi il fissativo attraverso l'ago, il ventricolo sinistro e l'intero sistema vascolare utilizzando una pompa. Questo fissa tutti i tessuti in modo rapido e uniforme e provoca l'eutanasia.
- Raccogliere il tessuto dopo la perfusione, incubarlo in un volume 20x di paraformaldeide fredda al 4% in tampone PB e posizionarlo su un agitatore a 4 °C per una notte (Figura 1B). L'imaging è possibile per tessuti di dimensioni comprese tra 0,05 e 2 cm3. Per fazzoletti più grandi, tagliare in pezzi adatti.
- Il giorno successivo, lavare il fazzoletto 3 volte per 10 minuti ciascuno con tampone PB agitando a 40 giri/min a temperatura ambiente per rimuovere il PFA.
NOTA: A causa della tossicità del PFA, questi passaggi devono essere eseguiti all'interno di una cappa aspirante. Smaltire i rifiuti di PFA secondo le linee guida istituzionali. - Rimuovere il tampone PB in eccesso e aggiungere 20 volte il volume del tampone di conservazione crioprotettore21 (876 mM di saccarosio, 0,25 mM di polivinilpirrolidone, 40% (v/v) di glicole etilenico, disciolto in 50 mM di tampone PB) al campione agitando a 40 giri/min a 4 °C per 6-8 ore o fino a quando i tessuti non affondano sul fondo. Incubare i campioni a -20 °C per almeno 3 giorni per garantire un'efficace riduzione dell'autofluorescenza tissutale11. I campioni possono essere utilizzati fino a 5 anni se conservati a -20 °C.
NOTA: L'affondamento del tessuto indica una corretta penetrazione del tampone di conservazione nel tessuto.
2. Inclusione del campione
- Incorporare il tessuto in un blocco di agarosio per un taglio regolare con un vibratomo. Pre-attivare chimicamente l'agarosio mediante ossidazione con periodato e quindi ridurre con boroidruro per formare un legame incrociato con il tessuto per una migliore stabilità durante il processo di taglio. Eseguire i passaggi descritti di seguito.
- Preparare l'agarosio ossidato al 4% pesando 2,25 g di agarosio e aggiungere 0,21 g di NaIO4. Aggiungere 100 ml di PB all'agarosio e NaIO4. Agitare a temperatura ambiente in cappa aspirante per 2-3 h al riparo dalla luce.
NOTA: Passaggio critico: non superare l'agitazione per più di 3 ore o l'agarosio polimerizzerà male. - Recuperare l'agarosio mediante filtrazione a membrana in un filtro sottovuoto standard con membrana da 0,2 μm. Rimuovere il NaIO4 rimanente lavando 3 volte con 50 ml di tampone PB. Risospendere l'agarosio ossidato in 50 mL di tampone PB.
NOTA: Non sciogliere l'agarosio durante questa fase. L'agarosio può essere conservato a 4 °C per un massimo di 2-3 settimane al riparo dalla luce. - Preparare la soluzione di boroidruro aggiungendo 19 g di borace e 3 g di acido borico a 1 L di acqua. Mescolare fino a quando non si sarà sciolto e regolare il pH a 9-9,5 con 1 M NaOH. Su una piastra di agitazione in una cappa aspirante, riscaldare 100 mL di tampone borato a 40 °C. Aggiungere 0,2 g di NaBH4 e mescolare per 15-30 minuti proteggendo dalla luce.
NOTA: La soluzione di boroidruro può essere conservata per diverse settimane a temperatura ambiente al buio. Passaggio critico: quando si aggiunge NaBH4 al tampone di borato, si formerà del gas, quindi eseguire questo passaggio all'interno di una cappa aspirante. Non eseguire questa operazione in un contenitore ermeticamente sigillato perché potrebbe causare un'esplosione. - Prelevare il tessuto dal congelatore dopo un'incubazione sufficiente per ridurre l'autofluorescenza (≥ 3 giorni), rimuovere il tampone di conservazione del crioprotettore in eccesso e lavare il tessuto 3 volte per 10 minuti ciascuno con tampone PB agitando a 40 giri/min a temperatura ambiente (RT).
- Sciogliere circa 20 mL di sospensione di agarosio ossidato (preparata al punto 1) in un forno a microonde (700 W per ~30 s), lasciarla raffreddare abbastanza da poter essere maneggiata (~45 °C) e versarla in uno stampo di plastica.
NOTA: Passaggio critico: non aggiungere l'agarosio subito dopo averlo riscaldato nel microonde sul campione, ciò potrebbe portare alla distruzione del tessuto. - Usa una pinzetta per raccogliere delicatamente il fazzoletto e inserirlo rapidamente nella parte inferiore centrale dell'agarosio. Incubare lo stampo a RT per 15 minuti per consentire all'agarosio di polimerizzare.
- Dopo la polimerizzazione dell'agarosio, aprire lo stampo di plastica con un bisturi dai quattro angoli e raccogliere con cura il cubo di agarosio con le dita guantate.
- Immergere il campione in una soluzione di boroidruro (preparata nella fase 2) per 2-3 ore a 4 °C per avviare la reticolazione dell'agarosio ossidato e del tessuto. Ciò impedisce al tessuto di staccarsi durante il sezionamento.
NOTA: Passaggio critico: il campione immerso nel boroidruro deve essere incubato al riparo dalla luce a 4 °C. - Lavare il cubetto di agarosio con tampone PB 2 volte per 10 minuti. Girare il cubo di agarosio in modo che il tessuto si trovi nella parte superiore e attaccare il blocco a un vetrino magnetico (vetrino da microscopio con due magneti attaccati sul retro) utilizzando alcune gocce di adesivo istantaneo. La colla impiega 10-20 minuti per solidificarsi, quindi posizionare un fazzoletto di carta con gocce di tampone PB sopra l'agarosio assicurandosi che il campione rimanga adeguatamente idratato per tutto il tempo (Figura 1C).
3. Preparazione del microtomo e preparazione del terreno
- Posizionare la slitta magnetica sul metallo al centro della scatola del campione del tomografo.
- Riempire la scatola con tampone PB e posizionarla con cura sul tavolino e serrare la vite a sinistra per fissare la scatola del campione sul tavolino del tomografo.
NOTA: Non serrare eccessivamente le viti, altrimenti si danneggerà la scatola di plastica. - Posizionare la lama sul microtomo e spostare con cautela la posizione del campione vicino e sotto il microtomo. La superficie superiore dell'agarosio deve essere allo stesso livello della lama del microtomo.
NOTA: Posizionarlo troppo in basso farà perdere tempo di taglio, mentre posizionarlo troppo in alto rimuoverà una grossa fetta del blocco al primo taglio. - Sposta il tavolino per posizionare il cubo di agarosio al centro dell'obiettivo e fai clic ripetutamente sul pulsante Affetta nel software del tomografo fino a ottenere una fetta intatta dell'intero cubo di agarosio.
4. Assegnazione della superficie e acquisizione di immagini 2D
- Centrare la lente dell'obiettivo sul campione di tessuto nelle direzioni x e y.
- Aprire il software del laser e accendere il laser nel software. Quando si sente un ticchettio, accendere l'interruttore laser e regolare la lunghezza d'onda a 800 nm.
NOTA: La sorgente laser deve essere riscaldata per almeno 30 minuti, idealmente 1 ora, prima dell'imaging. - Chiudere le porte dell'armadio del microscopio, spegnere tutte le fonti di luce nella stanza, accendere i PMT e impostare la tensione a 750 V.
NOTA: Passaggio critico: i PMT sono molto sensibili alla luce, tenerla spenta a meno che le ante dell'armadio non siano chiuse e tutte le fonti di luce nella stanza siano spente. - Regolare le impostazioni del protocollo del software del tomografo come descritto e impostare l'otturatore del microscopio su automatico e la tensione su 20 e 3 rispettivamente per V1 e V2.
- Fare clic sul pulsante 2D nel software del tomografo per acquisire uno scatto del campione.
- Una volta visualizzata un'immagine con autofluorescenza visibile, regolare il piezo z per impostare il piano focale sulla superficie del cubo di agarosio. La prima scansione sarà a 20 μm sotto la superficie per acquisire un'immagine stabile del campione.
- Se non è visibile alcuno sfondo di autofluorescenza, aumentare il contrasto fino a quando non viene visualizzato un segnale. Può accadere che non ci sia alcun segnale visibile (ad esempio, a causa dell'agarosio sulla parte superiore del tessuto). In questo caso, regolare l'asse z utilizzando il dispositivo piezoelettrico z fino a raggiungere la superficie dell'agarosio. Potrebbe essere necessario un movimento manuale dell'obiettivo se la posizione desiderata è al di fuori della portata del dispositivo piezoelettrico z.
- Taglia più fette da 50 μm dopo aver trovato la superficie del campione e acquisisci immagini 2D dopo ogni taglio per assicurarti che il piano focale sia impostato correttamente sulla superficie del blocco.
5. Trovare i bordi dei campioni e impostare il laser al punto di partenza
- Sulla base delle coordinate xy dell'area di scansione laser (4 coordinate angolari), confinare l'area di imaging al tessuto con un'imaging minima del blocco di agarosio circostante.
- Spegnere i PMT, impostare la lunghezza d'onda del laser su 800 nm, aprire l'otturatore laser. La posizione della luce laser che entra nel blocco di agarosio è visibile come una macchia rossa di luce diffusa (Figura 2A, B).
Passaggio critico: prestare particolare attenzione con il laser di classe 4. Può danneggiare rapidamente i tessuti umani e gli occhi. Il personale ha bisogno di una formazione speciale per utilizzare uno strumento di questo tipo in modo sicuro. - Usa lo spot laser mentre sposti il tavolino per trovare le coordinate dei bordi del tessuto utilizzando la sequenza indietro, destra, davanti e sinistra.
- Ricontrolla le coordinate e posiziona lo spot laser nell'angolo anteriore destro, che è il punto di partenza predefinito per l'imaging del tessuto.
- Converti le coordinate dalla finestra della console in dimensioni di blocco riconoscibili dal computer, definite dai passaggi xy. Inserire queste misurazioni dei passi nel protocollo del software. Chiudere le ante dell'armadio.
6. 3D di scansione / sezionamento
- Modificare la lunghezza d'onda a 940 nm per eccitare GFP, mWasabi o YPet e PE.
NOTA: Il laser è invisibile all'occhio umano a questa lunghezza d'onda. Per migliorare il segnale-sfondo per GFP, mWasabi e il componente di emissione verde di TIMERbac, posizionare un filtro passa-banda stretto 510/20 nm davanti a PMT 2. Ciò riduce l'interferenza dell'autofluorescenza tissutale verde-gialla. - Impostare l'otturatore del microscopio in modalità automatica e le impostazioni della tensione dell'otturatore su 20 per V1 e 1,71 per V2.
- Regolare i seguenti tre parametri nel software: Spessore della sezione- per la maggior parte degli scopi, impostato su 50 μm. Per il fegato, impostare a 30 μm a causa della limitata profondità di imaging. Il numero di sezioni fisiche: determina la profondità totale del tessuto che verrà visualizzata, impostata di conseguenza. Il numero di aerei da catturare per ogni sezione: questo determinerà la risoluzione z. Utilizzare 5 piani (con una risoluzione verticale di 10 μm) per la maggior parte degli scopi. Per il fegato utilizzare 3 o meno aerei.
- Imposta il guadagno del laser. Questo deve essere testato empiricamente per diversi tessuti e profondità di scansione.
- Definire il percorso e il nome della cartella per la memorizzazione delle immagini acquisite e fornire le informazioni appropriate per il metafile di imaging.
- Ricontrolla tutti i valori inseriti.
- Fare clic sull'impostazione mosaico 3D per avviare il processo di scansione (Figura 1D).
- Al termine dell'imaging, raccogliere con cura le sezioni di tessuto dal serbatoio dell'acqua e conservarle in un tampone di conservazione a -20 °C fino a nuovo utilizzo (Figura 1E; Figura 2C, D).
7. Elaborazione delle immagini e analisi dei dati
- Scarica gli script MATLAB su un computer Linux da https://github.com/BumannLab/Li_BumannLab_2020. Copia gli script in una cartella, ad esempio /home/user/Program/.
NOTA: Il componente aggiuntivo Computer Vision Toolbox per MATLAB e Fiji (o ImageJ) deve essere installato su computer Linux affinché lo script sia completamente funzionante. - Unisci le immagini delle tessere come descritto di seguito.
- Trasferisci i dati dal server del tomografo al computer Linux.
- Apri lo script MATLAB StepOneStitchingAndArchive.m e individua la cartella di origine contenente i dati grezzi. Definire le cartelle di origine e di destinazione nello script.
- Passa alla scheda Editor e fai clic su Esegui. Le informazioni sullo stato di avanzamento vengono visualizzate nella finestra di comando. L'elaborazione include la lettura delle informazioni dal file Mosaic, la generazione dell'indice delle tessere prima dello stitching dell'immagine, la correzione dell'illuminazione di sfondo con Cidre22 (Figura 3) e la correzione dell'illuminazione tra strati ottici acquisiti a diverse profondità. Trova le immagini unite in una sottocartella chiamata stitchedImages_100 nella cartella di origine. Comprimere i dati grezzi per l'archiviazione a lungo termine utilizzando il comando tar e salvarli come file singolo con l'estensione tar.bz2 (Figura 1F).
- Esegui l'addestramento e la segmentazione del modello con la macchina vettoriale di supporto. Per addestrare la macchina a vettori di supporto ed eseguire la segmentazione, attenersi alla seguente procedura.
NOTA: Le immagini unite per ciascuno dei tre canali di fluorescenza sono memorizzate in 3 sottocartelle (denominate 1, 2 e 3, corrispondenti alla fluorescenza rossa, arancione e verde) di stitchedImages_100. Ogni sottocartella contiene tutte le immagini unite dallo stesso canale.- Visualizza l'anteprima delle immagini cucite. Apri un'immagine con lo stesso nome file da ogni sottocartella con Fiji (o ImageJ). Unisci i tre canali in un'unica immagine a colori. Regolare la luminosità di ciascun canale fino a quando non si vedono segnali chiari dai batteri e dall'autofluorescenza dei tessuti. Annotare il livello di intensità massimo regolato di ciascun canale per il passaggio successivo.
- Apri lo script MATLAB StepTwoSegmentationAndAnalysis.m e vai alla fonte. Definire la cartella di origine e i nomi delle immagini per il training. Per esempio
sourceD = '/' ;
red_name = [sourceD '1/section_020_01.tif']; green_name = [sourceD '2/section_020_01.tif'];
blue_name = [sourceD '3/section_020_01.tif']; - Vai alla scheda Editor e fai clic su Esegui.
- Apparirà una finestra di dialogo che richiede le soglie di colore, riempila in base al precedente controllo manuale con Fiji (passaggio 7.3.1).
- Seleziona le regioni per lo sfondo e le regioni di interesse (ad esempio, i batteri) in base alle finestre di dialogo. Verranno visualizzati grafici che mostrano il livello di training del modello e chiedono se è necessario aggiungere altre aree. Aggiungere altre regioni di interesse e di sfondo fino a quando i batteri possono essere chiaramente segmentati (Figura 4A, B).
- Assegna un nome al modello e salvalo. Lo script caricherà automaticamente le immagini e applicherà un filtro mediano (raggio 2 pixel).
- Il processo di segmentazione viene eseguito automaticamente e le informazioni sullo stato di avanzamento vengono visualizzate nella finestra di comando. Guarda l'andamento della segmentazione. Se la segmentazione non funziona bene con il modello su altre immagini unite, ripeti l'addestramento del modello per includere più regioni di sfondo e batteriche.
- Per vedere i risultati della segmentazione, controlla il file denominato Allpositions_filter3D.txt. Questo file contiene le coordinate del baricentro per ogni batterio o microcolonia batterica e la corrispondente intensità di fluorescenza integrata (Figura 1G).
- Rimuovere ulteriori artefatti con la rete neurale convoluzionale come descritto di seguito.
NOTA: Per alcuni batteri, la semplice segmentazione con la Support Vector Machine include ancora alcuni artefatti di imaging. Ciò è particolarmente vero per i batteri che esprimono YPet. La segmentazione basata su reti neurali può rimuovere in modo efficiente questi artefatti (Figura 4C). L'add-on Computer Vision Toolbox per MATLAB e Fiji (o ImageJ) deve essere installato su computer Windows affinché lo script sia completamente funzionante. Per informazioni sulla rete neurale sottostante vedere: https://ch.mathworks.com/help/deeplearning/ug/create-simple-deep-learning-network-for-classification.html- Prepara le immagini. Converti i file tif in file jpg con diverse regolazioni della luminosità utilizzando le Fiji. Identifica con la cura manuale almeno 600 batteri e 600 artefatti e salva le immagini ritagliate di questi oggetti in cartelle appropriate.
- Aprire lo script MATLAB CNNtestV4.m e individuare i file di origine contenenti le immagini degli oggetti del passaggio 7.4.1.
- Definire la cartella di immagini per il training. Per esempio
digitDatasetPath = fullfile ('D:\20200602_CNN','fortrainingPixel12Folder2');
Definisci il numero di immagini per l'addestramento, ad esempio numTrainFiles = 600. - Vai alla scheda Editor e fai clic su Esegui. Dopo l'addestramento, il file del modello CNN viene salvato come gregnet1.mat nella cartella di origine.
- Definire una cartella con le immagini di prova. Per esempio
digitDatasetPathTest = fullfile('D:\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
I risultati vengono salvati in un file YourFile.txt che contiene informazioni sul nome dell'immagine e sulla classificazione come batterio o artefatto. - Definisci la cartella con le immagini di prova. Per esempio
digitDatasetPathTest = fullfile('D:\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
I risultati verranno salvati in un file YourFile.txt, che contiene informazioni sul nome dell'immagine e sulla classificazione come batteri o artefatti.
- Stimare la dimensione delle microcolonie in base all'intensità di fluorescenza dei singoli batteri come descritto di seguito.
NOTA: La risoluzione spaziale del tomografo è insufficiente per risolvere le singole cellule batteriche all'interno di microcolonie densamente impacchettate. Tuttavia, il numero di batteri in ciascuna microcolonia può essere stimato in base alla fluorescenza totale della microcolonia in relazione alla fluorescenza dei singoli batteri. L'accuratezza di questa stima dipende dall'omogeneità dell'intensità della fluorescenza nella popolazione batterica.- Confermare l'identità degli eventi GFP-positivi nella tomografia STP colorando i componenti batterici come il lipopolisaccaride utilizzando l'immunoistochimica delle sezioni recuperate dal tomografo (Figura 1H; Figura 5).
- Identificare almeno 30 singoli batteri e recuperare la loro intensità di fluorescenza dai risultati della segmentazione corrispondenti. Utilizzare il valore di intensità mediana come riferimento per un singolo batterio. Calcolare il numero batterico di ciascuna microcolonia dividendo l'intensità totale di fluorescenza della microcolonia per il valore di riferimento per un singolo batterio (Figura 1I).
- Esegui la ricostruzione 3D con il software di visualizzazione come descritto di seguito.
- Preparazione dell'immagine
- Prendiamo le immagini del canale blu, che contengono segnali di seconda armonica del collagene fornendo un utile riferimento anatomico tra cui capsule tissutali, arterie e trabecole. Utilizzare immagini segmentate per i batteri come 2° canale.
- Raccogli le immagini dal 1° canale di 10 volte su entrambi gli assi x e y e salva le immagini ridimensionate in una nuova cartella. Crea 3 repliche di immagini ridimensionate all'interno della stessa cartella. Il nome delle nuove repliche deve mantenere la stessa sequenza. Ad esempio: section_001_01.tif, replica 1 con nome section_001_01-copy.tif, section_001_01-copy-copy.tif. Applicare questi passaggi anche alle immagini dal 2° canale. Il ridimensionamento produce file di dimensioni più gestibili. Le immagini triple smusseranno l'asse z.
- Unisci le immagini di ciascun canale per formare pile di immagini con le Fiji. Fare clic su Immagine > Pile > Strumenti > Ordinatore pile > Ordina per etichette.
- Eseguire il filtraggio 3D. Fare clic su Elabora filtri > > filtro 3D e impostare la dimensione del filtro Z su 6. Salva le pile di immagini.
- Eseguire la visualizzazione 3D come descritto di seguito.
- Aprire il pacchetto software di visualizzazione nella vista Arena (impostazione predefinita). Usa l'icona Cartella di controllo per aggiungere cartelle alla vista Arena. Fare doppio clic per aprire il file *.ims o *.tif (preparato nella versione 7.6.1).
- Utilizzando le proprietà Modifica>Immagine, regolare le rappresentazioni dei colori (LUT) dei diversi canali nella finestra Regolazione visualizzazione.
- Fare clic su Avanzate per impostare manualmente i valori min/max e un valore per la correzione gamma.
Fare clic sui nomi dei canali per modificare i nomi e le LUT. - Dopo aver regolato l'aspetto dell'immagine, esportate la vista corrente utilizzando lo strumento Istantanea.
- Utilizzare l'icona Animazione per rappresentare i dati 3D come un filmato. Usa il puntatore di navigazione per trovare una prospettiva/vista e ingrandisci e usa + Aggiungi per aggiungere fotogrammi chiave. Spostati in un'altra posizione e aggiungi il fotogramma chiave successivo. Premi il pulsante rosso di registrazione per creare il filmato. Salvarlo nella cartella di destinazione e nel tipo di file desiderati (Figura 1J).
- Preparazione dell'immagine
Risultati
La procedura descritta consente il rilevamento di singole cellule di Salmonella in interi organi di topo come milza, fegato, linfonodi mesenterici e cerotti di Peyer11 (Figura 5 e Figura 6). Rileva anche i parassiti Toxoplasma gondii nel cervello del topo12. Alcuni tessuti infetti, tra cui il fegato, le chiazze di Peyer e la milza, emettono una sostanziale autofluorescenza nell'intervallo verde-giallo. L'autofluorescenza è ulteriormente potenziata dalla fissazione con paraformaldeide, necessaria per preservare la struttura tissutale. La rilevazione della fluorescenza verde da GFP, mWasabi e la componente verde di TIMERbac su questo sfondo di autofluorescenza è migliorata mettendo un filtro passa-banda stretto a 510/20 nm (che trasmette la maggior parte delle emissioni di GFP ma blocca gran parte dello spettro di autofluorescenza) davanti al fotomoltiplicatore 2 (che raccoglie le emissioni verdi) e riducendo l'autofluorescenza tissutale conservando i tessuti fissati per 3 o più giorni in crioprotettore11. Tuttavia, i batteri dovrebbero comunque esprimere almeno alcune migliaia di copie per cellula di GFP o altre proteine fluorescenti. D'altra parte, livelli eccessivi di proteine fluorescenti dovrebbero essere evitati per ridurre al minimo i costi di fitness che potrebbero portare a una virulenza attenuata23.
La corretta segmentazione dei batteri fluorescenti nei tessuti può essere confermata dall'immunoistochimica delle sezioni di tessuto recuperate dopo l'imaging. In particolare, gli oggetti colorati con un anticorpo contro i componenti batterici della superficie come il lipopolisaccaride possono essere allineati con l'immagine di fluorescenza ottenuta mediante tomografia STP (Figura 5). È importante notare che alcune cellule di Salmonella colorate mancano di proteine fluorescenti e quindi di fluorescenza rilevabile sia nella microscopia confocale che nella tomografia STP. Queste cellule sono Salmonella che sono state uccise dal sistema immunitario dell'ospite, come dimostrato dalla selezione citofluorimetrica e dalle colture di crescita da singole cellule selezionate, nonché dalla stretta correlazione tra il numero di unità formanti colonie su piastre di agar e il numero di cellule fluorescenti di Salmonella come determinato dalla citometria a flusso24. Inoltre, la perdita di plasmidi può portare a cellule vitali non fluorescenti e questo deve essere testato mediante placcatura su terreni con e senza antibiotici appropriati corrispondenti al marcatore di selezione sul plasmide. Per i plasmidi derivati da pSC101, la perdita di plasmidi in vivo è rara19. Per la maggior parte delle cassette di espressione cromosomicamente integrate come sifB::gfp utilizzate in11, la perdita di espressione non è rilevabile in vivo. Se la segmentazione non è coerente con i dati immunoistochimici, la pipeline di segmentazione deve essere modificata.
La risoluzione della tomografia STP è insufficiente per risolvere le singole cellule batteriche all'interno di microcolonie densamente impacchettate. Tuttavia, l'intensità totale della fluorescenza della microcolonia consente di stimare il numero di cellule di Salmonella . Ciò richiede un ceppo fluorescente con livelli di fluorescenza altamente omogenei come Salmonella sifB::gfp11. Combinando il numero stimato di cellule di Salmonella per tutte le microcolonie e le singole cellule si ottengono cariche tissutali batteriche totali coerenti con metodi alternativi come la piastratura o la citometria a flusso di omogeneizzati tissutali11. La piastratura e la citometria a flusso non possono essere eseguite direttamente dagli stessi tessuti perché devono essere fissate per perfusione per la tomografia STP. Invece, devono essere fatti con animali aggiuntivi che non sono fissi. Se le cariche batteriche mediane, come determinato dai vari approcci, differiscono di oltre 3 volte, la vitalità dei batteri fluorescenti potrebbe essere compromessa (nel caso di unità formanti colonie inferiori) o alcuni batteri potrebbero aver perso il costrutto reporter fluorescente (nel caso di unità formanti colonie più elevate). Sarà necessario un esperimento di controllo per identificare la fonte di tali discrepanze.
La tomografia STP fornisce la localizzazione delle cellule batteriche all'interno della struttura 3D dei tessuti infetti. I secondi segnali armonici del collagene forniscono punti di riferimento anatomici come arterie e trabecole. Inoltre, le cellule ospiti possono essere colorate in vivo iniettando un anticorpo ai marcatori di superficie prima della perfusione (Figura 5 e Figura 6). Questa colorazione fornisce ulteriori punti di riferimento per i compartimenti tissutali e i microambienti specifici, compresi i focolai di infiammazione (i marcatori intracellulari e alcuni compartimenti con barriere diffusionali come la polpa bianca splenica o il cervello non sono facilmente accessibili, adatti a questa colorazione in vivo ). Questo approccio ha rivelato che la polpa bianca splenica è un compartimento tissutale che consente la sopravvivenza a lungo termine della Salmonella durante la chemioterapia antimicrobica11.
Infine, la replicazione della Salmonella a velocità moderate o lente può essere identificata e localizzata all'interno della struttura 3D dei tessuti utilizzando ceppi che esprimono il timerbac, un reporter a singola cellula per una velocità di replicazionedi 11,15.
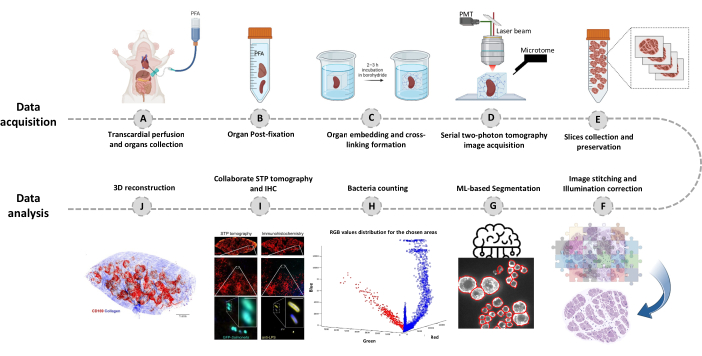
Figura 1: Procedura per l'imaging dell'intero organo mediante tomografia seriale a due fotoni (STP). (A-B) L'organo viene prelevato dopo perfusione transcardica e conservato per una notte in paraformaldeide al 4% (PFA) a 4 °C. (C-E) L'organo viene incorporato in agarosio ossidato e reticolato, quindi il tessuto viene scansionato e affettato utilizzando la tomografia STP. Le fette vengono raccolte per la successiva immunoistochimica. (F-J) Pipeline di analisi computazionale per la quantificazione del numero di batteri, la conferma di batteri fluorescenti e la ricostruzione 3D delle posizioni batteriche. Clicca qui per visualizzare una versione più grande di questa figura.

Figura 2: Configurazione della tomografia seriale a due fotoni (STP). (A) Tomografo che incorpora (B) imaging a 2 fotoni con (C) sezionamento seriale automatizzato dei tessuti. (D) Sezioni di tessuto raccolte per indagini di follow-up. Clicca qui per visualizzare una versione più grande di questa figura.
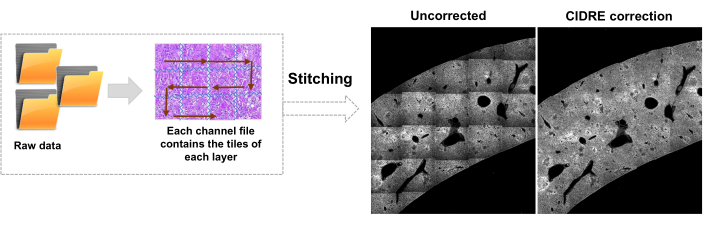
Figura 3: Cucitura delle piastrelle e correzione dell'illuminazione. Le piastrelle vengono cucite e l'illuminazione irregolare viene corretta utilizzando distribuzioni di intensità corrette utilizzando la minimizzazione dell'energia regolarizzata (CIDRE). Clicca qui per visualizzare una versione più grande di questa figura.
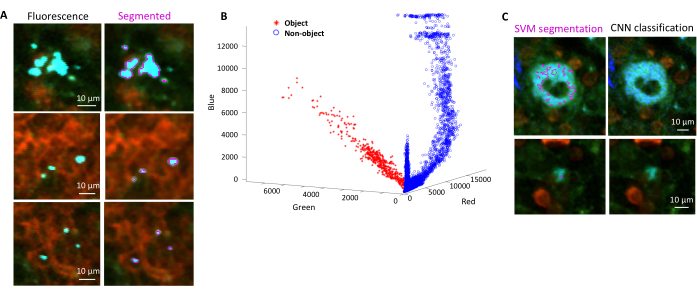
Figura 4: Segmentazione di Salmonella che esprime la proteina fluorescente verde (GFP) utilizzando una Support Vector Machine (SVM) e una rete neurale convoluzionale (CNN). (A) Immagini rappresentative di oggetti GFP segmentati da SVM (a sinistra) e immagini corrispondenti (a destra) con regioni segmentate da SVM (barra di scala: 10 μm). (B) Distribuzione dei valori raggruppati rosso-verde-blu (RGB) per le regioni segmentate. (C) Immagini rappresentative di oggetti non GFP erroneamente identificati dalla SVM come batteri (a sinistra). La CNN li liquida correttamente come sfondo (a destra, barra della scala: 10 μm). Clicca qui per visualizzare una versione più grande di questa figura.
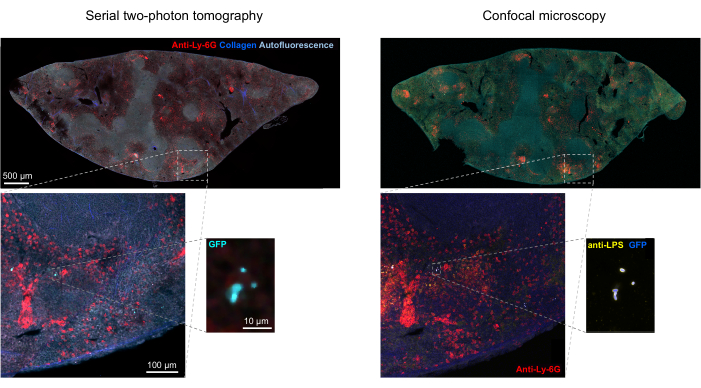
Figura 5: Rilevamento di Salmonella che esprime la proteina fluorescente verde (GFP) mediante tomografia e conferma mediante immunoistochimica. Immagini della stessa sezione acquisite mediante tomografia (a sinistra) o microscopia confocale dopo colorazione con un anticorpo contro il lipopolisaccaride di Salmonella (a destra). I neutrofili (rosso) sono stati colorati mediante iniezione in vivo di un anticorpo anti-Ly-6G marcato con PE prima della perfusione. Clicca qui per visualizzare una versione più grande di questa figura.
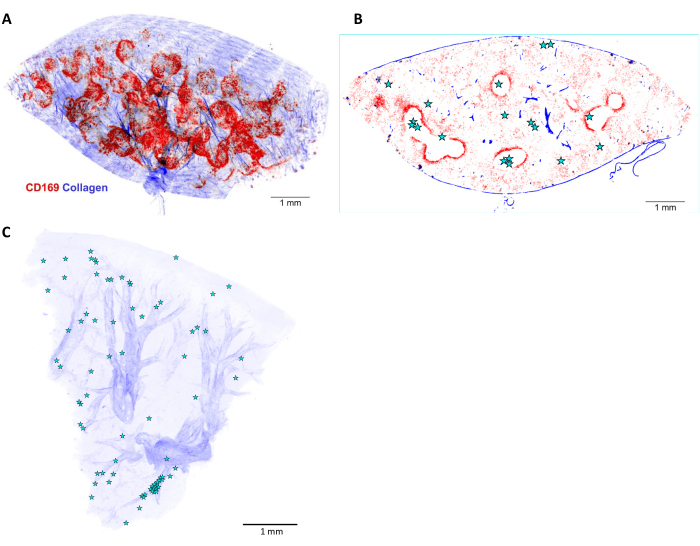
Figura 6: Ricostruzione 3D e localizzazione di Salmonella nella milza di topo infetta. (A) Ricostruzione tridimensionale (3D) di una fetta di milza di 5 mm di spessore e colorata in vivo prima della perfusione con anticorpo anti-CD169 (rosso). Il segnale blu rappresenta il collagene rilevato dalle seconde armoniche. (B) Un piano ottico della pila 3D mostrata in (A). Le posizioni delle cellule o delle microcolonie di Salmonella sono indicate da stelle. (C) Ricostruzione 3D delle posizioni (stelle) della Salmonella nel fegato infetto. Le arterie sono visibili in base alle loro guaine di collagene (blu). Barra della scala: 1 mm. Clicca qui per visualizzare una versione più grande di questa figura.
Discussione
Il contesto tissutale locale dei patogeni batterici è cruciale per determinare gli attacchi locali dell'ospite, gli adattamenti batterici, l'esito locale delle interazioni tra patogeni dell'ospite e la chemioterapia antimicrobica e i contributi individuali all'esito complessivo della malattia. L'imaging di batteri di dimensioni micrometriche in organi di dimensioni centimetriche è stato impegnativo. La tomografia seriale a due fotoni (STP) fornisce una risoluzione spaziale sufficiente per rilevare singole cellule batteriche in interi organi, sezionamento e imaging automatizzati e una produttività sufficiente (~1 organo al giorno)11. Mentre gli antigeni dell'ospite possono essere colorati in vivo, le cellule patogene dovrebbero esprimere proteine fluorescenti adeguate per garantire il rilevamento completo delle cellule patogene intracellulari. I set di dati risultanti (0,5-1,5 TeraByte per organo) pongono sfide sostanziali alle infrastrutture IT per l'analisi e l'archiviazione dei dati.
Ci sono diversi passaggi critici in questo metodo. In primo luogo, è necessario un ceppo patogeno con espressione rilevabile e omogenea della proteina fluorescente GFP o YFP. Idealmente, una cassetta di espressione cromosomica25 viene utilizzata per ridurre al minimo l'eterogeneità della fluorescenza dovuta alla variazione del numero di copie del plasmide. È richiesta un'intensità di fluorescenza sufficiente, ma devono essere evitati livelli eccessivi di proteina fluorescente per evitare compromessi di fitness del patogeno23. Livelli di espressione appropriati possono essere ottenuti selezionando un promotore appropriato e mettendo a punto il sito di legame ribosomiale25 o l'intera regione 5' non tradotta (UTR)26. In secondo luogo, la fissazione della perfusione dovrebbe comportare un lavaggio iniziale con tampone per rimuovere il maggior numero possibile di eritrociti dalla circolazione sanguigna. Questo è particolarmente critico per la milza e il fegato (anche se la rimozione completa degli eritrociti da questi organi è difficile). Gli eritrociti rimanenti assorbono la luce nella parte visibile dello spettro, compromettendo la qualità dell'imaging27. In terzo luogo, la conservazione dei tessuti fissati nel crioprotettore è fondamentale per ridurre l'autofluorescenza tissutale, che è particolarmente elevata nei tessuti infiammati e può mettere in ombra la fluorescenza relativamente debole delle cellule patogene11. In quarto luogo, l'efficace reticolazione del tessuto con il blocco di agarosio circostante è fondamentale per un taglio regolare del vibratomo senza che il tessuto salti fuori dal blocco di agarosio. In quinto luogo, i segnali fluorescenti e la loro identificazione come cellule patogene devono essere verificati in modo indipendente utilizzando approcci ortogonali come la colorazione con anticorpi contro componenti patogeni (come il lipopolisaccaride per i batteri Gram-negativi) e la microscopia confocale delle sezioni recuperate dal tomografo11. Alcuni tessuti infetti contengono particelle autofluorescenti con forma simile e spettri di fluorescenza sovrapposti che possono essere facilmente interpretati erroneamente come cellule patogene. In sesto luogo, la quantità di cellule patogene all'interno delle microcolonie dovrebbe essere confrontata con approcci ortogonali come la microscopia confocale per valutarne l'accuratezza. Le cariche batteriche complessive basate su questi calcoli devono essere verificate confrontandole con approcci ortogonali come la citometria a flusso e la piastra.
Importanti modifiche al protocollo STP, ampiamente utilizzato, includono il posizionamento di un filtro passa-banda stretto a 510/20 nm davanti al fotomoltiplicatore 211, per ridurre l'interferenza dell'autofluorescenza verde-gialla che è particolarmente forte nel fegato, nella milza e nelle chiazze di Peyer infette e infiammate. La forte autofluorescenza e l'aumento della dispersione della luce di tali organi rispetto al cervello (che domina altre applicazioni della STP) generano anche la necessità di una correzione più efficace per l'illuminazione irregolare. Come altra modifica, questo protocollo impiega l'approccio CIDRE22 per questo scopo (Figura 3) e la segmentazione dei batteri basata sull'intelligenza artificiale. Infine, il pretrattamento tissutale è stato modificato includendo una fase di incubazione nel crioprotettore a -20 °C che riduce l'autofluorescenza tissutale e quindi facilita il rilevamento di piccole cellule patogene con fluorescenza relativamente debole11.
La risoluzione dei problemi potrebbe essere necessaria se non è possibile rilevare alcun segnale patogeno o se la segmentazione produce una sensibilità insufficiente (troppe cellule patogene vengono perse) o una precisione insufficiente (troppe particelle di fondo vengono segmentate come cellule patogene). Se l'autofluorescenza tissutale di fondo è rilevabile ma ci sono troppo pochi segnali patogeni, i patogeni potrebbero contenere quantità insufficienti di proteine fluorescenti. Questo può essere testato utilizzando la microscopia confocale di sezioni di tessuto dello stesso tessuto infetto o la citometria a flusso di omogeneizzati tissutali19,28. Le ragioni sottostanti potrebbero essere livelli di espressione insufficienti o instabilità della cassetta di espressione. Le strategie di mitigazione potrebbero includere promotori alternativi per guidare l'espressione, l'adattamento dei codoni dei geni che codificano la proteina fluorescente per le specie patogene, l'impiego di costrutti episomiali con un numero di copie più elevato o la stabilizzazione delle cassette di espressione mediante integrazione cromosomica o complementazione bilanciata-letale29. Anche la scelta della proteina fluorescente è importante, ma il rilevamento è possibile con GFP.mut2, mWasabi, YPet e TIMERbac. Se la segmentazione è imprecisa, ciò potrebbe essere causato da una fluorescenza del patogeno troppo debole che potrebbe essere affrontata come descritto sopra, o da un fondo di autofluorescenza tissutale troppo elevato. Un'estesa perfusione della soluzione di lavaggio o un'incubazione prolungata nel tampone di conservazione immediatamente prima dell'inclusione nel blocco di agarosio e nella tomografia potrebbero risolvere questi problemi. Infine, è necessario un addestramento sufficiente della rete neurale per una classificazione precisa, ma un addestramento eccessivo può portare a un overfitting che compromette le prestazioni per i nuovi campioni.
Attualmente, nessun altro metodo è in grado di visualizzare interi organi con una risoluzione spaziale sufficiente in 3D per rilevare singoli batteri. I futuri miglioramenti nella pulizia dei tessuti e nella microscopia a foglio di luce potrebbero raggiungere una risoluzione simile. Ciò potrebbe consentire l'imaging a velocità più elevata e con più canali fluorescenti.
Un'importante limitazione dell'STP è la risoluzione dei pixel nel piano di ~0,5 μm e la risoluzione verticale da 5 a 10 μm, che è insufficiente per risolvere batteri vicini, ad esempio all'interno di una microcolonia densamente compatta. Tuttavia, è possibile recuperare sezioni di tessuto dopo la tomografia per la microscopia confocale secondaria ad alta risoluzione di parti di tessuto selezionate. Un'altra limitazione dell'STP è la disponibilità di soli tre canali di fluorescenza, che limita il numero di fluorofori che possono essere visualizzati contemporaneamente. Anche in questo caso, l'analisi secondaria delle sezioni di tessuto recuperate con metodi di multiplexing può rivelare la posizione e l'intensità di molti più marcatori per parti di tessuto selezionate. Queste informazioni potrebbero essere integrate nella struttura 3D complessiva del tessuto circostante come determinato con STP.
In conclusione, questo protocollo consente indagini dettagliate sulle interazioni ospite-patogeno a livello locale e di intero organo. Il protocollo dovrebbe essere facilmente adattabile ad altri agenti patogeni (a condizione che possano essere ottenuti come ceppi fluorescenti), ad altri organi e a diverse specie ospiti.
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Il lavoro è stato sostenuto dal Fondo nazionale svizzero per la ricerca scientifica 310030_156818, 310030_182315 e da NCCR_ 180541 AntiResist (to DB).
Materiali
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agarose Low Melt | Roth | Art. 6351.5 25g | |
| Boric acid | Sigma-Aldrich | 6768-500G | |
| Instant adhesive Loctite 435 | Henkel | ||
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Poly(ethylene glycol) | Sigma-Aldrich | P5413-1kg | |
| Polyvinylpyrrolidone | Sigma-Aldrich | PVP-100G | |
| Sodium borohydride | Sigma-Aldrich | 71321-25g | |
| Sodium hydroxide | Merck | 106453 | |
| Sodium periodate | Sigma-Aldrich | 311448-100G | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640-250G | |
| Sodium phosphate monobasic dihydrate | Sigma-Aldrich | 71500-1KG | |
| Sodium tetraborate | Sigma-Aldrich | 221732-100g | |
| Sucrose | AppliChem | A4734,1000 | |
| Tris-buffered saline (TBS) | Merck | T5912-1L | |
| Triton X-100 | Sigma-Aldrich | 9002-93-1 | |
| Vacuum filtration 500 | TPP | TPP99250 | |
| Equipment | |||
| Blade | Campden Instruments Limited | 01-01-4692 | |
| MAITAI Laser | Spectra-Physics | ||
| Peel away plastic mold | Sigma-Aldrich | E6032-1CS | |
| TissueCyte 1000 tomograph | TissueVision | ||
| Antibody/dyes | |||
| DAPI | Merck | D9542-5MG | |
| Primary antibodies | |||
| anti-LPS Salmonella, rabbit | Sifin | REF TS 1624 | |
| anti-CD169-PE, clone 3D6.112 | Biolegend | 142403 | |
| anti-Ly-6G-PE, clone 1A8 | Biolegend | 127608 | |
| Secondary antibodies | Invitrogen | ||
| chicken anti-rabbit Alexa 647 | Invitrogen | A-21443 | |
| Software | Company | Version | |
| Fiji | Image J | 1.54g or later | |
| MATLAB | MathWorks | 2017b/2018b or later | |
| Orchestrator (tomograph) | TissueVision | ||
| Visualization software Imaris | Oxford Instruments | 9.9.0 or later |
Riferimenti
- Bjarnsholt, T., et al. The importance of understanding the infectious microenvironment. Lancet Infect Dis. 22 (3), e88-e92 (2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Bumann, D., Cunrath, O. Heterogeneity of Salmonella-host interactions in infected host tissues. Curr Opin Microbiol. 39, 57-63 (2017).
- Hofmann, J., Keppler, S. J. Tissue clearing and 3D imaging - putting immune cells into context. J Cell Sci. 134 (15), jcs258494(2021).
- Blain, R., et al. A tridimensional atlas of the developing human head. Cell. 186 (26), 5910-5924.e17 (2023).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Meth. 9 (3), 255-258 (2012).
- Oh, S. W., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Kim, Y., et al. Brain-wide maps reveal stereotyped cell-type-based cortical architecture and subcortical sexual dimorphism. Cell. 171 (2), 456-469.e422 (2017).
- Matho, K. S., et al. Genetic dissection of the glutamatergic neuron system in cerebral cortex. Nature. 598 (7879), 182-187 (2021).
- Muñoz-Castañeda, R., et al. Cellular anatomy of the mouse primary motor cortex. Nature. 598 (7879), 159-166 (2021).
- Li, J., et al. Tissue compartmentalization enables Salmonella persistence during chemotherapy. Proc Natl Acad Sci U S A. 118 (51), e2113951118(2021).
- Dogga, S. K., et al. Importance of aspartyl protease 5 in the establishment of the intracellular niche during acute and chronic infection of Toxoplasma gondii. Mol Microbiol. 118 (6), 601-622 (2022).
- Cormack, B. P., Valdivia, R. H., Falkow, S. FACS-optimized mutants of the green fluorescent protein (GFP). Gene. 173 (1 Spec No), 33-38 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Claudi, B., et al. Phenotypic variation of Salmonella in host tissues delays eradication by antimicrobial chemotherapy. Cell. 158 (4), 722-733 (2014).
- Ai, H. W., Olenych, S. G., Wong, P., Davidson, M. W., Campbell, R. E. Hue-shifted monomeric variants of Clavularia cyan fluorescent protein: identification of the molecular determinants of color and applications in fluorescence imaging. BMC Biol. 6, 13(2008).
- Drobizhev, M., Makarov, N. S., Tillo, S. E., Hughes, T. E., Rebane, A. Two-photon absorption properties of fluorescent proteins. Nat Methods. 8 (5), 393-399 (2011).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Burton, N. A., et al. Disparate impact of oxidative host defenses determines the fate of Salmonella during systemic infection in mice. Cell Host&Microbe. 15 (1), 72-83 (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio Protoc. 11 (5), e3988(2021).
- de Olmos, J., Hardy, H., Heimer, L. The afferent connections of the main and the accessory olfactory bulb formations in the rat: an experimental HRP-study. J Comp Neurol. 181 (2), 213-244 (1978).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nat Methods. 12 (5), 404-406 (2015).
- Wendland, M., Bumann, D. Optimization of GFP levels for analyzing Salmonella gene expression during an infection. FEBS Lett. 521 (1-3), 105-108 (2002).
- Barat, S., et al. Immunity to intracellular Salmonella depends on surface-associated antigens. PLoS Pathog. 8 (10), e1002966(2012).
- Rollenhagen, C., Sorensen, M., Rizos, K., Hurvitz, R., Bumann, D. Antigen selection based on expression levels during infection facilitates vaccine development for an intracellular pathogen. Proc Natl Acad Sci U S A. 101 (23), 8739-8744 (2004).
- Chen, F., Cocaign-Bousquet, M., Girbal, L., Nouaille, S. 5'UTR sequences influence protein levels in Escherichia coli by regulating translation initiation and mRNA stability. Front Microbiol. 13, 1088941(2022).
- Weissleder, R. A clearer vision for in vivo imaging. Nat Biotechnol. 19 (4), 316-317 (2001).
- Bumann, D. Examination of Salmonella gene expression in an infected mammalian host using the green fluorescent protein and two-colour flow cytometry. Mol.Microbiol. 43 (5), 1269-1283 (2002).
- Nakayama, K., Kelly, S. M., Curtiss III, R. J. B. t Construction of an Asd+ expression-cloning vector: stable maintenance and high level expression of cloned genes in a Salmonella. vaccine strain. 6 (6), 693-697 (1988).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon