Method Article
微生物感染症の高解像度三次元全臓器断層撮影法
要約
ここでは、感染時の病原菌の臓器全体にわたる検出と蛍光レポーター活性の定量化を可能にする手順について説明します。
要約
ほとんどの感染症は、複雑な解剖学的構造と局所的に変化する宿主生理機能を持つ三次元の宿主組織内で発生します。この多様な環境内での病原体細胞の位置付けは、病原細胞のストレスレベル、反応、運命、および疾患の全体的な進行と治療の失敗への寄与に大きく影響します。しかし、cmサイズの宿主臓器内にμmサイズの病原体細胞を位置特定することは技術的な困難さのため、この研究分野は比較的未開拓でした。ここでは、この課題を解決する方法を紹介します。私たちは、連続2光子断層撮影法とAI強化画像解析を用いて、感染マウスの脾臓全体、肝葉、リンパ節全体にわたって個々の サルモネラ 細胞を特定します。蛍光レポーターと in vivo 抗体の投与により、単一 サルモネラ 菌細胞の複製速度、特定の免疫細胞との局所的な相互作用、および抗生物質に対する細菌の応答を決定できます。これらの方法論は、3次元組織コンテキスト内での感染症、その予防、および治療の包括的な調査への道を開きます。
概要
感染は、複雑な解剖学的構造と区画化された生理機能を持つ組織で発生します。感染組織に共存する多様な微小環境は、局所的な病原体サブセットの運命と、それらが全体的な疾患転帰に寄与することを決定することができる1,2,3。しかし、cmサイズの組織における微生物病原体の包括的な3Dマッピングは依然として困難です4。脳やその他の臓器のイメージングは、実験戦略5を絶えず改善している非常に活発な研究分野ですが、多くの方法では、μmサイズの細菌性病原体を自信を持って特定するために必要なサブμmの分解能が依然として不足しています。対照的に、シリアル2光子(STP)トモグラフィー6は、サブμmの面内分解能で組織全体の自動マルチカラー変形フリーイメージングを可能にし、完全な体積データセットを生成します。この方法は、ビブラトームを使用した組織の繰り返しの物理的切片化と、赤外光による出現するブロック面の断続的な2光子イメージングを組み合わせたものです。STPトモグラフィーは、脳内の細い軸索をマッピングして接続マップを確立するために広く使用されています7,8,9,10。
STPトモグラフィーは、トモグラフィーを使用して、感染組織全体の個々の微生物病原体細胞(サルモネラ菌、トキソプラズマ)の3Dマッピングも可能にします11,12。第2高調波生成は、動脈の周りや脾臓の小柱などの線維性バンドにコラーゲン鞘を明らかにし、解剖学的状況を提供します。in vivoで注入した蛍光抗体は、宿主細胞の染色に使用でき、個々の病原体細胞と好中球などの浸潤免疫細胞との間の相互作用を明らかにすることができます。ここでは、組織の処理、イメージング、照明補正によるイメージングタイルのスティッチング、3次元での画像の積層、機械学習ツールを使用したセグメンテーションを含むパイプラインについて説明します。このパイプラインは、個々の病原体、細胞、およびマイクロコロニーの宿主コンテキスト内の3D位置を生成します。マイクロコロニー内の個々の細胞の数を数えることは、解像度の限界のために依然として困難ですが、そのような数はマイクロコロニーの統合された明るさに基づいて推定することができます。このパイプラインは、組換えGFPまたはYFP発現病原体が利用可能な場合、他の感染モデルに容易に適応させることができます。
プロトコル
ここに記載されているすべての動物実験は、当局によって承認されており(ライセンス2239、Kantonales Veterinäramt Basel)、現地のガイドライン(Tierschutz-Verordnung、バーゼル)およびスイスの動物保護法(Tierschutz-Gesetz)に従っています。
1. 感染組織の調製と保管
- 選択した感染モデルを使用します。ここでは、雌雄ともに10〜16週齢のマウスに、静脈内注射により~1,000コロニー形成単位(CFU)を、丸い針による胃内投与により5×107CFU を感染させます。適切なマウス系統には、BALB/cおよびC57BL/6が含まれます。
注:同じアプローチは、他の宿主種にも簡単に適応できます。 - 病原体が蛍光タンパク質を発現し、未染色組織での同定を可能にすることを確認します。GFP.mut213、YPet14、TIMERbac15、またはmWasabi16を発現する病原体は、全て940nmの励起17を用いて可視化することができる。フローサイトメトリー19で容易に検出できるレベルでmCherry18を発現するサルモネラ菌は、800 nmの励起を使用してイメージングできますが、感染した脾臓と肝臓ではS/S/Backgroundの比率が低く、かなりの自家蛍光を示します。
- フローサイトメトリーによる定量に基づいて、パラホルムアルデヒドを中性pHで固定した後、すべての蛍光タンパク質が蛍光強度の>70%を保持することを確認します。ここでは、感染したマウス組織、すなわち脾臓、肝臓、リンパ節、およびパイエル板が使用されます。
- 灌流の10分前にフィコエリトリン(PE)標識抗体を注入することにより、 in vivo で宿主細胞表面マーカーを染色します。好中球を染色するには、100 μL の PBS に 4 μg の抗 Ly-6G-PE を静脈内注入します。脾臓の辺縁領域マクロファージを染色するには、100 μL の PBS に 4 μg の抗 CD169-PE を注入します ( 材料の表を参照)。
- 15 mLの氷冷50 mMリン酸緩衝液(PB;10 mM NaH2PO4、40 mMNa2HPO4、pH 7.4)で標準的な心血管灌流20で感染組織を固定し、続いてPBに35 mLの4%パラホルムアルデヒド(PFA;固定剤)をPBで固定します(図1A)。
- 簡単に言えば、100 mg / kgケタミンと16 mg / kgキシラジンの腹腔内注射により、感染した動物に深部麻酔を投与します。つま先つま先挟み反射の喪失を確認することにより、適切な麻酔を確保します。
- 70%エタノールを使用して毛皮を消毒し、滅菌ペーパータオルで乾かし、ピンセットとはさみで胸を外科的に開きます。心臓の左心室に滅菌針を留置し、滅菌ハサミで右心房を開き、ポンプを使用して最初のバッファーと固定剤を針、左心室、および血管系全体に流します。これにより、すべての組織が迅速かつ均一に固定され、安楽死を引き起こします。
- 灌流後に組織を採取し、PBバッファー中の20倍容量の4%冷パラホルムアルデヒドでインキュベートし、4°Cのシェーカーに一晩置きます(図1B)。0.05〜2cm3のサイズ範囲の組織に対してイメージングが可能です。大きな組織の場合は、適切な部分にカットします。
- 翌日、ティッシュをPBバッファーでそれぞれ10分間3回洗浄し、室温で40rpmで振とうしてPFAを除去します。
注:PFAの毒性のため、これらの手順はドラフト内で実行する必要があります。PFA廃棄物は、機関のガイドラインに従って処分してください。 - 余分なPBバッファーを取り除き、20倍の量の凍結保護剤保存バッファー21 (876 mMスクロース、0.25 mMポリビニルピロリドン、40%(v / v)エチレングリコール、50 mM PBバッファーに溶解)をサンプルに加え、4°Cで40 rpmで6〜8時間、または組織が底に沈むまで振とうします。サンプルを-20°Cで少なくとも3日間インキュベートして、組織の自家蛍光を効果的に減少させます11。サンプルは、-20°Cで保存した場合、最大5年間使用できます。
注:組織の沈下は、保存バッファーが組織に適切に浸透していることを示しています。
2. サンプルの埋め込み
- 組織をアガロースブロックに埋め込むと、ビブラトームでスムーズに切断できます。アガロースを過ヨウ素酸で酸化して化学的に活性化し、次に水素化ホウ素で還元して組織との架橋を形成し、切断プロセス中の安定性を向上させます。以下で説明する手順を実行します。
- 2.25 gのアガロースを秤量して4%酸化アガロースを調製し、0.21 gのNaIO4を加えます。100 mLのPBをアガロースとNaIO4に加えます。ヒュームフード内で室温で2〜3時間攪拌し、光から保護します。
注:重要なステップ:3時間以上攪拌しないと、アガロースの重合が不十分になります。 - アガロースを0.2 μmのメンブレンを備えた標準的な真空フィルターでメンブレンろ過により回収します。残りのNaIO4 を50 mLのPBバッファーで3回洗浄して除去します。酸化したアガロースを50 mLのPBバッファーに再懸濁します。
注意: この段階ではアガロースを溶かさないでください。アガロースは、光から保護されて、最大2〜3週間4°Cで保存できます。 - 19gのホウ砂と3gのホウ酸を1Lの水に加えて、水素化ホウ素溶液を調製します。溶解するまで撹拌し、1 M NaOHでpHを9〜9.5に調整します。ドラフト内の攪拌プレートで、100 mLのホウ酸緩衝液を40°Cに加熱します。 0.2 gのNaBH4 を加え、光から保護しながら15〜30分間攪拌します。
注:水素化ホウ素溶液は、暗所で室温で数週間保存できます。重要なステップ:ホウ酸バッファーにNaBH4 を添加すると、ガスが形成されるため、このステップはヒュームフード内で行ってください。この手順は、爆発につながる可能性があるため、密閉された容器で行わないでください。 - 自家蛍光を低減するのに十分なインキュベーション(≥3日間)した後、フリーザーから組織を取り出し、余分な凍結保護剤保存バッファーを取り除き、室温(RT)で40rpmで振とうしながらPBバッファーで組織をそれぞれ10分間3回洗浄します。
- 約20 mLの酸化アガロース懸濁液(ステップ1で調製)をマイクロ波(700 Wで~30秒)で溶かし、取り扱いができる程度に冷却(~45 °C)して、プラスチック型に流し込みます。
注:重要なステップ:サンプル上のマイクロ波で加熱した直後にアガロースを追加しないでください、これは組織を破壊することにつながる可能性があります。 - ピンセットを使用してティッシュをそっと持ち上げ、アガロースの下部中央にすばやく挿入します。型をRTで15分間インキュベートし、アガロースを重合させます。
- アガロース重合後、プラスチック型を四隅からメスで開き、手袋をはめた指でアガロースキューブを慎重につまみます。
- サンプルを水素化ホウ素溶液(ステップ2で調製)に4°Cで2〜3時間浸し、酸化したアガロースと組織の架橋を開始します。これにより、切片化中に組織が剥がれるのを防ぎます。
注:重要なステップ:水素化ホウ素に浸漬したサンプルは、4°Cで光から保護してインキュベートする必要があります。 - アガロースキューブをPBバッファーで2回10分間洗浄します。組織が上側になるようにアガロースキューブを回し、数滴の瞬間接着剤を使用してブロックを磁気スライド(背面に2つの磁石が取り付けられた顕微鏡スライド)に取り付けます。接着剤が固まるのに10〜20分かかるため、PBバッファーを滴下したペーパーティッシュをアガロースの上に置き、サンプルが全体を通して十分に水和するようにします(図1C)。
3. ミクロトームの準備とステージセッティング
- 断層撮影装置のサンプルボックスの中央にある金属に磁気スライドを置きます。
- ボックスにPBバッファーを充填し、ステージ上に慎重に配置し、左側のネジを締めてサンプルボックスを断層撮影のステージに固定します。
注意: ネジを締めすぎると、プラスチックの箱が損傷します。 - ブレードをミクロトームに置き、サンプルの位置をミクロトームの近くと下に慎重にシフトします。アガロースの上面は、ミクロトームのブレードと同じレベルにある必要があります。
注意: 低すぎる配置は切断時間を無駄にし、高すぎる配置は最初の切断でブロックの大きな塊を削除します。 - ステージを動かしてアガロースキューブを対物レンズの中央に配置し、断層撮影ソフトウェアの [スライス ]ボタンを繰り返しクリックして、アガロースキューブ全体の無傷のスライスを取得します。
4. サーフェスの割り当てと2D画像の取得
- 対物レンズを組織サンプルの中央にX方向とY方向に配置します。
- レーザーソフトウェアを開き、ソフトウェアでレーザーをオンにします。カチカチという音が聞こえたら、レーザースイッチをオンにし、波長を800nmに調整します。
注:レーザー光源は、イメージングする前に少なくとも30分間、理想的には1時間ウォームアップする必要があります。 - 顕微鏡のキャビネットのドアを閉じ、部屋のすべての光源をオフにし、PMTをオンにして電圧を750Vに設定します。
注:重要なステップ:PMTは光に非常に敏感であるため、キャビネットのドアが閉じられ、部屋のすべての光源がオフになっていない限り、光をオフにしてください。 - 断層撮影ソフトウェアのプロトコル設定を概説されているように調整し、顕微鏡のシャッターを自動に、電圧をV1とV2の電圧をそれぞれ20と3に設定します。
- 断層撮影ソフトウェアの 2Dボタンをクリックして 、サンプルのスナップをキャプチャします。
- 自家蛍光が見える画像が表示されたら、Z-ピエゾを調整して、アガロースキューブの表面に焦点面を設定します。最初のスキャンは、サンプルの安定した画像を取得するために、表面下20μmになります。
- 自家蛍光の背景が見えない場合は、信号が現れるまでコントラストを上げます。目に見える信号がないことがあります(つまり、組織の上部にあるアガロースが原因です)。この場合、Zピエゾデバイスを使用して、Z軸がアガロース表面に到達するまで調整します。目的の位置がZピエゾデバイスの範囲外にある場合は、レンズの手動移動が必要になることがあります。
- サンプルの表面を見つけた後、複数の50 μmスライスをカットし、カットのたびに2D画像を取得して、焦点面がブロック面表面に適切に設定されていることを確認します。
5.サンプルのエッジを見つけ、レーザーを始点に設定します
- レーザースキャン領域のxy座標(4つのコーナー座標)に基づいて、イメージング領域を組織に限定し、周囲のアガロースブロックのイメージングを最小限に抑えます。
- 光電子増倍管をオフにし、レーザー波長を800nmに設定し、レーザーシャッターを開いてください。アガロースブロックに入射するレーザー光の位置は、散乱光の赤い点として見えます(図2A、B)。
重要なステップ:クラス4レーザーには特に注意してください。人間の組織や目に急速に損傷を与える可能性があります。このような機器を安全に操作するには、スタッフに特別なトレーニングが必要です。 - ステージをシフトしながらレーザースポットを使用し、背面、右、前面、左のシーケンスを使用して組織エッジの座標を見つけます。
- 座標を再確認し、レーザースポットを組織のイメージングのデフォルトの出発点である右前隅に配置します。
- コンソール ウィンドウからの座標を、xy-steps によって定義される機械で認識可能なブロック サイズに変換します。これらのステップ測定値をソフトウェアのプロトコルに入力します。キャビネットのドアを閉じます。
6. 3Dスキャン/セクショニング
- 波長を940nmに変更して、GFP、mWasabi、またはYPet、およびPEを励起します。
注意: この波長では、レーザーは人間の目には見えません。GFP、mWasabi、およびTIMERbacの緑色発光成分のS/N比を向上させるには、PMT 2の510/20 nm前に狭帯域パスフィルタを配置します。これにより、緑黄色の組織自家蛍光による干渉が減少します。 - マイクロスコープのシャッターを自動モードに切り替え、シャッター電圧設定をV1の場合は20、V2の場合は1.71に設定します。
- ソフトウェアで次の3つのパラメータを調整します: セクションの厚さ - ほとんどの目的で、50 μmに設定します。肝臓の場合は、イメージング深度が限られているため、30 μmに設定します。物理セクションの数 - これにより、画像化される組織の総深さが決定され、それに応じて設定されます。各セクションでキャプチャする平面の数 - これにより、z解像度が決まります。ほとんどの目的に5つの平面(10μmの垂直分解能が得られます)を使用します。肝臓には3つ以下の平面を使用してください。
- レーザーゲインを設定します。これは、さまざまな組織とスキャン深度について経験的にテストする必要があります。
- 取得した画像を格納するためのフォルダー パスと名前を定義し、イメージング メタファイルに適切な情報を提供します。
- 入力したすべての値を再確認してください。
- 3Dモザイク設定をクリックして、スキャンプロセスを開始します(図1D)。
- イメージングが完了したら、水タンクから組織切片を慎重に収集し、さらに使用するまで-20°Cの保存バッファーに保存します(図1E;図2C,D)。
7. 画像処理とデータ解析
- MATLAB スクリプトを https://github.com/BumannLab/Li_BumannLab_2020 から Linux コンピューターにダウンロードします。スクリプトをフォルダ(例:/home/user/Program/)にコピーします。
注: スクリプトを完全に機能させるには、MATLAB と Fiji (または ImageJ) 用の Computer Vision Toolbox アドオンを Linux コンピューターにインストールする必要があります。 - 以下で説明するように、タイル画像をステッチします。
- 断層撮影サーバーからLinuxコンピューターにデータを転送します。
- MATLAB スクリプト StepOneStitchingAndArchive.m を開き、生データを含むソース フォルダーを見つけます。スクリプトでソース フォルダーと宛先フォルダーを定義します。
- [エディター] タブに切り替えて、[ 実行] をクリックします。進行状況の情報は、コマンド ウィンドウに表示されます。この処理には、Mosaicファイルからの情報の読み取り、画像スティッチング前のタイルインデックスの生成、Cidre22 による背景照明補正(図3)、および異なる深度で取得された光学層間の照明補正が含まれます。ソースフォルダ内のstitchedImages_100というサブフォルダでステッチされた画像を見つけます。tar コマンドを使用して、生データを長期保存用に圧縮し、ファイル名拡張子 tar.bz2 で 1 つのファイルとして保存します (図 1F)。
- サポートベクターマシンを使用してモデルのトレーニングとセグメンテーションを実行します。サポート ベクター マシンをトレーニングし、セグメンテーションを実行するには、次の手順に従います。
注:3つの蛍光チャネルのそれぞれのステッチ画像は、stitchedImages_100の3つのサブフォルダ(1、2、および3、赤、オレンジ、および緑の蛍光に対応する)に保存されます。各サブフォルダには、同じチャネルからスティッチされたすべての画像が含まれています。- ステッチされた画像をプレビューします。Fiji(またはImageJ)の各サブフォルダから同じファイル名の画像を1つ開きます。3 つのチャネルを 1 つのカラー イメージに結合します。細菌や組織の自家蛍光から明確なシグナルが見られるまで、各チャンネルの明るさを調整します。次のステップのために、各チャンネルの調整された最大強度レベルに注意してください。
- MATLAB スクリプト StepTwoSegmentationAndAnalysis.m を開き、ソースを参照します。トレーニングのソース フォルダーとイメージ名を定義します。例えば
ソースD = '/' ;
red_name = [sourceD '1/section_020_01.tif'];green_name = [sourceD '2/section_020_01.tif'];
blue_name = [sourceD '3/section_020_01.tif']; - [エディター] タブに移動し、[ 実行] をクリックします。
- 色のしきい値を求めるダイアログボックスが表示されるので、フィジーでの以前の手動チェック(ステップ7.3.1)に基づいて入力します。
- ダイアログに従って、背景の領域と関心のある領域(バクテリアなど)を選択します。モデルがどの程度適切にトレーニングされているかを示すグラフが表示され、さらに領域を追加する必要があるかどうかを尋ねられます。細菌が明確にセグメント化されるまで、関心領域とバックグラウンドを追加します(図4A、B)。
- モデルに名前を付けて保存します。スクリプトは自動的に画像を読み込み、中央値フィルター(半径2ピクセル)を適用します。
- セグメンテーション プロセスが自動的に実行され、進行状況情報がコマンド ウィンドウに表示されます。セグメンテーションの進行状況を監視します。セグメンテーションが他のステッチされた画像のモデルでうまく機能しない場合は、モデルのトレーニングを繰り返して、より多くの背景領域と細菌領域を含めます。
- セグメンテーションの結果を確認するには、Allpositions_filter3D.txt という名前のファイルを確認します。このファイルには、各細菌または細菌のマイクロコロニーの重心の座標と、対応する積算蛍光強度が含まれています(図1G)。
- 以下で説明するように、畳み込みニューラルネットワークを使用して追加のアーティファクトを削除します。
注:一部の細菌については、Support Vector Machineを使用した単純なセグメンテーションには、まだいくつかのイメージングアーティファクトが含まれています。これは、YPet発現細菌に特に当てはまります。ニューラルネットワークベースのセグメンテーションは、これらのアーティファクトを効率的に除去できます(図4C)。スクリプトを完全に機能させるには、MATLAB と Fiji (または ImageJ) 用の Computer Vision Toolbox アドオンを Windows コンピューターにインストールする必要があります。基になるニューラル ネットワークの詳細については、https://ch.mathworks.com/help/deeplearning/ug/create-simple-deep-learning-network-for-classification.html を参照してください。- 画像を準備します。フィジーを使用して、さまざまな明るさ調整でtifファイルをjpgファイルに変換します。手動キュレーションにより、少なくとも600のバクテリアと600のアーティファクトを特定し、これらのオブジェクトの安全にトリミングされた画像を適切なフォルダーに保管します。
- MATLAB スクリプト CNNtestV4.m を開き、手順 7.4.1 のオブジェクト イメージを含むソース ファイルを参照します。
- トレーニング用の画像フォルダーを定義します。例えば
digitDatasetPath = フルファイル ('D:\20200602_CNN','fortrainingPixel12Folder2');
トレーニング用の画像の数を定義します (例: numTrainFiles = 600)。 - [エディター] タブに移動し、[ 実行] をクリックします。トレーニング後、CNN モデル ファイルは gregnet1.mat としてソース フォルダーに保存されます。
- テスト画像を含むフォルダーを定義します。例えば
digitDatasetPathTest = fullfile('D:\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
結果は、画像名とバクテリアまたはアーティファクトとしての分類に関する情報を含むファイルYourFile.txtに保存されます。 - テスト画像を含むフォルダを定義します。例えば
digitDatasetPathTest = fullfile('D:\20200602_CNN\fortrainingPixel12Folder2\R2500G550B450yfp').
結果は、画像名とバクテリアまたはアーティファクトとしての分類に関する情報を含むファイルYourFile.txtに保存されます。
- 以下に説明するように、単一細菌の蛍光強度に基づいてマイクロコロニーのサイズを推定します。
注:断層撮影装置の空間分解能は、密集したマイクロコロニー内の個々の細菌細胞を分離するには不十分です。ただし、各マイクロコロニー内の細菌の数は、単一の細菌の蛍光に対するマイクロコロニーの総蛍光に基づいて推定できます。この推定精度は、細菌集団全体の蛍光強度の均一性に依存します。- STPトモグラフィーで取得した切片の免疫組織化学を用いてリポ多糖などの細菌成分を染色することにより、STPトモグラフィーにおけるGFP陽性イベントの同定を確認します(図1H; 図 5)。
- 少なくとも 30 種類の単一細菌を同定し、対応するセグメンテーション結果からそれらの蛍光強度を取り出します。強度の中央値を 1 つの細菌の基準として利用します。マイクロコロニーの総蛍光強度を単一の細菌の基準値で割ることにより、各マイクロコロニーの細菌数を計算します(図1I)。
- 以下に説明するように、視覚化ソフトウェアを使用して3D再構成を実行します。
- 画像の準備
- 青チャンネルの画像には、組織嚢、動脈、小柱などの有用な解剖学的参照を提供するコラーゲンの第2高調波信号が含まれています。バクテリア用にセグメント化された画像を 2番目の チャネルとして使用します。
- 1番目の チャンネルの画像を X 軸と Y 軸の両方で 10 倍にビン化し、縮小した画像を新しいフォルダーに保存します。同じフォルダ内で縮小された画像を3回複製します。新しいレプリケートの名前は、同じシーケンスを維持する必要があります。例: section_001_01.tif、名前が section_001_01-copy.tif、section_001_01-copy-copy.tif の 1 をレプリケートします。これらの手順を 2番目の チャネルの画像にも適用します。ダウンサイジングにより、ファイルサイズがより管理しやすくなります。トリプル画像はZ軸を滑らかにします。
- 各チャネルの画像をマージして、フィジーと画像スタックを形成します。 [イメージ] > [スタック] > [ツール] > [スタック ソーター] > [ラベルで並べ替え] をクリックします。
- 3Dフィルタリングを実行します。 「3D フィルター>「処理フィルター」>フィルターをクリックし、「 Z フィルターサイズ」を 6 に設定します。画像スタックを保存します。
- 以下で説明するように、3Dビジュアライゼーションを実行します。
- 視覚化ソフトウェア パッケージをアリーナ ビュー (デフォルト設定) で開きます。ウォッチフォルダアイコンを使用して、アリーナビューにフォルダを追加します。ダブルクリックして、*.imsまたは*.tifファイル(7.6.1で準備)を開きます。
- Edit>Image Propertiesを使用して、Display Adjustmentウィンドウでさまざまなチャンネルの色表現(LUT)を調整します。
- [詳細設定]をクリックして、最小/最大値とガンマ補正の値を手動で設定します。
チャネル名をクリックして、名前と LUT を変更します。 - 画像の外観を調整したら、スナップショットツールを使用して現在のビューをエクスポートします。
- アニメーションアイコンを使用して、3D データをムービーとして表現します。ナビゲーションポインターを使用してパースペクティブ/ビューを見つけてズームし、+ 追加を使用してキーフレームを追加します。別の位置に移動し、次のキーフレームを追加します。 赤の録画 ボタンを押して、ムービーを作成します。目的の宛先フォルダとファイルタイプに保存します(図1J)。
- 画像の準備
結果
記載されている手順により、脾臓、肝臓、腸間膜リンパ節、およびパイエル板11などのマウス臓器全体の個々のサルモネラ細胞の検出が可能になります11(図5および図6)。また、マウスの脳12でトキソプラズマ原虫も検出します。肝臓、パイエル板、脾臓などの一部の感染組織は、緑黄色の範囲でかなりの自家蛍光を発します。自家蛍光は、組織構造を保存するために必要なパラホルムアルデヒドによる固定によってさらに増強されます。この自家蛍光バックグラウンドに対するGFP、mWasabi、およびTIMERbacの緑色成分からの緑色蛍光の検出は、光電子増倍管2(緑色発光を収集する)の前に510/20 nmの狭帯域通過フィルター(ほとんどのGFP発光を透過するが、自家蛍光スペクトルの大部分を遮断する)を置き、固定組織を凍結保護剤11に3日以上保存することにより組織の自家蛍光を減らすことによって改善される.しかし、細菌は依然としてGFPまたは他の蛍光タンパク質の細胞あたり少なくとも数千コピーを発現するはずです。一方、過剰な蛍光タンパク質レベルは、病原性の減衰につながる可能性のあるフィットネスコストを最小限に抑えるために避けるべきである23。
組織中の蛍光細菌の正しいセグメンテーションは、イメージング後に取得した組織切片の免疫組織化学によって確認できます。具体的には、リポ多糖などの細菌表面成分に対する抗体で染色した物体を、STPトモグラフィーで得られた蛍光画像と一致させることができます(図5)。染色されたサルモネラ細胞の中には、蛍光タンパク質を欠くものがあり、共焦点顕微鏡とSTPトモグラフィーの両方で検出可能な蛍光を発することに注意することが重要です。これらの細胞は、フローサイトメトリーによる選別と単一選別細胞からの増殖培養、および寒天プレート上のコロニー形成ユニットの数とフローサイトメトリーによって決定される蛍光サルモネラ細胞の数との間に密接な相関があることで示されるように、宿主免疫系によって死滅されたサルモネラ菌です24.さらに、プラスミドの喪失は非蛍光性の生細胞をもたらす可能性があり、これはプラスミド上の選択マーカーに対応する適切な抗生物質の有無にかかわらず培地にプレーティングすることによって試験する必要があります。pSC101由来プラスミドの場合、in vivoでのプラスミド損失はまれです19。11で使用されているsifB::gfpのようなほとんどの染色体統合発現カセットでは、発現喪失はin vivoでは検出できません。セグメンテーションが免疫組織化学データと一致しない場合は、セグメンテーションパイプラインを変更する必要があります。
STPトモグラフィーの分解能は、密集したマイクロコロニー内の個々の細菌細胞を分離するには不十分です。しかし、マイクロコロニーの全蛍光強度により、 サルモネラ 菌の数を推定することができます。これには、 サルモネラ菌sifB::gfp11のような非常に均質な蛍光レベルを持つ蛍光株が必要です。すべてのマイクロコロニーおよび単一細胞の サルモネラ 細胞の推定数を組み合わせると、組織ホモジネートのプレーティングまたはフローサイトメトリーなどの代替方法と一致する総細菌組織負荷が得られる11。プレーティングとフローサイトメトリーは、STPトモグラフィーのために灌流固定する必要があるため、同じ組織から直接行うことはできません。それどころか、固定されていない追加の動物を使って行う必要があります。さまざまなアプローチによって決定される細菌負荷の中央値が3倍以上異なる場合、蛍光細菌の生存率が損なわれる可能性がある(コロニー形成単位が低い場合)か、一部の細菌が蛍光レポーターコンストラクトを失った(コロニー形成単位が高い場合)可能性があります。このような不一致の原因を特定するためには、対照実験が必要になります。
STPトモグラフィーは、感染組織の3D構造内の細菌細胞の局在化を提供します。コラーゲンの第2高調波信号は、動脈や小柱などの解剖学的ランドマークを提供します。さらに、宿主細胞は、灌流前に表面マーカーに抗体を注入することにより、 in vivo で染色できます(図5 および 図6)。この染色は、組織コンパートメントや炎症病巣などの特定の微小環境に追加のランドマークを提供します(細胞内マーカーや、脾臓の白い歯髄や脳などの拡散バリアを持つ一部のコンパートメントは簡単にアクセスできないため、この in vivo 染色に適しています)。このアプローチにより、脾臓の白い歯髄が、抗菌化学療法中の サルモネラ 菌の長期生存を可能にする組織コンパートメントであることが明らかになりました11。
最後に、中程度または遅い速度で複製するサルモネラ菌は、複製速度11,15の単一細胞レポーターであるタイマーbacを発現する株を使用して、組織の3D構造内で同定および局在化することができる。
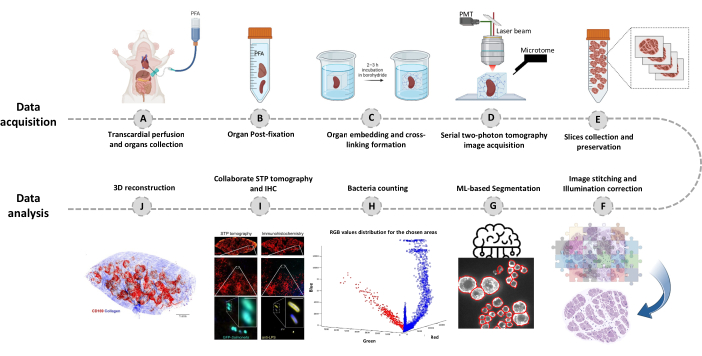
図1:連続2光子(STP)トモグラフィーを用いた全臓器イメージングの手順(A-B) 臓器を経心灌流後に採取し、4%パラホルムアルデヒド(PFA)に4°Cで一晩保存します。(C-E) 臓器を酸化アガロースに包埋して架橋した後、組織をスキャンし、STPトモグラフィーを使用してスライスします。スライスは、その後の免疫組織化学のために収集されます。(F-J)細菌数の定量化、蛍光細菌の確認、細菌位置の3D再構成のための計算機解析パイプライン。この図の拡大版を表示するには、ここをクリックしてください。

図2:シリアル2光子(STP)トモグラフィーセットアップ(A)断層撮影は、(B)2光子イメージングと(C)自動シリアル組織切片を組み込んだものです。(D)追跡調査のために収集された組織切片。この図の拡大版を表示するには、ここをクリックしてください。
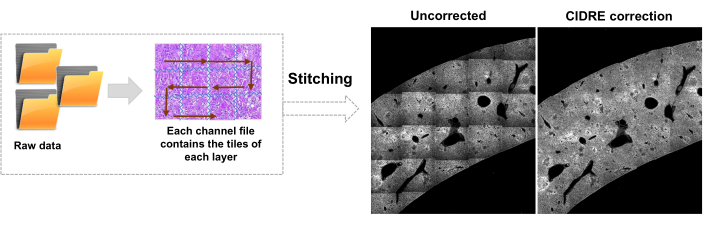
図3:タイルのステッチと照明の補正。 タイルはステッチされ、不均一な照明は、正則化エネルギー最小化(CIDRE)を使用して補正された強度分布を使用して補正されます。 この図の拡大版を表示するには、ここをクリックしてください。
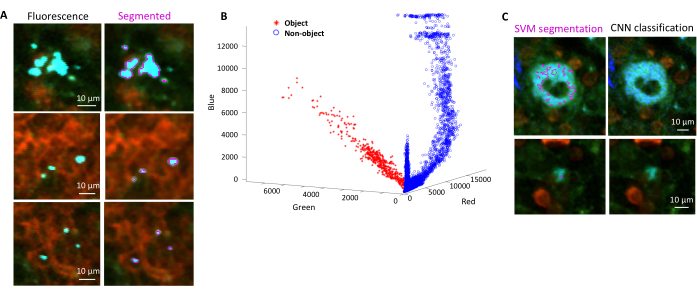
図4:サポートベクターマシン(SVM)と畳み込みニューラルネットワーク(CNN)を使用した緑色蛍光タンパク質(GFP)を発現する サルモネラ菌 のセグメンテーション。 (A)SVMでセグメント化されたGFPオブジェクト(左)とSVMでセグメント化された領域を持つ対応する画像(右)の代表的な画像(スケールバー:10μm)。(B)セグメント化された地域のクラスター化された赤-緑-青(RGB)値の分布。(C)SVMが細菌と誤認した非GFP物体の代表画像(左)。CNN は、それらを背景として正しく無視します (右、スケール バー: 10 μm)。 この図の拡大版を表示するには、ここをクリックしてください。
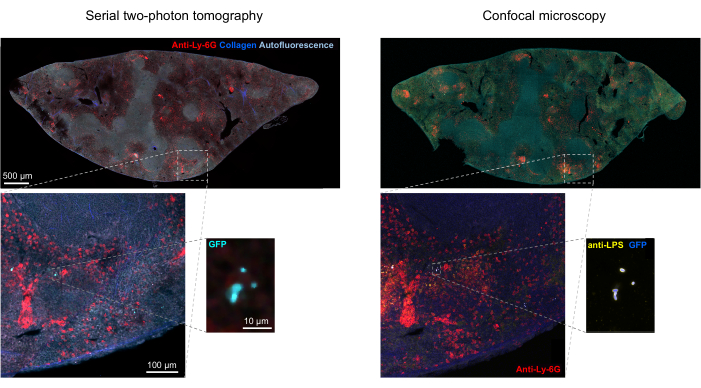
図5:トモグラフィーによる緑色蛍光タンパク質(GFP)を発現するサルモネラ菌の検出と免疫組織化学による確認。サルモネラリポ多糖に対する抗体で染色した後、トモグラフィーまたは共焦点顕微鏡法で取得した同じ切片の画像(左)。好中球(赤)は、灌流前にPE標識抗Ly-6G抗体をin vivo注射して染色しました。この図の拡大版を表示するには、ここをクリックしてください。
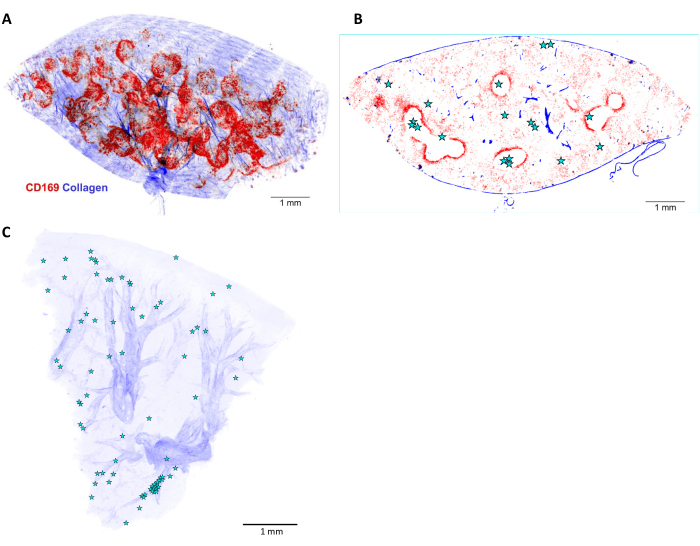
図6:感染したマウス脾臓における サルモネラ 菌の3D再構築と局在化。 (A)厚さ5mmの脾臓切片を、抗CD169抗体(赤)による灌流前に in vivo で染色した3次元(3D)再構成。青色の信号は、第2高調波によって検出されたコラーゲンを表します。(B) (A)に示す3Dスタックの1つの光学面。 サルモネラ 菌細胞またはマイクロコロニーの位置は星で示されています。(C)感染した肝臓の サルモネラ 菌の位置(星)の3D再構築。動脈は、コラーゲン鞘(青)に基づいて見えます。スケールバー:1 mm。 この図の拡大版を表示するには、ここをクリックしてください。
ディスカッション
細菌性病原体の局所組織状況は、局所的な宿主攻撃、細菌の適応、宿主病原体との相互作用と抗菌化学療法の局所的な結果、および全体的な疾患結果に対する個々の寄与を決定するために重要です。マイクロメートルサイズの細菌をセンチメートルサイズの臓器にイメージングすることは困難でした。シリアル 2 光子 (STP) トモグラフィーは、臓器全体の個々の細菌細胞を検出するのに十分な空間分解能、自動化された切片化とイメージング、および十分なスループット (1 日あたり ~1 臓器) を提供します11。宿主抗原は in vivoで染色できますが、病原体細胞は、細胞内病原体細胞を包括的に検出するために、適切な蛍光タンパク質を発現する必要があります。結果として得られるデータセット(臓器あたり0.5〜1.5テラバイト)は、データ分析と保存のためのITインフラストラクチャに大きな課題をもたらします。
この方法には、いくつかの重要な手順があります。まず、蛍光タンパク質GFPまたはYFPの検出可能で均質な発現を持つ病原体株が必要です。理想的には、染色体発現カセット25 を使用して、プラスミドのコピー数変動による蛍光の不均一性を最小化します。十分な蛍光強度が必要であるが、病原体の適応度障害を避けるために、過剰なレベルの蛍光タンパク質を避けるべきである23。適切な発現レベルは、適切なプロモーターの選択およびリボソーム結合部位25 または全体の5'非翻訳領域(UTR)26の微調整によって得ることができる。第二に、灌流固定には、血液循環からできるだけ多くの赤血球を除去するためのバッファーによる最初の洗浄を含める必要があります。これは、脾臓と肝臓にとって特に重要です(ただし、これらの臓器から赤血球を完全に除去することは困難です)。残りの赤血球は、スペクトルの可視部分で光を吸収し、イメージング品質を損なう27。第三に、凍結保護剤に固定組織を保存することは、炎症組織で特に高く、病原体細胞の比較的弱い蛍光を覆い隠す可能性のある組織の自家蛍光を減らすために重要である11。第四に、組織と周囲のアガロースブロックへの効果的な架橋は、組織がアガロースブロックから飛び出すことなくスムーズなビブラトーム切断を行うために重要です。第五に、蛍光シグナルおよびそれらが病原体細胞として同定されることは、病原体成分に対する抗体(グラム陰性菌のリポ多糖類など)による染色や、断層撮影装置11から取り出した切片の共焦点顕微鏡法などの直交的アプローチを用いて、独立して検証されなければならない。感染組織の中には、形状が似ていて蛍光スペクトルが重なり合う自己蛍光粒子が含まれているものもあり、病原体細胞と誤解されやすいことがあります。第六に、マイクロコロニー内の病原体細胞の量を共焦点顕微鏡法などの直交的アプローチと比較して、精度を評価する必要があります。これらの計算に基づく全体的な細菌負荷は、フローサイトメトリーやプレーティングなどの直交的なアプローチと比較することで検証する必要があります。
広く使用されているSTPプロトコルの重要な変更には、光電子増倍管211の前に510/20 nmの狭帯域パスフィルターを配置することが含まれ、感染して炎症を起こした肝臓、脾臓、およびパイエルパッチで特に強い緑黄色の自家蛍光の干渉を減らすためである。このような臓器は、脳(STPの他の用途が主流)と比較して、自家蛍光が強く、光散乱が増加するため、照明の不均一に対するより効果的な補正の必要性も生じています。別の変更として、このプロトコルは、この目的のためにCIDREアプローチ22 (図3)とAIベースの細菌のセグメンテーションを採用しています。最後に、組織の前処理は、-20°Cの凍結保護剤にインキュベーションステップを含めることによって変更され、これにより組織の自家蛍光が減少し、したがって比較的弱い蛍光を有する小さな病原体細胞の検出が容易になった11。
病原体シグナルを検出できない場合、またはセグメンテーションによって感度が不十分(見逃される病原体細胞が多すぎる)または精度が不十分(病原体細胞としてセグメント化されるバックグラウンド粒子が多すぎる)場合は、トラブルシューティングが必要になることがあります。バックグラウンド組織の自家蛍光が検出可能であるにもかかわらず、病原体のシグナルが少なすぎる場合、病原体に含まれる蛍光タンパク質の量が不十分である可能性があります。これは、同じ感染組織からの組織切片の共焦点顕微鏡法または組織ホモジネートのフローサイトメトリーを用いて試験することができる19,28。根本的な理由は、発現レベルが不十分であるか、発現カセットが不安定であることである可能性があります。緩和戦略には、発現を促進するための代替プロモーター、病原体種の蛍光タンパク質をコードする遺伝子のコドン適応、コピー数の多いエピソームコンストラクトの採用、または染色体統合またはバランス致死相補による発現カセットの安定化が含まれる可能性がある29。蛍光タンパク質の選択も重要ですが、GFP.mut2、mWasabi、YPet、TIMERbacで検出が可能です。セグメンテーションが不正確な場合、これは、上記のように対処できる病原体の蛍光が弱すぎるか、組織の自家蛍光バックグラウンドが高すぎることが原因である可能性があります。アガロースブロックおよびトモグラフィーに包埋する直前に、洗浄液を広範囲に灌流するか、保存バッファーで長時間インキュベートすることで、これらの問題を解決できる可能性があります。最後に、正確な分類にはニューラルネットワークの十分なトレーニングが必要ですが、過度のトレーニングは、新しいサンプルのパフォーマンスを損なう過剰適合につながる可能性があります。
現在、個々の細菌を検出するために、臓器全体を3Dで十分な空間分解能で画像化できる方法は他にありません。組織透明化とライトシート顕微鏡法の将来の改善により、同様の解像度が得られる可能性があります。これにより、より高速で、より多くの蛍光チャンネルでのイメージングが可能になるかもしれません。
STPの重要な制限は、面内ピクセル解像度が~0.5 μm、垂直解像度が5〜10 μmであることであり、密集したマイクロコロニー内など、密集した細菌の分離には不十分です。しかし、トモグラフィー後に組織切片を回収し、選択した組織部分の二次高解像度共焦点顕微鏡法を行うことが可能です。STPのもう一つの制限は、蛍光チャネルが3つしかないため、同時にイメージングできる蛍光色素の数が制限されていることです。ここでも、マルチプレックス法で取得した組織切片を二次解析することで、選択した組織部分のさらに多くのマーカーの位置と強度を明らかにすることができます。この情報は、STPで決定されるように、周囲の組織の全体的な3D構造に統合できます。
結論として、このプロトコルは、局所および全臓器レベルでの宿主と病原体の相互作用の詳細な調査を可能にします。このプロトコルは、他の病原体(蛍光株として入手できる場合)、他の臓器、および異なる宿主種に容易に適応できる必要があります。
開示事項
著者は何も開示していません。
謝辞
この研究は、スイス国立科学財団 310030_156818、310030_182315、および NCCR_ 180541 AntiResist (DB へ) の支援を受けました。
資料
| Name | Company | Catalog Number | Comments |
| Chemicals | |||
| Agarose Low Melt | Roth | Art. 6351.5 25g | |
| Boric acid | Sigma-Aldrich | 6768-500G | |
| Instant adhesive Loctite 435 | Henkel | ||
| Paraformaldehyde | Sigma-Aldrich | P6148 | |
| Poly(ethylene glycol) | Sigma-Aldrich | P5413-1kg | |
| Polyvinylpyrrolidone | Sigma-Aldrich | PVP-100G | |
| Sodium borohydride | Sigma-Aldrich | 71321-25g | |
| Sodium hydroxide | Merck | 106453 | |
| Sodium periodate | Sigma-Aldrich | 311448-100G | |
| Sodium phosphate dibasic | Sigma-Aldrich | 71640-250G | |
| Sodium phosphate monobasic dihydrate | Sigma-Aldrich | 71500-1KG | |
| Sodium tetraborate | Sigma-Aldrich | 221732-100g | |
| Sucrose | AppliChem | A4734,1000 | |
| Tris-buffered saline (TBS) | Merck | T5912-1L | |
| Triton X-100 | Sigma-Aldrich | 9002-93-1 | |
| Vacuum filtration 500 | TPP | TPP99250 | |
| Equipment | |||
| Blade | Campden Instruments Limited | 01-01-4692 | |
| MAITAI Laser | Spectra-Physics | ||
| Peel away plastic mold | Sigma-Aldrich | E6032-1CS | |
| TissueCyte 1000 tomograph | TissueVision | ||
| Antibody/dyes | |||
| DAPI | Merck | D9542-5MG | |
| Primary antibodies | |||
| anti-LPS Salmonella, rabbit | Sifin | REF TS 1624 | |
| anti-CD169-PE, clone 3D6.112 | Biolegend | 142403 | |
| anti-Ly-6G-PE, clone 1A8 | Biolegend | 127608 | |
| Secondary antibodies | Invitrogen | ||
| chicken anti-rabbit Alexa 647 | Invitrogen | A-21443 | |
| Software | Company | Version | |
| Fiji | Image J | 1.54g or later | |
| MATLAB | MathWorks | 2017b/2018b or later | |
| Orchestrator (tomograph) | TissueVision | ||
| Visualization software Imaris | Oxford Instruments | 9.9.0 or later |
参考文献
- Bjarnsholt, T., et al. The importance of understanding the infectious microenvironment. Lancet Infect Dis. 22 (3), e88-e92 (2022).
- Azimi, S., Lewin, G. R., Whiteley, M. The biogeography of infection revisited. Nat Rev Microbiol. 20 (10), 579-592 (2022).
- Bumann, D., Cunrath, O. Heterogeneity of Salmonella-host interactions in infected host tissues. Curr Opin Microbiol. 39, 57-63 (2017).
- Hofmann, J., Keppler, S. J. Tissue clearing and 3D imaging - putting immune cells into context. J Cell Sci. 134 (15), jcs258494 (2021).
- Blain, R., et al. A tridimensional atlas of the developing human head. Cell. 186 (26), 5910-5924.e17 (2023).
- Ragan, T., et al. Serial two-photon tomography for automated ex vivo mouse brain imaging. Nat Meth. 9 (3), 255-258 (2012).
- Oh, S. W., et al. A mesoscale connectome of the mouse brain. Nature. 508 (7495), 207-214 (2014).
- Kim, Y., et al. Brain-wide maps reveal stereotyped cell-type-based cortical architecture and subcortical sexual dimorphism. Cell. 171 (2), 456-469.e422 (2017).
- Matho, K. S., et al. Genetic dissection of the glutamatergic neuron system in cerebral cortex. Nature. 598 (7879), 182-187 (2021).
- Muñoz-Castañeda, R., et al. Cellular anatomy of the mouse primary motor cortex. Nature. 598 (7879), 159-166 (2021).
- Li, J., et al. Tissue compartmentalization enables Salmonella persistence during chemotherapy. Proc Natl Acad Sci U S A. 118 (51), e2113951118 (2021).
- Dogga, S. K., et al. Importance of aspartyl protease 5 in the establishment of the intracellular niche during acute and chronic infection of Toxoplasma gondii. Mol Microbiol. 118 (6), 601-622 (2022).
- Cormack, B. P., Valdivia, R. H., Falkow, S. FACS-optimized mutants of the green fluorescent protein (GFP). Gene. 173 (1 Spec No), 33-38 (1996).
- Nguyen, A. W., Daugherty, P. S. Evolutionary optimization of fluorescent proteins for intracellular FRET. Nat Biotechnol. 23 (3), 355-360 (2005).
- Claudi, B., et al. Phenotypic variation of Salmonella in host tissues delays eradication by antimicrobial chemotherapy. Cell. 158 (4), 722-733 (2014).
- Ai, H. W., Olenych, S. G., Wong, P., Davidson, M. W., Campbell, R. E. Hue-shifted monomeric variants of Clavularia cyan fluorescent protein: identification of the molecular determinants of color and applications in fluorescence imaging. BMC Biol. 6, 13 (2008).
- Drobizhev, M., Makarov, N. S., Tillo, S. E., Hughes, T. E., Rebane, A. Two-photon absorption properties of fluorescent proteins. Nat Methods. 8 (5), 393-399 (2011).
- Shaner, N. C., et al. Improved monomeric red, orange and yellow fluorescent proteins derived from Discosoma sp. red fluorescent protein. Nat Biotechnol. 22 (12), 1567-1572 (2004).
- Burton, N. A., et al. Disparate impact of oxidative host defenses determines the fate of Salmonella during systemic infection in mice. Cell Host&Microbe. 15 (1), 72-83 (2014).
- Wu, J., et al. Transcardiac perfusion of the mouse for brain tissue dissection and fixation. Bio Protoc. 11 (5), e3988 (2021).
- de Olmos, J., Hardy, H., Heimer, L. The afferent connections of the main and the accessory olfactory bulb formations in the rat: an experimental HRP-study. J Comp Neurol. 181 (2), 213-244 (1978).
- Smith, K., et al. CIDRE: an illumination-correction method for optical microscopy. Nat Methods. 12 (5), 404-406 (2015).
- Wendland, M., Bumann, D. Optimization of GFP levels for analyzing Salmonella gene expression during an infection. FEBS Lett. 521 (1-3), 105-108 (2002).
- Barat, S., et al. Immunity to intracellular Salmonella depends on surface-associated antigens. PLoS Pathog. 8 (10), e1002966 (2012).
- Rollenhagen, C., Sorensen, M., Rizos, K., Hurvitz, R., Bumann, D. Antigen selection based on expression levels during infection facilitates vaccine development for an intracellular pathogen. Proc Natl Acad Sci U S A. 101 (23), 8739-8744 (2004).
- Chen, F., Cocaign-Bousquet, M., Girbal, L., Nouaille, S. 5'UTR sequences influence protein levels in Escherichia coli by regulating translation initiation and mRNA stability. Front Microbiol. 13, 1088941 (2022).
- Weissleder, R. A clearer vision for in vivo imaging. Nat Biotechnol. 19 (4), 316-317 (2001).
- Bumann, D. Examination of Salmonella gene expression in an infected mammalian host using the green fluorescent protein and two-colour flow cytometry. Mol.Microbiol. 43 (5), 1269-1283 (2002).
- Nakayama, K., Kelly, S. M., Curtiss III, R. J. B. t. Construction of an Asd+ expression-cloning vector: stable maintenance and high level expression of cloned genes in a Salmonella. vaccine strain. 6 (6), 693-697 (1988).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved