Method Article
Registro y análisis de la dinámica de conjuntos neuronales multimodales a gran escala en una matriz de microelectrodos de alta densidad integrada en CMOS
En este artículo
Resumen
Aquí, empleamos HD-MEA para profundizar en la dinámica computacional de conjuntos neuronales a gran escala, particularmente en el hipocampo, los circuitos del bulbo olfativo y las redes neuronales humanas. La captura de la actividad espacio-temporal, combinada con herramientas computacionales, proporciona información sobre la complejidad de los conjuntos neuronales. El método mejora la comprensión de las funciones cerebrales, lo que podría identificar biomarcadores y tratamientos para trastornos neurológicos.
Resumen
Las redes neuronales a gran escala y sus complejos microcircuitos distribuidos son esenciales para generar la percepción, la cognición y el comportamiento que emergen de los patrones de actividad neuronal espaciotemporal. Estos patrones dinámicos que emergen de grupos funcionales de conjuntos neuronales interconectados facilitan cálculos precisos para procesar y codificar información neuronal multiescala, impulsando así funciones cerebrales superiores. Para sondear los principios computacionales de la dinámica neuronal que subyacen a esta complejidad e investigar el impacto multiescala de los procesos biológicos en la salud y la enfermedad, las grabaciones simultáneas a gran escala se han convertido en instrumentales. Aquí, se emplea una matriz de microelectrodos de alta densidad (HD-MEA) para estudiar dos modalidades de dinámica neuronal: circuitos del hipocampo y del bulbo olfativo a partir de cortes de cerebro de ratón ex vivo y redes neuronales a partir de cultivos celulares in vitro de células madre pluripotentes inducidas humanas (iPSC). La plataforma HD-MEA, con 4096 microelectrodos, permite grabaciones no invasivas, multisitio y sin etiquetas de patrones de disparo extracelular de miles de conjuntos neuronales simultáneamente a una alta resolución espacio-temporal. Este enfoque permite la caracterización de varias características electrofisiológicas de toda la red, incluidos los patrones de actividad de picos de una o varias unidades y las oscilaciones de potencial de campo local. Para analizar estos datos neuronales multidimensionales, hemos desarrollado varias herramientas computacionales que incorporan algoritmos de aprendizaje automático, detección y clasificación automática de eventos, teoría de grafos y otros análisis avanzados. Al complementar estos canales computacionales con esta plataforma, proporcionamos una metodología para estudiar la dinámica grande, multiescala y multimodal desde los ensamblajes de células hasta las redes. Esto puede potencialmente avanzar en nuestra comprensión de las funciones cerebrales complejas y los procesos cognitivos en la salud y la enfermedad. El compromiso con la ciencia abierta y los conocimientos sobre la dinámica neuronal computacional a gran escala podrían mejorar el modelado inspirado en el cerebro, la computación neuromórfica y los algoritmos de aprendizaje neuronal. Además, la comprensión de los mecanismos subyacentes de los cálculos neuronales a gran escala deteriorados y su dinámica de microcircuitos interconectados podría conducir a la identificación de biomarcadores específicos, allanando el camino para herramientas de diagnóstico más precisas y terapias dirigidas para trastornos neurológicos.
Introducción
Los conjuntos neuronales, a menudo denominados ensamblajes celulares, son fundamentales en la codificación neuronal, facilitando cálculos intrincados para procesar información neuronal multiescala 1,2,3. Estos conjuntos sustentan la formación de redes neuronales expansivas y sus microcircuitos matizados4. Dichas redes y sus patrones oscilatorios impulsan funciones cerebrales avanzadas, incluida la percepción y la cognición. Si bien una amplia investigación ha explorado tipos neuronales específicos y vías sinápticas, sigue siendo difícil comprender más profundamente cómo las neuronas forman conjuntos celulares de forma colaborativa e influyen en el procesamiento de información espaciotemporal a través de circuitos y redes.
Los cortes cerebrales agudos ex vivo son herramientas electrofisiológicas fundamentales para el estudio de circuitos neuronales intactos, ya que ofrecen un entorno controlado para sondear los patrones de actividad oscilatoria de la función neuronal, la transmisión sináptica y la conectividad, con implicaciones en las pruebas farmacológicas y el modelado de enfermedades 6,7,8. Este protocolo de estudio destaca dos circuitos cerebrales clave: el hipocampo-cortical (HC) involucrado en los procesos de aprendizaje y memoria 9,10, y el bulbo olfatorio (OB) responsable de la discriminación de olores 11,12,13. En estas dos regiones, la neurogénesis adulta genera continuamente nuevas neuronas funcionales a lo largo de la vida en los cerebros de los mamíferos14. Ambos circuitos demuestran patrones de actividad neuronal dinámicos multidimensionales y plasticidad inherente que participan en el recableado de la red neuronal existente y facilitan estrategias alternativas de procesamiento de información cuando es necesario15,16.
Los modelos agudos de corte cerebral ex vivo son indispensables para profundizar en la funcionalidad cerebral y comprender los mecanismos de la enfermedad a nivel de microcircuitos. Sin embargo, los cultivos celulares in vitro derivados de redes neuronales de células madre pluripotentes inducidas humanas (iPSC) ofrecen una vía prometedora de investigación traslacional, ya que conectan a la perfección los hallazgos de los experimentos con animales con el posible tratamiento clínico humano17,18. Estos ensayos in vitro centrados en el ser humano sirven como una plataforma fiable para evaluar la toxicidad farmacológica, lo que permite un cribado preciso de fármacos y fomenta la investigación de estrategias terapéuticas innovadoras basadas en células19,20. Reconociendo el papel fundamental del modelo neuronal iPSC, hemos dedicado el tercer módulo de este estudio de protocolo a investigar a fondo las características funcionales de sus redes derivadas y a afinar los protocolos de cultivo celular asociados.
Estos módulos neuronales electrogénicos se han estudiado comúnmente utilizando técnicas como el calcio (imágenes de Ca2+), los registros de patch-clamp y las matrices de microelectrodos de baja densidad (LD-MEA). Si bien las imágenes de Ca2+ ofrecen un mapeo de la actividad de una sola célula, es un método basado en el etiquetado celular que se ve obstaculizado por su baja resolución temporal y los desafíos en los registros a largo plazo. Los LD-MEAs carecen de precisión espacial, mientras que el patch-clamp, al ser una técnica invasiva de un solo sitio y laboriosa, a menudo produce una baja tasa de éxito 21,22,23. Para abordar estos desafíos y sondear de manera efectiva la actividad de toda la red, las grabaciones neuronales simultáneas a gran escala han surgido como un enfoque fundamental para comprender los principios computacionales de la dinámica neuronal que subyacen a la complejidad cerebral y sus implicaciones en la salud y la enfermedad24,25.
En este protocolo JoVE, demostramos un método de registro neuronal a gran escala basado en la MEA DE ALTA DENSIDAD (HD-MEA) para capturar la actividad neuronal espaciotemporal a través de varias modalidades cerebrales, incluidos los circuitos del hipocampo y del bulbo olfativo de cortes agudos de cerebro de ratón ex vivo (Figuras 1A-C) y redes neuronales humanas derivadas de iPSC in vitro (Figuras 1D-E), previamente informadas por nuestro grupo y otros colegas26,27,28,29,30,31,32,33,34,35. El HD-MEA, basado en la tecnología de semiconductores complementarios de óxido metálico (CMOS), cuenta con circuitos y amplificación en chip, lo que permite grabaciones de menos de un milisegundo en una matriz de 7 mm2 tamaño36. Este enfoque no invasivo captura patrones de disparo extracelular multisitio y sin etiquetas de miles de conjuntos neuronales simultáneamente utilizando 4096 microelectrodos a una alta resolución espacio-temporal, revelando la intrincada dinámica de los potenciales de campo local (LFP) y la actividad de pico multiunidad (MUA)26,29.
Dada la inmensidad de los datos generados por esta metodología, es esencial contar con un marco analítico sofisticado, pero plantea desafíos37. Hemos desarrollado herramientas computacionales que abarcan la detección automática de eventos, la clasificación, la teoría de grafos, el aprendizaje automático y otras técnicas avanzadas (Figura 1F)26,29,38,39. Al integrar el HD-MEA con estas herramientas analíticas, se diseña un enfoque holístico para sondear la intrincada dinámica desde los ensamblajes celulares individuales hasta las redes neuronales más amplias a través de diversas modalidades neuronales. Este enfoque combinado profundiza nuestra comprensión de la dinámica computacional en las funciones cerebrales normales y ofrece información sobre las anomalías presentes en condiciones patológicas28. Además, los conocimientos de este enfoque pueden impulsar avances en el modelado inspirado en el cerebro, la computación neuromórfica y los algoritmos de aprendizaje neuronal. En última instancia, este método es prometedor para descubrir los mecanismos centrales detrás de las interrupciones de la red neuronal, identificar potencialmente biomarcadores y guiar la creación de herramientas de diagnóstico precisas y tratamientos dirigidos para afecciones neurológicas.
Protocolo
Todos los experimentos se realizaron de acuerdo con las regulaciones europeas y nacionales aplicables (Tierschutzgesetz) y fueron aprobados por la autoridad local (Landesdirektion Sachsen; 25-5131/476/14).
1. Cortes cerebrales ex vivo de circuitos hipocampal-corticales y del bulbo olfativo en HD-MEA
- Preparación de soluciones experimentales de corte y grabación (Figura 2A)
- El día del experimento, prepare 0,5 L de solución de corte con alto contenido de sacarosa y 1 L de solución de registro de líquido cefalorraquídeo artificial (LCRa) (Tabla 1A,B).
- Agregue todos los productos químicos sólidos a un matraz aforado seco, luego llene parte del camino con agua bidestilada (dd).
- Agregue MgCl2 y CaCl2 de soluciones madre de 1 M, luego llene el resto con agua dd. Comience a revolver constantemente con un agitador magnético hasta que los sólidos visibles se hayan disuelto ~ 5 min.
- Utilice un osmómetro de punto de congelación para validar la osmolaridad entre 350-360 mOsm para la solución de corte con alto contenido de sacarosa y 315-325 mOsm para la solución de registro de aCSF.
- Utilice un medidor de pH para validar el pH entre 7,3 y 7,4 para la solución de corte con alto contenido de sacarosa y entre 7,25 y 7,35 para la solución de registro de aCSF. Comience a burbujear continuamente con 95% deO2 y 5% de CO2.
- Coloque la solución de corte con alto contenido de sacarosa en hielo durante al menos 30 minutos antes de cortar y comience a burbujear continuamente con 95% de O2 y 5% de CO2.
- Después de 10 min de carbogenación, llene un vaso de precipitados de 50 ml con 30 ml de solución de corte y guárdelo en el congelador (-20 °C) durante 20-30 min o hasta que esté parcialmente congelado.
NOTA: Todas las soluciones deben prepararse frescas para cada experimento. El agua dd utilizada aquí es agua ultrapura esterilizada en autoclave almacenada a temperatura ambiente (RT). La cantidad de solución preparada debe adaptarse a la pregunta específica del estudio.
- El día del experimento, prepare 0,5 L de solución de corte con alto contenido de sacarosa y 1 L de solución de registro de líquido cefalorraquídeo artificial (LCRa) (Tabla 1A,B).
- Preparación de las áreas de trabajo de corte de cerebro (Figura 2A)
- Lleva al animal a la sala experimental.
NOTA: En este protocolo, se utilizaron ratones hembra C57BL/J6 de 8 a 16 semanas de edad como se describió anteriormente 26,29,32. Se debe permitir que el animal se aclimate durante al menos 30 minutos después del transporte. Los traslados a larga distancia (es decir, entre institutos) deben evitarse el mismo día del experimento. La edad, el sexo y la cepa del animal deben determinarse en función de la pregunta específica del estudio. - Mientras el animal se está aclimatando y la solución con alto contenido de sacarosa se está enfriando, coloque las herramientas necesarias en cada espacio de trabajo designado (consulte la Tabla de materiales).
- Prepare el espacio de trabajo de recuperación y mantenimiento de cortes de cerebro. Llene la cámara de recuperación de cortes con una solución de registro de LCR carbugenada y coloque la cámara en el baño de agua ajustado a 32 °C. Mantenga una carbogenación continua durante todo el experimento.
- Prepare el espacio de trabajo de preparación de cortes de cerebro. Configure el vibrátomo: coloque la cuchilla en el soporte de la cuchilla del vibrátomo y calibre el vibratomo con la configuración correcta (velocidad de desplazamiento de la hoja: 0,20 mm/s, amplitud de altura: 95 μm, ángulo de la hoja: 45°). Llene la bandeja de hielo vibratoto con hielo y la bandeja tampón con una solución de corte con alto contenido de sacarosa y comience a carbogenar la solución en la bandeja tampón.
- Preparar el espacio de trabajo de preparación cerebral. Llene la placa de Petri de vidrio de 150 mm con hielo y coloque una placa de cultivo de plástico de 90 mm con papel de filtro en su interior. Llene la placa de cultivo de plástico con una solución de corte con alto contenido de sacarosa y comience a carbogenar. Agregue una gota de superpegamento a la placa de muestra enfriada y coloque el molde de agarosa.
NOTA: El molde de agarosa se prepara al menos el día anterior con un 3% de agarosa en agua en un molde de cerebro de ratón personalizado. - Por último, prepare el espacio de trabajo de extracción cerebral. Cubra el papel de aluminio con papel de seda, recupere un vaso de precipitados de 50 ml que contenga aguanieve de solución de corte con alto contenido de sacarosa y agregue isoflurano a la cámara de anestesia.
NOTA: La anestesia se agregará a la cámara de anestesia ~ 1 minuto antes de la colocación del animal. Un vaso de precipitados de 50 ml con 30 ml de granizado de solución de corte con alto contenido de sacarosa se retirará del congelador a -20 °C ~ 2 min antes de la decapitación.
- Lleva al animal a la sala experimental.
- Extracción y corte del cerebro de ratón
NOTA: Todo este procedimiento debe realizarse lo más rápido posible para evitar la falta de oxigenación del cerebro. La extracción del cerebro solo debe tomar de 1 a 2 minutos desde la decapitación hasta la inmersión en el fango de la solución de corte con alto contenido de sacarosa.- Anestesiar al animal con la dosis adecuada de isoflurano (0,5 mL/1 L cámara de anestesia). Determine la profundidad de la anestesia a través de un pellizco en la pata; Confirme la falta de reflejo de retirada de la pata antes de continuar.
- Transfiera al animal al papel de seda en el espacio de trabajo de extracción de cerebro y decapitánelo con tijeras quirúrgicas.
- Inserte las tijeras de iris en el tronco encefálico y mantenga las tijeras inferiores al ras de la calvaria. Cortar a lo largo de la sutura sagital hasta llegar a la sutura coronal. Coloque las tijeras de iris en las cuencas de los ojos y corte a través de la sutura metópica. Use pinzas curvas para mover los lados de la pantorrilla hacia abajo, exponiendo todo el cerebro.
NOTA: Tenga cuidado tanto con las tijeras de iris como con las pinzas para no perforar el cerebro mientras corta las suturas. - Deslice el cerebro con el borde romo de las pinzas curvas en el vaso de precipitados de 50 ml con 30 ml de granizado de solución de corte con alto contenido de sacarosa. Déjalo reposar durante 1 min.
- Transfiera el cerebro a la placa de cultivo de plástico de 90 mm con solución de corte carbogenada refrigerada en el espacio de trabajo de preparación del cerebro. Orientar el cerebro para colocarlo en el molde de agarosa.
- Agrega un pequeño punto de superpegamento al extremo rostral del molde de agarosa. Coloca el cerebro en el molde con la espátula. Asegúrese de que el cerebro esté colocado con el lado dorsal hacia abajo para cortarlo horizontalmente.
NOTA: La ubicación del pegamento en el molde cambiará según la región de interés (ROI). En el caso de las cortes del hipocampo cortical (HC) y del bulbo olfatorio (OB), asegúrese de que el OB esté estabilizado y que los lados del cerebro permanezcan libres de pegamento. Demasiado pegamento afectará la calidad del corte y causará desgarros durante el corte del vibráomo. - Mueva la placa de la muestra a la bandeja de amortiguación, mueva la cuchilla a su posición con el ángulo correcto y aumente la altura de la bandeja de la muestra para acercar la cuchilla lo más posible al cerebro.
- Cortar a intervalos de 300 μm a una velocidad de 0,20 mm/s de tejidos HC y OB, y luego recogerlos después de cada ronda de corte con una pipeta Pasteur de vidrio.
- Deje las rodajas en la cámara de recuperación llena de líquido cefalorraquídeo en un baño de agua a 32 °C durante 45 minutos, seguido de 1 h a temperatura ambiente. Asegúrese de que las rodajas no se superpongan y estén completamente expuestas a la solución carbógena.
NOTA: Asegúrese de mantener una carbogenación continua de todas las soluciones y de las cámaras mencionadas que contengan la solución. Se puede usar un regulador de presión para mantener una carbogenación constante.
2. Red neuronal humana in vitro basada en iPSC en HD-MEA
NOTA: Todas las neuronas iPSC utilizadas en este estudio se obtienen comercialmente (ver Tabla de Materiales). Estas células humanas se diferenciaron de las líneas celulares iPS estables que se derivaron de sangre periférica humana o fibroblastos.
- Recubrimiento de chips HD-MEA para cultivos celulares iPSC humanos in vitro (Figura 2B)
- Coloque el chip HD-MEA en la plataforma de grabación de adquisición, llene el depósito con PBS y pruebe el chip antes del recubrimiento. Inicie el software Brainwave. Seleccione Archivo > nueva sesión de grabación. Configure los parámetros de grabación para que tengan una frecuencia de grabación de 50 Hz y una frecuencia de muestreo de 18 kHz/electrodo. Cambie el desplazamiento del amplificador para calibrar el chip. Consulte la Tabla 2 para obtener consejos para la solución de problemas.
NOTA: Los parámetros de frecuencia de grabación y frecuencia de muestreo dependerán del tipo de datos y de los requisitos individuales del sistema. - Esterilizar y preacondicionar los HD-MEA.
- Debajo del capó, limpie el chip y el anillo de vidrio con un pañuelo humedecido con etanol al 96 % (EtOH), luego coloque cada dispositivo en una placa de Petri estéril de 100 mm x 20 mm y llene el depósito de MEA con EtOH al 70 % durante 20 minutos.
- Aspire el EtOH y lave el depósito con agua filtrada estéril 3 veces. Añadir 1 ml de medio de preacondicionamiento e incubar durante la noche a 37 °C y 5% deCO2.
NOTA: Los medios de preacondicionamiento deben ser una solución a base de sal para que la superficie HD-MEA sea más hidrófila. Esto puede incluir medios completos de BrainPhys (BP) previamente preparados (no >3 meses de antigüedad) (Tablas 1C).
- Recubrimiento HD-MEAs. Al día siguiente, aspire los medios de preacondicionamiento. Añadir 1 ml de 0,1 mg/ml de poli-dl-ornitina (PDLO) para cubrir toda la zona activa. Incubar a 37 °C durante la noche en una incubadora.
- Prepare y caliente los medios para RT. Los protocolos aquí explotan neuronas iPSC humanas funcionales de dos fuentes comerciales; Por lo tanto, los componentes de los medios varían para cada proveedor. Un protocolo se describe en las Tablas 1C, D).
- Aspirar PDLO, lavar 3 veces con agua dd y dejar secar las virutas bajo el capó durante 10 min.
- Llene una placa de Petri de 35 mm x 10 mm con agua dd filtrada estéril y colóquela junto a la viruta para mantener la humedad adecuada y evitar la evaporación de las células sembradas en los siguientes pasos.
- Coloque el chip HD-MEA en la plataforma de grabación de adquisición, llene el depósito con PBS y pruebe el chip antes del recubrimiento. Inicie el software Brainwave. Seleccione Archivo > nueva sesión de grabación. Configure los parámetros de grabación para que tengan una frecuencia de grabación de 50 Hz y una frecuencia de muestreo de 18 kHz/electrodo. Cambie el desplazamiento del amplificador para calibrar el chip. Consulte la Tabla 2 para obtener consejos para la solución de problemas.
- Recubrimiento y mantenimiento de neuronas iPSC humanas en HD-MEAs (Figura 2B)
- Descongele y diluya las células hasta las células deseadas por concentración de microlitros (es decir, 1000 células/μL para obtener 50.000 células de densidad en una gota de 50 μL en el HD-MEA) (Tabla 1C).
- Pipetear la suspensión celular en la superficie del área activa de la viruta utilizando medios de punteado de alta laminina (Tabla 1D).
- Incubar a 37 °C con 5% de CO2 durante 45-60 min.
- Llene suavemente 2 ml de medio en el depósito de HD-MEA (Tabla 1C).
- Realice un cambio de medio del 100 % en el día 1 (DIV1) después de la siembra utilizando medios RT (Tabla 1C). Cambie el 50% de los medios cada 3-4 días. Mantener los HD-MEAs incubados a 37 °C con 5% de CO2 durante todo el experimento.
NOTA: Pipetear suavemente para evitar que se desprendan las células. Compruebe el color del medio para ver si hay contaminación. El intervalo y la cantidad de cambio de medio se pueden determinar mediante preguntas de estudio individuales o necesidades/especificaciones de la celda. - Opcional: Compruebe el progreso del crecimiento del cultivo celular entre DIV4-DIV8 bajo un microscopio vertical de contraste de interferencia diferencial (DIC) después de limpiar la platina con EtOH al >70%.
3. Grabaciones neuronales a gran escala ex-vivo e in vitro con HD-MEAs
- Preparación del espacio de trabajo de grabación de cortes de cerebro (Figura 2A)
- Mientras se recuperan los cortes de cerebro, coloque las herramientas necesarias en cada espacio de trabajo designado (consulte la Tabla de materiales).
NOTA: La configuración del sistema principal debe optimizarse y probarse mucho antes del día experimental del corte de cerebro. El sistema de perfusión (líneas de entrada, líneas de salida de la bomba, tubería y puesta a tierra) debe probarse con PBS o aCSF y un HD-MEA en la plataforma de grabación para garantizar una señal limpia, una mayor relación señal-ruido y la ausencia de ruido de perfusión. - Cubra el chip HD-MEA con 0,1 mg/ml de PDLO para mejorar el acoplamiento tejido-chip e incube a 37 °C durante 20 min.
- Durante la incubación de las virutas, llene el sistema de perfusión basado en la gravedad y las líneas con aCSF de registro. Asegurar la carbogenación continua del sistema de perfusión. Ajuste un caudal de 4,5 ml/min y una temperatura de 37 °C.
- Coloque el chip HD-MEA en la plataforma de grabación de adquisición, llene el depósito con aCSF, pruebe el sistema de perfusión y solucione los problemas de ruido restante del sistema.
- Inicie el software Brainwave. Seleccione Archivo > nueva sesión de grabación. Configure los parámetros de grabación para que tengan una frecuencia de grabación de 1 Hz y una frecuencia de muestreo de 14 kHz/electrodo. Cambie el desplazamiento del amplificador para calibrar el chip. Consulte la Tabla 2 para obtener consejos para la solución de problemas.
NOTA: Los parámetros de frecuencia de grabación y frecuencia de muestreo dependerán del tipo de datos y de los requisitos individuales del sistema.
- Inicie el software Brainwave. Seleccione Archivo > nueva sesión de grabación. Configure los parámetros de grabación para que tengan una frecuencia de grabación de 1 Hz y una frecuencia de muestreo de 14 kHz/electrodo. Cambie el desplazamiento del amplificador para calibrar el chip. Consulte la Tabla 2 para obtener consejos para la solución de problemas.
- Asegúrese de que el área de grabación esté oscura a través de un sistema de iluminación de la habitación o una jaula sombreada en la mesa óptica.
- Alinee el microscopio estereoscópico con el depósito del chip HD-MEA y el área activa para la adquisición de imágenes.
- Coloque el ancla en el depósito de virutas para equilibrar.
NOTA: Anchor es un arpa de platino hecha a medida con alambres mínimos para promover la oxigenación; sin embargo, algunos comerciales están disponibles. - Añadir compuestos farmacológicos a los tubos de perfusión adecuados.
NOTA: En este protocolo, se obtuvieron registros inducidos farmacológicamente tanto espontáneos como de 100 μM de 4-aminopiridina (4-AP) como se describió anteriormente. Los compuestos farmacológicos se pueden adaptar a la pregunta específica del estudio. - En el espacio de trabajo de preparación de cortes de cerebro, coloque una nueva placa de cultivo de plástico de 90 mm en una placa de Petri de vidrio de 150 mm. Agregue aCSF y comience a carbogenar.
- Mientras se recuperan los cortes de cerebro, coloque las herramientas necesarias en cada espacio de trabajo designado (consulte la Tabla de materiales).
- Grabaciones de todo el circuito a partir de cortes HC y OB utilizando HD-MEA
NOTA: El acoplamiento de la rebanada debe realizarse lo más rápido posible para evitar la falta de oxigenación de la rebanada. El acoplamiento solo debe tomar ~ 1 minuto desde la colocación inicial de la rebanada microdiseccionada en el área activa del chip hasta el inicio final del sistema de perfusión.- Retire la rebanada de la cámara de recuperación de rebanadas de cerebro con una pipeta de vidrio y colóquela en una placa de cultivo de plástico de 90 mm con carbogenación continua. Con una herramienta de microdisección, aísle el HC u OB del tejido circundante del corte de cerebro.
- Mueva los cortes agudos aislados de HC u OB con una pipeta de vidrio al depósito HD-MEA. Alinee suavemente la rebanada en el área activa de MEA con un cepillo fino. Aspire bien todas las soluciones del chip HD-MEA con un sistema de aspiración.
- Coloque el anclaje suavemente sobre la rebanada con pinzas.
NOTA: El anclaje debe colocarse sin movimiento de la rebanada para evitar la pérdida de acoplamiento. - Agregue suavemente la solución al depósito de virutas y ponga en marcha el sistema de perfusión.
NOTA: Asegure el flujo laminar desde la entrada de perfusión y la salida de la bomba para obtener parámetros de registro óptimos. - Asegúrese de que el área de grabación esté adecuadamente atenuada a través del sistema de iluminación de la sala o con una jaula sombreada en una configuración de mesa óptica.
- Deje que el corte se aclimate durante 10 minutos antes de comenzar las grabaciones o la modulación farmacológica adicional.
- Inicie el software Brainwave. Seleccione Archivo > nueva sesión de grabación. Configure los parámetros de grabación para que tengan una frecuencia de grabación de 1 Hz y una frecuencia de muestreo de 14 kHz/electrodo. Cambie el desplazamiento del amplificador para calibrar el chip.
NOTA: Como se indicó anteriormente en la sección 3.1.4.1, al realizar pruebas del sistema, asegúrese de aplicar estos mismos parámetros de registro. - Pulse Grabar para comenzar la adquisición con las condiciones experimentales preestablecidas.
- Inmediatamente después de la grabación final, capture imágenes de luz del corte agudo del cerebro. Mueva la rebanada de regreso a la cámara de recuperación de rebanadas, retire cualquier material orgánico acoplado a la viruta con un cepillo y continúe con la siguiente rebanada. Limpie los HD-MEA como se describe en la sección 3.4.
- Preparación del espacio de trabajo de grabación iPSC humano y grabaciones de toda la red en HD-MEA (Figura 2B)
NOTA: Cambie el medio el día antes de la grabación o inmediatamente después de la grabación de iPSC humana (Tabla 1C). En los estudios que utilizaron las neuronas funcionales, los medios se cambiaron cada 4 días, y a los 4, 8, 16 y 24 DIV los medios se cambiaron inmediatamente después de los registros de iPSC.- Garantice un entorno de trabajo estéril limpiando la plataforma de adquisición HD-MEA con >70% de EtOH.
- Coloque suavemente una tapa con base de polidimetilsiloxano (PDMS) con referencia en el anillo HD-MEA debajo del capó. Mueva el chip HD-MEA al espacio de trabajo de grabación iPSC y conecte el chip HD-MEA a la plataforma de adquisición.
- Asegúrese de que el área de grabación esté adecuadamente atenuada a través de un sistema de iluminación de la habitación o una jaula sombreada en una mesa óptica.
- Deje que el chip HD-MEA se equilibre durante 10 minutos antes de comenzar las grabaciones o la modulación farmacológica adicional.
- Inicie el software Brainwave. Seleccione Archivo > nueva sesión de grabación. Configure los parámetros de grabación para que tengan una frecuencia de grabación de 50 Hz y una frecuencia de muestreo de 18 kHz/electrodo. Cambie el desplazamiento del amplificador para calibrar el chip.
NOTA: Como se indicó anteriormente en la sección 2.1.1, al realizar una prueba del sistema antes del recubrimiento y el enchapado, asegúrese de aplicar estos mismos parámetros de registro. - Registre la actividad de disparo espontáneo o las respuestas inducidas farmacológicamente de la red de iPSC humana en cada día del plan experimental (es decir, 4, 8, 16, 24 DIV).
NOTA: No permita que el chip permanezca fuera de la incubadora durante >30 minutos para mantener una temperatura y humedad estables y evitar cualquier choque de temperatura en las células. - Incubar HD-MEAs a 37 °C con 5% de CO2 durante el transcurso del experimento.
- Una vez finalizado el experimento, fije la red neuronal en los chips y tiña para obtener más imágenes ópticas o limpie los HD-MEA directamente, como se describe en el paso 3.4.
- Limpieza de chips HD-MEA
- Después del experimento, deseche la solución de acuerdo con la eliminación adecuada de los desechos y enjuague con agua dd.
- Agregue el detergente de su elección, limpie el área activa y todo el depósito con un hisopo y deseche el detergente. Vuelva a llenar con detergente, incube durante 20 minutos y luego deseche el detergente.
- Enjuague bien con agua de laboratorio. Luego, enjuague 3-4 veces con agua dd.
- Utilice presión de aire para secar completamente el chip HD-MEA.
4. Análisis de grabaciones neuronales a gran escala a partir de HD-MEAs
NOTA: Si bien el paso 4.1 es específico del software Brainwave, el paso 4.2 se puede modificar en función del tipo de dispositivo HD-MEA disponible comercialmente de cada usuario.
- Preprocesamiento de datos sin procesar y detección de eventos
- Abra un archivo de datos sin procesar grabado (.brw) en el software Brainwave. Seleccione Análisis > Detección de LFP o Detección de picos.
NOTA: La detección LFP emplea un filtrado IIR con un filtro Butterworth de paso bajo de4º orden (1-100 Hz). Los algoritmos de umbral duro incluyen un umbral alto de 150 μV, un umbral bajo de -150 μV, una ventana de energía entre 70 y 120 ms, un período refractario de 10 ms y una duración máxima del evento de 1 s. La detección de picos simples y MUA emplea un filtrado IIR con un filtro Butterworth de paso alto de4º orden (300-3500 Hz). Se aplica un algoritmo de TEPT con un factor de desviación estándar de 8, un período de vida útil máximo de 2 ms y un período refractario de 1 ms. - Para las grabaciones de circuitos HC y OB, agregue la opción Espacio de trabajo avanzado en el archivo de eventos detectado (.bxr) para importar la imagen de luz estructural capturada desde el microscopio estereoscópico. Al examinar los circuitos de HC a gran escala, cree capas estructurales que contengan la circunvolución dentada (DG), el hilio, Cornu Ammonis 1 (CA1), Cornu Ammonis 3 (CA3), la corteza entorrinal (EC) y la corteza perirrinal (PC). Al examinar los circuitos de OB a gran escala, cree capas estructurales que contengan la capa nerviosa olfatoria (ONL), la capa glomerular (GL), la capa plexiforme externa (EPL), la capa de células mitrales (MCL) y la capa de células granulosas (GCL). Considere la EPL y la MCL como la capa de proyección (PL), incluida la corteza olfativa (OCx).
- Abra un archivo de datos sin procesar grabado (.brw) en el software Brainwave. Seleccione Análisis > Detección de LFP o Detección de picos.
- Procesamiento de datos con una canalización computacional de Python personalizada
- Eliminación de ruido
- Lea el archivo .bxr utilizando un script de Python escrito a medida 26,29,32 y un paquete de python h5py 3.6.0.
- Extraiga los trenes de picos pertenecientes a las grabaciones de la red iPSC y los trenes de eventos LFP pertenecientes a las grabaciones del circuito de corte cerebral HC y OB.
- Caracterice los eventos con un número total de electrodos activos inferior al 0,1 % o al 10 % de los electrodos activos promedio por evento promedio o los eventos detectados que caen fuera de un rango de velocidad de disparo estadísticamente razonable como eventos aleatorios y elimínelos. Además, aplique valores de umbral de amplitud y duración del evento.
NOTA: Para el rango de velocidad de disparo, se consideran 0,1-15 picos/s y 0,1-60 eventos LFP/min. Estos son valores de umbral de tasa de ejemplo utilizados para los conjuntos de datos analizados. Los umbrales de velocidad, amplitud y duración dependerán de los datos individuales. - Guarde los datos del tren de eventos resultante con la información espaciotemporal adjunta en formato de archivo .npy.
- Rastergramas
- Lea archivos .npy y .bxr de eventos filtrados y genere una gráfica ráster utilizando la función Matplotlib pyplot (https://matplotlib.org/3.5.3/api/_as_gen/matplotlib.pyplot.html).
- Además, para los registros de cortes de cerebro con especificidad de capa, ordene y agrupe los ID de los electrodos en función de las capas producidas en el paso 4.1.2.
- Actividad media de disparo
- Procese los datos de series temporales del archivo .bxr, calculando la velocidad media de disparo de cada electrodo (número de eventos/tiempo de grabación).
- Construya una matriz de datos en la que las filas y columnas representen las coordenadas de los electrodos en la matriz HD-MEA 64 x 64, donde cada valor de la matriz signifique la velocidad media de disparo.
- Emplee una biblioteca de trazado como imshow de Matplotlib o las funciones de mapa de calor de Seaborn en Python.
- Emplee el mapa de colores "calientes" aquí, creando un mapa de calor informativo que encapsula visualmente la distribución espacial de las velocidades medias de disparo en toda la guía de electrodos.
- Trazas de forma de onda representativas
- Lea los datos de series temporales del archivo .brw y genere una traza de forma de onda utilizando la función pyplot de Matplotlib. (https://matplotlib.org/3.5.3/api/_as_gen/matplotlib.pyplot.html).
- Ingrese el ID de electrodo deseado, el intervalo de tiempo y la banda de frecuencia para obtener una traza de forma de onda representativa. Las bandas de frecuencia definidas en estos análisis incluyen oscilaciones LFP de baja frecuencia (1-100 Hz) con bandas de frecuencia filtradas de paso de banda δ, θ, β y γ; ondulaciones de onda aguda (ROE) (140-220 Hz); y simple de alta frecuencia y MUA (300-3500 Hz). Las bandas de frecuencia δ, θ, β y γ son 1-4 Hz, 5-12 Hz, 13-35 Hz y 35-100 Hz respectivamente.
- Densidad espectral de potencia
- Lea los datos de series temporales del archivo .brw y calcule los periodogramas para discernir las frecuencias dominantes subyacentes a la actividad oscilatoria dentro de cada serie temporal.
- Construya espectrogramas de pseudo-color de la dinámica frecuencia-tiempo.
NOTA: Los espectros se calculan utilizando el método de Welch utilizando la transformada rápida de Fourier de los LFP registrados para estimar la densidad de potencia espectral41. - Ingrese la identificación del electrodo deseado, el intervalo de tiempo y la banda de frecuencia para obtener un mapa de densidad espectral. Las bandas de frecuencias definidas en estos análisis incluyen las descritas en el paso 4.2.4.
- Conectividad funcional
- Para las grabaciones de circuitos de corte de cerebro, siga los pasos 4.2.6.2-4.2.6.4.
- Lea los datos de series temporales del archivo .brw y calcule la covarianza cruzada entre pares de electrodos activos en la matriz de 64 x 64 empleando el coeficiente de correlación de Pearson (PCC)42.
- Ajuste un modelo autorregresivo vectorial a la serie temporal utilizando la causalidad multivariante de Granger para cuantificar la influencia de una serie temporal sobre otra.
- Aplique la función de transferencia dirigida (DTF) para evaluar el flujo de información direccional dentro de los enlaces correlacionados.
NOTA: La conectividad funcional en la red multicapa se establece estableciendo un umbral de valor de correlación basado en los que están por encima de la media y dos desviaciones estándar de todos los valores de covarianza cruzada43,44. - Para la grabación de iPSC, siga los pasos 4.2.6.6-4.2.6.8.
- Lea los datos de los trenes de picos del archivo .bxr y calcule una matriz de 64 x 64 de los coeficientes de correlación PCC entre todas las combinaciones de trenes de picos agrupados utilizando funciones spike_train_correlation (https://elephant.readthedocs.io/en/v0.7.0/reference/spike_train_correlation.html).
NOTA: La conectividad funcional en la red multicapa se establece estableciendo un umbral de valor de correlación basado en los que están por encima de la media y dos desviaciones estándar de todos los valores de covarianza cruzada. - Además, implementar filtros espacio-temporales (STF) y umbrales de latencia dependientes de la distancia (DdLT) en la matriz de conectividad para eliminar las posibles conexiones emparejadas que superen la velocidad máxima de propagación (establecida en 400 mm/s)45.
- Extraiga picos negativos de matrices de correlación cruzada resultantes con operaciones de filtrado y umbral para identificar las conexiones inhibitorias utilizando el algoritmo de histograma de correlación cruzada filtrado y normalizado (FNCCH)45.
- Transforme cada matriz de conectividad en un archivo de gráfico dinámico (.gexf).
- Mapas de conectividad de red
- Laboratorio de datos abiertos en el programa Gephi versión 9.2 (https://gephi.org) para el gráfico dinámico para trazar intervalos de tiempo específicos.
- Aplique Diseño geográfico en la ventana de diseño para la representación cartográfica espacial.
- Coloque restricciones de parámetros en Rango de grados y Grosor de borde para comparar.
- Asigne el color del nodo, el tamaño del borde y el tamaño del grado para una mejor visualización.
- Eliminación de ruido
Resultados
Mapeo espacio-temporal multimodelo y extracción de características de disparo oscilatorio
Para cuantificar los eventos de LFP y picos en toda la red que surgieron de conjuntos neuronales dinámicos, investigamos patrones de disparo sincrónicos a gran escala en circuitos HC y OB y redes iPSC humanas. Los circuitos de corte de cerebro registrados en el paso 3.2 y las redes iPSC registradas en el paso 3.3 se analizaron de acuerdo con los pasos 4.1-4.2 del protocolo. En primer lugar, se realizaron la detección de eventos y la eliminación de ruido para todos los conjuntos de datos registrados y se resolvieron regionalmente de acuerdo con las especificaciones del circuito. A continuación, se trazó un mapeo espacial topográfico pseudo-color de los patrones medios de disparo de LFP y picos a gran escala, rastergramas de eventos detectados y trazas representativas de 5-s de formas de onda filtradas (Figuras 3 A-I). El mapeo topográfico de pseudocolor de los patrones de LFP y velocidad de disparo de picos a gran escala se superpuso en las respectivas imágenes ópticas capturadas por el microscopio de HC (Figura 3A), OB (Figura 3B) y la red neuronal iPSC humana (Figura 3C). Esto permite la investigación de patrones y respuestas oscilatorias individuales basadas en circuitos y redes. Los rastergramas HC y OB contienen recuentos de eventos LFP detectados ordenados en las capas DG, Hilus, CA3, CA1, EC y PC del circuito HC y las capas ONL, OCx, GL, PL y GCL de la red OB durante un intervalo de tiempo de 60 s (Figuras 3D, E). El rastergrama iPSC humano muestra los eventos de pico detectados sincrónicamente de la red cultivada interconectada durante un intervalo de tiempo de 20 s (Figura 3G). A continuación, las trazas de eventos representativos de 5s de sitios de registro HD-MEA a gran escala muestran un rango de frecuencias oscilatorias registradas en los circuitos HC (es decir, electrodo seleccionado en CA3) (Figura 3G) y OB (es decir, electrodo seleccionado en GL) (Figura 3H) y actividad de estallido de picos multiunidad en la red iPSC humana de cuatro electrodos activos seleccionados en la matriz (Figura 3I). Estas señales ejemplares muestran firmas de bioseñales, incluidas oscilaciones LFP de baja frecuencia (1-100 Hz) con bandas de frecuencia filtradas de paso de banda δ, θ, β y γ; ondulaciones de onda aguda (ROE) (140-220 Hz); y simple de alta frecuencia y MUA (300-3500 Hz). Finalmente, se empleó el análisis de densidad espectral de potencia (PSD) para cuantificar simultáneamente la magnitud de potencia de una banda oscilatoria específica en el circuito HC y OB interconectado registrado desde HD-MEA (Figuras 3J, K).
Conectoma funcional multimodal para toda la red
Para inferir la conectividad a gran escala de las redes neuronales multicapa a partir de patrones de disparo simultáneo de conjuntos neuronales activos simultáneamente, se calculó la covarianza cruzada entre pares de electrodos activos en eventos detectados de acuerdo con el paso 4.2.6 del protocolo. Aquí, el coeficiente de correlación se clasificó en función de las capas en el circuito HC y OB o se desclasificó en la red iPSC y luego se almacenó en una matriz simétrica. Los conectomas funcionales de los circuitos HC y OB se generaron aplicando la causalidad multivariante de Granger y la función de transferencia dirigida (DTF) para cuantificar la influencia de una serie temporal sobre otra y evaluar el flujo de información direccional dentro de los enlaces correlacionados en las distintas redes. El mapeo del conectoma de HC (Figura 4A) y OB (Figura 4B) y la visualización de la red se realizaron utilizando el programa Gephi versión 9.2 (https://gephi.org). Se impusieron restricciones de parámetros similares a los enlaces funcionales para comparar los circuitos de corte de cerebro HC y OB e ilustraron 100 s de la conectividad funcional de los eventos LFP detectados. Los nodos se escalan de acuerdo con el grado de fuerza, con el color nodal que indica la capa y el color del enlace que identifica las conexiones dentro y entre capas. Los conectomas funcionales de las redes iPSC humanas se generaron mediante la aplicación de filtros espacio-temporales (STF) y umbrales de latencia dependientes de la distancia (DdLT) para mejorar la selección de enlaces significativos y refinar la identificación de conexiones significativas mediante la aplicación de un análisis de histograma de correlación cruzada filtrado y normalizado (FNCCH). Mapeo de conectoma de redes iPSC humanas en todo el chip HD-MEA (Figura 4C) visualización realizada con Gephi. El color nodal indica la entrada excitatoria o inhibitoria, y el color del enlace identifica las conexiones.
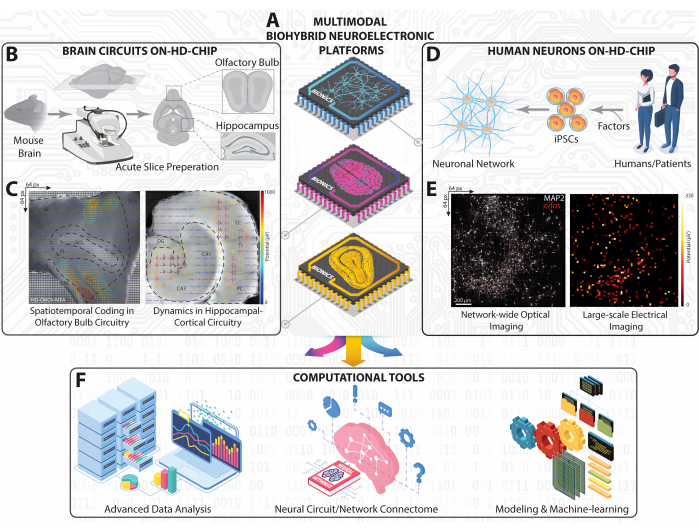
Figura 1: Visión general de la plataforma experimental y computacional en HD-MEA a gran escala. (A) Representación esquemática isométrica de nuestras plataformas neuroelectrónicas biohíbridas multimodales realizadas con HD-MEA basadas en CMOS para capturar la dinámica neuronal de los circuitos y redes neuronales de HC, OB y iPSC humanas. (B) Flujo de trabajo esquemático para el corte del cerebro del ratón y su paisaje de trabajo para obtener cortes de HC y OB. (C) Representaciones topográficas de los patrones de disparo a gran escala registrados simultáneamente a partir de los cortes completos de HC y OB superpuestos con las formas de onda extracelulares extraídas a las imágenes ópticas del corte. (D) Representación esquemática de la red neuronal iPSC obtenida de humanos. (E) Micrografías de fluorescencia que muestran c-fos celulares y MAP-2 somático/dendrítico de toda la red neuronal humana en el chip HD-MEA (izquierda) emparejadas con todo el mapa de actividad de disparo promedio (derecha). (F) Marco computacional que incluye análisis avanzado de datos, mapeo de conectividad y herramientas de aprendizaje automático de IA para analizar datos neuronales multidimensionales obtenidos de grabaciones a gran escala en HD-MEA. Haga clic aquí para ver una versión más grande de esta figura.

Figura 2: Diseños de los espacios de trabajo de preparación y registro de cortes de cerebro ex vivo y cultivos de iPSC humanos in vitro . (A) Flujo de trabajo esquemático que ilustra la configuración para la preparación de cortes de HC y OB, con las herramientas y el equipo necesarios en cada espacio de trabajo. (B) Representación esquemática para la preparación del cultivo humano de iPSC, incluidas las herramientas y dispositivos necesarios. Se incluye una lista completa de materiales en los pasos 1.2.2, 2.1, 2.2, 3.1.1, 3.3 y la Tabla de materiales. Haga clic aquí para ver una versión más grande de esta figura.
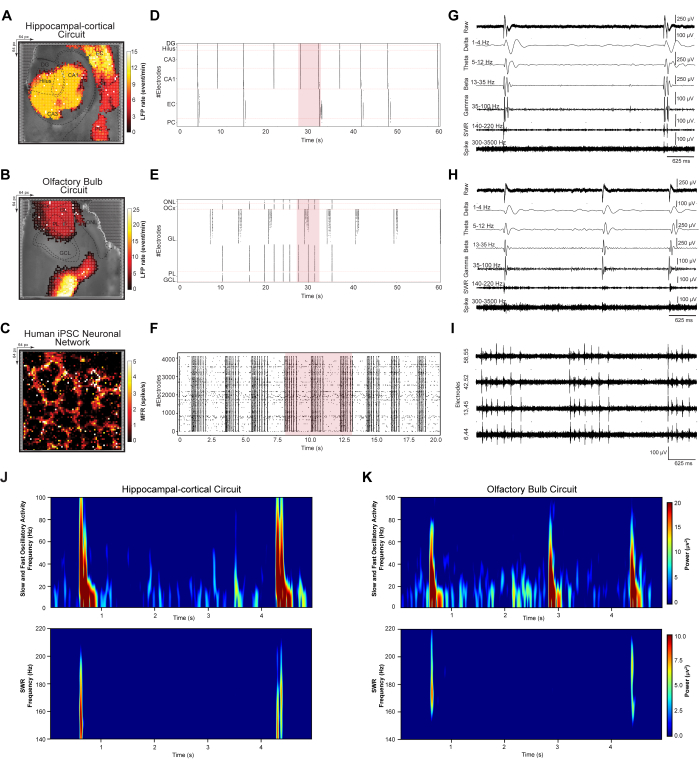
Figura 3: Mapeo y extracción de patrones espaciotemporales de la dinámica de redes. (A-C) Mapas espaciales de LFP media y tasa de pico, calculados a lo largo de grabaciones de cinco minutos, superpuestos a la imagen de luz del microscopio. (D-F) Gráficos ráster que representan eventos LFP detectados y sin ruido en una submuestra de datos de 60 segundos y picos en una submuestra de datos de 20 segundos. (G-I) Extracción representativa de trazas de forma de onda de un segmento de 5 segundos de la submuestra de datos de la gráfica ráster (resaltada en rojo en la gráfica ráster), que se muestra como bandas oscilatorias LFP sin procesar (1-100 Hz); δ (1-4 Hz), θ (5-12 Hz), β (13-35 Hz) y γ (35-100 Hz) bandas de frecuencia; ROE (140-220 Hz); y picos simples y MUA de alta frecuencia (300-3500 Hz). (J,K) Mapas de densidad espectral de potencia de LFPs oscilatorios rápidos y lentos (1-100 Hz) y ROE (140-220 Hz). Haga clic aquí para ver una versión más grande de esta figura.
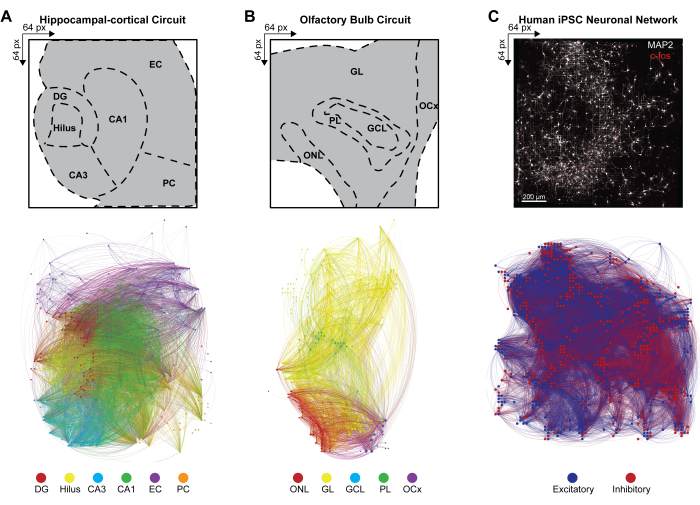
Figura 4: Organización de conectomas funcionales multimodales en toda la red. (A-C) Gephi mapea que ilustra la conectividad funcional nodal, donde los nodos corresponden a una de las leyendas de la barra de color de ejemplo (abajo), mientras que los enlaces (o bordes) están sombreados para que coincidan con los nodos de conexión. Las leyendas de ejemplo para las capas (A) HC, (B) OB y (C) iPSC se muestran en una matriz de 64 x 64. Las capas HC y OB se trazan en un intervalo de tiempo de 100 s para reducir eficazmente el número de nodos y vínculos visibles con fines de visualización. Haga clic aquí para ver una versión más grande de esta figura.
Tabla 1: Soluciones para la preparación de cortes de cerebro y medios para cultivos neuronales iPSC. (A) Solución de corte con alto contenido de sacarosa para la preparación de cortes de cerebro ex vivo. (B) solución de registro de aCSF para la preparación y grabación de cortes de cerebro ex vivo. (C-D) Protocolo de medios iPSC neuronales humanos, donde (C) es el medio completo de BrainPhys utilizado para la descongelación celular, el recubrimiento de chips HD-MEA y el mantenimiento de HD-MEA cultivado, y (D) el medio de punteado utilizado para la colocación de placas celulares HD-MEA. Haga clic aquí para descargar esta tabla.
Tabla 2: Solución de problemas comunes de adquisición de grabaciones HD-MEA. Una lista de problemas comunes, sus posibles causas y soluciones de solución de problemas relacionados con los chips HD-MEA, la plataforma de grabación, el ruido del sistema y el software. Haga clic aquí para descargar esta tabla.
Discusión
La intrincada dinámica de la actividad neuronal espacio-temporal, que emerge de conjuntos neuronales interconectados, ha sido durante mucho tiempo un tema de intriga en la neurociencia. Las metodologías tradicionales, como la pinza de parche, la MEA estándar y las imágenes de Ca2+, han proporcionado información valiosa sobre la complejidad del cerebro. Sin embargo, a menudo se quedan cortos a la hora de capturar la dinámica computacional integral de toda la red 21,22,23. El protocolo técnico de la plataforma HD-MEA, tal y como se detalla en este estudio de JoVE, representa un importante salto adelante, ya que ofrece una visión panorámica de la dinámica neuronal a través de diversas modalidades, desde ensamblajes celulares hasta redes expansivas (es decir, cortes agudos de cerebro de ratón ex vivo y redes iPSC humanas in vitro)26,29,30,32.
Los cortes agudos de cerebro de ratón ex vivo han sido una herramienta fundamental en la investigación neuronal, facilitando las investigaciones moleculares y a nivel de circuito 6,7. Sin embargo, el reto de mantener la viabilidad de los tejidos ha sido un cuello de botella persistente. El protocolo delineado en este estudio introduce modificaciones críticas para optimizar la calidad y longevidad de estos cortes para explotar sus beneficios en la plataforma HD-MEA. Este protocolo subraya la importancia de: i) Lograr la uniformidad del corte, para lo cual se prefiere el uso de un vibratomo a un picador de tejidos debido a su precisión y al daño tisular minimizado, a pesar de la compensación de tiempos de corte más largos. ii) Asegurar una carbogenación constante durante todo el proceso, desde la extracción hasta el registro, para mantener la viabilidad de los tejidos. iii) Regular la temperatura y permitir un tiempo de recuperación adecuado antes de la grabación. iv) Utilizar un bloque o molde de agarosa para estabilizar el cerebro, evitar el desgarro y minimizar el contacto con el pegamento. v) Mantener caudales óptimos de aCSF carbogenado dentro del reservorio HD-MEA para garantizar la salud de la rebanada y evitar problemas como el desacoplamiento, el ruido y la deriva (Tabla 2).
Tanto para los cortes de cerebro de ratón como para las preparaciones de iPSC humanas, la mejora del acoplamiento de la interfaz electrodo-tejido es primordial 30,46,47. Nuestro protocolo subraya la importancia de utilizar la molécula promotora de adhesión Poli-dl-ornitina (PDLO). Esta molécula no solo aumenta el área de superficie para detectar señales eléctricas, sino que también aumenta la conductividad eléctrica46. Al hacerlo, promueve la adhesión celular, el crecimiento y el desarrollo de propiedades funcionales de la red. Dicha optimización desempeña un papel fundamental en la mejora de la eficacia de la plataforma HD-MEA. Esto, a su vez, garantiza un análisis preciso y coherente de los conectomas ex-vivo e in vitro a microescala y sus secuencias de disparo espacio-temporales. En particular, se ha demostrado que el PDLO supera a otros sustratos como la polietileneimina (PEI) y la poli-l-ornitina (PLO) en la promoción de la actividad de disparo espontáneo y la capacidad de respuesta a los estímulos eléctricos en cultivos neuronales. Además, el PDLO se ha utilizado para la funcionalización de superficies en el HD-MEA y se ha demostrado que mejora la interfaz de acoplamiento electrodo-corte y aumenta la relación señal-ruido en cortes OB y HC26,29. La adición de un anclaje de platino hecho a medida aumenta aún más el acoplamiento de la interfaz electrodo-corte, lo que lleva a grabaciones con una mayor relación señal-ruido.
La utilización de HD-MEA tanto para cortes de cerebro de ratón ex vivo como para redes iPSC humanas in vitro introduce un método experto en explorar dinámicas extensas, multiescala y multimodales. Sin embargo, este enfoque innovador plantea desafíos considerables, especialmente en la gestión de datos 48,49,50,51. Una sola grabación HD-MEA adquirida a una frecuencia de muestreo de 18 kHz/electrodo genera la asombrosa cantidad de 155 MB/s de datos. El volumen de datos aumenta rápidamente cuando se tienen en cuenta múltiples cortes, diversas condiciones farmacológicas o períodos de registro prolongados. Esta afluencia de información requiere infraestructuras de almacenamiento sólidas y herramientas informáticas avanzadas para agilizar el procesamiento. La capacidad de la plataforma HD-MEA para recopilar simultáneamente datos de miles de conjuntos neuronales es tanto una bendición como un obstáculo. Proporciona una visión suprema de la dinámica computacional de las funciones cerebrales, pero también requiere un marco analítico refinado. En este protocolo JoVE, hemos proporcionado ejemplos de estrategias computacionales, incluida la detección de eventos a gran escala, la clasificación, la teoría de grafos, el análisis de frecuencia y el aprendizaje automático. Estos métodos subrayan los intensos esfuerzos realizados para abordar los desafíos del análisis de datos neuronales complejos. No obstante, todavía hay un espacio considerable para el desarrollo de herramientas computacionales más avanzadas para analizar estos conjuntos de datos neuronales multidimensionales. Armada con las herramientas y metodologías adecuadas, se magnifica el potencial de la plataforma HD-MEA, ofreciendo una visión profunda de las complejidades de las funciones cerebrales tanto en condiciones saludables como patológicas.
En esencia, la plataforma HD-MEA, cuando se integra con los protocolos detallados y las herramientas computacionales discutidas, ofrece un enfoque transformador para comprender el intrincado funcionamiento del cerebro. Al capturar dinámicas a gran escala, multiescala y multimodales, proporciona información invaluable sobre procesos como el aprendizaje, la memoria y el procesamiento de la información. Además, su aplicación en redes iPSC humanas in vitro tiene el potencial de revolucionar el cribado de fármacos y la medicina personalizada. Sin embargo, si bien esta plataforma representa un avance significativo en la investigación de la neurociencia, es crucial reconocer y abordar los desafíos técnicos inherentes. Con el perfeccionamiento continuo y la integración de herramientas computacionales avanzadas, la plataforma HD-MEA está preparada para marcar el comienzo de una nueva era de herramientas de diagnóstico precisas, la identificación de biomarcadores específicos y terapias dirigidas para trastornos neurológicos.
Divulgaciones
Los autores declaran no tener intereses contrapuestos ni financieros.
Agradecimientos
Este estudio contó con el apoyo de fondos institucionales (DZNE), la Asociación Helmholtz dentro del Fondo de Validación Helmholtz (HVF-0102) y la Escuela Internacional de Posgrado de Biomedicina y Bioingeniería de Dresde (DIGS-BB). También nos gustaría agradecer a la plataforma de pruebas de comportamiento con animales en el DZNE-Dresden (Alexander Garthe, Anne Karasinsky, Sandra Günther y Jens Bergmann) por su apoyo. Nos gustaría reconocer que una parte de la Figura 1 se creó utilizando la plataforma BioRender.com.
Materiales
| Name | Company | Catalog Number | Comments |
| 150 mm Glass Petri Dish | generic | generic | Brain Preparation Workspace, Brain Slice Recording Workspace |
| 0.22 μm Sterile Filter Unit | Assorted | Assorted | Assorted |
| 90 mm Plastic Culture Dish | TPP | 93100 | Brain Preparation Workspace, Brain Slice Recording Workspace |
| Agarose | Roth | 6351.5 | Brain Preparation Workspace |
| Agarose Mold | CUSTOM | CUSTOM | Brain Preparation Workspace; Custom designed 3D Printer Design, available upon request |
| Aluminum Foil | generic | generic | Brain Extraction Workspace |
| Anesthesia chamber | generic | generic | Brain Extraction Workspace; Assorted Beaker, Bedding etc |
| Ascorbic Acid | Sigma Aldrich | A4544-25G | Solution Preparation Workspace |
| Assorted Beakers | generic | generic | Solution Preparation Workspace; 50 mL |
| Assorted Luers | Cole Parmer | 45511-00 | Brain Slice Recording Workspace |
| Assorted Volumetric flasks | generic | generic | Solution Preparation Workspace; 500 mL, 1 L |
| B27 Supplement | Life Technologies | 17504-044 | BrainXell Commercial Supplier Protocol |
| BDNF | Peprotech | 450-02 | BrainXell Commercial Supplier Protocol |
| Biological Safety Cabinet with UV Lamp | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| BrainPhys Neuronal Medium | STEMCELL Technologies | 05790 | CDI, and BrainXell Commerical Supplier Protocol |
| Brainwave Software | 3Brain AG | Version 4 | Brain Slice and Human iPSC Recording Workspace |
| BrainXell Glutamatergic Neuron Assay | BrainXell | BX-0300 | BrainXell Commercial Supplier Protocol |
| CaCl2 | Sigma Aldrich | 21115-100ML | Solution Preparation Workspace |
| Carbogen | generic | generic | All Workspaces; 95%/5% O2 and CO2 mixture |
| Cell Culture Incubator | Assorted | Assorted | Assorted |
| CMOS-based HD-MEA chip | 3Brain AG | CUSTOM | Brain Slice and Human iPSC Recording Workspace |
| Conical Tubes, 50 mL, Falcon (Centrifuge Tubes) | STEMCELL Technologies | 38010 | CDI Commerical Supplier Protocol |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | Brain Slice Recording Workspace |
| Curved Forceps | FST | 11052-10 | Brain Extraction Workspace |
| DMEM/F12 Medium | Life Technologies | 11330-032 | BrainXell Commercial Supplier Protocol |
| Dulbecco’s Phosphate Buffered Saline without Ca2+ and Mg2+ (D-PBS) | STEMCELL Technologies | 37350 | CDI Commerical Supplier Protocol |
| Filter Paper | Macherey-Nagel | 531 011 | Brain Preparation Workspace |
| Fine Brush | Leonhardy | 773 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Forceps | VITLAB | 67895 | Brain Slice Recording Workspace |
| GDNF | Peprotech | 450-10 | BrainXell Commercial Supplier Protocol |
| Geltrex | Life Technologies | A1413201 | BrainXell Commercial Supplier Protocol |
| Glass pasteur pipette | Roth | 4518 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Glucose | Sigma Aldrich | G7021-1KG | Solution Preparation Workspace |
| GlutaMAX | Life Technologies | 35050-061 | BrainXell Commercial Supplier Protocol |
| Gravity-based Perfusion System | ALA | VC3-8xG | Brain Slice Recording Workspace |
| HD-MEA Recording platform | 3Brain AG | CUSTOM | Brain Slice and Human iPSC Recording Workspace |
| Heater | Warner Instruments | TC-324C | Brain Slice Recording Workspace |
| Hemocytometer or Automated Cell Counter | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| Hypo Needles | Warner Instruments | 641489 | Brain Slice Recording Workspace |
| iCell GlutaNeurons Kit, 01279 | CDI | R1061 | CDI Commerical Supplier Protocol |
| Iris Scissors | Vantage | V95-304 | Brain Extraction Workspace |
| Isoflurane | Baxter | HDG9623 | Brain Extraction Workspace |
| KCl | Sigma Aldrich | P5405-250G | Solution Preparation Workspace |
| Laminin | Sigma-Aldrich | L2020 | CDI Commerical Supplier Protocol |
| Liquid Nitrogen Storage Unit | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| Magnetic Stirrer | generic | generic | Solution Preparation Workspace |
| Metal Screws | Thorlabs | HW-KIT2/M | Brain Slice Recording Workspace |
| MgCl2 | Sigma Aldrich | M1028-100ML | Solution Preparation Workspace |
| MgSO4 | Sigma Aldrich | 63138-250G | Solution Preparation Workspace |
| Microdissection Tool Holder | Braun | 4606108V | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Microdissection Tool Needle | Braun | 9186166 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Modular Stereomicroscope | Leica | CUSTOM | Brain Slice Recording Workspace; custom specifications and modifications |
| N2 Supplement | Life Technologies | 17502-048 | CDI, and BrainXell Commercial Supplier Protocol |
| NaCl | Sigma Aldrich | S3014-1KG | Solution Preparation Workspace |
| NaH2PO4 | Sigma Aldrich | S0751-100G | Solution Preparation Workspace |
| NaHCO3 | Sigma Aldrich | S5761-500G | Solution Preparation Workspace |
| Neurobasal Medium | Life Technologies | 21103-049 | BrainXell Commercial Supplier Protocol |
| Optical Cage System | Thorlabs | Assorted | Brain Slice Recording Workspace |
| Optical Table w/Breadboard | Thorlabs | SDA7590 | Brain Slice Recording Workspace |
| PDLO | Sigma Aldrich | P0671 | HD-MEA Coating, Brain Slice Recording Workspace |
| Penicillin-streptomycin, 100x | Thermo Fisher Scientific | 15140-122 | CDI Commerical Supplier Protocol |
| Pipette tips | TipONE | S1120-8810 | Brain Slice Recording Workspace |
| Pipettors | Assorted | Assorted | Assorted |
| Platinum Anchor | CUSTOM | CUSTOM | Brain Slice Recording Workspace |
| Polyethylene Tubing | Assorted | Assorted | Brain Slice Recording Workspace |
| Pump | MasterFlex | 78018-22 | Brain Slice Recording Workspace |
| Razor Blade | Apollo | 10179960 | Brain Preparation Workspace |
| Reference Electrode Cell Culture Cap | CUSTOM | CUSTOM | Human iPSC Recording Workspace; Custom designed 3D Printer Design, available upon request |
| Rubber Pipette Bulb | Duran Wheaton Kimble | 292000205 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Serological Pipettes, 1 mL, 2 mL, 5 mL, 10 mL, 25 mL | Assorted | Assorted | Assorted |
| Slice Recovery Chamber | CUSTOM | CUSTOM | Brain Slice Recovery Workspace; Custom designed 3D Printer Design, available upon request |
| Spatula | ISOLAB | 047.06.150 | Brain Preparation Workspace |
| Sucrose | Sigma Aldrich | 84100-1KG | Solution Preparation Workspace |
| Super Glue | UHU | 358221 | Brain Slice Preparation Workspace |
| Surgical Scissors | Peters Instruments | BC 344 | Brain Extraction Workspace |
| Tabletop Centrifuge | Assorted | Assorted | Assorted |
| TGF-β1 | Peprotech | 100-21C | BrainXell Commercial Supplier Protocol |
| Tissue Paper | generic | generic | Brain Extraction Workspace |
| Trypan Blue | STEMCELL Technologies | 07050 | CDI Commerical Supplier Protocol |
| Upright Microscope | Olympus | CUSTOM | Imaging Workspace; Custom specifications and modifications |
| Vacusip | Integra | 159010 | Brain Slice Recording Workspace |
| Vibratome | Leica | VT1200s | Brain Slice Preparation Workspace; Includes: Specimen plate, buffer tray, ice tray, specimen plate holding tool, vibratome blade adjusting tool |
| Vibratome Blade | Personna | N/A | Brain Slice Preparation Workspace |
| Water Bath | Lauda | L000595 | Brain Slice Recovery Workspace |
Referencias
- Hebb, D. O. . The Organization of Behavior; A Neuropsychological Theory. , (1949).
- Cossart, R., Garel, S. Step by step: cells with multiple functions in cortical circuit assembly. Nat Rev Neurosci. 23, 395-410 (2022).
- Carrillo-Reid, L., Yuste, R. Playing the piano with the cortex: role of neuronal ensembles and pattern completion in perception and behavior. Curr Opin Neurobiol. 64, 89-95 (2020).
- Buzsáki, G. Large-scale recording of neuronal ensembles. Nat Neurosci. 7, 446-451 (2004).
- Buzsáki, G. Neural Syntax: Cell assemblies, synapsembles, and readers. Neuron. 68 (3), 362-385 (2010).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Cho, S., Wood, A., Bowlby, M. Brain slices as models for neurodegenerative disease and screening platforms to identify novel therapeutics. Curr Neuropharmacol. 5 (1), 19-33 (2007).
- Bliss, T. V. P., Collingridge, G. L. A synaptic model of memory: long-term potentiation in the hippocampus. Nature. 361, 31-39 (1993).
- Anderson, P., Morris, R., Amaral, D., Bliss, T., O'Keefe, L. . The Hippocampus Book. , (2006).
- Lisman, J., et al. Viewpoints: how the hippocampus contributes to memory, navigation and cognition. Nat Neurosci. 20, 1434-1447 (2017).
- Mori, K., Nagao, H., Yoshihara, Y. The olfactory bulb: Coding and processing of odor molecule information. Science. 286 (5440), 711-715 (1999).
- Buck, L., Axel, R. A novel multigene family may encode odorant receptors: A molecular basis for odor recognition. Cell. 65 (1), 175-187 (1991).
- Bushdid, C., Magnasco, M. O., Vosshall, L. B., Keller, A. Humans can discriminate more than 1 trillion olfactory stimuli. Science. 343 (6177), 1370-1372 (2014).
- Kempermann, G. Why new neurons? Possible functions for adult hippocampal neurogenesis. J Neurosci. 23 (3), 635-638 (2003).
- Aimone, J. B., Wiles, J., Gage, F. H. Computational influence of adult neurogenesis on memory encoding. Neuron. 61 (2), 187-202 (2009).
- Nithianantharajah, J., Hannan, A. J. Enriched environments, experience-dependent plasticity and disorders of the nervous system. Nat Rev Neurosci. 7, 697-709 (2006).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Espuny-Camacho, I., et al. Pyramidal neurons derived from human pluripotent stem cells integrate efficiently into mouse brain circuits in vivo. Neuron. 77 (3), 440-456 (2013).
- Rajamohan, D., et al. Current status of drug screening and disease modelling in human pluripotent stem cells. Bioessays. 35 (3), 281-298 (2013).
- Heilker, R., Traub, S., Reinhardt, P., Schöler, H. R., Sterneckert, J. iPS cell derived neuronal cells for drug discovery. Trends Pharmacol Sci. 35 (10), 510-519 (2014).
- Zhao, S. R., Mondéjar-Parreño, G., Li, D., Shen, M., Wu, J. C. Technical applications of microelectrode array and patch clamp recordings on human induced pluripotent stem cell-derived cardiomyocytes. J Vis Exp. (186), e64265 (2022).
- Hamill, O. P., McBride, D. W. Induced membrane hypo/hyper-mechanosensitivity: A limitation of patch-clamp recording. Annu Rev Physiol. 59, 621-631 (1997).
- Manz, K. M., Siemann, J. K., McMahon, D. G., Grueter, B. A. Patch-clamp and multi-electrode array electrophysiological analysis in acute mouse brain slices. STAR Protoc. 2 (2), 100442 (2021).
- Lee, C. H., Park, Y. K., Lee, K. Recent strategies for neural dynamics observation at a larger scale and wider scope. Biosens Bioelectron. 240, 115638 (2023).
- Urai, A. E., Doiron, B., Leifer, A. M., Churchland, A. K. Large-scale neural recordings call for new insights to link brain and behavior. Nat Neurosci. 25 (1), 11-19 (2022).
- Hu, X., Khanzada, S., Klütsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834 (2022).
- Amin, H., Marinaro, F., Tonelli, D. D. P., Berdondini, L. Developmental excitatory-to-inhibitory GABA-polarity switch is disrupted in 22q11.2 deletion syndrome: A potential target for clinical therapeutics. Sci Rep. 7 (1), 15752 (2017).
- Amin, H., Nieus, T., Lonardoni, D., Maccione, A., Berdondini, L. High-resolution bioelectrical imaging of Aβ-induced network dysfunction on CMOS-MEAs for neurotoxicity and rescue studies. Sci Rep. 7 (1), 2460 (2017).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471 (2023).
- Amin, H., et al. Electrical responses and spontaneous activity of human iPS-derived neuronal networks characterized for 3-month culture with 4096-electrode arrays. Front Neurosci. 10, 121 (2016).
- Lonardoni, D., et al. Recurrently connected and localized neuronal communities initiate coordinated spontaneous activity in neuronal networks. PLoS Comput Biol. 13 (7), e1005672 (2017).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. 2022 44th Annual International Conference of the IEEE Engineering in Medicine & Biology Society (EMBC). , 3111-3114 (2022).
- Rossi, L., Emery, B. A., Khanzada, S., Hu, X., Amin, H. Pharmacologically and electrically-induced network-wide activation of olfactory bulb with large-scale biosensor. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Emery, B. A., et al. Recording network-based synaptic transmission and LTP in the hippocampal network on a large-scale biosensor. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays). Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Berdondini, L., et al. Active pixel sensor array for high spatio-temporal resolution electrophysiological recordings from single cell to large scale neuronal networks. Lab Chip. 9, 2644-2651 (2009).
- Siegle, J. H., Hale, G. J., Newman, J. P., Voigts, J. Neural ensemble communities: open-source approaches to hardware for large-scale electrophysiology. Curr Opin Neurobiol. 32, 53-59 (2015).
- Amin, H., Maccione, A., Zordan, S., Nieus, T., Berdondini, L. High-density MEAs reveal lognormal firing patterns in neuronal networks for short and long term recordings. 2015 7th International IEEE/EMBS Conference on Neural Engineering (NER). , 1000-1003 (2015).
- Altuntac, E., et al. Bottom-up neurogenic-inspired computational model. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Maccione, A., et al. A novel algorithm for precise identification of spikes in extracellularly recorded neuronal signals. J Neurosci Methods. 177 (1), 241-249 (2009).
- Welch, P. D. The use of fast Fourier transform for the estimation of power spectra: A method based on time averaging over short, modified periodograms. IEEE Transactions on Audio and Electroacoustics. 15 (2), 70-73 (1967).
- Eggermont, J. J., Munguia, R., Pienkowski, M., Shaw, G. Comparison of LFP-based and spike-based spectro-temporal receptive fields and cross-correlation in cat primary auditory cortex. PLoS One. 6 (5), e20046 (2011).
- Damos, P. Using multivariate cross correlations, Granger causality and graphical models to quantify spatiotemporal synchronization and causality between pest populations. BMC Ecol. 16, 33 (2016).
- Kaminski, M. J., Blinowska, K. J. A new method of the description of the information flow in the brain structures. Biol Cybern. 65, 203-210 (1991).
- Pastore, V. P., Massobrio, P., Godjoski, A., Martinoia, S. Identification of excitatory-inhibitory links and network topology in large-scale neuronal assemblies from multi-electrode recordings. PLoS Comput Biol. 14 (8), e1006381 (2018).
- Amin, H., Dipalo, M., De Angelis, F., Berdondini, L. Biofunctionalized 3D nanopillar arrays fostering cell guidance and promoting synapse stability and neuronal activity in networks. ACS Appl Mater Interfaces. 10 (17), 15207-15215 (2018).
- Woeppel, K., Yang, Q., Cui, X. T. Recent advances in neural electrode-tissue interfaces. Curr Opin Biomed Eng. 4, 21-31 (2017).
- Steinmetz, N. A., Koch, C., Harris, K. D., Carandini, M. Challenges and opportunities for large-scale electrophysiology with Neuropixels probes. Curr Opin Neurobiol. 50, 92-100 (2018).
- Siegle, J. H., Hale, G. J., Newman, J. P., Voigts, J. Neural ensemble communities: open-source approaches to hardware for large-scale electrophysiology. Curr Opin Neurobiol. 32, 53-59 (2015).
- Freeman, J. Open source tools for large-scale neuroscience. Curr Opin Neurobiol. 32, 156-163 (2015).
- Stevenson, I. H., Kording, K. P. How advances in neural recording affect data analysis. Nat Neurosci. 14 (2), 139-142 (2011).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados