Method Article
Enregistrement et analyse de la dynamique d’ensemble neuronal multimodal à grande échelle sur un réseau de microélectrodes haute densité intégré au CMOS
Dans cet article
Résumé
Ici, nous utilisons la HD-MEA pour approfondir la dynamique computationnelle des ensembles neuronaux à grande échelle, en particulier dans l’hippocampe, les circuits de bulbes olfactifs et les réseaux neuronaux humains. La capture de l’activité spatio-temporelle, combinée à des outils informatiques, fournit des informations sur la complexité des ensembles neuronaux. La méthode améliore la compréhension des fonctions cérébrales, identifiant potentiellement des biomarqueurs et des traitements pour les troubles neurologiques.
Résumé
Les réseaux neuronaux à grande échelle et leurs microcircuits distribués complexes sont essentiels pour générer la perception, la cognition et le comportement qui émergent des modèles d’activité neuronale spatio-temporelle. Ces modèles dynamiques émergeant de groupes fonctionnels d’ensembles neuronaux interconnectés facilitent les calculs précis pour le traitement et le codage d’informations neuronales multi-échelles, entraînant ainsi des fonctions cérébrales supérieures. Pour sonder les principes informatiques de la dynamique neuronale sous-jacents à cette complexité et étudier l’impact multi-échelle des processus biologiques sur la santé et la maladie, les enregistrements simultanés à grande échelle sont devenus essentiels. Ici, un réseau de microélectrodes à haute densité (HD-MEA) est utilisé pour étudier deux modalités de dynamique neuronale : les circuits de l’hippocampe et du bulbe olfactif à partir de coupes de cerveau de souris ex-vivo et les réseaux neuronaux de cultures cellulaires in vitro de cellules souches pluripotentes induites humaines (iPSC). La plateforme HD-MEA, avec 4096 microélectrodes, permet des enregistrements non invasifs, multisites et sans marquage des modèles de tir extracellulaires de milliers d’ensembles neuronaux simultanément à haute résolution spatio-temporelle. Cette approche permet de caractériser plusieurs caractéristiques électrophysiologiques à l’échelle du réseau, y compris les modèles d’activité de pic à une ou plusieurs unités et les oscillations de potentiel de champ local. Pour examiner ces données neuronales multidimensionnelles, nous avons développé plusieurs outils informatiques intégrant des algorithmes d’apprentissage automatique, la détection et la classification automatiques des événements, la théorie des graphes et d’autres analyses avancées. En complétant ces pipelines de calcul avec cette plateforme, nous fournissons une méthodologie pour étudier la dynamique à grande échelle, multi-échelle et multimodale des assemblages cellulaires aux réseaux. Cela peut potentiellement faire progresser notre compréhension des fonctions cérébrales complexes et des processus cognitifs dans la santé et la maladie. L’engagement en faveur de la science ouverte et des connaissances sur la dynamique neuronale computationnelle à grande échelle pourrait améliorer la modélisation inspirée du cerveau, l’informatique neuromorphique et les algorithmes d’apprentissage neuronal. En outre, la compréhension des mécanismes sous-jacents des calculs neuronaux altérés à grande échelle et de leur dynamique interconnectée des microcircuits pourrait conduire à l’identification de biomarqueurs spécifiques, ouvrant la voie à des outils de diagnostic plus précis et à des thérapies ciblées pour les troubles neurologiques.
Introduction
Les ensembles neuronaux, souvent appelés assemblages cellulaires, sont essentiels au codage neuronal, facilitant les calculs complexes pour le traitement des informations neuronales multi-échelles 1,2,3. Ces ensembles sous-tendent la formation de réseaux neuronaux expansifs et de leurs microcircuits nuancés4. Ces réseaux et leurs modèles oscillatoires entraînent des fonctions cérébrales avancées, y compris la perception et la cognition. Bien que des recherches approfondies aient exploré des types de neurones et des voies synaptiques spécifiques, une compréhension plus approfondie de la façon dont les neurones forment en collaboration des assemblages cellulaires et influencent le traitement spatio-temporel de l’information à travers les circuits et les réseaux reste insaisissable5.
Les coupes cérébrales aiguës ex-vivo sont des outils électrophysiologiques essentiels pour l’étude des circuits neuronaux intacts, offrant un cadre contrôlé pour sonder les modèles d’activité oscillatoire de la fonction neuronale, de la transmission synaptique et de la connectivité, avec des implications dans les tests pharmacologiques et la modélisation des maladies 6,7,8. Ce protocole d’étude met en évidence deux circuits cérébraux clés : l’hippocampe-cortical (HC) impliqué dans les processus d’apprentissage et de mémoire 9,10, et le bulbe olfactif (OB) responsable de la discrimination des odeurs 11,12,13. Dans ces deux régions, de nouveaux neurones fonctionnels sont continuellement générés par la neurogenèse adulte tout au long de la vie dans le cerveau des mammifères14. Les deux circuits présentent des modèles d’activité neuronale dynamiques multidimensionnelles et une plasticité inhérente qui participent au recâblage du réseau neuronal existant et facilitent les stratégies alternatives de traitement de l’information si nécessaire15,16.
Les modèles de coupe de cerveau aiguë ex-vivo sont indispensables pour approfondir la fonctionnalité cérébrale et comprendre les mécanismes pathologiques au niveau des microcircuits. Cependant, les cultures cellulaires in vitro dérivées des réseaux neuronaux de cellules souches pluripotentes induites humaines (iPSC) offrent une voie prometteuse de recherche translationnelle, reliant de manière transparente les résultats des expériences animales au traitement clinique humain potentiel17,18. Ces tests in vitro centrés sur l’humain constituent une plate-forme fiable pour évaluer la toxicité pharmacologique, permettre un criblage précis des médicaments et faire avancer la recherche sur des stratégies thérapeutiques cellulaires innovantes19,20. Reconnaissant le rôle central du modèle neuronal iPSC, nous avons consacré le troisième module de cette étude de protocole à étudier en profondeur les caractéristiques fonctionnelles de ses réseaux dérivés et à affiner les protocoles de culture cellulaire associés.
Ces modules neuronaux électrogènes ont été couramment étudiés à l’aide de techniques telles que le calcium (imagerie Ca2+), les enregistrements patch-clamp et les réseaux de microélectrodes à faible densité (LD-MEA). Bien que l’imagerie Ca2+ offre une cartographie de l’activité d’une seule cellule, il s’agit d’une méthode basée sur le marquage cellulaire entravée par sa faible résolution temporelle et les défis liés aux enregistrements à long terme. Les LD-MEA manquent de précision spatiale, tandis que le patch-clamp, étant une technique invasive et laborieuse, donne souvent un faible taux de réussite 21,22,23. Pour relever ces défis et sonder efficacement l’activité à l’échelle du réseau, les enregistrements neuronaux simultanés à grande échelle sont apparus comme une approche essentielle pour comprendre les principes informatiques de la dynamique neuronale sous-jacente à la complexité du cerveau et leurs implications sur la santé et la maladie24,25.
Dans ce protocole JoVE, nous démontrons une méthode d’enregistrement neuronal à grande échelle basée sur la MEA à haute densité (HD-MEA) pour capturer l’activité neuronale spatio-temporelle à travers diverses modalités cérébrales, y compris les circuits de bulbe hippocampique et olfactif à partir de coupes aiguës de cerveau de souris ex-vivo (figures 1A-C) et les réseaux neuronaux humains in-vitro dérivés d’iPSC (figures 1D-E), précédemment rapportés par notre groupe et d’autres collègues26,27,28,29,30,31,32,33,34,35. Le HD-MEA, construit sur la technologie CMOS (Complementary-Metal-Oxide-Semiconductor), dispose de circuits et d’amplification sur puce, permettant des enregistrements inférieurs à la milliseconde sur une matrice de 7 mm2 taille36. Cette approche non invasive capture des modèles de tir extracellulaires multisites et sans marquage à partir de milliers d’ensembles neuronaux simultanément à l’aide de 4096 microélectrodes à haute résolution spatio-temporelle, révélant la dynamique complexe des potentiels de champ locaux (LFP) et de l’activité de pic multi-unités (MUA)26,29.
Compte tenu de l’immensité des données générées par cette méthodologie, un cadre analytique sophistiqué est essentiel, mais pose des défis37. Nous avons développé des outils informatiques qui englobent la détection automatique des événements, la classification, la théorie des graphes, l’apprentissage automatique et d’autres techniques avancées (Figure 1F)26,29,38,39. En intégrant le HD-MEA à ces outils analytiques, une approche holistique est conçue pour sonder la dynamique complexe des assemblages cellulaires individuels aux réseaux neuronaux plus larges à travers diverses modalités neuronales. Cette approche combinée approfondit notre compréhension de la dynamique computationnelle dans les fonctions cérébrales normales et offre un aperçu des anomalies présentes dans les conditions pathologiques28. De plus, les connaissances issues de cette approche peuvent propulser des progrès dans la modélisation inspirée du cerveau, l’informatique neuromorphique et les algorithmes d’apprentissage neuronal. En fin de compte, cette méthode est prometteuse pour découvrir les mécanismes fondamentaux derrière les perturbations des réseaux neuronaux, identifier potentiellement des biomarqueurs et guider la création d’outils de diagnostic précis et de traitements ciblés pour les maladies neurologiques.
Protocole
Toutes les expériences ont été réalisées conformément aux réglementations européennes et nationales applicables (Tierschutzgesetz) et ont été approuvées par l’autorité locale (Landesdirektion Sachsen ; 25-5131/476/14).
1. Coupes cérébrales ex-vivo de circuits hippocampiques-corticaux et olfactifs sur HD-MEA
- Préparation de solutions expérimentales de découpe et d’enregistrement (Figure 2A)
- Le jour de l’expérience, préparer 0,5 L de solution de coupe à haute teneur en saccharose et 1 L de solution d’enregistrement du liquide céphalorachidien artificiel (LCRa) (tableau 1A,B).
- Ajouter tous les produits chimiques solides dans une fiole jaugée sèche, puis remplir une partie de l’eau distillée doublement (dd).
- Ajouter le MgCl2 et le CaCl2 à partir de solutions mères à 1 M, puis remplir le reste d’eau dd. Commencer à remuer constamment avec un agitateur magnétique jusqu’à ce que les solides visibles soient dissous ~5 min.
- Utilisez un osmomètre de point de congélation pour valider l’osmolarité entre 350-360 mOsm pour la solution de coupe à haute teneur en saccharose et 315-325 mOsm pour la solution d’enregistrement aCSF.
- Utilisez un pH-mètre pour valider le pH entre 7,3-7,4 pour la solution de bouturage à haute teneur en saccharose et 7,25-7,35 pour la solution d’enregistrement du LCR-a. Commencez à bouillonner continuellement avec 95 % d’O2 et 5 % de CO2.
- Placez la solution de coupe à haute teneur en saccharose sur de la glace pendant au moins 30 minutes avant de trancher et commencez à bouillonner continuellement avec 95 % d’O2 et 5 % de CO2.
- Après 10 min de carbogénation, remplissez un bécher de 50 ml avec 30 ml de solution de coupe et conservez-le au congélateur (-20 °C) pendant 20 à 30 minutes ou jusqu’à ce qu’il soit partiellement congelé.
REMARQUE : Toutes les solutions doivent être préparées fraîches pour chaque expérience. L’eau dd utilisée ici est de l’eau ultrapure autoclavée stockée à température ambiante (RT). La quantité de solution préparée doit être adaptée à la question d’étude spécifique.
- Le jour de l’expérience, préparer 0,5 L de solution de coupe à haute teneur en saccharose et 1 L de solution d’enregistrement du liquide céphalorachidien artificiel (LCRa) (tableau 1A,B).
- Préparation des espaces de travail de la tranche de cerveau (Figure 2A)
- Amenez l’animal dans la salle d’expérimentation.
REMARQUE : Dans ce protocole, des souris femelles C57BL/J6 âgées de 8 à 16 semaines ont été utilisées comme décrit précédemment 26,29,32. L’animal doit s’acclimater pendant au moins 30 minutes après le transport. Les transferts longue distance (c.-à-d. entre établissements) doivent être évités le jour même de l’expérience. L’âge, le sexe et la souche de l’animal doivent être déterminés en fonction de la question spécifique de l’étude. - Pendant que l’animal s’acclimate et que la solution à haute teneur en saccharose refroidit, placez les outils requis dans chaque espace de travail désigné (voir la table des matériaux).
- Préparez l’espace de travail de récupération et de maintenance de la tranche de cerveau. Remplissez la chambre de récupération de la tranche avec une solution d’enregistrement du CSF aC carbogéné et placez la chambre dans le bain-marie réglé à 32 °C. Maintenir une carbogénisation continue tout au long de l’expérience.
- Préparer l’espace de travail de préparation de la tranche de cerveau. Configurer le vibratome - placez la lame dans le porte-lame du vibratome et calibrez le vibratome aux réglages corrects (vitesse de déplacement de la lame : 0,20 mm/s, amplitude de hauteur : 95 μm, angle de la lame : 45°). Remplissez le bac à glaçons vibratome avec de la glace et le bac tampon avec une solution de coupe à haute teneur en saccharose et commencez à carbogéner la solution dans le bac tampon.
- Préparez l’espace de travail de préparation du cerveau. Remplissez la boîte de Pétri en verre de 150 mm de glace et placez une boîte de culture en plastique de 90 mm avec du papier filtre à l’intérieur. Remplissez le plat de culture en plastique avec une solution de coupe à haute teneur en saccharose et commencez à carbogéner. Ajoutez une goutte de super colle sur la plaque d’échantillon réfrigérée et fixez le moule à agarose.
REMARQUE : Le moule à agarose est préparé au moins la veille avec 3% d’agarose dans de l’eau dans un moule à cerveau de souris personnalisé. - Enfin, préparez l’espace de travail d’extraction du cerveau. Couvrir le papier d’aluminium de papier de soie, récupérer un bécher de 50 ml contenant de la gadoue de solution de coupe à haute teneur en saccharose et ajouter de l’isoflurane dans la chambre d’anesthésie.
REMARQUE : L’anesthésie sera ajoutée à la chambre d’anesthésie ~1 min avant le placement de l’animal. Un bécher de 50 mL avec 30 mL de boudoue de solution de coupe à haute teneur en saccharose sera retiré du congélateur à -20 °C ~2 min avant la décapitation.
- Amenez l’animal dans la salle d’expérimentation.
- Extraction et découpage du cerveau de souris
REMARQUE : Toute cette procédure doit être effectuée le plus rapidement possible pour éviter un manque d’oxygénation du cerveau. L’ablation du cerveau ne devrait prendre que 1 à 2 minutes entre la décapitation et l’immersion dans la gadoue de solution de coupe à haute teneur en saccharose.- Anesthésier l’animal avec la dose appropriée d’isoflurane (chambre d’anesthésie de 0,5 mL/1 L). Déterminer la profondeur de l’anesthésie par un pincement de patte ; confirmer l’absence de réflexe de retrait de la patte avant de continuer.
- Transférez l’animal sur le papier de soie dans l’espace de travail d’extraction du cerveau et décapitez-le avec des ciseaux chirurgicaux.
- Insérez les ciseaux à iris dans le tronc cérébral et gardez les ciseaux inférieurs au ras du calvaria. Coupez le long de la suture sagittale jusqu’à ce que la suture coronale soit atteinte. Placez des ciseaux à iris dans les orbites et coupez à travers la suture métopique. Utilisez une pince incurvée pour déplacer les côtés de la calvaria vers le bas, exposant tout le cerveau.
REMARQUE : Faites attention avec les ciseaux à iris et les pinces pour ne pas perforer le cerveau lorsque vous coupez les sutures. - Faites glisser le cerveau avec le bord émoussé de la pince incurvée dans le bécher de 50 mL avec 30 mL de gadoue de solution de coupe à haute teneur en saccharose. Laissez reposer 1 min.
- Transférer le cerveau dans la boîte de culture en plastique de 90 mm avec une solution de coupe de carbbogène réfrigérée dans l’espace de travail de préparation du cerveau. Orientez le cerveau pour le positionner dans le moule d’agarose.
- Ajoutez un petit point de super colle à l’extrémité rostrale du moule à agarose. Placez le cerveau dans le moule avec la spatule. Assurez-vous que le cerveau est placé avec le côté dorsal vers le bas pour un tranchage horizontal.
REMARQUE : L’emplacement de la colle dans le moule changera en fonction de la région d’intérêt (ROI). Pour les coupes hippocampiques-corticales (HC) et de bulbe olfactif (OB), assurez-vous que le OB est stabilisé et que les côtés du cerveau restent exempts de colle. Trop de colle affectera la qualité du tranchage et provoquera des déchirures lors du tranchage du vibratome. - Déplacez la plaque d’échantillon dans le plateau tampon, déplacez la lame en position avec le bon angle et augmentez la hauteur du plateau tampon pour amener la lame aussi près que possible du cerveau.
- Trancher à une vitesse de 0,20 mm/s des intervalles de 300 μm de tissus HC et OB, puis les prélever après chaque tour de tranchage avec une pipette Pasteur en verre.
- Laisser les tranches dans la chambre de récupération remplie de LCR dans un bain-marie à 32 °C pendant 45 min, puis 1 h à RT.
REMARQUE : Assurez-vous de maintenir une carbogénisation continue de toutes les solutions et de toutes les chambres mentionnées contenant la solution. Un régulateur de pression peut être utilisé pour maintenir une carbogénisation constante.
2. Réseau neuronal humain in vitro basé sur des iPSC sur HD-MEA
REMARQUE : Tous les neurones iPSC utilisés dans cette étude sont obtenus commercialement (voir le tableau des matériaux). Ces cellules humaines se différenciaient des lignées cellulaires iPS stables dérivées du sang périphérique humain ou des fibroblastes.
- Revêtement de puces HD-MEA pour les cultures de cellules iPSC humaines in vitro (Figure 2B)
- Placez la puce HD-MEA sur la plate-forme d’acquisition d’enregistrement, remplissez le réservoir de PBS et testez la puce avant le revêtement. Démarrez le logiciel Brainwave. Sélectionnez Fichier > Nouvelle session d’enregistrement. Réglez les paramètres d’enregistrement pour avoir une fréquence d’enregistrement de 50 Hz et une fréquence d’échantillonnage de 18 kHz/électrode. Modifiez le décalage de l’amplificateur pour calibrer la puce. Reportez-vous au Tableau 2 pour obtenir des conseils de dépannage.
REMARQUE : Les paramètres de fréquence d’enregistrement et de fréquence d’échantillonnage dépendent du type de données et des exigences de chaque système. - Stériliser et pré-conditionner les MD-MEA.
- Sous le capot, essuyez la puce et l’anneau de verre avec un mouchoir imbibé d’éthanol à 96 % (EtOH), puis placez chaque dispositif dans une boîte de Pétri stérile de 100 mm x 20 mm et remplissez le réservoir MEA avec de l’EtOH à 70 % pendant 20 min.
- Aspirez l’EtOH et lavez le réservoir avec de l’eau stérile filtrée 3 fois. Ajouter 1 mL de milieu de préconditionnement et incuber toute la nuit à 37 °C et 5 % de CO2.
REMARQUE : Le média de préconditionnement doit être une solution à base de sel pour rendre la surface HD-MEA plus hydrophile. Il peut s’agir de milieux complets BrainPhys (BP) préalablement préparés (âgés de moins de >3 mois) (tableaux 1C).
- Enrobez les HD-MEA. Le lendemain, aspirer le milieu de préconditionnement. Ajouter 1 mL de poly-dl-ornithine (PDLO) à 0,1 mg/mL pour enrober toute la zone active. Incuber à 37 °C pendant la nuit dans un incubateur.
- Préparez et réchauffez le support sur RT. Les protocoles ici exploitent les neurones iPSC humains fonctionnels à partir de deux sources commerciales ; Ainsi, les composants multimédias varient pour chaque fournisseur. Un protocole est décrit dans les tableaux 1C et D.
- Aspirer le PDLO, laver 3 fois à l’eau DD et laisser sécher les copeaux sous le capot pendant 10 min.
- Remplissez une boîte de Pétri de 35 mm x 10 mm avec de l’eau stérile filtrée et placez-la à côté de la puce pour maintenir une humidité appropriée et éviter l’évaporation des cellules ensemencées dans les étapes suivantes.
- Placez la puce HD-MEA sur la plate-forme d’acquisition d’enregistrement, remplissez le réservoir de PBS et testez la puce avant le revêtement. Démarrez le logiciel Brainwave. Sélectionnez Fichier > Nouvelle session d’enregistrement. Réglez les paramètres d’enregistrement pour avoir une fréquence d’enregistrement de 50 Hz et une fréquence d’échantillonnage de 18 kHz/électrode. Modifiez le décalage de l’amplificateur pour calibrer la puce. Reportez-vous au Tableau 2 pour obtenir des conseils de dépannage.
- Placage et maintenance des neurones iPSC humains dans les MDE-MEA (Figure 2B)
- Décongeler et diluer les cellules jusqu’à la concentration désirée par microlitre (c.-à-d. 1000 cellules/μL pour obtenir une densité cellulaire de 50 000 dans une goutte de 50 μL sur l’AMH-HD) (tableau 1C).
- Pipeter la suspension cellulaire à la surface de la zone active de la puce à l’aide d’un milieu à points à haute teneur en laminine (tableau 1D).
- Incuber à 37 °C avec 5 % de CO2 pendant 45 à 60 min.
- Remplir délicatement 2 mL de milieu dans le réservoir de MD-MEA (tableau 1C).
- Effectuer un changement de milieu de 100 % le jour 1 (DIV1) après l’ensemencement à l’aide d’un milieu RT (Tableau 1C). Changez 50% des supports tous les 3-4 jours. Maintenir les MD-MEA incubés à 37 °C avec 5 % de CO2 pendant toute la durée de l’expérience.
REMARQUE : Pipeter doucement pour éviter de déloger les cellules. Vérifiez la couleur du support pour toute contamination. L’intervalle et la quantité de changement de milieu peuvent être déterminés par des questions d’étude individuelles ou des besoins/spécifications des cellules. - Facultatif : Vérifiez la progression de la croissance de la culture cellulaire entre DIV4 et DIV8 sous un microscope à contraste interférentiel différentiel (DIC) droit après avoir nettoyé la platine avec >70 % d’EtOH.
3. Enregistrements neuronaux ex-vivo et in vitro à grande échelle avec des AME-HD
- Préparation de l’espace de travail d’enregistrement des tranches de cerveau (Figure 2A)
- Pendant que les tranches de cerveau se rétablissent, placez les outils requis dans chaque espace de travail désigné (voir la table des matériaux).
REMARQUE : La configuration principale du système doit être optimisée et testée bien avant le jour de l’expérimentation de la tranche de cerveau. Le système de perfusion (conduites d’entrée, conduites de sortie de pompe, tubulure et mise à la terre) doit être testé avec du PBS ou de l’aCSF et un HD-MEA sur la plate-forme d’enregistrement pour garantir un signal propre, un rapport signal-bruit accru et l’absence de bruit de perfusion. - Enduire la puce HD-MEA de 0,1 mg/mL de PDLO pour améliorer le couplage tissu-puce et incuber à 37 °C pendant 20 min.
- Pendant l’incubation de la puce, remplissez le système de perfusion gravitaire et les lignes avec un LCR enregistreur. Assurer une carbogénisation continue du système de perfusion. Réglez un débit de 4,5 mL/min et une température de 37 °C.
- Placez la puce HD-MEA sur la plate-forme d’enregistrement d’acquisition, remplissez le réservoir d’aCSF, testez le système de perfusion et dépannez tout bruit de système restant.
- Démarrez le logiciel Brainwave. Sélectionnez Fichier > Nouvelle session d’enregistrement. Réglez les paramètres d’enregistrement pour qu’ils aient une fréquence d’enregistrement de 1 Hz et une fréquence d’échantillonnage de 14 kHz/électrode. Modifiez le décalage de l’amplificateur pour calibrer la puce. Reportez-vous au Tableau 2 pour obtenir des conseils de dépannage.
REMARQUE : Les paramètres de fréquence d’enregistrement et de fréquence d’échantillonnage dépendent du type de données et des exigences de chaque système.
- Démarrez le logiciel Brainwave. Sélectionnez Fichier > Nouvelle session d’enregistrement. Réglez les paramètres d’enregistrement pour qu’ils aient une fréquence d’enregistrement de 1 Hz et une fréquence d’échantillonnage de 14 kHz/électrode. Modifiez le décalage de l’amplificateur pour calibrer la puce. Reportez-vous au Tableau 2 pour obtenir des conseils de dépannage.
- Assurez-vous que la zone d’enregistrement est sombre grâce à un système d’éclairage de la pièce ou à une cage ombragée sur la table optique.
- Alignez le stéréomicroscope avec le réservoir de puce HD-MEA et la zone active pour l’acquisition d’images.
- Placez l’ancrage dans le réservoir à copeaux pour équilibrer.
REMARQUE : L’ancre est une harpe en platine sur mesure avec un minimum de fils pour favoriser l’oxygénation ; cependant, certains commerciaux sont disponibles. - Ajouter des composés pharmacologiques dans les tubes de perfusion appropriés.
REMARQUE : Dans ce protocole, des enregistrements spontanés et des enregistrements pharmacologiques de 100 μM de 4-aminopyridine (4-AP) ont été obtenus comme décrit précédemment. Les composés pharmacologiques peuvent être adaptés à la question spécifique de l’étude. - Dans l’espace de préparation des tranches de cerveau, placez une nouvelle boîte de culture en plastique de 90 mm dans une boîte de Pétri en verre de 150 mm. Ajouter un LCR et commencer le carbogénage.
- Pendant que les tranches de cerveau se rétablissent, placez les outils requis dans chaque espace de travail désigné (voir la table des matériaux).
- Enregistrements à l’échelle du circuit à partir de tranches HC et OB à l’aide de HD-MEA
REMARQUE : Le couplage de la tranche doit être effectué le plus rapidement possible pour éviter un manque d’oxygénation de la tranche. Le couplage ne devrait prendre que ~1 min entre la mise en place initiale de la tranche microdisséquée sur la zone active de la puce et le démarrage final du système de perfusion.- Retirer la tranche de la chambre de récupération de la tranche de cerveau à l’aide d’une pipette en verre et la placer dans une boîte de culture en plastique de 90 mm à carbogénation continue. À l’aide d’un outil de microdissection, isolez le HC ou le OB du tissu de coupe cérébrale environnant.
- Déplacez les tranches aiguës HC ou OB isolées avec une pipette en verre dans le réservoir HD-MEA. Alignez délicatement la tranche sur la zone active MEA avec un pinceau fin. Aspirez toutes les solutions du puits de puce HD-MEA avec un système d’aspiration.
- Placez délicatement l’ancre sur le dessus de la tranche à l’aide d’une pince.
REMARQUE : L’ancrage doit être placé sans mouvement de tranche pour éviter la perte d’attelage. - Ajoutez doucement la solution dans le réservoir à copeaux et démarrez le système de perfusion.
REMARQUE : Assurez-vous d’un flux laminaire à partir de l’entrée de perfusion et de la sortie de la pompe pour des paramètres d’enregistrement optimaux. - Assurez-vous que la zone d’enregistrement est correctement atténuée par le système d’éclairage de la pièce ou avec une cage ombragée sur une table optique.
- Laissez la tranche s’acclimater pendant 10 minutes avant de commencer les enregistrements ou la modulation pharmacologique supplémentaire.
- Démarrez le logiciel Brainwave. Sélectionnez Fichier > Nouvelle session d’enregistrement. Réglez les paramètres d’enregistrement pour qu’ils aient une fréquence d’enregistrement de 1 Hz et une fréquence d’échantillonnage de 14 kHz/électrode. Modifiez le décalage de l’amplificateur pour calibrer la puce.
NOTA : Comme indiqué précédemment dans la section 3.1.4.1, lors des tests du système, assurez-vous d’appliquer ces mêmes paramètres d’enregistrement. - Appuyez sur Enregistrer pour commencer l’acquisition avec les conditions expérimentales prédéfinies.
- Immédiatement après l’enregistrement final, capturez une imagerie lumineuse de la tranche cérébrale aiguë. Ramenez la tranche dans la chambre de récupération de la tranche, retirez toute matière organique couplée à la copeau avec une brosse et continuez avec la tranche suivante. Nettoyez les MDE-AEM comme décrit dans la section 3.4.
- Préparation de l’espace de travail d’enregistrement iPSC humain et des enregistrements à l’échelle du réseau sur HD-MEA (Figure 2B)
REMARQUE : Changez de support la veille de l’enregistrement ou immédiatement après l’enregistrement de l’iPSC humain (Tableau 1C). Dans les études utilisant les neurones fonctionnels, les médias ont été changés tous les 4 jours, et sur 4, 8, 16 et 24 DIV, les médias sont changés immédiatement après les enregistrements iPSC.- Assurez un environnement de travail stérile en nettoyant la plate-forme d’acquisition HD-MEA avec >70 % d’EtOH.
- Placez délicatement un capuchon à base de polydiméthylsiloxane (PDMS) avec référence sur l’anneau HD-MEA sous le capot. Déplacez la puce HD-MEA vers l’espace d’enregistrement iPSC et connectez-la à la plate-forme d’acquisition.
- Assurez-vous que la zone d’enregistrement est suffisamment atténuée par un système d’éclairage de pièce ou une cage ombragée sur une table optique.
- Laissez la puce HD-MEA s’équilibrer pendant 10 minutes avant de commencer les enregistrements ou la modulation pharmacologique supplémentaire.
- Démarrez le logiciel Brainwave. Sélectionnez Fichier > Nouvelle session d’enregistrement. Réglez les paramètres d’enregistrement pour avoir une fréquence d’enregistrement de 50 Hz et une fréquence d’échantillonnage de 18 kHz/électrode. Modifiez le décalage de l’amplificateur pour calibrer la puce.
REMARQUE : Comme indiqué précédemment dans la section 2.1.1, lors d’un test du système avant le revêtement et le placage, assurez-vous d’appliquer ces mêmes paramètres d’enregistrement. - Enregistrer l’activité de décharge spontanée ou les réponses pharmacologiquement induites par le réseau de CSPi humaines chaque jour du plan expérimental (c.-à-d. 4, 8, 16, 24 DIV).
REMARQUE : Ne laissez pas la puce à l’extérieur de l’incubateur pendant >30 min pour maintenir une température et une humidité stables et éviter tout choc thermique aux cellules. - Incuber des MDE-MEA à 37 °C avec 5 % de CO2 au cours de l’expérience.
- Une fois l’expérience terminée, fixer le réseau neuronal sur les puces et colorer pour une imagerie optique plus poussée ou nettoyer directement les MDE-MEA, comme décrit à l’étape 3.4.
- Nettoyage des puces HD-MEA
- Après l’expérience, jetez la solution selon une élimination appropriée des déchets et rincez à l’eau dd.
- Ajoutez le détergent de votre choix, nettoyez la zone active et tout le réservoir avec un coton-tige et jetez le détergent. Remplissez de détergent, laissez incuber pendant 20 min, puis jetez le détergent.
- Rincez abondamment à l’eau de laboratoire. Ensuite, rincez 3-4 fois avec de l’eau dd.
- Utilisez la pression d’air pour sécher complètement la puce HD-MEA.
4. Analyse d’enregistrements neuronaux à grande échelle provenant d’AME-HD
REMARQUE : Bien que l’étape 4.1 soit spécifique au logiciel Brainwave, l’étape 4.2 peut être modifiée en fonction du type d’appareil HD-MEA disponible dans le commerce de chaque utilisateur.
- Prétraitement des données brutes et détection d’événements
- Ouvrez un fichier de données brutes enregistré (.brw) dans le logiciel Brainwave. Sélectionnez Analyse > détection LFP ou détection de pointe.
REMARQUE : La détection LFP utilise un filtrage IIR avec un filtre Butterworth passe-bas de 4eordre ( 1-100 Hz). Les algorithmes de seuil dur comprennent un seuil élevé de 150 μV, un seuil bas de -150 μV, une fenêtre d’énergie comprise entre 70 et 120 ms, une période réfractaire de 10 ms et une durée maximale de l’événement de 1 s. La détection des pointes simples et MUA utilise un filtrage IIR avec un filtre Butterworth passe-haut de 4eordre ( 300-3500 Hz). Un algorithme de TSPT est appliqué avec un facteur d’écart-type de 8, une période de vie maximale de 2 ms et une période réfractaire de 1 ms. - Pour les enregistrements de circuits HC et OB, ajoutez l’option Espace de travail avancé dans le fichier d’événements détectés (.bxr) pour importer l’image de lumière structurelle capturée à partir du stéréomicroscope. Lors de l’examen des circuits HC à grande échelle, créez des couches structurelles contenant le gyrus denté (DG), le hilus, le Cornu Ammonis 1 (CA1), le Cornu Ammonis 3 (CA3), le cortex entorhinal (EC) et le cortex périrhinal (PC). Lors de l’examen des circuits OB à grande échelle, créez des couches structurelles contenant la couche nerveuse olfactive (ONL), la couche glomérulaire (GL), la couche plexiforme externe (EPL), la couche de cellules mitrales (MCL) et la couche de cellules granulaires (GCL). Considérons l’EPL et le MCL comme la couche de projection (PL), y compris le cortex olfactif (OCx).
- Ouvrez un fichier de données brutes enregistré (.brw) dans le logiciel Brainwave. Sélectionnez Analyse > détection LFP ou détection de pointe.
- Traitement des données avec un pipeline de calcul Python personnalisé
- Débruitage
- Lisez le fichier .bxr à l’aide d’un script Python 26,29,32 et d’un package python h5py 3.6.0 écrit sur mesure.
- Extraire les trains de pointes relatifs aux enregistrements du réseau iPSC et les trains d’événements LFP relatifs aux enregistrements des circuits de tranches de cerveau HC et OB.
- Caractériser les événements dont le nombre total d’électrodes actives est inférieur à 0,1 % ou 10 % de la moyenne des électrodes actives par événement moyen ou les événements détectés se situant en dehors d’une plage de cadence de tir statistiquement raisonnable en tant qu’événements aléatoires et les éliminer. En outre, appliquez des valeurs de seuil d’amplitude et de durée d’événement.
REMARQUE : Pour la plage de cadence de tir, 0,1-15 pointes/s et 0,1-60 événements LFP/min sont pris en compte. Il s’agit d’exemples de valeurs de seuil de débit utilisées pour les jeux de données analysés. Les seuils de débit, d’amplitude et de durée dépendent des données individuelles. - Enregistrez les données d’entraînement d’événements résultantes avec les informations spatio-temporelles qui les accompagnent au format de fichier .npy.
- Rastergrammes
- Lisez les fichiers d’événements .npy et .bxr filtrés et générez un tracé raster à l’aide de la fonction pyplot de Matplotlib (https://matplotlib.org/3.5.3/api/_as_gen/matplotlib.pyplot.html).
- De plus, pour les enregistrements de tranches de cerveau avec une spécificité de couche, triez et regroupez les ID d’électrodes en fonction des couches produites à l’étape 4.1.2.
- Activité de tir moyenne
- Traiter les données de séries chronologiques du fichier .bxr, en calculant la cadence de décharge moyenne de chaque électrode (nombre d’événements/durée d’enregistrement).
- Construisez une matrice de données où les lignes et les colonnes représentent les coordonnées des électrodes dans le réseau HD-MEA 64 x 64, où chaque valeur de matrice signifie la vitesse de décharge moyenne.
- Utilisez une bibliothèque de traçage telle que les fonctions imshow de Matplotlib ou de carte thermique de Seaborn en Python.
- Utilisez la carte de couleurs « chaudes » ici, créant une carte thermique informative qui encapsule visuellement la distribution spatiale des taux de décharge moyens sur le réseau d’électrodes.
- Traces de forme d’onde représentatives
- Lisez les données de séries chronologiques à partir du fichier .brw et générez une trace de forme d’onde à l’aide de la fonction pyplot de Matplotlib. (https://matplotlib.org/3.5.3/api/_as_gen/matplotlib.pyplot.html).
- Entrez l’ID d’électrode, le intervalle de temps et la bande de fréquence souhaités pour une trace de forme d’onde représentative. Les bandes de fréquences définies dans ces analyses comprennent les oscillations LFP basse fréquence (1-100 Hz) avec des bandes de fréquences filtrées passe-bande δ, θ, β et γ ; ondulations d’ondes aiguës (TOS) (140-220 Hz) ; et simple haute fréquence et MUA (300-3500 Hz). Les bandes de fréquences δ, θ, β et γ sont respectivement de 1 à 4 Hz, de 5 à 12 Hz, de 13 à 35 Hz et de 35 à 100 Hz.
- Densité spectrale de puissance
- Lisez les données de séries chronologiques du fichier .brw et calculez les périodogrammes pour discerner les fréquences dominantes sous-jacentes à l’activité oscillatoire dans chaque série chronologique.
- Construire des spectrogrammes pseudo-couleur de la dynamique fréquence-temps.
NOTE : Les spectres sont calculés à l’aide de la méthode de Welch en utilisant la transformée de Fourier rapide des LFP enregistrés pour estimer la densité de puissance spectrale41. - Entrez l’ID d’électrode souhaité, le intervalle de temps et la bande de fréquence pour une carte de densité spectrale. Les bandes de fréquences définies dans ces analyses comprennent celles décrites à l’étape 4.2.4.
- Connectivité fonctionnelle
- Pour les enregistrements de circuits de tranches de cerveau, suivez les étapes 4.2.6.2 à 4.2.6.4.
- Lisez les données de séries chronologiques du fichier .brw et calculez la covariance croisée entre les paires d’électrodes actives dans le réseau 64 x 64 en utilisant le coefficient de corrélation de Pearson (PCC)42.
- Ajuster un modèle vectoriel autorégressif à la série chronologique en utilisant la causalité de Granger multivariée pour quantifier l’influence d’une série chronologique sur une autre.
- Appliquer la fonction de transfert dirigé (DTF) pour évaluer le flux d’informations directionnelles au sein des liens corrélés.
REMARQUE : La connectivité fonctionnelle dans le réseau multicouche est établie en fixant un seuil de valeur de corrélation basé sur celles au-dessus de la moyenne et deux écarts-types de toutes les valeurs de covariance croisée43,44. - Pour l’enregistrement iPSC, suivez les étapes 4.2.6.6 à 4.2.6.8.
- Lire les données des spiketrains à partir du fichier .bxr et calculer une matrice 64x64 des coefficients de corrélation PCC entre toutes les combinaisons de trains de spikes binned à l’aide de fonctions spike_train_correlation (https://elephant.readthedocs.io/en/v0.7.0/reference/spike_train_correlation.html).
REMARQUE : La connectivité fonctionnelle dans le réseau multicouche est établie en définissant un seuil de valeur de corrélation basé sur celles supérieures à la moyenne et deux écarts-types de toutes les valeurs de covariance croisée. - En outre, mettre en œuvre des procédures de filtrage des filtres spatio-temporels (STF) et des seuils de latence en fonction de la distance (DdLT) sur la matrice de connectivité afin d’éliminer les connexions appariées potentielles dépassant la vitesse de propagation maximale (fixée à 400 mm/s)45.
- Extrayez les pics négatifs des matrices de corrélation croisée résultantes avec des opérations de filtrage et de seuillage pour identifier les connexions inhibitrices à l’aide de l’algorithme d’histogramme de corrélation croisée filtré et normalisé (FNCCH)45.
- Transformez chaque matrice de connectivité en un fichier de graphe dynamique (.gexf).
- Cartes de connectivité réseau
- Laboratoire de données ouvertes dans le programme Gephi version 9.2 (https://gephi.org) pour le graphe dynamique afin de tracer des intervalles de temps spécifiques.
- Appliquez la disposition géographique dans la fenêtre de disposition pour le mappage spatial.
- Placez des contraintes de paramètre sur la plage de degrés et le poids des arêtes à des fins de comparaison.
- Attribuez une couleur de nœud, une taille d’arête et une taille de degré pour une meilleure visualisation.
- Débruitage
Résultats
Cartographie spatio-temporelle multimodèle et extraction de caractéristiques de tir oscillatoire
Pour quantifier les événements LFP et spike à l’échelle du réseau qui ont émergé d’ensembles neuronaux dynamiques, nous avons étudié les modèles de décharge synchrones à grande échelle dans les circuits HC et OB et les réseaux iPSC humains. Les circuits de coupe de cerveau enregistrés à l’étape 3.2 et les réseaux iPSC enregistrés à l’étape 3.3 ont été analysés conformément aux étapes 4.1 à 4.2 du protocole. Tout d’abord, la détection et le débruitage des événements ont été effectués pour tous les ensembles de données enregistrés et résolus régionalement selon les spécifications du circuit. Ensuite, une cartographie spatiale topographique pseudo-couleur des modèles moyens de tir LFP et de pointe à grande échelle, des rastergrammes d’événements détectés et des traces représentatives 5-s de formes d’onde filtrées ont été tracées (figures 3 A-I). La cartographie topographique pseudo-couleur des modèles de LFP et de taux de décharge de pointe à grande échelle a été superposée aux images optiques capturées au microscope respectives de HC (Figure 3A), OB (Figure 3B) et réseau neuronal iPSC humain (Figure 3C). Cela permet d’étudier les modèles et les réponses oscillatoires individuels basés sur les circuits et les réseaux. Les rastergrammes HC et OB contiennent le nombre d’événements LFP détectés triés sur les couches DG, Hilus, CA3, CA1, EC et PC du circuit HC et les couches ONL, OCx, GL, PL et GCL du réseau OB sur un intervalle de temps de 60 s (figures 3D, E). Le rastergramme iPSC humain affiche les événements de pic détectés synchrones du réseau culturel interconnecté sur une plage de temps de 20 s (Figure 3G). Ensuite, les traces d’événements représentatives 5s provenant de sites d’enregistrement HD-MEA à grande échelle montrent une gamme de fréquences oscillatoires enregistrées dans les circuits HC (c’est-à-dire l’électrode sélectionnée dans CA3) (Figure 3G) et OB (c’est-à-dire l’électrode sélectionnée dans GL) (Figure 3H) et une activité d’éclatement de pics multi-unités dans le réseau d’iPSC humain à partir de quatre électrodes actives sélectionnées dans le réseau (Figure 3I). Ces signaux exemplaires montrent des signatures de biosignaux, y compris des oscillations LFP basse fréquence (1-100 Hz) avec des bandes de fréquences filtrées δ, θ, β et γ filtrées ; ondulations d’ondes aiguës (TOS) (140-220 Hz) ; et simple haute fréquence et MUA (300-3500 Hz). Enfin, l’analyse de la densité spectrale de puissance (DSP) a été utilisée pour quantifier simultanément l’amplitude de puissance d’une bande oscillatoire spécifique dans le circuit HC et OB interconnecté enregistré à partir de la MD-MEA (figures 3J et K).
Connectome fonctionnel multimodal à l’échelle du réseau
Pour déduire la connectivité à grande échelle des réseaux neuronaux multicouches à partir de modèles de décharge simultanée d’ensembles neuronaux actifs simultanés, la covariance croisée entre les paires d’électrodes actives dans les événements détectés a été calculée selon l’étape 4.2.6 du protocole. Ici, le coefficient de corrélation a été trié en fonction des couches dans les circuits HC et OB ou non trié dans le réseau iPSC, puis stocké dans une matrice symétrique. Des connectomes fonctionnels des circuits HC et OB ont été générés en appliquant la causalité multivariée de Granger et la fonction de transfert dirigé (DTF) pour quantifier l’influence d’une série chronologique sur une autre et évaluer le flux d’information directionnel au sein des liens corrélés dans les réseaux distincts. La cartographie du connectome de HC (Figure 4A) et OB (Figure 4B) et la visualisation du réseau ont été réalisées à l’aide de la version 9.2 du programme Gephi (https://gephi.org). Des contraintes de paramètres similaires ont été placées sur les liens fonctionnels pour comparer les circuits de coupe de cerveau HC et OB et ont illustré 100 s de la connectivité fonctionnelle des événements LFP détectés. Les nœuds sont mis à l’échelle en fonction de leur degré d’intensité, la couleur nodale indiquant la couche et la couleur du lien identifiant les connexions intra et intercouches. Les connectomes fonctionnels des réseaux iPSC humains ont été générés en appliquant des filtres spatio-temporels (STF) et des seuils de latence dépendants de la distance (DdLT) pour améliorer la sélection des liens significatifs et affiner l’identification des connexions significatives en appliquant une analyse d’histogramme de corrélation croisée filtrée et normalisée (FNCCH). Cartographie du connectome des réseaux iPSC humains sur l’ensemble de la visualisation de la puce HD-MEA (Figure 4C) réalisée à l’aide de Gephi. La couleur nodale indique une entrée excitatrice ou inhibitrice, et la couleur de liaison identifie les connexions.
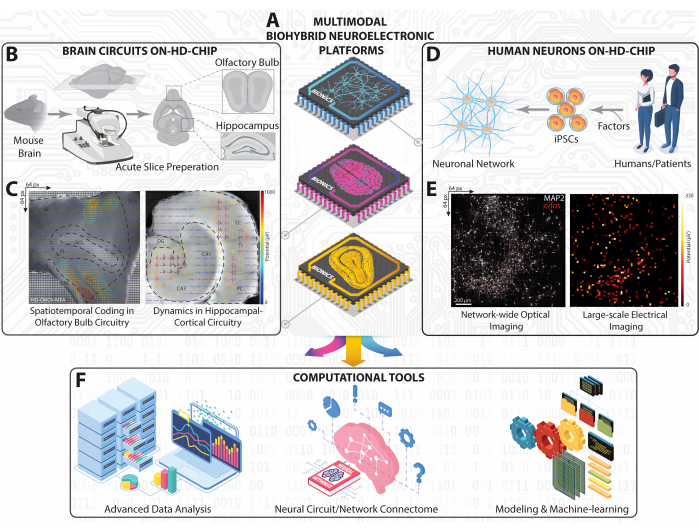
Figure 1 : Vue d’ensemble de la plateforme expérimentale et computationnelle sur la HD-MEA à grande échelle. (A) Représentation schématique isométrique de nos plateformes neuroélectroniques biohybrides multimodales réalisées avec la HD-MEA basée sur CMOS pour capturer la dynamique neuronale des circuits et réseaux neuronaux HC, OB et iPSC humains. (B) Flux de travail schématique pour le découpage du cerveau de la souris et son environnement de travail pour obtenir des coupes HC et OB. (C) Représentations topographiques des modèles de tir à grande échelle enregistrés simultanément à partir des coupes HC et OB entières superposées aux formes d’onde extracellulaires extracellulaires extraites aux images optiques de coupe. (D) Représentation schématique du réseau neuronal iPSC obtenu à partir d’humains. (E) Micrographies à fluorescence montrant le c-fos cellulaire et le MAP-2 somatique/dendritique de l’ensemble du réseau neuronal humain sur puce HD-MEA (à gauche) appariés à l’ensemble de la carte d’activité de tir moyenne (à droite). (F) Cadre de calcul comprenant des outils avancés d’analyse de données, de cartographie de la connectivité et d’apprentissage automatique de l’IA pour analyser les données neuronales multidimensionnelles obtenues à partir d’enregistrements à grande échelle sur des MDE-AEM. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Dispositions des espaces de travail de préparation et d’enregistrement de la culture de CSPi ex-vivo et de la culture humaine in vitro. (A) Flux de travail schématique illustrant la configuration de la préparation des coupes HC et OB, avec les outils et l’équipement requis dans chaque espace de travail. (B) Représentation schématique pour la préparation de la culture humaine des CSPi, y compris les outils et dispositifs nécessaires. Une liste complète des matériaux est incluse aux étapes 1.2.2, 2.1, 2.2, 3.1.1, 3.3 et dans le tableau des matériaux. Veuillez cliquer ici pour voir une version agrandie de cette figure.
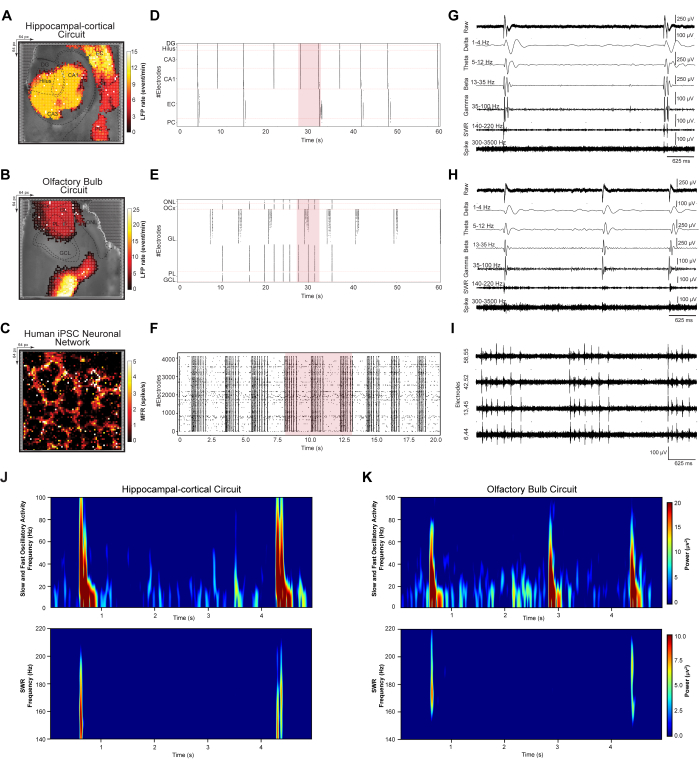
Figure 3 : Cartographie et extraction des modèles spatio-temporels de la dynamique des réseaux. (A-C) Cartes spatiales de la LFP moyenne et du taux de pointe, calculées sur des enregistrements de cinq minutes, superposées à l’image lumineuse du microscope. (D-F) Tracés raster représentant les événements LFP détectés et débruités dans un sous-échantillon de données de 60 secondes et les pics dans un sous-échantillon de données de 20 secondes. (G-I) Extraction de traces de forme d’onde représentatives à partir d’un segment de 5 secondes du sous-échantillon de données du diagramme matriciel (surligné en rouge dans le graphique matriciel), affiché sous forme de bandes oscillatoires LFP brutes (1-100 Hz) ; δ (1-4 Hz), θ (5-12 Hz), β (13-35 Hz) et γ (35-100 Hz) ; ROS (140-220 Hz) ; et le pic haute fréquence simple et MUA (300-3500 Hz). (J,K) Cartes de densité spectrale de puissance des LFP oscillatoires rapides et lentes (1-100 Hz) et du TOS (140-220 Hz). Veuillez cliquer ici pour voir une version agrandie de cette figure.
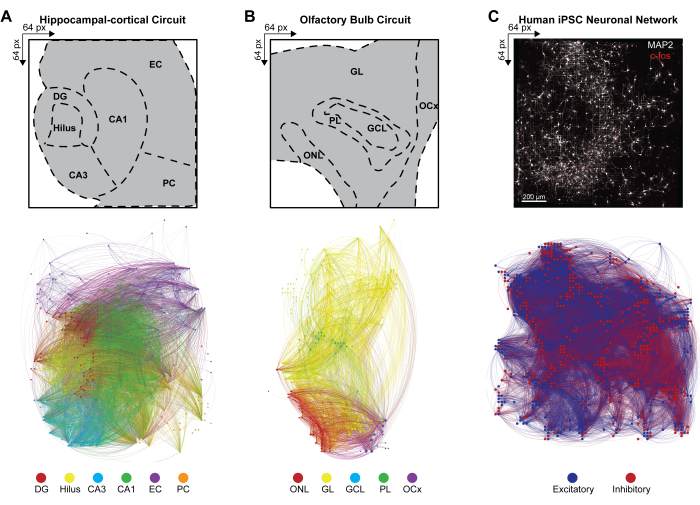
Figure 4 : Organisation des connectomes fonctionnels multimodaux à l’échelle du réseau. (A-C) Cartes Gephi illustrant la connectivité fonctionnelle des nœuds, où les nœuds correspondent à l’une des légendes de la barre de couleur (ci-dessous), tandis que les liens (ou arêtes) sont ombrés pour correspondre aux nœuds de connexion. Des exemples de légendes pour (A) HC, (B) OB et (C) iPSC sont affichés sur un tableau de 64 x 64. Les couches HC et OB sont tracées sur une plage de temps de 100 s pour réduire efficacement le nombre de nœuds et de liens visibles à des fins de visualisation. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Tableau 1 : Solutions pour la préparation de coupes de cerveau et les milieux pour les cultures neuronales iPSC. (A) Solution de coupe à haute teneur en saccharose pour la préparation de tranches de cerveau ex-vivo. (B) une solution d’enregistrement du LCR pour la préparation et l’enregistrement de tranches de cerveau ex-vivo. (C-D) Protocole de milieu iPSC neuronal humain, où (C) est le milieu complet de BrainPhys utilisé pour la décongélation cellulaire, le revêtement de puce HD-MEA et la maintenance de la HD-MEA cultivée, et (D) le milieu de pointage utilisé pour le placage cellulaire HD-MEA. Veuillez cliquer ici pour télécharger ce tableau.
Tableau 2 : Résolution des problèmes courants d’acquisition d’enregistrements HD-MEA. Liste des problèmes courants, de leurs causes potentielles et des solutions de dépannage liées aux puces HD-MEA, à la plate-forme d’enregistrement, au bruit du système et aux logiciels. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
La dynamique complexe de l’activité neuronale spatio-temporelle, émergeant d’ensembles neuronaux interconnectés, a longtemps été un sujet d’intrigue en neurosciences. Les méthodologies traditionnelles, telles que le patch-clamp, le MEA standard et l’imagerie Ca2+, ont fourni des informations précieuses sur la complexité du cerveau. Cependant, ils ne parviennent souvent pas à capturer la dynamique de calcul complète à l’échelle du réseau 21,22,23. Le protocole technique de la plateforme HD-MEA, tel que détaillé dans cette étude JoVE, représente un bond en avant significatif, offrant une vue panoramique de la dynamique neuronale à travers diverses modalités, des assemblages cellulaires aux réseaux expansifs (c’est-à-dire les tranches aiguës de cerveau de souris ex-vivo et les réseaux iPSC humains in vitro)26,29,30,32.
Les coupes aiguës de cerveau de souris ex-vivo ont été un outil fondamental dans la recherche neuronale, facilitant les études moléculaires et au niveau des circuits 6,7. Cependant, le défi du maintien de la viabilité des tissus a été un goulot d’étranglement persistant. Le protocole décrit dans cette étude introduit des modifications critiques pour optimiser la qualité et la longévité de ces coupes afin d’exploiter leurs avantages sur la plateforme HD-MEA. Ce protocole souligne l’importance de - i) Obtenir l’uniformité des tranches, pour lequel l’utilisation d’un vibratome est préférée à un broyeur de tissus en raison de sa précision et de ses dommages tissulaires minimisés, malgré le compromis de temps de tranchage plus longs. ii) Assurer une carbogénation constante tout au long du processus, de l’extraction à l’enregistrement, pour maintenir la viabilité des tissus. iii) Réguler la température et permettre un temps de récupération adéquat avant l’enregistrement. iv) Utiliser un bloc ou un moule d’agarose pour stabiliser le cerveau, prévenir les déchirures et minimiser le contact avec la colle. v) Maintenir des débits optimaux de LCR carbogéné dans le réservoir de méant-mélène HD pour assurer la santé des tranches tout en évitant les problèmes tels que le découplage, le bruit et la dérive (tableau 2).
Pour les coupes de cerveau de souris et les préparations d’iPSC humaines, l’amélioration du couplage électrode-tissu est primordiale 30,46,47. Notre protocole souligne l’importance d’utiliser la molécule favorisant l’adhésion Poly-dl-ornithine (PDLO). Cette molécule augmente non seulement la surface de détection des signaux électriques, mais augmente également la conductivité électrique46. Ce faisant, il favorise l’adhésion cellulaire, la croissance et le développement des propriétés fonctionnelles du réseau. Cette optimisation joue un rôle central dans l’amélioration de l’efficacité de la plateforme HD-MEA. Ceci, à son tour, garantit une analyse précise et cohérente des connectomes ex-vivo et in vitro à l’échelle microscopique et de leurs séquences de tir spatio-temporelles. Il a notamment été démontré que PDLO surpasse d’autres substrats comme la polyéthylèneimine (PEI) et la poly-l-ornithine (PLO) pour favoriser l’activité de décharge spontanée et la réactivité aux stimuli électriques dans les cultures neuronales. De plus, le PDLO a été utilisé pour la fonctionnalisation de surface sur le HD-MEA et s’est avéré améliorer l’interface de couplage électrode-tranche et augmenter le rapport signal/bruit dans les coupes OB et HC26,29. L’ajout d’un ancrage en platine sur mesure augmente encore le couplage électrode-tranche, ce qui permet d’obtenir des enregistrements avec un rapport signal/bruit plus élevé.
L’utilisation de la HD-MEA pour les tranches de cerveau de souris ex-vivo et les réseaux iPSC humains in vitro introduit une méthode apte à explorer la dynamique extensive, multi-échelle et multimodale. Cette approche innovante soulève toutefois des défis considérables, notamment en matière de gestion des données 48,49,50,51. Un seul enregistrement HD-MEA acquis à une fréquence d’échantillonnage de 18 kHz/électrode génère 155 Mo/s de données. Le volume de données augmente rapidement lorsque l’on tient compte de plusieurs tranches, de diverses conditions pharmacologiques ou de périodes d’enregistrement prolongées. Un tel afflux d’informations nécessite des infrastructures de stockage robustes et des outils de calcul avancés pour un traitement rationalisé. La capacité de la plate-forme HD-MEA à collecter simultanément des données à partir de milliers d’ensembles neuronaux est à la fois une aubaine et un obstacle. Il fournit des informations suprêmes sur la dynamique computationnelle des fonctions cérébrales, mais il nécessite également un cadre analytique affiné. Dans ce protocole JoVE, nous avons fourni des exemples de stratégies informatiques, notamment la détection d’événements à grande échelle, la classification, la théorie des graphes, l’analyse fréquentielle et l’apprentissage automatique. Ces méthodes soulignent les efforts intensifs déployés pour relever les défis de l’analyse de données neuronales complexes. Néanmoins, il reste encore beaucoup de place pour le développement d’outils informatiques plus avancés pour analyser ces ensembles de données neuronales multidimensionnelles. Armée des outils et des méthodologies appropriés, le potentiel de la plateforme HD-MEA est amplifié, offrant des informations approfondies sur les subtilités des fonctions cérébrales dans des conditions saines et pathologiques.
Essentiellement, la plateforme HD-MEA, lorsqu’elle est intégrée aux protocoles détaillés et aux outils informatiques discutés, offre une approche transformatrice pour comprendre le fonctionnement complexe du cerveau. En capturant des dynamiques à grande échelle, multi-échelles et multimodales, il fournit des informations précieuses sur des processus tels que l’apprentissage, la mémoire et le traitement de l’information. De plus, son application dans les réseaux de CSPi humaines in vitro a le potentiel de révolutionner le dépistage des médicaments et la médecine personnalisée. Cependant, bien que cette plateforme représente une avancée significative dans la recherche en neurosciences, il est crucial de reconnaître et de relever les défis techniques inhérents. Grâce à l’amélioration continue et à l’intégration d’outils informatiques avancés, la plateforme HD-MEA est prête à inaugurer une nouvelle ère d’outils de diagnostic précis, d’identification de biomarqueurs spécifiques et de thérapies ciblées pour les troubles neurologiques.
Déclarations de divulgation
Les auteurs ne déclarent aucun intérêt concurrent ou financier.
Remerciements
Cette étude a été soutenue par des fonds institutionnels (DZNE), l’Association Helmholtz dans le cadre du Fonds de validation Helmholtz (HVF-0102) et l’École supérieure internationale de biomédecine et de bioingénierie de Dresde (DIGS-BB). Nous tenons également à remercier la plateforme pour les tests comportementaux sur les animaux de la DZNE-Dresde (Alexander Garthe, Anne Karasinsky, Sandra Günther et Jens Bergmann) pour son soutien. Nous tenons à souligner qu’une partie de la figure 1 a été créée à l’aide de la plateforme BioRender.com.
matériels
| Name | Company | Catalog Number | Comments |
| 150 mm Glass Petri Dish | generic | generic | Brain Preparation Workspace, Brain Slice Recording Workspace |
| 0.22 μm Sterile Filter Unit | Assorted | Assorted | Assorted |
| 90 mm Plastic Culture Dish | TPP | 93100 | Brain Preparation Workspace, Brain Slice Recording Workspace |
| Agarose | Roth | 6351.5 | Brain Preparation Workspace |
| Agarose Mold | CUSTOM | CUSTOM | Brain Preparation Workspace; Custom designed 3D Printer Design, available upon request |
| Aluminum Foil | generic | generic | Brain Extraction Workspace |
| Anesthesia chamber | generic | generic | Brain Extraction Workspace; Assorted Beaker, Bedding etc |
| Ascorbic Acid | Sigma Aldrich | A4544-25G | Solution Preparation Workspace |
| Assorted Beakers | generic | generic | Solution Preparation Workspace; 50 mL |
| Assorted Luers | Cole Parmer | 45511-00 | Brain Slice Recording Workspace |
| Assorted Volumetric flasks | generic | generic | Solution Preparation Workspace; 500 mL, 1 L |
| B27 Supplement | Life Technologies | 17504-044 | BrainXell Commercial Supplier Protocol |
| BDNF | Peprotech | 450-02 | BrainXell Commercial Supplier Protocol |
| Biological Safety Cabinet with UV Lamp | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| BrainPhys Neuronal Medium | STEMCELL Technologies | 05790 | CDI, and BrainXell Commerical Supplier Protocol |
| Brainwave Software | 3Brain AG | Version 4 | Brain Slice and Human iPSC Recording Workspace |
| BrainXell Glutamatergic Neuron Assay | BrainXell | BX-0300 | BrainXell Commercial Supplier Protocol |
| CaCl2 | Sigma Aldrich | 21115-100ML | Solution Preparation Workspace |
| Carbogen | generic | generic | All Workspaces; 95%/5% O2 and CO2 mixture |
| Cell Culture Incubator | Assorted | Assorted | Assorted |
| CMOS-based HD-MEA chip | 3Brain AG | CUSTOM | Brain Slice and Human iPSC Recording Workspace |
| Conical Tubes, 50 mL, Falcon (Centrifuge Tubes) | STEMCELL Technologies | 38010 | CDI Commerical Supplier Protocol |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | Brain Slice Recording Workspace |
| Curved Forceps | FST | 11052-10 | Brain Extraction Workspace |
| DMEM/F12 Medium | Life Technologies | 11330-032 | BrainXell Commercial Supplier Protocol |
| Dulbecco’s Phosphate Buffered Saline without Ca2+ and Mg2+ (D-PBS) | STEMCELL Technologies | 37350 | CDI Commerical Supplier Protocol |
| Filter Paper | Macherey-Nagel | 531 011 | Brain Preparation Workspace |
| Fine Brush | Leonhardy | 773 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Forceps | VITLAB | 67895 | Brain Slice Recording Workspace |
| GDNF | Peprotech | 450-10 | BrainXell Commercial Supplier Protocol |
| Geltrex | Life Technologies | A1413201 | BrainXell Commercial Supplier Protocol |
| Glass pasteur pipette | Roth | 4518 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Glucose | Sigma Aldrich | G7021-1KG | Solution Preparation Workspace |
| GlutaMAX | Life Technologies | 35050-061 | BrainXell Commercial Supplier Protocol |
| Gravity-based Perfusion System | ALA | VC3-8xG | Brain Slice Recording Workspace |
| HD-MEA Recording platform | 3Brain AG | CUSTOM | Brain Slice and Human iPSC Recording Workspace |
| Heater | Warner Instruments | TC-324C | Brain Slice Recording Workspace |
| Hemocytometer or Automated Cell Counter | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| Hypo Needles | Warner Instruments | 641489 | Brain Slice Recording Workspace |
| iCell GlutaNeurons Kit, 01279 | CDI | R1061 | CDI Commerical Supplier Protocol |
| Iris Scissors | Vantage | V95-304 | Brain Extraction Workspace |
| Isoflurane | Baxter | HDG9623 | Brain Extraction Workspace |
| KCl | Sigma Aldrich | P5405-250G | Solution Preparation Workspace |
| Laminin | Sigma-Aldrich | L2020 | CDI Commerical Supplier Protocol |
| Liquid Nitrogen Storage Unit | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| Magnetic Stirrer | generic | generic | Solution Preparation Workspace |
| Metal Screws | Thorlabs | HW-KIT2/M | Brain Slice Recording Workspace |
| MgCl2 | Sigma Aldrich | M1028-100ML | Solution Preparation Workspace |
| MgSO4 | Sigma Aldrich | 63138-250G | Solution Preparation Workspace |
| Microdissection Tool Holder | Braun | 4606108V | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Microdissection Tool Needle | Braun | 9186166 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Modular Stereomicroscope | Leica | CUSTOM | Brain Slice Recording Workspace; custom specifications and modifications |
| N2 Supplement | Life Technologies | 17502-048 | CDI, and BrainXell Commercial Supplier Protocol |
| NaCl | Sigma Aldrich | S3014-1KG | Solution Preparation Workspace |
| NaH2PO4 | Sigma Aldrich | S0751-100G | Solution Preparation Workspace |
| NaHCO3 | Sigma Aldrich | S5761-500G | Solution Preparation Workspace |
| Neurobasal Medium | Life Technologies | 21103-049 | BrainXell Commercial Supplier Protocol |
| Optical Cage System | Thorlabs | Assorted | Brain Slice Recording Workspace |
| Optical Table w/Breadboard | Thorlabs | SDA7590 | Brain Slice Recording Workspace |
| PDLO | Sigma Aldrich | P0671 | HD-MEA Coating, Brain Slice Recording Workspace |
| Penicillin-streptomycin, 100x | Thermo Fisher Scientific | 15140-122 | CDI Commerical Supplier Protocol |
| Pipette tips | TipONE | S1120-8810 | Brain Slice Recording Workspace |
| Pipettors | Assorted | Assorted | Assorted |
| Platinum Anchor | CUSTOM | CUSTOM | Brain Slice Recording Workspace |
| Polyethylene Tubing | Assorted | Assorted | Brain Slice Recording Workspace |
| Pump | MasterFlex | 78018-22 | Brain Slice Recording Workspace |
| Razor Blade | Apollo | 10179960 | Brain Preparation Workspace |
| Reference Electrode Cell Culture Cap | CUSTOM | CUSTOM | Human iPSC Recording Workspace; Custom designed 3D Printer Design, available upon request |
| Rubber Pipette Bulb | Duran Wheaton Kimble | 292000205 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Serological Pipettes, 1 mL, 2 mL, 5 mL, 10 mL, 25 mL | Assorted | Assorted | Assorted |
| Slice Recovery Chamber | CUSTOM | CUSTOM | Brain Slice Recovery Workspace; Custom designed 3D Printer Design, available upon request |
| Spatula | ISOLAB | 047.06.150 | Brain Preparation Workspace |
| Sucrose | Sigma Aldrich | 84100-1KG | Solution Preparation Workspace |
| Super Glue | UHU | 358221 | Brain Slice Preparation Workspace |
| Surgical Scissors | Peters Instruments | BC 344 | Brain Extraction Workspace |
| Tabletop Centrifuge | Assorted | Assorted | Assorted |
| TGF-β1 | Peprotech | 100-21C | BrainXell Commercial Supplier Protocol |
| Tissue Paper | generic | generic | Brain Extraction Workspace |
| Trypan Blue | STEMCELL Technologies | 07050 | CDI Commerical Supplier Protocol |
| Upright Microscope | Olympus | CUSTOM | Imaging Workspace; Custom specifications and modifications |
| Vacusip | Integra | 159010 | Brain Slice Recording Workspace |
| Vibratome | Leica | VT1200s | Brain Slice Preparation Workspace; Includes: Specimen plate, buffer tray, ice tray, specimen plate holding tool, vibratome blade adjusting tool |
| Vibratome Blade | Personna | N/A | Brain Slice Preparation Workspace |
| Water Bath | Lauda | L000595 | Brain Slice Recovery Workspace |
Références
- Hebb, D. O. . The Organization of Behavior; A Neuropsychological Theory. , (1949).
- Cossart, R., Garel, S. Step by step: cells with multiple functions in cortical circuit assembly. Nat Rev Neurosci. 23, 395-410 (2022).
- Carrillo-Reid, L., Yuste, R. Playing the piano with the cortex: role of neuronal ensembles and pattern completion in perception and behavior. Curr Opin Neurobiol. 64, 89-95 (2020).
- Buzsáki, G. Large-scale recording of neuronal ensembles. Nat Neurosci. 7, 446-451 (2004).
- Buzsáki, G. Neural Syntax: Cell assemblies, synapsembles, and readers. Neuron. 68 (3), 362-385 (2010).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Cho, S., Wood, A., Bowlby, M. Brain slices as models for neurodegenerative disease and screening platforms to identify novel therapeutics. Curr Neuropharmacol. 5 (1), 19-33 (2007).
- Bliss, T. V. P., Collingridge, G. L. A synaptic model of memory: long-term potentiation in the hippocampus. Nature. 361, 31-39 (1993).
- Anderson, P., Morris, R., Amaral, D., Bliss, T., O'Keefe, L. . The Hippocampus Book. , (2006).
- Lisman, J., et al. Viewpoints: how the hippocampus contributes to memory, navigation and cognition. Nat Neurosci. 20, 1434-1447 (2017).
- Mori, K., Nagao, H., Yoshihara, Y. The olfactory bulb: Coding and processing of odor molecule information. Science. 286 (5440), 711-715 (1999).
- Buck, L., Axel, R. A novel multigene family may encode odorant receptors: A molecular basis for odor recognition. Cell. 65 (1), 175-187 (1991).
- Bushdid, C., Magnasco, M. O., Vosshall, L. B., Keller, A. Humans can discriminate more than 1 trillion olfactory stimuli. Science. 343 (6177), 1370-1372 (2014).
- Kempermann, G. Why new neurons? Possible functions for adult hippocampal neurogenesis. J Neurosci. 23 (3), 635-638 (2003).
- Aimone, J. B., Wiles, J., Gage, F. H. Computational influence of adult neurogenesis on memory encoding. Neuron. 61 (2), 187-202 (2009).
- Nithianantharajah, J., Hannan, A. J. Enriched environments, experience-dependent plasticity and disorders of the nervous system. Nat Rev Neurosci. 7, 697-709 (2006).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Espuny-Camacho, I., et al. Pyramidal neurons derived from human pluripotent stem cells integrate efficiently into mouse brain circuits in vivo. Neuron. 77 (3), 440-456 (2013).
- Rajamohan, D., et al. Current status of drug screening and disease modelling in human pluripotent stem cells. Bioessays. 35 (3), 281-298 (2013).
- Heilker, R., Traub, S., Reinhardt, P., Schöler, H. R., Sterneckert, J. iPS cell derived neuronal cells for drug discovery. Trends Pharmacol Sci. 35 (10), 510-519 (2014).
- Zhao, S. R., Mondéjar-Parreño, G., Li, D., Shen, M., Wu, J. C. Technical applications of microelectrode array and patch clamp recordings on human induced pluripotent stem cell-derived cardiomyocytes. J Vis Exp. (186), e64265 (2022).
- Hamill, O. P., McBride, D. W. Induced membrane hypo/hyper-mechanosensitivity: A limitation of patch-clamp recording. Annu Rev Physiol. 59, 621-631 (1997).
- Manz, K. M., Siemann, J. K., McMahon, D. G., Grueter, B. A. Patch-clamp and multi-electrode array electrophysiological analysis in acute mouse brain slices. STAR Protoc. 2 (2), 100442 (2021).
- Lee, C. H., Park, Y. K., Lee, K. Recent strategies for neural dynamics observation at a larger scale and wider scope. Biosens Bioelectron. 240, 115638 (2023).
- Urai, A. E., Doiron, B., Leifer, A. M., Churchland, A. K. Large-scale neural recordings call for new insights to link brain and behavior. Nat Neurosci. 25 (1), 11-19 (2022).
- Hu, X., Khanzada, S., Klütsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834 (2022).
- Amin, H., Marinaro, F., Tonelli, D. D. P., Berdondini, L. Developmental excitatory-to-inhibitory GABA-polarity switch is disrupted in 22q11.2 deletion syndrome: A potential target for clinical therapeutics. Sci Rep. 7 (1), 15752 (2017).
- Amin, H., Nieus, T., Lonardoni, D., Maccione, A., Berdondini, L. High-resolution bioelectrical imaging of Aβ-induced network dysfunction on CMOS-MEAs for neurotoxicity and rescue studies. Sci Rep. 7 (1), 2460 (2017).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471 (2023).
- Amin, H., et al. Electrical responses and spontaneous activity of human iPS-derived neuronal networks characterized for 3-month culture with 4096-electrode arrays. Front Neurosci. 10, 121 (2016).
- Lonardoni, D., et al. Recurrently connected and localized neuronal communities initiate coordinated spontaneous activity in neuronal networks. PLoS Comput Biol. 13 (7), e1005672 (2017).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. 2022 44th Annual International Conference of the IEEE Engineering in Medicine & Biology Society (EMBC). , 3111-3114 (2022).
- Rossi, L., Emery, B. A., Khanzada, S., Hu, X., Amin, H. Pharmacologically and electrically-induced network-wide activation of olfactory bulb with large-scale biosensor. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Emery, B. A., et al. Recording network-based synaptic transmission and LTP in the hippocampal network on a large-scale biosensor. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays). Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Berdondini, L., et al. Active pixel sensor array for high spatio-temporal resolution electrophysiological recordings from single cell to large scale neuronal networks. Lab Chip. 9, 2644-2651 (2009).
- Siegle, J. H., Hale, G. J., Newman, J. P., Voigts, J. Neural ensemble communities: open-source approaches to hardware for large-scale electrophysiology. Curr Opin Neurobiol. 32, 53-59 (2015).
- Amin, H., Maccione, A., Zordan, S., Nieus, T., Berdondini, L. High-density MEAs reveal lognormal firing patterns in neuronal networks for short and long term recordings. 2015 7th International IEEE/EMBS Conference on Neural Engineering (NER). , 1000-1003 (2015).
- Altuntac, E., et al. Bottom-up neurogenic-inspired computational model. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Maccione, A., et al. A novel algorithm for precise identification of spikes in extracellularly recorded neuronal signals. J Neurosci Methods. 177 (1), 241-249 (2009).
- Welch, P. D. The use of fast Fourier transform for the estimation of power spectra: A method based on time averaging over short, modified periodograms. IEEE Transactions on Audio and Electroacoustics. 15 (2), 70-73 (1967).
- Eggermont, J. J., Munguia, R., Pienkowski, M., Shaw, G. Comparison of LFP-based and spike-based spectro-temporal receptive fields and cross-correlation in cat primary auditory cortex. PLoS One. 6 (5), e20046 (2011).
- Damos, P. Using multivariate cross correlations, Granger causality and graphical models to quantify spatiotemporal synchronization and causality between pest populations. BMC Ecol. 16, 33 (2016).
- Kaminski, M. J., Blinowska, K. J. A new method of the description of the information flow in the brain structures. Biol Cybern. 65, 203-210 (1991).
- Pastore, V. P., Massobrio, P., Godjoski, A., Martinoia, S. Identification of excitatory-inhibitory links and network topology in large-scale neuronal assemblies from multi-electrode recordings. PLoS Comput Biol. 14 (8), e1006381 (2018).
- Amin, H., Dipalo, M., De Angelis, F., Berdondini, L. Biofunctionalized 3D nanopillar arrays fostering cell guidance and promoting synapse stability and neuronal activity in networks. ACS Appl Mater Interfaces. 10 (17), 15207-15215 (2018).
- Woeppel, K., Yang, Q., Cui, X. T. Recent advances in neural electrode-tissue interfaces. Curr Opin Biomed Eng. 4, 21-31 (2017).
- Steinmetz, N. A., Koch, C., Harris, K. D., Carandini, M. Challenges and opportunities for large-scale electrophysiology with Neuropixels probes. Curr Opin Neurobiol. 50, 92-100 (2018).
- Siegle, J. H., Hale, G. J., Newman, J. P., Voigts, J. Neural ensemble communities: open-source approaches to hardware for large-scale electrophysiology. Curr Opin Neurobiol. 32, 53-59 (2015).
- Freeman, J. Open source tools for large-scale neuroscience. Curr Opin Neurobiol. 32, 156-163 (2015).
- Stevenson, I. H., Kording, K. P. How advances in neural recording affect data analysis. Nat Neurosci. 14 (2), 139-142 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon