Method Article
Registrazione e analisi della dinamica di ensemble neuronali multimodali su larga scala su array di microelettrodi ad alta densità integrati in CMOS
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Qui, impieghiamo l'HD-MEA per approfondire le dinamiche computazionali di insiemi neuronali su larga scala, in particolare nell'ippocampo, nei circuiti del bulbo olfattivo e nelle reti neuronali umane. L'acquisizione dell'attività spazio-temporale, combinata con strumenti computazionali, fornisce informazioni sulla complessità dell'insieme neuronale. Il metodo migliora la comprensione delle funzioni cerebrali, identificando potenzialmente biomarcatori e trattamenti per i disturbi neurologici.
Abstract
Le reti neuronali su larga scala e i loro complessi microcircuiti distribuiti sono essenziali per generare percezione, cognizione e comportamento che emergono da modelli di attività neuronale spazio-temporale. Questi modelli dinamici che emergono da gruppi funzionali di insiemi neuronali interconnessi facilitano calcoli precisi per l'elaborazione e la codifica di informazioni neurali multiscala, guidando così le funzioni cerebrali superiori. Per sondare i principi computazionali delle dinamiche neurali alla base di questa complessità e studiare l'impatto multiscala dei processi biologici nella salute e nella malattia, le registrazioni simultanee su larga scala sono diventate strumentali. Qui, un array di microelettrodi ad alta densità (HD-MEA) viene impiegato per studiare due modalità di dinamica neurale: circuiti del bulbo ippocampale e olfattivo da fette di cervello di topo ex-vivo e reti neuronali da colture cellulari in vitro di cellule staminali pluripotenti indotte umane (iPSC). La piattaforma HD-MEA, con 4096 microelettrodi, consente registrazioni non invasive, multi-sito e prive di etichette di modelli di attivazione extracellulare da migliaia di insiemi neuronali simultaneamente ad alta risoluzione spazio-temporale. Questo approccio consente la caratterizzazione di diverse caratteristiche elettrofisiologiche a livello di rete, tra cui modelli di attività di spiking a singola o multi-unità e oscillazioni del potenziale di campo locale. Per esaminare questi dati neurali multidimensionali, abbiamo sviluppato diversi strumenti computazionali che incorporano algoritmi di apprendimento automatico, rilevamento e classificazione automatica degli eventi, teoria dei grafi e altre analisi avanzate. Integrando queste pipeline computazionali con questa piattaforma, forniamo una metodologia per studiare le dinamiche grandi, multiscala e multimodali dagli assemblaggi di celle alle reti. Questo può potenzialmente far progredire la nostra comprensione delle complesse funzioni cerebrali e dei processi cognitivi in salute e malattia. L'impegno per la scienza aperta e le intuizioni sulle dinamiche neurali computazionali su larga scala potrebbero migliorare la modellazione ispirata al cervello, il calcolo neuromorfico e gli algoritmi di apprendimento neurale. Inoltre, la comprensione dei meccanismi alla base dei calcoli neurali su larga scala compromessi e delle loro dinamiche microcircuitali interconnesse potrebbe portare all'identificazione di biomarcatori specifici, aprendo la strada a strumenti diagnostici più accurati e terapie mirate per i disturbi neurologici.
Introduzione
Gli insiemi neuronali, spesso definiti assemblaggi cellulari, sono fondamentali nella codifica neurale, facilitando calcoli complessi per l'elaborazione di informazioni neurali multiscala 1,2,3. Questi insiemi sono alla base della formazione di reti neuronali espansive e dei loro microcircuiti sfumati4. Tali reti e i loro modelli oscillatori guidano funzioni cerebrali avanzate, tra cui la percezione e la cognizione. Mentre un'ampia ricerca ha esplorato specifici tipi neuronali e percorsi sinaptici, una comprensione più profonda di come i neuroni formano in modo collaborativo gli assemblaggi cellulari e influenzano l'elaborazione delle informazioni spazio-temporali attraverso circuiti e reti rimaneelusiva.
Le fette di cervello acute ex-vivo sono strumenti elettrofisiologici fondamentali per lo studio dei circuiti neurali intatti, offrendo un ambiente controllato per sondare i modelli di attività oscillatoria della funzione neurale, della trasmissione sinaptica e della connettività, con implicazioni nei test farmacologici e nella modellazione della malattia 6,7,8. Questo protocollo di studio evidenzia due circuiti cerebrali chiave: l'ippocampo-corticale (HC) coinvolto nei processi di apprendimento e memoria 9,10 e il bulbo olfattivo (OB) responsabile della discriminazione degli odori 11,12,13. In queste due regioni, nuovi neuroni funzionali sono continuamente generati dalla neurogenesi adulta per tutta la vita nel cervello dei mammiferi14. Entrambi i circuiti dimostrano modelli di attività neurale dinamica multidimensionale e plasticità intrinseca che partecipano al ricablaggio della rete neurale esistente e facilitano strategie alternative di elaborazione delle informazioni quando richiesto15,16.
I modelli acuti ex-vivo di fette cerebrali sono indispensabili per approfondire la funzionalità del cervello e comprendere i meccanismi della malattia a livello di microcircuito. Tuttavia, le colture cellulari in vitro derivate da reti neuronali di cellule staminali pluripotenti indotte umane (iPSC) offrono una strada promettente per la ricerca traslazionale, collegando senza soluzione di continuità i risultati degli esperimenti sugli animali al potenziale trattamento clinico umano17,18. Questi saggi in vitro incentrati sull'uomo fungono da piattaforma affidabile per la valutazione della tossicità farmacologica, consentendo uno screening preciso dei farmaci e promuovendo la ricerca su strategie terapeutiche innovative basate sulle cellule19,20. Riconoscendo il ruolo fondamentale del modello neuronale iPSC, abbiamo dedicato il terzo modulo di questo studio del protocollo ad indagare a fondo le caratteristiche funzionali delle sue reti derivate e a mettere a punto i protocolli di coltura cellulare associati.
Questi moduli neurali elettrogenici sono stati comunemente studiati utilizzando tecniche come il calcio (imaging Ca2+), registrazioni patch-clamp e array di microelettrodi a bassa densità (LD-MEA). Sebbene l'imaging Ca2+ offra la mappatura dell'attività di una singola cellula, si tratta di un metodo basato sull'etichettatura cellulare ostacolato dalla sua bassa risoluzione temporale e dalle sfide nelle registrazioni a lungo termine. Le LD-MEA mancano di precisione spaziale, mentre il patch-clamp, essendo una tecnica invasiva a sito singolo e laboriosa, spesso produce un basso tasso di successo 21,22,23. Per affrontare queste sfide e sondare efficacemente l'attività a livello di rete, le registrazioni neurali simultanee su larga scala sono emerse come un approccio fondamentale per comprendere i principi computazionali delle dinamiche neurali alla base della complessità cerebrale e le loro implicazioni nella salute e nella malattia24,25.
In questo protocollo JoVE, dimostriamo un metodo di registrazione neurale su larga scala basato sulla MEA ad alta densità (HD-MEA) per catturare l'attività neuronale spazio-temporale attraverso varie modalità cerebrali, inclusi i circuiti del bulbo ippocampale e olfattivo da fette acute di cervello di topo ex-vivo (Figure 1A-C) e reti neuronali umane derivate da iPSC umane in vitro (Figure 1D-E), precedentemente riportate dal nostro gruppo e da altri colleghi26,27,28,29,30,31,32,33,34,35. L'HD-MEA, basato sulla tecnologia CMOS (Complementary Metal-Oxide-Semiconductor), vanta circuiti e amplificazione su chip, che consentono registrazioni inferiori al millisecondo su un array da 7 mm2 di dimensioni36. Questo approccio non invasivo cattura modelli di attivazione extracellulare multi-sito e label-free da migliaia di insiemi neuronali contemporaneamente utilizzando 4096 microelettrodi ad alta risoluzione spazio-temporale, rivelando le intricate dinamiche dei potenziali di campo locali (LFP) e dell'attività di picco multiunità (MUA)26,29.
Data la vastità dei dati generati da questa metodologia, un quadro analitico sofisticato è essenziale, ma pone delle sfide37. Abbiamo sviluppato strumenti computazionali che comprendono il rilevamento automatico degli eventi, la classificazione, la teoria dei grafi, l'apprendimento automatico e altre tecniche avanzate (Figura 1F)26,29,38,39. Integrando l'HD-MEA con questi strumenti analitici, viene ideato un approccio olistico per sondare le intricate dinamiche dai singoli assemblaggi cellulari alle reti neurali più ampie attraverso diverse modalità neurali. Questo approccio combinato approfondisce la nostra comprensione delle dinamiche computazionali nelle normali funzioni cerebrali e offre approfondimenti sulle anomalie presenti in condizioni patologiche28. Inoltre, le intuizioni di questo approccio possono spingere i progressi nella modellazione ispirata al cervello, nel calcolo neuromorfico e negli algoritmi di apprendimento neurale. In definitiva, questo metodo è promettente nello scoprire i meccanismi principali alla base delle interruzioni della rete neurale, identificando potenzialmente i biomarcatori e guidando la creazione di strumenti diagnostici precisi e trattamenti mirati per le condizioni neurologiche.
Protocollo
Tutti gli esperimenti sono stati eseguiti in conformità con le normative europee e nazionali applicabili (Tierschutzgesetz) e sono stati approvati dall'autorità locale (Landesdirektion Sachsen; 25-5131/476/14).
1. Sezioni di cervello ex-vivo da circuiti del bulbo ippocampale-corticale e olfattivo su HD-MEA
- Preparazione di soluzioni sperimentali di taglio e registrazione (Figura 2A)
- Il giorno sperimentale, preparare 0,5 L di soluzione di taglio ad alto contenuto di saccarosio e 1 L di soluzione di registrazione del liquido cerebrospinale artificiale (aCSF) (Tabella 1A,B).
- Aggiungere tutti i prodotti chimici solidi in un matraccio tarato asciutto, quindi riempire parte del percorso con acqua bidistillata (dd).
- Aggiungere MgCl2 e CaCl2 dalle soluzioni madre 1 M, quindi riempire il resto con acqua dd. Iniziare a mescolare costantemente con un agitatore magnetico fino a quando i solidi visibili non si sono sciolti ~ 5 min.
- Utilizzare un osmometro del punto di congelamento per convalidare l'osmolarità tra 350-360 mOsm per la soluzione di taglio ad alto contenuto di saccarosio e 315-325 mOsm per la soluzione di registrazione dell'aCSF.
- Utilizzare un pHmetro per convalidare il pH compreso tra 7,3-7,4 per la soluzione di taglio ad alto contenuto di saccarosio e tra 7,25-7,35 per la soluzione di registrazione aCSF. Iniziare a gorgogliare continuamente con il 95% di O2 e il 5% di CO2 .
- Mettere la soluzione da taglio ad alto contenuto di saccarosio sul ghiaccio per almeno 30 minuti prima di affettare e iniziare a gorgogliare continuamente con il 95% di O2 e il 5% di CO2 .
- Dopo 10 minuti di carbogenazione, riempire un becher da 50 ml con 30 ml di soluzione da taglio e conservarlo nel congelatore (-20 °C) per 20-30 minuti o fino a completo congelamento.
NOTA: Tutte le soluzioni devono essere preparate fresche per ogni esperimento. L'acqua dd utilizzata qui è acqua ultrapura sterilizzata in autoclave conservata a temperatura ambiente (RT). La quantità di soluzione preparata deve essere adattata alla specifica domanda di studio.
- Il giorno sperimentale, preparare 0,5 L di soluzione di taglio ad alto contenuto di saccarosio e 1 L di soluzione di registrazione del liquido cerebrospinale artificiale (aCSF) (Tabella 1A,B).
- Preparazione delle aree di lavoro della fetta di cervello (Figura 2A)
- Porta l'animale nella stanza degli esperimenti.
NOTA: In questo protocollo, topi femmina C57BL/J6 di età compresa tra 8 e 16 settimane sono stati utilizzati come descritto in precedenza 26,29,32. L'animale deve essere lasciato acclimatare per almeno 30 minuti dopo il trasporto. I trasferimenti a lunga distanza (cioè tra istituti) dovrebbero essere evitati lo stesso giorno dell'esperimento. L'età, il sesso e il ceppo dell'animale devono essere determinati in base alla specifica domanda di studio. - Mentre l'animale si acclimata e la soluzione ad alto contenuto di saccarosio si raffredda, posizionare gli strumenti necessari in ogni area di lavoro designata (vedi Tabella dei materiali).
- Prepara lo spazio di lavoro per il recupero e la manutenzione delle fette cerebrali. Riempire la camera di recupero delle fette con una soluzione di registrazione aCSF carbogenata e posizionare la camera nel bagnomaria impostato a 32 °C. Mantenere la carbogenazione continua per tutta la durata dell'esperimento.
- Prepara l'area di lavoro per la preparazione delle fette di cervello. Impostare il vibratomo - posizionare la lama nel supporto della lama del vibratomo e calibrare il vibratomo sulle impostazioni corrette (velocità di traslazione della lama: 0,20 mm/s, ampiezza dell'altezza: 95 μm, angolo della lama: 45°). Riempire la vaschetta del ghiaccio del vibratomo con ghiaccio e la vaschetta tampone con una soluzione da taglio ad alto contenuto di saccarosio e iniziare a carbogenare la soluzione nella vaschetta tampone.
- Prepara lo spazio di lavoro per la preparazione del cervello. Riempire di ghiaccio la capsula di Petri in vetro da 150 mm e posizionare all'interno una capsula di plastica da 90 mm con carta da filtro. Riempi il piatto di coltura in plastica con una soluzione da taglio ad alto contenuto di saccarosio e inizia a carbogenare. Aggiungi una goccia di super colla alla piastra del campione raffreddata e attacca lo stampo di agarosio.
NOTA: La muffa di agarosio viene preparata almeno il giorno prima con il 3% di agarosio in acqua in uno stampo personalizzato per il cervello di topo. - Infine, prepara lo spazio di lavoro per l'estrazione del cervello. Coprire un foglio di alluminio con carta velina, recuperare un becher da 50 ml contenente granita di soluzione di taglio ad alto contenuto di saccarosio e aggiungere isoflurano alla camera di anestesia.
NOTA: L'anestesia verrà aggiunta alla camera di anestesia ~ 1 minuto prima del posizionamento dell'animale. Un becher da 50 ml con 30 ml di granita di soluzione di taglio ad alto contenuto di saccarosio verrà rimosso dal congelatore a -20 °C ~ 2 minuti prima della decapitazione.
- Porta l'animale nella stanza degli esperimenti.
- Estrazione e affettatura del cervello di topo
NOTA: L'intera procedura deve essere eseguita il più rapidamente possibile per evitare una mancanza di ossigenazione al cervello. La rimozione del cervello dovrebbe richiedere solo 1-2 minuti dalla decapitazione all'immersione nella granita della soluzione di taglio ad alto contenuto di saccarosio.- Anestetizzare l'animale con il dosaggio appropriato di isoflurano (camera di anestesia da 0,5 ml/1 L). Determinare la profondità dell'anestesia pizzicando una zampa; Confermare l'assenza del riflesso di ritiro della zampa prima di procedere.
- Trasferisci l'animale sulla carta velina nell'area di lavoro per l'estrazione del cervello e decapitalo con le forbici chirurgiche.
- Inserire le forbici dell'iride nel tronco encefalico e tenere le forbici inferiori a filo con la calvaria. Tagliare lungo la sutura sagittale fino a raggiungere la sutura coronale. Posiziona le forbici per l'iride nelle orbite e taglia la sutura metopica. Usa una pinza curva per spostare i lati della calvaria verso il basso, esponendo l'intero cervello.
NOTA: Fare attenzione sia con le forbici dell'iride che con le pinze per non perforare il cervello durante il taglio delle suture. - Far scorrere il cervello con il bordo smussato della pinza curva nel becher da 50 ml con 30 ml di granita di soluzione di taglio ad alto contenuto di saccarosio. Lasciare agire per 1 min.
- Trasferire il cervello nella piastra di coltura in plastica da 90 mm con soluzione di taglio carbogenata refrigerata nell'area di lavoro di preparazione del cervello. Orientare il cervello per il posizionamento nella muffa dell'agarosio.
- Aggiungi un piccolo punto di super colla all'estremità rostrale dello stampo di agarosio. Mettere il cervello nello stampo con la spatola. Assicurati che il cervello sia posizionato con il lato dorsale rivolto verso il basso per affettare orizzontalmente.
NOTA: La posizione della colla nello stampo cambierà a seconda della regione di interesse (ROI). Per le fette di ippocampo-corticale (HC) e del bulbo olfattivo (OB), assicurarsi che l'OB sia stabilizzato e che i lati del cervello rimangano liberi da colla. Troppa colla influirà sulla qualità dell'affettatura e causerà strappi durante l'affettatura del vibratomo. - Spostare la piastra del campione nel vassoio del tampone, spostare la lama in posizione con l'angolazione corretta e aumentare l'altezza del vassoio del tampone per portare la lama il più vicino possibile al cervello.
- Affettare a intervalli di 300 μm a una velocità di 0,20 mm/s i tessuti HC e OB, quindi raccoglierli dopo ogni ciclo di affettatura con una pipetta Pasteur in vetro.
- Lasciare le fette nella camera di recupero riempita di aCSF a bagnomaria a 32 °C per 45 minuti, seguiti da 1 ora a RT. Assicurarsi che le fette non si sovrappongano e siano completamente esposte alla soluzione carbogenata.
NOTA: Assicurarsi di mantenere la carbogenazione continua di tutte le soluzioni e di tutte le camere menzionate contenenti la soluzione. Un regolatore di pressione può essere utilizzato per sostenere una carbogenazione costante.
2. Rete neuronale umana in vitro basata su iPSC su HD-MEA
NOTA: Tutti i neuroni iPSC utilizzati in questo studio sono ottenuti commercialmente (vedere Tabella dei materiali). Queste cellule umane si sono differenziate da linee cellulari iPS stabili derivate da sangue periferico umano o fibroblasti.
- Rivestimento di chip HD-MEA per colture cellulari iPSC umane in vitro (Figura 2B)
- Posizionare il chip HD-MEA sulla piattaforma di registrazione di acquisizione, riempire il serbatoio con PBS e testare il chip prima del rivestimento. Avvia il software Brainwave. Selezionare File > Nuova sessione di registrazione. Impostare i parametri di registrazione in modo che abbiano una frequenza di registrazione di 50 Hz e una frequenza di campionamento di 18 kHz/elettrodo. Modificare l'offset dell'amplificatore per calibrare il chip. Vedere la Tabella 2 per suggerimenti sulla risoluzione dei problemi.
NOTA: I parametri della frequenza di registrazione e della frequenza di campionamento dipendono dal tipo di dati e dai requisiti di sistema individuali. - Sterilizzare e precondizionare gli HD-MEA.
- Sotto il cofano, pulire il chip e l'anello di vetro con un fazzoletto inumidito con etanolo al 96% (EtOH), quindi posizionare ciascun dispositivo in una capsula di Petri sterile da 100 mm x 20 mm e riempire il serbatoio MEA con EtOH al 70% per 20 minuti.
- Aspirare l'EtOH e lavare il serbatoio con acqua sterile e filtrata per 3 volte. Aggiungere 1 mL di terreno di precondizionamento e incubare per una notte a 37 °C e 5% di CO2 .
NOTA: Il materiale di precondizionamento deve essere una soluzione a base di sale per rendere la superficie HD-MEA più idrofila. Ciò può includere terreni completi BrainPhys (BP) precedentemente preparati (non >3 mesi) (Tabelle 1C).
- Rivestire HD-MEA. Il giorno successivo, aspirare i terreni di precondizionamento. Aggiungere 1 mL di poli-dl-ornitina (PDLO) 0,1 mg/mL per rivestire l'intera area attiva. Incubare a 37 °C per una notte in incubatrice.
- Preparare e riscaldare i supporti su RT. I protocolli qui sfruttano neuroni iPSC umani funzionali provenienti da due fonti commerciali; Pertanto, i componenti dei supporti variano per ogni fornitore. Un protocollo è descritto in (Tabelle 1C, D).
- Aspirare il PDLO, lavare 3 volte con acqua e lasciare asciugare i trucioli sotto il cofano per 10 min.
- Riempire una capsula di Petri di 35 mm x 10 mm con acqua sterile filtrata e posizionarla accanto al chip per mantenere la giusta umidità ed evitare l'evaporazione delle cellule seminate nei passaggi successivi.
- Posizionare il chip HD-MEA sulla piattaforma di registrazione di acquisizione, riempire il serbatoio con PBS e testare il chip prima del rivestimento. Avvia il software Brainwave. Selezionare File > Nuova sessione di registrazione. Impostare i parametri di registrazione in modo che abbiano una frequenza di registrazione di 50 Hz e una frequenza di campionamento di 18 kHz/elettrodo. Modificare l'offset dell'amplificatore per calibrare il chip. Vedere la Tabella 2 per suggerimenti sulla risoluzione dei problemi.
- Placcatura e mantenimento di neuroni iPSC umani in HD-MEA (Figura 2B)
- Scongelare e diluire le cellule alle cellule desiderate per concentrazione di microlitro (cioè 1000 cellule/μL per ottenere una densità di 50.000 cellule in una goccia di 50 μL sull'HD-MEA) (Tabella 1C).
- Pipettare la sospensione cellulare sulla superficie dell'area attiva del truciolo utilizzando terreni di punteggiatura ad alto contenuto di laminina (Tabella 1D).
- Incubare a 37 °C con il 5% di CO2 per 45-60 min.
- Riempire delicatamente 2 mL di terreno nel serbatoio HD-MEA (Tabella 1C).
- Eseguire il cambio del 100% del terreno il giorno 1 (DIV1) dopo la semina utilizzando il terreno RT (Tabella 1C). Cambiare il 50% dei supporti ogni 3-4 giorni. Mantenere gli HD-MEA incubati a 37 °C con il 5% di CO2 per tutta la durata dell'esperimento.
NOTA: Pipettare delicatamente per evitare che le cellule si spostino. Controllare il colore del supporto per eventuali contaminazioni. L'intervallo e la quantità di sostituzione del terreno possono essere determinati dalle singole domande dello studio o dalle esigenze/specifiche delle cellule. - Opzionale: controllare l'andamento della crescita della coltura cellulare tra DIV4 e DIV8 con un microscopio verticale a contrasto di interferenza differenziale (DIC) dopo aver pulito il tavolino con EtOH al >70%.
3. Registrazioni neurali ex-vivo e in vitro su larga scala con HD-MEA
- Preparazione dell'area di lavoro per la registrazione delle sezioni cerebrali (Figura 2A)
- Mentre le fette di cervello si stanno riprendendo, posiziona gli strumenti necessari in ogni area di lavoro designata (vedi Tabella dei materiali).
NOTA: La configurazione del sistema principale deve essere ottimizzata e testata ben prima del giorno sperimentale della fetta di cervello. Il sistema di perfusione (linee di ingresso, linee di uscita della pompa, tubi e messa a terra) deve essere testato con PBS o aCSF e un HD-MEA sulla piattaforma di registrazione per garantire un segnale pulito, un maggiore rapporto segnale-rumore e l'assenza di rumore di perfusione. - Rivestire il chip HD-MEA con 0,1 mg/mL di PDLO per migliorare l'accoppiamento tessuto-chip e incubare a 37 °C per 20 min.
- Durante l'incubazione del chip, riempire il sistema di perfusione basato sulla gravità e le linee con la registrazione di aCSF. Garantire la carbogenazione continua del sistema di perfusione. Impostare una portata di 4,5 mL/min e una temperatura di 37 °C.
- Posizionare il chip HD-MEA sulla piattaforma di registrazione di acquisizione, riempire il serbatoio con aCSF, testare il sistema di perfusione e risolvere eventuali disturbi residui del sistema.
- Avvia il software Brainwave. Selezionare File > Nuova sessione di registrazione. Impostare i parametri di registrazione in modo che abbiano una frequenza di registrazione di 1 Hz e una frequenza di campionamento di 14 kHz/elettrodo. Modificare l'offset dell'amplificatore per calibrare il chip. Vedere la Tabella 2 per suggerimenti sulla risoluzione dei problemi.
NOTA: I parametri della frequenza di registrazione e della frequenza di campionamento dipendono dal tipo di dati e dai requisiti di sistema individuali.
- Avvia il software Brainwave. Selezionare File > Nuova sessione di registrazione. Impostare i parametri di registrazione in modo che abbiano una frequenza di registrazione di 1 Hz e una frequenza di campionamento di 14 kHz/elettrodo. Modificare l'offset dell'amplificatore per calibrare il chip. Vedere la Tabella 2 per suggerimenti sulla risoluzione dei problemi.
- Assicurarsi che l'area di registrazione sia buia attraverso un sistema di illuminazione della stanza o una gabbia ombreggiata sul tavolo ottico.
- Allineare lo stereomicroscopio con il serbatoio del chip HD-MEA e l'area attiva per l'acquisizione delle immagini.
- Posizionare l'ancora nel serbatoio dei trucioli per equilibrare.
NOTA: Anchor è un'arpa in platino realizzata su misura con fili minimi per favorire l'ossigenazione; tuttavia, sono disponibili alcuni commerciali. - Aggiungere composti farmacologici alle provette di perfusione appropriate.
NOTA: In questo protocollo, sono state ottenute registrazioni farmacologicamente indotte sia spontanee che da 100 μM di 4-aminopiridina (4-AP) come descritto in precedenza. I composti farmacologici possono essere adattati alla specifica domanda di studio. - Nell'area di lavoro per la preparazione delle fette cerebrali, posizionare una nuova piastra di coltura in plastica da 90 mm in una capsula di Petri in vetro da 150 mm. Aggiungere aCSF e iniziare a carbogenare.
- Mentre le fette di cervello si stanno riprendendo, posiziona gli strumenti necessari in ogni area di lavoro designata (vedi Tabella dei materiali).
- Registrazioni a livello di circuito da slice HC e OB utilizzando HD-MEA
NOTA: L'accoppiamento della fetta deve essere eseguito il più rapidamente possibile per evitare una mancanza di ossigenazione della fetta. L'accoppiamento dovrebbe richiedere solo ~1 minuto dal posizionamento iniziale della fetta microsezionata sull'area attiva del chip all'avvio finale del sistema di perfusione.- Rimuovere la fetta dalla camera di recupero della fetta di cervello con una pipetta di vetro e posizionarla in un piatto di plastica da 90 mm con carbogenazione continua. Utilizzando uno strumento di microdissezione, isolare l'HC o l'OB dal tessuto cerebrale circostante.
- Spostare le fette acute isolate di HC o OB con una pipetta di vetro nel serbatoio HD-MEA. Allineare delicatamente la fetta sull'area attiva MEA con un pennello fine. Aspirare bene tutte le soluzioni dal chip HD-MEA con un sistema di aspirazione.
- Posiziona delicatamente l'ancora sopra la fetta usando una pinza.
NOTA: L'ancoraggio deve essere posizionato senza movimento di fetta per evitare la perdita di accoppiamento. - Aggiungere delicatamente la soluzione al serbatoio dei trucioli e avviare il sistema di perfusione.
NOTA: Garantire il flusso laminare dall'ingresso di perfusione e dall'uscita della pompa per parametri di registrazione ottimali. - Assicurarsi che l'area di registrazione sia adeguatamente oscurata attraverso il sistema di illuminazione della stanza o con una gabbia ombreggiata su un tavolo ottico.
- Lasciare acclimatare la fetta per 10 minuti prima di iniziare le registrazioni o un'ulteriore modulazione farmacologica.
- Avvia il software Brainwave. Selezionare File > Nuova sessione di registrazione. Impostare i parametri di registrazione in modo che abbiano una frequenza di registrazione di 1 Hz e una frequenza di campionamento di 14 kHz/elettrodo. Modificare l'offset dell'amplificatore per calibrare il chip.
NOTA: Come indicato in precedenza nella sezione 3.1.4.1, durante l'esecuzione dei test di sistema, assicurarsi di applicare gli stessi parametri di registrazione. - Premere Record per iniziare l'acquisizione con le condizioni sperimentali preimpostate.
- Subito dopo la registrazione finale, acquisisci l'immagine della luce della fetta di cervello acuta. Riportare la fetta nella camera di recupero della fetta, rimuovere con una spazzola l'eventuale materiale organico accoppiato al truciolo e continuare con la fetta successiva. Pulire gli HD-MEA come descritto nella sezione 3.4.
- Preparazione dell'area di lavoro di registrazione iPSC umana e registrazioni a livello di rete su HD-MEA (Figura 2B)
NOTA: Cambiare supporto il giorno prima della registrazione o immediatamente dopo la registrazione di iPSC umano (Tabella 1C). Negli studi che hanno utilizzato i neuroni funzionali, i terreni sono stati cambiati ogni 4 giorni e su 4, 8, 16 e 24 DIV i terreni sono stati cambiati immediatamente dopo le registrazioni iPSC.- Garantire un ambiente di lavoro sterile pulendo la piattaforma di acquisizione HD-MEA con >70% di EtOH.
- Posizionare delicatamente un tappo a base di polidimetilsilossano (PDMS) con riferimento sull'anello HD-MEA sotto il cofano. Spostare il chip HD-MEA nell'area di registrazione iPSC e collegare il chip HD-MEA alla piattaforma di acquisizione.
- Assicurarsi che l'area di registrazione sia adeguatamente oscurata attraverso un sistema di illuminazione della stanza o una gabbia ombreggiata su un tavolo ottico.
- Lasciare che il chip HD-MEA si equilibri per 10 minuti prima di iniziare le registrazioni o la modulazione farmacologica aggiuntiva.
- Avvia il software Brainwave. Selezionare File > Nuova sessione di registrazione. Impostare i parametri di registrazione in modo che abbiano una frequenza di registrazione di 50 Hz e una frequenza di campionamento di 18 kHz/elettrodo. Modificare l'offset dell'amplificatore per calibrare il chip.
NOTA: Come indicato in precedenza nella sezione 2.1.1, durante l'esecuzione di un test del sistema prima del rivestimento e della placcatura, assicurarsi di applicare gli stessi parametri di registrazione. - Registrare l'attività di attivazione spontanea o le risposte farmacologicamente indotte dalla rete iPSC umana in ogni giorno del piano sperimentale (ad esempio, 4, 8, 16, 24 DIV).
NOTA: Non lasciare che il chip rimanga all'esterno dell'incubatrice per >30 minuti per mantenere la temperatura e l'umidità stabili e prevenire shock termici alle cellule. - Incubare HD-MEA a 37 °C con il 5% di CO2 nel corso dell'esperimento.
- Dopo il completamento dell'esperimento, fissare la rete neuronale sui chip e colorare per ulteriori immagini ottiche o pulire direttamente gli HD-MEA, come descritto nel passaggio 3.4.
- Pulizia dei chip HD-MEA
- Dopo l'esperimento, scartare la soluzione secondo il corretto smaltimento dei rifiuti e risciacquare con acqua e acqua.
- Aggiungere il detersivo desiderato, pulire l'area attiva e l'intero serbatoio con un cotton fioc e gettare il detersivo. Rabboccare con il detersivo, incubare per 20 minuti, quindi gettare il detersivo.
- Risciacquare accuratamente con acqua di laboratorio. Quindi, risciacquare 3-4 volte con acqua e acqua.
- Utilizzare la pressione dell'aria per asciugare completamente il chip HD-MEA.
4. Analisi di registrazioni neurali su larga scala da HD-MEA
NOTA: Mentre il passaggio 4.1 è specifico del software Brainwave, il passaggio 4.2 può essere modificato in base al tipo di dispositivo HD-MEA disponibile in commercio di ciascun utente.
- Pre-elaborazione dei dati grezzi e rilevamento degli eventi
- Aprire un file di dati grezzi registrati (.brw) nel software Brainwave. Selezionare Analisi > Rilevamento LFP o Rilevamento picchi.
NOTA: Il rilevamento LFP utilizza il filtro IIR con un filtro Butterworth passa-basso del 4° ordine (1-100 Hz). Gli algoritmi di soglia rigida includono una soglia alta di 150 μV, una soglia bassa di -150 μV, una finestra di energia compresa tra 70-120 ms, un periodo refrattario di 10 ms e una durata massima dell'evento di 1 s. Il rilevamento di picchi singoli e MUA impiega il filtraggio IIR con un filtro Butterworth passa-alto del 4° ordine (300-3500 Hz). Viene applicato un algoritmo PTSD con un fattore di deviazione standard di 8, un periodo di vita di picco di 2 ms e un periodo refrattario di 1 ms. - Per le registrazioni dei circuiti HC e OB, aggiungere l'opzione Area di lavoro avanzata nel file degli eventi rilevati (.bxr) per importare l'immagine della luce strutturale catturata dallo stereomicroscopio. Quando si esaminano i circuiti HC su larga scala, creare strati strutturali contenenti il giro dentato (DG), l'ilo, Cornu Ammonis 1 (CA1), Cornu Ammonis 3 (CA3), la corteccia entorinale (EC) e la corteccia peririnale (PC). Quando si esaminano circuiti OB su larga scala, creare strati strutturali contenenti lo strato nervoso olfattivo (ONL), lo strato glomerulare (GL), lo strato plessiforme esterno (EPL), lo strato di cellule mitrali (MCL) e lo strato di cellule granulari (GCL). Considera l'EPL e l'MCL come lo strato di proiezione (PL), inclusa la corteccia olfattiva (OCx).
- Aprire un file di dati grezzi registrati (.brw) nel software Brainwave. Selezionare Analisi > Rilevamento LFP o Rilevamento picchi.
- Elaborazione dei dati con una pipeline computazionale Python personalizzata
- Riduzione del rumore
- Leggi il file .bxr usando uno script Pythonpersonalizzato 26,29,32 e il pacchetto python h5py 3.6.0.
- Estrai i treni di picchi relativi alle registrazioni di rete iPSC e i treni di eventi LFP relativi alle registrazioni del circuito di slice cerebrale HC e OB.
- Caratterizzare gli eventi con un numero totale di elettrodi attivi inferiore allo 0,1% o il 10% della media degli elettrodi attivi per evento medio o gli eventi rilevati che non rientrano in un intervallo di frequenza di accensione statisticamente ragionevole come eventi casuali e rimuoverli. Inoltre, applicare i valori di soglia dell'ampiezza e della durata dell'evento.
NOTA: Per l'intervallo di frequenza di sparo, vengono considerati 0,1-15 picchi/s e 0,1-60 eventi LFP/min. Questi sono esempi di valori di soglia di velocità utilizzati per i set di dati analizzati. Le soglie di frequenza, ampiezza e durata dipenderanno dai singoli dati. - Salvare i dati del treno di eventi risultanti con le informazioni spaziotemporali di accompagnamento in formato di file .npy.
- Rastergrammi
- Leggere i file .npy e .bxr degli eventi filtrati e generare un grafico raster utilizzando la funzione pyplot Matplotlib (https://matplotlib.org/3.5.3/api/_as_gen/matplotlib.pyplot.html).
- Inoltre, per le registrazioni di sezioni cerebrali con specificità di strato, ordinare e raggruppare gli ID degli elettrodi in base agli strati prodotti nel passaggio 4.1.2.
- Attività media di cottura
- Elabora i dati delle serie temporali dal file .bxr, calcolando la frequenza media di accensione di ciascun elettrodo (numero di eventi/tempo di registrazione).
- Costruire una matrice di dati in cui le righe e le colonne rappresentano le coordinate degli elettrodi nell'array HD-MEA 64 x 64, dove ogni valore della matrice indica la velocità media di accensione.
- Utilizza una libreria di plottaggio come imshow di Matplotlib o le funzioni della mappa di calore di Seaborn in Python.
- Utilizza la mappa dei colori "caldi" qui, creando una mappa di calore informativa che incapsula visivamente la distribuzione spaziale delle velocità medie di accensione attraverso l'array di elettrodi.
- Tracce di forme d'onda rappresentative
- Leggi i dati delle serie temporali dal file .brw e genera una traccia della forma d'onda utilizzando la funzione pyplot di Matplotlib. (https://matplotlib.org/3.5.3/api/_as_gen/matplotlib.pyplot.html).
- Immettere l'ID dell'elettrodo desiderato, l'intervallo di tempo e la banda di frequenza per una traccia della forma d'onda rappresentativa. Le bande di frequenza definite in queste analisi includono oscillazioni LFP a bassa frequenza (1-100 Hz) con bande di frequenza filtrate passa-δ, θ, β e γ; ondulazioni d'onda acuta (SWR) (140-220 Hz); e ad alta frequenza singola e MUA (300-3500 Hz). Le bande di frequenza δ, θ, β e γ sono rispettivamente 1-4 Hz, 5-12 Hz, 13-35 Hz e 35-100 Hz.
- Densità spettrale di potenza
- Leggere i dati delle serie temporali dal file .brw e calcolare i periodogrammi per discernere le frequenze dominanti alla base dell'attività oscillatoria all'interno di ciascuna serie temporale.
- Costruire spettrogrammi pseudo-colore della dinamica frequenza-tempo.
NOTA: Gli spettri sono calcolati utilizzando il metodo di Welch utilizzando la trasformata di Fourier veloce delle LFP registrate per stimare la densità di potenza spettrale41. - Immettere l'ID dell'elettrodo desiderato, l'intervallo di tempo e la banda di frequenza per una mappa di densità spettrale. Le bande di frequenza definite in queste analisi comprendono quelle descritte al punto 4.2.4.
- Connettività funzionale
- Per le registrazioni del circuito brain slice, seguire i passaggi 4.2.6.2-4.2.6.4.
- Leggi i dati delle serie temporali dal file .brw e calcola la covarianza incrociata tra coppie di elettrodi attivi nell'array 64 x 64 utilizzando il coefficiente di correlazione di Pearson (PCC)42.
- Adattare un modello autoregressivo vettoriale alla serie temporale utilizzando la causalità di Granger multivariata per quantificare l'influenza di una serie temporale su un'altra.
- Applicare la funzione di trasferimento diretto (DTF) per valutare il flusso di informazioni direzionali all'interno dei collegamenti correlati.
NOTA: La connettività funzionale nella rete multistrato viene stabilita impostando una soglia di valore di correlazione basata su quelli sopra la media e due deviazioni standard di tutti i valori di covarianza incrociata43,44. - Per la registrazione iPSC, seguire i passaggi 4.2.6.6-4.2.6.8.
- Leggere i dati dei treni di spike dal file .bxr e calcolare una matrice 64x64 dei coefficienti di correlazione PCC tra tutte le combinazioni di treni di spike binned utilizzando le funzioni di spike_train_correlation (https://elephant.readthedocs.io/en/v0.7.0/reference/spike_train_correlation.html).
NOTA: La connettività funzionale nella rete multilivello viene stabilita impostando una soglia di valore di correlazione basata su quelli sopra la media e due deviazioni standard di tutti i valori di covarianza incrociata. - Inoltre, implementare procedure di filtraggio dei filtri spazio-temporali (STF) e delle soglie di latenza dipendenti dalla distanza (DdLT) sulla matrice di connettività per eliminare le potenziali connessioni accoppiate che superano la velocità massima di propagazione (impostata a 400 mm/s)45.
- Estrarre i picchi negativi dalle matrici di correlazione incrociata risultanti con operazioni di filtraggio e soglie per identificare le connessioni inibitorie utilizzando l'algoritmo FNCCH (Filtered and Normalized Cross-Correlation Histogram)45.
- Trasformare ogni matrice di connettività in un file grafico dinamico (con estensione gexf).
- Mappe di connettività di rete
- Laboratorio open data nel programma Gephi versione 9.2 (https://gephi.org) per il grafico dinamico per tracciare intervalli di tempo specifici.
- Applicare Geo Layout nella finestra di layout per la mappatura spaziale.
- Posizionare i vincoli di parametro su Intervallo gradi (Degree Range ) e Spessore spigolo (Edge Weight ) per il confronto.
- Assegna il colore nodale, la dimensione del bordo e la dimensione del grado per una migliore visualizzazione.
- Riduzione del rumore
Risultati
Mappatura spazio-temporale multimodello ed estrazione di caratteristiche di attivazione oscillatoria
Per quantificare gli eventi LFP e spike a livello di rete emersi da insiemi neuronali dinamici, abbiamo studiato modelli di attivazione sincroni su larga scala nei circuiti HC e OB e nelle reti iPSC umane. I circuiti di slice cerebrale registrati dal passaggio 3.2 e le reti iPSC registrate dal passaggio 3.3 sono stati analizzati secondo i passaggi 4.1-4.2 del protocollo. In primo luogo, il rilevamento degli eventi e la riduzione del rumore sono stati eseguiti per tutti i set di dati registrati e risolti a livello regionale in base alle specifiche del circuito. Successivamente, sono state tracciate mappature spaziali topografiche pseudo-colore di LFP medie su larga scala e modelli di sparo a picco, rastergrammi di eventi rilevati e tracce rappresentative 5-s di forme d'onda filtrate (Figure 3 A-I). La mappatura topografica pseudo-colore dei modelli di LFP su larga scala e di frequenza di attivazione dei picchi è stata sovrapposta alle rispettive immagini ottiche catturate al microscopio di HC (Figura 3A), OB (Figura 3B) e rete neuronale iPSC umana (Figura 3C). Ciò consente lo studio di modelli e risposte oscillatorie basati su singoli circuiti e reti. I rastergrammi HC e OB contengono i conteggi degli eventi LFP rilevati ordinati sui livelli DG, Hilus, CA3, CA1, EC e PC del circuito HC e sui livelli ONL, OCx, GL, PL e GCL della rete OB in un intervallo di tempo di 60 secondi (Figure 3D,E). Il rastergramma iPSC umano visualizza gli eventi di picco rilevati sincroni della rete colturata interconnessa in un intervallo di tempo di 20 s (Figura 3G). Successivamente, le tracce rappresentative degli eventi 5s provenienti da siti di registrazione HD-MEA su larga scala mostrano una gamma di frequenze oscillatorie registrate nei circuiti HC (cioè elettrodo selezionato in CA3) (Figura 3G) e OB (cioè elettrodo selezionato in GL) (Figura 3H) e l'attività di spike bursting multiunità nella rete iPSC umana da quattro elettrodi attivi selezionati nell'array (Figura 3I). Questi segnali esemplari mostrano firme di biosegnali, tra cui oscillazioni LFP a bassa frequenza (1-100 Hz) con bande di frequenza filtrate passa-δ, θ, β e γ; ondulazioni d'onda acuta (SWR) (140-220 Hz); e ad alta frequenza singola e MUA (300-3500 Hz). Infine, l'analisi della densità spettrale di potenza (PSD) è stata impiegata per quantificare simultaneamente l'ampiezza di potenza di una specifica banda oscillatoria nel circuito HC e OB interconnesso registrato da HD-MEA (Figure 3J,K).
Connettoma funzionale multimodale a livello di rete
Per dedurre la connettività su larga scala delle reti neurali multistrato da modelli di attivazione simultanea di insiemi neuronali attivi contemporaneamente, la covarianza incrociata tra coppie di elettrodi attivi negli eventi rilevati è stata calcolata secondo il passaggio 4.2.6 del protocollo. In questo caso, il coefficiente di correlazione è stato ordinato in base agli strati nel circuito HC e OB o non ordinato nella rete iPSC e quindi memorizzato in una matrice simmetrica. I connettomi funzionali dei circuiti HC e OB sono stati generati applicando la causalità di Granger multivariata e la funzione di trasferimento diretto (DTF) per quantificare l'influenza di una serie temporale su un'altra e valutare il flusso di informazioni direzionali all'interno dei collegamenti correlati nelle reti distinte. La mappatura del connettoma di HC (Figura 4A) e OB (Figura 4B) e la visualizzazione della rete sono state eseguite utilizzando il programma Gephi versione 9.2 (https://gephi.org). Vincoli di parametri simili sono stati posti sui collegamenti funzionali per confrontare i circuiti di taglio cerebrale HC e OB e hanno illustrato 100 s della connettività funzionale degli eventi LFP rilevati. I nodi sono scalati in base all'intensità in gradi con il colore nodale che indica lo strato e il colore del collegamento che identifica le connessioni intra e interstrato. I connettomi funzionali delle reti iPSC umane sono stati generati applicando filtri spazio-temporali (STF) e soglie di latenza dipendenti dalla distanza (DdLT) per migliorare la selezione di collegamenti significativi e perfezionare l'identificazione di connessioni significative applicando l'analisi dell'istogramma di correlazione incrociata filtrata e normalizzata (FNCCH). Mappatura del connettoma delle reti iPSC umane sull'intera visualizzazione del chip HD-MEA (Figura 4C) eseguita utilizzando Gephi. Il colore nodale indica l'input eccitatorio o inibitorio, mentre il colore del collegamento identifica le connessioni.
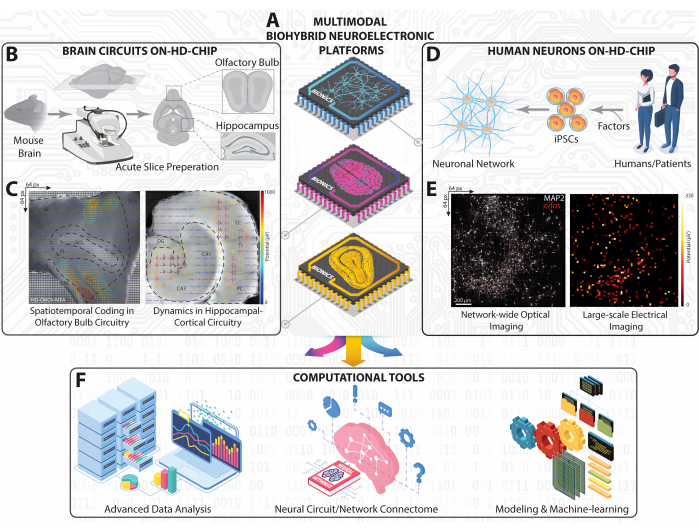
Figura 1: Panoramica della piattaforma sperimentale e computazionale su HD-MEA su larga scala. (A) Rappresentazione schematica isometrica delle nostre piattaforme neuroelettroniche bioibride multimodali realizzate con HD-MEA basate su CMOS per catturare le dinamiche neurali da circuiti e reti neuronali HC, OB e iPSC umane. (B) Flusso di lavoro schematico per lo slicing del cervello del topo e il suo workscape per ottenere fette HC e OB. (C) Rappresentazioni topografiche dei pattern di sparo su larga scala registrati simultaneamente dall'intera sezione HC e OB sovrapposta alle immagini ottiche della sezione con le forme d'onda extracellulari estratte dalla sezione. (D) Rappresentazione schematica della rete neuronale iPSC ottenuta dall'uomo. (E) Micrografie a fluorescenza che mostrano i c-fos cellulari e la MAP-2 somatica/dendritica dell'intera rete neuronale umana su chip HD-MEA (a sinistra) abbinata all'intera mappa media dell'attività di attivazione (a destra). (F) Framework computazionale che include analisi avanzata dei dati, mappatura della connettività e strumenti di apprendimento automatico AI per analizzare i dati neurali multidimensionali ottenuti da registrazioni su larga scala su HD-MEA. Fare clic qui per visualizzare una versione più grande di questa figura.

Figura 2: Layout per spazi di lavoro per la preparazione e la registrazione di colture di colture di iPSC umane ex-vivo e in vitro . (A) Flusso di lavoro schematico che illustra la configurazione per la preparazione delle sezioni HC e OB, con gli strumenti e le attrezzature necessarie in ogni area di lavoro. (B) Rappresentazione schematica per la preparazione di colture di iPSC umane, inclusi gli strumenti e i dispositivi necessari. Un elenco completo dei materiali è incluso nei passaggi 1.2.2, 2.1, 2.2, 3.1.1, 3.3 e nell'Indice dei materiali. Fare clic qui per visualizzare una versione più grande di questa figura.
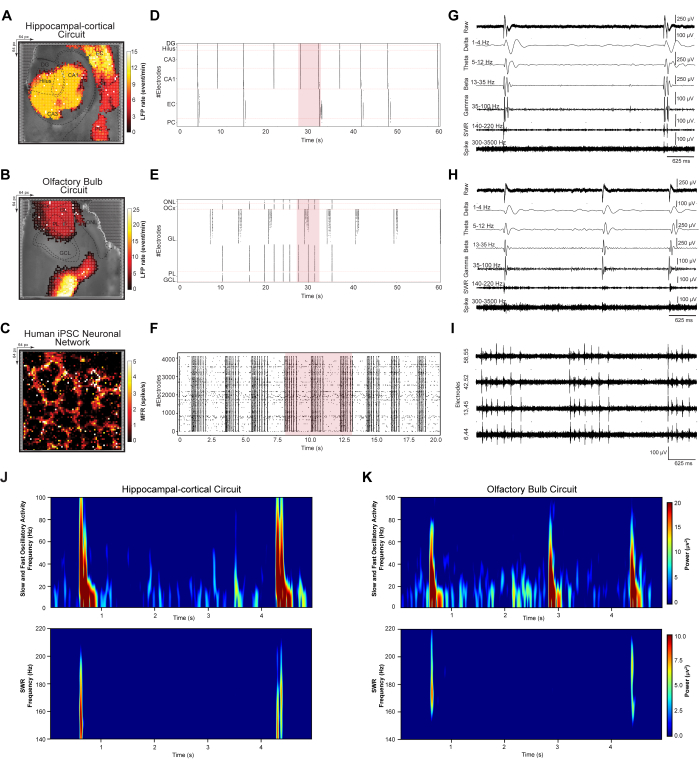
Figura 3: Mappatura ed estrazione di modelli spazio-temporali delle dinamiche di rete. (A-C) Mappe spaziali LFP medie e spike rate, calcolate su registrazioni di cinque minuti, sovrapposte all'immagine di luce del microscopio. (D-F) Grafici raster che mostrano gli eventi LFP rilevati e denoised in un sottocampione di dati di 60 secondi e i picchi in un sottocampione di dati di 20 secondi. (G-I) Estrazione di tracce di forme d'onda rappresentative da un segmento di 5 secondi del sottocampione di dati del grafico raster (evidenziato in rosso nel grafico raster), visualizzato come bande oscillatorie LFP grezze (1-100 Hz); δ (1-4 Hz), θ (5-12 Hz), β (13-35 Hz) e γ (35-100 Hz); SWR (140-220 Hz); e picchi singoli e MUA ad alta frequenza (300-3500 Hz). (J,K) Mappe di densità spettrale di potenza di LFP oscillatorie veloci e lente (1-100 Hz) e SWR (140-220 Hz). Fare clic qui per visualizzare una versione più grande di questa figura.
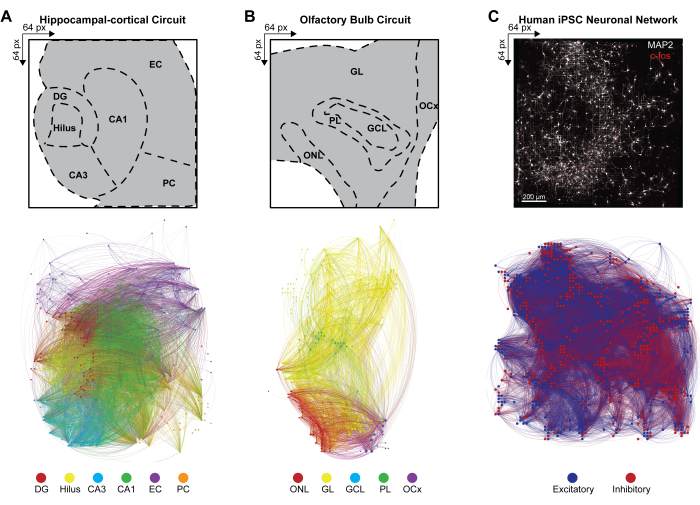
Figura 4: Organizzazione di connettomi funzionali multimodali a livello di rete. (A-C) Mappe Gephi che illustrano la connettività funzionale nodale, in cui i nodi corrispondono a una delle legende della barra dei colori di esempio (sotto), mentre i collegamenti (o bordi) sono ombreggiati in modo che corrispondano ai nodi di connessione. Le legende di esempio per i layer (A) HC, (B) OB e (C) iPSC vengono visualizzate su un array 64 x 64. I layer HC e OB vengono tracciati su un intervallo di tempo di 100 secondi per ridurre efficacemente il numero di nodi e collegamenti visibili a scopo di visualizzazione. Fare clic qui per visualizzare una versione più grande di questa figura.
Tabella 1: Soluzioni per la preparazione di fette cerebrali e terreni per colture neuronali iPSC. (A) Soluzione da taglio ad alto contenuto di saccarosio per la preparazione di fette cerebrali ex-vivo. (B) soluzione di registrazione aCSF per la preparazione e la registrazione di fette cerebrali ex-vivo. (C-D) Protocollo dei terreni iPSC neuronali umani, dove (C) è il terreno completo BrainPhys utilizzato per lo scongelamento cellulare, il rivestimento del chip HD-MEA e il mantenimento dell'HD-MEA in coltura, e (D) il terreno di punteggiatura utilizzato per la placcatura cellulare HD-MEA. Clicca qui per scaricare questa tabella.
Tabella 2: Risoluzione dei problemi comuni di acquisizione delle registrazioni HD-MEA. Un elenco di problemi comuni, le loro potenziali cause e soluzioni per la risoluzione dei problemi relativi ai chip HD-MEA, alla piattaforma di registrazione, al rumore di sistema e al software. Clicca qui per scaricare questa tabella.
Discussione
Le intricate dinamiche dell'attività neuronale spazio-temporale, che emergono da insiemi neuronali interconnessi, sono state a lungo oggetto di intrighi nelle neuroscienze. Le metodologie tradizionali, come il patch-clamp, la MEA standard e l'imaging Ca2+, hanno fornito preziose informazioni sulla complessità del cervello. Tuttavia, spesso non sono in grado di catturare le dinamiche computazionali complete a livello di rete 21,22,23. Il protocollo tecnico della piattaforma HD-MEA, come dettagliato in questo studio JoVE, rappresenta un significativo balzo in avanti, offrendo una visione panoramica delle dinamiche neurali attraverso diverse modalità, dagli assemblaggi cellulari alle reti espansive (ad esempio, fette di cervello di topo acute ex-vivo e reti iPSC umane in vitro)26,29,30,32.
Fette di cervello di topo acute, ex-vivo, sono state uno strumento fondamentale nella ricerca neuronale, facilitando le indagini a livello molecolare e circuitale 6,7. Tuttavia, la sfida di mantenere la vitalità dei tessuti è stata un collo di bottiglia persistente. Il protocollo delineato in questo studio introduce modifiche critiche per ottimizzare la qualità e la longevità di queste fette per sfruttarne i vantaggi sulla piattaforma HD-MEA. Questo protocollo sottolinea l'importanza di: i) Raggiungere l'uniformità delle fette, per le quali l'uso di un vibratomo è preferito rispetto a un tritatutto per tessuti grazie alla sua precisione e alla riduzione al minimo del danno tissutale, nonostante il compromesso di tempi di affettatura più lunghi. ii) Garantire una carbogenazione costante durante tutto il processo, dall'estrazione alla registrazione, per mantenere la vitalità dei tessuti. iii) Regolare la temperatura e consentire un adeguato tempo di recupero prima della registrazione. iv) Utilizzo di un blocco o di uno stampo di agarosio per stabilizzare il cervello, prevenire lo strappo e ridurre al minimo il contatto con la colla. v) Mantenere portate ottimali di aCSF carbogenato all'interno del serbatoio HD-MEA per garantire la salute della fetta evitando problemi come il disaccoppiamento, il rumore e la deriva (Tabella 2).
Sia per le fette di cervello di topo che per le preparazioni di iPSC umane, il miglioramento dell'accoppiamento dell'interfaccia elettrodo-tessuto è fondamentale 30,46,47. Il nostro protocollo sottolinea l'importanza di utilizzare la molecola che promuove l'adesione Poli-dl-ornitina (PDLO). Questa molecola non solo aumenta la superficie per il rilevamento dei segnali elettrici, ma aumenta anche la conduttività elettrica46. In questo modo, promuove l'adesione cellulare, la crescita e lo sviluppo di proprietà funzionali della rete. Tale ottimizzazione svolge un ruolo fondamentale nel migliorare l'efficacia della piattaforma HD-MEA. Questo, a sua volta, garantisce un'analisi accurata e coerente dei connettomi ex-vivo e in vitro su microscala e delle loro sequenze di sparo spazio-temporali. In particolare, è stato dimostrato che il PDLO supera altri substrati come la polietilenimmina (PEI) e la poli-l-ornitina (PLO) nel promuovere l'attività di attivazione spontanea e la risposta agli stimoli elettrici nelle colture neuronali. Inoltre, PDLO è stato utilizzato per la funzionalizzazione superficiale sull'HD-MEA e ha dimostrato di migliorare l'interfaccia di accoppiamento elettrodo-slice e di aumentare il rapporto segnale-rumore in entrambe le fette OB e HC26,29. L'aggiunta di un ancoraggio in platino costruito su misura aumenta ulteriormente l'accoppiamento dell'interfaccia elettrodo-fetta, portando a registrazioni con un rapporto segnale/rumore più elevato.
L'utilizzo di HD-MEA sia per fette di cervello di topo ex-vivo che per reti iPSC umane in vitro introduce un metodo abile nell'esplorazione di dinamiche estese, multiscala e multimodali. Questo approccio innovativo, tuttavia, comporta notevoli sfide, soprattutto nella gestione dei dati 48,49,50,51. Una singola registrazione HD-MEA acquisita a 18 kHz/frequenza di campionamento dell'elettrodo genera ben 155 MB/s di dati. Il volume di dati aumenta rapidamente quando si tiene conto di più sezioni, condizioni farmacologiche diverse o periodi di registrazione prolungati. Un tale afflusso di informazioni richiede solide infrastrutture di archiviazione e strumenti computazionali avanzati per un'elaborazione semplificata. La capacità della piattaforma HD-MEA di raccogliere simultaneamente dati da migliaia di insiemi neuronali è sia un vantaggio che un ostacolo. Fornisce informazioni supreme sulle dinamiche computazionali delle funzioni cerebrali, ma richiede anche un quadro analitico raffinato. In questo protocollo JoVE, abbiamo fornito esempi di strategie computazionali, tra cui il rilevamento di eventi su larga scala, la classificazione, la teoria dei grafi, l'analisi della frequenza e l'apprendimento automatico. Questi metodi sottolineano gli intensi sforzi compiuti per affrontare le sfide dell'analisi di dati neurali complessi. Ciononostante, c'è ancora molto spazio per lo sviluppo di strumenti computazionali più avanzati per analizzare questi set di dati neurali multidimensionali. Armato degli strumenti e delle metodologie appropriate, il potenziale della piattaforma HD-MEA viene amplificato, offrendo approfondimenti sulla complessità delle funzioni cerebrali sia in condizioni sane che patologiche.
In sostanza, la piattaforma HD-MEA, se integrata con i protocolli dettagliati e gli strumenti computazionali discussi, offre un approccio trasformativo alla comprensione dell'intricato funzionamento del cervello. Catturando dinamiche su larga scala, multiscala e multimodali, fornisce informazioni preziose su processi come l'apprendimento, la memoria e l'elaborazione delle informazioni. Inoltre, la sua applicazione nelle reti iPSC umane in vitro ha il potenziale per rivoluzionare lo screening dei farmaci e la medicina personalizzata. Tuttavia, sebbene questa piattaforma rappresenti un progresso significativo nella ricerca neuroscientifica, è fondamentale riconoscere e affrontare le sfide tecniche intrinseche. Con il continuo perfezionamento e l'integrazione di strumenti computazionali avanzati, la piattaforma HD-MEA è pronta a inaugurare una nuova era di strumenti diagnostici precisi, l'identificazione di biomarcatori specifici e terapie mirate per i disturbi neurologici.
Divulgazioni
Gli autori dichiarano di non avere interessi concorrenti o finanziari.
Riconoscimenti
Questo studio è stato sostenuto da fondi istituzionali (DZNE), dall'Associazione Helmholtz nell'ambito del Fondo di convalida Helmholtz (HVF-0102) e dalla Scuola di specializzazione internazionale per la biomedicina e la bioingegneria di Dresda (DIGS-BB). Vorremmo anche ringraziare la piattaforma per i test comportamentali sugli animali presso la DZNE-Dresden (Alexander Garthe, Anne Karasinsky, Sandra Günther e Jens Bergmann) per il loro supporto. Vorremmo riconoscere che una parte della Figura 1 è stata creata utilizzando la piattaforma BioRender.com.
Materiali
| Name | Company | Catalog Number | Comments |
| 150 mm Glass Petri Dish | generic | generic | Brain Preparation Workspace, Brain Slice Recording Workspace |
| 0.22 μm Sterile Filter Unit | Assorted | Assorted | Assorted |
| 90 mm Plastic Culture Dish | TPP | 93100 | Brain Preparation Workspace, Brain Slice Recording Workspace |
| Agarose | Roth | 6351.5 | Brain Preparation Workspace |
| Agarose Mold | CUSTOM | CUSTOM | Brain Preparation Workspace; Custom designed 3D Printer Design, available upon request |
| Aluminum Foil | generic | generic | Brain Extraction Workspace |
| Anesthesia chamber | generic | generic | Brain Extraction Workspace; Assorted Beaker, Bedding etc |
| Ascorbic Acid | Sigma Aldrich | A4544-25G | Solution Preparation Workspace |
| Assorted Beakers | generic | generic | Solution Preparation Workspace; 50 mL |
| Assorted Luers | Cole Parmer | 45511-00 | Brain Slice Recording Workspace |
| Assorted Volumetric flasks | generic | generic | Solution Preparation Workspace; 500 mL, 1 L |
| B27 Supplement | Life Technologies | 17504-044 | BrainXell Commercial Supplier Protocol |
| BDNF | Peprotech | 450-02 | BrainXell Commercial Supplier Protocol |
| Biological Safety Cabinet with UV Lamp | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| BrainPhys Neuronal Medium | STEMCELL Technologies | 05790 | CDI, and BrainXell Commerical Supplier Protocol |
| Brainwave Software | 3Brain AG | Version 4 | Brain Slice and Human iPSC Recording Workspace |
| BrainXell Glutamatergic Neuron Assay | BrainXell | BX-0300 | BrainXell Commercial Supplier Protocol |
| CaCl2 | Sigma Aldrich | 21115-100ML | Solution Preparation Workspace |
| Carbogen | generic | generic | All Workspaces; 95%/5% O2 and CO2 mixture |
| Cell Culture Incubator | Assorted | Assorted | Assorted |
| CMOS-based HD-MEA chip | 3Brain AG | CUSTOM | Brain Slice and Human iPSC Recording Workspace |
| Conical Tubes, 50 mL, Falcon (Centrifuge Tubes) | STEMCELL Technologies | 38010 | CDI Commerical Supplier Protocol |
| Crocodile Clip Grounding Cables | JWQIDI | B06WGZG17W | Brain Slice Recording Workspace |
| Curved Forceps | FST | 11052-10 | Brain Extraction Workspace |
| DMEM/F12 Medium | Life Technologies | 11330-032 | BrainXell Commercial Supplier Protocol |
| Dulbecco’s Phosphate Buffered Saline without Ca2+ and Mg2+ (D-PBS) | STEMCELL Technologies | 37350 | CDI Commerical Supplier Protocol |
| Filter Paper | Macherey-Nagel | 531 011 | Brain Preparation Workspace |
| Fine Brush | Leonhardy | 773 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Forceps | VITLAB | 67895 | Brain Slice Recording Workspace |
| GDNF | Peprotech | 450-10 | BrainXell Commercial Supplier Protocol |
| Geltrex | Life Technologies | A1413201 | BrainXell Commercial Supplier Protocol |
| Glass pasteur pipette | Roth | 4518 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Glucose | Sigma Aldrich | G7021-1KG | Solution Preparation Workspace |
| GlutaMAX | Life Technologies | 35050-061 | BrainXell Commercial Supplier Protocol |
| Gravity-based Perfusion System | ALA | VC3-8xG | Brain Slice Recording Workspace |
| HD-MEA Recording platform | 3Brain AG | CUSTOM | Brain Slice and Human iPSC Recording Workspace |
| Heater | Warner Instruments | TC-324C | Brain Slice Recording Workspace |
| Hemocytometer or Automated Cell Counter | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| Hypo Needles | Warner Instruments | 641489 | Brain Slice Recording Workspace |
| iCell GlutaNeurons Kit, 01279 | CDI | R1061 | CDI Commerical Supplier Protocol |
| Iris Scissors | Vantage | V95-304 | Brain Extraction Workspace |
| Isoflurane | Baxter | HDG9623 | Brain Extraction Workspace |
| KCl | Sigma Aldrich | P5405-250G | Solution Preparation Workspace |
| Laminin | Sigma-Aldrich | L2020 | CDI Commerical Supplier Protocol |
| Liquid Nitrogen Storage Unit | Assorted | Assorted | HD-MEA Coating, Plating, Mainainance Workspace |
| Magnetic Stirrer | generic | generic | Solution Preparation Workspace |
| Metal Screws | Thorlabs | HW-KIT2/M | Brain Slice Recording Workspace |
| MgCl2 | Sigma Aldrich | M1028-100ML | Solution Preparation Workspace |
| MgSO4 | Sigma Aldrich | 63138-250G | Solution Preparation Workspace |
| Microdissection Tool Holder | Braun | 4606108V | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Microdissection Tool Needle | Braun | 9186166 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Modular Stereomicroscope | Leica | CUSTOM | Brain Slice Recording Workspace; custom specifications and modifications |
| N2 Supplement | Life Technologies | 17502-048 | CDI, and BrainXell Commercial Supplier Protocol |
| NaCl | Sigma Aldrich | S3014-1KG | Solution Preparation Workspace |
| NaH2PO4 | Sigma Aldrich | S0751-100G | Solution Preparation Workspace |
| NaHCO3 | Sigma Aldrich | S5761-500G | Solution Preparation Workspace |
| Neurobasal Medium | Life Technologies | 21103-049 | BrainXell Commercial Supplier Protocol |
| Optical Cage System | Thorlabs | Assorted | Brain Slice Recording Workspace |
| Optical Table w/Breadboard | Thorlabs | SDA7590 | Brain Slice Recording Workspace |
| PDLO | Sigma Aldrich | P0671 | HD-MEA Coating, Brain Slice Recording Workspace |
| Penicillin-streptomycin, 100x | Thermo Fisher Scientific | 15140-122 | CDI Commerical Supplier Protocol |
| Pipette tips | TipONE | S1120-8810 | Brain Slice Recording Workspace |
| Pipettors | Assorted | Assorted | Assorted |
| Platinum Anchor | CUSTOM | CUSTOM | Brain Slice Recording Workspace |
| Polyethylene Tubing | Assorted | Assorted | Brain Slice Recording Workspace |
| Pump | MasterFlex | 78018-22 | Brain Slice Recording Workspace |
| Razor Blade | Apollo | 10179960 | Brain Preparation Workspace |
| Reference Electrode Cell Culture Cap | CUSTOM | CUSTOM | Human iPSC Recording Workspace; Custom designed 3D Printer Design, available upon request |
| Rubber Pipette Bulb | Duran Wheaton Kimble | 292000205 | Brain Slice Preparation Workspace, Brain Slice Recording Workspace |
| Serological Pipettes, 1 mL, 2 mL, 5 mL, 10 mL, 25 mL | Assorted | Assorted | Assorted |
| Slice Recovery Chamber | CUSTOM | CUSTOM | Brain Slice Recovery Workspace; Custom designed 3D Printer Design, available upon request |
| Spatula | ISOLAB | 047.06.150 | Brain Preparation Workspace |
| Sucrose | Sigma Aldrich | 84100-1KG | Solution Preparation Workspace |
| Super Glue | UHU | 358221 | Brain Slice Preparation Workspace |
| Surgical Scissors | Peters Instruments | BC 344 | Brain Extraction Workspace |
| Tabletop Centrifuge | Assorted | Assorted | Assorted |
| TGF-β1 | Peprotech | 100-21C | BrainXell Commercial Supplier Protocol |
| Tissue Paper | generic | generic | Brain Extraction Workspace |
| Trypan Blue | STEMCELL Technologies | 07050 | CDI Commerical Supplier Protocol |
| Upright Microscope | Olympus | CUSTOM | Imaging Workspace; Custom specifications and modifications |
| Vacusip | Integra | 159010 | Brain Slice Recording Workspace |
| Vibratome | Leica | VT1200s | Brain Slice Preparation Workspace; Includes: Specimen plate, buffer tray, ice tray, specimen plate holding tool, vibratome blade adjusting tool |
| Vibratome Blade | Personna | N/A | Brain Slice Preparation Workspace |
| Water Bath | Lauda | L000595 | Brain Slice Recovery Workspace |
Riferimenti
- Hebb, D. O. . The Organization of Behavior; A Neuropsychological Theory. , (1949).
- Cossart, R., Garel, S. Step by step: cells with multiple functions in cortical circuit assembly. Nat Rev Neurosci. 23, 395-410 (2022).
- Carrillo-Reid, L., Yuste, R. Playing the piano with the cortex: role of neuronal ensembles and pattern completion in perception and behavior. Curr Opin Neurobiol. 64, 89-95 (2020).
- Buzsáki, G. Large-scale recording of neuronal ensembles. Nat Neurosci. 7, 446-451 (2004).
- Buzsáki, G. Neural Syntax: Cell assemblies, synapsembles, and readers. Neuron. 68 (3), 362-385 (2010).
- Huang, Y., Williams, J. C., Johnson, S. M. Brain slice on a chip: opportunities and challenges of applying microfluidic technology to intact tissues. Lab Chip. 12 (12), 2103-2117 (2012).
- Cho, S., Wood, A., Bowlby, M. Brain slices as models for neurodegenerative disease and screening platforms to identify novel therapeutics. Curr Neuropharmacol. 5 (1), 19-33 (2007).
- Bliss, T. V. P., Collingridge, G. L. A synaptic model of memory: long-term potentiation in the hippocampus. Nature. 361, 31-39 (1993).
- Anderson, P., Morris, R., Amaral, D., Bliss, T., O'Keefe, L. . The Hippocampus Book. , (2006).
- Lisman, J., et al. Viewpoints: how the hippocampus contributes to memory, navigation and cognition. Nat Neurosci. 20, 1434-1447 (2017).
- Mori, K., Nagao, H., Yoshihara, Y. The olfactory bulb: Coding and processing of odor molecule information. Science. 286 (5440), 711-715 (1999).
- Buck, L., Axel, R. A novel multigene family may encode odorant receptors: A molecular basis for odor recognition. Cell. 65 (1), 175-187 (1991).
- Bushdid, C., Magnasco, M. O., Vosshall, L. B., Keller, A. Humans can discriminate more than 1 trillion olfactory stimuli. Science. 343 (6177), 1370-1372 (2014).
- Kempermann, G. Why new neurons? Possible functions for adult hippocampal neurogenesis. J Neurosci. 23 (3), 635-638 (2003).
- Aimone, J. B., Wiles, J., Gage, F. H. Computational influence of adult neurogenesis on memory encoding. Neuron. 61 (2), 187-202 (2009).
- Nithianantharajah, J., Hannan, A. J. Enriched environments, experience-dependent plasticity and disorders of the nervous system. Nat Rev Neurosci. 7, 697-709 (2006).
- Takahashi, K., et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors. Cell. 131 (5), 861-872 (2007).
- Espuny-Camacho, I., et al. Pyramidal neurons derived from human pluripotent stem cells integrate efficiently into mouse brain circuits in vivo. Neuron. 77 (3), 440-456 (2013).
- Rajamohan, D., et al. Current status of drug screening and disease modelling in human pluripotent stem cells. Bioessays. 35 (3), 281-298 (2013).
- Heilker, R., Traub, S., Reinhardt, P., Schöler, H. R., Sterneckert, J. iPS cell derived neuronal cells for drug discovery. Trends Pharmacol Sci. 35 (10), 510-519 (2014).
- Zhao, S. R., Mondéjar-Parreño, G., Li, D., Shen, M., Wu, J. C. Technical applications of microelectrode array and patch clamp recordings on human induced pluripotent stem cell-derived cardiomyocytes. J Vis Exp. (186), e64265 (2022).
- Hamill, O. P., McBride, D. W. Induced membrane hypo/hyper-mechanosensitivity: A limitation of patch-clamp recording. Annu Rev Physiol. 59, 621-631 (1997).
- Manz, K. M., Siemann, J. K., McMahon, D. G., Grueter, B. A. Patch-clamp and multi-electrode array electrophysiological analysis in acute mouse brain slices. STAR Protoc. 2 (2), 100442 (2021).
- Lee, C. H., Park, Y. K., Lee, K. Recent strategies for neural dynamics observation at a larger scale and wider scope. Biosens Bioelectron. 240, 115638 (2023).
- Urai, A. E., Doiron, B., Leifer, A. M., Churchland, A. K. Large-scale neural recordings call for new insights to link brain and behavior. Nat Neurosci. 25 (1), 11-19 (2022).
- Hu, X., Khanzada, S., Klütsch, D., Calegari, F., Amin, H. Implementation of biohybrid olfactory bulb on a high-density CMOS-chip to reveal large-scale spatiotemporal circuit information. Biosens Bioelectron. 198, 113834 (2022).
- Amin, H., Marinaro, F., Tonelli, D. D. P., Berdondini, L. Developmental excitatory-to-inhibitory GABA-polarity switch is disrupted in 22q11.2 deletion syndrome: A potential target for clinical therapeutics. Sci Rep. 7 (1), 15752 (2017).
- Amin, H., Nieus, T., Lonardoni, D., Maccione, A., Berdondini, L. High-resolution bioelectrical imaging of Aβ-induced network dysfunction on CMOS-MEAs for neurotoxicity and rescue studies. Sci Rep. 7 (1), 2460 (2017).
- Emery, B. A., Hu, X., Khanzada, S., Kempermann, G., Amin, H. High-resolution CMOS-based biosensor for assessing hippocampal circuit dynamics in experience-dependent plasticity. Biosens Bioelectron. 237, 115471 (2023).
- Amin, H., et al. Electrical responses and spontaneous activity of human iPS-derived neuronal networks characterized for 3-month culture with 4096-electrode arrays. Front Neurosci. 10, 121 (2016).
- Lonardoni, D., et al. Recurrently connected and localized neuronal communities initiate coordinated spontaneous activity in neuronal networks. PLoS Comput Biol. 13 (7), e1005672 (2017).
- Emery, B. A., et al. Large-scale multimodal recordings on a high-density neurochip: Olfactory bulb and hippocampal networks. 2022 44th Annual International Conference of the IEEE Engineering in Medicine & Biology Society (EMBC). , 3111-3114 (2022).
- Rossi, L., Emery, B. A., Khanzada, S., Hu, X., Amin, H. Pharmacologically and electrically-induced network-wide activation of olfactory bulb with large-scale biosensor. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Emery, B. A., et al. Recording network-based synaptic transmission and LTP in the hippocampal network on a large-scale biosensor. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Hierlemann, A., Frey, U., Hafizovic, S., Heer, F. Growing cells atop microelectronic chips: Interfacing electrogenic cells in vitro with CMOS-based microelectrode arrays). Proceedings of the IEEE. 99 (2), 252-284 (2011).
- Berdondini, L., et al. Active pixel sensor array for high spatio-temporal resolution electrophysiological recordings from single cell to large scale neuronal networks. Lab Chip. 9, 2644-2651 (2009).
- Siegle, J. H., Hale, G. J., Newman, J. P., Voigts, J. Neural ensemble communities: open-source approaches to hardware for large-scale electrophysiology. Curr Opin Neurobiol. 32, 53-59 (2015).
- Amin, H., Maccione, A., Zordan, S., Nieus, T., Berdondini, L. High-density MEAs reveal lognormal firing patterns in neuronal networks for short and long term recordings. 2015 7th International IEEE/EMBS Conference on Neural Engineering (NER). , 1000-1003 (2015).
- Altuntac, E., et al. Bottom-up neurogenic-inspired computational model. 2023 IEEE BioSensors Conference (BioSensors). , 1-4 (2023).
- Maccione, A., et al. A novel algorithm for precise identification of spikes in extracellularly recorded neuronal signals. J Neurosci Methods. 177 (1), 241-249 (2009).
- Welch, P. D. The use of fast Fourier transform for the estimation of power spectra: A method based on time averaging over short, modified periodograms. IEEE Transactions on Audio and Electroacoustics. 15 (2), 70-73 (1967).
- Eggermont, J. J., Munguia, R., Pienkowski, M., Shaw, G. Comparison of LFP-based and spike-based spectro-temporal receptive fields and cross-correlation in cat primary auditory cortex. PLoS One. 6 (5), e20046 (2011).
- Damos, P. Using multivariate cross correlations, Granger causality and graphical models to quantify spatiotemporal synchronization and causality between pest populations. BMC Ecol. 16, 33 (2016).
- Kaminski, M. J., Blinowska, K. J. A new method of the description of the information flow in the brain structures. Biol Cybern. 65, 203-210 (1991).
- Pastore, V. P., Massobrio, P., Godjoski, A., Martinoia, S. Identification of excitatory-inhibitory links and network topology in large-scale neuronal assemblies from multi-electrode recordings. PLoS Comput Biol. 14 (8), e1006381 (2018).
- Amin, H., Dipalo, M., De Angelis, F., Berdondini, L. Biofunctionalized 3D nanopillar arrays fostering cell guidance and promoting synapse stability and neuronal activity in networks. ACS Appl Mater Interfaces. 10 (17), 15207-15215 (2018).
- Woeppel, K., Yang, Q., Cui, X. T. Recent advances in neural electrode-tissue interfaces. Curr Opin Biomed Eng. 4, 21-31 (2017).
- Steinmetz, N. A., Koch, C., Harris, K. D., Carandini, M. Challenges and opportunities for large-scale electrophysiology with Neuropixels probes. Curr Opin Neurobiol. 50, 92-100 (2018).
- Siegle, J. H., Hale, G. J., Newman, J. P., Voigts, J. Neural ensemble communities: open-source approaches to hardware for large-scale electrophysiology. Curr Opin Neurobiol. 32, 53-59 (2015).
- Freeman, J. Open source tools for large-scale neuroscience. Curr Opin Neurobiol. 32, 156-163 (2015).
- Stevenson, I. H., Kording, K. P. How advances in neural recording affect data analysis. Nat Neurosci. 14 (2), 139-142 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon