Method Article
Enfoque microfluídico para resolver la secreción simultánea y secuencial de citocinas de células polifuncionales individuales
* Estos autores han contribuido por igual
En este artículo
Resumen
El protocolo describe una plataforma microfluídica avanzada para medir cuantitativamente la dinámica de secreción de citocinas de células mononucleares individuales de sangre periférica humana. La plataforma mide hasta tres citocinas en paralelo (IL-6, TNFα e IL-1β) para cada célula individual estimulada con lipopolisacárido como ejemplo.
Resumen
Las infecciones, las enfermedades autoinmunes y las respuestas inmunológicas deseadas y adversas al tratamiento pueden dar lugar a una respuesta compleja y dinámica de citocinas in vivo. Esta respuesta implica que numerosas células inmunitarias secretan varias citocinas para orquestar la reacción inmunitaria. Sin embargo, la dinámica de secreción, las cantidades y la co-ocurrencia de las diferentes citocinas por parte de varios subtipos de células siguen siendo poco conocidas debido a la falta de herramientas adecuadas para estudiarlas. Aquí, describimos un protocolo utilizando una plataforma de gotas microfluídicas que permite la medición cuantitativa resuelta en el tiempo de la dinámica de secreción de varias citocinas en paralelo a nivel de una sola célula. Esto es posible gracias a la encapsulación de células individuales en gotas microfluídicas junto con un inmunoensayo multiplexado para la cuantificación paralela de las concentraciones de citocinas, su inmovilización para la obtención de imágenes fluorescentes dinámicas y el análisis de las imágenes respectivas para derivar las cantidades y la dinámica secretadas. El protocolo describe la preparación de nanopartículas magnéticas funcionalizadas, experimentos de calibración, preparación celular y la encapsulación de las células y nanopartículas en gotas para imágenes fluorescentes y posterior análisis de imágenes y datos utilizando el ejemplo de células mononucleares de sangre periférica humana estimuladas por lipopolisacáridos. La plataforma presentada identificó un comportamiento distinto de secreción de citocinas para células individuales y cosecretoras, caracterizando la heterogeneidad fenotípica esperada en la muestra celular medida. Además, la naturaleza modular del ensayo permite su adaptación y aplicación para estudiar una variedad de proteínas, citocinas y muestras de células, lo que podría conducir a una comprensión más profunda de la interacción entre los diferentes tipos de células inmunitarias y el papel de las diferentes citocinas secretadas dinámicamente para dar forma a la respuesta inmunitaria estrictamente regulada. Estos nuevos conocimientos podrían ser particularmente interesantes en los estudios de desregulaciones inmunitarias o en la identificación de poblaciones objetivo en el desarrollo de terapias y fármacos.
Introducción
Las infecciones a menudo causan reacciones complejas en el huésped que involucran los sistemas inmunes innato y adaptativo 1,2. Tras la infección o el reconocimiento de agentes infecciosos, las células huésped pueden producir una amplia gama de quimioterapia y citocinas, que son pequeñas proteínas conocidas como comunicadores críticos y moduladores del sistema inmunitario3. Las citocinas proinflamatorias se liberan temprano después de la infección para iniciar la respuesta inmunitaria, seguidas más tarde por citocinas antiinflamatorias, que son fundamentales para prevenir el daño tisular y las enfermedades crónicas o autoinflamatorias posteriores. Este equilibrio entre la eliminación de amenazas y la protección de los tejidos se manifiesta como un amplio repertorio de citocinas que ejercen diferentes funciones durante la infección, lo que permite un ajuste fino de la respuesta 4,5. Dentro de esta mezcla, se pueden observar firmas únicas en función del patógeno y las señales que inducen, la ubicación del tejido y las células inmunitarias de las que se originan. Sin embargo, la liberación de citocinas también parece constituir un proceso biológico multifuncional único para cada población celular, diverso en la dinámica de secreción y la respuesta individual. Esta heterogeneidad se ha descrito en la literatura desde hace muchos años, por ejemplo, entre las subpoblaciones de células T 6,7, donde las investigaciones sobre enfermedades autoinflamatorias e infecciones graves por COVID-19 mostraron una gran diversidad funcional de marcadores inflamatorios dentro y entre pacientes 8,9. Recientemente, el advenimiento de la secuenciación de una sola célula puso de manifiesto la alta plasticidad y la diafonía entre subpoblaciones dentro de microambientes inmunes que no eran evidentes anteriormente, lo que indica que los métodos de una sola célula son necesarios para capturar esta heterogeneidad10,11. Si bien se están desarrollando nuevos métodos para analizar el transcriptoma, el análisis fenotípico sigue siendo un desafío, ya que requiere mediciones simultáneas, cuantitativas y resueltas en el tiempo de la secreción de proteínas a nivel de una sola célula. Tales mediciones nos permiten investigar las identidades celulares secretoras, la dinámica y los patrones de secreción (lento/rápido, temprano/tardío, simultáneo/secuencial) para un repertorio o un panel de citocinas. Al permitir el estudio cuantitativo y temporal de la dinámica de la liberación de citocinas durante una respuesta inmunitaria, los conocimientos resultantes podrían permitir comprender el conjunto celular y la respuesta inducida.
En los protocolos estándar, las citocinas generalmente se detectan en el sobrenadante de las suspensiones celulares y el suero mediante un ensayo de inmunoabsorción ligado a enzimas (ELISA), lo que produce cantidades secretadas en gran medida. Las mediciones masivas no permiten cuantificar las cantidades de citocinas producidas por cada célula, un problema especialmente destacado en muestras de células heterogéneas. Los métodos alternativos, como la tinción de citocinas intracelulares, el ensayo de inmunopunto ligado a enzimas (ELISpot) o los ensayos micrograbados (por ejemplo, Isoplexis) detectan las citocinas expresadas por células individuales, pero solo proporcionan mediciones de puntos finales12,13. Esto significa que se ignora la dinámica de secreción y los cambios que pueden ocurrir en el patrón de secreción celular durante el tiempo de incubación. Además, las mediciones de los puntos finales no pueden diferenciar entre la secreción simultánea y secuencial de citocinas, por lo que el verdadero alcance de la polifuncionalidad simultánea de las células inmunitarias en la secreción de citocinas sigue sin estar claro con estos métodos.
La resolución de una sola célula se puede lograr utilizando la microfluídica de gotas para generar y procesar compartimentos físicos del tamaño de un picolitro con el fin de estudiar las células inmunitarias en sus fenotipos únicos de secreción de citocinas a nivel de una sola célula14,15. Estos compartimentos consisten en emulsiones de agua en aceite y se pueden generar utilizando chips microfluídicos16,17. De hecho, los ensayos microfluídicos basados en gotas han demostrado una extrema versatilidad para permitir el análisis de diferentes muestras y repertorios biológicos a nivel de una sola célula y su integración con los procesos anteriores (procesamiento de células y reactivos) y posteriores (clasificación de células individuales, proteómica o secuenciación)18,19,20,21,22. En particular, las configuraciones que permiten la inmovilización de gotas permiten la medición de la funcionalidad de una sola célula a lo largo del tiempo, lo que es valioso para el análisis de la secreción de proteínas18. Además, la integración de ensayos cuantitativos multiplexados facilita investigaciones adicionales en dimensiones previamente inaccesibles, en procesos como la cosecreción y la identificación de células inmunitarias polifuncionales23,24.
En este protocolo, describimos un flujo de trabajo de una sola célula basado en gotas inmovilizadas para detectar, cuantificar y medir temporalmente la secreción de hasta tres citocinas en paralelo a partir de células individuales17,23. La tecnología ofrece la capacidad de monitorear las respuestas de citocinas de más de 20.000 células en paralelo.
El flujo de trabajo presentado consiste en la encapsulación microfluídica de células inmunitarias individuales y nanopartículas funcionalizadas en gotas de agua en aceite de 60 pL. La inmovilización de >100.000 gotas en una cámara de observación y la microscopía de fluorescencia resuelta en el tiempo permiten medir la dinámica de la secreción de citocinas dentro de cada gota y cada citocina (Figura 1A). Para cada célula individual dentro de una gota, la secreción de citocinas se mide mediante un inmunoensayo sándwich, donde las nanopartículas magnéticas funcionalizadas con un anticuerpo de captura específico se unen a la citocina secretada, lo que lleva a la posterior reubicación y unión de anticuerpos de detección marcados con fluorescencia (Figura 1B, C). Se forma una línea de perlas alineando las nanopartículas magnéticas, a las que se puede cuantificar la reubicación de la fluorescencia en presencia de citocinas. Aquí, la reubicación de la fluorescencia se define como la intensidad de fluorescencia promedio encontrada en la línea de perlas dividida por la intensidad de fluorescencia promedio de la gota restante. Este ensayo se puede multiplexar para varias citocinas mezclando lotes de nanopartículas funcionalizadas de manera diferente y los respectivos anticuerpos de detección marcados en diferentes canales de fluorescencia23, lo que da como resultado reubicaciones de fluorescencia específicas en los diferentes canales. Con la ayuda de un script de análisis personalizado, se pueden extraer valores de reubicación de fluorescencia y las imágenes se pueden convertir en perfiles dinámicos de secreción para cada célula y citocina individual. Por lo tanto, los conjuntos de datos resultantes producen numerosas lecturas, como la medición cuantitativa de la secreción a lo largo del tiempo, la identificación de subpoblaciones cosecretoras y las distribuciones de las células según las cantidades, tasas y combinaciones de citocinas secretadas.
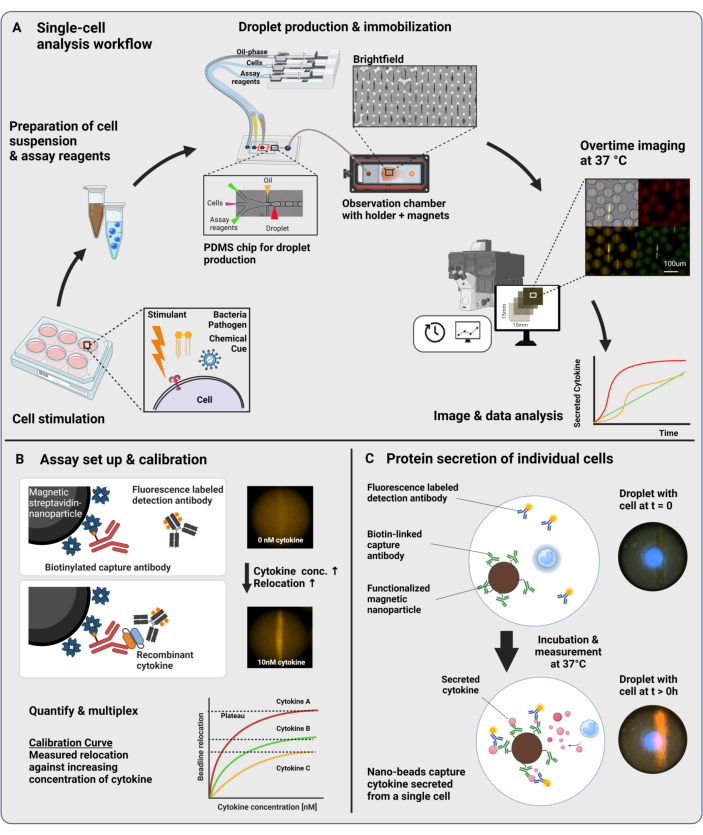
Figura 1: Flujo de trabajo y principio de ensayo. (A) Descripción general del flujo de trabajo para analizar las células secretoras de citocinas después de la estimulación. Las suspensiones unicelulares y las nanopartículas magnéticas se preparan y encapsulan en emulsiones aceite/agua de 60 pL de volumen (gotas). Las gotas se inmovilizan y las nanopartículas se alinean dentro de un campo magnético antes de la medición durante un máximo de 4 h cada 30 minutos. Finalmente, se analizan las imágenes y se extraen los parámetros de cada gota, punto de tiempo y canal fluorescente. Esta cifra ha sido modificada de17. (B) Principio del bioensayo de sándwich de gotas. Las nanopartículas funcionalizadas se unen a las citocinas secretadas, lo que conduce a la posterior reubicación de los anticuerpos de detección marcados con fluorescencia a las nanopartículas. Esta reubicación de la fluorescencia se cuantifica y valida con experimentos de calibración realizados con citocinas recombinantes. La mezcla de diferentes nanopartículas funcionalizadas permite las mediciones multiplexadas de hasta tres citoquinas simultáneamente. (C) En los experimentos basados en células, las gotas se siguen durante el tiempo de medición y las células secretoras se identifican mediante un aumento con el tiempo de la reubicación de la fluorescencia en las nanopartículas. Los esquemas no están a la altura. Figura creada con BioRender.com. Haga clic aquí para ver una versión más grande de esta figura.
Protocolo
Todos los experimentos se realizaron bajo el acuerdo de ética EK202-N-56 y fueron aprobados por la comisión de ética de ETH Zurich. La manipulación de las células humanas se realizó en una cabina de flujo laminar contenida en un laboratorio de nivel 2 de bioseguridad.
NOTA: En las siguientes secciones se detalla el protocolo para medir la secreción de citocinas resueltas en el tiempo a nivel de una sola célula. El procedimiento descrito aquí se aplica a la estimulación de células mononucleares de sangre periférica (PBMC) con lipopolisacárido (LPS) y la medición paralela de las citocinas IL-6, TNFα e IL-1β. Sin embargo, si es necesario, el protocolo se puede adaptar a otros tipos de células, estimulantes y citoquinas.
1. Fabricación de la cámara de observación
NOTA: Para evitar el movimiento de las gotas durante la toma de imágenes, se prepara una cámara de observación con una altura que es aproximadamente un 10% menor que el diámetro de la gota.
- Preparación de la cinta de corte de doble cara y la corredera de vidrio superior
- Dibuje o cargue el diseño deseado del recorte de la cámara en la pestaña Diseño del software de corte. Para conocer las dimensiones específicas utilizadas aquí, consulte la Figura 2E.
- Fije la cinta adhesiva de doble cara de 32 μm de grosor a la alfombrilla de corte adhesiva con cinta adhesiva y coloque la alfombrilla de corte en la máquina de corte automática.
- Corta el diseño de la cámara de la cinta, mientras prestas atención a cortar los bordes largos de la cámara en la misma dirección para facilitar el desprendimiento en el paso 1.3.
- Guarde los recortes de cinta a temperatura ambiente para almacenarlos a largo plazo. Para el almacenamiento a corto plazo, guárdelos a -20 °C y retírelos poco antes del paso 1.3. para facilitar su manejo.
- Taladre dos orificios de aproximadamente 1 mm de diámetro en el centro de un portaobjetos de microscopio estándar (76 mm x 26 mm x 1 mm), con una distancia entre los dos orificios de aproximadamente 3,5 cm.
- Limpieza y activación por plasma de portaobjetos de vidrio
- Limpie un portaobjetos de vidrio con agujeros y otro sin agujeros con jabón. Enjuague bien con agua destilada y seque con toallitas de precisión sin pelusa.
- Coloque los portaobjetos de vidrio en un limpiador de plasma y trate con plasma las superficies superiores a 55 W durante 10 minutos. Retire los portaobjetos de vidrio y continúe con el paso 1.3.
- Montaje de la cámara
- Coloque el portaobjetos de vidrio con orificios sobre una superficie limpia con el lado activado por plasma hacia arriba, sin tocar la superficie activada.
- Retire la capa protectora de un lado de la cinta adhesiva de doble cara, en la misma dirección de corte. Sin tocar, alinee el corte de la cinta con los bordes del portaobjetos de vidrio y los orificios perforados, y coloque lentamente la cinta en contacto con el portaobjetos de vidrio comenzando por el borde corto.
NOTA: Preste atención a no generar estiramientos o pliegues en la cinta, ya que esto dará como resultado alturas de cámara incorrectas. Dado que este paso es propenso a errores y requiere cierta experiencia práctica, es aconsejable preparar varios portaobjetos de vidrio en paralelo. - Retire la segunda capa protectora de la cinta, nuevamente en la dirección de corte, y coloque la segunda corredera de vidrio sin orificios con la superficie activada hacia abajo. Presione toda la superficie de los dos portaobjetos de vidrio colocando una tabla plana en la parte superior y presionando hacia abajo con la fuerza de la parte superior del cuerpo durante unos 10 s.
- Después de ensamblar las dos correderas de vidrio, voltee la cámara para que los dos orificios queden frente a usted. Pegue los nanopuertos a los dos orificios colocando una pequeña cantidad de pegamento curable por UV en el anillo debajo del puerto y colocando el puerto encima del orificio en el portaobjetos de vidrio. Agregue un anillo de pegamento curable UV alrededor del puerto y cure el pegamento con una lámpara UV. La cámara debería tener ahora el aspecto que se muestra en la Figura 2E. Continúe inmediatamente con el paso 1.4.
NOTA: La luz ultravioleta puede dañar los ojos y la piel. Use el equipo de protección adecuado.
- Recubrimiento fluorófilo de la superficie de la cámara
NOTA: Este paso debe realizarse dentro de 1 h después del tratamiento con plasma de los portaobjetos de vidrio (paso 1.2.2) para garantizar una buena eficiencia del recubrimiento.- Prepare 1 mL de solución de fluorosilano al 1% (1H, 1H, 2H, 2H-perfluorodecirltriclorosilano) en aceite fluorado (HFE-7500) y llénelo en una jeringa. Empuje la solución de recubrimiento a través de un filtro de jeringa de PTFE y una aguja de 27G x 0,75 pulgadas conectada a un microtubo de PTFE de 0,3 mm x 0,76 mm, dentro de la cámara de observación.
- Después de 1 minuto de incubación, enjuague la solución de recubrimiento fuera de la cámara usando presión de nitrógeno bajo una campana extractora. Enjuague la cámara con aceite fluorado (solo HFE-7500) utilizando otro conjunto de jeringa.
- Guarde la cámara llena de aceite fluorado con entradas cerradas a temperatura ambiente (RT). Después de cada experimento, lave las células y las gotas directamente para garantizar una buena conservación del recubrimiento.
NOTA: El protocolo se puede pausar aquí, y las cámaras se pueden almacenar y reutilizar durante varios meses.
- Soporte de cámara con imanes
- Para la alineación de las nanopartículas magnéticas, aplique un campo magnético estático a la cámara de observación durante la encapsulación de gotas y la obtención de imágenes. Para ello, coloque la cámara en un soporte de microscopía personalizado impreso en 3D (véase la Figura 2D y el archivo que se encuentra en Bounab et al.17 Datos complementarios 4) que contiene dos imanes de neodimio a lo largo de los lados largos de la cámara.
2. Funcionalización de nanopartículas
NOTA: El proceso de funcionalización de nanopartículas es similar para cada citocina, la única diferencia es la adición de anticuerpos de captura específicos de citocinas. La funcionalización de cada citocina se realiza en diferentes tubos de reacción individuales en paralelo. Antes de este protocolo, el anticuerpo de captura de TNFα y el anticuerpo de detección de IL-1β se marcaban internamente con biotina y Alexa Fluor 647, respectivamente. La conjugación se realizó de acuerdo con el protocolo del fabricante que se encuentra en el sitio web del proveedor (ver enlaces en la Tabla de Materiales) y los anticuerpos se alícuotas y almacenaron a -20 °C.
- Añadir 50 μL de nanopartículas funcionalizadas con estreptavidina (diámetro (Ø) 300 nm) en el tubo destinado a la detección de TNFα, 50 μL para la IL-1β y 100 μL para la IL-6. Diluir la solución de nanopartículas 1:1 (v/v) en solución salina tamponada con fosfato (PBS).
- Añadir a cada tubo 1/20 (v/v) del volumen respectivo de los anticuerpos de captura biotinilados (concentraciones de stock de 0,5 mg/mL) e incubar durante 30 min en RT.
NOTA: Cuando agregue pequeños volúmenes a la solución de nanopartículas, deposite el volumen en la parte superior del tubo y lávelo varias veces con la mayor parte de la solución. Esto asegura una mezcla adecuada y evita la formación de agregados. - Añadir 1/100 (v/v) de 1 mM de solución de D-biotina al tubo e incubar durante 5 min a RT. Esto da como resultado una concentración final de biotina de 10 μM.
NOTA: El exceso de biotina bloquea los lados de unión libre en las nanopartículas y reduce la formación de agregados no deseados. - Recoja las partículas sosteniendo un imán de neodimio cerca del tubo. Espere hasta que el sobrenadante esté claro y deseche el sobrenadante.
NOTA: Los imanes utilizados a lo largo del ensayo exhiben fuerzas de atracción muy fuertes, que pueden causar daños físicos si dos imanes se unen accidentalmente. - Para reducir la adsorción inespecífica a la superficie de la nanopartícula, resuspenda inmediatamente las nanopartículas en 0,5 veces el volumen final del paso 2.1 de Pluronic F-127 (10%) y 0,5 veces el volumen PBS. Incubar la solución durante 30 min en RT.
- Recoja las partículas con el imán, deseche el sobrenadante y vuelva a suspender en 1 vez el volumen del tampón de almacenamiento (RPMI 1640, 5% de reemplazo de suero knockout, 1% de Pen/Strep, 1% de albúmina sérica humana recombinante (HSA), 25 mM de HEPES, 0,1% de Pluronic F-127). Incubar la solución durante 30 min en RT.
NOTA: El protocolo se puede pausar aquí, y las partículas ahora se pueden almacenar hasta por 1 semana a 4 °C. - Inmediatamente antes de la encapsulación, vuelva a suspender las partículas mediante pipeteo y mezcle las nanopartículas conjugadas en una proporción de 2:1:1 (v/v) para IL-6:TNFα:IL-1β, respectivamente.
NOTA: Las diferentes proporciones de nanopartículas funcionalizadas dependen del par de anticuerpos utilizado para cada citocina y se han determinado experimentalmente a través de muestras de calibración para obtener un rango dinámico óptimo. - Lavar con medios completos (RPMI 1640, 10% FBS, 1% Pen/Strep, 25 mM HEPES) recogiendo las partículas con el imán, desechando el sobrenadante y volviendo a suspender. Repita este paso, pero vuelva a suspender solo en 0,5 veces el volumen de los medios completos del paso 2.7.
- Agregue los anticuerpos de detección IL-6, TNFα e IL-1β marcados de manera diferente a la solución para alcanzar una concentración final de 10 nM cada uno. La solución ya está lista para ser utilizada en los experimentos con gotas.
3. Preparación de la célula
NOTA: Las PBMC se aislaron de un abrigo buffy recibido del banco de sangre de Zúrich. Las células se congelaron y se almacenaron en crioviales (1 x 107 células/vial) en nitrógeno líquido durante varios meses.
- Descongelación de células
- 1 h antes de comenzar el experimento, deje que el medio completo y el tampón MACS (2 mM EDTA, 0,5% BSA en DPBS, filtrado estéril) se calienten en RT. Prepare el tubo que contiene las células añadiendo 9 mL de medio completo en un tubo de 15 mL y manteniéndolo en el baño de agua a 37 °C.
- Recupere un criovial PBMC (que contiene ~ 1 x 107 celdas) de su almacenamiento en nitrógeno líquido. Agite el tubo criogénico en el baño de agua a 37 °C hasta que solo quede una pequeña cantidad de hielo.
- Limpie el tubo con 70% de EtOH y transfiéralo a la cabina de flujo laminar. Añada 1 ml de medio completo precalentado al criovial, mezcle suavemente y transfiera todas las células al tubo que contiene el medio completo tibio. El criovial se puede lavar con 1 mL de medio completo tibio para recuperar el máximo número de células.
- Centrifugar las células a 500 x g durante 5 min en RT, desechar el sobrenadante y volver a suspender suavemente con una pipeta el pellet de células con 1 mL de medio completo. Agregue 9 mL de medio completo.
- Centrifugar las células a 500 x g durante 5 min en RT. Deseche el sobrenadante y vuelva a suspender como se ha hecho anteriormente en 1 mL de medio completo.
- Cuente las celdas usando el contador de celdas disponible. En este caso, se utilizó un contador de células automatizado. Las células se contaron mezclando 10 μL de suspensión celular con 10 μL de azul de tripán y transfiriendo 10 μL de la mezcla al portaobjetos de recuento celular.
- Tinción y bloqueo de FcR
- Calcule el número total de células y el volumen necesario para resuspender las células a 2 x 106 células vivas/mL. Prepare la solución de tinción celular (CellTrace Violet) diluyendo el material (5 mM) 1000 veces en PBS (concentración de trabajo de 5 μM).
- Centrifugar las células a 500 x g durante 5 min en RT. Deseche el sobrenadante y vuelva a suspender las células en el volumen calculado de solución de tinción celular preparado en el paso 3.2.1. Incubar las células a 37 °C durante 5 min.
- Al final de la incubación, enfríe el colorante restante en la solución agregando medios completos (al menos 2 veces el volumen de la solución de tinte). Gire las celdas a 500 x g durante 5 min en RT.
- Deseche el sobrenadante, vuelva a suspender el pellet celular en 60 μL de tampón MACS y agregue 20 μL de bloque de FcR humano por 1 x 107 células. Incubar las células durante 10 min en RT.
- Llene el tubo hasta 10 ml con MACS Buffer y centrifuga las células a 500 x g durante 5 min en RT.
- Deseche el sobrenadante y vuelva a suspender las células en 1 ml de medio completo Cuente las células como se describe en el paso 3.1.6.
- Estimulación celular con LPS
- Usando el recuento de células, diluya las células a 1 x 106 células/mL y transfiera 2 mL de células a cada pocillo en una placa de 6 pocillos de unión ultrabaja.
- Diluya el LPS en medios completos y añádalo al pocillo que contiene las células para obtener una concentración final de LPS de 1 μg/mL. Incubar las células durante 6 h a 37 °C.
- Preparación para la encapsulación
- Al final del tiempo de estimulación, transfiera la suspensión celular a un nuevo tubo de 15 mL.
- Agregue 1 mL de medio completo en el pocillo vacío. Con un raspador de células, separe las células restantes. Transfiera las células a un nuevo tubo de 15 ml. Lave el pocillo con 1 mL de medio completo y transfiéralo a otro tubo de 15 mL.
- Centrifugar los dos tubos a 500 x g durante 5 min a RT y transferir 1 mL de la solución sobrenadante sin diluir (del primer tubo que contiene las células no lavadas) a un nuevo tubo para su posterior análisis si es necesario (p. ej., ELISA).
- Desecha el resto de los sobrenadantes.
- Vuelva a suspender los gránulos en 0,5 ml de medio completo, combine las células del mismo pocillo y transfiéralas a un tubo de centrífuga. Cuente las celdas como se describe en el paso 3.1.6.
- Centrifugar las células a 500 x g durante 5 min a RT y desechar la mayor parte del sobrenadante (dejando aproximadamente 100 μL). Sin volver a suspender el pellet, agregue con mucho cuidado 200 μL de medio completo.
- Deseche el sobrenadante. Vuelva a suspender las células en medios completos a una concentración de 6,6 a 13,3 x 106 células/ml para lograr un número medio de células por gota de λ = 0,2-0,4 para la encapsulación, tal como se define en el paso 8.6.
NOTA: Los pasos 3.4.6 y 3.4.7 deben realizarse inmediatamente antes de la encapsulación para evitar la secreción de citocinas en el sobrenadante. El número de células por gota sigue una distribución de Poisson: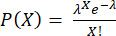 , donde P muestra la fracción de gotas que contienen X células y λ es el número medio de células por gota.
, donde P muestra la fracción de gotas que contienen X células y λ es el número medio de células por gota.
4. Encapsulación y producción de gotas
NOTA: La encapsulación de células en gotas es posible mediante un chip generador de gotas microfluídicas, cuya fabricación se describe con gran detalle en otra parte17. Las alternativas están disponibles comercialmente (ver ejemplo en la Tabla de Materiales). Un diseño de chip generador de gotas adecuado tiene dos entradas para las fases acuosas, una entrada para la fase oleosa y una salida para las gotas generadas. Además, un chip generador de gotas comercial adecuado debería permitir la producción de agua en gotas de aceite fluorado de 40-60 pL de volumen. El protocolo descrito aquí da como resultado emulsiones de agua/aceite (gotas) con un diámetro de 50 μm. El uso de varias opciones para modificar el protocolo puede dar lugar a gotas más grandes o más pequeñas.
- Preparación de la bomba de jeringa (Figura 2A)
- Llene una jeringa de 1 ml con 500 μL de fase continua que consiste en un 2% de 008-tensioactivo fluorado en aceite fluorado HFE-7500. Conecte una aguja de 27G x 0,75 pulgadas a un microtubo de PTFE de 0,30 mm x 0,76 mm y monte el conjunto en la jeringa y, posteriormente, en la bomba de jeringa.
NOTA: Asegúrese de que no quede aire en la jeringa o cánula, ya que esto evita que los caudales sean constantes. - Prepare dos conectores de punta de pipeta hechos a medida para las fases acuosas (Figura 2B): Haga un orificio con un punzón de biopsia de Ø0,75 mm en el centro de un recorte de PDMS de ~5 mm de altura con Ø6 mm. Tire de ~3 cm de tubo de PTFE (0,56 mm de diámetro interior, 1,07 mm de diámetro exterior) a través del orificio del recorte del PDMS y empuje el conjunto hacia la parte superior de una punta de pipeta de 200 μL. Conecte el otro lado del tubo a una aguja de 23Gx 1.25 pulgadas. Selle el conector esparciendo pegamento curable por UV sobre la pipeta y cúrelo con luz UV.
NOTA: Dado que la luz ultravioleta es dañina para los ojos, use gafas que bloqueen los rayos UV para protegerse. - Llene dos jeringas de 1 ml con 500 μl de aceite mineral ligero, conecte dos agujas de 23 g con los accesorios hechos a medida y monte ambas en la bomba de jeringa.
- Aspire 30 μL de nanopartícula y 30 μL de solución celular en las puntas de pipeta de las fases acuosas utilizando el software de control de la bomba de jeringa.
- Prepare una cámara de observación limpiando la superficie con agua para eliminar la suciedad y el polvo y séquela con toallitas de precisión. Sujete la cámara en el soporte de cámara impreso equipado con dos imanes de neodimio.
NOTA: Asegúrese de que los imanes apunten en la dirección correcta (atrayéndose entre sí) para formar un agregado alargado. - Inclinar ligeramente la cámara (30°). Abra ambos puertos y conecte una toalla de papel en el puerto superior para absorber el exceso de fase exterior durante el llenado.
- Llene una jeringa de 1 ml con 500 μL de fase continua que consiste en un 2% de 008-tensioactivo fluorado en aceite fluorado HFE-7500. Conecte una aguja de 27G x 0,75 pulgadas a un microtubo de PTFE de 0,30 mm x 0,76 mm y monte el conjunto en la jeringa y, posteriormente, en la bomba de jeringa.
- Producción de gotas y llenado de cámaras
- Conecte la fase continua a través de un tubo a la entrada superior del chip microfluídico (Figura 2A, C, F). Enjuague la viruta durante unos 30 s con fase continua utilizando un caudal de 1800 μL/h.
- Conecte las puntas de pipeta de las soluciones acuosas a las dos entradas intermedias (Figura 2A, C, F).
- Inicie el flujo de la solución acuosa con 200 μL/h cada uno y deje que los canales y la salida se llenen de líquido. Cuando se utilizan nanopartículas magnéticas, una solución homogénea de color marrón rojizo debe salir de la salida del chip.
- Una vez que el líquido aparezca en la salida, inicie el flujo de la fase oleosa fluorada a 800 μL/h y espere hasta que se establezca una producción de gotas estable, confirmada por la salida de una solución homogénea, gris y brillante en la salida.
- Una vez que se establece una producción estable de gotas, recoja las gotas producidas conectando microtubos de PTFE (0,3 mm de diámetro interior x 0,76 mm de diámetro exterior) al puerto de salida y diríjalos a una cámara de observación pasando el microtubo a través del módulo de férula de un accesorio de una sola pieza apretado con los dedos (Figura 2A).
- Si se produce una producción adecuada de gotas, un líquido homogéneo y brillante debe llenar la cámara con un frente recto de abajo hacia arriba.
- Una vez que la cámara esté llena, detenga el flujo y cierre los puertos con tapones de puerto usando una presión apretada con los dedos.
NOTA: Preste atención a no cerrar la cámara demasiado herméticamente. El atrapamiento o la afluencia de aire pueden provocar movimientos de las gotas y, por lo tanto, comprometer el seguimiento durante la medición. - Después de la producción de gotas, enjuague el chip con aceite fluorado y sople cualquier recordatorio de líquido con nitrógeno para preservar su función. Los chips se pueden reutilizar varias veces y almacenar durante meses, siempre que no se obstruyan.
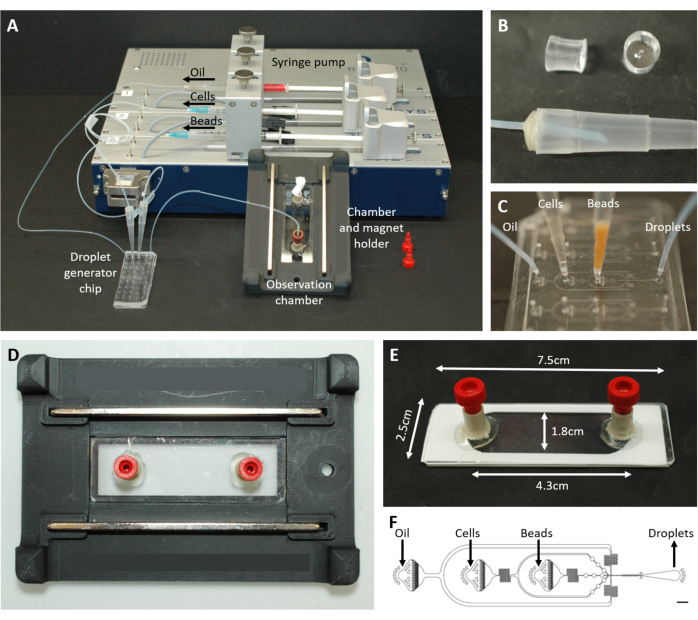
Figura 2: Descripción general de la configuración microfluídica. (A) Configuración para la encapsulación de gotas con la bomba de jeringa, el chip de generación de gotas y la cámara de observación y el soporte del microscopio. (B) Imagen del conector PDMS perforado (arriba) para formar un conector a una punta de pipeta de 200 μL (abajo), como se describe en el paso del protocolo 4.1.2. (C) Imágenes de la conexión de los tubos y las puntas de pipeta al chip de generación de gotas. (D) Imagen de la cámara colocada dentro del soporte de microscopio personalizado impreso en 3D con dos imanes en la parte superior e inferior. (E) Foto de la cámara de observación (con cinta blanca a modo de ilustración). (F) Disposición del chip microfluídico para la creación de gotas (barra de escala: 750 μm). Esta cifra ha sido modificada de17. Haga clic aquí para ver una versión más grande de esta figura.
5. Adquisición y medición de imágenes
NOTA: La adquisición de imágenes se realiza en un microscopio de epifluorescencia invertida estándar encerrado en una incubadora, lo que permite mediciones a 37 °C. Los ajustes aquí descritos son específicos para un microscopio Nikon Eclipse Ti2 que funciona con el software NIS Elements (V. 5.30.04) equipado con una cámara Orca Fusion, pero generalmente se adaptan a cualquier otro microscopio y cámara de fluorescencia.
- Ajuste de los parámetros de medición
- Para establecer el tamaño de la imagen, seleccione un tamaño de matriz de 10 x 10 imágenes. Esta matriz contendrá aproximadamente entre 50.000 y 70.000 gotas. Utilice una superposición del 1% y active la fusión para la unión de imágenes.
- Para establecer el número de canales medidos, seleccione el canal DAPI para la detección de células, FITC, TRITC, los canales Cy5 para la detección de citocinas (beadlines) y el canal BF para la detección de gotas. Utilice la agrupación en bins de píxeles 2 x 2 y 16 bits para la profundidad de bits. Adapte la configuración de la cámara para lograr valores de píxeles de intensidad en gota que no alcancen el máximo de la cámara para cada canal de fluorescencia.
NOTA: Los tiempos de exposición exactos y las intensidades de lámpara para cada canal dependen del modelo y los reactivos utilizados y se establecen antes de generar las curvas de calibración (paso 7). El uso de los mismos ajustes de adquisición en la calibración y las mediciones de celdas es importante para una cuantificación precisa. - Para configurar la medición resuelta en el tiempo, seleccione una medición cada 30 minutos para un total de 9 mediciones.
NOTA: Los parámetros de medición pueden diferir para las células utilizadas, los estimulantes, los reactivos, las citocinas medidas, la temperatura de incubación y los modelos de microscopio.
- Inicio de la medición
- Monte el soporte de la cámara en el microscopio con una platina de formato de placa de pocillo (Figura 2D) y cambie al canal de campo claro (BF) utilizando el objetivo 10x.
- Concéntrese en las gotas inmovilizadas en BF y asegúrese de que el conjunto esté montado en un plano perfecto girando y ajustando si es necesario. Muévase al centro de la cámara para los pasos siguientes.
- Active el sistema de enfoque automático (PFS) y ajústelo al plano de medición óptimo en el canal BF para que los bordes de las gotas aparezcan como círculos negros y nítidos que se puedan distinguir fácilmente de la fase oleosa y el fondo.
NOTA: Las mediciones también son posibles sin un sistema de enfoque automatizado, pero si el microscopio está equipado con uno, recomendamos encarecidamente usarlo. Esto mejora la calidad de medición de imágenes grandes y muy unidas. - Recorra todos los canales de fluorescencia y establezca el plano de medición óptimo para cada uno. Para las mediciones de reubicación en los canales FITC, TRITC y Cy5, asegúrese de que el agregado de nanopartículas esté perfectamente enfocado, para el canal DAPI, asegúrese de que las celdas estén enfocadas.
NOTA: Los planos focales óptimos y los valores z pueden diferir para todos los canales medidos. Asegúrese de guardar los desplazamientos PFS individuales para cada canal. - Antes de comenzar la medición, revise todos los canales para verificar los focos individuales y espere 5 minutos para equilibrar, ya que puede ocurrir movimiento inicialmente mientras las soluciones se calientan.
- Inicie la medición. Después de generar la primera imagen, compruebe si hay irregularidades (enfoque, gotas en movimiento, canales incorrectos, etc.). Reinicie la adquisición si es necesario, o en el caso de aire, vuelva a llenar la cámara (comience desde el paso 4.1.4). Deje que el ensamblaje muestre las gotas durante 4 h.
6. Análisis de imágenes
- Instale el software de análisis de imágenes (DropMap Analyzer App v 4.023) en MatLab (https://github.com/ESPCI-LCMD/MiMB) y transfiera el archivo .nd2 generado desde el experimento a un equipo de análisis.
- Abra la aplicación. Seleccione la configuración especificada, de lo contrario, deje el valor predeterminado: CH1: DAPI, WD (gota entera) seleccionado; CH2: FITC, BL (línea de cuentas) seleccionados; CH3: TRITC, BL seleccionados; CH4: Cy5, BL seleccionado; Diámetro máximo de caída (μm): 70; Detección de caídas: Completo; Seguimiento: Sí. Pulse el botón Inicio (icono de fruta) para seleccionar la ubicación del archivo .nd2 e iniciar el análisis.
- Después de unos minutos, el programa mostrará una sección de ejemplo de la imagen (Figura 3A). Presione la barra espaciadora hasta que encuentre uno adecuado para la detección de gotas, luego presione Enter. En la misma sección de la imagen, dibuje un rectángulo en un área representativa para encontrar parámetros de umbral para detectar gotas.
- Después de unos minutos, se abrirá otra ventana que muestra la distribución de intensidad del canal DAPI. Arrastre y suelte el control deslizante para detectar solo la señal de las células manchadas y haga clic en Listo en la esquina superior derecha.
- Después de segmentar la imagen en gotas individuales con diámetros menores que el diámetro máximo de gota (μm), el programa ahora realizará los siguientes pasos para cada gota, punto de tiempo y canal de fluorescencia sin más intervención del usuario (consulte la Figura 3).
- El software calcula los píxeles movidos de la gota entre puntos de tiempo (las gotas que se mueven más de 40 píxeles se excluyen automáticamente).
- El software mide el valor medio de fluorescencia de toda la gota, detectando y midiendo la intensidad media de la línea de perlas encontrando el píxel más brillante en una línea horizontal y promediando todas las intensidades de píxeles en una línea vertical de arriba a abajo de la gota. Esto se hace automáticamente y se utiliza para calcular los valores promedio de reubicación de la línea de cuentas (Figura 3B) de acuerdo con la ecuación:
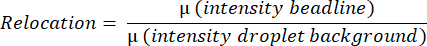
- El software calcula el porcentaje del total de píxeles en el área de la gota por encima del umbral establecido en el canal DAPI.
- El archivo .xslx resultante contendrá las siguientes columnas de interés para un análisis más detallado: DropIdX (ID de la gota rastreada a lo largo del tiempo), TrueCentroid_ t*2-1 y t+2 (coordenadas x e y, respectivamente, del centro de la gota para el punto de tiempo t), DiameterMicrons (diámetro de la gota en μm), TrackingMove (número de píxeles movidos durante todo el tiempo de medición), FluoChannel_BL_Ratio_t (valor de reubicación para FluoChannel en el punto de tiempo t), DAPI_WD_PosPxlCount_t (número de píxeles por encima del umbral en toda la gota en el canal DAPI en el punto de tiempo t).
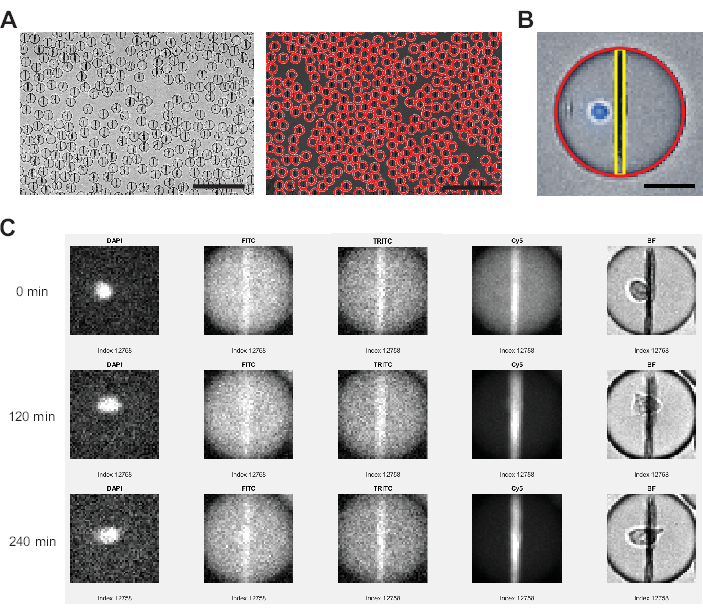
Figura 3: Análisis de imágenes realizado por el software de análisis de imágenes. (A) Las gotas se detectan en el canal de campo claro (BF) utilizando una transformación de Hough, marcando cada gota con un círculo rojo. Barras de escala: 200 μm. (B) Dentro de cada gota, la línea de perlas de nanopartículas se identifica a través de los píxeles más brillantes en el plano horizontal y las intensidades de fluorescencia promediadas para todos los píxeles que abarcan de arriba a abajo de la gota. Además, la celda se identifica a través de un porcentaje de píxeles >0 por encima del umbral para toda el área de la gota. Barra de escala: 20 μm. (C) El software del analizador compara la intensidad de fluorescencia de las nanopartículas con el fondo de las gotas para los canales FITC, TRITC y Cy5 en todos los puntos de tiempo medidos para cada gota individual. Se muestran los puntos de tiempo 0, 4 (120 min) y 9 (240 min). Para comprobar manualmente la correcta detección de gotas y células, también se muestran los canales DAPI y BF. Haga clic aquí para ver una versión más grande de esta figura.
7. Calibración
NOTA: Para una lectura cuantitativa, la calibración de las concentraciones de citocinas a los valores de reubicación de fluorescencia debe realizarse una vez, ya que pueden ocurrir diferencias entre diferentes configuraciones experimentales. Todos los pasos necesarios se detallan en las secciones de protocolo anteriores a las que se hace referencia.
- Prepare las nanopartículas como se describe en el paso 2.
- Reconstituya las proteínas recombinantes IL-6, TNFα e IL-1β humanas de acuerdo con las instrucciones del fabricante.
NOTA: Asegúrese de que las alícuotas congeladas se descongelen solo una vez y se usen con prontitud. - Prepare una serie de diluciones de 2 veces para las tres proteínas juntas utilizando medios completos (10% FBS, 1% Pen/Strep, 25 mM HEPES) con una concentración inicial de 80 nM hasta 0,625 nM.
- Realice la encapsulación como se describe en el paso 4 con nanopartículas funcionalizadas en la primera y RPMI solo en la segunda fase acuosa. Esta medición sirve como blanco y la desviación estándar medida se utiliza para el análisis de datos posterior.
- Espere 5 minutos y obtenga una imagen de las gotas como se describe en el paso 5. Tome 3 imágenes con un tamaño de matriz de 2 x 2 en los respectivos canales de fluorescencia.
- Repita los pasos 7.4 y 7.5 con todas las soluciones de calibración preparadas, comenzando con la concentración más baja y terminando con la concentración más alta.
- Analice las imágenes como se describe en el paso 6. No utilice la opción WD para el canal DAPI y establezca Tracking en No.
- El análisis genera valores de reubicación de fluorescencia para cada gota medida en una imagen. Extraiga la mediana y la desviación estándar de cada canal de fluorescencia. Promedie la mediana y la desviación estándar de cada imagen medida por concentración.
- Genere una curva de calibración trazando la reubicación mediana promediada frente a las concentraciones medidas de cada proteína recombinante.
- Ajuste las curvas mediante una asociación monofásica:
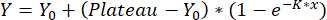 ,
,
con Y = reubicación en x, Y0 = reubicación de la medición en blanco y x la concentración utilizada. La curva de calibración obtenida se utiliza para cuantificar los valores de reubicación como se describe en el paso 8.
NOTA: Solo ajuste los valores hasta la reubicación medida más alta y excluya los valores de concentraciones más altas con una reubicación medida más baja. Se espera una disminución en los valores de reubicación medidos a concentraciones más altas, y se produce debido al efecto Hook y a la limitada capacidad de unión de las nanopartículas.
8. Análisis de datos
- Excluya las gotas con un valor de TrackingMove superior a 10, es decir, que se hayan movido más de 10 píxeles durante el transcurso de la medición.
- Identifique las gotas que contienen células teñidas (canal DAPI) en el primer punto de tiempo ordenando las gotas con valores superiores a 0 en la columna DAPI_WD_PosPxlPercent_1.
- Identifique las gotas que contienen células secretoras aplicando los siguientes 3 criterios a la reubicación de fluorescencia de cada canal de fluorescencia (columnas FluoChannel_BL_Ratio_t).
- Identificación de gotas con valores de reubicación crecientes, mediante la clasificación de una pendiente positiva durante el tiempo de medición.
- Identificación de gotas con valores de reubicación que alcanzan el límite de detección (LOD). Se selecciona una gota cuando la reubicación máxima de fluorescencia a lo largo del tiempo de medición es superior a la LOD, calculada como se describe en la parte25:
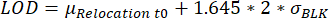 , donde μReubicación t0 es la mediana de todos los valores de reubicación en el punto de tiempo 0 y σBLK la desviación estándar del blanco medidos durante la calibración, cada uno de ellos es específico de la citocina.
, donde μReubicación t0 es la mediana de todos los valores de reubicación en el punto de tiempo 0 y σBLK la desviación estándar del blanco medidos durante la calibración, cada uno de ellos es específico de la citocina. - Verificar que el aumento en el valor de reubicación es significativo comprobando que el cambio entre la reubicación de fluorescencia máxima y mínima medida durante el tiempo de medición es superior a:
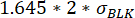 .
.
- Identifique las células cosecretoras cumpliendo los criterios descritos en el paso 8.3. para más de un canal de fluorescencia simultáneamente.
- Repita el paso 8.3. para todas las gotas que no contengan ninguna célula (DAPI_WD_PosPxlPercent_1 = 0). Utilice estas gotas para calcular el porcentaje de falsos positivos.
- Determine el valor λ preciso de la medición seleccionando aleatoriamente de 200 a 500 gotas e inspeccionándolas con la función Verificar y clasificar del software de análisis de imágenes. Cuente el número de células de estas gotas y calcule:
λ = número de células contadas / número de gotas analizadas - Calcule el número total de celdas encapsuladas para la medición mediante:
Recuento total de células = λ × número de gotas analizadas - Calcule el porcentaje de células secretoras utilizando el recuento de células determinado. Además, calcule el porcentaje de falsos positivos para cada citocina (generalmente menos del 3%-5% en relación con el número de positivos reales por canal) y utilícelos como control interno para la consistencia experimental y la reproducibilidad.
- Para calcular las concentraciones de citocinas secretadas, convierta los valores de reubicación en concentración utilizando las ecuaciones de calibración establecidas en el paso 7.10.
- Calcule la tasa de secreción (SR) entre puntos de tiempo utilizando la siguiente ecuación.
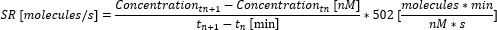
- Calcule la tasa de secreción promedio durante la medición promediando las tasas de secreción individuales entre puntos de tiempo. Si se alcanzó la reubicación máxima medible antes del final de la medición, ajuste la concentración a la concentración máxima medible (este valor es específico de la citocina y corresponde a la concentración máxima medida y utilizada en la curva de calibración en el paso 6.10) y no calcule más concentración. Calcule la tasa de secreción y el promedio solo hasta este punto de tiempo.
NOTA: Si se detectan menos de 50 células secretoras en un canal de fluorescencia, las gotas deben examinarse visualmente a través de la función de verificación y clasificación y las gotas que tengan agregados de fluorescencia o nanopartículas se pueden excluir del análisis. - Para extraer más parámetros de la curva de secreción de cada célula, realice un ajuste de mínimos cuadrados a la curva de tiempo-concentración para cada célula y citocina utilizando un script de Python personalizado (disponible a pedido). La función ajustada es una curva sigmoidal siguiendo la fórmula que se describe a continuación (los ajustes con R2<0,95 se excluyen de los siguientes pasos):
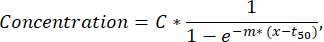
donde C corresponde a la meseta de concentración [nM], t50 al desplazamiento del semimáximo [s] y m a la pendiente del cerro [min-1]. A partir de estos parámetros, se extraen los siguientes descriptores de curva como se describe a continuación.- Cmax [nM]: Concentración máxima medida.
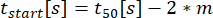 : Momento de inicio de la secreción, donde el ajuste alcanza el 10% de C.
: Momento de inicio de la secreción, donde el ajuste alcanza el 10% de C.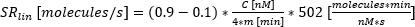 : tasa de secreción como la pendiente lineal aproximada de la curva entre el 10% y el 90% de la curva de tiempo-concentración.
: tasa de secreción como la pendiente lineal aproximada de la curva entre el 10% y el 90% de la curva de tiempo-concentración.
Resultados
La plataforma funcional de una sola célula presentada permitió la medición de varios parámetros. En primer lugar, y de forma similar a las técnicas estándar, la frecuencia de las células secretoras se representa al final de la medición (Figura 4A). Tras la estimulación con 1 μg/mL de lipopolisacárido (LPS) durante 6 h de células mononucleares de sangre periférica (PBMC), el 5,81% de las células segregaron IL-6 (n= 1270), el 4,55% TNFα (n= 995) y el 6,06% IL-1β (n= 1326).
Para cuantificar la secreción de citocinas, se generaron curvas de calibración con concentraciones conocidas de citocinas recombinantes (Figura 4B). Estas curvas de calibración permiten la cuantificación de las concentraciones de citocinas en gotas a lo largo del tiempo. De manera ejemplar, la concentración promedio de IL-6 en gotas alcanzó una meseta después de 90 min para PBMC estimulada por LPS, mientras que la concentración promedio de IL-1β en gotas aumentó más rápidamente a partir de 90 min, mostrando la resolución dinámica de la plataforma y la posibilidad de extraer subpoblaciones celulares que secretan citocinas específicas (Figura 4C). A medida que la concentración cambia entre los puntos de medición, es posible calcular las tasas de secreción dinámica por citocina. Teniendo en cuenta la tasa de secreción promedio para cada citocina (Figura 4D), las células secretoras de IL-6 mostraron una disminución constante en la tasa de secreción promedio, mientras que las células secretoras de TNFα e IL-1β mostraron un aumento en la tasa de secreción después de 90 minutos de tiempo de medición y una segunda disminución después de 150 minutos.
Además, es posible agrupar las células en subpoblaciones en función de las citocinas secretadas y cosecretadas (Figura 4E). Aquí, la IL-6 y el TNFα son secretadas por el 30,2% y el 26,4% de las células que secretan IL-6 o TNFα, respectivamente, mientras que las células de IL-1β secretoras únicas constituyeron el 68,8% de todas las células secretoras de IL-1β. Además, se pueden resolver los efectos de la cosecreción sobre las concentraciones secretadas y las tasas de secreción (Figura 4F). Al observar las células secretoras de IL-6, se secretaron diferentes cantidades de IL-6 si las células producían adicionalmente TNFα o IL-1β. De manera similar, la distribución de las tasas de secreción promediadas durante la medición difirió estadísticamente entre las células que secretaron solo IL-6 o IL-6 junto con TNFα (tasas de secreción más altas) e IL-1β (tasas de secreción de IL-6 más bajas).
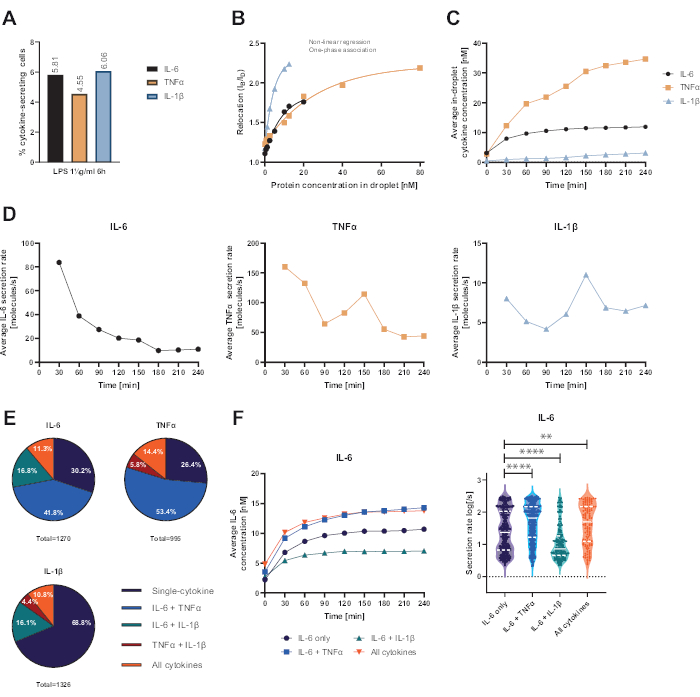
Figura 4: Resultados representativos de IL-6, TNFα e IL-1β secretando PBMC después de 6 h de estimulación con 1 μg/mL de LPS. (A) Porcentaje de PBMC secretor de IL-6, TNFα e IL-1β al final de la medición de 4 h. (B) Las curvas de calibración de citocinas multiplexadas se generan con concentraciones conocidas de citocinas recombinantes. Esto permite la cuantificación de experimentos celulares calculando a partir del valor de reubicación la concentración de citoquinas dentro de la gota. Los puntos se ajustaron mediante un ajuste de curva de asociación monofásico no lineal, r2=0,9926 (IL-6), 0,9901 (TNFα), 0,9990 (IL-1β). (C) Concentraciones medias secretadas de IL-6, TNFα e IL-1β liberadas por la secreción de PBMC durante el tiempo de medición de 4 h. (D) Tasas medias de secreción de IL-6, TNFα e IL-1β durante el tiempo de medición de 4 h. (E) Porcentaje relativo de células co-secretoras que secretan IL-6, TNFα o IL-1β y combinaciones de las mismas. Normalizado a todas las células secretoras detectadas para cada citocina. (F) Concentraciones promediadas de IL-6 durante el tiempo de medición y las distribuciones de tasa de secreción promedio (log) para células secretoras de IL-6 con resolución de co-secreción (n = 383 para IL-6 solamente, n = 531 para IL-6 + TNFα, n = 213 para IL-6 + IL-1β y n = 143 para IL-6 + TNFα + IL-1β). Las diferencias estadísticas en las distribuciones de las tasas de secreción se evaluaron mediante pruebas de Kolmogorov-Smirnov bilaterales, no pareadas, no paramétricas con un 95% de confianza, se representan los valores p. ** (p <0,002) y **** (p <0,0001). La línea completa representa la mediana y la línea punteada los cuartiles. n celdas totales = 21 866. Haga clic aquí para ver una versión más grande de esta figura.
Para extraer información adicional a nivel de una sola célula, se puede ajustar una función sigmoide a los puntos de concentración-tiempo de cada célula y citocina (Figura 5). En la Figura 5A se muestra un conjunto de datos ejemplares de concentración en el tiempo para una celda y el ajuste sigmoidal correspondiente. Aquí, el procedimiento de ajuste de mínimos cuadrados produce los siguientes parámetros: C, que corresponde al valor de la meseta superior de la curva, t50 que cuantifica el desplazamiento en el tiempo de la curva desde cero, y la pendiente de Hill m, que describe la pendiente de la parte ascendente de la curva sigmoidea con valores de concentración del 10% y el 90% alcanzados a lo largo de la medición. A partir de estos parámetros de ajuste, se pueden extraer algunos descriptores de curva como se explica en el paso 7.12. produciendo el Cmax, el valor de concentración más alto de los datos, tstart, el tiempo de inicio de la secreción, definido como alcanzar el 10% del valor de concentración de la meseta superior, y SRlin, la tasa de secreción durante la parte ascendente de la curva.
Para clasificar las subpoblaciones celulares, los descriptores de la curva obtenidos de todos los ajustes de una sola célula se clasificaron en tres categorías cada uno: los valores de Cmax se agruparon en bajo, medio y alto para tinicio a temprano, medio y tardío y SRlin en secretores lentos, medios y rápidos. Para ilustrar esta clasificación, se muestran cuatro curvas de secreción de una sola célula ejemplares y sus correspondientes descriptores de curva (Figura 5A-D), donde la curva A exhibe las características de un secretor bajo temprano de tasa media, la curva B es un secretor temprano, lento y alto, la curva C un secretor alto rápido temprano y la curva D muestra una secreción baja tardía. Es importante tener en cuenta que los límites para estos criterios son específicos de cada célula, citocina y parámetro de ensayo, y deben adaptarse a cada pregunta de investigación. Además, solo se consideró la secreción de IL-6 de PBMC después de la estimulación de LPS de 1 μg/mL durante 6 h, lo que significa que la mayoría de las células fueron secretoras tempranas y altas con un 80% y un 79%, respectivamente (Figura 5E-F). En cuanto a la tasa de secreción, se observó una respuesta bipolar con un 55% de las células secretoras de IL-6 que son secretoras lentas y un 39% que son secretoras rápidas (Figura 5G).
Para caracterizar aún más el comportamiento de la secreción, se trazaron los descriptores de la curva para cada célula entre sí y se extrajeron diferentes grupos (Figura 5H-J). No se da una correlación clara entre elinicio de t y el Cmáx (Figura 5H): las dos poblaciones más grandes fueron secretores bajos tempranos y secretores altos independientes del inicio de la secreción. Teniendo en cuenta la relación entre elinicio de t y laSR lin (Figura 5I), la mayoría de las células fueron secretoras lentas tempranas con una clara población de secredores altos tempranos y pocos secretores lentos/medianos a tardíos. Con respecto a las correlaciones entre SRlin y Cmax (Figura 5J), casi no hubo secretores rápidos bajos a medios, con solo una población mayor de secretores bajos rápidos. Además, había una gran población de secretores rápidos que no dependían de la concentración máxima medida, y dos poblaciones de secretores altos secretaban lento o rápido. En resumen, se puede concluir que la investigación de la relación entre los descriptores de la curva para células individuales produce un análisis mucho más detallado y potencialmente puede extraer nuevos hallazgos biológicos a partir de mediciones de secreción de una sola célula.
Con el análisis presentado anteriormente, extrajimos la dinámica de secreción de las células co-secretoras (Figura 6). Dos curvas de ejemplo muestran diferentes dinámicas de co-secreción de IL-6 y TNFα de dos células individuales con un inicio simultáneo de ambas citocinas (Figura 6A), o un inicio de secreción secuencial, con IL-6 secretada primero (Figura 6B). Para clasificar todas las células co-secretoras, se definió un retraso de secreción de 60 min, donde todas las células que inician la secreción dentro de este rango se consideran secretoras simultáneas y todas las células con retrasos más largos se consideran secretoras secuenciales. Este análisis también permitió observar qué citocina se secretó primero. Para IL-6 y TNFα, se observó principalmente cosecreción simultánea en el 76% de las células (Figura 6C), mientras que para IL-6 e IL-1β se observó cosecreción secuencial en el 86% de las células, siendo IL-6 la primera citocina secretada en la mayoría de los casos (Figura 6D).
Al observar el momento de inicio de la secreción de las diferentes citocinas para todas las células cosecretoras individuales, no se observó una correlación clara entre los tiempos de inicio de la secreción en los experimentos realizados. Para la co-secreción de IL-6 y TNFα (Figura 6E), se presentó un grupo vertical más grande alrededor de 0 min, correspondiente a las células co-secretoras más prevalentes a partir de IL-6. Para la cosecreción de IL-6 e IL-1β (Figura 6F), la mayoría de las células comenzaron a secretar IL-6 alrededor del inicio de la medición, mientras que la IL-1β se secretó principalmente más tarde. En resumen, el análisis aquí presentado permitió la identificación de diferentes subpoblaciones secretoras y dinámicas complejas de cosecreción de citocinas.
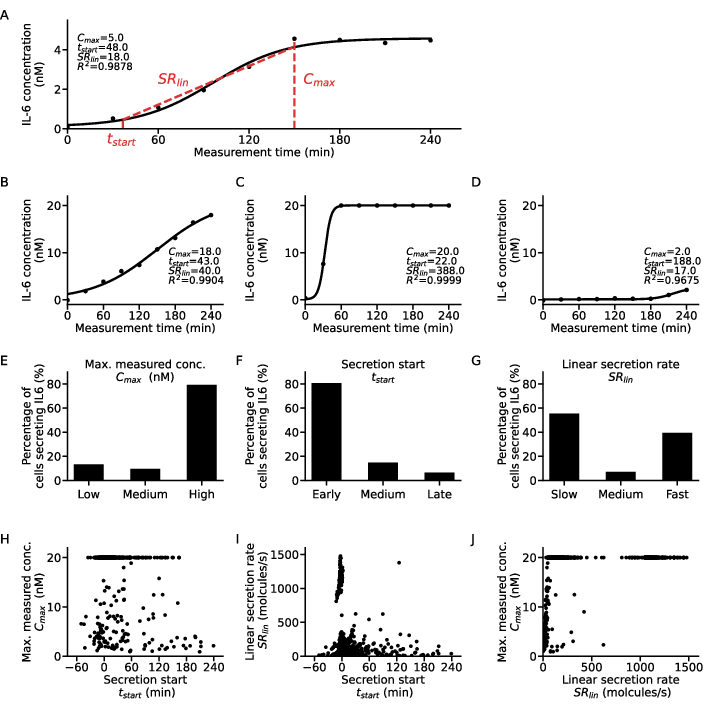
Figura 5: Análisis detallado de diferentes patrones dinámicos de secreción para curvas de células secretoras de IL-6 individuales. (A) Datos representativos de la concentración de citocinas de una sola célula durante el tiempo de medición con la curva sigmoide ajustada y los parámetros extraídos. (B-D) Tres curvas de concentración de citocinas unicelulares ejemplares para los diferentes tipos de secretores de citocinas encontrados para la secreción de IL-6 después de la estimulación con LPS. (E-G) Porcentajes de células secretoras de IL-6 que se clasifican en los diferentes tipos de secretores con los siguientes criterios (n=633): E. Cmax: bajo <5 nM, alto >19,5 nM, F.t inicio: temprano <30 min, tarde >120min, G. SRlin: lento <250 moléculas/s, rápido >750 moléculas/s. (H-J) Relación entre los tres descriptores de la curva de secreción Cmáx., tstart y SRlin para cada celda individual (n = 633). La gran población a Cmáx. = 20 nM es el resultado de alcanzar el límite superior de detección del ensayo. Haga clic aquí para ver una versión más grande de esta figura.
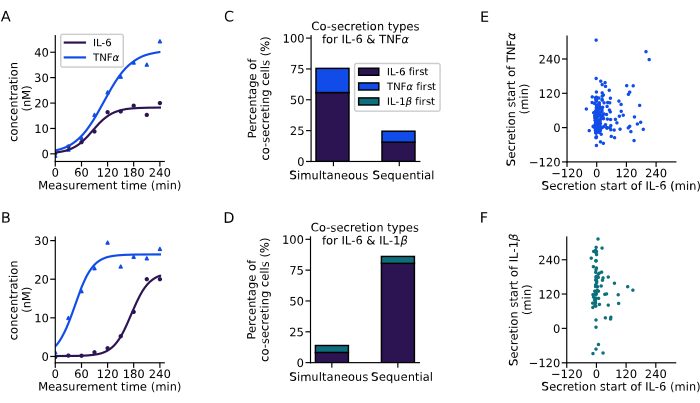
Figura 6: Extracción de patrones de cosecreción a partir de curvas de concentración de una sola célula. (A-B) Curvas de concentración representativas para células individuales que cosecretan IL-6 y TNFα (A) simultáneamente y (B) secuencialmente, respectivamente. (C-D) Porcentaje de células que presentan cosecreción simultánea y secuencial de IL-6 y TNFα (n = 249), o IL-6 e IL-1β (n = 72), respectivamente. La secreción secuencial se define a través del retraso entre el inicio de la secreción de citocinas de más de 60 min. Los colores indican cuál de las citocinas comenzó primero la secreción. (E-F) Relación entre los tiempos de inicio de la secreción de las diferentes citocinas para cada célula secretora (nIL6-TNFα=249, nIL6-IL1β=72). Haga clic aquí para ver una versión más grande de esta figura.
Discusión
La liberación y secreción de citocinas se investigan con frecuencia en inmunología y medicina clínica3. La secreción desequilibrada de citoquinas puede provocar efectos perjudiciales para los pacientes que padecen infecciones, pero también en enfermedades neurológicas, inflamación o cáncer 26,27,28. A pesar de que su importancia en la salud y la enfermedad está bien establecida, el estudio de las citocinas y sus células secretoras sigue siendo un desafío, ya que las metodologías actuales no son capaces de detectar y cuantificar con precisión las citocinas que se originan en una sola célula de una manera resuelta en el tiempo. Para el flujo de trabajo que aquí se presenta, se utilizó un protocolo de estimulación establecido con PBMC y se midió su secreción de IL-6, TNF-α e IL-1β. La elección de utilizar PBMCs en lugar de subpoblaciones individuales purificadas surgió de la aplicación previa para investigar los síndromes de liberación de citocinas (SRC)23, una condición caracterizada por concentraciones plasmáticas muy elevadas de citocinas proinflamatorias, incluyendo IL-6, TNF-α e IL-1β29. Dado que el SRC no suele estar ligado a una sola población, utilizamos las PBMC ya que estarían presentes in vivo. Sin embargo, las subpoblaciones celulares pueden ser purificadas y evaluadas individualmente, si la cuestión científica lo exige. El tiempo de incubación, las condiciones de estimulación y los rangos de ensayos dinámicos se optimizaron para medir la secreción de las tres citocinas de interés. El flujo de trabajo y los datos presentados aquí demuestran cómo configurar, calibrar, cuantificar, medir y analizar la secreción de múltiples citocinas en una sola célula resuelta en el tiempo. Este protocolo proporciona un modelo sobre cómo el análisis multifuncional de la secreción de citocinas podría permitir la gran diversidad funcional y dinámica de las citocinas secretadas en los pacientes.
Varios aspectos cruciales del protocolo de ensayo descrito permiten una lectura biológica única. En primer lugar, la encapsulación de una sola célula en gotas microfluídicas permitió la extracción de datos de cada célula individual. Los eventos de múltiples encapsulaciones de celdas se pueden detectar y clasificar mediante análisis de imágenes, dependiendo de la pregunta de investigación. En segundo lugar, la inclusión de varios inmunoensayos fluorescentes independientes en gotas y la alineación de las nanopartículas funcionalizadas permitieron la medición cuantitativa de hasta tres concentraciones de citocinas en paralelo. Esta multiplexación permitió el análisis de los patrones de cosecreción de citocinas a nivel de una sola célula. En tercer lugar, la inmovilización de las gotas permitió la medición y correlación temporal de la secreción de citocinas para cada célula secretora y permitió distinguir la secreción simultánea de la secuencial. La resolución temporal proporcionó datos de forma única sobre los patrones de secreción y las subpoblaciones de diferentes tipos de secretores. Por último, el análisis de imágenes paralelizadas permitió la extracción y el seguimiento eficientes de grandes cantidades de datos de mediciones con más de 20.000 células individuales. La extracción a partir de curvas de secreción individuales permitió además el descubrimiento de subpoblaciones fenotípicas y funcionalidades.
Además de su lectura única, el ensayo también tiene ventajas técnicas sobre el análisis de citocinas estándar. Gracias al pequeño tamaño de los compartimentos de encapsulación de alrededor de 60 pL, se pueden detectar cantidades absolutas de citoquinas secretadas directamente desde la fuente biológica con límites de detección adecuados para la secreción celular. La miniaturización del ensayo también utiliza cantidades más pequeñas de biorreactivos costosos. Además, la instalación requiere muy poco equipo especializado, que a menudo ya está disponible en los laboratorios de biología y bioingeniería. Los microscopios de fluorescencia están ampliamente disponibles, y las bombas de jeringa se usan con frecuencia en los laboratorios de bioingeniería o se pueden comprar a un costo relativamente bajo. Si hay un cultivo celular, el coste del equipo completo necesario para llevar a cabo los experimentos es de unos 148.000 euros, la mayor parte aportados por el microscopio epifluorescente automatizado (130.000 euros). Sin embargo, un instrumento de este tipo se puede encontrar a menudo en los laboratorios biológicos, y el resto del coste se distribuye entre la bomba de jeringa (13.000 euros, pero hay alternativas más baratas disponibles) y equipos más pequeños. La fabricación del chip de gotas y de la cámara de observación está muy bien descrita17 y puede realizarse fuera de un entorno de sala limpia con la infraestructura necesaria, como hornos y limpiadores de plasma presentes en la mayoría de los laboratorios de bioingeniería. Alternativamente, hay diferentes proveedores disponibles para suministrar a los laboratorios interesados chips generadores de gotas. Debido a los pequeños volúmenes necesarios, el ensayo es rentable y fácil de configurar.
Para garantizar el más alto grado de reproducibilidad, identificamos algunos pasos críticos para el éxito del protocolo. Un problema común para los usuarios primerizos es el movimiento de las gotas durante la medición. Si bien el software de análisis puede rastrear gotas individuales hasta cierto punto, el movimiento excesivo conduce a la pérdida de resolución de una sola célula y resultados inexactos. El movimiento se puede evitar mediante el uso de cámaras de medición herméticas adecuadas, el tamaño de gota y el tamaño de la cámara correctos, un breve período de equilibrio antes de comenzar la medición y una concentración adecuada de surfactante. Otro paso crítico es el enfoque preciso antes de comenzar la medición. Un enfoque inadecuado conduce a valores de reubicación de fluorescencia significativamente más bajos y a la subestimación de la cantidad de citocinas secretadas. Por último, dependiendo de la pregunta y el protocolo en cuestión, el tiempo correcto entre los diferentes pasos es de suma importancia para la reproducibilidad. Especialmente, el tiempo de espera entre el llenado de la cámara y el inicio de la medición debe ser constante, de lo contrario, se podría perder la ventana de medición de las citocinas secretadas.
Las limitaciones de la tecnología presentada incluyen la capacidad restringida de manipular aún más las células después de la encapsulación. Por lo tanto, actualmente no es posible agregar o eliminar estimulantes, anticuerpos o reactivos adicionales. Además, dado que las células están encapsuladas en su biorreactor aislado, no pueden producirse interacciones entre las células (señales paracrinas o basadas en contacto) durante la medición. Esta limitación puede superarse parcialmente con incubaciones a granel previas. Además, también es posible aumentar los efectos autocrinos de las citocinas secretadas y estos efectos no se pueden cuantificar o excluir con certeza, ya que solo se miden las citocinas secretadas detectadas por anticuerpos. Por lo tanto, la visión aislada sobre la secreción de citocinas siempre debe describirse en el contexto de la pregunta y la aplicación correspondientes. Sin embargo, esta limitación también podría utilizarse para el estudio detallado de múltiplos encapsulados, dobletes y tripletes si es de interés. Esto proporcionaría una configuración interesante útil para investigar el contacto célula-célula o preguntas basadas en paracrinos. Por último, también el rango dinámico del ensayo es limitado y necesita adaptación a la aplicación específica. Aquí, hemos adaptado el rango dinámico del ensayo a la cantidad secretada esperada de las citocinas medidas.
Para avanzar aún más en las capacidades y aplicabilidad del ensayo, se podrían abordar varios desarrollos en el futuro, en aspectos biológicos, técnicos y de análisis de datos. Desde el punto de vista biológico, la medición de citocinas adicionales, otras proteínas secretadas, marcadores metabólicos o de superficie celular podría integrarse adaptando el ensayo. Además, este ensayo podría integrarse en un flujo de trabajo junto con otros ensayos basados en células para ampliar las lecturas (por ejemplo, tinción o secuenciación por citometría de flujo). Además, la usabilidad del ensayo podría simplificarse, por ejemplo, mediante la creación de un chip microfluídico integrado para la creación y observación de gotas, lo que podría permitir una aplicación más amplia fuera de los laboratorios de bioingeniería en un entorno clínico. En cuanto al análisis de datos, la extracción y el seguimiento de la información de las imágenes podrían ampliarse mejorando la automatización y utilizando enfoques de aprendizaje automático, por ejemplo, para detectar la presencia y la posición de la(s) célula(s) y la línea de perlas en cada gota sin etiquetado fluorescente. De este modo, se abrirían canales fluorescentes adicionales que podrían utilizarse para inmunoensayos, lo que daría lugar a la medición de aún más citocinas en paralelo.
El ensayo presentado y los protocolos y análisis asociados se pueden aplicar a diversos casos de uso potenciales relacionados con la dinámica de la secreción de citocinas. Más específicamente, el ensayo podría abordar cuestiones inmunológicas fundamentales, como la identificación del tipo de célula y los perfiles de secreción de citocinas específicos de la activación, la polifuncionalidad de las células secretoras de citocinas o la temporalidad y los mecanismos de mantenimiento de los equilibrios de citocinas. Además, en aplicaciones clínicas, la plataforma podría permitir desentrañar el papel de las citocinas durante las respuestas inflamatorias activas o crónicas, como se observó en COVID-1930, o proporcionar una herramienta para estratificar a los pacientes y personalizar los tratamientos basados en firmas únicas, como en la autoinflamación31. En conclusión, la evaluación cuantitativa resuelta en el tiempo de la secreción de citocinas de células individuales es un método muy necesario, ya que dilucida cómo un fármaco en particular, una infección, una alteración genética o una estimulación ex vivo induce una respuesta particular.
Divulgaciones
Se han patentado aspectos específicos como las medidas de las células.
Agradecimientos
Este proyecto contó con el apoyo de la subvención #2021-349 del Área de Enfoque Estratégico Salud Personalizada y Tecnologías Relacionadas (PHRT) del Dominio ETH (Institutos Federales Suizos de Tecnología), la subvención inicial del Consejo Europeo de Investigación (subvención #803,336) y la Fundación Nacional Suiza para la Ciencia (subvención #310030_197619). Además, agradecemos a Guilhem Chenon y Jean Baudry por su trabajo y desarrollo del analizador DropMap inicial.
Materiales
| Name | Company | Catalog Number | Comments |
| 008-FluoroSurfactant | RAN Biotechnologies | 008-FluoroSurfactant-10G | |
| 2-Stream flow-focusing droplet maker, 30 µm nozzle, PFOS hydrophobic surface treatment | Wunderli chips | ||
| Alexa Fluor 647 NHS Ester | ThermoFisher | A20006 | https://www.thermofisher.com/ch/en/home/references/protocols/cell-and-tissue-analysis/labeling-chemistry-protocols/fluorescent-amine-reactive-alexa-fluor-dye-labeling-of-igm-antibodies.html |
| Anti-Human IL-1β (Monoclonal Mouse), AF647 labelled in-house | PeproTech | 500-M01B | |
| ARcare92524 double-sided adhesive tape | Adhesvies Reasearch | ARcare92524 | |
| Bio-Adembeads Streptavidin plus 300nm | Ademtech | Cat#03233 | |
| Biotinylated Goat Anti-Human IL-1β | PeproTech | 500-P21BGBT | |
| Bovine Serum Albumin (BSA) | Sigma-Aldrich | A3059 | |
| Cell Scraper | TPP | 99002 | |
| CellTrace Violet Cell Proliferation Kit | Invitrogen | C34557 | Cell staining solution |
| Chromafil Xtra PTFE-45/25 syringe filters | Macherey-Nagel | 729205 | |
| Costar 6-well Clear Flat Bottom Ultra-Low Attachment | Corning | 3471 | |
| Countess Cell Counting Chamber Slides | Invitrogen | C10283 | |
| D-Biotin | Fluorochem | M02926 | |
| DPBS, no calcium, no magnesium | Gibco | 14196-094 | |
| epT.I.P.S. Standard 2-200 µl | Eppendorf | 30000889 | |
| Ethylenediaminetetraacetic acid disodium salt solution | Sigma-Aldrich | 3690 | |
| EZ-LINK-NHS-PEG4-Biotin | ThermoFisher | A39259 | https://www.thermofisher.com/order/catalog/product/20217 |
| FcR Blocking Reagent, human | Miltenyi Biotec | 130-059-901 | |
| Fetal Bovine Serum | Gibco | 10270-106 | |
| Handy dish soap | Migros | 5.01002E+11 | |
| HEPES (1 M) | Gibco | 15630-080 | |
| HFE-7500 Oil 3M TM Novec | Fluorochem | B40045191 | |
| Idex F-120 Fingertight One-Piece Fitting, Standard Knurl, Natural PEEK, 1/16" OD Tubing, 10-32 Coned | Cole-Parmer | GZ-02014-15 | |
| IL-6 Monoclonal Antibody (MQ2-13A5 - Rat), FITC | ThermoFisher | 11-7069-81 | |
| IL-6 Monoclonal Antibody (MQ2-39C3), Biotin | ThermoFisher | 13-7068-85 | |
| KnockOut Serum Replacement | ThermoFisher | 10828-010 | |
| Loctite AA 3491 curable UV glue | Henkel AG & Co | 3491 | |
| Microscope slides (76x26x1mm, clear white) | Menzel Gläser | ||
| Mineral oil light | Sigma-Aldrich | 330779 | |
| NanoPort Assembly Headless, 10-32 Coned, for 1/16" OD | Idex | N-333 | |
| Neodymium block magnet | K&J Magnetics | BZX082 | |
| Omnifix-F Spritze, 1 ml, LS | Braun | 9161406V | |
| Penicillin-Streptomycin (10,000 U/mL) | Gibco | 15140-122 | |
| Phosphate buffered saline | Sigma-Aldrich | P4417 | |
| Pluronic F-127, 0.2 µm filtered (10% Solution in Water) | ThermoFisher | P6866 | |
| Precision wipes | Kimtech Science | 5511 | |
| PTFE microtubing 0.30 × 0.76 mm | FisherScientific | 1191-9445 | |
| PTFE microtubing 0.56 × 1.07 mm | FisherScientific | 1192-9445 | |
| Recombinant Human IL-1β | Peprotech | Cat#200-01B | |
| Recombinant Human IL-6 | Peprotech | Cat#200-06 | |
| Recombinant human serum albumine (HSA) | Sigma-Aldrich | A9731 | |
| Recombinant Human TNF-α | Peprotech | Cat#300-01A | |
| Reusable biopsy punch diameter 0.75 mm and 6 mm | Stiefel | 504529 and 504532 | |
| RPMI 1640 Medium, no phenol red | Gibco | 11835-030 | |
| Standard LPS, E. coli K12 | InvivoGen | tlrl-eklps | |
| Sterican needles 23 G for 0.56 mm diameter microtubing | FisherScientific | 15351547 | |
| Sterican needles 27 G for 0.30mm diameter microtubing | FisherScientific | 15341557 | |
| TNF alpha Monoclonal Antibody (MAb11), PE | ThermoFisher | 12-7349-81 | |
| TNF-alpha Monoclonal Antibody (MAb1), biotinylated in-house | ThermoFisher | 14-7348-85 | |
| Trypan Blue Stain (0.4%) for use with the Countess Automated Cell Counter | Invitrogen | T10282 | |
| Vacuum Filtration "rapid"-Filtermax | TPP | 99500 | |
| Devices | |||
| Cameo 4 automatic cutting machine | Silhouette | ||
| Cetoni Base 120 + 3x NEMESYS Low Pressure Syringe Pumps | Cetoni | NEM-B101-03 A | |
| Countess II Automated Cell Counter | ThermoFisher | ||
| Inverted Epi-fluorescence microscope Ti2 | Nikon | ECLIPSE Ti2-E, Ti2-E/B*1 | |
| OKO Lab Cage Incubator, dark panels | OKO Lab | ||
| ORCA-Fusion Digital CMOS camera | Hammatsu | C14440 | |
| SOLA Light Engine | Lumencor | sola 80-10247 |
Referencias
- Chen, L., et al. Inflammatory responses and inflammation-associated diseases in organs. Oncotarget. 9 (6), 7204-7218 (2017).
- Cicchese, J. M., et al. Dynamic balance of pro- and anti-inflammatory signals controls disease and limits pathology. Immunol Rev. 285 (1), 147-167 (2018).
- Liu, C., et al. Cytokines: From clinical significance to quantification. Adv Sci. 8 (15), e2004433 (2021).
- Rojas, J. M., Avia, M., Martín, V., Sevilla, N. IL-10: A multifunctional cytokine in viral infections. J Immunol Res. 2017, 6104054 (2017).
- Kohanawa, Y. M. A regulatory effect of the balance between TNF-α and IL-6 in the granulomatous and inflammatory response to Rhodococcus aurantiacus infection in mice. J Immunol. 177 (1), 642-650 (2006).
- Geginat, J., et al. Plasticity of human CD4 T cell subsets. Front Immunol. 5, 630 (2014).
- Sallusto, F. Heterogeneity of human CD4+ T cells against microbes. Ann Rev Immunol. 34 (1), 317-334 (2016).
- Chetaille Nézondet, A. L., Poubelle, P. E., Pelletier, M. The evaluation of cytokines to help establish diagnosis and guide treatment of autoinflammatory and autoimmune diseases. J Leukocyte Biol. 108 (2), 647-657 (2020).
- Sims, J. T., et al. Characterization of the cytokine storm reflects hyperinflammatory endothelial dysfunction in COVID-19. J Allergy Clin Immunol. 147 (1), 107-111 (2021).
- Yasen, A., et al. Single-cell RNA sequencing reveals the heterogeneity of infiltrating immune cell profiles in the hepatic cystic echinococcosis microenvironment. Infection and Immunity. 89 (12), (2021).
- Jiang, Y., et al. Single-cell RNA sequencing highlights intratumor heterogeneity and intercellular network featured in adamantinomatous craniopharyngioma. Sci Adv. 9 (15), (2023).
- Tanguay, S., Killion, J. J. Direct comparison of ELISPOT and ELISA-based assays for detection of individual cytokine-secreting cells. Lymphokine Cytokine Res. 13 (4), 259-263 (1994).
- Bucheli, O. T. M., Sigvaldadóttir, I., Eyer, K. Measuring single-cell protein secretion in immunology: Technologies, advances, and applications. Eur J Immunol. 51 (6), 1334-1347 (2021).
- Brower, K. K., et al. Double emulsion flow cytometry with high-throughput single droplet isolation and nucleic acid recovery. Lab Chip. 20 (12), 2062-2074 (2020).
- Brower, K. K., et al. Double emulsion picoreactors for high-throughput single-cell encapsulation and phenotyping via FACS. Anal Chem. 92 (19), 13262-13270 (2020).
- Luo, X., Chen, J. Y., Ataei, M., Lee, A. Microfluidic compartmentalization platforms for single cell analysis. Biosensors. 12 (2), 58 (2022).
- Bounab, Y., et al. Dynamic single-cell phenotyping of immune cells using the microfluidic platform DropMap. Nat Protoc. 15 (9), 2920-2955 (2020).
- Eyer, K., et al. Single-cell deep phenotyping of IgG-secreting cells for high-resolution immune monitoring. Nat Biotechnol. 35 (10), 977-982 (2017).
- Gaa, R., et al. Versatile and rapid microfluidics-assisted antibody discovery. mAbs. 13 (1), 198130 (2021).
- Gerard, A., et al. High-throughput single-cell activity-based screening and sequencing of antibodies using droplet microfluidics. Nat Biotechnol. 38 (6), 715-721 (2020).
- De Jonghe, J., et al. spinDrop: a droplet microfluidic platform to maximise single-cell sequencing information content. Nat Comm. 14 (1), 4788 (2023).
- Wheeler, M. A., et al. Droplet-based forward genetic screening of astrocyte-microglia cross-talk. Science. 379 (6636), 1023-1030 (2023).
- Portmann, K., Linder, A., Oelgarth, N., Eyer, K. Single-cell deep phenotyping of cytokine release unmasks stimulation-specific biological signatures and distinct secretion dynamics. Cell Rep Meth. 3 (7), 100502 (2023).
- Portmann, K., Linder, A., Eyer, K. Stimulation-induced cytokine polyfunctionality as a dynamic concept. eLife. 12, 89781 (2023).
- Armbruster, D. A., Pry, T. Limit of blank, limit of detection and limit of quantitation. Clin Biochem Rev. 29, S49-S52 (2008).
- Yang, J., et al. New insight into neurological degeneration: Inflammatory cytokines and blood-brain barrier. Front Mol Neurosci. 15, 1013933 (2022).
- Kim, P. S., Ahmed, R. Features of responding T cells in cancer and chronic infection. Curr Opin Immunol. 22 (2), 223-230 (2010).
- Becher, B., Spath, S., Goverman, J. Cytokine networks in neuroinflammation. Nat Rev Immunol. 17 (1), 49-59 (2017).
- Cosenza, M., Sacchi, S., Pozzi, S. Cytokine release syndrome associated with T-cell-based therapies for hematological malignancies: Pathophysiology, clinical presentation, and treatment. Int J Mol Sci. 22 (14), 7652 (2021).
- Hu, B., Huang, S., Yin, L. The cytokine storm and COVID-19. J Med Virol. 93 (1), 250-256 (2021).
- Marcuzzi, A., et al. Autoinflammatory diseases and cytokine storms-Imbalances of innate and adaptative immunity. Int J Mol Sci. 22 (20), 11241 (2021).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados