Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Estudio de la conectividad metabólica del cerebro mediante tomografía dinámica por emisión de positrones con 2-desoxi-2-[18F]fluoro-D-glucosa a nivel de un solo sujeto
En este artículo
Resumen
La adquisición de datos de tomografía dinámica por emisión de positrones (PET) y su reconstrucción en marcos de tiempo permite el análisis de la conectividad metabólica del cerebro a nivel de un solo sujeto. Describimos un método para adquirir datos PET dinámicos de [18F]FDG del cerebro de rata y obtener una matriz de conectividad a través de la extracción de curvas de tiempo-actividad de volúmenes de interés.
Resumen
Hasta el día de hoy, la conectividad metabólica del cerebro se estudia principalmente a nivel grupal a través de la adquisición de datos de tomografía por emisión de positrones estática (PET) de múltiples sujetos. Actualmente, nuestros grupos de investigación están estudiando los cambios en la conectividad metabólica a través de múltiples puntos temporales después de una hemorragia intracerebral a nivel intrasujeto en ratas. Para investigar la conectividad metabólica del cerebro dentro del sujeto, se requiere información temporal de la absorción del marcador en diferentes regiones del cerebro, lo que se puede lograr a través de la PET dinámica. En esta publicación, damos una descripción detallada de nuestro protocolo de adquisición y análisis de datos.
Los datos PET dinámicos del cerebro de rata se adquieren en un sistema PET preclínico dedicado que utiliza 2-desoxi-2-[18F]fluoro-D-glucosa ([18F]FDG) como trazador. El marcador se inyecta por vía intravenosa en forma de bolo al inicio de la TEP. Durante los 60 minutos de adquisición, los animales son sedados con medetomidina.
Después de la adquisición, los datos PET se reconstruyen en treinta marcos de tiempo de 2 minutos utilizando un algoritmo de reconstrucción iterativo (Maximum-Likelihood Expectation-Maximization). Se utiliza un atlas parcelado que consta de múltiples volúmenes de interés (VOI) para extraer curvas de tiempo-actividad de cada VOI, que luego se utilizan para calcular el coeficiente de correlación de Pearson entre cada par de VOI.
Este protocolo PET dinámico permite la evaluación de las diferencias de conectividad metabólica entre dos exploraciones individuales, en lugar de entre grupos de exploraciones. Este enfoque permite el estudio de los cambios en la conectividad metabólica dentro de un solo sujeto a través de diferentes puntos de tiempo, o para la comparación de la conectividad metabólica de un individuo con una base de datos normal. Tales comparaciones podrían ser útiles para rastrear la progresión de la enfermedad o ayudar en el diagnóstico de trastornos neurológicos caracterizados por la interrupción de la comunicación entre las regiones del cerebro, como la epilepsia o la demencia.
Introducción
La tomografía por emisión de positrones (PET) es una técnica de imagen molecular comúnmente utilizada en la investigación y en entornos clínicos. Debido al desarrollo de varios trazadores PET, el PET se puede utilizar para estudiar la fisiopatología de la enfermedad y monitorear la progresión de la enfermedad y la respuesta a los tratamientos1. Uno de los radiotrazadores más utilizados es el 2-desoxi-2-[18F]fluoro-D-glucosa ([18F]FDG), que permite obtener imágenes del metabolismo de la glucosa, indicativo de la activación celular. Se utiliza en oncología para el diagnóstico, la estadificación y el pronóstico; en neurología, comúnmente en el contexto de enfermedades neurodegenerativas como la demencia; y en cardiología, para diagnosticar enfermedades como la sarcoidosis, por citar solo algunos ejemplos 1,2,3.
La evaluación de la conectividad metabólica cerebral, obtenida a partir de los datos de [18F]FDG PET, se refiere a las relaciones funcionales entre la absorción de trazadores en diferentes regiones del cerebro. Este enfoque permite el cálculo de una "matriz de conectividad" mediante la selección de un conjunto de regiones cerebrales, que pueden proporcionar información sobre cómo interactúan y funcionan juntas las diferentes partes del cerebro. Este tipo de análisis es particularmente útil para estudiar la función cerebral en la salud y la enfermedad, incluidas afecciones como la demencia, la epilepsia y otros trastornos neurológicos 4,5.
El primer estudio que evaluó la conectividad metabólica cerebral ya se remonta a la década de 19806, pero los investigadores exploraron principalmente la conectividad estructural del cerebro, también conocida como "conectoma"7, mediante imágenes de resonancia magnética ponderada por difusión (DW-MRI). Además, la conectividad funcional mediante técnicas como la resonancia magnética funcional (fMRI), la electroencefalografía (EEG) y la magnetoencefalografía (MEG) ha sido ampliamente investigada durante varias décadas 8,9.
Recientemente, se ha recuperado el interés por estudiar la conectividad metabólica del cerebro utilizando [18F]FDG PET, no solo por sí solo, sino también en combinación con otras formas de conectividad cerebral10. Sin embargo, debido a la naturaleza "estática" inherente de las imágenes PET (en contraste con, por ejemplo, la resonancia magnética funcional), la gran mayoría de los resultados basados en PET de redes cerebrales se basan en análisis a nivel de grupo, donde las correlaciones entre las regiones cerebrales se calculan a nivel intersujeto. Esta limitación hace imposible un análisis intrasujeto de las imágenes PET, que es esencial para los estudios longitudinales que pueden rastrear los cambios a lo largo del tiempo dentro del mismo individuo4. Por lo tanto, el desarrollo de métodos que permitan el análisis de un solo sujeto, como la conectividad molecular dinámica basada en PET, es una dirección de investigación importante en la investigación del cerebro que investiga los trastornos de la red, ya que abre la puerta al uso del análisis de redes moleculares en la práctica clínica. Por lo tanto, se utilizaron datos dinámicos de PET en nuestro estudio preclínico.
Actualmente, nuestros grupos de investigación están llevando a cabo un estudio que examina los cambios en la conectividad metabólica después de una hemorragia intracerebral a nivel intrasujeto en múltiples puntos temporales utilizando el modelo de colagenasa de rata11. Para investigar la conectividad metabólica del cerebro intrasujeto, se requiere información temporal de la absorción del trazador en diferentes regiones del cerebro, que se puede obtener a través de la PET dinámica. En las siguientes secciones, damos una descripción detallada del protocolo de adquisición y análisis de datos.
Protocolo
Todos los procedimientos están de acuerdo con las directrices europeas (directiva 2010/63/UE), y el protocolo fue aprobado por el Comité de Ética Animal local de la Universidad de Gante (ECD 23/33). Doce ratas Sprague Dawley (seis hembras y seis machos) fueron incluidas en el estudio. Sus tomografías por emisión de positrones se obtuvieron utilizando el siguiente protocolo en múltiples puntos de tiempo, desde 2 semanas antes hasta 18 semanas después de una hemorragia intracerebral inducida. En el momento de la primera exploración, todos los animales tenían 18 semanas de edad y las hembras pesaban 244,8 ± 10,1 g (media ± DE), mientras que los machos pesaban 363,6 ± 13,3 g.
Asegúrese de que los materiales radiactivos solo sean trabajados y manipulados por personal capacitado. Mantenga la dosis administrada al personal y a los animales tan baja como sea razonablemente posible (ALARA).
1. Adquisición de datos
NOTA: Consulte la Tabla de materiales para obtener detalles sobre el generador de imágenes PET preclínico y el software utilizado para la adquisición de datos. El generador de imágenes es un escáner PET de 45 detectores (dispuestos en 5 anillos) que cubre un campo de visión axial (FOV) de 13 cm y un FOV transaxial de 7,6 cm, utilizando cristales LYSO y detectores SiPM. El sistema muestra una resolución espacial de 850 μm, una sensibilidad del 12% y una resolución de energía del 12,6%12. Los siguientes pasos se escribieron con esto en mente.
- Prive de comida a los animales al menos 6 horas antes de la exploración retirando toda la comida de su jaula. Una vez que el animal haya pasado por un período de ayuno suficiente, comience los preparativos para la adquisición de datos de la siguiente manera.
- Encienda una almohadilla térmica y deje que se caliente a 30-35 °C.
- Anestesiar al animal con isoflurano colocando al animal en una cámara de anestesia gaseosa llena de isoflurano al 5% enO2 médico a un caudal de 2.000 mL/min. Ayuda a mantener la temperatura corporal del animal colocando una lámpara calefactora a 30 cm de la cámara. El animal está debidamente anestesiado cuando la frecuencia respiratoria está entre 0,5 y 1 Hz.
- Pesa al animal.
- Coloque al animal sobre la almohadilla térmica. Utilice un cono nasal para administrar isoflurano al 2% en O2 médico a un caudal de 500 mL/min. Ajustar la dosis de isoflurano según sea necesario para mantener al animal correctamente anestesiado.
- Afeita una sección del cuello del animal entre los omóplatos.
- Preparación de la vena de cola para la inyección de trazadores:
- Gire al animal sobre su lado izquierdo o derecho y use la lámpara de calefacción para calentar su cola, causando vasodilatación.
- Tome aproximadamente 20 cm de tubo BTPE-10. Con un portaagujas, rompa el eje de una aguja de 30 G e inserte 1-2 mm de su extremo posterior en el tubo de PE. Llene una jeringa de insulina de 1 ml con solución salina e inserte la punta de la aguja de 3 a 5 mm en el tubo de PE. Llene previamente el tubo de PE con solución salina de la jeringa llena y mantenga la jeringa de solución salina conectada al tubo de PE.
- Limpiar la cola del animal con etanol. Retire la lámpara de calefacción.
- Inserte la punta de la aguja del tubo de PE en una vena de cola lateral. Confirme la colocación correcta de la aguja: busque sangre que ingrese al tubo de PE cuando se retira el émbolo, ausencia de resistencia durante la inyección y formación de ampollas subcutáneas (que indican inyección paravenosa) en el lugar de la inyección. Si la entrada no ha sido exitosa, retire la aguja de la cola y vuelva a intentarlo en una sección más proximal.
- Asegure la aguja con una gota de pegamento instantáneo en el sitio de penetración y pegue el tubo de PE a la cola.
NOTA: Evite el movimiento del tubo de PE. Si en algún momento entra sangre en el tubo de PE, enjuáguelo inmediatamente con solución salina para evitar obstrucciones.
- Cambiar a la sedación con medetomidina:
- Coloque al animal en posición prona sobre la estera calefactora. Pon una gota de gel para ojos en cada ojo para evitar que se sequen.
- Prepare el tubo de PE (40–100 cm) para conectarlo a una bomba de infusión que se coloca cerca del escáner PET. Con un portaagujas, rompa el eje de una aguja de 30 G e inserte 1-2 mm de su dorso en el tubo de PE. Llene una jeringa de plástico de 1 ml con una aguja de 30 g con al menos 0,2 ml de medetomidina (por animal) e inserte la punta de la aguja de 3 a 5 mm en el tubo de PE. Llene previamente el tubo de PE con medetomidina de la jeringa.
- Coloque la jeringa con medetomidina en la bomba de infusión. Encienda la bomba y seleccione la jeringa correcta haciendo clic en Buscar jeringa | BD Plastic | 1 mL.
- Expulse bolos de 0,005 mL con la bomba hasta que una gota emerja de la aguja en el otro extremo del tubo de PE. Asegúrese de que no haya burbujas de aire en el tubo de PE.
- Inserte la aguja del tubo de PE 2-3 mm por vía subcutánea entre los omóplatos del animal. Asegure la aguja con una gota de pegamento instantáneo en el sitio de penetración y pegue suavemente el tubo de PE a la espalda del animal.
- Inyectar un bolo de volumen v de medetomidina (tasa: 3 mL/min) en función del peso del animal:
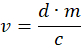
Donde d = 0,05 mg/kg (la dosis de medetomidina en mg por kg de peso animal), m es el peso del animal en kg, y c es la concentración de medetomidina en mg/mL. - Suspender la administración de isoflurano 10 min después de la administración del bolo de medetomidina, ajustando la dosis de isoflurano según sea necesario para mantener una anestesia estable durante este período de espera.
NOTA: El isoflurano combinado con medetomidina disminuye significativamente la frecuencia respiratoria del animal. Es esencial una estrecha vigilancia hasta que cese la administración de isoflurano. - Cinco minutos después de suspender el tratamiento con isoflurano, iniciar una infusión continua de medetomidina a una velocidad de:
rcont = r • m
Donde r = 0,1 mg/(kg·h); la cantidad de medetomidina en mg por peso corporal del animal en kg/h), y m es el peso del animal en kg.
NOTA: Se necesita un período de equilibrio de 25 minutos adicionales después de iniciar la infusión continua (por lo tanto, 30 minutos después de suspender el isoflurano) antes de que se pueda iniciar la exploración para asegurarse de que el sistema del animal está libre de isoflurano. Durante este tiempo, continúe con los siguientes pasos.
- Posicionamiento de los animales en el escáner PET:
- Prepare la cama del escáner con una almohadilla térmica, un sensor de presión y pañuelos de papel. Utilice el sensor para controlar la frecuencia respiratoria del animal y los pañuelos de papel para absorber la orina excretada por el animal.
NOTA: Debido a la medetomidina, el animal suele excretar grandes cantidades de orina. - Transfiera con cuidado al animal a la cama del escáner, asegurándose de que la línea de medetomidina y el catéter de la vena de la cola permanezcan seguros. Coloque la cabeza del animal lo más recta posible usando un cono de nariz para ayudar si es necesario. Asegure la cabeza con cinta adhesiva una vez colocada correctamente.
- Encienda la almohadilla térmica y verifique que el sistema de monitoreo de la frecuencia respiratoria esté funcionando correctamente, ajustando la posición del sensor de presión si es necesario.
- Prepare la cama del escáner con una almohadilla térmica, un sensor de presión y pañuelos de papel. Utilice el sensor para controlar la frecuencia respiratoria del animal y los pañuelos de papel para absorber la orina excretada por el animal.
- De diez a cinco minutos antes de comenzar la TEP, llene una jeringa de insulina con aproximadamente 20 MBq de 2-desoxi-2-[18F]fluoro-D-glucosa ([18F]FDG) diluida en 0,1–0,8 mL de solución salina.
PRECAUCIÓN: El flúor-18 (18F) es un radioisótopo de flúor con una vida media de 109,8 min. Siga los protocolos locales de seguridad radiológica cuando manipule material radiactivo. - Abra el software del escáner y configure los siguientes parámetros de escaneo: Seleccione 18F como isótopo (importante para la corrección de la desintegración), introduzca la actividad medida de la jeringa que contiene el radiotrazador (en MBq medido por el calibrador de dosis) y el tiempo de medición (la información se guardará en los metadatos, que son necesarios para convertir los datos a otras unidades, como los valores de absorción estándar). Seleccione el protocolo General y marque Clasificación automática después de la adquisición. Establezca la duración del escaneo en 1 h y ajuste la región de interés (ROI) para que contenga todo el cerebro de la rata (normalmente posición 2-5).
- Una vez transcurridos 30 min desde la interrupción de la administración de isoflurano, iniciar la PET:
- Una vez más, confirme la colocación correcta en la vena por aspiración seguida de una inyección de una pequeña cantidad de solución salina como se describe en el paso 1.7.4.
- Reemplace la jeringa de solución salina por la jeringa de radiosonda. Al retirar la jeringa del tubo de PE, es posible que la sangre entre en el tubo de PE y expulse la solución salina. Inserte la aguja de la jeringa de radiotrazadora rápidamente sin perforar el tubo de PE.
- Inicie el escaneo con el software del escáner.
- Una vez que comience la adquisición, inyecte el marcador en forma de bolo durante aproximadamente 0,5 a 2 s. Reemplace inmediatamente la jeringa radiosonda vacía por la jeringa de solución salina y enjuague el tubo de PE con aproximadamente 0.1 mL de solución salina. Use un pañuelo desechable para atrapar las gotas de radiosonda que puedan escapar durante el intercambio de jeringas.
NOTA: La tasa de conteo mostrada por el software del escáner debe aumentar rápidamente a 4.5-6.5 Mcps después de la inyección del trazador. El hecho de que la tasa de recuento esté inicialmente significativamente por debajo de 4 Mcps y aumente posteriormente indica una inyección de trazador (parcialmente) paravenosa. - Evalúe la actividad restante que no se inyectó en el animal midiendo la actividad de la jeringa trazadora y de los tejidos y guantes potencialmente contaminados. Introduzca esa actividad y la hora de la medición en el software del escáner para guardarla en los metadatos.
NOTA: Es típica una actividad restante de 0,1 a 1,5 MBq, dependiendo de la cantidad de trazador que se escapó al cambiar el marcador por la jeringa de solución salina.
- Una vez finalizada la exploración, expulse la cama con el animal del escáner.
NOTA: El animal puede reaccionar a los estímulos o despertarse antes de que la medetomidina haya sido antagonizada. Manipule al animal con cuidado y esté preparado para movimientos bruscos durante los siguientes pasos. También hay que tener en cuenta que el animal es radiactivo. Por lo tanto, trate de completar los siguientes pasos lo más rápido y cuidadosamente posible. - Retire la aguja y el tubo de PE de la cola del animal con un soporte para agujas. Aplique un pañuelo de papel o una compresa en el sitio de la punción para detener el posible sangrado.
- Suspenda la administración de medetomidina y retire la aguja y el tubo de PE del cuello del animal.
- Coloque al animal en una jaula libre de otros animales, usando un pañuelo de papel para atrapar la orina (radiactiva) que pueda excretarse mientras mueve al animal. Administrar 1 mL de solución salina por vía subcutánea en su espalda utilizando una jeringa de insulina de 1 mL para promover la rehidratación.
- Revertir la sedación con una inyección subcutánea de atipamezol.
- Calcule el volumen v apropiado utilizando la siguiente ecuación:
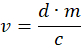
Donde d = 0,5 mg/kg, es decir , la dosis de medetomidina en mg por kg de peso animal, m es el peso del animal en kg y c es la concentración de atipamezol en mg/mL. - Para animales de peso ligero que resultan en pequeños volúmenes calculados, diluya el atipamezol con solución salina para permitir una dosis más precisa. Llene una jeringa de insulina con 4 • v de atipamezol y 12• v de solución salina. Inyecte 1/4 de la solución resultante por vía subcutánea en el lomo del animal, cerca del cuello.
- Calcule el volumen v apropiado utilizando la siguiente ecuación:
- Vigile al animal visualmente hasta que se despierte, lo que debería tardar de 1 a 5 minutos. Una vez que el animal sea capaz de mantenerse erguido y moverse libremente, devuélvalo al alojamiento grupal. Proporciónale al animal comida y agua.
2. Reconstrucción de datos y control de calidad
NOTA: Utilizando el hardware y el software disponibles en nuestro estudio, todos los datos de PET se corrigieron para la desintegración de radionúclidos, y los sinogramas adquiridos se reconstruyeron con el algoritmo tridimensional de maximización de expectativas de subconjuntos ordenados (OSEM-3D) utilizando una ventana del 30% alrededor del fotopico de 511 keV. El software de reconstrucción OSEM utilizó su configuración predeterminada de 10 subconjuntos con 20 millones de eventos por subconjunto. Las imágenes se reconstruyeron en una matriz transversal de 192 x 192 x 384 con vóxeles de imagen cúbica de 0,4 mm. No se realizó ninguna corrección de atenuación.
- Para verificar que la inyección del trazador se realizó correctamente, verifique la tasa de conteo detectada durante la adquisición de 60 minutos (es decir, la curva de tiempo-actividad (TAC)). Confirme que hay un pico dentro del primer minuto de la adquisición, inmediatamente después de que se haya inyectado el trazador.
- Reconstrucción estática: Reconstruya los datos adquiridos utilizando un algoritmo OSEM utilizando 30 iteraciones, un tamaño de vóxel isométrico de 400 μm y una ventana de energía del 30% centrada en el fotopico de 511 keV.
- Una vez completada la reconstrucción, inspeccione la imagen resultante en busca de artefactos.
- Reconstrucción dinámica: Reconstruya los datos adquiridos en marcos de tiempo de treinta y 2 minutos utilizando la misma configuración que para la reconstrucción estática.
3. Análisis de datos
NOTA: Los siguientes pasos 3.1 y 3.2 se realizan en un entorno de software biomédico dedicado a la cuantificación de datos de PET, utilizando la Herramienta de Registro y Fusión de Imágenes (PFUS) y la Herramienta de Modelado Cinético General (PKIN).
- En PFUS:
- En la subpágina Cargar imágenes de entrada:
- Cargue el archivo DICOM de la reconstrucción dinámica utilizando el botón de carga INP o arrastrando y soltando el archivo en el lienzo. Utilice el control de visualización de imagen general en la parte superior derecha para navegar por segmentos y marcos de tiempo y ajustar la barra de colores. Si es necesario, ajuste la orientación de la imagen a la orientación estándar mediante el botón Cambiar la orientación de la imagen de entrada en la barra lateral derecha.
- Recorte y promedio de fotogramas: haga clic en la flecha en la parte inferior derecha para mostrar los controles ocultos para sumar y recortar y marque Recortar. En el lienzo, defina los límites para recortar ajustando los bordes y arrastrando el centro del cuadro de recorte; Asegúrese de que todo el cerebro y un poco de almohadilla, que contiene partes de los ojos y la médula espinal, estén contenidos. Una vez que el cuadro de recorte esté definido correctamente, haga clic en el botón de recorte para recortar la imagen. Calcule el promedio de fotogramas ponderados en el tiempo en todos los fotogramas de tiempo haciendo clic en el botón naranja de promedio y espere a que el resultado se agregue automáticamente a las imágenes de entrada, lo que da como resultado dos imágenes de entrada en total: los datos dinámicos recortados con los marcos de tiempo y el promedio ponderado en el tiempo recortado de los mismos datos.
- Seleccione Deformar como método coincidente o presione el botón si ya está seleccionado para pasar a la normalización basada en plantillas.
- En la subpágina Referencia y coincidencia:
- Seleccione la imagen media ponderada en el tiempo como imagen de entrada (normalmente se selecciona automáticamente desde el último paso).
- Cargue una referencia haciendo clic en el botón para seleccionar una plantilla de normalización como imagen de referencia. Seleccione Rata FDG (W. Schiffer).
- Haga coincidir la entrada con la plantilla haciendo clic en el botón Coincidencia de entrada | Entrada: Ajuste manual en la barra lateral de la derecha. Arrastre y gire la imagen de entrada para que coincida aproximadamente con la plantilla. Asegúrese de que Rata esté seleccionada como especie, el método de registro sea Deformar, luego haga clic en el botón Coincidir con la corriente para llegar a la siguiente subpágina.
- En el resultado coincidente de la subpágina:
- Verifique el resultado y ajuste la coincidencia manualmente si es necesario. Haga clic en Aplicar transformación actual a todo para que coincida también con la imagen de entrada que consta de los fotogramas de tiempo.
- Haga clic en el botón verde VOI para pasar al siguiente paso de procesamiento.
- En la subpágina Análisis de VOI:
- Seleccione la imagen que contiene los marcos de tiempo como imagen A en el menú inferior derecho.
- Vaya a la pestaña Plantilla y seleccione Rata Px (W. Schiffer) en la pestaña Atlas , o cargue un atlas personalizado y seleccione los volúmenes deseados. Para aplicar las VOI, haga clic en el botón Contorno en la parte inferior.
- Haga clic en el botón situado encima de la pestaña Plantilla para calcular los TAC y enviarlos a las herramientas PKIN.
- En la subpágina Cargar imágenes de entrada:
- En PKIN:
- En la parte inferior derecha, seleccione TACs para Display Type (Tipo de visualización ) para visualizar los TACs en kBq/mL de las VOI seleccionadas previamente.
- Haga clic con el botón derecho del ratón en el lienzo con los TAC y haga clic en Tabla de valores de las curvas visibles. Copie la tabla en una hoja de cálculo para su posterior análisis.
- Calcule la correlación de Pearson entre todos los emparejamientos de VOI, utilizando datos desde el sexto hasta el último período de tiempo (= 10-60 min después del inicio de la exploración), para obtener una matriz de correlación. Un script de Python llamado "correlation_matrix.py" que cumple este propósito se incluye en el Archivo Suplementario 1.
- Calcule si la correlación de Pearson es significativa utilizando el estadístico de la prueba t:
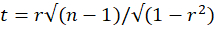 con n - 2 grados de libertad, donde r es el coeficiente de correlación de Pearson y n = 25 el número de marcos de tiempo utilizados para calcular la correlación de Pearson. Aplique una corrección adecuada para comparaciones múltiples y compare el estadístico con el valor crítico de una distribución t de dos colas (p = 0,05) con n - 2 grados de libertad. Considere que la correlación de Pearson entre dos VOI es significativa si el valor de la prueba t corregida es menor que el valor crítico.
con n - 2 grados de libertad, donde r es el coeficiente de correlación de Pearson y n = 25 el número de marcos de tiempo utilizados para calcular la correlación de Pearson. Aplique una corrección adecuada para comparaciones múltiples y compare el estadístico con el valor crítico de una distribución t de dos colas (p = 0,05) con n - 2 grados de libertad. Considere que la correlación de Pearson entre dos VOI es significativa si el valor de la prueba t corregida es menor que el valor crítico.
Resultados
Una vez finalizado el escaneo, se puede investigar el TAC de la tasa detectada durante la adquisición para comprobar la correcta inyección y absorción del trazador. La Figura 1 muestra un TAC resultante de todo el campo de visión del escáner después de una inyección y adquisición exitosa del trazador (panel A), y un TAC resultante después de una inyección de trazador parcialmente paravenosa (panel B). En el caso exitoso, la tasa de...
Discusión
El protocolo proporcionado aquí guía a los usuarios a través del proceso de adquisición de datos PET dinámicos de 1 h utilizando [18F]FDG como trazador en ratas. Al final, se obtiene una matriz de correlación de VOIs, que se puede utilizar para evaluar la conectividad metabólica a nivel de un solo sujeto. Los investigadores experimentados pueden ajustar el protocolo para que se adapte a sus necesidades específicas en varios puntos, por ejemplo, mediante el uso de un ra...
Divulgaciones
Los autores no tienen conflictos de intereses que revelar.
Agradecimientos
Este trabajo fue apoyado por una beca de investigación de la Fundación Flamenca de Investigación [G0A7422N].
Materiales
| Name | Company | Catalog Number | Comments |
| Antisedan | Orion Pharma | Atipamezole hydrochloride 5 mg/mL | |
| BD Micro-Fine+ insulin syringe 1 mL | BD | 324827 | 0.33 mm (29G) x 12.7 mm |
| BD Microlance 3 Needles 30 G x 1/2" | BD | 304000 | 30 G x 1/2"; 0,3 x 13 mm |
| BD Plastipak syringe 1 mL | BD | 303172 | for infusion pump |
| BTPE-10 Polyethylene tubing | Instech | 0.11x.024in (.28x60mm) | |
| Domitor | Orion Pharma | 1070499 | Medetomidine hydrochloride 1 mg/mL |
| Fusion 100 infusion pump | Chemyx Inc. | 07100 | Newer model available: Fusion 100X |
| Isoflutek 1000 mg/g | Alivira | Isoflurane | |
| MOLECUBES β-CUBE with CUBEFLOW software | MOLECUBES NV | Preclinical PET scanner | |
| PMOD Software version 4.4 | Bruker Corporation | http://www.pmod.com; quantification of PET data | |
| Saline | B. Braun | 394496 | NaCl 0.9% |
| Vidisic eye gel | Vidisic | Carbomerum 980 2 mg/g |
Referencias
- Wilson, H., Pagano, G., Politis, M. Dementia spectrum disorders: lessons learnt from decades with PET research. J Neural Transm. 126 (3), 233-251 (2019).
- Singnurkar, A., Poon, R., Metser, U. Comparison of 18F-FDG-PET/CT and 18F-FDG-PET/MR imaging in oncology: a systematic review. Ann Nucl Med. 31 (5), 366-378 (2017).
- Youssef, G., et al. The use of 18F-FDG PET in the diagnosis of cardiac sarcoidosis: a systematic review and metaanalysis including the Ontario experience. J Nucl Med. 53 (2), 241-248 (2012).
- Carli, G., Tondo, G., Boccalini, C., Perani, D. Brain molecular connectivity in neurodegenerative conditions. Brain Sci. 11 (4), 433 (2021).
- Sala, A., Perani, D. Brain molecular connectivity in neurodegenerative diseases: Recent advances and new perspectives using positron emission tomography. Front Neurosci. 13, 617 (2019).
- Horwitz, B., Duara, R., Rapoport, S. I. Intercorrelations of glucose metabolic rates between brain regions: Application to healthy males in a state of reduced sensory input. J Cereb Blood Flow Metab. 4 (4), 484-499 (1984).
- Sporns, O., Tononi, G., Kötter, R. The human connectome: A structural description of the human brain. PLOS Comput Biol. 1 (4), e42 (2005).
- Biswal, B., Zerrin Yetkin, F., Haughton, V. M., Hyde, J. S. Functional connectivity in the motor cortex of resting human brain using echo-planar MRI. Magn Reson Med. 34 (4), 537-541 (1995).
- Sanchez-Catasus, C. A., Diercke, R. A. J. O., de Vries, E. F. J., van Waarde, A., Leenders, K. L., et al. Use of nuclear medicine molecular neuroimaging to model brain molecular connectivity. PET and SPECT in neurology. , 181-207 (2021).
- Wehrl, H. F., et al. Simultaneous PET-MRI reveals brain function in activated and resting state on metabolic, hemodynamic and multiple temporal scales. Nat Med. 19 (9), 1184-1189 (2013).
- Rosenberg, G. A., Mun-Bryce, S., Wesley, M., Kornfeld, M. Collagenase-induced intracerebral hemorrhage in rats. Stroke. 21 (5), 801-807 (1990).
- Krishnamoorthy, S., et al. Performance evaluation of the MOLECUBES β-CUBE - a high spatial resolution and high sensitivity small animal PET scanner utilizing monolithic LYSO scintillation detectors. Phys Med Biol. 63 (15), 155013 (2018).
- Schiffer, W. K., Mirrione, M. M., Dewey, S. L. Optimizing experimental protocols for quantitative behavioral imaging with 18F-FDG in rodents. J Nucl Med. 48 (2), 277-287 (2007).
- Fueger, B. J., et al. Impact of animal handling on the results of 18F-FDG PET studies in mice. J Nucl Med. 47 (6), 999-1006 (2006).
- Gold, M. E. L., Norell, M. A., Budassi, M., Vaska, P., Schulz, D. Rapid 18F-FDG uptake in brain of awake, behaving rat and anesthetized chicken has implications for behavioral PET studies in species with high metabolisms. Front Behav Neurosci. 12, 115 (2018).
- Miranda, A., et al. PET imaging of freely moving interacting rats. NeuroImage. 191, 560-567 (2019).
- Miranda, A., et al. Awake 18F-FDG PET imaging of memantine-induced brain activation and test-retest in freely running mice. J Nucl Med. 60 (6), 844-850 (2019).
- Jahreis, I., Bascuñana, P., Ross, T. L., Bankstahl, J. P., Bankstahl, M. Choice of anesthesia and data analysis method strongly increases sensitivity of 18F-FDG PET imaging during experimental epileptogenesis. PLOS ONE. 16 (11), e0260482 (2021).
- Pawela, C. P., et al. A protocol for use of medetomidine anesthesia in rats for extended studies using task-induced BOLD contrast and resting-state functional connectivity. NeuroImage. 46 (4), 1137-1147 (2009).
- Rubinov, M., Sporns, O. Complex network measures of brain connectivity: Uses and interpretations. NeuroImage. 52 (3), 1059-1069 (2010).
- Engel, J., et al. Connectomics and epilepsy. Curr Opin Neurol. 26 (2), 186 (2013).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoThis article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados