Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Untersuchung der metabolischen Konnektivität des Gehirns mit Hilfe der dynamischen Positronen-Emissions-Tomographie von 2-Desoxy-2-[18F]Fluoro-D-Glukose auf Einzelprobenebene
In diesem Artikel
Zusammenfassung
Die Erfassung von Daten der dynamischen Positronen-Emissions-Tomographie (PET) und die Rekonstruktion in Zeitrahmen ermöglicht die Analyse der metabolischen Hirnkonnektivität auf der Ebene eines einzelnen Probanden. Wir beschreiben eine Methode zur Erfassung von [18F]FDG-dynamischen PET-Daten des Rattengehirns und erhalten eine Konnektivitätsmatrix durch die Extraktion von Zeit-Aktivitäts-Kurven der interessierenden Volumina.
Zusammenfassung
Bis heute wird die metabolische Konnektivität des Gehirns meist auf Gruppenebene durch die Erfassung von statischen Positronen-Emissions-Tomographie (PET)-Daten mehrerer Probanden untersucht. Unsere Forschungsgruppen untersuchen derzeit Veränderungen der metabolischen Konnektivität über mehrere Zeitpunkte nach einer intrasubjektiven Blutung bei Ratten. Um die intrasubjektive metabolische Konnektivität des Gehirns zu untersuchen, werden zeitliche Informationen über die Traceraufnahme in verschiedenen Hirnregionen benötigt, die durch dynamische PET erreicht werden können. In dieser Veröffentlichung geben wir eine detaillierte Beschreibung unseres Datenerfassungs- und Analyseprotokolls.
Dynamische PET-Daten des Rattengehirns werden mit einem speziellen präklinischen PET-System unter Verwendung von 2-Desoxy-2-[18F]fluor-D-glucose ([18F]FDG) als Tracer erfasst. Der Tracer wird zu Beginn des PET-Scans als Bolus intravenös injiziert. Während der 60-minütigen Einnahme werden die Tiere mit Medetomidin sediert.
Nach der Erfassung werden die PET-Daten mit Hilfe eines iterativen Rekonstruktionsalgorithmus (Maximum-Likelihood Expectation-Maximization) in Zeitrahmen von 32 Minuten rekonstruiert. Ein parzellierter Atlas, der aus mehreren interessierenden Volumina (VOIs) besteht, wird verwendet, um Zeit-Aktivitäts-Kurven jedes VOI zu extrahieren, die dann zur Berechnung des Pearson-Korrelationskoeffizienten zwischen den einzelnen VOI-Paaren verwendet werden.
Dieses dynamische PET-Protokoll ermöglicht die Beurteilung von Unterschieden in der metabolischen Konnektivität zwischen zwei einzelnen Scans und nicht zwischen Gruppen von Scans. Dieser Ansatz ermöglicht die Untersuchung von Veränderungen der metabolischen Konnektivität innerhalb eines einzelnen Probanden über verschiedene Zeitpunkte hinweg oder den Vergleich der metabolischen Konnektivität einer Person mit einer normalen Datenbank. Solche Vergleiche könnten nützlich sein, um das Fortschreiten der Krankheit zu verfolgen oder die Diagnose von neurologischen Störungen zu unterstützen, die durch eine gestörte Kommunikation zwischen Gehirnregionen gekennzeichnet sind, wie Epilepsie oder Demenz.
Einleitung
Die Positronen-Emissions-Tomographie (PET) ist ein molekulares Bildgebungsverfahren, das sowohl in der Forschung als auch im klinischen Umfeld eingesetzt wird. Aufgrund der Entwicklung verschiedener PET-Tracer kann PET zur Untersuchung der Pathophysiologie von Krankheiten und zur Überwachung des Krankheitsverlaufs und des Ansprechens auf Behandlungen verwendet werden1. Einer der am weitesten verbreiteten Radiotracer ist 2-Desoxy-2-[18F]fluor-D-Glukose ([18F]FDG), der die Abbildung des Glukosestoffwechsels ermöglicht, was auf eine zelluläre Aktivierung hinweist. Es wird in der Onkologie zur Diagnose, zum Staging und zur Prognose eingesetzt. in der Neurologie, häufig im Zusammenhang mit neurodegenerativen Erkrankungen wie Demenz; und in der Kardiologie, um Erkrankungen wie Sarkoidose zu diagnostizieren, um nur einige Beispielezu nennen 1,2,3.
Die Bewertung der metabolischen Konnektivität des Gehirns, die aus [18F]FDG PET-Daten gewonnen wurde, bezieht sich auf die funktionellen Beziehungen zwischen der Traceraufnahme in verschiedenen Hirnregionen. Dieser Ansatz ermöglicht die Berechnung einer "Konnektivitätsmatrix", indem eine Reihe von Gehirnregionen ausgewählt wird, die Aufschluss darüber geben, wie verschiedene Teile des Gehirns interagieren und zusammenarbeiten. Diese Art der Analyse ist besonders nützlich für die Untersuchung der Gehirnfunktion bei Gesundheit und Krankheit, einschließlich Erkrankungen wie Demenz, Epilepsie und anderen neurologischen Störungen 4,5.
Die erste Studie zur Erfassung der metabolischen Konnektivität des Gehirns stammt bereits aus den 1980er Jahren6, aber die Forscher untersuchten die strukturelle Konnektivität des Gehirns, die auch als "Konnektom" bezeichnet wird7, hauptsächlich mit Hilfe der diffusionsgewichteten Magnetresonanztomographie (DW-MRT). Darüber hinaus wird die funktionelle Konnektivität mit Techniken wie funktioneller MRT (fMRT), Elektroenzephalographie (EEG) und Magnetenzephalographie (MEG) seit mehreren Jahrzehnten umfassend untersucht 8,9.
In jüngster Zeit gibt es ein wiedergewonnenes Interesse an der Untersuchung der metabolischen Hirnkonnektivität mit [18F]FDG PET, nicht nur allein, sondern auch in Kombination mit anderen Formen der Gehirnkonnektivität10. Aufgrund der inhärenten "statischen" Natur von PET-Bildern (im Gegensatz zu z.B. funktioneller MRT) basiert die überwiegende Mehrheit der Ergebnisse von Gehirnnetzwerk-PET jedoch auf Analysen auf Gruppenebene, bei denen Korrelationen zwischen Gehirnregionen auf Intersubjektebene berechnet werden. Diese Einschränkung macht eine Analyse von PET-Bildern innerhalb des Probanden unmöglich, was für Längsschnittstudien unerlässlich ist, die Veränderungen im Laufe der Zeit innerhalb derselben Person verfolgen können4. Daher ist die Entwicklung von Methoden, die eine Einzelsubjektanalyse ermöglichen, wie z.B. die dynamische PET-basierte molekulare Konnektivität, eine wichtige Forschungsrichtung in der Hirnforschung zur Erforschung von Netzwerkstörungen, da sie die Tür für den Einsatz der molekularen Netzwerkanalyse in der klinischen Praxis öffnet. Daher wurden in unserer präklinischen Studie dynamische PET-Daten verwendet.
Unsere Forschungsgruppen führen derzeit eine Studie durch, in der Veränderungen der metabolischen Konnektivität nach einer intrasubjektiven Blutung über mehrere Zeitpunkte hinweg mit dem Ratten-Kollagenase-Modell11 untersucht werden. Um die intrasubjektive metabolische Konnektivität des Gehirns zu untersuchen, werden zeitliche Informationen über die Traceraufnahme in verschiedenen Hirnregionen benötigt, die durch dynamische PET gewonnen werden können. In den folgenden Abschnitten geben wir eine detaillierte Beschreibung des Datenerfassungs- und Analyseprotokolls.
Protokoll
Alle Verfahren entsprechen den europäischen Richtlinien (Richtlinie 2010/63/EU), und das Protokoll wurde von der lokalen Tierethischen Kommission der Universität Gent (ECD 23/33) genehmigt. Zwölf Sprague Dawley-Ratten (sechs weiblich, sechs männlich) wurden in die Studie eingeschlossen. Ihre PET-Scans wurden unter Verwendung des folgenden Protokolls zu mehreren Zeitpunkten von 2 Wochen vor bis 18 Wochen nach einer induzierten intrazerebralen Blutung durchgeführt. Zum Zeitpunkt der ersten Untersuchung waren alle Tiere 18 Wochen alt und die Weibchen wogen 244,8 ± 10,1 g (mittlere ± SD), während die Männchen 363,6 ± 13,3 g wogen.
Stellen Sie sicher, dass radioaktive Materialien nur von geschultem Personal bearbeitet und gehandhabt werden. Halten Sie die Dosis für Personal und Tiere so niedrig wie vernünftigerweise erreichbar (ALARA).
1. Datenerfassung
HINWEIS: In der Materialtabelle finden Sie Einzelheiten zum präklinischen PET-Imager und zur Software, die für die Datenerfassung verwendet werden. Der Imager ist ein PET-Scanner mit 45 Detektoren (angeordnet in 5 Ringen), der ein axiales Sichtfeld (FOV) von 13 cm und ein transaxiales FOV von 7,6 cm abdeckt und LYSO-Kristalle und SiPM-Detektoren verwendet. Das System zeigt eine räumliche Auflösung von 850 μm, eine Empfindlichkeit von 12 % und eine Energieauflösung von 12,6 %12. Die folgenden Schritte wurden vor diesem Hintergrund geschrieben.
- Entziehen Sie den Tieren mindestens 6 Stunden vor dem Scannen das Futter, indem Sie sämtliches Futter aus dem Käfig nehmen. Sobald das Tier eine ausreichende Fastenzeit durchlaufen hat, beginnen Sie wie folgt mit den Vorbereitungen für die Datenerfassung.
- Schalten Sie ein Heizkissen ein und lassen Sie es auf 30–35 °C erwärmen.
- Betäuben Sie das Tier mit Isofluran, indem Sie das Tier in eine Gasanästhesiekammer legen, die mit 5 % Isofluran in medizinischem O2 bei einer Flussrate von 2.000 mL/min gefüllt ist. Helfen Sie dabei, die Körpertemperatur des Tieres zu halten, indem Sie eine Heizlampe 30 cm von der Kammer entfernt aufstellen. Das Tier wird richtig betäubt, wenn die Atemfrequenz bei 0,5–1 Hz liegt.
- Wiegen Sie das Tier.
- Lege das Tier auf das Heizkissen. Verwenden Sie einen Nasenkonus, um 2% Isofluran in medizinischem O2 bei einer Flussrate von 500 mL/min zu verabreichen. Passen Sie die Isofluran-Dosis nach Bedarf an, um das Tier ordnungsgemäß zu betäuben.
- Rasieren Sie einen Teil des Halses des Tieres zwischen den Schulterblättern.
- Vorbereitung der Schwanzvene für die Tracer-Injektion:
- Drehen Sie das Tier auf die linke oder rechte Seite und erwärmen Sie mit der Heizlampe den Schwanz, was zu einer Vasodilatation führt.
- Nehmen Sie ca. 20 cm BTPE-10-Schlauch. Brechen Sie mit einem Nadelhalter den Schaft einer 30 G Nadel ab und stecken Sie 1–2 mm des hinteren Endes in den PE-Schlauch. Füllen Sie eine 1-ml-Insulinspritze mit Kochsalzlösung und führen Sie die Nadelspitze 3–5 mm in den PE-Schlauch ein. Füllen Sie den PE-Schlauch mit Kochsalzlösung aus der gefüllten Spritze vor und lassen Sie die Kochsalzspritze mit dem PE-Schlauch verbunden.
- Reinigen Sie den Schwanz des Tieres mit Ethanol. Entfernen Sie die Heizlampe.
- Führen Sie die Nadelspitze des PE-Schlauchs in eine seitliche Schwanzvene ein. Vergewissern Sie sich, dass die Nadel richtig platziert ist: Achten Sie darauf, dass Blut in den PE-Schlauch eindringt, wenn der Kolben zurückgezogen wird, dass es während der Injektion keinen Widerstand gibt und dass sich an der Injektionsstelle eine subkutane Blase bildet (was auf eine paravenöse Injektion hinweist). Wenn der Einstieg nicht erfolgreich war, entfernen Sie die Nadel aus dem Schwanz und versuchen Sie es erneut an einem proximalen Abschnitt.
- Befestigen Sie die Nadel mit einem Tropfen Sekundenkleber an der Einstichstelle und kleben Sie den PE-Schlauch an den Schwanz.
HINWEIS: Vermeiden Sie Bewegungen des PE-Schlauchs. Wenn zu irgendeinem Zeitpunkt Blut in den PE-Schlauch eindringt, spülen Sie ihn sofort mit Kochsalzlösung aus, um ein Verstopfen zu vermeiden.
- Umstellung auf Medetomidin-Sedierung:
- Legen Sie das Tier in Bauchlage auf die Heizmatte. Geben Sie einen Tropfen Augengel auf jedes Auge, um ein Austrocknen zu verhindern.
- Bereiten Sie einen PE-Schlauch (40–100 cm) vor, der an eine Infusionspumpe angeschlossen werden soll, die in der Nähe des PET-Scanners platziert ist. Brechen Sie mit einem Nadelhalter den Schaft einer 30 G Nadel ab und stecken Sie 1–2 mm der Rückseite in den PE-Schlauch. Füllen Sie eine 1-ml-Kunststoffspritze mit einer 30-g-Nadel mit mindestens 0,2 mL Medetomidin (pro Tier) und führen Sie die Nadelspitze 3–5 mm in den PE-Schlauch ein. Füllen Sie den PE-Schlauch mit Medetomidin aus der Spritze vor.
- Legen Sie die Spritze mit Medetomidin in die Infusionspumpe. Schalten Sie die Pumpe ein und wählen Sie die richtige Spritze aus, indem Sie auf Spritze suchen | BD Kunststoff | 1 ml.
- Stoßen Sie Boli von 0,005 ml mit der Pumpe aus, bis ein Tropfen aus der Nadel am anderen Ende des PE-Schlauchs austritt. Stellen Sie sicher, dass keine Luftblasen in den PE-Schläuchen vorhanden sind.
- Führen Sie die Nadel des PE-Schlauches 2–3 mm subkutan zwischen die Schulterblätter des Tieres ein. Befestigen Sie die Nadel mit einem Tropfen Sekundenkleber an der Einstichstelle und kleben Sie den PE-Schlauch vorsichtig auf den Rücken des Tieres.
- Injizieren Sie einen Bolus des Volumens v von Medetomidin (Rate: 3 ml/min) basierend auf dem Gewicht des Tieres:
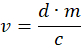
Dabei ist d = 0,05 mg/kg (die Dosis von Medetomidin in mg pro kg Tiergewicht), m ist das Gewicht des Tieres in kg und c ist die Konzentration von Medetomidin in mg/ml. - Beenden Sie die Verabreichung von Isofluran 10 Minuten nach der Verabreichung des Medetomidin-Bolus und passen Sie die Isofluran-Dosis nach Bedarf an, um während dieser Wartezeit eine stabile Anästhesie aufrechtzuerhalten.
HINWEIS: Isofluran in Kombination mit Medetomidin verlangsamt die Atemfrequenz des Tieres erheblich. Eine engmaschige Überwachung ist unerlässlich, bis die Verabreichung von Isofluran eingestellt wird. - Fünf Minuten nach dem Absetzen von Isofluran beginnen Sie eine kontinuierliche Infusion von Medetomidin mit einer Geschwindigkeit von:
rcont = r • m
Dabei ist r = 0,1 mg/(kg·h); Die Menge an Medetomidin in mg pro Körpergewicht des Tieres in kg/h), und m ist das Gewicht des Tieres in kg.
HINWEIS: Eine Äquilibrierungsphase von weiteren 25 Minuten nach Beginn der kontinuierlichen Infusion (also 30 Minuten nach Absetzen von Isofluran) ist erforderlich, bevor der Scan eingeleitet werden kann, um sicherzustellen, dass das System des Tieres frei von Isofluran ist. Fahren Sie während dieser Zeit mit den folgenden Schritten fort.
- Positionierung der Tiere im PET-Scanner:
- Bereiten Sie das Scannerbett mit einem Heizkissen, einem Drucksensor und Papiertaschentüchern vor. Verwenden Sie den Sensor, um die Atemfrequenz des Tieres zu überwachen, und die Papiertücher zur Aufnahme des vom Tier ausgeschiedenen Urins.
HINWEIS: Aufgrund des Medetomidins scheidet das Tier in der Regel große Mengen Urin aus. - Bringen Sie das Tier vorsichtig auf das Scannerbett und stellen Sie sicher, dass die Medetomidinleitung und der Schwanzvenenkatheter sicher bleiben. Positionieren Sie den Kopf des Tieres so gerade wie möglich und verwenden Sie bei Bedarf einen Nasenkonus zur Unterstützung. Sichern Sie den Kopf mit Klebeband, sobald er richtig positioniert ist.
- Schalten Sie das Heizkissen ein und überprüfen Sie, ob das Atemfrequenzüberwachungssystem ordnungsgemäß funktioniert, und passen Sie bei Bedarf die Positionierung des Drucksensors an.
- Bereiten Sie das Scannerbett mit einem Heizkissen, einem Drucksensor und Papiertaschentüchern vor. Verwenden Sie den Sensor, um die Atemfrequenz des Tieres zu überwachen, und die Papiertücher zur Aufnahme des vom Tier ausgeschiedenen Urins.
- Füllen Sie eine Insulinspritze zehn bis fünf Minuten vor Beginn des PET-Scans mit ca. 20 MBq 2-Desoxy-2-[18F]fluor-D-glucose ([18F]FDG), verdünnt in 0,1–0,8 mL Kochsalzlösung.
ACHTUNG: Fluor-18 (18F) ist ein Fluor-Radioisotop mit einer Halbwertszeit von 109,8 min. Befolgen Sie beim Umgang mit radioaktivem Material die örtlichen Strahlenschutzprotokolle. - Öffnen Sie die Scanner-Software und konfigurieren Sie die folgenden Scan-Parameter: Wählen Sie 18F als Isotop (wichtig für die Zerfallskorrektur), geben Sie die gemessene Aktivität der Spritze mit dem Radiotracer (in MBq , gemessen mit dem Dosiskalibrator) und den Zeitpunkt der Messung ein (die Informationen werden in den Metadaten gespeichert, die zur Umrechnung der Daten in andere Einheiten, wie z. B. Standardaufnahmewerte, benötigt werden). Wählen Sie das Protokoll Allgemein und aktivieren Sie Automatische Sortierung nach der Erfassung. Legen Sie die Scandauer auf 1 h fest und passen Sie den Bereich of Interest (ROI) so an, dass er das gesamte Rattengehirn enthält (in der Regel Position 2-5).
- Sobald 30 Minuten seit dem Stopp der Isofluran-Verabreichung vergangen sind, starten Sie den PET-Scan:
- Auch hier ist die korrekte Platzierung in der Vene durch Aspiration und anschließende Injektion einer kleinen Menge Kochsalzlösung zu bestätigen, wie in Schritt 1.7.4 beschrieben.
- Ersetzen Sie die Kochsalzspritze durch die Radiotracer-Spritze. Beim Entfernen der Spritze aus dem PE-Schlauch ist es möglich, dass Blut in den PE-Schlauch eindringt und Kochsalzlösung verdrängt. Führen Sie die Nadel der Radiotracer-Spritze schnell ein, ohne den PE-Schlauch zu durchstechen.
- Starten Sie den Scan mit der Scanner-Software.
- Sobald die Aufnahme beginnt, injizieren Sie den Tracer als Bolus über etwa 0,5–2 s. Ersetzen Sie sofort die leere Radiotracer-Spritze durch die Kochsalzspritze und spülen Sie den PE-Schlauch mit ca. 0,1 mL Kochsalzlösung. Verwenden Sie ein Taschentuch, um alle Tropfen Radiotracer aufzufangen, die während des Spritzenwechsels austreten könnten.
HINWEIS: Die von der Scannersoftware angezeigte Zählrate sollte nach der Tracerinjektion schnell auf 4,5 bis 6,5 Mcps ansteigen. Die anfänglich deutlich unter 4 Mcps liegende und danach ansteigende Zählrate deutet auf eine (teilweise) paravenöse Tracerinjektion hin. - Beurteilen Sie die verbleibende Aktivität, die dem Tier nicht injiziert wurde, indem Sie die Aktivität der Prüfspritze und des potenziell kontaminierten Gewebes und der Handschuhe messen. Geben Sie diese Aktivität und den Zeitpunkt der Messung in die Scannersoftware ein, um sie in den Metadaten zu speichern.
HINWEIS: Typisch ist eine verbleibende Aktivität von 0,1–1,5 MBq, abhängig davon, wie viel Tracer beim Wechsel des Tracers gegen die Kochsalzspritze entwichen ist.
- Sobald der Scan abgeschlossen ist, werfen Sie das Bett mit dem Tier aus dem Scanner aus.
HINWEIS: Das Tier kann auf Reize reagieren oder aufwachen, bevor Medetomidin antagonisiert wurde. Gehen Sie vorsichtig mit dem Tier um und seien Sie bei den folgenden Schritten auf plötzliche Bewegungen vorbereitet. Beachten Sie auch, dass das Tier radioaktiv ist. Versuchen Sie daher, die folgenden Schritte so schnell und sorgfältig wie möglich durchzuführen. - Entfernen Sie die Nadel und den PE-Schlauch mit einem Nadelhalter vom Schwanz des Tieres. Legen Sie ein Taschentuch oder eine Kompresse auf die Einstichstelle, um mögliche Blutungen zu stoppen.
- Beenden Sie die Verabreichung von Medetomidin und entfernen Sie die Nadel und den PE-Schlauch vom Hals des Tieres.
- Setzen Sie das Tier in einen Käfig, der frei von anderen Tieren ist, und verwenden Sie ein Taschentuch, um (radioaktiven) Urin aufzufangen, der beim Bewegen des Tieres ausgeschieden werden kann. Verabreichen Sie 1 ml Kochsalzlösung subkutan in den Rücken mit einer 1 ml Insulinspritze, um die Rehydratation zu fördern.
- Kehren Sie die Sedierung mit einer subkutanen Injektion von Atipamezol um.
- Berechnen Sie das entsprechende Volumen v mit der folgenden Gleichung:
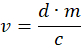
Dabei ist d = 0,5 mg/kg, d. h. die Dosis von Medetomidin in mg pro kg Tiergewicht, m ist das Gewicht des Tieres in kg und c ist die Konzentration von Atipamezol in mg/ml. - Bei leichten Tieren, die zu kleinen berechneten Volumina führen, verdünnen Sie das Atipamezol mit Kochsalzlösung, um eine genauere Dosierung zu ermöglichen. Füllen Sie eine Insulinspritze mit 4 V Atipamezol und 12 V Kochsalzlösung. Injizieren Sie 1/4 der resultierenden Lösung subkutan in den Rücken des Tieres in der Nähe des Halses.
- Berechnen Sie das entsprechende Volumen v mit der folgenden Gleichung:
- Überwachen Sie das Tier visuell, bis es aufwacht, was 1-5 Minuten dauern sollte. Sobald das Tier in der Lage ist, sich aufrecht zu halten und sich frei zu bewegen, bringen Sie es in die Gruppenhaltung zurück. Versorgen Sie das Tier mit Futter und Wasser.
2. Datenrekonstruktion und Qualitätsprüfung
HINWEIS: Unter Verwendung der in unserer Studie verfügbaren Hard- und Software wurden alle PET-Daten um den Radionuklidzerfall korrigiert, und die erworbenen Sinogramme wurden mit dem 3-dimensionalen (OSEM-3D) Algorithmus zur Maximierung der erwarteten Teilmenge (OSEM-3D) unter Verwendung eines 30%-Fensters um den 511 keV-Photopeak rekonstruiert. Die OSEM-Rekonstruktionssoftware verwendete ihre Standardeinstellungen von 10 Teilmengen mit 20 Millionen Ereignissen pro Teilmenge. Die Bilder wurden in eine 192 x 192 x 384 große Quermatrix mit kubischen Bildvoxeln von 0,4 mm rekonstruiert. Es wurde keine Dämpfungskorrektur durchgeführt.
- Um zu überprüfen, ob die Tracer-Injektion erfolgreich war, überprüfen Sie die während der 60-minütigen Erfassung erkannte Zählrate (d. h. die Zeit-Aktivitäts-Kurve (TAC)). Vergewissern Sie sich, dass innerhalb der ersten Minute nach der Erfassung, unmittelbar nach der Injektion des Tracers, ein Peak auftritt.
- Statische Rekonstruktion: Rekonstruieren Sie die erfassten Daten mit einem OSEM-Algorithmus unter Verwendung von 30 Iterationen, einer isometrischen Voxelgröße von 400 μm und einem Energiefenster von 30%, das auf dem 511 keV-Photopeak zentriert ist.
- Sobald die Rekonstruktion abgeschlossen ist, untersuchen Sie das resultierende Bild auf Artefakte.
- Dynamische Rekonstruktion: Rekonstruieren Sie die erfassten Daten in Zeitrahmen von 32 Minuten mit den gleichen Einstellungen wie bei der statischen Rekonstruktion.
3. Datenanalyse
HINWEIS: Die folgenden Schritte 3.1 und 3.2 werden in einer biomedizinischen Softwareumgebung für die Quantifizierung von PET-Daten unter Verwendung des Image Registration and Fusion Tool (PFUS) und des General Kinetic Modeling Tool (PKIN) durchgeführt.
- Bei PFUS:
- Auf der Unterseite Eingabebilder laden:
- Laden Sie die DICOM-Datei der dynamischen Rekonstruktion, indem Sie entweder die INP-Ladetaste verwenden oder die Datei per Drag & Drop auf die Leinwand ziehen. Verwenden Sie das Steuerelement Allgemeine Bildanzeige oben rechts, um durch Segmente und Zeitrahmen zu navigieren und die Farbleiste anzupassen. Passen Sie bei Bedarf die Bildausrichtung auf die Standardausrichtung an, indem Sie die Schaltfläche Eingabebildausrichtung ändern in der rechten Seitenleiste verwenden.
- Zuschneiden und Rahmendurchschnitt: Klicken Sie auf den Pfeil rechts unten, um die ausgeblendeten Steuerelemente für das Summieren und Zuschneiden anzuzeigen, und aktivieren Sie Zuschneiden. Definieren Sie im Canvas die Grenzen für den Zuschnitt, indem Sie die Kanten anpassen und die Mitte des Zuschneidefelds ziehen. Stellen Sie sicher, dass das gesamte Gehirn und ein wenig Polsterung, die Teile der Augen und des Rückenmarks enthält, enthalten sind. Sobald das Zuschneidefeld richtig definiert ist, klicken Sie auf die Schaltfläche zum Zuschneiden , um das Bild zuzuschneiden. Berechnen Sie den Durchschnitt der zeitgewichteten Frames über alle Zeitrahmen, indem Sie auf die orangefarbene Schaltfläche "Durchschnitt" klicken, und warten Sie, bis das Ergebnis automatisch zu den Eingabebildern hinzugefügt wird, was zu insgesamt zwei Eingabebildern führt: den zugeschnittenen dynamischen Daten mit den Zeitrahmen und dem zugeschnittenen zeitgewichteten Durchschnitt derselben Daten.
- Wählen Sie Verformen als Abgleichsmethode aus, oder drücken Sie die Schaltfläche, wenn diese bereits ausgewählt ist, um mit der vorlagenbasierten Normalisierung fortzufahren.
- Auf der Unterseite Referenz & Matching:
- Wählen Sie das zeitgewichtete Durchschnittsbild als Eingabebild aus (es wird normalerweise automatisch aus dem letzten Schritt ausgewählt).
- Laden Sie eine Referenz, indem Sie auf die Schaltfläche klicken, um eine Normalisierungsvorlage als Referenzbild auszuwählen. Wählen Sie Rat FDG (W. Schiffer).
- Ordnen Sie die Eingabe der Vorlage zu, indem Sie auf die Schaltfläche Eingabeabgleich | Eingabe: Manuell in der Seitenleiste rechts anpassen. Ziehen und drehen Sie das Eingabebild so, dass es ungefähr der Vorlage entspricht. Stellen Sie sicher, dass Ratte als Spezies ausgewählt ist, die Registrierungsmethode ist Verformung, und klicken Sie dann auf die Schaltfläche Aktueller Übereinstimmung , um zur nächsten Unterseite zu gelangen.
- Auf der Unterseite Matching-Ergebnis:
- Überprüfen Sie das Ergebnis und passen Sie die Übereinstimmung bei Bedarf manuell an. Klicken Sie auf Aktuelle Transformation auf alle anwenden , um auch das Eingabebild abzugleichen, das aus den Zeitrahmen besteht.
- Klicken Sie auf die grüne Schaltfläche VOIs , um zum nächsten Verarbeitungsschritt zu gelangen.
- Auf der Unterseite VOI-Analyse:
- Wählen Sie das Bild, das die Zeitrahmen enthält, als Bild A im Menü unten rechts aus.
- Gehen Sie zur Registerkarte Vorlage und wählen Sie auf der Registerkarte Atlas die Option Px Rat (W. Schiffer) aus, oder laden Sie einen benutzerdefinierten Atlas und wählen Sie die gewünschten Volumina aus. Um die VOIs anzuwenden, klicken Sie unten auf die Schaltfläche Gliederung.
- Klicken Sie auf die Schaltfläche über der Registerkarte Vorlage, um TACs zu berechnen und an PKIN-Tools zu senden.
- Auf der Unterseite Eingabebilder laden:
- In PKIN:
- Wählen Sie unten rechts TACs für Anzeigetyp aus, um die TACs in kBq/mL der zuvor ausgewählten VOIs zu visualisieren.
- Klicken Sie mit der rechten Maustaste auf den Zeichenbereich mit den TACs , und klicken Sie auf Wertetabelle der sichtbaren Kurven. Kopieren Sie die Tabelle zur weiteren Analyse in eine Tabelle.
- Berechnen Sie die Pearson-Korrelation zwischen allen Paarungen von VOIs unter Verwendung von Daten vom sechsten bis letzten Zeitrahmen (= 10–60 Minuten nach dem Scan-Start), um eine Korrelationsmatrix zu erhalten. Ein Python-Skript namens "correlation_matrix.py", das diesen Zweck erfüllt, ist in der ergänzenden Datei 1 enthalten.
- Berechnen Sie, ob die Pearson-Korrelation signifikant ist, indem Sie die t-Test-Statistik verwenden:
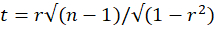 mit n - 2 Freiheitsgraden, wobei r der Pearson-Korrelationskoeffizient und n = 25 die Anzahl der Zeitrahmen ist, die für die Berechnung der Pearson-Korrelation verwendet werden. Wenden Sie eine geeignete Korrektur für Mehrfachvergleiche an und vergleichen Sie die Statistik mit dem kritischen Wert einer zweiseitigen t-Verteilung (p = 0,05) mit n - 2 Freiheitsgraden. Betrachten Sie die Pearson-Korrelation zwischen zwei VOIs als signifikant, wenn der korrigierte t-Testwert kleiner als der kritische Wert ist.
mit n - 2 Freiheitsgraden, wobei r der Pearson-Korrelationskoeffizient und n = 25 die Anzahl der Zeitrahmen ist, die für die Berechnung der Pearson-Korrelation verwendet werden. Wenden Sie eine geeignete Korrektur für Mehrfachvergleiche an und vergleichen Sie die Statistik mit dem kritischen Wert einer zweiseitigen t-Verteilung (p = 0,05) mit n - 2 Freiheitsgraden. Betrachten Sie die Pearson-Korrelation zwischen zwei VOIs als signifikant, wenn der korrigierte t-Testwert kleiner als der kritische Wert ist.
Ergebnisse
Sobald der Scan abgeschlossen ist, kann der TAC der während der Erfassung erkannten Rate untersucht werden, um eine korrekte Tracerinjektion und -aufnahme zu überprüfen. Abbildung 1 zeigt eine TAC, die sich aus dem gesamten FOV des Scanners nach einer erfolgreichen Tracerinjektion und -erfassung ergibt (Bild A), und eine TAC, die sich aus einer teilweise paravenösen Tracerinjektion ergibt (Bild B). Im erfolgreichen Fall steigt die Zählr...
Diskussion
Das hier bereitgestellte Protokoll führt den Benutzer durch den Prozess der Erfassung dynamischer PET-Daten von 1 h unter Verwendung von [18F]FDG als Tracer bei Ratten. Am Ende erhält man eine Korrelationsmatrix von VOIs, die zur Beurteilung der metabolischen Konnektivität auf der Ebene eines einzelnen Probanden verwendet werden kann. Erfahrene Forscher können das Protokoll an verschiedenen Stellen an ihre spezifischen Bedürfnisse anpassen, z. B. durch die Verwendung eine...
Offenlegungen
Die Autoren haben keine Interessenkonflikte offenzulegen.
Danksagungen
Diese Arbeit wurde durch ein Forschungsstipendium der Flämischen Forschungsstiftung [G0A7422N] unterstützt.
Materialien
| Name | Company | Catalog Number | Comments |
| Antisedan | Orion Pharma | Atipamezole hydrochloride 5 mg/mL | |
| BD Micro-Fine+ insulin syringe 1 mL | BD | 324827 | 0.33 mm (29G) x 12.7 mm |
| BD Microlance 3 Needles 30 G x 1/2" | BD | 304000 | 30 G x 1/2"; 0,3 x 13 mm |
| BD Plastipak syringe 1 mL | BD | 303172 | for infusion pump |
| BTPE-10 Polyethylene tubing | Instech | 0.11x.024in (.28x60mm) | |
| Domitor | Orion Pharma | 1070499 | Medetomidine hydrochloride 1 mg/mL |
| Fusion 100 infusion pump | Chemyx Inc. | 07100 | Newer model available: Fusion 100X |
| Isoflutek 1000 mg/g | Alivira | Isoflurane | |
| MOLECUBES β-CUBE with CUBEFLOW software | MOLECUBES NV | Preclinical PET scanner | |
| PMOD Software version 4.4 | Bruker Corporation | http://www.pmod.com; quantification of PET data | |
| Saline | B. Braun | 394496 | NaCl 0.9% |
| Vidisic eye gel | Vidisic | Carbomerum 980 2 mg/g |
Referenzen
- Wilson, H., Pagano, G., Politis, M. Dementia spectrum disorders: lessons learnt from decades with PET research. J Neural Transm. 126 (3), 233-251 (2019).
- Singnurkar, A., Poon, R., Metser, U. Comparison of 18F-FDG-PET/CT and 18F-FDG-PET/MR imaging in oncology: a systematic review. Ann Nucl Med. 31 (5), 366-378 (2017).
- Youssef, G., et al. The use of 18F-FDG PET in the diagnosis of cardiac sarcoidosis: a systematic review and metaanalysis including the Ontario experience. J Nucl Med. 53 (2), 241-248 (2012).
- Carli, G., Tondo, G., Boccalini, C., Perani, D. Brain molecular connectivity in neurodegenerative conditions. Brain Sci. 11 (4), 433 (2021).
- Sala, A., Perani, D. Brain molecular connectivity in neurodegenerative diseases: Recent advances and new perspectives using positron emission tomography. Front Neurosci. 13, 617 (2019).
- Horwitz, B., Duara, R., Rapoport, S. I. Intercorrelations of glucose metabolic rates between brain regions: Application to healthy males in a state of reduced sensory input. J Cereb Blood Flow Metab. 4 (4), 484-499 (1984).
- Sporns, O., Tononi, G., Kötter, R. The human connectome: A structural description of the human brain. PLOS Comput Biol. 1 (4), e42 (2005).
- Biswal, B., Zerrin Yetkin, F., Haughton, V. M., Hyde, J. S. Functional connectivity in the motor cortex of resting human brain using echo-planar MRI. Magn Reson Med. 34 (4), 537-541 (1995).
- Sanchez-Catasus, C. A., Diercke, R. A. J. O., de Vries, E. F. J., van Waarde, A., Leenders, K. L., et al. Use of nuclear medicine molecular neuroimaging to model brain molecular connectivity. PET and SPECT in neurology. , 181-207 (2021).
- Wehrl, H. F., et al. Simultaneous PET-MRI reveals brain function in activated and resting state on metabolic, hemodynamic and multiple temporal scales. Nat Med. 19 (9), 1184-1189 (2013).
- Rosenberg, G. A., Mun-Bryce, S., Wesley, M., Kornfeld, M. Collagenase-induced intracerebral hemorrhage in rats. Stroke. 21 (5), 801-807 (1990).
- Krishnamoorthy, S., et al. Performance evaluation of the MOLECUBES β-CUBE - a high spatial resolution and high sensitivity small animal PET scanner utilizing monolithic LYSO scintillation detectors. Phys Med Biol. 63 (15), 155013 (2018).
- Schiffer, W. K., Mirrione, M. M., Dewey, S. L. Optimizing experimental protocols for quantitative behavioral imaging with 18F-FDG in rodents. J Nucl Med. 48 (2), 277-287 (2007).
- Fueger, B. J., et al. Impact of animal handling on the results of 18F-FDG PET studies in mice. J Nucl Med. 47 (6), 999-1006 (2006).
- Gold, M. E. L., Norell, M. A., Budassi, M., Vaska, P., Schulz, D. Rapid 18F-FDG uptake in brain of awake, behaving rat and anesthetized chicken has implications for behavioral PET studies in species with high metabolisms. Front Behav Neurosci. 12, 115 (2018).
- Miranda, A., et al. PET imaging of freely moving interacting rats. NeuroImage. 191, 560-567 (2019).
- Miranda, A., et al. Awake 18F-FDG PET imaging of memantine-induced brain activation and test-retest in freely running mice. J Nucl Med. 60 (6), 844-850 (2019).
- Jahreis, I., Bascuñana, P., Ross, T. L., Bankstahl, J. P., Bankstahl, M. Choice of anesthesia and data analysis method strongly increases sensitivity of 18F-FDG PET imaging during experimental epileptogenesis. PLOS ONE. 16 (11), e0260482 (2021).
- Pawela, C. P., et al. A protocol for use of medetomidine anesthesia in rats for extended studies using task-induced BOLD contrast and resting-state functional connectivity. NeuroImage. 46 (4), 1137-1147 (2009).
- Rubinov, M., Sporns, O. Complex network measures of brain connectivity: Uses and interpretations. NeuroImage. 52 (3), 1059-1069 (2010).
- Engel, J., et al. Connectomics and epilepsy. Curr Opin Neurol. 26 (2), 186 (2013).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten