È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Studio della connettività cerebrale metabolica utilizzando la tomografia a emissione di positroni dinamica 2-deossi-2-[18F]Fluoro-D-glucosio a livello di singolo soggetto
In questo articolo
Riepilogo
L'acquisizione di dati di tomografia dinamica a emissione di positroni (PET) e la ricostruzione in intervalli di tempo consente l'analisi della connettività cerebrale metabolica a livello di singolo soggetto. Descriviamo un metodo per acquisire dati PET dinamici [18F]FDG del cervello di ratto e ottenere una matrice di connettività attraverso l'estrazione di curve tempo-attività di volumi di interesse.
Abstract
Ad oggi, la connettività cerebrale metabolica è per lo più studiata a livello di gruppo attraverso l'acquisizione di dati di tomografia a emissione di positroni statici (PET) di più soggetti. I nostri gruppi di ricerca stanno attualmente studiando i cambiamenti nella connettività metabolica in più punti temporali a seguito di un'emorragia intracerebrale a livello intrasoggettivo nei ratti. Per studiare la connettività cerebrale metabolica intrasoggettiva, sono necessarie informazioni temporali dell'assorbimento del tracciante in diverse regioni cerebrali, che possono essere ottenute attraverso la PET dinamica. In questa pubblicazione, forniamo una descrizione dettagliata del nostro protocollo di acquisizione e analisi dei dati.
I dati dinamici della PET del cervello di ratto sono acquisiti su un sistema PET preclinico dedicato utilizzando il 2-deossi-2-[18F]fluoro-D-glucosio ([18F]FDG) come tracciante. Il tracciante viene iniettato per via endovenosa come bolo all'inizio della scansione PET. Durante l'acquisizione di 60 minuti, gli animali vengono sedati con medetomidina.
Dopo l'acquisizione, i dati PET vengono ricostruiti in intervalli di tempo di trenta 2 minuti utilizzando un algoritmo di ricostruzione iterativo (Maximum-Likelihood Expectation-Maximization). Un atlante parcellato costituito da più volumi di interesse (VOI) viene utilizzato per estrarre le curve tempo-attività di ciascun VOI, che vengono poi utilizzate per calcolare il coefficiente di correlazione di Pearson tra ciascuna coppia di VOI.
Questo protocollo PET dinamico consente la valutazione delle differenze di connettività metabolica tra due singole scansioni, piuttosto che tra gruppi di scansioni. Questo approccio consente lo studio dei cambiamenti nella connettività metabolica all'interno di un singolo soggetto in diversi punti temporali o il confronto della connettività metabolica di un individuo con un normale database. Tali confronti potrebbero essere utili per monitorare la progressione della malattia o aiutare nella diagnosi di disturbi neurologici caratterizzati da una comunicazione interrotta tra le regioni del cervello, come l'epilessia o la demenza.
Introduzione
La tomografia a emissione di positroni (PET) è una tecnica di imaging molecolare comunemente utilizzata nella ricerca e in ambito clinico. A causa dello sviluppo di vari traccianti PET, la PET può essere utilizzata per studiare la fisiopatologia della malattia e monitorare la progressione della malattia e la risposta ai trattamenti1. Uno dei radiotraccianti più utilizzati è il 2-deossi-2-[18F]fluoro-D-glucosio ([18F]FDG), che consente l'imaging del metabolismo del glucosio, indicativo dell'attivazione cellulare. Viene utilizzato in oncologia per la diagnosi, la stadiazione e la prognosi; in neurologia, comunemente nel contesto di malattie neurodegenerative come la demenza; e in cardiologia, per diagnosticare condizioni come la sarcoidosi, per citare solo alcuni esempi 1,2,3.
La valutazione della connettività cerebrale metabolica, ottenuta dai dati PET [18F]FDG, si riferisce alle relazioni funzionali tra l'assorbimento del tracciante in diverse regioni cerebrali. Questo approccio consente il calcolo di una "matrice di connettività" selezionando un insieme di regioni del cervello, che possono fornire informazioni su come diverse parti del cervello interagiscono e funzionano insieme. Questo tipo di analisi è particolarmente utile per studiare la funzione cerebrale in salute e in malattia, comprese condizioni come la demenza, l'epilessia e altri disturbi neurologici 4,5.
Il primo studio che ha valutato la connettività cerebrale metabolica risale già agli anni '806, ma i ricercatori hanno esplorato principalmente la connettività cerebrale strutturale, nota anche come "connettoma"7, per mezzo della risonanza magnetica pesata in diffusione (DW-MRI). Inoltre, la connettività funzionale che utilizza tecniche come la risonanza magnetica funzionale (fMRI), l'elettroencefalografia (EEG) e la magnetoencefalografia (MEG) è stata ampiamente studiata per diversi decenni 8,9.
Recentemente, c'è un rinnovato interesse nello studio della connettività cerebrale metabolica utilizzando la PET [18F]FDG, non solo da sola, ma anche in combinazione con altre forme di connettività cerebrale10. Tuttavia, a causa della natura intrinseca "statica" delle immagini PET (in contrasto, ad esempio, con la risonanza magnetica funzionale), la stragrande maggioranza dei risultati basati sulla PET della rete cerebrale si basa sull'analisi a livello di gruppo, in cui le correlazioni tra le regioni cerebrali sono calcolate a livello intersoggettivo. Questa limitazione rende impossibile un'analisi all'interno del soggetto delle immagini PET, che è essenziale per gli studi longitudinali in grado di tracciare i cambiamenti nel tempo all'interno dello stesso individuo4. Pertanto, lo sviluppo di metodi che consentono l'analisi di un singolo soggetto, come la connettività molecolare dinamica basata sulla PET, è un'importante direzione di ricerca nella ricerca sul cervello che indaga i disturbi di rete, poiché apre le porte all'uso dell'analisi di rete molecolare nella pratica clinica. Pertanto, nel nostro studio preclinico sono stati utilizzati dati PET dinamici.
I nostri gruppi di ricerca stanno attualmente conducendo uno studio che esamina i cambiamenti nella connettività metabolica a seguito di un'emorragia intracerebrale a livello intrasoggettivo in più punti temporali utilizzando il modello di collagenasi di ratto11. Per studiare la connettività cerebrale metabolica intrasoggettiva, sono necessarie informazioni temporali dell'assorbimento del tracciante in diverse regioni cerebrali, che possono essere ottenute attraverso la PET dinamica. Nelle sezioni seguenti, forniamo una descrizione dettagliata del protocollo di acquisizione e analisi dei dati.
Protocollo
Tutte le procedure sono conformi alle linee guida europee (direttiva 2010/63/UE) e il protocollo è stato approvato dal Comitato Etico Animale locale dell'Università di Gand (ECD 23/33). Dodici ratti Sprague Dawley (sei femmine, sei maschi) sono stati inclusi nello studio. Le loro scansioni PET sono state ottenute utilizzando il seguente protocollo in più punti temporali che vanno da 2 settimane prima a 18 settimane dopo un'emorragia intracerebrale indotta. Al momento della prima scansione, tutti gli animali avevano 18 settimane di età e le femmine pesavano 244,8 ± 10,1 g (media ± DS) mentre i maschi pesavano 363,6 ± 13,3 g.
Assicurarsi che i materiali radioattivi vengano lavorati e maneggiati solo da personale addestrato. Mantenere la dose per il personale e gli animali al livello più basso ragionevolmente ottenibile (ALARA).
1. Acquisizione dati
NOTA: Vedere la Tabella dei materiali per i dettagli sull'imager PET preclinico e sul software utilizzato per l'acquisizione dei dati. L'imager è uno scanner PET a 45 rivelatori (disposti in 5 anelli) che coprono un campo visivo assiale (FOV) di 13 cm e un FOV transassiale di 7,6 cm, utilizzando cristalli LYSO e rivelatori SiPM. Il sistema mostra una risoluzione spaziale di 850 μm, una sensibilità del 12% e una risoluzione energetica del 12,6%12. I passaggi seguenti sono stati scritti tenendo presente questo.
- Privare gli animali del cibo almeno 6 ore prima della scansione rimuovendo tutto il cibo dalla loro gabbia. Una volta che l'animale è stato sottoposto a un periodo di digiuno sufficiente, iniziare i preparativi per l'acquisizione dei dati come segue.
- Accendi un termoforo e lascialo riscaldare fino a 30–35 °C.
- Anestetizzare l'animale con isoflurano posizionandolo in una camera di anestesia gassosa riempita con isoflurano al 5% in O2 medicale a una velocità di flusso di 2.000 ml/min. Aiuta a mantenere la temperatura corporea dell'animale posizionando una lampada riscaldante a 30 cm dalla camera. L'animale viene adeguatamente anestetizzato quando la frequenza respiratoria è di 0,5-1 Hz.
- Pesare l'animale.
- Posiziona l'animale sul termoforo. Utilizzare un cono nasale per somministrare isoflurano al 2% nell'O2 medicale a una velocità di flusso di 500 ml/min. Regolare la dose di isoflurano secondo necessità per mantenere l'animale adeguatamente anestetizzato.
- Radere una sezione del collo dell'animale tra le scapole.
- Preparazione della vena caudale per l'iniezione del tracciante:
- Gira l'animale sul lato sinistro o destro e usa la lampada riscaldante per riscaldargli la coda, provocando vasodilatazione.
- Prendi circa 20 cm di tubo BTPE-10. Utilizzando un portaaghi, rompere l'asta di un ago da 30 G e inserire 1-2 mm della sua estremità posteriore nel tubo in PE. Riempire una siringa da insulina da 1 ml con soluzione fisiologica e inserire la punta dell'ago per 3-5 mm nel tubo in PE. Preriempire il tubo PE con soluzione fisiologica dalla siringa piena e mantenere la siringa salina collegata al tubo PE.
- Pulisci la coda dell'animale con etanolo. Rimuovere la lampada riscaldante.
- Inserire la punta dell'ago del tubo in PE in una vena di coda laterale. Verificare il corretto posizionamento dell'ago: verificare la presenza di sangue che entra nel tubo in PE quando lo stantuffo viene tirato indietro, l'assenza di resistenza durante l'iniezione e la formazione di bolle sottocutanee (che indicano l'iniezione paravenosa) nel sito di iniezione. Se l'inserimento non è andato a buon fine, rimuovere l'ago dalla coda e riprovare in una sezione più prossimale.
- Fissare l'ago con una goccia di colla istantanea nel sito di penetrazione e fissare il tubo in PE con del nastro adesivo alla coda.
NOTA: Evitare il movimento del tubo in PE. Se in qualsiasi momento il sangue entra nel tubo in PE, sciacquarlo immediatamente con soluzione fisiologica per evitare l'intasamento.
- Passa alla sedazione con medetomidina:
- Posizionare l'animale in posizione prona sul tappetino riscaldante. Metti una goccia di gel per gli occhi su ogni occhio per evitare che si secchino.
- Preparare un tubo in PE (40-100 cm) da collegare a una pompa per infusione posizionata vicino allo scanner PET. Utilizzando un portaaghi, rompere l'asta di un ago da 30 G e inserire 1-2 mm della sua parte posteriore nel tubo in PE. Riempire una siringa di plastica da 1 ml con un ago da 30 G con almeno 0,2 ml di medetomidina (per animale) e inserire la punta dell'ago da 3-5 mm nel tubo in PE. Preriempire il tubo in PE con medetomidina dalla siringa.
- Posizionare la siringa con medetomidina nella pompa per infusione. Accendere la pompa e selezionare la siringa corretta facendo clic su Trova siringa | BD Plastic | 1 ml.
- Espellere boli di 0,005 mL utilizzando la pompa fino a quando non fuoriesce una goccia dall'ago all'altra estremità del tubo in PE. Assicurarsi che non siano presenti bolle d'aria nel tubo in PE.
- Inserire l'ago del tubo in PE per 2-3 mm per via sottocutanea tra le scapole dell'animale. Fissare l'ago con una goccia di colla istantanea nel sito di penetrazione e fissare delicatamente il tubo in PE alla schiena dell'animale.
- Iniettare un bolo di volume v di medetomidina (tasso: 3 ml/min) in base al peso dell'animale:
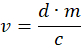
Dove d = 0,05 mg/kg (la dose di medetomidina in mg per kg di peso animale), m è il peso dell'animale in kg e c è la concentrazione di medetomidina in mg/mL. - Interrompere la somministrazione di isoflurano 10 minuti dopo la somministrazione del bolo di medetomidina, aggiustando la dose di isoflurano se necessario per mantenere stabile l'anestesia durante questo periodo di attesa.
NOTA: L'isoflurano combinato con medetomidina rallenta significativamente la frequenza respiratoria dell'animale. Un attento monitoraggio è essenziale fino alla cessazione della somministrazione di isoflurano. - Cinque minuti dopo l'interruzione dell'isoflurano, iniziare un'infusione continua di medetomidina a una velocità di:
rcont = r • m
dove r = 0,1 mg/(kg·h); la quantità di medetomidina in mg per peso corporeo dell'animale in kg/h), e m è il peso dell'animale in kg.
NOTA: È necessario un periodo di equilibrio di ulteriori 25 minuti dopo l'inizio dell'infusione continua (quindi 30 minuti dopo l'interruzione dell'isoflurano) prima che la scansione possa essere avviata per garantire che il sistema dell'animale sia privo di isoflurano. Durante questo periodo, procedere con i seguenti passaggi.
- Posizionamento degli animali nello scanner PET:
- Preparare il piano dello scanner con un termoforo, un sensore di pressione e fazzoletti di carta. Utilizzare il sensore per monitorare la frequenza respiratoria dell'animale e i fazzoletti di carta per assorbire l'urina escreta dall'animale.
NOTA: A causa della medetomidina, l'animale in genere espelle grandi quantità di urina. - Trasferire con cautela l'animale sul letto dello scanner, assicurandosi che la linea di medetomidina e il catetere della vena caudale rimangano ben saldi. Posiziona la testa dell'animale il più dritta possibile usando un cono per il naso per assistenza, se necessario. Fissare la testina con del nastro adesivo una volta posizionata correttamente.
- Accendere il termoforo e verificare che il sistema di monitoraggio della frequenza respiratoria funzioni correttamente, regolando il posizionamento del sensore di pressione se necessario.
- Preparare il piano dello scanner con un termoforo, un sensore di pressione e fazzoletti di carta. Utilizzare il sensore per monitorare la frequenza respiratoria dell'animale e i fazzoletti di carta per assorbire l'urina escreta dall'animale.
- Da dieci a cinque minuti prima di iniziare la scansione PET, riempire una siringa da insulina con circa 20 MBq di 2-deossi-2-[18F]fluoro-D-glucosio ([18F]FDG) diluiti in 0,1-0,8 mL di soluzione fisiologica.
ATTENZIONE: Il fluoro-18 (18F) è un radioisotopo del fluoro con un'emivita di 109,8 min. Seguire i protocolli locali di sicurezza dalle radiazioni quando si maneggia materiale radioattivo. - Aprire il software dello scanner e configurare i seguenti parametri di scansione: selezionare 18F come isotopo (importante per la correzione del decadimento), inserire l'attività misurata della siringa contenente il radiotracciante (in MBq misurata dal calibratore di dose) e il tempo di misurazione (le informazioni verranno salvate nei metadati, necessari per convertire i dati in altre unità come i valori di assorbimento standard). Selezionare il protocollo generale e selezionare Ordinamento automatico dopo l'acquisizione. Impostare la durata della scansione su 1 ora e regolare la regione di interesse (ROI) in modo che contenga l'intero cervello del ratto (in genere posizione 2-5).
- Una volta trascorsi 30 minuti dall'interruzione della somministrazione di isoflurano, iniziare la scansione PET:
- Anche in questo caso, confermare il corretto posizionamento in vena mediante aspirazione seguita dall'iniezione di una piccola quantità di soluzione fisiologica, come descritto al punto 1.7.4.
- Sostituire la siringa salina con la siringa radiotracciante. Quando si rimuove la siringa dal tubo in PE, è possibile che il sangue entri nel tubo in PE, spingendo fuori la soluzione fisiologica. Inserire rapidamente l'ago della siringa del radiotracciante senza forare il tubo in PE.
- Avviare la scansione utilizzando il software dello scanner.
- Una volta iniziata l'acquisizione, iniettare il tracciante come bolo in circa 0,5-2 s. Sostituire immediatamente la siringa del radiotracciante vuota con la siringa salina e lavare il tubo in PE con circa 0,1 mL di soluzione fisiologica. Utilizzare un fazzoletto per raccogliere eventuali gocce di radiotracciante che potrebbero fuoriuscire durante la sostituzione della siringa.
NOTA: La velocità di conteggio visualizzata dal software dello scanner dovrebbe aumentare rapidamente a 4,5–6,5 Mcps dopo l'iniezione del tracciante. Il tasso di conta inizialmente significativamente inferiore a 4 Mcps e successivamente crescente indica un'iniezione di tracciante (parzialmente) paravenoso. - Valutare l'attività residua che non è stata iniettata nell'animale misurando l'attività della siringa tracciante e dei tessuti e dei guanti potenzialmente contaminati. Inserire l'attività e l'ora della misurazione nel software dello scanner per salvarla nei metadati.
NOTA: Un'attività residua di 0,1-1,5 MBq è tipica, a seconda della quantità di tracciante fuoriuscita quando si sostituisce il tracciante con la siringa salina.
- Una volta completata la scansione, espellere il letto con l'animale dallo scanner.
NOTA: L'animale può reagire agli stimoli o svegliarsi prima che la medetomidina sia stata antagonizzata. Maneggiare l'animale con cura e prepararsi a movimenti improvvisi durante i passaggi successivi. Inoltre, tieni presente che l'animale è radioattivo. Pertanto, cerca di completare i seguenti passaggi nel modo più rapido e accurato possibile. - Rimuovere l'ago e il tubo in PE dalla coda dell'animale utilizzando un portaago. Applicare un fazzoletto o un impacco sul sito di puntura per fermare il potenziale sanguinamento.
- Interrompere la somministrazione di medetomidina e rimuovere l'ago e il tubo in PE dal collo dell'animale.
- Collocare l'animale in una gabbia priva di altri animali, utilizzando un fazzoletto per raccogliere l'urina (radioattiva) che può essere espulsa durante lo spostamento dell'animale. Somministrare 1 mL di soluzione fisiologica per via sottocutanea nella parte posteriore utilizzando una siringa da insulina da 1 mL per favorire la reidratazione.
- Invertire la sedazione con un'iniezione sottocutanea di atipamezolo.
- Calcola il volume v appropriato utilizzando la seguente equazione:
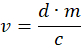
Dove d = 0,5 mg/kg, ossia la dose di medetomidina in mg per kg di peso animale, m è il peso dell'animale in kg e c è la concentrazione di atipamezolo in mg/mL. - Per gli animali leggeri che producono piccoli volumi calcolati, diluire l'atipamezolo con soluzione fisiologica per consentire una dose più accurata. Riempire una siringa da insulina con 4 v di atipamezolo e 12 v di soluzione fisiologica. Iniettare 1/4 della soluzione risultante per via sottocutanea nella schiena dell'animale vicino al collo.
- Calcola il volume v appropriato utilizzando la seguente equazione:
- Monitora visivamente l'animale fino a quando non si sveglia, il che dovrebbe richiedere 1-5 minuti. Una volta che l'animale è in grado di mantenersi in posizione eretta e di muoversi liberamente, riportalo nella stabulazione del gruppo. Fornisci all'animale cibo e acqua.
2. Ricostruzione dei dati e controllo qualità
NOTA: Utilizzando l'hardware e il software disponibili nel nostro studio, tutti i dati PET sono stati corretti per il decadimento del radionuclide e i sinogrammi acquisiti sono stati ricostruiti con l'algoritmo 3-dimensionale di massimizzazione dell'aspettativa del sottoinsieme ordinato (OSEM-3D) utilizzando una finestra del 30% intorno al fotopicco di 511 keV. Il software di ricostruzione OSEM utilizzava le impostazioni predefinite di 10 sottoinsiemi con 20 milioni di eventi per sottoinsieme. Le immagini sono state ricostruite in una matrice trasversale di 192 x 192 x 384 con voxel cubici di 0,4 mm. Non è stata eseguita alcuna correzione dell'attenuazione.
- Per verificare che l'iniezione del tracciante sia andata a buon fine, controllare la velocità di conteggio rilevata durante l'acquisizione di 60 minuti (ad esempio, la curva tempo-attività (TAC)). Verificare che vi sia un picco entro il primo minuto dall'acquisizione, immediatamente dopo l'iniezione del tracciante.
- Ricostruzione statica: ricostruire i dati acquisiti utilizzando un algoritmo OSEM utilizzando 30 iterazioni, una dimensione isometrica del voxel di 400 μm e una finestra di energia del 30% centrata sul fotopicco di 511 keV.
- Una volta completata la ricostruzione, ispezionare l'immagine risultante per verificare la presenza di artefatti.
- Ricostruzione dinamica: ricostruisce i dati acquisiti in intervalli di tempo di trenta 2 minuti utilizzando le stesse impostazioni della ricostruzione statica.
3. Analisi dei dati
NOTA: I seguenti passaggi 3.1 e 3.2 vengono eseguiti in un ambiente software biomedico dedicato alla quantificazione dei dati PET, utilizzando lo strumento di registrazione e fusione delle immagini (PFUS) e lo strumento di modellazione cinetica generale (PKIN).
- In PFUS:
- Nella sottopagina Carica immagini di input:
- Caricare il file DICOM della ricostruzione dinamica utilizzando il pulsante di caricamento INP o trascinando e rilasciando il file nell'area di disegno. Utilizzare il controllo di visualizzazione dell'immagine Generale in alto a destra per navigare tra le sezioni e gli intervalli di tempo e regolare la barra dei colori. Se necessario, regolare l'orientamento dell'immagine sull'orientamento standard utilizzando il pulsante Modifica orientamento immagine di input nella barra laterale destra.
- Ritaglio e media dei fotogrammi: fai clic sulla freccia in basso a destra per visualizzare i controlli nascosti per la somma e il ritaglio e seleziona Ritaglia. Nell'area di disegno, definisci i limiti per il ritaglio regolando i bordi e trascinando il centro della casella di ritaglio; Assicurati che l'intero cervello e un po' di imbottitura, contenente parti degli occhi e del midollo spinale, siano contenuti. Una volta che la casella di ritaglio è stata definita correttamente, fare clic sul pulsante di ritaglio per ritagliare l'immagine. Calcola la media dei fotogrammi ponderata nel tempo su tutti gli intervalli di tempo facendo clic sul pulsante arancione della media e attendi che il risultato venga aggiunto automaticamente alle immagini di input, ottenendo due immagini di input in totale: i dati dinamici ritagliati con gli intervalli di tempo e la media ponderata nel tempo ritagliata degli stessi dati.
- Selezionare Deformazione come metodo di corrispondenza o premere il pulsante se già selezionato per passare alla normalizzazione basata su modello.
- Nella sottopagina Riferimento e corrispondenza:
- Selezionare l'immagine media ponderata nel tempo come immagine di input (normalmente viene selezionata automaticamente dall'ultimo passaggio).
- Caricare un riferimento facendo clic sul pulsante per selezionare un modello di normalizzazione come immagine di riferimento. Seleziona Rat FDG (W. Schiffer).
- Abbina l'input al modello facendo clic sul pulsante per Corrispondenza input | Input: regola manualmente nella barra laterale a destra. Trascina e ruota l'immagine di input in modo che corrisponda approssimativamente al modello. Assicurati che Rat sia selezionato come specie, che il metodo di registrazione sia Deform, quindi fai clic sul pulsante Match Current per accedere alla sottopagina successiva.
- Nella sottopagina Risultato corrispondente:
- Controlla il risultato e regola la corrispondenza manualmente se necessario. Fare clic su Applica trasformazione corrente a tutto per far corrispondere anche l'immagine di input costituita dagli intervalli di tempo.
- Fare clic sul pulsante verde VOIs per passare alla fase di elaborazione successiva.
- Nella sottopagina Analisi VOI:
- Seleziona l'immagine contenente i tempi come immagine A nel menu in basso a destra.
- Vai alla scheda Modello e seleziona Px Rat (W. Schiffer) nella scheda Atlas , oppure carica un atlante personalizzato e seleziona i volumi desiderati. Per applicare i VOI, fare clic sul pulsante Struttura in basso.
- Fare clic sul pulsante sopra la scheda Modello per calcolare i TAC e inviarli agli strumenti PKIN.
- Nella sottopagina Carica immagini di input:
- In PKIN:
- In basso a destra, selezionare TAC per Tipo di visualizzazione per visualizzare i TAC in kBq/mL dei VOI selezionati in precedenza.
- Fare clic con il pulsante destro del mouse sull'area di disegno con i TAC e fare clic su Tabella dei valori delle curve visibili. Copia la tabella in un foglio di calcolo per ulteriori analisi.
- Calcola la correlazione di Pearson tra tutte le coppie di VOI, utilizzando i dati dal sesto all'ultimo intervallo di tempo (= 10-60 minuti dopo l'inizio della scansione), per ottenere una matrice di correlazione. Uno script Python chiamato "correlation_matrix.py" che soddisfa questo scopo è incluso nel file supplementare 1.
- Calcola se la correlazione di Pearson è significativa utilizzando la statistica del test t:
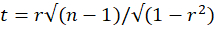 con n - 2 gradi di libertà, dove r è il coefficiente di correlazione di Pearson e n = 25 il numero di intervalli di tempo utilizzati per calcolare la correlazione di Pearson. Applicare una correzione appropriata per confronti multipli e confrontare la statistica con il valore critico di una distribuzione t a due code (p = 0,05) con n - 2 gradi di libertà. Si consideri significativa la correlazione di Pearson tra due VOI se il valore del test t corretto è inferiore al valore critico.
con n - 2 gradi di libertà, dove r è il coefficiente di correlazione di Pearson e n = 25 il numero di intervalli di tempo utilizzati per calcolare la correlazione di Pearson. Applicare una correzione appropriata per confronti multipli e confrontare la statistica con il valore critico di una distribuzione t a due code (p = 0,05) con n - 2 gradi di libertà. Si consideri significativa la correlazione di Pearson tra due VOI se il valore del test t corretto è inferiore al valore critico.
Risultati
Una volta completata la scansione, è possibile esaminare il TAC della velocità rilevata durante l'acquisizione per verificare la corretta iniezione e assorbimento del tracciante. La Figura 1 mostra un TAC risultante dall'intero FOV dello scanner dopo un'iniezione e un'acquisizione di traccianti riuscite (pannello A) e un TAC risultante dopo un'iniezione di tracciante parzialmente paravenoso (pannello B). In caso di successo, il tasso di co...
Discussione
Il protocollo qui fornito guida gli utenti attraverso il processo di acquisizione di dati PET dinamici di 1 ora utilizzando [18F]FDG come tracciante nei ratti. Alla fine, si ottiene una matrice di correlazione dei VOI, che può essere utilizzata per valutare la connettività metabolica a livello di singolo soggetto. I ricercatori esperti possono adattare il protocollo alle loro esigenze specifiche in vari punti, ad esempio utilizzando un radiotracciante, un tempo di acquisizio...
Divulgazioni
Gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo lavoro è stato sostenuto da una borsa di ricerca della Fondazione per la ricerca fiamminga [G0A7422N].
Materiali
| Name | Company | Catalog Number | Comments |
| Antisedan | Orion Pharma | Atipamezole hydrochloride 5 mg/mL | |
| BD Micro-Fine+ insulin syringe 1 mL | BD | 324827 | 0.33 mm (29G) x 12.7 mm |
| BD Microlance 3 Needles 30 G x 1/2" | BD | 304000 | 30 G x 1/2"; 0,3 x 13 mm |
| BD Plastipak syringe 1 mL | BD | 303172 | for infusion pump |
| BTPE-10 Polyethylene tubing | Instech | 0.11x.024in (.28x60mm) | |
| Domitor | Orion Pharma | 1070499 | Medetomidine hydrochloride 1 mg/mL |
| Fusion 100 infusion pump | Chemyx Inc. | 07100 | Newer model available: Fusion 100X |
| Isoflutek 1000 mg/g | Alivira | Isoflurane | |
| MOLECUBES β-CUBE with CUBEFLOW software | MOLECUBES NV | Preclinical PET scanner | |
| PMOD Software version 4.4 | Bruker Corporation | http://www.pmod.com; quantification of PET data | |
| Saline | B. Braun | 394496 | NaCl 0.9% |
| Vidisic eye gel | Vidisic | Carbomerum 980 2 mg/g |
Riferimenti
- Wilson, H., Pagano, G., Politis, M. Dementia spectrum disorders: lessons learnt from decades with PET research. J Neural Transm. 126 (3), 233-251 (2019).
- Singnurkar, A., Poon, R., Metser, U. Comparison of 18F-FDG-PET/CT and 18F-FDG-PET/MR imaging in oncology: a systematic review. Ann Nucl Med. 31 (5), 366-378 (2017).
- Youssef, G., et al. The use of 18F-FDG PET in the diagnosis of cardiac sarcoidosis: a systematic review and metaanalysis including the Ontario experience. J Nucl Med. 53 (2), 241-248 (2012).
- Carli, G., Tondo, G., Boccalini, C., Perani, D. Brain molecular connectivity in neurodegenerative conditions. Brain Sci. 11 (4), 433 (2021).
- Sala, A., Perani, D. Brain molecular connectivity in neurodegenerative diseases: Recent advances and new perspectives using positron emission tomography. Front Neurosci. 13, 617 (2019).
- Horwitz, B., Duara, R., Rapoport, S. I. Intercorrelations of glucose metabolic rates between brain regions: Application to healthy males in a state of reduced sensory input. J Cereb Blood Flow Metab. 4 (4), 484-499 (1984).
- Sporns, O., Tononi, G., Kötter, R. The human connectome: A structural description of the human brain. PLOS Comput Biol. 1 (4), e42 (2005).
- Biswal, B., Zerrin Yetkin, F., Haughton, V. M., Hyde, J. S. Functional connectivity in the motor cortex of resting human brain using echo-planar MRI. Magn Reson Med. 34 (4), 537-541 (1995).
- Sanchez-Catasus, C. A., Diercke, R. A. J. O., de Vries, E. F. J., van Waarde, A., Leenders, K. L., et al. Use of nuclear medicine molecular neuroimaging to model brain molecular connectivity. PET and SPECT in neurology. , 181-207 (2021).
- Wehrl, H. F., et al. Simultaneous PET-MRI reveals brain function in activated and resting state on metabolic, hemodynamic and multiple temporal scales. Nat Med. 19 (9), 1184-1189 (2013).
- Rosenberg, G. A., Mun-Bryce, S., Wesley, M., Kornfeld, M. Collagenase-induced intracerebral hemorrhage in rats. Stroke. 21 (5), 801-807 (1990).
- Krishnamoorthy, S., et al. Performance evaluation of the MOLECUBES β-CUBE - a high spatial resolution and high sensitivity small animal PET scanner utilizing monolithic LYSO scintillation detectors. Phys Med Biol. 63 (15), 155013 (2018).
- Schiffer, W. K., Mirrione, M. M., Dewey, S. L. Optimizing experimental protocols for quantitative behavioral imaging with 18F-FDG in rodents. J Nucl Med. 48 (2), 277-287 (2007).
- Fueger, B. J., et al. Impact of animal handling on the results of 18F-FDG PET studies in mice. J Nucl Med. 47 (6), 999-1006 (2006).
- Gold, M. E. L., Norell, M. A., Budassi, M., Vaska, P., Schulz, D. Rapid 18F-FDG uptake in brain of awake, behaving rat and anesthetized chicken has implications for behavioral PET studies in species with high metabolisms. Front Behav Neurosci. 12, 115 (2018).
- Miranda, A., et al. PET imaging of freely moving interacting rats. NeuroImage. 191, 560-567 (2019).
- Miranda, A., et al. Awake 18F-FDG PET imaging of memantine-induced brain activation and test-retest in freely running mice. J Nucl Med. 60 (6), 844-850 (2019).
- Jahreis, I., Bascuñana, P., Ross, T. L., Bankstahl, J. P., Bankstahl, M. Choice of anesthesia and data analysis method strongly increases sensitivity of 18F-FDG PET imaging during experimental epileptogenesis. PLOS ONE. 16 (11), e0260482 (2021).
- Pawela, C. P., et al. A protocol for use of medetomidine anesthesia in rats for extended studies using task-induced BOLD contrast and resting-state functional connectivity. NeuroImage. 46 (4), 1137-1147 (2009).
- Rubinov, M., Sporns, O. Complex network measures of brain connectivity: Uses and interpretations. NeuroImage. 52 (3), 1059-1069 (2010).
- Engel, J., et al. Connectomics and epilepsy. Curr Opin Neurol. 26 (2), 186 (2013).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon