Se requiere una suscripción a JoVE para ver este contenido. Inicie sesión o comience su prueba gratuita.
Method Article
Activación cardíaca retrospectiva con un prototipo de tomógrafo computarizado de rayos X para animales pequeños
En este artículo
Resumen
Proporcionamos una descripción completa del método de activación cardíaca retrospectiva intrínseca del CrumpCAT, un prototipo de escáner de tomografía computarizada (TC) de rayos X para animales pequeños diseñado y construido en nuestra institución de investigación.
Resumen
El CrumpCAT es un prototipo de escáner de tomografía computarizada (TC) de rayos X para animales pequeños desarrollado en nuestra institución de investigación. El detector CMOS con una velocidad de fotogramas máxima de 29 Hz y fuentes similares de rayos X de tungsteno con energías que oscilan entre 50 kVp y 80 kVp se utilizan ampliamente en los instrumentos de TC de rayos X preclínicos disponibles en el mercado. Esto hace que el trabajo descrito sea muy relevante para otras instituciones, a pesar de la opinión generalizada de que estos detectores no son adecuados para controlar las altas frecuencias cardíacas de los ratones (~600 latidos/min). El escáner cuenta con imágenes de resolución media (200 μm) y alta (125 μm), fluoroscopia, activación respiratoria retrospectiva y activación cardíaca retrospectiva, con reconstrucción de imágenes de proyección iterativa o filtrada. Entre estas características, la compuerta cardíaca es la característica más útil para estudiar las funciones cardíacas in vivo, ya que elimina eficazmente la borrosidad de la imagen causada por el movimiento respiratorio y cardíaco.
Aquí, describimos nuestro método para la tomografía computarizada cardíaca retrospectiva preclínica, destinada a avanzar en la investigación sobre la función y el análisis de la estructura cardíaca in vivo . El método de activación cardíaca adquiere un gran número de proyecciones en el menor tiempo de exposición práctico (~20 ms) y luego extrae retrospectivamente las señales respiratorias y cardíacas de los cambios temporales en secuencias de proyección brutas. Estas señales se utilizan para rechazar las proyecciones que pertenecen a la fase de inspiración de alta velocidad de movimiento del ciclo respiratorio y para dividir las proyecciones restantes en 12 grupos, cada uno correspondiente a una fase del ciclo cardíaco. Cada grupo se reconstruye de forma independiente utilizando un método iterativo para producir una imagen volumétrica para cada fase cardíaca, lo que da como resultado un conjunto de datos de cuatro dimensiones (4D).
Estas imágenes de fase se pueden analizar de forma colectiva o individual, lo que permite una evaluación detallada de la función cardíaca. Demostramos la efectividad de ambos enfoques de la función de activación cardíaca del prototipo del escáner a través de resultados representativos de imágenes in vivo .
Introducción
La investigación con animales pequeños a menudo emplea una combinación de modalidades de imagen no invasivas, siendo la tomografía computarizada (TC) de rayos X, una opción destacada debido a su madurez, rentabilidad, velocidad 1,2 y capacidad para proporcionar información complementaria junto con otras modalidades como la tomografía por emisión de positrones (PET)2,3 y la tomografía computarizada por emisión de fotón único (SPECT)2,4. Sin embargo, al igual que otras técnicas de imagen, la TC es susceptible a los artefactos de movimiento fisiológico causados por el latido del corazón o la respiración, que introducen desenfoque y limitan la precisión de la investigación.
Para abordar esta limitación, el desenfoque del movimiento respiratorio y cardíaco se puede mitigar a través de una técnica conocida como compuerta 5,6,7,8, donde la adquisición de datos se sincroniza con fases específicas del ciclo cardíaco o respiratorio (o puertas). Un enfoque para lograr esto, conocido como compuerta prospectiva 3,6, consiste en conectar sensores al animal para proporcionar señales de compuerta en tiempo real a un escáner compatible. Si bien es efectivo, este método requiere mucho trabajo y tiempo, especialmente cuando se colocan sensores en el pecho y las patas de animales pequeños como ratones, lo que limita la escala de los estudios. Alternativamente, la activación retrospectiva intrínseca 7,9,10,11 implica la adquisición de datos de series temporales sin el uso de sensores, sino mediante la identificación de características en los datos que permiten la clasificación retrospectiva de los resultados en función de su fase en el ciclo cardíaco o respiratorio. Este enfoque ofrece resultados comparables a los de las compuertas prospectivas, pero sin la necesidad de hardware adicional ni el esfuerzo que implica la conexión del sensor de pulso y, por lo tanto, simplifica enormemente los protocolos experimentales.
En nuestro método para la toma de imágenes preclínicas de TC cardíaca, utilizamos la activación retrospectiva intrínseca para extraer los ciclos respiratorios y cardíacos de las variaciones de amplitud en las regiones de las proyecciones de rayos X que exhiben los cambios más significativos entre fotogramas sucesivos. Para facilitar este proceso, se co-registra una plantilla de tórax de ratón en la primera proyección posteroanterior utilizando Información Mutua12. Una vez que la plantilla está en su lugar, las intensidades de píxeles en una ventana cerca del diafragma se suman para generar una señal respiratoria sustituta, mientras que las que están cerca del miocardio se suman para derivar la señal cardíaca sustituta. A continuación, estas señales se filtran de paso de banda en el dominio del tiempo, y a cada fotograma del conjunto de datos se le asigna un número de fase fraccionaria (entre 0 y 1) en función de su fase respiratoria y cardíaca. Esto permite la selección o el rechazo de proyecciones de acuerdo con sus valores de fase. Normalmente, se conservan los fotogramas correspondientes a la fase de fin de espiración del ciclo respiratorio (0,15 ≤ fase < 0,85), mientras que los de la fase de inspiración, donde el movimiento es más pronunciado, se descartan. Los fotogramas restantes se agrupan en 12 fases cardíacas, cada una de las cuales representa 1/12 (0,083) del ciclo cardíaco y se reconstruyen en imágenes 3D utilizando un método iterativo (Ordered Subset Expectation Maximization [OSEM])13,14. Todo el proceso se resume en la Figura 1.
Protocolo
Los protocolos experimentales con animales fueron revisados y aprobados por el Comité Institucional de Cuidado y Uso de Animales de la Universidad de California, Los Ángeles (UCLA). En este protocolo se utilizaron ratones C57BL/6J (8 semanas de edad, machos, 24-26 g). El tomógrafo utilizado en este estudio es el CrumpCAT (Figura 2), un prototipo desarrollado en nuestra institución de investigación para la investigación preclínica, que nos proporciona el control y la flexibilidad necesarios para optimizar los protocolos de adquisición y reconstrucción. El método supone que los ratones anestesiados tendrán una frecuencia cardíaca no superior a 600 latidos/min y una frecuencia respiratoria entre 20 y 180 respiraciones/min15.
1. Configuración del equipo
- Opere la fuente de rayos X a un voltaje máximo de 50 kVp con corriente continua de 200 μA.
- Establezca el agrupamiento de píxeles de la cámara de rayos X en 2 y el tiempo de exposición en su valor práctico más corto (20 ms de tiempo de exposición más 14 ms de tiempo de lectura para un tiempo de muestreo total de 34 ms).
NOTA: La opción sin discretización (discretización 1) tiene una velocidad de fotogramas más lenta y, por lo tanto, no se puede utilizar para adquisiciones controladas. La velocidad máxima de fotogramas es de unos 30 fotogramas/s.
2. Preparación animal
- Anestesiar ratones con una mezcla de oxígeno y gas isoflurano a una concentración del 2,0%.
- Mantenga a los animales bajo anestesia durante 10 minutos antes de la tomografía computarizada, para asegurarse de que los signos vitales estén estables y que los animales estén relajados para evitar cualquier movimiento corporal durante la exploración. Juzgue la profundidad de la anestesia de un ratón por la falta del reflejo de pellizco del dedo del pie al pellizcar firmemente la membrana entre los dedos con una uña.
- Para que las imágenes con compuerta cardíaca visualicen las cavidades cardíacas, inyecte por vía intravenosa 100 μL de agente de contraste para TC a través de la vena de la cola inmediatamente antes de la imagen para TC (Figura 3A).
- Coloque el ratón anestesiado dentro de la cámara de imágenes de TC (Figura 3B).
3. Adquisición de datos
- Encienda el software CrumpCAT.
- Tomografía computarizada sin compuerta con alta resolución (125 μm, binning 1) (Figura 4A)
- Introduzca un ID de estudio en la interfaz de usuario.
- Seleccione Mouse Hi-Res en el menú desplegable Protocolo .
- Haga clic en el botón Escanear de la interfaz de usuario para adquirir 720 proyecciones con un tiempo de exposición de 80 ms/proyección.
- TC no compuerta con resolución media (200 μm, binning 2) (Figura 4B)
- Introduzca un ID de estudio en la interfaz de usuario.
- Seleccione Estándar de mouse en el menú desplegable Protocolo .
- Haga clic en el botón Escanear de la interfaz de usuario para adquirir 720 proyecciones con un tiempo de exposición de 100 ms/proyección.
- Tomografía computarizada con compuerta (200 μm, agrupación 2) (Figura 4C)
- Introduzca un ID de estudio en la interfaz de usuario.
- Seleccione Cardiac Gating en el menú desplegable Protocolo .
- Haga clic en el botón Escanear de la interfaz de usuario y adquiera 21.600 proyecciones con un tiempo de exposición de 20 ms/proyección.
4. Preprocesamiento de datos
NOTA: Los pasos de preprocesamiento solo son necesarios para las adquisiciones controladas. Todos estos pasos se realizan automáticamente mediante el software de reconstrucción y no se requiere la intervención del operador.
- Extracción de señales
- Para el co-registro de la plantilla de tórax, deje que una pequeña imagen (plantilla) que represente de manera tosca un tórax de ratón con las costillas, el corazón, los pulmones y el hígado se registre automáticamente en la primera proyección de rayos X, maximizando la Información Mutua12 entre la proyección y la plantilla (Figura 5).
NOTA: Solo se realizan operaciones de conversión en la plantilla y la plantilla de registro conjunto se utiliza para identificar regiones de interés (ROI) en todas las proyecciones. - Para la extracción de señales respiratorias, permita que se designe un ROI rectangular (ROI-1) en la plantilla para representar el diafragma y la señal respiratoria que se generará sumando las intensidades de píxeles dentro de ROI-1 para cada proyección (Figura 5).
- Para la extracción de la señal cardíaca, se designe un segundo rectángulo ROI (ROI-2) en la plantilla cerca del corazón, y la señal cardíaca generada sumando las intensidades de píxeles en ROI-2 para cada proyección (Figura 5).
NOTA: La plantilla de tórax se registra conjuntamente solo en la primera proyección para identificar qué filas y columnas de imagen (ventana) se deben usar para la extracción de señales. Esa ventana es lo suficientemente ancha como para abarcar todas las proyecciones siempre que el animal esté razonablemente centrado, como es el caso cuando se utiliza nuestro lechopara animales 16.
- Para el co-registro de la plantilla de tórax, deje que una pequeña imagen (plantilla) que represente de manera tosca un tórax de ratón con las costillas, el corazón, los pulmones y el hígado se registre automáticamente en la primera proyección de rayos X, maximizando la Información Mutua12 entre la proyección y la plantilla (Figura 5).
- Filtro de paso de banda
- Permite generar un filtro de paso de banda combinando dos funciones Sinc con diferentes frecuencias de corte en el dominio temporal17, que corresponden a filtros de paso bajo en el dominio de frecuencia como se describe a continuación.
- Deje que el software aplique el filtro de paso de banda en la señal cardíaca utilizando una banda de frecuencia de [300, 600] min-1 (Figura 6A).
NOTA: La señal cardíaca sin filtrar (Figura 6B) todavía contiene una contribución significativa del movimiento respiratorio. - Del mismo modo, permita filtrar la señal respiratoria utilizando una banda de frecuencia de [20, 300] min-1. Los armónicos superiores se conservan para producir una señal que no es simplemente sinusoidal e identifica más claramente las dos fases principales: inspiración y espiración.
NOTA: En este punto, tanto las señales respiratorias como las cardíacas se pueden interpolar fácilmente y considerar funciones continuas de tiempo con el fin de calcular el número de fase fraccional.
- Asignación de fases
- Deje que el software identifique el comienzo de cada ciclo (respiratorio o cardíaco) encontrando los cruces por cero de la primera derivada de la señal. Cada paso por cero corresponde a un máximo en la señal y marca el inicio de un nuevo ciclo y el final del anterior. Para ver un ejemplo, vea la señal respiratoria en la Figura 7.
- Permite asignar un valor fraccionario de fase (entre 0 y 1) a cada proyección para cada una de las dos señales (respiratoria o cardíaca).
NOTA: Por definición, el punto de inicio tiene un valor de fase fraccionaria de cero (para el ciclo actual) o uno (para el ciclo anterior).
- Máscaras de selección
- Permite crear 12 máscaras binarias (una por fase cardíaca) para seleccionar las proyecciones que pertenecen a cada fase. Cada máscara contiene 21.600 entradas (una por proyección) que son 0 o 1, lo que significa rechazar o mantener esa proyección, respectivamente.
- Para cada máscara (fase), observe que el software indica si una proyección se mantiene (1) o se descarta (0) de acuerdo con su número de fase fraccionaria. Para la fase 0, se mantienen las proyecciones con un valor de fase cardíaca en el intervalo [0, 1/12]. Para la fase 1, se mantienen los valores de la fase cardíaca en el intervalo [1/12, 2/12], y así sucesivamente.
- Para cada mascarilla (fase), tenga en cuenta que cualquier proyección con una fase respiratoria inferior a 0,15 o superior a 0,85 se rechaza (0 en la mascarilla) porque pertenece a la fase de inspiración, que tiene el mayor movimiento (Figura 8A). La figura 8B muestra la asignación de la fase cardíaca a las proyecciones durante los primeros 2 s.
NOTA: No se introdujo ningún sesgo significativo en este proceso de selección, ya que el número de proyecciones por fase se mantuvo relativamente constante en 1.800 ± 194.
5. Reconstrucción de la imagen
- En el caso de las tomografías computarizadas sin compuertas, reconstruya las imágenes con una resolución media (200 μm) o alta (125 μm) mediante el algoritmo de retroproyección filtrada (Feldkamp18) con un filtro Shepp-Logan.
- En el caso de las tomografías computarizadas con activación cardíaca, se reconstruya cada imagen de fase cardíaca utilizando el algoritmo OSEMiterativo 13, con 12 subconjuntos y 8 iteraciones, teniendo en cuenta solo las proyecciones seleccionadas que contribuyen a una fase determinada (como se indica en una máscara binaria de selección de fase).
- Para reducir el ruido, aplique un filtro mediano posterior a la reconstrucción en la dimensión temporal (es decir, a través de imágenes) y un filtro gaussiano 3D ligero en la dimensión espacial (σ = 38 μm).
NOTA: Para la reconstrucción de imágenes con compuerta respiratoria, utilice todas las fases cardíacas indistintamente. La corrección del endurecimiento de la viga (corrección del agua) se aplica a cada reconstrucción.
6. Evaluación por imagen y cuantificación del volumen del ventrículo izquierdo (VI)
- Abra la imagen de TC en un visor DICOM como Amide19 (Figura 9A).
- Mejore el contraste de la imagen visible ajustando el rango del valor CT (medido en la unidad de Hounsfield [HU]) como [-500, 500] (Figura 9A).
- Evaluación de la calidad de la imagen
- Extraiga un ROI para la región calcificada, que se define como la región alrededor del máximo con un valor CT mayor o igual al 85% del valor máximo de HU (Figura 9B).
- Utilice la relación contraste-ruido (CNR)20,21 como métrica para evaluar la calidad de la imagen y su capacidad para identificar estructuras pequeñas, como pequeñas calcificaciones:
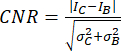 ,
,
donde I y σ representan la intensidad media y la desviación estándar de una región: calcificación (subíndice C) o fondo (subíndice B).
- Cuantificación del volumen del ventrículo izquierdo
- Dibuje un ROI a mano alzada en 3D para identificar el VI en cada fase (Figura 10A).
- Cuantifique el volumen del VI calculando vóxeles con un valor CT umbral de 730 HU (Figura 10B). En la Figura 10C se muestra un gráfico de los volúmenes de VI.
NOTA: El umbral del valor CT no es absoluto; depende del agente de contraste utilizado, el tamaño del animal, la cepa, el estado de salud y el tiempo entre la inyección y la tomografía computarizada. Amide es un software de distribución libre desarrollado en nuestro instituto, pero hay otros visores de imágenes más sofisticados disponibles (por ejemplo, ORS Dragonfly22).
Resultados
En primer lugar, comparamos imágenes de TC con y sin compuerta para visualizar la calcificación cardíaca en ratones (machos, 30-32 g). El modelo murino de calcificación cardíaca fue creado mediante la inducción de lesión cardíaca por congelación-descongelación rápida del tejido cardíaco (criolesión), como se describió anteriormente23. Con los protocolos de TC sin compuertas, las calcificaciones cardíacas se identificaron más claramente en la imagen...
Discusión
La implementación de hardware específica descrita aquí es un sistema de TC de rayos X hecho a medida exclusivo de nuestro instituto, pero el detector específico se usa ampliamente en los instrumentos de TC de rayos X preclínicos disponibles comercialmente, lo que hace que el trabajo descrito sea relevante para otras instituciones. Este sistema es funcionalmente el prototipo de dos subsistemas de microCT de rayos X in vivo disponibles comercialmente y ampliamente utilizados<...
Divulgaciones
El Dr. Richard Taschereau es consultor de Sofie Biosciences y Xodus Imaging. El Dr. Arion F. Chatziioannou es uno de los fundadores de Sofie Biosciences.
Agradecimientos
Agradecemos a todos los miembros del Centro de Tecnología de Imágenes Preclínicas Crump de UCLA por su ayuda y apoyo. En particular, agradecemos a Mikayla Tamboline e Isabel Day por preparar a los animales para las imágenes de TC cardíaca y agradecemos a Sophie Shumilov por generar algunas de las ROI del ventrículo izquierdo durante el estudio. También agradecemos a los Dres. Arjun Deb y Yijie Wang (UCLA) por proporcionar los modelos murinos de lesión cardíaca isquémica aguda para la obtención de imágenes de microTC de calcificación cardíaca. Este trabajo cuenta con el apoyo de la Subvención del Centro Oncológico de los NIH (2 P30 CA016042-44).
Materiales
| Name | Company | Catalog Number | Comments |
| C57BL/6J mice | Jackson Laboratory | 664 | Male, 8 weeks old, 24-26 g |
| Dexela camera | Varex | 1512 | Detector, 20 ms exposure, 74.8/149.6 µm pixel |
| VivoVist | Nanoprobes | 1301-5X0.25ML | CT Contrast agent |
| X-ray source | Moxtek | TUB00082 | 50 kV peak, 200 µA, 1.0 mm-thick Al filter |
Referencias
- Schambach, S. J., Bag, S., Schilling, L., Groden, C., Brockmann, M. A. Application of micro-CT in small animal imaging. Methods. 50 (1), 2-13 (2010).
- Koba, W., Jelicks, L. A., Fine, E. J. MicroPET/SPECT/CT imaging of small animal models of disease. Am J Pathol. 182 (2), 319-324 (2013).
- Hutchins, G. D., Miller, M. A., Soon, V. C., Receveur, T. Small animal PET imaging. ILAR J. 49 (1), 54-65 (2008).
- Franc, B. L., Acton, P. D., Mari, C., Hasegawa, B. H. Small-animal SPECT and SPECT/CT: important tools for preclinical investigation. J Nucl Med. 49 (10), 1651-1663 (2008).
- Badea, C. T. H. L., Hedlund, L. W., Johnson, G. A. Micro-CT with respiratory and cardiac gating: Micro-CT with respiratory and cardiac gating. Med Phys. 31 (12), 3324-3329 (2004).
- Guo, X., Johnston, S. M., Qi, Y., Johnson, G. A., Badea, C. T. 4D micro-CT using fast prospective gating. Phys Med Biol. 57 (1), 257 (2011).
- Drangova, M., Ford, N. L., Detombe, S. A., Wheatley, A. R., Holdsworth, D. W. Fast retrospectively gated quantitative four-dimensional (4D) cardiac micro computed tomography imaging of free-breathing mice. Invest Radiol. 42 (2), 85-94 (2007).
- Blocker, S. J., Holbrook, M. D., Mowery, Y. M., Sullivan, D. C., Badea, C. T. The impact of respiratory gating on improving volume measurement of murine lung tumors in micro-CT imaging. PLoS One. 15 (2), e0225019 (2020).
- Bartling, S. H., et al. Retrospective motion gating in small animal CT of mice and rats. Invest Radiol. 42 (10), 704-714 (2007).
- Kuntz, J., et al. Fully automated intrinsic respiratory and cardiac gating for small animal CT. Phys Med Biol. 55 (7), 2069 (2010).
- Hahn, A., Sauppe, S., Lell, M., Kachelrieß, M. Automatic intrinsic cardiac and respiratory gating from cone-beam CT scans of the thorax region. SPIE Proc. Med Imaging. 9783, 200-205 (2016).
- Maes, F., Vandermeulen, D., Suetens, P. Medical image registration using mutual information. Proc IEEE. 91 (10), 1699-1722 (2003).
- Sheng, J., Chen, B., Ma, Y., Shi, Y. A novel reconstruction approach combining OSEM and split Bregman method for low dose CT. Biomedical Signal Processing and Control. 62, 102095 (2020).
- Romdhane, H., Cherni, M. A., Sallem, D. B. On the efficiency of OSEM algorithm for tomographic lung CT images reconstruction. 2016 Second International Image Processing, Applications and Systems (IPAS). , (2016).
- Ewald, A. J., Werb, Z., Egeblad, M. Monitoring of vital signs for long-term survival of mice under anesthesia. Cold Spring Harb Protoc. 2, 5563 (2011).
- Suckow, C., et al. Multimodality rodent imaging chambers for use under barrier conditions with gas anesthesia. Mol Imaging Biol. 11 (2), 100-106 (2009).
- Owen, M. . Practical signal processing. , 81 (2007).
- Feldkamp, L. A., Davis, L. C., Kress, J. W. Practical cone-beam algorithm. J Opt Soc Am. A-optics Image Sci Vision. 1 (6), 612-619 (1984).
- Loening, A. M., Gambhir, S. S. AMIDE: A free software tool for multimodality medical image analysis. Mol Imaging. 2 (3), 131-137 (2003).
- Rodriguez-Molares, A., et al. The generalized contrast-to-noise ratio: A formal definition for lesion detectability. IEEE Trans Ultrason Ferroelectr Freq Control. 67 (4), 745-759 (2020).
- Patterson, M. S., Foster, F. S. The improvement and quantitative assessment of B-mode images produced by an annular array/cone hybrid. Ultrason Imaging. 5 (3), 195-213 (1983).
- . Dragonfly 2022.2 Available from: https://www.theobjects.com/dragonfly (2022)
- Pillai, I. C. L., et al. Cardiac fibroblasts adopt osteogenic fates and can be targeted to attenuate pathological heart calcification. Cell Stem Cell. 20 (2), 218-232 (2017).
- Li, S., et al. A humanized monoclonal antibody targeting an ectonucleotidase rescues cardiac metabolism and heart function after myocardial infarction. Cell Rep Med. 5 (11), 101795 (2024).
- Nadkarni, R., Clark, D. P., Allphin, A. J., Badea, C. T. Investigating deep learning strategies for fast denoising of 5D cardiac photon-counting micro-CT images. Phys Med Biol. 69 (20), 205010 (2024).
Reimpresiones y Permisos
Solicitar permiso para reutilizar el texto o las figuras de este JoVE artículos
Solicitar permisoExplorar más artículos
This article has been published
Video Coming Soon
ACERCA DE JoVE
Copyright © 2025 MyJoVE Corporation. Todos los derechos reservados