Method Article
Développement de l'imagerie automatisée et d'analyse pour les écrans chimiques du poisson zèbre.
Dans cet article
Résumé
Nous rapportons le développement d'un système automatisé pour l'imagerie et l'analyse des embryons de poisson zèbre transgénique dans des plaques multipuits. Ceci démontre la capacité de mesurer les effets dose-dépendante d'une petite molécule, la BCI, le Fibroblast Growth Factor expression du gène rapporteur et de fournir la technologie pour établir des écrans à haut débit chimiques du poisson zèbre.
Résumé
Nous démontrons l'application de la base d'images à haute teneur de dépistage (HCS) une méthodologie pour identifier de petites molécules qui peuvent moduler la voie FGF / RAS / MAPK dans des embryons de poissons zèbres. L'embryon de poisson zèbre est un système idéal pour les écrans chimiques in vivo à haute teneur. L'embryon de 1 jour est d'environ vieille 1mm de diamètre et peuvent être facilement rangés dans des boîtes de 96 puits, un format standard pour le criblage haut débit. Durant la première journée de développement, les embryons sont transparents avec la plupart des principaux organes présents, permettant ainsi la visualisation de la formation des tissus durant l'embryogenèse. L'automatisation complète d'écrans chimiques poisson zèbre est toujours un défi, cependant, en particulier dans le développement de l'acquisition automatique d'images et d'analyse. Nous avons déjà généré une ligne journaliste transgénique qui exprime la protéine fluorescente verte (GFP) sous le contrôle de l'activité du FGF et démontré leur utilité dans les écrans chimiques
Protocole
1) Mettre en place des accouplements de poisson zèbre et le traitement de BCI.
- Deux jours avant le traitement, identifier les homozygotes mâles Tg (dusp6: d2eGFP) PT6 poisson zèbre et le lieu individuellement dans une réservoirs d'accouplement. Ajouter séparateur et un type de poisson sauvage AB * femelle et laisser dans des cages d'accouplement pendant la nuit.
- Le lendemain matin enlever le séparateur, et de recueillir des embryons après 1 heure. Transfert des embryons à des plats 100mm avec l'E3 (5 mM de NaCl, 0,17 mM de KCl, 0,33 mM de CaCl2, 0,33 mM MgSO4), solution et incuber à 28 ° C pendant 6 heures. Retirer morts et non fécondés embryons, ajouter la solution E3 frais, et incuber pendant la nuit.
- Trier embryons transgéniques pour le stade similaire (24hpf) et expression de la GFP uniforme. Embryons tableau individuellement dans chaque puits d'une plaque de 96 puits.
- Retirer l'excédent de solution E3 avec une micropipette. Ajouter 200 ul de solution E3 avec une pipette multi-canaux dans chaque puits.
- Ajouter les composés énumérés ci-après:
Les concentrations finales sont 1 uM, 5 um, 10 uM, 20μM, épaisseur 25 BCI dans le DMSO à 1%. Recouvrir la plaque dans du papier aluminium et laisser incuber pendant 6 heures à 28 ° C.1 2 3 4 5 6 7 8 9 10 11 12 Une 2μl
DMSO2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
DMSOB 2μl
DMSO2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
DMSOC 2μl
DMSO2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
DMSOD 2μl
DMSO2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
DMSOE 2μl
DMSO2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
DMSOF 2μl
DMSO2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
DMSOG 2μl
DMSO2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mM)2μl
DMSOH 2μl
DMSO2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
BCl
(0.1mm)2μl
BCl
(0.5mm)2μl
BCl
(1 mM)2μl
BCl
(2mm)2μl
BCl
(2.5mm)2μl
DMSO
2) l'imagerie automatisée des plaques de 96 puits et d'analyse.
- Ajouter 10 pi de tricaïne (0,61 mm) à chaque puits pour anesthésier embryons 30hpf.
- Charge plaque dans Molecular appareils ultra Imagexpress.
- Ouvrez le logiciel Metaxpress. Choisissez une configuration avec un objectif 4x et un diamètre maximum de sténopé, configurer laser autofocus pour détecter plaque inférieure, et de déterminer optimale plan Z pour détecter des régions d'intérêt. Acquérir les images au excitation / émission longueurs d'onde de 488/525 nm (GFP). Set zone de numérisation comme un site unique de 2000x2000 pixels (4 x 4 mm), sans binning. Configurer la puissance du laser et le gain de telle sorte que les régions plus brillants dans les images de contrôle positif ne saturent pas. Confirmer l'acquisition d'image correcte dans plusieurs puits de contrôle positif et négatif.
- Démarrer l'analyse assiette. L'instrument sera automatiquement acquérir des images de tous les puits et les archiver dans une base SQL server.
- Upload images en utilisant Definiens Developer le module d'application Cellenger et un filtre d'importation qui reconnaît le format d'image et de la structure des fichiers générés par la plateforme Molecular Devices Imagexpress Ultra.
- Traiter les images avec un algorithme personnalisé destiné Cognition Network Technology (également appelé un jeu de règles). L'algorithme, qui nous personnalisée développée basée sur un ensemble de règles récemment publié 2, est conçu pour identifier automatiquement l'embryon, le jaune, et la tête, et de quantifier la luminosité et le domaine de la GFP exprimant structures de la tête. Les puits dans lesquels aucun embryon n'est détecté ou si la tête ne peut être affecté sans ambiguïté (par exemple, avec une toxicité élevée ou composé d'images floues) sont exclus de l'analyse. Configurer l'algorithme utilisant plusieurs images de contrôle négatif (traités par le véhicule) et des images de contrôle positif (BCI traités) de telle sorte que la tête et le jaune sont correctement identifiés dans les deux. Régler une «tête de structures« seuil de détection comme des multiples d'intensité jaune signifie que la détection des structures de la tête lumineuse correspond à l'observation visuelle. Définir les paramètres d'exportation. Dans cet exemple, le paramètre le plus informatif a été la luminosité intégrée totale de la GFP-positives dans la tête des régions ci-après appelé «tête de luminosité totale des structures» (figure 1).
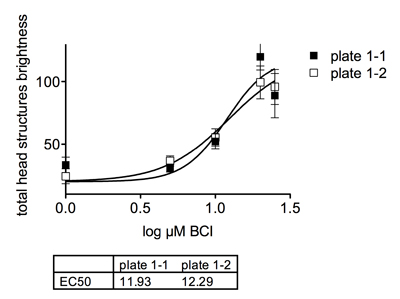
Figure 1
Graphique depicitng les effets dose-dépendante de la BCI sur l'expression de GFP dans les embryons transgéniques. - Exécuter l'algorithme sur toutes les images plaque à l'aide du planificateur intégré d'emplois. Le logiciel Definiens utilise l'informatique distribuée sur plusieurs processeurs («moteurs»), permettant l'analyse simultanée de plusieurs images. L'algorithme calcule les mesures numériques basés sur les résultats de l'analyse d'image, qui sont enregistrées dans la base de données avec une copie de l'image traitée, avec l'analyse appliquée.
Les résultats représentatifs:
Après toutes les images sont analysées, les données peuvent être visualisées au sein Cellenger ou exportées vers un logiciel tiers (Excel, GraphPad Prism) pour une analyse approfondie et de représentation graphique. La figure 2 montre les images archivées numériser à partir d'un véhicule et un traité BCI-bien traités, avec et sans CNT analyse appliquée. Figure 1 documents un effet dose-dépendant de la BCI sur l'expression du gène rapporteur GFP dans les Tg (dusp6: d2EGFP) PT6 embryons. La concentration nécessaire pour obtenir une réponse maximale moitié (50) était d'environ 12 um. 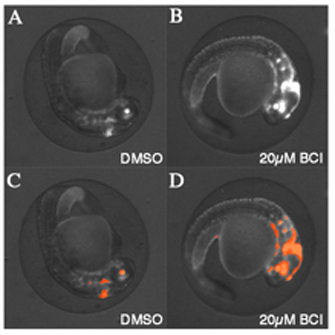
Figure 2
Capture d'image automatisée et l'analyse des embryons de poissons zèbres transgéniques. (A) du véhicule DMSO traitée Tg (dusp6: d2eGFP) PT6 embryon au 30hpf. (B) des embryons transgéniques traitées avec 20μM BCI montrent des augmentations dans l'expression de la GFP. (C) image de l'embryon à partir de (A) après l'exécution de Definiens ruleset de trouver expression de la GFP dans la tête. Taches orange indiquent où le logiciel mesure l'intensité de la GFP. (D) image de l'embryon à partir de (B) après l'exécution de Definiens ruleset de trouver expression de la GFP dans la tête. Taches orange indiquent où le logiciel mesure l'intensité de la GFP. Notez que le jeu de règles a été en mesure de trouver un espace élargi de l'expression de GFP dans les embryons traités.
Discussion
Un des principaux facteurs limitant le débit d'écrans chimiques poisson zèbre est l'absence de méthodologie pour traiter systématiquement les plaques 96 puits pour l'imagerie et l'analyse. Parce que les images des organismes multicellulaires complexes, faible contraste, et de nature hétérogène, les algorithmes existants d'analyse d'image ne parviennent pas à détecter et quantifier des structures spécifiques au sein de l'organisme. La plupart des écrans chimiques publiée le poisson-zèbre donc impliquer un examen visuel de chaque puits par un personnel dévoué pendant toute la procédure. Cela empêche généralement la génération de lectures numériques et un archivage complet des images d'écran et les données. Par ailleurs, l'évaluation manuelle élimine plusieurs avantages clés de l'image basée sur le dépistage, à savoir la capacité d'effectuer des analyses rétrospectives de performances de l'écran, pour examiner les changements phénotypiques qui ne sont pas l'objectif principal de la campagne de dépistage (par exemple, la toxicité ou des anomalies du développement) et de traverser référence les données de dépistage avec des écrans passées ou à venir en utilisant la bibliothèque même composé.
Dans ce rapport, nous mettons en évidence la méthodologie pour l'imagerie automatisée et d'analyse des embryons de poissons zèbres en plaques multipuits, sans intervention de l'utilisateur. Nous capture d'image automatisée sur un laser ultra ImageXpress confocale à balayage lecteur (Molecular Devices, Sunnyvale, CA) à l'image de Tg (dusp6: d2EGFP) PT6 embryons en plaques de 96 puits et développé un algorithme d'analyse d'images basée sur la technologie Definiens «Cognition réseau que la GFP quantifiés expression dans les têtes des embryons transgéniques. La méthode livré des réponses classées et quantifié l'activité in vivo d'une petite molécule d'activateur de FGF de signalisation. Des résultats similaires ont été obtenus avec la II épifluorescence basé ArrayScan (Cellomics Inc, Pittsburgh, PA) documentant qu'il est possible de mettre en oeuvre la capture d'image automatisée des embryons de poissons zèbres sur une variété de lecteurs de plaques disponibles dans le commerce 2. Le développement de la méthodologie pour analyser automatiquement les images organisme multicellulaire éliminé le besoin de notation visuelle par un observateur humain et a permis à la génération et l'archivage des données numériques et des images pour l'analyse rétrospective et des comparaisons avec des écrans de demain.
Remerciements
Ce travail est financé par le NICHD / NIH (1R01HD053287) pour Hukriede, NCI / NIH (P01 CA78039) Vogt, et NHLBI / NIH (1R01HL088016) pour Tsang.
matériels
| Name | Company | Catalog Number | Comments |
| Tricaine (MS222) | Sigma-Aldrich | Cat.# A-5040 |
Références
- Molina, G. A., Watkins, S. C., Tsang, M. Generation of FGF reporter transgenic zebrafish and their utility in chemical screens. BMC Dev Biol. 7, 62-62 (2007).
- Vogt, A. Automated image-based phenotypic analysis in zebrafish embryos. Dev Dyn. 238, 656-663 (2009).
- Molina, G. Zebrafish chemical screening reveals an inhibitor of Dusp6 that expands cardiac cell lineages. Nat Chem Biol. 5, 680-687 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon