Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Colonie de cellules de formage (CFC) Dosage pour les cellules hématopoïétiques humaines
Dans cet article
Résumé
Les cellules formant colonie (CFC) de dosage est un dosage in vitro dans les progéniteurs hématopoïétiques, qui forment des colonies dans un milieu semi-solide. Une combinaison de la morphologie des colonies, la morphologie cellulaire et la cytométrie de flux sont utilisés pour évaluer la capacité des progéniteurs à proliférer et se différencier ainsi les différentes lignées hématopoïétiques.
Résumé
Humaines hématopoïétiques cellules souches / progénitrices sont généralement obtenus à partir de la moelle osseuse, sang du cordon ou du sang périphérique et sont utilisées pour étudier l'hématopoïèse et la leucémogenèse. Ils ont la capacité de se différencier en lignées lymphoïdes et myéloïdes. Les cellules formant colonie (CFC) de dosage est utilisé pour étudier la prolifération et la différenciation des progéniteurs tendance hématopoïétique par leur capacité à former des colonies dans un milieu semi-solide. Le nombre et la morphologie des colonies formées par un nombre fixe de cellules d'entrée fournir des informations préliminaires sur la capacité des progéniteurs de différencier et de proliférer. Les cellules peuvent être récoltées à partir des colonies individuelles ou de la plaque entière pour mieux évaluer leur nombre et les états de différenciation par cytométrie en flux et l'évaluation morphologique de Giemsa lames colorées. Ce test est utile pour évaluer la différenciation myéloïde mais pas lymphoïdes. Le myéloïde terme dans ce contexte est utilisé dans son sens large pour englober lignées granulocytaire, monocytaire, érythroïdes et mégacaryocytaires.
Nous avons utilisé ce test pour évaluer les effets des oncogènes sur la différenciation des primaires humaines dérivées de cellules CD34 + du sang périphérique. Pour cette fin sont les cellules transduites avec le contrôle de construire soit rétroviraux ou une construction exprimant l'oncogène d'intérêt, dans ce cas-NUP98 HOXA9. Nous employons un vecteur rétroviral couramment utilisés, MSCV-IRES-GFP, qui exprime un ARNm bicistronique qui produit le gène d'intérêt et un marqueur de la GFP. Les cellules sont pré-activés par de plus en plus la présence de cytokines pendant deux jours avant transduction rétrovirale. Après deux jours, les cellules GFP + sont isolés par tri cellulaire par fluorescence (FACS) et mélangé avec un milieu semi-solide contenant méthylcellulose complétées par des cytokines et incubés jusqu'au colonies apparaissent à la surface, généralement 14 jours. Le nombre et la morphologie des colonies sont documentés. Les cellules sont ensuite retirés des plaques, lavés, comptés, et soumis à la cytométrie en flux et l'examen morphologique. La cytométrie en flux avec des anticorps spécifiques à la surface cellulaire des marqueurs exprimés au cours de l'hématopoïèse fournit des informations sur la lignée et le stade de maturation. Des études morphologiques des cellules individuelles au microscope après coloration de Wright-Giemsa fournir des informations supplémentaires à l'égard de la lignée et la maturation. Comparaison des cellules transduites avec le vecteur contrôle vide à ceux transduites avec un oncogène révèle les effets de l'oncogène sur la différenciation hématopoïétique.
Protocole
1. PREPARATION DES REACTIFS
- Préparer des solutions stériles stock de 1000x de FMS liées tyrosine kinase 3 (FLT-3) ligand, granulocytes / macrophage colony-stimulating factor (GM-CSF), stem cell factor (SCF), la thrombopoïétine (TPO), l'interleukine (IL) -3 , et l'IL-6 conformément aux instructions du fabricant. Préparer une solution stérile d'actions Retronectin (1mg/ml) aussi selon les instructions du fabricant. Divisez ces solutions de stock en petites fractions aliquotes et de garder à -20 ° C pour éviter répétés de gel-dégel.
- Préparer FBS 2% en IMDM.
- Préparer milieu IMDM complet avec des concentrations finales de 20% de FBS, 2 mM de glutamine, 100 unités / ml de pénicilline / streptomycine (PS). Ajouter les cytokines suivantes juste avant utilisation: 100 ng / ml FLT-3 ligand, 20 ng / ml de GM-CSF, 100 ng / ml EFC, 100 ng / ml de TPO, 50 ng / ml d'IL-3, et 100 ng / ml IL-6.
- Préparer 1% de BSA dans du HBSS (Ca & Mg-libre, sans rouge de phénol) et BSA 2% en D-PBS (Ca & Mg-libres). Stériliser les solutions en passant par des filtres de 0,22 um seringue et conserver à 4 ° C.
- Préparer EDTA à 0,02% dans HBSS et stocker à 4 ° C.
- Préparer GALV pseudotypé rétrovirus codant l'ADN d'intérêt et de stocker en aliquots à -80 ° C. Cette procédure est en dehors de la portée de cet article, mais il est décrit ailleurs (1-3).
Pour la coloration de Giemsa
- Juste avant l'utilisation, filtrer la solution de Wright / Giemsa à deux reprises à 1 um papiers filtres dans une bouteille en verre propre.
- Immédiatement avant utilisation, préparer Wright / Giemsa tampon pH 6,4, en mélangeant 50 ml de la filtrée Wright / Giemsa solution de teinture et 200 ml de solution tampon phosphate, pH 6,4 - Giordano formule.
- Préparer une solution tampon phosphate, pH 6,0 en dissolvant 27,3 g de KH 2 PO 4 et 4,62 g de Na 2 HPO 4 en 3,5 LH 2 O.
- Préparer méthanol à 50% dans H 2 O.
2. PROCÉDURE
La décongélation et la pré-activation des cellules CD34 +
- Décongelez un flacon de cellules CD34 + congelés rapidement à 37 ° C en agitant jusqu'à ce qu'un cristal de glace dernier petit est à gauche et le transfert de la suspension cellulaire (1 ml) à un tube conique de 50 ml. Rincer délicatement les cellules restantes hors de la fiole avec 1 ml de la température ambiante 2% de FBS / IMDM et l'ajouter goutte à goutte sur le tube de 50 ml, tandis que tourbillonnant doucement. Attendre 3 minutes. Ensuite, ajoutez lentement 2 ml de 2% de FBS / IMDM, tout en mélangeant doucement, et attendre 3 minutes. Répétez la procédure en ajoutant 2% de FBS / IMDM qui est le même volume que la suspension cellulaire diluée à intervalles de 3 min, en remuant délicatement entre les deux ajouts, jusqu'à ce que le volume final atteint 32 ml.
- Centrifuger à 250 xg pendant 10 minutes à température ambiante et éliminer le surnageant en laissant derrière autour de 0,5 ml.
- Laver le culot par mise en suspension dans 20 ml de 2% de FBS / IMDM et centrifugation à 200 xg pendant 8 minutes à température ambiante.
- Enlever le surnageant et les cellules en suspension en milieu IMDM complet à environ 0,5 x 10 6 cellules / ml. Prenez 10 pi de la suspension de cellules dans un microtube, mélanger avec le même volume de solution de bleu trypan (0,4%) et de compter à l'aide d'un hémocytomètre.
- Diluer les cellules à 0,1-0,15 x 10 6 cellules / ml par addition de milieu IMDM complet additionné de cytokines (voir 1.3)
- Culture des cellules dans un incubateur humidifié 37 ° C avec 5% de CO 2 pendant 2 jours.
Transduction rétrovirale par le virus de pré-chargement
- Le jour de la transduction, compter les cellules avant de commencer le virus de préchargement pour déterminer le nombre de puits d'être préparé.
- Diluer la solution mère Retronectin à 25 ug / ml et ajouter 400 ul à chaque puits d'une de 24 puits non traités plaque de culture de tissus. Incuber pendant 2 heures à température ambiante dans la hotte à flux laminaire de biosécurité à la pré-couche.
- Retirer la solution Retronectin et ajouter 400 ul de BSA 2% en D-PBS dans chaque puits. Incuber pendant 30 minutes à température ambiante.
- Dégel des solutions stock de virus et de garder sur la glace. Retirer la solution de BSA, ajoutez 0,5 ml de préparation de virus dans chaque puits, et centrifuger à 2200 xg à 4 ° C pendant 15 minutes.
- Retirer la solution de virus provenant de puits et de répéter le chargement de virus décrit dans la version 2.10 plus de trois fois.
- Rincez chaque puits de froid HBSS 0,5 ml (+ Ca et Mg) ou IMDM.
- Recueillir les CD34 pré-activé des cellules + par centrifugation à 200 xg pendant 8 minutes à température ambiante.
- Resuspendre les cellules à 0,1 à 0,15 x 10 6 cellules par 1,5 ml de milieu IMDM complet avec des cytokines.
- Ajouter 1,5 ml de suspension cellulaire dans chaque puits et incuber dans un incubateur humidifié 37 ° C avec 5% de CO 2 pendant 2 jours.
Le tri de cellules et la collecte de cellules transduites
- Doucement mais bien de suspendre les cellules dans le dessus de la plaque chargée viruss et filtrer à travers un filtre cellulaire CellTrics 50 um dans un tube conique. Prenez 10 pi de la suspension de cellules dans un microtube, mélanger avec un volume égal de solution de bleu trypan et compter à l'aide d'un hémocytomètre. Rincez chaque puits avec 0,8 ml d'EDTA 0,02% à froid / HBSS (Ca & Mg-libres sans rouge de phénol) et ajouter au tube même collection à travers un filtre cellulaire CellTrics 50 pm.
- Centrifuger les cellules à 200 xg pendant 8 minutes à 4 ° C, et laver le culot avec le froid HBSS (Ca & Mg-libres sans rouge de phénol) par centrifugation à 200 xg pendant 8 minutes à 4 ° C.
- Reprendre le culot dans 1% de BSA / HBSS (Ca & Mg-libres sans rouge de phénol) à une concentration de 15 X 10 6 / ml ou un volume minimum de 0.250 ml pour le tri cellulaire. Gardez les cellules sur la glace dans l'obscurité. Préparer un tube contenant 20% de FBS froide / IMDM / glutamine / PS moyennes pour la collecte des cellules triées. Le tri est effectué à la base Trieuse vitesse élevée des cellules de l'Alvin J. Siteman Cancer Center à Washington University School of Medicine en utilisant un MoFlo trieuse à haute vitesse (Dako, Glostrup, Danemark).
CFC de dosage
- Décongelez le nombre requis de trois aliquotes ml de milieu de dosage de CFC. Vortex vigoureusement pour mélanger et laisser les tubes reposer au moins 5 minutes pour laisser les bulles présentes à la naissance à la surface avant d'ajouter les cellules.
- Pour obtenir la concentration de cellules pour chaque échantillon trié, divisez le nombre de cellules fournies par le centre de tri par le volume de suspension cellulaire approximative. Prenez-virus 3000 transduites, les cellules triées dans un microtube stérile contenant 2% de FBS froide / IMDM. Pré-régler le volume de 2% de FBS / IMDM sorte que le volume final de suspension sera d'environ 0,3 ml.
- Suspendre les cellules et le transfert de l'ensemble 0,3 ml de suspension cellulaire à une aliquote de 3 ml de Methocult GF + H4435, qui se compose de méthylcellulose à 1%, 30% de FBS, 1% de BSA, 10 -4 M de 2-mercaptoéthanol, 2 mM L-glutamine, 50 ng / ml EFC, 20 ng / ml de GM-CSF, 20 ng / ml d'IL-3, 20 ng / ml d'IL-6, 20 ng / ml de G-CSF, et de 3 unités / ml en IMDM érythropoïétine. Homme Méthylcellulose milieux enrichis (R & D Systems) qui se compose de la méthylcellulose 1,3%, 25% de FBS, 1% de BSA, 5 x 10 -5 M de 2-mercaptoéthanol, 2 mM L-glutamine, 50 ng / ml EFC, 20 ng / ml de GM-CSF, 20 ng / ml d'IL-3, 20 ng / ml d'IL-6, 20 ng / ml de G-CSF, et de 3 unités / ml en IMDM érythropoïétine peuvent être également utilisés.
- Vortex vigoureusement pour rendre le mélange parfaitement montée et la chute de 3-4 fois. Laisser le tube immobile pendant 3 min.
- Joindre un calibre 16 à bout émoussé l'aiguille d'une seringue de 3 ml et tirer en hausse de 2,2 ml. Ne pas établir de grosses bulles; les expulser au début en appuyant sur une couple de fois. Poussez 1,1 ml chacun en deux 30 mm non traitée plat et étaler le mélange uniformément en tournant.
- Placez les plaques reproduire dans une plaque de 100 mm avec un plat d'eau contenant 3 ml d'eau stérile. Culture pour 14 - 17 jours.
- Caractériser et le score des colonies en fonction de leur morphologie avec un microscope inversé à un grossissement de 40x dans un plat culture marquée par une grille de notation. Aux fins de notre analyse, les colonies sont classés en 3 catégories: pure érythroïdes, myélomonocytaire, et mixtes. Voir les instructions du fabricant pour la colonie sous-classification plus loin.
- La plaque de CFC ensemble de dosage peuvent être numérisés sans grossissement en utilisant un scanner régulièrement à 600 ppp, et de faible puissance (40x) microphotographies des colonies peuvent être prises à l'aide d'un microscope inversé équipé d'une caméra couleur.
- Pour une analyse plus poussée de la différenciation et la prolifération, les cellules de la plaque de dosage CFC ensemble sont récupérés par mise en suspension dans plusieurs volumes de la température ambiante 2% de FBS / IMDM. Après centrifugation à 300 xg pendant 10 min à 4 ° C, les cellules sont remises en suspension dans 2% de FBS / IMDM, compté, et soit colorées avec des anticorps pour la cytométrie de flux ou transférés à diapositives à l'aide d'une centrifugeuse Cytospin pour la coloration de Giemsa.
Coloration des anticorps et de cytométrie en flux
- Centrifuger les cellules à 200 xg pendant 8 min à 4 ° C et remettre en suspension dans du sérum de souris à froid 50% de la normale dans HBSS (Ca & Mg-libres sans rouge de phénol).
- Placer environ 5 x 10 5 cellules dans 80 ul par tube (12 x 75 mm à fond rond tubes) et de garder sur la glace. Ajouter le mélange d'anticorps (environ 20 ul mélange ou une solution de contrôle négatif sans anticorps anti-contrôles) et mélanger en tapotant légèrement les tubes. Préparer des tubes de contrôle d'étalonnage sans anticorps ou avec un anticorps anti-CD45 pour chaque fluorochrome. Placer les tubes sur la glace dans l'obscurité et incuber pendant 20 minutes. Mélanger en tapotant légèrement les tubes après les 10 premières minutes. Après l'incubation de 20 min, remplir les tubes avec HBSS froid et centrifuger à 200 xg pendant 8 min à 4 ° C. Reprendre le culot dans 0,5% paraformaldéhyde froid / HBSS.
Les mélanges suivants d'anticorps sont généralement utilisés pour notre analyse. Pour la différenciation myéloïde: CD11b (phycoérythrine conjuguée clone ICRF44) / CD33 (allophycocyanin conjugué clone WM53) / CD45 (phycoérythrine-Cy7-congjugated clone HI30). Pour la différenciation érythroïde: CD71 (phycoérythrine conjuguée clone M-A712) / CD235a (allophycocyanine conjugué clone GA-R2) / CD45 (phycoérythrine-Cy7-congjugated clone HI30). - La cytométrie en flux est réalisée au cœur Trieuse vitesse élevée des cellules de l'Alvin J. Siteman Cancer Center à Washington University School of Medicine sur un cytomètre de flux FACScan amélioré à 5 couleurs et deux lasers (BD Biosciences) et analysées en utilisant CellQuest (BD Biosciences) ou FlowJo v7.2.4 (Arbre Star, Inc, Ashland, OR, USA) de logiciels.
La coloration de Giemsa
- Environ 30 000 cellules récupérées à partir des plaques d'essai décrites ci-dessus CFC sont en suspension dans 0,3 ml de 2% de FBS / IMDM et transféré à une diapositive à l'aide d'une centrifugeuse Cytospin à 1000 rpm pendant 10 min à température ambiante.
- Mettre en place des neuf navires coloration des solutions contenant dans l'ordre suivant.
- Méthanol absolu
- Wright / Giemsa solution stock
- Wright / Giemsa tampon, pH 6,4
- Méthanol à 50% dans H 2 O
- H 2 O
- H 2 O
- Tampon phosphate, pH 6,0
- H 2 O
- H 2 O
- Placer les lames dans un transporteur diapositives et les tremper dans du méthanol absolu (# 1) pendant 2 min et éponger hors méthanol en excès.
- Immédiatement, tremper le transporteur glisser dans Wright / Giemsa solution stock (# 2) pendant 5 min.
- Transférer le support dans Wright / Giemsa tampon, pH 6,4 (# 3) et incuber pendant 10 min.
- Tremper le transporteur à deux reprises en 50% de méthanol (# 4), 10 fois en H 2 O (# 5), un autre 10 fois en H 2 O (# 6), puis 5 fois dans un tampon phosphate, pH 6,0 (# 7) .
- Transfert du transporteur en H 2 O (# 8) et incuber pendant 2 min. Répéter le lavage dans H 2 O (# 9).
- Autoriser les diapositives à l'air complètement sec dans le support. Retirez chaque diapositive et essuyez le dos de la lame avec du méthanol imbibés Kimwipes pour enlever les taches. Déposer une goutte de Cytoseal 60 et un couvercle en verre pour sceller.
- Une formule leucocytaire de 500 cellules est effectué pour chaque diapositive colorés au Giemsa en utilisant un microscope Olympus BX51. Les cellules sont divisées en cinq catégories: les cellules primitives comprennent explosions et promyélocytes; cellules myéloïdes intermédiaires comprennent myélocytes et métamyélocytes; cellules myéloïdes matures comprennent des bandes, des polynucléaires neutrophiles, les monocytes et les macrophages; intermédiaires comprennent les cellules érythroïdes cellules avec hemoglobinization intermédiaires, et les cellules érythroïdes matures comprennent cellules avec hemoglobinization plein. Photomicrographies sont prises avec un appareil photo Olympus Dp71 avec un objectif 60x du pétrole.
3.0) des résultats représentatifs:
- Dosage de CFC: Certains de la population cellulaire d'entrée vont proliférer, se différencier, et forment des colonies sur le milieu semi-solide lors de la période d'incubation de 14 à 17 jours. Dans cette expérience, il est évident que l'expression de l'oncogène NUP98-HOXA9 provoqué la formation de premier plan rouge (colonies érythroïdes) (figure 1) (3).
- Immunofluorescence et cytométrie de flux: immunophénotypage par cytométrie en flux des cellules prélevées à partir des plaques d'essai CFC fournit des informations sur l'état de différenciation de la population cellulaire. En utilisation combinée des anticorps contre les marqueurs de différenciation de certains, la lignée et le degré de maturation des cellules peuvent être évalués. Dans ce cas CD235a a été utilisé pour identifier les cellules érythroïdes. Comme le montre la figure 2A, NUP98-HOXA9 causé une augmentation du pourcentage de cellules érythroïdes CD235a +, mais la luminosité de l'expression par ces cellules CD235a a été diminuée par rapport aux contrôles, indiquant une inhibition de la maturation érythroïde. Dans la figure 2B, cellules myéloïdes sont identifiés en vertu de leur positivité CD33. NUP98-HOXA9 a provoqué une diminution du nombre de cellules CD33 +, avec une diminution de la luminosité de l'expression CD11b, compatible avec l'inhibition de la maturation myéloïde (3). Ainsi, les résultats de cytométrie en flux montrent que NUP98-HOXA9 causé une hyperplasie érythroïde avec l'inhibition de la maturation à la fois myéloïdes et érythroïdes.
- Coloration de Giemsa: L'examen morphologique des cellules prélevées à partir des plaques d'essai CFC fournit des informations sur la lignée et le degré de maturation de la population cellulaire. Les conclusions obtenues morphologiquement peuvent différer de celles obtenues par des études de cytométrie en flux, et donc ces méthodes sont complémentaires les unes aux autres (3). L'expérience est répétée au moins 3 fois, et chaque fois qu'une numération différentielle de 500 cellules est réalisée à distinguer cinq catégories de cellules, comme décrit ci-dessus. Des exemples de ces cellules sont fait remarquer dans la figure 3. Les résultats sont compilés et analysés afin de déterminer s'il existe des différences statistiquement significatives entre l'oncogène transduites échantillon et le contrôle. Dans ce cas, les résultats ont montré que NUP98-HOXA9 provoqué une augmentation globale du nombres de cellules, avec hyperplasie érythroïde et l'inhibition de la maturation érythroïde et deux myéloïde (3) (non représentée).
Les légendes des figures

Figure 1: Les effets de la NUP98-HOXA9 sur la morphologie humaine CFC.
Primaires humaines CD34 + ont été transduites avec un retrovirus soit un contrôle MSCV-IRES-GFP vecteur ou vecteur exprimant NUP98-HOXA9, et les cellules ont été triés pour la positivité GFP. Un millier de cellules ont été ensemencées dans chacune des deux plaques en double pour dosage de CFC et de l'expérience a été répétée trois fois quatre indépendants. Représentant plaques sans grossissement (à gauche) et microphotographies de faible puissance du représentant colonies érythroïdes (à droite) sont représentés (3).
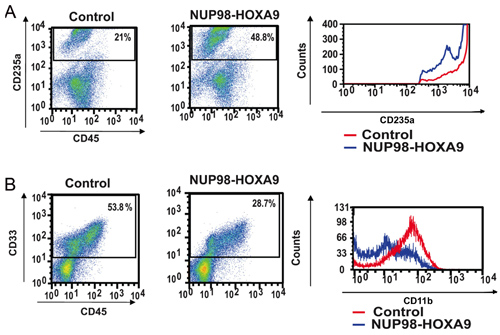
Figure 2: La cytométrie en flux montre perturbation des humains CD34 + primaires différenciation cellulaire par NUP98-HOXA9.
(A) par cytométrie en flux pour la différenciation érythroïde: Cellules des plaques de CFC (voir Figure 1) ont été récoltées et colorées avec des anticorps à CD45 et CD235a. Le CD235a + porte était tracée sur un histogramme (à droite) pour montrer l'expression de CD235a par rapport aux cellules de contrôle (B) par cytométrie en flux pour la différenciation myéloïde: Cellules des plaques de CFC ont été récoltées et colorées avec CD45 et CD33. Le CD33 + porte a été tracée sur un histogramme pour montrer l'expression CD11b par rapport au contrôle (panneau de droite). Les pourcentages de cellules relevant de chaque porte sont représentées (3).
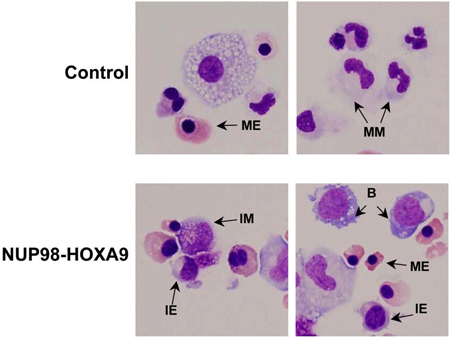
Figure 3: La morphologie cellulaire montre la perturbation de la différenciation par NUP98-HOXA9 par rapport à la lutte antivectorielle.
Frottis ont été préparés à partir Cytospin plaques CFC et colorées au Giemsa. Photomicrographies ont été prélevés dans des champs représentatifs avec un objectif 60x du pétrole. B: explosion; MM: myéloïdes matures, IM: myéloïde intermédiaires, ME: matures érythroïdes, IE: intermédiaire érythroïdes (3).
Discussion
Le dosage de CFC a été largement utilisée pour déterminer la prolifération et la différenciation des modèles de progéniteurs hématopoïétiques et d'étudier les effets des oncogènes (4, 5). Il a l'avantage sur les cultures de liquide d'être un test clonal, telles que les colonies constituent la descendance d'un ancêtre unique et peut être retiré individuellement pour une analyse ultérieure. La limitation du dosage de CFC est qu'elle n'est pas adéquate pour la détection de progé...
Déclarations de divulgation
Remerciements
matériels
| Name | Company | Catalog Number | Comments |
| IMDM | Life Technologies | 12440 | |
| FBS | Stem Cell Technologies | 06150 | |
| L-Glutamine | Life Technologies | 25030 | |
| Penicillin/Streptomycin (PS) | Life Technologies | 15140 | |
| FLT-3 ligand | PeproTech Inc | 300-19 | |
| GM-CSF | PeproTech Inc | 300-03 | |
| SCF | PeproTech Inc | 300-07 | |
| TPO | PeproTech Inc | 300-18 | |
| IL3 | PeproTech Inc | 200-03 | |
| IL6 | PeproTech Inc | 200-06 | |
| Bovine Serum Albumin | Sigma-Aldrich | A7030 | |
| EDTA | Fisher Scientific | BP118 | |
| Retronectin | Takara Bio Inc | T100A | |
| HBSS | Life Technologies | 14175 | |
| PBS | Life Technologies | 14200 | |
| Trypan Blue solution (0.4%) | Sigma-Aldrich | T8154 | |
| Methocult GF+ H4435 | Stem Cell Technologies | 04445 | |
| Human Methylcellulose Enriched Media | R&D Systems | HSC005 | |
| Wright/ Giemsa stain | Harleco | 64571 | |
| Phosphate Buffer Solution, pH 6.4 - Giordano formula | Ricca Chemical Company | 1450 | |
| Methanol | Fisher Scientific | A412-4 | |
| Cytoseal 60 | Thermo Fisher Scientific, Inc. | 8310 | |
| Normal Mouse Serum | Rockland Immunochemicals | D208 | |
| Anti-Human CD11b phyc–rythrin-conjugated | BD Biosciences | 555388 | |
| Anti-Human CD33 allophycocyanin-conjugated | BD Biosciences | 551378 | |
| Anti-Human CD45 phyc–rythrin-Cy7-congjugated | BD Biosciences | 557748 | |
| Anti-Human CD71 phyc–rythrin-conjugated | BD Biosciences | 555537 | |
| Anti-Human CD235a allophycocyanin-conjugated | BD Biosciences | 551336 | |
| Anti-Human CD45 phyc–rythrin-Cy7-congjugated | BD Biosciences | 557748 | |
| 24-well non-treated tissue culture plates | BD Biosciences | 35-1147 | |
| 30 mm non-treated dish | Stem Cell Technologies | 27150 | |
| 100 mm tissue culture dish | Fisher Scientific | 08-757-12 | |
| Gridded scoring dishes | Stem Cell Technologies | 27500 | |
| 15 ml centrifuge tubes | BD Biosciences | 35-2097 | |
| 50 ml centrifuge tubes | BD Biosciences | 35-2070 | |
| Syringes 3 ml | Stem Cell Technologies | 28240 | |
| 16 gauge blunt-end, 1½ inch needle | Stem Cell Technologies | 28110 | |
| 50 μm CellTrics cell filter | Partec | 04-004-2327 | |
| Hemocytometer | Fisher Scientific | 0267110 | |
| TPX sample chambers | Thermo Fisher Scientific, Inc. | A78710018 | |
| Fisherbrand Superfrost/Plus Microscope Slides, Precleaned | Fisher Scientific | 12-550-15 | |
| Shandon filter cards | Thermo Fisher Scientific, Inc. | 5991022 | |
| Shandon cytospin slide holder | Thermo Fisher Scientific, Inc. | 59920063 | |
| Shandon Complete Staining Assembly 100 | Thermo Fisher Scientific, Inc. | 100 | |
| Kimwipes | Kimberly-Clark Corporation | 34155 | |
| 1 μm filter paper | VWR international | 28307-134 | |
| Inverted microscope | Nikon Instruments | Diaphot | |
| Microscope camera | Nikon Instruments | DS-F11 | |
| Microscope | Olympus Corporation | BX51 | |
| Microscope camera | Olympus Corporation | DP71 | |
| Scanner | Microtek | Scanmaker 4 | |
| Vortex mixer | Fisher Scientific | 12-812 | |
| Tissue culture incubator | Sanyo | MCO-18AIC | |
| Cytospin | Shandon, Inc. | Cytospin 2 | |
| Bench-top centrifuge | Eppendorf | 5810-R | |
| Water purification system | Barnstead | Nanopure-Diamond |
Références
- Takeda, A., Goolsby, C., Yaseen, N. R. NUP98-HOXA9 induces long-term proliferation and blocks differentiation of primary human CD34+ hematopoietic cells. Cancer Res. , 66-6628 (2006).
- Yassin, E. R., Abdul-Nabi, A. M., Takeda, A., Yaseen, N. R. Effects of the NUP98-DDX10 oncogene on primary human CD34+ cells: role of a conserved helicase motif. Leukemia. , (2010).
- Yassin, E. R., Sarma, N. J., Abdul-Nabi, A. M., Dombrowski, J., Han, Y., Takeda, A., Yaseen, N. R. Dissection of the transformation of primary human hematopoietic cells by the oncogene NUP98-HOXA9. PLoS One. 4, e6719-e6719 (2009).
- Nissen-Druey, C., Tichelli, A., Meyer-Monard, S. Human hematopoietic colonies in health and disease. Acta Haematol. 113, 5-96 (2005).
- Pereira, C., Clarke, E., Damen, J. Hematopoietic colony-forming cell assays. Methods Mol Biol. 407, 177-208 (2007).
- Coulombel, L. Identification of hematopoietic stem/progenitor cells: strength and drawbacks of functional assays. Oncogene. 23, 7210-7222 (2004).
- Hogge, D. E., Lansdorp, P. M., Reid, D., Gerhard, B., Eaves, C. J. Enhanced detection, maintenance, and differentiation of primitive human hematopoietic cells in cultures containing murine fibroblasts engineered to produce human steel factor, interleukin-3, and granulocyte colony-stimulating factor. Blood. 88, 3765-3773 (1996).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon