Method Article
L'empreinte génétique des Mycobacterium leprae Souches à l'aide variable Tandem Repeat Nombre (VNTR) - Analyse de la longueur des fragments (FLA)
Dans cet article
Résumé
La lèpre, causée par Mycobacterium leprae, Est encore endémique dans de nombreux endroits. Afin d'en apprendre davantage sur la propagation et le mode de transmission de la lèpre, il est important de déterminer quelle souche de M. leprae A infecté un patient. Nombre variable de répétitions en tandem (VNTR) frappe est un tel procédé.
Résumé
L'étude de la transmission de la lèpre est particulièrement difficile car l'agent causal, Mycobacterium leprae, ne peuvent pas être cultivées en laboratoire. Les seules sources de la bactérie sont malades de la lèpre et de tatous infectés expérimentalement et des souris nude. Ainsi, de nombreuses méthodes utilisées en épidémiologie moderne ne sont pas disponibles pour l'étude de la lèpre. Malgré un vaste programme mondial de la drogue traitement de la lèpre en œuvre par l'OMS une, la lèpre reste endémique dans de nombreux pays, avec environ 250 000 nouveaux cas chaque année. 2 L'ensemble de M. leprae génome a été cartographié 3,4 et de nombreux locus ont été identifiés qui ont répété les segments de deux ou plusieurs paires de bases (appelés micro et minisatellites). 5 souches cliniques de M. leprae peut varier dans le nombre de segments répétés en tandem (courtes répétitions en tandem, STR) à plusieurs de ces loci. 5,6,7 répéter en tandem en nombre variable (VNTR) 5 analyse a été utilisée pour distinguer les différentes souches du bacille de la lèpre. Certains des loci semblent être plus stables que d'autres, montrant moins de variation dans le nombre le répète, tandis que d'autres semblent évoluer plus rapidement, parfois chez le même patient. Alors que la variabilité de certains VNTR a soulevé des questions quant à leur aptitude à 7,8,9 typage des souches, les nouvelles données indiquent que l'analyse de loci multiples, qui sont diversifiés dans leur stabilité, peut être utilisé comme un outil précieux épidémiologiques. Plusieurs locus VNTR analyse (MLVA) 10 a été utilisé pour étudier l'évolution de la lèpre et la transmission dans plusieurs pays dont la Chine 11,12, au Malawi 8, les Philippines 10,13, et le Brésil 14. MLVA implique plusieurs étapes. Tout d'abord, l'ADN bactérien est extrait avec l'ADN du tissu hôte à partir de biopsies cliniques ou frottis fente (SSS). 10 Le locus désiré sont ensuite amplifiés de l'ADN extrait par réaction en chaîne de polymérase (PCR). Amorces marqués à la fluorescence pour 4-5 loci différents sont utilisés par réaction, avec 18 loci étant amplifié dans un total de quatre réactions. 10 Les produits de PCR peut être soumis à une électrophorèse sur gel d'agarose pour vérifier la présence des segments d'ADN désirée, puis soumis à une analyse de longueur des fragments fluorescents (FLA) par électrophorèse capillaire. ADN de bactéries de tatou passages avec un nombre connu de copies répéter pour chaque locus est utilisé comme contrôle positif. Les chromatogrammes FLA sont ensuite examinés en utilisant le logiciel du scanner Peak et de longueur des fragments est converti en nombre de copies VNTR (allèle). Enfin, les haplotypes VNTR sont analysés pour les modèles, et lorsqu'ils sont combinés avec les données cliniques des patients peuvent être utilisés pour suivre la distribution des types de souches.
Protocole
Le but de cet article la vidéo est de donner un aperçu du flux de travail avec le format des données et l'interprétation pour les chercheurs qui peuvent être simplement de commencer ce type de travail (figure 1). Il comprend la démonstration de techniques, des protocoles simplifiés et des conseils pratiques décrites dans des ouvrages publiés antérieurement. 5,10
Work Flow général et des installations de laboratoire:
Il devrait y avoir au moins 3 zones de travail séparées pour ce genre de recherche. Le laboratoire devrait avoir 1) une zone pré-PCR avec un capuchon de PCR (boîte à air propre ou zone de travail isolée) pour la préparation de l'amorce (de dilution, la préparation et le mélange aliquote), 2) une bio-sécurité séparée du cabinet pour la manipulation et l'ajout de l'ADN aux mélanges de PCR, et 3) une zone de travail post-PCR pour la préparation et le chargement des gels et de la préparation des échantillons pour FLA. Les amorces et les échantillons d'ADN doivent être conservés dans des congélateurs et des réfrigérateurs séparés. La contamination Primer est un des problèmes principaux et les plus persistantes dans le travail de laboratoire de ce type. Pipettes utilisées pour les apprêts et PCR mélange ne doit PAS être utilisé pour l'ADN. Il devrait y avoir des ensembles distincts de pipettes pour les pré-PCR, PCR et les aires de travail post-PCR. Généralement, un chercheur peut traiter 12 à 18 échantillons dans une période de 12 à 24 heures en utilisant l'équipement de laboratoire standard.
Avant le début de chaque phase de travail:
- Porter une blouse de laboratoire et des gants. Les gants doivent être portés en tout temps des échantillons biologiques, les amorces, l'ADN et le bromure d'éthidium sont traitées. Changer de gants souvent.
- Travailler sous une hotte PCR cabinet, enceinte de sécurité biologique ou de la zone de travail propre.
- Utilisez un papier banc de frais ou pad.
- Mettre en place des poubelles adapté à des liquides et des embouts de pipette.
- Essuyez avec l'intérieur de la hotte PCR et pipettes avec 70% d'éthanol.
- Pipettes Sujet et la zone de travail à la lumière UV pendant 15 minutes avant la PCR mis en place. (Lumière ultraviolette contaminants réticule l'ADN de surface.)
- Toujours utiliser des aérosols embouts de pipette de prévention pour les matériaux contenant de l'ADN, amorces et les réactifs de PCR.
- Centrifuger tous les tubes / bandes / plaques contenant du liquide avant l'ouverture pour empêcher la fuite d'aérosols et la contamination croisée par la manipulation.
1. M. préparation de l'ADN leprae
Les échantillons cliniques contenant M. leprae sont obtenus à partir de patients atteints de lèpre qui visitent les cliniques peau. Routine d'échantillons de diagnostic peuvent être biopsies cutanées, taches cutanées fente ou écouvillons nasaux. Généralement, biopsies cutanées ou de frottis fentes sont les meilleurs pour l'épidémiologie moléculaire, car ils sont propres et contiennent des quantités suffisantes de M. leprae. L'utilisation de ces matériaux pour la recherche doivent être approuvés conformément aux directives institutionnelles.
- Préserver une biopsie ou une fente échantillons de prélèvement cutané dans un flacon à bouchon à vis avec 1 ml d'éthanol à 70%.
- Centrifuger chaque échantillon à une vitesse de paillasse centrifugeuse variable à 12 000 xg pendant 15 minutes.
- Enlever le surnageant et le placer dans un tube à centrifuger. Si nécessaire, il peut être utile de ré-centrifugeuse ces échantillons de tissus et de récupérer l'ADN supplémentaires ou plus tard.
- Ajouter 500 ul de tampon phosphate salin (PBS) pour les échantillons de tissus et laisser tremper pendant 1 heure pour l'échange de conservation éthanol résiduel et réhydrater l'échantillon.
- Centrifuger les échantillons dans une centrifugeuse de paillasse à 12.000 xg pendant 20 minutes. Jeter la solution de PBS dans un conteneur à déchets partiellement rempli avec une solution désinfectante.
- Extraire l'ADN des bactéries en utilisant le sang Qiagen DNeasy et kit de tissus en suivant les directives prescrites.
- L'ADN extrait devrait être répartie dans 4 flacons. Store 2 aliquotes à -80 ° C pour une utilisation future si / quand la reproductibilité des résultats est nécessaire ou lorsque les nouvelles technologies deviennent disponibles. Magasin une aliquote à -20 ° C et une à 4 ° C pour une utilisation plus immédiate.
- Il est prudent de préparer un «vide d'extraction" avec les échantillons de tissus. L'ébauche est soumise à tous les mêmes traitements décrits ci-dessus, mais est réalisée sans tissu. PCR le blanc d'extraction avec les échantillons du patient afin de garantir que la technique d'extraction de l'opérateur est correcte et que les réactifs sont exempts de contamination par l'ADN.
2. Préparation Primer
- Les amorces pour les loci pour lesquels il existe des données les plus complètes de la souche type sont répertoriés dans le tableau 1. Quatre ou cinq amorces sont combinés pour PCR multiplex. Une amorce pour chaque locus porte une étiquette de 5 'chimiques fluorescents qui seront détectés pendant l'analyse par électrophorèse capillaire de longueur de fragment (FLA).
- Pour chaque combinaison de PCR multiplex, l'amorce marqués et correspondant amorce reverse pour chaque locus sont combinés dans des tubes Eppendorf distinctes: amorces sens dans un tube, inverse dans l'autre. Stock amorces sont préparées comme des solutions 100 microns (Figure 2a), dont une partieest dilué 10x à une concentration de 10 uM en utilisant TE (Tris-EDTA 1x, pH 8,0). Aliquotes restantes des 100 solutions apprêt uM sont stockés à -20 ° C pour une utilisation ultérieure.
- Des quantités égales de chaque amorce 10 uM sont mélangés résultant en des concentrations finales de 2 pm chaque amorce. Lorsque les combinaisons d'amorces sont ajoutés au mélange de PCR (tableau 3), la concentration finale de chaque amorce chute à 0,2 pM.
Lorsque les amorces combinés sont ajoutés aux mélanges de PCR (tableau 3), les concentrations finales tomber à 0,2 um chacun.
3. Amplifier l'ADN bactérien par PCR multiplex
- PCR Setup
- Notez le nombre d'échantillons qui seront utilisées et préparer une feuille de calcul avec les réactifs nécessaires et leurs volumes avant de collecter les plastiques et les matériaux nécessaires d'autres.
- Etiqueter les tubes stériles / bandes / plaques à être utilisé pour la PCR avec des nombres d'échantillons. N'oubliez pas d'inclure des contrôles positifs et négatifs.
- Essuyez le thermocycleur avec une solution de nettoyage (comme Decon ELIMINase) et le programme qu'il conformément au Tableau 2. Changer de gants après le nettoyage et avant de manipuler des amorces et des autres réactifs.
- Préparer PCR Mastermix selon le tableau 3 et étiqueter les tubes PCR / bandes / plaque avec la combinaison d'amorces et les numéros d'échantillon à utiliser.
- Aliquote de 18μl Mastermix dans chaque tube d'échantillon PCR étiquetés ou bien. Changer de gants.
- Essuyez séparé de bio-sécurité armoire et pipettes avec 70% d'éthanol pour aider à nettoyer et désinfecter la zone de travail et des outils, puis soumise la zone de travail et des pipettes à la lumière UV pendant 15 minutes à réticuler tous les contaminants ADN. Changer de gants.
- Dans le nettoyage bio-sécurité armoire, ajoutez 2μl de la matrice d'ADN à l'Mastermix dans chaque tube de PCR / plaque ainsi d'acquérir un volume total de 20 pi (Figure 2b). Utilisez des conseils de prévention des aérosols pipette pour toutes les matières liquides.
- Centrifuger les tubes PCR / bandes / plaques brièvement pour mélanger le contenu.
- Placer les tubes d'échantillon / bandes / plaque dans le thermocycleur et démarrer le programme de PCR.
- Lorsque le programme est terminé, retirer les produits du thermocycleur et stocker à 4 ° C jusqu'à électrophorèse.
4. L'électrophorèse sur gel des produits de PCR
* Ce protocole a été la norme pendant de nombreuses années et est une étape facultative qui peut être utilisé si la confirmation des produits de PCR est souhaitée avant de les envoyer pour le FLA.
- Dans une zone de travail post-PCR séparées, préparer un gel d'agarose 2%. Utilisez 2.0g agarose powder/100ml de 1x TBE (Tris / borate / EDTA) solution tampon dans un flacon. Chauffer le mélange pendant environ 1,5-2 minutes dans un four à micro-ondes. Agiter le contenu, le chauffage à nouveau si nécessaire pour dissoudre l'agarose. Laisser refroidir légèrement et versez le gel sous une forme à l'aide d'un peigne avec des puits suffisant pour les échantillons.
- Retirer les produits de PCR à partir de 4 ° C et centrifuger pendant 30 secondes environ.
- Mélanger 2-5uL de produit PCR avec 0,5 1 microlitre de tampon de gel de chargement 5x ou 6x (tampon de charge est disponible auprès de diverses entreprises, ou peuvent être mélangés dans le laboratoire. Recettes sont disponibles en ligne.)
- Chargez le puits du gel avec le mélange tampon d'échantillon / chargement 6μl. Ajouter à une échelle moléculaire pour un puits (de préférence une échelle 20 pb).
- Exécutez le gel à 100V pendant environ 90 minutes.
- Faire tremper le gel dans une solution de bromure d'éthidium pendant 15-30 minutes, puis dans l'eau distillée ultrapure pour une quantité égale de temps.
- Le gel d'image tandis que la lumière UV est appliquée. Il devrait y avoir une bande pour chacun des segments d'ADN de 4-5 dans la combinaison (figure 3).
- Éliminer le gel conforme à l'institution dangereuse politique de matériaux. Solution de bromure d'éthidium peut être utilisé à plusieurs reprises avant l'élimination appropriée.
5. Préparation des échantillons pour l'analyse de longueur des fragments (FLA)
- Dans un tube propre Eppendorf, préparer un mélange maître contenant 12μl de Salut-Di solution de formamide à partir d'un aliquote fraîche et 0.3μl de GeneScan -500 standards LIZ dimensionnement (à la fois d'Applied Biosystems) pour chaque échantillon à tester. (Salut-Di formamide dénature chimiquement les brins d'ADN avant d'électrophorèse capillaire, ce qui élimine le besoin de chauffage.) Utilisation d'un 96-puits optique plaque de réaction de la qualité, 12.3μl partie aliquote du mélange formamide-LIZ dans chaque puits étant utilisée pour des échantillons et mis en la plaque de côté.
- Faites un plan de la plaque pour vos dossiers indiquant quelles échantillon d'ADN est dans chaque puits (figure 4a).
- Utilisation de tubes / bandes ou une plaque de 96 puits, ajouter 1 microlitre de produit de PCR (de la partie 3) à 59μl d'eau de qualité PCR créer une dilution 1:60 du produit de PCR. Les dilutions peuvent être ajustés en fonction de la puissance du signal à partir des données FLA ou la luminosité de bandes de gel. Même à des concentrations inférieures de l'ADN peutêtre suffisante (1:120 ou 1:180).
- Ajouter 1 microlitre du produit dilué par PCR à la bonne et dans la plaque contenant le mélange de formamide-LIZ.
- Rapidité et efficacité peut être grandement améliorée si le PCR a également été fait dans une plaque de 96 puits. On peut simplement aligner 3 de telles plaques dans l'ordre: Planche 1 avec des produits de PCR, Planche 2 avec de l'eau stérile pour la dilution des produits de PCR, et la plaque 3 avec le formamide et l'échelle de dimensionnement pour l'analyse de la longueur de fragment. Une pipette multicanaux permet un processus rapide de préparation FLA.
Analyse des échantillons via l'analyseur génétique
- Calibrer l'analyseur génétique pour détecter la flurophores appliquée par les instructions du fabricant. Veillez à utiliser une teinture-ensemble qui inclut LIZ.
- Reconstituer l'eau de rinçage des contenants et d'ajouter de nouvelles tampon courant avec de l'EDTA (1x) (Applied Biosystems) pour amortir les chambres.
- Ajouter un septum pré-fente de silicium à la plaque et placer la plaque dans le plateau de maintien conçu. Appuyez sur le "Plateau" bouton sur l'analyseur génétique pour amener le passeur avant. Bac Placer sur échantillonneur automatique et fermer la porte à l'analyseur génétique.
- Créer ou importer un tableur pour l'analyse. Fichiers importables ont une extension. PLT et sont dans l'onglet format délimité. Les fichiers peuvent être modifiés dans Excel (Microsoft) ou des programmes semblables.
- Les échantillons sont injectés dans le capillaire (50 cm de longueur, POP-7 polymère) en appliquant une tension d'injection de 1,6 kV pendant 15 s. L'électrophorèse capillaire fonctionne à une tension de 15 kV à 60 ° C pendant 1800 secondes. L'ensemble du processus prend environ 45 minutes.
- Une fois une course est terminée, les données pour chaque échantillon analysé sont convertis en fichiers avec une extension. FSA et placés dans un dossier plaque (figure 4b). Chaque fichier de données est d'environ 100 Ko de taille et peut être stocké sur un lecteur flash ou zippé et envoyé par courriel. Les fichiers de données peuvent être visualisées en utilisant un logiciel approprié, tel que GeneMapper ABI ou Scanner Peak.
6. Analyse des résultats de la longueur des fragments
- L'analyse des données de fluorescence électrophorèse capillaire nécessite un logiciel spécial. Si un tel logiciel n'est pas disponible, rendez-vous le site d'Applied Biosystems et téléchargez Scanner Peak. Le logiciel est gratuit et fonctionne très bien. https://products.appliedbiosystems.com/ab/en/US/adirect/ab?cmd=catNavigate2&catID=603624
- Ouvrez Scanner Peak et «Commencer un nouveau projet» suivi par «Ajouter des fichiers». Chargez le sélectionné. FSA fichiers de données dans le programme, y compris les contrôles positifs et négatifs.
- Sélectionnez (mettez en surbrillance) et "analyser" tous les fichiers chargés. Soyez sûr de chaque échantillon est réglé sur Standard Taille ": GS500 (-250) et Méthode d'analyse:. Dimensionnement Default-PP de presse« Analyze ».
- Examiner la taille en paires de base de chaque pic de couleur produite par les fragments d'ADN dans les échantillons.
- Notez chaque valeur de la taille maximale de paires de base et la comparer à la pointe de contrôle positif.
- Les pics dans les échantillons de contrôle positif devrait se comparent favorablement avec les numéros de paires de bases et les numéros correspondants de répétitions en tandem figurant dans les tableaux 4-7.
- Déterminer le nombre de courts segments de répétition en tandem sont présents dans chaque allèle de l'échantillon par comparaison avec le contrôle positif. Nous utilisons NHDP63 qui a été séquencé pour le nombre de copies à chaque locus VNTR être examinés. 4
- Entrez les données enregistrées dans un tableur pour des analyses comparatives et / ou mathématiques. VNTR «empreintes» ou haplotypes, sont des chaînes d'allèles à des loci définies qui sont caractéristiques d'un M. leprae souche.
7. Les résultats représentatifs
L'électrophorèse sur gel des produits de PCR, nous l'espérons produire une bande pour chaque locus dans la combinaison d'amorces (figure 3). Dans la figure 3, il ya deux sections pour le gel: la section du haut est une combinaison des échantillons PCR et la partie inférieure Combinaison 2 échantillons. Chaque section contient une échelle 20 paires de bases moléculaires, suivi par les produits de PCR obtenus à partir de 8 échantillons de patients. Une combinaison a aussi un contrôle négatif et enfin un contrôle positif (NHDP63 souche). (Les commandes de combinaison 2 étaient sur un gel différent.) Notez que la plupart des échantillons afficher clairement 5 bandes, une pour chaque locus dans la combinaison. Dans certains cas, les bandes peuvent être trop proches les unes à apparaître comme des lieux séparés entraînant l'apparition de seulement 4 bandes.
Tailles amplicon attendus sont énumérés dans le tableau 1. Notre laboratoire utilise NHDP63 souche de M. leprae comme un contrôle positif. Deux types de segments de répéter ont été étudiés: loci microsatellites (avec 1-5 répétitions de base) et loci minisatellites (avec des segments de plus de 5 paires de bases répétées plusieurs fois). 5
Interprétation des fichiers de données de l'électrophorèse capillaire repose sur deux normes: une interne fragment d'ADN dimensionnement standard appelée GeneScan-500LIZ (ABI) et un échantillon de contrôle externes positifs de l'ADN bactérien amplifié.
Les chromatogrammes FLA, visualisées à l'aide du logiciel du scanner Peak, peut être vu dans les figures 5a, 5b et 6.
Scanner Peak fournit des données sur la taille amplicon (axe x en paires de bases) et la force du signal (axe des y). (Figure 5b) des données supplémentaires sur l'aire du pic, etc sont également disponibles, bien que la taille et la hauteur du pic des valeurs sont les plus importants. Pics à moins de 100 unités de hauteur sont généralement considérés comme trop faible d'un signal pour être fiables.
La figure 6 compare le contrôle positif (NHDP63) et deux échantillons de patients, montrant une variation dans le nombre de répétitions en tandem au locus (RGT) 9. Dans le contrôle positif, la suite (GTA) est répété 10 fois. Le VNTR NHDP63 et taille de l'amplicon ont été vérifiées par séquençage des gènes. 4 Patient amplicon de PCR est de 3 bp plus petit que le contrôle positif indiquant il ya seulement 9 unités de répétition, alors que le patient 6 a un amplicon qui est de 3 bp plus grand que NHDP63 révélant 11 répétitions de (RGT). Patient 2 avaient un faible indice bactérien (IB) avec peu ou pas de PCR de l'ADN de réplication, donc pas de signal de FLA.
Difficulté à FLA interprétation des données se produit parfois à la suite de «bégaiement». Lors de la réaction PCR, l'ADN polymérase peut produire des fragments qui sont une ou plusieurs répète plus ou moins courtes de l'allèle source. Ce sont généralement reconnus comme des pics de hauteur inférieure entourant le pic principal. Ils seront "sur l'échelle" qui est, le nombre exact de paires de bases du segment répété sur grand ou plus petit que le pic principal. Le résultat est une famille de pics de la même couleur. La figure 7 montre cela avec le locus (TA) 10.
Une autre difficulté, parfois rencontrés avec FLA implique '+ A "ou" A-tailing », dans lequel l'ADN polymérase ajoute une seule base [habituellement adénine (A)] à l'extrémité 3' du segment d'ADN copié. Cela montre en FLA comme un pic vers la droite d'un pic principal ou bégaiement qui est une paire de base plus grand que le pic adjacent comme le montre la Figure 8. Il ne doit pas être confondu avec un pic principal ou bégaiement. Un pic principal et son A-queue sont considérées comme une seule espèce. La queue A-ne modifie pas le nombre de répétitions VNTR. (Certains kits ADN polymérase sont conçus spécifiquement pour promouvoir une-tailing pour réduire cet effet de confusion. Promouvoir complète A-tailing tend à produire un seul pic, plutôt que d'une paire de pointes.)
L'extraction d'ADN et des produits de PCR contiennent à la fois M. leprae et l'ADN humain, cependant, à l'exception de (TA) 18, les amorces sont assez spécifiques qu'ils ne amplifier l'ADN bactérien, et il ya peu ou pas d'amplification de l'ADN humain. (TA) 18 produit souvent un pic à 242 paires de base qui est de l'ADN humain et n'est pas vu dans les échantillons de l'ADN tatou passages (figure 9).
La durée de conservation des amorces est généralement bonne à condition qu'ils soient conservés à -20 ° C, seulement enlevé pour une utilisation immédiate, puis conservés à 4 ° C jusqu'à la consommation. Primaires devrait être stable à 4 ° C pendant 1-2 semaines. Même si des combinaisons d'amorces pour la PCR est un peu de temps, il est recommandé que seuls combinaison d'amorces pour utilisation immédiate être préparé. Combinaisons d'amorces semblent se dégrader quelque peu lorsqu'il est stocké pendant des périodes prolongées.
TE devraient être aliquotés de stocks plus importants, et des aliquotes peuvent être stockées soit congelés ou à température ambiante. Agrandir aliquotes de 200-400μl sont bons pour diluer les stocks de primer séchés ou concentrés (figure 2a). Les petites aliquotes de 10-50 ul sont utiles pour compléter les volumes de combinaisons d'amorces 3 et 4. TE est peu coûteux et aliquotes doivent être jetés après usage.
Kits enzymatiques Multiplex sont assez chers et doivent être conservés congelés (-20 ° C) jusqu'à utilisation. Après la préparation de PCR, toutes les solutions multiplex inutilisés devraient être retournés immédiatement à 4 ° C. La petite Qiagen PCR multiplex kit est livré avec 3 tubes de mélange de multiplex, chacun contenant 0.85ml (850μl) de solution; suffisant pour environ 65-70 PCR.
Généralement, il est préférable d'éviter la répétition, les changements de température importants pour les matériaux utilisés dans ce type de travaux de laboratoire, y compris les échantillons d'ADN, des amorces et des solutions kit multiplex. Tous les matériaux doivent être conservés à -20 ° C jusqu'à ce que nécessaire, puis conservé à 4 ° C jusqu'à la consommation. Stockage à long terme de l'ADN devrait être à -80 ° C.
Tous les matériaux contenant passé tissus, des amorces et / ou de l'ADN doivent être autoclavés et éliminés lorsqu'ils ne sont plus d'aucune utilité. Tous les échantillons traitésau bromure d'éthidium ou le formamide devrait être traité comme un déchet dangereux et éliminés conformément aux dangereux de l'institution politique de matériaux.
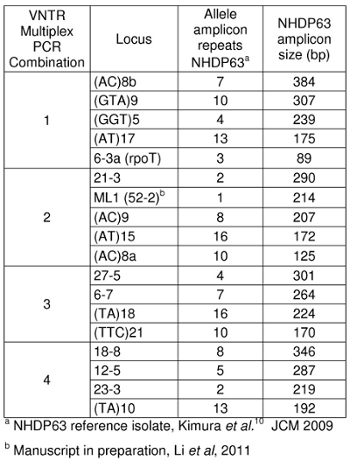
Tableau 1: Tailles Amplicon pour la souche NHDP63
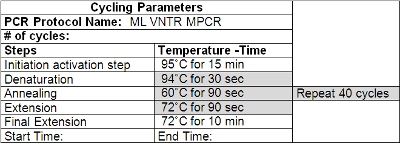
Tableau 2: Paramètres de cyclisme VNTR PCR

Tableau 3: Préparation de la PCR
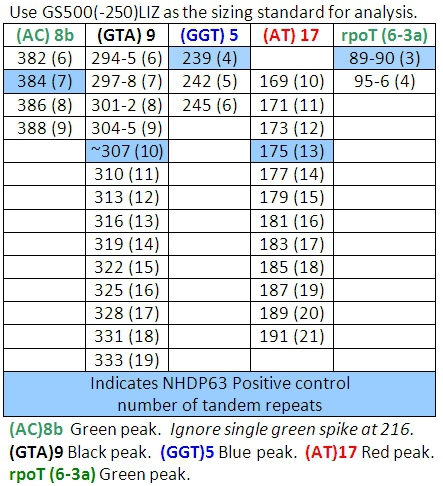
Tableau 4: Appels allèle de la combinaison 1
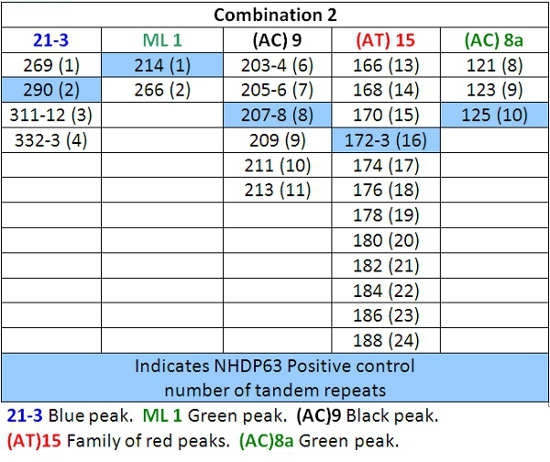
Tableau 5: Appels allèle Combinaison 2
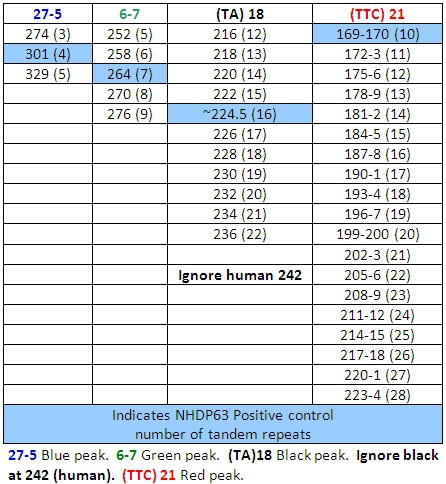
Tableau 6: Appels allèle Combinaison 3
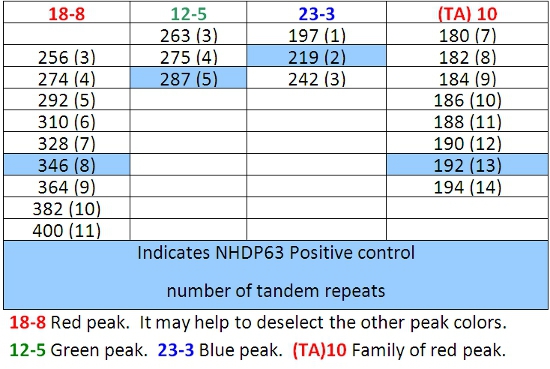
Tableau 7: Appels d'allèles pour la combinaison 4

Figure 1: Diagramme VNTR-FLA Process Flow
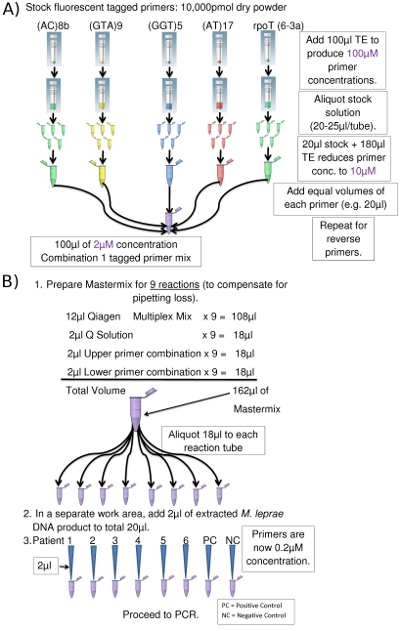
Figure 2. (A) Préparation des amorces Combinaison Upper 1 (courtoisie Eppendorf tube cathodique d' www.clker.com ). (B) l'installation de PCR (pour 8 PCR) (courtoisie Eppendorf tube cathodique d'www.clker.com)
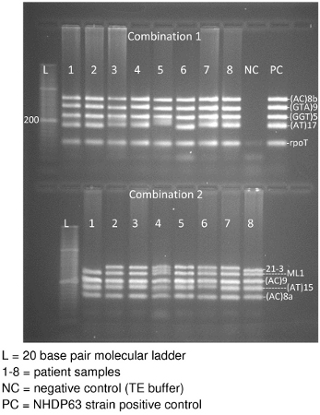
Figure 3. Gel d'agarose 1 et 2 Combinaisons ADN VNTR produit PCR
Figure 4. (A). FLA planche cartographique (b) FLA fichiers de données: *. FSA
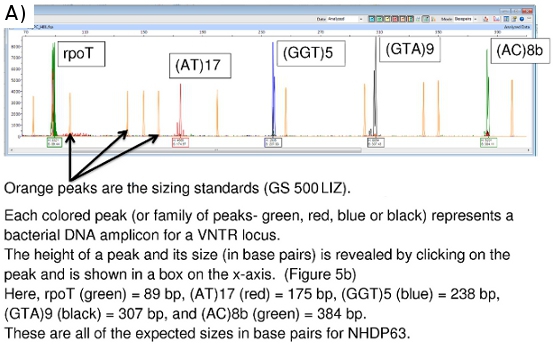
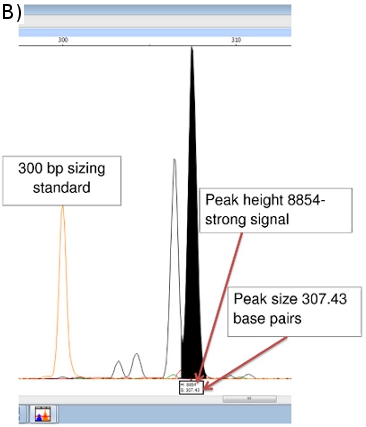
Figure 5. (A) Chromatogramme FLA du contrôle positif (NHDP63) pour les loci VNTR Combinaison 1. (B) les données du scanner de pointe pour la taille et l'abondance des amplicons (RGT) 9
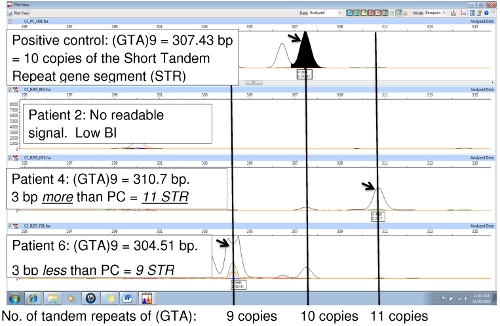
Figure 6. Comparaison des échantillons PCR pour le PC (NHDP63) pour le locus (RGT) 9
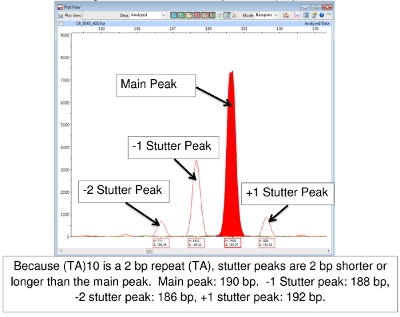
Figure 7. Main et Peaks Stutter pour (TA) 10
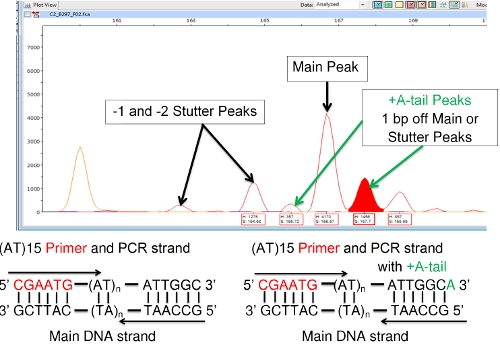
Figure 8: A + (A-queue) pics adjacents aux principaux sommets ou de bégaiement.
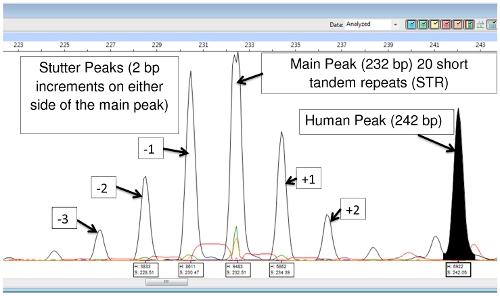
Figure 9: (TA) 18 pic principal, Stutter Peaks et Peak ADN humain
Figure 10:. Différenciation des souches (A) de M. leprae à partir des données VNTR différenciation des souches (B) de M. leprae basés sur l'ADN MLVA empreintes digitales
Discussion
La collecte d'échantillons de peau de patients lépreux nécessite cliniciens qualifiés ou techniciens travaillant dans des cliniques de peau. Les travailleurs de laboratoire manipulant ces échantillons doit prendre grand soin de porter des blouses de laboratoire, des gants et des lunettes de protection et de travailler dans un cabinet de bio-sécurité lors de la manipulation des échantillons de tissus humains infectés ou tatou. Désinfection des surfaces et des outils est également critique. Travailler dans un endroit propre, stérile de bio-sécurité armoire est important pour éviter la contamination des échantillons d'ADN.
Extraction de l'ADN est devenu relativement facile grâce au développement de kits d'extraction d'entreprises telles que Qiagen. Des directives doivent être suivies attentivement. Tous les échantillons doivent être conservés au froid lorsqu'il n'est pas utilisé. Évitez répétées, des changements de température extrêmes pour les échantillons.
Amorces d'ADN utilisées dans ce travail peut être commandé auprès de diverses entreprises qui ont été cités dans la liste des références bibliographiques à la fin du présent document. Il faut prendre soin de travailler avec des amorces dans un environnement de l'ADN libre, afin d'éviter toute contamination. Primaires viennent sous forme de poudre sèche et doit être mélangé avec TE et dilué à une concentration 100 microns, puis séparés en petites quantités (figure 2a). Les solutions de travail des amorces sont encore dilués à des concentrations 10 uM. Encore une fois, ne pas utiliser des échantillons d'ADN dans les zones / hottes où l'on utilise des amorces et préparés.
Les produits de PCR doivent également être conservés au froid lorsqu'il n'est pas utilisé.
Un gel d'agarose 3% la préparation donnera une meilleure séparation bande d'ADN, mais prend plus de temps à courir. Pour des raisons purement qualitative, 2% est généralement suffisant. Solution de bromure d'éthidium utilisé pour la coloration de l'ADN dans les gels d'agarose est une génotoxine très actif qui est absorbé par la peau. Manipuler cette solution et les gels colorés avec de l'utiliser le plus grand soin et toujours en étant sûr de porter des gants. Laver soigneusement les mains après avoir manipulé des échantillons d'ADN ou de matériaux de bromure d'éthidium. Eliminer la solution une coloration au bromure d'éthidium, les laver et les gels selon les directives institutionnelles. Les gels sont pas systématiquement requis; cette étape peut être éliminé une fois les méthodes FLA ont été établis. Il est temps et consommer réactif.
La solution de formamide utilisées dans la préparation des échantillons pour FLA est également très toxique et doit être manipulé avec soin. Se laver les mains après son utilisation. Débarrassez-vous de la suite des directives institutionnelles.
Lecture des résultats du FLA en utilisant le logiciel du scanner de pointe peuvent être difficiles. Un ensemble de base des appels allèle (nombre de répétitions en tandem) a été développée au CSU (tableaux 4-7). (Il convient de noter que tableaux 4-7 peut-être pas tout compris. Comme d'autres souches de M. leprae sont étudiés, avec des allèles nombre de copies en dehors des plages inscrites peuvent être trouvés.) Un défi particulier consiste à "bégayer" pics. Ce sont des familles de pics, en particulier des déclarations de soupçon qui n'impliquent que deux à trois répétitions de paires de bases, telles que (TA) 10. Parfois, la sélection des pics corriger à lire est difficile (figure 7). Dans cette figure, le pic dans le contrôle positif à 190 pb est le pic principal. Inférieure Peaks en hauteur qui sont 2, 4 ou même 6 paires de base plus grande ou plus petite que le pic principal sont appelés «pics bégaiement." A-tailing peut aussi causer de la confusion. A-tailing est décrit plus haut dans le texte et la figure 8. Enfin, si les produits de PCR sont très abondants dans les échantillons, les pics peuvent apparaître avec un pic double. Dans ce cas, lisez le centre de la forme de pointe. (Voir Figure 6, Patient 6.)
La gestion des données peut être une tâche redoutable pour ce type de travail. Il est important d'enregistrer toutes les informations pertinentes pour toutes les expériences telles que: la date d'une tâche ou une procédure, l'opérateur, bandes / plaques cartes, des ordres FLA, des conditions de PCR et les recettes, les dates des gels et des photographies, des températures de stockage, des dilutions matrice d'ADN, FLA électroniques lieux de stockage de fichiers, organisation, etc Bonne gestion des données et peut sauver des heures de temps passé à chercher des éléments d'information dans le futur.
Le travail de laboratoire décrit ici a été en cours depuis plusieurs années à la Colorado State University et à d'autres endroits dans le monde. La grande photo de ce que l'ensemble des données recueillies signifie et comment elle peut être d'un usage futur commence à émerger. Figures 10A et 10B montrent comment ces empreintes d'ADN peuvent être utilisés pour distinguer les différentes M. leprae tensions entre les pays (10A), ou même entre les familles (10B). Cas de la famille et la communauté liée a été démontré pour mener M. leprae d'similaires ou identiques types de souches VNTR. L'espoir est que cela peut être possible d'avoir un aperçu plus loin dans le mode (s) de transmission de la lèpre afin qu'un système de détection précoce des réseaux de transmission peuvent être développés pour ces mos de personnest à risque, et que la pharmacothérapie curative peut commencer avant des dommages neurologiques permanents et dermatologiques est fait.
Déclarations de divulgation
Remerciements
Le financement a été fourni par le NIH / NIAID octroi RO1-AI-63457 et de l'ARRA subventions complément RO1-AI-63457 S1. Nous reconnaissons les contributions de tous les membres actuels et passés du groupe de laboratoires et des collaborateurs.
matériels
| Name | Company | Catalog Number | Comments |
| Nom du réactif | Société | Numéro de catalogue | Commentaires |
|---|---|---|---|
| Sang DNeasy et Kit de tissus | Qiagen | 69504 | |
| Multiplex PCR Kit | Qiagen | 206143 | |
| DNA Primers | Divers | ||
| Gel d'agarose | Divers | ||
| 5x ou 6x tampon de chargement de gel | New England Biolabs | B7021S | Recettes disponibles pour rendre votre propre * |
| Solution de bromure d'éthidium | Divers | Diluer du con. | |
| Salut-Di Formamide Solution | Applied Biosystems | 4311320 | |
| Gene scan -500 LIZ | Applied Biosystems | 4322682 |
* Http://biowww.net/buffer-reagent/6X-Gel-loading-buffer-bromophenol-blue-sucrose.html
| Équipement | Commentaires |
|---|---|
| 2 Réfrigérateurs | 4 ° C: 1 pour les échantillons d'ADN, une pour les amorces et les réactifs Multiplex |
| 1 four micro-ondes | Ou de façon appropriée pour le chauffage et l'eau d'agarose pour faire des gels |
| 1 autoclave | Autocuiseur ou pour stériliser des matériaux |
| Machine à PCR | Thermocycleur |
| 2 jeux de pipettes | 0,5 à 10 ul, 10-100 ul, 20-200 ul, 1000 ul 1 jeu pour les apprêts, 1 jeu de l'ADN |
| Submarine boîte électrophorèse sur gel | Avec des formes et des peignes pour les gels préparation. |
| Alimentation d'électrophorèse | Avec des câbles pour la connexion à la boîte de gel |
| Bloc de chaleur | Pour tubes Eppendorf |
| Centrifugeuse | Pour les tubes Eppendorf / bandes |
| Système d'électrophorèse capillaire (analyseur génétique) | Ou l'accès à une institution qui peut effectuer cette analyse |
| Plastiques | Jetable d'aérosol de pipette, tubes Eppendorf, des bandes de 8 puits, des plaques de 96 puits, dont certains de qualité optique |
| Un ordinateur avec connexion Internet |
Références
- World Health Organization. Chemotherapy of leprosy for control programmes. Technical Report Series. 675, 18-22 (1982).
- Cole, S. T. Massive gene decay in the leprosy bacillus. Nature. 409, 1007-1011 (2001).
- . Leproma Web Server. Leproma Web Server [Internet]. , (2004).
- Groathouse, N. A. Multiple Polymorphic Loci for Molecular Typing of Strains of Mycobacterium leprae. J. of Clinical Microbiology. 42, 1666-1672 (2004).
- Young, S. K. Use of Short Tandem Repeat Sequences to Study Mycobacterium leprae in Leprosy Patients in Malawi and India. Public Library of Science: Neglected Tropical Diseases. 2, E214-E214 (2008).
- Monot, M. Are Variable-Number Tandem Repeats Appropriate for Genotyping Mycobacterium leprae. J. of Clinical Microbiology. 46, 2291-2297 (2008).
- Kimura, M. Rapid Variable Number Tandem-Repeat Genotyping for Mycobacterium leprae Clinical Specimens. J. of Clinical Microbiology. 47, 1757-1766 (2009).
- Weng, X. i. a. o. m. a. n. Identification and Distribution of Mycobacterium leprae Genotypes in a Region of High Leprosy Prevalence in China: a 3-Year Molecular Epidemiological Study. J. of Clinical Microbiology. 45, 1728-1734 (2007).
- Weng, X. Transmission of leprosy in Qiubei County, Yunnan, China: Insights from an eight year molecular epidemiology investigation. Infection, Genetics and Evolution. , (2010).
- Sakamuri, R. M. Population-Based Molecular Epidemiology of Leprosy in Cebu, Philippines. J. of Clinical Microbiology. 47, 2844-2854 (2009).
- Fontes, A. N. B. Genetic diversity of Mycobacterium leprae isolates from Brazilian leprosy patients. Leprosy Review. 80, 302-315 (2009).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon