Method Article
DNA fingerprinting di Mycobacterium leprae Ceppi Utilizzando variabile Tandem Repeat Number (VNTR) - Lunghezza Analisi Fragment (FLA)
In questo articolo
Riepilogo
La lebbra, causata da Mycobacterium leprae, È ancora endemica in molti luoghi. Al fine di conoscere la diffusione e la modalità di trasmissione della lebbra, è importante per determinare quale ceppo di M. leprae Ha infettato un paziente. Un numero variabile di ripetizioni tandem (VNTR) digitazione è uno di questi metodi.
Abstract
Lo studio della trasmissione della lebbra è particolarmente difficile dato che l'agente eziologico, Mycobacterium leprae, non può essere coltivato in laboratorio. Le uniche fonti dei batteri sono malati di lebbra, e armadilli sperimentalmente infetti e topi nudi. Così, molti dei metodi utilizzati in epidemiologia moderna non sono disponibili per lo studio della lebbra. Nonostante un ampio programma globale di trattamento farmacologico per la lebbra attuato dalla WHO 1, la lebbra rimane endemica in molti paesi con circa 250.000 nuovi casi ogni anno. 2 L'intera M. leprae genoma è stata mappata 3,4 e molti loci sono stati identificati che si sono ripetuti segmenti di 2 o più paia di basi (chiamate micro-e minisatelliti). 5 ceppi clinici di M. leprae possono variare nel numero di segmenti ripetuti in tandem (ripetizioni in tandem corto, STR) a molti di questi loci. 5,6,7 numero variabile di ripetizione in tandem (VNTR) 5 analisi è stato utilizzato per distinguere i diversi ceppi di bacilli della lebbra. Alcuni dei loci sembrano essere più stabili di altri, mostrando meno variazioni di numeri si ripetono, mentre altri sembrano cambiare più rapidamente, a volte nello stesso paziente. Mentre la variabilità di alcuni VNTRs ha portato fino domande riguardanti la loro idoneità per la tipizzazione ceppo 7,8,9, i dati emersi suggeriscono che loci multipli, che sono diversi nella loro stabilità, può essere utilizzato come un valido strumento epidemiologico. Multiple locus VNTR analisi (MLVA) 10 è stato usato per studiare l'evoluzione lebbra e la trasmissione in diversi paesi tra cui Cina 11,12, Malawi 8, le Filippine 10,13, e in Brasile 14. MLVA prevede più passaggi. In primo luogo, DNA batterico viene estratto insieme al DNA tessuto ospite da biopsie clinici o macchie della pelle fessura (SSS). 10 I loci desiderati sono poi amplificati dal DNA estratto tramite reazione a catena della polimerasi (PCR). Primers marcati con fluorescenza per 4-5 loci diversi sono usati per reazione, con 18 loci essere amplificato in un totale di quattro reazioni. 10 I prodotti di PCR può essere sottoposto a elettroforesi su gel di agarosio per verificare la presenza di segmenti di DNA desiderato, quindi sottoposti ad analisi a fluorescenza della lunghezza dei frammenti (FLA) usando elettroforesi capillare. DNA da batteri armadillo diversi passaggi con un numero definito di copie ripetere per ogni locus viene utilizzato come controllo positivo. I cromatogrammi FLA vengono poi esaminati usando il software dello scanner e di picco della lunghezza dei frammenti viene convertito in numero di copie VNTR (allele). Infine, gli aplotipi VNTR sono analizzati per i modelli, e se combinata con i dati clinici del paziente possono essere utilizzati per monitorare la distribuzione dei tipi di deformazione.
Protocollo
Lo scopo di questo articolo video è di fornire una panoramica del flusso di lavoro con il formato dei dati e l'interpretazione di ricercatori che possono essere solo a partire da questo tipo di lavoro (Figura 1). Contiene la dimostrazione di tecniche, protocolli semplificati e consigli pratici descritti in opere precedentemente pubblicate. 5,10
Flusso di lavoro generale e laboratori:
Ci dovrebbero essere almeno 3 aree di lavoro separate per questo tipo di ricerca. Il laboratorio dovrebbe avere 1) una pre-PCR con un cappuccio PCR (casella aria pulita o area di lavoro isolato) per la preparazione del fondo (diluizione, preparazione aliquota e la miscelazione), 2) un separato bio-sicurezza armadio per la gestione e l'aggiunta di DNA alle miscele PCR, e 3) un post-PCR area di lavoro per la preparazione e il caricamento e gel per la preparazione di campioni per FLA. Primer e campioni di DNA devono essere conservati in congelatori e frigoriferi separati. Contaminazione fondo è uno dei problemi principali e più persistenti nel lavoro di laboratorio di questo tipo. Pipette di primer e PCR mix NON deve essere utilizzato per il DNA. Non ci dovrebbero essere gruppi separati di pipette per la pre-PCR, PCR e post-PCR aree di lavoro. In generale, un ricercatore in grado di elaborare 12-18 i campioni in un periodo di 12-24 ore con attrezzature standard di laboratorio.
Prima di iniziare ogni fase del lavoro:
- Indossare un camice da laboratorio e guanti. I guanti devono essere indossati in qualsiasi momento campioni biologici, primer, DNA e bromuro di etidio vengono gestiti. Cambiare i guanti frequentemente.
- Lavoro in una cappa di PCR armadio, la biosicurezza armadio o area di lavoro pulita.
- Utilizzare una carta nuova panchina o pad.
- Impostare contenitori dei rifiuti per liquidi e puntali.
- Pulire con l'interno della cappa PCR e pipette con il 70% di etanolo.
- Pipette soggetto e l'area di lavoro alla luce UV per 15 minuti prima impostazione della PCR. (Luce ultravioletta legami crociati DNA contaminanti di superficie.)
- Usare sempre aerosol suggerimenti prevenzione pipetta per materiali contenenti DNA, primer e reagenti PCR.
- Centrifuga qualsiasi tubi / strisce / piastre contenenti liquido prima di aprire per prevenire fuoriuscita di aerosol e contaminazione incrociata da manipolazione.
1. M. leprae DNA preparazione
Campioni clinici contenenti M. leprae sono ottenuti da malati di lebbra che visitano cliniche della pelle. Campioni diagnostici di routine possono essere biopsie cutanee, macchie della pelle fessura o tamponi nasali. In generale, biopsie o macchie della pelle fessure sono i migliori per l'epidemiologia molecolare, perché sono puliti e contengono quantità sufficienti di M. leprae. L'uso di questi materiali per la ricerca deve essere approvato secondo le linee guida istituzionali.
- Preservare la biopsia o fessura campioni striscio pelle in un tappo a vite flacone con 1 ml di etanolo al 70%.
- Centrifugare ogni campione in una velocità variabile da banco centrifugare a 12000 xg per 15 minuti.
- Rimuovere il surnatante e metterlo in una provetta. Se necessario, può essere utile a ri-centrifuga questi campioni e recupero del tessuto supplementare o del DNA in seguito.
- Aggiungere 500 ml di tampone fosfato (PBS) per i campioni di tessuto e immergerlo per 1 ora per lo scambio di conservanti residui di etanolo e reidratare il campione.
- Centrifugare i campioni in una centrifuga da banco a 12.000 xg per 20 minuti. Gettare la soluzione PBS in un contenitore di rifiuti parzialmente riempito con soluzione disinfettante.
- Estrarre il DNA batterico con il sangue e il kit Qiagen DNEasy tessuto seguendo le linee guida prescritte.
- DNA estratto deve essere aliquotati in 4 fiale. Conservare 2 aliquote a -80 ° C per utilizzo futuro se / quando la riproducibilità dei risultati dei test è necessario o quando si rendono disponibili nuove tecnologie. Memorizzare una aliquota a -20 ° C e uno a 4 ° C per un uso più immediato.
- E 'prudente preparare un' vuoto di estrazione 'insieme ai campioni di tessuto. Il vuoto è sottoposto a tutti gli stessi trattamenti di cui sopra, ma viene eseguita senza tessuto. PCR il vuoto di estrazione con i campioni dei pazienti per garantire che la tecnica di estrazione dell'operatore sia corretto e che i reagenti sono esenti da contaminazione del DNA.
2. Primer di preparazione
- Primer per i loci per i quali ci sono i più numerosi dati di tipo ceppo sono elencati nella Tabella 1. Quattro o 5 primer sono combinati per multiplex PCR. Un fondo per ogni locus porta etichetta fluorescente chimica a 5 ', che sarà automaticamente rilevato durante l'analisi capillare della lunghezza dei frammenti elettroforesi (FLA).
- Per ogni combinazione multiplex PCR, il primer marcati e corrispondente fondo inversa per ogni locus sono combinati in separati provette Eppendorf: primer in avanti in una provetta, invertire in un altro. Primer magazzino sono preparati come 100μM soluzioni (Figura 2a), una parte dei qualiè diluito 10x per una concentrazione di 10 mM con TE (1x Tris-EDTA, pH 8,0). Aliquote delle soluzioni rimanenti 100 mM fondo sono conservati a -20 ° C per un uso successivo.
- Uguali quantità di ciascun primer 10 micron vengono miscelati con conseguente 2μM concentrazioni finali di ciascun primer. Quando le combinazioni fondo vengono aggiunti alla miscela di PCR (Tabella 3), la concentrazione finale di ciascun primer scende a 0,2 micron.
Quando il primer combinato vengono aggiunti alla miscela PCR (Tabella 3), concentrazioni finali goccia a 0.2μM ciascuno.
3. Amplificazione del DNA batterico mediante PCR multiplex
- PCR Setup
- Registrare il numero di campioni che verranno utilizzati e preparare un foglio di lavoro con i reagenti necessari ed i loro volumi prima di raccogliere la plastica e altri materiali necessari.
- Etichettare le provette sterili / strisce / piastre da utilizzare per la PCR con i numeri del campione. Ricordati di includere controlli positivi e negativi.
- Pulire il termociclatore con una soluzione detergente (come Decon ELIMINase) e programmarlo in base alla tabella 2. Cambiare i guanti dopo la pulizia e prima di maneggiare fondi e altri reagenti.
- Preparare PCR Mastermix secondo la Tabella 3 ed etichettare le provette PCR / strisce / piastra con la combinazione di primer e numeri campione da utilizzare.
- 18μl aliquota Mastermix ad ogni etichetta provetta PCR o bene. Cambiare i guanti.
- Pulire un separato biosicurezza cabinet e pipette con il 70% di etanolo per pulire e disinfettare l'area di lavoro e strumenti, poi soggetta l'area di lavoro e pipette ai raggi UV per 15 minuti a cross-link eventuali contaminanti DNA. Cambiare i guanti.
- Nel pulito bio-safe armadio, aggiungere 2μl del modello del DNA per il Mastermix in ogni tubo di PCR / pozzetti di acquisire un volume totale di 20μl (Figura 2b). Usa aerosol puntali prevenzione per tutti i materiali liquidi.
- Centrifugare le provette PCR / strisce / piastre brevemente per miscelare il contenuto.
- Posizionare i tubi di campione / strisce / piastra nel termociclatore e avviare il programma PCR.
- Quando il programma è completo, rimuovere i prodotti dal termociclatore e conservare a 4 ° C fino elettroforesi.
4. Elettroforesi su gel dei prodotti di PCR
* Questo protocollo è stato standard per molti anni ed è un passo opzionale che può essere impiegato se la conferma di prodotti di PCR è voluto prima di inviarli per FLA.
- In un apposito post-PCR area di lavoro, preparare un gel di agarosio al 2%. Usa 2.0g di agarosio powder/100ml 1x TBE (Tris / Borato / EDTA) soluzione tampone in un pallone. Scaldare la miscela per circa 1,5-2 minuti in un forno a microonde. Agitare il contenuto, il riscaldamento di nuovo, se necessario, per sciogliere l'agarosio. Raffreddare leggermente e versare il gel in un modulo utilizzando un pettine con i pozzi a sufficienza per i campioni.
- Rimuovere i prodotti di PCR da 4 ° C e centrifugare per circa 30 secondi.
- Mix 2 5μl del prodotto di PCR con 0.5-1ml di tampone gel 5x o 6x caricamento (buffer di caricamento è a disposizione di diverse aziende, o possono essere mescolati in laboratorio. Ricette sono disponibili online.)
- Caricare i pozzetti del gel con il campione 6μl / caricamento mix buffer. Aggiungi una scala molecolare ad un pozzetto (preferibilmente a 20 bp scala).
- Attivare il gel a 100V per circa 90 minuti.
- Immergere il gel nella soluzione di bromuro di etidio per 15-30 minuti, poi in ultrapura acqua distillata per un pari ammontare di tempo.
- Immagine del gel mentre la luce UV viene applicato. Ci dovrebbe essere una band per ciascuno dei segmenti di DNA 4-5 nella combinazione (Figura 3).
- Smaltire il gel secondo la politica di pericolosi dell'istituzione materiali. Soluzione di bromuro di etidio può essere utilizzato più volte prima di corretto smaltimento.
5. La preparazione di campioni per l'analisi frammento lunghezza (FLA)
- In un tubo Eppendorf pulito, preparare una miscela master contenente 12μl di Hi-Di soluzione formammide da una nuova aliquota e 0.3μl di GeneScan -500 LIZ dimensionamento standard (sia da Applied Biosystems) per ogni campione da analizzare. (Hi-Di formammide denatura chimicamente i filamenti di DNA prima di elettroforesi capillare, eliminando la necessità di riscaldamento.) Utilizzando un 96-ottico ben piastra di reazione di qualità, 12.3μl aliquota del formammide-LIZ miscela in ciascun pozzetto utilizzato per i campioni e impostare il piatto da parte.
- Fare una mappa piastra per i record che indica quale campione di DNA è in ogni pozzetto (Figura 4a).
- Utilizzando tubi / strisce o una piastra a 96 pozzetti, aggiungere 1ml di prodotto di PCR (da parte 3) a 59μl di acqua di qualità PCR creando una diluizione 1:60 del prodotto della PCR. Diluizioni possono essere regolati sulla base di potenza del segnale a partire dai dati FLA o la luminosità di bande gel. Concentrazioni ancora più basse di DNA puòessere sufficiente (1:120 o 1:180).
- Aggiungere 1ml di prodotto diluito PCR per la corretta bene nel piatto contenente la formammide-LIZ miscela.
- Velocità e l'efficienza può essere notevolmente migliorata se la PCR è stato fatto anche in una piastra da 96 pozzetti. Si può semplicemente allineare 3 piastre quali in sequenza: Piatto 1 con prodotti di PCR, Tavola 2 con acqua sterile per la diluizione dei prodotti di PCR, e 3 con piastra formammide e scaletta di dimensionamento per l'analisi dei frammenti di lunghezza. Una pipetta multicanale permette un rapido processo di preparazione FLA.
Analisi del campione tramite Genetic Analyzer
- Calibrare l'analizzatore genetico per rilevare la flurophores applicata per le istruzioni del produttore. Assicurarsi di utilizzare un colorante-set che include LIZ.
- Rifornire l'acqua di risciacquo contenitori e aggiungere nuovi buffer Correndo con EDTA (1x) (Applied Biosystems) al buffer camere.
- Aggiungi un pre-fessura setto silicio al piatto e posizionare la piastra nel cassetto tenendo progettato. Premere il pulsante 'vassoio' sul Genetic Analyzer per portare avanti il campionatore automatico. Vassoio posto sul autocampionatore e chiudere la porta alla Genetic Analyzer.
- Creare o importare un foglio di calcolo per l'analisi. File importabili hanno estensione. Plt e sono in formato delimitato da tabulazioni. I file possono essere modificati in Excel (Microsoft) o programmi simili.
- I campioni sono iniettati nel capillare (50 cm di lunghezza, POP-7 polimero) applicando una tensione di iniezione di 1,6 kV per 15 s. L'elettroforesi capillare funziona a una tensione di 15 kV a 60 ° C per 1800 secondi. L'intero processo dura circa 45 minuti.
- Una volta che una corsa è completa, i dati per ogni campione analizzato vengono convertiti in file con estensione. Fsa e collocati in una cartella di file piatto (Figura 4b). Ogni file di dati è di circa 100 KB e può essere memorizzato su un flash drive o zip e via e-mail. I file di dati possono essere visualizzati utilizzando il software adatto, come GeneMapper ABI o Scanner Peak.
6. Analisi dei risultati della lunghezza dei frammenti
- L'analisi dei dati di fluorescenza elettroforesi capillare richiede un software speciale. Se tale software non è disponibile, il sito web Applied Biosystems e scaricare Scanner Peak. Il software è gratuito e funziona abbastanza bene. https://products.appliedbiosystems.com/ab/en/US/adirect/ab?cmd=catNavigate2&catID=603624
- Aprire Scanner Peak e 'Start New Project' seguito da 'Aggiungi file'. Caricare il selezionata. Fsa file di dati nel programma, inclusi i controlli positivi e negativi.
- Selezionare (evidenziare) e "analizzare" tutti i file caricati. Assicurarsi che ogni campione è impostato su "Formato standard: GS500 (-250) e Metodo di analisi:. Dimensionamento Default-pp Premere 'analizzare'.
- Esaminare le dimensioni in paia di basi di ogni picco colorate prodotte dai frammenti di DNA nei campioni.
- Registrare ogni valore di picco dimensioni in paia di basi e di confrontarlo con il picco di controllo positivo.
- Picchi nei campioni di controllo positivo deve reggono bene il confronto con il numero di coppie di basi e corrispondente numero di ripetizioni in tandem elencati nelle tabelle 4-7.
- Determinare il numero di brevi segmenti di ripetizione in tandem sono presenti in ogni allele campione rispetto al controllo positivo. Noi usiamo NHDP63 che è stato sequenziato per il numero di copie VNTR ad ogni locus in esame 4.
- Inserisci i dati registrati in un foglio di calcolo per analisi comparative e / o matematica. VNTR 'impronte digitali' o aplotipi, sono stringhe di alleli a loci definiti che sono caratteristici di un M. leprae ceppo.
7. Rappresentante Risultati
Elettroforesi su gel dei prodotti di PCR, si spera, producono una banda per ogni locus nella combinazione primer (Figura 3). Nella Figura 3, ci sono 2 sezioni per il gel: la sezione superiore ha Combinazione 1 campioni PCR e la porzione inferiore Combinazione 2 campioni. Ogni sezione contiene una coppia di 20 scala molecolare di base, seguiti da prodotti di PCR ottenuti da 8 campioni dei pazienti. 1 combinazione ha anche un controllo negativo e, infine, un controllo positivo (NHDP63 ceppo). (I controlli per la combinazione 2 erano su un gel differenti.) Si noti che la maggior parte dei campioni in modo visibile 5 bande, 1 per ogni locus nella combinazione. In alcuni casi, le bande possono essere troppo vicini tra loro di apparire come luoghi separati con conseguente comparsa di solo 4 bande.
Dimensioni attese degli ampliconi sono elencati nella Tabella 1. Il nostro laboratorio utilizza NHDP63 ceppo di M. leprae come controllo positivo. Due tipi di segmenti di ripetere sono stati studiati: loci microsatelliti (con 1-5 ripetizioni di base) e loci minisatellite (con segmenti superiori a 5 paia di basi ripetute più volte). 5
Interpretazione dei file di dati da elettroforesi capillare si basa su due norme: uno interno frammenti di DNA dimensionamento standard chiamata GeneScan-500LIZ (ABI) e un campione di controllo positivo esterno amplificato di DNA batterico.
Il cromatogrammi FLA, visualizzare con il software di picco Scanner, può essere visto in Figure 5a, 5b e 6.
Scanner Peak fornisce i dati sulle dimensioni amplicone (asse x in paia di basi) e la potenza del segnale (asse y). (Figura 5b) Ulteriori dati sulla superficie del picco, ecc sono inoltre disponibili, anche se le dimensioni e valori di altezza di picco sono più importanti. Picchi meno di 100 unità in altezza sono di solito considerato un segnale troppo debole per essere affidabile.
Figura 6 confronta il controllo positivo (NHDP63) e due campioni dei pazienti, mostrando una variazione del numero di ripetizioni in tandem locus (GTA) 9. Nel controllo positivo, la sequenza (GTA) è ripetuta 10 volte. I VNTR NHDP63 e le dimensioni amplicone sono stati verificati attraverso il sequenziamento del gene. 4 del paziente amplicone PCR è di 3 bp più piccolo del controllo positivo che indica che sono solo 9 unità ripetitive, mentre la paziente 6 ha un amplicone che è di 3 bp più grande NHDP63 rivelando di 11 ripete (GTA). Paziente 2 era basso indice batterica (BI) con poco o nessun replicazione del DNA PCR, quindi nessun segnale FLA.
Difficoltà di interpretazione FLA dati avviene a volte a causa di 'balbettare'. Durante la reazione di PCR, la DNA polimerasi può produrre frammenti che sono 1 o più ripetizioni più o meno lungo di quanto l'allele fonte. Questi sono solitamente imputati come i picchi di minore altezza che circonda la vetta principale. Saranno 'in scaletta la', cioè il numero corretto di coppie di basi del segmento ripetere più grande o più piccolo del picco principale. Il risultato è una famiglia di picchi dello stesso colore. Figura 7 mostra questo con locus (TA) 10.
Un'altra difficoltà che si manifestano con FLA comporta 'A +' o 'A-tailing', in cui la DNA polimerasi aggiunge una singola base [di solito adenina (A)] per terminare la 3 'del segmento di DNA copiato. Questo dimostra in FLA come un picco a destra di un picco principale o balbuzie che è di 1 coppia di basi più grande del picco adiacente, come mostrato nella Figura 8. Non deve essere confuso con un picco principale o balbuzie. A picco principale e la sua A-coda sono considerati una singola specie. L'A-coda non altera il numero di ripetizioni VNTR. (Alcuni kit DNA polimerasi sono destinate a promuovere specificamente A-tailing per ridurre questo effetto di confondimento. Promuovere la completa A-tailing tende a produrre un unico picco, piuttosto che un paio di picchi.)
L'estrazione del DNA e prodotti di PCR contengono sia M. leprae e DNA umano, ma con l'eccezione di (TA) 18, i primer sono sufficientemente specifica che solo amplificare il DNA batterico, e non vi è poca o nessuna amplificazione del DNA umano. (TA) 18 produce spesso un picco a 242 paia di basi che viene dal DNA umano e non è visto nei campioni di DNA armadillo diversi passaggi (Figura 9).
La durata di conservazione di primer è generalmente buona a patto che siano mantenuti a -20 ° C, solo rimosse per l'uso immediato, e poi conservati a 4 ° C fino al consumo. Primer dovrebbe essere stabile a 4 ° C per 1-2 settimane. Anche se creando combinazioni di primer per PCR è po 'di tempo, si raccomanda che solo combinazione fondo sufficiente per un uso immediato essere preparati. Combinazioni di fondo sembrano degradarsi in qualche modo, se conservati per periodi prolungati.
TE dovrebbe essere aliquotati da grandi scorte, e aliquote possono essere conservate congelate oa temperatura ambiente. Maggiori aliquote di 200-400μl sono buoni per diluire le scorte fondo essiccati o concentrati (Figura 2a). Piccole aliquote di 10-50 microlitri sono utili per integrare i volumi del fondo combinazioni 3 e 4. TE è poco costoso e aliquote deve essere eliminata dopo l'uso.
Kit di enzimi multiplex sono abbastanza costosi e devono essere tenuti congelati (-20 ° C) fino al momento dell'uso. A seguito di preparazione della PCR, eventuali soluzioni multiplex non utilizzata deve essere immediatamente rigettati in 4 ° C. Il piccolo Qiagen PCR multiplex kit viene fornito con 3 tubi di mix multiplex, ciascuno dei quali contiene 0.85ml (850μl) di soluzione, sufficienti per circa il 65-70 PCR.
In generale, è meglio evitare il ripetersi, sbalzi di temperatura maggiore per i materiali utilizzati in questo tipo di attività di laboratorio tra cui i campioni di DNA, primer e le soluzioni kit multiplex. Tutti i materiali devono essere conservati a -20 ° C fino al momento, e poi conservato a 4 ° C fino al consumo. Conservazione a lungo termine del DNA deve essere a -80 ° C.
Tutti i materiali contenenti speso tessuto, primer e / o il DNA deve essere autoclavato ed eliminato quando non sono più di alcuna utilità. Tutti i campioni trattaticon bromuro di etidio o formammide dovrebbero essere trattati come rifiuti pericolosi e smaltiti in conformità con la politica pericolosi dell'istituzione materiali.
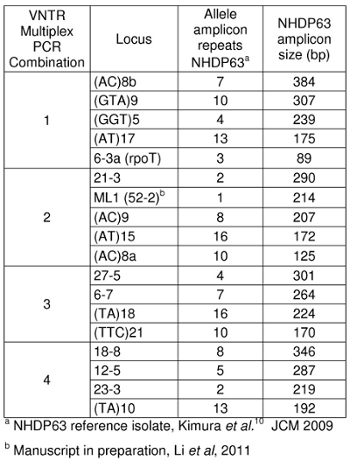
Tabella 1: dimensioni Amplicon per ceppo NHDP63
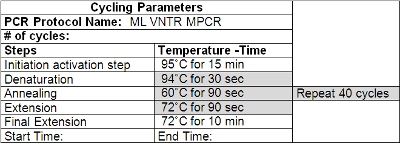
Tabella 2: Ciclismo Parametri per VNTR PCR

Tabella 3: Preparazione della PCR
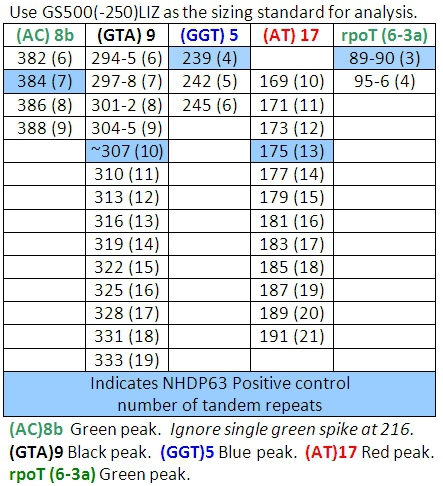
Tabella 4: le chiamate allele per Combinazione 1
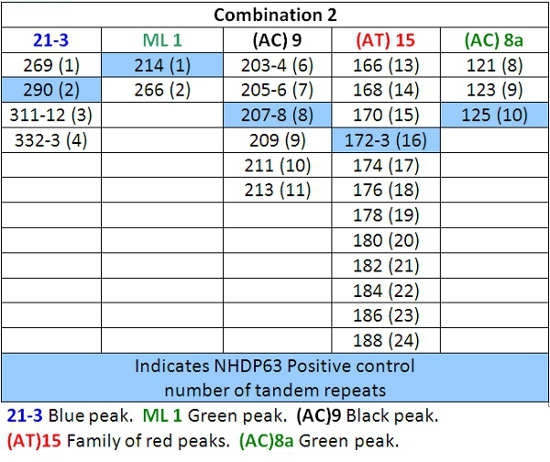
Tabella 5: le chiamate alleli per Combinazione 2
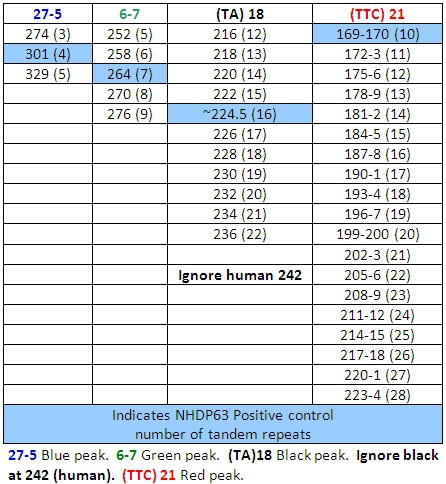
Tabella 6: le chiamate alleli per Combinazione 3
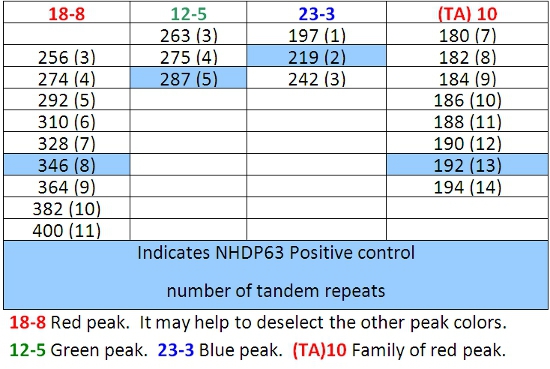
Tabella 7: le chiamate alleli per combinazione 4

Figura 1: VNTR-FLA diagramma di flusso
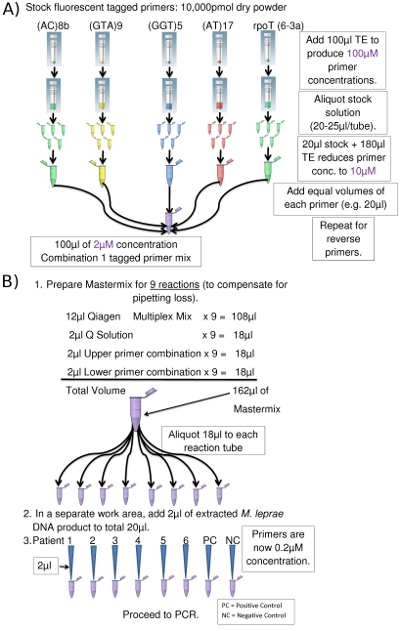
Figura 2. (A) Preparazione di primer Combinazione 1 superiore (per gentile concessione provetta Eppendorf foto di www.clker.com ). (B) installazione PCR (PCR per 8) (per gentile concessione provetta Eppendorf foto di www.clker.com)
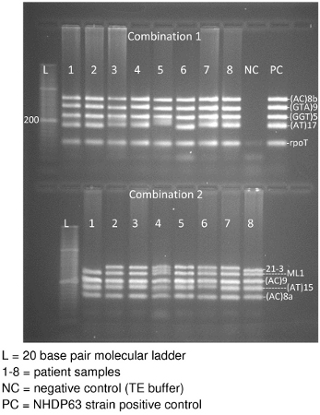
Figura 3. Gel di combinazioni 1 e 2 prodotto DNA VNTR PCR
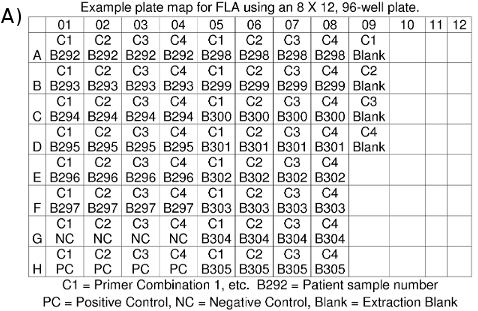
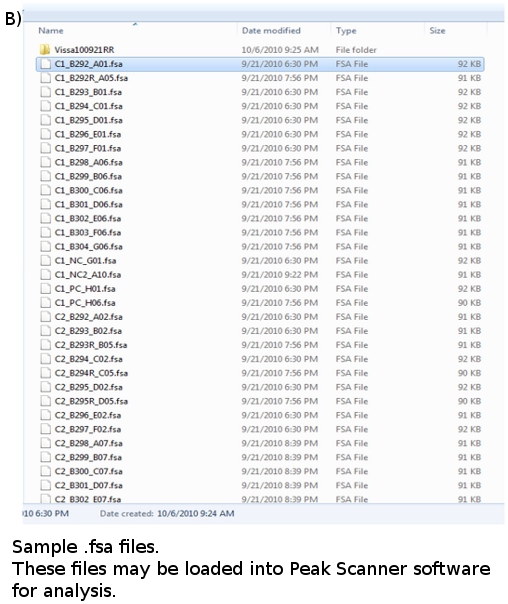
Figura 4. . (A) FLA Piastra Mappa (b) FLA file di dati: *. fsa
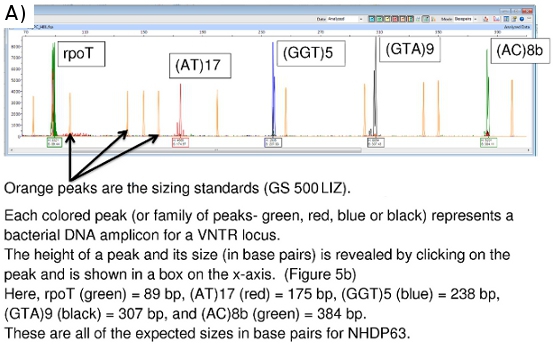
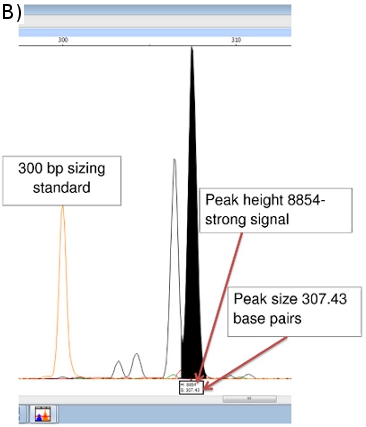
Figura 5. (A) Cromatogramma FLA di controllo positivo (NHDP63) per la combinazione 1 loci VNTR. (B) i dati scanner di picco per le dimensioni e l'abbondanza di amplicone (GTA) 9
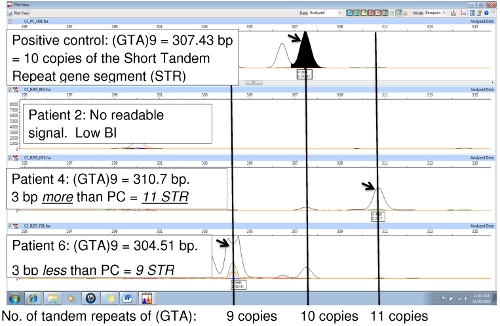
Figura 6. Confronto di campioni PCR al PC (NHDP63) per locus (GTA) 9
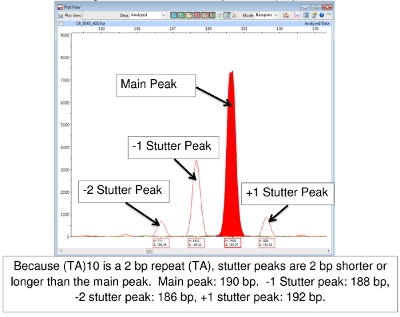
Figura 7. Principale e Peaks Stutter per (TA) 10
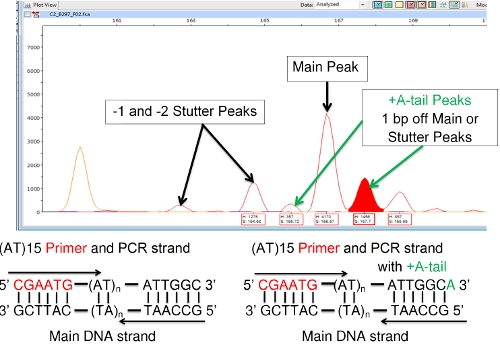
Figura 8: + A (A-coda) Peaks Adiacente Peaks principale o Stutter.
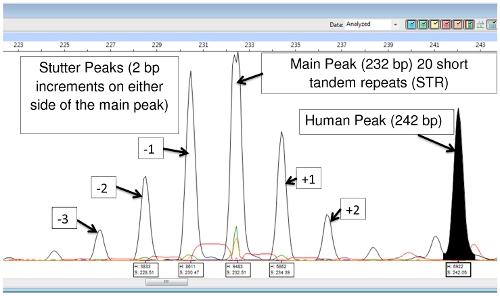
Figura 9: (TA) 18 vetta principale, Picchi Stutter e Peak DNA umano
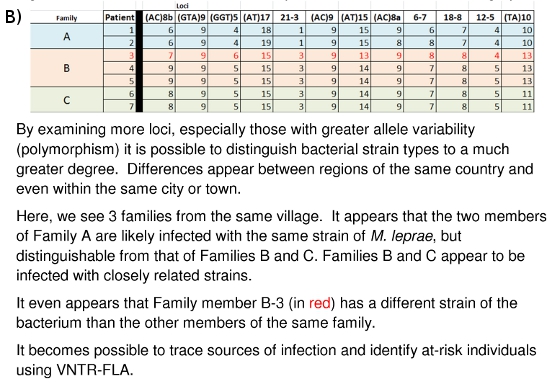
Figura 10:. (A) Differenziazione Strain di M. leprae sulla base dei dati VNTR (B) Differenziazione Strain di M. leprae sulla base delle impronte digitali MLVA DNA
Discussione
La raccolta di campioni di pelle di malati di lebbra richiede medici specializzati o tecnici che lavorano nelle cliniche della pelle. Personale di laboratorio maneggiare questi campioni devono prestare particolare attenzione ad indossare camici, guanti e occhiali protettivi e di lavorare in un bio-sicurezza gabinetto durante la manipolazione di campioni di tessuti umani infetti o armadillo. Disinfezione delle superfici e degli strumenti è anche critico. Lavorare in un ambiente pulito, sterile bio-safe mobile è importante per evitare la contaminazione dei campioni di DNA.
L'estrazione del DNA è diventato relativamente facile grazie allo sviluppo di kit di estrazione da aziende come Qiagen. Indicazioni devono essere seguite attentamente. Tutti i campioni devono essere conservati al freddo, quando non in uso. Evitare il ripetersi, sbalzi di temperatura per i campioni.
Primer DNA usato in questo lavoro può essere ordinato presso diverse aziende che sono state citate nella lista dei riferimenti bibliografici alla fine di questo documento. Si deve prestare attenzione a lavorare con primer in un ambiente DNA libero al fine di evitare la contaminazione. Primer vengono come polvere e deve essere miscelato con TE e diluito ad una concentrazione 100μM, poi separati in piccole quantità (Figura 2a). Soluzioni di lavoro di primer sono ulteriormente diluito a concentrazioni 10μM. Anche in questo caso, non utilizzare campioni di DNA in aree / cappe in cui vengono utilizzati primer e preparati.
I prodotti di PCR devono essere conservati al freddo, quando non in uso.
Un 3% di agarosio preparazione gel darà una migliore separazione banda del DNA, ma richiede più tempo per l'esecuzione. Per scopi puramente qualitativi, 2% è solitamente sufficiente. Soluzione di bromuro di etidio utilizzato per la colorazione del DNA in gel di agarosio è un genotoxin altamente attivo che viene assorbito attraverso la pelle. Gestire questa soluzione e gel colorati con essa utilizzando grande attenzione e sempre con la certezza di indossare i guanti. Lavarsi accuratamente le mani dopo aver toccato qualsiasi DNA campioni o materiali di bromuro di etidio. Smaltire soluzione colorante etidio bromuro, lavare e gel secondo le linee guida istituzionali. Gel non sono normalmente necessarie; questo passo può essere eliminata una volta metodi FLA sono state stabilite. E 'tempo e consumare reagenti.
La soluzione formamide utilizzati nella preparazione dei campioni per la FLA è anche altamente tossico e deve essere maneggiato con cura. Lavarsi le mani dopo l'uso. Disporre di esso le linee guida istituzionali.
Lettura dei risultati di FLA utilizzando il software di picco Scanner può essere impegnativo. Un insieme di base delle chiamate allele (numero di ripetizioni in tandem) è stato sviluppato a CSU (Tabelle 4-7). (Va notato che le tabelle 4-7 non può essere tutto compreso. Come altri ceppi di M. leprae sono studiati, alleli con numero di copie di fuori degli intervalli elencati possono essere trovati.) Una sfida particolare consiste 'balbettare' picchi. Si tratta di famiglie di picchi, in particolare di segnalazioni di operazioni sospette che coinvolgono solo 2-3 ripete coppia di basi, come ad esempio (TA) 10. Volte la scelta della punta giusta per leggere è difficile (Figura 7). In questa figura, il picco nel controllo positivo a 190 bp è la vetta principale. Picchi inferiori in altezza che sono 2, 4 o anche 6 coppie di basi più grande o più piccola della vetta principale si chiamano 'picchi balbettare.' A-tailing può anche causare confusione. A-tailing è descritto in precedenza nel testo e nella Figura 8. Infine, se i prodotti di PCR sono molto abbondanti nei campioni, i picchi possono apparire con un picco doppio. In questo caso, leggere il centro della forma di picco. (Vedi Figura 6, paziente 6.)
La gestione dei dati può essere un compito arduo per questo tipo di lavoro. E 'importante registrare tutte le informazioni pertinenti per tutti gli esperimenti, quali: data di un compito o di procedura, l'operatore, striscia / piastra mappe, ordini FLA, condizioni di PCR e le ricette, le date di gel e fotografie, temperature di conservazione, diluizioni modello di DNA, FLA percorsi di memorizzazione di file elettronici, organizzazione ecc Buono e gestione dei dati consente di risparmiare ore di tempo dedicato alla ricerca di informazioni specifiche per il futuro.
Il lavoro di laboratorio qui descritta è andata avanti per diversi anni presso la Colorado State University e in altri luoghi in tutto il mondo. L'immagine grande di quello che tutti i dati raccolti mezzi e come può essere di uso futuro sta iniziando ad emergere. Figure 10A e 10B dimostrare come questi le impronte digitali del DNA può essere utilizzato per distinguere differenti M. leprae tensioni tra paesi (10A) o anche tra le famiglie (10B). In materia di famiglia e di comunità collegate hanno dimostrato di portare M. leprae di tipo VNTR simili o identici ceppo. La speranza è che possa essere possibile ottenere una visione più completa la modalità (s) di trasmissione di lebbra in modo che un sistema di individuazione precoce delle reti di trasmissione possono essere sviluppate per quelle persone most a rischio, e che la terapia farmacologica curativa può iniziare prima di danni neurologici permanenti e dermatologici è fatto.
Divulgazioni
Riconoscimenti
Il finanziamento è stato fornito dal NIH / NIAID concedere RO1-AI-63457 e ARRA concedere supplemento RO1-AI-63457 S1. Riconosciamo il contributo di tutti i membri attuali e passati del gruppo di laboratorio e collaboratori.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
|---|---|---|---|
| Sangue DNeasy e Kit Tissue | Qiagen | 69504 | |
| Multiplex PCR Kit | Qiagen | 206143 | |
| DNA primer | Vari | ||
| Gel di agarosio | Vari | ||
| 5x o 6x Gel Loading Buffer | New England Biolabs | B7021S | Ricette a disposizione per rendere il vostro proprio * |
| Bromuro di etidio soluzione | Vari | Diluire da truffa. | |
| Hi-Di Formammide Solution | Applied Biosystems | 4311320 | |
| Gene Scan -500 LIZ | Applied Biosystems | 4322682 |
* Http://biowww.net/buffer-reagent/6X-Gel-loading-buffer-bromophenol-blue-sucrose.html
| Attrezzatura | Commenti |
|---|---|
| 2 frigoriferi | 4 ° C: 1 per i campioni di DNA, 1 per primer e reagenti Multiplex |
| 1 forno a microonde | O modo adatto per il riscaldamento agarosio e acqua per fare gel |
| 1 autoclave | O pentola a pressione per la sterilizzazione dei materiali |
| PCR macchina | Termociclatore |
| 2 set di pipette | 0,5-10 microlitri, 10-100 microlitri, 20-200 microlitri, 1000 ml 1 set per primer, 1 set di DNA |
| Sottomarino gel elettroforesi scatola | Con forme e pettini per la preparazione gel. |
| Elettroforesi alimentazione | Con cavi per il collegamento alla casella gel |
| Calore blocco | Per tubi Eppendorf |
| Centrifuga | Per tubi Eppendorf / strisce |
| Sistema di elettroforesi capillare (analisi genetiche) | O l'accesso a un istituto in grado di eseguire questa analisi |
| Plastica | Aerosol monouso puntali, provette Eppendorf, strisce da 8 pozzetti, piastre a 96 pozzetti, alcune delle qualità ottica |
| Computer con collegamento Internet |
Riferimenti
- World Health Organization. Chemotherapy of leprosy for control programmes. Technical Report Series. 675, 18-22 (1982).
- Cole, S. T. Massive gene decay in the leprosy bacillus. Nature. 409, 1007-1011 (2001).
- . Leproma Web Server. Leproma Web Server [Internet]. , (2004).
- Groathouse, N. A. Multiple Polymorphic Loci for Molecular Typing of Strains of Mycobacterium leprae. J. of Clinical Microbiology. 42, 1666-1672 (2004).
- Young, S. K. Use of Short Tandem Repeat Sequences to Study Mycobacterium leprae in Leprosy Patients in Malawi and India. Public Library of Science: Neglected Tropical Diseases. 2, E214-E214 (2008).
- Monot, M. Are Variable-Number Tandem Repeats Appropriate for Genotyping Mycobacterium leprae. J. of Clinical Microbiology. 46, 2291-2297 (2008).
- Kimura, M. Rapid Variable Number Tandem-Repeat Genotyping for Mycobacterium leprae Clinical Specimens. J. of Clinical Microbiology. 47, 1757-1766 (2009).
- Weng, X. i. a. o. m. a. n. Identification and Distribution of Mycobacterium leprae Genotypes in a Region of High Leprosy Prevalence in China: a 3-Year Molecular Epidemiological Study. J. of Clinical Microbiology. 45, 1728-1734 (2007).
- Weng, X. Transmission of leprosy in Qiubei County, Yunnan, China: Insights from an eight year molecular epidemiology investigation. Infection, Genetics and Evolution. , (2010).
- Sakamuri, R. M. Population-Based Molecular Epidemiology of Leprosy in Cebu, Philippines. J. of Clinical Microbiology. 47, 2844-2854 (2009).
- Fontes, A. N. B. Genetic diversity of Mycobacterium leprae isolates from Brazilian leprosy patients. Leprosy Review. 80, 302-315 (2009).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon