Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Fluorescence Biomembrane la Force Probe: quantification simultanée de Kinetics récepteur-ligand et induits Reliure-signalisation intracellulaire sur une cellule unique
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
We describe a technique for concurrently measuring force-regulated single receptor-ligand binding kinetics and real-time imaging of calcium signaling in a single T lymphocyte.
Résumé
Interactions récepteur-ligand à membrane médiation de nombreuses fonctions cellulaires. Cinétique de liaison et la signalisation en aval déclenchée par ces interactions moléculaires sont probablement affectés par l'environnement mécanique dans lequel la liaison et de signalisation ont lieu. Une étude récente a montré que la force mécanique peut réguler reconnaissance de l'antigène et par le déclenchement du récepteur des cellules T (TCR). Ceci a été rendu possible grâce à une nouvelle technologie développée et la fluorescence Qualifié de sonde de force de biomembrane (FBFP), qui combine la spectroscopie de force seule molécule avec la microscopie de fluorescence. Utiliser une cellule ultra-doux rouge sang humain comme le capteur de force sensible, une caméra à haute vitesse et en temps réel des techniques de suivi de l'imagerie, l'FBFP est de ~ 1 PN (10 -12 N), ~ 3 nm et ~ 0,5 ms en vigueur, la résolution spatiale et temporelle. Avec la FBFP, on peut mesurer précisément la cinétique simples de liaison récepteur-ligand en vertu du règlement de la force et cal intracellulaire simultanément l'image-liaison déclenchécium de signalisation sur une seule cellule vivante. Cette nouvelle technologie peut être utilisée pour étudier une autre interaction et la signalisation du récepteur-ligand membrane dans d'autres cellules sous régulation mécanique.
Introduction
Cellule à cellule et cellule-matrice extracellulaire à (ECM) est médiée par l'adhérence de liaison entre les récepteurs de surface cellulaire, des protéines de l'ECM, et / ou des lipides 1. Reliure permet cellules pour former des structures fonctionnelles 1, ainsi que de reconnaître, de communiquer, et réagissent à l'environnement 1-3. Contrairement aux protéines solubles (par exemple, cytokines et facteurs de croissance) qui se lient à partir d'un à trois dimensions (3D) en phase liquide sur les récepteurs de surface cellulaire, des récepteurs d'adhésion cellulaire forment des liaisons avec leurs ligands à travers une fente de jonction étroit pour combler deux surfaces opposées qui limitent moléculaire diffusion dans une dimensions (2D) Interface 7.4 deux. Contrairement à la cinétique 3D qui sont couramment mesurée par des tests de liaison traditionnels (par exemple, la résonance plasmonique de surface ou SPR), la cinétique 2D ont être quantifiés avec des techniques spécialisées telles que la microscopie à force atomique (AFM) 8-10, chambre 11,12 couler, 13,14 micropipette, optiquepincettes 15 et sonde de force de biomembrane (BFP) 16-21.
Plus que de simplement fournir liaison physique pour la cohésion cellulaire, des molécules d'adhésion sont une composante majeure de la machinerie de signalisation de la cellule pour communiquer avec son environnement. Il ya eu un intérêt croissant pour comprendre comment l'engagement ligand de molécules d'adhésion initie la signalisation intracellulaire et comment le signal initial est transduit intérieur de la cellule. Intuitivement, propriétés de liaison récepteur-ligand peut influer sur les signaux qu'il induit. Cependant, il est difficile de disséquer les relations mécanistes entre l'interaction extracellulaire et des événements de signalisation intracellulaire utilisant ensemble traditionnel des essais biochimiques en raison de leurs nombreuses limites, par exemple, une résolution temporelle pauvres et l'absence totale de la résolution spatiale. Des méthodes qui permettent à la fois biophysique (2D cinétique liaison récepteur-ligand) et biochimiques (signalisation) des observations sur direct existantcellules comprennent des substrats de rigidité accordable 22, élastomère tableaux pilier 23 et dispositifs chambre d'écoulement / microfluidiques intégrés avec une capacité de fluorescence 24-26. Cependant, des lectures de signalisation et de liaison au récepteur-ligand doivent être obtenus séparément (le plus souvent par des méthodes différentes), ce qui rend difficile de disséquer les relations temporelles et spatiales des caractéristiques d'adhérence avec les événements de signalisation.
BFP classique est une spectroscopie de force à la résolution spatio-temporelle ultrasensible haute 17. Il utilise une cellule souple rouge sang (RBC) comme un capteur de force, ce qui permet la mesure de la cinétique d'une seule molécule 2D, les propriétés mécaniques et les changements de conformation 14,16,19-21,27-29. Un BFP basée imagerie de fluorescence (FBFP) corrèle la cinétique de liaison récepteur-ligand avec la signalisation cellulaire de liaison déclenché à l'échelle de molécule unique. Avec cette configuration, à des activités de signalisation de cellule in situ dans le cadre de méca de surfacecal stimulation a été observée dans les lymphocytes T 27. Le FBFP est polyvalent et peut être utilisé pour des études de l'adhésion cellulaire et de la signalisation à médiation par d'autres molécules dans d'autres cellules.
Protocole
Ce protocole suit les directives de et a été approuvé par le comité d'éthique de la recherche humaine de l'Institut de technologie de Géorgie.
1. hématies humaines isolement, Biotinylation et osmolarité Ajustement
Note: Etape 1.1 doit être effectuée par un professionnel, comme une infirmière médical qualifié, avec un Institutional Review Board protocole approuvé.
- Obtenir 8-10 pi (une goutte) de sang du bout du doigt et ajouter à 1 ml de tampon carbonate / bicarbonate (tableau 1 et 2). Vortexer doucement ou d'une pipette le mélange et centrifuger pendant 1 min à 900 x g. Rejeter le surnageant et laver une fois de plus.
- Dans un petit bêcher peser 3,5 à 4 mg de biotine-NHS-PEG3500 lieur (tableau 1). Dissoudre dans le tampon de carbonate / bicarbonate de rendre la concentration finale de 3 mg / ml.
- Mélanger 171 pi de tampon carbonate / bicarbonate, 10 pi de paquet RBC et 1049 pi deLa biotine-NHS-PEG3500 solution de liaison et mettre en incubation à température ambiante pendant 30 min. Laver une fois avec le RBC tampon carbonate / bicarbonate et ensuite deux fois avec du tampon N2-5% (Tableau 1 et 2).
- Pendant ce temps, placer la bouteille de liaison avec bouchon dévissé dans un dessiccateur à vide en verre rempli d'agents desséchants dans le fond et le vide pendant 5 min, et remplissez le dessiccateur avec de l'argon. Serrer le bouchon et de prendre la bouteille. Sceller la bouteille avec un film de paraffine plastique (tableau 1), le placer dans un récipient rempli d'agents desséchants sur le fond et la ranger dans -20 ° C.
Remarque: Les étapes qui impliquent l'utilisation de biotine-NHS PEG3500-linker, y compris 1.2-1.4 (sauf pour l'incubation et laver à 1.3), doivent être effectuées aussi vite que possible. - Diluer nystatine dans le tampon de N2-5% pour obtenir une concentration finale de 40 pg / ml. Mélanger 5 ul de RBC biotinylé avec 71,4 ul de la nystatine (tableau 1) et on incube la solution pendant 1 heure à 0 &# 176; C. Laver deux fois avec N2-5% de tampon et de magasin avec N2-5% de tampon + 0,5% de BSA (tableau 1) dans le réfrigérateur (4 ° C).
2. Glass Bead Silanisation
- Nettoyage des perles Surface
- Peser 50 mg de billes de verre en poudre et remettre en suspension dans 500 ul eux d'eau DI.
- Mélanger 0,5 ml de 30% de H 2 O 2 (tableau 1) avec 9,5 ml d'eau désionisée dans un bêcher de 50 ml, puis ajouter 2 ml de NH4OH concentré (tableau 1) et porter cette solution à une chaudière sur une plaque chaude .
- Ajouter les perles de verre à la solution bouillante et continuer à faire bouillir pendant encore 5 min. Mélanger doucement la solution tous min.
- Après ébullition, transférer ~ 5 ml de cette suspension de billes chaud dans un tube de 15 ml de micro-centrifugeuse et le haut avec de l'eau DI RT. Centrifuger à 3500 g pendant 5 min, retirer et jeter le surnageant.
- Transfert encore 5 ml de suspension de billes chaude et ajouter aux billes lavées, Compléter avec plus d'eau DI, bien mélanger et centrifuger à nouveau. Répétez cette procédure jusqu'à environ 50 ml d'eau DI est utilisé, ce qui sera un total de 4 à 5 fois de lavage.
- Transférer la suspension de billes dans un flacon de 1 ml. Répéter le lavage des billes avec du methanol (tableau 1) par centrifugation à 17 000 xg pendant 5 min à 3 heures, et enfin remettre en suspension les billes dans 1 ml de methanol à 100%.
- Perle Surface Thiolation
- Pour un tube à centrifuger de 50 ml d'ajouter 45,6 ml de methanol, 0,4 ml d'acide acétique (tableau 1), 1,85 ml d'eau désionisée, 1,15 ml de 3-mercaptopropyltriméthoxysilane (MPTMS) (tableau 1) et 1 ml de perles suspension préparée à 2,1, puis incuber à température ambiante pendant 3 h.
- Après la réaction, enlever tous les réactifs en lavant une fois avec du méthanol frais, et remettre en suspension les perles dans 500 ul de méthanol. Répartir cette suspension de billes de verre concentrée en un ensemble de 20 flacons de verre sèches et propres avecles bouchons. Evaporer le méthanol à l'aide d'un jet d'argon sec et faire tourner lentement les flacons de façon à rendre une mince couche de perles sèches sur les côtés de chaque flacon.
- Placez les flacons de perles dans un four de séchage préchauffé à 120 ° C pendant 5 min puis sortez et rapidement placer le bouchon (s) lâchement sur. Placer les flacons dans un dessiccateur sous vide en verre rempli d'un dessiccant dans le fond et le dessiccateur sous vide avec une pompe à vide jusqu'à refroidissement.
- Purger le dessiccateur aspirateur d'argon sec pour apporter le dessiccateur à la pression atmosphérique normale. Retirez le couvercle du dessiccateur et rapidement re-serrer le bouchon (s) sur le flacon. Sceller les fioles avec un film de paraffine en plastique et de les stocker à température ambiante dans une boîte de stockage sec et sombre.
- Lors de l'utilisation immédiate, prendre un flacon de perles sèches et laver une fois avec un tampon phosphate (tableaux 1 et 2), remettre en suspension dans 50 pi de tampon phosphate et conserver à 4 ° C. Cette préparation de perles concentré sera désignécomme des «perles de MPTMS" dans les étapes suivantes.
Remarque: Avec un stockage, une des perles de MPTMS pourraient rester fonctionnel pour un maximum de trois mois.
3. Perle fonctionnalisation
- Revêtement de manière covalente protéines sur des billes
- Prenez un certain volume (par ex, 2,5 pi) de la protéine stock et mélanger avec un volume égal de tampon carbonate / bicarbonate de faire Solution 1.
Remarque: le volume dépend de la concentration des stocks et la densité des sites finale souhaitée de la protéine sur la surface des perles. - Dans un petit bêcher peser 2-3 mg de MAL-PEG3500-NHS lieur (tableau 1) et dissoudre avec du tampon carbonate / bicarbonate pour atteindre une concentration finale de 0,231 mg / ml.
- Mélanger une solution avec un volume égal de la solution de liant préparé en 3.1.2. Incuber le mélange à la température ambiante pendant 30 minutes pour faire la solution 2.
- Pendant ce temps, placer la bouteille de liaison avec bouchon dévissé dans le vide de verre dessiccateur filled avec desséchants dans le fond et le vide pendant 5 min, et remplissez le dessiccateur avec de l'argon. Serrer le bouchon et de prendre la bouteille. Sceller la bouteille avec un film de paraffine en plastique, placez-le dans un récipient rempli d'agents desséchants sur le fond et la ranger dans -20 ° C.
Remarque: Les étapes qui impliquent l'utilisation de MAL-PEG3500-NHS linker, y compris 3.1.2-3.1.4 (sauf pour l'incubation en 3.1.3), doivent être accomplis, aussi vite que possible. - Mélanger 5 ul de MPTMS perles avec la solution 2 et ajouter un tampon au phosphate (tableau 1) pour obtenir un volume final de 250 ul.
- Incuber les perles nuit à température ambiante, laver 3 fois avec un tampon phosphate, et remettre en suspension dans 100 pi de tampon phosphate et conserver à 4 ° C.
- Prenez un certain volume (par ex, 2,5 pi) de la protéine stock et mélanger avec un volume égal de tampon carbonate / bicarbonate de faire Solution 1.
- Protéines Préparation / Perles streptavidine (SA) revêtus
- Suivre le protocole 3.1.1-3.1.4.
- Mélanger 5 ul de MPTMS perles avec la solution 2 et 5 ul de 4 mg / ml de streptavidine-maléimide (MAL-SA) (tableau 1) Solution et puis ajouter un tampon phosphate pour obtenir un volume final de 250 pi.
- Incuber les perles nuit à température ambiante, laver 3 fois avec un tampon phosphate, et enfin remettre en suspension dans 100 pi de tampon phosphate et conserver à 4 ° C.
- Revêtement Streptavidin sur Perles de verre
- Mélanger 5 ul de billes MPTMS avec 5 pi de 4 mg / ml, solution SA et ajouter 140 pi de tampon phosphate.
- Incuber les perles nuit à température ambiante, laver 3 fois avec un tampon phosphate, et remettre en suspension dans 50 pi de tampon phosphate et conserver à 4 ° C.
- Le revêtement des Perles SA revêtue d'une protéine biotinylée
- Mélanger 5 ul de billes revêtues avec la protéine SA (volume en fonction de la densité souhaitée de revêtement) et ajouter un tampon phosphate pour obtenir un volume final à 100 ul soit.
- Incuber le mélange pendant une nuit à 4 ° C ou pendant 3 heures à température ambiante, laver 3 fois avec un tampon phosphate, et re-suspension dans 50 ul phosphate tampon et stocker à 4 ° C.
4. Préparation des cellules
Remarque: Pour purifier les cellules, suivre les protocoles de purification de cellules standards correspondant au type de cellules dans l'utilisation, par exemple les cellules T 27 ou certaines lignées cellulaires 21,29.
- Pour les expériences FBFP, une fois que la suspension de cellules est préparé, ajouter Fura2-AM (Tableau 1) dissous dans du DMSO dans la suspension de cellules pour atteindre une concentration finale de 2 uM, incuber pendant 30 min à température ambiante, puis laver une fois. Gardez cette suspension cellulaire par fluorescence chargé dans l'obscurité jusqu'à utilisation.
5. Préparation pour Micropipettes et une Chambre cellulaire
- Préparation Micropipettes
- Tubes Coupe longue capillaires de verre (Tableau 1) avec un coupe-verre en petits morceaux d'environ 3 pouces de longueur. Monter une pièce sur l'extracteur micropipette (tableau 1), cliquez sur le "Pull" maistonne sorte que le milieu du capillaire sera chauffée par la machine et le capillaire sera tiré sur les deux extrémités de faire deux capillaires avec des bouts pointus (micropipettes premières).
Remarque: En suivant la ligne directrice de produit, la morphologie souhaitée de la pipette brute a 6-8 mm cône et 0,1-0,5 um pointe. - Monter une pipette brut sur le support de pipette de la forge de micropipette (tableau 1). Chaleur pour faire fondre la sphère de verre sur la forge. Insérer la pointe de la pipette cru à l'intérieur de la sphère de verre. Refroidir la sphère de verre et tirez la pipette brute pour casser de l'extérieur et de laisser sa pointe à l'intérieur de la sphère. Répétez cette procédure jusqu'à ce que l'orifice de la buse désirée est obtenue.
Remarque: Les exemples d'un diamètre intérieur de la pointe de micropipette: 2,0-2,4 um pour un RBC, ~ 1,5 um pour une perle, ~ 2-4 pm pour une cellule T et -7 um pour une cellule d'hybridome.
- Tubes Coupe longue capillaires de verre (Tableau 1) avec un coupe-verre en petits morceaux d'environ 3 pouces de longueur. Monter une pièce sur l'extracteur micropipette (tableau 1), cliquez sur le "Pull" maistonne sorte que le milieu du capillaire sera chauffée par la machine et le capillaire sera tiré sur les deux extrémités de faire deux capillaires avec des bouts pointus (micropipettes premières).
- Construire une chambre de la cellule
Remarque: la chambre de cellule est construit sur la base d'une maison-foue chambre titulaire, qui se compose de deux pièces d'équerres métalliques (cuivre / aluminium) et d'une poignée qui les relie entre eux (figure 1D). - Coupez un 40 mm x 22 mm x 0,2 mm lamelle en utilisant un coupe-verre en deux morceaux de 40 mm x 11 mm x 0,2 mm (lamelle 1 et 2). Colle lamelle couvre-1 par de la graisse sur le côté supérieur de la porte de la chambre dans la façon dont il relie les deux équerres métalliques, et de même lamelle colle 2 à la face inférieure, qui forme une chambre de cellule parallèles lamelle (Figure 1D).
- Utiliser une pipette pour injecter 200 ul de tampon expérimental entre les deux lamelles. Assurez-vous que le tampon attache à deux lamelles. Tournez doucement et agiter la chambre de laisser le tampon toucher les deux extrémités de la chambre.
- Injecter délicatement l'huile minérale dans les deux côtés de la chambre flanquant la zone tampon expérimentale scellant ainsi le tampon de l'air. Injecter des suspensions de perles de sonde (par exemple, des perles pMHC-couché), les globules rouges et les cibles(par exemple, des cellules T) dans les régions supérieure, intermédiaire et inférieure de la zone tampon, respectivement.
6. expérience BFP
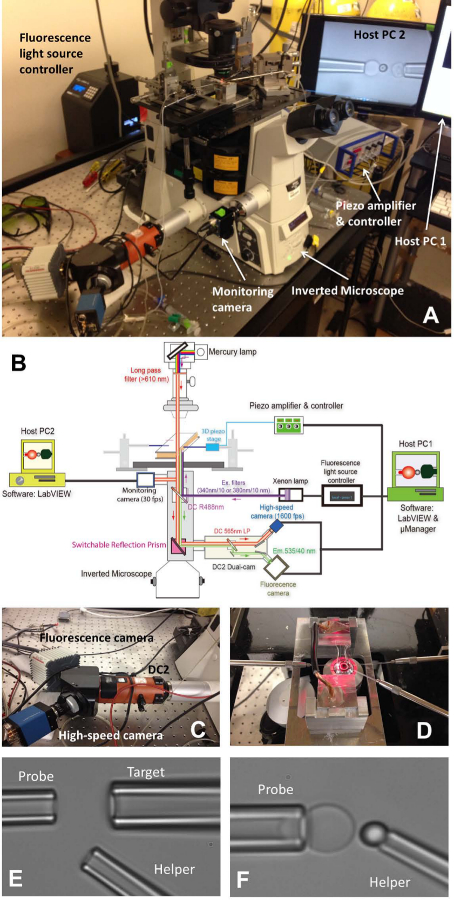
Figure 1: assemblage FBFP (A) Un aperçu de l'image du système matériel FBFP.. (B) Un dessin schématique du système de matériel FBFP. (C) Le système dual-cam "DC2" (orange) sur lequel la caméra à grande vitesse (bleu) et un appareil photo de fluorescence (blanc) ont été montés. (D) de la platine du microscope qui adapte une chambre d'essai et trois systèmes de manipulation de micropipette. Des micrographies de réglage BFP dans une chambre expérimentale (E et F). (E) Micropipettes montrant l'assemblage de la pipette de la sonde (à gauche), cible pipette (en haut à droite) et aide pipette (r inférieureroite). (F) Sonde placement de perles. Un cordon de sonde a été manipulé par une pipette d'aide et attaché à un sommet RBC pour former une sonde de force. S'il vous plaît cliquez ici pour voir une version plus grande de cette figure.
- Allumez le microscope (tableau 1) et la source lumineuse. Placez la chambre sur la scène principale du microscope (figure 1A, D).
- Installez les trois micropipettes de BFP (figure 1D Gauche:. Sonde, pour prendre un RBC, à droite: cible, pour saisir une cellule ou une perle, en bas à droite: aide, pour saisir une perle).
- Utilisation d'un micro-injecteur (tableau 1) pour remblayer une micropipette avec un tampon expérimental. Enlevez le porte-pipette (tableau 1) et maintenez-le à une place inférieure pour permettre de gouttes d'eau à partir de la pointe. Insérer rapidement la micropipette dans la pointe de support et assurez-vous qu'aucune bulle d'air pénètre dans la micropipetteau cours de l'insertion. Serrer la vis du support.
- Montez chaque titulaire de la pipette sur son micro-manipulateur correspondant. Pousser la micropipette vers la chambre de sorte que leurs conseils entrent dans la zone tampon de la chambre. Ajustez la position de la micropipette et les trouver sous le champ de vision du microscope.
- Déplacez-vous dans l'étape de support de chambre pour trouver les colonies de trois éléments (les globules rouges, les objectifs et les perles de sonde) un par un. Ajustez la position de la micropipette correspondante en tournant les boutons des manipulateurs de laisser la pointe de l'approche de micropipette une cellule / perle. Aspirer la cellule / perle en ajustant la pression à l'intérieur de la micropipette correspondant. Tous trois seront micropipettes capturer leurs éléments correspondants.
- Déplacez-vous dans l'étape de support de chambre pour trouver un espace ouvert loin des colonies des éléments injectés où l'expérience sera effectuée. Mettez le mode visuel de microscope pour visualiser l'image sur l'échantillonprogramme uter sur l'écran de l'ordinateur. Déplacer tous les trois éléments sur les embouts de pipette dans le champ de vision du programme.
- Alignez le cordon de sonde et RBC, et manœuvrer avec précaution le cordon de la sonde à l'apex de la RBC, empiéter brièvement la perle sur la RBC et doucement se rétracter. Régler la pression de la micropipette d'aide pour souffler doucement le cordon loin, de sorte qu'il sera laissé collé sur le sommet RBC (figure 1F). Eloignez-vous de la micropipette d'aide et aligner la cible et de la sonde talon (figure 2A).
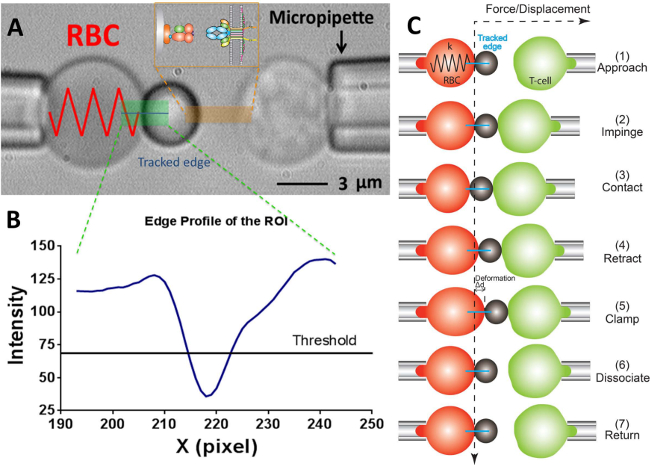
Figure 2:. Régime BFP et son cycle d'essai (A) Vidéo-micrographie illustrant une sonde de force (à gauche) et un T-cellule cible (à droite) aspiré par leur capteur de force stationnaire respective pipettes.The se compose d'un RBC gonflé et un joint ligand portant bourrelet. Le récepteur palier-Des cellules T (cible) est monté sur une face de translation piézo-électrique aligné à la sonde. Le retour sur investissement est indiquée en vert. Le tracker de bord est indiqué dans une ligne bleue. L'insert montre le ligand (pMHC, côté talon) et le récepteur (TCR, côté T-cellules) paire sur les deux surfaces opposées dans la zone marquée en orange. (B) Le profil d'intensité de bord de bourrelet (A). La région de ROI dans la direction- x est tracé comme axe des x (en nombre de pixels) et l'intensité lumineuse (en valeur d'échelle de gris) en moyenne par binning 30 pixels sur l'y direction-. (C) La déviation du RBC et la position de la bille et la cible (cellules T) dans un cycle d'essai de dosage force de serrage. Les lignes pointillées verticales et horizontales indiquent la position de l'apex RBC et le déroulement dans le temps zéro de la force, respectivement. Le tracker de bord de ligne de la déformation RBC est représenté en bleu dans chaque panneau. Les mêmes encore moins d'étapes sont adoptées dans la fréquence d'adhérencetest (qui n'a pas les étapes de "serrage" et "dissocier") et le dosage de fluctuation thermique (qui manque l'étape de "dissocier").
- Dans le programme, dans la fenêtre de champ de vision en utilisant les outils dans le programme pour mesurer les rayons respectifs de la micropipette de sonde (R p), le RBC (R 0), la zone de contact circulaire entre la RBC et sonde bourrelet (R c) , qui permet l'estimation de la constante de ressort de la RBC (k) par l'équation suivante 17,30,
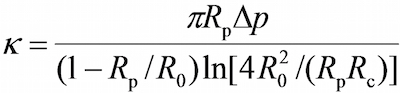
Δ où p est la pression d'aspiration à l'extrémité sonde de pipette.
Remarque: Il résulte de la loi de Hooke ce que la force de liaison, F, peut être quantifiée par le produit de la constante de ressort et le déplacement de la bille de palpage (d), soit, F = d (Figure 2C). - Entrez la constante souhaitée de printemps RBC dans le programme (S'il vous plaît se référer au chapitre Protocole 6.6. La constante de ressort est généralement fixé à 0,25 ou 0,3 PN / nm pour le test de fréquence de dosage vigueur pince et l'adhérence et 0,1 PN / nm pour le test de fluctuation thermique) , qui renvoie une pression d'aspiration nécessaire dans l'unité de centimètres d'eau. Ajustez la hauteur du réservoir d'eau qui relie à la pipette de la sonde jusqu'à ce que la pression d'aspiration souhaitée est atteinte.
- Tracez une ligne horizontale à travers le sommet de RBC, qui donnera une courbe dans la fenêtre adjacente indiquant la luminosité (valeur d'échelle de gris) de chaque pixel le long de cette ligne. Faites glisser la ligne de seuil pour être à peu près la moitié de la profondeur de la courbe (figure 2A, B).
Remarque: Le point minimum de la courbe de luminosité en dessous de la ligne de seuil indique la position de la limite de perles, ainsi qu'un minimum local est autorisé. Si deux ou plusieurs minima locaux sont à l'heure actuelle, ilindique que l'image est pas optimale (probablement en raison de l'image étant hors du foyer, ou d'un mauvais alignement entre le bourrelet de la sonde et la RBC). - Sélectionnez le mode d'expérience souhaitée: test de fluctuation thermique, l'adhérence analyse de fréquence ou de dosage force de serrage. Définissez les paramètres comme désiré (par exemple, la force d'impact = 15 pN, le taux de charge = 1000 PN / s, temps de contact = 1 sec, la force de serrage = 20 PN (pour le dosage de serrage de la force), etc.).
- Cliquez sur "Démarrer", qui permet au programme de déplacer la pipette cible et conduire la cible dans et hors de contact avec la sonde (voir la section Les résultats représentatifs pour plus de détails). La collecte des données est effectuée en parallèle, qui enregistre la position du cordon de sonde en temps réel. Arrêtez le programme en cliquant sur le bouton "Stop expérience», date à laquelle une fenêtre va sortir pour permettre la sauvegarde des données acquises.
7. Fluorescence BFP (FBFP) expérience
- Pour utiliser le Fluorescence fonction du système BFP, allumer la source de lumière d'excitation (tableau 1) et la caméra à fluorescence (tableau 1), qui sont commandés par un programme distinct (tableau 1). Au programme, sélectionnez les paramètres pour l'imagerie de fluorescence, y compris le gain, l'exposition, les canaux d'excitation (dans ce cas, 340 nm et 380 nm lumière), etc. Suivez toutes les préparations dans le protocole expérimental BFP, y compris l'alignement de la sonde et la cible, ce qui permettra pour la visualisation de l'image fluorescente en direct de la cellule cible excité par 340 nm ou 380 nm excitation lumineuse.
- Utilisez l'outil de découpe à peu près la section la zone dans laquelle la cellule restera au cours de la période d'enregistrement.
Remarque: En raison de l'utilisation du cycle approche contact-rétraction, la cellule sera en mouvement vers l'avant et vers l'arrière de façon répétitive, ainsi la zone en coupe est beaucoup plus grande que la cellule elle-même. - Cliquez sur «Enregistrer» pour permettre à l'un de 340 nmd 380 nm lumière pour exciter alternativement le colorant fluorescent intracellulaire (Fura2), et une paire d'images de fluorescence correspondant sera alternativement enregistré environ une fois par seconde. Simultanément cliquez sur "Démarrer" dans le programme pour commencer l'expérience BFP pour l'interaction moléculaire de l'analyse et de l'expérience de l'imagerie de fluorescence pour surveiller la signalisation du calcium intracellulaire. Le système produira un fichier de données brutes pour la liaison récepteur-ligand (voir la figure 6A ci-dessous) et une série d'images fluorescentes en format .tiff pour les signaux de calcium.
8. Analyse des données
- Analyse BFP données
- L'analyse des données pour l'analyse de la fréquence d'adhérence
- Inspecter séquentiellement "force en fonction du temps" le signal de chaque cycle et simplement enregistrer cycles qui contiennent un événement d'adhérence et qui ne le font pas, et résument pour obtenir une fréquence moyenne d'adhérence.
- Recueillir la force de rupture de chaque événement d'adhérence, quiest la valeur de crête de la force linéaire rampe avant obligataire rupture. Après avoir recueilli une quantité suffisante de forces de rupture dans une fourchette de taux de rampe, dériver la répartition de la force de rupture à chaque taux de rampe à partir de laquelle est dérivée la force dépendant hors vitesse de dissociation du récepteur-ligand en utilisant la force dynamique analyse par spectroscopie 18,31.
- L'analyse des données pour l'analyse de fluctuation thermique
- Inspecter séquentiellement le signal de phase de chaque cycle de serrage, qui contient probablement plusieurs association et de dissociation liaison événements. Utiliser le niveau de fluctuation thermique en phase serrage (l'écart-type moyen d'un intervalle glissant de 70 points temporels séquentiels de la position de la bille) dans la "force en fonction du temps" de signal en tant que guide pour distinguer les événements d'association et de dissociation liaison, depuis une liaison formation correspond à une diminution de la fluctuation thermique.
- Désigner l'intervalle entre le moment de la dissociation de la liaison (lorsque le therfluctuation mal reprend au niveau normal) à l'instant de la prochaine formation d'une liaison que le temps d'attente, et de désigner la durée de la liaison de son association à la dissociation que la durée de vie des obligations, qui sont tous deux recueillis lors de l'inspection des données. Calculer le temps moyen d'attente et la durée de vie moyen des obligations, qui reflètent respectivement l'inverse de sur-taux et celle de taux hors sous zéro vigueur 16,30.
- L'analyse des données pour l'analyse force de serrage
- paramètres d'enregistrement de tous les événements de vie, y compris la force moyenne et la durée de la liaison avec le numéro de séquence ainsi que l'heure de début et l'heure de fin, ce qui permettra de dessiner une courbe cumulative à vie (par exemple, la figure 6C, la courbe jaune).
- Prélever une quantité suffisante d'événements de vie dans une gamme de forces. Regroupez-les dans des bacs différents de force, qui va produire une durée de vie moyenne dans chaque bac de la force, et le rendement tout à fait un «durée de vie moyenne vs. vigueur "courbe (Figure 4).
- L'analyse des données pour l'analyse de la fréquence d'adhérence
- fluorescence de calcium Analyse des données d'imagerie
- Réglez le seuil d'intensité jusqu'à ce que les images de fluorescence montrent un contour clair de la cellule dans les deux canaux 340 nm et 380 nm, sans bruit de fond (figure 5A, B). Ensuite, examiner le Ca 2+ trame de signal intracellulaire par image avec un pseudo couleur indiquant le niveau d'intensité (figure 6B), qui est dérivée basée sur le rapport d'intensité de 340 nm / 380 nm, pour générer le "Ca 2+ intensité normalisée vs . courbe de temps "(Figure 6C). Utilisez les images de fluorescence pseudo-couleur pour produire un film qui affiche le deuxième taux de fluorescence par seconde.
Résultats
La technique BFP a été mis au point par le laboratoire Evans à 1995 17. Cet outil de picoforce a été largement utilisé pour mesurer les interactions de protéines immobilisées sur des surfaces, de manière à analyser la cinétique à deux dimensions de molécules d'adhérence interagissant avec leurs ligands 16,19,20, 30, pour mesurer l'élasticité moléculaire 21,29, et de déterminer la protéine de conformation change 21. Pour un FBFP, un ensemble suppléme...
Discussion
Une expérience réussie FBFP implique quelques considérations critiques. Tout d'abord, pour le calcul de la force pour être fiable, la micropipette, la RBC, et le cordon de la sonde doivent être alignées aussi près que possible coaxial. La projection de l'intérieur de la pipette RBC devrait être d'environ une sonde de diamètre de la pipette de sorte que le frottement entre le RBC et la pipette est négligeable. Pour une RBC humain typique, le diamètre de la pipette est optimal de 2,0 à 2,4 um, ce ...
Déclarations de divulgation
The authors have nothing to disclose.
Remerciements
Research related to this paper and the development of the fBFP technology in the Zhu lab were supported by NIH grants AI044902, AI077343, AI038282, HL093723, HL091020, GM096187, and TW008753. We thank Evan Evans for inventing this empowering experimental tool, and members of the Evans lab, Andrew Leung, Koji Kinoshita, Wesley Wong, and Ken Halvorsen, for helping us to build the BFP. We also thank other Zhu lab members, Fang Kong, Chenghao Ge and Kaitao Li, for their helps in the instrumentation development.
matériels
| Name | Company | Catalog Number | Comments |
| Sodium Phosphate Monobasic Monohydrate (NaH2PO4 • H2O) | Sigma-Aldrich | S9638 | Phosphate buffer preparation |
| Anhy. Sodium Phosphate Dibasic (Na2HPO4) | Sigma-Aldrich | S7907 | Phosphate buffer preparation |
| Sodium Carbonate (Na2CO3) | Sigma-Aldrich | S2127 | Carbonate/bicarbonate buffer preparation |
| Sodium Bicarbonate (NaHCO3) | Sigma-Aldrich | S5761 | Carbonate/bicarbonate buffer preparation |
| Sodium chloride (NaCl) | Sigma-Aldrich | S7653 | N2-5% buffer preparation |
| Potassium chloride (KCl) | Sigma-Aldrich | P9541 | N2-5% buffer preparation |
| Potassium phosphate monobasic (KH2PO4) | Sigma-Aldrich | P5655 | N2-5% buffer preparation |
| Sucrose | Sigma-Aldrich | S0389 | N2-5% buffer preparation |
| MAL-PEG3500-NHS | JenKem | A5002-1 | Bead functionalization |
| Biotin-PEG3500-NHS | JenKem | A5026-1 | RBC biotinylation |
| Nystatin | Sigma-Aldrich | N6261 | RBC osmolarity adjustment |
| Ammonium Hydroxide (NH4OH) | Sigma-Aldrich | A-6899 | Glass bead silanization |
| Methanol | BDH | 67-56-1 | Glass bead silanization |
| 30% Hydrogen Peroxide (H2O2) | J. T. Barker | Jan-86 | Glass bead silanization |
| Acetic Acid (Glacial) | Sigma-Aldrich | ARK2183 | Glass bead silanization |
| 3-Mercaptopropyltrimethoxysilane (MPTMS) | Uct Specialties, llc | 4420-74-0 | Glass bead functionalization |
| Borosilicate Glass beads | Distrilab Particle Technology | 9002 | Glass bead functionalization |
| Streptavidin−Maleimide | Sigma-Aldrich | S9415 | Glass bead functionalization |
| BSA | Sigma-Aldrich | A0336 | Ligand functionalizing |
| Fura2-AM | Life Technologies | F-1201 | Intracellular calcium fluorescence dye loading |
| Dimethyl sulfoxide (DMSO) | Sigma-Aldrich | D2650 | Intracellular calcium fluorescence dye loading |
| Quantibrite PE Beads | BD Biosciences | 340495 | Density quantification |
| Flow Cytometer | BD Biosciences | BD LSR II | Density quantification |
| Capillary Tube 0.7-1.0 mm x 30 inches | Kimble Chase | 46485-1 | Micropipette making |
| Flaming/Brown Micropipette Puller | sutter instrument | P-97 | Micropipette making |
| Pipette microforce | Narishige | MF-900 | Micropipette making |
| Mineral Oil | Fisher Scientific | BP2629-1 | Chamber assembly |
| Microscope Cover Glass | Fisher Scientific | 12-544-G | Chamber assembly |
| Micro-injector | World Precision Instruments | MF34G-5 | Chamber assembly |
| 1 ml syringe | BD | 309602 | chamber assembly |
| Micropipette holder | Narishige | HI-7 | Chamber assembly |
| Home-designed mechanical parts and adaptors fabrications using CNC machining. | Biophysics Instrument | All parts are customized according to the CAD designs. | BFP system |
| Microscope (TiE inverted) | Nikon | MEA53100 | BFP system |
| Objective CFI Plan Fluor 40x (NA 0.75, WD 0.72 mm, Spg) | Nikon | MRH00401 | BFP system |
| Camera, GE680, 640 x 480, GigE, 1/3" CCD, mono | Graftek Imaging | 02-2020C | BFP system |
| Prosilica GC1290 - ICX445, 1/3", C-Mount, 1280 x 960, Mono., CCD, 12 Bit ADC | Graftek Imaging | 02-2185A | BFP system |
| Manual submicron probehead with high resolution remote control | Karl Suss | PH400 | BFP system |
| Anti-vibration table (5’ x 3’) | TMC | 77049089 | BFP system |
| 3D manual translational stage | Newport | 462-XYZ-M | |
| SolidWorks 3D CAD software | SOLIDWORKS Corp. | Version 2012 SP5 | BFP system |
| LabVIEW software | National Instruments | Version 2009 | BFP system, BFP program |
| 3D piezo translational stage | Physik Instrumente | M-105.3P | BFP system |
| Linear piezo accuator | Physik Instrumente | P-753.1CD | BFP system |
| Micromanager software | Version 1.4 | fBFP system, fluorescence imaging program | |
| Dual Cam (DC-2) | Photometrics | 77054724 | fBFP system |
| Dual Cam emission filter (T565LPXR) | Photometrics | 77054725 | fBFP system |
| Fluorescence Camera | Hamamatsu | ORCA-R2 C10600-10B | fBFP system |
| Plastic paraffin film (Parafilm) | Bemis Company, Inc | PM996 | bottle sealing |
| Carbonate/bicarbonate buffer (pH 8.5) | 8.4 g/L sodium carbonate (Na2CO3), 10.6 g/L sodium bicarbonate (NaHCO3) | ||
| Phosphate buffer (pH 6.5-6.8) | 27.6 g/L NaPhosphate monobasic (NaH2PO4 • H2O), 28.4 g/L Anhy. NaPhosphate dibasic (Na2HPO4) | ||
| N2-5% buffer (pH 7.2) | 20.77 g/L potassium chloride (KCl), 2.38 g/L sodium chloride (NaCl), 0.13 g/L potassium phosphate monobasic (KH2PO4), 0.71 g/L anhy. sodium phosphate dibasic (Na2HPO4), 9.70 g/L sucrose |
Références
- Aplin, A. E., Howe, A., Alahari, S. K., Juliano, R. L. Signal transduction and signal modulation by cell adhesion receptors: the role of integrins, cadherins, immunoglobulin-cell adhesion molecules, and selectins. Pharmacological reviews. 50, 197-263 (1998).
- Davis, M. M., Bjorkman, P. J. T-cell antigen receptor genes and T-cell recognition. Nature. 334, 395-402 (1988).
- Dado, D., Sagi, M., Levenberg, S., Zemel, A. Mechanical control of stem cell differentiation. Regenerative medicine. 7, 101-116 (2012).
- Edwards, L. J., Zarnitsyna, V. I., Hood, J. D., Evavold, B. D., Zhu, C. Insights into T cell recognition of antigen: significance of two-dimensional kinetic parameters. Frontiers in immunology. 3, 86 (2012).
- Zhu, C., Jiang, N., Huang, J., Zarnitsyna, V. I., Evavold, B. D. Insights from in situ analysis of TCR-pMHC recognition: response of an interaction network. Immunological reviews. 251, 49-64 (2013).
- Huang, J., Meyer, C., Zhu, C. T. T cell antigen recognition at the cell membrane. Molecular immunology. 52, 155-164 (2012).
- Zarnitsyna, V., Zhu, C. T. T cell triggering: insights from 2D kinetics analysis of molecular interactions. Physical biology. 9, 045005 (2012).
- Binnig, G., Quate, C. F., Gerber, C. Atomic Force Microscope. Physical Review Letters. 56, 930-933 (1986).
- Marshall, B. T., et al. Direct observation of catch bonds involving cell-adhesion molecules. Nature. 423, 190-193 (2003).
- Kong, F., Garcia, A. J., Mould, A. P., Humphries, M. J., Zhu, C. Demonstration of catch bonds between an integrin and its ligand. The Journal of cell biology. 185, 1275-1284 (2009).
- Yago, T., et al. Catch bonds govern adhesion through L-selectin at threshold shear. The Journal of cell biology. 166, 913-923 (2004).
- Yago, T., et al. Platelet glycoprotein Ibalpha forms catch bonds with human WT vWF but not with type 2B von Willebrand disease vWF. The Journal of clinical investigation. 118, 3195-3207 (2008).
- Chesla, S. E., Selvaraj, P., Zhu, C. Measuring two-dimensional receptor-ligand binding kinetics by micropipette. Biophysical journal. 75, 1553-1572 (1998).
- Huang, J., et al. The kinetics of two-dimensional TCR and pMHC interactions determine T-cell responsiveness. Nature. 464, 932-936 (2010).
- Heinrich, V., Wong, W. P., Halvorsen, K., Evans, E. Imaging biomolecular interactions by fast three-dimensional tracking of laser-confined carrier particles. Langmuir : the ACS journal of surfaces and colloids. 24, 1194-1203 (2008).
- Chen, W., Evans, E. A., McEver, R. P., Zhu, C. Monitoring receptor-ligand interactions between surfaces by thermal fluctuations. Biophysical journal. 94, 694-701 (2008).
- Evans, E., Ritchie, K., Merkel, R. Sensitive force technique to probe molecular adhesion and structural linkages at biological interfaces. Biophysical. 68, 2580-2587 (1995).
- Evans, E., Leung, A., Heinrich, V., Zhu, C. Mechanical switching and coupling between two dissociation pathways in a P-selectin adhesion bond. Proceedings of the National Academy of Sciences of the United States of America. 101, 11281-11286 (2004).
- Ju, L., Dong, J. -. f., Cruz, M. A., Zhu, C. The N-terminal Flanking Region of the A1 Domain Regulates the Force-dependent Binding of von Willebrand Factor to Platelet Glycoprotein Ib. Journal of Biological Chemistry. 288, (2013).
- Chen, W., Lou, J., Zhu, C. Forcing switch from short- to intermediate- and long-lived states of the alphaA domain generates LFA-1/ICAM-1 catch bonds. The Journal of biological chemistry. 285, 35967-35978 (2010).
- Chen, W., Lou, J., Evans, E. A., Zhu, C. Observing force-regulated conformational changes and ligand dissociation from a single integrin on cells. The Journal of cell biology. 199, 497-512 (2012).
- Judokusumo, E., Tabdanov, E., Kumari, S., Dustin, M. L., Kam, L. C. Mechanosensing in T lymphocyte activation. Biophysical journal. 102, L5-L7 (2012).
- Bashour, K. T., et al. CD28 and CD3 have complementary roles in T-cell traction forces. Proceedings of the National Academy of Sciences of the United States of America. 111, 2241-2246 (2014).
- Nesbitt, W. S., et al. Distinct glycoprotein Ib/V/IX and integrin alpha IIbbeta 3-dependent calcium signals cooperatively regulate platelet adhesion under flow. The Journal of biological chemistry. 277, 2965-2972 (2002).
- Mazzucato, M., Pradella, P., Cozzi, M. R., De Marco, L., Ruggeri, Z. M. Sequential cytoplasmic calcium signals in a 2-stage platelet activation process induced by the glycoprotein Ibalpha mechanoreceptor. Blood. 100, 2793-2800 (2002).
- Lefort, C. T., Ley, K. Neutrophil arrest by LFA-1 activation. Frontiers in immunology. 3, 157 (2012).
- Liu, B., Chen, W., Evavold, B. D., Zhu, C. Accumulation of dynamic catch bonds between TCR and agonist peptide-MHC triggers T cell signaling. Cell. 157, 357-368 (2014).
- Lou, J., et al. Flow-enhanced adhesion regulated by a selectin interdomain hinge. The Journal of cell biology. 174, 1107-1117 (2006).
- Fiore, V. F., Ju, L., Chen, Y., Zhu, C., Barker, T. H. Dynamic catch of a Thy-1-alpha5beta1+syndecan-4 trimolecular complex. Nature communications. 5, 4886 (2014).
- Chen, W., Zarnitsyna, V. I., Sarangapani, K. K., Huang, J., Zhu, C. Measuring Receptor-Ligand Binding Kinetics on Cell Surfaces: From Adhesion Frequency to Thermal Fluctuation Methods. Cellular and molecular bioengineering. 1, 276-288 (2008).
- Marshall, B. T., Sarangapani, K. K., Lou, J., McEver, R. P., Zhu, C. Force history dependence of receptor-ligand dissociation. Biophysical. 88, 1458-1466 (2005).
- Xiang, X., et al. Structural basis and kinetics of force-induced conformational changes of an alphaA domain-containing integrin. PloS one. 6, e27946 (2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon