Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Structure-fonction des études chez la souris à l'aide de cellules souches embryonnaires recombinase médiation cassette d'échange
Dans cet article
Résumé
Les protéines contiennent souvent plusieurs domaines qui peuvent exercer différentes fonctions cellulaires. knock-out gène (KO) ne considèrent pas cette diversité fonctionnelle. Nous rapportons ici un échange de cassette à médiation recombinaison (CREM) approche structure-fonction dans les cellules à base de souches embryonnaires KO qui permet la dissection moléculaire de différents domaines fonctionnels ou variants d'une protéine.
Résumé
génie génétique dans des embryons de souris ou de cellules souches embryonnaires (mESCs) permet l'étude de la fonction d'une protéine donnée. Les protéines sont les chevaux de trait de la cellule et sont souvent constitués de multiples domaines fonctionnels, qui peuvent être influencés par des modifications post-traductionnelles. L'épuisement de la protéine entière chez les souris knock-out conditionnel ou constitutive (KO) ne prend pas en compte cette diversité fonctionnelle et de la réglementation. Une ligne MESC et un modèle de souris dérivé, dans lequel un site d'amarrage pour l'échange de cassette de recombinaison à médiation par FLPE (CREM) a été insérée dans le locus ROSA26 (R26), a été précédemment rapporté. Nous présentons ici une approche structure-fonction qui permet la dissection moléculaire des différentes fonctionnalités d'une protéine multidomaine. A cet effet, les souris compatibles CREM doivent être croisées avec des souris KO et KO mESCs compatibles CREM doivent être isolés. Ensuite, un panel de constructions de sauvetage putatifs peut être introduit dans le locus R26 via CREM targeting. Les cDNA de sauvetage candidats peuvent être facilement insérés entre les sites CREM du vecteur de ciblage en utilisant le clonage de recombinaison. Ensuite, KO mESCs sont transfectées avec le vecteur de ciblage en combinaison avec un plasmide d'expression de recombinase FLPE. CREM le gène réactive résistance à la néomycine promoteur moins dans les sites d'accueil Rosa26 et permet la sélection de l'événement correct de ciblage. De cette manière, l'efficacité élevée de ciblage proches de 100% sont obtenus, ce qui permet l'insertion de plusieurs constructions de sauvetage putatifs d'une manière semi-débit élevé. Enfin, peut être testé une multitude de constructions de secours entraînées R26 pour leur capacité à sauver le phénotype qui a été observé dans mESCs KO parental. Nous présentons une étude de validation de principe structure-fonction dans p120 caténine (p120ctn) KO mESCs en utilisant la différenciation endoderme dans les corps embryoïdes (EbS) comme lecture phénotypiques. Cette approche permet d'identifier des domaines importants, en aval des voies putatives, et point pertinent maladiemutations qui sous-tendent KO pour une phénotypes protéine donnée.
Introduction
On estime que les génomes de mammifères contiennent environ 20 000 gènes codant pour des protéines. L'épissage alternatif et modifications post-traductionnelles augmenter encore le répertoire de protéines. Les protéines ont une structure modulaire 1 et contiennent souvent de multiples domaines d'interaction, qui permettent leur recrutement dans différents complexes de protéines et leur participation à de multiples processus cellulaires 2. Un exemple est la protéine multifonctionnelle appelée p120ctn. p120ctn est codée par le gène Ctnnd1 et se compose d'un grand domaine de répétition de tatou centrale flanquée par une extrémité N-terminale et une région C-terminale. Le domaine de tatou p120ctn se lie à un domaine juxtamembranaire hautement conservée de cadhérines classiques, qui sont impliqués dans l'adhésion cellulaire des cellules, mais il se lie également au répresseur de la transcription Kaiso. Le domaine N-terminal de p120ctn interagit avec différentes kinases, les phosphatases, les petites GTPases Rho et associée aux microtubules proteins 3. Il est intéressant, en raison de l' épissage alternatif, isoformes p120ctn peuvent être générés à partir de quatre codons de départ alternatifs 4. p120ctn isoforme 1A est la plus longue, elle est traduite à partir de la plus 5' codon de départ et contient la longueur complète segment N-terminal. Dans p120ctn isoformes 3 et 4, ce segment N-terminal est supprimé partiellement et complètement, respectivement. Comprendre le rôle précis des protéines (ou isoformes de protéines) et leurs domaines dans différentes fonctions cellulaires reste un défi.
Le ciblage de gènes dans mESCs permet l'étude de la fonction d'une protéine à travers la deletion génétique du gène correspondant et a largement contribué à l'identification des gènes et les voies de développement importants et importantes maladies. Cette percée en génétique inverse a été le résultat des progrès dans les domaines de l' isolement MESC et ciblage de gène en raison de la recombinaison homologue 5 . La recombinaison homologue est un procédé dans lequel des fragments d'ADN sont échangés entre deux fragments nucléiques similaires ou identiques après cassures de l'ADN (DS) double brin. En règle générale, HR est inefficace parce que les pauses dsDNA sont rares. Récemment, l'efficacité de gène dirigé vers une homologie de ciblage pourrait être augmentée en utilisant des nucléases spécifiques à un site 6, 7, mais , malheureusement, elles sont sujettes à des effets hors-cible 8. Une technique plus fiable pour permettre le ciblage de gène est CREM, qui est basée sur des systèmes de recombinaison spécifiques au site tels que Cre / loxP ou FLPE / FRT. LoxP et la séquence Frt se trouvent dans le bactériophage P1 et Saccharomyces cerevisiae, respectivement, et se composent de 34 pb, comprenant une séquence de 8 pb asymétrique qui détermine l'orientation du site. D'autre part, l'orientation, par exemple, deux sites loxP dans un tronçon d'ADN déterminera si l'ADN floxés devient excisé ou inversed lors d'une recombinaison à médiation par Cre 9. En outre, la Cre peut également induire une translocation si deux sites sont situés sur des chromosomes différents. CREM profite des sites de recombinaison hétérospécifiques qui ne sont pas provoquer une réaction croisée et qui sont incorporés dans un locus génomique. En présence d'un plasmide donneur qui contient un fragment d'ADN flanqué par les mêmes sites hétérospécifiques, la recombinase va insérer ce fragment d'ADN dans le locus génomique compatible CREM en raison de translocation à double simultanée (Figure 1). Ici, seuls correctement clones CREM ciblés peuvent rendre la résistance aux médicaments grâce à un promoteur sur le vecteur entrant qui restaure un « piégé », sans promoteur gène de résistance à la néomycine (Neo R) présente dans le génome R26 des cellules d'accueil (Figure 1) 10, 11. Il en résulte une efficacité de ciblage très élevé, souvent près de 100% 11, </ sup> 12. En conclusion, le ciblage par CREM-est très efficace et peut être utilisé pour les études de structure-fonctions; cependant, il a besoin d'un locus génomique précalculé.
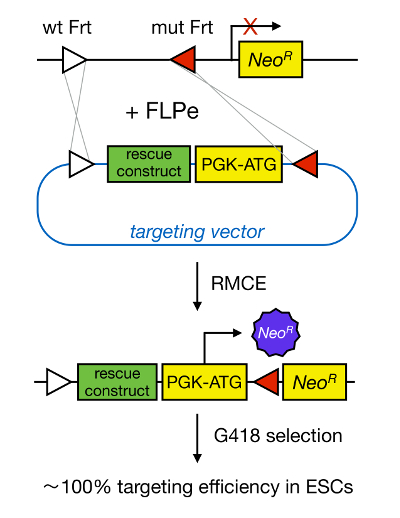
Figure 1. Représentation schématique de ciblage à médiation par CREM. CREM permet l'échange de segments d'ADN à partir d'un vecteur de ciblage entrant à un locus génomique définie si les deux hébergent deux sites hétérospécifiques Frt (représentés par des triangles blancs et rouges). En outre, le locus génomique conçu contient un promoteur et un gène de résistance à la néomycine tronquée (Neo R). En fournissant un promoteur et codon de départ dans le fragment d'ADN entrant, seuls les événements de recombinaison restauration correcte résistance à la néomycine, entraînant des rendements élevés de ciblage. S'il vous plaît cliquer ici pour voir une version plus grande de tsa figure.
L'ingénierie des génomes en mESCs permet la génération de souris compatible CREM. En 1981, deux groupes ont réussi à capturer des cellules pluripotentes à partir de la masse cellulaire interne (ICM) de blastocystes et à les maintenir en culture 13, 14. mESCs sont capables d'auto-renouvellement et de différenciation dans tous les types de cellules embryonnaires et adultes, y compris la lignée germinale. Par conséquent, le ciblage génique dans mESCs permet des études génétiques inverse par le développement de constitutifs ou conditionnels (en utilisant le système Cre / LoxP) souris KO. Cependant, la manière classique pour isoler des cellules ES de souris est très inefficace. Plusieurs améliorations importantes ont fortement augmenté le taux de succès pour dériver des lignes Mesc, y compris l'utilisation d'un sérum de remplacement défini (SR) moyen 15, en alternant entre milieu MESC contenant du sérum bovin SR et fœtal (FBS) 16, et l'utilisation de pharmacocomposés logiques tels que pluripotin ou 17 2i. Pluripotin, une petite molécule synthétique, permet la propagation de mESCs dans un état indifférencié en l'absence de facteur inhibiteur de leucémie (LIF) et les fibroblastes embryonnaires de souris (MEF) 18. Enfin, il a été démontré que mESCs peuvent être isolés avec un rendement très élevé (proche de 100%) lorsqu'une SR / FBS protocole moyen d'alternance est combiné avec du LIF et pluripotin 19, 20. Ces protocoles permettent l'isolement efficace de KO mESCs de CREM compatibles qui peuvent ensuite être utilisés pour des études structure-fonction.
Le présent document décrit une méthode que l'on permet d'identifier les domaines clés ou les résidus dans une protéine qui sont responsables des processus cellulaires spécifiques. À cette fin, un pipeline de technologies de pointe qui permettent l'isolement MESC efficace, le ciblage ensemble vecteur, et le ciblage MESC a été créeré. En tant que tel, de grands panneaux avec des isoformes de protéines, des mutants de domaine et effecteurs en aval peuvent être introduits dans KO mESCs et peuvent être évalués pour leur capacité à sauver le phénotype KO in vitro.
Protocole
Toutes les expériences sur des souris ont été menées conformément à la réglementation des animaux dans les institutions, nationales et européennes.
1. Isolement de mESCs KO CREM compatibles
- Breed souris hétérozygotes avec des souris KO compatible CREM, tels que des souris ROSALUC 10 ou souris ROSA26-COPSi 21. Les deux souris compatibles CREM ont été maintenues sur un mélange 129 / C57BL6 / fond suisse.
NOTE: Le croisement avec des souris hétérozygotes KO est conseillé de surmonter chez la souris mortalité embryonnaire homozygotes KO. - Utiliser une PCR pour sélectionner les souris hétérozygotes KO contenant une cassette d'CREM dans le locus 12 R26.
- Race souris hétérozygotes CREM compatible, KO avec des souris KO hétérozygotes et isoler CREM-compatible, blastocystes homozygotes KO.
- Mise en place de conjugaisons temps le soir et vérifier les fiches copulation le lendemain matin.
REMARQUE: Les bouchons sont faits de sécrétions coagulées des glandes coagulantes et vésiculaires du mâle.Ces bouchons remplissent le vagin de la femelle et persistent pendant 8 - 24 h après la reproduction. les femmes sont enfichés considérées comme portant 0,5 embryons dpc (jours de post coïtum).- Pour vérifier les bouchons, soulevez la femelle par la base de sa queue et en examiner son ouverture vaginale pour une masse blanchâtre. Répartir les lèvres de la vulve légèrement avec une sonde coudée lorsque le bouchon est difficile à voir. femelles bouchés séparés de leur mâle.
- Recueillir blastocystes à 3,5 dpc.
- Euthanasier femelles gravides par la méthode approuvée (par exemple, la dislocation cervicale). Faire une incision médioventral et disséquer l'utérus et oviducte (encore attaché à l'autre) en utilisant des ciseaux et des pinces fines.
- Plier une aiguille de calibre 26 dans un angle de 45 °. Attacher une seringue de 1 ml rempli avec du milieu M2 pour cette aiguille courbée et l'utiliser pour rincer les blastocystes de l'utérus dans le couvercle d'une boîte de 10 cm.
- Insérer l'aiguille dans l'extrémité de l'utérus qui est le plus proche del'oviducte. Maintenir l'aiguille en place avec des pinces fines tout en poussant le piston plongeur; le gonflement de l'utérus indique un rinçage avec succès.
- Utiliser une pipette de bouche (d'un diamètre de 100 - 200 um) pour recueillir tous les embryons et les laver deux fois dans une goutte de milieu frais M2. Immédiatement après les laver, transférer les blastocystes aux plaques de culture (voir ci-dessous).
NOTE: La dissection et la manipulation des blastocystes de doit être fait dans le flux d'air laminaire.
- Mise en place de conjugaisons temps le soir et vérifier les fiches copulation le lendemain matin.
- Isoler mESCs CREM compatible KO
- Préparer une plaque de 12 puits avec de la mitomycine-C-traités DR4 MEF (voir le tableau des matériaux) un jour avant l'isolement du blastocyste.
REMARQUE: Ces MEF ont été isolés à partir de Tg (DR4) 1Jae / souris J qui contiennent quatre gènes de médicaments sélectionnable et confèrent une résistance à la néomycine, la puromycine, l' hygromycine, et 6-thioguanine 22.- Manteau de toutes les plaques de culture avec 0,1% de gélatine. Ajouter 0,1% de gélatineles plaques de culture, incuber pendant 5 minutes à 37 ° C dans 5% de CO 2, et aspirer la solution de gélatine. Graine d' un quart d'un flacon de P2 MEFs dans une plaque à 12 puits et les cultiver dans 2 ml de milieu MEF (voir le tableau 1, la Table des matières) pour une monocouche confluente 19.
- Les inactiver avec de la mitomycine-C (10 ug / ml) pendant 3 h et les laver deux fois avec une solution saline tamponnée au phosphate (PBS) 19.
- En utilisant une pipette à la bouche, la plaque blastocystes sur des plaques gélatinisés 12 puits (1 puits / embryon), avec les MEFs traités mitomycine-C dans un milieu de cellules SR-ES (2 mL / puits) complété avec soit 2 uM pluripotin ou avec 2i (1 uM inhibiteur de Erk PD0325901 et 3 uM inhibiteur de GSK3 CHIR99021). Incuber à 37 ° C dans 5% de CO 2.
- Actualisez le milieu cellulaire SR-ES (complété par pluripotin ou 2i) tous les 2 - 3 jours.
- Examinez chaque blastocyste sous un stereomicroportée à un grossissement de 4,0X et vérifier l'éclosion et l'attachement à la couche MEF.
REMARQUE: Lorsque la trappe de blastocystes, ils perdent la zone pellucide qui les encapsule. Wells avec blastocystes doivent être vivant seules rafraîchi en utilisant la bouche pipetage. - Choisissez des excroissances individuels ICM (en utilisant un stéréomicroscope) après 10 - 12 jours de culture à l'aide d'une pipette P10 avec embouts jetables. Transférer l'excroissance dans environ 10 ul de milieu à une forme de V, plaque à 96 puits contenant 30 pl / puits de PBS (à température ambiante).
- Ajouter 50 ul de trypsine à 0,25% à chaque puits en utilisant une pipette multicanaux et incuber pendant 3 minutes à 37 ° C dans 5% de CO 2.
- Ajouter 100 pi de FBS milieu contenant MESC; dissocier l'ICM excroissances dans des cellules individuelles par pipetage 10-15 fois; et transférer les cellules dissociées à la mitomycine-C-traitée, plaques de 96 puits MEF qui ont été préparées un jour avant les colonies sont prélevées de l'ICM.
- A partir de cette étape en avant, omi t pluripotin ou 2i du milieu MESC. Le lendemain, changer le moyen de FBS- à SR milieu contenant MESC (100 pl / puits).
- Développez les lignes Mesc établies à partir 96- au format 24 puits 19.
- Laver les cellules avec 200 ul de PBS, ajouter 50 ul de trypsine, et incuber pendant 5 minutes à 37 ° C dans 5% de CO Ajouter 100 ul de milieu à base de MESC FBS. dissocier en pipetant 10 - 15 fois à l'aide d'une pipette multicanaux; et transférer les cellules dissociées à la mitomycine-C-traitée, à 24 puits des plaques MEF.
- Changer de milieu à base de SR-le lendemain. Développez les mESCs de façon similaire au format de 24- 6 puits. Faire 3 - 4 congélations à partir d' un confluentes plaque à 6 puits 19.
- Identifier CREM compatible, mESCs homozygotes KO en utilisant des amorces de PCR pour le locus R26 23 et allèle KO de choix (dans ce cas, p120ctn, pour p120ctn allèles PCRs nuls et floxés ont été décrits avantlass = "xref"> 12).
- Préparer une plaque de 12 puits avec de la mitomycine-C-traités DR4 MEF (voir le tableau des matériaux) un jour avant l'isolement du blastocyste.

Tableau 1. Milieux de culture. Tous les milieux ont été stockés à 4 ° C et réchauffé à 37 ° C 30 min avant utilisation.
2. Génération d'un vecteur de ciblage compatible CREM L'utilisation Recombinaison clonage
- Cloner le sauvetage construit dans des vecteurs compatibles recombinaison à l' aide de l' enzyme de restriction (RE) à base de ou à base de PCR 24 des techniques de clonage. Assurez-vous que les contiennent un codon ADNc d'arrêt.
- Conception des amorces marquées AttB 24. Assurez-vous que l'amorce avant contient les éléments suivants: un tronçon de GGGG, un site de AttB1, un segment de liaison, une séquence Kozak, et environ 25 nucleotides de l'ADNc de sauvetage (en commençant par son ATG). Assurez-vous que l'amorce inverse a une composition similaire: un tronçon de GGGG, un site attB2, un éditeur de liens, et environ 25 NUCleotides d'ADNc de secours (complément inverse).
- Amplifier l'ADNc de sauvetage par PCR pour obtenir des ADNc flanqué AttB.
- Effectuer une réaction de 10 uL BP avec 100 ng d'ADNc flanqué AttB et 150 ng de vecteur donneur compatible recombinaison, qui contient un gène de résistance à la kanamycine.
- Transformer 5 ul du mélange de BP dans Escherichia MC1061-chocs compétente de chaleur coli bactéries (E. coli) (similaires à celles décrites à l' étape 2.3).
- Identifier les colonies contenant les vecteurs de sauvetage corrects contenant de l'ADNc (similaires à celles décrites à l'étape 2.4)
- Effectuer une réaction LR de 10 ul en utilisant 100 ng de sauvetage vecteur contenant l'ADNc; 150 ng de vecteur excisé par Cre-pRMCE-DV1 11 (LMBP 8195); et 2 pl de mélange de recombinase, qui contient une intégrase phagique codée et excisionase et un facteur hôte d'intégration bactérienne. Incuber pendant 2 heures à 25 ° C.
NOTE: Une réaction LR est une recombinaison réagirions dans laquelle un clone d'entrée contenant des sites attL et un vecteur de destination contenant des sites attR sont recombinées par le mélange d'enzymes LR clonase. Il en résulte un clone d'expression contenant des sites attB flanquant le gène d'intérêt. - Transformer 5 ul des mélanges de LR dans MC1061 de E. coli bactéries-chocs compétente de chaleur.
- Ajouter 5 ul de mélanges LR à un nervurée, jupe, tube à bouchon à vis de 2 ml avec 40 ul de bactéries compétentes E.-choc thermique coli et incuber pendant 20 min sur de la glace. Incuber pendant 5 min à 37 ° C.
- Ajouter 1 ml de milieu de bouillon de Luria (LB) et incuber pendant 1 h à 37 ° C. Plaque de 50 ul sur ampicilline (Amp; 100 pg / ml) contenant des plaques de gélose et faire croître pendant une nuit à 37 ° C.
- Identifier les colonies avec le vecteur de ciblage correct.
- Choisissez 5 colonies au hasard en utilisant une pointe de P200. Transférer la pointe à un tube à essai en verre contenant du 2 - 5 ml de milieu LB et faire croître pendant une nuit à 37 ° C.
- Extracter l'ADN plasmidique à partir des cultures bactériennes en utilisant des kits disponibles dans le commerce.
- Valider les hachages RE et le séquençage. Couper 0,5 à 2 pg de plasmide en utilisant 0,2 ul de RE (20 U / pl) et 1 pi de tampon 10x correspondant dans une réaction de 10 uL. Incuber pendant au moins 1 h à 37 ° C et séparé sur un gel d'agarose à 1%. Sélectionnez les colonies avec le modèle prédit de fragments d'ADN.
- Analyser les vecteurs confirmés (50 ng / ul) avec Tlox amorces F (ATG ATC TCT GGA TCC CCA TC) et R IRES (GGG GCG GAA TTC GAT ATC AAG) (5 pmol / pl) en utilisant le séquençage de Sanger.
3. MESC de CREM à médiation par ciblage de sauvetage Construit au R26 Locus
- Lancer une culture de mESCs KO CREM compatible et passage les au moins deux fois dans un milieu MESC FAE à base de FBS. Diviser les mESCs sur une plaque gélatinisée 6 puits.
- Le lendemain, rafraîchir les cellules, à environ 50% de confluence, avec 1,5ml de milieu à base de MESC FBS et transfecter les mESCs avec un pRMCE-DV1 vecteur de ciblage Cre-excisé contenant de l'ADNc de sauvetage.
- Faites un mélange d'ADN. Ajouter 1 pg du vecteur de ciblage et 1 pg du plasmide d'expression FLPE 25 à 250 ul de milieu DMEM pur.
- Faites un mélange de lipofection. Ajouter 7 ul de réactif de transfection à base de lipofection (par exemple, Lipofectamine 2000) à 250 ul de milieu DMEM pur et incuber pendant 5 minutes à la température ambiante (RT).
NOTE: CREM similaires ciblant l' efficacité ont été obtenues à l' aide d' autres réactifs à base lipofection (par exemple, Lipofectamine LTX et Effectene). - Mélanger le mélange d'ADN avec le mélange de lipofection et incuber pendant 20 min à température ambiante. Pipeter le mélange de transfection sur les mESCs rafraîchie. Tourbillonner doucement et laisser une nuit.
- Un jour après la transfection, diviser tous les mESCs de la plaque à 6 puits à une boîte de culture de 10 cm avec une couche confluente de MEFs DR4 et 10 ml de FBS à base de mmoyen ESC.
- Deux jours après la transfection, sélectionner des clones Mesc avec l'échange de cassette à médiation FLPE correcte par addition de 0,2 mg / mL de G418 (100x) dans le milieu.
REMARQUE: Faire une courbe kill pour chaque lot de G418 pour identifier la plus faible concentration de G418 qui tue tous les mESCs. - Actualiser les mESCs quotidien avec milieu contenant G418 MESC. Les colonies doivent apparaître au bout de 7 - 10 jours, alors choisissez et élargir ceux-ci comme à l'étape 1.4.
- Vérifiez les clones corrects par PCR 11.
4. Différenciation des mESCs dans les corps embryoïdes (EbS)
- Lancer une culture de KO mESCs avec des constructions de secours entraînées R26 et les passage au moins deux fois dans un milieu MESC FAE à base de FBS. Passage des mESCs une fois sur des plaques 6 puits gélifiés pour se débarrasser des FAE.
- Laver avec du PBS et trypsiniser les cultures Mesc presque confluentes. Plate dissociées mESCs dans différentes dilutions (1/20 et 1/40) sur la non-adhérent, animal de compagnie de qualité bactérienneri-plats en milieu de différenciation.
- Laisser se former dans EBs ces plats pendant 30 jours. Actualiser le moyen tous les 2 - 3 jours selon la procédure suivante: transférer la suspension EB à un tube de 50 ml, laisser les EBs régler par gravité, éliminer le surnageant, ajouter du milieu frais, et transférer la suspension EB retour à une qualité bactérienne plat.
- Analyser les mESCs ciblées et EBS par microscopie à immunofluorescence et électronique à transmission en utilisant des protocoles qui ont été décrits précédemment 12.
Résultats
La procédure d'isoler les lignes compatibles CREM KO Mesc est représenté sur la figure 2. Deux consécutifs sont nécessaires Élevages pour obtenir des blastocystes KO compatible CREM. Tout d'abord, les souris KO hétérozygotes sont croisées avec des souris compatible CREM pour obtenir, souris hétérozygotes KO compatible CREM. Ces souris sont ensuite utilisées pour chronométrés avec d'autres conjugaisons souris KO hétérozygotes pour obtenir 3,5 d...
Discussion
Notre méthode d'isolement MESC est convivial et ne nécessite pas de compétences avancées ou de l'équipement, comme la microchirurgie de blastocystes. Ainsi, cette technologie est accessible à une grande partie de la communauté scientifique. Toute personne ayant une expérience de culture cellulaire de base peut se propager ICM excroissances et établir des lignes de mESCs. Cependant, le rinçage et la manipulation des blastocystes nécessite une certaine pratique. Une pipette de bouche est utilisé pour t...
Déclarations de divulgation
Les auteurs n'ont rien à dévoiler.
Remerciements
Nous remercions Jinke D'Hont, Frederique Van Rockeghem, Natalie Farla, Kelly Lemeire et Riet De Rycke pour leur excellent support technique. Nous remercions également Eef Parthoens, Evelien Van Hamme et Amanda Goncalves du Core Facility Bioimaging du Centre de recherche Inflammation pour leur aide d'experts. Nous reconnaissons les membres de notre groupe de recherche pour des discussions utiles. Ce travail a été soutenu par la Politique scientifique (Belspo Pôles d'attraction interuniversitaires - Prix IAP VII-07 DevRepair; https://devrepair.be), par la Fondation Médicale Reine Elisabeth, Belgique (GSKE 2008-2010; http: // www .fmre-gske.be), et par les actions de recherche concertée (GOA) 01G01908 de l'Université de Gand, Belgique (http://www.ugent.be/en/ghentuniv). SG est un stagiaire post-doctoral du Fonds de recherche Flandre (FWO-V).
matériels
| Name | Company | Catalog Number | Comments |
| ROSALUC Mice | made in house | frozen sperm available upon request | |
| R26-iPSC mice | made in house | frozen sperm available upon request | |
| Vessel probe | Fine Science Tools | 10160-13 | to check for copulation plugs |
| M2 medium | Sigma-Aldrich | M7167 | make aliquots and store at -20 °C |
| Fine forceps (Dumont #5 Standard tip Student forceps) | Fine Science Tools | 11251-10 | spray with 70% EtOH before use (do not autoclave) |
| 23 G needles | Fine-ject | 8697 | |
| 1-mL syringes | Soft-ject | 6680 | |
| 60-mm bacterial grade plates (for flushing) | Gosselin | BB60-01 | |
| Mouth pipette | made in house | see discussion | |
| Mouse embryonic fibroblasts (MEFs, TgN (DR4)1 Jae strain) | ATTC | SCRC-1045 | |
| TgN (DR4)1 Jae mice | The Jackson Laboratory | 3208 | |
| Mitomycin C | Sigma-Aldrich | M0503 | |
| Phosphate buffered saline (PBS) without calcium or magnesium | Gibco | 14190-094 | |
| Tg(DR4)1Jae/J mice | JAX | 3208 | mice that contain four drug-selectable genes and DR4 MEFS confers resistance to neomycin, puromycin, hygromycin and 6-thioguanine |
| 0.1% Gelatin | Sigma-Aldrich | G1393 | Dissolve 0.5 g in 500 mL distilled water, autoclave and store at 4 °C. |
| Trypsin (0.25%) | Gibco | 25200-056 | |
| 2 μM pluripotin | Cayman Chemical | 10009557 | |
| pRMCE-DV1 | BCCM/LMBP collection | LMBP 08870 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| cre-excised pRMCE-DV1 | BCCM/LMBP collection | LMBP 08195 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| pCAG-FlpE-IRES-Puro-pA | Addgene | 20733 | |
| heat-shock competent DH5α bacteria | made in house | ||
| Gateway pDONR221 vector | Thermo Fisher | 12536-017 | contains kanamycin-resistance gene |
| BP clonase II mix | Thermo Fisher | 11789-020 | |
| LR clonase II mix | Thermo Fisher | 11791-020 | |
| Luria Broth (LB) | |||
| Ampicillin | |||
| Applied Biosystems 3730XL DNA Analyzer | Thermo Fisher | 3730XL | |
| G418 | Thermo Fisher | 11811-023 | |
| Lipofectamine 2000 transfection reagent | Thermo Fisher | 11668027 | |
| Lipofectamine LTX transfection reagent | Thermo Fisher | 15338100 | |
| Effectene transfection reagent | Qiagen | 301425 | |
| GATEWAY pENTR 1A vector | Thermo Fisher | A10462 | recombination-compatible vector |
| mouse monoclonal anti-p120ctn antibody | BD Transduction Laboratories | 610134 | |
| mouse monoclonal anti-Ecadherin antibody | BD Transduction Laboratories | 610181 | |
| General equipment | |||
| Sterile dissection tools | fine scissors and forceps for dissecting the uterus | ||
| Sterile pipettes: 5 mL, 10 mL and 25 mL | |||
| 15-mL and 50-mL conical centrifuge tubes | |||
| 96-well culture plates V-shaped bottom and flat bottom) | |||
| Culture dishes: 24 wells, 12 wells and 6 wells | |||
| Multichannel pipettes (to pipette 30, 50, 100 and 200 μL) | |||
| Sterile multichannel reservoirs | |||
| Access to a laminar air flow | |||
| Access to an incubator at 37 °C with 5% CO2 | |||
| Access to an inverted microscope | |||
| Access to a bench-top centrifuge | |||
| Access to a stereo microscope with transmitted-light | |||
| Culture media | |||
| MEF Medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| Dulbecco’s modified Eagle’s medium (DMEM) | Gibco | 41965-062 | |
| 10% fetal bovine serum (FBS) | Sigma-Aldrich | F-7524 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| Sodium pyruvate (0.4 mM) | Gibco | 11360-039 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| SR-based mESC medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| DMEM/F12 | Gibco | 31330-038 | mixed in a 1:1 ratio |
| 15% knock-out serum replacement (SR) | Gibco | 10828–028 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| 0.1 mM non-essential amino acids | Gibco | 11140-050 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| β-mercaptoethanol (0.1 mM) | Sigma-Aldrich | M 3148 | |
| 2,000 U/mL recombinant mouse LIF | (IRC/VIB Protein Service facility) | ||
| FBS-based mESC medium (similar to SR-based mESC medium) | stored at 4°C; warm 30 min at 37°C before use | ||
| Knockout DMEM | Gibco | 10829-018 | |
| 15% FBS | Hyclone | SH30070.03E | |
| Differention Medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| Iscove's Modified Dulbecco's Medium (IMDM) | Gibco | 21980-032 | |
| 15% FBS | Hyclone | SH30070.03E | |
| 5% CD Hybridoma Medium(1x) liquid | Gibco | 11279-023 | |
| 2 mM L-glutamine | Gibco | 25030-024 | |
| 0.4 mM 1-thioglycerol | Sigma-Aldrich | M-6145 | |
| 50 μg/mL ascorbic acid | Sigma-Aldrich | A-4544 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| 2i | |||
| 1 μM Erk inhibitor PD0325901 | Axon Medchem | Axon 1408 | |
| 3 μM Gsk3 inhibitor CHIR99021 | Axon Medchem | Axon 1386 |
Références
- Gul, I. S., Hulpiau, P., Saeys, Y., van Roy, F. Metazoan evolution of the armadillo repeat superfamily. Cell Mol Life Sci. , (2016).
- Valenta, T., Hausmann, G., Basler, K. The many faces and functions of beta-catenin. EMBO J. 31, 2714-2736 (2012).
- Pieters, T., van Hengel, J., van Roy, F. Functions of p120ctn in development and disease. Front Biosci. 17, 760-783 (2012).
- Keirsebilck, A., et al. Molecular cloning of the human p120ctn catenin gene (CTNND1): Expression of multiple alternatively spliced isoforms. Genomics. 50, 129-146 (1998).
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6, 507-512 (2005).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Yang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 154, 1370-1379 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31, 822-826 (2013).
- Branda, C. S., Dymecki, S. M. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell. 6, 7-28 (2004).
- Sandhu, U., et al. Strict control of transgene expression in a mouse model for sensitive biological applications based on RMCE compatible ES cells. Nucleic Acids Res. 39, 1 (2010).
- Haenebalcke, L., et al. Efficient ROSA26-based conditional and/or inducible transgenesis using RMCE-compatible F1 hybrid mouse embryonic stem cells. Stem Cell Rev. 9, 774-785 (2013).
- Pieters, T., et al. p120 Catenin-Mediated Stabilization of E-Cadherin Is Essential for Primitive Endoderm Specification. PLoS Genet. 12, e1006243 (2016).
- Evans, M. J., Kaufman, M. H. Establishment in culture of pluripotential cells from mouse embryos. Nature. 292, 154-156 (1981).
- Martin, G. R. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells. Proc Natl Acad Sci U S A. 78, 7634-7638 (1981).
- Cheng, J., Dutra, A., Takesono, A., Garrett-Beal, L., Schwartzberg, P. L. Improved generation of C57BL/6J mouse embryonic stem cells in a defined serum-free media. Genesis. 39, 100-104 (2004).
- Bryja, V., et al. An efficient method for the derivation of mouse embryonic stem cells. Stem Cells. 24, 844-849 (2006).
- Ying, Q. L., et al. The ground state of embryonic stem cell self-renewal. Nature. 453, 519-523 (2008).
- Chen, S., et al. Self-renewal of embryonic stem cells by a small molecule. Proc Natl Acad Sci U S A. 103, 17266-17271 (2006).
- Pieters, T., et al. Efficient and User-Friendly Pluripotin-based Derivation of Mouse Embryonic Stem Cells. Stem Cell Rev. 8, (2012).
- Yang, W., et al. Pluripotin combined with leukemia inhibitory factor greatly promotes the derivation of embryonic stem cell lines from refractory strains. Stem Cells. 27, 383-389 (2009).
- Haenebalcke, L., et al. The ROSA26-iPSC Mouse: A Conditional, Inducible, and Exchangeable Resource for Studying Cellular (De)Differentiation. Cell Rep. 3, (2013).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25, 3745-3746 (1997).
- Nyabi, O., et al. Efficient mouse transgenesis using Gateway-compatible ROSA26 locus targeting vectors and F1 hybrid ES cells. Nucleic Acids Res. 37, 55 (2009).
- Katzen, F. Gateway((R)) recombinational cloning: a biological operating system. Expert Opin Drug Discov. 2, 571-589 (2007).
- Schaft, J., Ashery-Padan, R., vander Hoeven, F., Gruss, P., Stewart, A. F. Efficient FLP recombination in mouse ES cells and oocytes. Genesis. 31, 6-10 (2001).
- Spencer, H. L., et al. E-cadherin inhibits cell surface localization of the pro-migratory 5T4 oncofetal antigen in mouse embryonic stem cells. Mol Biol Cell. 18, 2838-2851 (2007).
- Stryjewska, A., et al. Zeb2 Regulates Cell Fate at the Exit from Epiblast State in Mouse Embryonic Stem Cells. Stem Cells. , (2016).
- Nagy, A., Gertsenstein, M., Vintersten, K., Behringer, R. . Manipulating the mouse embryo: A laboratory manual. , (2003).
- van den Berghe, V., et al. Directed migration of cortical interneurons depends on the cell-autonomous action of Sip1. Neuron. 77, 70-82 (2013).
- Li, J., et al. The EMT transcription factor Zeb2 controls adult murine hematopoietic differentiation by regulating cytokine signaling. Blood. , (2016).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon