Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Struktur-Funktions-Studien in embryonalen Maus-Stammzellen Rekombinase-vermittelten Kassettenaustausch
In diesem Artikel
Zusammenfassung
Proteine enthalten oft mehrere Domänen, die verschiedene zelluläre Funktionen ausüben kann. Gen-Knock-outs (KO) nicht über diese funktionelle Vielfalt berücksichtigen. Hier berichten wir über eine Rekombination vermittelten Kassettenaustausch (RMCE) -basierte Struktur-Funktions-Ansatz in KO embryonalen Stammzellen, die für die molekulare Präparation von verschiedenen funktionellen Domänen oder Varianten eines Proteins ermöglicht.
Zusammenfassung
Gentechnik in Maus-Embryonen oder embryonale Stammzellen (mESCs) ermöglicht die Untersuchung der Funktion eines bestimmten Proteins. Proteine sind die Arbeitspferde der Zelle und bestehen oft aus mehreren funktionellen Domänen, die durch posttranslationale Modifikationen beeinflusst werden kann. Die Verarmung des gesamten Proteins in bedingten oder konstitutiv Knock-out (KO) Mäusen nimmt nicht berücksichtigt diese funktionale Vielfalt und Regulierung. Eine MESC Linie und ein abgeleitetes Mausmodell, in dem eine Andockstelle für FLPe Rekombination vermittelten Kassettenaustausch (RMCE) innerhalb des ROSA26 (R26) Locus eingefügt wurde, wurde bereits berichtet. Hier berichten wir über eine Struktur-Funktions-Ansatz, der für die molekulare Zerlegung der verschiedenen Funktionalitäten eines Multi-Domain Protein ermöglicht. Zu diesem Zweck RMCE-kompatible Mäuse müssen mit KO-Mäusen gekreuzt werden und dann RMCE-kompatibel KO mESCs isoliert werden müssen. Als nächstes kann ein Panel von putativen rescue-Konstrukten in die R26-Locus über RMCE targeti eingeführt werdenng. Die Kandidaten rescue cDNAs kann leicht zwischen RMCE Seiten des Targeting-Vektors unter Verwendung von Rekombinations-Klonierung eingefügt werden. Als nächstes wird KO mESCs mit dem Targeting-Vektor in Kombination transfiziert mit einer FLPe Rekombinase Expressionsplasmid. RMCE reaktiviert die promotorlose Neomycin-Resistenz-Gene in den ROSA26 Andockstellen und ermöglicht die Auswahl des korrekten Targeting-Ereignisses. Auf diese Weise werden hohe Wirkungsgrad Targeting von nahezu 100% erzielt, so dass zum Einsetzen mehrerer putativen rescue Konstrukte in eine halb-hohen Durchsatz Weise. Schließlich kann eine Vielzahl von R26 getriebenen Rettungs Konstrukte auf ihre Fähigkeit getestet werden, um den Phänotyp zu retten, die in Eltern KO mESCs beobachtet wurde. Wir präsentieren eine Proof-of-Principle-Struktur-Wirkungs-Studie in p120 Catenin (p120ctn) KO mESCs mit endoderm Differenzierung in Embryoidkörpern (EVG) als phänotypische Anzeige. Dieser Ansatz ermöglicht die Identifizierung von wichtigen Domänen, putative nachgeschaltete Wege und krankheitsrelevanten PunktMutationen, die KO-Phänotypen für ein bestimmtes Protein zu Grunde liegt.
Einleitung
Es wird geschätzt, dass etwa 20.000 Säugergenomen Protein-kodierenden Gene enthalten. Alternatives Spleißen und posttranslationale Modifikationen erhöhen die Proteinrepertoires. Proteine haben einen modularen Aufbau 1 und häufig mehrere Wechselwirkungsdomänen enthalten, die ihre Einstellung 2 in verschiedene Proteinkomplexe und ihre Beteiligung an mehreren zellulären Prozesse ermöglichen. Ein Beispiel dafür ist das multifunktionale Protein namens p120ctn. p120ctn wird durch das CTNND1 Gen codiert und besteht aus einer großen zentralen Domäne Armadillo - Repeat flankiert von einem N-terminalen und C-terminalen Region. Die Armadillo-Domäne von p120ctn bindet an eine hoch konservierten Juxtamembrandomäne der klassischen Cadherine, die in Zell-Zell-Adhäsion beteiligt sind, aber es bindet auch an den Repressor Kaiso. Die N-terminale Domäne von p120ctn interagiert mit verschiedenen Kinasen, Phosphatasen, kleinen RhoGTPasen und Mikrotubuli-assoziierte proteins 3. Interessanterweise als Ergebnis des alternativen Spleißens, p120ctn Isoformen können 4 aus vier alternativen Startcodons erzeugt werden. p120ctn Isoform 1A ist die längste, wie es aus dem am meisten 5' translatiert wird Startcodon und enthält die Volllängen-N-terminales Segment. In p120ctn Isoformen 3 und 4 ist dieses N-terminale Segment teilweise gelöscht und vollständig sind. die genaue Rolle von Proteinen (oder Protein-Isoformen) und die Domänen in verschiedenen zellulären Funktionen zu verstehen, bleibt eine Herausforderung.
Gen-Targeting in mESCs ermöglicht die Untersuchung der Funktion eines Proteins durch die genetische Deletion des entsprechenden Gens und hat weitgehend dazu beigetragen, die Identifizierung von entwicklungs wichtig und krankheitsrelevanten Genen und Wegen. Dieser Durchbruch in der reversen Genetik war das Ergebnis der Fortschritte in den Bereichen der MESC Isolierung und Gene aufgrund homologer Rekombination Targeting 5 . Homologe Rekombination ist ein Verfahren, bei der DNA-Fragmenten zwischen zwei ähnlichen oder identischen Nukleins Einheiten nach doppelsträngige (ds) DNA-Brüchen ausgetauscht werden. Normalerweise ist HR ineffizient, weil dsDNA Pausen selten sind. Seit kurzem wird die Effizienz der Homologie-directed gene targeting könnte unter Verwendung von ortsspezifischen Nucleasen erhöht 6, 7, aber unglücklicherweise sind diese anfällig für Nebeneffekte 8. Eine zuverlässigere Technik zu ermöglichen, Gen-Targeting ist RMCE, die auf ortsspezifische Rekombinationssysteme, wie Cre / loxP oder FLPe / FRT-basiert. LoxP und Frt - Sequenz werden in Bakteriophage P1 und Saccharomyces cerevisiae, bzw., und aus 34 bp, einschließlich einer asymmetrischen 8 - bp - Sequenz gefunden, die die Orientierung der Website festlegt. Auf der anderen Seite ist die Orientierung von, zum Beispiel, zwei loxP-Stellen innerhalb einer DNA-Strecke werden bestimmen, ob die floxed DNA exzidiert wird oder i9 nversed nach Cre-vermittelter Rekombination. Darüber hinaus kann Cre induziert auch eine Translokation, wenn zwei Standorte auf verschiedene Chromosomen befinden. RMCE nutzt artfremden Rekombinationsstellen, die nicht kreuzreagieren und das in einer genomischen Locus eingebettet sind. In Anwesenheit eines Donator - Plasmid , das ein DNA - Fragment mit den gleichen hetero Stellen flankierte enthält, fügt die Rekombinase dieses DNA - Fragment in den kompatibelen RMCE genomischen Locus durch doppelte versetzte Translokation (Abbildung 1). Hier wird nur richtig RMCE gezielte Klone können drug resistance dank eines Promotors auf dem ankommenden Vektor rendern , die ein „gefangen“ , stellt promotorlose Neomycinresistenzgen (Neo R) in der R26 Genom der Docking - Zellen (Abbildung 1) 10, 11. Daraus ergibt sich eine sehr hohe Targeting Effizienz, oft nahe 100% 11 </ sup> 12. Abschließend ist RMCE basierte Targeting sehr effizient und kann für Struktur-Funktionen Studien verwendet werden; Jedoch erfordert es eine vorgefertigte genomischen Locus.
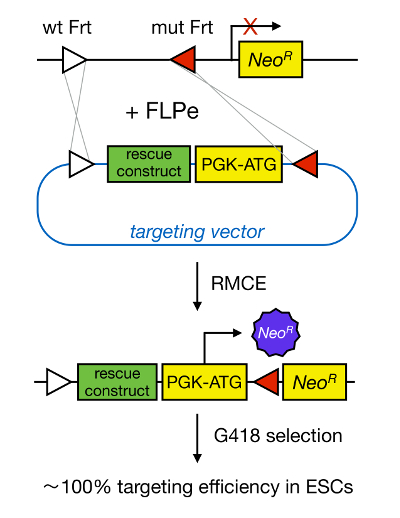
Abbildung 1 : Schematische Darstellung der RMCE Vermittelte Targeting. RMCE ermöglicht den Austausch von DNA-Segmenten aus einem ankommenden Targeting-Vektor zu einem definierten genomischen Locus, wenn sowohl zwei hetero FRT-Stellen Harbor (dargestellt durch weiße und rote Dreiecke). Darüber hinaus enthält das manipulierte genomischen Locus und ein promotorloses Neomycin-Resistenz verkürztes (Neo R) -Gen. Durch die Bereitstellung von Wiederherstellung einen Promotor und Startcodon in dem ankommenden DNA-Fragmente, nur korrekte Rekombinationsereignisse Neomycin-Resistenz, was zu hohen Effizienzen Targeting. Bitte klicken Sie hier , um eine größere Version t anzuzeigenseine Figur.
Genomtechnik in mESCs ermöglicht die Erzeugung von RMCE-kompatible Mäuse. Im Jahr 1981 gelang es zwei Gruppen von pluripotenten Zellen der inneren Zellmasse (ICM) von Blastozysten in Erfassung und in Kultur 13, 14 aufrechtzuerhalten. mESCs der Lage ist, die Selbsterneuerung und Differenzierung in alle Arten von embryonalen und adulten Zellen, einschließlich der Keimzelllinie. Daher Gene in mESCs Targeting ermöglichen Reverse genetische Studien über die Entwicklung von konstitutiven oder bedingten KO-Mäusen (die Cre / loxP-System verwendet wird). Allerdings ist die klassische Art und Weise Maus-ES-Zellen zu isolieren, ist sehr ineffizient. Verschiedene Verbesserungen haben große stark erhöht die Erfolgsrate für die Ableitung MESC Linien, einschließlich der Verwendung eines definierten serum-Ersatz (SR) Medium 15, im Wechsel zwischen MESC Medium SR und fötales Rinderserum (FBS) , 16, und die Verwendung von pharmakodynamischen enthaltendlogische Verbindungen wie pluripotin oder 2i 17. Pluripotin, ein kleines synthetisches Molekül, ermöglicht die Ausbreitung von mESCs in einem undifferenzierten Zustand in Abwesenheit von Leukämie - Hemmfaktor (LIF) und embryonalen Mausfibroblasten (MEFs) 18. Schließlich hat es sich gezeigt , dass mESCs kann mit einem sehr hohen Wirkungsgrad ( in der Nähe von 100%) isoliert werden , wenn ein SR / FBS - Medium Wechsel - Protokoll mit LIF kombiniert und pluripotin 19, 20. Diese Protokolle ermöglichen die effiziente Isolierung von RMCE-kompatibel KO mESCs, die anschließend für Struktur-Funktions-Studien verwendet werden können.
Dieses Dokument beschreibt ein Verfahren, das man die Schlüssel-Domänen oder Reste innerhalb eines Proteins zu identifizieren, ermöglicht es, die für spezifische zelluläre Prozesse verantwortlich sind. Zu diesem Zweck wird eine Pipeline von fortschrittlichen Technologien, die eine effiziente MESC Isolierung ermöglichen, Targeting-Vektor Montage und MESC Targeting war erstellend. Als solche großen Platten mit Proteinisoformen, Domain - Mutanten und Effektoren können in KO mESCs eingeführt werden können und für ihre Fähigkeit , die in - vitro - KO - Phänotyp zu retten ausgewertet werden.
Access restricted. Please log in or start a trial to view this content.
Protokoll
Alle Versuche an Mäusen wurden nach institutionellen, nationalen und europäischen Tiere Vorschriften durchgeführt.
1. Isolierung von RMCE-kompatibel KO mESCs
- Breed heterozygot KO - Mäuse mit RMCE-kompatible Mäuse, wie ROSALUC Mäuse 10 oder ROSA26-iPS - Mäuse 21. Beide RMCE-kompatible Mäuse wurden auf einem Misch 129 / C57BL6 / Swiss Hintergrund gehalten.
HINWEIS: Kreuzung mit heterozygot KO-Mäusen wird empfohlen, embryonale Letalität in homozygot KO-Mäusen zu überwinden. - Verwenden PCR heterozygot KO - Mäuse wählen 12 eine RMCE Kassette im R26 - Locus enthält.
- Breed RMCE-kompatibel, heterozygot KO-Mäuse mit heterozygot KO-Mäusen und isolieren RMCE-kompatibel, homozygot KO Blastozysten.
- Legen Sie Zeit Paarungen am Abend und prüfen für Kopulation am nächsten Morgen eingesteckt wird.
HINWEIS: Die Stecker sind aus geronnenem Absonderungen aus den koagulierenden und vesikuläre Drüsen der männlichen gemacht.Diese Stecker füllen die Vagina des Weibchens und bleiben für 8-24 Stunden nach der Züchtung. Plugged Frauen gelten als 0,5 dpc (Tage post coitum) Embryonen zu tragen.- Um Stecker zu prüfen, heben Sie das Weibchen durch die Basis ihres Schwanzes und durch ihre vaginale Öffnung für eine weißliche Masse untersuchen. Verbreitet die Lippen der Vulva leicht mit einer abgewinkelten Sonde, wenn der Stopfen nur schwer zu sehen. Separate gesteckt Frauen von ihren männlichen.
- Sammeln Sie Blastozysten bei 3,5 dpc.
- Euthanize trächtige Weibchen von den zugelassenen Verfahren (zB Genickbruch). Machen Sie einen midventral Schnitt und sezieren die Gebärmutter und Eileiter (noch aneinander befestigt) mit feinen Schere und Pinzette.
- Biegen, um eine 26-Gauge-Nadel in einem Winkel von 45 °. Anhänge eine 1-ml-Spritze mit M2-Medium auf diese gebogene Nadel gefüllt und es verwendet, die Blastozysten aus dem Gebärmutter in den Deckel einer 10-cm-Schale zu spülen.
- Führen Sie die Nadel in das Ende des Uterus, die am nächstendie Eileiter. Halten Sie die Nadel an Ort und Stelle mit einer feinen Pinzette während Schieben des Kolbens; zeigt eine erfolgreiche Spülung der Gebärmutter Schwellung.
- Verwenden, um eine Mundpipette (mit einem Durchmesser von 100 bis 200 um) alle Embryonen zu sammeln und waschen, sie zweimal in einem Tropfen frischen Mediums M2. Unmittelbar nach dem Waschen sie übertragen die Blastozysten zu den Kulturplatten (siehe unten).
HINWEIS: Die Präparation und Handhabung von Blastozysten sollte in laminarer Luftströmung erfolgen.
- Legen Sie Zeit Paarungen am Abend und prüfen für Kopulation am nächsten Morgen eingesteckt wird.
- Isolieren RMCE-kompatibel KO mESCs
- Vorbereiten einer 12-Well - Platte mit Mitomycin-C-behandelten DR4 MEFs (siehe Tabelle of Materials) einen Tag vor der Blastozyste Isolation.
ANMERKUNG: Diese MEFs wurden isoliert aus Tg (DR4) 1Jae / J - Mäusen , die vier arzneimittel selektierbare Gene enthalten , und verleihen eine Resistenz gegen Neomycin, Puromycin, Hygromycin, und 6-Thioguanin 22.- Coat alle Kulturplatten mit 0,1% Gelatine. In 0,1% Gelatinefür 5 min bei 37 ° C in 5% CO 2 und aspirieren die Gelatinelösung zu den Kulturplatten, inkubieren. Seed ein Viertel einer Phiole P2 MEFs in einer 12-Well - Platte und wächst sie in 2 ml MEF - Medium (siehe Tabelle 1, die Tabelle der Materialien) zu einer konfluenten Monoschicht 19.
- Inaktivieren sie mit Mitomycin C (10 ug / ml) für 3 h und wäscht sie zweimal mit phosphatgepufferter Kochsalzlösung (PBS) 19.
- Verwendung einer Mundpipette Platte, die die Blastozysten auf gelatinisierte 12-well-Platten (1 Vertiefung / Embryo), mit Mitomycin-C-behandelten MEFs in SR-ES-Zell-Medium (2 ml / Vertiefung), ergänzt mit entweder 2 uM pluripotin oder mit 2i (1 & mgr; M Inhibitor Erk PD0325901 und 3 uM GSK3-Inhibitor CHIR99021). Inkubieren bei 37 ° C in 5% CO 2.
- Aktualisieren Sie die SR-ES-Zellmedium (ergänzt mit pluripotin oder 2i) alle 2 - 3 Tage.
- Untersuchen Sie jeden Blastozyste unter einem stereomicroAnwendungsbereich bei 4,0X Vergrößerung und prüfen die Schraffur und die Befestigung an der MEF-Schicht.
HINWEIS: Wenn Blastozysten Luke, sie verlieren die Zona pellucida, die sie kapselt. Wells mit ungebunden Blastozysten müssen das Pipettieren mit dem Mund aufgefrischt werden verwendet wird. - Wählen Sie einzelne ICM Auswüchsen (ein Stereomikroskop mit) nach 10 bis 12 Tagen Kultur eine P10-Pipette mit Einwegspitzen verwenden. Übertragen, das Auswachsen in etwa 10 & mgr; l Medium zu einer V-förmigen, 96-Well-Platte, enthaltend 30 & mgr; l / Vertiefung PBS (bei Raumtemperatur).
- Je 50 ul 0,25% Trypsin in jede Vertiefung einer Multikanalpipette und Inkubation für 3 min bei 37 ° C in 5% CO 2.
- In 100 ul FBS enthaltenden MESC Medium; die ICM Auswüchse in einzelne Zellen dissoziiert von 10-15 mal pipettiert; und übertragen Sie die dissoziierten Zellen zu Mitomycin-C-behandeln 96-Well-Platten MEF, die einen Tag vorbereitet wurden, bevor die ICM Kolonien gepickt werden.
- Von diesem Schritt vorwärts, omi t pluripotin oder 2i vom MESC Medium. Am nächsten Tag, ändern Sie das Medium aus FBS- auf SR-haltigen MESC Medium (100 & mgr; l / Well).
- Erweitern Sie die etablierten MESC Linien von 96- bis 24-Well - Format 19.
- Waschen Sie die Zellen mit 200 ul PBS, werden 50 & mgr; l Trypsin und Inkubation für 5 min bei 37 ° C in 5% CO 100 l FBS-basiertes MESC Mediums. dissoziieren durch Pipettieren von 10 - 15 mal unter Verwendung einer Mehrkanalpipette; und übertragen die dissoziierten Zellen zu Mitomycin-C-behandelten 24-Well-Platten MEF.
- Wechseln Sie auf SR-basierten Medium am nächsten Tag. Erweitern Sie die mESCs in ähnlicher Weise von 24- bis 6-Well-Format. Machen Sie 3 bis 4 Frost aus einer konfluenten 6-Well - Platte 19.
- Identifizieren RMCE-kompatibel, homozygot KO mESCs unter Verwendung von PCR - Primern für die R26 - Locus 23 und KO - Allel der Wahl (in diesem Fall p120ctn; PCRs für p120ctn null und floxed Allele wurde beschrieben , bevorlass = "xref"> 12).
- Vorbereiten einer 12-Well - Platte mit Mitomycin-C-behandelten DR4 MEFs (siehe Tabelle of Materials) einen Tag vor der Blastozyste Isolation.

Tabelle 1. Nährmedien. Alle Medien wurden bei 4 ° C gelagert und erwärmt auf 37 ° C 30 min vor der Verwendung.
2. Erzeugung eines RMCE-kompatiblen Vektor-Targeting Rekombinationsklonierung Verwendung
- Klon , der die Rettungs Konstrukte in Rekombinations-kompatible Vektoren Restriktionsenzym (RE) -basierte oder 24 Klonierungstechniken-PCR basiert. Stellen Sie sicher, dass die cDNAs ein Stop-Codon enthalten.
- Design AttB-markierte Primer 24. Sicherzustellen, dass der Vorwärtsprimer enthält die folgenden Elemente: a stretch GGGG, eine Stelle attB1, ein Linker, eine Kozak-Sequenz und etwa 25 Nucleotide der cDNA rescue (beginnend mit ATG). Achten Sie darauf, dass der Reverse-Primer eine ähnliche Zusammensetzung hat: eine GGGG Strecke, eine attB2 Website, einen Linker, und etwa 25 Nukleotides von Rettungs cDNA (reverse Komplement).
- Amplify die Rettung cDNA über PCR AttB-flankierten cDNA zu erhalten.
- Durchführen einer 10-ul BP-Reaktion mit 100 ng AttB-flankierten cDNA und 150 ng der Rekombination-kompatiblen Donorvektor, der ein Kanamycin-Resistenz-Gen enthält.
- Transformieren 5 ul der BP in Mischungen Hitzeschock-kompetente MC1061 Escherichia coli (E. coli) Bakterien (ähnlich wie in Schritt 2.3 beschrieben).
- Identifizieren Kolonien mit dem korrekten rescue cDNA enthaltenden Vektoren (ähnlich wie in Schritt 2.4 beschrieben)
- Durchführen einer 10-ul LR-Reaktion unter Verwendung von 100 ng von Rettungs- cDNA enthaltenden Vektor; 150 ng des Cre-exzidiert pRMCE DV1-Vektor 11 (LMBP 8195); und 2 ul Rekombinase-Mischung, die enthält eine Phagen-codierte Integrase und Excisionase und ein bakterielles Integration Host Factor. bei 25 ° C für 2 h inkubiert.
HINWEIS: Eine LR-Reaktion ist eine Rekombination reagierenIons in der ein Eintrag Klon attL-Stellen und einen Zielvektor, enthaltend attR-Stellen enthalten, wird durch den LR CLONASE Enzymmix rekombiniert. Dies führt zu einem Ausdruck Klon, attB-Stellen das Gen von Interesse flankieren. - Transformieren 5 ul der LR Mischungen in Hitzeschock-kompetenten MC1061 E. coli - Bakterien.
- Zugabe von 5 & mgr; l LR - Mischungen zu einem gerippten, umging, 2-ml - Schraubdeckel Rohr mit 40 ul Hitzeschock-kompetenter E. - coli - Bakterien und Inkubation für 20 min auf Eis. Inkubieren Sie für 5 min bei 37 ° C.
- 1 ml Luria-Brühe (LB) Medium und Inkubation für 1 h bei 37 ° C. Platte 50 & mgr; l auf Ampicillin (Amp; 100 ug / ml) enthaltenden Agarplatten wachsen und über Nacht bei 37 ° C.
- Identifizieren Sie die Kolonien mit dem richtigen Zielvektor.
- Pick-5 Kolonien zufällig eine p200 Spitze verwenden. Übertragen, um die Spitze zu einem Glasteströhrchen, das 2 bis 5 ml LB-Medium wachsen und über Nacht bei 37 ° C.
- ExTrakt den Plasmid-DNA aus den Bakterienkulturen im Handel erhältliche Kits.
- Validieren RE verdaut und Sequenzierung verwendet. Geschnitten 0,5-2 ug Plasmid unter Verwendung von 0,2 & mgr; L RE (20 U / ul) und 1 ul des entsprechenden 10-fach-Puffers in einer 10-ul-Reaktion. mindestens 1 h inkubieren bei 37 ° C und getrennt auf einem 1% Agarosegel. Wählen für Kolonien, die mit dem vorhergesagten Muster von DNA-Fragmenten.
- Analysieren der Vektoren bestätigt (50 ng / & mgr; l) mit Tlox F (ATC ATG TCT GGA TCC CCA TC) und IRES R (GGG GCG GAA TTC GAT ATC AAG) Primer (5 pmol / & mgr; l) unter Verwendung von Sanger-Sequenzierung.
3. RMCE vermittelte MESC Rescue Targeting Konstrukten der R26 Locus
- Starten Sie eine Kultur der RMCE-kompatibel KO mESCs und Passage sie mindestens zweimal auf MEFs in-FBS Basis MESC Medium. Teilen Sie die mESCs auf einem verkleistert 6-Well-Platte.
- Am nächsten Tag, aktualisieren Sie die Zellen, bei etwa 50% Konfluenz, mit 1,5ml FBS-basierte MESC Medium und transfektieren die mESCs mit einem Cre-exzidiert pRMCE DV1-Targeting-Vektor rescue cDNA enthält.
- Machen Sie einen DNA-Mix. 1 ug Targeting - Vektor und 1 & mgr; g FLPe-Expressionsplasmid 25 bis 250 & mgr; l reinen DMEM - Medium.
- Machen Sie eine Lipofektion Mix. ADD 7 uL Lipofektion basierte Transfektionsreagenz (zB Lipofectamine 2000) zu 250 & mgr; l reinem DMEM - Medium , und Inkubation für 5 min bei Raumtemperatur (RT).
Hinweis: ähnliche RMCE Targeting - Effizienz wurden mit anderen Lipofektion Basis erhalten Reagenzien (zB Lipofectamin LTX und Effectene). - Mischen Sie das DNA-Gemisch mit der Lipofektion Mischung und Inkubation für 20 min bei RT. Pipette, um die Transfektion Mischung auf die aufgefrischt mESCs. Vorsichtig schwenken und lassen Sie über Nacht.
- Ein Tag nach der Transfektion aufgeteilt alle mESCs aus der 6-Well-Platte auf eine 10-cm-Kulturschale mit einer konfluenten Schicht von DR4 MEFs und 10 ml FBS-basiertem mESC Medium.
- Zwei Tage nach der Transfektion auszuwählen MESC Klont mit dem korrekten FLPe-vermittelten Kassettenaustausch durch Zugabe von 0,2 mg / ml G418 (100-fach) auf das Medium.
HINWEIS: Stellen Sie eine Kill-Kurve für jede Charge von G418 die niedrigste Konzentration von G418 zu identifizieren, die alle mESCs töten. - Aktualisieren Sie die mESCs täglich mit G418-haltigem MESC Medium. 10 Tage, so dass diese auswählen und erweitern gemäß Schritt 1.4 - Kolonien sollen nach 7 erscheinen.
- Bestätigen Sie die richtige Klone mittels PCR 11.
4. Differenzierung von mESCs in Embryoid Bodies (EBs)
- Starten Sie eine Kultur der KO mESCs mit R26 getriebenen Rettungs Konstrukte und Passage sie mindestens zweimal auf MEFs in-FBS Basis MESC Medium. Passage die mESCs einmal auf verkleistert 6-Well-Platten erhalten der MEFs befreien.
- Waschen mit PBS und trypsinize die fast-konfluenten MESC Kulturen. Platte dissoziiert mESCs in verschiedenen Verdünnungen (1/20 und 1/40) auf nicht-adhärenten, bakteriell-grade petri-Gerichte in Differenzierungsmedium.
- Lassen Sie EBs in diese Gerichte für 30 Tage zu bilden. Aktualisieren Sie das Medium alle 2 - 3 Tage nach dem folgende Verfahren: Übertragung der EB Suspension auf ein 50-ml-Röhrchen, lassen Sie die EBs durch die Schwerkraft, Entfernen des Überstandes, fügen Sie frisches Medium, und übertragen Sie die EB Suspension zurück zu einer bakteriellen Qualität Gericht.
- Analysiert die gezielten mESCs und EBs durch Immunofluoreszenz und Transmissionselektronenmikroskopie unter Verwendung von Protokollen , die zuvor 12 beschrieben wurden.
Access restricted. Please log in or start a trial to view this content.
Ergebnisse
Das Verfahren RMCE-kompatiblen KO MESC Linien zu isolieren , ist in Abbildung 2 dargestellt. Zwei aufeinanderfolgende Züchtungen sind erforderlich RMCE-kompatibel KO Blastozysten zu erhalten. Zunächst werden heterozygot KO-Mäuse mit RMCE-kompatibelen Mäusen gekreuzt RMCE-kompatibel, heterozygot KO-Mäuse zu erhalten. Diese Mäuse werden dann für zeitlich Paarungen mit anderen heterozygot KO-Mäusen verwendet, zu erhalten, 3,5-dpc, RMCE-kompatibel, homozygot KO Blast...
Access restricted. Please log in or start a trial to view this content.
Diskussion
Unsere MESC Isolationsmethode ist benutzerfreundlich und erfordert keine fortgeschrittenen Fähigkeiten oder Ausrüstung, wie Mikrochirurgie von Blastozysten. Somit ist diese Technologie zu einem großen Teil der wissenschaftlichen Gemeinschaft zugänglich. Jeder, der Grundzellkultur Erfahrung kann ICM Auswüchse propagieren und mESCs Linien etablieren. Allerdings erfordert die Spülung und die Handhabung von Blastozysten etwas Übung. Ein Mundpipette wird verwendet Blastozysten zu übertragen und besteht aus einer Mikr...
Access restricted. Please log in or start a trial to view this content.
Offenlegungen
Die Autoren haben nichts zu offenbaren.
Danksagungen
Wir danken Jinke D'Hont, Frederique Van Rockeghem, Natalie Farla, Kelly Lemeire und Riet De Rycke für ihre hervorragende technische Unterstützung. Wir danken auch Eef Parthoens, Evelien Van Hamme und Amanda Goncalves vom Bioimaging Core Facility des Inflammation Research Center für ihre Unterstützung von Experten. Wir erkennen an Mitglieder unserer Forschungsgruppe für wertvolle Diskussionen. Diese Arbeit wurde von der belgischen Wissenschaftspolitik unterstützt (BELSPO Interuniversitäre Anziehungsschwerpunkte - Preis IAP VII-07 DevRepair; https://devrepair.be), von der Königin Elisabeth-Stiftung für Medizin, Belgien (GSKE 2008-2010; http: // www .fmre-gske.be) und der konzertierten Forschungsaktionen (GOA 01G01908) der Universität Gent, Belgien (http://www.ugent.be/en/ghentuniv). SG ist ein Postdoctoral Fellow der Flandern Forschungsfonds (FWO-V).
Access restricted. Please log in or start a trial to view this content.
Materialien
| Name | Company | Catalog Number | Comments |
| ROSALUC Mice | made in house | frozen sperm available upon request | |
| R26-iPSC mice | made in house | frozen sperm available upon request | |
| Vessel probe | Fine Science Tools | 10160-13 | to check for copulation plugs |
| M2 medium | Sigma-Aldrich | M7167 | make aliquots and store at -20 °C |
| Fine forceps (Dumont #5 Standard tip Student forceps) | Fine Science Tools | 11251-10 | spray with 70% EtOH before use (do not autoclave) |
| 23 G needles | Fine-ject | 8697 | |
| 1-mL syringes | Soft-ject | 6680 | |
| 60-mm bacterial grade plates (for flushing) | Gosselin | BB60-01 | |
| Mouth pipette | made in house | see discussion | |
| Mouse embryonic fibroblasts (MEFs, TgN (DR4)1 Jae strain) | ATTC | SCRC-1045 | |
| TgN (DR4)1 Jae mice | The Jackson Laboratory | 3208 | |
| Mitomycin C | Sigma-Aldrich | M0503 | |
| Phosphate buffered saline (PBS) without calcium or magnesium | Gibco | 14190-094 | |
| Tg(DR4)1Jae/J mice | JAX | 3208 | mice that contain four drug-selectable genes and DR4 MEFS confers resistance to neomycin, puromycin, hygromycin and 6-thioguanine |
| 0.1% Gelatin | Sigma-Aldrich | G1393 | Dissolve 0.5 g in 500 mL distilled water, autoclave and store at 4 °C. |
| Trypsin (0.25%) | Gibco | 25200-056 | |
| 2 μM pluripotin | Cayman Chemical | 10009557 | |
| pRMCE-DV1 | BCCM/LMBP collection | LMBP 08870 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| cre-excised pRMCE-DV1 | BCCM/LMBP collection | LMBP 08195 | public available from the BCCM/LMBP collection (http://bccm.belspo.be) |
| pCAG-FlpE-IRES-Puro-pA | Addgene | 20733 | |
| heat-shock competent DH5α bacteria | made in house | ||
| Gateway pDONR221 vector | Thermo Fisher | 12536-017 | contains kanamycin-resistance gene |
| BP clonase II mix | Thermo Fisher | 11789-020 | |
| LR clonase II mix | Thermo Fisher | 11791-020 | |
| Luria Broth (LB) | |||
| Ampicillin | |||
| Applied Biosystems 3730XL DNA Analyzer | Thermo Fisher | 3730XL | |
| G418 | Thermo Fisher | 11811-023 | |
| Lipofectamine 2000 transfection reagent | Thermo Fisher | 11668027 | |
| Lipofectamine LTX transfection reagent | Thermo Fisher | 15338100 | |
| Effectene transfection reagent | Qiagen | 301425 | |
| GATEWAY pENTR 1A vector | Thermo Fisher | A10462 | recombination-compatible vector |
| mouse monoclonal anti-p120ctn antibody | BD Transduction Laboratories | 610134 | |
| mouse monoclonal anti-Ecadherin antibody | BD Transduction Laboratories | 610181 | |
| General equipment | |||
| Sterile dissection tools | fine scissors and forceps for dissecting the uterus | ||
| Sterile pipettes: 5 mL, 10 mL and 25 mL | |||
| 15-mL and 50-mL conical centrifuge tubes | |||
| 96-well culture plates V-shaped bottom and flat bottom) | |||
| Culture dishes: 24 wells, 12 wells and 6 wells | |||
| Multichannel pipettes (to pipette 30, 50, 100 and 200 μL) | |||
| Sterile multichannel reservoirs | |||
| Access to a laminar air flow | |||
| Access to an incubator at 37 °C with 5% CO2 | |||
| Access to an inverted microscope | |||
| Access to a bench-top centrifuge | |||
| Access to a stereo microscope with transmitted-light | |||
| Culture media | |||
| MEF Medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| Dulbecco’s modified Eagle’s medium (DMEM) | Gibco | 41965-062 | |
| 10% fetal bovine serum (FBS) | Sigma-Aldrich | F-7524 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| Sodium pyruvate (0.4 mM) | Gibco | 11360-039 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| SR-based mESC medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| DMEM/F12 | Gibco | 31330-038 | mixed in a 1:1 ratio |
| 15% knock-out serum replacement (SR) | Gibco | 10828–028 | |
| L-glutamine (2 mM) | Gibco | 25030-024 | |
| 0.1 mM non-essential amino acids | Gibco | 11140-050 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| β-mercaptoethanol (0.1 mM) | Sigma-Aldrich | M 3148 | |
| 2,000 U/mL recombinant mouse LIF | (IRC/VIB Protein Service facility) | ||
| FBS-based mESC medium (similar to SR-based mESC medium) | stored at 4°C; warm 30 min at 37°C before use | ||
| Knockout DMEM | Gibco | 10829-018 | |
| 15% FBS | Hyclone | SH30070.03E | |
| Differention Medium | stored at 4 °C; warm 30 min at 37 °C before use | ||
| Iscove's Modified Dulbecco's Medium (IMDM) | Gibco | 21980-032 | |
| 15% FBS | Hyclone | SH30070.03E | |
| 5% CD Hybridoma Medium(1x) liquid | Gibco | 11279-023 | |
| 2 mM L-glutamine | Gibco | 25030-024 | |
| 0.4 mM 1-thioglycerol | Sigma-Aldrich | M-6145 | |
| 50 μg/mL ascorbic acid | Sigma-Aldrich | A-4544 | |
| penicillin (100 U/mL) | Gibco | 15140-122 | |
| streptomycin (100 µg/mL) | Gibco | 15140-122 | |
| 2i | |||
| 1 μM Erk inhibitor PD0325901 | Axon Medchem | Axon 1408 | |
| 3 μM Gsk3 inhibitor CHIR99021 | Axon Medchem | Axon 1386 |
Referenzen
- Gul, I. S., Hulpiau, P., Saeys, Y., van Roy, F. Metazoan evolution of the armadillo repeat superfamily. Cell Mol Life Sci. , (2016).
- Valenta, T., Hausmann, G., Basler, K. The many faces and functions of beta-catenin. EMBO J. 31, 2714-2736 (2012).
- Pieters, T., van Hengel, J., van Roy, F. Functions of p120ctn in development and disease. Front Biosci. 17, 760-783 (2012).
- Keirsebilck, A., et al. Molecular cloning of the human p120ctn catenin gene (CTNND1): Expression of multiple alternatively spliced isoforms. Genomics. 50, 129-146 (1998).
- Capecchi, M. R. Gene targeting in mice: functional analysis of the mammalian genome for the twenty-first century. Nat Rev Genet. 6, 507-512 (2005).
- Wang, H., et al. One-step generation of mice carrying mutations in multiple genes by CRISPR/Cas-mediated genome engineering. Cell. 153, 910-918 (2013).
- Yang, H., et al. One-step generation of mice carrying reporter and conditional alleles by CRISPR/Cas-mediated genome engineering. Cell. 154, 1370-1379 (2013).
- Fu, Y., et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol. 31, 822-826 (2013).
- Branda, C. S., Dymecki, S. M. Talking about a revolution: The impact of site-specific recombinases on genetic analyses in mice. Dev Cell. 6, 7-28 (2004).
- Sandhu, U., et al. Strict control of transgene expression in a mouse model for sensitive biological applications based on RMCE compatible ES cells. Nucleic Acids Res. 39, 1(2010).
- Haenebalcke, L., et al. Efficient ROSA26-based conditional and/or inducible transgenesis using RMCE-compatible F1 hybrid mouse embryonic stem cells. Stem Cell Rev. 9, 774-785 (2013).
- Pieters, T., et al. p120 Catenin-Mediated Stabilization of E-Cadherin Is Essential for Primitive Endoderm Specification. PLoS Genet. 12, e1006243(2016).
- Evans, M. J., Kaufman, M. H. Establishment in culture of pluripotential cells from mouse embryos. Nature. 292, 154-156 (1981).
- Martin, G. R. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells. Proc Natl Acad Sci U S A. 78, 7634-7638 (1981).
- Cheng, J., Dutra, A., Takesono, A., Garrett-Beal, L., Schwartzberg, P. L. Improved generation of C57BL/6J mouse embryonic stem cells in a defined serum-free media. Genesis. 39, 100-104 (2004).
- Bryja, V., et al. An efficient method for the derivation of mouse embryonic stem cells. Stem Cells. 24, 844-849 (2006).
- Ying, Q. L., et al. The ground state of embryonic stem cell self-renewal. Nature. 453, 519-523 (2008).
- Chen, S., et al. Self-renewal of embryonic stem cells by a small molecule. Proc Natl Acad Sci U S A. 103, 17266-17271 (2006).
- Pieters, T., et al. Efficient and User-Friendly Pluripotin-based Derivation of Mouse Embryonic Stem Cells. Stem Cell Rev. 8, (2012).
- Yang, W., et al. Pluripotin combined with leukemia inhibitory factor greatly promotes the derivation of embryonic stem cell lines from refractory strains. Stem Cells. 27, 383-389 (2009).
- Haenebalcke, L., et al. The ROSA26-iPSC Mouse: A Conditional, Inducible, and Exchangeable Resource for Studying Cellular (De)Differentiation. Cell Rep. 3, (2013).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25, 3745-3746 (1997).
- Nyabi, O., et al. Efficient mouse transgenesis using Gateway-compatible ROSA26 locus targeting vectors and F1 hybrid ES cells. Nucleic Acids Res. 37, 55(2009).
- Katzen, F. Gateway((R)) recombinational cloning: a biological operating system. Expert Opin Drug Discov. 2, 571-589 (2007).
- Schaft, J., Ashery-Padan, R., vander Hoeven, F., Gruss, P., Stewart, A. F. Efficient FLP recombination in mouse ES cells and oocytes. Genesis. 31, 6-10 (2001).
- Spencer, H. L., et al. E-cadherin inhibits cell surface localization of the pro-migratory 5T4 oncofetal antigen in mouse embryonic stem cells. Mol Biol Cell. 18, 2838-2851 (2007).
- Stryjewska, A., et al. Zeb2 Regulates Cell Fate at the Exit from Epiblast State in Mouse Embryonic Stem Cells. Stem Cells. , (2016).
- Nagy, A., Gertsenstein, M., Vintersten, K., Behringer, R. Manipulating the mouse embryo: A laboratory manual. , Cold Spring Harbor Laboratory Press. Third edn (2003).
- van den Berghe, V., et al. Directed migration of cortical interneurons depends on the cell-autonomous action of Sip1. Neuron. 77, 70-82 (2013).
- Li, J., et al. The EMT transcription factor Zeb2 controls adult murine hematopoietic differentiation by regulating cytokine signaling. Blood. , (2016).
Access restricted. Please log in or start a trial to view this content.
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten