Method Article
Cell Culture on Silicon Nitride Membranes and Cryopreparation for Synchrotron X-ray Fluorescence Nano-analyse
Dans cet article
Résumé
Présenté ici est un protocole pour la culture cellulaire sur les membranes de nitride de silicium et de plongée-congélation avant l'imagerie de fluorescence de rayon X avec une nanosonde cryogénique synchrotron de rayon X. Lorsque seule une nano-analyse à température ambiante est fournie, les échantillons congelés peuvent être lyophilisés. Il s'agit d'étapes critiques pour obtenir des informations sur la composition élémentaire intracellulaire.
Résumé
On sait très peu de choses sur la distribution des ions métalliques au niveau subcellulaire. Cependant, ces éléments chimiques ont des fonctions réglementaires essentielles et leur homéostasie perturbée est impliquée dans diverses maladies. Les nanosons de fluorescence synchrotron à rayons X à la fine pointe de la technologie fournissent la sensibilité et la résolution spatiale requises pour élucider la distribution et la concentration bidimensionnelles (2D) et tridimensionnelles (3D) des métaux à l'intérieur de cellules entières à la niveau d'organelle. Cela ouvre de nouveaux domaines scientifiques passionnants de recherche sur le rôle des métaux dans la physiopathologie de la cellule. La préparation cellulaire est une procédure clé et souvent complexe, en particulier pour l'analyse de base. Bien que les techniques de fluorescence aux rayons X soient maintenant répandues et que diverses méthodes de préparation aient été utilisées, très peu d'études ont étudié la préservation de la teneur élémentaire des cellules, au mieux, et aucun protocole détaillé sur le plan pas à pas pour la cryopréparation de des cellules adhérentes pour les nanosondes de fluorescence des rayons X ont été libérées jusqu'à présent. Il s'agit d'une description d'un protocole qui fournit la préparation cellulaire progressive pour la cryofixation rapide pour permettre la nano-analyse de fluorescence de rayons X synchrotron des cellules dans un état hydraté gelé quand un environnement cryogénique et le transfert est disponible. Dans le cas où la nano-analyse doit être effectuée à température ambiante, une procédure supplémentaire pour le lyophilisation de la préparation cellulaire adhérente cryofixe est fournie. Les protocoles proposés ont été utilisés avec succès dans des travaux précédents, plus récemment dans l'étude de la distribution intracellulaire 2D et 3D d'un composé organométallique dans les cellules cancéreuses du sein.
Introduction
Les nanosons de fluorescence des rayons X synchrotron (SR-XRF) nouvellement conçus permettent de visualiser la distribution subcellulaire des éléments d'une manière entièrement quantitative. À titre d'exemple, cette capacité analytique permet d'examiner l'utilisation de nanoparticules1 ou de molécules organométalliques telles que les complexes à base d'osmium2,fournissant un aperçu de l'utilisation intracellulaire de molécules à base de métaux ayant de puissantes propriétés anticancéreuses. En tant que technique multi-éléments, SR-XRF3 avec une nanoprobe fournit un moyen de quantifier et de localiser simultanément les éléments intracellulaires les plus importants biologiquement, y compris le phosphore, le soufre, le potassium, le calcium, le fer, le cuivre et le zinc. En effet, l'utilisation de rayons X durs fournit une grande profondeur de pénétration pour l'image de cellules entières hydratées congelées d'une manière sans étiquette. En outre, offrant l'accès à la k-edge de la plupart des éléments d'intérêt, la fluorescence aux rayons X est excité le plus efficacement. L'utilisation d'approches cryogéniques permet de réduire les dommages causés par les radiations et d'optimiser la préservation de la structure cellulaire et la distribution élémentaire.
La plupart des techniques d'analyse spatialement résolues disponibles pour étudier les métaux dans les cellules sont des techniques de surface nécessitant des sections très minces et plates de cellules à produire. Il s'agit principalement de la microscopie électronique de transmission de balayage avec l'analyse de rayons X dispersive séviscreuse d'énergie (STEM-EDX), la microscopie électronique de transmission à transmission d'énergie (EF-TEM) et la spectrométrie de masse d'ions secondaires à l'échelle nanométrique (nanoSIMS). Cette dernière ne peut pas être effectuée sur des sections de cellules congelées et hydratées, tandis que la cryo-analyse peut être effectuée avec une microscopie électronique avec une résolution spatiale inégalée mais une faible sensibilité élémentaire. L'émission de rayons X induite par les particules (PIXE) a permis l'étude des distributions élémentaires dans des cellules entières. Il a l'avantage d'être entièrement quantitatif avec une sensibilité élémentaire juste à l'échelle du micron et même à la résolution submicron4, mais souffre de dommages causés par les radiations et le manque de capacités cryogéniques pour étudier les cellules hydratées congelées. Toutes ces techniques analytiques se complètent dans l'imagerie élémentaire des cellules, mais pour toutes les techniques, la procédure de préparation de l'échantillon est une étape cruciale. Il faut garder simple de limiter la contamination possible ainsi que la redistribution élémentaire et / ou des fuites pour obtenir des résultats significatifs. Comme démontré dans la microscopie électronique, un flux de travail cryogénique, y compris la cryo-immobilisation de la cellule et le cryotransfert à un stade de cryoscanning, permet une conservation élémentaire optimale à des niveaux subcellulaires aussi près que possible de l'état natif5,6,7,8,9,10. Cette compréhension a été mise en œuvre avec succès dans le développement de la microscopie à rayons X cryo-douce synchrotron (p. ex., microscopes complets sur le terrain et microscopes à balayage) pour produire une imagerie ultrastructurale de cellules entières hydratées congelées en 2D ou en 3D. Divers flux de travail cryogéniques ont été développés11 pour les microscopes à rayons X souples à Beamline 2.1 (XM-2) de l'Advanced Light Source au Lawrence Berkeley National Laboratory12, beamline U41-XM à l'anneau de stockage d'électrons BESSY II (Allemagne)13, faisceau MISTRAL de la source lumineuse ALBA (Espagne)14, et à Beamline B24 de la source de lumière Diamond15, entre autres. Un flux de travail similaire s'est récemment avéré être la méthode de préparation et de conservation la plus fiable pour l'analyse élémentaire intracellulaire à l'aide de microsondes à rayons X16,17.
Bien que les techniques de nanoprobe à rayons X commencent à être largement utilisées pour l'analyse élémentaire cellulaire, en particulier avec l'avènement des capacités sR-XRF cryogéniques, aucun protocole pas à pas n'a été diffusé jusqu'à présent à la communauté de la recherche. Ici, une procédure détaillée est fournie pour préparer les cellules adhérentes cryofixes cultivées comme monocouches sur des membranes de nitride de silicium à analyser dans des conditions cryogéniques. Une étape de séchage au gel à appliquer après le protocole au cas où l'analyse des rayons X doit être effectuée à température ambiante est également fournie. Tandis que le protocole proposé a été avec succès employé avec les cellules humaines de cancer du sein MD-MB-2312 et le gel-séchage a été démontré entre autres sur des neurones de souris18,20,21,il peut être facilement étendu à divers types de cellules humaines ou animales.
Protocole
Les procédures expérimentales ont été approuvées par le comité de soins aux animaux de la Division des sciences de la vie du CEA (CETEA, A14-006). Elles ont été menées conformément à la législation Français et à la directive du Conseil communautaire européen du 24 novembre 1986 (86/609/CEE).
1. Préparation de soutien de la membrane de silicium (Si3N4)
REMARQUE : Parce que la membrane est fragile et délicate, son support (cadre en silicium de 200 m d'épaisseur) doit être manipulé en douceur, idéalement avec une pince à épiler en carbone mince ou des pinces Dumont #5, Straight Self-closing fine tips. Ce protocole utilisait des membranes de nitride de silicium avec un cadre de 5 mm x 5 mm et une taille de membrane de 1,5 mm x 1,5 mm. La membrane doit être préparée à environ 12 h avant de commencer l'expérience (c.-à-d. l'ensemencement cellulaire). Les membranes peuvent être préparées à la fin de la journée et laissées sécher toute la nuit sous une hotte à débit laminaire de classe II afin qu'elles soient prêtes à l'utiliser le lendemain matin. Une épaisseur de cadre en silicium de 200 m est la norme pour la plupart des entreprises qui vendent des fenêtres de nitride de silicium. Si le produit utilisé dans ce protocole n'est pas disponible, une taille de membrane de l'ordre de 0,5 à 1,5 mm peut être utilisée avec un cadre standard de 5 mm x 5 mm. La plus grande taille de membrane est préférée quand la tomographie de rayon X sera employée. Les fenêtres de nitride de silicium de type grille TEM d'une taille de membrane de 0,5 mm et d'une épaisseur de 50 nm peuvent également être utilisées.
- Ouvrez la capsule contenant le support membranaire Si3N4 (Figure 1). Presser doucement la capsule afin de desserrer légèrement le support.
- Tenez l'un des coins du cadre en silicium à l'aide de la pince à épiler mince. Veillez à ne pas toucher la membrane Si3N4 au centre. La membrane de 200 ou 500 nm d'épaisseur peut être facilement endommagée.
- À l'aide de la pince fine, placez délicatement le support membranaire Si3N4 dans un plat stérile petri en verre, surface plane de la fenêtre de nitride de silicium orientée vers le haut (c.-à-d., la cavité faisant face au fond du plat).
- Retirer le couvercle du plat Petri et laisser les membranes sous la lumière UV pendant 25 à 30 min sous l'armoire à débit laminaire.
REMARQUE : La lumière UVC (254 nm) est généralement fixée à 200 W/cm2. - Mettre 10 l de poly-L-lysine sur la membrane. La goutte doit bien couvrir la membrane Si3N4 et peut se propager un peu sur le cadre de silicium. Laissez-le à 37 oC pendant 25 min dans l'incubateur standard de culture tissulaire à 100% d'humidité relative et 95% d'air, 5% de CO2.
REMARQUE : Dans ce cas-ci un revêtement de poly-L-lysine a été employé pour les cellules de cancer du sein de MDA-MB-231. Selon le type de ligne cellulaire, divers revêtements peuvent être utilisés, et cette étape doit être optimisée en conséquence. - Dans une plaque stérile de 48 puits, remplissez différents puits de 200 à 250 l d'eau ultra-pure et ultra-trace filtrée à travers un filtre stérile de 0,22 m. Typiquement, chaque puits peut être utilisé pour rincer jusqu'à 2/3 membranes. À l'aide d'une pince fine, ramasser le support de la membrane à un coin de son cadre en silicium. Rincer doucement la membrane en la submergeant verticalement de 10 s dans trois puits successifs.
REMARQUE : Les supports membranaires sont sortis de l'incubateur et peuvent être traités à température ambiante, avec la température et l'humidité définies par une hotte à débit laminaire de classe II. - Placez le support de membrane verticalement dans un puits vide d'une plaque stérile de puits 96, couvrez-la et laissez-la sécher toute la nuit sous une hotte à débit laminaire de classe II.
2. Ensemencement cellulaire
- Dans une plaque stérile de 4 puits, placez les membranes avec leur côté plat vers lehaut.
- Les cellules MDA-MB-231 sont maintenues dans une culture monocouche dans le DMEM avec le phénol rouge/Glutamax I, complétée avec 10% de sérum foetal de veau et 1% de pénicilline et de streptomycine à 37 oC dans un incubateur humidifié à l'air CO2 de 5%.
- Lorsque les cellules atteignent 60 à 70% de confluence enlever le support du plat ou flacon.
- Laver 1x avec 10 ml de phosphate tamponné de Dulbecco salin sans Ca2 ou Mg2 .
- Ajoutez 3 mL/T75 flacon de la solution trypsine/EDTA de 0,05 % et assurez-vous que la monocouche entière est recouverte de la solution trypsine.
- Incuber pendant 3 à 5 min à 37 oC jusqu'à ce que les cellules commencent à se détacher. Il faut prendre soin de ne pas sur-trypsiniser les cellules et de ne pas forcer les cellules à se détacher prématurément.
- Ajouter 8 ml de DMEM complété par 10% de sérum fœtal de veau et 1% de pénicilline et de streptomycine ou de médias complets et recueillir les cellules par pipetting. Le sérum dans les médias neutralisera la trypsine.
- Faire tourner à 250 x g pendant 3 min à température ambiante. Aspirez le supernatant.
- Ajouter 8 ml de puisette complète fraîche au tube de 15 ml contenant la pastille cellulaire, et pipet les cellules de haut en bas jusqu'à ce que les cellules sont dispersées dans une suspension de cellule unique.
- Comptez les cellules à l'aide d'un hémocytomètre et diluez-vous à une concentration de 5 x 106 cellules par mL dans un média complet (DMEM avec du phenol rouge/1 % d'un 200 mM L-alanyl-L-glutamine dipeptide dans 0,85% solution NaCl complétée par 10% de sérum de veau fœtal et 1% de pénicice et de streptomycine).
- Prenez 10 l de la suspension de la cellule MDA-MB-231 et déposez-la sur la membrane. Cela correspond à 50 000 cellules/10 L pour MDA-MB-231. La goutte doit bien couvrir la membrane Si3N4 et peut se propager un peu sur le cadre en silicium. Il faut prendre soin de ne pas toucher la membrane Si3N4 avec la pointe de la micropipette.
REMARQUE : Selon le type de lignée cellulaire et d'expériences ou de mesures, la densité cellulaire peut varier et doit être testée en conséquence. Ici, la densité cellulaire proposée pour l'ensemencement de la membrane Si3N4 a été jugée optimale pour les conditions expérimentales et d'autres nano-analyses SR-XRF des cellules MDA-MB-2312. - Pour les neurones hippocampiques (HN), retirez le tissu cérébral de l'hippocampe des souris embryonnaires de 18,5 jours et digérez-le en trypsine de 0,25% dans Hepes-HBSS (5,3 mM KCl, 0,44 mM KH2PO4, 137,9 mM NaCl, 0,34 mM NaH2PO4, 5,56 mM de glucose) à 37 oC pour 15 min18,19.
- À l'aide d'une pipette P1000 avec une pointe P1000 et une pointe P200, effectuer la dissociation mécanique en dessinant et en libérant le contenu du cône avec la pipette plusieurs fois. Pendant cette étape, veillez à ne pas créer de bulles d'air dans le milieu, car les bulles d'air sont toxiques pour les neurones.
- Attendez quelques minutes jusqu'à ce que l'agrégat se dépose au fond du tube.
- Transférer le supernatant contenant les cellules dispersées dans un tube stérile d'Eppendorf. Laisser 25 euros de milieu de culture contenant l'agrégat.
- Comptez les cellules dissociées à l'aide d'un hémocytomètre. Les neurones Isolés de HN sont plaqués à une concentration de 7 x 104 cellules cm-2 sur la membrane de nitride de silicium poly-L-lysine (1 mg/mL)-enduit.
- Seulement pour les membranes avec HN, incuber les neurones dans le premier DMEM complété avec 10% de sérum bovin fœtal. Un h après le placage HN dans DMEM, le milieu est changé en milieu de placage neurobasal (200 mM L-alanyl-L-glutamine dipeptide en 0,85% solution NaCl, et B27 supplément d 1/50 dilué en Neurobasal)18,19.
- Pour les cellules MDA-MB-231, placez les supports membranaires à 37 oC dans l'incubateur (100 % d'humidité relative, 95 % d'air et 5 % de CO2)pendant 25 min. Cela permet aux cellules de se déposer et de commencer à se fixer au substrat. Cela peut être adapté en fonction de la lignée cellulaire utilisée.
- Ajouter 1 ml du milieu de culture complet requis (DMEM avec Phenol Red/1% d'un dipeptide L-alanyl-L-glutamine de 200 mM dans une solution NaCl de 0,85% complétée par 10 % de sérum fœtal de veau et 1 % de pénicilline et de streptomycine) dans chaque puits des cellules MDA-MB-231 en plaçant la pointe de pipette contre la paroi du puits en plastique et en libérant le milieu très lentement pendant qu'il couvre la membrane.
- Placez la membrane verticale contre la paroi de la plaque de 4 puits afin d'enlever les bulles d'air emprisonnées dans la cavité du puits de la membrane Si3N4 (Figure 2). Pour ce faire, utilisez des pinces fines et repoussez la bulle très doucement, en se déplaçant parallèlement au cadre arrière Si3N4 pour éviter de toucher et d'endommager la membrane.
- Remettez la membrane horizontalement au fond du puits et laissez la plaque de puits 4 dans l'incubateur pendant le temps requis en fonction du taux de croissance de la lignée cellulaire utilisée. Les cellules MDA-MB-231 ont été incubées pendant la nuit.
3. Traitement ou changement moyen
- Retirer le milieu de la plaque de 4 puits.
- Rincer une fois avec 1 ml de solution PBS à 37 oC. Jeter le PBS et ajouter 1 ml de milieu frais complet réchauffé en présence ou en absence (contrôles) du traitement désiré à l'aide d'une pointe de pipette de 1 ml, libérant le liquide très lentement contre la paroi de la plaque de puits. La membrane Si3N4 doit être lentement submergée sans aucune perturbation pour éviter le mouvement de la membrane ou le levage.
4. Cryo-immobilisation de la préparation cellulaire par le plongeon-congélation
REMARQUE : À la fin du temps d'incubation requis, en présence ou en absence de traitement, les cellules doivent être soigneusement rincées et cryofixes. Environ 30 minutes avant de commencer à rincer et à effacer la préparation cellulaire avant de plonger- geler, d'abord mettre en place et refroidir la machine automatique de congélation à plongée. Lorsque vous manipulez des cryogènes, l'utilisation de gants cryogéniques appropriés, de lunettes de sécurité, de chaussures fermées et d'une blouse de laboratoire est nécessaire. L'azote liquide doit être transporté dans les Dewars appropriés, et le lieu de travail doit être suffisamment ventilé avec la présence d'un moniteur d'oxygène. Idéalement, un faible niveau d'hygrométrie de 20 à 30 % aide à limiter la contamination par la glace des matériaux, des Dewars et des cryogènes, ce qui nuit à la vitrification des échantillons (c.-à-d. une couche de glace amorphe). Idéalement, selon le niveau d'expérience du chercheur, jusqu'à 10 à 12 échantillons pour une seule séance peuvent être préparés à l'aide de la même tasse d'éthane liquide cryogène secondaire pour la vitrification. Entre les séances, le congélateur à plongée automatique nécessite une procédure de cuisson automatique de 1 h. Idéalement, les échantillons devraient être traités dans des conditions d'incubation identiques. Néanmoins, les contrôles peuvent être traités en premier, suivis par les échantillons avec une condition de traitement particulière.
REMARQUE : Pour le plongeon-congélation, les étapes suivantes s'appliquent à la fois aux cellules MDA-MB-231 ou à HN.
- Configurer le cryoplunger pour la cryofixation rapide des cellules.
- Allumez le congélateur à plongée automatique.
- Entrez les paramètres (p. ex., température, pourcentage d'humidité, temps de ballonnement si le ballonnement automatique est utilisé, et position pour soulever l'échantillon à la surface du cryogène pour faciliter le transfert vers un contenant cryogénique) directement à partir de la console et des paramètres menu de paramètres. Dans le cas présent, les paramètres de la chambre d'humidité ont été fixés à 37 oC et 80 % d'humidité.
REMARQUE : De meilleurs résultats de vitrification ont été obtenus pour ce protocole et la formation image de rayon X avec le blotting manuel rapide et soigneux. Ainsi, le protocole n'utilise pas un programme de séquence de ballonnement automatique. - Attachez la chambre humidificateur et afin de préserver l'humidité d'abord le remplir à l'aide d'une seringue avec 60 ml d'eau double distillée, puis 20 ml comme demandé sur la console de congélation à plongée automatique.
REMARQUE : Évitez d'utiliser de l'eau ultrapure, car elle peut endommager le système de vaporisateur. Fermez la vanne et laissez le tube attaché à l'arrière de l'humidificateur. - Installez la tasse d'éthane noir dans son support et couvrez-la avec les bouchons en plastique.
- Remplissez le dewar de la chambre froide avec LN2, l'amenant au niveau de la grille dans la zone de travail.
- Mettez une cryo-boîte dédiée pour stocker les membranes après la cryofixation dans le conteneur de transfert conservé dans l'emplacement dédié dans la zone de travail EM-GP et à proximité du porte-gobelets éthane.
REMARQUE : La cryo-boîte dédiée est un développement interne à la nanoprobe beamline ID16A du rayonnement synchrotron européen à Grenoble. Des dessins avec spécifications sont disponibles sur demande (Figure 3). Ils peuvent être stockés quatre à la fois dans un tube conique de 50 ml pour le stockage à long terme dans un LN2 Dewar. Une autre possibilité consiste à utiliser un petit tube pcR pcR mince de 0,2 ml avec des bouchons de dôme pour stocker un seul support de membrane Si3N4. Vous devrez percer un trou de 2 mm dans la partie supérieure du tube mural à l'aide d'une aiguille à seringue chauffée afin de permettre à LN2 de remplir le tube. - Remplissez le contenant de transfert avec lN2 et couvrez-le du couvercle en aluminium dédié. Continuer à remplir la chambre froide avec LN2 (généralement 2 L est nécessaire) en gardant à 100% l'écran de moniteur de niveau LN2 sur la console. Attendez que la température finale requise soit atteinte.
- Retirez le bouchon en plastique et recouvrez la tasse d'éthane avec le liquéfieur relié à la bouteille d'éthane. Attendez que la température de la tasse d'éthane équilibre jusqu'au point de température fixé. Lorsqu'il est atteint, commencer à utiliser le cryogène secondaire (c.-à-d., éthane liquéfié).
REMARQUE : Le point d'ensemble utilisé était de -180 oC, légèrement au-dessus du point de fusion de l'éthane (-182,8 oC). Vous n'avez pas besoin de pré-refroidir le liquéfieur d'éthane, car il peut être une source de formation de gel et de contamination de la tasse d'éthane. - Ouvrez la soupape principale de bouteille d'éthane de haute pureté et ouvrez très lentement le régulateur de pression jusqu'à ce que vous obniez un brouillard lent d'éthane. Maintenir ce débit très faible jusqu'à ce que l'éthane liquide s'accumule. Remplissez la tasse jusqu'à son bord supérieur. Fermer le régulateur de pression et la vanne principale de la bouteille d'éthane. Retirez soigneusement le liquéfieur Leica et laissez-le de côté sur un petit support en polystyrène sous le capot de fumée. Gardez la zone de travail recouverte d'un bouchon de polystyrène noir fourni avec la machine pour prévenir la contamination par le gel de la zone de travail et du contenant d'éthane.
- Juste avant le ballonnement manuel de l'échantillon, retirez le bouchon en polystyrène noir et du menu de la console de presse "Chambre basse", qui met la chambre environnementale en contact avec la zone de travail cryogénique.
- Préparez-vous à effacer l'échantillon.
- Préparer le tampon adéquat pour enlever les traces de sels du milieu de culture. Pour ce protocole, tampon d'acétate d'ammonium a été utilisé pour rincer les cellules MDA-MB-231.
REMARQUE: Tampon d'acétate d'ammonium est adapté pour la plupart des types de cellules, et il n'ajoute pas au signal de fluorescence des rayons X (compte tenu des éléments avec Z 'gt; 9). Certaines lignées cellulaires particulières telles que les cellules neuronales peuvent nécessiter l'utilisation d'un tampon dédié. Par exemple, pour les neurones corticaux primaires, une solution saline composée de 1,8 volume de 0,5 M Na2HPO4 et 1,9 volume de 0,5 M NaH2PO4 peut être utilisé15. D'autre part, le phosphore ou le chlore contenu dans le tampon contribuera au spectre XRF. Cette limitation des fausses lignes d'émission de rayons X doit être gardée à l'esprit en fonction des éléments d'intérêt à détecter. - Préparer une solution d'acétate d'ammonium de 150 mM à partir d'une solution ultrapure d'acétate d'ammonium et vérifier le pH (7,0 à 7,3) et l'osmolarité (270 à 300 mOsm/kg)
REMARQUE : L'osmolarité mentionnée ci-dessus est équivalente à la saline tampon de phosphate de Dulbecco (D-PBS) sans calcium et magnésium et peut être vérifiée à l'aide d'un micro-osmomètre. - Remplissez le nombre requis de puits à partir d'une plaque en plastique de 12 puits avec le tampon d'acétate d'ammonium.
- Couper un quart de papier filtre pour le ballonnement, soit à partir du papier filtre no 1 avec un trou prédécoupé, soit du papier filtre poinçonné manuellement d'un diamètre de 55 mm avec un trou central de 15 mm.
- Sortez l'échantillon requis stocké dans l'incubateur à 37 oC au dernier moment avant de rincer et de plonger la membrane.
- Débloquez les pinces à l'aide de l'anneau de pince noire des forceps à libération rapide (généralement une pince à pinces médicales de haute précision de l'anneau de serrage Dumont) et saisissez le support de membrane Si3N4 de la culture bien.
REMARQUE : Saisissez le milieu du cadre en silicium, en gardant la pointe de la pince près de la membrane. Déplacez l'anneau de pince noire vers le bas pour les premières bandes pour verrouiller les pinces. - Immerger le support de la membrane Si3N4 verticalement dans la solution tampon d'acétate d'ammonium maintenue à 37 oC pendant 5 s.
REMARQUE : Le support doit rester vertical dans le tampon. Notez que la solution tampon dans chaque puits de la plaque peut être utilisée pour un jusqu'à trois membranes pour les mêmes conditions d'incubation. - Blot manuellement avec du papier filtre pour drainer l'excès tampon de la solution de rinçant la membrane (Figure 4) afin de laisser une couche mince et homogène de l'acétate d'ammonium solution aqueuse couvrant les cellules.
REMARQUE : Pour ce faire, appuyez d'abord sur l'arrière de la fenêtre sur le papier filtre pour enlever presque tout le tampon aqueux restant dans le puits et l'arrière de la membrane. Deuxièmement, effacer le côté avant, en commençant par les deux côtés de la pince à épiler, puis chaque côté du cadre (Figure 4). Ne touchez jamais la membrane. L'excès de tampon drainé peut être surveillé avec l'auréole formé sur le papier filtre. - Ouvrez la porte de la chambre environnementale et montez rapidement les pinces, la glissant dans le verrouillage des forceps, et fermez la porte (figure 5).
- Appuyez sur "Blot/A plunge". Les pinces contenant la membrane Si3N4 seront rapidement plongées dans le cryogène.
- Retirez le couvercle du contenant de transfert avec des forceps pré-réfrigérés.
- Appuyez sur "Transfert". La membrane Si3N4 sera légèrement déplacée au-dessus du cryogène.
- En un seul mouvement rapide, déconnectez les pinces en les faisant glisser hors du verrouillage des forceps et inclinez légèrement hors du verrouillage pour les amener directement dans une fente vide de la cryo-boîte dans le conteneur de transfert rempli de LN2. Relâchez l'anneau de pince noire pour libérer la membrane (Figure 5).
REMARQUE: Le conteneur de transfert doit toujours être recouvert de LN2. Lorsqu'une recharge est nécessaire, recouvrir la tasse d'éthane du couvercle en plastique fourni avec la machine pour éviter de mélanger l'éthane Etaïe. - Couvrir le récipient de transfert avec un couvercle et utiliser une petite tasse en polystyrène blanc remplie de LN2 pour le transférer dans une boîte en polystyrène remplie de LN2.
REMARQUE : La cryo-boîte ou le tube contenant les membranes peut ensuite être stocké dans des tubes coniques de 50 ml remplis de LN2 et transférés dans un stockage à long terme LN2 Dewar. Avant de commencer à plonger, congelez l'échantillon suivant, réchauffez toutes les pinces froides et givrées à l'air d'un sèche-cheveux ou d'un séchoir à plaques chaudes/cryotools (45 oC) afin d'éviter toute contamination par des cristaux de glace.
- Préparer le tampon adéquat pour enlever les traces de sels du milieu de culture. Pour ce protocole, tampon d'acétate d'ammonium a été utilisé pour rincer les cellules MDA-MB-231.
5. Le séchage des cellules surgelées cultivées sur des membranes de nitride de silicium
REMARQUE : Pour le lyophilisation, les étapes suivantes s'appliquent aux cellules MDA-MB-231 et HN. Pour refroidir le séchoir à congélation, vous devrez attendre environ 40 min à 1 h.
- Configurer la sécheuse à congélation
- Allumez la puissance avec l'interrupteur de bascule situé sur le panneau arrière de l'instrument.
- Commencer à entrer les paramètres suivant le menu LCD: Segment 1 ' 2 h à -120 'C; Rampe de segment 2 à 2 h de -120 oC à -80 oC; Segment 3 à 2 h à -80 oC; Rampe de segment 4 à 2 h de -80 oC à 50 oC; Segment 5 à 2 h à -50 oC; Rampe de segment 6 à 6 h de -50 oC à 30 oC.
- À la fin de la configuration du paramètre, enregistrer les réglages, fermer le couvercle de la chambre et appuyez sur "START".
- L'unité pompera jusqu'à 1,10-5 mbar. Lorsque cette pression est atteinte, la ligne de commande de l'écran affichera "Start Cooling Now, START to continue".
- Remplissez régulièrement l'azote liquide Dewar pour refroidir la scène en dessous du réglage de la température triple point.
REMARQUE : La température du triple point de l'étape est fixée à -140 oC. Avant de charger l'échantillon pour ce protocole, il est préférable d'attendre environ 1 h et une température de -160 oC. - L'affichage affichera "Press ENTER" lorsqu'il est prêt à "Charger l'échantillon".
- Refroidissez-vous à la température de l'azote liquide dans un LN2 rempli de polystyrène Dewar, le support de transfert d'échantillon fourni par le fournisseur, et les deux supplémentaires cylindriques en laiton Si3N4 porte-membrane.
- Montez le support en laiton à membrane Si3N4 sur le dessus du support de transfert d'échantillon fourni par le fournisseur dans le dewar en polystyrène(figure 6A). Maintenez le niveau de LN2 à environ 1/2 mm au-dessous du bord supérieur de la première pièce en laiton.
- Procurez-vous un support d'échantillon de membrane Si3N4 à partir de la cryo-boîte ou du tube PCR à l'aide d'une pince pré-réfrigérée à l'inox ou au téflon.
- Déposez la membrane avec le côté de l'échantillon de cellules face vers le haut dans la cavité numérotée de support de laiton.
- Couvrir l'assemblage de la deuxième pièce en laiton comme couvercle (Figure 6C).
REMARQUE : Nous avons conçu deux disques en laiton d'une épaisseur de 5 mm chacun, d'un diamètre de 50 mm et d'un trou central de 11 mm de diamètre. Le premier disque en laiton a 14 emplacements rectangulaires usés (8 mm x 6 mm) pour accueillir les supports (5 mm x 5 mm). Chaque fente a un puits plat et poli avec une profondeur de 2 mm. Le deuxième disque en laiton est plat pour couvrir le support en laiton à membrane Si3N4 et agit comme un boîtier de piège à froid. - Précool la tige de transfert dans la boîte en mousse de polystyrène remplie LN2 et l'utiliser pour verrouiller l'assemblage complet (Figure 6D, E).
- Appuyez sur "ENTER" sur le panneau avant du séchoir à congélation.
- Les pompes turbo et rotatives s'arrêteront et la chambre purgée avec du gaz azoté sec pour permettre l'ouverture du couvercle de la chambre.
- Transférer immédiatement l'assemblage de transfert d'échantillon avec la tige de transfert à ressort dans la chambre de sécheuse à gel et le couper sur le stade froid LN2 en cuivre.
REMARQUE : Laissez l'assemblage complet avec la tige de transfert dans la chambre. - Fermez immédiatement le couvercle de la chambre de sécheuse à gel et appuyez sur «START» pour continuer le cycle de séchage.
- Remplissez le réservoir LN2 de la sécheuse à congélation manuellement toutes les 2 h.
REMARQUE : Un système de remplissage Automatique LN2 peut être connecté à ce réservoir. - À la fin du cycle de séchage, appuyez sur «STOP» pour évacuer la chambre et retirez l'assemblage complet pour accéder aux échantillons lyophilisés.
Résultats
Une vue vidéo typique de microscope de MDA-MB-231 hydratée congelée qui ont été sous-cultivées sur un support de membrane si3N4 enduit de poly-L-lysine est montrée dans la figure 7A. La vue optique de l'échantillon dans la chambre à vide a été obtenue en mode de réflexion à l'aide du microscope vidéo en ligne dédié de la ligne de faisceau ID16A de l'ESRF22. Bien que la microscopie électronique ou douce aux rayons X exige que la couche de glace qui intègre la cellule soit aussi mince que possible (généralement 0,5 m), les rayons X durs (keV) ont l'avantage d'une profondeur de pénétration beaucoup plus élevée et d'un dépôt de dose plus faible. L'épaisseur de la glace peut donc être plus grande, généralement 10 m, y compris la cellule, de sorte que la glace qui intègre la cellule est de quelques mètres d'épaisseur. Ceci peut être estimé par l'intensité mesurée de rayon X dans la transmission comparée à l'intensité sans l'échantillon, en tenant compte de l'absorption de la membrane Si3N4 épaisse de 500 nm. Cette épaisseur de glace peut être atteinte par le ballonnement manuel tel que décrit dans le protocole actuel. Dans la région des anneaux Newton, l'épaisseur de la glace peut être encore plus mince (non mesurée).
La cartographie élémentaire de la fluorescence des rayons X de la cellule hydratée congelée est indiquée à la figure 7B avec les distributions représentatives d'éléments physiologiques tels que le potassium (K), le soufre (S) et le zinc (Zn). Ces cartes représentent la masse aréale élémentaire (c.-à-d. la masse projetée élémentaire). Bien qu'elles ne soient pas effectuées en l'espèce, ces cartes peuvent être normalisées grâce à l'imagerie de contraste de phase basée sur la propagation des rayons X qui fournit l'estimation de la masse projetée de l'échantillon23. Comme l'ont rapporté de nombreuses études, l'ion K hautement diffusible dans les cellules conservées dans leur état quasi-indigène a été supposé être homogènement distribué dans toute la cellule23,24,16. Comme le montre les images élémentaires de fluorescence 2D de la fluorescence des rayons X de la figure 7B, l'élément S étroitement lié a été réparti uniformément dans la cellule, de la même façon que K, et représente une bonne estimation du profil de masse cellulaire. La distribution de Zn avait un signal plus élevé dans le noyau que dans le cytosol et a clairement décrit le noyau. On peut noter que de petites régions enrichies de Zn peuvent être détectées à la résolution spatiale (50 nm) dans la région nucléaire.
Les nanosondes de rayons X existantes ou celles à construire ne s'adaptent pas nécessairement aux capacités cryogéniques. Dans ce cas, la meilleure alternative pour obtenir des images de fluorescence de rayon X des cellules aux résolutions spatiales de sous-100 nm est d'exécuter une procédure de gel-séchage décrite dans ce protocole après le plongeon-congélation de la cellule. La figure 8A montre une vue typique de microscopie de champ lumineux des neurones hippocampaux primaires de souris lyophilisés résultants directement cultivés sur la membrane Si3N4. Dans ce cas, s'ils sont stockés dans une chambre propre et desséchée, les échantillons peuvent être préparés une ou deux semaines à l'avance et être observés avec un microscope optique droit ordinaire pour l'enregistrement des régions d'intérêt. Il faut prendre soin d'éviter l'exposition à l'humidité ambiante, car elle peut être captée par l'échantillon lyophilisé et entraîner des dommages sous le nanofaisceau de rayons X. Cette procédure a été appliquée avec succès à des cellules très sensibles (c.-à-d., cellules neuronales) et des résultats encore meilleurs ont été obtenus avec d'autres types plus robustes de cellules, telles que les cellules cancéreuses. En ce qui concerne les cellules gelées à plongée, les images de fluorescence aux rayons X de K, S et Zn sur l'ensemble des cellules lyophilisées sont semblables à celles décrites ci-dessus. Ils sont représentatifs des distributions élémentaires que l'on trouve dans divers types de cellules lyophilisées à une résolution spatiale de 50 à 100 nm. Bien que le lyophilisation des cellules entières soit une alternative pour préserver l'intégrité élémentaire, il est au détriment d'une parfaite préservation de la morphologie cellulaire16, en particulier les membranes cellulaires.

Figure 1 : Support typique de l'échantillon pour la nano-analyse de la fluorescence aux rayons X. Un support de membrane De3N4 dans sa capsule protectrice. Ce type de substrat peut être utilisé à la fois pour l'analyse de la température ambiante (préparation cellulaire de gel-plunge suivie d'un processus de congélation à basse température et sous vide) ou pour l'analyse cryogénique de la fluorescence des rayons X. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
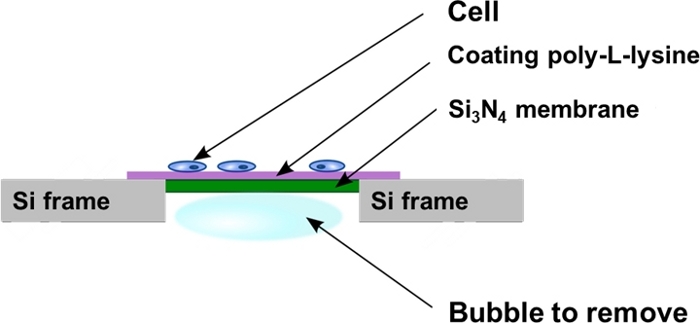
Figure 2 : Vue schématique des fenêtres de nitride de silicium après l'ensemencement cellulaire. Les cellules sont cultivées directement sur la surface plane recouverte de polylysine du support membranaire Si3N4. Parfois, les bulles d'air peuvent être emprisonnées dans la cavité arrière du support membranaire Si3N4 et doivent être enlevées comme décrit dans le protocole. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 3 : Cryo-box imprimé en 3D pour le stockage à long terme des supports à membrane Si3N4 congelés dans l'azote liquide Dewar. (A) Cryo-box démonté avec le récipient et les bouchons (partie inférieure) et (B) la cryo-boîte assemblée avec des bouchons verrouillés. Les bouchons peuvent être manipulés avec les pinces, l'ouverture ou le verrouillage par rotation. Un plan détaillé pour l'impression 3D est disponible sur demande auprès de l'ESRF ID16A. La conception a été faite pour accueillir les grilles tem de nitride de silicium. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 4 : Blotting des cellules cultivées sur Si3N4. Avant de plonger-congélation de la monocouche cellulaire cultivée sur une membrane Si3N4 doit être rincé dans la solution d'acétate d'ammonium (A) et soigneusement effacé manuellement à l'aide de papier filtre (B). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 5 : Machine EM-GP à semi-cverre de plongée automatique. (A) Le congélateur à plongée automatique. (B) Chambre environnementale avec les pinces enfermée. (C) La tasse d'éthane recouverte du liquéfieur Leica relié à une bouteille d'éthane. (D) L'enceinte de congélation de plongée montrant la tasse noire pleine d'éthane liquéfié et la cryo-boîte pour le stockage ultérieur dans LN2 des membranes Vitrified Si3N4. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 6 : Assemblage de cryotransfert d'échantillon pour la procédure de séchage par congélation. (A) Le premier récipient en laiton pour les membranes Si3N4 est monté sur le support de transfert d'échantillon fourni par le fournisseur de sécheuse à congélation. (B) et (C) montrent que le deuxième disque plat en laiton est utilisé comme couverture et agit comme un enclos à froid à insérer dans l'enceinte sous vide de la sécheuse à froid. (D) L'assemblage complet avec la tige de transfert à ressort. (E) Le porte-échantillon portant la préparation cellulaire vitrifiée cultivée sur la membrane Si3N4 doit être inséré davantage dans le séchoir à congélation Refroidi LN2. Toutes les étapes pour monter l'assemblage sont effectuées en LN2 dans une boîte en polystyrène. Pour plus de clarté, toutes les images ont été produites en l'absence de LN2. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.

Figure 7 : Images de fluorescence cryo-x d'une cellule hydratée congelée à l'aide d'une nanosonde à rayons X dur. (A) Vue en ligne typique en mode réflexion à l'aide du microscope vidéo optique dédié de la ligne de faisceau ESRF ID16A. Après le ballonnement manuel, une épaisseur totale de glace d'environ 5 à 10 m a été atteinte, ce qui permet une vue claire des cellules hydratées congelées. Une région avec des anneaux Newton indicatif de glace même beaucoup plus mince est perceptible. (B) Distributions cellulaires cellulaires de fluorescence cryo-x représentativedes des éléments physiologiques potassium (K), soufre (S) et zinc (Zn). Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
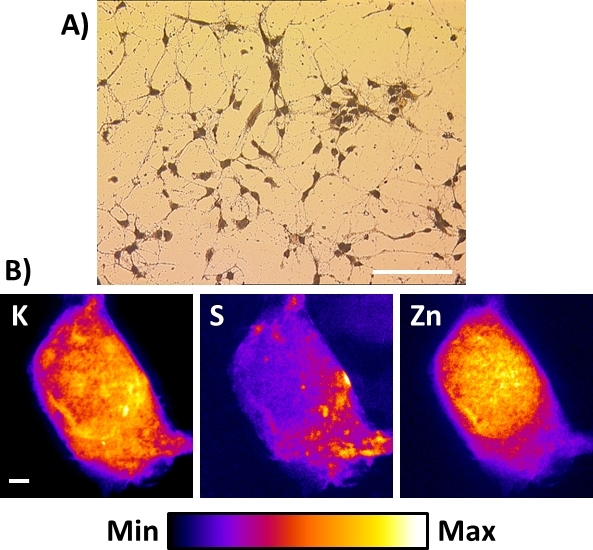
Figure 8 : Images de fluorescence aux rayons X d'une cellule neuronale lyophilisées à l'aide d'une nanosonde à rayons X dure. (A) Vue typique de microscopie de champ lumineux de cellules neuronales corticaux lyophilisées résultantes directement cultivées sur la membrane Si3N4. Barre à l'échelle de 200 m (B) Images représentatives de fluorescence des rayons X de la température ambiante d'un seul neurone hippocampique lyophilisé montrant la distribution d'éléments physiologiques potassium (K), soufre (S) et zinc (Zn). Barre d'échelle de 2 m. Veuillez cliquer ici pour voir une version plus grande de ce chiffre.
Discussion
La microscopie cryo-électron (cryo-EM) a remporté le prix Nobel de chimie 2017 et, à ce titre, le développement de J. Dubochet sur la vitrification du matériel biologique pour la détermination de la structure à haute résolution des biomolécules dans la solution25. Comme indiqué par Dubochet dans sa conférence Nobel "Savoir comment vitrifier une goutte d'eau est une chose, la préparation d'un échantillon biologique pour l'observation biologique en est une autre"25. Les étapes de cryopréparation sont maintenant considérées comme la technique standard pour atténuer les dommages causés par la dose de rayonnement et étudier les cellules proches de leur état d'origine. La préparation reste cependant fastidieuse. C'est parce que la microscopie électronique, en raison de sa résolution spatiale inégalée, est sensible à tout artefact ultrastructural qui se produit pendant la préparation de l'échantillon. Les cryonanoprobes synchrotron s'approchent maintenant de difficultés similaires pour atteindre des résolutions spatiales aussi basses que 13 nm dans la plage de rayons X à haute énergie26. La microscopie à rayons X durs peut analyser des cellules entières tandis que la microscopie électronique souffre de la faible profondeur de pénétration des électrons permettant d'observer uniquement des tranches de cellules très fines.
Les monocouches de cellules sont assez minces pour qu'en plongeant dans l'éthane liquide, les taux de refroidissement requis pour la vitrification de l'eau soient atteints. En théorie, des taux de refroidissement aussi élevés que 108 K/s sont possibles en utilisant le gel à haute pression27 qui permet la vitrification des spécimens trop épais pour le gel de plongée. Un taux de refroidissement de 105 K/s, nécessaire pour permettre la vitrification complète de l'échantillon à la pression ambiante28, est atteint de façon reproductible à l'aide de la machine automatique de congélation de plongée et des paramètres présentés ici. Cela permet à un chercheur de vitrifier de minces spécimens biologiques (lt;10 m) tels qu'une monocouche de cellules12,13,14,15,29,30 par le plongeon-congélation dans l'éthane liquide.
Un défi important avec ce protocole est également de préserver autant que possible l'intégrité chimique du contenu intracellulaire pour fournir des distributions élémentaires fiables dans la cellule en 2D ou 3D. Comme publié ailleurs2,16,17,31, dans le cas de l'imagerie élémentaire au niveau subcellulaire, l'analyse des cellules hydratées congelées devrait être considérée. Dans le cas contraire, la combinaison de la congélation et du séchage des cellules peut être utilisée pour l'analyse de la température ambiante. Pour ce dernier, la glace amorphe est enlevée par le processus de sublimation, tandis que les molécules d'eau liées sont enlevées par le processus de desorption. Ce processus peut être loin d'être idéal par rapport aux échantillons hydratés congelés en raison de l'altération possible des membranes cellulaires et de la morphologie de certaines structures subcellulaires32. En outre, pour les études de spéciation, l'extraction de l'eau peut conduire à des artefacts de spéciation métallique. Pourtant, il a été couronné de succès et la meilleure alternative aux échantillons hydratés congelés pour l'imagerie élémentaire à des niveaux de moins de 100 nm2,16,17,18,20,33,34,35,36.
Comme il a été rapporté37, la qualité des préparations cellulaires cryoconservées peut être évaluée par le rapport potassium-sodium K/Na. Malheureusement, il ne peut pas encore être déterminé avec la nanosonde à rayons X dur utilisé ici, en raison de la faible coupure d'énergie du détecteur de dérive de silicium utilisé pour détecter les photons de fluorescence des rayons X des éléments (E 1,3 keV magnésium). En effet, un ratio K/Na élevé (-10) qui peut être mesuré à l'aide de TOF-SIMS, EPMA, ou microprobe nucléaire PIXE16,37 est révélateur de l'intégrité chimique préservée de la cellule par rapport à la K / Na prévu de 25 dans une cellule vivante37. Cela peut être soutenu par un ratio Cl/K faible concomitante38. Pourtant, la vitrification imparfaite, en particulier si la vitesse de refroidissement de l'échantillon est trop faible, peut conduire à la formation de grands cristaux de glace qui peuvent endommager les membranes cellulaires et les organites, modifiant ainsi la distribution des éléments chimiques. Bien qu'il n'y ait pas de procédure courante pour surveiller ces dommages potentiels et l'impact sur la distribution intracellulaire, les rapports élémentaires ci-dessus et la possibilité d'imager la cellule à haute résolution en utilisant le contraste de phase de rayons X ou la microscopie cryo-douce de rayon X peuvent être les meilleures approches pour soutenir la bonne conservation des compartiments intracellulaires avec la conservation concomitante de l'intégrité élémentaire. La combinaison de ces techniques et l'utilisation de microscopes optiques à fluorescence cryocorrelative nouvellement développés aideront à évaluer dans quelle mesure ces dommages se produisent et affectent la distribution élémentaire intracellulaire.
Dans l'ensemble, un protocole détaillé et complet pour préparer des échantillons cellulaires pour la nano-analyse de fluorescence des rayons X synchrotron est présenté. C'est un bon point de départ pour la communauté de la recherche, aidant à résoudre la question difficile de la façon de préparer des échantillons cellulaires appropriés pour l'imagerie élémentaire 2D et 3D à (cryo) nanosondes à rayons X durs. Ces approches peuvent être fusionnées avec des capacités de fluorescence optique et de microscopie électronique pour l'imagerie chimique et structurale corrélative en profondeur des cellules.
Déclarations de divulgation
Les auteurs n'ont aucun conflit d'intérêts.
Remerciements
Les expériences sur la ligne de faisceau de nano-imagerie ID16A ont été réalisées dans le cadre des propositions ESRF LS2430, LS2303 et LS2765.
matériels
| Name | Company | Catalog Number | Comments |
| Ammonium Acetate solution, BioUltra, for molecular biology, ~5M in H2O | SIGMA | 09691-250mL | One can prepare the required solution from high-grade ammonium acetate powder and ultrapure water, pH and osmolarity needs to be adjusted anyway. |
| B27 supplement, 50x | Life Technologies, Invitrogen | 17504-044 | for hippocampal neuron culture |
| Dulbecco’s Phosphate Buffered Saline, DPBS, ([-] CaCl2, [-] MgCl2) | GIBCO | 14190-094 | cell culture |
| DMEM with Phenol Red/Glutamax I (Medium ATCC modification) | GIBCO | 21885025 | cell culture |
| Dulbecco’s modified Eagle medium (DMEM) | Life Technologies, Invitrogen | 31966-02 | for hippocampal neuron culture |
| Dumont Tweezers #5, Straight Self-closing, 0.05x0.01mm Tips, Biology | World Precision Instrument | 501202 | |
| Emitech K750X Peltier-Cooled EM Freeze Dryer | Quorum Technology | EK3147 | |
| Ethane N45 | Air Liquid | p0505s05r0a001 | C2H6 > 99,995 % |
| Fetal Bovine Serum, Performance Plus, certified One Shot format, US origin | GIBCO | A31604-02 | cell culture |
| HBSS 10x | Life Technologies, Invitrogen | 14185-052 | for hippocampal neuron culture |
| Leica GP quick-release forceps | Leica | 16706435 | |
| MDA-MB-231 cell line, an epithelial, human adenocarcinoma breast cancer cell | ATCC | ATCC HTB-26 | cell culture |
| Neurobasal medium | Life Technologies, Invitrogen | 21103-049 | for hippocampal neuron culture |
| Nunc 4-Well Plate | Thermo Fisher | 176740 | cell culture |
| Osmo1 Single-Sample Micro-Osmometer | Advanced Instruments | Osmo1 | Alternative can be found at Fisher scientific (Wescor Inc. VAPRO® Vapor Pressure Osmometer) |
| Penicillin-Streptomycin | SIGMA | P4333 | cell culture |
| poly-L-lysine | SIGMA | P4707 | Other type of coating can be used that is dependent of the cell type to be cultured on the membrane, other adhesion factors such as fibronectin, collagen, polyornithine can be tested accordingly. Cell can be cultured directly on silicon nitride membrane, but the latter are slightly hydrophobic and adhesion factors are recommended unless the membrane are processed to be hydrophilic (glow plasma discharged). |
| Plunge freezing robot Leica EM GP main unit | Leica | 16706401 | Alternative for automated plunger are the Vitrobot Mark IV (FEI), CryoPlunge 3 (Gatan), MS-002 Rapid Immersion Freezer (EMS). Manual home-made system can be used but an environment-controlled chamber is an asset for plunge-freezing. |
| Silicon nitride membrane (Si3N4) | Silson Ltd. | SiRN-5.0(o)-200-1.5-500-NoHCl | The proposed silicon nitride membrane type is optimised for analysis at ID16A ESRF X-ray nanoprobe, The 500 nm thickness of the membrane was chosen being more robust for cellular manipulation and cryofixation detailed within this protocol. Membrane with thickness of 200 nm or below can also be used although quite fragile, and other design of silicon nitride membrane can be purchased (for example TEM compatible membrane...) from Sislon or other company such as Norcada, SPI supplies, Ted Pella, EMS, LabTech, Neyco... |
| Trypan blue solution 0.4% | GIBCO | 15250061 | cell culture |
| Trypsin-EDTA, 0.05% | GIBCO | 25300-054 | cell culture |
| Ultratrace Elemental Analysis Grade, Ultrapure Water | Fisher Chemicals | W9-1 | MilliQ water can be used but has to be tested for trace element level of contamination using for example ICP-MS analysis. |
| Whatman No. 1 filter paper with precut hole | Leica | 16706440 | Alternative filter paper may be used and must have an outer diameter of 55 mm, the Punch for filter paper system from Leica (ref.16706443) can be used. |
Références
- Lewis, D. J., et al. Intracellular synchrotron nanoimaging and DNA damage/genotoxicity screening of novel lanthanide-coated nanovectors. Nanomedicine. 5 (10), 1547-1557 (2010).
- Fus, F., et al. The intracellular localization of osmocenyl-tamoxifen derivatives in hormone-independent breast cancer cells revealed by 2D and 3D nano X-ray fluorescence imaging. Angwendte Chemie. , (2019).
- Janssens, K., Adams, F., Rindby, A. Microscopic X-Ray Fluorescence Analysis. , Wiley. Chichester, UK. (2000).
- Carmona, A., et al. Uranium exposure of human dopaminergic cells results in low cytotoxicity, accumulation within sub-cytoplasmic regions, and down regulation of MAO-B. Neurotoxicology. 68, 177-188 (2018).
- Leapman, R. D., Hunt, J. A., Buchanan, R. A., Andrews, S. B. Measurement of low calcium concentrations in cryosectioned cells by parallel-EELS mapping. Ultramicroscopy. 49 (1-4), 225-234 (1993).
- Saubermann, A. J., Echlin, P., Peters, P. D., Beeuwkes, R. Application of scanning electron microscopy to X-ray analysis of frozen hydrated sections. I. Specimen handling techniques. Journal of Cell Biology. 88 (2), 257-267 (1981).
- Saubermann, A. J., Heyman, R. V. Quantitative digital X-ray imaging using frozen hydrated and frozen dried tissue sections. Journal of Microscopy. 146, Pt2 169-182 (1987).
- Wroblewski, J., Roomans, G. M. X-ray microanalysis of single and cultured cells. Scanning Electron Microscopy. , Pt 4 1875-1882 (1984).
- Wroblewski, J., Müller, R. M., Wroblewski, R., Roomans, G. M. Quantitative X-ray microanalysis of semi-thick cryosections. Histochemistry. 77 (4), 447-463 (1983).
- Zierold, K. Cryopreparation of mammalian tissue for X-ray microanalysis in STEM. Journal of Microscopy. 125, Pt2 149-156 (1982).
- Harkiolaki, M., et al. Cryo-soft X-ray tomography: Using soft X-rays to explore the ultrastructure of whole cells. Emerging Topics in Life Sciences. 2 (1), 81-92 (2018).
- McDermott, G., Le Gros, M. A., Knoechel, C. G., Uchida, M., Larabell, C. A. Soft X-ray tomography and cryogenic light microscopy: the cool combination in cellular imaging. Trends in Cell Biology. 19 (11), 587-595 (2009).
- Schneider, G., et al. Three-dimensional cellular ultrastructure resolved by X-ray microscopy. Nature Methods. 7 (12), 985-987 (2010).
- Sorrentino, A., et al. MISTRAL: a transmission soft X-ray microscopy beamline for cryo nano-tomography of biological samples and magnetic domains imaging. Journal of Synchrotron Radiation. 22 (4), 1112-1117 (2015).
- Carzaniga, R., Domart, M. C., Duke, E., Collinson, L. M. Correlative cryo-fluorescence and cryo-soft X-ray tomography of adherent cells at European synchrotrons. Methods in Cell Biology. 124, Academic Press. 151-175 (2014).
- Perrin, L., Carmona, A., Roudeau, S., Ortega, R. Evaluation of sample preparation methods for single cell quantitative elemental imaging using proton or synchrotron radiation focused beams. Journal of Analytical Atomic Spectrometry. 30 (12), 2525-2532 (2015).
- Jin, Q., et al. Preserving elemental content in adherent mammalian cells for analysis by synchrotron-based x-ray fluorescence microscopy. Journal of Microscopy. 265 (1), 81-93 (2017).
- Daoust, A., et al. Impact of manganese on primary hippocampal neurons from rodents. Hippocampus. 24 (5), 598-610 (2014).
- Daoust, A., et al. Manganese Cytotoxicity Assay on Hippocampal Neuronal Cell Culture. Bio-protocol. 5 (1), 1368(2015).
- Gibon, J., et al. The over-expression of TRPC6 channels in HEK-293 cells favours the intracellular accumulation of zinc. Biochimica et Biophysica Acta (BBA)-Biomembranes. 1808 (12), 2807-2818 (2011).
- Hasna, J., Bohic, S., Lemoine, S., Blugeon, C., Bouron, A. Zinc Uptake and Storage During the Formation of the Cerebral Cortex in Mice. Molecular Neurobiology. , 1-13 (2019).
- Villar, F., et al. Nanopositioning for the ESRF ID16A Nano-Imaging Beamline. Synchrotron Radiation News. 31 (5), 9-14 (2018).
- Kosior, E., et al. Combined use of hard X-ray phase contrast imaging and X-ray fluorescence microscopy for subcellular metal quantification. Journal of Structural Biology. 177 (2), 239-247 (2012).
- Bohic, S., et al. Synchrotron hard X-ray microprobe: fluorescence imaging of single cells. Applied Physics Letters. 78, 3544-3546 (2001).
- Dubochet, J. On the Development of Electron Cryo-Microscopy (Nobel Lecture). Angwendte Chemie. 57 (34), 10842-10846 (2018).
- Da Silva, J. C., et al. Efficient concentration of high-energy X-rays for diffraction-limited imaging resolution. Optica. 4 (5), 492-495 (2017).
- Studer, D., Humbel, B. M., Chiquet, M. Electron microscopy of high-pressure frozen samples: bridging the gap between cellular ultrastructure and atomic resolution. Histochemistry and Cell Biology. 130 (5), 877-889 (2008).
- Moor, H. Theory and pratice of high pressure freezing. Cryotechniques in Biological Electron Microscopy. Steinbrecht, R. A., Zierold, K. , Springer. 175-191 (1987).
- Gilkey, J. C., Staehelin, L. A. Advances in ultrarapid freezing for the preservation of cellular ultrastructure. Journal of Electron Microscopy Techniques. 3 (2), 177-210 (1986).
- Ferreira, J. L., Matthews-Palmer, T. R., Beeby, M. Electron Cryo-Tomography. Cellular Imaging. , Springer, Cham. 61-94 (2018).
- Colvin, R. A., Jin, Q., Lai, B., Kiedrowski, L. Visualizing metal content and intracellular distribution in primary hippocampal neurons with synchrotron X-ray fluorescence. PLoS One. 11 (7), 0159582(2016).
- Vavpetič, P., et al. Elemental distribution and sample integrity comparison of freeze-dried and frozen-hydrated biological tissue samples with nuclear microprobe. Nuclear Instruments and Methods in Physics Research Section B: Beam Interactions with Materials and Atoms. 348, 147-151 (2015).
- Guerquin-Kern, J. L., Bordat, C. Cryo-preparation procedures for elemental imaging by sims and eftem. Handbook of Cryo-Preparation Methods for Electron Microscopy. , CRC Press. 499-536 (2008).
- Gramaccioni, C., et al. Nanoscale quantification of intracellular element concentration by X-ray fluorescence microscopy combined with X-ray phase contrast nanotomography. Applied Physics Letters. 112 (5), 053701(2018).
- Perrin, L., et al. Zinc and Copper Effects on Stability of Tubulin and Actin Networks in Dendrites and Spines of Hippocampal Neurons. ACS Chemical Neurosciences. 8 (7), 1490-1499 (2017).
- Ortega, R., et al. α-synuclein over-expression induces increased iron accumulation and redistribution in iron-exposed neurons. Molecular Neurobiology. 53 (3), 1925-1934 (2016).
- Fartmann, M., et al. Quantitative imaging of atomic and molecular species in cancer cultures with TOF-SIMS and Laser-SNMS. Applied Surface Sciences. 231 (2), 428-431 (2004).
- Pålsgård, E., Lindh, U., Roomans, G. M. Comparative study of freeze-substitution techniques for X-ray microanalysis of biological tissue. Microscopy Research and Techniques. 28 (3), 254-258 (1994).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon