Un abonnement à JoVE est nécessaire pour voir ce contenu. Connectez-vous ou commencez votre essai gratuit.
Method Article
Extraction et visualisation d’agrégats protéiques après traitement d’Escherichia coli avec un facteur de stress protéotoxique
Dans cet article
Résumé
Ce protocole décrit l’extraction et la visualisation de protéines agrégées et solubles d’Escherichia coli après traitement avec un antimicrobien protéotoxique. Suivre cette procédure permet une comparaison qualitative de la formation d’agrégats protéiques in vivo dans différentes souches bactériennes et/ou entre les traitements.
Résumé
L’exposition des organismes vivants à des stress environnementaux et cellulaires provoque souvent des perturbations dans l’homéostasie des protéines et peut entraîner une agrégation des protéines. L’accumulation d’agrégats de protéines dans les cellules bactériennes peut entraîner des altérations significatives du comportement phénotypique cellulaire, notamment une réduction des taux de croissance, de la résistance au stress et de la virulence. Plusieurs procédures expérimentales existent pour l’examen de ces phénotypes médiés par les facteurs de stress. Cet article décrit un essai optimisé pour l’extraction et la visualisation de protéines agrégées et solubles de différentes souches d’Escherichia coli après traitement avec un antimicrobien contenant de l’argent-ruthénium. Ce composé est connu pour générer des espèces réactives de l’oxygène et provoque une agrégation protéique généralisée.
La méthode combine une séparation basée sur la centrifugation des agrégats de protéines et des protéines solubles des cellules traitées et non traitées avec une séparation et une visualisation ultérieures par électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide (SDS-PAGE) et coloration de Coomassie. Cette approche est simple, rapide et permet une comparaison qualitative de la formation d’agrégats protéiques dans différentes souches d’E. coli. La méthodologie a un large éventail d’applications, y compris la possibilité d’étudier l’impact d’autres antimicrobiens protéotoxiques sur l’agrégation in vivo des protéines dans un large éventail de bactéries. De plus, le protocole peut être utilisé pour identifier les gènes qui contribuent à une résistance accrue aux substances protéotoxiques. Les bandes de gel peuvent être utilisées pour l’identification ultérieure des protéines particulièrement sujettes à l’agrégation.
Introduction
Les bactéries sont inévitablement exposées à une myriade de stress environnementaux, y compris un faible pH (par exemple, dans l’estomac des mammifères)1,2, des espèces réactives de l’oxygène et du chlore (ROS / RCS) (par exemple, lors d’une explosion oxydative dans les phagocytes)3,4,5, des températures élevées (par exemple, dans les sources chaudes ou pendant un choc thermique)6,7, et plusieurs antimicrobiens puissants (par exemple, AGXX utilisé dans ce protocole)8. Les protéines sont particulièrement vulnérables à l’un de ces facteurs de stress, et l’exposition peut provoquer un dépliage / un mauvais repliement des protéines qui s’agréger ensuite. Tous les organismes emploient des systèmes de protection qui leur permettent de faire face au mauvais repliement des protéines9. Cependant, un stress sévère peut submerger la machinerie de contrôle de la qualité des protéines et perturber la structure secondaire et / ou tertiaire des protéines, ce qui finit par inactiver les protéines. En conséquence, les agrégats de protéines peuvent gravement altérer les fonctions cellulaires critiques nécessaires à la croissance et à la survie bactériennes, à la résistance au stress et à la virulence10. Par conséquent, la recherche axée sur l’agrégation des protéines et ses conséquences sur les bactéries est un sujet pertinent en raison de son impact potentiel sur le contrôle des maladies infectieuses.
Le déploiement et l’agrégation des protéines induites par la chaleur sont souvent réversibles7. En revanche, d’autres stress protéotoxiques, tels que le stress oxydatif, peuvent provoquer des modifications irréversibles des protéines par l’oxydation de chaînes latérales d’acides aminés spécifiques, entraînant un dépliage / mauvais repliement des protéines et, éventuellement, une agrégation des protéines4. La formation induite par le stress d’agrégats protéiques insolubles a été largement étudiée dans le contexte des chaperons moléculaires et de leurs fonctions protectrices chez les levures et les bactéries11,12,13. Plusieurs protocoles ont été publiés qui utilisent une variété de techniques biochimiques pour l’isolement et l’analyse des agrégats de protéines insolubles14,15,16,17. Les protocoles existants ont principalement été utilisés pour étudier l’agrégation des protéines bactériennes lors d’un choc thermique et/ou l’identification de chaperons moléculaires. Bien que ces protocoles aient certainement été une avancée dans le domaine, il y a quelques inconvénients majeurs dans les procédures expérimentales car ils nécessitent (i) un grand volume de culture bactérienne allant jusqu’à10 L 14,17, (ii) des processus de perturbation physique compliqués, y compris l’utilisation de perturbateurs cellulaires, Français presse et / ou sonication14,15,17, ou (iii) des répétitions chronophages étapes de lavage et d’incubation15,16,17.
Cet article décrit un protocole modifié qui vise à remédier aux limites des approches précédentes et permet l’analyse de la quantité d’agrégats de protéines formés dans deux souches différentes d’Escherichia coli après traitement avec un revêtement de surface antimicrobien protéotoxique. Le revêtement est composé de métal-argent (Ag) et de ruthénium (Ru) conditionnés avec de l’acide ascorbique, et son activité antimicrobienne est obtenue par la génération d’espèces réactives de l’oxygène8,18. On trouvera ici une description détaillée de la préparation de la culture bactérienne après traitement avec le composé antimicrobien et une comparaison de l’état d’agrégation des protéines lors de l’exposition de deux souches d’E. coli présentant des profils de sensibilité distincts à l’augmentation de la concentration de l’antimicrobien. La méthode décrite est peu coûteuse, rapide et reproductible et peut être utilisée pour étudier l’agrégation des protéines en présence d’autres composés protéotoxiques. En outre, le protocole peut être modifié pour analyser l’impact des délétions de gènes spécifiques sur l’agrégation des protéines dans une variété de bactéries différentes.
Protocole
1. Traitement du stress des souches d’E. coli MG1655 et CFT073
- Inoculer 5 mL de bouillon de lysogénie (LB) avec une seule colonie de souche commensale d’E. coli MG1655 et de souche uropathogène E. coli (UPEC) CFT073, respectivement, et incuber pendant 14 à 16 h (pendant la nuit) à 37 °C et 300 tr/min.
REMARQUE: Escherichia coli CFT073 est un agent pathogène humain. La manipulation du CFT073 doit être effectuée avec des mesures de biosécurité appropriées dans un laboratoire certifié de niveau de biosécurité 2. - Diluer chaque souche dans une fiole de 500 mL contenant70 mL de 3-(N-morpholino)acide propanesulfonique (MOPS)-glucose (MOPS-g)(tableau 1)à une densité optique à 600 nm (OD600)valeur de 0,1. Incuber à 37 °C et 300 tr/min jusqu’à ce que la phase médiane soit atteinte (OD600 = 0,5-0,55).
- Transférer 20 mL de chaque culture dans trois flacons pré-avertis de 125 mL et incuber à 37 °C et 300 tr/min pendant 2 min.
REMARQUE: Comme le traitement rapide des échantillons est nécessaire, ne manipulez pas plus de 6 cultures à la fois. - Préparer une solution de composé antimicrobien dans un milieu MOPS-g à une concentration de 2 mg/mL. Ajouter l’antimicrobien à chaque culture pour atteindre les concentrations indiquées. Pour le contrôle non traité, ajoutez le volume requis de milieu MOPS-g.
REMARQUE: Vortex la solution antimicrobienne de 2 mg / mL pour permettre une distribution uniforme des particules composées et éviter la sédimentation. - Incuber les cultures pendant 45 min à 37 °C et 300 tr/min.
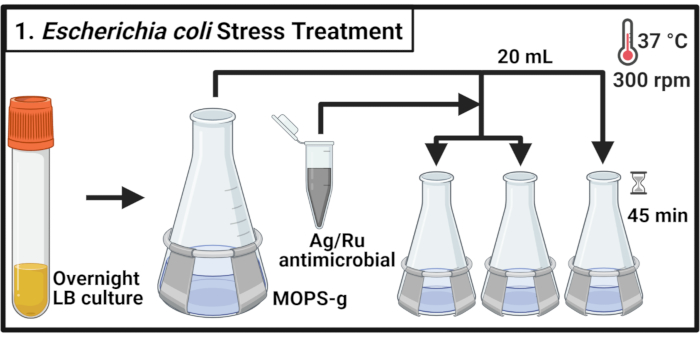
Figure 1: Traitement du stress d’Escherichia coli. Les cultures bactériennes sont cultivées dans MOPS-g et traitées avec les concentrations indiquées de l’antimicrobien contenant de l’argent-ruthénium lorsque la phase mi-logarithmique est atteinte. Abréviations : LB = bouillon de lysogénie ; Ag-Ru = argent-ruthénium; MOPS-g = 3-( N-morpholino)acide propanesulfonique (MOPS)-glucose. Veuillez cliquer ici pour voir une version agrandie de cette figure.
2. Prélèvement d’échantillons de cellules bactériennes
- Après 45 min de traitement du stress, déterminer l’OD600 de chaque culture. Pour chaque échantillon, récolter des cellules équivalant à 4 mL de DO600 = 1 dans des tubes centrifuges de 15 mL par centrifugation pendant 15 min à 3 000 × g et 4 °C.
- Retirer complètement le surnageant et ressusser les pastilles cellulaires dans 50 μL de tampon de lyse glacée (Tableau 1). Incuber les échantillons pendant 30 min sur de la glace.
REMARQUE: Cette étape de lyse dégrade la couche de peptidoglycane. Utilisez toujours un tampon de lyse fraîchement préparé. - Transférer les échantillons dans des tubes de microcentrifugation de 1,7 mL. Congeler à -80 °C jusqu’à une utilisation ultérieure.
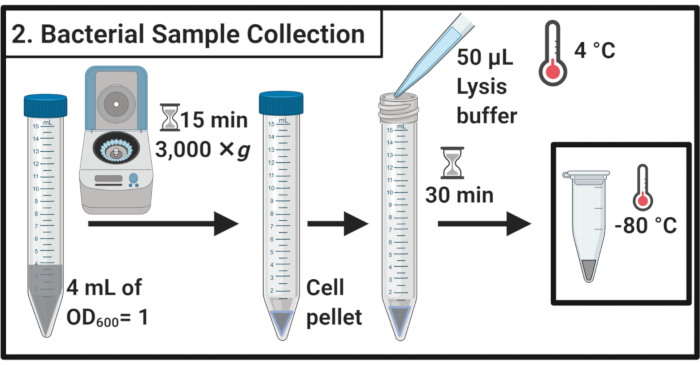
Figure 2: Prélèvement d’échantillons bactériens. Les échantillons de cellules sont prélevés par centrifugation et mis en caisse dans un tampon de lyse suivi d’un stockage à -80 °C. Veuillez cliquer ici pour voir une version plus grande de cette figure.
3. Extraction des agrégats de protéines insolubles
- Décongeler des échantillons sur la glace.
REMARQUE: Le cycle de gel-dégel contribue à la lyse cellulaire. - Ajouter 360 μL de tampon glacé A(tableau 1)et mélanger doucement par pipetage.
REMARQUE: Le choc osmotique contribuera également à la lyse cellulaire. - Transférer l’échantillon dans un tube de microcentrifugation de 2 mL contenant environ 200 μL de billes de verre de 0,5 mm. Incuber pendant 30 min à 8 °C dans un thermomixeur en agitant à 1 400 tr/min.
REMARQUE: Cette étape entraîne la perturbation physique de la cellule. Un inhibiteur de la protéase peut être utilisé pour minimiser la dégradation des protéines. La perturbation peut être effectuée à 4 °C. Notez que l’utilisation de billes de verre a été rapportée pour induire l’agrégation d’un petit sous-ensemble de protéines dans la levure19. - Incuber pendant 5 min sur de la glace sans secouer pour déposer les perles de verre. Transférer 200 μL de lysate cellulaire dans des tubes de microcentrifugation de 1,7 mL.
REMARQUE: Évitez le transfert des perles de verre. - Centrifuger à 16 000 × g et 4 °C pendant 20 min. Recueillir le surnageant, qui contient des protéines solubles, et passer à la rubrique 4.
- Resuspendez la pastille dans 200 μL de tampon glacé A (Tableau 1) à l’aide de la pipette. Centrifuger à 16 000 × g et 4 °C pendant 20 min. Retirez soigneusement le surnageant.
- Ajouter 200 μL de tampon glacé B (voir le tableau 1 et le tableau des matériaux)et resuspendez soigneusement la pastille par pipetage.
REMARQUE: Le détergent non ionique solubilise les protéines membranaires. - Répéter la centrifugation à 16 000 × g et 4 °C pendant 20 min. Retirez soigneusement le surnageant.
- Resuspendez la pastille dans 200 μL de tampon froid A (Tableau 1) par pipetage. Centrifuger à 16 000 × g et 4 °C pendant 20 min. Retirez complètement le surnageant.
- Resuspendez la pastille dans 100 μL de 1x tampon d’échantillon SDS réducteur (Tableau 1) et faites bouillir pendant 5 min à 95 °C dans un thermomélangeur.
- Conservez l’échantillon à -20 °C pour procéder ultérieurement ou chargez immédiatement sur un gel de polyacrylamide SDS pour la séparation.
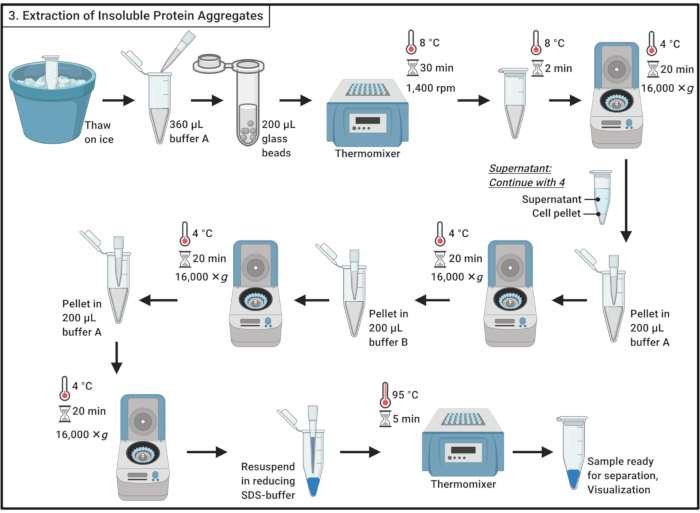
Figure 3: Extraction d’agrégats protéiques insolubles. L’extraction des agrégats protéiques implique une série d’étapes, y compris la perturbation cellulaire, la séparation des agrégats protéiques des protéines solubles, la solubilisation des protéines membranaires et le lavage. Abréviation : FDS = dodécylsulfate de sodium. Veuillez cliquer ici pour voir une version agrandie de cette figure.
4. Préparation d’échantillons de protéines solubles
- Mélanger 1 volume d’acide trichloroacétique (TCA) à 100 % avec 4 volumes d’échantillon de protéines solubles de l’étape 3.6.
REMARQUE : La manipulation du TCA nécessite une hotte aspirante et un équipement de protection individuelle ainsi qu’une procédure approuvée d’élimination des déchets. - Incuber pendant 10 min à 4 °C pour permettre la précipitation des protéines.
REMARQUE: Le précipité blanc apparaîtra très bientôt. - Centrifuger pour précipiter à 21 000 × g et 4 °C pendant 5 min et retirer le surnageant. Lavez la pastille avec 200 μL d’acétone glacée pour éliminer les débris cellulaires. Centrifuger à 21 000 × g et 4 °C pendant 5 min et retirer le surnageant. Répétez ces actions à l’étape 4.3 trois fois au total.
- Placez les tubes de microcentrifugation à couvercle ouvert dans un thermomélangeur à 37 °C pour retirer l’acétone restante de la pastille.
REMARQUE: Une incubation de plus de 5 minutes peut réduire la solubilité de la pastille de protéine. - Ajouter 100 μL de tampon SDS réducteur 1x(tableau 1)et dissoudre complètement la pastille. Faire bouillir l’échantillon pendant 5 min à 95 °C.
- Conserver l’échantillon à -20 °C pour procéder ultérieurement ou charger immédiatement sur un gel de polyacrylamide FDS pour la séparation.
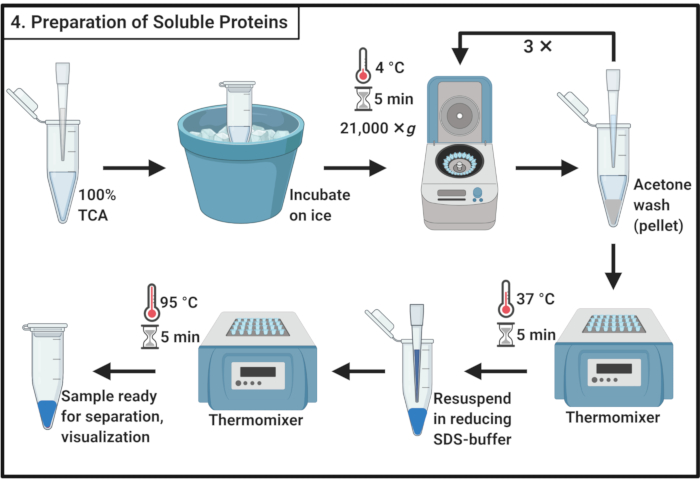
Figure 4: Préparation des protéines solubles. La préparation de protéines solubles implique une étape de précipitation avec de l’acide trichloroacétique et un lavage répété avec de l’acétone glacée. Abréviations : TCA = acide trichloroacétique; FDS = dodécylsulfate de sodium. Veuillez cliquer ici pour voir une version agrandie de cette figure.
5. Séparation et visualisation des agrégats de protéines extraites à l’aide de SDS-PAGE
- Préparez un gel de polyacrylamide SDS à 12 %.
- Pour deux gels séparateurs, pipeter 5,1 mL d’eau double distillée (ddH2O), 3,75 mL de Tris-HCl (pH 8,8), 7,5 mL de SDS à 20 % (p/v), 6 mL de solution d’acrylamide/bisacrylamide à 30 % (29:1), 75 mL de persulfate d’ammonium à 10 % p/v et 10 mL de tétraméthyléthylènediamine (TEMED) dans un tube de centrifugeuse de 15 mL et mélanger doucement sans introduire de bulles d’air. Versez le gel à l’aide d’une pipette de 1 mL dans les plaques de verre, en laissant les 2 cm supérieurs libres du mélange. Ajouter 70% d’éthanol sur le dessus du gel séparateur et permettre une interface uniforme entre les deux couches.
- Après polymérisation du gel séparateur, préparer le gel empilable en pipetant 1,535 mL deddH2O, 625 mL de Tris-HCl (pH 6,8), 12,5 mL de FDS à 20 % (p/v), 335 mL de solution d’acrylamide/bisacrylamide à 30 % (29:1), 12,5 mL de persulfate d’ammonium à 10 % p/v et 2,5 mL de TEMED. Retirez l’éthanol des gels séparateurs et ajoutez la solution de gel empilable. Insérez un peigne avec le nombre de poches souhaité sans introduire de bulles d’air. Laisser la polymérisation pendant 20-30 min.
- Chargez 4 μL de chaque échantillon et échelle protéique dans des puits séparés et faites couler le(s) gel(s) dans un tampon de tris-glycine(tableau1) à 144 V pendant 45 minutes à température ambiante.
REMARQUE: Arrêtez le gel lorsque la bande de bromophénol est sur le point de migrer hors du gel. - Tacher le(s) gel(s) dans une solution Fairbanks pré-avertie A (Tableau 1) pendant 30 min sur une bascule.
- Décolorer le(s) gel(s) dans une solution Fairbanks pré-pré-avertie D (Tableau 1) jusqu’à l’arrière-plan souhaité (par exemple, pendant la nuit) sur une bascule.
Figure 5: Séparation et visualisation des protéines. Les échantillons sont séparés par SDS-PAGE et visualisés par coloration de Coomassie. Abréviation : SDS-PAGE = électrophorèse sur gel de dodécylsulfate de sodium-polyacrylamide. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
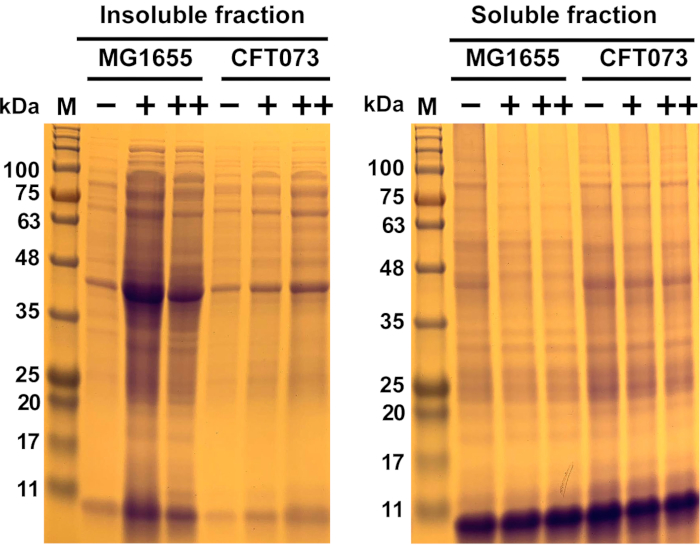
Figure 6: Résultats représentatifs de l’agrégation protéique induite par les antimicrobiens dans la souche commensale Mg1655 d’Escherichia coli et la souche CFT073 de l’UPEC. Les souches d’E. coli MG1655 et CFT073 ont été cultivées à 37 °C et de 300 tr/min àOD 600= 0,5-0,55 dans un milieu MOPS-g avant d’être t...
Discussion
Ce protocole décrit une méthodologie optimisée pour l’analyse de la formation d’agrégats protéiques après traitement de différentes souches d’E. coli avec un antimicrobien protéotoxique. Le protocole permet l’extraction simultanée de fractions protéiques insolubles et solubles à partir de cellules E. coli traitées et non traitées. Par rapport aux protocoles existants pour l’isolement des agrégats de protéines à partir des cellules14,
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail a été soutenu par les fonds de démarrage de l’Illinois State University School of Biological Sciences, l’Illinois State University New Faculty Initiative Grant et la subvention NIAID R15AI164585 (à J.-U. D.). G.M.A. a été soutenu par l’Illinois State University Undergraduate Research Support Program (à G.M.A.). K. P. H. a été soutenu par une bourse RISE fournie par le Service allemand d’échanges universitaires (DAAD). Les auteurs remercient le Dr Uwe Landau et le Dr Carsten Meyer de Largentech Vertriebs GmbH pour avoir fourni la poudre AGXX. Les figures 1, Figure 2, Figure 3, Figure 4et Figure 5 ont été générées avec Biorender.
matériels
| Name | Company | Catalog Number | Comments |
| Chemicals/Reagents | |||
| Acetone | Fisher Scientific | 67-64-1 | |
| 30% Acrylamide/Bisacrylamide solution 29:1 | Bio-Rad | 1610156 | |
| Ammonium persulfate | Millipore Sigma | A3678-100G | |
| Benzonase nuclease | Sigma | E1014-5KU | |
| Bluestain 2 Protein ladder, 5-245 kDa | GoldBio | P008-500 | |
| β-mercaptoethanol | Millipore Sigma | M6250-100ML | |
| Bromophenol blue | GoldBio | B-092-25 | |
| Coomassie Brilliant Blue R-250 | MP Biomedicals LLC | 821616 | |
| D-Glucose | Millipore Sigma | G8270-1KG | |
| D-Sucrose | Acros Organics | 57-50-1 | |
| Ethylenediamine tetra acetic acid (EDTA) | Sigma-Aldrich | SLBT9686 | |
| Glacial Acetic acid | Millipore Sigma | ARK2183-1L | |
| Glycerol, 99% | Sigma-Aldrich | G5516-1L | |
| Glycine | GoldBio | G-630-1 | |
| Hydrochloric acid, ACS reagent | Sigma-Aldrich | 320331-2.5L | |
| Isopropanol (2-Propanol) | Sigma | 402893-2.5L | |
| LB broth (Miller) | Millipore Sigma | L3522-1KG | |
| LB broth with agar (Miller) | Millipore Sigma | L2897-1KG | |
| Lysozyme | GoldBio | L-040-25 | |
| 10x MOPS Buffer | Teknova | M2101 | |
| Nonidet P-40 | Thomas Scientific | 9036-19-5 | |
| Potassium phosphate, dibasic | Sigma-Aldrich | P3786-1KG | |
| Potassium phosphate, monobasic | Acros Organics | 7778-77-0 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771-500G | |
| Tetramethylethylenediamine (TEMED) | Millipore Sigma | T9281-50ML | |
| Thiamine | Sigma-Aldrich | T4625-100G | |
| 100% Trichloroacetic acid | Millipore Sigma | T6399-100G | |
| Tris base | GoldBio | T-400-1 | |
| Material/Equipment | |||
| Centrifuge tubes (15 mL) | Alkali Scientific | JABG-1019 | |
| Erlenmeyer flask (125 mL) | Carolina | 726686 | |
| Erlenmeyer flask (500 mL) | Carolina | 726694 | |
| Freezer: -80 °C | Fisher Scientific | ||
| Glass beads (0.5 mm) | BioSpec Products | 1107-9105 | |
| Microcentrifuge | Hermle | Z216MK | |
| Microcentriguge tubes (1.7 mL) | VWR International | 87003-294 | |
| Microcentriguge tubes (2.0 mL) | Axygen Maxiclear Microtubes | MCT-200-C | |
| Plastic cuvettes | Fischer Scientific | 14-377-012 | |
| Power supply | ThermoFisher Scientific | EC105 | |
| Rocker | Alkali Scientific | RS7235 | |
| Shaking incubator (37 °C) | Benchmark Scientific | ||
| Small glass plate | Bio-Rad | 1653311 | |
| Spacer plates (1 mm) | Bio-Rad | 1653308 | |
| Spectrophotometer | Thermoscientific | 3339053 | |
| Tabletop centrifuge for 15 mL centrifuge tubes | Beckman-Coulter | ||
| Vertical gel electrophoresis chamber | Bio-Rad | 1658004 | |
| Vortexer | Fisher Vortex Genie 2 | 12-812 | |
| Thermomixer | Benchmark Scientific | H5000-HC | |
| 10 well comb | Bio-Rad | 1653359 |
Références
- Dahl, J. -. U., et al. HdeB functions as an acid-protective chaperone in bacteria. Journal of Biological Chemistry. 290 (1), 65-75 (2015).
- Foit, L., George, J. S., Zhang, B. W., Brooks, C. L., Bardwell, J. C. A. Chaperone activation by unfolding. Proceedings of the National Academy of Sciences of the United States of America. 110 (14), 1254-1262 (2013).
- Sultana, S., Foti, A., Dahl, J. -. U. Bacterial defense systems against the neutrophilic oxidant hypochlorous acid. Infection and Immunity. 88 (7), 00964 (2020).
- Dahl, J. -. U., Gray, M. J., Jakob, U. Protein quality control under oxidative stress conditions. Journal of Molecular Biology. 427 (7), 1549-1563 (2015).
- Groitl, B., Dahl, J. -. U., Schroeder, J. W., Jakob, U. Pseudomonas aeruginosa defense systems against microbicidal oxidants. Molecular Microbiology. 106 (3), 335-350 (2017).
- Casadevall, A. Thermal restriction as an antimicrobial function of fever. PLoS Pathogens. 12 (5), 1005577 (2016).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Van Loi, V., Busche, T., Preuß, T., Kalinowski, J., Bernhardt, J. The AGXX ® antimicrobial coating causes a thiol-specific oxidative stress response and protein S-bacillithiolation in Staphylococcus aureus. Frontiers in Microbiology. 9, 3037 (2018).
- Anfinsen, C. B., Scheraga, H. A. Experimental and theoretical aspects of protein folding. Advances in Protein Chemistry. 29, 205-300 (1975).
- Schramm, F. D., Schroeder, K., Jonas, K. Protein aggregation in bacteria. FEMS Microbiology Reviews. 44 (1), 54-72 (2020).
- Tomoyasu, T., Mogk, A., Langen, H., Goloubinoff, P., Bukau, B. Genetic dissection of the roles of chaperones and proteases in protein folding and degradation in the Escherichia coli cytosol. Molecular Microbiology. 40 (2), 397-413 (2001).
- Gray, M. J., et al. Polyphosphate is a primordial chaperone. Molecular Cell. 53 (5), 689-699 (2014).
- Weids, A. J., Ibstedt, S., Tamás, M. J., Grant, C. M. Distinct stress conditions result in aggregation of proteins with similar properties. Scientific Reports. 6, 24554 (2016).
- Mogk, A., et al. Identification of thermolabile Escherichia coli proteins: prevention and reversion of aggregation by DnaK and ClpB. EMBO Journal. 18 (24), 6934-6949 (1999).
- Fay, A., Glickman, M. S. An essential nonredundant role for mycobacterial DnaK in native protein folding. PLoS Genetics. 10 (7), 1004516 (2014).
- Schramm, F. D., Heinrich, K., Thüring, M., Bernhardt, J., Jonas, K. An essential regulatory function of the DnaK chaperone dictates the decision between proliferation and maintenance in Caulobacter crescentus. PLoS Genetics. 13 (12), 1007148 (2017).
- Maisonneuve, E., Fraysse, L., Moinier, D., Dukan, S. Existence of abnormal protein aggregates in healthy Escherichia coli cells. Journal of Bacteriology. 190 (3), 887-893 (2008).
- Heiss, A., Freisinger, B., Held-Föhn, E. Enhanced antibacterial activity of silver-ruthenium coated hollow microparticles. Biointerphases. 12 (5), (2017).
- Papnayotou, I., Sun, B., Roth, A. F., Davis, N. G. Protein aggregation induced during glass bead lysis of yeast. Yeast. 27 (10), 801-816 (2010).
- Chuang, S. E., Blattner, F. R. Characterization of twenty-six new heat shock genes of Escherichia coli. Journal of Bacteriology. 175 (16), 5242-5252 (1993).
- Imlay, J. A. The molecular mechanisms and physiological consequences of oxidative stress: Lessons from a model bacterium. Nature Reviews Microbiology. 11 (7), 443-454 (2013).
- Mühlhofer, M., et al. The heat shock response in yeast maintains protein homeostasis by chaperoning and replenishing proteins. Cell Reports. 29 (13), 4593-4607 (2019).
- Chandrangsu, P., Rensing, C., Helmann, J. D. Metal homeostasis and resistance in bacteria. Nature Reviews Microbiology. 15, 338-350 (2017).
- Stevens, M., et al. HSP60/10 chaperonin systems are inhibited by a variety of approved drugs, natural products, and known bioactive molecules. Bioorganic and Medicinal Chemistry Letters. 29 (9), 1106-1112 (2019).
- Schramm, F. D., Schroeder, K., Alvelid, J., Testa, I., Jonas, K. Growth-driven displacement of protein aggregates along the cell length ensures partitioning to both daughter cells in Caulobacter crescentus. Molecular Microbiology. 111 (6), 1430-1448 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationThis article has been published
Video Coming Soon