È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Estrazione e visualizzazione di aggregati proteici dopo il trattamento di Escherichia coli con un fattore di stress proteotossico
In questo articolo
Riepilogo
Questo protocollo descrive l'estrazione e la visualizzazione di proteine aggregate e solubili da Escherichia coli dopo il trattamento con un antimicrobico proteotossico. Seguire questa procedura permette un confronto qualitativo della formazione di aggregati proteici in vivo in diversi ceppi batterici e/o tra trattamenti.
Abstract
L'esposizione degli organismi viventi a stress ambientali e cellulari spesso causa interruzioni nell'omeostasi proteica e può provocare l'aggregazione proteica. L'accumulo di aggregati proteici nelle cellule batteriche può portare ad alterazioni significative nel comportamento fenotipico cellulare, tra cui una riduzione dei tassi di crescita, resistenza allo stress e virulenza. Esistono diverse procedure sperimentali per l'esame di questi fenotipi mediati da fattori di stress. Questo documento descrive un test ottimizzato per l'estrazione e la visualizzazione di proteine aggregate e solubili da diversi ceppi di Escherichia coli dopo il trattamento con un antimicrobico contenente argento-rutenio. Questo composto è noto per generare specie reattive dell'ossigeno e provoca un'aggregazione proteica diffusa.
Il metodo combina una separazione basata sulla centrifugazione di aggregati proteici e proteine solubili da cellule trattate e non trattate con successiva separazione e visualizzazione mediante elettroforesi su gel di dodecilsalfato-poliacrilammide (SDS-PAGE) e colorazione di Coomassie. Questo approccio è semplice, veloce e consente un confronto qualitativo della formazione di aggregati proteici in diversi ceppi di E. coli. La metodologia ha una vasta gamma di applicazioni, tra cui la possibilità di studiare l'impatto di altri antimicrobici proteotossici sull'aggregazione proteica in vivo in una vasta gamma di batteri. Inoltre, il protocollo può essere utilizzato per identificare i geni che contribuiscono ad aumentare la resistenza alle sostanze proteossiche. Le bande di gel possono essere utilizzate per la successiva identificazione di proteine particolarmente inclini all'aggregazione.
Introduzione
I batteri sono inevitabilmente esposti a una miriade di stress ambientali, tra cui pH basso (ad esempio, nello stomaco dei mammiferi)1,2,specie reattive dell'ossigeno e del cloro (ROS / RCS) (ad esempio, durante lo scoppio ossidativo nei fagociti)3,4,5, temperature elevate (ad esempio, nelle sorgenti calde o durante lo shock termico)6,7e diversi potenti antimicrobici (ad esempio, AGXX utilizzato in questo protocollo)8. Le proteine sono particolarmente vulnerabili a uno qualsiasi di questi fattori di stress e l'esposizione può provocare un-/misfolding proteico che poi semina l'aggregazione. Tutti gli organismi impiegano sistemi protettivi che consentono loro di far fronte al misfolding proteico9. Tuttavia, uno stress grave può sopraffare il meccanismo di controllo della qualità delle proteine e interrompere la struttura secondaria e / o terziaria delle proteine, che alla fine inattiva le proteine. Di conseguenza, gli aggregati proteici possono compromettere gravemente le funzioni cellulari critiche necessarie per la crescita e la sopravvivenza batterica, la resistenza allo stress e la virulenza10. Pertanto, la ricerca incentrata sull'aggregazione proteica e le sue conseguenze nei batteri è un argomento rilevante a causa del suo potenziale impatto sul controllo delle malattie infettive.
Lo sviluppo e l'aggregazione delle proteine indotte dal calore sono spesso reversibili7. Al contrario, altri stress proteotossici, come lo stress ossidativo, possono causare modificazioni proteiche irreversibili attraverso l'ossidazione di specifiche catene laterali di aminoacidi con conseguente un-/misfolding proteico e, infine, aggregazione proteica4. La formazione indotta da stress di aggregati proteici insolubili è stata ampiamente studiata nel contesto degli chaperoni molecolari e delle loro funzioni protettive in lieviti e batteri11,12,13. Sono stati pubblicati diversi protocolli che utilizzano una varietà di tecniche biochimiche per l'isolamento e l'analisi di aggregati proteici insolubili14,15,16,17. I protocolli esistenti sono stati utilizzati principalmente per studiare l'aggregazione proteica batterica in caso di shock termico e/o identificazione di chaperoni molecolari. Mentre questi protocolli sono stati certamente un progresso nel campo, ci sono alcuni importanti inconvenienti nelle procedure sperimentali perché richiedono (i) un grande volume di coltura batterica fino a 10 L14,17, (ii) complicati processi di interruzione fisica, incluso l'uso di disgregatori cellulari, stampa francese e / o sonicazione14,15,17o (iii) lunghi processi ripetuti fasi di lavaggio e incubazione15,16,17.
Questo documento descrive un protocollo modificato che mira ad affrontare i limiti degli approcci precedenti e consente l'analisi della quantità di aggregati proteici formati in due diversi ceppi di Escherichia coli dopo il trattamento con un rivestimento superficiale antimicrobico proteotossico. Il rivestimento è composto da metallo-argento (Ag) e rutenio (Ru)-condizionati con acido ascorbico, e la sua attività antimicrobica è ottenuta dalla generazione di specie reattive dell'ossigeno8,18. Di seguito è riportata una descrizione dettagliata della preparazione della coltura batterica dopo il trattamento con il composto antimicrobico e un confronto dello stato di aggregazione proteica in caso di esposizione di due ceppi di E. coli con distinti profili di suscettibilità all'aumento della concentrazione dell'antimicrobico. Il metodo descritto è economico, veloce e riproducibile e può essere utilizzato per studiare l'aggregazione proteica in presenza di altri composti proteotossici. Inoltre, il protocollo può essere modificato per analizzare l'impatto che specifiche delezioni geniche hanno sull'aggregazione proteica in una varietà di batteri diversi.
Protocollo
1. Trattamento da stress dei ceppi di E. coli MG1655 e CFT073
- Inoculare 5 mL di brodo di lipogenesi (LB) con una singola colonia di E. coli ceppo MG1655 e uropatogeno E. coli (UPEC) ceppo CFT073, rispettivamente, e incubare per 14-16 ore (durante la notte) a 37 °C e 300 rpm.
NOTA: Escherichia coli CFT073 è un agente patogeno umano. La manipolazione di CFT073 deve essere eseguita con adeguate misure di biosicurezza in un laboratorio certificato di livello 2 di biosicurezza. - Diluire ogni ceppo in un matraccio da 500 mLcontenente 70 mL di acido 3-( N-morfolino)propanisolfonico (MOPS)-glucosio (MOPS-g) (Tabella 1) da mezzo a una densità ottica a 600 nm (OD600) valore di 0,1. Incubare a 37 °C e 300 giri/min fino a raggiungere la fase mid-log (OD600 = 0,5-0,55).
- Trasferire 20 mL di ciascuna coltura in tre palloni da 125 mL preriscaldati e incubare a 37 °C e 300 giri/min per 2 min.
NOTA: poiché è richiesta un'elaborazione tempestiva dei campioni, gestire non più di 6 colture alla volta. - Preparare una soluzione di composto antimicrobico in mezzo MOPS-g ad una concentrazione di 2 mg/ml. Aggiungere l'antimicrobico a ciascuna coltura per raggiungere le concentrazioni indicate. Per il controllo non trattato, aggiungere il volume richiesto di MOPS-g medium.
NOTA: Vortex la soluzione antimicrobica da 2 mg/mL per consentire una distribuzione uniforme delle particelle composte ed evitare la sedimentazione. - Incubare le colture per 45 min a 37 °C e 300 giri/min.
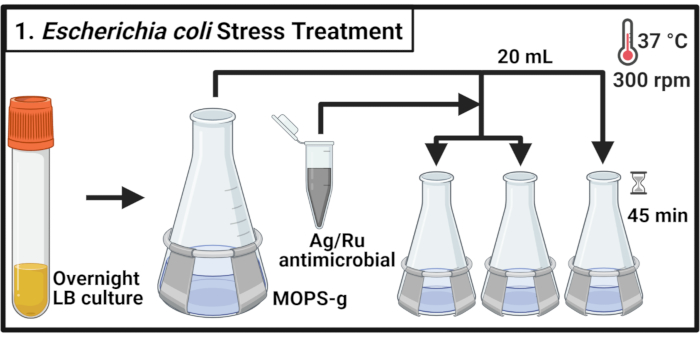
Figura 1: Trattamento da stress da Escherichia coli. Le colture batteriche vengono coltivate in MOPS-g e trattate con le concentrazioni indicate dell'antimicrobico contenente argento-rutenio quando viene raggiunta la fase mid-log. Abbreviazioni: LB = brodo di lipogenesi; Ag-Ru = argento-rutenio; MOPS-g = acido3-( N-morfolino)propanisolfonico (MOPS)-glucosio. Fare clic qui per visualizzare una versione più grande di questa figura.
2. Raccolta di campioni di cellule batteriche
- Dopo 45 minuti di trattamento dello stress, determinare l'OD600 di ogni coltura. Per ogni campione, cellule di raccolta equivalenti a 4 mL di OD600 = 1 in tubi centrifughi da 15 mL mediante centrifugazione per 15 minuti a 3.000 × g e 4 °C.
- Rimuovere completamente il surnatante e riaspentare i pellet cellulari in 50 μL di tampone di lisi ghiacciato (Tabella 1). Incubare i campioni per 30 minuti sul ghiaccio.
NOTA: questa fase di lisi degrada lo strato di peptidoglicano. Utilizzare sempre un tampone di lisi appena preparato. - Trasferire i campioni in tubi microcentrifuga da 1,7 mL. Congelare a -80 °C fino a nuovo utilizzo.
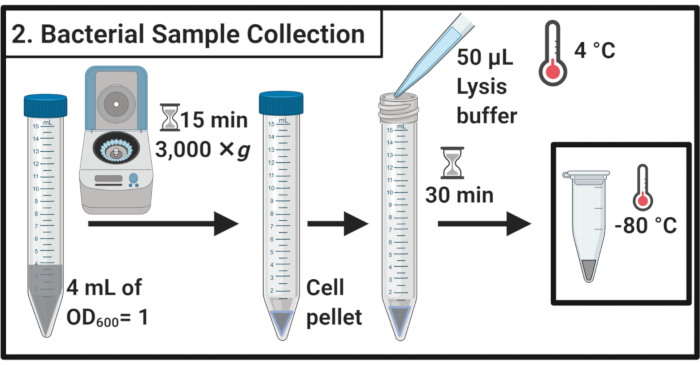
Figura 2: Raccolta di campioni batterici. I campioni di cellule vengono raccolti mediante centrifugazione e risuscisi in tampone di lisi seguito da stoccaggio a -80 °C. Fare clic qui per visualizzare una versione più grande di questa figura.
3. Estrazione degli aggregati proteici insolubili
- Campioni di scongelamento su ghiaccio.
NOTA: Il ciclo di congelamento-disgelo contribuisce alla lisi cellulare. - Aggiungere 360 μL di tampone ghiacciato A (Tabella 1) e mescolare delicatamente mediante pipettaggio.
NOTA: Lo shock osmotico contribuirà anche alla lisi cellulare. - Trasferire il campione in un tubo microcentrifuga da 2 mL contenente ~ 200 μL di perle di vetro da 0,5 mm. Incubare per 30 minuti a 8 °C in un bimbyxer con agitazione a 1.400 giri/min.
Nota : questo passaggio provoca l'interruzione fisica della cella. Un inibitore della proteasi può essere utilizzato per ridurre al minimo la degradazione delle proteine. L'interruzione può essere eseguita a 4 °C. Si noti che l'uso di perle di vetro è stato segnalato per indurre l'aggregazione di un piccolo sottoinsieme di proteine nel lievito19. - Incubare per 5 minuti sul ghiaccio senza agitare per sistemare le perle di vetro. Trasferire 200 μL del liscerato cellulare in tubi microcentrifuga da 1,7 mL.
NOTA: Evitare il trasferimento delle perle di vetro. - Centrifuga a 16.000 × g e 4 °C per 20 min. Raccogliere il surnatante, che contiene proteine solubili, e procedere al paragrafo 4.
- Spese di sospensione del pellet in 200 μL di tampone ghiacciato A (Tabella 1) utilizzando la pipetta. Centrifuga a 16.000 × g e 4 °C per 20 min. Rimuovere con attenzione il surnatante del tutto.
- Aggiungere 200 μL di tampone ghiacciato B (vedere Tabella 1 e Tabella dei materiali)e risuscidare accuratamente il pellet mediante pipettaggio.
NOTA: Il detergente non ionico solubilizza le proteine di membrana. - Ripetere la centrifugazione a 16.000 × g e 4 °C per 20 min. Rimuovere con attenzione il surnatante.
- Rinsodare il pellet in 200 μL di tampone freddo A (Tabella 1) mediante pipettaggio. Centrifuga a 16.000 × g e 4 °C per 20 min. Rimuovere completamente il surnatante.
- Risospenare il pellet in 100 μL di tampone per campioni SDS 1x riducente(Tabella 1)e far bollire per 5 minuti a 95 °C in un bimbyxer.
- Conservare il campione a -20 °C per procedere successivamente o caricare immediatamente su un gel di poliacrilammide SDS per la separazione.
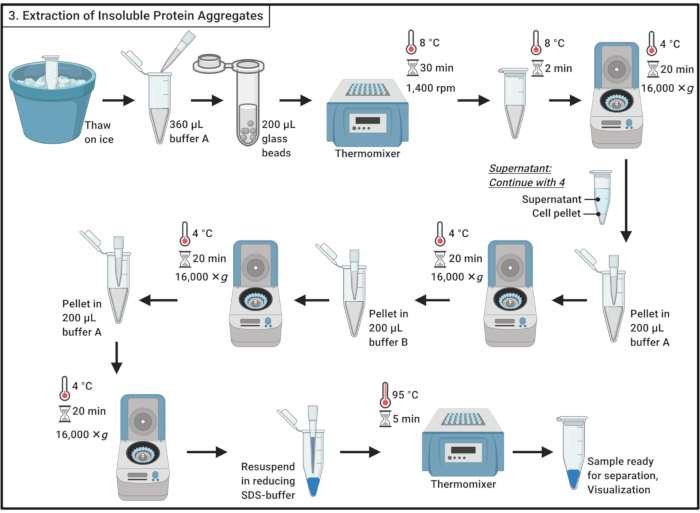
Figura 3: Estrazione di aggregati proteici insolubili. L'estrazione di aggregati proteici comporta una serie di passaggi tra cui la distruzione cellulare, la separazione degli aggregati proteici dalle proteine solubili, la solubilizzazione delle proteine di membrana e il lavaggio. Abbreviazione: SDS = sodio dodecil solfato. Fare clic qui per visualizzare una versione più grande di questa figura.
4. Preparazione del campione proteico solubile
- Mescolare 1 volume di acido tricloroacetico al 100% (TCA) con 4 volumi di campione proteico solubile dal punto 3.6.
NOTA: La gestione del TCA richiede una cappa aspirante e dispositivi di protezione individuale e una procedura di smaltimento dei rifiuti approvata. - Incubare per 10 minuti a 4 °C per consentire la precipitazione delle proteine.
NOTA: il precipitato bianco apparirà molto presto. - Centrifugare per precipitare a 21.000 × g e 4 °C per 5 minuti e rimuovere il surnatante. Lavare il pellet con 200 μL di acetone ghiacciato per rimuovere i detriti cellulari. Centrifugare a 21.000 × g e 4 °C per 5 minuti e rimuovere il surnatante. Ripetere queste azioni nel passaggio 4.3 per un totale di tre volte.
- Posizionare i tubi microcentrifuga con coperchi aperti in un termomixer a 37 °C per rimuovere l'acetone rimanente dal pellet.
NOTA: L'incubazione di oltre 5 minuti può ridurre la solubilità del pellet proteico. - Aggiungere 100 μL di tampone SDS riducente 1x (Tabella 1) e sciogliere completamente il pellet. Far bollire il campione per 5 minuti a 95 °C.
- Conservare il campione a -20 °C per procedere successivamente o caricare immediatamente su un gel di poliacrilammide SDS per la separazione.
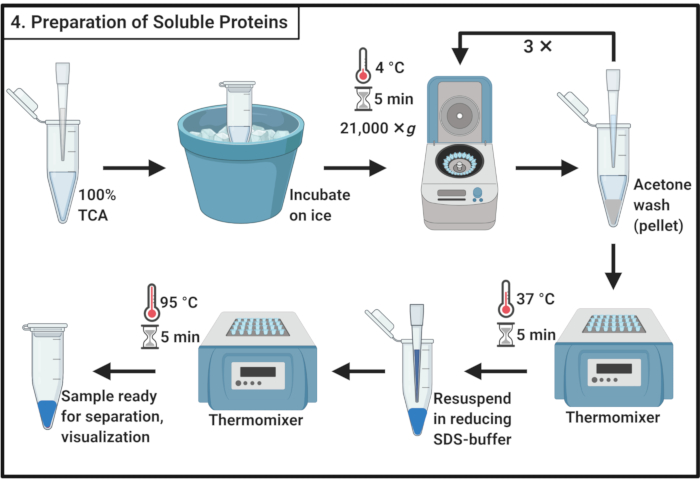
Figura 4: Preparazione di proteine solubili. La preparazione della proteina solubile comporta una fase di precipitazione con acido tricloroacetico e lavaggio ripetuto con acetone ghiacciato. Abbreviazioni: TCA = acido tricloroacetico; SDS = sodio dodecil solfato. Fare clic qui per visualizzare una versione più grande di questa figura.
5. Separazione e visualizzazione di aggregati proteici estratti utilizzando SDS-PAGE
- Preparare un gel SDS-poliacrilammide al 12%.
- Per due gel di separazione, pipettare 5,1 mL di acqua distillata doppia (ddH2O), 3,75 mL di Tris-HCl (pH 8,8), 7,5 mL di SDS al 20% (p/v), 6 mL di soluzione di acrilammide/bisacrilammide al 30% (29:1), 75 mL di persolfato di ammonio al 10% p/v e 10 mL di tetrametiletilendiammina (TEMED) in un tubo centrifugo da 15 mL e mescolare delicatamente senza introdurre bolle d'aria. Versare il gel utilizzando un pipetto da 1 ml all'interno delle lastre di vetro, lasciando la parte superiore di 2 cm libera dalla miscela. Aggiungere il 70% di etanolo sulla parte superiore del gel separatore e consentire un'interfaccia uniforme tra i due strati.
- Dopo la polimerizzazione del gel separatore, preparare il gel impilante pipettando 1,535 mL di ddH2O, 625 mL di soluzione tris-HCl (pH 6,8), 12,5 mL di 20% (p/v) SDS, 335 mL di soluzione di acrilammide/bisacrilammide al 30% (29:1), 12,5 mL di persolfato di ammonio al 10% p/v e 2,5 mL di TEMED. Rimuovere l'etanolo dai gel di separazione e aggiungere la soluzione di gel impilante. Inserire un pettine con il numero desiderato di tasche senza introdurre bolle d'aria. Consentire la polimerizzazione per 20-30 min.
- Caricare 4 μL di ogni campione e scala proteica in pozzetto separati ed eseguire il gel (s) nel tampone di corsa Tris-Glycine (Tabella 1) a 144 V per 45 min a temperatura ambiente.
NOTA: Interrompere il gel quando la banda di bromofenoli sta per migrare fuori dal gel. - Macchiare il gel in una soluzione Fairbanks A preriscaldita(Tabella 1)per 30 minuti su un bilanciere.
- Scolorire il gel o i gel in una soluzione Fairbanks preriscaldita D (Tabella 1) fino allo sfondo desiderato (ad esempio, durante la notte) su un bilanciere.
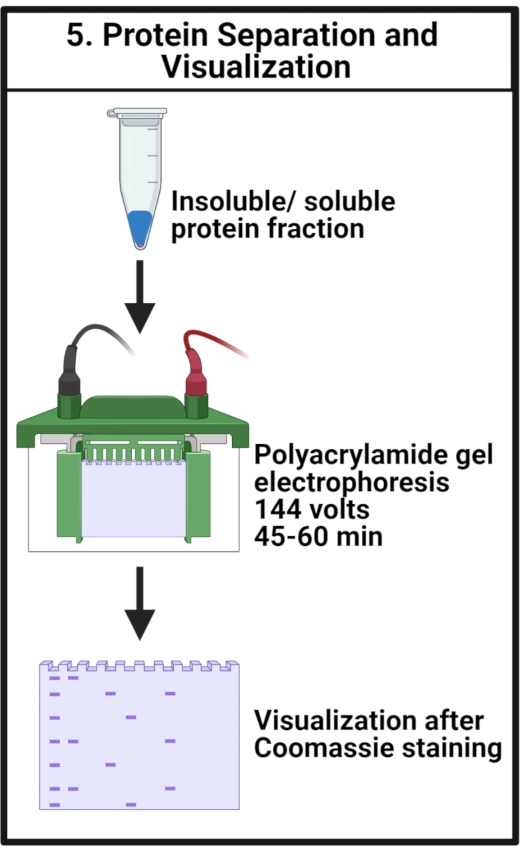
Figura 5: Separazione e visualizzazione delle proteine. I campioni sono separati da SDS-PAGE e visualizzati dalla colorazione Coomassie. Abbreviazione: SDS-PAGE = elettroforesi su gel di sodio dodecilsolfato-poliacrilammide. Fare clic qui per visualizzare una versione più grande di questa figura.
Risultati
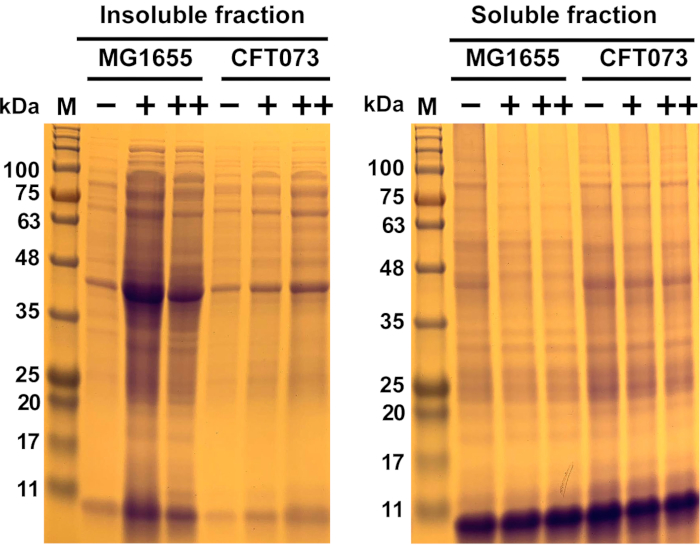
Figura 6: Risultati rappresentativi dell'aggregazione proteica indotta da antimicrobici in Escherichia coli commensale ceppo MG1655 e UPEC ceppo CFT073. I ceppi di E. coli MG1655 e CFT073 sono stati coltivati a 37 °C e 300 rpm a OD600= 0,5-0,55 in mezzi MOPS-g prima di essere trattati con le concentrazioni indicate (-, 0 mg/mL; +...
Discussione
Questo protocollo descrive una metodologia ottimizzata per l'analisi della formazione di aggregati proteici dopo il trattamento di diversi ceppi di E. coli con un antimicrobico proteotossico. Il protocollo consente l'estrazione simultanea di frazioni proteiche insolubili e solubili da cellule di E. coli trattate e non trattate. Rispetto ai protocolli esistenti per l'isolamento di aggregati proteici dalle cellule14,15,
Divulgazioni
Gli autori non hanno nulla da rivelare.
Riconoscimenti
Questo lavoro è stato supportato dai fondi di avvio della Illinois State University School of Biological Sciences, dall'Illinois State University New Faculty Initiative Grant e dalla sovvenzione NIAID R15AI164585 (a J.-U. D.). G.M.A. è stato supportato dall'Illinois State University Undergraduate Research Support Program (a G.M.A.). K. P. H. è stato supportato da una borsa di studio RISE fornita dal German Academic Exchange Service (DAAD). Gli autori ringraziano il Dr. Uwe Landau e il Dr. Carsten Meyer di Largentech Vertriebs GmbH per aver fornito la polvere AGXX. Le figure 1, figura 2, figura 3, figura 4e figura 5 sono state generate con Biorender.
Materiali
| Name | Company | Catalog Number | Comments |
| Chemicals/Reagents | |||
| Acetone | Fisher Scientific | 67-64-1 | |
| 30% Acrylamide/Bisacrylamide solution 29:1 | Bio-Rad | 1610156 | |
| Ammonium persulfate | Millipore Sigma | A3678-100G | |
| Benzonase nuclease | Sigma | E1014-5KU | |
| Bluestain 2 Protein ladder, 5-245 kDa | GoldBio | P008-500 | |
| β-mercaptoethanol | Millipore Sigma | M6250-100ML | |
| Bromophenol blue | GoldBio | B-092-25 | |
| Coomassie Brilliant Blue R-250 | MP Biomedicals LLC | 821616 | |
| D-Glucose | Millipore Sigma | G8270-1KG | |
| D-Sucrose | Acros Organics | 57-50-1 | |
| Ethylenediamine tetra acetic acid (EDTA) | Sigma-Aldrich | SLBT9686 | |
| Glacial Acetic acid | Millipore Sigma | ARK2183-1L | |
| Glycerol, 99% | Sigma-Aldrich | G5516-1L | |
| Glycine | GoldBio | G-630-1 | |
| Hydrochloric acid, ACS reagent | Sigma-Aldrich | 320331-2.5L | |
| Isopropanol (2-Propanol) | Sigma | 402893-2.5L | |
| LB broth (Miller) | Millipore Sigma | L3522-1KG | |
| LB broth with agar (Miller) | Millipore Sigma | L2897-1KG | |
| Lysozyme | GoldBio | L-040-25 | |
| 10x MOPS Buffer | Teknova | M2101 | |
| Nonidet P-40 | Thomas Scientific | 9036-19-5 | |
| Potassium phosphate, dibasic | Sigma-Aldrich | P3786-1KG | |
| Potassium phosphate, monobasic | Acros Organics | 7778-77-0 | |
| Sodium dodecyl sulfate (SDS) | Sigma-Aldrich | L3771-500G | |
| Tetramethylethylenediamine (TEMED) | Millipore Sigma | T9281-50ML | |
| Thiamine | Sigma-Aldrich | T4625-100G | |
| 100% Trichloroacetic acid | Millipore Sigma | T6399-100G | |
| Tris base | GoldBio | T-400-1 | |
| Material/Equipment | |||
| Centrifuge tubes (15 mL) | Alkali Scientific | JABG-1019 | |
| Erlenmeyer flask (125 mL) | Carolina | 726686 | |
| Erlenmeyer flask (500 mL) | Carolina | 726694 | |
| Freezer: -80 °C | Fisher Scientific | ||
| Glass beads (0.5 mm) | BioSpec Products | 1107-9105 | |
| Microcentrifuge | Hermle | Z216MK | |
| Microcentriguge tubes (1.7 mL) | VWR International | 87003-294 | |
| Microcentriguge tubes (2.0 mL) | Axygen Maxiclear Microtubes | MCT-200-C | |
| Plastic cuvettes | Fischer Scientific | 14-377-012 | |
| Power supply | ThermoFisher Scientific | EC105 | |
| Rocker | Alkali Scientific | RS7235 | |
| Shaking incubator (37 °C) | Benchmark Scientific | ||
| Small glass plate | Bio-Rad | 1653311 | |
| Spacer plates (1 mm) | Bio-Rad | 1653308 | |
| Spectrophotometer | Thermoscientific | 3339053 | |
| Tabletop centrifuge for 15 mL centrifuge tubes | Beckman-Coulter | ||
| Vertical gel electrophoresis chamber | Bio-Rad | 1658004 | |
| Vortexer | Fisher Vortex Genie 2 | 12-812 | |
| Thermomixer | Benchmark Scientific | H5000-HC | |
| 10 well comb | Bio-Rad | 1653359 |
Riferimenti
- Dahl, J. -. U., et al. HdeB functions as an acid-protective chaperone in bacteria. Journal of Biological Chemistry. 290 (1), 65-75 (2015).
- Foit, L., George, J. S., Zhang, B. W., Brooks, C. L., Bardwell, J. C. A. Chaperone activation by unfolding. Proceedings of the National Academy of Sciences of the United States of America. 110 (14), 1254-1262 (2013).
- Sultana, S., Foti, A., Dahl, J. -. U. Bacterial defense systems against the neutrophilic oxidant hypochlorous acid. Infection and Immunity. 88 (7), 00964 (2020).
- Dahl, J. -. U., Gray, M. J., Jakob, U. Protein quality control under oxidative stress conditions. Journal of Molecular Biology. 427 (7), 1549-1563 (2015).
- Groitl, B., Dahl, J. -. U., Schroeder, J. W., Jakob, U. Pseudomonas aeruginosa defense systems against microbicidal oxidants. Molecular Microbiology. 106 (3), 335-350 (2017).
- Casadevall, A. Thermal restriction as an antimicrobial function of fever. PLoS Pathogens. 12 (5), 1005577 (2016).
- Richter, K., Haslbeck, M., Buchner, J. The heat shock response: life on the verge of death. Molecular Cell. 40 (2), 253-266 (2010).
- Van Loi, V., Busche, T., Preuß, T., Kalinowski, J., Bernhardt, J. The AGXX ® antimicrobial coating causes a thiol-specific oxidative stress response and protein S-bacillithiolation in Staphylococcus aureus. Frontiers in Microbiology. 9, 3037 (2018).
- Anfinsen, C. B., Scheraga, H. A. Experimental and theoretical aspects of protein folding. Advances in Protein Chemistry. 29, 205-300 (1975).
- Schramm, F. D., Schroeder, K., Jonas, K. Protein aggregation in bacteria. FEMS Microbiology Reviews. 44 (1), 54-72 (2020).
- Tomoyasu, T., Mogk, A., Langen, H., Goloubinoff, P., Bukau, B. Genetic dissection of the roles of chaperones and proteases in protein folding and degradation in the Escherichia coli cytosol. Molecular Microbiology. 40 (2), 397-413 (2001).
- Gray, M. J., et al. Polyphosphate is a primordial chaperone. Molecular Cell. 53 (5), 689-699 (2014).
- Weids, A. J., Ibstedt, S., Tamás, M. J., Grant, C. M. Distinct stress conditions result in aggregation of proteins with similar properties. Scientific Reports. 6, 24554 (2016).
- Mogk, A., et al. Identification of thermolabile Escherichia coli proteins: prevention and reversion of aggregation by DnaK and ClpB. EMBO Journal. 18 (24), 6934-6949 (1999).
- Fay, A., Glickman, M. S. An essential nonredundant role for mycobacterial DnaK in native protein folding. PLoS Genetics. 10 (7), 1004516 (2014).
- Schramm, F. D., Heinrich, K., Thüring, M., Bernhardt, J., Jonas, K. An essential regulatory function of the DnaK chaperone dictates the decision between proliferation and maintenance in Caulobacter crescentus. PLoS Genetics. 13 (12), 1007148 (2017).
- Maisonneuve, E., Fraysse, L., Moinier, D., Dukan, S. Existence of abnormal protein aggregates in healthy Escherichia coli cells. Journal of Bacteriology. 190 (3), 887-893 (2008).
- Heiss, A., Freisinger, B., Held-Föhn, E. Enhanced antibacterial activity of silver-ruthenium coated hollow microparticles. Biointerphases. 12 (5), (2017).
- Papnayotou, I., Sun, B., Roth, A. F., Davis, N. G. Protein aggregation induced during glass bead lysis of yeast. Yeast. 27 (10), 801-816 (2010).
- Chuang, S. E., Blattner, F. R. Characterization of twenty-six new heat shock genes of Escherichia coli. Journal of Bacteriology. 175 (16), 5242-5252 (1993).
- Imlay, J. A. The molecular mechanisms and physiological consequences of oxidative stress: Lessons from a model bacterium. Nature Reviews Microbiology. 11 (7), 443-454 (2013).
- Mühlhofer, M., et al. The heat shock response in yeast maintains protein homeostasis by chaperoning and replenishing proteins. Cell Reports. 29 (13), 4593-4607 (2019).
- Chandrangsu, P., Rensing, C., Helmann, J. D. Metal homeostasis and resistance in bacteria. Nature Reviews Microbiology. 15, 338-350 (2017).
- Stevens, M., et al. HSP60/10 chaperonin systems are inhibited by a variety of approved drugs, natural products, and known bioactive molecules. Bioorganic and Medicinal Chemistry Letters. 29 (9), 1106-1112 (2019).
- Schramm, F. D., Schroeder, K., Alvelid, J., Testa, I., Jonas, K. Growth-driven displacement of protein aggregates along the cell length ensures partitioning to both daughter cells in Caulobacter crescentus. Molecular Microbiology. 111 (6), 1430-1448 (2019).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon