Method Article
Amélioration du contenu tumoral grâce à la macrodissection tumorale
Dans cet article
Résumé
Ce protocole présente une méthode pour augmenter le pourcentage de teneur tumorale des échantillons de tissus incorporés à la paraffine fixée au formol.
Résumé
La présence de tissus non tumoraux contaminants dans les tissus incorporés dans la paraffine fixée au formol (FFPE) peut grandement saper les études génomiques. Nous décrivons ici la macrodissection, une méthode conçue pour augmenter le pourcentage de contenu tumoral d’un échantillon de tissu en enlevant et en éliminant les tissus indésirables avant d’effectuer des extractions d’acides nucléiques en aval. Les blocs de tissus FFPE ont été sectionnés pour produire des sections de tissus montées sur lame de 4 à 5 μm. Une section représentative a été soumise pour la coloration à l’hématoxyline et à l’éosine (H & E) et a ensuite été examinée par un pathologiste certifié par le conseil. Au cours de l’examen, le pathologiste a identifié et marqué les régions du tissu tumoral dans le H & E. Une fois terminé, le H& E marqué a été utilisé pour guider la résection des sections non colorées en série du même bloc de tissu. Pour démontrer les effets de la macrodissection, l’ARN extrait de lymphomes diffus à grandes cellules B (LDGCB) macrodisséqués et non disséqués appariés a été exécuté sur un test d’expression génique numérique capable de déterminer le sous-type de LDGCB et le statut de translocation BCL2. Les résultats ont montré que la macrodissection modifiait le sous-type ou les appels d’état de translocation BCL2 dans 60 % des échantillons examinés. En conclusion, la macrodissection est une méthode simple et efficace pour effectuer un enrichissement tumoral avant les extractions d’acides nucléiques, dont le produit peut ensuite être utilisé en toute confiance dans des études génomiques en aval.
Introduction
Les tissus incorporés dans la paraffine fixée au formol (FFPE), collectés dans le cadre du processus normal de diagnostic clinique et conservés dans les dépôts de tissus cliniques, représentent une vaste ressource pour la recherche humaine, y compris la recherche sur le cancer1. Au fur et à mesure que notre compréhension des maladies humaines s’approfondit, il devient de plus en plus clair que les maladies, auparavant considérées comme des entités uniques basées sur des caractéristiques morphologiques et immunophénotypiques, sont en fait composées de sous-types moléculaires distincts qui nécessitent des tests de sous-typage moléculaire. Par conséquent, les essais génomiques à haute sensibilité capables de discerner ces sous-types sont devenus de plus en plus importants2. Bien que les tissus FFPE soient réputés pour être peu compatibles avec les techniques génomiques en raison de problèmes liés à la fixation, à mesure que la technologie et les protocoles évoluent, ces techniques deviennent de plus en plus compatibles avec ce format tissulaire cliniquement omniprésent 3,4,5. Cependant, les tissus FFPE sont souvent des mélanges de matériaux tumoraux et non tumoraux, où la présence de matériel non tumoral est souvent indésirable et peut, s’il est présent dans une proportion élevée, saper et affecter de manière significative les résultats des analysesgénomiques 6. En effet, une teneur minimale en tumeur de 60% est fréquemment utilisée pour de telles analyses, où les tissus qui ne répondent pas à ce seuil peuvent être exclus, bien qu’ils remplissent par ailleurs les critères de l’étude7. Cela peut être particulièrement problématique dans les contextes de maladies rares, où les tissus des patients sont précieux et difficiles à collecter en grand nombre.
La macrodissection est une méthode qui minimise les effets d’une faible teneur en tumeur en réduisant la quantité de tissu normal3. L’élimination de ce matériel non tumoral confondant avant l’extraction des acides nucléiques peut augmenter considérablement la teneur en pourcentage de la tumeur et donc la pureté tumorale des acides nucléiques extraits. La résection tissulaire repose de manière critique sur un examen pathologique expert, dans lequel la région tumorale est identifiée et encerclée sur une section de tissu coloré en hématoxyline et éosine (H & E) fraîchement générée par un pathologiste certifié par le conseil8. Le H &E encerclé est ensuite utilisé pour guider l’enlèvement et la collecte des tissus indésirables et cibles, respectivement. Ce protocole décrit les étapes de la macrodissection de l’examen pathologique à la collecte de tissus effectuée au laboratoire de base technique de la aids and Cancer Specimen Resource (ACSR) de la Mayo Clinic.
Protocole
Tous les échantillons ont été prélevés et utilisés conformément aux protocoles approuvés de la Mayo Clinic IRB (PR16-000507 et PR2207-02).
1. Préparation de l’échantillon
- Allumez le bain-marie à flottaison tissulaire. Réglez la température à 39 °C et laissez l’eau arriver à température. Faites tremper les bâtons de collection en bois dans le bain-marie.
- Identifier et récupérer les blocs de tissu FFPE à sectionner.
- Pré-étiquette des lames de microscope à l’aide d’un marqueur permanent de qualité histologique qui peut résister aux lavages au solvant.
- Utilisez un microtome pour sectionner les blocs FFPE. Couper au moins 2 sections pleine face à une épaisseur de 4 à 5 μm par section pour chaque bloc (figure 1A).
- Transférer le ruban de coupes de tissu fraîchement coupé dans le bain de flottaison de tissu préchauffé pour le montage de la glissière (Figure 1B, C).
REMARQUE: L’eau chaude aide à « repasser » les rides dans les sections (Figure 1C). - En manipulant chaque section de manière séquentielle, utilisez des pinces pour détacher une seule section du ruban (Figure 1D).
- Collectez la section unique sur une lame de microscope pré-étiquetée.
- Immergez la lame du microscope à un angle sous la section, en positionnant la lame de manière à ce que le bord de la section de tissu touche la lame (Figure 1E).
- Une fois en contact avec la section tissulaire, retirez lentement la glissière du bain de flottaison pour permettre à la section tissulaire de se redresser contre la lame lorsqu’elle émerge de l’eau (Figure 1F).
- Montez 1 section de tissu par lame et répétez jusqu’à ce que toutes les sections aient été collectées.
- Laissez les sections de tissu montées sur glissière sécher complètement à température ambiante (RT).
2. Examen pathologique
- Effectuer une coloration H&E sur une section de tissu représentative pour chaque bloc9.
- Soumettez les H &E fraîchement colorés pour examen pathologique par un pathologiste certifié par le conseil.
REMARQUE: Au cours de l’examen, le pathologiste détermine et enregistre le pourcentage de contenu tumoral dans chaque tissu et entoure la zone tumorale sur chaque diapositive H & E (voir Figure 2 et Figure 3, Ligne 1). Le tableau 1 présente le pourcentage de teneur tumorale par cellularité déterminé lors de l’examen pathologique des H&E pour les échantillons A-E présentés à la figure 3, ligne 1. Les sections avec <60% de contenu tumoral nécessitent une macro-dissection7. Le nombre de sections nécessaires à l’extraction des acides nucléiques dépend de la taille de la zone tumorale encerclée. Si des sections insuffisantes ont été coupées dans la section 1 du protocole et que d’autres coupes sont possibles, il peut être nécessaire de couper des sections supplémentaires.
3. Déparaffinisation
- Dans une hotte, préremplissez deux boîtes de teinture en verre avec du d-limonène de qualité histologique non dilué ou un solvant à base de d-limonène et 1 boîte à coloration en verre avec de l’éthanol de qualité moléculaire non dilué à l’épreuve de 200.
ATTENTION : Évitez le contact du d-limonène avec la peau et les yeux, évitez l’inhalation de vapeur ou de brouillard et tenez-vous à l’écart des sources d’inflammation. Gardez l’éthanol à l’abri de la chaleur, des étincelles et des flammes nues, évitez les déversements et le contact avec la peau ou les yeux, ventilez bien et évitez de respirer des vapeurs.
REMARQUE : Remplissez suffisamment les plats pour immerger les glissières en rack (Figure 4A); 250 mL sont nécessaires pour remplir les plats de coloration de 20 diapositives montrés. Remplacez les lavages au d-limonène et à l’éthanol après chaque 40 lames. Le D-limonène (C10H16) est un agent de déparaffinage alternatif, tout aussi efficace et moins toxique que le xylène, qui devient de plus en plus courant dans les méthodologies histologiques et produit un acide nucléique de bonne qualité après l’extraction 10,11,12,13,14. Bien que ce protocole puisse être réalisé en utilisant des alternatives plus biofriendly15, leurs effets, le cas échéant, sur la qualité des acides nucléiques extraits restent à déterminer. - Rangez les glissières montées en tissu FFPE non coloré dans les glissières en verre Coplin (Figure 4A, encart).
- Immerger les glissières rackées dans d-Limonène laver 1 pendant 2 min; agiter doucement pendant les 20 premières secondes.
REMARQUE: Pour minimiser le report entre les lavages, lorsque vous retirez le rack de glissières d’un lavage, laissez le rack s’écouler brièvement avant de tamponner doucement le fond du rack sur du papier de soie pour éliminer l’excès de lavage. - Immerger les lames en rack dans un lavage au D-Limonène non dilué 2 pendant 2 min; agiter doucement pendant les 20 premières secondes. Retirez, égouttez et tamponnez à nouveau le rack.
- Immergez les glissières en rack dans le lavage à l’éthanol pendant 2 min; agiter doucement pendant les 20 premières secondes. Retirez la grille et placez-la sur un tissu absorbant pour la drainer. Laissez les lames sécher à l’air libre pendant au moins 10 min, mais pas plus de 2 h.
3. Macrodissection
- Sur le banc, préremplissez un bocal en verre Coplin avec 50 mL de glycérol à 3 % dans de l’eau sans DNase/RNase
REMARQUE: Remplacez le lavage au glycérol après chaque 40 lames. - Pré-étiqueter et préremplir des microtubes de 1,5 mL avec 160 μL de tampon de digestion tissulaire par microtube.
REMARQUE: Les extractions d’acides nucléiques effectuées après macrodissection ont utilisé un kit d’extraction d’ADN / ARN FFPE (Table des matériaux). Ainsi, le tampon de digestion tissulaire utilisé dans ce protocole comprenait 10 μL de protéinase K et 150 μL de tampon PKD. - Tracez les marques pathologiques sur le H & E à l’arrière des lames tissulaires déparaffinées.
REMARQUE : Comparativement aux tissus non déparaffinés (figure 3, rangée 2 et figure 4B), les tissus déparaffinés sont blancs et très visibles (figure 3, ligne 3 et figure 4C). C’est cette visibilité accrue et cette discernabilité des caractéristiques tissulaires déparaffinées qui permettent la macrodissection. Placez le H&E face vers le bas sur le banc et placez l’avant de la glissière déparaffinisée contre l’arrière du pathologiste apparié examiné H&E (Figure 4D). Aligner le tissu déparaffinisé avec le tissu H&E (Figure 4E). Le traçage des marques du pathologiste est une étape critique dans le processus de macrodissection, et il faut prendre soin de reproduire ces marques aussi précisément que possible. Cela peut être particulièrement difficile pour les tissus petits et/ou déconnectés tels que les échantillons B, D et E (Figure 3, Figure 4E et Figure 4E encadré i). Pour faciliter le traçage, il faut utiliser un marqueur d’encre fine ou ultrafine (Figure 4E, encadré ii) pour tracer les marques dessinées par le pathologiste. Les lingettes à l’éthanol peuvent être utiles pour éliminer les erreurs et permettre le retraçage si nécessaire. - Tournez le tissu de lame déparaffinisé maintenant marqué vers le haut et tracez la ligne du marqueur avec le coin d’une lame de rasoir propre pour pré-couper les bords de la zone tumorale.
- En manipulant chaque lame séquentiellement, trempez les lames déparaffinées dans la solution de glycérol à 3%. Assurez-vous que le tissu est complètement immergé avant de retirer lentement la lame (Figure 4F).
REMARQUE: Le but de la trempette au glycérol est d’humidifier le tissu pour faciliter la collecte des tissus, mais aussi de réduire l’accumulation de charge statique qui peut provoquer une répulsion entre le tissu et le microtube en plastique dans lequel le tissu collecté doit être placé. - Essuyez doucement l’arrière de la glissière avec un mouchoir en papier pour éliminer l’excès de solution de glycérol et posez la glissière sur le banc, essuyée face vers le bas. Laissez les tissus sécher brièvement pendant 1 à 2 minutes.
REMARQUE: Le transfert de l’excès de glycérol dans le processus d’extraction peut affecter négativement le rendement et la qualité des extractions d’acides nucléiques. Les tissus doivent être légèrement humides mais pas visiblement humides lorsqu’ils sont collectés. - Selon l’endroit où se trouve la zone tumorale d’intérêt sur la lame, utilisez le bord plat de la lame de rasoir pour (a) collecter directement le tissu tumoral, en utilisant le rasoir pour gratter / collecter le tissu d’intérêt de la lame, ou (b) enlever et jeter le tissu non tumoral d’abord avant de prélever le tissu tumoral d’intérêt (Figure 3).
REMARQUE : Les tissus prélevés ont tendance à s’accumuler ou à s’enrouler au bas de la lame (Figure 4G) - Utilisez un bâton en bois pour retirer le tissu prélevé de la lame (figure 4H et encart) et transférez-le dans le microtube pré-étiqueté et prérempli approprié (figure 4I).
REMARQUE: Le tampon de digestion sert à « tirer » le tissu du pic en bois dans le liquide. - Procéder à l’extraction des acides nucléiques.
REMARQUE: Les extractions d’acides nucléiques ont été effectuées à l’aide du kit d’extraction FFPE ADN/ARN conformément aux instructions du fabricant, et les acides nucléiques résultants ont été quantifiés à l’aide d’un spectrophotomètre UV-vis. L’ARN obtenu a été exécuté sur le test DLBCL90 basé sur le profilage numérique de l’expression génique16.
Résultats
Au total, 5 blocs tissulaires FFPE du lymphome diffus à grandes cellules B (LDGCB) ont été sectionnés, et les sections résultantes ont été macrodisséquées ou non avant l’extraction des acides nucléiques. L’ARN extrait a été exécuté sur le test DLBCL9016. Des échantillons macrodisséqués ont été exécutés deux fois, une fois en utilisant 5 μL de concentration d’ARN stock mais pas plus de 300 ng d’entrée totale d’ARN et une fois en utilisant 5 μL de stock d’ARN dilué pour correspondre aux entrées d’ARN de leurs homologues respectifs non disséqués. Les résultats de DLBCL90 sont présentés dans le tableau 2.
Le LDGCB est composé de 3 sous-types distincts de cellules d’origine (COO) avec une réactivité thérapeutique différente, à savoir GCB, ABC et un groupe intermédiaire connu sous le nom de non classé ou UNC17,18. Des translocations impliquant MYC, BCL2 et/ou BCL6 seuls ou en combinaison (double ou triple hit) sont également fréquemment observées dans le LDGCB, en particulier dans le sous-typeGCB 19. Le test DLBCL90 est une extension de son prédécesseur, le test clinique Lymph2Cx, et est donc capable de déterminer le sous-type de DLBCL COO, mais a été principalement développé pour identifier des échantillons présentant des translocations à double succès (DH) impliquant BCL2 en utilisant l’expression numérique des gènes comme alternative à l’hybridation in situ par fluorescence (FISH)16,20. Les résultats du tableau 2 montrent que la macrodissection a changé soit le statut coo, soit le statut DHITsig nécessite 60% (3/5) des échantillons examinés.
La macrodissection de l’échantillon A n’a eu aucun effet sur l’appel COO mais a changé l’appel DHITsig de NEG à UNCLASS, et ce changement a été observé indépendamment de l’entrée d’ARN de l’échantillon macrodisséqué et avec des scores de probabilité similaires (0,224, 0,254). En revanche, la macrodissection de l’échantillon C n’a eu aucun effet sur l’appel DHITsig, mais a changé l’appel COO de GCB à UNC. Encore une fois, ce changement a été observé indépendamment de l’entrée d’ARN de l’échantillon macrodisséqué. Cependant, à 0,117, la probabilité d’appel COO était plus proche du seuil d’appel de 0,1 pour l’échantillon macrodisséqué avec une entrée d’ARN réduite. Semblable à l’échantillon A, la macrodissection de l’échantillon E n’a eu aucun effet sur l’appel COO mais a modifié l’appel DHITsig. Cependant, pour l’échantillon E, l’appel est passé de UNCLASS à NEG et l’a fait indépendamment de l’entrée d’ARN de l’échantillon macrodisséqué avec des appels de probabilité raisonnablement similaires (0,849, 0,833). Notamment, ce changement d’appel à DHITsig NEG est biologiquement logique étant donné que l’échantillon E a été trouvé à ABC-DLBCL, et que des translocations à double impact impliquant BCL2 ont été signalées comme étant exclusivement observées dans GCB-DLBCL19.

Figure 1 : Génération de coupes de tissu montées sur glissière à l’aide d’un microtome. (A) Bloc de tissu FFPE maintenu en place par le mandrin du microtome et coupé pour produire un ruban de sections de tissu FFPE séquentielles. (B) À l’aide de bâtonnets de bois pré-trempés, le ruban est recueilli dans le microtome et transféré dans un bain-marie chaud. (C) La chaleur de l’eau aide à aplanir les plis dans le ruban de tissu. (D) Les coupes de tissu FFPE individuelles sont retirées du ruban tissulaire en plaçant des pinces fermées à la jonction de deux sections et en ouvrant doucement les pinces, ce qui sépare les sections les unes des autres. (E) Les sections sont recueillies dans l’eau en immergeant une lame de verre à un angle et en déplaçant doucement le côté vers la section de tissu jusqu’à ce que le bord de la section touche la lame de verre. (F) Une fois que la glissière et la section se touchent, retirez lentement la lame de l’eau, ce qui permet à la section de tissu de tomber au ras de la glissière. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 2 : Examen pathologique et histologique d’une section colorée à l’hématoxyline et à l’éosine (H&E). (A,B) La section de tissu H&E est soumise à un examen microscopique par un pathologiste certifié par le conseil. (C,D) Une fois que le pathologiste a examiné l’ensemble du tissu et constaté qu’il ne s’agit pas à 100% de tissu tumoral, le pathologiste utilisera un marqueur pour encercler la zone tumorale du tissu. (E,F) Tenir le H & E marqué contre le bloc de tissu FFPE d’origine montre que tout le tissu n’est pas du matériel tumoral. Veuillez cliquer ici pour voir une version agrandie de cette figure.
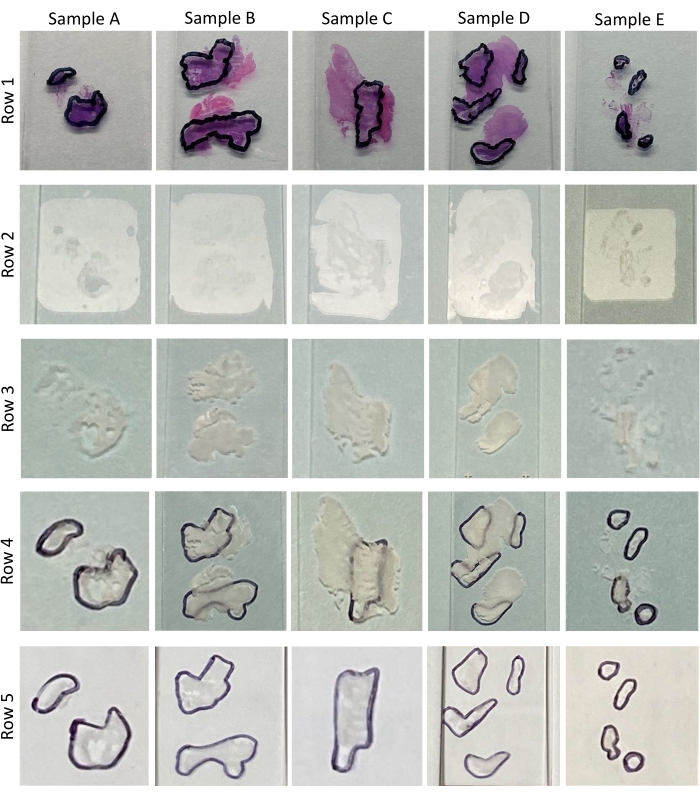
Figure 3 : Échantillons de tissus. Cette image montre les H&E pathologiquement examinés et marqués par la tumeur (rangée 1), les coupes de tissu FFPE non traitées montées sur lame (rangée 2), les sections de tissu FFPE déparaffinisées montées sur lame (rangée 3), les sections de tissu FFPE déparaffinisées montées sur lame avec des marques de pathologie tracées à l’arrière de la diapositive (rangée 4), les sections de tissu FFPE désparaffinisées et macrodisséquées montées sur lame (ligne 5) pour les 5 échantillons (A-E) utilisés pour démontrer ce protocole. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 4 : Déparaffinisation et macro-dissection des coupes de tissus FFPE : (A) Dans une hotte aspirante, les tissus FFPE montés dans un rack coulissant sont lavés dans deux lavages au d-limonène et un lavage à l’éthanol. (B,C) Après le lavage, toute la paraffine a été enlevée et seul le tissu reste sur la lame, qui est maintenant blanche et très visible par rapport à son homologue prélavé. (D) La section de tissu déséparaffinisée montée sur lame est placée face cachée au dos de son H&E marqué apparié. (E) Les marques de la zone tumorale sur le H&E sont ensuite tracées à l’arrière de la lame déparaffinisée à l’aide d’un marqueur permanent finement ou ultrafin (F) La section de tissu déparaffinisée marquée montée sur lame est ensuite trempée dans un lavage au glycérol pour amortir la section de tissu pour la collecte. La lame est retirée lentement du glycérol et l’arrière de la lame est essuyé avec un mouchoir en papier pour éliminer l’excès de glycérol avant de poser le tissu de la lame face vers le haut sur le banc. (G) En utilisant le côté plat d’une lame de rasoir propre, le tissu est macrodisséqué et le tissu indésirable à l’extérieur des marques du pathologiste est jeté avant de recueillir le tissu d’intérêt, qui se rassemble le long du bord de la lame. (H) Un bâton en bois est utilisé pour retirer le tissu prélevé du bord de la lame. (I) Le tissu est ensuite transféré dans un tampon de digestion tissulaire pré-rempli de microtubes prémarqué et est prêt pour l’extraction des acides nucléiques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| EXEMPLE D’ID | Tissu entier (a) | Zone encerclée du tissu (b) | Teneur tumorale globale du tissu entier (c) | Augmentation du pli de la teneur tumorale par macrodissection (d) | Non macrodisséqué (e) | Macrodisséqué (f) | ||||
| % Tumeur viable | % Autres | % Tumeur viable | % Autres | Nombre de lames de 5 μm extraites | ARN conc (ng/μL) | Nombre de lames de 5 μm extraites | CONC ARN (ng/μL) | |||
| Exemple A | 60 | 40 | 75 | 25 | 45 | 1.7 | 2 | 19.0 | 4 | 58.3 |
| Échantillon B | 60 | 40 | 65 | 35 | 39 | 1.7 | 1 | 34.0 | 2 | 60.0 |
| Exemple C | 40 | 60 | 65 | 35 | 26 | 2.5 | 1 | 13.7 | 2 | 46.2 |
| Échantillon D | 35 | 65 | 90 | 10 | 32 | 2.9 | 1 | 57.3 | 2 | 60.0 |
| Échantillon E | 20 | 80 | 30 | 70 | 6 | 5.0 | 3 | 25.2 | 3 | 44.6 |
Tableau 1 : Données de l’examen de la pathologie. Le tableau montre (a) le pourcentage de tumeur viable dans l’ensemble de la section tissulaire par zone, (b) le pourcentage de tumeur viable dans la zone encerclée/marquée par le pathologiste lors de l’examen par cellularité, (c) la cellularité tumorale globale estimée de l’ensemble du tissu (a x b), (d) l’augmentation estimée du pli de la cellularité tumorale obtenue avec la macrodissection, e et f) le nombre de coupes de tissus ffPE non colorés de 5 μm prélevées sur lames et les concentrations d’ARN résultantes pour les échantillons appariés non macrodisséqués et macrodisséqués. % Autre fait référence à tous les autres tissus présents dans un échantillon donné qui n’est pas du tissu tumoral et peut inclure du tissu conjonctif, des fibroblastes stromaux, des vaisseaux sanguins ainsi que d’autres éléments stromaux inhérents. Veuillez cliquer ici pour télécharger ce tableau.
| EXEMPLE D’ID | Entrée d’ARN (ng) | Appel DLBCL90 COO | Probabilité d’appel DLBCL90 | Appel DHITsig | Probabilité de pos DHITsig | Probabilité DHITsig neg | |
| Non macrodisséqué | Exemple A | 95.0 | Le | 0.000 | Neg | 0.135 | 0.865 |
| Échantillon B | 170.0 | Le | 0.000 | Neg | 0.032 | 0.968 | |
| Exemple C | 68.5 | Le | 0.028 | Neg | 0.033 | 0.967 | |
| Échantillon D | 286.5 | Abc | 0.998 | Neg | 0.002 | 0.998 | |
| Échantillon E | 126.0 | Abc | 0.989 | DÉCLASSER | 0.212 | 0.788 | |
| Macrodissectionné | Exemples A_M | 291.7 | Le | 0.000 | DÉCLASSER | 0.224 | 0.776 |
| Exemples B_M | 300.0 | Le | 0.000 | Neg | 0.016 | 0.984 | |
| Exemples de C_M | 231.2 | DÉCLASSER | 0.210 | Neg | 0.015 | 0.985 | |
| Exemples D_M | 300.0 | Abc | 0.999 | Neg | 0.002 | 0.998 | |
| Exemple de E_M | 223.2 | Abc | 0.987 | Neg | 0.151 | 0.849 | |
| Macrodissectionné & ARN dilué | Exemples A_M | 95.0 | Le | 0.000 | DÉCLASSER | 0.254 | 0.746 |
| Exemples B_M | 170.0 | Le | 0.000 | Neg | 0.023 | 0.977 | |
| Exemples de C_M | 68.5 | DÉCLASSER | 0.117 | Neg | 0.027 | 0.973 | |
| Exemples D_M | 286.5 | Abc | 0.999 | Neg | 0.002 | 0.998 | |
| Exemple de E_M | 126.0 | Abc | 0.995 | Neg | 0.167 | 0.833 | |
Tableau 2 : Résultats du test d’expression génique numérique DLBCL90. Cinq échantillons (A-E) n’ont pas été macrodisséqués ou macrodisséqués avant que les extractions d’acides nucléiques ne soient effectuées. L’ARN obtenu a été exécuté sur le test DLBCL90, pour lequel le volume maximal d’entrée d’ARN est de 5 μL. Des échantillons non macrodisséqués ont été exécutés en utilisant 5 μL d’ARN stock. Chaque échantillon macrodisséqué a été exécuté deux fois en utilisant (a) 5 μL d’ARN stock, sauf si des aliquotes de 60 ng/μL étaient possibles et (b) 5 μL d’ARN stock dilué pour correspondre aux concentrations/entrées de leurs homologues non macrodisséqués. Le suffixe _M indique que cet échantillon a été macrodisséqué. Veuillez cliquer ici pour télécharger ce tableau.
Discussion
Les tissus FFPE sont souvent des mélanges hétérogènes de tissus tumoraux et non tumoraux. Les tests génomiques à haute sensibilité sont de plus en plus répandus dans les milieux cliniques et de recherche, mais peuvent être confondus par la présence de tissus non tumoraux contaminants. En effet, une teneur minimale en tumeur de 60% est fréquemment recommandée pour les études génomiques. Le pourcentage de tumeur peut être déterminé par la zone de tissu occupée par le matériau tumoral ou par la proportion de cellules tumorales dans le tissu. Bien que la tumeur par zone soit une mesure couramment utilisée pour la pureté tumorale, elle ne décrit pas toujours une description précise du tissu. Considérons deux tissus, tous deux avec 1000 cellules, dont 500 sont des cellules tumorales. Dans le tissu A, les 500 cellules non tumorales sont des cellules stromales avec des volumes similaires à ceux de la cellule tumorale. Dans ce tissu, le pourcentage de tumeur peut être considéré comme 50% à la fois par la cellularité et la zone. Dans le tissu B, les 500 cellules non tumorales sont des cellules graisseuses dont les volumes sont 4 fois supérieurs à ceux de la cellule tumorale. Dans ce tissu, le pourcentage de tumeur est encore de 50% par cellularité mais de 20% par surface. Un troisième tissu, le tissu C, est composé de 500 cellules tumorales plus 400 cellules graisseuses et 800 cellules stromales avec des volumes qui sont 4x et 0,5x celui des cellules tumorales, respectivement. Étant donné que 100 cellules graisseuses équivaut au volume de 800 cellules stromales, le pourcentage de tumeur du tissu C est de 29% par cellularité (500/1700) mais toujours de 20% par surface. Le tissu D est également composé de la tumeur, de la graisse et des cellules stromales avec des rapports de volume de 1x, 4x et 0,1x. Cependant, le nombre de cellules est de 400, 10 et 720, respectivement. Ainsi, le pourcentage de tumeur du tissu D est de 35% par cellularité (400/1130) mais de 78% par surface. Ces exemples sont trop simplistes et ne reflètent pas les compositions tissulaires du monde réel, mais transmettent clairement l’importance de la composition tissulaire et la différence entre le contenu tumoral par zone et par cellularité. Il est important de noter que lorsqu’il s’agit d’enrichir le contenu tumoral pour l’extraction des acides nucléiques en aval, la cellularité tumorale est l’attribut le plus important en raison du potentiel de confusion accru de l’extraction de matériel génomique à partir de plus de cellules non tumorales que de cellules tumorales. Cela souligne non seulement la nécessité d’évaluer le contenu tumoral des tissus en termes de pourcentage de cellularité, mais également la nécessité d’exciser les tissus indésirables afin de minimiser les effets négatifs potentiels du tissu non tumoral. Il existe plusieurs méthodes disponibles pour l’enrichissement des tissus, les principales étant la macrodissection et la microdissection.
La macrodissection, la méthode décrite dans ce protocole, est relativement rapide, simple et ne nécessite pas d’équipement coûteux ou spécialisé. Bien que la macrodissection puisse grandement améliorer le contenu tumoral, il est important de comprendre qu’elle n’élimine pas complètement le matériel non tumoral. Le but de la macrodissection est d’enrichir suffisamment le tissu d’intérêt par l’exclusion des tissus indésirables afin de réduire le « bruit » provenant des tissus indésirables, ce qui peut à son tour renforcer le signal d’intérêt du tissu d’intérêt. Ainsi, l’enrichissement tumoral médié par macrodissection est un moyen d’améliorer le rapport signal/bruit afin de mieux détecter les marqueurs d’intérêt, en particulier les marqueurs moléculaires spécifiques à la tumeur avec une faible abondance ou une mauvaise expression. Cependant, la macrodissection a des limites en raison du manque de précision offert par les outils grossiers tels que les lames de rasoir et est sensible aux problèmes de précision découlant de l’épaisseur de ligne du marqueur du pathologiste, ainsi qu’aux erreurs potentielles lors du traçage des démarcations H & E des pathologistes. Comme mentionné ci-dessus, il n’est pas possible d’atteindre une pureté tumorale de 100% en raison de la présence d’éléments stromaux inhérents et induisant la tumeur (c’est-à-dire le tissu conjonctif, les fibroblastes stromaux, les vaisseaux sanguins, les lymphocytes réactifs bénins, les macrophages) intégrés dans la tumeur elle-même. En effet, de nombreuses tumeurs malignes invasives ou infiltrantes diffuses induisent une réponse stromale desmoplastique robuste, résultant en grappes de cellules tumorales qui sont intimement mélangées avec des fibroblastes stromaux et d’autres types de cellules non néoplasiques; où les tumeurs associées à ce schéma de réaction stromale, telles que les tissus cancéreux du pancréas21, peuvent bénéficier davantage d’une microdissection guidée numériquement que d’une macrodissection manuelle.
La microdissection manuelle est réalisée au microscope pour faciliter l’identification, la dissection et l’isolement de cellules ou de populations spécifiques de tissus à l’aide d’une aiguille ou d’un scalpel et présente l’avantage d’une précision accrue par rapport à la macrodissection22. Cependant, la microdissection manuelle est un processus laborieux qui manque de la finesse nécessaire pour les tissus complexes avec un faible contenu tumoral ou des caractéristiques complexes incompatibles avec la dissection manuelle. Ces tissus peuvent être disséqués à l’aide de méthodes automatisées de haute précision telles que la microdissection par capture laser. En effet, il a été démontré que la microdissection guidée numériquement donne un pourcentage plus élevé de contenu tumoral par rapport à la macrodissection manuelle dans les tissus cancéreux du pancréas23. Cependant, les inconvénients de ces méthodes automatisées de haute précision, tels que le besoin d’équipements spécialisés et coûteux et de personnes hautement qualifiées, ont entravé son intégration dans les flux de travail. Une étude de de Bruin et al. comparant les effets de la macrodissection et de la microdissection par capture laser (LCM) sur le profilage de l’expression génique a révélé que les échantillons de LCM avaient de faibles rendements totaux en ARN (30 ng en moyenne) et nécessitaient deux cycles d’amplification de l’ARNm afin d’atteindre le seuil d’entrée de préparation de la bibliothèque d’ADNc24. Les auteurs ont constaté que les profils d’expression génique LCM résultants étaient affectés par les cycles d’amplification de l’ARNm plus que les profils macrodisséqués étaient affectés par des contributions stromales non tumorales et ont conclu que la macrodissection pouvait être utilisée de manière adéquate pour générer des données fiables surl’expression génique 24.
Un avantage significatif du profilage de l’expression génique numérique NanoString, en particulier lorsque vous travaillez avec de l’ARN dérivé de FFPE fortement dégradé, est qu’il ne nécessite pas de processus dépendants enzymatiques tels que l’amplification de l’ARN ou la préparation de bibliothèques d’ADNc. Cependant, les essais sont généralement optimisés pour les entrées comprises entre 50 et 300 ng de l’ARNtotal 25,26, ce qui, d’après les résultats de de Bruin et al.24, peut ne pas être compatible avec les tissus microdisséqués sans augmenter l’apport tissulaire; une demande défavorable à une époque où les échantillons de tissus sont de plus en plus prélevés sous forme de petites biopsies plutôt que de résections chirurgicales. Les entrées d’ARN utilisées pour le test DLBCL90 variaient de 68,5 à 300 ng pour les tissus macrodisséqués et non disséqués. Les résultats montrent que la macrodissection a entraîné des changements d’appel dans 60% des échantillons examinés et que ces changements ont été observés indépendamment de l’entrée d’ARN des échantillons macrodisséqués. Cependant, la probabilité COO pour l’entrée d’ARN faible a empiété sur le seuil d’appel de probabilité COO GCB/UNC, où les seuils sont de 0 à <0,1 pour GCB, 0,1-0,9 pour UNC et >0,9 à 1,0 pour les appels ABC20. Les principaux sous-types de COO DLBCL sont GCB et ABC, qui représentent 41% et 44% de tous les cas de DLBCL, l’UNC représentant un groupe intermédiaire des deux et ABC étant leplus agressif 20,27. Ainsi, bien que le changement d’appel du chef de l’exploitation lors de la macrodissection de l’échantillon C n’ait pas entraîné un changement franc du sous-type de chef de l’exploitation de GCB à ABC, le changement de GCB à UNC peut suggérer un changement vers une maladie plus agressive. De plus, des études récentes indiquent que le sous-type UNC n’est pas simplement un sous-type intermédiaire et qu’il peut potentiellement posséder des attributs thérapeutiques exploitables spécifiques au sous-type28. De même, la macrodissection des échantillons A et E n’a pas provoqué de changements francs dans les appels DHITsig de DH négatif à DH positif, ou vice versa. Cependant, les mouvements d’un échantillon GCB (échantillon A) de NEG à UNCLASS et d’un échantillon ABC (échantillon E) de UNCLASS à NEG lors de la macrodissection sont biologiquement appropriés car les translocations à double succès impliquant BCL2 sont signalées comme étant un phénomène exclusivement GCB19. Bien que les translocations soient traditionnellement et omniprésentes détectées par FISH en milieu clinique, il existe un élan croissant pour identifier une méthode alternative moins impliquée et plus longue pour leur détection. Le test DLBCL90 est un outil important qui répond à ce besoin, où la justification de son utilisation est renforcée par la découverte que ce test est capable de détecter des translocations cryptiques aux sondes FISH utilisées dans les diagnostics cliniques29.
Le protocole de macrodissection décrit ci-dessus décrit une méthode simple qui permet aux chercheurs d’augmenter la teneur en tumeurs d’échantillons de tissus qui tomberaient normalement en dessous des seuils d’inclusion d’études couramment utilisés. L’inclusion de la macrodissection dans un flux de travail d’étude permet aux chercheurs de sauver des tissus peu denses en tumeurs de l’exclusion de l’étude en augmentant leur contenu tumoral. À son tour, cela permet une confiance accrue dans le fait que l’ARN et l’ADN résultants représentent la tumeur faisant l’objet d’une enquête génomique. Bien qu’il existe d’autres méthodes plus précises de dissection tissulaire, pour les tumeurs qui se développent de manière plus expansive, non infiltrante, en forme de feuille ou solide, la macrodissection est probablement suffisante. Les résultats présentés ici soulignent l’importance de la pureté tumorale dans les tests génomiques et la macrodissection en tant qu’outil fiable pour y parvenir.
Déclarations de divulgation
Les auteurs n’ont rien à divulguer.
Remerciements
Ce travail est soutenu par AIDS and Cancer Specimen Resource (ACSR, UM1 CA181255-2), financé par les NIH, dans le cadre de son programme scientifique sur les échantillons biologiques. La vidéo a été filmée et le montage de post-production a été effectué par Mayo Clinic Media Services.
matériels
| Name | Company | Catalog Number | Comments |
| 200-proof ethanol | Decon | 2701 | |
| AllPrep DNA/RNA FFPE Kit | Qiagen | 80234 | DNA/RNA FFPE extraction kit |
| Coplin pots | Various | x | |
| DLBCL90 probes | NanoString | various | Digital gene expression profiling based DLBCL90 assay |
| d-Limonene | VWR | 89376-092 | |
| Forceps | Various | x | |
| Glass micrscope slides | FisherBrand | 12-550-15 | |
| Glycerol | VWR | 0854-1L | |
| Master kits | NanoString | various | |
| Microtome | Leica | RM2265 | |
| Microtubes | Ambion | AM12400 | |
| NanoDrop One | Thermo Scientific | ND-ONE-W | Spectrophotometer for DNA, RNA and protein qualitation |
| nCounter | NanoString | x | Digital gene expression profiling platform used to run the DLBCL90 assay |
| Permanent marker | Electrib Microscope Sciences | 72109-12 | |
| Razor blade dispenser | Electrib Microscope Sciences | 71985-10 | |
| Razor blades | Electrib Microscope Sciences | 71985-23 | |
| Tissue digestion buffer | Qiagen | 80234 | |
| Ultrapure water | VWR | SH30538.02 | |
| Waterbath | Triangle Biomedical Sciences | TFB-120 | |
| Wooden stick | FisherBrand | 22363158 |
Références
- Mathieson, W., Thomas, G. A. Why formalin-fixed, paraffin-embedded biospecimens must be used in genomic medicine: An evidence-based review and conclusion. Journal of Histochemistry and Cytochemistry. 68 (8), 543-552 (2020).
- Robetorye, R. S., Maguire, A., Rosenthal, A. C., Rimsza, L. M. Profiling of lymphoma from formalin-fixed paraffin-embedded tissue. Seminars in Hematology. 56 (1), 46-51 (2019).
- Moorcraft, S. Y., Gonzalez, D., Walker, B. A. Understanding next generation sequencing in oncology: A guide for oncologists. Critical Reviews in Oncology/Hematology. 96 (3), 463-474 (2015).
- Haile, S., et al. Automated high throughput nucleic acid purification from formalin-fixed paraffin-embedded tissue samples for next generation sequence analysis. PLoS One. 12 (6), 0178706(2017).
- Oh, E., et al. Comparison of accuracy of whole-exome sequencing with formalin-fixed paraffin-embedded and fresh frozen tissue samples. PLoS One. 10 (12), 0144162(2015).
- Holley, T., et al. Deep clonal profiling of formalin fixed paraffin embedded clinical samples. PLoS One. 7 (11), 50586(2012).
- Network T.C.G.A.R. TCGA Tissue sample requirements: High quality requirements yield high quality data. , Available from: https://www.cancer.gov/about-nci/organization/ccg/structural-genomics/tcga/studied-cancers (2021).
- Javey, M., et al. Innovative tumor tissue dissection tool for molecular oncology diagnostics. The Journal of Molecular Diagnostics: JMD. 23 (4), 399-406 (2021).
- Feldman, A. T., Wolfe, D. Tissue processing and hematoxylin and eosin staining. Methods in Molecular Biology. 1180, 31-43 (2014).
- Duan, Q., Zhang, H., Zheng, J., Zhang, L. Turning cold into hot: Firing up the tumor microenvironment. Trends in Cancer. 6 (7), 605-618 (2020).
- Kim, Y. W., et al. Safety evaluation and risk assessment of d-Limonene. Journal of Toxicology and Environmental Health Part B: Critical Reviews. 16 (1), 17-38 (2013).
- Foti, C., et al. Occupational contact dermatitis to a limonene-based solvent in a histopathology technician. Contact Dermatitis. 56 (2), 109-112 (2007).
- Meuse, C. W., Barker, P. E. Quantitative infrared spectroscopy of formalin-fixed, paraffin-embedded tissue specimens: paraffin wax removal with organic solvents. Applied Immunohistochemistry and Molecular Morphology. 17 (6), 547-552 (2009).
- Schmeller, J., et al. Setting out the frame conditions for feasible use of FFPE derived RNA. Pathology - Research and Practice. 215 (2), 381-386 (2019).
- Prema, V., et al. Biofriendly substitutes for xylene in deparaffinization. Journal of Pharmacy and Bioallied Sciences. 12, Suppl 1 623-630 (2020).
- Ennishi, D., et al. Double-hit gene expression signature defines a distinct subgroup of germinal center B-cell-like diffuse large B-cell lymphoma. Journal of Clinical Oncology. 37 (3), 190-201 (2019).
- Alizadeh, A. A., et al. Distinct types of diffuse large B-cell lymphoma identified by gene expression profiling. Nature. 403 (6769), 503-511 (2000).
- Rosenwald, A., et al. The use of molecular profiling to predict survival after chemotherapy for diffuse large-B-cell lymphoma. The New England Journal of Medicine. 346 (25), 1937-1947 (2002).
- Scott, D. W., et al. High-grade B-cell lymphoma with MYC and BCL2 and/or BCL6 rearrangements with diffuse large B-cell lymphoma morphology. Blood. 131 (18), 2060-2064 (2018).
- Scott, D. W., et al. Determining cell-of-origin subtypes of diffuse large B-cell lymphoma using gene expression in formalin-fixed paraffin-embedded tissue. Blood. 123 (8), 1214-1217 (2014).
- Heinrich, M. A., Mostafa, A., Morton, J. P., Hawinkels, L., Prakash, J. Translating complexity and heterogeneity of pancreatic tumor: 3D in vitro to in vivo models. Advanced Drug Delivery Reviews. 174, 265-293 (2021).
- Erickson, H. S., Gillespie, J. W., Emmert-Buck, M. R. Tissue microdissection. Methods in Molecular Biology. 424, 433-448 (2008).
- Geiersbach, K., et al. Digitally guided microdissection aids somatic mutation detection in difficult to dissect tumors. Cancer Genetics. 209 (1-2), 42-49 (2016).
- de Bruin, E. C., et al. Macrodissection versus microdissection of rectal carcinoma: minor influence of stroma cells to tumor cell gene expression profiles. BMC Genomics. 6, 142(2005).
- Ramsower, C. A., et al. Clinical laboratory validation of the MCL35 assay for molecular risk stratification of mantle cell lymphoma. Journal of Hematopathology. 13 (4), 231-238 (2020).
- Maguire, A., et al. Enhanced DNA repair and genomic stability identify a novel HIV-related diffuse large B-cell lymphoma signature. International Journal of Cancer. 145 (11), 3078-3088 (2019).
- Rosenwald, A., Staudt, L. M. Gene expression profiling of diffuse large B-cell lymphoma. Leukemia & Lymphoma. 44, Suppl 3 41-47 (2003).
- Wright, G. W., et al. A probabilistic classification tool for genetic subtypes of diffuse large B cell lymphoma with therapeutic implications. Cancer Cell. 37 (4), 551-568 (2020).
- Hilton, L. K., et al. The double-hit signature identifies double-hit diffuse large B-cell lymphoma with genetic events cryptic to FISH. Blood. 134 (18), 1528-1532 (2019).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon