Method Article
Évaluation rapide de la qualité des protéines membranaires par chromatographie d’exclusion de taille fluorescente
Dans cet article
Résumé
Le présent protocole décrit une procédure pour effectuer une chromatographie d’exclusion de taille fluorescente (FSEC) sur les protéines membranaires afin d’évaluer leur qualité pour l’analyse fonctionnelle et structurale en aval. Des résultats CSEM représentatifs recueillis pour plusieurs récepteurs couplés aux protéines G (RCPG) dans des conditions solubilisées et sans détergent sont présentés.
Résumé
Au cours de l’élucidation structurelle des protéines membranaires et de la caractérisation biophysique, il est courant d’essayer de nombreuses constructions protéiques contenant différentes étiquettes, troncations, délétions, insertions de partenaires de fusion et mutations stabilisatrices pour en trouver une qui n’est pas agrégée après extraction de la membrane. De plus, le criblage tampon pour déterminer le détergent, l’additif, le ligand ou le polymère qui fournit la condition la plus stabilisatrice pour la protéine membranaire est une pratique importante. La caractérisation précoce de la qualité des protéines membranaires par chromatographie d’exclusion de taille fluorescente fournit un outil puissant pour évaluer et classer différentes constructions ou conditions sans avoir besoin de purification des protéines, et cet outil minimise également les besoins en échantillons. Les protéines membranaires doivent être marquées par fluorescence, généralement en les exprimant avec une étiquette GFP ou similaire. La protéine peut être solubilisée directement à partir de cellules entières, puis grossièrement clarifiée par centrifugation; Par la suite, la protéine est transmise dans une colonne d’exclusion de taille et une trace fluorescente est collectée. Ici, une méthode d’exécution de l’ESCR et des données CSEM représentatives sur les cibles GPCR du récepteur sphingosine-1-phosphate (S1PR1) et du récepteur de la sérotonine (5HT2AR) sont présentées.
Introduction
La chromatographie d’exclusion de taille (SEC), également connue sous le nom de chromatographie par filtration sur gel, est couramment utilisée en science des protéines1. Au cours de la SEC, les protéines sont séparées en fonction de leur rayon hydrodynamique, qui est fonction de la taille et de la forme de la protéine2. En bref, cette séparation est obtenue en appliquant les échantillons de protéines sous flux sur un lit garni de billes poreuses qui agissent comme un tamis moléculaire. Les billes utilisées sont souvent de l’agarose réticulée avec une gamme définie de tailles de pores pour permettre aux protéines d’entrer ou d’être exclues des pores des billes 3,4,5,6,7. Les protéines avec des rayons hydrodynamiques plus petits passent une plus grande proportion de temps dans les pores et, par conséquent, circulent à travers le lit garni à un rythme plus lent, tandis que les protéines plus grosses passent une plus grande proportion de temps à l’extérieur des billes (le volume exclu) et se déplacent à travers le lit garni à un rythme plus rapide. SEC peut être utilisé comme étape de purification des protéines lorsqu’une colonne préparatoire est utilisée1. Lorsqu’une colonne analytique est utilisée, la SEC peut être utilisée pour analyser la qualité et les propriétésdes protéines 2. Par exemple, les agrégats de protéines qui peuvent être présents dans un échantillon et qui indiquent des protéines de mauvaise qualité ont tendance à être très gros, ce qui signifie qu’ils ne voyagent que dans le volume exclu et, par conséquent, sont élués de la colonne au point le plus précoce; Ce volume est appelé volume de colonne vide ou volume vide. De plus, des étalons de poids moléculaire peuvent être utilisés pour calibrer la colonne, ce qui permet d’interpoler une masse moléculaire estimée de la protéine d’intérêt à partir d’une courbe standard.
En règle générale, l’absorbance des protéines à 280 nm est utilisée pour surveiller l’élution des protéines à partir d’une colonne d’exclusion de taille. Cela limite l’utilisation de la SEC comme outil d’analyse jusqu’à ce que la protéine d’intérêt soit en grande partie exempte de protéines contaminantes, par exemple, à la dernière étape de la purification des protéines. Cependant, la SEC fluorescente (FSEC) utilise une protéine d’intérêt qui est marquée par fluorescence. Par conséquent, un signal fluorescent peut être utilisé pour surveiller spécifiquement l’élution de la protéine d’intérêt en présence d’autres protéines ou même de mélanges bruts 8,9. De plus, comme les signaux fluorescents sont très sensibles, une analyse réussie peut être effectuée sur des échantillons avec des quantités de protéines extrêmement faibles. La protéine d’intérêt est souvent marquée par fluorescence en incluant une protéine fluorescente verte (GFP) ou une étiquette GFP améliorée (eGFP) dans la construction d’expression. Le signal fluorescent peut ensuite être surveillé par excitation à 395 nm ou 488 nm et détection de l’émission fluorescente à 509 nm ou 507 nm pour GFP ou eGFP, respectivement10.
L’avantage de l’utilisation d’un signal fluorescent pour surveiller l’élution des protéines à partir d’une colonne SEC fait de la CEMN un outil précieux pour analyser les échantillons de protéines membranaires lorsque les niveaux d’expression sont particulièrement faibles par rapport aux protéines solubles. Fondamentalement, la qualité et les propriétés des protéines membranaires peuvent être analysées directement après solubilisation à partir de lysats bruts sans qu’il soit nécessaire d’optimiser le processus de purificationau préalable 11,12. Pour ces raisons, le CEMN peut être utilisé pour analyser rapidement la qualité de la protéine membranaire tout en explorant les différents facteurs qui peuvent être nécessaires pour améliorer le comportement de la protéine membranaire en solution. Par exemple, il est courant d’essayer de nombreuses constructions contenant différentes étiquettes, troncations, délétions, insertions de partenaires de fusion et mutations stabilisatrices pour en trouver une qui n’est pas agrégée après extraction de la membrane13,14. De plus, le criblage tampon pour déterminer le détergent, l’additif, le ligand ou le polymère qui fournit l’état le plus stabilisateur pour la protéine membranaire peut définir la meilleure composition tampon pour la purification des protéines ou pour assurer la stabilité pour les utilisations en aval, telles que les essais biophysiques ou la caractérisation structurale.
Ainsi, l’objectif global de la méthode FSEC est de recueillir un profil d’élution sur colonne SEC pour une protéine membranaire cible d’intérêt. De plus, comme la fluorescence est utilisée, cette trace SEC est collectée le plus tôt possible dans l’optimisation des constructions et des conditions avant toute purification prolongée. La trace FSEC peut être utilisée comme outil comparatif pour juger de la probabilité de succès de la purification d’une protéine membranaire avec différentes conditions tampons ou constructions protéiques membranaires. De cette façon, la collecte de profils FSEC peut être utilisée comme un processus itératif rapide pour arriver à une conception de construction optimale et à une composition tampon avant de dépenser des efforts pour générer les quantités de protéines pures requises pour d’autres méthodes d’analyse.
Protocole
1. Préparation du détergent et du tampon pour l’ESCN
- Préparez une solution mère de détergent.
- Pour préparer une solution mère de 20 mL, peser 4 g de poudre de dodécyl maltoside (DDM) et 0,4 g de poudre d’hémisuccinate de cholestéryle (CHS) et obtenir 20 mL avec duH2O distillé de qualité laboratoire.
- Après avoir ajouté tous les composants, mélanger avec une inversion bout à bout à 4 °C jusqu’à ce que les composants soient complètement solubilisés. Un mélange de bout en bout pendant la nuit à 4 °C est recommandé.
- Aliquote et stocker les stocks de détergent à −20 °C jusqu’à utilisation. Si le stock doit être utilisé immédiatement, entreposez-le sur de la glace.
NOTA : Le stock de détergent standard utilisé dans ce travail était un mélange de 20 % (p/v) de DDM et de 2 % (p/v) de CHS (voir le tableau des matières). Différents détergents peuvent être utilisés (p. ex. lauryl maltose néopentylglycol; LMNG), ou l’utilisation d’une extraction sans détergent avec des polymères tels que l’acide styrène-maléique (SMA) peut être testée. Cela doit être décidé lors de la conception des conditions expérimentales à tester.
- Préparez un tampon de solubilisation.
- Préparer un tampon de solubilisation en combinant le poids ou le volume correct des composants pour obtenir une concentration finale de 100 mM HEPES, 200 mM de NaCl, 20 % (v/v) de glycérol et 1x cocktail inhibiteur de protéase (voir le tableau des matériaux) dans un bécher.
REMARQUE : Dans la présente étude, la préparation de 50 mL de tampon de solubilisation était suffisante pour traiter cinq échantillons. - Ajouter un volume de 0,7 (p. ex. 35 mL si vous faites 50 mL de tampon) deH2O distillé de qualité laboratoire dans le bécher.
- Mélanger sur un agitateur magnétique et, à l’aide d’un pH-mètre, ajuster le pH tampon à 7,5 par addition goutte à goutte de NaOH concentré.
- À l’aide d’une éprouvette graduée, remplir le tampon jusqu’au volume final requis avec duH2O distillé de qualité laboratoire.
- Préparer un tampon de solubilisation en combinant le poids ou le volume correct des composants pour obtenir une concentration finale de 100 mM HEPES, 200 mM de NaCl, 20 % (v/v) de glycérol et 1x cocktail inhibiteur de protéase (voir le tableau des matériaux) dans un bécher.
- Préparez le tampon d’exécution SEC.
- Préparer le tampon courant SEC en combinant le poids ou le volume correct des composants pour obtenir une concentration finale de 100 mM HEPES, 150 mM NaCl et 10% (v/v) de glycérol dans un bécher.
REMARQUE : Dans la présente étude, la préparation de 600 mL de tampon SEC était suffisante pour effectuer cinq échantillons. - Ajouter un volume de 0,7 (p. ex. 420 mL si vous faites 600 mL de tampon) deH2Odistillé de qualité laboratoire dans le bécher.
- Mélanger sur un agitateur magnétique et, à l’aide d’un pH-mètre, ajuster le pH tampon à 7,5 par addition goutte à goutte de NaOH concentré.
- À l’aide d’une éprouvette graduée, remplir le tampon jusqu’au volume final requis avec duH2O distillé de qualité laboratoire.
- Filtrer le tampon SEC à travers un filtre à pores de 0,45 μm sous vide (voir le tableau des matériaux).
- Une fois que le tampon a traversé le filtre, dégazez-le en le laissant sous vide jusqu’à ce qu’il n’apparaisse plus de bulles lorsqu’il est secoué.
- Ajouter une concentration finale de 0,03 % (p/v) de DDM et de 0,003 % (p/v) de CHS au tampon SEC en ajoutant le volume requis du stock de détergent préparé à l’étape 1 (p. ex., 0,9 mL si vous fabriquez 600 mL de tampon SEC).
- Pré-refroidir le tampon avant utilisation.
REMARQUE: Différents tampons peuvent être utilisés en fonction des conditions testées. Par exemple, si vous testez l’effet de différents détergents sur la protéine d’intérêt, un tampon avec le même détergent que celui utilisé pour solubiliser la protéine devrait idéalement être fabriqué. Si vous testez des conditions sans détergent avec SMA, le détergent doit être complètement omis du tampon SEC. Cependant, consultez la section de discussion pour plus de détails sur les modifications du protocole pour le dépistage des détergents.
- Préparer le tampon courant SEC en combinant le poids ou le volume correct des composants pour obtenir une concentration finale de 100 mM HEPES, 150 mM NaCl et 10% (v/v) de glycérol dans un bécher.
2. Préparation des échantillons pour la CEMN
- Préparez les granulés de cellule.
REMARQUE : Le point de départ de l’essai est la récolte de la pastille cellulaire à partir d’une culture d’expression cellulaire en suspension de la protéine d’intérêt marquée GFP (ou marquée par fluorescence). Les moments et les conditions exacts de récolte dépendront de la protéine exprimée, de la lignée cellulaire utilisée, des conditions dans lesquelles les cellules ont été cultivées et de la méthode par laquelle l’expression de la protéine a été induite. Ces détails dépassent le cadre de ce protocole. Dans cette étude, 0,5 à 1 g de pastille cellulaire Sf21 a été utilisée par condition à tester, ce qui correspond à 25-50 mL de culture 2-3 jours après l’infection avec environ 4 x 106 cellules viables / mL d’une dilution 1:20 du baculovirus P2. Veuillez noter que le protocole décrit ici a été testé et s’est avéré tout aussi efficace avec des poids humides similaires de granulés cellulaires provenant d’autres lignées eucaryotes (par exemple, HEK293E).- Lorsque les cellules des cultures en suspension sont prêtes à être récoltées, transférer des aliquotes de culture de 25 à 50 mL dans des tubes coniques de 50 mL.
- Contrebalancer les tubes et utiliser une centrifugeuse de paillasse dans un seau pivotant (voir le tableau des matériaux) à 2 000 x g pendant 15 minutes à température ambiante pour enduire les cellules.
- Retirez et jetez le surnageant de culture en le versant doucement, ou utilisez une pipette de 50 ml avec une charge de pipette si la pastille de cellule est particulièrement lâche.
- Si les cellules doivent être utilisées immédiatement pour l’analyse, placez la pastille de cellule sur de la glace et passez directement à l’étape 5. Si les cellules doivent être conservées pour une utilisation ultérieure, congelez-les en les plaçant à -80 °C.
- Si la pastille de cellule a été conservée à -80 °C, décongelez-la rapidement en l’incubant à température ambiante pendant 15 minutes ou jusqu’à ce que l’échantillon ne soit plus congelé. Déplacez immédiatement l’échantillon sur la glace après cette étape.
- Remettez l’échantillon en suspension et solubilisez.
- Ajouter 2 mL du tampon de solubilisation (étape 1.2) à la pastille de cellule.
- Incuber avec une inversion bout à bout à 4 °C pendant 15-30 minutes jusqu’à homogénéité.
- Ajouter le stock de détergent prémélangé (étape 1.1) (p. ex., 100 μL de 20 % de DDM/2 % de CHS) pour obtenir une concentration finale de 1 % DDM/0,1 % CHS.
- Solubiliser pendant 30 min avec inversion bout à bout à 4 °C.
REMARQUE : Si cela est souhaitable, plusieurs conditions peuvent être testées en parallèle. Le nombre d’échantillons parallèles pouvant être traités simultanément dépendra du système disponible pour exécuter l’expérience SEC. Dans la configuration décrite ici, il était possible de traiter jusqu’à cinq échantillons à la fois.
- Effectuez une étape de centrifugation à basse vitesse.
- Centrifuger l’échantillon dans une centrifugeuse de paillasse prérefroidie (4 °C) dans un seau pivotant à 2 000 x g pendant 15 min.
- Effectuez une étape de centrifugation à grande vitesse.
- Transférer délicatement le surnageant de la centrifugation à basse vitesse vers des tubes d’ultracentrifugation (p. ex. tubes de 0,5 à 2 mL) à l’aide d’une aiguille émoussée fixée à une seringue de 5 mL, en prenant soin de ne pas perturber la pastille de l’essorage à basse vitesse.
- Équilibrer les paires de tubes à 0,05 g près et les placer dans un rotor à ultracentrifugation à angle fixe (voir le tableau des matériaux).
- Centrifuger à 4 °C pendant 30 min à 250 000 x g.
3. Chromatographie d’exclusion de taille (SEC)
- Préparez le système de chromatographie liquide à protéines rapides (FPLC) et équilibrez la colonne.
- Préparez le système en suivant les instructions du fabricant (voir le tableau des matériaux), par exemple, en remplissant le système avec un tampon SEC et en purgeant les pompes d’air.
- Connectez la colonne SEC au FPLC, en veillant à ce qu’aucun air ne pénètre dans la colonne. Ceci est accompli en appliquant une contre-pression à la colonne SEC avec une seringue remplie d’eau (fixée au fond de la colonne) afin d’effectuer une connexion goutte à goutte avec le chemin d’écoulement du système FPLC.
- Prééquilibrer la colonne SEC en la lavant d’abord dans 1,5 colonne (36 mL pour une colonne de 24 mL) de H2O distillé et filtré de qualité laboratoire, puis dans 1,5 volume de colonne de tampon SEC au débit et à la pression recommandés pour la colonne (voir le tableau des matériaux).
REMARQUE : Le système FPLC utilisé dans cette étude pour effectuer l’exécution de l’ESCR se trouvait dans un environnement froid et comportait une vanne à boucle à cinq positions et un collecteur de fraction à plaques à six positions. Cette configuration permet de charger cinq échantillons et de les exécuter séquentiellement dans la même colonne de manière automatisée sans nécessiter d’intervention manuelle entre les exécutions. Pour mener les expériences SEC, une colonne préemballée de 24 mL disponible dans le commerce a été utilisée (voir le tableau des matériaux), qui contenait une résine permettant de résoudre les protéines de la gamme de poids moléculaire de 10 à 600 kDa. Si la protéine d’intérêt est particulièrement grande, une matrice de colonne alternative pourrait être utilisée à la place, permettant la séparation de protéines jusqu’à 5 000 kDa de poids moléculaire. Veuillez noter que la présence de micelles détergentes/lipidiques augmentera la taille globale de la protéine membranaire de >150 kDa, selon la protéine et le détergent utilisés.
- Appliquez l’exemple à la colonne et exécutez l’expérience SEC.
- Transférer le surnageant de l’étape de centrifugation à grande vitesse vers une seringue de 1 mL à l’aide d’une aiguille émoussée fixée à la seringue. Cela permet de récupérer l’échantillon du tube centrifuge sans perturber la pastille.
- Définissez la boucle d’exemple à charger. Remplir excessivement une boucle d’échantillon de 500 μL en injectant 600 à 700 μL de l’échantillon de la seringue dans l’orifice de chargement. Selon le système utilisé, cette étape de chargement peut être programmée dans la méthode pour s’assurer qu’aucune erreur n’est commise.
- Au cours de la méthode, injecter l’échantillon de la boucle dans la colonne en le vidant avec 4 mL de tampon SEC au débit et à la pression recommandés pour la colonne (voir le tableau des matériaux).
- Exécutez la colonne au même débit jusqu’à ce que 1,5 volume de colonne (36 mL pour une colonne de 24 mL) du tampon soit passé.
- À 0,25 du volume de la colonne (6 mL pour une colonne de 24 mL), commencez à recueillir 0,2 mL de fractions afin de recueillir 90 fractions.
REMARQUE : Comme le volume vide de la colonne devrait être de 0,3 volume de colonne, commencer la collecte des fractions immédiatement avant cela garantit que l’élution de toutes les protéines est surveillée, y compris toute protéine présente dans le volume vide.
4. Collecte et analyse de traces fluorescentes
- Transférer les échantillons du collecteur de fractions (étape 3.2.5) sur une plaque de 96 puits et lire le signal fluorescent.
- Avant de prendre une lecture de fluorescence, diluez les échantillons collectés. À l’aide d’une pipette multicanaux, transférer 90 μL deH2Odistillé de qualité laboratoire d’un réservoir à chaque puits d’une plaque opaque à fond plat de 96 puits (voir le tableau des matériaux).
- Si des fractions ont été recueillies dans un bloc de 96 puits à l’étape 3.2.5, utiliser une pipette multicanal pour transférer 10 μL des fractions SEC du bloc à la plaque opaque à fond plat à 96 puits, et mélanger en pipetant de haut en bas. Sinon, si des fractions SEC ont été recueillies dans des tubes individuels à l’étape 3.2.5, transférer 10 μL de chaque fraction dans la plaque opaque à fond plat à 96 puits une par une, en pipetant de haut en bas chaque fois pour mélanger les échantillons.
- Placez la plaque opaque à fond plat à 96 puits dans le lecteur de plaque et mesurez la fluorescence. Si GFP est l’étiquette fluorescente (utilisée ici), réglez l’excitation aussi près que possible de 488 nm et détectez l’émission fluorescente aussi près que possible de 507 nm.
NOTE: La dilution requise avant de prendre la lecture de fluorescence dépendra de la quantité totale de protéine d’intérêt présente dans la culture d’expression et de la sensibilité du lecteur de plaque utilisé. Dans les exemples présentés dans cette étude, les échantillons ont été dilués 10 fois dans de l’eau. Comme suggestion de point de départ, les fractions doivent être diluées 5 à 10 fois dans de l’eau ou un tampon avant la détection. Si le signal de trace FSEC enregistré est particulièrement faible, des dilutions plus petites ou même un échantillon non dilué peuvent être utilisées à la place. Le dispositif utilisé pour la détection dans cette étude était un lecteur de plaques capable d’excitation à 488 nm et de détecter l’émission fluorescente à 507 nm (voir le tableau des matériaux).
- Tracez les traces FSEC.
- Exportez les données du lecteur de plaques dans un format brut (texte brut ou fichier de valeurs séparées par des virgules). Ces données brutes seront tracées sur l’axe Y de la trace FSEC.
- Pour l’axe des abscisses, calculez le volume auquel chaque fraction a été collectée. Le premier puits doit être le volume d’élution auquel la première fraction a été recueillie pour tenir compte du délai de fractionnement (6 mL dans cet exemple). Les fractions suivantes doivent augmenter séquentiellement du volume de fraction recueilli (0,2 mL dans cet exemple).
- Une fois les données de l’axe X et de l’axe Y calculées, copiez et collez les données dans le logiciel graphique (voir Tableau des matériaux) afin de tracer le signal fluorescent dans chaque puits par rapport au volume auquel il a été élué de la colonne.
Remarque : La figure 1 montre une représentation schématique des étapes requises pour exécuter une expérience FSEC.
- Analysez les traces FSEC.
- Évaluer la quantité d’élution de protéines au volume vide (environ 8 mL pour une colonne de 24 mL), ce qui indique que la protéine est de très grande taille et qu’elle est probablement dépliée ou agrégée.
- Évaluez la quantité de protéines éluées dans les pics ultérieurs, qui indiquent une protéine repliée. On s’attend à ce que cela se situe entre 10 mL et 16 mL pour une colonne de 24 mL selon la taille de la protéine (lorsqu’elle est associée à une micelle détergente/lipidique). Portez une attention particulière à la forme du pic, en particulier s’il s’agit d’un pic large ou divisé de plus de 3 à 4 mL (pour une colonne de 24 mL), car cela indique un échantillon polydispersé.
- Calculez le rapport entre la hauteur du pic monomère et la hauteur du pic du vide en divisant le signal maximal enregistré dans le pic monomère par le signal maximal dans le pic vide.
NOTE: Cette valeur représente l’indice de monodispersité et permet une mesure quantitative de la qualité des protéines; Des valeurs plus élevées indiquent la meilleure qualité possible, tandis que les valeurs inférieures à 1 indiquent des échantillons problématiques, car ils contiennent plus de protéines agrégées que de protéines repliées. - Si plusieurs essais FSEC doivent être comparés et que la caractéristique la plus importante est la quantité de monomère dans chaque cas, tracez les traces comme le signal brut enregistré par le lecteur de plaques (par exemple, RFU).
REMARQUE: Si une comparaison de la quantité de monomère par rapport à la protéine dépliée est plus importante, le signal doit être normalisé à un pourcentage du signal total en utilisant les lectures minimale et maximale dans la trace, ce qui accentuera les différences dans le rapport entre les protéines agrégées et monomères entre les traces.
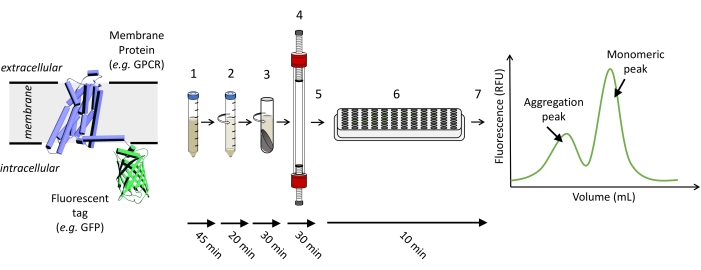
Figure 1 : Représentation schématique des étapes requises pour exécuter une expérience FSEC. (1) Les cellules qui expriment la protéine marquée par fluorescence d’intérêt sont solubilisées. (2) La solubilisation brute est d’abord clarifiée avec une rotation à basse vitesse, suivie de (3) une rotation à grande vitesse. (4) Le surnageant de l’échantillon clarifié est chargé et exécuté sur une colonne SEC appropriée, et (5) les fractions sont collectées. (6) Des échantillons des fractions sont transférés dans une plaque de 96 puits, et un signal fluorescent GFP est détecté à l’aide d’un lecteur de plaque pour (7) tracer la trace FSEC. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Résultats
Tout d’abord, la plage dynamique et les limites inférieures de détection eGFP pour le lecteur de plaques utilisé dans cette étude ont été étudiées. Un étalon eGFP purifié de concentration connue a été dilué dans un volume final de 50 μL à 50 ng·μL−1, 25 ng·μL−1, 12,5 ng·μL−1, 6,25 ng·μL−1, 3,125 ng·μL−1, 1,5625 ng·μL−1, 0,78125 ng·μL−1 et 0,390625 ng·μL−1, et la fluorescence a été lue en utilisant une excitation de 488 nm et une émission de 507 nm (Figure 2 ). Cette expérience a indiqué que le lecteur de plaques avait une limite de détection inférieure de 30 ng de protéine marquée eGFP par puits et une plage dynamique allant jusqu’à 500 ng de protéine marquée eGFP par puits avant la saturation du signal. En utilisant la valeur de la limite inférieure et en supposant que l’élution de la protéine est limitée à 0,33 du volume de la colonne, aussi peu que 1,28 μg de protéine d’intérêt marquée eGFP est nécessaire pour le chargement de la colonne SEC afin qu’un signal FSEC détectable soit observé.
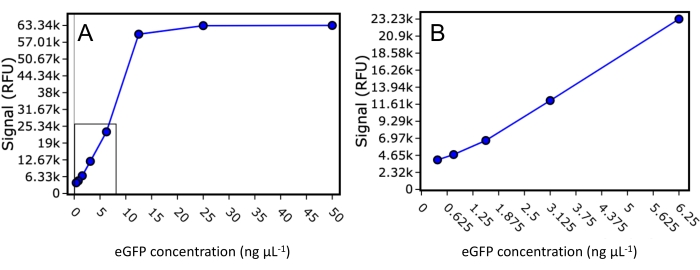
Figure 2 : courbe standard eGFP. Diagramme de dispersion du signal fluorescent pour l’étalon eGFP purifié dilué à 50 ng·μL−1, 25 ng·μL−1, 12,5 ng·μL−1, 6,25 ng·μL−1, 3,125 ng·μL−1, 1,5625 ng·μL−1, 0,78125 et 0,390625 ng·μL−1. (A) Toutes les dilutions sont affichées, y compris celles avec le signal saturé. (B) Un graphique de dispersion zoomé comprenant uniquement les étalons qui se situent dans la plage dynamique du lecteur de plaques. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Deuxièmement, la colonne de 24 mL utilisée pour cette étude a été calibrée avec des étalons de poids moléculaire. En utilisant le même tampon et les mêmes conditions de fonctionnement que pour l’analyse CEMN, les étalons de poids moléculaire bleu dextran (>2 000 kDa), ferritine (440 kDa), aldolase (158 kDa), conalbumine (75 kDa) et ovalbumine (43 kDa) ont été injectés individuellement et traversés la colonne, et des traces d’élution ont été recueillies à 280 nm d’absorption. Les volumes d’élution enregistrés étaient respectivement de 8,9 mL, 12,4 mL, 15,2 mL, 16,9 mL et 18 mL. Lorsque ces volumes d’élution ont été convertis en Kav (équation 1) et tracés en fonction des masses moléculaires logarithmiques, une courbe standard a pu être ajustée. Cela a permis d’estimer le poids moléculaire des RCPG testés dans cette étude par interpolation de la courbe standard (Figure 3). Par exemple, la trace FSEC du récepteur de sérotonine 2A (5HT2A R) des RCPG après solubilisation dans le détergent DDM indiquait un volume d’élution de 13,4 mL. Ce volume d’élution 5HT2AR se situe entre les volumes d’élution enregistrés pour la ferritine et l’aldolase et fournit un poids moléculaire estimé à environ 300 kDa. La construction 5HT 2A R utilisée dans cette étude est d’environ 50 kDa (y compris l’étiquette eGFP), ce qui signifie que si l’on suppose que 5HT2AR est monomère,250kDa de poids moléculaire pourraient être attribués à la micelle détergent/lipidique DDM. L’équation de conversion des volumes d’élution est la suivante (équation 1):
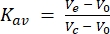 (Équation 1)
(Équation 1)
où V e est le volume d’élution, V 0 est le volume vide de colonne et Vc est le volume total de la colonne.
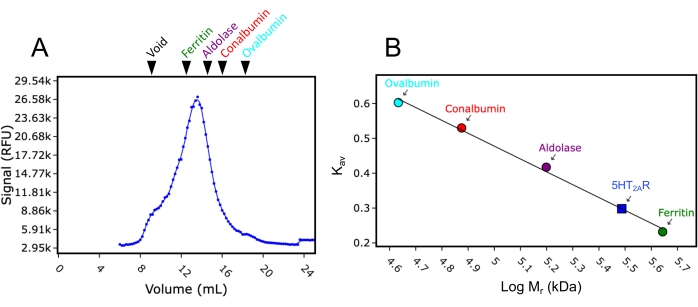
Figure 3 : Courbe d’étalonnage de la colonne SEC à l’aide des étalons de poids moléculaire. (A) Une trace FSEC représentative de 5HT2AR solubilisée dans la DDM, avec les positions d’élution relatives des étalons de poids moléculaire bleu dextran (Void), ferritine, aldolase, conalbumine et ovalbumine marquées. La ferritine, l’aldolase, la conalbumine et l’ovalbumine sont colorées respectivement en vert, violet, rouge et cyan. (B) Courbe d’étalonnage du poids moléculaire en utilisant les positions d’élution des étalons après conversion en Kav (équation 1) tracée en fonction du logarithme du poids moléculaire (Mr). Le Mr de 5HT2AR en DDM a été interpolé à partir de la courbe à l’aide de Kav et est affiché sur la courbe (carré bleu). Veuillez cliquer ici pour voir une version agrandie de cette figure.
L’ESCM a ensuite été utilisée pour évaluer la qualité et les propriétés du récepteur de la sphingosine-1-phosphate des RCPG (S1PR1)15. Les cellules d’insectes exprimant la S1PR 1 humaine marquée GFP ont été traitées pour la CEMN comme décrit dans le protocole (Figure 1).
Tout d’abord, les conditions optimales d’extraction membranaire ont été explorées en testant les détergents DDM et LMNG contre une extraction sans détergent avec SMA (Figure 4A). L’indice de monodispersité a été utilisé pour évaluer la qualité de l’échantillon de protéines et le rapport entre la protéine dans le vide (rétention de ~8 mL de la colonne) par rapport à l’échantillon monodispersé (rétention de colonne de 14-15 mL). L’échantillon solubilisé dans le LMNG présentait un profil FSEC supérieur avec une meilleure forme de pic de monomère et un pic d’agrégat protéique plus faible, ce qui indique que la solubilisation et la purification dans le LMNG étaient les conditions les plus stabilisatrices pour cette protéine membranaire. En revanche, l’échantillon solubilisé dans le DDM avait un profil CEMN relativement plus faible, avec un pic agrégé plus important et un pic monomère plus large, indiquant une polydispersité dans l’échantillon.
Deuxièmement, l’effet de l’ajout de ligand sur le profil de l’ESCN a été étudié en ajoutant de la sphingosine-1-phosphate (S1P) à l’échantillon pendant la solubilisation. Dans ce cas, le DDM a été utilisé comme réactif de solubilisation, et les traces de S1PR1 en présence et en absence de S1P ont été comparées (figure 4B). L’échantillon solubilisé en présence de S1P a montré une trace FSEC supérieure avec une agrégation réduite. Cela indiquait que la purification en présence d’un ligand était avantageuse pour améliorer la qualité de l’échantillon de protéines en stabilisant le récepteur en solution, mais était également avantageuse en tant que marqueur de substitution de l’activité protéique, car les résultats suggéraient que la protéine était correctement repliée et compétente pour la liaison au ligand.
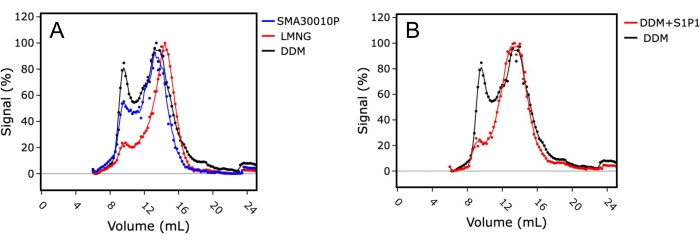
Figure 4 : ESCR utilisant un extrait brut de S1PR 1 indiquant les conditions optimales d’extraction membranaire et de liaison au ligand. (A) Comparaison des traces de S1PR1 solubilisées dans un copolymère styrène-acide maléique (SMA; bleu), lauryl maltose néopentyl glycol (LMNG; rouge) ou dodécyl maltoside (DDM; noir). (B) Comparaison des traces FSEC de S1PR 1 solubilisées dans le DDM en présence (rouge) ou en absence (noir) de l’agoniste sphingosine-1-phosphate (S1P). Veuillez cliquer ici pour voir une version agrandie de cette figure.
La CEMN a également été utilisée pour étudier la stabilité à long terme du 5HT2AR dans différentes conditions. Le 5HT 2A R humain marqué GFP a été solubilisé à partir de membranes cellulaires d’insectes dans un détergent (DDM) ou un polymèreSMAet analysé par FSEC à plusieurs moments après stockage à 4 °C ou à température ambiante (figure 5). Après plusieurs jours d’incubation, le 5HT2AR dans le DDM a montré une baisse significative de la hauteur du pic monomère à l’une ou l’autre température, et des augmentations significatives du pic global ont été observées. En revanche, 5HT2AR dans la particule lipidique SMA (SMALP) n’a pas montré de baisse significative de la hauteur du pic monomère au cours de l’expérience, indiquant que la protéine dans SMALP est restée stable plus longtemps, même à des températures défavorables. Cela peut être important lorsque l’on considère des préparations protéiques pour des applications biophysiques en aval telles que des expériences de résonance plasmonique de surface (SPR) pour lesquelles il est nécessaire que l’échantillon soit stable et actif sur une longue période de temps pour la réussite des expériences de liaison.
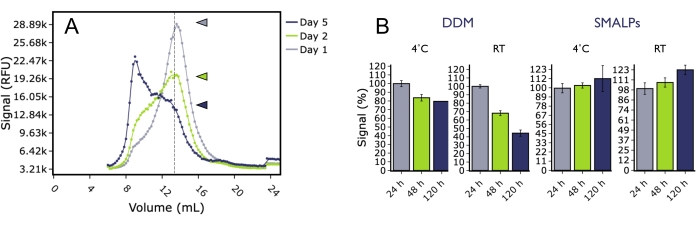
Figure 5 : Effet du temps et de la température sur la qualité du 5HT 2A R extrait de la membrane à l’aide de DDM ou de SMALP, tel qu’analysé par FSEC. (A) Traces FSEC représentatives de 5HT2AR solubilisées dans DDM et stockées à température ambiante pendant 1 jour (gris), 2 jours (vert) ou 5 jours (bleu). (B) Histogrammes de la hauteur normalisée du pic monomère pour les échantillons DDM stockés à 4 °C (bleu) ou RT (vert) par rapport aux échantillons SMALP stockés à 4 °C (gris) ou RT (noir). Les barres d’erreur sont représentatives du SEM. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Discussion
Les approches systématiques génériques de criblage d’état avec FSEC qui sont présentées ici permettent l’optimisation rapide des paramètres de solubilisation et de purification pour la production de protéines membranaires. Cela signifie que des protéines membranaires stables et fonctionnellement actives peuvent être produites rapidement pour des études biophysiques et structurelles. De plus, le CEMN peut être exécuté à l’aide d’équipement de laboratoire qui est probablement déjà en place dans les laboratoires de protéines membranaires, et il n’est donc pas nécessaire d’acheter un instrument spécialisé pour exécuter les essais.
Étapes critiques
Le temps écoulé entre le moment de la solubilisation des cellules dans le détergent et le moment où l’échantillon est transmis dans la colonne SEC (étapes 2.1.5 à 3.2.5) est critique dans le temps, et il ne doit pas y avoir de pause entre ces étapes. Toutes les étapes doivent être effectuées à 4 °C ou sur la glace, et le temps nécessaire pour effectuer ces étapes doit être réduit au minimum. Ces contraintes de temps et de température sont nécessaires pour enregistrer le profil CEMN de la protéine membranaire avant tout déploiement ou dégradation potentiel. Une fois la protéine membranaire solubilisée, le risque de dépliage, d’agrégation et de dégradation est plus grand, même à 4 °C. Idéalement, tous les échantillons pour lesquels les traces de l’ESCN doivent être comparées devraient être transmis dans la colonne SEC dans le même laps de temps après l’étape de solubilisation. Dans la pratique, cela est difficile, en particulier si les échantillons sont passés séquentiellement sur une seule colonne, mais il est possible de collecter jusqu’à cinq traces SEC à moins de 3 heures d’intervalle, et dans ce laps de temps, il ne devrait pas y avoir de dégradation significative.
Dépannage
Si, lors de l’expérience CEMN, le signal fluorescent est faible ou inexistant, il est possible que la protéine membranaire d’intérêt n’ait pas exprimé la lignée cellulaire choisie, ait une très faible expression de la lignée cellulaire choisie ou n’ait pas été solubilisée dans le détergent choisi. Si les échantillons étaient dilués avant de recueillir le signal de fluorescence et d’enregistrer la trace de l’ESCR, une première étape simple consisterait à essayer une dilution plus faible ou aucune dilution des fractions SEC. Si cela ne donne toujours pas de trace FSEC interprétable, l’expression et la solubilisation de la protéine doivent être vérifiées.
L’analyse de l’expression protéique peut être réalisée en vérifiant la fluorescence de l’échantillon après l’étape 2.2.2. S’il y a un signal fluorescent très faible ou nul de cet échantillon (p. ex., un signal très proche du fond), il y a probablement un problème avec l’expression de la protéine. Des mesures peuvent être prises pour améliorer les niveaux d’expression de la protéine membranaire, telles que le passage à une autre lignée cellulaire ou l’ajustement des conditions de croissance, l’induction de l’expression et le temps entre l’induction / infection / transfection et la récolte. Cependant, une expression protéique particulièrement faible peut indiquer une protéine membranaire instable et, par conséquent, un mauvais choix de construction.
Si l’expression a été vérifiée et qu’il y a un signal fluorescent clair au-dessus du bruit de fond avant FSEC, l’efficacité de solubilisation peut être vérifiée en mesurant le signal fluorescent restant de l’échantillon après l’étape 2.4.3 (protéine membranaire soluble) par rapport à l’échantillon après l’étape 2.2.2 (protéines totales). Il est courant que l’efficacité de solubilisation soit de 20% à 30% tout en permettant une analyse et une purification réussies de la protéine membranaire. Toutefois, si l’efficacité de solubilisation est inférieure à 20 %, un détergent différent pour la solubilisation ou des conditions de solubilisation différentes peuvent être nécessaires. Si les tentatives d’amélioration de la solubilisation échouent, cela peut indiquer une protéine membranaire particulièrement instable et, par conséquent, un mauvais choix de construction.
Si un pic d’élution très tardive est observé dans la trace de l’ESCR (p. ex., 18-24 mL), cela indique que la protéine fluorescente a un poids moléculaire de protéine beaucoup plus faible que prévu. Cela peut être causé par la dégradation de la protéine membranaire d’intérêt, ce qui entraîne une GFP « libre ». Il faut vérifier si la protéine est intacte avant et après solubilisation en utilisant la fluorescence GFP dans le gel. Si la protéine d’intérêt semble se dégrader ou être protéolysée, la quantité d’inhibiteur de la protéase peut être multipliée par deux à quatre. Cependant, une sensibilité élevée aux protéases ou aux protéines dégradées avant même la solubilisation peut indiquer une protéine particulièrement instable et, par conséquent, un mauvais choix de construction.
Modifications et applications ultérieures de la CEMN
Généralement, l’étiquette fluorescente utilisée dans l’ESCG est GFP ou eGFP, comme décrit dans ce protocole. Cependant, de nombreuses étiquettes de protéines fluorescentes différentes sont disponibles. Le choix de l’étiquette fluorescente à utiliser dépend de la présence d’un lecteur de plaques capable d’obtenir les paramètres d’excitation et d’émission corrects pour enregistrer le signal fluorescent pour l’étiquette fluorescente sélectionnée et d’un fluorophore avec peu ou pas de changement de rendement quantique dans différentes conditions environnementales. De plus, la CEMN ne se limite pas aux protéines fluorescentes, mais peut également fonctionner aussi bien avec une protéine qui a été marquée avec un colorant fluorescent. Par exemple, un colorant NTA pourrait être utilisé, ce qui se lierait favorablement aux constructions protéiques membranaires marquées à l’histidine. De plus, soit un anticorps marqué par fluorescence, marqué chimiquement avec un colorant fluorescent et spécifique pour lier la protéine membranaire d’intérêt, soit une étiquette de purification incluse dans la construction de la protéine membranaire pourrait indirectement marquer une cible pour FSEC.
Lors de la sélection des détergents à l’aide de FSEC, il est possible de choisir si le tampon utilisé pour exécuter la colonne SEC doit contenir le détergent correspondant dans lequel la protéine a été solubilisée ou si un détergent standard doit être utilisé dans toutes les séries. Une représentation plus précise du comportement de la protéine sera obtenue si toute l’expérience est réalisée avec le détergent correspondant. Cependant, cela peut prendre beaucoup de temps et gaspiller du détergent si la colonne doit être rééquilibrée dans un nouveau détergent avant chaque essai. De plus, comme l’objectif principal du criblage des détergents est de comparer les traces, les tendances resteront dans les traces même si les conditions ne sont pas idéales. Ainsi, un compromis peut être atteint selon lequel la protéine est solubilisée dans le détergent d’intérêt, mais la colonne est exécutée dans un tampon standard avec un seul détergent sur toutes les séries (par exemple, DDM)11, ce qui peut économiser du temps et des consommables de détergent.
En modifiant l’équipement FLPC utilisé, le débit du protocole FSEC peut être considérablement augmenté et l’exigence d’échantillon peut être minimisée. Par exemple, un système FPLC ou HPLC pourrait être équipé d’un échantillonneur automatique, d’une colonne d’analyse de volume de lit plus petite (telle qu’une colonne SEC analytique de 3,2 mL) et d’un détecteur fluorescent en ligne pour surveiller les traces FSEC continues directement à partir de la colonne. La configuration résultante permettrait d’effectuer plus d’exécutions FSEC dans un laps de temps plus court et supprimerait l’étape de traçage manuel, permettant ainsi de tester un plus grand nombre de conditions dans un laps de temps plus court. De plus, les besoins en échantillons seraient encore réduits, car moins d’échantillons devraient être préparés et chargés dans la colonne de l’OCEMN pour chaque essai. Cela ouvrirait la voie à la réduction des cultures d’expression à un format à base de plaques, car il faudrait si peu de matériel pour l’analyse.
Forces et faiblesses de la CEMF par rapport à d’autres méthodes
Un inconvénient de la CEMN est que les constructions de protéines membranaires doivent être conçues pour introduire le marqueur fluorescent, et lors de l’introduction, il y a une faible possibilité que l’emplacement de l’étiquette puisse interférer avec la fonction ou le repliement de la protéine membranaire d’intérêt. De plus, le protocole CEMN, tel que décrit ici, surveille les caractéristiques d’une protéine membranaire en présence de lysat cellulaire, qui est un mélange brut de protéines. Le comportement d’une protéine membranaire dans cet environnement peut être différent de celui lorsque la protéine membranaire d’intérêt est soumise à une colonne SEC préparative à la fin de la purification lorsqu’elle est complètement isolée des autres protéines. De plus, l’ESCR fournit une mesure quelque peu qualitative de la qualité des protéines. Cependant, en convertissant la trace FSEC en un indice de monodispersité, comme décrit à l’étape 4.3.3 du protocole, une mesure quantitative de la qualité des protéines peut être obtenue.
La CEMN n’est pas la seule méthode qui peut être utilisée dans l’analyse précoce des constructions protéiques membranaires, des conditions de solubilisation et de la composition tampon de purification. Les approches alternatives présentent à la fois des avantages et des inconvénients par rapport à FSEC. Par exemple, il existe des essais de thermostabilité à base de fluorophore, en particulier l’utilisation du colorant 7-diéthylamino-3-(4′-maléimidylphényl)-4-méthylcoumarine (CPM)16,17. L’avantage de cette méthode est que, contrairement au FSEC, qui fournit une mesure qualitative de la qualité des protéines, les tests de thermostabilité fournissent une mesure quantitative sous la forme d’une température de fusion relative. De plus, il n’est pas nécessaire d’introduire une étiquette fluorescente sur la construction protéique. Cependant, les inconvénients des tests de thermostabilité par rapport à l’ESCM sont que la protéine purifiée doit être utilisée et que le test n’est pas compatible avec toutes les constructions protéiques, car il repose sur les positions avantageuses des résidus de cystéine natifs dans la protéine repliée.
Une autre méthode qui présente des similitudes avec les tests de thermostabilité à base de FSEC et de fluorophore est un test qui mesure la sensibilité à la température d’une protéine membranaire. Dans ce test, la protéine est mise au défi avec différentes températures, et la protéine qui reste en solution après la centrifugation est détectée. La détection dans cette méthode a été effectuée de plusieurs façons, y compris la mesure de la fluorescence dans la solution18, la fluorescence d’un gel SDS-PAGE bande19, ou l’intensité du signal dans un Western blot20. Cependant, un inconvénient important de ces approches est que le test est très laborieux et sujet à un bruit élevé dans les résultats, car chaque point de température individuel doit être collecté indépendamment.
Enfin, plusieurs techniques biophysiques plus avancées peuvent être utilisées pour évaluer la qualité des protéines membranaires de la même manière que FSEC, par exemple, l’analyse de dispersion induite par l’écoulement21, la thermophorèse microscopique22 ou SPR. Bien que les approches soient très puissantes, l’inconvénient de ces méthodes est la nécessité de disposer d’instruments hautement spécialisés pour effectuer les analyses.
En conclusion, la CEMF fournit un outil inestimable pour les campagnes de production de protéines membranaires, et bien qu’elle ne soit pas la seule option, elle présente plusieurs avantages distincts par rapport aux autres méthodes, énumérées ci-dessus. La validation croisée des résultats par des essais orthogonaux est toujours recommandée, et aucune des méthodes discutées ci-dessus ne s’exclut mutuellement.
Déclarations de divulgation
Peak Proteins est un organisme de recherche sous contrat qui fournit l’expression des protéines, la purification, la spectrométrie de masse et la détermination structurelle moyennant des frais.
Remerciements
Nous tenons à remercier toute l’équipe de Peak Proteins pour son aide et son soutien. De la part de l’équipe de science cellulaire, nous aimerions remercier Ian Hampton pour ses précieuses idées et ses conseils dans l’expression des cellules d’insectes. Nous tenons à remercier Mark Abbott d’avoir fourni les ressources et l’occasion de poursuivre ce projet.
matériels
| Name | Company | Catalog Number | Comments |
| 1 mL and 5 mL plastic syringes | Generic | - | Syringes for transfer of samples |
| 10x EDTA Free Protease inhibitor cocktail | Abcam | ab201111 | Protease inhibitors |
| 15 mL tubes | Generic | - | 15 mL tubes for pellet preparation and solubilisation |
| 2 mL ultra-centrifuge tubes | Beckman Coulter | 344625 | Tubes for ultra-centrifuge rotor |
| 50 mL tubes | Generic | - | 50 mL tubes for cell harvest |
| 96 deep-well blocks | Greiner | 15922302 | For collecting 0.2 mL SEC fractions |
| ÄKTA V9-L loop valve | Cytiva | 29011358 | 5 posiiton loop valve for the ÄKTA FPLC system |
| ÄKTA F9-C fraction collector | Cytiva | 29027743 | 6 position plate fraction collector for the ÄKTA FPLC system |
| ÄKTA pure 25 L | Cytiva | 29018224 | FPLC system for running the experiment |
| Benchtop centrifuge (e.g. Fisherbrand GT4 3L) | Fisher Scientific | 15828722 | Centrifuge for low-speed spin |
| Blunt end filling needles | Generic | - | For transfer of samples |
| Bottle top vacuum filter | Corning | 10005490 | Bottle top vacuum filter for filtering SEC buffers |
| Cholesteryl hemisuccinate (CHS) | Generon | CH210-5GM | Additive for detergent solubilisation |
| Disposable multichannel reseviour | Generic | - | Resevior for addition of water or buffer to 96-well micro-plate |
| Dodecyl maltoside (DDM) | Glycon | D97002-C-25g | Detergent for solubilisation |
| eGFP protein standards | BioVision | K815-100 | eGFP standards for fluorescent calibration curve |
| Glycerol | Thermo Scientific | 11443297 | Glycerol for buffer preparation |
| HEPES | Thermo Scientific | 10411451 | HEPES for buffer preparation |
| High molecular weight SEC calibration standards kit | Cytiva | 28403842 | Molecular weight calibration kit for SEC |
| Lauryl maltose neopentylglycol (LMNG) | Generon | NB-19-0055-5G | Detergent for solubilisation |
| Low molecular weight SEC calibration standards kit | Cytiva | 28403841 | Molecular weight calibration kit for SEC |
| MLA-130 ultra-centrifuge rotor | Beckman Coulter | 367114 | Rotor for ultracentrifuge that fits 2 mL capacity tubes |
| Opaque 96-well flat-bottom micro-plate | Corning | 10656853 | 96-well for reading fluorescent signal in plate reader |
| Optima MAX-XP ultra-centrifuge | Beckman Coulter | 393315 | Centrifuge for high-speed spin |
| pH meter | Generic | - | For adjusting the pH of buffers during preparation |
| Prism | GraphPad | - | Graphing software for plotting traces |
| Rotary mixer | Fisher Scientific | 12027144 | Mixer for end over end mixing in the cold |
| Sodium chloride | Fisher Scientific | 10316943 | Sodium chloride for buffer preparation |
| Sodium hydroxide | Fisher Scientific | 10488790 | Sodium hydroxide for buffer preparation |
| Spectramax ID3 Plate Reader | Molecular Devices | 735-0391 | Micro-plate reader capable of reading fluorescence |
| Stirrer plate | Generic | - | For stirring buffers during preparation |
| Styrene maleic acid (SMA) | Orbiscope | SMALP 300 | Polymer for detergent free extraction |
| Superdex 200 Increase 10/300 GL | Cytiva | 28990944 | SEC column for running the experiment. The bed volume of this column is 24 mL. The recommended flow rate for this column in 0.9 ml/min (in water at 4 °C). The maximum pressure limit for this column is 5 MPa. |
| Vacuum pump | Sartorius | 16694-2-50-06 | For filtering and degassing buffers |
Références
- Burgess, R. R. A brief practical review of size exclusion chromatography: Rules of thumb, limitations, and troubleshooting. Protein Expression and Purification. 150, 81-85 (2018).
- Erickson, H. P. Size and shape of protein molecules at the nanometer level determined by sedimentation, gel filtration, and electron microscopy. Biological Procedures Online. 11 (1), 32-51 (2009).
- Lathe, G. H., Ruthven, C. R. J. The separation of substances and estimation of their relative molecular sizes by the use of columns of starch in water. Biochemical Journal. 62 (4), 665-674 (1956).
- Lathe, G. H., Ruthven, C. R. The separation of substances on the basis of their molecular weights, using columns of starch and water. Biochemical Journal. 60 (4), (1955).
- Polson, A. Fractionation of protein mixtures on columns of granulated agar. Biochimica et Biophysica Acta. 50 (3), 565-567 (1961).
- Hjertén, S., Mosbach, R. 34;Molecular-sieve" chromatography of proteins on columns of cross-linked polyacrylamide. Analytical Biochemistry. 3 (2), 109-118 (1962).
- Porath, J., Flodin, P. Gel filtration: A method for desalting and group separation. Nature. 183 (4676), 1657-1659 (1959).
- Kawate, T., Gouaux, E. Fluorescence-detection size-exclusion chromatography for precrystallization screening of integral membrane proteins. Structure. 14 (4), 673-681 (2006).
- Hattori, M., Hibbs, R. E., Gouaux, E. A Fluorescence-detection size-exclusion chromatography-based thermostability assay for membrane protein precrystallization screening. Structure. 20 (8), 1293-1299 (2012).
- Shimomura, O., Johnson, F. H., Saiga, Y. Extraction, purification and properties of aequorin, a bioluminescent protein from the luminous hydromedusan, Aequorea. Journal of Cellular and Comparative Physiology. 59 (3), 223-239 (1962).
- Drew, D., et al. GFP-based optimization scheme for the overexpression and purification of eukaryotic membrane proteins in Saccharomyces cerevisiae. Nature Protocols. 3 (5), 784-798 (2008).
- Newstead, S., Kim, H., von Heijne, G., Iwata, S., Drew, D. High-throughput fluorescent-based optimization of eukaryotic membrane protein overexpression and purification in Saccharomyces cerevisiae. Proceedings of the National Academy of Sciences of the United States of America. 104 (35), 13936-13941 (2007).
- Vaidehi, N., Grisshammer, R., Tate, C. G. How can mutations thermostabilize G-protein-coupled receptors. Trends in Pharmacological Sciences. 37 (1), 37-46 (2016).
- Hardy, D., Bill, R. M., Jawhari, A., Rothnie, A. J. Overcoming bottlenecks in the membrane protein structural biology pipeline. Biochemical Society Transactions. 44 (3), 838-844 (2016).
- Hanson, M. A., et al. Crystal structure of a lipid G protein-coupled receptor. Science. 335 (6070), 851-855 (2012).
- Alexandrov, A. I., Mileni, M., Chien, E. Y. T., Hanson, M. A., Stevens, R. C. Microscale fluorescent thermal stability assay for membrane proteins. Structure. 16 (3), 351-359 (2008).
- Harborne, S. P. D., King, M. S., Kunji, E. R. S. Thermostability assays: A generic and versatile tool for studying the functional and structural properties of membrane proteins in detergents. Biophysical Journal. 118 (4), 105-121 (2020).
- Nji, E., Chatzikyriakidou, Y., Landreh, M., Drew, D. An engineered thermal-shift screen reveals specific lipid preferences of eukaryotic and prokaryotic membrane proteins. Nature Communications. 9 (1), 4253 (2018).
- Harborne, S. P. D., et al. IMPROvER: The Integral Membrane Protein Stability Selector. Scientific Reports. 10 (1), 15165 (2020).
- Ashok, Y., Nanekar, R., Jaakola, V. -. P. Defining thermostability of membrane proteins by western blotting. Protein Engineering Design and Selection. 28 (12), 539-542 (2015).
- Pedersen, M. E., Østergaard, J., Jensen, H. Flow-induced dispersion analysis (FIDA) for protein quantification and characterization. Methods Mol Biol. 1972, 109-123 (2019).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nature Communications. 1 (1), 100 (2010).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon