Method Article
Automatisation du criblage par cryo-microscopie électronique sur plusieurs grilles à l’aide de Smart Leginon
* Ces auteurs ont contribué à parts égales
Dans cet article
Résumé
Le criblage multi-grilles par cryo-microscopie électronique (cryoEM) est souvent un processus fastidieux qui demande des heures d’attention. Ce protocole montre comment configurer une collection Leginon standard et Smart Leginon Autoscreen pour automatiser ce processus. Ce protocole peut être appliqué à la majorité des grilles de feuilles de trous cryoEM.
Résumé
Les progrès des techniques de cryo-microscopie électronique (cryoEM) au cours de la dernière décennie ont permis aux biologistes structuraux de résoudre régulièrement des complexes de protéines macromoléculaires à une résolution proche de l’atome. Le flux de travail général de l’ensemble du pipeline cryoEM implique l’itération entre la préparation des échantillons, la préparation de la grille cryoEM et le criblage des échantillons/grilles avant de passer à la collecte de données haute résolution. L’itération entre la préparation de l’échantillon/de la grille et le criblage est généralement un goulot d’étranglement majeur pour les chercheurs, car chaque expérience itérative doit optimiser la concentration de l’échantillon, les conditions tampons, le matériau de la grille, la taille du trou de la grille, l’épaisseur de la glace et le comportement des particules de protéines dans la glace, entre autres variables. De plus, une fois ces variables déterminées de manière satisfaisante, les grilles préparées dans des conditions identiques varient considérablement quant à leur capacité à être collectées ou non, de sorte qu’il est recommandé de procéder à des séances de sélection supplémentaires avant de sélectionner les grilles optimales pour la collecte de données à haute résolution. Ce processus de préparation et de criblage des échantillons/grilles consomme souvent plusieurs dizaines de grilles et plusieurs jours de temps d’opérateur au microscope. De plus, le processus de dépistage est limité à la disponibilité de l’opérateur/microscope et à l’accessibilité du microscope. Nous montrons ici comment utiliser Leginon et Smart Leginon Autoscreen pour automatiser la majorité du criblage de la grille cryoEM. Autoscreen combine l’apprentissage automatique, les algorithmes de vision par ordinateur et les algorithmes de manipulation du microscope pour éliminer le besoin d’une intervention manuelle constante de l’opérateur. Autoscreen peut charger et imager de manière autonome des grilles avec une imagerie multi-échelle à l’aide d’un système automatisé de cassettes d’échange d’échantillons, ce qui permet un criblage de grille sans surveillance pour une cassette entière. En conséquence, le temps de l’opérateur pour le criblage de 12 grilles peut être réduit à ~10 min avec Autoscreen contre ~6 h avec les méthodes précédentes qui sont entravées par leur incapacité à tenir compte de la grande variabilité entre les grilles. Ce protocole présente d’abord la configuration et les fonctionnalités de base de Leginon, puis démontre la fonctionnalité Autoscreen étape par étape, de la création d’une session modèle à la fin d’une session de criblage automatisé à 12 grilles.
Introduction
La cryo-microscopie électronique à particule unique (cryoEM) permet de déterminer la structure à résolution quasi atomique des complexes macromoléculaires purifiés. Une expérience de cryoEM à particules uniques ne nécessite qu’une ou deux grilles bien choisies sélectionnées dans un ensemble beaucoup plus large de grilles avec des conditions d’échantillon et de grille variables. Le criblage au microscope pour examiner ces grilles implique l’imagerie de chaque grille à plusieurs grossissements pour déterminer quelle grille répond à la plupart des exigences clés pour la collecte de données à haute résolution, y compris l’épaisseur de la glace, des zones suffisantes pour la collecte complète des données, la pureté des protéines, la concentration des protéines, la stabilité des protéines et les problèmes d’orientation préférés minimaux1. L’optimisation pour ces exigences clés implique souvent un retour d’information entre le criblage au microscope et les conditions de préparation telles que la production de protéines, la sélection du tampon, les détergents potentiels et le typede grille 2,3,4 (Figure 1). Le criblage de grille conventionnel est effectué manuellement ou semi-manuellement avec des logiciels tels que Leginon5, SerialEM6 et EPU7. Le criblage conventionnel oblige l’opérateur du microscope à passer des heures au microscope pour cribler plusieurs grilles, ce qui crée un goulot d’étranglement important dans le flux de travail haute résolution à particules uniques en occupant l’opérateur avec des opérations par cœur plutôt qu’avec l’optimisation de l’échantillon/de la grille.
Auparavant, Smart Leginon Autoscreen et le logiciel d’apprentissage automatique sous-jacent, Ptolemy, ont été introduits, et leurs méthodes et algorithmes sous-jacents ainsi que des exemples ont été décrits 8,9. Plusieurs autres progiciels sont capables de réaliser un criblage multi-grilles entièrement automatisé10, notamment SmartScope11, Smart EPU12 et CryoRL13,14. Pour résoudre le goulot d’étranglement du criblage, Smart Leginon permet à l’utilisateur de configurer d’abord les paramètres de criblage dans une session de microscope modèle, puis d’utiliser les paramètres de cette session de modèle comme modèle pour filtrer la cassette complète de grilles dans le chargeur automatique de microscope. Tout travail manuel pendant le criblage de la cassette est éliminé, ce qui permet à la boucle de rétroaction d’optimisation de se dérouler beaucoup plus efficacement.
Dans ce protocole, le flux de travail complet de Smart Leginon Autoscreen est décrit afin que le lecteur puisse effectuer un criblage cryoEM multi-grilles entièrement automatisé de manière indépendante. Pour ceux qui découvrent Leginon, la première section du protocole décrit l’utilisation conventionnelle de Leginon. Ces connaissances sont composées de plusieurs années d’expérience sur plusieurs microscopes à chargement automatique, qui sont ensuite développées dans la section Smart Leginon suivante du protocole. D’autres vidéos tutorielles peuvent être trouvées sur https://memc.nysbc.org.
Protocole
Pour suivre ce protocole, illustré à la figure 2, Leginon 3.6+ doit être installé sur l’ordinateur du microscope et sur un poste de travail Linux supplémentaire, et Ptolemy doit être installé sur le poste de travail Linux. Ce protocole a été développé sur plusieurs années à l’aide des microscopes Thermo Fisher Scientific (TFS) Glacios et Krios. Ce protocole suppose que le lecteur a déjà configuré Leginon, Appion15, la base de données associée, les étalonnages du microscope, effectué des alignements directs sur le microscope et mis en place deux applications Leginon : une pour la collecte standard de particules uniques et une pour la collecte de particules uniques avec Ptolémée. Les informations pour la configuration de Leginon sont disponibles ici : https://emg.nysbc.org/redmine/projects/leginon/wiki/Leginon_Manual. Les informations pour la mise en place de Ptolémée au sein de Leginon sont disponibles ici : https://emg.nysbc.org/redmine/projects/leginon/wiki/Multi-grid_autoscreening. Téléchargez Leginon de http://leginon.org et Ptolémée de https://github.com/SMLC-NYSBC/ptolemy. Leginon est sous licence Apache, version 2.0, et Ptolémée est sous licence CC BY-NC 4.0.
1. Utilisation de Leginon
- Démarrer Leginon
- Sur l’ordinateur Windows microscope, fermez tous les clients Leginon, puis rouvrez-le. Dans la station de travail Linux, ouvrez une fenêtre de terminal et tapez start-leginon.py ou l’alias approprié du système pour démarrer Leginon.
- Dans la nouvelle fenêtre Configuration de Leginon , sélectionnez Créer une session et cliquez sur Suivant.
- Sélectionnez le projet dans la liste déroulante et cliquez sur Suivant.
- Laissez le nom tel quel, sélectionnez le support approprié pour la configuration du microscope, puis cliquez sur Suivant.
- Pour la description, saisissez les informations pertinentes telles que le nom du microscope, la description de la grille/de l’échantillon et la description de l’expérience, puis cliquez sur Suivant.
- Pour le répertoire d’images, assurez-vous que le système de fichiers approprié est sélectionné et que le chemin d’accès complet est approprié pour l’enregistrement des images, puis cliquez sur Suivant.
- Sous Se connecter aux clients, cliquez sur Modifier. Dans le menu déroulant, sélectionnez tous les ordinateurs qui doivent être connectés et cliquez sur le bouton + pour chacun, puis cliquez sur OK et Suivant.
- Entrez la taille d’ouverture C2 correcte et cliquez sur Suivant. Cette valeur se trouve dans l’onglet Apertures du logiciel TFS TUI.
- Interface Leginon
- Sélectionnez Application dans la barre d’outils et cliquez sur Exécuter.
- Sélectionnez l’application appropriée dans le menu déroulant (cliquez sur Afficher tout si nécessaire). Définissez main sur l’ordinateur Leginon et scope et caméra sur cet ordinateur respectif, puis cliquez sur Exécuter. Le côté gauche de la fenêtre principale de Leginon sera rempli de nœuds.
REMARQUE : Le panneau de gauche affiche tous les nœuds Leginon. Les nœuds d’icône de caméra verts sont les images qui seront enregistrées : Grille, Carré, Trou et Exposition. Les nœuds avec le signe cible sont des images à faible grossissement pour cibler les images à plus fort grossissement. Les nœuds violets de la caméra sont les nœuds programmés pour trouver la hauteur z eucentrique et la mise au point eucentrique. De plus, il existe des nœuds pour aligner le pic sans perte, surveiller le cycle de tampon, surveiller le remplissage d’azote liquide, collecter des images de correction de gain, calculer l’épaisseur de la glace (IceT) et naviguer dans la grille à travers différents grossissements à l’aide de la platine et du décalage d’image. - Gestionnaire de préréglages
- Cliquez sur le nœud Presets_Manager. Dans ce nœud, cliquez sur l’icône du bas pour importer des préréglages ou sur l’icône au-dessus de celle-ci pour créer un nouveau préréglage à partir de l’état actuel du microscope.
- Si vous cliquez sur l’icône du bas, une fenêtre Importer des préréglages s’ouvre. Sélectionnez la GDT et la caméra numérique appropriées, puis cliquez sur Rechercher et sélectionnez la session la plus récente avec les préréglages souhaités. Mettez en surbrillance tous les préréglages souhaités et cliquez sur Importer, puis sur Terminé.
REMARQUE : le nœud Gestionnaire de préréglages doit maintenant répertorier tous les préréglages importés et créés. Il est recommandé d’avoir des préréglages pour plusieurs grossissements et mises au point, y compris gr : Grossissement de la grille, sq : Grossissement carré, hln : Grossissement du trou, ventilateur : Auto-focus, fcn : Mise au point centrale, enn : Grossissement de l’exposition (le « n » à la traîne fait référence à la nanosonde). Les paramètres prédéfinis typiques pour chaque grossissement sont indiqués dans le Tableau 1, le Tableau 2 et le Tableau 3. Notez que ce protocole utilise une taille d’ouverture C2 de 70 μm pour le Glacios, de 50 μm pour le Krios avec un Selectris X et un Falcon 4i, et de 100 μm pour le Krios avec un BioQuantum avec un K3.
- Navigation et hauteur eucentrique
- Pour vous familiariser avec le contrôle du microscope via Leginon et pour définir la hauteur z de la grille, allez dans le nœud Navigation, sélectionnez le préréglage gr en haut et cliquez sur la flèche rouge à droite pour envoyer les paramètres prédéfinis au microscope. Le microscope devrait se mettre à jour après 1 à 2 s. Une fois la mise à jour effectuée, cliquez sur le bouton de l’appareil photo à droite pour acquérir une image.
- À l’aide de l’outil curseur, sélectionnez un carré de grille pour déplacer la scène. Cliquez sur le grossissement carré , puis sur la flèche rouge pour envoyer au microscope, puis cliquez sur le bouton de l’appareil photo pour acquérir une image.
- Accédez au nœud Z_Focus et cliquez sur le bouton Simuler la cible en haut, près du milieu des boutons. Pendant la collecte des images pour la mise au point de l’inclinaison de la scène, passez en mode corrélation et observez le pic pour vous assurer qu’il se trouve dans le coin de l’image de corrélation. Une fois la mise au point terminée, assurez-vous que la scène est réglée à la hauteur z de la grille.
- Correction du coma
NOTE : Cette sous-section suppose que des alignements directs ont déjà été effectués et que des corrections de coma n’ont pas été effectuées.- Naviguez jusqu’à une zone de la grille qui produit des anneaux Thon transparents, tels qu’un substrat de carbone.
REMARQUE : Un réseau transversal peut être utilisé si la collecte doit être effectuée sur une grille dorée. - Dans les paramètres Beam_Tilt_Image, assurez-vous que l’ordre des préréglages inclut uniquement fcn avec quatre directions d’inclinaison à un angle de 0,005 radians.
- Cliquez sur Simuler la cible pour créer un tableau Zemlin. Cliquez sur Tableau sur le côté gauche de la fenêtre principale pour afficher le tableau.
- Corrigez le coma en comparant les transformées de Fourier gauche et droite entre elles et les transformées de Fourier en haut et en bas entre elles. Si les paires d’images ne sont pas identiques, cliquez d’abord sur l’icône du curseur à droite des ajustements d’image, puis cliquez légèrement décentré dans l’image Tableau dans le sens de la différence et attendez qu’un nouvel ensemble de transformées de Fourier soit collecté. Répétez jusqu’à ce que les transformées de Fourier soient identiques.
REMARQUE : chaque clic sur Tableau prend plusieurs secondes et aucun clic supplémentaire ne doit être effectué pendant ce temps.
- Naviguez jusqu’à une zone de la grille qui produit des anneaux Thon transparents, tels qu’un substrat de carbone.
- Gagner des références
REMARQUE : Ignorez cette section si l’appareil photo dispose de références matérielles automatiques.- Dans le nœud Navigation, envoyez un préréglage à faible grossissement, tel que gr, et accédez à une zone où il n’y a pas d’obstruction à la trajectoire du faisceau.
- Vérifiez que la position de la platine se trouve à un endroit non obstrué par le trajet du faisceau en prenant une image à grossissement moyen à l’aide du préréglage sq ou hln .
- Envoyez le préréglage à fort grossissement au microscope.
- Dans les paramètres du nœud Correction, sélectionnez les informations appropriées sur l’instrument et configurez la configuration de la caméra pour qu’elle corresponde aux paramètres de collection.
- Collectez une image de référence sombre en fermant les vannes de colonne du microscope, puis dans le nœud Correction, sélectionnez Sombre et les deux canaux dans les menus déroulants en haut et cliquez sur le bouton Acquérir la caméra à droite.
- Une fois terminé, sélectionnez Bright dans le menu déroulant et cliquez sur Acquérir. Leginon ouvrira automatiquement les vannes de colonne.
- Vérifiez que le gain a été correctement collecté en sélectionnant Corrigé dans le menu déroulant, en cliquant sur Acquérir et en observant l’image résultante.
- Image de référence de l’épaisseur de la glace
- Si le microscope dispose d’un filtre d’énergie, dans les paramètres du nœud IceT, cochez Collecter l’image de l’épaisseur de la glace, entrez 395 pour le chemin libre moyen et remplissez le reste des valeurs pour la configuration.
- Si le microscope n’a pas de filtre d’énergie, dans le nœud Navigation, envoyez le préréglage enn au microscope et cliquez sur Acquérir. Notez la valeur moyenne des pixels sur le côté gauche. Dans les paramètres du nœud IceT, cochez Calculer l’épaisseur de la glace à partir de la diffusion limitée à l’ouverture, entrez 1055 pour le coefficient ALS et la valeur moyenne des pixels mesurée.
REMARQUE : Les valeurs 395 et 1055 ont été déterminées pour le TFS Krios et le Glacios, respectivement, comme décrit précédemment16, et peuvent devoir être recalibrées pour différentes configurations de microscope.
- Étalonnage de la dose d’image
- Dans le Preset_Manager, sélectionnez le préréglage enn et cliquez sur le bouton de l’appareil photo (Acquérir l’image de dose pour le préréglage sélectionné). Vérifiez la dose mesurée sur le fond. Si elle est proche de la valeur attendue (généralement entre 30 et 70), cliquez sur OUI.
- Alignements prédéfinis
- Dans le Preset_Manager, vérifiez tous les préréglages de grossissement élevé (enn, fcn et fan) pour vous assurer que le décalage de l’image et le décalage du faisceau sont de 0, 0.
- Sur l’ordinateur du microscope, accédez à une zone de carbone.
- Sur l’ordinateur Leginon dans le nœud Navigation, acquérez une image avec le préréglage gr .
- Trouvez un objet d’intérêt et accédez à cet emplacement à l’aide de l’outil de curseur.
- Acquérez une image avec le préréglage hln et déplacez une partie unique de cet objet d’intérêt vers le centre à l’aide de l’outil curseur.
- Acquérez une image avec le préréglage enn et déplacez-la vers la même partie unique de l’objet d’intérêt à l’aide de l’outil curseur.
- Sélectionnez le décalage d’image dans le menu déroulant et acquérez une image avec le préréglage hln. Déplacer vers la même partie unique de l’objet d’intérêt avec l’outil curseur.
- Dans le Presets_Manager, sélectionnez le préréglage hln , cliquez sur lebouton s ettings et importez le décalage de l’image à partir de la navigation en cliquant sur la flèche verte gauche à côté des valeurs de décalage de l’image .
- Répétez les étapes 2.9.7 et 2.9.8 pour les préréglages sq et gr .
- Atlas quadrillé
- Sur l’ordinateur du microscope, fermez les vannes de la colonne et rétractez l’ouverture de l’objectif. Accédez au nœud Grid_Targeting. Dans les paramètres, modifiez l’étiquette de la grille. Choisissez le rayon souhaité de l’atlas (le rayon maximum est de 0,0009 m). Cliquez sur OK. Cliquez ensuite sur le bouton Calculer le calculateur de l’atlas en haut et cliquez sur le bouton vert Jouer ('Soumettre les cibles').
- Dans le nœud Square_Targeting, les images de la grille seront collectées et assemblées pour former un atlas. Effectuez un zoom avant et arrière à l’aide du menu déroulant et ajustez le contraste et la luminosité. Utilisez les barres de défilement pour vous déplacer dans la grille.
- Une fois l’atlas collecté, insérez l’ouverture de l’objectif si vous le souhaitez.
- Si le microscope est équipé d’un filtre énergétique, sélectionnez une cible de référence au centre d’un carré cassé, appuyez sur le bouton Lecture et procédez à l’alignement ZLP dans la sous-section suivante. Sinon, ignorez l’étape d’alignement ZLP.
- Alignement ZLP
- Dans les paramètres du nœud Align_ZLP, sélectionnez la position d’étapen pour déplacer la cible de référence et sélectionnez le gestionnaire de préréglages comme déménageur. Désélectionnez le conditionneur de dérivation, puis appuyez sur OK.
REMARQUE : L’alignement ZLP doit maintenant être configuré de manière à ce que le microscope se déplace périodiquement vers la cible de référence et exécute la routine d’alignement ZLP de la caméra. Des temps de réalignement ZLP de 30 min et 60 min sont généralement sûrs pour les filtres énergétiques Gatan BioQuantum et TFS Selectris X, respectivement. Ces valeurs varient en fonction des conditions du filtre énergétique, y compris une humidité constante, une température constante, une isolation du champ électromagnétique et une isolation des vibrations.
- Dans les paramètres du nœud Align_ZLP, sélectionnez la position d’étapen pour déplacer la cible de référence et sélectionnez le gestionnaire de préréglages comme déménageur. Désélectionnez le conditionneur de dérivation, puis appuyez sur OK.
- Configuration du ciblage du modèle de trou
- Dans le nœud Square_Targeting, sélectionnez plusieurs cibles d’acquisition , puis appuyez sur Lecture.
- Dans les paramètres du nœud Hole_Targeting, assurez-vous que les options Autoriser la vérification utilisateur des cibles sélectionnées et Mettre en file d’attente sont cochées. Consultez également Ignorer le détecteur de trous automatisé pour le moment. Cliquez sur Appliquer, puis sur OK.
- Dans la fenêtre principale, utilisez Ctrl-Maj-clic droit pour supprimer toutes les cibles. Sélectionnez le curseur d’acquisition et placez les cibles. Sélectionnez le curseur de focus et placez une cible de focus entre les cibles d’acquisition. Cliquez sur Lecture.
- Pour l’image Hole_Targeting suivante, décochez l’option Ignorer l’outil de recherche automatique de trous dans les paramètres, puis cliquez sur Appliquer et OK. Supprimez les cibles automatiques avec Ctrl-Maj-clic droit.
- Sélectionnez l’outil règle et mesurez le diamètre à travers un trou. Dans les paramètres du gabarit, remplacez le diamètre final du gabarit par le diamètre du trou mesuré. Ne modifiez pas le diamètre du gabarit d’origine. Cliquez sur Tester. Si les pics brillants ne sont pas au centre de chaque trou, augmentez le diamètre final du gabarit. Lorsque vous avez terminé, cliquez sur OK.
- Dans les paramètres de seuil, choisissez une valeur pour A qui segmente les trous individuellement lorsque vous cliquez sur Tester . Cliquez sur OK lorsque vous êtes satisfait.
- Dans les paramètres Objets blob, saisissez les valeurs et cliquez sur Tester. La valeur Nombre maximal d’objets blob est 1, donc un seul objet blob s’affiche. Cliquez sur OK.
- Dans Paramètres de treillis, utilisez l’outil Règle pour mesurer la distance entre deux trous (centre à centre). Entrez la valeur dans Espacement et cliquez sur Tester. La goutte se transformera en un point de réseau. Cliquez sur OK.
- Accédez aux paramètres d’acquisition et optimisez les cibles d’acquisition à l’aide des seuils d’épaisseur de glace et du bouton Tester le ciblage. Obtenez des informations sur l’épaisseur de la glace en survolant les points du réseau.
- Si les cibles d’acquisition ne sont pas satisfaisantes, utilisez l’outil règle pour mesurer la distance et l’angle entre le point du treillis et l’emplacement souhaité pour une cible d’acquisition. Supprimez les points du modèle de cible d’acquisition précédents. Cliquez sur Remplissage automatique, mettez 4 pour le nombre de cibles et modifiez le rayon et l’angle aux valeurs mesurées. Cliquez sur OK. Cochez la case Appliquer le seuil d’épaisseur de glace sur les cibles d’acquisition convoluées par modèle.
- Une fois satisfait des points du réseau et des seuils d’épaisseur de la glace, cliquez sur le bouton Soumettre les cibles .
- Répétez l’une des étapes ci-dessus si nécessaire pour chaque carré sélectionné. Soumettez l’ensemble de la file d’attente avec le bouton Soumettre les cibles en file d’attente une fois que toutes les cibles carrées sont soumises.
- Leginon commencera à faire la mise au point et à imager chaque ensemble de cibles. Dans le nœud Z_Focus, assurez-vous que la hauteur eucentrique est correctement trouvée.
- Configuration du ciblage du modèle d’exposition
- Dans le nœud Ciblage de l’exposition, les images d’agrandissement des trous s’affichent. Utilisez Ctrl-Maj-clic droit pour supprimer les cibles automatiques.
- Mesurez le diamètre d’un trou avec l’outil règle. Dans les paramètres du modèle, saisissez le diamètre dans Diamètre final du modèle et cliquez sur Tester. Un pic devrait maintenant être au centre de chaque trou. Ajustez les valeurs de diamètre si nécessaire.
- Dans les paramètres de seuil, ajustez la valeur A jusqu’à ce que l’image de test binarisée affiche des zones blanches uniquement à l’endroit où se trouvent les trous.
- Dans les paramètres Objets blob, cliquez sur Tester. Une goutte par trou segmenté doit apparaître. Augmentez la bordure pour supprimer les taches des bords de l’image, si vous le souhaitez.
- Dans les paramètres de Treillis, cliquez sur Tester. Ajustez les paramètres jusqu’à ce que toutes les gouttes se soient transformées en points de treillis. Cliquez sur OK.
- Cliquez sur l’outil règle et mesurez la distance entre deux points du réseau. Dans les paramètres Treillis, définissez l’espacement sur cette distance.
- Survolez chaque point du réseau pour voir l’intensité moyenne, l’épaisseur moyenne, l’intensité de l’écart type et l’épaisseur de l’écart type. Notez les intensités de chaque point du réseau et utilisez-les pour définir les paramètres d’épaisseur de glace souhaités dans les paramètres d’acquisition.
- Mesurez la distance et l’angle d’un point de treillis au centre de 4 trous avec l’outil règle. Dans les paramètres d’acquisition, supprimez les cibles de focus actuelles. Cliquez sur Remplissage automatique et modifiez le rayon et l’angle en fonction des valeurs mesurées. Cliquez sur Ciblage de test, cliquez sur OK, puis sur Envoyer les cibles.
REMARQUE : Leginon trouvera le foyer eucentrique (nœud Focus) et collectera les expositions, qui apparaîtront dans le nœud Exposition. - Une fois que toutes les cibles sont imagées, accédez au nœud Exposure_Targeting pour voir l’image du trou suivant. Dans les paramètres, décochez Autoriser la vérification utilisateur des cibles sélectionnées. Décochez également les cases Mettre les cibles en file d’attente et Ignorer l’outil de recherche de trous automatisé. Cliquez sur OK , puis sur Envoyer les cibles.
REMARQUE : Leginon collectera automatiquement les images en fonction des paramètres configurés ci-dessus. Voir les images et les métadonnées dans Appion.
- Des modifications peuvent être apportées lors de la collecte automatisée. Par exemple, modifiez la plage de défocalisation de la collection à tout moment en modifiant le préréglage enn dans le Preset_Manager.
- Si la collection doit être arrêtée, mettez fin à la file d’attente en cliquant sur les boutons Abandonner et Abandonner la file d’attente dans les nœuds Trou et Exposition.
- Une fois la collecte terminée, allez dans Application et cliquez sur Arrêter, puis allez dans Fichier et cliquez sur Quitter.
2. Utilisation de Smart Leginon Autoscreen
- Création d’une session de modèle Smart Leginon
- Suivez les instructions de la section 1 pour démarrer Leginon.
- Accédez à Application et cliquez sur Exécuter. Dans la fenêtre Exécuter l’application , sélectionnez l’application Ptolémée (sélectionnez Afficher tout si nécessaire). Réglez main sur l’ordinateur Leginon et scope et caméra sur cet ordinateur respectif.
- Dans le Preset_Manager, importez les préréglages comme décrit à l’étape 1.2.3.
- Configurez les paramètres du nœud.
- Dans les paramètres du nœud Square_Targeting, assurez-vous que les options Trier les cibles par chemin le plus court et Activer le ciblage automatique sont cochées (Figure supplémentaire 1A).
- Dans les paramètres du nœud Carré, assurez-vous que l’option Attendre que le nœud traite l’image est cochée. Ajoutez le préréglage Square à la liste de droite dans le menu déroulant s’il n’y est pas déjà. Dans Paramètres avancés, cochez la case Réglez ces ouvertures pendant l’imagerie et assurez-vous que les valeurs des deux ouvertures sont correctes (Figure supplémentaire 1B).
- Dans les paramètres du nœud Hole_Targeting, cochez Autoriser la vérification utilisateur des cibles sélectionnées. Décochez les cases Mettre les cibles en file d’attente et Ignorer le détecteur de trous automatisé (Figure supplémentaire 2A).
- Dans le paramètre Nœud de perçage, cochez Attendre qu’un nœud traite l’image et le paramètre prédéfini de perçage se trouve dans la liste de droite. Dans Paramètres avancés, cochez la case Réglez ces ouvertures pendant l’imagerie et assurez-vous que les valeurs des deux ouvertures sont correctes (Figure supplémentaire 2B).
- Dans les paramètres du nœud Exposure_Targeting, cochez Autoriser la vérification utilisateur des cibles sélectionnées. Décochez les cases Mettre en file d’attente les cibles et Ignorer l’outil de recherche de trous automatisé (Figure 3A supplémentaire).
- Dans les paramètres du nœud Exposition, assurez-vous que l’option Attendre qu’un nœud traite l’image n’est pas cochée, que le préréglage Exposition est répertorié à droite, et dans les paramètres avancés, cochez la case Définir ces ouvertures pendant l’imagerie et assurez-vous que les valeurs des deux ouvertures sont correctes (Figure supplémentaire 3B).
- Dans les paramètres du nœud de mise au point, assurez-vous que l’option Attendre qu’un nœud traite l’image n’est pas cochée, que le préréglage de mise au point automatique est répertorié à droite et que la précision de mise au point automatique souhaitée est définie sur 4 x 10-6 m (Figure supplémentaire 4A).
- Dans la séquence de mise au point du nœud Focus (à côté du bouton des paramètres), activez uniquement deux étapes de mise au point automatique de l’inclinaison du faisceau (Figure supplémentaire 4B,C).
- Dans les paramètres du nœud Z_Focus, assurez-vous que l’option Attendre qu’un nœud traite l’image n’est pas cochée, que le préréglage Trou est répertorié à droite et que la précision de mise au point automatique souhaitée est de 5 x 10-5 m (Figure supplémentaire 5A).
- Dans la séquence de mise au point du nœud Z_Focus, activez uniquement deux étapes d’inclinaison de la platine à faible grossissement (Figure supplémentaire 5B,C).
- Déterminez la hauteur z de la grille comme décrit à l’étape 1.2.4.
- Collectez un atlas comme décrit à l’étape 1.2.10.
- Configurez les paramètres du chercheur carré.
- Une fois l’atlas collecté, Ptolémée localisera les carrés dans le nœud Square_Targeting. Chaque carré affichera un cercle bleu, appelé blob. En survolant chaque blob, Leginon signale sa taille telle que calculée par Ptolémée. Notez les plus gros et les plus petits blobs.
- Dans les paramètres Seuil, modifiez la plage de filtrage minimale et maximale pour inclure les carrés souhaitables et exclure les carrés indésirables.
- Cliquez sur le bouton Rechercher des carrés dans la barre d’outils supérieure. Ajustez la plage de filtre jusqu’à ce que l’option Rechercher les carrés cible bien.
- Dans les paramètres d’acquisition, choisissez des valeurs pour le nombre maximal de cibles et le nombre de groupes cibles à échantillonner. Ces paramètres définiront le nombre de carrés et de groupes de carrés ciblés.
- Une fois satisfait des paramètres, cliquez sur le bouton Lecture . Un exemple d’atlas après la configuration est présenté à la figure supplémentaire 6.
- Configurez les paramètres de la recherche de trous.
- Dans le nœud Hole_Targeting, utilisez l’outil règle pour mesurer le diamètre d’un trou.
- Dans les paramètres du modèle, saisissez le diamètre dans Diamètre final du modèle et cliquez sur Tester. Ajustez le diamètre jusqu’à ce que tous les trous aient des pics blancs brillants au centre.
- Dans les paramètres de seuil, cliquez sur Tester. Ajustez la valeur A jusqu’à ce que l’image binarisée affiche des zones blanches uniquement à l’endroit où se trouvent les trous.
- Dans les paramètres Objets blob, choisissez d’exclure les cibles de bordure en utilisant la règle pour déterminer une distance minimale par rapport au bord et en saisissant cette valeur. Les blobs peuvent être filtrés en fonction de leur taille, de leur rondeur et du nombre souhaité. Survolez les blobs pour montrer leurs valeurs. Cliquez sur Test pour inspecter les valeurs actuelles.
- Dans les paramètres Treillis, saisissez le rayon des trous et l’espacement entre les trous (utilisez l’outil de mesure), puis cliquez sur le bouton 42 pour mesurer la valeur d’intensité de référence d’une zone de vide (trou vide ou film de support cassé).
- Dans les paramètres d’acquisition, cochez Utiliser le sous-ensemble des cibles d’acquisition et définissez la valeur Échantillon maximal sur un petit nombre, tel que 2. Définissez une large gamme de moyennes d’épaisseur de glace et d’écarts-types (mesurez ces valeurs en survolant les cibles). Cliquez sur Tester le ciblage pour randomiser la sélection de cibles en fonction des valeurs ci-dessus.
- Cliquez sur le bouton Lecture lorsque vous êtes satisfait de tous les paramètres. Leginon effectuera l’étape Z_Focus et récupérera la première cible. Un exemple d’image après la configuration est illustré à la figure supplémentaire 7.
- Configurez les paramètres de ciblage de l’exposition.
- Dans les paramètres de trou, définissez le script Shell sur le chemin de script hl_finding.sh dans l’installation de Ptolemy. Définissez le score minimum à accepter sur ≤0. Saisissez le rayon des trous (utilisez l’outil de mesure), puis cliquez sur le bouton 42 pour mesurer la valeur d’intensité de référence d’une zone de vide (trou vide ou film de support cassé). Cliquez sur Tester pour trouver le réseau de trous.
- Dans les paramètres d’acquisition, cochez Utiliser le sous-ensemble des cibles d’acquisition et définissez la valeur Échantillon maximal sur un petit nombre, tel que 4, à collecter sur un sous-ensemble de trous pour le tramage. Définissez une large gamme de moyennes d’épaisseur de glace et d’écarts-types (mesurez ces valeurs en survolant les cibles).
- Cliquez sur le bouton Lecture lorsque vous êtes satisfait de tous les paramètres. Leginon effectuera une mise au point eucentrique et collectera des images à fort grossissement, qui peuvent être vues dans le nœud Exposition . Un exemple d’image après la configuration est illustré à la figure supplémentaire 8.
- Vérifiez l’image Exposure_Targeting suivante pour voir si les paramètres ci-dessus sont toujours suffisants. Une fois que vous êtes satisfait, décochez la case Autoriser la vérification par l’utilisateur des cibles sélectionnées dans les paramètres Ciblage de l’exposition et Ciblage des trous.
REMARQUE : Le dépistage devrait maintenant se dérouler sans surveillance pour la grille actuelle. Cette session sera utilisée comme session de modèle pour toutes les grilles.
- Une fois le filtrage de la grille terminé, cliquez sur Fichier > Quitter pour fermer Leginon.
- Configurer Smart Leginon Autoscreen
- Dans une fenêtre de terminal, exécutez le autoscreen.py de Smart Leginon.
- Sélectionnez l’interface graphique, saisissez une liste d’emplacements de grille séparés par des virgules à sélectionner, saisissez full pour le workflow, saisissez le nom de la session modèle sur lequel baser les nouvelles sessions (cela se trouve dans Appion imageviewer) et saisissez la valeur z-height de la session modèle (Figure supplémentaire 9).
- Une interface graphique s’ouvre pour permettre d’entrer le nom de session pour chaque grille et de sélectionner leurs associations de projet respectives (Figure supplémentaire 10).
REMARQUE : Smart Leginon Autoscreen utilisera désormais les paramètres de session du modèle pour filtrer automatiquement chaque grille et passer d’une grille à l’autre sans surveillance. - Suivez la collecte à Leginon, Appion et l’ordinateur du microscope, ou laissez le microscope complètement sans surveillance.
REMARQUE : Une fois toutes les grilles criblées, Smart Leginon fermera les vannes de colonne sur le microscope.
Résultats
En suivant le protocole, les séances de dépistage cryoEM peuvent être exécutées automatiquement et avec succès pour la majorité (80 % à 90 %) des grilles et des conditions trouées. Plusieurs exemples et expériences ont été présentés précédemment 8,9 pour démontrer les résultats attendus de sessions Smart Leginon Autoscreen réussies. Une session Autoscreen réussie commence par ~10 min de configuration et aboutit généralement à une cassette complète de 12 grilles criblées automatiquement après environ 6 h (30 min par grille) où 3 à 5 carrés de tailles différentes et 3 à 5 trous par carré sont imagés à fort grossissement, permettant à l’utilisateur de déterminer rapidement les caractéristiques de l’échantillon sur chaque grille et d’itérer rapidement dans les conditions échantillon/grille (Figure 3). Parfois, les sessions échouent, généralement parce qu’Autoscreen cible les carrés cassés, n’interprète pas correctement les grands gradients d’épaisseur de glace sur la grille ou sur les carrés, ou ne parvient pas à identifier correctement les trous sur les grilles de carbone. De plus, les fuites de mémoire potentielles peuvent provoquer un plantage de Leginon en raison d’une utilisation excessive de la mémoire, qui peut être résolu en libérant de la RAM, ou en redémarrant l’ordinateur, ou amélioré en ajoutant plus de RAM à l’ordinateur.
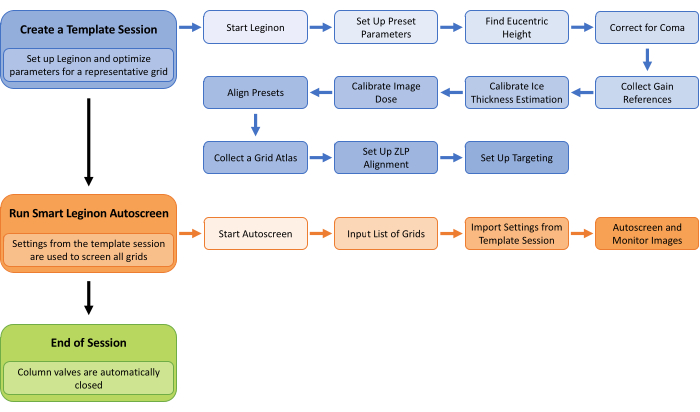
Figure 1 : flux de travail Smart Leginon Autoscreen. Un aperçu de haut niveau du flux de travail Smart Leginon Autoscreen. Tout d’abord, une session de modèle est créée en sélectionnant les paramètres d’une grille représentative dans le lot de grilles à filtrer. La configuration de Leginon et la création d’une session de modèle prennent moins de 45 minutes. Deuxièmement, Autoscreen est configuré pour utiliser les paramètres de session de modèle pour filtrer toutes les grilles de la cassette. La configuration d’Autoscreen prend moins de 10 minutes. Enfin, Autoscreen met fin à la séance de projection. Veuillez cliquer ici pour voir une version agrandie de cette figure.
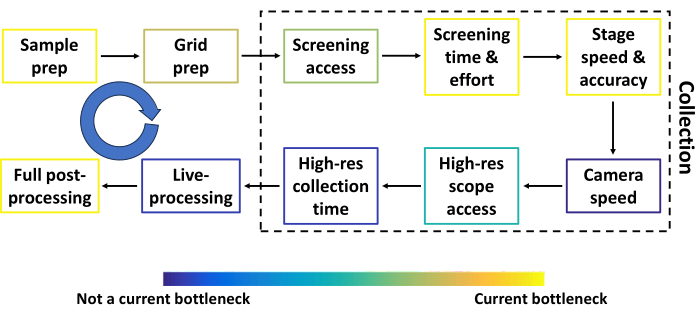
Figure 2 : Pipeline cryoEM conventionnel à particule unique avant le criblage automatisé. Les étapes les plus courantes du pipeline cryoEM conventionnel à particules uniques avant le criblage automatisé, ainsi que les composants qui peuvent être améliorés. Chaque étape est colorée pour se rapprocher du goulot d’étranglement par rapport aux autres. La flèche circulaire bleue représente plusieurs boucles de rétroaction entre la plupart des étapes. Le débit à plusieurs étapes dépend fortement de l’échantillon, du financement et de l’emplacement du chercheur. Veuillez cliquer ici pour voir une version agrandie de cette figure.

Figure 3 : Résultats représentatifs de Smart Leginon Autoscreen. Images multi-échelles représentatives suivant le protocole Smart Leginon Autoscreen collectées sur un cryoTEM TFS Krios avec un filtre d’énergie BioQuantum et une caméra K3. (A) Une image composite de l’atlas montrant une vue d’ensemble d’une grille cryoEM. (B-F) Images multi-échelles des emplacements indiqués dans l’atlas quadrillé. Les images à faible grossissement dans la première rangée, les images à grossissement moyen dans la deuxième rangée et les images à fort grossissement dans la troisième rangée ont chacune été sélectionnées automatiquement pour obtenir des informations sur l’échantillon des carrés de glace minces à épais. L’épaisseur de la glace estimée par Leginon est indiquée sur le fond. Les barres d’échelle sont de 500 μm en (A) et 10 μm pour la première rangée, de 5 μm pour la deuxième rangée et de 100 nm pour la troisième rangée pour (B-F). Cette figure a été modifiée avec la permission de Cheng et al.8. Veuillez cliquer ici pour voir une version agrandie de cette figure.
| gr : Grille | sq : Carré | hln : Trou | ventilateur : Mise au point automatique | fcn : Focus central | enn : Exposition | |
| Grossissement | 210 | 2600 | 6700 | 120000 | 120000 | 120000 |
| Defocus | -0.0002 | -0.00015 | -0.00015 | -2 x 10-06 | -7 x 10-07 | -2,5 x 10-06 |
| Taille du spot | 5 | 5 | 4 | 2 | 2 | 2 |
| Intensité | 1.1 | 0.83 | 0.65 | 0.44 | 0.44 | 0.45 |
| Dimension | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 4096 x 4096 |
| Compenser | 0, 0 | 0, 0 | 0, 0 | 0, 0 | 512, 512 | 0, 0 |
| Binning | 4 x 4 | 4 x 4 | 4 x 4 | 4 x 4 | 2 x 2 | 1 x 1 |
| Temps d’exposition (ms) | 200 | 500 | 500 | 500 | 500 | 1000 |
| Pré-exposition (s) | 0 | 0 | 0 | 0 | 0 | 0 |
| Dose (e/Å2) | -- | -- | -- | 36.5 | 36.5 | 64.7 |
| Enregistrer des images brutes | Non | Non | Non | Non | Non | Oui |
Tableau 1 : Paramètres prédéfinis pour le criblage de la grille cryoEM au Simons Electron Microscopy Center (SEMC) à l’aide d’un cryoTEM Glacios avec une caméra Falcon 3EC. Les paramètres de chaque préréglage couramment utilisé sur un cryoTEM Glacios avec une caméra Falcon 3EC au SEMC sont affichés. Différents microscopes auront des grossissements variables disponibles et différentes expériences utiliseront des paramètres variables tels que la défocalisation et le temps d’exposition.
| gr : Grille | sq : Carré | hln : Trou | ventilateur : Mise au point automatique | fcn : Focus central | enn : Exposition | |
| Grossissement | 64 | 1700 | 2850 | 75000 | 75000 | 75000 |
| Defocus | 0 | -5 x 10-05 | -5 x 10-05 | -1 x 10-06 | -7 x 10-07 | -2 x 10-06 |
| Taille du spot | 6 | 9 | 9 | 6 | 6 | 7 |
| Intensité | 0.001 | 1,65 x 10-05 | 1,5 x 10-05 | 4,3 x 10-07 | 4,3 x 10-07 | 5,5 x 10-07 |
| Largeur du filtre d’énergie | -- | -- | -- | 20 | 20 | 20 |
| Dimension | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 1024 x 1024 | 2048 x 2048 | 4096 x 4096 |
| Compenser | 0, 0 | 0, 0 | 0, 0 | 0, 0 | 0, 0 | 0, 0 |
| Binning | 4 x 4 | 4 x 4 | 4 x 4 | 4 x 4 | 2 x 2 | 1 x 1 |
| Temps d’exposition (ms) | 500 | 2000 | 1000 | 500 | 300 | 8700 |
| Pré-exposition (s) | 0 | 0 | 0 | 0 | 0 | 0 |
| Dose (e/Å2) | -- | -- | -- | -- | -- | 47.4 |
| Enregistrer des images brutes | Non | Non | Non | Non | Non | Oui |
Tableau 2 : Paramètres prédéfinis pour le criblage de la grille cryoEM au SEMC à l’aide d’un cryoTEM Krios avec une caméra Selectris X et Falcon 4i. Les paramètres de chaque préréglage couramment utilisé sur un Krios avec un filtre d’énergie Selectris X et une caméra Falcon 4i au SEMC sont affichés. Différents microscopes auront des grossissements variables disponibles et différentes expériences utiliseront des paramètres variables tels que la défocalisation et le temps d’exposition.
| gr : Grille | sq : Carré | hln : Trou | ventilateur : Mise au point automatique | fcn : Focus central | enn : Exposition | |
| Grossissement | 1550 | 940 | 2250 | 81000 | 81000 | 81000 |
| Defocus | 0 | -5 x 10-05 | -5 x 10-05 | -1 x 10-06 | -7 x 10-07 | -2 x 10-06 |
| Taille du spot | 4 | 8 | 7 | 6 | 6 | 6 |
| Intensité | 0.0015 | 0.00017 | 7,3 x 10-05 | 1,3 x 10-06 | 1,3 x 10-06 | 9,2 x 10-07 |
| Largeur du filtre d’énergie | -- | -- | 50 | 20 | 20 | 20 |
| Dimension | 1024 x 1024 | 1440 x 1024 | 1440 x 1024 | 1440 x 1024 | 1008 x 1008 | 5760 x 4092 |
| Compenser | 0, 0 | 0, 0 | 0, 0 | 0, 0 | 936, 519 | 0, 0 |
| Binning | 4 x 4 | 8 x 8 | 8 x 8 | 8 x 8 | 4 x 4 | 2 x 2 |
| Temps d’exposition (ms) | 250 | 600 | 600 | 500 | 500 | 2100 |
| Pré-exposition (s) | 0 | 0 | 0 | 0 | 0 | 0 |
| Dose (e/Å2) | -- | -- | -- | -- | -- | 51 |
| Enregistrer des images brutes | Non | Non | Non | Non | Non | Oui |
Tableau 3 : Paramètres prédéfinis pour le criblage de la grille cryoEM au SEMC à l’aide d’un cryoTEM Krios avec une caméra BioQuantum et K3. Les paramètres de chaque préréglage couramment utilisé sur un Krios avec un filtre d’énergie BioQuantum et une caméra K3 au SEMC sont affichés. Différents microscopes auront des grossissements variables disponibles et différentes expériences utiliseront des paramètres variables tels que la défocalisation et le temps d’exposition.
Figure supplémentaire 1 : Paramètres de ciblage carré et paramètres de carré pour Smart Leginon. (A) Paramètres de ciblage Square. (B) Paramètres carrés. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 2 : Paramètres de ciblage des trous et Paramètres des trous pour Smart Leginon. (A) Paramètres de ciblage des trous. (B) Paramètres de trous. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 3 : Paramètres de ciblage de l’exposition et paramètres d’exposition pour Smart Leginon. (A) Paramètres de ciblage de l’exposition. (B) Paramètres d’exposition. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 4 : Paramètres de mise au point et paramètres de séquence de mise au point pour Smart Leginon. (A) Paramètres de mise au point. (B) Paramètres de la séquence de mise au point (Défocalisation1). (C) Paramètres de la séquence de mise au point (Défocalisation2). Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 5 : Paramètres de Z_Focus et paramètres de séquence Z_Focus pour Smart Leginon. (A) Z_Focus paramètres. (B) Z_Focus réglages de séquence (Stage_Tilt_Rough). (C) Z_Focus Paramètres de séquence (Stage_Tilt_Fine). Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 6 : Un exemple d’atlas après la configuration des paramètres de Square_Targeting Smart Leginon. Les cercles bleus sont des taches, les signes plus verts sont des lieux d’acquisition et le « x » brun est l’emplacement actuel de la scène. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 7 : Un exemple d’atlas après la configuration des paramètres de Hole_Targeting Smart Leginon. Les signes plus violets sont des emplacements en treillis, les signes plus verts avec des boîtes sont des emplacements d’acquisition et le signe plus bleu est l’emplacement principal. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 8 : Un exemple d’atlas après la configuration des paramètres de Exposure_Targeting Smart Leginon. Les cercles bleus sont des taches, les signes plus verts sont des emplacements d’acquisition et le signe plus bleu est l’emplacement du focus. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 9 : Configuration du terminal Smart Leginon Autoscreen. Veuillez cliquer ici pour télécharger ce fichier.
Figure supplémentaire 10 : Configuration de l’interface graphique de Smart Leginon Autoscreen. Veuillez cliquer ici pour télécharger ce fichier.
Discussion
Dans ce protocole, nous décrivons le pipeline de Smart Leginon Autoscreen, ainsi que l’utilisation de base de Leginon pour ceux qui découvrent le logiciel de collecte. La cryoEM à particule unique est en passe de devenir la technique de résolution de la structure protéique tridimensionnelle (3D) la plus productive d’ici la fin de 202417. Le pipeline cryoEM à particules uniques se compose de plusieurs étapes qui sont constamment optimisées pour augmenter la qualité et le débit des données. La figure 2 montre les étapes les plus courantes (préparation des échantillons, préparation de la grille, temps et efforts de criblage, temps de collecte haute résolution, traitement en direct et post-traitement complet) ainsi que d’autres composants du pipeline qui peuvent être améliorés (accès au microscope de criblage, vitesse et précision de la platine, vitesse de la caméra et accès au microscope haute résolution). Les résultats de la plupart des étapes deviennent des boucles de rétroaction dans les étapes précédentes (flèches bleues dans la figure 2), ce qui rend l’ensemble du pipeline hautement interdépendant. Chaque étape de la figure 2 est colorée pour se rapprocher du goulot d’étranglement par rapport aux autres. Smart Leginon Autoscreen réduit considérablement le temps et les efforts de l’opérateur pour le criblage de 12 grilles de 6 h à moins de 10 min, soulageant ainsi ce goulot d’étranglement et permettant un retour plus rapide sur la préparation des échantillons/grilles (Figure 3).
Le protocole comporte plusieurs étapes critiques, illustrées à la figure 1. Il est essentiel que la grille utilisée pour créer la session de modèle soit représentative des grilles restantes à filtrer. Il est important de noter que Leginon se souvient de tous les paramètres de l’ensemble du processus de configuration pour créer une session de modèle (étapes bleues dans la figure 1), ce qui permet de configurer des sessions de modèle récurrentes plus rapidement à chaque fois. Lors de la création d’une session de modèle, l’étape la plus critique consiste à configurer le ciblage à tous les grossissements afin que les paramètres et les seuils reflètent la variation attendue sur les grilles à cribler. Les différents boutons « Test » permettent d’être efficace dans ce processus de configuration. Lors d’une session Autoscreen, il est essentiel de surveiller les premières grilles d’Appion pour détecter rapidement tout problème et le résoudre dans Leginon dès que possible.
Le flux de travail typique du SEMC consiste à alimenter les données Autoscreen dans CryoSPARC Live18 et à utiliser ces informations supplémentaires pour informer les boucles de rétroaction dans la préparation des échantillons/grilles. Pendant les journées intensives d’optimisation cryoEM des chercheurs-opérateurs, les informations sur les conditions de l’échantillon et de la grille sont réinjectées dans la préparation de l’échantillon et de la grille pendant qu’Autoscreen continue de cribler les grilles. Cela permet de geler et de cribler plusieurs dizaines de grilles par semaine8.
Smart Leginon Autoscreen fonctionne pour la majorité (80 % à 90 %) des grilles et des conditions trouées observées au SEMC. Les 10 à 20 % restants des grilles comprennent celles qui ne fonctionnent parfois pas bien - des grilles avec une différence de contraste minimale entre les trous et le substrat ; grilles avec des trous et un espacement plus petits (par exemple, 0,6/0,8) - et des grilles où le ciblage sur plusieurs grilles est souvent peu pratique - Spotiton/Chameleon19,20 grilles qui consistent en des bandes d’échantillon à travers la grille ; Grilles en dentelle. Notez que la collection de grilles inclinées avec Autoscreen est en cours de développement mais n’est pas encore disponible. Il peut être possible de modifier le protocole pour qu’il fonctionne avec des grilles Spotiton/Chameleon en imageant d’abord manuellement les zones de la bande pour déterminer des seuils de paramètres étroits, puis en essayant de regrouper des carrés plus grands et plus petits, respectivement, à l’étape 2.1.7.4, puis en sélectionnant des cibles dans le groupe avec de la glace. L’objectif de cette modification est de faire en sorte que Smart Leginon sépare les cases vides et non vides en deux groupes. Si des paramètres sont trouvés, ils peuvent ne pas bien s’étendre aux grilles restantes à cribler. Il peut également être possible de modifier le protocole pour qu’il fonctionne avec des grilles en dentelle en supprimant le script hl_finding.sh à l’étape 2.1.9.1 et en configurant les paramètres pour cibler les zones plus claires/plus sombres comme vous le souhaitez. Le taux de réussite de cette modification peut varier d’une grille à l’autre en fonction de l’épaisseur de la glace et du matériau de la grille.
Le dépannage pendant une session Autoscreen est possible et parfois approprié. Des modifications des paramètres prédéfinis (par exemple, défocalisation) et de ciblage (par exemple, les seuils de ciblage des trous) peuvent être apportées lors de la collecte automatisée. Pendant la collecte d’une session Autoscreen, une session de grille ne peut pas être annulée car elle se terminera autoscreen.py. Cependant, les boutons Abandonner dans les nœuds de ciblage peuvent être utilisés pour ignorer n’importe quelle partie d’une grille ou une grille entière. Parfois, autoscreen.py peut utiliser trop de mémoire et se figer, offrant deux options : « forcer à quitter » ou « attendre ». Si l’option « forcer l’arrêt » est sélectionnée, l’ensemble du script se termine, ce qui oblige l’utilisateur à réexécuter le script à appliquer aux grilles restantes pour le filtrage. Si 'attendre' est sélectionné, le script continuera et les paramètres peuvent être modifiés pour éviter un gel futur, par exemple, en désactivant l’affichage de l’image dans le nœud Exposition, en diminuant la taille des pixels dans l’atlas ou en exécutant un script d’effacement de la mémoire. Si le programme se bloque sans offrir les deux options, les erreurs de mémoire peuvent ne pas se résoudre d’elles-mêmes, ce qui entraîne une interruption de l’acquisition. L’option « forcer à quitter » peut être utile dans ce cas.
Smart Leginon Autoscreen est utilisé régulièrement chez SEMC. Alors que les goulots d’étranglement dans le pipeline cryoEM à particule unique continuent d’être réduits, l’adoption de cryoEM continuera d’augmenter pour aider à répondre aux questions biologiques. Ce protocole est un pas dans la direction de l’optimisation de l’ensemble du pipeline en fournissant une voie claire pour réduire considérablement les boucles de rétroaction.
Déclarations de divulgation
Les auteurs déclarent qu’ils n’ont pas d’intérêts financiers concurrents.
Remerciements
Une partie de ce travail a été réalisée au Simons Electron Microscopy Center du New York Structural Biology Center, avec le soutien de la Simons Foundation (SF349247), du NIH (U24 GM129539) et de l’Assemblée de l’État de New York.
matériels
| Name | Company | Catalog Number | Comments |
| Glacios cryoTEM | Thermo Fisher Scientific | GLACIOSTEM | FEG, 200 keV, Falcon 3EC camera |
| Krios cryoTEM | Thermo Fisher Scientific | KRIOSG4TEM | XFEG, 300 keV, Gatan BioQuantum energy filter, Gatan K3 camera |
| Leginon | Simons Electron Microscopy Center | http://leginon.org | |
| Ptolemy | Simons Machine Learning Center | https://github.com/SMLC-NYSBC/ptolemy |
Références
- Noble, A. J., et al. Routine single particle CryoEM sample and grid characterization by tomography. eLife. 7, e34257 (2018).
- Weissenberger, G., Henderikx, R. J. M., Peters, P. J. Understanding the invisible hands of sample preparation for cryo-EM. Nature Methods. 18 (5), 463-471 (2021).
- Carragher, B., et al. Current outcomes when optimizing 'standard' sample preparation for single-particle cryo-EM. Journal of Microscopy. 276 (1), 39-45 (2019).
- Kampjut, D., Steiner, J., Sazanov, L. A. Cryo-EM grid optimization for membrane proteins. iScience. 24 (3), 102139 (2021).
- Suloway, C., et al. Automated molecular microscopy: The new Leginon system. Journal of Structural Biology. 151 (1), 41-60 (2005).
- Mastronarde, D. N. Automated electron microscope tomography using robust prediction of specimen movements. Journal of Structural Biology. 152 (1), 36-51 (2005).
- Koh, A., et al. Routine collection of high-resolution cryo-EM datasets using 200 KV transmission electron microscope. Journal of Visualized Experiments. 181, 63519 (2022).
- Cheng, A., et al. Fully automated multi-grid cryoEM screening using Smart Leginon. IUCrJ. 10 (1), 77-89 (2023).
- Kim, P. T., Noble, A. J., Cheng, A., Bepler, T. Learning to automate cryo-electron microscopy data collection with Ptolemy. IUCrJ. 10 (1), 90-102 (2023).
- Bepler, T., et al. Smart data collection for CryoEM. Journal of Structural Biology. 214 (4), 107913 (2022).
- Bouvette, J., Huang, Q., Riccio, A. A., Copeland, W. C., Bartesaghi, A., Borgnia, M. J. Automated systematic evaluation of cryo-EM specimens with SmartScope. eLife. 11, e80047 (2022).
- Deng, Y., Grollios, F., Kohr, H., van Knippenberg, B., Janus, M., Caglar, F. Smart EPU: SPA Getting Intelligent. Microscopy and Microanalysis. 27 (S1), 454-455 (2021).
- Fan, Q., et al. CryoRL: Reinforcement learning enables efficient cryo-EM data collection. arXiv. , (2022).
- Li, Y., et al. Optimized path planning surpasses human efficiency in cryo-EM imaging. bioRxiv. Biophysics. , (2022).
- Lander, G. C., et al. Appion: An integrated, database-driven pipeline to facilitate EM image processing. Journal of Structural Biology. 166 (1), 95-102 (2009).
- Rice, W. J., et al. Routine determination of ice thickness for cryo-EM grids. Journal of Structural Biology. 204 (1), 38-44 (2018).
- Russo, C. J. The potential for seeing molecules in cells, CZ Imaging Institute's Hardware Frontiers for CryoET Workshop. CZ Imaging Institute's Hardware Frontiers for CryoET Workshop. , (2023).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Budell, W. C., Allegri, L., Dandey, V., Potter, C. S., Carragher, B. Cryo-electron microscopic grid preparation for time-resolved studies using a novel robotic system, Spotiton. Journal of Visualized Experiments. 168, 62271 (2021).
- Darrow, M. C., Booth, T., Moore, J. P., Doering, K., Thaw, P., King, R. S. Enabling a paradigm shift in CryoEM sample preparation with chameleon. Microscopy and Microanalysis. 27 (S1), 524-525 (2021).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon