Method Article
Une méthode efficace d’extraction de l’épithélium de la trompe de Fallope humaine pour des analyses de cellules uniques
Dans cet article
Résumé
Ce protocole décrit une méthode d’une journée pour l’isolement des cellules épithéliales des trompes de Fallope humaines. Les cellules épithéliales isolées peuvent être plaquées en culture bidimensionnelle (2D) ou dissociées en suspensions unicellulaires et utilisées dans des expériences en aval, y compris la cytométrie en flux et le séquençage de l’ARN unicellulaire.
Résumé
Les trompes de Fallope humaines sont intrinsèques à la reproduction. Le but des trompes de Fallope est de permettre le transit des spermatozoïdes, de l’ovule et, si la fécondation réussit, de l’embryon. Les cellules épithéliales qui tapissent la surface interne des trompes de Fallope font partie intégrante des processus normaux et anormaux des trompes de Fallope, y compris l’initiation de la maladie. Après la ménopause, les trompes de Fallope cessent d’avoir un rôle important dans le corps et la composition des cellules intra-épithéliales change. Nous décrivons une méthode dans laquelle ces cellules épithéliales peuvent être isolées à partir de trompes de Fallope fraîches avec une contamination minimale des cellules stromales dans une suspension unicellulaire. Ces cellules peuvent être cultivées en culture ou utilisées pour des analyses plus approfondies telles que la cytométrie en flux et le séquençage de l’ARN sur cellule unique. Ce protocole d’isolement peut être réalisé en 4 à 6 h et produit des cellules viables qui peuvent être utilisées pour une analyse en aval immédiat. Ce protocole efficace facilite l’isolement des cellules épithéliales des trompes de Fallope avec une population épithéliale enrichie.
Introduction
Les trompes de Fallope sont constituées de plusieurs parties. De l’ovaire à l’utérus, la trompe de Fallope est composée des fimbriae, de l’ampoule, de l’isthme et de la partie intramurale. Les fimbriae s’étendent à partir de l’extrémité de la trompe de Fallope où elles saisissent l’ovule libéré par l’ovaire. L’ovule se déplace ensuite à travers l’ampoule, où il est le plus susceptible d’être fécondé, jusqu’à l’isthme et enfin est transféré à l’utérus par la partie intra-muros1. La muqueuse la plus interne de la trompe de Fallope qui facilite le transport de l’ovule est constituée d’une couche d’épithélium luminal, comprenant des cellules ciliées et sécrétoires. Les cellules ciliées ont tendance à être plus concentrées au niveau des fimbriae2. Ils jouent un rôle essentiel dans le déplacement physique de l’ovule à travers la trompe de Fallope, de l’ovaire à l’utérus. Leurs appendices permettent aux cellules ciliées non seulement de faire avancer l’ovule, mais aussi d’éliminer le stress génotoxique après l’ovulation3.
Les cellules épithéliales sécrétoires de la trompe de Fallope sécrètent un liquide qui aide à la nutrition et à l’assemblage des gamètes4. La proportion de cellules ciliées et sécrétoires le long de l’épithélium de la trompe de Fallope diffère dans l’état post-ménopausique avec une diminution des cellules ciliées car la trompe de Fallope ne remplit plus une fonction critique dans le transport5. De plus, en l’absence d’œstrogènes, on pense que les trompes de Fallope deviennent des résidusde 1,6. Cette perte de cellules ciliées dans la trompe de Fallope est supposée augmenter le risque de développer des carcinomes séreux7. De plus, on pense que les cellules épithéliales sécrétoires des trompes de Fallope donnent lieu à des lésions de carcinome intra-épithélial tubaire séreux (STIC), un précurseur bien connu du sous-type le plus agressif de cancers de l’ovaire tubulaire, le carcinome séreux de haut grade 7,8.
Le but de ce protocole est d’isoler les cellules épithéliales des trompes de Fallope humaines et de les dissocier en suspensions unicellulaires. Ce protocole permet d’obtenir une population épithéliale unicellulaire enrichie qui peut être utilisée pour de nombreuses analyses. Comme le montre ce manuscrit, nous avons effectué une analyse par cytométrie en flux et plaqué des cellules en 2D après isolement. L’analyse par cytométrie en flux démontre la présence de cellules uniques, qui sont pour la plupart viables et de nature épithéliale. Dans ces analyses, nous avons inclus quatre marqueurs, e506 pour la viabilité, EpCAM pour les cellules épithéliales, CD45 pour les cellules immunitaires et CD10 pour les cellules stromales. Les cellules mortes ont été exclues à l’aide du marqueur de viabilité e506, et les cellules immunitaires ont été exclues à l’aide de CD45. Il est possible que la suspension ait une population de cellules immunitaires ; cependant, pour obtenir une population relativement pure de cellules épithéliales, les cellules CD45 positives peuvent être appauvries à l’aide d’un kit de déplétion CD45. De plus, lorsqu’elles sont mises en culture, les cellules CD45-positives ne prolifèrent souvent pas. Les cellules isolées par cette méthode et cultivées en 2D montrent des populations épithéliales adhérentes de type pavé. Cette méthode peut être utilisée pour générer des préparations cellulaires, qui peuvent être développées dans des banques d’ARN unicellulaires.
La recherche visant à définir la lignée cellulaire des épithéliums des trompes de Fallope, les altérations de ces lignées au cours des différentes phases de la vie reproductive et les événements déclencheurs conduisant à une transformation et à une genèse malignes est devenue plus importante au cours des quatre dernières années 6,9,10,11,12 . Ce protocole profitera considérablement à la recherche dans ce domaine en fournissant un moyen efficace d’isoler les épithéliums des trompes de Fallope et de les transformer en cellules uniques.
Protocole
Ce protocole a été adapté d’une méthode d’isolement de cellules épithéliales utérines décrite précédemment13,14. Des échantillons frais de trompes de Fallope anonymisées ont été prélevés selon notre protocole #10-0727 approuvé par l’IRB de l’Université de Californie à Los Angeles (UCLA) et digérés en une suspension unicellulaire dans un délai d’environ 4 à 6 heures.
1. Prélèvement et préparation des trompes de Fallope
- Prélever des trompes de Fallope humaines fraîches (figure 1A) dans des milieux de dissociation (DMEM [4,5 g/L de D-glucose] contenant 10 % de sérum de veau fœtal, 5 mL de L-glutamine et 5 mL de pénicilline/streptomycine) dans des tubes de 15 mL ou 50 mL et transporter sur de la glace jusqu’au laboratoire.
- Transférez les trompes de Fallope dans une boîte de Pétri stérile contenant un milieu de dissociation frais.
- À l’aide d’un microscope à dissection, disséquez la graisse, le tissu conjonctif et tous les vaisseaux entourant les tubes à l’aide d’une pince à pointe fine et de ciseaux Vannas Tubingen (figure 1B).
- Sous le microscope de dissection, coupez le plan coronal des trompes de Fallope avec des ciseaux pour former de petits cylindres de ~3 à 5 mm de diamètre chacun (Figure 1C), à l’aide de la pince à pointe fine pour stabiliser le tissu.
2. Isolement et dissociation des cellules épithéliales
- Aspirez soigneusement le milieu de dissociation du plat.
- Lavez les morceaux de tissu 2x avec une solution saline tamponnée au phosphate (PBS) froide en pipetant doucement ~2 mL pour couvrir les morceaux et en tournant légèrement la plaque.
- Aspirez le dernier lavage 1x PBS, incubez les morceaux de tissu pendant 5 min dans de l’acide éthylènediaminetétraacétique (EDTA) froid de 5 mM à température ambiante, et répétez l’opération pour un total de deux incubations. Aspirer les 5mM d’EDTA après chaque incubation.
- Mettre les fragments de tissu en suspension dans de la trypsine à 1 % froide et une solution saline équilibrée de Hanks (HBSS) et les incuber à 4 °C pendant 40 minutes (figure 1D).
- Aspirez la trypsine à 1 % / HBSS et lavez les morceaux de tissu 2 fois dans un milieu de dissociation à froid pour inactiver la trypsine.
- Incuber les fragments de tissu dans 2 mL de milieu de dissociation avec 0,4 mg de DNase pendant au moins 30 min à température ambiante.
REMARQUE : Une période d’incubation plus longue peut être nécessaire, mais ne dépassez pas 45 minutes avant de commencer l’étape 2.7. - Sous un microscope de dissection, alors que vous êtes encore dans le milieu avec de la DNase, maintenez en place un fragment de la trompe de Fallope, la lumière parallèle au fond de la boîte, en place avec une pince. De l’autre main, appuyez à plusieurs reprises sur le morceau de la trompe de Fallope avec une pince pour expulser les cellules épithéliales de la lumière du tube. Confirmez qu’un halo de cellules est visible autour du tissu lorsque les cellules sont libérées de la lumière du tube. Répétez l’opération pour chaque fragment de trompe de Fallope dans la boîte (Figure 1E).
- Transférez le milieu contenant les cellules dans un tube stérile de 15 ml, lavez la boîte de Pétri avec un milieu de dissociation au moins 2 fois et combinez les lavages dans le tube de 15 ml. Jetez les fragments du tube stromal.
- Récolter les cellules par centrifugation à 500 × g pendant 5 min. Aspirer le surnageant et remettre la pastille en suspension dans 1 mL de milieu de dissociation.
- Compter les cellules en aliquotant 10 μL de la suspension cellulaire et en mélangeant 1:1 avec du bleu trypan. Pipeter 10 μL du mélange dans une lame de comptage à chambre et l’insérer dans un compteur de cellules (figure 1F).
3. Digestion en suspension unicellulaire
- Remettre les cellules en suspension dans 9 ml de milieu de dissociation, 1 ml de collagénase à 8 mg/ml (type 1) et 0,4 mg de DNase. Incuber à 37 °C pendant 30 à 45 min dans un agitateur orbital à 200 tr/min (Figure 1G).
- Récolter les cellules par centrifugation à 500 × g pendant 5 min. Aspirez le surnageant et remettez la pastille en suspension dans 5 mL de milieu de dissociation pour éliminer la collagénase.
- Passez la suspension cellulaire à travers une crépine de 100 μm dans un tube de 50 mL (Figure 1H).
- Récolter les cellules par centrifugation à 500 × g pendant 5 min.
- Si la pastille cellulaire apparaît rouge, effectuez la lyse des globules rouges (GR) comme décrit aux étapes 3.6 à 3.8. Si la pastille de cellule n’est pas rouge, passez à l’étape 3.9.
- Diluer 500 μL de tampon de lyse des globules rouges dans 4,5 mL d’eau ultrapure.
- Aspirez le milieu, ajoutez 5 mL de tampon de lyse des globules rouges dilué et incubez sur de la glace pendant 3 min.
- Pour arrêter la lyse des globules rouges, ajoutez 45 ml de 1x PBS dans le tube de 50 ml.
- Récolter les cellules par centrifugation à 500 × g pendant 5 min.
- Comptez les cellules en aliquotant 10 μL de la suspension cellulaire et en la mélangeant 1:1 avec du bleu de trypan. Pipeter 10 μL du mélange dans une lame de comptage en chambre et l’insérer dans le compteur de cellules (Figure 1I).
4. Coloration par cytométrie en flux
- Aliquote de 250 000 cellules dans deux tubes distincts en polystyrène à fond rond de 5 ml pour le contrôle primaire des anticorps conjugués et des isotypes à utiliser pour la cytométrie en flux de marqueurs de surface.
REMARQUE : Il est possible d’utiliser moins de cellules pour prendre en charge le nombre de cellules et le nombre de cellules nécessaires à l’analyse en aval. Pour obtenir des résultats d’écoulement optimaux, un minimum de 50 000 cellules doit être inclus dans l’analyse. - Diluer l’anticorps (p. ex., EpCAM et CD10 anti-humains) et l’isotype selon la dilution recommandée par le fabricant dans 1x PBS. Inclure un colorant de viabilité fixable (e506) et un anticorps anti-CD45 humain conjugué pour éliminer les cellules immunitaires humaines vivantes. Préparez suffisamment de mélange d’anticorps pour au moins 50 μL par tube. Laisser dans l’obscurité sur de la glace jusqu’à l’étape 4.4.
- Lavez les cellules avec 1 mL de 1x PBS. Récoltez les cellules par centrifugation à 500 × g pendant 5 min à 4 °C.
- Aspirez le 1x PBS. Remettez la pastille en suspension dans le panel d’anticorps ou d’isotype et incubez à 4 °C pendant 15 min dans l’obscurité.
- Après l’incubation, diluer dans 1 mL de 1x PBS et récolter les cellules par centrifugation à 500 × g pendant 5 min à 4 °C.
- Aspirez le 1x PBS et remettez en suspension la pastille cellulaire dans 200 μL de 1x PBS.
5. Collecte de données de cytométrie en flux
- Connectez-vous à la machine FACS.
REMARQUE : Nous présentons le protocole en utilisant le cytomètre en flux référencé (voir le tableau des matériaux). Pour les autres cytomètres en flux, respectez les instructions du fabricant. Le réglage de la tension du cytomètre en flux a été préconfiguré pour des performances optimales avec les PBMC humains, éliminant ainsi le besoin d’ajuster la tension avant chaque exécution FACS. - Si la machine est en mode d’acquisition , passez à l’étape 5.3. Si la machine est en mode d’analyse de données , passez en mode acquisition en appuyant sur le bouton d’alimentation en haut à droite. Lorsqu’une fenêtre contextuelle s’affiche, appuyez sur le bouton qui indique le mode d’acquisition.
- En bas à gauche, si l’appareil indique Étalonnage requis, passez à l’étape 5.3.1. Si l’appareil indique Étalonnage correct, passez à l’étape 5.4.
- Pour calibrer la machine, sortez les billes d’étalonnage du réfrigérateur à 4 °C et du vortex.
- Placez 1 goutte de billes d’étalonnage dans un tube à fond rond en polystyrène de 5 ml et placez le tube dans le support de tube unique.
- Appuyez sur le bouton du code-barres en haut à droite et scannez la bouteille de perle d’étalonnage. Une fenêtre contextuelle apparaîtra. Si tout est configuré, cliquez sur Oui | OK et laissez le calibrage se dérouler.
- Si l’étalonnage réussit, déconnectez-vous et reconnectez-vous en mode d’acquisition . Passez à l’étape 5.4.
- Cliquez sur Ouvrir | nouvel espace de travail en haut à gauche.
- Sélectionnez la grille de refroidissement 5 ; Sélectionnez trois des puits. Sélectionnez le premier puits et donnez-lui une description (par exemple, FTX Ab). Sélectionnez le puits suivant et donnez-lui une description unique (par exemple, FTX Iso). Sélectionnez le dernier puits et donnez-lui la description eau de Javel.
REMARQUE : Incluez toujours un échantillon d’eau de Javel tous les 4 à 5 échantillons expérimentaux pour assurer un nettoyage rapide des échantillons de cellules. - Sélectionnez tous les puits et configurez les conditions expérimentales comme suit : Paramètres de l’instrument : PBMC humains ; Débit : élevé ; Échantillon de mélange : mélange doux ; Mode : standard ; Volume total de l’échantillon 200 μL ; Prélèvement d’échantillon 150 μL ; Annotation : anticorps d’entrée et leur marquage fluorophore.
- Sélectionnez Modifier | options et donnez un nom au fichier sous description| appliquer | ok.
- Sortez la grille du réfrigérateur et placez-la sur le plateau avec le code-barres face à la machine. Placez les tubes d’échantillons dans le rack pour refléter l’ordre des échantillons à l’écran et appuyez sur le bouton de lecture pour démarrer l’expérience.
- Une fois l’expérience terminée, branchez la clé USB, sélectionnez Fichier | copier, développez le dossier privé, sélectionnez l’expérience, puis cliquez sur copier et éjecter.
- Après la collecte des données, utilisez un logiciel pour analyser la fiche technique.
- Commencez par le contrôle d’isotype. Utilisez l’outil polygone pour sélectionner des cellules sur le graphique de diffusion directe (FSC) par rapport à la diffusion latérale (SSC).
- Éliminez les cellules mortes en remplaçant l’axe Y par e506 et l’axe X par FSC. Utilisez l’outil polygone pour sélectionner la population de cellules vivantes négative à e506.
- Double-cliquez sur la population sélectionnée. Changez l’axe Y en CD45 humain. Utilisez l’outil polygone pour sélectionner la population de cellules. Il s’agit de la population de cellules CD45 négatives humaines.
- Double-cliquez sur la population de cellules CD45 négatives. Remplacez l’axe Y par histogramme et l’axe X par EpCAM humain. Utilisez l’outil de sélection de plage pour cliquer sur la fin de l’histogramme et sélectionner l’espace vide jusqu’à la fin du graphique. Cela représente la population positive à l’EpCAM. Répéter l’opération pour la coloration humaine au CD10.
- Faites glisser le contrôle de l’isotype sous l’espace de travail anticorps. Les cellules CD45 positives seront exclues et les populations EpCAM positives et CD10 positives seront sélectionnées.
- Pour créer des histogrammes représentatifs, sélectionnez l’éditeur de mise en page. Faites glisser les histogrammes souhaités vers la page de mise en page. Pour comparer la coloration de l’isotype et celle des anticorps, superposez l’histogramme de l’isotype à l’histogramme des anticorps. Gate les cellules positives en fonction du contrôle de l’isotype.
6. Immunocytochimie
- Cultivez des cellules après l’étape finale d’isolement dans la lame de verre de chambre souhaitée jusqu’à ce qu’elles confluent.
- Pour fixer les cellules, ajoutez 500 μL de paraformaldéhyde (PFA) froid à 4 % Incuber à température ambiante pendant 10 min.
- Aspirez les 4 % de PFA et lavez 3x avec 1x PBS.
- Pour perméabiliser les cellules, ajoutez au moins 200 μL de Triton-X à 0,25 % dans du PBS et incubez à température ambiante pendant 10 min.
- Pendant la perméabilisation, préparez le tampon bloquant (4 % de sérum de chèvre normal [NGS] pour les anticorps non produits chez la chèvre).
- Aspirez le Triton-X à 0,25 % et lavez-le 3 fois avec 1 PBS.
- Ajouter au moins 200 μL de tampon de blocage (4 % NGS dans le PBS). Bloquer pendant 1 h à température ambiante.
- Préparez des dilutions d’anticorps primaires dans 1 % NGS dans 1x PBS selon les recommandations du fabricant.
- Après le blocage, ajoutez au moins 200 μL de la solution d’anticorps primaires. Incuber à température ambiante pendant 2 h.
REMARQUE : Certains anticorps primaires peuvent préférer une incubation nocturne à 4 °C ; Consultez la fiche technique de l’anticorps. - Préparez des solutions d’anticorps secondaires dans 1 % NGS dans 1x PBS. Enveloppez les tubes dans du papier d’aluminium et préparez les solutions dans l’obscurité car les anticorps secondaires sont sensibles à la lumière. Conserver dans l’obscurité jusqu’à ce qu’il soit prêt.
- Après l’incubation primaire de l’anticorps, laver 3 fois avec 1 PBS.
- Ajouter au moins 200 μL de la solution secondaire d’anticorps. Incuber dans l’obscurité à température ambiante pendant 1 h.
REMARQUE : Au cours des étapes suivantes, essayez de garder la diapositive dans l’obscurité autant que possible. - Après l’incubation secondaire de l’anticorps, laver 3 fois avec 1 fois PBS.
- Retirez les chambres, soit en les retirant simplement à la main, soit à l’aide de l’outil fourni avec les glissières. Suivez les instructions imprimées sur l’emballage de la lame de chambre pour le retrait.
- Ajoutez une goutte de sérum de montage 4',6-diamidino-2-phénylindole (DAPI) et placez une lamelle sur le dessus. Limitez le nombre de bulles et assurez-vous que toutes les cellules sont couvertes. Étape facultative : utilisez du vernis à ongles sur les bords pour sceller la lamelle.
- Posez la diapositive à plat dans une boîte à lames pour qu’elle sèche et placez-la à 4 °C jusqu’à l’imagerie.
- Image sur un microscope équipé d’une fluorescence correcte basée sur la conjugaison d’anticorps secondaires pour garantir une image nette.
Résultats
Nous avons inclus sept collections de trompes de Fallope où nous avons isolé une population de cellules épithéliales enrichie (figure 2A et figure supplémentaire S1A). Pour évaluer la viabilité et l’enrichissement en cellules épithéliales de cette méthode de suspension unicellulaire, une cytométrie en flux a été réalisée. Pour mesurer la viabilité cellulaire, les cellules ont été colorées avec le marqueur de viabilité, e506. Cela a également permis d’éliminer tous les débris et cellules mortes lors de l’analyse.
Pour déterminer la composition des cellules isolées par cette méthode, les échantillons ont été colorés à l’aide d’un marqueur de cellules épithéliales (EpCAM), d’un marqueur de cellules stromales (CD10) et d’un marqueur de cellules immunitaires (CD45). Comme le montre la figure 2B, nous avons isolé une population de cellules épithéliales viable et enrichie à partir de tissus de trompes de Fallope. La viabilité de l’échantillon était en moyenne de 82 %. Pour tous les échantillons, après avoir éliminé les cellules CD45 positives, nous avons observé une population de cellules épithéliales enrichie, les cellules positives à EpCAM représentant en moyenne 80 % de l’échantillon. Il y a eu une certaine contamination des cellules stromales, mais seulement 7,8 % en moyenne. La figure 2C montre des cellules isolées en 2D 4 à 6 jours après le placage. Il est évident que les cellules épithéliales ont été isolées car elles formaient des amas cohérents et adhérents ressemblant à des pavés. Dans la figure supplémentaire S1B, les cellules ont été mises en boîte après isolement et cultivées pendant 2 à 6 jours. L’immunocytochimie a été utilisée pour caractériser les cellules en croissance. La plupart des cellules de la culture étaient positives pour EpCAM et le marqueur cellulaire sécrétoire, PAX8. Seules quelques cellules ont été identifiées comme positives à la vimentine. Des cellules ciliées ont été observées en culture, comme le montre la vidéo supplémentaire S1.
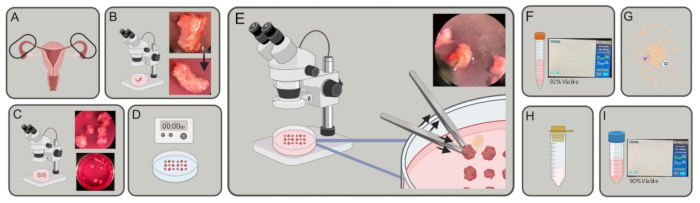
Figure 1 : Schéma expérimental. (A) Les trompes de Fallope sont acquises. (B) L’excès de graisse et de tissu conjonctif est enlevé. (C) Les tubes sont coupés en morceaux de ~3-5 mm. (D) Les morceaux sont lavés dans du PBS, puis incubés dans de l’EDTA 2x pendant 5 min, et incubés dans de la trypsine à 1 % pendant 40 min à 4 °C. (E) À l’aide de deux paires de pinces, l’une pour tenir et l’autre pour pousser, expulsez les cellules épithéliales des morceaux de la trompe de Fallope. (F) Transférez les cellules dans un tube de 15 ml, tournez et comptez. (G) Remettre en suspension dans le DMEM, la collagénase et la DNase. Digestion pendant 30-45 min. (H) Filtrer avec une passoire de 100 μm. (I) Récolte par centrifugation et comptage. Veuillez cliquer ici pour voir une version agrandie de cette figure.
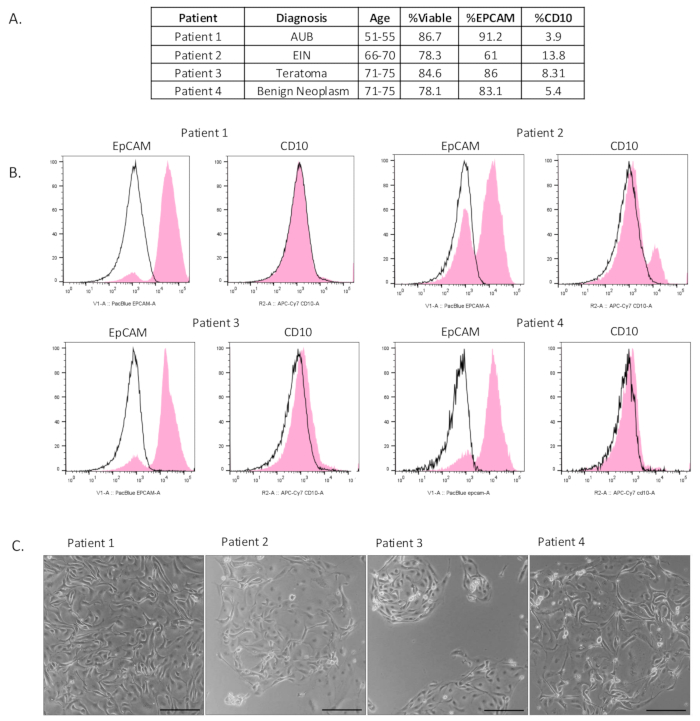
Figure 2 : Caractérisation de cellules épithéliales isolées. (A) Tableau des informations cliniques du patient et des résultats de la cytométrie en flux. (B) Une cytométrie en flux a été réalisée après l’isolement pour montrer qu’une population enrichie de cellules épithéliales (EpCAM-positive) avait été isolée. Le contrôle négatif de l’isotype est indiqué en noir et le résultat de l’expérience est indiqué en rose. (C) Les cellules ont été placées après isolement. Les photos ont été prises 4 à 6 jours après le placage. Barres d’échelle = 100 μm. Abréviations : AUB = Saignement utérin anormal ; EIN = Néoplasie intra-épithéliale de l’endomètre. Veuillez cliquer ici pour voir une version agrandie de cette figure.
Figure supplémentaire S1 : Caractérisation supplémentaire de cellules épithéliales isolées en culture. (A) Tableau des informations cliniques sur le patient. (B) Des cellules isolées ont été cultivées et colorées pour EpCAM, un marqueur cellulaire sécrétoire (PAX8) et un marqueur cellulaire stromal (Vimentin) 2 à 6 jours après le placage pour caractériser le type de cellule en culture. Barres d’échelle = 50 μm. Abréviations : EIN : Néoplasie intra-épithéliale de l’endomètre ; DAPI = 4',6-diamidino-2-phénylindole. Veuillez cliquer ici pour télécharger cette figure.
Vidéo supplémentaire S1 : Une vidéo montrant des cellules ciliées en train de battre a été prise à un grossissement de 30x pour montrer les cellules ciliées en culture. Veuillez cliquer ici pour télécharger cette vidéo.
Discussion
L’étude de l’épithélium des trompes de Fallope suscite un intérêt considérable, car les trompes de Fallope jouent un rôle important dans la reproduction et sont le site d’origine de la plupart des HGSOC. À cette fin, de nombreux chercheurs ont décrit des protocoles permettant d’isoler les cellules des trompes de Fallope dans des modèles humains et murins10,11,12,15,16,17,18,19,20,21. La méthode que nous décrivons pour extraire et enrichir les cellules épithéliales des trompes de Fallope s’ajoute aux protocoles existants d’isolement des cellules des trompes de Fallope. Bien qu’il existe des chevauchements au sein de ces protocoles, il existe deux types généraux de méthodes signalées chez la souris et chez l’homme. La première consiste à hacher et à digérer par voie enzymatique l’ensemble de la trompe de Fallope qui donne une suspension cellulaire totale 10,12,15,16,17. La seconde consiste à muer avec agitation ou grattage, ce qui donne des feuilles de tissu 11,18,19,20,21. Les deux méthodes permettent d’analyser les cellules épithéliales via des expériences en aval telles que la cytométrie en flux, le séquençage et la culture in vitro. Un avantage majeur de notre méthode est que le protocole produit une population de cellules de trompes de Fallope qui sont déjà enrichies pour les cellules épithéliales par la digestion enzymatique et les étapes de poussée mécaniques. Cette population enrichie en épithéliums peut être digérée davantage, ce qui donne une suspension unicellulaire qui peut être utilisée pour de nombreuses applications telles que la cytométrie en flux, la culture 2D, l’immunocytochimie et le séquençage de l’ARN unicellulaire.
Les principales étapes qui ont un impact sur le rendement cellulaire comprennent la durée pendant laquelle les fragments de la trompe de Fallope incubent dans 1 % de trypsine/HBSS et de DMEM/DNase. Une surincubation dans l’une ou l’autre solution dégradera les cellules et diminuera considérablement la viabilité des cellules. Cependant, un temps d’incubation insuffisant inhibera la capacité du chercheur à expulser de nombreuses cellules épithéliales au cours de l’étape 2.6 du protocole, car elles continueront d’adhérer les unes aux autres. Il est également important d’utiliser une solution de trypsine à 1 %, car des concentrations plus faibles et plus élevées de solutions de trypsine réduisent le rendement de cellules épithéliales viables des trompes de Fallope. En combinant l’isolement chimique et mécanique et une étape de digestion (étape 3.1 du protocole), nous pouvons raccourcir de manière exponentielle la période nécessaire pour passer d’une suspension tissulaire à une suspension unicellulaire. Cela garantit une bonne viabilité et le temps nécessaire pour effectuer l’analyse en aval le même jour.
À l’étape 1.4 du protocole, il est essentiel de s’assurer que les morceaux de trompe de Fallope coupés ont une épaisseur de 3 à 5 mm. Si les morceaux sont trop gros, il sera difficile d’effectuer l’étape 2.7 du protocole et finira par diminuer le rendement cellulaire car un temps d’incubation plus long dans DMEM/DNase sera nécessaire.
Bien que la suspension cellulaire soit enrichie pour les cellules épithéliales, la contamination des cellules stromales est inévitable. Si la préparation doit être constituée de cellules purement épithéliales, le tri à l’aide de la cytométrie en flux et des marqueurs que nous avons décrits peut être effectué pour isoler les cellules épithéliales et épuiser les cellules stromales.
Des trompes de Fallope post-ménopausées ont été utilisées dans cette étude. Cependant, cette méthode a été utilisée avec succès dans notre laboratoire sur les trompes de Fallope préménopausées. La principale différence entre les trompes de Fallope préménopausiques et postménopausiques est la composition des cellules épithéliales ciliées et sécrétoires1. Ce protocole fonctionne pour tous les stades de reproduction.
Ce protocole efficace facilitera l’étude des types cellulaires de l’épithélium de la trompe de Fallope, y compris la délimitation des lignées cellulaires, leurs changements dynamiques pendant les cycles de reproduction ainsi qu’après la ménopause, et leur rôle dans l’initiation du cancer de l’ovaire séreux de haut grade.
Déclarations de divulgation
Les auteurs n’ont aucun conflit d’intérêts à déclarer.
Remerciements
Les auteurs Ruegg L, James-Allan LB et DiBernardo G sont partiellement soutenus par les projets 1I01BX006019-01A2 de la Greater Los Angeles Veterans Association et I01BX006411-01 à Memarzadeh S. L’auteur Ochoa C est soutenu par UCLA Eli et Edythe Broad Center of Regenerative Medicine and Stem Cell Research Rose Hills Foundation Graduate Scholarship Training Program. Nous tenons à remercier le Translational Pathology Core Laboratory de l’UCLA, et plus particulièrement Ko Kiehle et Chloe Yin, pour leur aide dans l’approvisionnement en tissus. Nous tenons également à remercier Ken Yamauchi et le noyau de microscopie du BSCRC pour leur aide en matière d’imagerie. La figure 1 a été créée avec BioRender.com (numéro d’accord JL27QWDYNT). Enfin, nous tenons à remercier Felicia Cordea et la plateforme de cytométrie en flux du BSCRC pour leur aide en cytométrie en flux.
matériels
| Name | Company | Catalog Number | Comments |
| 1% Trypsin | Thermo Scientific | J63993.09 | |
| 100 µm Cell Strainer | Corning | 431752 | |
| 4% Paraformaldehyde | Electron Microscopy Sciences | 15710-S | |
| 5 mL round bottom tubes | Corning | 352008 | |
| 60 mm cell culture plate | Corning | CLS430589-500EA | |
| 6-well Cell Culture Plate | Corning | 353046 | |
| Anti-CD10 | BioLegend | 312212 | |
| Anti-CD326 (Ep-CAM) | BioLegend | 324218 | |
| Anti-CD45 | BioLegend | 304026 | |
| Anti-EpCAM | Abcam | ab223582 | |
| Anti-IgG2a PerCP | BioLegend | 400250 | |
| Anti-PAX8 | Sigma-Aldrich | 363M-15 | |
| Anti-Vimentin | Agilent Technologies | M072501-2 | |
| Chamber Slide System | Thermo Scientific | 154917PK | |
| Collagenase | Thermo Scientific | 17100017 | |
| DMEM | Thermo Scientific | 10569-010 | |
| DNase I | Sigma | 10104159001 | |
| EDTA | Sigma | 3690 | |
| eFluor 506 | Invitrogen | 65-0866-14 | |
| FBS | Sigma | F2442 | |
| Fine point forceps | VWR | 102091-526 | Any finepoint forceps of your choice will work |
| Fixable Viability Dye eFluor 506 | Invitrogen | 65-0866-14 | |
| FlowJo software version 9 | BD Biosciences | Data analysis software | |
| GlutaMAX | Gibco | 35050-061 | |
| HBSS | Thermo Scientific | 14175-095 | |
| MACSQuant Analyzer 10 flow cytometer | Miltenyi Biotec | ||
| MACSQuant Calibration Beads | Miltenyi Biotec | 130-093-607 | |
| Mammocult | Stemcell Technologies | 5620 | |
| Normal Goat Serum | Fisher Scientific | PI31873 | |
| PBS | Thermo Scientific | 14190-144 | |
| Penicillin-Streptomyocin | Gibco | 15140-122 | |
| PerCP Conjugated CD45 | BioLegend | 304026 | |
| Red Blood Cell lysis buffer | Tonbo Biosciences | TNB-4300-L100 | |
| Triton X-100 | Thermo Scientific | BP151-100 | |
| Vannas-Tubingen Spring Scissors | Fine Science Tools | 15003-08 | |
| VECTASHIELD with DAPI | Fisher Scientific | NC9524612 |
Références
- Crow, J., Amso, N. N., Lewin, J., Shaw, R. W. Morphology and ultrastructure of fallopian tube epithelium at different stages of the menstrual cycle and menopause. Hum Reprod. 9 (12), 2224-2233 (1994).
- Patek, E., Nilsson, L., Johannisson, E. Scanning electron microscopic study of the human fallopian tube. Report II. Fetal life, reproductive life, and postmenopause. Fert Steril. 23 (10), 719-733 (1972).
- Coan, M., et al. Exploring the role of fallopian ciliated cells in the pathogenesis of high-grade serous ovarian cancer. Int J Mol Sci. 19 (9), 2512(2018).
- Leese, H., Tay, J., Reischl, J., Downing, S. Formation of fallopian tubal fluid: role of a neglected epithelium. Reproduction. 121 (3), 339-346 (2001).
- Donnez, J., Casanas-Roux, F., Ferin, J., Thomas, K. Changes in ciliation and cell height in human tubal epithelium in the fertile and post-fertile years. Maturitas. 5 (1), 39-45 (1983).
- Lengyel, E., et al. A molecular atlas of the human postmenopausal fallopian tube and ovary from single-cell RNA and ATAC sequencing. Cell Rep. 41 (12), 111838-111838 (2022).
- Tao, T., et al. Loss of tubal ciliated cells as a risk for "ovarian" or pelvic serous carcinoma. Am J Cancer Res. 10 (11), 3815(2020).
- Li, J., Fadare, O., Xiang, L., Kong, B., Zheng, W. Ovarian serous carcinoma: recent concepts on its origin and carcinogenesis. J Hematol Oncol. 5 (1), 8(2012).
- Hu, Z., et al. The repertoire of serous ovarian cancer non-genetic heterogeneity revealed by single-cell sequencing of normal fallopian tube epithelial cells. Cancer Cell. 37 (2), 226-242.e7 (2020).
- Dinh, H. Q., et al. Single-cell transcriptomics identifies gene expression networks driving differentiation and tumorigenesis in the human fallopian tube. Cell Rep. 35 (2), 108978(2021).
- Ulrich, N., et al. Cellular heterogeneity of human fallopian tubes in normal and hydrosalpinx disease states identified using scRNA-seq. Dev Cell. 57 (7), 914-929.e7 (2022).
- Brand, J., et al. Fallopian tube single cell analysis reveals myeloid cell alterations in high-grade serous ovarian cancer. iScience. 27 (3), 108990-108990 (2024).
- Memarzadeh, S., et al. Cell-autonomous activation of the PI3-kinase pathway initiates endometrial cancer from adult uterine epithelium. Proc Natl Acad Sci USA. 107 (40), 17298-17303 (2010).
- Cunha, G. R. Stromal induction and specification of morphogenesis and cytodifferentiation of the epithelia of the mullerian ducts and urogenital sinus during development of the uterus and vagina in mice. J Exp Zool. 196 (3), 361-369 (1976).
- Xie, Y., Park, E. -S., Xiang, D., Li, Z. Long-term organoid culture reveals enrichment of organoid-forming epithelial cells in the fimbrial portion of mouse fallopian tube. Stem Cell Res. 32 (1873-5061), 51-60 (2018).
- Karst, A. M., Drapkin, R. Primary culture and immortalization of human fallopian tube secretory epithelial cells. Nat Protoc. 7 (9), 1755-1764 (2012).
- Ford, M. J., Harwalkar, K., Yamanaka, Y. Protocol to generate mouse oviduct epithelial organoids for viral transduction and whole-mount 3D imaging. STAR Protocols. 3 (1), 101164(2022).
- Radecki, K. C., Lorenson, M. Y., Carter, D. G., Walker, A. M. Microdissection and dissociation of the murine oviduct: Individual segment identification and single cell isolation. J Vis Exp. (177), e63168(2021).
- Feng, L., et al. Protocol for the detection of organoid-initiating cell activity in patient-derived single fallopian tube epithelial cells. Methods Mol Bio. 2429, 445-454 (2022).
- Kessler, M., et al. The Notch and Wnt pathways regulate stemness and differentiation in human fallopian tube organoids. Nat Commun. 6 (1), 8989(2015).
- Fotheringham, S., Levanon, K., Drapkin, R. Ex vivo culture of primary human fallopian tube epithelial cells. J Vis Exp. (51), e2728(2011).
Réimpressions et Autorisations
Demande d’autorisation pour utiliser le texte ou les figures de cet article JoVE
Demande d’autorisationExplorer plus d’articles
This article has been published
Video Coming Soon