A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
שקיעת שיווי משקל של חלבון ממברנה הקטן oligomer יוצרי: אפקט של היסטידין protonation על pentameric יציבות
In This Article
Summary
Sedimentation equilibrium (SE) can be used to study protein-protein interactions in a physiological environment. This manuscript describes the use of this technique to determine the effect of pH on the stability of a homo-pentamer formed by the small hydrophobic (SH) protein encoded by the human syncytial respiratory virus (hRSV).
Abstract
ultracentrifugation אנליטית (AUC) יכול לשמש כדי לחקור אינטראקציות הפיכים בין מקרו-מולקולות על פני טווח רחב של עוצמות אינטראקציה ובתנאים פיסיולוגיים. זה עושה AUC שיטת הבחירה להעריך stoichiometry ותרמודינאמיקה של הומו והטרו-עמותה שחולפות והפיכים בתהליכים ביוכימיים כמותית. באופנות של שיווי משקל שקיעה (SE), איזון בין דיפוזיה והשקיעה מספק פרופיל כפונקציה של מרחק הרדיאלי שתלויה בדגם ספציפי עמותה. במסמך זה, פרוטוקול SE מפורט מתואר כדי לקבוע את אנרגיית קשר הגודל ומונומר-מונומר של oligomer חלבון קרום קטן באמצעות ultracentrifuge אנליטיים. AUC-ES הוא ללא תווית, המבוסס רק על עקרונות פיזיים, וניתן להשתמש בו בשני חלבונים מסיסים וקרום המים. דוגמא מוצגת של האחרון, החלבון הקטן הידרופובי (SH) בוירוס syncytial נשימה האנושי (hRSV), פוליפפטיד חומצת אמינו עם 65-תחום α-סליל יחיד הטרנסממברני (TM) שיוצר תעלות יונים pentameric. נתונים מבניים המבוססים על NMR מראה כי חלבון SH שתי protonatable השאריות שלו בתחום הטרנסממברני שמכוונים מול לומן של הערוץ. ניסויי SE תוכננו כדי לקבוע כיצד pH משפיע קבוע עמותה וגודל oligomeric חלבון SH של. בעוד שצורת pentameric נשמרה בכל המקרים, קבוע הקשר שלה הופחת ב- pH הנמוך. נתונים אלה הם בהסכם עם תלות דומה pH נצפתה לפעילות ערוץ SH, עולה בקנה אחד עם נטייה lumenal של שתי השאריות שלו בחלבון SH. האחרונים עשויים לחוות דחייה אלקטרוסטטית ויציבות oligomer מופחתת ב- pH הנמוך. לסיכום, שיטה זו ישימה בכל פעם שמידע כמותי על עדינים חלבונים שינויי עמותה בתנאים פיסיולוגיים צריכה להימדד.
Introduction
ultracentrifugation אנליטית 1-5 הוא אחת מהשיטות החשובות ביותר ללמוד אינטראקציות של מקרו-מולקולות בתנאים פיסיולוגיים, להיות נגיש לשני אינטראקציות חלשות וחזקות. השיטה היא ללא תווית ומשתמשת קליטה או הפרעות של אור, והוא יכול לשמש גם מערכות אופטיות הקרינה לגשת טווחי ריכוז על פני כמה סדרי הגודל 6.
שיטה זו שימושית במיוחד מכיוון שרוב התהליכים ביוכימיים תלויים באינטראקציות הפיכה. Stoichiometry ועוצמתו של אינטראקציות אלה צריכים להיות מאופיינים כמותית להבין תהליכים ביולוגיים, ומספר השיטות קיים למטרה זו 7, 8. עם זאת, אינטראקציות חולפות קשות ללמוד 9.
הבחירה של שיטה לאפיון אינטראקציות macromolecular תלויה באופי סטטי או הדינמי שלה. במקרה הראשון, sedim entation מהירות (SV) משמשת, שבה השיעור של תחבורת רדיאלי נמדד ומתחמים הם מופרדים על בסיס ההבדלים במסה ובצורה קלילה.
בניגוד לכך, עמותות דינמיות שהן הפיכים בקנה המידה של הניסוי הזמן לא ניתן להפריד באופן פיזי. במקרה זה, עצמי או הטרו-אינטראקציות מובילות לאינטראקציות שאינן קוולנטיים נמצאות בשיווי משקל זה תלוי בסך הכל ריכוז החלבון. ניתן ללמוד אינטראקציות דינמיות אלה על ידי שני שיווי משקל שקיעה (SE) ומהירות שקיעה (SV) 10. עם זאת, השיטה הראשונה היא פשוטה לביצוע, והוא מתואר כאן. בSE, צנטריפוגה מתבצעת במהירות נמוכה מספיק כדי שהוא הגיע לשיווי משקל בין דיפוזיה ושקיעה. בשלב זה, פרופיל שיווי המשקל של אות אופטי (UV-VIS) כפונקציה של מרחק הרדיאלי, ניתן לנתח באמצעות מודלים תרמודינמיים שנקבע מראש לעמותות 11.
ve_content "> במאמר הנוכחי, מחקר שיווי משקל שקיעה מוצג העמותה העצמית של חלבון קרום ויראלי שיוצר יון ערוצים. בגלל הידרופוביות שלה, הניסוי מנוהל בנוכחות של חומר ניקוי, ובמקרה זה את הצפיפות ממס צריך להיות מתאים לזה של חומר הניקוי. עם זאת, הפרוטוקול מתואר היית זהה במקרה של חלבון מסיס במים, פרט לכך שלא תהיה צורך התאמת צפיפות ממס.החלבון משמש מקודד בוירוס syncytial הנשימה האנושי (hRSV), pneumovirus עטוף במשפחת paramyxoviridae שגורמת למחלות בדרכי נשימה תחתונה בתינוקות, קשישים ואוכלוסיות מדוכאי חיסון ברחבי העולם 12. עד 64 מיליון מקרים מדווחים של זיהום hRSV ו160,000 מקרי מוות מתרחשים בכל שנה.
הגנום hRSV מעתיק 11 חלבונים, ביניהם שלושה חלבוני קרום F, G, וקטן הידרופובי (SH). חלבון SH מעורבבפתוגנזה של זיהום RSV. RSV חסר את גן SH (RSVΔSH) היה בת-קיימא, שנגרם היווצרות syncytia וגדל, כמו גם את וירוס wild-type (WT) 13-16. עם זאת, וירוס RSVΔSH משוכפל פי 10 פחות יעיל מאשר WT בדרך הנשימה העליונה 15, 16. כמו כן, וירוס RSVΔSH היה מופחת בעכבר vivo ודגמי שימפנזה 13, 17.
החלבון הוא SH (תת-קבוצה RSV) 64 או 65 חומצות אמינו (תת-הקבוצה B RSV) סוג II חלבון קרום נפרד שמתרכז בעיקר בקרומים של תא Golgi 18 ארוך. חלבון SH יש אחד שחזה סליל תחום הטרנסממברני (TM) 19 אשר נשמרת 20,21 מאוד. תחומים extramembrane C- וN-terminal מכוונים lumenally / extracellularly וcytoplasmically, בהתאמה.
שני תחום TM הסינתטי (שאריות 18-43) וחלבון SH אורך מלא הוכחו בצורת homopentamers במגוון רחב של חומרי ניקוי. טופס homopentameric אחראי על פעילות ערוץ בbilayers שומנים מישוריים 22,23. הכיוון הנכון של מונומרים TM בbilayer השומנים נקבע תחילה באמצעות dichroism האתר הספציפי אינפרא אדום 23, אשר הראה-22 להיות בlumenal, קרובה להבין-סליל, אורינטציה. אותו כיוון תחום TM אושר על ידי מחקרי NMR ששחזרו את pentameric סליל-חבילה של החלבון באורך מלא בdodecylphosphocholine (DPC) מיצלות 22. במודל 'micelle' זה, תחום TM סליל a- יחיד היה מוקף N-סופני על ידי סליל, ו- C-סופני על ידי b-סיכת ראש ממושכת. שתי שאריות protonatable חלבון SH של, שלו 22 ו-51, נמצאים בתחום TM (האוריינטציה lumenally), ובקצה של סיכת ראש β C-מסוף extramembrane (רחוק מנקבובי הערוץ), בהתאמה. בenviro bicellarnment, לעומת זאת, α-helix TM משתרע עד-51, ושניהם השאריות שלו נגישות ללומן של הערוץ 24. מבנה ערוץ מאמצת ארכיטקטורה כמו משפך-22, שבו האזור הצר (Ser-29 לCys-45) 22 עטור שרשרות צד הידרופובי (Ile-32, Ile-36, Ile-40 וLeu-44), ו Ile-36 מגדיר את הנקודה הצרה ביותר בלומן הערוץ. שלו-22 ממוקם בפתיחה הגדולה ביותר של משפך זה, ואילו 51-הוא בקצה של הפתיחה הקטנה ביותר.
בעבודה הנוכחית, צנטריפוגה אנליטיות במצב שיווי משקל שקיעה נעשתה שימוש כדי לקבוע אם protonation משפיע על היציבות של pentamer חלבון SH. במקרה זה, חלבון SH היה solubilized בחומר ניקוי C14-betaine, אשר שימש בעבר כדי להראות שצורות חלבון SH oligomers pentameric 22.
Protocol
פרוטוקול זה מבוסס על המקורות הבאים, שאמורים להיות מופנה לפרטים ושיקולים מיוחדים 3, 25-28 יותר.
1. התאמת צפיפות של מיצלות חומר הניקוי עם 2 H 2 O
הערה: הצפיפות של פתרון החיץ צריכה להיות מותאמת לצפיפות של מיצלות חומר הניקוי. סוכני התאמת צפיפות נפוצים כוללים 2 H 2 O, H 2 O 18, 2 H 2 O 18, גליצרול וסוכרוז 29. יש H 2 O 18 באותה הצפיפות כמו 2 H 2 O ועשוי להיות בחירה טובה יותר אם deuteration של פרוטונים להחלפה בחלבון אינו רצוי. בהליך זה, הצפיפות של 3 -propanesulfonate חומרי ניקוי (N, N-dimethylmyristylammonio) (C14SB) ב 50 מ"מ טריס pH 7.3, 100 mM NaCl יהיה שווה עם 2 H 2 O. כניחוש ראשוני ריכוזים הבאים2 H 2 O ישמש: 10, 30, ו -50% v / v.
1.1. לדוגמא הכנה
- הכן את פתרונות המניות הבאות ומסנן לעקר דרך פילטר מזרק 0.2 מיקרומטר: 50 מ"ל 500 מ"מ טריס pH 7.3 ו 1 M NaCl (פתרון חיץ 10X); 1 מ"ל 250 מ"מ C14SB (תמיסת ניקוי 50X).
- הכן 200 μl של פתרון מדגם על ידי ערבוב 20 μl 10X פתרון חיץ, 4 μl פתרון 50X חומר ניקוי, 20 μl 2 H 2 O (99.9%), ו -156 μl deionized H 2 O. הכן גם 200 μl של פתרון התייחסות על ידי ערבוב 20 μl פתרון חיץ 10X, 20 μl 2 H 2 O (99.9%), ו -160 μl deionized H 2 O.
- חזור על שלב 1.1.2 לריכוזי 2 O 2 H האחרים, כלומר 30% ו -50%, התאמת 2 H 2 O ו- H 2 O הכמויות כראוי.
1.2. הרכבה של 6 ערוציםטעינת תאים ומדגם UC לתוך התאים.
הערה: ישנם שני סוגים של תאי AUC בהתאם לשיטת טעינת מדגם. תאים ללא מילוי חיצוני להיות טעונים לפני איטום התא, ואילו חיצוני המילוי ניתן לטעון תאים לאחר שהתאים אטומים. הרכבה של תא AUC חיצוני מילוי תוארה בעבר 3. בפרוטוקול זה, ההרכבה של תא AUC 6 ערוצים ללא מילוי חיצוני מתוארת. ההבדל העיקרי הוא שיש לו טבעות ברגים בשני הצדדים שצריכים להדק בנפרד, וזה לא צריך אטמי דיור (איור. 1). ההבדל בשלבי הרכבה מודגשים להלן.
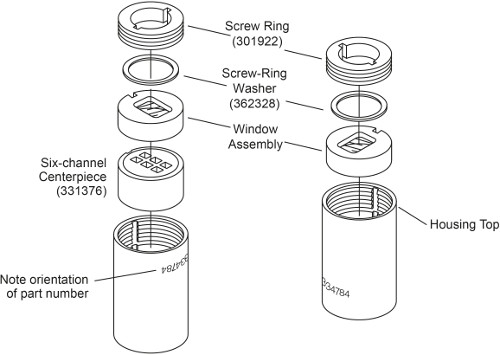
איור 1. התפוצץ נוף של תא AUC 6 ערוצים ללא מילוי חיצוני. נתון זה שונה מBeckman Coulter-50 Ti ו- 60 Ti Analytical רוטור, תאים, ומדריך למשתמש לאזן.
- הכן שתי אסיפות חלון לכל תא AUC עם חלון ספיר במקום חלון קוורץ (איור. 2). מניחים את אטם החלון לתוך חלון בעל. מעט לכופף את חלון האונייה ולמקם אותו לתוך מחזיק החלון כך שהפער נוצר הפוך לחור מנעול חלון בעל. הנח את חלון הספיר בתוך חלון האונייה, יישור הסימן עם חור מנעול חלון בעל.
הערה:. חלון קוורץ הוא דחיסה וכך יהיה לייצר שבירת אור יותר במהירות גבוהה 28, 30 לכן למדידות הפרעה מעל 30000 סל"ד, כגון בניסוי התאמת צפיפות זו, חלונות ספיר משמשים. חלון ספיר הוא כבד יותר מחלון קוורץ ויש "X" חרוט על הצד שלה.
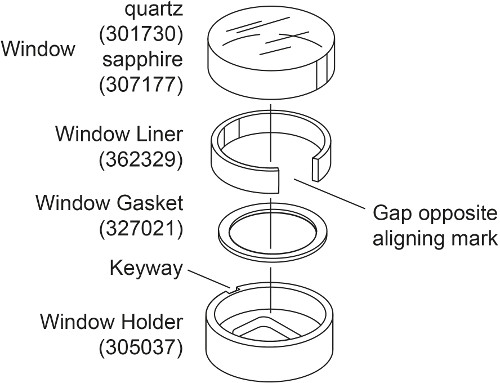
o תצוגת איור 2. התפוצץf חלון ההרכבה. נתון זה שונה מBeckman Coulter-50 Ti ו- 60 Ti אנליטית רוטור, תאים, ומדריך למשתמש לאזן
- הנח את דיור התא עם מספר חלק ההפוך. עם keyways מיושר עם מפתח הדיור, להחליק לתוך תא הדיור ראשית מרכזי 6 מגזר עם צד משופע כלפי מטה, ואחריו הרכבה חלון אחד עם החלון הפונה כלפי מטה (איור. 1, משמאל).
- קל מעיל מכונת הכביסה אשכולות טבעת בורג ובורג עם טבעת spinkote. הנח מכונת כביסה טבעת ברגים בחלק העליון של חלון ההרכבה. התקן את טבעת הבורג לדיור החלון עם "OUT" המילה מול מחוץ. יד להדק את טבעת הבורג ידי שימוש באפשרות יישור תא.
- באמצעות מפתח המומנט, להדק את טבעת הבורג לאינץ 'קילו רק 60.
- הנח את התא עם מספר החלק הזקוף ואת מיקומו בגיל 12-הצהריים. LOAD 120 התייחסות μl לשורות שמאל ו -110 מדגם μl לשורות ימין. ודא שכל דגימה והתייחסות היא זיווג נכון.
הערה: נפח דגימה מדויק אינו קריטי, אך ההתייחסות צריכה להיות מעט יותר נפח מהמדגם (μl 5-10) כך המניסקוס המדגם יהיה שונה. - חלק בזהירות לתוך חלון ההרכבה דיור תא אחד עם החלון פונה כלפי מטה (איור. 1, מימין). תשמור על עצמך לא להפריע תא יתר על המידה ולשפוך את התוכן.
- חזור על שלב 1.2. 3 ו -1.2. 4, הידוק טבעת הבורג השנייה עד 120 אינץ 'קילו. הפוך את התא ומחדש להדק את טבעת הבורג הראשונה ל -120 אינץ 'קילו.
- טען את התאים לתוך הרוטור, להתקין את הרוטור לצנטריפוגה ולהתקין את monochromator פי הוראות היצרן 28.
הערה: פרטים על st זהניתן למצוא גם ep בהתייחסות זו 3.
1.3. הגדרת מדידת הפרעה
- להפעיל את תוכנת ממשק משתמש למכשיר AUC ולבצע התקנת לייזר וכיול רדיאלי עבור כל תא בסל"ד 3000, על פי הוראות היצרן, אשר ניתן לסכם בקצרה בשלב הבא.
- הושג 1.3.1.1 לאחר ואקום מספק (<100 מיקרון), להפעיל את צנטריפוגות ב 3000 סל"ד. התצוגה מקדימה של תבנית ההתאבכות בתוכנת ממשק המשתמש ולהתאים את הפרמטרים הלייזר כדי להשיג את הניגוד הגבוה ביותר.
- צור קובץ חדש שהוקם (קובץ | קובץ חדש) המפרט "שיווי משקל" ומדידת "הפרעה". הגדר את שיטת שיווי משקל שקיעה (כפתור "שיטה") לרוץ ב45000 סל"ד או המהירות הגבוהה ביותר הצפויות לדגימות חלבון, לפי הגבוה מביניהם, עם טמפרטורת ריצה ב 20 ° C ולאסוף סריקת 1 בכל 15 דקות. Monitoהתקדמות r שיווי משקל על ידי פתיחת קבצי נתונים בHeteroAnalysis ובחירת פונקציה "התאמה" אחרי לפחות 12 שעות (כ הלילה, איור. 3).
הערה: פונקציה דומה זמינה גם בSEDFIT (אפשרויות | אפשרויות Loading | גישת מבחן לשיווי המשקל).

איור 3. תוצאה מתפקוד HeteroAnalysis התאמה. פונקצית ההתאמה יכולה לשמש כדי לעקוב אחר התקדמות שיווי משקל על ידי השוואה בין RMSD סריקות הרצופות והסריקה האחרונה. דוגמא זו מראה השגת שיווי המשקל לאחר 8 שעות כפי שצוינה על ידי ערכי RMSD אסימפטוטי לציר ה- X.
1.4. ניתוח נתונים
- עבור כל קבוצה של דגימות, עלילה המדרון של פרופיל הפצת רדיאלי נגד ריכוז 2 O 2 H.
הערה: החלוקה תהיה מעריכי ap רדוד מאוד שליניאריות גישות. ליירט ציר ה- X מתאים לריכוז 2 O 2 H ההתאמה. - לקבלת תוצאות מדויקות יותר, לבצע את הניסוי במספר חזרות. לחלופין, לחזור על הניסוי עם מגוון מצומצם של 2 ריכוז O H 2.
2. שיווי משקל שקיעה של SH במיצלות C14SB
2.1. פרמטרים הפעילו
- חישוב צפיפות חיץ וצמיגות, נפח ספציפי חלקי חלבון ומהירות צנטריפוגה באמצעות SEDNTERP. כדי לחשב את צפיפות וצמיגות חיץ, Compute בחר בסעיף "מאגר נתונים בחר" והזן את רכיבי החיץ בהתאם, כולל הריכוז O D 2.
- 2.1.1.1 כדי לחשב נפח חלבון חלקי ספציפי, Compute בחר בסעיף "V-בר" והזן את רצף החומצות האמיניות של חלבון. ציין את גודל oligomeric הצפוי הגבוה ביותר ב" תן oligomer ממונומר זה: N = "שדה, במקרה זה N = 5. חשב את המהירות על ידי הזנת ערכים בתחום RPM בחלון הראשי עד σ ≈ 1; זה כלל אצבע כדי להבטיח צורה מעריכית טובה של פרופיל הפצת רדיאלי 25.
הערה: ρ = 1.03839 g / ml, η = 1.0267 CP = .7569 / g, סל"ד 1 = 16000 ω מיליליטר: הערכים מחושבים לצורך הניסוי הזה היה כדלקמן. - לחשב מהירויות שלאחר מכן לבצע כדי להבטיח הבדל מספיק בין פרופיל ההפצה במהירות אחת ו-25 הבאים.
הערה: זה יכול להיעשות גם מהפונקציה "הערכת הרוטור שיווי משקל במהירויות פונקציה" בSEDFIT, אשר לוקחת בחשבון את עמודת הפתרון (נפח מילוי).
2.2. הכנות לדוגמא
- הכן פתרון 1 מיליליטר התייחסות עם 5 מ"מ C14SB ו32.3% 2 H 2 O כפי שיקבע מניסוי התאמת צפיפות (סעיף 1), על ידי ערבוב 100 μl solutio 10X חיץn (שלב 1.1.1), 20 μl פתרון 50X חומר הניקוי (שלב 1.1.1), 323 μl 2 H 2 O (99.9%) וμl 527 deionized H 2 O.
- לפזר lyophilized, פפטידים SH-מטוהר HPLC (ביטוי וטיהור שתואר קודם לכן 31) בממס מתאים כגון v / אצטוניטריל המימי v מתנול או 50%. למדוד A280 של פפטידים מומסים בmicrolitre בקנה מידה UV / Vis ספקטרופוטומטר וaliquot לשלוש דגימות לתת 280, 12mm = 0.3, 0.5, 0.8 ו( 280, 10 מ"מ = 0.25, 0.417, ו0.67) כל העת בדילול ל 130 μl. Lyophilize דגימות הלילה וגלול ב130 פתרון μl התייחסות (צעד 2.2.1) לתת פתרונות לדוגמא.
הערה: חלבון SH ניתן לאתר מUV / Vis הספיגה ב 280 ננומטר מכיוון שהוא מכיל שאריות TRP וTyr. חלבונים ללא שאריות ריחניות יכולים להיות מזוהים על ידי תיוגם עם chromophore מתאים, באמצעות מוטציה TRP המכיל, או באמצעות להפריעמדידות פליטות במקום ספיגה. - בצע את השלבים בסעיף 1.2 להרכבת תא AUC 6 ערוצים עם חלונות קוורץ. טען את מדגם הריכוז הגבוה ביותר (280, 12 מ"מ = 0.8) בערוץ הקרוב ביותר למרכז הרוטור ומדגם הריכוז הנמוך ביותר (280, 12 מ"מ = 0.3) רחוק ממרכז הרוטור.
2.3. הגדרת מדידות ספיגה
- צור קובץ חדש שהוקם (קובץ | קובץ חדש) המפרט "שיווי משקל" ומדידה "ספיגה". ציין 280 ננומטר כמו אורך גל הגלאי.
- לבצע כיול רדיאלי ב 3000 סל"ד ידי בדיקת "כיול מחוגי לפני הסריקה הראשונה" באפשרויות סריקה, המפרט איסוף נתונים ברזולוציה נמוכה, למשל עם גודל צעד מחוגי = 0.01 סנטימטר, משכפל = 3 (ברזולוציה נמוכה, מהיר), וביצוע סריקה בודדת . לאחר הסריקה הושלמה, בטל את האפשרות.
- הגדר את שיטת שיווי משקל שקיעה ("Methoד כפתור ") לרוץ במהירות הראשונה מחושבת בשלב 2.1.3, על 20 מעלות צלזיוס, ולאסוף סריקה 1 בכל 30 דקות. בכל תאים "פרט", ציין איסוף נתונים ברזולוציה נמוכה כמו בשלב 2.3.2. לעקוב אחר התקדמות של שיווי משקל על ידי פתיחת קבצי נתונים בHeteroAnalysis ובחירת פונקציה "התאמה" אחרי לפחות 18 שעות (כ הלילה, איור. 4).
הערה: השגת שיווי המשקל עלולה לקחת זמן משמעותי יותר למהירות הראשונה, ואילו במהירויות שלאחר מכן ייקחו פחות זמן.
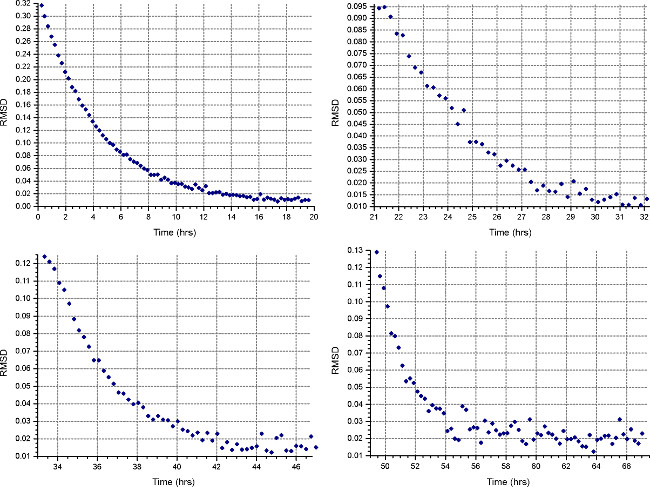
איור 4. תוצאות מפונקצית HeteroAnalysis התאמה. המהירויות הראשונות ושנייה (למעלה משמאל והמימין) נראה שהגיע לשיווי משקל, אבל עדיף לחכות עוד כמה שעות כדי להיות בטוח. לשם השוואה, במהירויות השלישית והרביעית (למטה משמאל ומימין) בבירור הגיעו לשיווי משקלבזמן קצר יותר. לחץ כאן כדי לצפות בגרסה גדולה יותר של דמות זו.
- ברגע שכבר הגיע לשיווי משקל, לאסוף סריקה בודדת ברזולוציה גבוהה, למשל בגודל צעד מחוגי = 0.001 סנטימטר, משכפל = 10 (ברזולוציה גבוהה, איטי).
- לאחר הסריקה הושלמה, חזור על שלב 2.3.3 ו2.3.4 למהירות הבאה.
- לחלופין, כאשר את הזמן הנדרש כדי להגיע לשיווי משקל לכל מהירות ידוע (מחושב או מניסיון), להגדיר את סריקת שיטת שיווי משקל שקיעה לכלול את כל המהירויות המחושבות בשלב 2.1.3 ולאסוף סריקה 1 לאחר זמן איזון לכל מהירות. במקרה זה, לציין איסוף נתונים ברזולוציה גבוהה בכל תאים "פרט".
2.4. ניתוח נתונים בSEDFIT וSEDPHAT
הערה: לפרטים ושיקולים בניתוח נתונים קורא נוספים נקרא to באתר האינטרנט הבא: www.analyticalultracentrifugation.com.
- סריקות פתוחות ברזולוציה הגבוהה בSEDFIT (נתונים | נתונים שיווי משקל שקיעת עומס) ולפצל את הנתונים ל 3 ערוצים (אלה מתאימות לריכוזים שונים עבור כל דגימה; אפשרויות | אפשרויות Loading | שמירת 6 ערוצי גלם נתונים בתת 3).
- Re-פתוחים קבצי נתונים ששייכים לאותו מדגם וריכוז זהה אך במהירויות שונות בSEDFIT. התאם את המניסקוס (קו אדום אנכי), תחתון תא (קו כחול אנכי) וגבולות ראויים (קווים ירוקים אנכיים), ולייצא את הנתונים לשימוש בSEDPHAT (נתונים | יצוא נתונים לSEDPHAT). קלט הפרמטרים מחושבים בשלב 2.1.1, כמו גם סוג הרוטור וסוג מרכזי כפי שהתבקש. חזור על פעולה זו עבור כל מדגם וריכוז.
- פתח את כל הנתונים מהמדגם זהה (כל ריכוזים ומהירויות) בSEDPHAT ולמלא בפרמטרים של ניסוי; דוגמא מוצגת באיור. 5.
הערה: כאשר D 2 O מתווסף למאגר, דב ' eration של פרוטונים להחלפה באופן משמעותי יכול לשנות את המשקל המולקולרי של החלבון, במיוחד עבור חלבונים מסיסים במים. חלבונים בממברנה, במיוחד אלה קטנים כמו חלבון SH, פחות מושפעות משום אזורים משובצי קרום מוגנים מתמורה. כדי לתקן זאת, קלט "חיץ D חלק mol".
הערה: בשלב זה מומלץ לשמור את בסיס הנתונים בעריכה בנפרד על ידי בחירת נתונים תפריט | להעתיק את כל הנתונים ושמירה בניו Config. - בחר דגם ולמלא בGlobal פרמטרים למודל ש.
הערה: כדוגמא, המודל "מונומר-n-Mer עצמי האגודה" והפרמטרים שלה מוצגים באיור. 6.
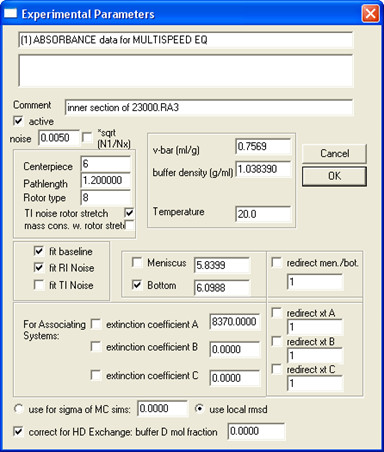
איור 5. דוגמא על איך למלא בפרמטרים של ניסוי.
קבצים / ftp_upload / 52,404 / 52404fig6.jpg "/>
איור 6. דוגמא על איך למלא בGlobal פרמטרים למודל מונומר-n-Mer עצמי איגוד.
- הפעל גלובלי Fit ידי בחירת Fit תפריט | Global Fit ולחכות עד החזרה לנכון. הערה למטה (או לקחת צילום מסך של) את התוצאות הראויה, במיוחד צ'י מרובע המופחת הגלובלי ולהתחבר K ערכים. לחלץ נתונים אחרים, כגון נתונים בכושר ושאריות הולמים מתפריט העתקה והצגה | הצגת מידע תרמודינמיים.
- חזור אל Global פרמטרים ולבדוק M (1) כדי להתאים את המשקל המולקולרי מונומר וחזור על שלב 2.4.5. רשום את המשקל המולקולרי המצויד וצ'י מרובע מופחת גלובלי.
- חזור על שלב 2.4.3 ל2.4.6 עבור כל דגם להיבדק ולהשוות את איכות ההתאמה של כל דגם על ידי השוואת הערך מופחת הגלובלי צ'י מרובע כמו גם שאריות הולמת.
הערה: שאריות הולמים קטנות ואקראיות, בדרך כלל, מציינת בכושר טוב, והמודל המתאים ביותר הייתיש צ'י מרובע המופחת הגלובלי הקטן ביותר. המשקל המצויד מונומר המולקולרי וערך החי בריבוע שלה לא צריכים להיות שונה באופן מהותי מזה של המשקל המולקולרי הקבוע (התיאורטי). - לחשב את רווח הסמך ליומן שהושג על ידי Ka ערך על ידי סטטיסטיקות בחירה הראשונות | צ'י מרובע קריטית לתחזיות משטח שגיאה ומזין את מרווח הביטחון הרצוי. בשלב הבא, ללכת לסטטיסטיקה | צור הקרנת משטח שגיאת 1-ממדית וביטול בחירה להיכנס קה בהדו-שיח גלובלי פרמטרים כדי לקבל את הערכים החיים בריבוע ליומן Ka.
הערה: קוראים מומלץ להתייעץ המקורות הבאים (http://www.analyticalultracentrifugation.com/ sedphat / statistics.htm) לפרטים נוספים על השיטה 32, כמו גם איור של שיטה זו 33.
תוצאות
פרופיל רדיאלי ההפצה של חומרי ניקוי מיצלות C14SB ב 50 מ"מ טריס, 100 מ"מ NaCl pH 7.3 צורות מעריכי מאוד רדודים שיכול להיות מצויד למודל ליניארי (איור 7 א). השיפוע של הפצה זו קשור ביחס הפוך לריכוז D 2 O (איור 7). הנקודה שבה השיפוע הוא אפס, כלומר, D ההתאמה ...
Discussion
מאמר זה מספק פרוטוקול ניסוי להכנת מדגם וניתוח של oligomerization של חלבון קרום קטן בחומר ניקוי באמצעות שיקוע שיווי משקל. הפרוטוקול המתואר באותה מידה תקף -ואז simpler- לחלבונים מסיסים, כמו לא נדרש צעד התאמת צפיפות. ואכן, המערכת היוותה ידי תערובת של חומרי ניקוי וחלבון. לערוך מחקרי ...
Disclosures
The authors have nothing to disclose.
Acknowledgements
This work has been funded by the National Research Foundation grant NRF-CRP4-2008-02 (J.T.) and Tier 1 grant RG 51/13.
Materials
| Name | Company | Catalog Number | Comments |
| 3-(N,N-dimethylmyristylammonio)propanesulfonate | Sigma | T0807 | |
| Deuterium oxide 99.8% | Cambridge Isotope | DLM-4-99.8 | |
| An-50 Ti Rotor, Analytical, 8-Place | Beckman Coulter | 363782 | |
| An-60 Ti Rotor, Analytical, 4-Place | Beckman Coulter | 361964 | |
| Cell housing | Beckman Coulter | 334784 | |
| 12 mm six-channel centerpiece, epon charcoal-filled | Beckman Coulter | 331376 | |
| Window holder | Beckman Coulter | 305037 | |
| Window gasket | Beckman Coulter | 327021 | |
| Window liner | Beckman Coulter | 362329 | |
| Sapphire window | Beckman Coulter | 307177 | |
| Quartz window | Beckman Coulter | 301730 | |
| Screw-ring washer | Beckman Coulter | 362328 | |
| Screw ring | Beckman Coulter | 301922 | |
| Spinkote | Beckman Coulter | 306812 | |
| Torque stand assembly | Beckman Coulter | 361318 | |
| Counterbalance | Beckman Coulter | 360219 | |
| Cell alignment tool | Beckman Coulter | 362340 | |
| SEDNTERP | http://bitcwiki.sr.unh.edu/index.php/Main_Page | ||
| HeteroAnalysis | http://www.biotech.uconn.edu/auf/?i=aufftp | ||
| SEDFIT | http://www.analyticalultracentrifugation .com/sedfit.htm | ||
| SEDPHAT | http://www.analyticalultracentrifugation .com/sedphat/default.htm |
References
- Laue, T. M., Stafford, W. F. Modern applications of analytical ultracentrifugation. Annu. Rev. Biophys. Biomol. Struct. 28, 75-100 (1999).
- Lebowitz, J., Lewis, M. S., Schuck, P. Modern analytical ultracentrifugation in protein science: A tutorial review. Protein Sci. 11, 2067-2079 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. . Assembly, Loading, and Alignment of an Analytical Ultracentrifuge Sample Cell. , e1530 (2009).
- Rivas, G., Stafford, W., Minton, A. P. Characterization of heterologous protein-protein interactions using analytical ultracentrifugation. Methods-a Companion to Methods in Enzymology. 19, 194-212 (1999).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for association and assembly the study of protein. Curr. Opin. Chem. Biol. 10, 430-436 (2006).
- MacGregor, I. K., Anderson, A. L., Laue, T. M. Fluorescence detection for the XLI analytical ultracentrifuge. Biophys. Chem. 108, 165-185 (2004).
- Phizicky, E. M., Fields, S. Protein-Protein Interactions - Methods for Detection and Analysis. Microbiol. Rev. 59, 94-123 (1995).
- Alexandrov, A. A facile method for high-throughput co-expression of protein pairs. Mol. Cell. Proteomics. 3, 934-938 (2004).
- Nooren, I. M. A., Thornton, J. M. Structural characterisation and functional significance of transient protein-protein interactions. J. Mol. Biol. 325, 991-1018 (2003).
- Ebel, C. Sedimentation velocity to characterize surfactants and solubilized membrane proteins. Methods. 54, 56-66 (2011).
- Minton, A. P. Quantitative characterization of reversible macromolecular associations via sedimentation equilibrium: an introduction. Exp. Mol. Med. 32, 1-5 (2000).
- Dowell, S. F. Respiratory syncytial virus is an important cause of community-acquired lower respiratory infection among hospitalized adults. J. Infect. Dis. 174, 456-462 (1996).
- Bukreyev, A., Whitehead, S. S., Murphy, B. R., Collins, P. L. Recombinant respiratory syncytial virus from which the entire SH gene has been deleted grows efficiently in cell culture and exhibits site-specific attenuation in the respiratory tract of the mouse. J. Virol. 71, 8973-8982 (1997).
- Fuentes, S., Tran, K. C., Luthra, P., Teng, M. N., He, B. Function of the respiratory syncytial virus small hydrophobic protein. J. Virol. 81, 8361-8366 (2007).
- Jin, H. Recombinant respiratory syncytial viruses with deletions in the NS1, NS2, SH, and M2-2 genes are attenuated in vitro and in vivo. Virology. 273, 210-218 (2000).
- Karron, R. A. Respiratory syncytial virus (RSV) SH and G proteins are not essential for viral replication in vitro: clinical evaluation and molecular characterization of a cold-passaged, attenuated RSV subgroup B. Proc. Nat. Acad. Sci. USA. 94, 13961-13966 (1997).
- Whitehead, S. S. Recombinant respiratory syncytial virus bearing a deletion of either the NS2 or SH gene is attenuated in chimpanzees. J. Virol. 73, 3438-3442 (1999).
- Rixon, H. W. The small hydrophobic (SH) protein accumulates within lipid-raft structures of the Golgi complex during respiratory syncytial virus infection. J. Gen. Virol. 85, 1153-1165 (2004).
- Collins, P. L., Mottet, G. Membrane orientation and oligomerization of the small hydrophobic protein of human respiratory syncytial virus. J. Gen. Virol. 74, 1445-1450 (1993).
- Collins, P. L., Olmsted, R. A., Johnson, P. R. The small hydrophobic protein of human respiratory syncytial virus: comparison between antigenic subgroups A and B. J. Gen. Virol. 71, 1571-1576 (1990).
- Chen, M. D., Vazquez, M., Buonocore, L., Kahn, J. S. Conservation of the respiratory syncytial virus SH gene. J. Infect. Dis. 182, 1228-1233 (2000).
- Gan, S. W. The small hydrophobic protein of the human respiratory syncytial virus forms pentameric ion channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Gan, S. W., Ng, L., Xin, L., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein Sci. 17, 813-820 (2008).
- Li, Y. Inhibition of the Human Respiratory Syncytial Virus Small Hydrophobic Protein and Structural variations in a bicelle environment. J. Virol. 88 (22), 11899-914 (2014).
- Burgess, N. K., Stanley, A. M., Fleming, K. G. Determination of membrane protein molecular weights and association equilibrium constants using sedimentation equilibrium and sedimentation velocity. Meth. Cell. Biol. 84, 181-211 (2008).
- Cole, J. L., Lary, J. W., Moody, T. P., Laue, T. M. Analytical Ultracentrifugation: Sedimentation Velocity and Sedimentation Equilibrium. Meth. Cell. Biol. 84, 143-179 (2008).
- Fleming, K. G. Determination of membrane protein molecular weight using sedimentation equilibrium analytical ultracentrifugation. Curr. Protoc. Prot. Sci. 53, 17.12.11-17.12.13 (2008).
- . . An-50 Ti and An-60 Ti Analytical Rotor, Cells, and Counterbalance. , (2005).
- Mayer, G. Studying membrane proteins in detergent solution by analytical ultracentrifugation: Different methods for density matching. Prog. Colloid Polym. Sci. 113, 176-181 (1999).
- Laue, T. Ch. 20.3. Current Protocols in Protein Science. 20, 20.23.21-20.23.13 (2001).
- Gan, S. W. The Small Hydrophobic Protein Of The Human Respiratory Syncytial Virus Forms Pentameric Ion Channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Bevington, P. R., Robinson, D. K. . Data reduction and error analysis for the physical sciences. 336, (1969).
- Schuck, P., Radu, C. G., Ward, E. S. Sedimentation equilibrium analysis of recombinant mouse FcRn with murine IgG1. Molecular Immunology. 36, 1117-1125 (1999).
- Gan, S. W., Vararattanavech, A., Nordin, N., Eshaghi, S., Torres, J. A cost-effective method for simultaneous homo-oligomeric size determination and monodispersity conditions for membrane proteins. Anal. Biochem. 416, 100-106 (2011).
- Montserret, R. NMR structure and ion channel activity of the p7 protein from hepatitis C virus). J. Biol. Chem. 285, 31446-31461 (2010).
- Stouffer, A. L., DeGrado, W. F., Lear, J. D. Analytical Ultracentrifugation Studies of the Influenza M2 Homotetramerization Equilibrium in Detergent Solutions. Progr Colloid Polym Sci. 131, 108-115 (2006).
- Sorkin, A., von Zastrow, M. Signal transduction and endocytosis: Close encounters of many kinds. Nat. Rev. Mol. Cell Biol. 3, 600-614 (2002).
- Gan, S. W., Ng, L., Lin, X., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein science : a publication of the Protein Society. 17, 813-820 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved