Zum Anzeigen dieser Inhalte ist ein JoVE-Abonnement erforderlich. Melden Sie sich an oder starten Sie Ihre kostenlose Testversion.
Method Article
Sedimentationsgleichgewicht eines Klein Oligomer bildenden Membrane Protein: Wirkung von Histidin Protonierung auf pentamere Stabilität
In diesem Artikel
Zusammenfassung
Sedimentation equilibrium (SE) can be used to study protein-protein interactions in a physiological environment. This manuscript describes the use of this technique to determine the effect of pH on the stability of a homo-pentamer formed by the small hydrophobic (SH) protein encoded by the human syncytial respiratory virus (hRSV).
Zusammenfassung
Analytische Ultrazentrifugation (AUC) kann verwendet werden, um reversible Wechselwirkungen zwischen Makromolekülen über einen weiten Bereich von Wechselwirkungsstärken und unter physiologischen Bedingungen untersucht werden. Dies macht AUC eine Methode der Wahl die quantitative Erfassung Stöchiometrie und Thermodynamik von Homo- und Hetero-Vereinigung, die vorübergehende und reversible in biochemischen Prozessen sind. In der Modalität Sedimentationsgleichgewichts (SE), ein Gleichgewicht zwischen Diffusion und Sedimentation ein Profil als eine Funktion der radialen Distanz, die an einem bestimmten Assoziationsmodell abhängt. Hierin wird eine detaillierte SE Protokoll beschrieben, um die Größe und die Monomer-Monomer-Assoziationsenergie eines kleinen Membranprotein-Oligomer mittels einer analytischen Ultrazentrifuge bestimmen. AUC-ES ist markierungsfreie, nur auf physikalischen Prinzipien beruhen, und kann auf beiden wasserlöslich und Membranproteinen verwendet werden. Ein Beispiel des letzteren gezeigt ist, die kleinen hydrophoben (SH) Protein im menschlichen Respiratory Syncytial Virus (HRSV), Ein 65-Aminosäuren-Polypeptid mit einer einzigen α-Helix-Transmembran (TM), die gegen pentamere Ionenkanäle bildet. NMR basierenden Strukturdaten zeigt, daß SH-Protein hat zwei protonierbare His-Resten in seiner Transmembrandomäne, die dem Lumen zugewandt orientiert des Kanals. SE-Experimente wurden entworfen, um festzustellen, wie pH Assoziationskonstante und die oligomere Größe SH Proteins beeinflusst. Während die pentameren Form wurde in allen Fällen erhalten, wurde sein Assoziationskonstante bei niedrigen pH reduziert. Diese Daten sind in Übereinstimmung mit einem ähnlichen pH-Abhängigkeit für SH Kanalaktivität beobachtet, die mit einer Lumen Orientierung der beiden His-Reste in SH-Protein. Letztere können die elektrostatische Abstoßung und reduziert Oligomer Stabilität bei niedrigen pH zu erleben. Zusammenfassend ist das Verfahren anwendbar, wenn die quantitative Informationen über feine Assoziation von Proteinen Veränderungen in physiologischen Bedingungen gemessen werden.
Einleitung
Analytische Ultrazentrifugation 1-5 ist eine der wichtigsten Methoden, um Wechselwirkungen von Makromolekülen, die unter physiologischen Bedingungen zu studieren, zugänglich sowohl schwache und starke Wechselwirkung. Die Methode ist markierungsfreie und nutzt Lichtabsorption oder Störungen und sogar Fluoreszenz optische Systeme verwendet werden, um Konzentrationsbereiche über mehrere Größenordnungen 6 zuzugreifen.
Dieses Verfahren ist besonders nützlich, da die meisten biochemischen Prozessen abhängen reversible Interaktionen. Die Stöchiometrie und die Stärke dieser Wechselwirkungen müssen quantitativ charakterisiert, um biologische Prozesse zu verstehen, und eine Reihe von Methoden für diesen Zweck 7, 8 vorhanden sind. Allerdings sind vorübergehende Wechselwirkungen schwer zu untersuchen 9.
Die Wahl eines Verfahrens zur makromolekularen Wechselwirkungen charakterisieren hängt von seiner statischen oder dynamischen Natur. Im ersten Fall, SEDIM entation Geschwindigkeit (SV) verwendet wird, wobei die Rate der radialen Transport gemessen und Komplexe werden auf der Grundlage von Unterschieden in Auftriebs Masse und Form fraktioniert.
Im Gegensatz dazu können dynamische Verbände, die auf der Zeitskala des Experiments reversible nicht physisch getrennt sein. In diesem Fall kann selbst- oder hetero-Wechselwirkungen, die zu nicht-kovalente Wechselwirkungen in einem Gleichgewicht stehen, die auf der Gesamtproteinkonzentration abhängt. Diese dynamischen Wechselwirkungen kann sowohl Sedimentationsgleichgewicht (SE) und Sedimentation Geschwindigkeit (SV) 10 untersucht werden. Jedoch ist das erste Verfahren einfacher durchzuführen, und wird hier beschrieben. In SE wird eine Zentrifugation bei einer ausreichend niedrigen Geschwindigkeit durchgeführt, so dass ein Gleichgewicht zwischen Diffusion und Sedimentation erreicht. An diesem Punkt wird das Gleichgewichtsprofil eines optischen Signals (UV-VIS) als eine Funktion des radialen Abstands, können mit vorgegebenen thermodynamischen Modellen für Verbände 11 analysiert werden.
ve_content "> In der vorliegenden Arbeit wird eine Sedimentationsgleichgewichts Untersuchung der Selbstassoziation von einem viralen Membranprotein, Ionenkanäle bildet. Aufgrund ihrer Hydrophobizität präsentiert wird das Experiment in Gegenwart eines Detergens ausgeführt wird, und in diesem Fall die Dichte Lösungsmittel auf, dass der Reinigungsmittel abgestimmt werden. Das beschriebene Protokoll würde im Falle eines wasserlöslichen Proteins identisch ist, außer daß kein Lösungsmittel Dichteanpassung erforderlich wäre.Das verwendete Protein ist in der menschlichen Respiratory Syncytial Virus (HRSV), ein umhülltes Pneumovirus der Paramyxoviridae Familie, die Erkrankung der unteren Atemwege bei Säuglingen und älteren Menschen immun Populationen weltweit 12 bewirkt kodiert. Bis zu 64 Millionen Fälle von HRSV Infektion und 160.000 Todesfälle pro Jahr.
HRSV Genom transkribiert 11 Proteinen, einschließlich der drei Membranproteine F, G und kleine hydrophobe (SH). SH-Proteins beteiligt istbei der Pathogenese von RSV-Infektion. RSV fehlt das SH-Gen (RSVΔSH) war lebensfähig, verursacht die Bildung von Syncytien und wuchs als auch der Wildtyp (WT) Virus 13-16. Jedoch repliziert RSVΔSH Virus 10-mal weniger wirksam als das WT in den oberen Atemwegen 15, 16. Außerdem wurde RSVΔSH Virus in in vivo-Maus und Schimpansen Modelle 13, 17 gedämpft.
Der SH-Protein ist ein 64 (RSV Untergruppe A) oder 65 (RSV Untergruppe B) Aminosäuren lang Typ II integrales Membranprotein, die meist an den Membranen des Golgi-Kompartiment 18 ansammelt. SH-Protein hat eine einzige sagte eine Helix-Transmembran (TM) Domäne 19, die hoch konserviert ist 20,21. Die C- und N-terminalen Domänen extramembranären lumenally / extrazellulär cytoplasmatisch, jeweils ausgerichtet sind.
Sowohl synthetische TM-Domäne (Reste 18-43) Und in voller Länge SH Protein wurde gezeigt, dass homopentamers in einer Vielzahl von Reinigungsmitteln zu bilden. Die homopentameric Form ist für die Kanalaktivität in planaren Lipid-Doppelschichten 22,23 verantwortlich. Die korrekte Orientierung der TM-Monomeren in der Lipiddoppelschicht wurde zuerst bestimmt unter Verwendung von ortsspezifischen Infrarot-Dichroismus 23, die zeigten, His-22 in einer luminalen Seite nahe inter-helikalen, Orientierung. Das gleiche TM Domänenorientierung wurde durch NMR-Untersuchungen, die pentameren eine -helikale Bündel des gesamten Proteins in Dodecylphosphocholin (DPC) rekonstruiert Micellen 22 bestätigt. In diesem Modell "Mizelle", ein einzelnes a- helikalen Transmembrandomäne wurde N-terminal durch eine a-Helix durch eine erweiterte b-Hairpin flankiert, und C-terminal. Die beiden protonierbaren Reste von SH-Protein, His-22 und His-51, sind in der TM-Domäne (lumenally orientiert) angeordnet ist, und an der Spitze des extramembranären C-terminalen β Hairpins (vom Kanalpore) sind. In einem bicellar environment erstreckt sich jedoch das TM α-Helix bis His-51, und beide His-Resten zugänglich dem Lumen des Kanals 24 sind. Die Kanalstruktur nimmt eine trichterartige Architektur 22, wobei der engere Bereich (Ser-29 bis Cys-45) 22 ist mit hydrophoben Seitenketten (Ile-32 Ile-36 Ile-40 und Leu-44) und ausgekleidet Ile-36 ist der engsten Stelle in der Kanal Lumen. His-22 mit der größten Öffnung des Trichters, während His-51 ist an der Spitze der kleinsten Öffnung.
In der vorliegenden Arbeit, analytischen Zentrifugation in einer Sedimentationsgleichgewicht Modus verwendet wurde, um festzustellen, ob seine Protonierung die Stabilität des SH-Protein Pentamer betrifft. In diesem Fall wurde SH-Protein in C14-Betain-Wasch, die zuvor verwendet wurde, um die sh Proteinformen pentameren Oligomere 22 zeigen solubilisiert.
Protokoll
Dieses Protokoll wird auf die folgenden Ressourcen, die für weitere Informationen und spezielle Überlegungen 3, 25 bis 28 bezeichnet sind, beruht.
1 Dichteanpassung der Detergensmicellen mit 2 H 2 O
Anmerkung: Die Dichte der Pufferlösung muss auf die Dichte der Detergensmicellen angepaßt werden. Gemeinsame Dichte einstellende Mittel schließen 2 H 2 O, H 2 18 O, 2 H 2 18 O, Glycerin und Saccharose 29. H 2 18 O hat die gleiche Dichte wie 2 H 2 O, und kann eine bessere Wahl sein, wenn Deuterierung von austauschbaren Protonen im Protein nicht erwünscht ist. In diesem Verfahren wird die Dichte von 3- (N, N-dimethylmyristylammonio) -propanesulfonate (C14SB) Waschmittel in 50 mM Tris pH 7,3, 100 mM NaCl mit 2 H 2 O. abgestimmt werden Als Startwert die folgenden Konzentrationen von2 H 2 O verwendet werden: 10, 30 und 50% v / v.
1.1. Probenvorbereitung
- Die folgenden Stammlösungen und Filter durch einen 0,2 um Spritzenfilter sterilisieren: 50 ml 500 mM Tris pH 7,3 und 1 M NaCl (10X-Pufferlösung); 1 ml 250 mM C14SB (50X Reinigungslösung).
- Bereiten 200 ul Probenlösung durch Mischen von 20 ul 10x-Puffer-Lösung, 4 ul 50X Waschmittellösung, 20 ul 2 H 2 O (99,9%), und 156 & mgr; l deionisiertes H 2 O. Planen auch 200 ul Referenzlösung durch Mischen von 20 ul 10x-Puffer-Lösung, 20 & mgr; 2 H 2 O (99,9%), und 160 & mgr; l deionisiertes H 2 O.
- Wiederholen Sie Schritt 1.1.2 für die anderen 2 H 2 O-Konzentrationen, das heißt 30% bis 50%, Einstellen der 2 H 2 O und H 2 O beträgt entsprechend.
1.2. Versammlung der 6-Kanal-AUC-Zellen und Probenbeladung in die Zellen.
Hinweis: Es gibt zwei Arten von AUC Zelle in Abhängigkeit von der Probenladeverfahren. Zellen ohne äußere Füllung hat vor dem Abdichten der Zelle geladen werden, während externe Befüllung Zellen geladen, nachdem die Zellen zu verschließen. Montage eines externen füllen AUC Zelle zuvor 3 beschrieben. In diesem Protokoll wird die Anordnung einer 6-Kanal-AUC Zelle ohne externe Füllung beschrieben. Der Hauptunterschied ist, dass es Schraubenringe auf beiden Seiten die getrennt angezogen werden müssen, und es braucht nicht Gehäusestecker (Fig. 1). Der Unterschied in der Montageschritte sind unten hervorgehoben.
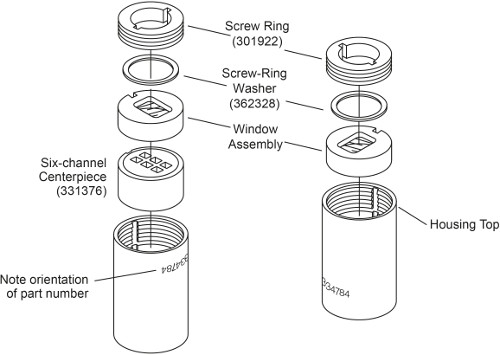
Abbildung 1: Explosionszeichnung eines 6-Kanal-AUC Zelle ohne äußere Füllung. Diese Zahl hat sich von Beckman Coulter An-50 Ti modifiziert und An-60 Ti Analytical Rotor, Zellen und Front Bedienungsanleitung.
- Bereiten Sie zwei Glasscheiben für jeden AUC Zelle mit Saphirfenster anstelle von Quarzfenster (Abb. 2). Legen Sie die Fensterdichtung in das Fenster Halter. Biegen Sie das Fenster Liner und legen Sie sie in das Fenster Halter, so dass der Zwischenraum gegenüber dem Fenster Halter Nut gebildet. Stellen Sie das Saphirfenster in das Fenster Liner, die Ausrichtung der Marke mit dem Fensterhalter Nut.
Hinweis:. Das Quarzfenster komprimierbar und damit mehr Lichtbrechung bei hoher Geschwindigkeit 28, 30 daher für Interferenzmessungen über 30.000 Umdrehungen pro Minute, wie in dieser Dichte passenden Experiment zu erzeugen, werden Saphirfenstern verwendet. Ein Saphirfenster ist schwerer als Quarzfenster und hat eine "X" auf die Seite geätzt sind.
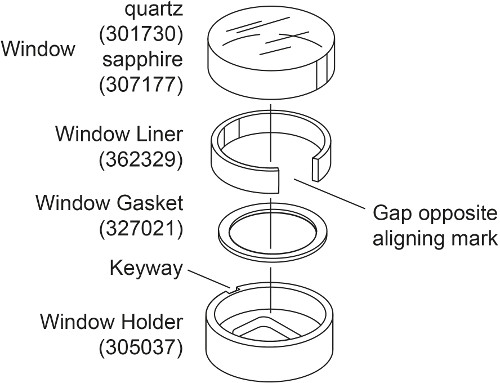
Abbildung 2. Explosions of der Fensteranordnung. Diese Zahl hat sich von Beckman Coulter An-50 Ti und An-60 Ti Analytische Rotor, Zellen und Front Bedienungsanleitung geändert worden
- Setzen Sie den Zellengehäuse mit der Teilenummer auf den Kopf. Mit den Keilnuten mit dem Gehäuse Schlüssel ausgerichtet ist, schieben Sie in die Zelle Gehäuse zunächst ein 6-Sektor Herz mit abgeschrägten Seite nach unten, gefolgt von einer Fensteranordnung mit dem Fenster nach unten (Abb. 1, links).
- Bestreichen Sie die Schraube Ring Gewinde und Schraubenringscheibe mit Spinkote. Legen Sie eine Schraube Ringscheibe auf der Fensteranordnung. Die Schraube Ring in das Fenster Gehäuse mit dem Wort "OUT" nach außen zeigt. Hand ziehen Sie die Schraube Ring mit der Zell Ausrichthilfe.
- Mit dem Drehmomentschlüssel die Schraube Ring nur 60 inch-pounds.
- Legen Sie die Zelle mit der Teilenummer aufrecht und bei 12-Uhr positioniert. Load 120 ul Bezugnahme in den linken Reihen und 110 ul-Probe in die richtigen Zeilen. Stellen Sie sicher, dass jede Probe und Referenz korrekt gepaart.
Hinweis: Genaue Probenvolumen ist nicht kritisch, aber die Bezugs muss etwas mehr Volumen als die Probe (5-10 ul) haben, so dass die Probenmeniskus unterscheidbar sein. - Schieben Sie vorsichtig in das Zellengehäuse eine Fensteranordnung mit dem Fenster nach unten (Abb. 1, rechts). Darauf achten, dass die Zelle übermäßig zu stören und den Inhalt zu verschütten.
- Wiederholen Sie Schritt 1.2. 3 und 1.2. 4 Festziehen der zweiten Schraubring zu 120 Zoll-Pfund. Kehren Sie die Zelle und ziehen Sie die erste Schraubring zu 120 Zoll-Pfund.
- Laden Sie die Zellen in den Rotor, installieren Sie den Rotor in der Zentrifuge und installieren Sie den Monochromator nach den Anweisungen des Herstellers 28.
Hinweis: Details zu diesem step können auch in dieser Referenz 3 gefunden werden.
1.3. Einrichten Interferenzmessung
- Starten Sie die Benutzerschnittstellensoftware für die AUC Instrument und führen Laseraufbau und radiale Kalibrierung für jede Zelle bei 3000 rpm, entsprechend den Anweisungen des Herstellers, die im folgenden kurz Schritt zusammengefasst werden.
- 1.3.1.1 Nach ausreichender Unterdruck erreicht ist (<100 Mikrometer), starten Sie die Zentrifuge bei 3000 Umdrehungen pro Minute. Vorschau des Interferenzmusters in der Benutzerschnittstellen-Software und stellen Sie die Laserparameter, um den höchsten Kontrast zu erhalten.
- Erstellen Sie eine neue Datei einrichten (Datei | Neue Datei) die Angabe "Equilibrium" und "Interference" Messung. Richten Sie eine Sedimentationsgleichgewichtsmethode ("Methode" Taste), um bei 45.000 Umdrehungen pro Minute oder die höchste Geschwindigkeit für Proteinproben zu erwarten, je nachdem, was höher ist bei 20 ° C ausgeführt wird, mit Lauftemperatur und sammeln ein Scan alle 15 Minuten. Monitor Gleichgewicht Fortschritt durch Öffnen der Dateien in HeteroAnalysis und Auswählen von Funktionen "Spiel" nach mindestens 12 Stunden (etwa über Nacht, Abb. 3).
Hinweis: eine ähnliche Funktion ist auch in SEDFIT (Optionen | Loading Optionen | Test Annäherung an das Gleichgewicht).

Abbildung 3. Ergebnis aus HeteroAnalysis Spiel-Funktion. Die Spiel-Funktion kann verwendet werden, um das Gleichgewicht Fortschritte durch einen Vergleich RMSD zwischen aufeinander folgenden Scans und der letzten Untersuchung zu überwachen. Dieses Beispiel zeigt, nach Gleichgewichtseinstellung nach 8 h hat RMSDs asymptotisch zur X-Achse angegeben.
1.4. Datenanalyse
- Für jeden Satz von Proben, plotten die Steigung der radialen Verteilungsprofil gegen den 2 H 2 O-Konzentration.
Hinweis: Die Verteilung wird eine sehr flache exponentielle dass ap seinAnsätze Linearität. X-Achsen-Abschnitt entspricht der pass 2 H 2 O-Konzentration. - Verfeinern, führen das Experiment in mehreren Wiederholungen. Oder wiederholen Sie den Versuch mit einem engeren Bereich von 2 H 2 O-Konzentration.
2. Sedimentationsgleichgewicht von SH in C14SB Mizellen
2.1. Führen Parameter
- Berechnen Puffer Dichte und Viskosität, Protein partielle spezifische Volumen und die Zentrifugiergeschwindigkeit mithilfe SEDNTERP. Um Puffer Dichte und Viskosität, wählen Sie Berechnen im Abschnitt "Pufferdaten Wählen Sie" berechnen und geben Sie die Pufferkomponenten entsprechend, einschließlich der D 2 O-Konzentration.
- 2.1.1.1 Um im Abschnitt "V-bar" berechnen Protein teilweisen spezifischen Volumen, wählen Compute und geben Sie die Protein-Aminosäuresequenz. Geben Sie die höchste erwartete oligomere Größe in "Make ein Oligomer von diesem Monomer: N ="Feld, in diesem Fall N = 5. Berechnen Sie die Geschwindigkeit, indem Sie Werte in der RPM-Feld im Hauptfenster, bis σ ≈ 1; dies ist eine Faustregel, um eine gute exponentielle Form der radialen Verteilungsprofil 25 zu gewährleisten.
Anmerkung: Die für dieses Experiment berechneten Werte waren wie folgt: ρ = 1,03839 g / ml, η = 1,0267 cP = 0,7569 ml / g, ω 1 = 16000 rpm. - Berechnen nachfolgenden Geschwindigkeiten zu folgen, um eine ausreichende Differenz zwischen dem Verteilungsprofil mit einer Geschwindigkeit und die nächsten 25 zu gewährleisten.
Hinweis: Diese kann auch von der Funktion durchgeführt werden "schätzen Gleichgewicht Rotor beschleunigt Funktion" in SEDFIT, die berücksichtigt die Lösung Säule (Füllvolumen).
2.2. Probenvorbereitung
- Bereiten Sie 1 ml Referenzlösung mit 5 mM C14SB und 32,3% 2 H 2 O ab Dichte passenden Experiment (Abschnitt 1), durch Mischen von 100 ul 10x-Puffer solutio bestimmtn (Schritt 1.1.1), 20 ul 50X Reinigungslösung (Schritt 1.1.1), 323 ul 2 H 2 O (99,9%) und 527 & mgr; l deionisiertes H 2 O.
- Löse lyophilisiert, HPLC-gereinigt SH Peptide (Expression und Reinigung vorher 31 beschrieben) in einem geeigneten Lösungsmittel, wie Methanol oder 50% v / v wässrigem Acetonitril. Messen A280 der gelösten Peptide in einer Mikroliter-Maßstab UV / Vis-Spektrophotometer und Aliquot für drei Proben zu A 280 zu geben, 12mm = 0,3, 0,5 und 0,8 (A 280, 10 mm = 0,25, 0,417 und 0,67), die jeweils, wenn sie verdünnt 130 ul. Lyophilisieren die Proben über Nacht und resuspendieren in 130 ul Standardlösung (Schritt 2.2.1), um die Musterlösungen zu geben.
Anmerkung: SH-Protein kann von UV / Vis-Absorption bei 280 nm nachgewiesen, weil sie Trp und Tyr-Reste enthalten sein. Proteine ohne aromatischen Reste können durch Markieren mit einem geeigneten Chromophor mit einem Trp enthaltenden Mutante oder durch Verwendung stören erkannt werdennce Messungen anstelle von Absorption. - Führen Sie die Schritte in Abschnitt 1.2, um einen 6-Kanal-AUC Zelle mit Quarzfenster montieren. Legen Sie die höchste Konzentration Probe (A 280, 12 mm = 0,8) in den Kanal am nächsten an der Rotormitte und niedrigste Konzentration Probe (A 280, 12 mm = 0,3) am weitesten von der Mitte des Rotors.
2.3. Einrichten Absorptionsmessungen
- Erstellen Sie eine neue Datei einrichten (Datei | Neue Datei) die Angabe "Equilibrium" und "Absorption" Messung. Geben Sie 280 nm als Detektor Wellenlänge.
- Führen radialen Kalibrierung bei 3000 Umdrehungen pro Minute, indem Sie "Radial Kalibrierung vor dem ersten Scan" im Scan-Optionen, unter Angabe der Datenerhebung bei niedriger Auflösung, zB mit Radial-Schrittweite = 0,01 cm, Replikate = 3 (niedrige Auflösung, schnell), und die Ausführung eines Single Scan . Nachdem der Scan abgeschlossen ist, deaktivieren Sie die Option.
- Richten Sie eine Sedimentationsgleichgewichtsmethode ("Method "Taste), um mit der ersten Geschwindigkeit in Schritt 2.1.3 berechnet laufen, bei 20 ° C, und sammeln Sie ein Scan alle 30 Minuten. In allen Zellen "Detail", geben Sie die Datenerfassung mit niedriger Auflösung, wie in Schritt 2.3.2. Überwachen Gleichgewicht Fortschritt durch Öffnen der Dateien in HeteroAnalysis und Auswählen von Funktionen "Spiel" nach mindestens 18 Stunden (etwa über Nacht, Abb. 4).
Anmerkung: Gleichgewichtseinstellung kann wesentlich längere Zeit für die erste Geschwindigkeit zu nehmen, während die nachfolgenden Geschwindigkeiten wird weniger Zeit.
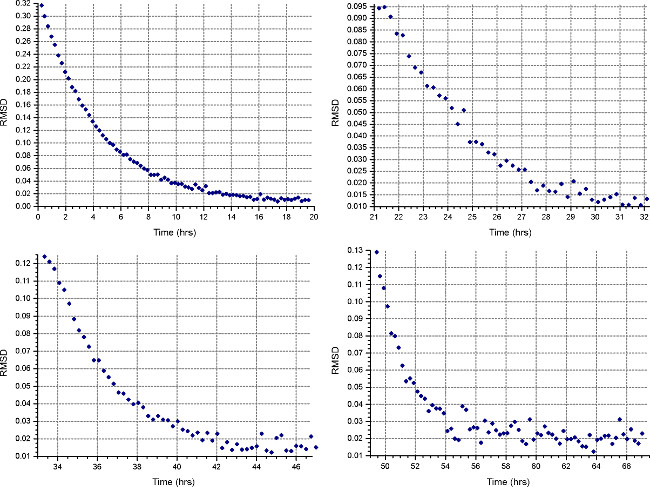
Abbildung 4. Die Ergebnisse HeteroAnalysis Spiel-Funktion. Die erste und zweite Drehzahlen (oben links und rechts) erscheinen, um ein Gleichgewicht erreicht haben, aber es ist besser, ein paar Stunden warten, um sicher zu sein. Im Vergleich dazu haben die dritten und vierten Gang (links unten und rechts) deutlich Gleichgewicht erreichtin kürzerer Zeit. Bitte klicken Sie hier, um eine größere Version dieses Bild anzuzeigen.
- Nachdem das Gleichgewicht erreicht ist, sammeln einen einzigen Scan mit hoher Auflösung, zB mit Radial Schrittgröße = 0.001 cm, Replikate = 10 (hohe Auflösung, langsam).
- Nachdem der Scan abgeschlossen ist, wiederholen Sie die Schritte 2.3.3 und 2.3.4 für die nächste Geschwindigkeit.
- Optional kann, wenn die erforderliche Zeit, um zu erreichen Gleichgewicht für jede Drehzahl ist bekannt (berechnet oder aus Erfahrung), die Sedimentationsgleichgewichtsmethode Scan eingestellt ist, um alle in Schritt 2.1.3 und berechnet Geschwindigkeiten sind bis 1 Scan nach der Gleichgewichtszeit für jeden zu sammeln Geschwindigkeit. In diesem Fall geben Sie hochauflösende Datenerhebung eines Zellen "Detail".
2.4. Datenanalyse in SEDFIT und SEDPHAT
Hinweis: Weitere Details und die Überlegungen bei der Datenanalyse Leser verwiesen to der folgenden Website: www.analyticalultracentrifugation.com.
- Öffnen Sie hochauflösende Scans in SEDFIT (Data | Last Sedimentationsgleichgewichtsdaten) und teilen Sie die Daten in 3 Kanäle (diese entsprechen unterschiedlichen Konzentrationen für jede Probe, Optionen | Loading Optionen | Speichern 6-Kanal-Rohdaten in 3 Untergruppen).
- Wieder offenen Dateien, die auf der gleichen Probe und derselben Konzentration jedoch mit unterschiedlichen Geschwindigkeiten in SEDFIT gehören. Stellen Sie den Meniskus (senkrechte rote Linie), Zellenboden (vertikale blaue Linie) und Pass Grenzen (vertikale grüne Linie), und exportieren Sie die Daten für den Einsatz in SEDPHAT (Data | Daten exportieren, um SEDPHAT). Eingabe der in Schritt 2.1.1 berechnet sowie Rotortyp und Kerntyp wie geforderten Parameter. Wiederholen Sie diesen Schritt für jede Probe und Konzentration.
- Öffnen Sie alle Daten aus der gleichen Probe (alle Konzentrationen und Geschwindigkeiten) in SEDPHAT und füllen Experiment-Parameter; ein Beispiel ist in Fig. 5.
Hinweis: wenn D 2 O in dem Puffer, deut hinzugefügt ration von austauschbaren Protonen könnte das Molekulargewicht des Proteins wesentlich verändern, insbesondere für wasserlösliche Proteine. Membranproteine, vor allem kleine wie SH-Protein, sind weniger betroffen, da Membran eingebetteten Regionen sind vom Umtausch geschützt. Um dies zu korrigieren, geben Sie die "Puffer D Molenbruch".
Hinweis: Bei diesem Schritt wird empfohlen, um die bearbeitete Datensatz einzeln, indem Sie im Menü Daten speichern | Kopieren aller Daten und Save As New Config. - Wählen Sie ein Modell und füllen Globale Parameter für dieses Modell.
Hinweis: Ein Beispiel sind die "Monomer-n-Mer Selbstassoziation" Modell und seine Parameter in Abb. 6.
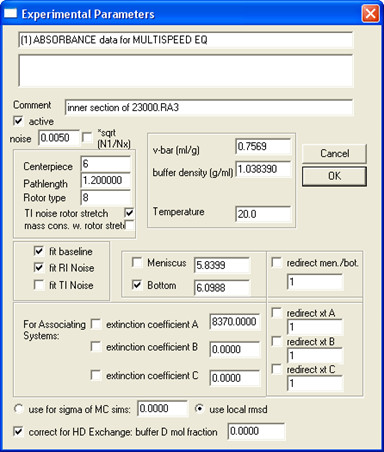
Abbildung 5. Ein Beispiel, wie man in der experimentellen Parameter zu füllen.
Dateien / ftp_upload / 52.404 / 52404fig6.jpg "/>
Abbildung 6. Ein Beispiel, wie man in Globale Parameter für die Monomer-n-Mer Selbstassoziation Modell zu füllen.
- Führen Sie einen globalen Fit über den Menü Fit | Die globale Fit und warten, bis die Passform konvergiert. Notieren Sie sich (oder nehmen Sie einen Screenshot) die passenden Ergebnisse, vor allem die globale reduzierte Chi-Quadrat-und log K a-Werte. Extrahieren Sie andere Daten wie Passdaten und passende Residuen aus dem Menü Kopieren und Anzeigen | Anzeigen Thermodynamische Informationen.
- Zurück zur Seite Globale Parameter und überprüfen Sie M (1), das Monomer Molekulargewicht passen, und wiederholen Sie Schritt 2.4.5. Notieren Sie die Einbaumolekulargewicht und die globale reduzierte Chi-Quadrat.
- Wiederholen Sie die Schritte 2.4.3 bis 2.4.6 für jedes Modell zu testen und vergleichen die Passqualität jedes Modell durch einen Vergleich der verringerten globalen Chi-Quadrat-Wert sowie passende Residuen.
Hinweis: Kleine und Zufallspass Residuen allgemein eine gute Passform, und das Modell, das am besten wäreden geringsten globalen reduziert Chi-Quadrat. Die Einbau Monomer Molekulargewicht und seine Chi-Quadrat-Wert sollte nicht wesentlich von dem des feststehenden (theoretische) Molekulargewicht unterscheiden. - Berechnen Sie das Vertrauensintervall für die erhaltenen log Ka von Wert von ersten Auswahl Statistik | Kritische Chi-Quadrat für Fehler Oberflächenprojektionen und die Eingabe des gewünschten Konfidenzintervalls. Als nächstes, um Statistik Forum | Gene 1-dimensionale Fehler Flächenprojektion und deaktivieren loggen Ka im Dialog Globale Parameter, die Chi-Quadrat-Werte für log Ka erhalten.
Hinweis: Leser wird empfohlen, die folgenden Quellen (http://www.analyticalultracentrifugation.com/ sedphat / statistics.htm) für weitere Informationen über die Verfahren 32 sowie Abbildung der Methode 33 zu konsultieren.
Ergebnisse
Die radiale Verteilungsprofil C14SB Detergensmicellen in 50 mM Tris, 100 mM NaCl pH 7,3 bildet einen sehr flachen Exponentialfunktion, die auf einem linearen Modell (7A) ausgestattet werden kann. Die Steigung dieser Verteilung wird invers mit D 2 O-Konzentration (Abbildung 7B) korreliert. Der Punkt, wo die Neigung Null ist, das heißt, die Anpassungs D 2 O-Konzentration, wurde gefunden, daß 32,3% sein.
Siehe Abbildung ...
Diskussion
Dieses Dokument enthält eine experimentelle Protokoll für die Probenvorbereitung und Analyse von Oligomerisierung von einem kleinen Membranprotein in Waschmittel mit Gleichgewicht Sedimentation. Das beschriebene Protokoll ist gleichermaßen gültig -und simpler- für lösliche Proteine, wie die Dichte Anpassungsschritt nicht erforderlich ist. Tatsächlich wird das System durch ein Gemisch aus Reinigungsmittel und Protein gebildet ist. Um Sedimentation Studien durchzuführen, muss das Reinigungsmittel unsichtbar für d...
Offenlegungen
The authors have nothing to disclose.
Danksagungen
This work has been funded by the National Research Foundation grant NRF-CRP4-2008-02 (J.T.) and Tier 1 grant RG 51/13.
Materialien
| Name | Company | Catalog Number | Comments |
| 3-(N,N-dimethylmyristylammonio)propanesulfonate | Sigma | T0807 | |
| Deuterium oxide 99.8% | Cambridge Isotope | DLM-4-99.8 | |
| An-50 Ti Rotor, Analytical, 8-Place | Beckman Coulter | 363782 | |
| An-60 Ti Rotor, Analytical, 4-Place | Beckman Coulter | 361964 | |
| Cell housing | Beckman Coulter | 334784 | |
| 12 mm six-channel centerpiece, epon charcoal-filled | Beckman Coulter | 331376 | |
| Window holder | Beckman Coulter | 305037 | |
| Window gasket | Beckman Coulter | 327021 | |
| Window liner | Beckman Coulter | 362329 | |
| Sapphire window | Beckman Coulter | 307177 | |
| Quartz window | Beckman Coulter | 301730 | |
| Screw-ring washer | Beckman Coulter | 362328 | |
| Screw ring | Beckman Coulter | 301922 | |
| Spinkote | Beckman Coulter | 306812 | |
| Torque stand assembly | Beckman Coulter | 361318 | |
| Counterbalance | Beckman Coulter | 360219 | |
| Cell alignment tool | Beckman Coulter | 362340 | |
| SEDNTERP | http://bitcwiki.sr.unh.edu/index.php/Main_Page | ||
| HeteroAnalysis | http://www.biotech.uconn.edu/auf/?i=aufftp | ||
| SEDFIT | http://www.analyticalultracentrifugation .com/sedfit.htm | ||
| SEDPHAT | http://www.analyticalultracentrifugation .com/sedphat/default.htm |
Referenzen
- Laue, T. M., Stafford, W. F. Modern applications of analytical ultracentrifugation. Annu. Rev. Biophys. Biomol. Struct. 28, 75-100 (1999).
- Lebowitz, J., Lewis, M. S., Schuck, P. Modern analytical ultracentrifugation in protein science: A tutorial review. Protein Sci. 11, 2067-2079 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. . Assembly, Loading, and Alignment of an Analytical Ultracentrifuge Sample Cell. , e1530 (2009).
- Rivas, G., Stafford, W., Minton, A. P. Characterization of heterologous protein-protein interactions using analytical ultracentrifugation. Methods-a Companion to Methods in Enzymology. 19, 194-212 (1999).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for association and assembly the study of protein. Curr. Opin. Chem. Biol. 10, 430-436 (2006).
- MacGregor, I. K., Anderson, A. L., Laue, T. M. Fluorescence detection for the XLI analytical ultracentrifuge. Biophys. Chem. 108, 165-185 (2004).
- Phizicky, E. M., Fields, S. Protein-Protein Interactions - Methods for Detection and Analysis. Microbiol. Rev. 59, 94-123 (1995).
- Alexandrov, A. A facile method for high-throughput co-expression of protein pairs. Mol. Cell. Proteomics. 3, 934-938 (2004).
- Nooren, I. M. A., Thornton, J. M. Structural characterisation and functional significance of transient protein-protein interactions. J. Mol. Biol. 325, 991-1018 (2003).
- Ebel, C. Sedimentation velocity to characterize surfactants and solubilized membrane proteins. Methods. 54, 56-66 (2011).
- Minton, A. P. Quantitative characterization of reversible macromolecular associations via sedimentation equilibrium: an introduction. Exp. Mol. Med. 32, 1-5 (2000).
- Dowell, S. F. Respiratory syncytial virus is an important cause of community-acquired lower respiratory infection among hospitalized adults. J. Infect. Dis. 174, 456-462 (1996).
- Bukreyev, A., Whitehead, S. S., Murphy, B. R., Collins, P. L. Recombinant respiratory syncytial virus from which the entire SH gene has been deleted grows efficiently in cell culture and exhibits site-specific attenuation in the respiratory tract of the mouse. J. Virol. 71, 8973-8982 (1997).
- Fuentes, S., Tran, K. C., Luthra, P., Teng, M. N., He, B. Function of the respiratory syncytial virus small hydrophobic protein. J. Virol. 81, 8361-8366 (2007).
- Jin, H. Recombinant respiratory syncytial viruses with deletions in the NS1, NS2, SH, and M2-2 genes are attenuated in vitro and in vivo. Virology. 273, 210-218 (2000).
- Karron, R. A. Respiratory syncytial virus (RSV) SH and G proteins are not essential for viral replication in vitro: clinical evaluation and molecular characterization of a cold-passaged, attenuated RSV subgroup B. Proc. Nat. Acad. Sci. USA. 94, 13961-13966 (1997).
- Whitehead, S. S. Recombinant respiratory syncytial virus bearing a deletion of either the NS2 or SH gene is attenuated in chimpanzees. J. Virol. 73, 3438-3442 (1999).
- Rixon, H. W. The small hydrophobic (SH) protein accumulates within lipid-raft structures of the Golgi complex during respiratory syncytial virus infection. J. Gen. Virol. 85, 1153-1165 (2004).
- Collins, P. L., Mottet, G. Membrane orientation and oligomerization of the small hydrophobic protein of human respiratory syncytial virus. J. Gen. Virol. 74, 1445-1450 (1993).
- Collins, P. L., Olmsted, R. A., Johnson, P. R. The small hydrophobic protein of human respiratory syncytial virus: comparison between antigenic subgroups A and B. J. Gen. Virol. 71, 1571-1576 (1990).
- Chen, M. D., Vazquez, M., Buonocore, L., Kahn, J. S. Conservation of the respiratory syncytial virus SH gene. J. Infect. Dis. 182, 1228-1233 (2000).
- Gan, S. W. The small hydrophobic protein of the human respiratory syncytial virus forms pentameric ion channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Gan, S. W., Ng, L., Xin, L., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein Sci. 17, 813-820 (2008).
- Li, Y. Inhibition of the Human Respiratory Syncytial Virus Small Hydrophobic Protein and Structural variations in a bicelle environment. J. Virol. 88 (22), 11899-914 (2014).
- Burgess, N. K., Stanley, A. M., Fleming, K. G. Determination of membrane protein molecular weights and association equilibrium constants using sedimentation equilibrium and sedimentation velocity. Meth. Cell. Biol. 84, 181-211 (2008).
- Cole, J. L., Lary, J. W., Moody, T. P., Laue, T. M. Analytical Ultracentrifugation: Sedimentation Velocity and Sedimentation Equilibrium. Meth. Cell. Biol. 84, 143-179 (2008).
- Fleming, K. G. Determination of membrane protein molecular weight using sedimentation equilibrium analytical ultracentrifugation. Curr. Protoc. Prot. Sci. 53, 17.12.11-17.12.13 (2008).
- . . An-50 Ti and An-60 Ti Analytical Rotor, Cells, and Counterbalance. , (2005).
- Mayer, G. Studying membrane proteins in detergent solution by analytical ultracentrifugation: Different methods for density matching. Prog. Colloid Polym. Sci. 113, 176-181 (1999).
- Laue, T. Ch. 20.3. Current Protocols in Protein Science. 20, 20.23.21-20.23.13 (2001).
- Gan, S. W. The Small Hydrophobic Protein Of The Human Respiratory Syncytial Virus Forms Pentameric Ion Channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Bevington, P. R., Robinson, D. K. . Data reduction and error analysis for the physical sciences. 336, (1969).
- Schuck, P., Radu, C. G., Ward, E. S. Sedimentation equilibrium analysis of recombinant mouse FcRn with murine IgG1. Molecular Immunology. 36, 1117-1125 (1999).
- Gan, S. W., Vararattanavech, A., Nordin, N., Eshaghi, S., Torres, J. A cost-effective method for simultaneous homo-oligomeric size determination and monodispersity conditions for membrane proteins. Anal. Biochem. 416, 100-106 (2011).
- Montserret, R. NMR structure and ion channel activity of the p7 protein from hepatitis C virus). J. Biol. Chem. 285, 31446-31461 (2010).
- Stouffer, A. L., DeGrado, W. F., Lear, J. D. Analytical Ultracentrifugation Studies of the Influenza M2 Homotetramerization Equilibrium in Detergent Solutions. Progr Colloid Polym Sci. 131, 108-115 (2006).
- Sorkin, A., von Zastrow, M. Signal transduction and endocytosis: Close encounters of many kinds. Nat. Rev. Mol. Cell Biol. 3, 600-614 (2002).
- Gan, S. W., Ng, L., Lin, X., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein science : a publication of the Protein Society. 17, 813-820 (2008).
Nachdrucke und Genehmigungen
Genehmigung beantragen, um den Text oder die Abbildungen dieses JoVE-Artikels zu verwenden
Genehmigung beantragenThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Alle Rechte vorbehalten