É necessária uma assinatura da JoVE para visualizar este conteúdo. Faça login ou comece sua avaliação gratuita.
Method Article
A sedimentação de equilíbrio de uma proteína de membrana de formação de oligómero pequeno: Efeito de Histidina A protonação em pentamérica Estabilidade
Neste Artigo
Resumo
Sedimentation equilibrium (SE) can be used to study protein-protein interactions in a physiological environment. This manuscript describes the use of this technique to determine the effect of pH on the stability of a homo-pentamer formed by the small hydrophobic (SH) protein encoded by the human syncytial respiratory virus (hRSV).
Resumo
Ultracentrifugação analítica (AUC) pode ser usado para estudar as interacções reversíveis entre macromoléculas sobre uma ampla gama de forças de interacção e em condições fisiológicas. Isso faz com que AUC um método de escolha para avaliar quantitativamente estequiometria e termodinâmica dos homo e hetero-associação que são transitórias e reversíveis nos processos bioquímicos. Na modalidade de equilíbrio de sedimentação (SE), um equilíbrio entre difusão e sedimentação fornece um perfil como uma função da distância radial que depende de um modelo de associação específica. Nisto, um protocolo detalhado SE é descrita para determinar a energia associação tamanho e monómero-monómero de uma pequena oligómero de proteína de membrana usando uma ultracentrífuga analítica. ES-AUC é livre de marcador, apenas com base em princípios físicos, e pode ser usado em ambas as proteínas solúveis da membrana e água. Um exemplo é mostrado deste último, o pequeno hidrofóbico (SH) nas proteínas do vírus sincicial respiratório humano (HRSV), Um polipeptídeo de ácido 65-amino com um único transmembranar (TM) de domínio helicoidal-α que forma canais de iões pentaméricos. Dados estrutural à base de RMN mostra que a proteína tem duas SH protonável resíduos de His no seu domínio transmembranar que são orientados de frente para o lúmen do canal. SE experimentos foram concebidos para determinar como pH afeta constante de associação e do tamanho da proteína oligomérica SH. Embora a forma pentamérica foi preservada em todos os casos, a sua constante de associação foi reduzida a pH baixo. Esses dados estão de acordo para uma dependência de pH semelhante ao observado para atividade canal SH, consistente com uma orientação do lúmen das duas Seus teor de resíduos no proteína SH. Este último pode experimentar repulsão eletrostática e estabilidade oligômero reduzida em pH baixo. Em resumo, este método é aplicável sempre que a informação quantitativa sobre proteína-proteína subtis alterações de associação em condições fisiológicas têm de ser medidos.
Introdução
Ultracentrifugação analítica 1-5 é um dos métodos mais importantes para estudar as interações de macromoléculas em condições fisiológicas, sendo acessível para ambas as interações fracas e fortes. O método é livre-label e utiliza a absorção de luz ou interferência, e até mesmo sistemas ópticos de fluorescência pode ser usado para acessar faixas de concentração ao longo de várias ordens de magnitude 6.
Este método é especialmente útil, pois a maioria dos processos bioquímicos dependem de interações reversíveis. A estequiometria e resistência destas interacções têm de ser quantitativamente caracterizada para compreender os processos biológicos, e um número de métodos existentes para este fim 7, 8. No entanto, as interacções transientes são difíceis de estudar 9.
A escolha de um método para caracterizar as interacções macromoleculares depende da sua natureza estática ou dinâmica. No primeiro caso, SEDIM entação velocidade (SV) é usada, em que a taxa de transporte radial é medida e complexos são fraccionados com base em diferenças na massa e forma flutuante.
Em contraste, as associações dinâmicas que são reversíveis na escala temporal da experiência não pode ser fisicamente separadas. Neste caso, auto ou hetero-interacções que as interacções não covalentes são em um equilíbrio que depende da concentração de proteína total. Estas interacções dinâmicas pode ser estudado por ambos equilíbrio de sedimentação (SE) e velocidade de sedimentação (SV) 10. No entanto, o primeiro método é mais simples de realizar e é descrito aqui. Em SE, a centrifugação é realizada a uma velocidade suficientemente baixa de modo a que se atingir um equilíbrio entre difusão e sedimentação. Neste ponto, o perfil de equilíbrio de um sinal óptico (UV-VIS), como uma função da distância radial, pode ser analisada utilizando modelos termodinâmicos pré-definido para associações 11.
ve_content "> No presente documento, um estudo de equilíbrio de sedimentação, é apresentado o auto-associação de uma proteína da membrana virai que forma canais de iões. Devido à sua hidrofobicidade, a experiência é executado em presença de detergentes, e no presente caso, a densidade de solvente tem de ser compensada com a do detergente. No entanto, o protocolo descrito seria idêntica no caso de uma proteína solúvel em água, excepto que sem correspondência de densidade solvente seria necessário.A proteína utilizada é codificado no vírus respiratório sincicial humano (hRSV), um pneumovirus envelopado na família paramyxoviridae que causa a doença do trato respiratório inferior em crianças, idosos e imunocomprometidos populações em todo o mundo 12. Até 64 milhões de casos notificados de infecção hRSV e 160.000 mortes ocorrem a cada ano.
O genoma hRSV transcreve 11 proteínas, incluindo as proteínas de membrana de três F, G, e pequeno hidrofóbico (SH). SH proteína está envolvidana patogênese da infecção pelo VSR. RSV sem o gene SH (RSVΔSH) era viável, causou a formação de sincícios e cresceu tão bem como o tipo selvagem (WT) vírus 13-16. No entanto, o vírus RSVΔSH replicado 10 vezes menos eficientemente do que o WT no tracto respiratório superior 15, 16. Além disso, o vírus RSVΔSH foi atenuada nos no rato vivo e modelos chimpanzé 13, 17.
A proteína é um SH (RSV subgrupo A) 64 ou 65 (RSV subgrupo B) aminoácidos de comprimento do tipo II de proteína integral da membrana que se acumula maioritariamente nas membranas do Golgi compartimento 18. SH proteína tem um único previu uma hélice transmembranar (TM) 19 de domínio que é altamente conservada 20,21. Os domínios extramembrane C- e N-terminais são orientados lumenally / extracelularmente e no citoplasma, respectivamente.
Ambos domínio TM sintético (resíduos 18-43) E SH proteína de comprimento completo foram mostrados para formar homopentamers em uma variedade de detergentes. A forma homopentameric é responsável pela atividade do canal em bicamadas lipídicas planas 22,23. A orientação correcta dos monómeros TM na bicamada lipídica foi primeiro determinada utilizando dicroismo local específico de infravermelhos 23, o qual mostrou His-22 para estar em uma luminal, perto de inter-helicoidal, a orientação. O domínio TM mesma orientação foi confirmada por estudos de RMN que reconstituídas a pentamérica um feixe helicoidal da proteína de comprimento completo em dodecylphosphocholine (DPC) 22 micelas. Neste modelo 'micela', um único domínio TM a- helicoidal foi flanqueada no terminal-N por uma a-hélice, e C-terminal por uma b-gancho de cabelo estendida. Os dois resíduos de proteína protonáveis SH, His-22 e His-51, estão localizados no domínio TM (orientada lumenally), e na extremidade do C-terminal extramembrane β hairpin (medida a partir da poro do canal), respectivamente. Em um ambie bicellarnment, no entanto, o TM α-hélice estende-se a His-51, e ambos os resíduos His são acessíveis para o lúmen do canal 24. A estrutura do canal adopta uma arquitectura em forma de funil 22, onde a região mais estreita (Ser-Cys-29 a 45) 22 está alinhada com cadeias laterais hidrofóbicas (Ile-32, Ile-36, Ile-40 e Leu-44), e Ile-36 define o ponto mais estreito no lúmen do canal. His-22 está localizado na maior abertura deste funil, enquanto que His-51 está na ponta de abertura mais pequena.
No presente documento, centrifugação analítica em equilíbrio de sedimentação de um modo foi usado para determinar se a sua protonação afecta a estabilidade do pentâmero da proteína de CS. Neste caso, SH proteína foi solubilizada em detergente de betaína-C14, que tem sido utilizado anteriormente para mostrar que as formas de proteínas pentaméricas oligómeros SH 22.
Protocolo
Este protocolo é baseado nos seguintes recursos, que devem ser encaminhados para mais detalhes e considerações especiais 3, 25-28.
1. Densidade de harmonização de micelas detergentes com 2 H2O
Nota: A densidade da solução tampão deve ser adaptada à densidade das micelas detergentes. Agentes de ajuste de densidade comuns incluem 2 H 2 O, 2 H 18 O, 2 H 2 O 18, glicerol e sacarose 29. H 2 O 18 tem a mesma densidade que 2 H 2 O e pode ser uma escolha melhor se deuteração de protões permutáveis na proteína não é desejado. Neste processo, a densidade de 3- -propanesulfonate (C14SB) detergente (N, N-dimethylmyristylammonio) em Tris 50 mM pH 7,3, NaCl 100 mM será combinado com 2 H 2 O. Como uma estimativa inicial as seguintes concentrações de2 H2O será utilizado: 10, 30, e 50% v / v.
1.1. Preparação de amostra
- Preparar as seguintes soluções de reserva e esterilizar filtro através de um filtro de seringa de 0,2 um: 50 ml de Tris 500 mM pH 7,3 e NaCl 1 M (tampão 10X); 1 mL C14SB 250 mM (solução detergente 50X).
- Prepare a 200 ul de solução de amostra através da mistura de 20 uL 10X tampão, 4 ul 50X solução detergente, 20 ul de H 2 O 2 (99,9%), e 156 ul de H2O desionizada Prepare também 200 jil de solução de referência, através da mistura de 20 uL solução tampão 10X, 20 ul de H 2 O 2 (99,9%), e 160 ul de H2O desionizada
- Repetir o passo 1.1.2 para os outros 2 H 2 O concentrações, isto é, 30% e 50%, ajustando as quantidades de H 2 O 2 e H 2 O apropriadamente.
1.2. Assembleia de 6 canais AUC células e as amostras de carregamento para dentro das células.
Nota: Existem dois tipos de células AUC, dependendo do método de carregamento de amostra. As células sem preenchimento externa tem de ser carregados antes da sua selagem da célula, ao passo que as células de enchimento externo pode ser carregado depois as células são selados. Montagem de uma célula de AUC externa de enchimento foi descrito anteriormente 3. Neste protocolo, a montagem de uma célula AUC 6 canais sem abastecimento externo é descrito. A principal diferença é que tem anéis de rosca de ambos os lados que têm de ser apertadas separadamente, e ele não precisa de tampões de habitação (Fig. 1). A diferença de etapas de montagem são destacadas a seguir.
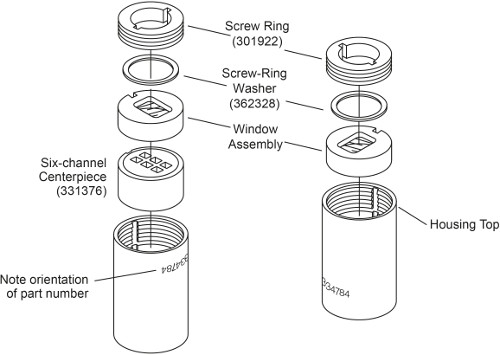
Figura 1. Vista explodida de uma célula AUC 6 canais sem abastecimento externo. Este valor foi modificado a partir do Beckman Coulter An-50 e An-Ti Ti 60 Analytical Rotor, Cells, e manual do usuário frontal.
- Preparar dois conjuntos de janelas para cada célula da AUC com janela de safira, em vez da janela de quartzo (Fig. 2). Coloque a junta da janela no suporte da janela. Ligeiramente dobrar o forro de janela e colocá-lo no suporte da janela de tal modo que a abertura é formada para a ranhura oposta titular janela. Coloque a janela de safira dentro do forro janela, alinhando a marca com a chaveta titular janela.
Nota:. A janela de quartzo é compressível e, portanto, irá produzir refracção mais luz a alta velocidade 28, 30 Portanto, para medições de interferência acima de 30000 rpm, como nesta experiência correspondente densidade, são utilizadas janelas de safira. Uma janela de safira, é mais pesado do que a janela de quartzo e tem um "X" para o lado gravado.
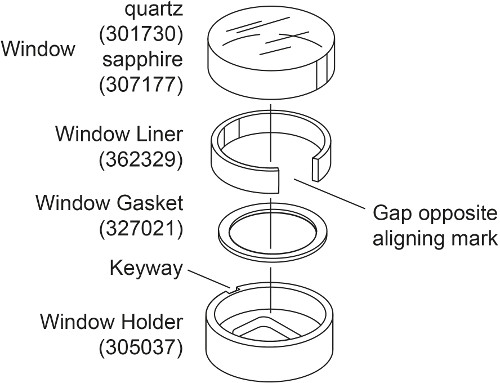
Figura 2. Vista explodida of o conjunto janela. Este valor foi modificado a partir do Beckman Coulter An-50 e An-Ti Ti 60 Analytical Rotor, de células e, manual do usuário Counterbalance
- Coloque o compartimento celular com o número de peça de cabeça para baixo. Com os keyways alinhadas com a chave da habitação, deslizar para dentro da caixa de células em primeiro lugar, uma peça central de 6 sector com o lado chanfrado para baixo, seguido por um conjunto de janela com a janela virada para baixo (Fig. 1, à esquerda).
- Cubra ligeiramente o roscas do anel de rosca e anel de parafuso arruela com spinkote. Coloque uma arruela anel de parafuso na parte superior do conjunto janela. Instale o anel de parafuso na habitação janela com a palavra "OUT" voltado para fora. Aperte o anel de parafuso utilizando a ferramenta de alinhamento celular.
- Usando a chave de torque, aperte o anel de parafuso para apenas 60 libras por polegada.
- Coloque a célula com o número de peça vertical e posicionadas em 12 horas. Load 120 ul de referência para as carreiras da esquerda e 110 ul de amostra para as carreiras da direita. Certifique-se de que cada amostra e referência está corretamente pareado.
Nota: O volume de amostra exacta não é crítica, mas a referência precisa de ter um pouco mais do que o volume da amostra (5-10 uL) de modo que o menisco da amostra sejam distintas. - Cuidadosamente deslize para dentro do conjunto habitacional celular uma janela com a janela virada para baixo (Fig. 1, à direita). Tome cuidado para não perturbar o celular excessivamente e derramar o conteúdo.
- Repita o passo 1.2. 3 e 1.2. 4, apertando o segundo anel de parafuso para 120 libras por polegada. Inverter o celular e voltar a apertar o primeiro anel de parafuso para 120 libras por polegada.
- Carregar as células dentro do rotor, instalar o rotor no centrifugador e instalar o monocromador de acordo com as instruções do fabricante 28.
Nota: Os detalhes deste step também pode ser encontrado nesta referência 3.
1.3. Configurando medição interferência
- Inicie o software de interface de usuário para o instrumento AUC e realizar a configuração de laser radial e calibração para cada célula a 3000 rpm, de acordo com as instruções do fabricante, que serão resumidos na etapa seguinte.
- 1.3.1.1 Depois de vácuo suficiente tiver sido atingido (<100 micra), executar a centrifugar a 3000 rpm. Visualize o padrão de interferência no software de interface de usuário e ajustar os parâmetros do laser para obter o mais alto contraste.
- Criar um novo ajustar o arquivo (File | New File) especificando "Equilibrium" e "interferência" de medição. Configurar um método de equilíbrio de sedimentação (botão "Method") para serem executadas em 45000 rpm ou a velocidade mais alta prevista para amostras de proteínas, o que for maior, com temperatura de execução a 20 ° C e coletar uma verificação a cada 15 minutos. Monitor equilíbrio andamento abrindo os arquivos de dados em HeteroAnalysis e selecionando a função "Match", depois de pelo menos 12 horas (aproximadamente durante a noite, Fig. 3).
Nota: a função similar também está disponível em SEDFIT (Opções | Carregando Opções | Teste abordagem para Equilibrium).

Figura 3. O resultado de função HeteroAnalysis Match. A função de fósforo pode ser usado para monitorar o progresso de equilíbrio, comparando RMSD entre as varreduras sucessivas e a última verificação. Este exemplo mostra para atingir o equilíbrio depois de 8 horas, tal como indicado por valores RMSD assimptóticas ao eixo-X.
1.4. Análise de dados
- Para cada conjunto de amostras, traçar o declive do perfil de distribuição radial contra o 2 H2O concentração.
Nota: A distribuição será um muito superficial exponencial que apabordagens linearidade. Intersecção do eixo-X corresponde à concentração de H 2 O 2 correspondentes. - Para resultados mais precisos, realizar o experimento em várias repetições. Alternativamente, repetir a experiência com uma faixa mais estreita de 2 H 2 O concentração.
2. Sedimentação equilíbrio do SH em micelas C14SB
2.1. Parâmetros de execução
- Calcular a densidade e viscosidade do tampão, a proteína volume específico parcial e velocidade de centrifugação usando SEDNTERP. Para calcular a densidade e viscosidade de buffer, selecione Compute na seção "Buffer de Dados Select" e insira os componentes do tampão em conformidade, incluindo a D 2 O concentração.
- 2.1.1.1 Para calcular proteína volume específico parcial, selecione Compute na secção de "V-bar" e digite a sequência de aminoácidos da proteína. Especifique o tamanho oligom�ica esperado mais alto em "Faça um oligômero deste monômero: N ="campo, neste caso N = 5. Calcule a velocidade ao entrar valores no campo RPM na janela principal até σ ≈ 1; esta é uma regra de ouro para garantir uma boa forma exponencial do perfil de distribuição radial 25.
Nota: Os valores calculados para esta experiência foi a seguinte: ρ = 1,03839 g / ml, 1,0267 η = cP = 0,7569 ml / g, 1 ω = 16000 rpm. - Calcule velocidades posteriores a seguir para garantir adequada diferença entre o perfil de distribuição em uma velocidade e no próximo 25.
Nota: Isto também pode ser feito a partir da função "Estimar equilíbrio rotor acelera função" em SEDFIT, que leva em conta a coluna de solução (volume de enchimento).
2.2. Preparações de amostras
- Prepare 1 ml de solução de referência com mM C14SB 5 e 32,3% 2 H 2 O, conforme determinado a partir de experimento correspondente densidade (seção 1), através da mistura de 100 l solutio 10X tampãon (passo 1.1.1), 20 ul de 50X solução detergente (passo 1.1.1), 323 ul de H 2 O 2 (99,9%) e 527 ul de H2O desionizada
- Dissolve-se liofilizado, SH péptidos purificados por HPLC (expressão e purificação descrito anteriormente 31) num solvente apropriado tal como metanol ou 50% v / v de acetonitrilo aquoso. Medir A280 dos péptidos dissolvidos em escala microlitro espectrofotómetro UV / Vis e alíquota para três amostras para dar um 280, 12 mm = 0,3, 0,5, e 0,8 (A280, 10 milímetros = 0,25, 0,417, e 0,67) quando diluída para cada 130 ul. Liofilizar as amostras durante a noite e ressuspender em 130 solução de referência ul (passo 2.2.1) para dar soluções de amostra.
Nota: proteína CS pode ser detectada a partir de UV / Vis a absorvância a 280 nm, porque contém resíduos de Tyr e Trp. Proteínas sem resíduos aromáticos podem ser detectados por marcá-los com um cromóforo adequado, utilizando um mutante contendo Trp, ou usando interferirnce em vez de medições de absorvância. - Siga as etapas na seção 1.2 para montar uma célula AUC 6 canais com janelas de quartzo. Carregar a amostra de maior concentração (280 A, 12 mm = 0,8) no canal mais próxima do centro do rotor e menor concentração da amostra (A280, 12 mm = 0,3) mais distante do centro do rotor.
2.3. Configurando medidas de absorbância
- Criar um novo ajustar o arquivo (File | New File) especificando "Equilibrium" e mensuração "Absorbance". Especificar 280 nm como o comprimento de onda do detector.
- Execute a calibração radial a 3000 rpm, verificando "calibração Radial antes da primeira varredura" em Opções de verificação, especificando a coleta de dados em baixa resolução, por exemplo, com tamanho do passo Radial = 0,01 centímetros, réplicas = 3 (baixa resolução, rápida), e executar uma única varredura . Após a verificação for concluída, desmarque a opção.
- Configurar um método de equilíbrio de sedimentação ("Method botão ") para ser executado na primeira velocidade calculada no passo 2.1.3, a 20 ° C, e recolher uma verificação a cada 30 minutos. Em "Detail" uns dos células, especifique a coleta de dados em baixa resolução como no passo 2.3.2. Monitorar o progresso de equilíbrio, abrindo os arquivos de dados em HeteroAnalysis e selecionando a função "Match", após pelo menos 18 horas (aproximadamente durante a noite, Fig. 4).
Nota: para atingir o equilíbrio podem demorar muito mais tempo para a primeira velocidade, enquanto velocidades subseqüentes levará menos tempo.
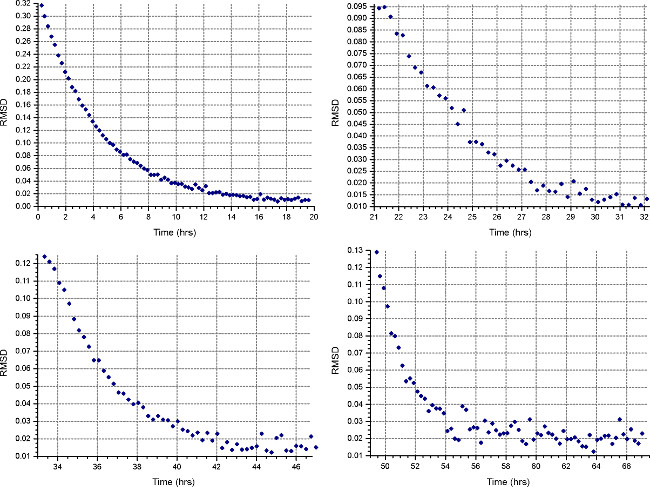
Figura 4. Os resultados de função HeteroAnalysis Match. A primeira e segunda velocidades (topo à esquerda e à direita) parecem ter atingido o equilíbrio, mas é melhor esperar mais algumas horas para ter certeza. Em comparação, a terceira e quarta velocidades (canto inferior esquerdo e direito) têm claramente atingido o equilíbrioem um tempo menor. Por favor, clique aqui para ver uma versão maior desta figura.
- Uma vez que o equilíbrio foi atingido, recolher uma única varredura em alta resolução, por exemplo, com o tamanho do passo Radial = 0,001 centímetros, réplicas = 10 (alta resolução, lento).
- Após a verificação for concluída, repita o passo 2.3.3 e 2.3.4 para a próxima velocidade.
- Opcionalmente, quando necessário o tempo para atingir o equilíbrio para cada velocidade é conhecida (ou calculada a partir da experiência), configurar o método de equilíbrio de sedimentação de verificação para incluir todas as velocidades calculadas no passo 2.1.3 e recolher uma varredura após o tempo de equilíbrio para cada velocidade. Neste caso, especifique a coleta de dados de alta resolução em "Detail" cada célula.
2.4. A análise dos dados em SEDFIT e SEDPHAT
Nota: Para mais detalhes e considerações em leitor de análise de dados é chamado to seguinte site: www.analyticalultracentrifugation.com.
- Abertas scans de alta resolução em SEDFIT (Dados | dados de equilíbrio de carga de sedimentação) e dividir os dados em três canais (estas correspondem a diferentes concentrações para cada amostra; Opções | Carregando Opções | Salve 6 canais de dados brutos em 3 subconjuntos).
- Re-abertos arquivos de dados que pertencem a uma mesma amostra e mesma concentração, mas diferentes velocidades em SEDFIT. Ajuste o menisco (linha vermelha vertical), fundo de células (linha vertical azul) e os limites de montagem (linhas verdes verticais), e exportar os dados para uso em SEDPHAT (Data | Exportação de dados para SEDPHAT). Entrada dos parâmetros calculados na etapa 2.1.1, assim como o tipo de rotor e tipo central, conforme solicitado. Repita este passo para cada amostra e concentração.
- Abra todos os dados da mesma amostra (todas as concentrações e velocidades) em SEDPHAT e preencha parâmetros da experiência; um exemplo é mostrado na Fig. 5.
Nota: quando D 2 O é adicionado no buffer, deut peração de protões permutáveis alterar significativamente o peso molecular da proteína, especialmente para as proteínas solúveis em água. As proteínas da membrana, especialmente as pequenas, como proteína SH, são menos afetados porque as regiões incorporado à membrana são protegidos de câmbio. Para corrigir isso, introduzir o "tampão D fração mol".
Nota: Nesta etapa é recomendado para salvar o conjunto de dados editados separadamente, selecionando menu Data | copiar todos os dados e salvar como Nova Configuração. - Selecione um modelo e preencha parâmetros globais para esse modelo.
Nota: A título de exemplo, o modelo "Monómero-n-Mer auto-associação" e os seus parâmetros são apresentados na Fig. 6.
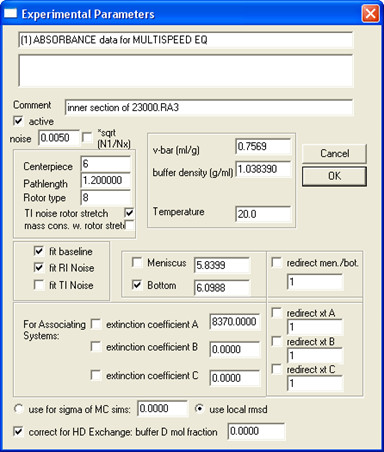
Figura 5. Um exemplo de como preencher parâmetros experimentais.
files / ftp_upload / 52404 / 52404fig6.jpg "/>
Figura 6. Um exemplo de como preencher parâmetros globais para o modelo monômero-n-Mer Auto-Associação.
- Executar um Fit global selecionando o menu Fit | Global de Fit e esperar até que os converge ajuste. Anote (ou tomar um screenshot) os resultados de ajuste, especialmente o qui-quadrado mundial reduziu e log K de valores. Extrato de outros dados, como dados de ajuste e resíduos de montagem a partir do menu de cópia e exibe | Mostrar informação termodinâmica.
- Retornar para parâmetros globais e verificar M (1) para acomodar o peso molecular monômero e repita o passo 2.4.5. Anote o peso molecular equipada e reduziu qui-quadrado global.
- Repita o passo 2.4.3 a 2.4.6 para cada modelo a ser testado e comparar a qualidade ajuste de cada modelo, comparando a redução do valor do qui-quadrado global, bem como resíduos de montagem.
Nota: As pequenas e aleatórias resíduos montagem geralmente indica um bom ajuste, e o modelo que se encaixa melhor fariatem a menor redução do qui-quadrado global. O peso molecular do monómero colocada e que o valor de qui-quadrado não devem diferir substancialmente da do peso molecular (teórico) fixo. - Calcule o intervalo de confiança para o registro obtido Ka por valor selecionando primeiro Estatísticas | Critical qui-quadrado para as projeções de superfície de erro e de introduzir o intervalo de confiança desejado. Em seguida, vá para Estatísticas | Gerar projeção superfície de erro 1-dimensional e desmarque log Ka em diálogo parâmetros globais para obter os valores de qui-quadrado para log Ka.
Nota: Os leitores são aconselhados a consultar as seguintes fontes (http://www.analyticalultracentrifugation.com/ sedphat / statistics.htm) para obter mais detalhes sobre o método 32, bem como ilustração deste método 33.
Resultados
O perfil radial de distribuição C14SB micelas detergentes em Tris 50 mM, NaCl 100 mM, pH 7,3, forma um exponenciais muito raso que poderiam ser ajustados a um modelo linear (Figura 7A). O declive desta distribuição está inversamente correlacionada com a concentração de D 2 O (Figura 7B). O ponto em que o declive é zero, ou seja, o correspondente D 2 O concentração, verificou-se ser de 32,3%.
Veja a Figura 7 ab...
Discussão
Este documento fornece um protocolo experimental para a preparação e análise de oligomerização de uma pequena proteína de membrana em detergente usando equilíbrio sedimentação da amostra. O protocolo descrito é igualmente válido para -e simpler- proteínas solúveis, como o passo correspondente da densidade não é necessária. Com efeito, o sistema é constituído por uma mistura de detergente e da proteína. Para realizar estudos de sedimentação, o detergente deve estar invisível para o campo gravitacion...
Divulgações
The authors have nothing to disclose.
Agradecimentos
This work has been funded by the National Research Foundation grant NRF-CRP4-2008-02 (J.T.) and Tier 1 grant RG 51/13.
Materiais
| Name | Company | Catalog Number | Comments |
| 3-(N,N-dimethylmyristylammonio)propanesulfonate | Sigma | T0807 | |
| Deuterium oxide 99.8% | Cambridge Isotope | DLM-4-99.8 | |
| An-50 Ti Rotor, Analytical, 8-Place | Beckman Coulter | 363782 | |
| An-60 Ti Rotor, Analytical, 4-Place | Beckman Coulter | 361964 | |
| Cell housing | Beckman Coulter | 334784 | |
| 12 mm six-channel centerpiece, epon charcoal-filled | Beckman Coulter | 331376 | |
| Window holder | Beckman Coulter | 305037 | |
| Window gasket | Beckman Coulter | 327021 | |
| Window liner | Beckman Coulter | 362329 | |
| Sapphire window | Beckman Coulter | 307177 | |
| Quartz window | Beckman Coulter | 301730 | |
| Screw-ring washer | Beckman Coulter | 362328 | |
| Screw ring | Beckman Coulter | 301922 | |
| Spinkote | Beckman Coulter | 306812 | |
| Torque stand assembly | Beckman Coulter | 361318 | |
| Counterbalance | Beckman Coulter | 360219 | |
| Cell alignment tool | Beckman Coulter | 362340 | |
| SEDNTERP | http://bitcwiki.sr.unh.edu/index.php/Main_Page | ||
| HeteroAnalysis | http://www.biotech.uconn.edu/auf/?i=aufftp | ||
| SEDFIT | http://www.analyticalultracentrifugation .com/sedfit.htm | ||
| SEDPHAT | http://www.analyticalultracentrifugation .com/sedphat/default.htm |
Referências
- Laue, T. M., Stafford, W. F. Modern applications of analytical ultracentrifugation. Annu. Rev. Biophys. Biomol. Struct. 28, 75-100 (1999).
- Lebowitz, J., Lewis, M. S., Schuck, P. Modern analytical ultracentrifugation in protein science: A tutorial review. Protein Sci. 11, 2067-2079 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. . Assembly, Loading, and Alignment of an Analytical Ultracentrifuge Sample Cell. , e1530 (2009).
- Rivas, G., Stafford, W., Minton, A. P. Characterization of heterologous protein-protein interactions using analytical ultracentrifugation. Methods-a Companion to Methods in Enzymology. 19, 194-212 (1999).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for association and assembly the study of protein. Curr. Opin. Chem. Biol. 10, 430-436 (2006).
- MacGregor, I. K., Anderson, A. L., Laue, T. M. Fluorescence detection for the XLI analytical ultracentrifuge. Biophys. Chem. 108, 165-185 (2004).
- Phizicky, E. M., Fields, S. Protein-Protein Interactions - Methods for Detection and Analysis. Microbiol. Rev. 59, 94-123 (1995).
- Alexandrov, A. A facile method for high-throughput co-expression of protein pairs. Mol. Cell. Proteomics. 3, 934-938 (2004).
- Nooren, I. M. A., Thornton, J. M. Structural characterisation and functional significance of transient protein-protein interactions. J. Mol. Biol. 325, 991-1018 (2003).
- Ebel, C. Sedimentation velocity to characterize surfactants and solubilized membrane proteins. Methods. 54, 56-66 (2011).
- Minton, A. P. Quantitative characterization of reversible macromolecular associations via sedimentation equilibrium: an introduction. Exp. Mol. Med. 32, 1-5 (2000).
- Dowell, S. F. Respiratory syncytial virus is an important cause of community-acquired lower respiratory infection among hospitalized adults. J. Infect. Dis. 174, 456-462 (1996).
- Bukreyev, A., Whitehead, S. S., Murphy, B. R., Collins, P. L. Recombinant respiratory syncytial virus from which the entire SH gene has been deleted grows efficiently in cell culture and exhibits site-specific attenuation in the respiratory tract of the mouse. J. Virol. 71, 8973-8982 (1997).
- Fuentes, S., Tran, K. C., Luthra, P., Teng, M. N., He, B. Function of the respiratory syncytial virus small hydrophobic protein. J. Virol. 81, 8361-8366 (2007).
- Jin, H. Recombinant respiratory syncytial viruses with deletions in the NS1, NS2, SH, and M2-2 genes are attenuated in vitro and in vivo. Virology. 273, 210-218 (2000).
- Karron, R. A. Respiratory syncytial virus (RSV) SH and G proteins are not essential for viral replication in vitro: clinical evaluation and molecular characterization of a cold-passaged, attenuated RSV subgroup B. Proc. Nat. Acad. Sci. USA. 94, 13961-13966 (1997).
- Whitehead, S. S. Recombinant respiratory syncytial virus bearing a deletion of either the NS2 or SH gene is attenuated in chimpanzees. J. Virol. 73, 3438-3442 (1999).
- Rixon, H. W. The small hydrophobic (SH) protein accumulates within lipid-raft structures of the Golgi complex during respiratory syncytial virus infection. J. Gen. Virol. 85, 1153-1165 (2004).
- Collins, P. L., Mottet, G. Membrane orientation and oligomerization of the small hydrophobic protein of human respiratory syncytial virus. J. Gen. Virol. 74, 1445-1450 (1993).
- Collins, P. L., Olmsted, R. A., Johnson, P. R. The small hydrophobic protein of human respiratory syncytial virus: comparison between antigenic subgroups A and B. J. Gen. Virol. 71, 1571-1576 (1990).
- Chen, M. D., Vazquez, M., Buonocore, L., Kahn, J. S. Conservation of the respiratory syncytial virus SH gene. J. Infect. Dis. 182, 1228-1233 (2000).
- Gan, S. W. The small hydrophobic protein of the human respiratory syncytial virus forms pentameric ion channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Gan, S. W., Ng, L., Xin, L., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein Sci. 17, 813-820 (2008).
- Li, Y. Inhibition of the Human Respiratory Syncytial Virus Small Hydrophobic Protein and Structural variations in a bicelle environment. J. Virol. 88 (22), 11899-914 (2014).
- Burgess, N. K., Stanley, A. M., Fleming, K. G. Determination of membrane protein molecular weights and association equilibrium constants using sedimentation equilibrium and sedimentation velocity. Meth. Cell. Biol. 84, 181-211 (2008).
- Cole, J. L., Lary, J. W., Moody, T. P., Laue, T. M. Analytical Ultracentrifugation: Sedimentation Velocity and Sedimentation Equilibrium. Meth. Cell. Biol. 84, 143-179 (2008).
- Fleming, K. G. Determination of membrane protein molecular weight using sedimentation equilibrium analytical ultracentrifugation. Curr. Protoc. Prot. Sci. 53, 17.12.11-17.12.13 (2008).
- . . An-50 Ti and An-60 Ti Analytical Rotor, Cells, and Counterbalance. , (2005).
- Mayer, G. Studying membrane proteins in detergent solution by analytical ultracentrifugation: Different methods for density matching. Prog. Colloid Polym. Sci. 113, 176-181 (1999).
- Laue, T. Ch. 20.3. Current Protocols in Protein Science. 20, 20.23.21-20.23.13 (2001).
- Gan, S. W. The Small Hydrophobic Protein Of The Human Respiratory Syncytial Virus Forms Pentameric Ion Channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Bevington, P. R., Robinson, D. K. . Data reduction and error analysis for the physical sciences. 336, (1969).
- Schuck, P., Radu, C. G., Ward, E. S. Sedimentation equilibrium analysis of recombinant mouse FcRn with murine IgG1. Molecular Immunology. 36, 1117-1125 (1999).
- Gan, S. W., Vararattanavech, A., Nordin, N., Eshaghi, S., Torres, J. A cost-effective method for simultaneous homo-oligomeric size determination and monodispersity conditions for membrane proteins. Anal. Biochem. 416, 100-106 (2011).
- Montserret, R. NMR structure and ion channel activity of the p7 protein from hepatitis C virus). J. Biol. Chem. 285, 31446-31461 (2010).
- Stouffer, A. L., DeGrado, W. F., Lear, J. D. Analytical Ultracentrifugation Studies of the Influenza M2 Homotetramerization Equilibrium in Detergent Solutions. Progr Colloid Polym Sci. 131, 108-115 (2006).
- Sorkin, A., von Zastrow, M. Signal transduction and endocytosis: Close encounters of many kinds. Nat. Rev. Mol. Cell Biol. 3, 600-614 (2002).
- Gan, S. W., Ng, L., Lin, X., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein science : a publication of the Protein Society. 17, 813-820 (2008).
Reimpressões e Permissões
Solicitar permissão para reutilizar o texto ou figuras deste artigo JoVE
Solicitar PermissãoThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. Todos os direitos reservados