JoVE 비디오를 활용하시려면 도서관을 통한 기관 구독이 필요합니다. 전체 비디오를 보시려면 로그인하거나 무료 트라이얼을 시작하세요.
Method Article
작은 올리고머 형성 막 단백질의 침전 평형 : 펜타 머 안정성에 히스티딘 양성자의 효과
요약
Sedimentation equilibrium (SE) can be used to study protein-protein interactions in a physiological environment. This manuscript describes the use of this technique to determine the effect of pH on the stability of a homo-pentamer formed by the small hydrophobic (SH) protein encoded by the human syncytial respiratory virus (hRSV).
초록
분석 용 초 원심 분리 (AUC)은 상호 작용의 세기의 광범위한 생리 학적 조건 하에서 고분자 사이의 가역적 인 상호 작용을 연구하는데 사용될 수있다. 이 AUC를 정량적으로 화학 양론과 호모 생화학 적 과정에 과도 되돌릴 수 있습니다 헤테로 협회의 열역학을 평가하기 위해 선택하는 방법을합니다. 침강 평형 양상 (SE)에서, 확산 침강 간의 균형은 특정 관계 모델에 따라 반경 방향 거리의 함수로서 프로파일을 제공한다. 여기서, SE 상세한 프로토콜 분석 초 원심 분리기를 사용하여 작은 막 단백질 올리고머의 크기와 연관 단량체 단량체 에너지를 결정하기 위해 설명된다. AUC-ES는 단지 물리적 원리에 기초하여, 라벨없는이며, 모두 수용성 단백질 및 막에 사용될 수있다. 예는 후자의 도시, 인간 호흡기 합 포체 바이러스의 작은 소수성 (SH) 단백질 (hRSV), 펜타 머 이온 채널을 형성하는 단일 나선 α 횡단 (TM) 도메인과 65 아미노산 폴리펩티드. 기반 구조 NMR 데이터는 SH 단백질이 채널의 루멘에 대향 배향의 막 횡단 도메인의 두 protonatable 그의 잔기를 갖는 것을 보여준다. SE 실험은 pH가 협회 상수와 SH 단백질의 올리고머 크기에 영향을 미치는 방법을 결정하기 위해 설계되었습니다. 펜타 머 형태는 모든 경우에 유지되었지만, 그 연관 상수는 낮은 pH에서 환원시켰다. 이러한 데이터는 SH 단백질의 두 그의 잔류의 내강 방향과 일치 SH 채널 활동에 대한 관찰과 유사한의 pH 의존성과 일치한다. 후자는 낮은 pH에서 정전 기적 반발력 감소 올리고머 안정성이 발생할 수 있습니다. 생리 학적 조건의 미묘한 단백질 - 단백질 연관 변경 정량적 정보를 측정 할 수있을 때마다 요약하면,이 방법은 적용 가능하다.
서문
분석 용 초 원심 1-5, 생리 학적 조건하에 거대 분자의 상호 작용을 연구 강약의 상호 작용 모두에 액세스 가능한 가장 중요한 방법 중 하나이다. 방법은 라벨없는이고 광 흡수 또는 간섭을 사용하며, 심지어 형광 광학 시스템 (6)의 크기는 몇 배 이상 농도 범위를 액세스하기 위해 사용될 수있다.
대부분의 생화학 적 과정이 가역적 인 상호 작용에 의존하기 때문에이 방법은 특히 유용합니다. 이러한 상호 작용의 강도 화학량 정량적 생물학적 과정을 이해하는 것을 특징으로 할 수 있고, 다수의 방법이 목적 7,8 존재한다. 그러나, 과도 상호 작용을 연구 9 어렵다.
거대 분자의 상호 작용을 특성화하는 방법의 선택은 정적 또는 동적 인 특성에 의존한다. 첫 번째 경우, sedim 정해져있는 속도 (SV)는 방사상 운송 속도가 측정되고, 복합체는 부력 질량 및 형상의 차이에 기초하여 분획 화되는 경우, 사용된다.
반면에, 실험의 시간 스케일에 가역적 동적 연결은 물리적으로 분리 될 수 없다. 이 경우, 자기 또는 비 - 공유 상호 작용으로 이어지는 헤테로 상호 작용은 총 단백질 농도에 의존 평형에있다. 이러한 동적 상호 작용 침강 평형 (SE) 및 침강 속도 (SV) (10) 모두 공부하실 수 있습니다. 그러나, 첫 번째 방법을 수행하는 단순하고 여기에 설명된다. 평형 확산 침강 사이 도달되도록 SE에서 원심 충분히 낮은 속도로 수행된다. 이 시점에서, 반경 방향 거리의 함수로서 광 신호 (UV-VIS)의 평형 프로필은, 연관 (11)에 대해 미리 설정된 열역학적 모델을 사용하여 분석 될 수있다.
ve_content "> 본 논문에서는 침강 평형 연구 때문에 그 소수성. 이온 채널을 형성 바이러스 막 단백질의 자기 연관으로 제시되고, 실험은 세제의 존재 하에서 실행하고,이 때의 밀도이다 용매 세제와 정합되어야한다. 그러나,이 프로토콜에는 용매 농도 정합이 요구되지 것 이외에는, 수용성 단백질의 경우에서와 동일한 기재.사용되는 단백질은 인간의 호흡기 세포 융합 바이러스 (hRSV),하기도 유아 질환, 노인과 면역 인구 전세계 12 원인 paramyxoviridae 제품군의 포락선 pneumovirus 인코딩됩니다. hRSV 감염의 최대 64,000,000보고 된 사례와 160,000 사망 매년 발생합니다.
게놈은 hRSV 세 막 단백질 F, G, 및 작은 소수성 (SH) 단백질을 포함한 11, 사본을 만듭니다. SH 단백질이 관여RSV 감염의 발병. SH 유전자 (RSVΔSH)를 결여 RSV는 실용적이었다 syncytia의 형성을 야기하고, 야생형 (WT) 바이러스뿐만 아니라 13-16 자랐다. 그러나 RSVΔSH 바이러스는 상부 호흡기 (15, 16)에보다 덜 효율적 WT 1/10 복제. 또한, RSVΔSH 바이러스는 생체 내 마우스와 침팬지 모델 13, 17에서 감쇠했다.
SH 단백질 64 (RSV 하위 그룹 A) 또는 65 (RSV의 서브 그룹 B) 아미노산으로 유형 골지 실 (18)의 막에 주로 축적 II 세포막 단백질이다. SH 단백질은 하나의 높은 (20, 21)를 보존하는 나선 막 횡단 (TM) 도메인 (19) 예측이있다. C- 및 N- 말단 extramembrane 도메인은 lumenally / 세포 외 및 세포질, 각각 지향하고 있습니다.
모두 합성 TM 도메인 (잔류 물 (18)-43)과 전체 길이 SH 단백질은 세제의 다양한 homopentamers를 형성하는 것으로 나타났다. homopentameric 형태는 평면 22,23 지질 이중층에서 채널 활동에 대한 책임이있다. 지질 이중층의 TM 단량체의 올바른 방향은 먼저 그의-22는, 내강에 가까운 방향, 간 나선형의 제품에 보였다 사이트 특정 적외선 이색 (23)를 사용하여 측정 하였다. 동일한 TM 도메인 배향 (DPC)에 dodecylphosphocholine 펜타 머에게 전장 단백질의 나선 다발 (22)을 재구성 미셀 NMR 연구에 의해 확인 하였다. 이 '미셀'모델에서, 단일의 헬리컬 A- TM 도메인 확장 헤어핀 B-C에 의해 말단 나선 의해 N 말단 어귀 하였다. SH 단백질의 두 protonatable 잔류, 그의-22과 그분의-51, TM 도메인 (lumenally 방향)에 위치하며, extramembrane의 C- 말단 β 머리핀의 끝에서 각각, (지금까지 채널 기공에서). bicellar ENVIRO에서nment 그러나, TM의 α 나선 그의-51까지 연장하고, 그 잔류 물은 두 채널 (24)의 내강에 접근 할 수있다. 22 소수성 사이드 체인 (일드-32, 일드-36, 일드-40와 레우-44), 그리고 줄 지어 채널 구조는 깔때기 모양의 구조 (22), 좁은 지역 채택 (의 Cys-45 빼앗아-29)은 일드-36는 채널 루멘의 좁은 지점을 정의합니다. 그의-51가 가장 작은 개구 선단에있는 반면, 그의-22은,이 깔때기의 가장 큰 개방에 있습니다.
본 논문에서는 침강 평형 모드에서 원심 분리 분석이 그의 양성자 SH 량체 단백질의 안정성에 영향을 미치는지를 결정하기 위해 사용되어왔다. 이 경우, SH 단백질은 그 단백질 SH 형 펜타 머 올리고머 22 보여 이전에 사용 된-C14 베타 인 세제, 가용화 하였다.
프로토콜
이 프로토콜은 자세한 내용과 특별한 고려 사항 3, 25 ~ 28에 대해 언급 할 수있는 다음 자원을 기반으로합니다.
H 2 O 2와 세제 미셀 1. 밀도 매칭
주 : 완충액의 농도는 세제 미셀 농도에 일치 될 필요가있다. 일반 밀도 조정 에이전트는 2 H 2 O, H 2 O (18), 2 H 2 O (18), 글리세롤과 자당 (29)를 포함한다. H 2 O (18)는 H 2 O 2와 같은 농도를 가지며, 교환 가능한 양성자 단백질의 중수 소화되지 않게 할 필요가있는 경우는 더 나은 선택 일 수있다. 이 절차에서, 50mM 트리스 pH가 7.3에서 3- (N, N-dimethylmyristylammonio) -propanesulfonate (C14SB)의 세제 농도는 100 mM의 NaCl을 2 H 2 O.과 일치 될 것이다 초기 추측으로 다음과 같은 농도H 2 O 2를 사용한다 : 10, 30, 50 % V / V.
1.1. 샘플 준비
- 원액을 다음과 필터가 0.2㎛ 시린지 필터를 통해 살균 준비 : 50 ㎖의 500 mM 트리스 pH를 7.3 M의 NaCl 및 1 (10X 완충 용액); 1 mL를 250 mM의 C14SB (50X 세제 용액).
- 20 ㎕의 10X 완충액 4 ㎕의 50X 세제 용액 20 μL H 2 O 2 (99.9 %), 156 μL를 혼합하여 시료 용액 200㎕를 준비하여 H 2 O를 탈 20 μL H 2 O 2 (99.9 %), 160 μL, 20 μL를 10X 완충 용액을 혼합함으로써 용액의 기준도 200 μl를 준비한 H 2 O를 탈
- 다른 2 H 2 O 농도 단계를 반복 1.1.2, 적절하게 2 H 2 O와 H 2 O 양을 조절, 30 % 및 50 % 즉.
1.2. 6 채널의 어셈블리UC 세포로 세포 및 샘플 로딩.
주의 : 샘플 로딩 방법에 따라 AUC 셀의 두 종류가있다. 셀을 밀봉 한 후 세포를로드 할 수있는 반면, 외부 입력 외부 충전없이 세포는 세포를 밀봉하기 전에 로딩되어야한다. 외부 입력 AUC 셀의 조립 이전에 3 기술되었다. 이 프로토콜에서, 외부 충전없이 6 채널 AUC 전지의 조립을 설명한다. 주요 차이점은 별도로 체결해야 양쪽 나사 링이 있는지이며 (도. 1) 플러그 하우징을 필요로하지 않는다. 조립 순서의 차이는 아래에 강조 표시됩니다.
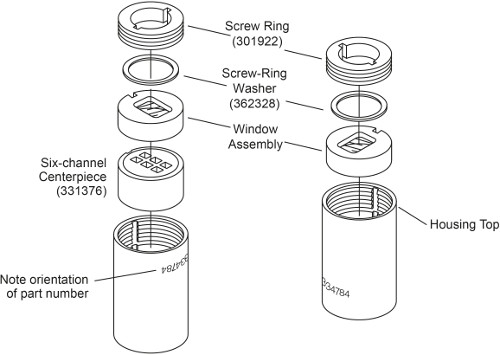
그림 1. 외부 충전없이 6 채널 AUC 셀의보기를 폭발했다.이 수치는 콜터-50 TI의 수정 및-60 티 Analyti되었습니다CAL 회, 세포 및 평형 사용자 설명서.
- 사파이어 창 대신 석영 창 (그림. 2) 각 AUC 셀에 대해 두 개의 창 어셈블리를 준비합니다. 창 홀더에 창 가스켓을 놓습니다. 약간 윈도우를 구부리고 라이너 갭 창 홀더 키홈 향하여 형성되도록 창 홀더에 배치. 창 홀더 열쇠 구멍에 마크를 정렬, 윈도우 라이너 내부의 사파이어 창을 놓습니다.
. 참고 사항 :이 밀도 일치하는 실험에서와 같이 30,000 rpm으로 위의 간섭 측정을위한 따라서 고속 (28), (30)에 더 많은 빛을 굴절을 생성 할 것이다, 따라서 석영 창을 압축하고, 사파이어 윈도우가 사용된다. 사파이어 창이 석영 창보다 무거운 그면에 에칭 "X"가.
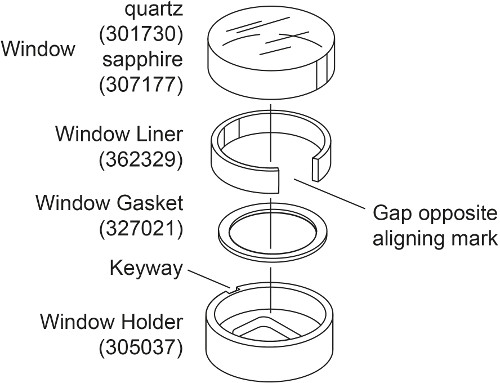
그림 2. 분해도 오F 창 조합.이 그림은 콜터-50 Ti 및-60 티타늄 분석 로터, 세포 및 평형 사용자 설명서에서 수정되었습니다
- 부품 번호 거꾸로와 셀 하우징을 놓습니다. 주택 키를 사용하여 정렬 된 열쇠 구멍으로, (왼쪽 그림. 1) 아래로 향하게 창 하나의 창 어셈블리 다음, 먼저 세포 하우징 아래로 경 사진면을 6 섹터 중심에 밀어 넣습니다.
- 가볍게 코트 spinkote로 나사 링 스레드와 나사 링 와셔. 윈도우 어셈블리의 상단에 나사 링 와셔를 놓습니다. 외부 직면하고있는 단어 "OUT"와 창 하우징에 나사 링을 설치합니다. 셀 정렬 도구를 사용하여 나사 링을 손으로 조.
- 토크 렌치를 사용하여 60 인치 - 파운드 나사 반지를 조입니다.
- 부품 번호 똑바로 세워 12 정오에 위치와 셀을 놓습니다. L왼쪽 행에 OAD 120 ㎕를 참조하고 오른쪽 행에 110 ㎕의 샘플. 각 샘플과 참조가 제대로 결합되어 있는지 확인합니다.
참고 : 정확한 샘플 량은 중요하지 않지만 샘플 메 니스 커스가 구별 될 수 있도록 참조 샘플 (5-10 μL)보다 약간 더 많은 양이 필요하다. - 조심스럽게 창 (그림. 1, 오른쪽) 아래로 향하도록 전지 하우징 하나의 창 조립품에 밀어 넣습니다. 지나치게 셀을 방해하고 내용을 흘리지 않도록주의하십시오.
- 단계를 반복 1.2. 3 1.2. 4, 120 인치 - 파운드 두 번째 나사 링을 체결. 세포 전환, 120 인치 - 파운드로 제 1 나사 링을 다시 조입니다.
- 회 전자에 세포를로드 원심 분리기 회 전자를 설치하고 제조업체의 지침 (28)에 따라 단색을 설치합니다.
참고 : 본 보안 목표 명세서에 대한 세부 사항을EP도이 문헌 3에서 찾을 수있다.
1.3. 간섭 측정 설정
- 간단히 다음 단계로 요약 될 제조업체의 지시에 따라, AUC 기기 용 사용자 인터페이스 소프트웨어를 시작하고, 3000 rpm에서 각 셀에 대해 사용 된 레이저 및 반경 보정을 수행.
- 충분한 진공 후 1.3.1.1은 (<100 미크론), 3000 rpm에서 원심 분리기를 실행에 도달했습니다. 사용자 인터페이스 소프트웨어에서의 간섭 패턴을 미리 높은 콘트라스트를 얻기 위해 레이저 파라미터를 조정한다.
- "균형"과 "간섭"측정을 지정 | (새 파일 파일) 새로운 설정 파일을 만듭니다. 침강 평형 방법 ( "방법"버튼) 20 ° C에서 실행 온도, 45,000 RPM 이상 중 단백질 시료에 대한 예상 최고 속도로 실행 한 검사를 수집하는 15 분 간격을 설정합니다. 과정 제어HeteroAnalysis 상기 데이터의 파일을 열어 12 시간 이상 이후에 "매치"기능을 선택하여 연구 진행 평형 (약 밤새도. 3).
참고 : 유사한 기능도 SEDFIT (옵션 |로드 옵션 | 평형에 시험 접근)에서 사용할 수 있습니다.

도 HeteroAnalysis match 함수 3. 결과. 대전 함수는 연속적인 검사 및 최종 검사 사이 RMSD 비교함으로써 평형 진행 상황을 모니터링 할 수있다. X 축에 점근 RMSD 값으로 표시된 바와 같이이 예제는 8 시간 후에 평형의 달성을 보여줍니다.
1.4. 데이터 분석
- 각 샘플 세트에 대해, H 2 O 2 농도에 대해서 방사상 분포 프로파일의 기울기를 플롯.
참고 : 분포가 매우 얕은 지수가 AP 될 것입니다proaches 선형성. X 축 절편은 일치하는 2 H 2 O 농도에 해당합니다. - 보다 정확한 결과를 위해, 몇 가지 반복 실험에 실험을 수행합니다. 대안으로, H 2 O 2 농도의 범위가 좁은 실험을 반복한다.
C14SB 미셀의 SH 2. 침전 평형
2.1. 실행 매개 변수
- SEDNTERP 버퍼를 사용하여 밀도 및 점도, 단백질 부분 체적 원심 분리 속도를 계산한다. D 2 O 농도를 포함하여, "버퍼 데이터 선택"절에서 버퍼 밀도와 점도를 선택 계산을 계산하고 이에 따라 버퍼 구성 요소를 입력합니다.
- 2.1.1.1는 "V-바"절에서 단백질 부분 특정 볼륨, 선택 계산을 계산하고 단백질의 아미노산 서열을 입력합니다. "N =이 단량체 올리고머를 만들기"에서 가장 높은 예상 올리고머 크기를 지정합니다필드,이 경우 N = 5. 메인 창에서 RPM 필드에 값을 입력하여 속도를 계산까지 σ ≈ 1; 이것은 방사상 분포 프로파일 (25)의 형상을 양호한 지수 보장 경험칙이다.
참고 : 다음과 같이이 실험에 대해 계산 된 값이었다 : ρ = 1.03839 g / ㎖, η = 1.0267 cP 인 = 0.7569 ㎖ / g, ω 1 = 16000 RPM. - 하나의 속도와 다음 25 배포 프로파일 사이에 충분한 차이를 확인하기 위해 수행 이후의 속도를 계산합니다.
참고 :이 또한 함수에서 수행 할 수 있습니다 계정에 솔루션 열 (충전 볼륨) 소요 SEDFIT에서 "평형 로터 기능을 속도 추정".
2.2. 샘플 준비
- 5 mM의 C14SB 1 ㎖의 표준 용액 및 32.3 % 2 H를 준비 2 O 100 μL 10X 버퍼 solutio을 혼합하여 밀도 매칭 실험 (섹션 1)에서 결정N (1.1.1), 20 μL 50X 세제 용액 (1.1.1), 323 μL 2 H 2 O (99.9 %)과 527 μl의 H 2 O를 탈
- 메탄올 또는 50 % v / V를 아세토 니트릴 등의 적절한 용매에 동결 건조, HPLC 정제 SH 펩티드 (이전 31 설명 발현 및 정제)를 용해. 각 희석하여 280을 수득 세 샘플 마이크로 리터 규모 UV / 비스 분광 광도계 분취 액에 용해 된 펩타이드의 A280을 측정, 12mm = 0.3, 0.5, 0.8 (280, 10mm = 0.25, 0.417, 0.67) 130 μL. 밤새 동결 건조 샘플을 재현 탁하고 130 μl의 표준 용액 (단계 2.2.1)에서 샘플 용액을 얻었다.
주 : Trp의 티르 잔류 물을 포함하고 있기 때문에 SH 단백질 280 nm에서 UV / 마주 흡광도에서 검출 될 수있다. 방향족 잔기가없는 단백질, 또는 방해하여, 적합한 발색단로 태깅 함유 Trp의 돌연변이를 사용하여 검출 될 수있다대신 흡광도의 후부 측정. - 석영 창문이있는 6 채널 AUC 셀을 조립 1.2 절의 단계를 따르십시오. 가장 가까운 회 센터에서 회 센터와 가장 낮은 농도 샘플 (280, 12mm = 0.3) 멀리로 (280, 12mm = 0.8) 채널을 높은 농도 샘플을로드합니다.
2.3. 흡광도 측정 설정
- "균형"과 "흡광도"측정을 지정 | (새 파일 파일) 새로운 설정 파일을 만듭니다. 검출기 파장 280 nm의를 지정합니다.
- 단일 스캔을 방사형 스텝 크기 = 0.01 cm, 복제합니다 = 3 (낮은 해상도, 빠른)와 예를 들어, 낮은 해상도에서 데이터 수집을 지정, 검색 옵션에서 "첫 번째 스캔하기 전에 레이디 얼 교정"을 확인하고 실행하여 3000 rpm에서 반경 보정을 수행합니다 . 스캔이 완료된 후에, 옵션 선택을 해제.
- "(운전 방식을 침강 평형 방법을 설정"버튼) D는 20 ° C에서, 단계 2.1.3에서 계산 된 최초의 속도로 실행하고, 1 스캔 30 분마다 수집합니다. 각 세포 ' "세부 사항"에서, 단계 2.3.2에서와 같이 낮은 해상도에서 데이터 수집을 지정합니다. 적어도 18 시간 후에 "일치"기능을 HeteroAnalysis 상기 데이터의 파일을 열어 선택하여 평형 과정을 모니터 (약 밤새도. 4).
주 : 이후의 속도는 시간이 덜 걸릴 것입니다 반면 균형의 달성은 제 속도를 실질적으로 긴 시간이 걸릴 수 있습니다.
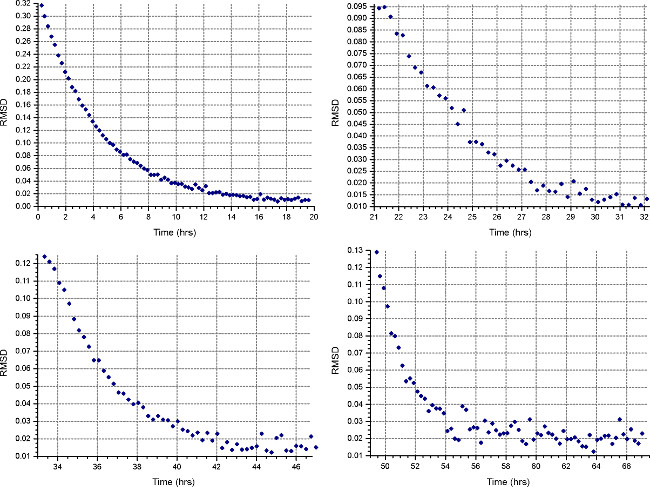
HeteroAnalysis match 함수에서 그림 4. 결과. 제 1 및 제 2 속도는 (상단 왼쪽 및 오른쪽)이 평형에 도달 한 것으로 보인다,하지만 확실하게 몇 시간을 더 기다려야하는 것이 좋습니다. 비교에서, 세 번째와 네 번째 속도 (하단 왼쪽 및 오른쪽)을 명확하게 평형에 도달짧은 시간에. 이 그림의 더 큰 버전을 보려면 여기를 클릭하십시오.
- 평형에 도달하면, 레이디 얼 스텝 크기 = 0.001 cm, 복제합니다 = 10 (높은 해상도, 느린)와 높은 해상도, 예에서 단일 스캔을 수집합니다.
- 스캔이 완료되면, 다음 단계 속도 2.3.4 및 2.3.3을 반복.
- 시간이 각각의 속도에 대한 평형 알려진 (계산 또는 경험)입니다 도달하기 위해 필요한 경우 선택적으로, 각 평형 시간 이후 1 스캔을 수집하는 단계 및 2.1.3에서 계산 된 모든 속도를 포함하는 침강 평형 방법 스캔을 설정 속도. 이 경우, 각 셀 ''정보 "의 고해상도 데이터 콜렉션을 지정.
2.4. SEDFIT 및 SEDPHAT에서 데이터 분석
참고 : 자세한 내용 및 데이터 분석 리더의 고려 사항은 t을 언급된다www.analyticalultracentrifugation.com : 다음 웹 사이트 오.
- 열기 고해상도 SEDFIT에서 스캔 (데이터 |로드 침강 평형 데이터) 및 3 채널 (; |로드 옵션 | 3 하위 집합에서 6 채널 원시 데이터를 저장 옵션이 서로 다른 각각의 샘플에 대한 농도에 해당)에 데이터를 분할합니다.
- 다시 열고 SEDFIT에서 동일한 샘플과 동일한 농도하지만 서로 다른 속도에 속한 데이터 파일. 메 니스 커스 (수직 레드 라인), 셀 하단 (세로 블루 라인) 및 피팅 제한 (수직 녹색 선)을 조정하고 SEDPHAT에 사용하기 위해 데이터를 내보낼 (데이터 | SEDPHAT하는 데이터 내보내기). 입력 요청과 같이 회전 자형과 중심 형뿐만 아니라, 단계 2.1.1에서 산출 파라미터. 모든 샘플과 농도에 대해이 단계를 반복합니다.
- SEDPHAT에서 동일한 샘플의 모든 데이터 (모든 농도 및 속도)을 열고 실험 매개 변수를 입력; 한 가지 예를 도시한다. 5.
참고 : D 2 O 버퍼, 신에 추가 될 때 교환 가능한 양성자 eration 크게 특히 수용성 단백질, 단백질의 분자량을 변경할 수있다. 막 임베디드 지역 교환으로부터 보호되기 때문에 막 단백질, SH 단백질 같은 특히 작은 것들, 덜 영향을 받는다. 이 문제를 해결하려면, 입력 "D에게 몰 분율 버퍼".
참고 :이 단계에서이 메뉴 데이터를 선택하여 개별적으로 편집 된 데이터 집합을 저장하는 것이 좋습니다 | 새로운 구성으로 복사 모든 데이터와 저장을. - 모델을 선택하고 그 모델에 대한 전역 매개 변수를 입력합니다.
참고 : 예를 들어, "N 모노머 - 메르 셀프 협회"모델 및 파라미터가도 2에 도시되어있다. 6.
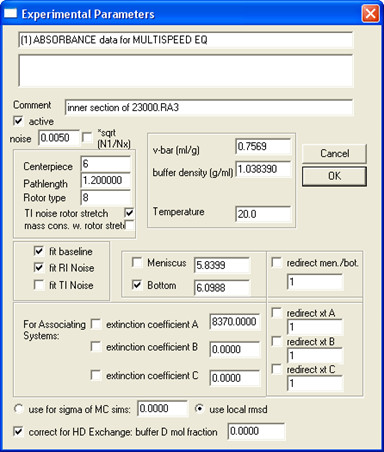
그림 5. 실험 매개 변수를 입력하는 방법에 대한 예.
파일 / ftp_upload / 52404 / 52404fig6.jpg "/>
그림 6. 모노머 - 앤 - 메르 셀프 협회 모델 전역 매개 변수를 입력하는 방법에 대한 예.
- 메뉴 맞춤 선택하여 글로벌 맞춤을 실행 | 글로벌 맞춤을하고 적합 수렴 할 때까지 기다립니다. 아래 참고 (또는 화면을 캡처) 피팅 결과를, 특히 글로벌 축소 카이 광장과 K에게 값을 기록합니다. 열역학 정보를 표시 |에 맞춤 데이터와 메뉴 복사 및 디스플레이에서 피팅 잔차와 같은 다른 데이터의 압축을 풉니 다.
- 전역 매개 변수로 돌아가서 (1) 단량체 분자량에 맞게 단계 2.4.5를 반복 M을 선택합니다. 장착 분자량 및 글로벌 축소 카이 제곱을합니다.
- 단계를 반복 2.4.3 각 모델이 테스트하기 2.4.6 및 감소 된 글로벌 카이 제곱 값뿐만 아니라 피팅 잔차를 비교하여 각 모델의 적합성 품질을 비교할 수 있습니다.
참고 : 작은 임의 피팅 잔차는 일반적으로 잘 맞는을 나타내고, 것 가장 적합한 모델작은 글로벌 축소 카이 제곱 있습니다. 장착 모노머 분자량과 카이 제곱 값은 고정 (이론) 분자량의 실질적 차이가 나지 않습니다. - 첫 번째 선택 통계에 의해 값에 의해 얻어진 로그 카에 대한 신뢰 구간을 계산 | 심각한 오류 표면 돌기에 대한 카이 제곱 원하는 신뢰 구간을 입력. 로그 카에 대한 카이 제곱 값을 얻기 위해 전역 매개 변수 대화 상자에서 카 로그 1 차원 오류 표면에 돌기를 생성하고 선택 취소 | 다음, 통계로 이동합니다.
주 : 독자는이 방법 (33)의 방법 (32)뿐만 아니라 그림에 대한 자세한 내용은 다음 소스를 (http://www.analyticalultracentrifugation.com/ sedphat / statistics.htm)를 참조하시기 바랍니다.
결과
50mM 트리스에서 C14SB 세제 미셀의 방사상 분포 프로필, 100 mM의 NaCl을 pH가 7.3 형태 선형 모델 (도 7A)에 장착 될 수있는 매우 얕은 지수. 이 분포의 기울기는 반대로 D 2 O 농도 (그림 7B)에 관련된다. 기울기가 0 인 점은, 정합 D 2 O 농도는 32.3 % 인 것으로 밝혀졌다, 즉.
아래 그림 7을 참조하십시오.
토론
이 논문은 샘플 준비 및 평형 침을 사용하여 세제의 작은 막 단백질의 올리고머의 분석을위한 실험 프로토콜을 제공합니다. 밀도 정합 단계가 필요하지 않은 것으로 설명 프로토콜은 동등하게 유효한 -and simpler- 가용성 단백질이다. 실제로, 시스템은 세제와 단백질의 혼합물로 구성된다. 이 부유 입자에 기여하지 않도록 침강 연구를 수행하기 위해, 세제 중력장에 보이지이어야한다. 따라서, 세제...
공개
The authors have nothing to disclose.
감사의 말
This work has been funded by the National Research Foundation grant NRF-CRP4-2008-02 (J.T.) and Tier 1 grant RG 51/13.
자료
| Name | Company | Catalog Number | Comments |
| 3-(N,N-dimethylmyristylammonio)propanesulfonate | Sigma | T0807 | |
| Deuterium oxide 99.8% | Cambridge Isotope | DLM-4-99.8 | |
| An-50 Ti Rotor, Analytical, 8-Place | Beckman Coulter | 363782 | |
| An-60 Ti Rotor, Analytical, 4-Place | Beckman Coulter | 361964 | |
| Cell housing | Beckman Coulter | 334784 | |
| 12 mm six-channel centerpiece, epon charcoal-filled | Beckman Coulter | 331376 | |
| Window holder | Beckman Coulter | 305037 | |
| Window gasket | Beckman Coulter | 327021 | |
| Window liner | Beckman Coulter | 362329 | |
| Sapphire window | Beckman Coulter | 307177 | |
| Quartz window | Beckman Coulter | 301730 | |
| Screw-ring washer | Beckman Coulter | 362328 | |
| Screw ring | Beckman Coulter | 301922 | |
| Spinkote | Beckman Coulter | 306812 | |
| Torque stand assembly | Beckman Coulter | 361318 | |
| Counterbalance | Beckman Coulter | 360219 | |
| Cell alignment tool | Beckman Coulter | 362340 | |
| SEDNTERP | http://bitcwiki.sr.unh.edu/index.php/Main_Page | ||
| HeteroAnalysis | http://www.biotech.uconn.edu/auf/?i=aufftp | ||
| SEDFIT | http://www.analyticalultracentrifugation .com/sedfit.htm | ||
| SEDPHAT | http://www.analyticalultracentrifugation .com/sedphat/default.htm |
참고문헌
- Laue, T. M., Stafford, W. F. Modern applications of analytical ultracentrifugation. Annu. Rev. Biophys. Biomol. Struct. 28, 75-100 (1999).
- Lebowitz, J., Lewis, M. S., Schuck, P. Modern analytical ultracentrifugation in protein science: A tutorial review. Protein Sci. 11, 2067-2079 (2002).
- Balbo, A., Zhao, H., Brown, P. H., Schuck, P. . Assembly, Loading, and Alignment of an Analytical Ultracentrifuge Sample Cell. , e1530 (2009).
- Rivas, G., Stafford, W., Minton, A. P. Characterization of heterologous protein-protein interactions using analytical ultracentrifugation. Methods-a Companion to Methods in Enzymology. 19, 194-212 (1999).
- Howlett, G. J., Minton, A. P., Rivas, G. Analytical ultracentrifugation for association and assembly the study of protein. Curr. Opin. Chem. Biol. 10, 430-436 (2006).
- MacGregor, I. K., Anderson, A. L., Laue, T. M. Fluorescence detection for the XLI analytical ultracentrifuge. Biophys. Chem. 108, 165-185 (2004).
- Phizicky, E. M., Fields, S. Protein-Protein Interactions - Methods for Detection and Analysis. Microbiol. Rev. 59, 94-123 (1995).
- Alexandrov, A. A facile method for high-throughput co-expression of protein pairs. Mol. Cell. Proteomics. 3, 934-938 (2004).
- Nooren, I. M. A., Thornton, J. M. Structural characterisation and functional significance of transient protein-protein interactions. J. Mol. Biol. 325, 991-1018 (2003).
- Ebel, C. Sedimentation velocity to characterize surfactants and solubilized membrane proteins. Methods. 54, 56-66 (2011).
- Minton, A. P. Quantitative characterization of reversible macromolecular associations via sedimentation equilibrium: an introduction. Exp. Mol. Med. 32, 1-5 (2000).
- Dowell, S. F. Respiratory syncytial virus is an important cause of community-acquired lower respiratory infection among hospitalized adults. J. Infect. Dis. 174, 456-462 (1996).
- Bukreyev, A., Whitehead, S. S., Murphy, B. R., Collins, P. L. Recombinant respiratory syncytial virus from which the entire SH gene has been deleted grows efficiently in cell culture and exhibits site-specific attenuation in the respiratory tract of the mouse. J. Virol. 71, 8973-8982 (1997).
- Fuentes, S., Tran, K. C., Luthra, P., Teng, M. N., He, B. Function of the respiratory syncytial virus small hydrophobic protein. J. Virol. 81, 8361-8366 (2007).
- Jin, H. Recombinant respiratory syncytial viruses with deletions in the NS1, NS2, SH, and M2-2 genes are attenuated in vitro and in vivo. Virology. 273, 210-218 (2000).
- Karron, R. A. Respiratory syncytial virus (RSV) SH and G proteins are not essential for viral replication in vitro: clinical evaluation and molecular characterization of a cold-passaged, attenuated RSV subgroup B. Proc. Nat. Acad. Sci. USA. 94, 13961-13966 (1997).
- Whitehead, S. S. Recombinant respiratory syncytial virus bearing a deletion of either the NS2 or SH gene is attenuated in chimpanzees. J. Virol. 73, 3438-3442 (1999).
- Rixon, H. W. The small hydrophobic (SH) protein accumulates within lipid-raft structures of the Golgi complex during respiratory syncytial virus infection. J. Gen. Virol. 85, 1153-1165 (2004).
- Collins, P. L., Mottet, G. Membrane orientation and oligomerization of the small hydrophobic protein of human respiratory syncytial virus. J. Gen. Virol. 74, 1445-1450 (1993).
- Collins, P. L., Olmsted, R. A., Johnson, P. R. The small hydrophobic protein of human respiratory syncytial virus: comparison between antigenic subgroups A and B. J. Gen. Virol. 71, 1571-1576 (1990).
- Chen, M. D., Vazquez, M., Buonocore, L., Kahn, J. S. Conservation of the respiratory syncytial virus SH gene. J. Infect. Dis. 182, 1228-1233 (2000).
- Gan, S. W. The small hydrophobic protein of the human respiratory syncytial virus forms pentameric ion channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Gan, S. W., Ng, L., Xin, L., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein Sci. 17, 813-820 (2008).
- Li, Y. Inhibition of the Human Respiratory Syncytial Virus Small Hydrophobic Protein and Structural variations in a bicelle environment. J. Virol. 88 (22), 11899-914 (2014).
- Burgess, N. K., Stanley, A. M., Fleming, K. G. Determination of membrane protein molecular weights and association equilibrium constants using sedimentation equilibrium and sedimentation velocity. Meth. Cell. Biol. 84, 181-211 (2008).
- Cole, J. L., Lary, J. W., Moody, T. P., Laue, T. M. Analytical Ultracentrifugation: Sedimentation Velocity and Sedimentation Equilibrium. Meth. Cell. Biol. 84, 143-179 (2008).
- Fleming, K. G. Determination of membrane protein molecular weight using sedimentation equilibrium analytical ultracentrifugation. Curr. Protoc. Prot. Sci. 53, 17.12.11-17.12.13 (2008).
- . . An-50 Ti and An-60 Ti Analytical Rotor, Cells, and Counterbalance. , (2005).
- Mayer, G. Studying membrane proteins in detergent solution by analytical ultracentrifugation: Different methods for density matching. Prog. Colloid Polym. Sci. 113, 176-181 (1999).
- Laue, T. Ch. 20.3. Current Protocols in Protein Science. 20, 20.23.21-20.23.13 (2001).
- Gan, S. W. The Small Hydrophobic Protein Of The Human Respiratory Syncytial Virus Forms Pentameric Ion Channels. J. Biol. Chem. 287, 24671-24689 (2012).
- Bevington, P. R., Robinson, D. K. . Data reduction and error analysis for the physical sciences. 336, (1969).
- Schuck, P., Radu, C. G., Ward, E. S. Sedimentation equilibrium analysis of recombinant mouse FcRn with murine IgG1. Molecular Immunology. 36, 1117-1125 (1999).
- Gan, S. W., Vararattanavech, A., Nordin, N., Eshaghi, S., Torres, J. A cost-effective method for simultaneous homo-oligomeric size determination and monodispersity conditions for membrane proteins. Anal. Biochem. 416, 100-106 (2011).
- Montserret, R. NMR structure and ion channel activity of the p7 protein from hepatitis C virus). J. Biol. Chem. 285, 31446-31461 (2010).
- Stouffer, A. L., DeGrado, W. F., Lear, J. D. Analytical Ultracentrifugation Studies of the Influenza M2 Homotetramerization Equilibrium in Detergent Solutions. Progr Colloid Polym Sci. 131, 108-115 (2006).
- Sorkin, A., von Zastrow, M. Signal transduction and endocytosis: Close encounters of many kinds. Nat. Rev. Mol. Cell Biol. 3, 600-614 (2002).
- Gan, S. W., Ng, L., Lin, X., Gong, X., Torres, J. Structure and ion channel activity of the human respiratory syncytial virus (hRSV) small hydrophobic protein transmembrane domain. Protein science : a publication of the Protein Society. 17, 813-820 (2008).
재인쇄 및 허가
JoVE'article의 텍스트 или 그림을 다시 사용하시려면 허가 살펴보기
허가 살펴보기더 많은 기사 탐색
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. 판권 소유